JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
Complementary Metal-Oxide-Semiconductor High-Density Microelectrode Array System을 사용한 급성 뇌 절편에서 고품질 발작 유사 활동
* 이 저자들은 동등하게 기여했습니다
요약
여기에서는 상보적 금속 산화물 반도체 고밀도 미세전극 어레이 시스템(CMOS-HD-MEAs)을 사용하여 생체 외 뇌 절편에서 발작과 같은 활동을 기록하는 프로토콜을 간략하게 설명합니다.
초록
상보적 금속-산화물-반도체 고밀도 미세전극 어레이(CMOS-HD-MEA) 시스템은 세포 배양 및 생체 외 뇌 절편의 신경 생리학적 활동을 전례 없는 전기생리학적 세부 사항으로 기록할 수 있습니다. CMOS-HD-MEA는 세포 배양에서 고품질 신경 단위 활동을 기록하도록 먼저 최적화되었지만, 급성 망막 및 소뇌 절편에서 고품질 데이터를 생성하는 것으로도 나타났습니다. 연구원들은 최근 CMOS-HD-MEA를 사용하여 급성 대뇌 피질 설치류 뇌 절편에서 국소 자기장 전위(LFP)를 기록했습니다. 관심 있는 LFP 중 하나는 발작과 유사한 활동입니다. 많은 사용자가 CMOS-HD-MEA를 사용하여 짧고 자발적인 간질형 방전을 일으켰지만, 고품질의 발작과 같은 활동을 안정적으로 생성하는 사용자는 거의 없습니다. 전기 노이즈, 수중 기록 챔버를 사용할 때 발작과 같은 활동을 생성하는 일관되지 않은 특성, 2D CMOS-MEA 칩이 뇌 절편 표면에서만 기록한다는 제한 등 많은 요인이 이러한 어려움에 기여할 수 있습니다. 이 프로토콜에 자세히 설명된 기술을 통해 사용자는 CMOS-HD-MEA 시스템을 사용하여 급성 뇌 절편에서 고품질 발작과 같은 활동을 지속적으로 유도하고 기록할 수 있습니다. 또한 이 프로토콜은 CMOS-HD-MEA 칩의 적절한 처리, 실험 중 용액 및 브레인 슬라이스 관리, 장비 유지 관리에 대해 설명합니다.
서문
수천 개의 기록 포인트(1,2)가 있는 MEA 칩과 데이터를 증폭하고 디지털화하기 위한 MEA 플랫폼을 포함하는 상업적으로 이용 가능한 고밀도 미세전극 어레이(HD-MEA) 시스템은 전기생리학 연구를 위한 새로운 도구입니다. 이러한 HD-MEA 시스템은 CMOS(Complementary Metal-Oxide-Semiconductor) 기술을 사용하여 세포 배양 및 생체 외 뇌 절편 제제의 고감도로 전기생리학적 데이터를 기록합니다. 이러한 MEA 시스템은 높은 전극 밀도와 고품질 신호 대 잡음비를 통해 신경 생리학 연구에 전례 없는 공간적, 시간적 해상도를 제공합니다3. 이 기술은 주로 세포외 활동 전위를 연구하는 데 사용되어 왔지만, 다양한 신경 세포 뇌 절편 제제(4,5,6,7,8,9,10,11,12,13,14,15)에서 고품질 국소 자기장 전위(LFP)를 캡처할 수도 있습니다 . 위에서 언급한 CMOS-HD-MEA 시스템의 고해상도 기록 기능으로 인해 사용자는 뛰어난 공간 정확도(16,17,18)로 전기생리학적 활동을 추적할 수 있습니다. 이 기능은 특히 네트워크 LFP 5,12,15,19,20,21의 전파 패턴을 추적하는 것과 관련이 있습니다. 따라서 CMOS-HD-MEA 시스템은 다양한 세포 배양 및 뇌 절편 제제의 생리학적 및 병리학적 활성의 증식 패턴에 대한 전례 없는 이해를 제공할 수 있습니다. 특히 CMOS-HD-MEA 시스템의 이러한 기능을 통해 연구자들은 서로 다른 뇌 영역의 발작 패턴을 동시에 대조하고 다양한 항간질 화합물이 이러한 패턴에 어떤 영향을 미치는지 분석할 수 있습니다. 이를 통해 ictogenesis 및 ictal propagation을 연구하고 약리학이 병리학적 네트워크 활동을 방해하는 방법을 이해하기 위한 혁신적인 방법을 제공합니다 7,10,14. 따라서 CMOS-HD-MEA 시스템의 이러한 새로운 기능은 신경 장애 연구에 크게 기여할 수 있을 뿐만 아니라 약물 발견 연구에도 도움이 될 수 있습니다 5,7,11,22. 우리는 CMOS-HD-MEA 시스템을 사용하여 발작과 같은 활동을 연구하는 방법에 대한 세부 정보를 제공하는 것을 목표로 합니다.
CMOS-HD-MEA 시스템을 사용하여 급성 뇌 절편의 간질 활동과 같은 LFP를 연구할 때 사용자는 쇠약해지는 전기 노이즈, 실험 중 절편을 건강하게 유지, 뇌 절편 표면에서만 기록하는 2차원(2D) CMOS-MEA 칩의 품질 신호 감지 등 많은 문제에 직면할 수 있습니다. 이 프로토콜은 실험에 사용되는 MEA 플랫폼 및 기타 장비를 적절하게 접지하기 위한 기본 단계를 설명하며, 이는 각 실험실 설정에 대한 개별 사용자 정의가 필요할 수 있는 중요한 단계입니다. 또한, CMOS-HD-MEA 시스템(23,24,25)과 함께 사용되는 수중 챔버에서 장시간 녹음하는 동안 뇌 절편을 건강하게 유지하는 데 도움이 되는 단계에 대해 논의합니다. 또한 뇌 절편 깊숙한 곳에서 기록하는 일반적인 전기생리학적 기록 방법과 달리 대부분의 CMOS-HD-MEA 시스템은 절편으로 침투하지 않는 2D 칩을 사용합니다. 따라서 이러한 시스템은 기록된 LFP 신호의 대부분을 생성하기 위해 건강한 신경 세포 외층이 필요합니다. 또 다른 과제로는 수천 개의 전극에서 생성되는 방대한 양의 데이터를 시각화하는 것이 있습니다. 이러한 문제를 극복하기 위해, 우리는 뇌 절편 전체에 전파되는 고품질 네트워크 간질 활동을 달성할 가능성을 높이는 간단하지만 효과적인 프로토콜을 권장합니다. 또한 데이터 시각화10을 돕기 위해 관련 리소스와 함께 개발한 공개적으로 사용 가능한 그래픽 사용자 인터페이스(GUI)에 대한 간략한 설명도 포함되어 있습니다.
이전의 간행물들은 MEA 기록 시스템(26,27,28,29)의 사용을 위한 관련 프로토콜을 제공하였다. 그러나 이 연구는 2D 칩이 있는 CMOS-HD-MEA 시스템을 사용하는 실험자, 특히 뇌 절편에서 고품질 간질 활동을 연구하려는 실험자를 지원하는 것을 목표로 합니다. 또한 발작과 유사한 활동 유도를 위한 가장 일반적인 두 가지 용액 조작, 즉 0 Mg2+ 및 4-AP 패러다임을 비교하여 사용자가 특정 응용 분야에 가장 적합한 경련 매체를 식별할 수 있도록 지원합니다. 이 프로토콜은 주로 발작과 유사한 활동의 생성에 초점을 맞추고 있지만, 뇌 절편을 사용하여 다른 전기 생리학적 현상을 탐색하도록 수정할 수 있습니다.
프로토콜
생쥐와 관련된 시술은 브리검 영 대학교의 IACUC(Institutional Animal Care and Use Committee)의 승인을 받았다. 수컷 및 암컷(n=8) C57BL/6 마우스를 P21 이상으로 숙성시킨 다음 실험에 사용하였다.
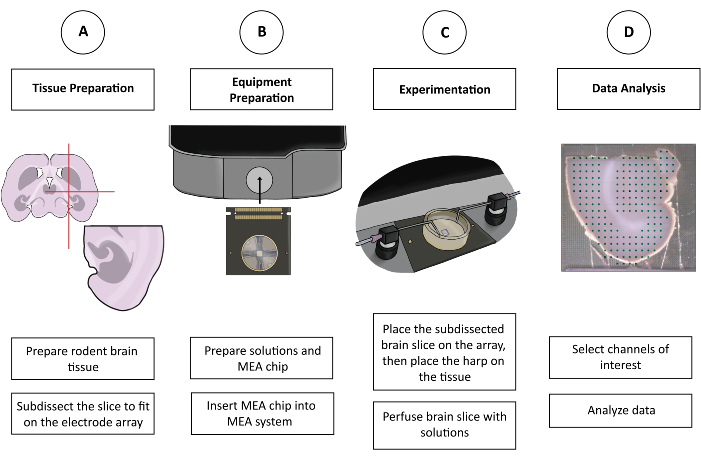
그림 1: CMOS-HD-MEA 실험의 개략도. (A) 뇌 절편은 선호하는 절단 방법으로 준비하고 MEA에 맞게 하위 절개합니다. (B) 용액과 CMOS-HD-MEA 칩을 준비합니다. (C) 하위 절개된 뇌 절편을 전극 어레이에 놓고 적절한 용액에 담근다. (D) 수집된 데이터에서 관련 채널을 선택합니다. 그런 다음 사용자가 선호하는 프로그램에서 분석을 위해 데이터를 준비합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| 이름 | 농도 (mM) | g/엘 | ||
| 염화나트륨(NaCl) | 126 | 7.36 | ||
| 염화칼륨(KCl) | 3.5 | 0.261 | ||
| 디 하이드로겐 인산 나트륨 (NaH2PO4) | 1.26 | 0.151 | ||
| 중탄산 나트륨 (NaHCO3) | 26 | 2.18 | ||
| 포도당 (C6H12O6) | 10 | 1.80 | ||
| 염화 마그네슘 (MgCl2) | 1 (1 M 재고부터) | 1mL | ||
| 염화칼슘(CaCl2) | 2 (1 M 재고에서) | 2mL | ||
표 1: aCSF 용액.
1. 솔루션 준비
- 실험 솔루션을 준비합니다.
- 인공 뇌척수액(aCSF) 1L를 준비합니다(자세한 내용은 표 1 참조).
- 경련 촉진제 용액 1L를 준비합니다.
참고: 이 프로토콜에서 발작과 같은 활성을 생성하는 데 사용되는 용액은 100μM 4-아미노피리딘(4-AP)이 있는 aCSF 또는 마그네슘 이온이 없는 aCSF를 사용했습니다. - 다공성 돌을 사용하여 사용하기 전에 최소 10분 동안 모든 용액을 탄화하십시오.
- 용액을 버리기 위한 비커를 구하십시오.
- aCSF 1L, 전경련제 용액 1L, 폐기 비커를 관류 시스템에 가까운 표면에 놓습니다.
- 절단 및 고정 챔버 솔루션 준비
- CaCl2 를 생략하고 3mM MgCl2 (1mM과 반대)를 추가하여 고유한 절단 솔루션을 만들고 0.5L의 aCSF를 준비합니다. 설치류 뇌 절편 준비에 사용할 수 있도록 이 용액을 급성 뇌 절편 준비 영역 근처에 두어 보관하십시오.
- 0.5L의 aCSF를 준비하고 실험에 사용하기 위해 슬라이스를 보관하는 데 사용할 조직 보유 챔버에 넣습니다. 다공성 돌을 조직 보유 챔버에 삽입하고 하위 절개 된 조각을 챔버에 놓기 전에 최소 10 분 동안 용액을 탄산염화합니다.
2. 설치류 뇌 슬라이스 준비하기
- 위에서 설명한 대로 모든 용액을 준비합니다(섹션 1 참조). 전처리 과정에서 모든 용액이 적절하게 탄화되었는지 확인하십시오.
참고: 절단 용액(섹션 1.2 참조)이 권장되지만 자당 절단 용액(30)과 같은 고품질 브레인 슬라이스를 얻기 위해 다른 절단 용액을 사용할 수 있습니다. - 진동하는 마이크로톰을 사용하여 350μm 설치류의 뇌 절편을 만듭니다. 이러한 기기를 사용하기 위한 프로토콜은 참조30,31,32,33에 나열되어 있습니다.
- MEA 칩의 기록 전극 영역에 맞도록 설치류 뇌 절편을 준비합니다( 그림 2 참조). 그림 1A 및 그림 2A 와 같이 크기 10의 수술용 칼날을 사용하여 뇌 절편을 앞뒤로 부드럽게 굴려 절편을 하위 절개합니다. 진동 마이크로톰 절단 챔버에서 하위 해부를 수행합니다. 하위 절개된 뇌 절편을 손상시키지 않도록 주의하십시오.
- 하위 절개된 조각을 aCSF로 채워진 조직 보유 챔버에 놓습니다. 하위 절개된 슬라이스를 추가하기 전에 챔버가 최소 10분 동안 탄화되었는지 확인하십시오.
3. 장비 준비
- MEA 칩 및 시스템 준비(그림 2 및 그림 3)
알림: 플라스틱 및 금속과 같은 단단한 재료는 칩 바닥에 강제로 잘 닿으면 MEA 칩을 쉽게 손상시킬 수 있습니다. 피펫을 사용하여 용액을 칩 안팎으로 옮길 때 칩 바닥을 피펫, 특히 칩 웰 측면의 기준 전극과 칩 웰 중앙의 기록 전극과 잘 접촉시키지 마십시오(그림 2C). 대신, 칩 웰의 바닥 위로 마우스를 가져가거나 칩 웰의 플라스틱 가장자리를 접촉하거나 근사화하여 솔루션을 추가합니다. 모든 용액을 쉽게 빼내려면 용액이 칩의 한쪽 면에 잘 고일 수 있도록 칩을 약간 기울여 풀링된 용액의 상단에서 제거하거나 정전기 방지 물티슈를 사용하여 용액이 남아 있는 부분을 가볍게 두드립니다. 칩을 잘 잡거나 칩 핀을 잡고 칩을 운반하지 마십시오(그림 2C). 칩 웰은 약 4mL의 용액을 담을 수 있습니다. 후속 단계에서는 달리 지정되지 않은 경우 약 2mL의 용액으로 칩 웰을 채웁니다.- 칩 준비를 시작하기 전에 다양한 작업을 위한 트랜스퍼 파이펫을 지정하십시오. 의도하지 않은 혼합을 방지하기 위해 하나의 이송 피펫에 에탄올, 다른 하나는 폐기물, 다른 하나는 aCSF, 다른 하나는 나머지 용액에 라벨을 부착합니다.
- MEA 칩의 웰을 190도 프루프 에탄올로 채워 칩 웰의 바닥이 완전히 덮이도록 합니다(그림 3). 에탄올을 30-60초 동안 그대로 둔 다음 폐기 피펫으로 제거합니다.
- MEA 칩의 웰을 aCSF로 채우고 폐기 피펫으로 제거하여 칩 웰에서 잔류 에탄올을 헹굽니다. 이전에 지정된 폐기물 및 aCSF 피펫을 사용하여 칩 웰에서 aCSF를 세 번 추가 및 제거합니다. 칩을 세 번 잘 세척한 후 aCSF를 넣고 30초 이상 그대로 두십시오.
알림: 기록 전극은 에탄올 및 aCSF로 헹군 후 aCSF가 최소 45분 동안 챔버에 남아 있을 때 소수성 상호 작용으로 인한 소음이 가장 적습니다. - MEA 칩을 도킹하기 전에 정전기 방지 물티슈에 190도 방지 에탄올을 적셔 칩의 핀을 닦습니다(그림 2C).
- MEA 칩을 MEA 플랫폼에 부드럽게 밀어 넣고 도킹 메커니즘을 작동시켜 칩을 제자리에 고정합니다.
- 기포가 있는지 기록 및 기준 전극을 확인합니다(그림 2C). 기포가 있으면 깨끗한 붓을 사용하여 전극을 가볍게 쓸어 제거합니다.
- CMOS-HD-MEA 소프트웨어(34 )를 사용하여 칩에 노이즈가 있는지 확인하고 전기적 간섭에 의해 야기된 기포, 비생물학적 진동 또는 스파이크에 대한 거짓 컬러 맵을 육안으로 스캔한다. 발생한 소음을 무효화하기 위해 MEA 시스템을 적절하게 접지하십시오.
알림: 접지 설정은 녹음 환경에 따라 다릅니다. 이 프로토콜의 실험을 위해 MEA 플랫폼과 관류 시스템이 접지되었습니다.
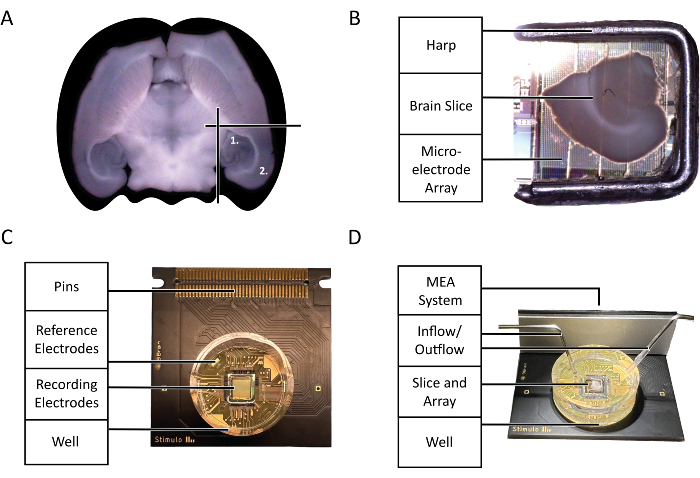
그림 2: 구성 및 기술 다이어그램. (A) 이 프로토콜에 의해 강조된 실험에 사용된 급성 마우스 뇌 절편의 선택 다이어그램. (1) 해마 영역 (2) 신피질 영역. (B) 미세전극 어레이(MEA)에 급성 마우스 뇌 절편과 하프의 적절한 배치. (C) 3Brain Accura CMOS-HD-MEA 칩의 해부학적 구조. (D) 관류 입구 및 출구의 적절한 구성. 입력은 칩 웰 깊숙이 있어야 하는 반면, 출력은 신선하고 산소가 공급된 aCSF의 일정한 흐름을 보장하기 위해 칩 웰 상단의 입구 반대쪽에 있어야 합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
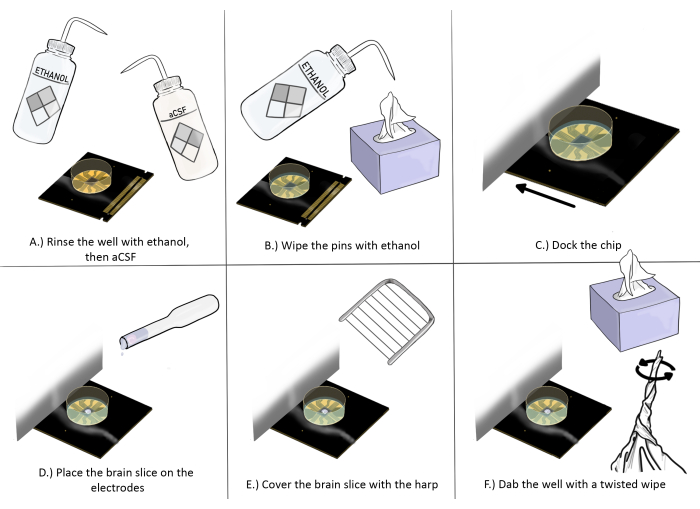
그림 3: 브레인 슬라이스 실험을 위한 칩 준비 및 배치의 개략도. (A) 칩을 에탄올로 한 번 헹군 다음 aCSF로 세 번 헹굽니다. (B) 정전기 방지 물티슈를 사용하여 에탄올로 핀을 닦습니다. (C) 칩을 도킹합니다. (D) 뇌 절편을 전극에 놓습니다. (E) 하프를 브레인 슬라이스에 놓습니다(적절한 배치 지침은 그림 2 참조). (F) 뇌 절편 근처의 기록 전극 우물 모서리를 비틀린 정전기 방지 물티슈로 두드립니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
4. 실험
- 브레인 슬라이스 배치
- MEA 플랫폼 근처의 계량 보트(또는 다른 깨끗한 표면)에 백금 하프를 놓습니다. 소수성 경향을 줄이기 위해 약 3mL의 aCSF로 하프를 덮으십시오.
- 가위를 사용하여 전사 피펫의 얇은 끝 부분을 제거합니다. 피펫 팁의 세 번째(약 1.5인치)를 잘라냅니다.
알림: 이 수정된 피펫은 채취 및 운송 중에 뇌 절편을 수축시키거나 손상시키지 않습니다. - 변형된 피펫을 사용하여 절편 보유 챔버에서 뇌 절편을 수집합니다. 브레인 슬라이스와 피펫의 용액을 칩 웰에 부드럽게 분배합니다. 슬라이스를 올바르게 배치하려면 전사 피펫에서 더 많은 aCSF를 부드럽게 방출하여 브레인 슬라이스의 위치를 조작하거나 부드러운 페인트 브러시를 사용하여 용액에 전류를 생성하여 브레인 슬라이스를 기록 전극으로 밀어 넣습니다. 손상을 최소화하기 위해 기록 전극 또는 브레인 슬라이스와의 접촉을 제한하십시오.
- 집게를 사용하여 실을 아래로 향하게 하여 하프를 뇌 절편 위에 부드럽게 올려 슬라이스를 기록 전극에 누릅니다. 전극 어레이를 하프와 접촉하지 마십시오. 프레임이 없는 면이 유입 바늘 쪽을 향하고 하프의 프레임이 녹음 전극과 접촉하지 않도록 하프의 방향을 지정합니다(그림 2C, D).
- 폐기된 피펫을 사용하여 과도한 aCSF를 제거합니다. 정전기 방지 물티슈를 사용하여 모서리를 비틀어 팁을 만든 다음 기록 전극, 브레인 슬라이스 또는 하프를 건드리지 않고 기록 전극 주변에 남아 있는 aCSF를 흡수하는 데 사용합니다(그림 2).
- 지정된 aCSF 피펫을 사용하여 뇌 절편을 덮을 수 있을 만큼의 충분한 탄산화 aCSF(약 2mL)를 빠르게 추가합니다.
- 4.1.5단계를 반복합니다. 및 4.1.6. 두 번 더.
- 웰이 대략 3/4, 약 3mL가 찰 때까지 웰을 탄수화된 aCSF로 채웁니다.
- 현미경이나 카메라로 MEA 칩의 뇌 절편 사진을 찍습니다. 사진의 해상도가 기록 전극 배열의 경계와 뇌 절편의 해부학적 구조를 볼 수 있을 만큼 충분히 높은지 확인합니다.
- 실험 실행
- 관류 시스템 운영
- 유입 및 유출 튜브를 aCSF로 채워진 비커에 넣고 유입 및 유출 바늘을 칩 웰에 넣습니다. 유입 바늘을 기록 전극 바로 바깥쪽의 칩 웰 바닥에 가깝게 놓습니다. 유출 바늘을 칩 웰의 상단 가까이에 놓고 가장자리를 향해 액체가 거의 칩 웰의 가장자리까지 올라오도록 하고 슬라이스가 실험 내내 잠기도록 합니다(그림 2D).
- 관류 유입을 5mL/분으로, 관류 유출을 7mL/분으로 설정합니다.
참고: 칩 웰에서 용액이 오버플로되는 것을 방지하고 브레인 슬라이스에 용액 전류를 생성하기 위해 유출 속도가 유입 속도를 초과하는 것이 좋습니다. - 유입과 유출을 켭니다. 바늘이 공기 대신 용액을 출력하기 시작할 때까지 칩 웰에서 유입 바늘을 제거합니다. 그런 다음 4.2.1.1단계에서 설명한 대로 바늘을 칩 내부의 원래 위치로 다시 놓습니다.
- 용액 히터를 사용하여 용액을 약 34-37 °C의 생리적 온도 또는 그 근처로 유지하십시오.
- aCSF가 뇌 절편에 관류되도록 10분 동안 둡니다. 이렇게 하면 슬라이스를 기록 환경에 맞게 조정할 수 있습니다.
- 10분이 경과한 후 유출 튜브를 폐기 비커로 이동합니다. 그런 다음 유입 튜브를 경련제 용액이 들어 있는 비커로 이동합니다. 비경련성 aCSF를 관류 시스템에서 폐기 비커로 10분 동안 씻어내십시오.
- 유출 튜브를 pro-convulsant 용액이 들어있는 비커로 옮깁니다.
- 실험이 끝날 때까지 전경련제 용액이 순환되도록 합니다.
- 슬라이스 기록 시간이 2시간을 초과하는 경우 활성 브레인 슬라이스의 포도당 소비를 상쇄하기 위해 다른 전경련제 용액을 준비하는 것이 좋습니다.
- 뇌 절편 교환
- 유입을 끕니다. 10-15초 후에 유출을 끕니다.
- 집게를 사용하여 하프를 제거하고 계량 보트나 다른 표면에 놓습니다.
- 개조된 피펫을 사용하여 뇌 절편을 추출하고 버립니다. 기준 전극 또는 기록 전극 어레이를 만지지 마십시오(그림 2C).
- 비경련성 aCSF 용액이 들어 있는 비커에 유입 튜브를 넣습니다. 폐기 비커에 유출 튜브를 넣습니다. 유입 및 유출 바늘을 칩에 잘 두십시오. 관류 시스템을 10분 동안 실행하여 칩 웰과 관류 시스템에서 잔류 경련 유발 용액을 모두 제거합니다. 새로운 브레인 슬라이스로 실험 프로세스를 다시 시작합니다(4.1단계에서 시작).
- 관류 시스템 운영
- 실험 완료
- 장비 청소
- 우물에서 하프, 브레인 슬라이스, 용액을 각각 꺼냅니다.
- MEA 칩의 도킹을 해제하고 깨끗한 표면에 놓고 칩에 탈이온수를 잘 채웁니다.
- aCSF 용액에 남아 있는 염분을 제거하기 위해 탈이온수를 버리십시오.
- 전사 피펫을 사용하여 칩을 세제 용액으로 잘 채웁니다. 전사 피펫의 끝을 칩 웰에 올려 놓고 기록 전극을 가리킵니다. 빠르고 반복적으로 세제를 짜서 전사 피펫에 풀어 1분 동안 기록 전극 위에 세제를 세게 세척합니다. 세제를 5-10분 동안 그대로 두십시오.
- 세제를 제거한 다음 탈이온수로 칩을 4-6번 잘 헹구어 세제 용액과 남아 있는 용질을 제거합니다.
- 칩 웰에서 모든 물을 제거한 다음 MEA 칩 위에 정전기 방지 물티슈를 놓고 칩이 잘 건조될 때까지 밤새 그대로 둡니다.
- MEA 칩이 이전에 놓여 있던 MEA 기록 플랫폼에 계량 보트 또는 다른 방수 플레이트를 놓습니다. 하프를 접시 중앙에 놓고 하프의 양쪽에 유입 및 유출 바늘을 놓습니다.
- 물 50mL, 140프루프 에탄올 50mL, 물 200mL를 각각 튜브를 통해 폐기 비커로 넣어 관류 시스템과 하프를 청소합니다.
알림: 이때 세척 시간을 줄이기 위해 관류 속도를 높일 수 있지만 홍수를 방지하기 위해 유출 속도는 항상 유입 속도를 초과해야 합니다. - 카보겐 스톤을 증류수 비커에 넣고 카보겐을 튜브를 통해 5분 동안 보내 청소합니다. 밤새 건조하고 깨끗한 표면에 놓고 먼지가 쌓이지 않도록 덮으십시오.
- 장비 청소
5. 데이터 분석
참고: 3Brain의 BrainWave, YASS(Yet Another Spike Sorter) 및 사용자 정의 Python 도구 34,35,36,37을 포함하여 CMOS-HD-MEA에 의해 생성된 전기생리학적 데이터를 분석하는 데 사용되는 다양한 분석 패키지가 있습니다. 그림 4와 그림 5에 제시된 데이터를 생성하기 위해 Xenon LFP 분석 플랫폼에서 사용하기 위해 BrainWave 데이터 파일 형식에서 데이터를 추출했습니다. 사용자 정의 Matlab 코드를 사용하여 그림 6의 데이터를 분석했습니다. Xenon LFP 분석 플랫폼용 프로토콜은 공개적으로 사용할 수 있습니다10. 다음 프로토콜 단계는 Brainwave 438로 만든 녹음에만 해당됩니다. 다른 시스템의 경우 해당 시스템과 관련된 지원 문서 34,35,36,37을 참조하십시오. 이 프로토콜로 데이터를 생성하기 위해 수행된 분석 단계의 개요는 다음과 같습니다. 자습서 비디오 및 모든 관련 코드 파일을 포함하여 데이터를 내보내고, 시각화하고, 분석하는 방법에 대한 자세한 내용은39를 참조하십시오.
- 분석을 위해 기록 파일을 내보냅니다.
- 다운샘플링할 데이터 기록 파일에 대한 폴더를 만듭니다.
- 4.1.9단계에서 촬영한 이미지를 잘라 뇌 슬라이스가 있는 기록 배열만 포함합니다.
- ExportToHDF5-ChannelSelection.py 파일(이 코드 파일은 GitHub의 xenon-lfp-analysis/code-files/3Brain-processing40에서 액세스할 수 있음)을 열고 창 상단에 나열된 URL을 복사하여 인터넷 브라우저에 붙여넣습니다.
- 데이터 기록 파일의 파일 경로를 입력하고 기록과 연결된 어레이의 자른 이미지를 업로드합니다.
- Select Channels for Export(내보낼 채널 선택)에서 올가미 선택 도구를 사용하여 배열의 잘린 이미지에 표시된 해마 영역과 신피질 영역을 선택합니다.
- 건너뛸 행 수와 건너뛸 열 수를 모두 2로 설정합니다.
참고: 녹음 파일은 종종 큽니다. 원하는 분석에 적합한 경우 내보낸 데이터를 다운샘플링할 수 있습니다. 공간적으로(기록 전극의 행과 열을 건너뛰기로) 및 시간적으로(더 낮은 샘플링 주파수로 다운샘플링하여) 다운샘플링하는 옵션이 있습니다. 분석에서 2개의 행과 2개의 열을 모두 건너뜁니다. 이를 통해 특정 채널로 편향되지 않고 데이터를 공간적으로 다운샘플링할 수 있습니다. 각 채널은 활동의 차이가 중요할 수 있을 만큼 충분히 멀리 떨어져 있습니다. 따라서 채널 평균화가 수행되지 않습니다. 전극의 행과 열을 건너뛸 때 선택되지 않은 채널의 데이터는 출력 파일을 내보낼 때 포함되지 않습니다. - 다운샘플링 주파수를 300Hz로 설정합니다.
- 채널 내보내기 버튼을 클릭하여 채널 선택 파일을 생성합니다.
알림: 소프트웨어는 채널 선택 파일을 데이터 녹음 파일이 저장된 폴더에 자동으로 저장합니다. - ExportToHDF5-ExtractDownsample.py 파일(GitHub의 xenon-lfp-analysis/code-files/3brain-processing40에서도 찾을 수 있음)을 열고 채널 선택 파일 및 데이터 기록 파일이 포함된 폴더의 파일 경로를 붙여넣은 다음 키보드에서 Enter 키를 눌러 데이터 기록 파일의 다운샘플링된 버전을 생성합니다.
- 분석 플랫폼 사용
- 컴퓨터의 명령 프롬프트에서 run_lfp_analysis 를 입력하고 Enter 키를 눌러 분석 플랫폼을 엽니다.
- 이미지와 다운샘플링된 녹음 파일의 파일 경로를 입력하고 제출을 클릭합니다.
- Select a Time Range for Analysis(분석을 위한 시간 범위 선택)에서 전체 기록을 선택합니다.
- Select Channels for Plots에서 해마를 Group-1로, 신피질을 Group-2로 강조 표시합니다.
- 채널 래스터에서 임계값을 0.06mV로, 기간을 0.02초로 설정합니다.
- Apply Settings and Generate Raster(설정 적용 및 래스터 생성)를 클릭합니다.
- 래스터 플롯을 사용하여 관심 있는 활동 패턴을 보여주는 다양한 채널을 탐색할 수 있습니다.
알림: 고품질 발작과 같은 활동(그림 4A-D) 대 최적이 아닌 활동(그림 4E-F)의 예에 대해서는 그림 4를 참조하십시오.
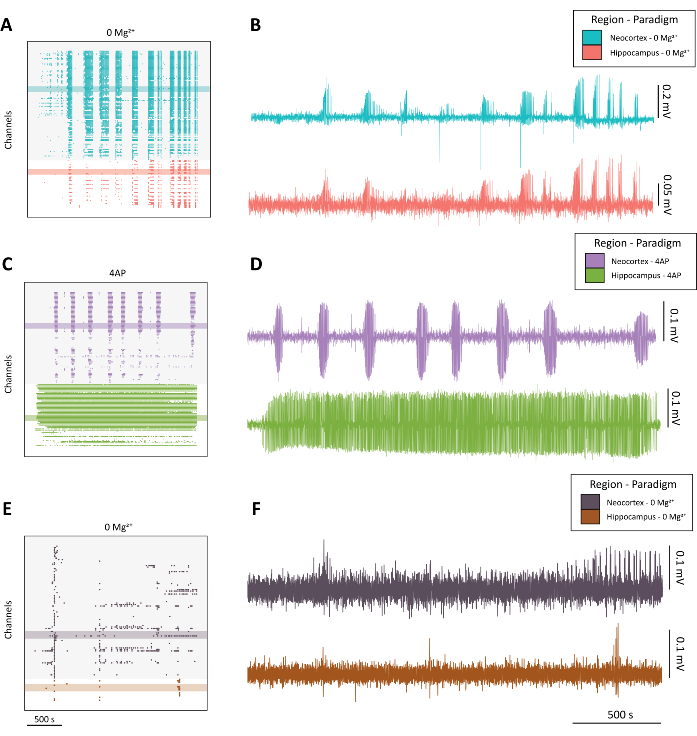
그림 4: 0 Mg2+ 및 4-AP 패러다임에서 진화하는 간질형 활성의 예. (A) 약 40분 동안 0 Mg2+ 의 aCSF를 적용한 래스터 플롯의 예. (B) 0 Mg2+ 패러다임의 간질 활성을 보여주는 신피질(파란색) 및 해마(빨간색)에서 가져온 전기생리학 추적의 예. (C) 약 40분 동안 100mM 4-AP 애플리케이션의 래스터 플롯 예. (D) 4-AP 적용으로 인한 간질 활성을 보여주는 신피질(보라색) 및 해마(녹색)에서 가져온 전기생리학 추적의 예. (E) 약 40분 동안 0 Mg2+ 의 aCSF 적용에서 얻은 래스터 플롯의 예는 다른 대표적인 추적에서 발견되는 발작과 같은 활동과 대조적으로 파열 활동을 보여줍니다. (F) 신피질(짙은 보라색) 및 해마(녹)에서 채취한 전기생리학적 흔적의 예로, B와 D에서 발견되는 양질의 발작과 같은 활성을 비교하기 위해 의도된 0 Mg2+ 패러다임의 최적이 아닌 활성을 보여줍니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
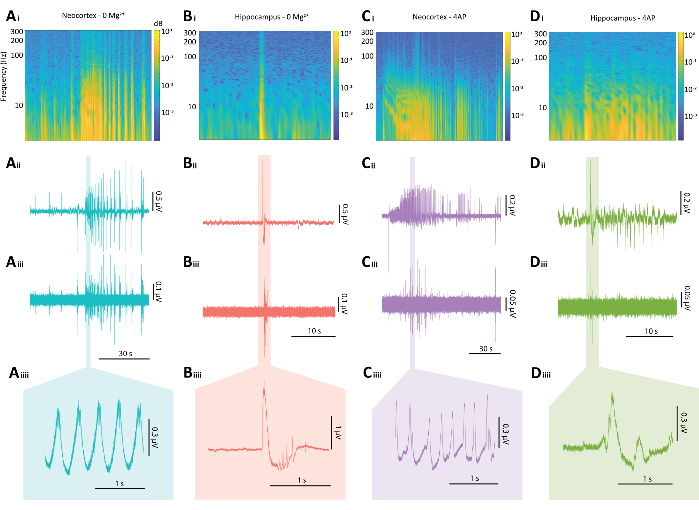
그림 5: 0 Mg2+ 및 4-AP 패러다임 모두에서 간질형 방전의 대표적인 결과. (A) (Ai) 발작 유사 사건의 스펙트로그램, (Aii) 관련 전기생리학적 추적, (Aiii) Aii의 추적에 적용된 80Hz 고역 통과 필터, (Aiiii) 및 Aii의 추적의 확대된 부분을 포함하는 0 Mg2+ 패러다임에 의해 유도된 전형적인 신피질 발작 유사 이벤트의 예시 플롯. (B) (Bi) 간질 파열의 스펙트로그램, (Bii) 관련 전기생리학적 추적, (Biii) Bii의 추적에 적용된 80Hz 고역 통과 필터, (Biiii) 및 Bii의 추적의 확대된 부분을 포함하는 0 Mg2+ 패러다임에 의해 유도된 전형적인 해마 간질 파열의 예시 플롯 (Ci) 간질 활성의 스펙트로그램, (Cii) 관련 전기생리학적 추적, (Ciii) Cii로부터의 추적에 적용된 80Hz 고역 통과 필터, (Ciiii) 및 Cii로부터의 추적의 확대된 부분 (D) (Di) 간질 활성의 스펙트로그램을 포함하는 4-AP 패러다임 하에서 해마 간질 파열의 예시 플롯, (Dii) 관련 전기생리학적 추적, (Diii) Dii로부터의 트레이스에 적용된 80Hz 고역 통과 필터, (Diiii) 및 Dii로부터의 트레이스의 확대된 부분. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
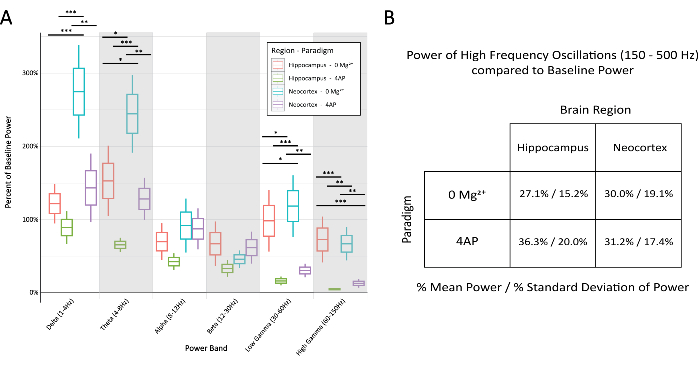
그림 6: 정형화된 간질 방전 중 패러다임과 뇌 영역에 걸친 다양한 대역의 기준선 전력 백분율 비교. (A) 간질 방전 중 전력은 대부분의 주파수 대역에서 패러다임과 뇌 영역 간에 유의한 차이를 보였다(Tukey 테스트를 사용한 2-way ANOVA, *P < 0.05, **P < 0.001, ***P < 0.0001). 각 상자의 중간 선은 평균, 상자의 테두리는 평균의 ±1 표준 오차(SEM), 가장 바깥쪽 선은 ±2 SEM을 나타냅니다. (B) 패러다임과 뇌 영역 모두 150Hz 이상의 고주파 활동과 관련된 대역에서 제한된 힘을 보여주었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
결과
여러 채널 1,4,5,10의 활동을 시각화할 때 표준적으로 사용되는 것처럼, 먼저 CMOS-HD-MEA로 획득한 데이터의 래스터 플롯을 생성하는 것이 좋습니다(그림 4A,C,E). 이 플롯은 각 채널을 y축에 표시하고 시간을 x축에 표시하여 각 브레?...
토론
이 프로토콜에는 CMOS-HD-MEA 사용자가 직면하는 일반적인 문제, 즉 뇌 절편 아래의 소음 발생 및 뇌 절편을 위한 건강한 환경 유지를 해결하는 급성 뇌 절편 관리와 관련된 특정 지침이 포함되어 있습니다. 슬라이스 아래의 소음 발생은 슬라이스가 어레이에 제대로 부착되지 않을 때 발생합니다. 브레인 슬라이스가 적절하게 부착되지 않으면 슬라이스 아래에 공기 주머니가...
공개
저자들은 이 연구 연구와 관련된 이해 상충이 없다고 선언합니다.
감사의 말
저자들은 이 원고를 편집해 준 전현직 Parrish 연구실 직원들에게 감사를 표한다. 또한 이 작업에 대한 피드백을 제공해 주신 3Brain의 Alessandro Maccione에게도 감사의 말씀을 전합니다. 이 연구는 AES/EF Junior Investigator Award와 Brigham Young University Colleges of Life Sciences and of Physical and Mathematical Sciences의 후원을 받았습니다.
자료
| Name | Company | Catalog Number | Comments |
| 2D Workbench | Cloudray | LM04CLLD26B | |
| 4-Aminopyridine | Sigma-Aldrich | 275875 | |
| Accura Chip | 3Brain | Accura HD-MEA | CMOS-HD-MEA chip |
| Agarose | Thermo Fisher Scientific | BP160-100 | |
| Vibration isolation table | Kinetic Systems | 91010124 | |
| Beaker for the slice holding chamber, 270 mL | VWR | 10754-772 | |
| BioCam | 3Brain | BioCAM DupleX | CMOS-HD-MEA platform |
| Brainwave Software | 3Brain | Version 4 | CMOS-HD-MEA software |
| Calcium Chloride | Thermo Fisher Scientific | BP510-500 | |
| Carbogen | Airgas | X02OX95C2003102 | |
| Carbogen | Airgas | 12005 | |
| Carbogen Stones | Supelco | 59277 | |
| Compresstome | Precissionary | VF-300-0Z | |
| Computer | Dell | Precission3650 | |
| Crocodile Clip Grounding Cables | JWQIDI | B06WGZG17W | |
| Detergent | Metrex | 10-4100-0000 | |
| D-Glucose | Macron Fine Chemicals | 4912-12 | |
| Dihydrogen Sodium Phosphate | Thermo Fisher Scientific | BP329-500 | |
| DinoCam | Dino-Lite | AM73915MZTL | |
| Ethanol | Thermo Fisher Scientific | A407P-4 | |
| Forceps | Fine Science Tools | 11980-13 | |
| Hot plate | Thermo Fisher Scientific | SP88857200 | |
| Ice Machine | Hoshizaki | F801MWH | |
| Inflow and outflow needles | Jensen Global | JG 18-3.0X | |
| Inline Solution Heater | Warner Instruments | SH-27B | |
| Isofluorine | Dechra | 08PB-STE22002-0122 | |
| Kim Wipes | Thermo Fisher Scientific | 06-666 | |
| Magnesium Chloride | Thermo Fisher Scientific | FLM33500 | |
| Micropipets | Gilson | F144069 | |
| Mili-Q Water Filter | Mili-Q | ZR0Q008WW | |
| Paintbrush | Daler Rowney | AF85 Round: 0 | |
| Paper Filter | Whatman | EW-06648-24 | |
| Parafilm | American National Can | PM996 | |
| Perfusion System | Multi Channel System | PPS2 | |
| Pipetor | Thermo Fisher Scientific | FB14955202 | |
| Platinum Harp | 3Brain | 3Brain | |
| Potassium Chloride | Thermo Fisher Scientific | P330-3 | |
| Razor blade | Personna | BP9020 | |
| Scale | Metter Toledo | AB204 | |
| Scissors | Solingen | 92008 | |
| Slice Holding Chamber | Custom | Custom | Custom 3D Printer Design, available upon request |
| Sodium Bicarbonate | Macron Fine Chemicals | 7412-06 | |
| Sodium Chloride | Thermo Fisher Scientific | S271-3 | |
| Temperature Control Box | Warner Instruments | TC344B | |
| Transfer Pipettes | Genesee Scientific | 30-200 | |
| Tubing | Tygon | B-44-3 TPE | |
| Vibratome VZ-300 | Precissionary | VF-00-VM-NC | |
| Weigh Boat | Electron Microscopy Sciences | 70040 |
참고문헌
- Obien, M. E. J., Frey, U. Large-scale, high-resolution microelectrode arrays for interrogation of neurons and networks. Adv Neurobiol. 22, 83-123 (2019).
- Schroter, M., et al. Functional imaging of brain organoids using high-density microelectrode arrays. MRS Bull. 47 (6), 530-544 (2022).
- Miccoli, B., et al. High-density electrical recording and impedance imaging with a multi-modal CMOS multi-electrode array chip. Front Neurosci. 13, 641 (2019).
- Emery, B. A., Hu, X., Khanzada, S., Kempermann, G., Amin, H. High-resolution CMOS-based biosensor for assessing hippocampal circuit dynamics in experience-dependent plasticity. Biosens Bioelectron. 237, 115471 (2023).
- Ferrea, E., et al. high-resolution electrophysiological imaging of field potentials in brain slices with microelectronic multielectrode arrays. Front Neural Circuits. 6, 80 (2012).
- Gagliano, G., et al. Non-linear frequency dependence of neurovascular coupling in the cerebellar cortex implies vasodilation-vasoconstriction competition. Cells. 11 (6), 1047 (2022).
- Goodchild, S. J., et al. Molecular pharmacology of selective Na(V)1.6 and dual Na(V)1.6/Na(V)1.2 channel inhibitors that suppress excitatory neuronal activity ex vivo. ACS Chem Neurosci. 15 (6), 1169-1184 (2024).
- Hu, X., Khanzada, S., Klutsch, D., Calegari, F., Amin, H. Implementation of biohybrid olfactory bulb on a high-density CMOS-chip to reveal large-scale spatiotemporal circuit information. Biosens Bioelectron. 198, 113834 (2022).
- Kim, S., et al. Alteration of neural network and hippocampal slice activation through exosomes derived from 5XFAD nasal lavage fluid. Int J Mol Sci. 24 (18), 14064 (2023).
- Mahadevan, A., Codadu, N. K., Parrish, R. R. Xenon LFP analysis platform is a novel graphical user interface for analysis of local field potential from large-scale MEA recordings. Front Neurosci. 16, 904931 (2022).
- Medrihan, L., Ferrea, E., Greco, B., Baldelli, P., Benfenati, F. Asynchronous GABA release is a key determinant of tonic inhibition and controls neuronal excitability: A study in the synapsin II-/- mouse. Cereb Cortex. 25 (10), 3356-3368 (2015).
- Monteverdi, A., Di Domenico, D., D'Angelo, E., Mapelli, L. Anisotropy and frequency dependence of signal propagation in the cerebellar circuit revealed by high-density multielectrode array recordings. Biomedicines. 11 (5), 1475 (2023).
- Obien, M. E. J., Hierlemann, A., Frey, U. Accurate signal-source localization in brain slices by means of high-density microelectrode arrays. Sci Rep. 9 (1), 788 (2019).
- Thouta, S., et al. Pharmacological determination of the fractional block of Nav channels required to impair neuronal excitability and ex vivo seizures. Front Cell Neurosci. 16, 964691 (2022).
- Tognolina, M., Monteverdi, A., D'Angelo, E. Discovering microcircuit secrets with multi-spot imaging and electrophysiological recordings: The example of cerebellar network dynamics. Front Cell Neurosci. 16, 805670 (2022).
- Hierlemann, A., Frey, U., Hafizovic, S., Heer, F. Growing cells atop microelectronic chips: Interfacing electrogenic cells in vitro with CMOS-based microelectrode arrays. Proceedings of the IEEE. 99 (2), 252-284 (2011).
- Maccione, A., et al. Experimental investigation on spontaneously active hippocampal cultures recorded by means of high-density MEAs: Analysis of the spatial resolution effects. Front Neuroeng. 3, 4 (2010).
- van Vliet, E., et al. Electrophysiological recording of re-aggregating brain cell cultures on multi-electrode arrays to detect acute neurotoxic effects. Neurotoxicology. 28 (6), 1136-1146 (2007).
- Emery, B. A., et al. Large-scale multimodal recordings on a high-density neurochip: Olfactory bulb and hippocampal networks. Annu Int Conf IEEE Eng Med Biol Soc. 2022, 3111-3114 (2022).
- Veleanu, M., et al. Modified climbing fiber/Purkinje cell synaptic connectivity in the cerebellum of the neonatal phencyclidine model of schizophrenia. Proc Natl Acad Sci U S A. 119 (21), e2122544119 (2022).
- Giansante, G., et al. Neuronal network activity and connectivity are impaired in a conditional knockout mouse model with PCDH19 mosaic expression. Mol Psychiatry. , (2023).
- Dossi, E., Blauwblomme, T., Nabbout, R., Huberfeld, G., Rouach, N. Multi-electrode array recordings of human epileptic postoperative cortical tissue. J Vis Exp. (92), e51870 (2014).
- Hajos, N., et al. Maintaining network activity in submerged hippocampal slices: importance of oxygen supply. Eur J Neurosci. 29 (2), 319-327 (2009).
- Hill, M. R., Greenfield, S. A. The membrane chamber: a new type of in vitro recording chamber. J Neurosci Methods. 195 (1), 15-23 (2011).
- Raimondo, J. V., et al. Methodological standards for in vitro models of epilepsy and epileptic seizures. A TASK1-WG4 report of the AES/ILAE Translational Task Force of the ILAE. Epilepsia. 58 (Suppl 4), 40-52 (2017).
- Hales, C. M., Rolston, J. D., Potter, S. M. How to culture, record and stimulate neuronal networks on micro-electrode arrays (MEAs). J Vis Exp. (39), 2056 (2010).
- Lin, C. H., Lee, J. K., LaBarge, M. A. Fabrication and use of microenvironment microarrays (MEArrays). J Vis Exp. (68), 4152 (2012).
- Panuccio, G., Colombi, I., Chiappalone, M. Recording and modulation of epileptiform activity in rodent brain slices coupled to microelectrode arrays. J Vis Exp. 135, 57548 (2018).
- Patel, C., Muthuswamy, J. High efficiency, site-specific transfection of adherent cells with siRNA using microelectrode arrays (MEA). J Vis Exp. 67, e4415 (2012).
- Ting, J. T., Daigle, T. L., Chen, Q., Feng, G. Acute brain slice methods for adult and aging animals: application of targeted patch clamp analysis and optogenetics. Methods Mol Biol. 1183, 221-242 (2014).
- Papouin, T., Haydon, P. G. Obtaining acute brain slices. Bio Protoc. 8 (2), e2699 (2018).
- Ting, J. T., et al. Preparation of acute brain slices using an optimized N-Methyl-D-glucamine protective recovery method. J Vis Exp. 132, 53825 (2018).
- Van Hoeymissen, E., Philippaert, K., Vennekens, R., Vriens, J., Held, K. Horizontal hippocampal slices of the mouse brain. J Vis Exp. (163), 61753 (2020).
- . 3Brain Available from: https://www.3brain.com/ (2022)
- Bridges, D. C., Tovar, K. R., Wu, B., Hansma, P. K., Kosik, K. S. MEA Viewer: A high-performance interactive application for visualizing electrophysiological data. PLoS One. 13 (2), e0192477 (2018).
- Hawrylycz, M., et al. Inferring cortical function in the mouse visual system through large-scale systems neuroscience. Proc Natl Acad Sci U S A. 113 (27), 7337-7344 (2016).
- Maccione, A., et al. Microelectronics, bioinformatics and neurocomputation for massive neuronal recordings in brain circuits with large scale multielectrode array probes. Brain Res Bull. 119 (Pt B), 118-126 (2015).
- . 3Brain Available from: https://www.3brain.com/products/software/brainwave4 (2022)
- Mahadevan, A. . Xenon LFP Analysis. , (2022).
- Mahadevan, A. . xenon-lfp-analysis github. , (2022).
- Codadu, N. K., et al. Divergent paths to seizure-like events. Physiol Rep. 7 (19), e14226 (2019).
- Kirsch, G. E., Drewe, J. A. Gating-dependent mechanism of 4-aminopyridine block in two related potassium channels. J Gen Physiol. 102 (5), 797-816 (1993).
- Levesque, M., Salami, P., Behr, C., Avoli, M. Temporal lobe epileptiform activity following systemic administration of 4-aminopyridine in rats. Epilepsia. 54 (4), 596-604 (2013).
- Myers, T. L., Gonzalez, O. C., Stein, J. B., Bazhenov, M. Characterizing concentration-dependent neural dynamics of 4-Aminopyridine-induced epileptiform activity. Epilepsy J. 4 (2), 128 (2018).
- Perreault, P., Avoli, M. Physiology and pharmacology of epileptiform activity induced by 4-aminopyridine in rat hippocampal slices. J Neurophysiol. 65 (4), 771-785 (1991).
- Rutecki, P. A., Lebeda, F. J., Johnston, D. 4-Aminopyridine produces epileptiform activity in hippocampus and enhances synaptic excitation and inhibition. J Neurophysiol. 57 (6), 1911-1924 (1987).
- Chen, Y., Chad, J. E., Cannon, R. C., Wheal, H. V. Reduced Mg2+ blockade of synaptically activated N-methyl-D-aspartate receptor-channels in CA1 pyramidal neurons in kainic acid-lesioned rat hippocampus. Neuroscience. 88 (3), 727-739 (1999).
- Fujiwara-Tsukamoto, Y., Isomura, Y., Takada, M. Comparable GABAergic mechanisms of hippocampal seizure-like activity in posttetanic and low-Mg2+ conditions. J Neurophysiol. 95 (3), 2013-2019 (2006).
- Swartzwelder, H. S., Anderson, W. W., Wilson, W. A. Mechanism of electrographic seizure generation in the hippocampal slice in Mg2+-free medium: the role of GABAa inhibition. Epilepsy Res. 2 (4), 239-245 (1988).
- Trevelyan, A. J., Graham, R. T., Parrish, R. R., Codadu, N. K. Synergistic positive feedback mechanisms underlying seizure initiation. Epilepsy Curr. 23 (1), 38-43 (2023).
- Croning, M. D., Haddad, G. G. Comparison of brain slice chamber designs for investigations of oxygen deprivation in vitro. J Neurosci Methods. 81 (1-2), 103-111 (1998).
- Hajos, N., Mody, I. Establishing a physiological environment for visualized in vitro brain slice recordings by increasing oxygen supply and modifying aCSF content. J Neurosci Methods. 183 (2), 107-113 (2009).
- Huang, Y., Williams, J. C., Johnson, S. M. Brain slice on a chip: opportunities and challenges of applying microfluidic technology to intact tissues. Lab Chip. 12 (12), 2103-2117 (2012).
- Andrew, R. D., et al. The critical role of spreading depolarizations in early brain injury: Consensus and contention. Neurocrit Care. 37 (Suppl 1), 83-101 (2022).
- Devonshire, I. M., Dommett, E. J., Grandy, T. H., Halliday, A. C., Greenfield, S. A. Environmental enrichment differentially modifies specific components of sensory-evoked activity in rat barrel cortex as revealed by simultaneous electrophysiological recordings and optical imaging in vivo. Neuroscience. 170 (2), 662-669 (2010).
- Parrish, R. R., Codadu, N. K., Mackenzie-Gray Scott, C., Trevelyan, A. J. Feedforward inhibition ahead of ictal wavefronts is provided by both parvalbumin- and somatostatin-expressing interneurons. J Physiol. 597 (8), 2297-2314 (2019).
- Wang, H., Jing, M., Li, Y. Lighting up the brain: genetically encoded fluorescent sensors for imaging neurotransmitters and neuromodulators. Curr Opin Neurobiol. 50, 171-178 (2018).
- Yaksi, E., Jamali, A., Diaz Verdugo, C., Jurisch-Yaksi, N. Past, present and future of zebrafish in epilepsy research. FEBS J. 288 (24), 7243-7255 (2021).
- He, M. F., et al. Ex vivo calcium imaging for drosophila model of epilepsy. J Vis Exp. 200, 65825 (2023).
- Driscoll, N., et al. Multimodal in vivo recording using transparent graphene microelectrodes illuminates spatiotemporal seizure dynamics at the microscale. Commun Biol. 4 (1), 136 (2021).
- Parrish, R. R., Grady, J., Codadu, N. K., Trevelyan, A. J., Racca, C. Simultaneous profiling of activity patterns in multiple neuronal subclasses. J Neurosci Methods. 303, 16-29 (2018).
- Valderhaug, V. D., et al. Criticality as a measure of developing proteinopathy in engineered human neural networks. bioRxiv. , (2020).
- Carleo, G., Lee, Y. -. S., Secondo, A., Miceli, F., Taglialatela, M. Multi-electrode array (MEASs) to investigate pathogenetic disease mechanisms and pharmacological properties in iPSC-derived neurons modelling neuropsychiatric diseases. , 667-672 (2022).
- Ruz, I. D., Schultz, S. R. Localising and classifying neurons from high density MEA recordings. J Neurosci Methods. 233, 115-128 (2014).
- Franke, F., Natora, M., Boucsein, C., Munk, M. H. J., Obermayer, K. An online spike detection and spike classification algorithm capable of instantaneous resolution of overlapping spikes. J Comput Neurosci. 29 (1-2), 127-148 (2010).
- Vollgraf, R., Obermayer, K. Improved optimal linear filters for the discrimination of multichannel waveform templates for spike-sorting applications. IEEE Signal Processing Letters. 13 (3), 121-124 (2006).
- Muller, J., et al. High-resolution CMOS MEA platform to study neurons at subcellular, cellular, and network levels. Lab Chip. 15 (13), 2767-2780 (2015).
- Mapelli, L., et al. implementation, and functional validation of a new generation of microneedle 3D high-density CMOS multi-electrode array for brain tissue and spheroids. bioRxiv. , (2022).
- Reddy, D. S., Kuruba, R. Experimental models of status epilepticus and neuronal injury for evaluation of therapeutic interventions. Int J Mol Sci. 14 (9), 18284-18318 (2013).
- Parrish, R. R., Trevelyan, A. J. Stress-testing the brain to understand its breaking points. J Physiol. 596 (11), 2033-2034 (2018).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유