Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Hochwertige anfallsähnliche Aktivität aus akuten Hirnschnitten unter Verwendung eines komplementären Metalloxid-Halbleiter-Mikroelektrodenarray-Systems mit hoher Dichte
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Hier skizzieren wir ein Protokoll für die Verwendung komplementärer Metalloxid-Halbleiter-Mikroelektroden-Array-Systeme mit hoher Dichte (CMOS-HD-MEAs) zur Aufzeichnung anfallsähnlicher Aktivität aus ex vivo Hirnschnitten.
Zusammenfassung
Komplementäre Metall-Oxid-Halbleiter-High-Density-Mikroelektroden-Array-Systeme (CMOS-HD-MEA) können die neurophysiologische Aktivität von Zellkulturen und Ex-vivo-Hirnschnitten in bisher unerreichter elektrophysiologischer Detailgenauigkeit aufzeichnen. CMOS-HD-MEAs wurden zunächst optimiert, um die Aktivität hochwertiger neuronaler Einheiten aus Zellkulturen aufzuzeichnen, es wurde aber auch gezeigt, dass sie qualitativ hochwertige Daten aus akuten Netzhaut- und Kleinhirnschnitten liefern. Forscher haben kürzlich CMOS-HD-MEAs verwendet, um lokale Feldpotentiale (LFPs) von akuten, kortikalen Nagetier-Hirnschnitten aufzuzeichnen. Ein LFP von Interesse ist die anfallsähnliche Aktivität. Während viele Anwender kurze, spontane epileptiforme Entladungen mit CMOS-HD-MEAs erzeugt haben, erzeugen nur wenige Anwender zuverlässig eine qualitativ hochwertige anfallsähnliche Aktivität. Viele Faktoren können zu dieser Schwierigkeit beitragen, darunter elektrisches Rauschen, die inkonsistente Art der Erzeugung anfallsähnlicher Aktivität bei der Verwendung von untergetauchten Aufnahmekammern und die Einschränkung, dass 2D-CMOS-MEA-Chips nur von der Oberfläche der Gehirnscheibe aufzeichnen. Die in diesem Protokoll beschriebenen Techniken sollten es den Benutzern ermöglichen, mit einem CMOS-HD-MEA-System konsistent eine qualitativ hochwertige anfallsähnliche Aktivität aus akuten Hirnschnitten zu induzieren und aufzuzeichnen. Darüber hinaus beschreibt dieses Protokoll die richtige Behandlung von CMOS-HD-MEA-Chips, das Management von Lösungen und Gehirnschnitten während des Experimentierens sowie die Wartung der Geräte.
Einleitung
Kommerziell erhältliche HD-MEA-Systeme (High-Density Microelectrode Array), die einen MEA-Chip mit Tausenden von Aufzeichnungspunkten 1,2 und eine MEA-Plattform zur Verstärkung und Digitalisierung der Daten umfassen, sind ein aufstrebendes Werkzeug für die elektrophysiologische Forschung. Diese HD-MEA-Systeme verwenden die komplementäre Metalloxid-Halbleiter-Technologie (CMOS), um elektrophysiologische Daten aus Zellkulturen und ex vivo Hirnschnittpräparaten mit hoher Empfindlichkeit aufzuzeichnen. Diese MEA-Systeme bieten eine beispiellose räumliche und zeitliche Auflösung für die neurophysiologische Forschung durch eine hohe Elektrodendichte und hochwertige Signal-Rausch-Verhältnisse3. Diese Technologie wurde hauptsächlich zur Untersuchung extrazellulärer Aktionspotentiale eingesetzt, kann aber auch hochwertige lokale Feldpotentiale (LFPs) aus verschiedenen neuronalen Hirnschnittpräparaten erfassen 4,5,6,7,8,9,10,11,12,13,14,15 . Durch die oben erwähnte hochauflösende Aufzeichnungsfähigkeit der CMOS-HD-MEA-Systeme können Anwender die elektrophysiologische Aktivität mit hoher räumlicher Genauigkeit verfolgen 16,17,18. Diese Fähigkeit ist besonders relevant für die Verfolgung von Ausbreitungsmustern der Netzwerk-LFPs 5,12,15,19,20,21. Daher können CMOS-HD-MEA-Systeme ein noch nie dagewesenes Verständnis der Ausbreitungsmuster physiologischer und pathologischer Aktivität aus verschiedenen Zellkultur- und Hirnschnittpräparaten liefern. Besonders hervorzuheben ist, dass diese Fähigkeiten von CMOS-HD-MEA-Systemen es Forschern ermöglichen können, Anfallsmuster verschiedener Gehirnregionen gleichzeitig zu vergleichen und zu testen, wie verschiedene antiepileptische Verbindungen diese Muster beeinflussen. Auf diese Weise bietet es eine innovative Methode zur Untersuchung der Iktogenese und der iktalen Ausbreitung und zum Verständnis, wie die Pharmakologie die Aktivität pathologischer Netzwerke stört 7,10,14. Daher können diese neuartigen Fähigkeiten von CMOS-HD-MEA-Systemen erheblich zur Erforschung neurologischer Erkrankungen beitragen und die Wirkstoffforschung unterstützen 5,7,11,22. Unser Ziel ist es, Details zur Verwendung von CMOS-HD-MEA-Systemen zur Untersuchung anfallsähnlicher Aktivität bereitzustellen.
Bei der Verwendung von CMOS-HD-MEA-Systemen zur Untersuchung von LFPs, wie z. B. der epileptiformen Aktivität in akuten Hirnschnitten, können Benutzer mit vielen Herausforderungen konfrontiert werden, darunter schwächendes elektrisches Rauschen, die Aufrechterhaltung der Gesundheit des Schnitts während des Experimentierens und die Erkennung eines Qualitätssignals von einem zweidimensionalen (2D) CMOS-MEA-Chip, der nur von der Oberfläche des Gehirnschnitts aufzeichnet. Dieses Protokoll beschreibt grundlegende Schritte für die ordnungsgemäße Erdung der MEA-Plattform und anderer Geräte, die für Experimente verwendet werden, ein entscheidender Schritt, der möglicherweise eine individuelle Anpassung für jede Laboreinrichtung erfordert. Darüber hinaus diskutieren wir Schritte, die dazu beitragen, den Gehirnschnitt während langer Aufzeichnungen in den getauchten Kammern, die mit CMOS-HD-MEA-Systemen verwendet werden, gesund zu halten 23,24,25. Darüber hinaus verwenden die meisten CMOS-HD-MEA-Systeme im Gegensatz zu den gängigeren elektrophysiologischen Aufzeichnungsmethoden, die aus den Tiefen des Hirnschnitts aufzeichnen, 2D-Chips, die nicht in den Schnitt eindringen. Daher benötigen diese Systeme eine gesunde neuronale äußere Schicht, um den Großteil der aufgezeichneten LFP-Signale zu erzeugen. Eine weitere Herausforderung ist die Visualisierung der riesigen Datenmengen, die von Tausenden von Elektroden erzeugt werden. Um diese Herausforderungen zu meistern, empfehlen wir ein einfaches, aber effektives Protokoll, das die Wahrscheinlichkeit erhöht, eine qualitativ hochwertige epileptiforme Netzwerkaktivität zu erreichen, die sich über den Gehirnschnitt ausbreitet. Wir enthalten auch eine kurze Beschreibung einer öffentlich zugänglichen grafischen Benutzeroberfläche (GUI), die wir mit den zugehörigen Ressourcen entwickelt haben, um die Datenvisualisierungzu unterstützen 10.
Frühere Veröffentlichungen haben verwandte Protokolle für die Verwendung von MEA-Aufzeichnungssystemen bereitgestellt 26,27,28,29. Diese Arbeit zielt jedoch darauf ab, Experimentatoren zu unterstützen, die CMOS-HD-MEA-Systeme mit 2D-Chips verwenden, insbesondere diejenigen, die qualitativ hochwertige epileptiforme Aktivität aus Hirnschnitten untersuchen möchten. Darüber hinaus vergleichen wir zwei der gebräuchlichsten Lösungsmanipulationen zur Induktion anfallsähnlicher Aktivität, nämlich das 0 Mg2+ und das 4-AP-Paradigma, um den Anwendern zu helfen, die am besten geeigneten Krampfmedien für ihre spezifische Anwendung zu identifizieren. Obwohl sich das Protokoll in erster Linie auf die Erzeugung von anfallsähnlicher Aktivität konzentriert, kann es modifiziert werden, um andere elektrophysiologische Phänomene mit Hilfe von Hirnschnitten zu untersuchen.
Access restricted. Please log in or start a trial to view this content.
Protokoll
Verfahren mit Mäusen wurden vom Institutional Animal Care and Use Committee (IACUC) an der Brigham-Young-Universität genehmigt. In den folgenden Experimenten wurden männliche und weibliche (n = 8) C57BL/6-Mäuse im Alter von mindestens P21 verwendet.
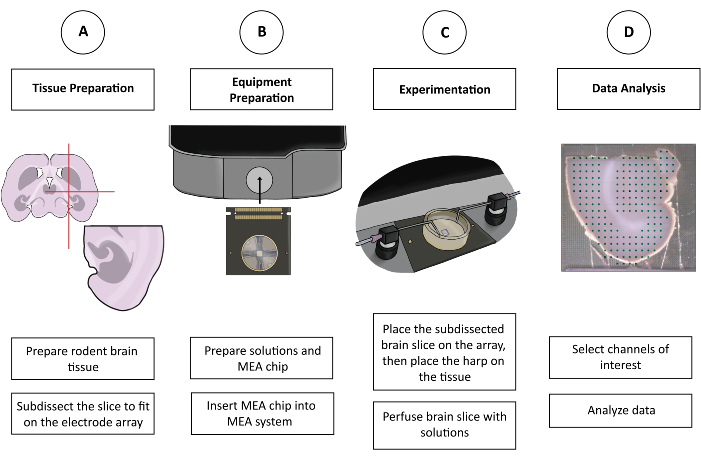
Abbildung 1: Schematische Darstellung von CMOS-HD-MEA-Experimenten. (A) Der Hirnschnitt wird mit der von einem bevorzugten Schnittmethode präpariert und so präpariert, dass er auf den MEA passt. (B) Bereiten Sie die Lösungen und den CMOS-HD-MEA-Chip vor. (C) Die subpräparierte Hirnscheibe wird auf das Elektrodenarray gelegt und in den entsprechenden Lösungen gebadet. (D) Aus den gesammelten Daten werden relevante Kanäle ausgewählt. Die Daten werden dann für die Analyse im bevorzugten Programm des Benutzers vorbereitet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Name | Konzentration (mM) | g/L | ||
| Natriumchlorid (NaCl) | 126 | 7.36 | ||
| Kaliumchlorid (KCl) | 3.5 | 0.261 | ||
| Dihydrogen-Natriumphosphat (NaH2PO4) | 1.26 | 0.151 | ||
| Natriumbicarbonat (NaHCO3) | 26 | 2.18 | ||
| Glukose (C6H12O6) | 10 | 1.80 | ||
| Magnesiumchlorid (MgCl2) | 1 (ab Lager 1 Mio.) | 1 mL | ||
| Calciumchlorid (CaCl2) | 2 (ab 1 M Lagerbestand) | 2 mL | ||
Tabelle 1: Liquorlösung.
1. Lösungen vorbereiten
- Bereiten Sie die experimentellen Lösungen vor.
- Bereiten Sie 1 l künstliche Zerebrospinalflüssigkeit (aCSF) vor (siehe Tabelle 1 für Details).
- Bereiten Sie 1 l prokonvulsive Lösung vor.
HINWEIS: Lösungen, die in diesem Protokoll zur Erzeugung anfallsähnlicher Aktivität verwendet werden, verwenden entweder aCSF mit 100 μM 4-Aminopyridin (4-AP) oder aCSF frei von Magnesiumionen. - Karbogenatisieren Sie alle Lösungen mindestens 10 Minuten vor Gebrauch mit porösen Steinen.
- Besorgen Sie sich ein Becherglas zum Verwerfen von Lösungen.
- 1 l aCSF, 1 l prokonvulsive Lösung und das Becherglas werden auf eine Oberfläche in der Nähe des Perfusionssystems gelegt.
- Vorbereitung der Schneid- und Warmhaltekammerlösungen
- Bereiten Sie 0,5 l aCSF vor, lassen Sie CaCl2 weg und fügen Sie 3 mM MgCl2 (im Gegensatz zu 1 mM) hinzu, um eine einzigartige Schneidlösung zu erhalten. Bewahren Sie diese Lösung für die Verwendung bei der Vorbereitung von Hirnschnitten bei Nagetieren auf, indem Sie sie in der Nähe des Vorbereitungsbereichs für akute Hirnschnitte platzieren.
- Bereiten Sie 0,5 l aCSF vor und legen Sie es in eine Gewebehaltekammer, die zur Aufbewahrung von Scheiben für die Verwendung in Experimenten verwendet wird. Legen Sie poröse Steine in die Haltekammer des Gewebes und karbogenieren Sie die Lösung mindestens 10 Minuten lang, bevor Sie die präparierten Scheiben in die Kammer legen.
2. Vorbereitung von Hirnschnitten von Nagetieren
- Bereiten Sie alle Lösungen wie oben beschrieben vor (siehe Abschnitt 1). Stellen Sie sicher, dass alle Lösungen während des Vorbereitungsprozesses ordnungsgemäß karbogenisiert werden.
HINWEIS: Die Schneidlösung (siehe Abschnitt 1.2) wird empfohlen, aber es können auch andere Schneidlösungen verwendet werden, um hochwertige Hirnschnitte zu erhalten, wie z. B. eine Saccharose-Schneidlösung30. - Verwenden Sie ein vibrierendes Mikrotom, um 350 μm große Hirnschnitte von Nagetieren herzustellen. Die Protokolle für die Verwendung dieser Geräte sind in den Referenzen 30,31,32,33 aufgeführt.
- Bereiten Sie die Hirnschnitte der Nagetiere so vor, dass sie auf den Aufzeichnungselektrodenbereich des MEA-Chips passen (siehe Abbildung 2). Verwenden Sie eine chirurgische Klinge der Größe 10, um die Scheiben zu präparieren, wie in Abbildung 1A und Abbildung 2A gezeigt, indem Sie die Klinge vorsichtig auf der Gehirnscheibe hin und her rollen. Führen Sie die Subdissektion in der Schneidkammer des vibrierenden Mikrotoms durch. Seien Sie vorsichtig, um den subpräparierten Hirnschnitt nicht zu beschädigen.
- Legen Sie die präparierten Scheiben in eine mit aCSF gefüllte Gewebehaltekammer. Stellen Sie sicher, dass die Kammer mindestens 10 Minuten lang karbogenisiert wurde, bevor Sie die präparierten Scheiben hinzufügen.
3. Vorbereitung der Ausrüstung
- Vorbereiten der MEA-Chips und des Systems (Abbildung 2 und Abbildung 3)
HINWEIS: Harte Materialien wie Kunststoffe und Metalle können den MEA-Chip leicht beschädigen, wenn sie den Boden des Chipschachts gewaltsam berühren. Wenn Sie Pipetten verwenden, um die Lösung in die oder aus den Chips zu übertragen, berühren Sie den Boden der Chip-Vertiefung nicht mit der Pipette, insbesondere nicht mit den Referenzelektroden an den Seiten der Chip-Vertiefung und den Aufzeichnungselektroden in der Mitte der Chip-Vertiefung (Abbildung 2C). Fügen Sie stattdessen die Lösung hinzu, indem Sie den Mauszeiger über den Boden des Chipfelds bewegen oder indem Sie die Kunststoffkanten des Chipfelds berühren oder annähern. Um die gesamte Lösung leicht herauszuziehen, kippen Sie den Chip leicht, damit sich die Lösung auf einer Seite der Chip-Vertiefung ansammeln kann, und entfernen Sie sie von der Oberseite der gepoolten Lösung, oder tupfen Sie mit einem antistatischen Tuch leicht auf die Stellen, an denen sich noch die Lösung befindet. Transportieren Sie den Chip nicht, indem Sie den Chip oder die Chipstifte gut festhalten (Abbildung 2C). Die Chip-Vertiefung kann ca. 4 mL Lösung aufnehmen. Füllen Sie die Chip-Vertiefung für die nachfolgenden Schritte mit ca. 2 ml Lösung, sofern nicht anders angegeben.- Bestimmen Sie Transferpipetten für verschiedene Aufgaben, bevor Sie mit der Chipvorbereitung beginnen. Markieren Sie eine Transferpipette für Ethanol, eine für Abfall, eine andere für aCSF und andere für alle verbleibenden Lösungen, um eine unbeabsichtigte Vermischung zu verhindern.
- Füllen Sie die Vertiefung des MEA-Chips mit 190-prozentigem Ethanol, so dass der Boden der Chip-Vertiefung vollständig bedeckt ist (Abbildung 3). Lassen Sie das Ethanol 30-60 s einwirken und entfernen Sie es dann mit einer Wegwerfpipette.
- Füllen Sie die Vertiefung des MEA-Chips mit aCSF und entfernen Sie es mit einer Entwurfpipette, um das restliche Ethanol aus der Chip-Vertiefung zu spülen. Fügen Sie aCSF dreimal hinzu und entfernen Sie es aus der Chip-Vertiefung, wobei Sie die zuvor vorgesehenen Abfall- und aCSF-Pipetten verwenden. Nachdem Sie den Chip dreimal gut gewaschen haben, fügen Sie aCSF hinzu und lassen Sie ihn mindestens 30 s stehen.
HINWEIS: Die Aufzeichnungselektroden weisen das geringste Rauschen durch hydrophobe Wechselwirkungen auf, wenn aCSF nach dem Spülen mit Ethanol und aCSF mindestens 45 Minuten lang in der Kammer verbleibt. - Befeuchten Sie vor dem Andocken des MEA-Chips ein antistatisches Tuch mit 190er Ethanol und wischen Sie damit die Stifte des Chips ab (Abbildung 2C).
- Schieben Sie den MEA-Chip vorsichtig in die MEA-Plattform und aktivieren Sie den Andockmechanismus, um den Chip zu verriegeln.
- Überprüfen Sie die Aufzeichnungs- und Referenzelektroden auf Blasen (Abbildung 2C). Wenn Blasen vorhanden sind, nehmen Sie einen sauberen Pinsel und streichen Sie leicht über die Elektroden, um sie zu entfernen.
- Prüfen Sie den Chip mit der CMOS-HD-MEA-Software34 auf Rauschen und scannen Sie die Falschfarbenkarte visuell auf Blasen, nichtbiologische Schwingungen oder Spitzen, die durch elektrische Störungen verursacht werden. Erden Sie das MEA-System entsprechend, um auftretende Geräusche zu vermeiden.
HINWEIS: Die Einrichtung der Erdung hängt von der Aufnahmeumgebung ab. Für die Experimente in diesem Protokoll wurden die MEA-Plattform und das Perfusionssystem geerdet.
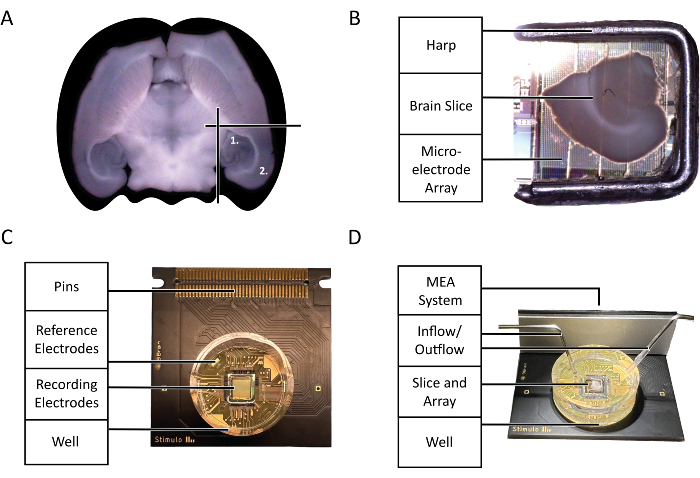
Abbildung 2: Konfigurations- und Technologiediagramme. (A) Diagramm der Auswahl der akuten Maushirnschnitte, die in dem in diesem Protokoll hervorgehobenen Experiment verwendet wurden. (1) Hippocampus-Region (2) Neokortex-Region. (B) Die richtige Platzierung eines akuten Maushirnschnitts und einer Harfe auf dem Mikroelektrodenarray (MEA). (C) Die Anatomie eines 3Brain Accura CMOS-HD-MEA Chips. (D) Die richtige Konfiguration der Perfusionsein- und -auslässe. Der Eingang sollte tief in der Chip-Vertiefung liegen, während sich der Ausgang auf der gegenüberliegenden Seite des Einlasses am oberen Ende der Chip-Vertiefung befinden sollte, um einen konstanten Fluss von frischem, sauerstoffhaltigem aCSF zu gewährleisten. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
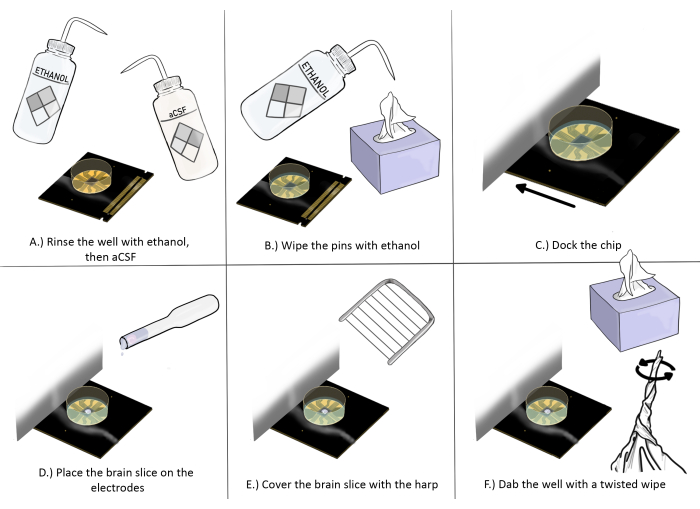
Abbildung 3: Schematische Darstellung der Chipvorbereitung und -platzierung für Hirnschnittexperimente. (A) Spülen Sie den Chip einmal gut mit Ethanol und dann dreimal mit aCSF. (B) Wischen Sie die Stifte mit Ethanol mit einem antistatischen Tuch ab. (C) Docken Sie den Chip an. (D) Platzieren Sie die Gehirnscheibe auf den Elektroden. (E) Platzieren Sie die Harfe auf der Gehirnscheibe (siehe Abbildung 2 für die richtige Platzierung). (F) Tupfen Sie die Ecke der Vertiefung der Aufnahmeelektroden in der Nähe der Gehirnscheibe mit einem verdrehten antistatischen Tuch ab. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
4. Experimentieren
- Platzieren der Gehirnscheibe
- Platzieren Sie eine Platinharfe in einem Wiegeschiff (oder einer anderen sauberen Oberfläche) in der Nähe der MEA-Plattform. Bedecken Sie die Harfe mit etwa 3 ml aCSF, um ihre hydrophoben Tendenzen zu reduzieren.
- Entferne mit einer Schere den dünnen Spitzenteil einer Transferpipette. Schneiden Sie ein Drittel, etwa 1,5 Zoll, der Pipettenspitze ab.
HINWEIS: Diese modifizierte Pipette verengt oder beschädigt die Hirnscheibe während der Entnahme und des Transports nicht. - Entnehmen Sie mit der modifizierten Pipette eine Gehirnscheibe aus der Schlitzhaltekammer. Geben Sie die Gehirnscheibe und die Lösung aus der Pipette vorsichtig in die Chip-Vertiefung. Um die Scheibe richtig zu positionieren, lösen Sie vorsichtig mehr aCSF aus einer Transferpipette, um die Position der Hirnscheibe zu manipulieren, oder verwenden Sie einen weichen Pinsel, um einen Strom in der Lösung zu erzeugen, der die Hirnscheibe auf die Aufzeichnungselektroden drückt. Begrenzen Sie den Kontakt mit den Aufzeichnungselektroden oder der Gehirnscheibe, um Schäden zu minimieren.
- Legen Sie die Harfe mit einer Pinzette vorsichtig mit den Fäden nach unten über die Hirnscheibe, um die Scheibe auf die Aufnahmeelektroden zu drücken. Vermeiden Sie es, den Elektrodenträger mit der Harfe in Berührung zu bringen. Richten Sie die Harfe so aus, dass die Seite ohne Rahmen zur Zuflussnadel zeigt und der Rahmen der Harfe keine der Aufnahmeelektroden berührt (Abbildung 2C, D).
- Nehmen Sie eine Discard-Pipette und entfernen Sie überschüssigen Liquor. Nehmen Sie ein antistatisches Tuch, drehen Sie eine Ecke, um eine Spitze zu bilden, und saugen Sie damit das verbleibende aCSF auf, das die Aufnahmeelektroden umgibt, ohne die Aufnahmeelektroden, die Gehirnscheibe oder die Harfe zu berühren (Abbildung 2).
- Fügen Sie mit einer dafür vorgesehenen aCSF-Pipette schnell so viel carbogenisierten aCSF hinzu, dass die Gehirnscheibe bedeckt ist, etwa 2 ml.
- Wiederholen Sie Schritt 4.1.5. und 4.1.6. noch zwei weitere Male.
- Füllen Sie die Vertiefung mit karbogeniertem aCSF, bis die Vertiefung etwa zu 3/4 gefüllt ist, etwa 3 ml.
- Machen Sie ein Foto des Gehirnschnitts auf dem MEA-Chip mit einem Mikroskop oder einer Kamera. Stellen Sie sicher, dass das Foto eine ausreichend hohe Auflösung hat, um die Ränder des Aufnahmeelektrodenarrays und die Anatomie des Gehirnschnitts zu sehen.
- Ausführen des Experiments
- Bedienung des Perfusionssystems
- Legen Sie die Zu- und Abflussrohre in das mit aCSF gefüllte Becherglas und die Zu- und Abflussnadeln in die Chip-Vertiefung. Platzieren Sie die Zuflussnadel nahe dem Boden der Chip-Vertiefung direkt außerhalb der Aufzeichnungselektroden. Platzieren Sie die Ausflussnadel nahe der Oberseite der Chip-Vertiefung in Richtung Rand, so dass die Flüssigkeit fast bis zum Rand der Chip-Vertiefung aufsteigt, etwa 4 ml, und die Scheibe während des gesamten Experiments untergetaucht ist (Abbildung 2D).
- Stellen Sie den Perfusionszufluss auf 5 mL/min und den Perfusionsausfluss auf 7 mL/min ein.
HINWEIS: Es wird empfohlen, dass die Ausflussrate die Zuflussrate übersteigt, um einen Überlauf der Lösung aus der Chipvertiefung zu vermeiden und einen Lösungsstrom über der Hirnschicht zu erzeugen. - Schalten Sie den Zu- und Abfluss ein. Entfernen Sie die Zuflussnadel aus der Spänevertiefung, bis die Nadel beginnt, Lösung anstelle von Luft auszugeben. Platzieren Sie dann die Nadel wieder in ihrer Position im Chip-Well, wie in Schritt 4.2.1.1 beschrieben.
- Verwenden Sie einen Lösungserhitzer, um die Lösung auf oder nahe der physiologischen Temperatur von etwa 34-37 °C zu halten.
- Lassen Sie den aLiquor 10 min lang über die Hirnscheibe perfundieren. Auf diese Weise kann sich das Slice an die Aufnahmeumgebung anpassen.
- Nach Ablauf von 10 Minuten den Abflussschlauch in den Entwurfbecher bringen. Bewegen Sie dann den Zuflussschlauch in das Becherglas mit der prokonvulsiven Lösung. Lassen Sie das nicht konvulsive aCSF 10 Minuten lang aus dem Perfusionssystem in das Discard-Becherglas spülen.
- Den Abflussschlauch in das Becherglas mit der krampflösenden Lösung geben.
- Lassen Sie die prokonvulsive Lösung zirkulieren, bis das Experiment beendet ist.
- Wenn die Aufzeichnungsdauer des Schnitts 2 Stunden überschreitet, sollten Sie eine andere prokonvulsive Lösung vorbereiten, um den Glukoseverbrauch des aktiven Hirnschnitts auszugleichen.
- Austausch von Gehirnschnitten
- Schalten Sie den Zufluss aus. Schalten Sie den Abfluss danach 10-15 s ab.
- Entferne die Harfe mit einer Pinzette und lege sie auf ein Waagschiffchen oder eine andere Oberfläche.
- Verwenden Sie eine modifizierte Pipette, um die Gehirnscheibe zu extrahieren und zu entsorgen. Berühren Sie nicht die Referenzelektroden oder das Aufzeichnungselektrodenarray (Abbildung 2C).
- Das Zulaufrohr wird in das Becherglas mit der nicht konvulsiven aCSF-Lösung eingeführt. Setzen Sie das Auslaufrohr in das Becherglas ein. Lassen Sie die Zu- und Abflussnadeln in der Chip-Vertiefung. Lassen Sie das Perfusionssystem 10 Minuten lang laufen, um alle Reste der prokonvulsiven Lösung aus der Chip-Vertiefung und dem Perfusionssystem zu entfernen. Beginnen Sie den Experimentierprozess erneut mit einem neuen Brain-Slice (beginnend bei Schritt 4.1).
- Bedienung des Perfusionssystems
- Beenden des Experiments
- Reinigung des Rigs
- Entferne die Harfe, die Hirnscheibe und die Lösung aus der Vertiefung.
- Docken Sie den MEA-Chip ab, legen Sie ihn auf eine saubere Oberfläche und füllen Sie den Chip gut mit entionisiertem Wasser.
- Entsorgen Sie das entionisierte Wasser, um alle Salze zu entfernen, die von den aCSF-Lösungen übrig geblieben sind.
- Füllen Sie die Chipmulde mit einer Transferpipette mit einer Reinigungslösung. Bewegen Sie die Spitze der Transferpipette in die Chipvertiefung und richten Sie sie auf die Aufzeichnungselektroden. Drücken Sie das Reinigungsmittel schnell und wiederholt in die Transferpipette und geben Sie es wieder ab, um das Reinigungsmittel 1 Minute lang kräftig über die Aufzeichnungselektroden zu spülen. Lassen Sie das Waschmittel 5-10 Minuten ruhen.
- Entfernen Sie das Reinigungsmittel und spülen Sie den Chip 4-6 Mal gut mit entionisiertem Wasser ab, um die Reinigungsmittellösung und alle verbleibenden gelösten Stoffe zu entfernen.
- Entfernen Sie das gesamte Wasser aus der Chip-Vertiefung, geben Sie dann ein antistatisches Tuch über den MEA-Chip und lassen Sie ihn über Nacht trocknen.
- Platzieren Sie eine Wägezelle oder eine andere wasserdichte Platte auf der MEA-Aufzeichnungsplattform, wo zuvor der MEA-Chip ruhte. Platzieren Sie die Harfe in der Mitte der Platte und platzieren Sie die Zu- und Abflussnadeln auf beiden Seiten der Harfe.
- Reinigen Sie das Perfusionssystem und die Harfe, indem Sie jeweils 50 ml Wasser, 50 ml 140-prozentiges Ethanol bzw. 200 ml Wasser durch den Schlauch in den Becher leiten.
HINWEIS: Zu diesem Zeitpunkt kann die Perfusionsgeschwindigkeit erhöht werden, um die Reinigungszeit zu verkürzen, aber die Abflussgeschwindigkeit muss immer die Zulaufgeschwindigkeit überschreiten, um eine Überflutung zu vermeiden. - Reinigen Sie die Carbogen-Steine, indem Sie sie in ein Becherglas mit destilliertem Wasser legen und den Carbogen 5 Minuten lang durch den Schlauch schicken. Legen Sie sie über Nacht auf eine trockene, saubere Oberfläche und decken Sie sie ab, um Staubansammlungen zu vermeiden.
- Reinigung des Rigs
5. Datenanalyse
HINWEIS: Es gibt eine Vielzahl von Analysepaketen, die für die Analyse elektrophysiologischer Daten verwendet werden, die von CMOS-HD-MEAs erzeugt werden, darunter BrainWave von 3Brain, Yet Another Spike Sorter (YASS) und benutzerdefinierte Python-Tools 34,35,36,37. Wir extrahierten Daten aus dem BrainWave-Datendateiformat für die Verwendung in der Xenon LFP-Analyseplattform, um die in Abbildung 4 und Abbildung 5 dargestellten Daten zu generieren. Benutzerdefinierter Matlab-Code wurde verwendet, um die Daten in Abbildung 6 zu analysieren. Protokolle für die Xenon LFP-Analyseplattform sind öffentlich verfügbar10. Die folgenden Protokollschritte sind spezifisch für Aufnahmen, die mit Brainwave 438 gemacht wurden; Für andere Systeme verweisen wir auf die unterstützende Dokumentation zu diesen Systemen 34,35,36,37. Im Folgenden finden Sie einen Überblick über die Analyseschritte, die zur Erzeugung der Daten mit diesem Protokoll unternommen wurden. Ausführliche Informationen zum Exportieren, Visualisieren und Analysieren von Daten, einschließlich Tutorial-Videos und aller relevanten Codedateien, finden Sie unter39.
- Exportieren Sie die Aufzeichnungsdatei zur Analyse.
- Erstellen Sie einen Ordner für die Datenaufzeichnungsdatei(en), die heruntergerechnet werden sollen.
- Schneiden Sie das Bild, das in Schritt 4.1.9 aufgenommen wurde, so zu, dass nur das Aufnahmearray mit den Gehirnschnitten enthalten ist.
- Öffnen Sie die ExportToHDF5-ChannelSelection.py Datei (diese Codedatei kann auf GitHub unter xenon-lfp-analysis/code-files/3Brain-processing40 aufgerufen werden), kopieren Sie die oben im Fenster aufgeführte URL und fügen Sie sie in einen Internetbrowser ein.
- Geben Sie den Dateipfad für die Datenaufzeichnungsdatei ein und laden Sie das zugeschnittene Bild des Arrays hoch, das mit der Aufzeichnung verknüpft ist.
- Verwenden Sie unter "Kanäle für den Export auswählen" das Lasso-Auswahlwerkzeug, um die hippokampalen und neokortikalen Regionen auszuwählen, die auf dem zugeschnittenen Bild des Arrays angezeigt werden.
- Legen Sie sowohl die Anzahl der zu überspringenden Zeilen als auch die Anzahl der zu überspringenden Spalten auf 2 fest.
HINWEIS: Aufnahmedateien sind oft groß; Wenn es für die gewünschte Analyse angemessen ist, können die exportierten Daten heruntergerechnet werden. Es gibt Optionen zum räumlichen (durch Überspringen von Zeilen und Spalten von Aufzeichnungselektroden) und zeitlichen (durch Downsampling auf eine niedrigere Abtastfrequenz) zum Downsampling. In der Analyse werden alle 2 Zeilen und 2 Spalten übersprungen. Dies ermöglicht ein räumliches Downsampling der Daten, ohne auf bestimmte Kanäle verzerrt zu sein. Jeder Kanal ist weit genug voneinander entfernt, dass die Unterschiede in der Aktivität zwischen ihnen erheblich sein können. Daher wird keine Kanalmittelung durchgeführt. Beim Überspringen von Zeilen und Spalten der Elektroden werden die Daten von nicht ausgewählten Kanälen beim Exportieren der Ausgabedatei nicht berücksichtigt. - Stellen Sie die Downsampling-Frequenz auf 300 Hz ein.
- Klicken Sie auf die Schaltfläche Kanäle exportieren , um die Kanalauswahldatei zu generieren.
HINWEIS: Die Software speichert die Kanalauswahldatei automatisch in dem Ordner, in dem die Datenaufzeichnungsdatei gespeichert ist. - Öffnen Sie die ExportToHDF5-ExtractDownsample.py Datei (auch auf GitHub unter xenon-lfp-analysis/code-files/3brain-processing40), fügen Sie den Dateipfad des Ordners ein, der die Kanalauswahldatei(en) und die Datenaufzeichnungsdatei(en) enthält, und drücken Sie die Eingabetaste auf der Tastatur, um die heruntergerechnete Version der Datenaufzeichnungsdatei zu generieren.
- Nutzung der Analyseplattform
- Geben Sie in der Eingabeaufforderung des Computers run_lfp_analysis ein und drücken Sie die Eingabetaste, um die Analyseplattform zu öffnen.
- Geben Sie die Dateipfade des Bildes und der heruntergerechneten Aufnahmedatei ein und klicken Sie auf Senden.
- Wählen Sie unter Zeitbereich für die Analyse auswählen die gesamte Aufzeichnung aus.
- Markieren Sie unter Select Channels for Plots den Hippocampus als Gruppe-1 und den Neokortex als Gruppe-2.
- Legen Sie unter Kanal-Raster den Schwellenwert auf 0,06 mV und die Zeitdauer auf 0,02 s fest.
- Klicken Sie auf Einstellungen anwenden und Raster generieren.
- Verwenden Sie das Rasterdiagramm, um verschiedene Kanäle zu erkunden, die interessante Aktivitätsmuster aufweisen.
HINWEIS: In Abbildung 4 finden Sie Beispiele für eine qualitativ hochwertige anfallsähnliche Aktivität (Abbildung 4A-D) im Vergleich zu einer suboptimalen Aktivität (Abbildung 4E-F).
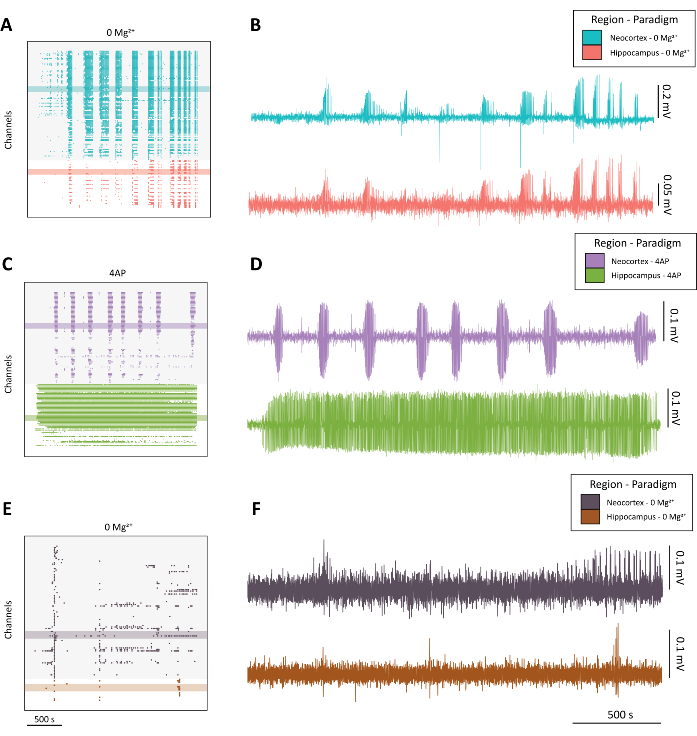
Abbildung 4: Beispiel für die Entwicklung der epileptiformen Aktivität aus den 0 Mg2+ und 4-AP Paradigmen. (A) Beispiel für ein Rasterdiagramm aus der Anwendung von aCSF mit 0 Mg2+ über ca. 40 min. (B) Beispiel für elektrophysiologische Spuren aus dem Neokortex (blau) und dem Hippocampus (rot), die die epileptiforme Aktivität aus dem 0 Mg2+ Paradigma zeigen. (C) Beispiel für ein Rasterdiagramm aus der Anwendung 100 mM 4-AP über ca. 40 min. (D) Beispiel für elektrophysiologische Spuren aus dem Neokortex (violett) und dem Hippocampus (grün), die die epileptiforme Aktivität aus der Anwendung von 4-AP zeigen. (E) Beispiel für ein Rasterdiagramm aus der Anwendung von aCSF mit 0 Mg2+ über ca. 40 Minuten, das eine Bursting-Aktivität im Gegensatz zu einer anfallsähnlichen Aktivität zeigt, wie sie in den anderen repräsentativen Spuren zu finden ist. (F) Beispiel für elektrophysiologische Spuren aus dem Neokortex (dunkelviolett) und dem Hippocampus (Rost), die eine suboptimale Aktivität aus dem 0 Mg2+ Paradigma zeigen, die zum Vergleich mit der Qualität der anfallsähnlichen Aktivität in B und D gedacht ist. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
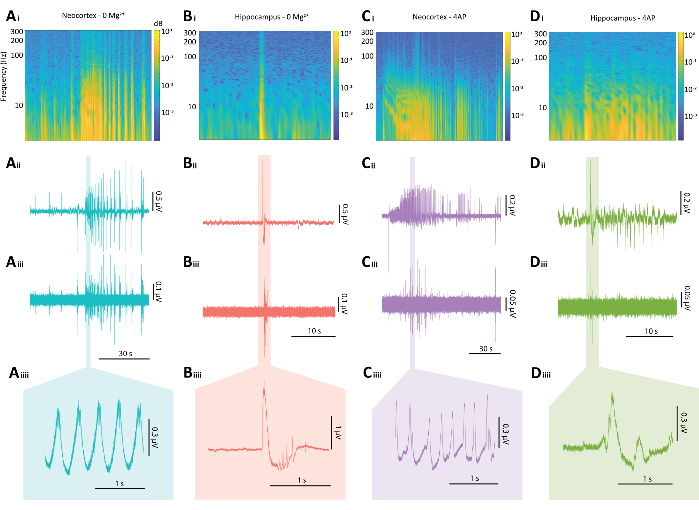
Abbildung 5: Repräsentative Ergebnisse epileptiformer Entladungen sowohl aus dem 0 Mg2+ als auch aus dem 4-AP-Paradigma. (A) Beispieldiagramme eines typischen neokortikalen anfallsähnlichen Ereignisses, das durch das 0 Mg2+ Paradigma induziert wird, einschließlich (Ai) eines Spektrogramms aus einem anfallsähnlichen Ereignis, (Aii) der zugehörigen elektrophysiologischen Spur, (Aiii) eines 80 Hz-Hochpassfilters, der auf die Spur von Aii angewendet wurde, (Aiiii) und eines vergrößerten Abschnitts der Spur von Aii. (B) Beispieldiagramme eines typischen hippokampalen epileptiformen Ausbruchs, der durch das 0 Mg2+ -Paradigma induziert wurde, einschließlich (Bi) eines Spektrogramms des epileptiformen Ausbruchs, (Bii) der zugehörigen elektrophysiologischen Spur, (Biii) eines 80-Hz-Hochpassfilters, der auf die Spur von Bii angewendet wurde, (Biiii) und eines vergrößerten Schnitts der Spur von Bii, (C) Beispieldiagramme eines typischen neokortikalen anfallsähnlichen Ereignisses, das durch das 4-AP-Paradigma induziert wird, einschließlich (Ci) ein Spektrogramm der epileptiformen Aktivität, (Cii) die zugehörige elektrophysiologische Spur, (Ciii) ein 80-Hz-Hochpassfilter, der auf die Spur von Cii aufgebracht wurde, (Ciiii) und ein vergrößerter Ausschnitt der Spur von Cii, (D) Beispieldiagramme eines hippokampalen epileptiformen Ausbruchs unter dem 4-AP-Paradigma, einschließlich (Di) eines Spektrogramms der epileptiformen Aktivität, (Dii) der zugehörigen elektrophysiologischen Spur, (Diii) ein 80-Hz-Hochpassfilter, der auf die Spur von Dii aufgebracht wird, (Diiii) und ein vergrößerter Ausschnitt der Spur von Dii. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
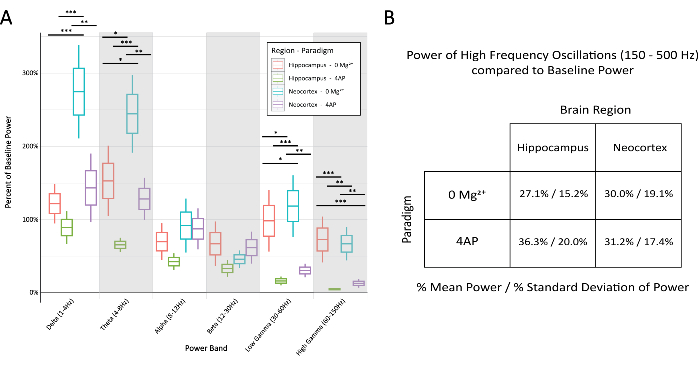
Abbildung 6: Vergleich des Prozentsatzes der Ausgangsleistung in den verschiedenen Banden über Paradigma und Hirnregion hinweg während stereotyper epileptiformer Entladungen. (A) Die Leistung während epileptiformer Entladungen unterschied sich signifikant zwischen den Paradigmen und Hirnregionen für die meisten Frequenzbänder (2-Wege-ANOVA mit Tukey-Test, *P < 0,05, **P < 0,001, ***P < 0,0001). Die mittlere Linie für jedes Kästchen stellt den Mittelwert dar, die Ränder des Kästchens ±1 Standardfehler des Mittelwerts (SEM) und die äußersten Linien ±2 SEM. (B) Sowohl Paradigmen als auch Hirnregionen zeigten eine begrenzte Leistung in Banden, die mit hochfrequenter Aktivität über 150 Hz zusammenhängen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Wie es bei der Visualisierung von Aktivitäten aus vielen Kanälen 1,4,5,10 üblich ist, finden wir es vorteilhaft, zunächst ein Rasterdiagramm der Daten zu erstellen, die wir mit dem CMOS-HD-MEA erfassen (Abbildung 4A,C,E). Dieses Diagramm kann eine Vogelperspektive der Aktivität in allen Aufzei...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Dieses Protokoll enthält spezifische Richtlinien für das Management von akuten Hirnschnitten, die sich mit häufigen Problemen befassen, mit denen CMOS-HD-MEA-Benutzer konfrontiert sind, nämlich die Geräuschentwicklung unter dem Hirnschnitt und die Aufrechterhaltung einer gesunden Umgebung für den Hirnschnitt. Die Rauschentwicklung unter dem Slice tritt auf, wenn der Slice nicht ordnungsgemäß am Array befestigt ist. Wird die Hirnscheibe nicht ausreichend verklebt, können sich unt...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren erklären, dass mit dieser Forschungsstudie keine Interessenkonflikte verbunden sind.
Danksagungen
Die Autoren danken ehemaligen und aktuellen Mitgliedern des Parrish-Labors für ihre Bearbeitung dieses Manuskripts. Wir möchten uns auch bei Alessandro Maccione von 3Brain für sein Feedback zu dieser Arbeit bedanken. Diese Arbeit wurde durch einen AES/EF Junior Investigator Award und durch die Brigham Young University Colleges of Life Sciences und of Physical and Mathematical Sciences finanziert.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| 2D Workbench | Cloudray | LM04CLLD26B | |
| 4-Aminopyridine | Sigma-Aldrich | 275875 | |
| Accura Chip | 3Brain | Accura HD-MEA | CMOS-HD-MEA chip |
| Agarose | Thermo Fisher Scientific | BP160-100 | |
| Vibration isolation table | Kinetic Systems | 91010124 | |
| Beaker for the slice holding chamber, 270 mL | VWR | 10754-772 | |
| BioCam | 3Brain | BioCAM DupleX | CMOS-HD-MEA platform |
| Brainwave Software | 3Brain | Version 4 | CMOS-HD-MEA software |
| Calcium Chloride | Thermo Fisher Scientific | BP510-500 | |
| Carbogen | Airgas | X02OX95C2003102 | |
| Carbogen | Airgas | 12005 | |
| Carbogen Stones | Supelco | 59277 | |
| Compresstome | Precissionary | VF-300-0Z | |
| Computer | Dell | Precission3650 | |
| Crocodile Clip Grounding Cables | JWQIDI | B06WGZG17W | |
| Detergent | Metrex | 10-4100-0000 | |
| D-Glucose | Macron Fine Chemicals | 4912-12 | |
| Dihydrogen Sodium Phosphate | Thermo Fisher Scientific | BP329-500 | |
| DinoCam | Dino-Lite | AM73915MZTL | |
| Ethanol | Thermo Fisher Scientific | A407P-4 | |
| Forceps | Fine Science Tools | 11980-13 | |
| Hot plate | Thermo Fisher Scientific | SP88857200 | |
| Ice Machine | Hoshizaki | F801MWH | |
| Inflow and outflow needles | Jensen Global | JG 18-3.0X | |
| Inline Solution Heater | Warner Instruments | SH-27B | |
| Isofluorine | Dechra | 08PB-STE22002-0122 | |
| Kim Wipes | Thermo Fisher Scientific | 06-666 | |
| Magnesium Chloride | Thermo Fisher Scientific | FLM33500 | |
| Micropipets | Gilson | F144069 | |
| Mili-Q Water Filter | Mili-Q | ZR0Q008WW | |
| Paintbrush | Daler Rowney | AF85 Round: 0 | |
| Paper Filter | Whatman | EW-06648-24 | |
| Parafilm | American National Can | PM996 | |
| Perfusion System | Multi Channel System | PPS2 | |
| Pipetor | Thermo Fisher Scientific | FB14955202 | |
| Platinum Harp | 3Brain | 3Brain | |
| Potassium Chloride | Thermo Fisher Scientific | P330-3 | |
| Razor blade | Personna | BP9020 | |
| Scale | Metter Toledo | AB204 | |
| Scissors | Solingen | 92008 | |
| Slice Holding Chamber | Custom | Custom | Custom 3D Printer Design, available upon request |
| Sodium Bicarbonate | Macron Fine Chemicals | 7412-06 | |
| Sodium Chloride | Thermo Fisher Scientific | S271-3 | |
| Temperature Control Box | Warner Instruments | TC344B | |
| Transfer Pipettes | Genesee Scientific | 30-200 | |
| Tubing | Tygon | B-44-3 TPE | |
| Vibratome VZ-300 | Precissionary | VF-00-VM-NC | |
| Weigh Boat | Electron Microscopy Sciences | 70040 |
Referenzen
- Obien, M. E. J., Frey, U. Large-scale, high-resolution microelectrode arrays for interrogation of neurons and networks. Adv Neurobiol. 22, 83-123 (2019).
- Schroter, M., et al. Functional imaging of brain organoids using high-density microelectrode arrays. MRS Bull. 47 (6), 530-544 (2022).
- Miccoli, B., et al. High-density electrical recording and impedance imaging with a multi-modal CMOS multi-electrode array chip. Front Neurosci. 13, 641(2019).
- Emery, B. A., Hu, X., Khanzada, S., Kempermann, G., Amin, H. High-resolution CMOS-based biosensor for assessing hippocampal circuit dynamics in experience-dependent plasticity. Biosens Bioelectron. 237, 115471(2023).
- Ferrea, E., et al. high-resolution electrophysiological imaging of field potentials in brain slices with microelectronic multielectrode arrays. Front Neural Circuits. 6, 80(2012).
- Gagliano, G., et al. Non-linear frequency dependence of neurovascular coupling in the cerebellar cortex implies vasodilation-vasoconstriction competition. Cells. 11 (6), 1047(2022).
- Goodchild, S. J., et al. Molecular pharmacology of selective Na(V)1.6 and dual Na(V)1.6/Na(V)1.2 channel inhibitors that suppress excitatory neuronal activity ex vivo. ACS Chem Neurosci. 15 (6), 1169-1184 (2024).
- Hu, X., Khanzada, S., Klutsch, D., Calegari, F., Amin, H. Implementation of biohybrid olfactory bulb on a high-density CMOS-chip to reveal large-scale spatiotemporal circuit information. Biosens Bioelectron. 198, 113834(2022).
- Kim, S., et al. Alteration of neural network and hippocampal slice activation through exosomes derived from 5XFAD nasal lavage fluid. Int J Mol Sci. 24 (18), 14064(2023).
- Mahadevan, A., Codadu, N. K., Parrish, R. R. Xenon LFP analysis platform is a novel graphical user interface for analysis of local field potential from large-scale MEA recordings. Front Neurosci. 16, 904931(2022).
- Medrihan, L., Ferrea, E., Greco, B., Baldelli, P., Benfenati, F. Asynchronous GABA release is a key determinant of tonic inhibition and controls neuronal excitability: A study in the synapsin II-/- mouse. Cereb Cortex. 25 (10), 3356-3368 (2015).
- Monteverdi, A., Di Domenico, D., D'Angelo, E., Mapelli, L. Anisotropy and frequency dependence of signal propagation in the cerebellar circuit revealed by high-density multielectrode array recordings. Biomedicines. 11 (5), 1475(2023).
- Obien, M. E. J., Hierlemann, A., Frey, U. Accurate signal-source localization in brain slices by means of high-density microelectrode arrays. Sci Rep. 9 (1), 788(2019).
- Thouta, S., et al. Pharmacological determination of the fractional block of Nav channels required to impair neuronal excitability and ex vivo seizures. Front Cell Neurosci. 16, 964691(2022).
- Tognolina, M., Monteverdi, A., D'Angelo, E. Discovering microcircuit secrets with multi-spot imaging and electrophysiological recordings: The example of cerebellar network dynamics. Front Cell Neurosci. 16, 805670(2022).
- Hierlemann, A., Frey, U., Hafizovic, S., Heer, F. Growing cells atop microelectronic chips: Interfacing electrogenic cells in vitro with CMOS-based microelectrode arrays. Proceedings of the IEEE. 99 (2), 252-284 (2011).
- Maccione, A., et al. Experimental investigation on spontaneously active hippocampal cultures recorded by means of high-density MEAs: Analysis of the spatial resolution effects. Front Neuroeng. 3, 4(2010).
- van Vliet, E., et al. Electrophysiological recording of re-aggregating brain cell cultures on multi-electrode arrays to detect acute neurotoxic effects. Neurotoxicology. 28 (6), 1136-1146 (2007).
- Emery, B. A., et al. Large-scale multimodal recordings on a high-density neurochip: Olfactory bulb and hippocampal networks. Annu Int Conf IEEE Eng Med Biol Soc. 2022, 3111-3114 (2022).
- Veleanu, M., et al. Modified climbing fiber/Purkinje cell synaptic connectivity in the cerebellum of the neonatal phencyclidine model of schizophrenia. Proc Natl Acad Sci U S A. 119 (21), e2122544119(2022).
- Giansante, G., et al. Neuronal network activity and connectivity are impaired in a conditional knockout mouse model with PCDH19 mosaic expression. Mol Psychiatry. , (2023).
- Dossi, E., Blauwblomme, T., Nabbout, R., Huberfeld, G., Rouach, N. Multi-electrode array recordings of human epileptic postoperative cortical tissue. J Vis Exp. (92), e51870(2014).
- Hajos, N., et al. Maintaining network activity in submerged hippocampal slices: importance of oxygen supply. Eur J Neurosci. 29 (2), 319-327 (2009).
- Hill, M. R., Greenfield, S. A. The membrane chamber: a new type of in vitro recording chamber. J Neurosci Methods. 195 (1), 15-23 (2011).
- Raimondo, J. V., et al. Methodological standards for in vitro models of epilepsy and epileptic seizures. A TASK1-WG4 report of the AES/ILAE Translational Task Force of the ILAE. Epilepsia. 58 (Suppl 4), 40-52 (2017).
- Hales, C. M., Rolston, J. D., Potter, S. M. How to culture, record and stimulate neuronal networks on micro-electrode arrays (MEAs). J Vis Exp. (39), 2056(2010).
- Lin, C. H., Lee, J. K., LaBarge, M. A. Fabrication and use of microenvironment microarrays (MEArrays). J Vis Exp. (68), 4152(2012).
- Panuccio, G., Colombi, I., Chiappalone, M. Recording and modulation of epileptiform activity in rodent brain slices coupled to microelectrode arrays. J Vis Exp. 135, 57548(2018).
- Patel, C., Muthuswamy, J. High efficiency, site-specific transfection of adherent cells with siRNA using microelectrode arrays (MEA). J Vis Exp. 67, e4415(2012).
- Ting, J. T., Daigle, T. L., Chen, Q., Feng, G. Acute brain slice methods for adult and aging animals: application of targeted patch clamp analysis and optogenetics. Methods Mol Biol. 1183, 221-242 (2014).
- Papouin, T., Haydon, P. G. Obtaining acute brain slices. Bio Protoc. 8 (2), e2699(2018).
- Ting, J. T., et al. Preparation of acute brain slices using an optimized N-Methyl-D-glucamine protective recovery method. J Vis Exp. 132, 53825(2018).
- Van Hoeymissen, E., Philippaert, K., Vennekens, R., Vriens, J., Held, K. Horizontal hippocampal slices of the mouse brain. J Vis Exp. (163), 61753(2020).
- 3Brain. , Available from: https://www.3brain.com/ (2022).
- Bridges, D. C., Tovar, K. R., Wu, B., Hansma, P. K., Kosik, K. S. MEA Viewer: A high-performance interactive application for visualizing electrophysiological data. PLoS One. 13 (2), e0192477(2018).
- Hawrylycz, M., et al. Inferring cortical function in the mouse visual system through large-scale systems neuroscience. Proc Natl Acad Sci U S A. 113 (27), 7337-7344 (2016).
- Maccione, A., et al. Microelectronics, bioinformatics and neurocomputation for massive neuronal recordings in brain circuits with large scale multielectrode array probes. Brain Res Bull. 119 (Pt B), 118-126 (2015).
- 3Brain. , BrainWave 4 https://www.3brain.com/products/software/brainwave4 (2022).
- Mahadevan, A. Xenon LFP Analysis. , https://xenon-lfp-analysis.readthedocs.io/en/latest/Installation.html (2022).
- Mahadevan, A. xenon-lfp-analysis github. , https://github.com/MicroBrew09/xenon-lfp-analysis (2022).
- Codadu, N. K., et al. Divergent paths to seizure-like events. Physiol Rep. 7 (19), e14226(2019).
- Kirsch, G. E., Drewe, J. A. Gating-dependent mechanism of 4-aminopyridine block in two related potassium channels. J Gen Physiol. 102 (5), 797-816 (1993).
- Levesque, M., Salami, P., Behr, C., Avoli, M. Temporal lobe epileptiform activity following systemic administration of 4-aminopyridine in rats. Epilepsia. 54 (4), 596-604 (2013).
- Myers, T. L., Gonzalez, O. C., Stein, J. B., Bazhenov, M. Characterizing concentration-dependent neural dynamics of 4-Aminopyridine-induced epileptiform activity. Epilepsy J. 4 (2), 128(2018).
- Perreault, P., Avoli, M. Physiology and pharmacology of epileptiform activity induced by 4-aminopyridine in rat hippocampal slices. J Neurophysiol. 65 (4), 771-785 (1991).
- Rutecki, P. A., Lebeda, F. J., Johnston, D. 4-Aminopyridine produces epileptiform activity in hippocampus and enhances synaptic excitation and inhibition. J Neurophysiol. 57 (6), 1911-1924 (1987).
- Chen, Y., Chad, J. E., Cannon, R. C., Wheal, H. V. Reduced Mg2+ blockade of synaptically activated N-methyl-D-aspartate receptor-channels in CA1 pyramidal neurons in kainic acid-lesioned rat hippocampus. Neuroscience. 88 (3), 727-739 (1999).
- Fujiwara-Tsukamoto, Y., Isomura, Y., Takada, M. Comparable GABAergic mechanisms of hippocampal seizure-like activity in posttetanic and low-Mg2+ conditions. J Neurophysiol. 95 (3), 2013-2019 (2006).
- Swartzwelder, H. S., Anderson, W. W., Wilson, W. A. Mechanism of electrographic seizure generation in the hippocampal slice in Mg2+-free medium: the role of GABAa inhibition. Epilepsy Res. 2 (4), 239-245 (1988).
- Trevelyan, A. J., Graham, R. T., Parrish, R. R., Codadu, N. K. Synergistic positive feedback mechanisms underlying seizure initiation. Epilepsy Curr. 23 (1), 38-43 (2023).
- Croning, M. D., Haddad, G. G. Comparison of brain slice chamber designs for investigations of oxygen deprivation in vitro. J Neurosci Methods. 81 (1-2), 103-111 (1998).
- Hajos, N., Mody, I. Establishing a physiological environment for visualized in vitro brain slice recordings by increasing oxygen supply and modifying aCSF content. J Neurosci Methods. 183 (2), 107-113 (2009).
- Huang, Y., Williams, J. C., Johnson, S. M. Brain slice on a chip: opportunities and challenges of applying microfluidic technology to intact tissues. Lab Chip. 12 (12), 2103-2117 (2012).
- Andrew, R. D., et al. The critical role of spreading depolarizations in early brain injury: Consensus and contention. Neurocrit Care. 37 (Suppl 1), 83-101 (2022).
- Devonshire, I. M., Dommett, E. J., Grandy, T. H., Halliday, A. C., Greenfield, S. A. Environmental enrichment differentially modifies specific components of sensory-evoked activity in rat barrel cortex as revealed by simultaneous electrophysiological recordings and optical imaging in vivo. Neuroscience. 170 (2), 662-669 (2010).
- Parrish, R. R., Codadu, N. K., Mackenzie-Gray Scott, C., Trevelyan, A. J. Feedforward inhibition ahead of ictal wavefronts is provided by both parvalbumin- and somatostatin-expressing interneurons. J Physiol. 597 (8), 2297-2314 (2019).
- Wang, H., Jing, M., Li, Y. Lighting up the brain: genetically encoded fluorescent sensors for imaging neurotransmitters and neuromodulators. Curr Opin Neurobiol. 50, 171-178 (2018).
- Yaksi, E., Jamali, A., Diaz Verdugo, C., Jurisch-Yaksi, N. Past, present and future of zebrafish in epilepsy research. FEBS J. 288 (24), 7243-7255 (2021).
- He, M. F., et al. Ex vivo calcium imaging for drosophila model of epilepsy. J Vis Exp. 200, 65825(2023).
- Driscoll, N., et al. Multimodal in vivo recording using transparent graphene microelectrodes illuminates spatiotemporal seizure dynamics at the microscale. Commun Biol. 4 (1), 136(2021).
- Parrish, R. R., Grady, J., Codadu, N. K., Trevelyan, A. J., Racca, C. Simultaneous profiling of activity patterns in multiple neuronal subclasses. J Neurosci Methods. 303, 16-29 (2018).
- Valderhaug, V. D., et al. Criticality as a measure of developing proteinopathy in engineered human neural networks. bioRxiv. , (2020).
- Multi-electrode array (MEASs) to investigate pathogenetic disease mechanisms and pharmacological properties in iPSC-derived neurons modelling neuropsychiatric diseases. Carleo, G., Lee, Y. -S., Secondo, A., Miceli, F., Taglialatela, M. 2022 IEEE International Conference on Metrology for Extended Reality, Artificial Intelligence and Neural Engineering (MetroXRAINE), , Rome, Italy. 667-672 (2022).
- Ruz, I. D., Schultz, S. R. Localising and classifying neurons from high density MEA recordings. J Neurosci Methods. 233, 115-128 (2014).
- Franke, F., Natora, M., Boucsein, C., Munk, M. H. J., Obermayer, K. An online spike detection and spike classification algorithm capable of instantaneous resolution of overlapping spikes. J Comput Neurosci. 29 (1-2), 127-148 (2010).
- Vollgraf, R., Obermayer, K. Improved optimal linear filters for the discrimination of multichannel waveform templates for spike-sorting applications. IEEE Signal Processing Letters. 13 (3), 121-124 (2006).
- Muller, J., et al. High-resolution CMOS MEA platform to study neurons at subcellular, cellular, and network levels. Lab Chip. 15 (13), 2767-2780 (2015).
- Mapelli, L., et al. implementation, and functional validation of a new generation of microneedle 3D high-density CMOS multi-electrode array for brain tissue and spheroids. bioRxiv. , (2022).
- Reddy, D. S., Kuruba, R. Experimental models of status epilepticus and neuronal injury for evaluation of therapeutic interventions. Int J Mol Sci. 14 (9), 18284-18318 (2013).
- Parrish, R. R., Trevelyan, A. J. Stress-testing the brain to understand its breaking points. J Physiol. 596 (11), 2033-2034 (2018).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten