È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Attività simile a una crisi epilettica di alta qualità da fette cerebrali acute utilizzando un sistema di array di microelettrodi ad alta densità a semiconduttore di ossido di metallo complementare
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Qui, descriviamo un protocollo per l'utilizzo di sistemi di array di microelettrodi ad alta densità di semiconduttori a ossido di metallo complementare (CMOS-HD-MEAs) per registrare l'attività simile a un sequestro da fette di cervello ex vivo .
Abstract
I sistemi di array di microelettrodi ad alta densità (CMOS-HD-MEA) complementari a base di metallo-ossido-semiconduttore possono registrare l'attività neurofisiologica da colture cellulari e fette di cervello ex vivo con un dettaglio elettrofisiologico senza precedenti. I CMOS-HD-MEA sono stati inizialmente ottimizzati per registrare l'attività delle unità neuronali di alta qualità da colture cellulari, ma hanno anche dimostrato di produrre dati di qualità da sezioni acute di retina e cerebellari. I ricercatori hanno recentemente utilizzato CMOS-HD-MEA per registrare i potenziali di campo locale (LFP) da fette di cervello di roditori corticali acuti. Un LFP di interesse è l'attività simile alle convulsioni. Mentre molti utenti hanno prodotto scariche epilettiformi brevi e spontanee utilizzando CMOS-HD-MEA, pochi utenti producono in modo affidabile un'attività simile a una crisi epilettica. Molti fattori possono contribuire a questa difficoltà, tra cui il rumore elettrico, la natura incoerente della produzione di attività simile a una crisi epilettica quando si utilizzano camere di registrazione sommerse e la limitazione che i chip CMOS-MEA 2D registrano solo dalla superficie della fetta cerebrale. Le tecniche descritte in questo protocollo dovrebbero consentire agli utenti di indurre e registrare costantemente un'attività simile a una crisi epilettica di alta qualità da fette cerebrali acute con un sistema CMOS-HD-MEA. Inoltre, questo protocollo delinea il corretto trattamento dei chip CMOS-HD-MEA, la gestione delle soluzioni e delle fette di cervello durante la sperimentazione e la manutenzione delle apparecchiature.
Introduzione
I sistemi HD-MEA (High-Density Microelectrode Array) disponibili in commercio, che includono un chip MEA con migliaia di punti di registrazione 1,2 e una piattaforma MEA per amplificare e digitalizzare i dati, sono uno strumento emergente per la ricerca elettrofisiologica. Questi sistemi HD-MEA utilizzano la tecnologia CMOS (Complementary Metal-Oxide-Semiconductor) per registrare dati elettrofisiologici con elevata sensibilità da colture cellulari e preparazioni di fette di cervello ex vivo. Questi sistemi MEA offrono una risoluzione spaziale e temporale senza precedenti alla ricerca neurofisiologica grazie all'elevata densità di elettrodi e ai rapporti segnale/rumoredi qualità 3. Questa tecnologia è stata utilizzata principalmente per studiare i potenziali d'azione extracellulari, ma può anche catturare potenziali di campo locale (LFP) di alta qualità da varie preparazioni di fette di cervello neuronale 4,5,6,7,8,9,10,11,12,13,14,15 . Grazie alla già citata capacità di registrazione ad alta risoluzione dei sistemi CMOS-HD-MEA, gli utenti possono monitorare l'attività elettrofisiologica con grande precisione spaziale 16,17,18. Questa capacità è particolarmente rilevante per il monitoraggio dei modelli di propagazione delle LFP di rete 5,12,15,19,20,21. Pertanto, i sistemi CMOS-HD-MEA possono fornire una comprensione senza precedenti dei modelli di propagazione dell'attività fisiologica e patologica da varie colture cellulari e preparazioni di fette di cervello. Di particolare rilievo, queste capacità dei sistemi CMOS-HD-MEA possono consentire ai ricercatori di contrastare i modelli di crisi epilettiche di diverse regioni del cervello contemporaneamente e di valutare come vari composti antiepilettici influenzano questi modelli. In questo modo, fornisce un metodo innovativo per studiare l'ictogenesi e la propagazione ictale e per comprendere come la farmacologia interrompe l'attività patologica della rete 7,10,14. Pertanto, queste nuove capacità dei sistemi CMOS-HD-MEA possono contribuire in modo significativo alla ricerca sui disturbi neurologici, oltre ad aiutare nella ricerca sulla scoperta di farmaci 5,7,11,22. Il nostro obiettivo è fornire dettagli sull'utilizzo dei sistemi CMOS-HD-MEA per studiare l'attività convulsiva.
Quando si utilizzano i sistemi CMOS-HD-MEA per studiare gli LFP, come l'attività epilettiforme nelle fette cerebrali acute, gli utenti possono affrontare molte sfide, tra cui il rumore elettrico debilitante, il mantenimento della salute della fetta durante la sperimentazione e il rilevamento di un segnale di qualità da un chip CMOS-MEA bidimensionale (2D) che registra solo dalla superficie della fetta cerebrale. Questo protocollo descrive i passaggi di base per la corretta messa a terra della piattaforma MEA e di altre apparecchiature utilizzate nella sperimentazione, un passaggio cruciale che può richiedere una personalizzazione individuale per ogni configurazione di laboratorio. Inoltre, discutiamo i passaggi per aiutare a mantenere sana la fetta di cervello durante lunghe registrazioni nelle camere sommerse utilizzate con i sistemi CMOS-HD-MEA 23,24,25. Inoltre, a differenza dei metodi di registrazione elettrofisiologica più comuni, che registrano dalle profondità della fetta cerebrale, la maggior parte dei sistemi CMOS-HD-MEA utilizza chip 2D che non penetrano nella fetta. Pertanto, questi sistemi richiedono uno strato esterno neuronale sano per produrre la maggior parte dei segnali LFP registrati. Altre sfide includono la visualizzazione dell'enorme quantità di dati generati da migliaia di elettrodi. Per superare queste sfide, raccomandiamo un protocollo semplice ma efficace che aumenti la probabilità di ottenere un'attività epilettiforme di rete di alta qualità che si propaga attraverso la fetta di cervello. Includiamo anche una breve descrizione di un'interfaccia utente grafica (GUI) disponibile al pubblico che abbiamo sviluppato con le risorse associate per facilitare la visualizzazione dei dati10.
Pubblicazioni precedenti hanno fornito protocolli correlati per l'uso dei sistemi di registrazione MEA 26,27,28,29. Tuttavia, questo lavoro mira ad assistere gli sperimentatori che utilizzano sistemi CMOS-HD-MEA con chip 2D, in particolare quelli che cercano di studiare l'attività epilettiforme di alta qualità da fette di cervello. Inoltre, confrontiamo due delle più comuni manipolazioni della soluzione per l'induzione di attività convulsive, vale a dire i paradigmi 0 Mg2+ e 4-AP, per aiutare gli utenti a identificare i mezzi convulsivanti più appropriati per la loro specifica applicazione. Sebbene il protocollo sia focalizzato principalmente sulla generazione di attività simile alle convulsioni, può essere modificato per esplorare altri fenomeni elettrofisiologici utilizzando fette di cervello.
Access restricted. Please log in or start a trial to view this content.
Protocollo
Le procedure che coinvolgono i topi sono state approvate dall'Institutional Animal Care and Use Committee (IACUC) della Brigham Young University. Negli esperimenti successivi sono stati utilizzati topi maschi e femmine (n = 8) C57BL/6 di età almeno pari a P21.
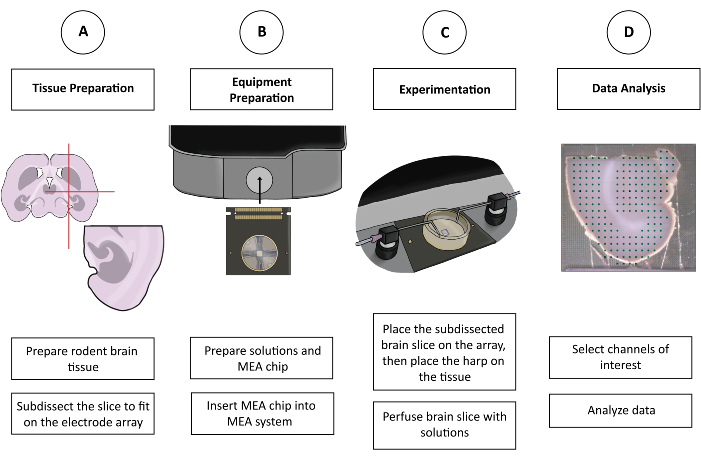
Figura 1: Figura schematica della sperimentazione CMOS-HD-MEA. (A) La fetta di cervello viene preparata con il metodo di taglio preferito e sezionata per adattarsi al MEA. (B) Preparare le soluzioni e il chip CMOS-HD-MEA. (C) La fetta di cervello sub-sezionata viene posizionata sull'array di elettrodi e immersa nelle soluzioni appropriate. (D) I canali pertinenti sono selezionati dai dati raccolti. I dati vengono quindi preparati per l'analisi nel programma preferito dall'utente. Clicca qui per visualizzare una versione più grande di questa figura.
| Nome | Concentrazione (mM) | g/L | ||
| Cloruro di sodio (NaCl) | 126 | 7.36 | ||
| Cloruro di potassio (KCl) | 3.5 | 0.261 | ||
| Diidrogeno Sodio Fosfato (NaH2PO4) | 1.26 | 0.151 | ||
| Bicarbonato di sodio (NaHCO3) | 26 | 2.18 | ||
| Glucosio (C6H12O6) | 10 | 1.80 | ||
| Cloruro di magnesio (MgCl2) | 1 (da 1 M di magazzino) | 1 ml | ||
| Cloruro di calcio (CaCl2) | 2 (da 1 M di magazzino) | 2 ml | ||
Tabella 1: soluzione di aCSF.
1. Preparazione delle soluzioni
- Preparare le soluzioni sperimentali.
- Preparare 1 litro di liquido cerebrospinale artificiale (aCSF) (vedere la Tabella 1 per i dettagli).
- Preparare 1 L di soluzione pro-convulsivante.
NOTA: Le soluzioni utilizzate per generare un'attività simile alle convulsioni in questo protocollo utilizzavano aCSF con 100 μM di 4-aminopiridina (4-AP) o aCSF privo di ioni magnesio. - Carbogenare tutte le soluzioni per almeno 10 minuti prima dell'uso utilizzando pietre porose.
- Procurati un becher per scartare le soluzioni.
- Porre 1 L di aCSF, 1 L di soluzione pro-convulsivante e il becher di scarto su una superficie vicina al sistema di perfusione.
- Preparazione delle soluzioni per la camera di taglio e di mantenimento
- Preparare 0,5 L di aCSF, omettendo CaCl2 e aggiungendo 3 mM di MgCl2 (invece di 1 mM) per creare una soluzione di taglio unica. Conservare questa soluzione per l'uso nella preparazione di fette di cervello di roditore posizionandola vicino all'area di preparazione della fetta di cervello acuta.
- Preparare 0,5 L di aCSF e metterlo in una camera di contenimento dei tessuti che verrà utilizzata per conservare le fette da utilizzare nella sperimentazione. Inserire i calcoli porosi nella camera di contenimento del tessuto e carbogenare la soluzione per almeno 10 minuti prima di posizionare le fette sub-sezionate nella camera.
2. Preparazione di fette di cervello di roditore
- Preparare tutte le soluzioni come descritto sopra (vedere paragrafo 1). Assicurarsi che tutte le soluzioni siano adeguatamente carbogenate durante il processo di preparazione.
NOTA: Si consiglia la soluzione di taglio (vedere paragrafo 1.2), ma è possibile utilizzare altre soluzioni di taglio per ottenere fette di cervello di qualità, come una soluzione di taglio di saccarosio30. - Usa un microtomo vibrante per creare fette di cervello di roditore da 350 μm. I protocolli per l'utilizzo di questi strumenti sono elencati nei riferimenti 30,31,32,33.
- Preparare le fette di cervello di roditore in modo che si adattino all'area dell'elettrodo di registrazione del chip MEA (vedere la Figura 2). Utilizzare una lama chirurgica di misura 10 per sezionare le fette, come mostrato nella Figura 1A e nella Figura 2A , facendo rotolare delicatamente la lama avanti e indietro sulla fetta del cervello. Eseguire la sotto-dissezione nella camera di taglio del microtomo vibrante. Prestare attenzione a non danneggiare la fetta di cervello sezionata.
- Posizionare le fette sub-sezionate in una camera di contenimento del tessuto riempita con aCSF. Assicurarsi che la camera sia stata carbogenata per almeno 10 minuti prima di aggiungere le fette sottosezionate.
3. Preparazione dell'attrezzatura
- Preparazione dei chip e del sistema MEA (Figura 2 e Figura 3)
NOTA: I materiali duri come la plastica e i metalli possono facilmente danneggiare il chip MEA se entrano in contatto con forza con la parte inferiore del chip. Quando si utilizzano pipette per trasferire la soluzione all'interno o all'esterno dei chip, non entrare in contatto con la parte inferiore del chip con la pipetta, in particolare gli elettrodi di riferimento ai lati del pozzetto del chip e gli elettrodi di registrazione al centro del pozzetto del chip (Figura 2C). Invece, aggiungi la soluzione passando il mouse sopra il fondo del pozzetto del chip o toccando o approssimando i bordi in plastica del pozzetto del chip. Per estrarre facilmente tutta la soluzione, inclinare leggermente il chip per consentire alla soluzione di accumularsi bene su un lato del chip e rimuoverlo dalla parte superiore della soluzione pooled, oppure utilizzare un panno antistatico per tamponare leggermente le aree che hanno ancora la soluzione. Non trasportare il truciolo tenendolo bene o tenendo bene i perni del truciolo (Figura 2C). Il pozzetto del chip può contenere circa 4 ml di soluzione. Per le fasi successive, riempire il pozzetto del chip con circa 2 mL di soluzione, se non diversamente specificato.- Designare le pipette di trasferimento per varie attività prima di iniziare la preparazione del chip. Etichettare una pipetta di trasferimento per l'etanolo, una per i rifiuti, un'altra per l'aCSF e altre per eventuali soluzioni rimanenti per evitare la miscelazione involontaria.
- Riempire il pozzetto del chip MEA con etanolo a prova 190 in modo che il fondo del pozzetto del chip sia completamente coperto (Figura 3). Lasciare riposare l'etanolo per 30-60 s, quindi rimuoverlo con una pipetta di scarto.
- Riempire il pozzetto del chip MEA con aCSF e rimuoverlo con una pipetta di scarto per sciacquare l'etanolo residuo dal pozzetto del chip. Aggiungere e rimuovere l'aCSF dal pozzetto del chip tre volte, utilizzando le pipette di scarto e aCSF precedentemente designate. Dopo aver lavato bene il truciolo per tre volte, aggiungere aCSF e lasciarlo riposare per almeno 30 s.
NOTA: Gli elettrodi di registrazione mostrano la minima quantità di rumore da interazioni idrofobiche quando l'aCSF rimane nella camera per almeno 45 minuti dopo il risciacquo con etanolo e aCSF. - Prima di agganciare il chip MEA, inumidire una salvietta antistatica con etanolo a prova di 190 e utilizzarla per pulire i pin del chip (Figura 2C).
- Far scorrere delicatamente il chip MEA nella piattaforma MEA e innestare il meccanismo di aggancio per bloccare il chip in posizione.
- Controllare la presenza di bolle negli elettrodi di registrazione e di riferimento (Figura 2C). Se sono presenti bolle, prendere un pennello pulito e passare leggermente sugli elettrodi per rimuoverle.
- Controllare la presenza di rumore nel chip utilizzando il software CMOS-HD-MEA34 ed eseguire la scansione visiva della mappa dei falsi colori alla ricerca di bolle, oscillazioni non biologiche o picchi causati da interferenze elettriche. Mettere a terra il sistema MEA in modo appropriato per annullare qualsiasi rumore riscontrato.
NOTA: La configurazione della messa a terra dipenderà dall'ambiente di registrazione. Per gli esperimenti di questo protocollo, la piattaforma MEA e il sistema di perfusione sono stati messi a terra.
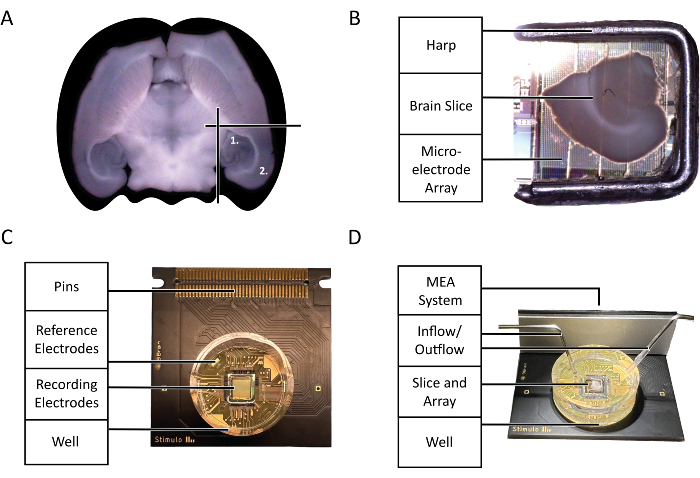
Figura 2: Diagrammi di configurazione e tecnologia. (A) Diagramma della selezione di fette acute di cervello di topo utilizzate nell'esperimento evidenziato da questo protocollo. (1) Regione dell'ippocampo, (2) Regione della neocorteccia. (B) Il corretto posizionamento di una fetta acuta di cervello di topo e di un'arpa sull'array di microelettrodi (MEA). (C) L'anatomia di un chip CMOS-HD-MEA 3Brain Accura. (D) La corretta configurazione degli ingressi e delle uscite di perfusione. L'ingresso dovrebbe essere in profondità nel pozzetto del truciolo, mentre l'uscita dovrebbe essere sul lato opposto dell'ingresso nella parte superiore del pozzetto del truciolo per garantire un flusso costante di aCSF fresco e ossigenato. Clicca qui per visualizzare una versione più grande di questa figura.
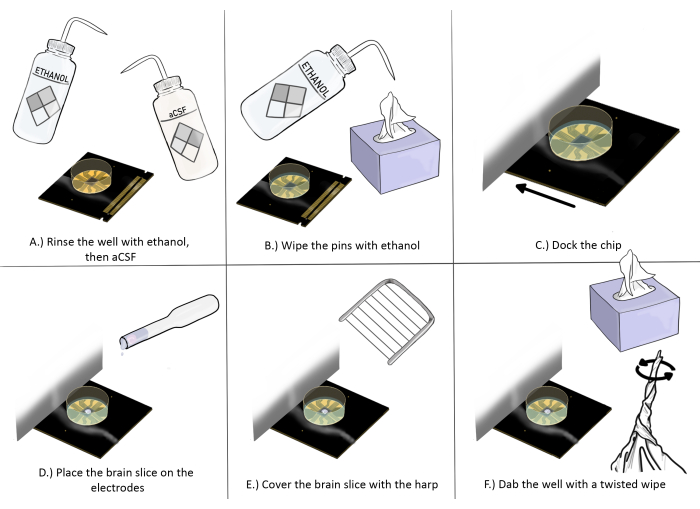
Figura 3: Diagramma schematico della preparazione e del posizionamento del chip per gli esperimenti di brain slice. (A) Sciacquare bene il chip con etanolo una volta, poi con aCSF tre volte. (B) Pulire i perni con etanolo utilizzando un panno antistatico. (C) Agganciare il chip. (D) Posizionare la fetta di cervello sugli elettrodi. (E) Posizionare l'arpa sulla fetta di cervello (vedere la Figura 2 per le linee guida per il posizionamento corretto). (F) Tamponare l'angolo del pozzetto degli elettrodi di registrazione vicino alla fetta del cervello con una salvietta antistatica attorcigliata. Clicca qui per visualizzare una versione più grande di questa figura.
4. Sperimentazione
- Posizionamento della fetta di cervello
- Posiziona un'arpa di platino in una barca di pesatura (o un'altra superficie pulita) vicino alla piattaforma MEA. Copri l'arpa con circa 3 ml di aCSF per ridurre le sue tendenze idrofobiche.
- Utilizzare le forbici per rimuovere la parte sottile del puntale di una pipetta di trasferimento. Tagliare un terzo, circa 1,5 pollici, del puntale della pipetta.
NOTA: Questa pipetta modificata non restringe o danneggia la fetta di cervello durante la raccolta e il trasporto. - Raccogliere una fetta di cervello dalla camera di contenimento delle fette con la pipetta modificata. Erogare delicatamente la fetta di cervello e l'eventuale soluzione nella pipetta nel pozzetto del chip. Per posizionare correttamente la fetta, rilasciare delicatamente più aCSF da una pipetta di trasferimento per manipolare la posizione della fetta cerebrale o utilizzare un pennello morbido per creare una corrente nella soluzione che spinge la fetta di cervello sugli elettrodi di registrazione. Limitare il contatto con gli elettrodi di registrazione o la fetta cerebrale per ridurre al minimo i danni.
- Usando una pinza, posiziona delicatamente l'arpa sopra la fetta cerebrale con i fili rivolti verso il basso per premere la fetta sugli elettrodi di registrazione. Evitare di entrare in contatto con l'array di elettrodi con l'arpa. Orientare l'arpa in modo che il lato senza telaio sia rivolto verso l'ago di afflusso e che il telaio dell'arpa non entri in contatto con nessuno degli elettrodi di registrazione (Figura 2C, D).
- Prendi una pipetta di scarto e rimuovi l'eccesso di aCSF. Prendi una salvietta antistatica, ruota un angolo per creare una punta e usala per assorbire l'aCSF rimanente che circonda gli elettrodi di registrazione senza toccare gli elettrodi di registrazione, la fetta cerebrale o l'arpa (Figura 2).
- Utilizzando una pipetta designata per aCSF, aggiungere rapidamente una quantità sufficiente di aCSF carbogenato a coprire la fetta di cervello, circa 2 ml.
- Ripetere il passaggio 4.1.5. e 4.1.6. altre due volte.
- Riempire il pozzetto con aCSF carbogenato fino a quando il pozzetto è pieno per circa 3/4, circa 3 ml.
- Scatta una foto della fetta di cervello sul chip MEA con un microscopio o una fotocamera. Assicurati che la foto abbia una risoluzione sufficientemente alta da vedere i bordi dell'array di elettrodi di registrazione e l'anatomia della fetta di cervello.
- Esecuzione dell'esperimento
- Funzionamento del sistema di perfusione
- Posizionare i tubi di afflusso e di deflusso nel becher riempito con aCSF e gli aghi di afflusso e deflusso nel pozzetto del chip. Posizionare l'ago di afflusso vicino alla parte inferiore del chip, appena fuori dagli elettrodi di registrazione. Posizionare l'ago di deflusso vicino alla parte superiore del chip verso il bordo in modo che il liquido salga quasi fino all'orlo del chip, circa 4 mL, e la fetta sia immersa durante l'esperimento (Figura 2D).
- Impostare l'afflusso di perfusione a 5 mL/min e il deflusso di perfusione a 7 mL/min.
NOTA: Si raccomanda che la velocità di deflusso superi la velocità di afflusso per evitare che la soluzione fuoriesca dal pozzetto del chip e per creare una corrente di soluzione sulla fetta del cervello. - Attiva l'afflusso e il deflusso. Rimuovere bene l'ago di afflusso dal chip fino a quando l'ago non inizia a emettere soluzione invece di aria. Quindi, riposizionare l'ago nella sua posizione all'interno del pozzetto del chip come descritto nel passaggio 4.2.1.1.
- Utilizzare un riscaldatore per soluzioni per mantenere la soluzione a una temperatura fisiologica o quasi, intorno ai 34-37 °C.
- Lasciare che l'aCSF persi sulla fetta di cervello per 10 minuti. Ciò consentirà alla sezione di adattarsi all'ambiente di registrazione.
- Trascorsi 10 minuti, spostare il tubo di deflusso nel becher di smaltimento. Quindi, spostare il tubo di afflusso nel becher contenente la soluzione pro-convulsivante. Lasciare che l'aCSF non convulsivante venga espulso dal sistema di perfusione nel becher di scarto per 10 minuti.
- Trasferire il tubo di deflusso nel becher contenente la soluzione proconvulsivante.
- Lasciare che la soluzione pro-convulsivante cicli fino al termine dell'esperimento.
- Se la durata della registrazione della fetta supera le 2 ore, prendere in considerazione la preparazione di un'altra soluzione pro-convulsivante per compensare il consumo di glucosio della fetta cerebrale attiva.
- Scambio di fette di cervello
- Disattiva l'afflusso. Chiudere il deflusso 10-15 s dopo.
- Usa una pinza per rimuovere l'arpa e posizionarla su una pesa o su un'altra superficie.
- Utilizzare una pipetta modificata per estrarre e scartare la fetta di cervello. Non toccare gli elettrodi di riferimento o l'array di elettrodi di registrazione (Figura 2C).
- Posizionare il tubo di afflusso nel becher contenente la soluzione non convulsivante di aCSF. Posizionare il tubo di deflusso nel becher di smaltimento. Lasciare gli aghi in entrata e in uscita nel pozzetto del chip. Far funzionare il sistema di perfusione per 10 minuti per rimuovere tutta la soluzione proconvulsivante residua dal pozzetto del chip e dal sistema di perfusione. Ricomincia il processo di sperimentazione con una nuova fetta di cervello (a partire dal passaggio 4.1).
- Funzionamento del sistema di perfusione
- Completamento dell'esperimento
- Pulizia del rig
- Rimuovi rispettivamente l'arpa, la fetta di cervello e la soluzione dal pozzo.
- Sganciare il chip MEA, posizionarlo su una superficie pulita e riempire bene il chip con acqua deionizzata.
- Scartare l'acqua deionizzata per eliminare i sali rimasti dalle soluzioni di aCSF.
- Utilizzando una pipetta di trasferimento, riempire bene il chip con una soluzione detergente. Posizionare la punta della pipetta di trasferimento nel pozzetto del chip e puntarla verso gli elettrodi di registrazione. Spremere e rilasciare rapidamente e ripetutamente il detersivo nella pipetta di trasferimento per lavare energicamente il detersivo sugli elettrodi di registrazione per 1 minuto. Lasciare riposare il detersivo per 5-10 min.
- Rimuovere il detersivo, quindi sciacquare bene il truciolo 4-6 volte con acqua deionizzata per rimuovere la soluzione detergente e gli eventuali soluti rimanenti.
- Rimuovere bene tutta l'acqua dal chip, quindi posizionare una salvietta antistatica sul chip MEA e lasciare agire per una notte per consentire al chip di asciugarsi bene.
- Posizionare una pesa a battente o un'altra piastra a tenuta stagna sulla piattaforma di registrazione MEA dove il chip MEA era precedentemente appoggiato. Posiziona l'arpa al centro del piatto e posiziona gli aghi di afflusso e di uscita su entrambi i lati dell'arpa.
- Pulire il sistema di perfusione e l'arpa inviando rispettivamente 50 ml di acqua, 50 ml di etanolo a prova 140 e 200 ml di acqua attraverso il tubo nel becher di smaltimento.
NOTA: A questo punto, la velocità di perfusione può essere aumentata per ridurre il tempo di pulizia, ma la velocità di deflusso deve sempre superare la velocità di afflusso per evitare allagamenti. - Pulire le pietre di carbogeno mettendole in un becher di acqua distillata e inviando il carbogeno attraverso il tubo per 5 minuti. Mettili a riposare su una superficie asciutta e pulita per una notte e coprili per evitare l'accumulo di polvere.
- Pulizia del rig
5. Analisi dei dati
NOTA: Esistono diversi pacchetti di analisi utilizzati per l'analisi dei dati elettrofisiologici prodotti dai CMOS-HD-MEA, tra cui BrainWave di 3Brain, Yet Another Spike Sorter (YASS) e gli strumenti Python personalizzati 34,35,36,37. Abbiamo estratto i dati dal formato di file di dati BrainWave per l'utilizzo nella piattaforma di analisi Xenon LFP per generare i dati presentati nella Figura 4 e nella Figura 5. Il codice Matlab personalizzato è stato utilizzato per analizzare i dati nella Figura 6. I protocolli per la piattaforma di analisi LFP allo xeno sono disponibili al pubblico10. I seguenti passaggi del protocollo sono specifici per le registrazioni effettuate con Brainwave 438; Per altri sistemi, fare riferimento alla documentazione di supporto relativa a tali sistemi 34,35,36,37. Di seguito viene fornita una panoramica delle fasi di analisi adottate per produrre i dati con questo protocollo. Per informazioni dettagliate su come esportare, visualizzare e analizzare i dati, inclusi i video di esercitazione e tutti i file di codice pertinenti, vedere39.
- Esporta il file di registrazione per l'analisi.
- Creare una cartella per i file di registrazione dei dati che verranno sottocampionati.
- Ritagliare l'immagine scattata durante il passaggio 4.1.9 per includere solo l'array di registrazione con le fette di cervello.
- Apri il file ExportToHDF5-ChannelSelection.py (questo file di codice è accessibile su GitHub in xenon-lfp-analysis/code-files/3Brain-processing40) e copia e incolla l'URL elencato nella parte superiore della finestra in un browser Internet.
- Immettere il percorso del file di registrazione dei dati e caricare l'immagine ritagliata dell'array associato alla registrazione.
- In Seleziona canali per l'esportazione, utilizzare lo strumento di selezione lazo per selezionare le regioni ippocampali e neocorticali mostrate nell'immagine ritagliata dell'array.
- Impostare sia il numero di righe da saltare che il numero di colonne da saltare su 2.
NOTA: I file di registrazione sono spesso di grandi dimensioni; Se appropriato per l'analisi desiderata, i dati esportati possono essere sottocampionati. Esistono opzioni per il downsampling spazialmente (saltando righe e colonne di elettrodi di registrazione) e temporalmente (downsampling a una frequenza di campionamento inferiore). Nell'analisi, ogni 2 righe e 2 colonne vengono saltate; Ciò consente il downsampling dei dati spazialmente senza essere distorti verso determinati canali. Ogni canale è abbastanza distante da far sì che le differenze di attività possano essere significative tra di loro; Pertanto, non viene eseguita alcuna media dei canali. Quando si saltano righe e colonne degli elettrodi, i dati dei canali non selezionati non vengono inclusi quando il file di output viene esportato. - Impostare la frequenza di downsampling su 300 Hz.
- Fare clic sul pulsante Esporta canali per generare il file di selezione dei canali.
NOTA: Il software salverà automaticamente il file di selezione del canale nella cartella in cui è salvato il file di registrazione dei dati. - Apri il file ExportToHDF5-ExtractDownsample.py (che si trova anche su GitHub sotto xenon-lfp-analysis/code-files/3brain-processing40), incolla il percorso del file della cartella contenente i file di selezione del canale e i file di registrazione dei dati e premi invio sulla tastiera per generare la versione sottocampionata del file di registrazione dei dati.
- Utilizzo della piattaforma di analisi
- Nel prompt dei comandi del computer, digita run_lfp_analysis e premi invio per aprire la piattaforma di analisi.
- Immettere i percorsi dei file dell'immagine e del file di registrazione sottocampionato e fare clic su Invia.
- In Seleziona un intervallo di tempo per l'analisi, selezionare l'intera registrazione.
- In Seleziona canali per i grafici, evidenziare l'ippocampo come Gruppo-1 e la neocorteccia come Gruppo-2.
- In Raster canale, impostare la soglia su 0,06 mV e la durata del tempo su 0,02 s.
- Fare clic su Applica impostazioni e generare raster.
- Utilizzare il grafico raster per esplorare diversi canali che mostrano i modelli di attività di interesse.
NOTA: Vedere la Figura 4 per esempi di attività simile a una crisi di alta qualità (Figura 4A-D) rispetto ad attività subottimale (Figura 4E-F).
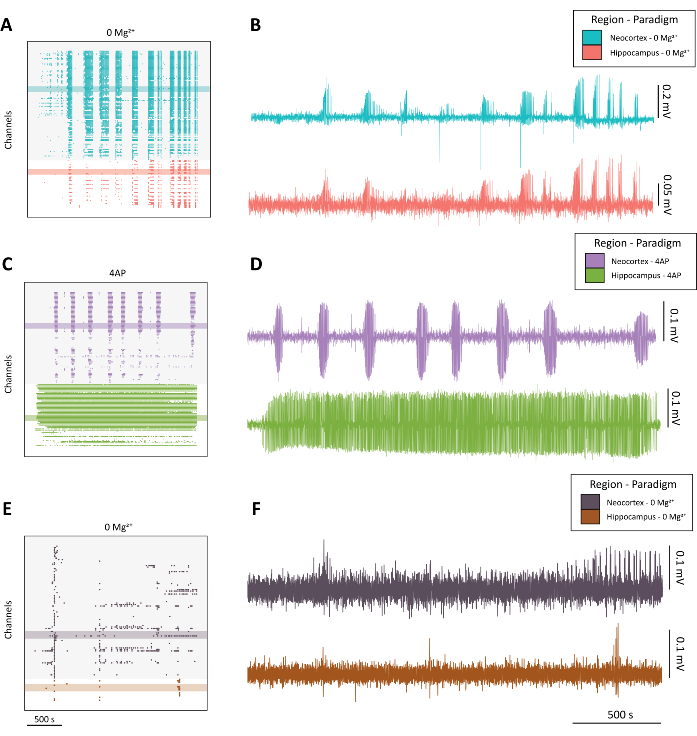
Figura 4: Esempio di evoluzione dell'attività epilettiforme dai paradigmi 0 Mg,2+ e 4-AP. (A) Esempio di grafico raster dall'applicazione di aCSF con 0 Mg2+ per circa 40 minuti. (B) Esempio di tracce elettrofisiologiche prelevate dalla neocorteccia (blu) e dall'ippocampo (rosso) che dimostrano l'attività epilettiforme dal paradigma 0 Mg2+ . (C) Esempio di grafico raster dall'applicazione 100 mM 4-AP per circa 40 minuti. (D) Esempio di tracce elettrofisiologiche prelevate dalla neocorteccia (viola) e dall'ippocampo (verde) che dimostrano l'attività epilettiforme dall'applicazione di 4-AP. (E) Esempio di grafico raster dall'applicazione di aCSF con 0 Mg2+ per circa 40 minuti che mostra l'attività di scoppio rispetto all'attività simile alle convulsioni come trovato nelle altre tracce rappresentative. (F) Esempi di tracce elettrofisiologiche prelevate dalla neocorteccia (viola scuro) e dall'ippocampo (ruggine) che dimostrano un'attività sub-ottimale dal paradigma 0 Mg2+ destinato al confronto con l'attività convulsiva di qualità trovata in B e D. Clicca qui per visualizzare una versione più grande di questa figura.
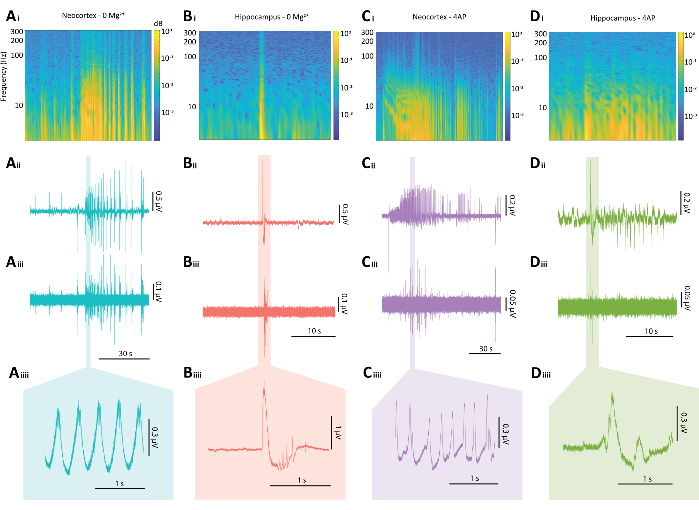
Figura 5: Risultati rappresentativi delle scariche epilettiformi da entrambi i paradigmi 0 Mg2+ e 4-AP. (A) Esempi di un tipico evento simile a una crisi neocorticale indotto dal paradigma 0 Mg2+ che include (Ai) uno spettrogramma da un evento simile a una crisi epilettica, (Aii) la traccia elettrofisiologica associata, (Aiii) un filtro passa-alto a 80 Hz applicato alla traccia da Aii, (Aiiii) e una sezione ingrandita della traccia da Aii. (B) Esempi di grafici di un tipico burst epilettiforme ippocampale indotto dal paradigma 0 Mg2+ tra cui (Bi) uno spettrogramma del burst epilettiforme, (Bii) la traccia elettrofisiologica associata, (Biii) un filtro passa-alto a 80 Hz applicato alla traccia da Bii, (Biiii) e una sezione ingrandita della traccia da Bii (C) Esempi di grafici di un tipico evento simile a una crisi neocorticale indotto dal paradigma 4-AP tra cui (Ci) uno spettrogramma dell'attività epilettiforme, (Cii) la traccia elettrofisiologica associata, (Ciii) un filtro passa-alto a 80 Hz applicato alla traccia da Cii, (Ciiii) e una sezione ingrandita della traccia da Cii (D) Esempi di grafici di un'esplosione epilettiforme ippocampale sotto il paradigma 4-AP che include (Di) uno spettrogramma dell'attività epilettiforme, (Dii) la traccia elettrofisiologica associata, (Diii) un filtro passa-alto a 80 Hz applicato alla traccia da Dii, (Diiii) e una sezione ingrandita della traccia da Dii. Clicca qui per visualizzare una versione più grande di questa figura.
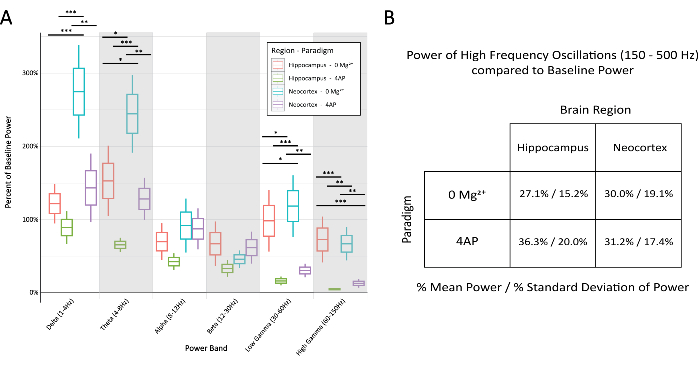
Figura 6: Confronto della percentuale della potenza basale nelle varie bande attraverso il paradigma e la regione del cervello durante le scariche epilettiformi stereotipate. (A) La potenza durante le scariche epilettiformi era significativamente diversa tra i paradigmi e le regioni cerebrali per la maggior parte delle bande di frequenza (ANOVA a 2 vie con test di Tukey, *P < 0,05, **P < 0,001, ***P < 0,0001). La linea centrale per ogni casella rappresenta la media, i bordi della casella ±1 errore standard della media (SEM) e le linee più esterne ±2 SEM. (B) Sia i paradigmi che le regioni del cervello hanno dimostrato una potenza limitata nelle bande relative all'attività ad alta frequenza superiore a 150 Hz. Clicca qui per visualizzare una versione più grande di questa figura.
Access restricted. Please log in or start a trial to view this content.
Risultati
Come è standard quando si visualizza l'attività da molti canali 1,4,5,10, troviamo vantaggioso generare prima un grafico raster dei dati che acquisiamo con il CMOS-HD-MEA (Figura 4A,C,E). Questo grafico può creare una vista a volo d'uccello dell'attività in tutti i canali di registrazione in ci...
Access restricted. Please log in or start a trial to view this content.
Discussione
Questo protocollo include linee guida specifiche relative alla gestione acuta della fetta cerebrale che affrontano i problemi comuni affrontati dagli utenti di CMOS-HD-MEA, vale a dire lo sviluppo di rumore sotto la fetta cerebrale e il mantenimento di un ambiente sano per la fetta cerebrale. Lo sviluppo di rumore sotto la fetta si verifica quando la fetta non aderisce correttamente all'array; Se la fetta di cervello non aderisce adeguatamente, possono formarsi sacche d'aria sotto la fet...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori dichiarano che non ci sono conflitti di interesse associati a questo studio di ricerca.
Riconoscimenti
Gli autori ringraziano gli ex e gli attuali membri del Parrish lab per le loro modifiche a questo manoscritto. Ringraziamo anche Alessandro Maccione di 3Brain per il suo feedback su questo lavoro. Questo lavoro è stato finanziato da un AES/EF Junior Investigator Award e dal Brigham Young University Colleges of Life Sciences e of Physical and Mathematical Sciences.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| 2D Workbench | Cloudray | LM04CLLD26B | |
| 4-Aminopyridine | Sigma-Aldrich | 275875 | |
| Accura Chip | 3Brain | Accura HD-MEA | CMOS-HD-MEA chip |
| Agarose | Thermo Fisher Scientific | BP160-100 | |
| Vibration isolation table | Kinetic Systems | 91010124 | |
| Beaker for the slice holding chamber, 270 mL | VWR | 10754-772 | |
| BioCam | 3Brain | BioCAM DupleX | CMOS-HD-MEA platform |
| Brainwave Software | 3Brain | Version 4 | CMOS-HD-MEA software |
| Calcium Chloride | Thermo Fisher Scientific | BP510-500 | |
| Carbogen | Airgas | X02OX95C2003102 | |
| Carbogen | Airgas | 12005 | |
| Carbogen Stones | Supelco | 59277 | |
| Compresstome | Precissionary | VF-300-0Z | |
| Computer | Dell | Precission3650 | |
| Crocodile Clip Grounding Cables | JWQIDI | B06WGZG17W | |
| Detergent | Metrex | 10-4100-0000 | |
| D-Glucose | Macron Fine Chemicals | 4912-12 | |
| Dihydrogen Sodium Phosphate | Thermo Fisher Scientific | BP329-500 | |
| DinoCam | Dino-Lite | AM73915MZTL | |
| Ethanol | Thermo Fisher Scientific | A407P-4 | |
| Forceps | Fine Science Tools | 11980-13 | |
| Hot plate | Thermo Fisher Scientific | SP88857200 | |
| Ice Machine | Hoshizaki | F801MWH | |
| Inflow and outflow needles | Jensen Global | JG 18-3.0X | |
| Inline Solution Heater | Warner Instruments | SH-27B | |
| Isofluorine | Dechra | 08PB-STE22002-0122 | |
| Kim Wipes | Thermo Fisher Scientific | 06-666 | |
| Magnesium Chloride | Thermo Fisher Scientific | FLM33500 | |
| Micropipets | Gilson | F144069 | |
| Mili-Q Water Filter | Mili-Q | ZR0Q008WW | |
| Paintbrush | Daler Rowney | AF85 Round: 0 | |
| Paper Filter | Whatman | EW-06648-24 | |
| Parafilm | American National Can | PM996 | |
| Perfusion System | Multi Channel System | PPS2 | |
| Pipetor | Thermo Fisher Scientific | FB14955202 | |
| Platinum Harp | 3Brain | 3Brain | |
| Potassium Chloride | Thermo Fisher Scientific | P330-3 | |
| Razor blade | Personna | BP9020 | |
| Scale | Metter Toledo | AB204 | |
| Scissors | Solingen | 92008 | |
| Slice Holding Chamber | Custom | Custom | Custom 3D Printer Design, available upon request |
| Sodium Bicarbonate | Macron Fine Chemicals | 7412-06 | |
| Sodium Chloride | Thermo Fisher Scientific | S271-3 | |
| Temperature Control Box | Warner Instruments | TC344B | |
| Transfer Pipettes | Genesee Scientific | 30-200 | |
| Tubing | Tygon | B-44-3 TPE | |
| Vibratome VZ-300 | Precissionary | VF-00-VM-NC | |
| Weigh Boat | Electron Microscopy Sciences | 70040 |
Riferimenti
- Obien, M. E. J., Frey, U. Large-scale, high-resolution microelectrode arrays for interrogation of neurons and networks. Adv Neurobiol. 22, 83-123 (2019).
- Schroter, M., et al. Functional imaging of brain organoids using high-density microelectrode arrays. MRS Bull. 47 (6), 530-544 (2022).
- Miccoli, B., et al. High-density electrical recording and impedance imaging with a multi-modal CMOS multi-electrode array chip. Front Neurosci. 13, 641(2019).
- Emery, B. A., Hu, X., Khanzada, S., Kempermann, G., Amin, H. High-resolution CMOS-based biosensor for assessing hippocampal circuit dynamics in experience-dependent plasticity. Biosens Bioelectron. 237, 115471(2023).
- Ferrea, E., et al. high-resolution electrophysiological imaging of field potentials in brain slices with microelectronic multielectrode arrays. Front Neural Circuits. 6, 80(2012).
- Gagliano, G., et al. Non-linear frequency dependence of neurovascular coupling in the cerebellar cortex implies vasodilation-vasoconstriction competition. Cells. 11 (6), 1047(2022).
- Goodchild, S. J., et al. Molecular pharmacology of selective Na(V)1.6 and dual Na(V)1.6/Na(V)1.2 channel inhibitors that suppress excitatory neuronal activity ex vivo. ACS Chem Neurosci. 15 (6), 1169-1184 (2024).
- Hu, X., Khanzada, S., Klutsch, D., Calegari, F., Amin, H. Implementation of biohybrid olfactory bulb on a high-density CMOS-chip to reveal large-scale spatiotemporal circuit information. Biosens Bioelectron. 198, 113834(2022).
- Kim, S., et al. Alteration of neural network and hippocampal slice activation through exosomes derived from 5XFAD nasal lavage fluid. Int J Mol Sci. 24 (18), 14064(2023).
- Mahadevan, A., Codadu, N. K., Parrish, R. R. Xenon LFP analysis platform is a novel graphical user interface for analysis of local field potential from large-scale MEA recordings. Front Neurosci. 16, 904931(2022).
- Medrihan, L., Ferrea, E., Greco, B., Baldelli, P., Benfenati, F. Asynchronous GABA release is a key determinant of tonic inhibition and controls neuronal excitability: A study in the synapsin II-/- mouse. Cereb Cortex. 25 (10), 3356-3368 (2015).
- Monteverdi, A., Di Domenico, D., D'Angelo, E., Mapelli, L. Anisotropy and frequency dependence of signal propagation in the cerebellar circuit revealed by high-density multielectrode array recordings. Biomedicines. 11 (5), 1475(2023).
- Obien, M. E. J., Hierlemann, A., Frey, U. Accurate signal-source localization in brain slices by means of high-density microelectrode arrays. Sci Rep. 9 (1), 788(2019).
- Thouta, S., et al. Pharmacological determination of the fractional block of Nav channels required to impair neuronal excitability and ex vivo seizures. Front Cell Neurosci. 16, 964691(2022).
- Tognolina, M., Monteverdi, A., D'Angelo, E. Discovering microcircuit secrets with multi-spot imaging and electrophysiological recordings: The example of cerebellar network dynamics. Front Cell Neurosci. 16, 805670(2022).
- Hierlemann, A., Frey, U., Hafizovic, S., Heer, F. Growing cells atop microelectronic chips: Interfacing electrogenic cells in vitro with CMOS-based microelectrode arrays. Proceedings of the IEEE. 99 (2), 252-284 (2011).
- Maccione, A., et al. Experimental investigation on spontaneously active hippocampal cultures recorded by means of high-density MEAs: Analysis of the spatial resolution effects. Front Neuroeng. 3, 4(2010).
- van Vliet, E., et al. Electrophysiological recording of re-aggregating brain cell cultures on multi-electrode arrays to detect acute neurotoxic effects. Neurotoxicology. 28 (6), 1136-1146 (2007).
- Emery, B. A., et al. Large-scale multimodal recordings on a high-density neurochip: Olfactory bulb and hippocampal networks. Annu Int Conf IEEE Eng Med Biol Soc. 2022, 3111-3114 (2022).
- Veleanu, M., et al. Modified climbing fiber/Purkinje cell synaptic connectivity in the cerebellum of the neonatal phencyclidine model of schizophrenia. Proc Natl Acad Sci U S A. 119 (21), e2122544119(2022).
- Giansante, G., et al. Neuronal network activity and connectivity are impaired in a conditional knockout mouse model with PCDH19 mosaic expression. Mol Psychiatry. , (2023).
- Dossi, E., Blauwblomme, T., Nabbout, R., Huberfeld, G., Rouach, N. Multi-electrode array recordings of human epileptic postoperative cortical tissue. J Vis Exp. (92), e51870(2014).
- Hajos, N., et al. Maintaining network activity in submerged hippocampal slices: importance of oxygen supply. Eur J Neurosci. 29 (2), 319-327 (2009).
- Hill, M. R., Greenfield, S. A. The membrane chamber: a new type of in vitro recording chamber. J Neurosci Methods. 195 (1), 15-23 (2011).
- Raimondo, J. V., et al. Methodological standards for in vitro models of epilepsy and epileptic seizures. A TASK1-WG4 report of the AES/ILAE Translational Task Force of the ILAE. Epilepsia. 58 (Suppl 4), 40-52 (2017).
- Hales, C. M., Rolston, J. D., Potter, S. M. How to culture, record and stimulate neuronal networks on micro-electrode arrays (MEAs). J Vis Exp. (39), 2056(2010).
- Lin, C. H., Lee, J. K., LaBarge, M. A. Fabrication and use of microenvironment microarrays (MEArrays). J Vis Exp. (68), 4152(2012).
- Panuccio, G., Colombi, I., Chiappalone, M. Recording and modulation of epileptiform activity in rodent brain slices coupled to microelectrode arrays. J Vis Exp. 135, 57548(2018).
- Patel, C., Muthuswamy, J. High efficiency, site-specific transfection of adherent cells with siRNA using microelectrode arrays (MEA). J Vis Exp. 67, e4415(2012).
- Ting, J. T., Daigle, T. L., Chen, Q., Feng, G. Acute brain slice methods for adult and aging animals: application of targeted patch clamp analysis and optogenetics. Methods Mol Biol. 1183, 221-242 (2014).
- Papouin, T., Haydon, P. G. Obtaining acute brain slices. Bio Protoc. 8 (2), e2699(2018).
- Ting, J. T., et al. Preparation of acute brain slices using an optimized N-Methyl-D-glucamine protective recovery method. J Vis Exp. 132, 53825(2018).
- Van Hoeymissen, E., Philippaert, K., Vennekens, R., Vriens, J., Held, K. Horizontal hippocampal slices of the mouse brain. J Vis Exp. (163), 61753(2020).
- 3Brain. , Available from: https://www.3brain.com/ (2022).
- Bridges, D. C., Tovar, K. R., Wu, B., Hansma, P. K., Kosik, K. S. MEA Viewer: A high-performance interactive application for visualizing electrophysiological data. PLoS One. 13 (2), e0192477(2018).
- Hawrylycz, M., et al. Inferring cortical function in the mouse visual system through large-scale systems neuroscience. Proc Natl Acad Sci U S A. 113 (27), 7337-7344 (2016).
- Maccione, A., et al. Microelectronics, bioinformatics and neurocomputation for massive neuronal recordings in brain circuits with large scale multielectrode array probes. Brain Res Bull. 119 (Pt B), 118-126 (2015).
- 3Brain. , BrainWave 4 https://www.3brain.com/products/software/brainwave4 (2022).
- Mahadevan, A. Xenon LFP Analysis. , https://xenon-lfp-analysis.readthedocs.io/en/latest/Installation.html (2022).
- Mahadevan, A. xenon-lfp-analysis github. , https://github.com/MicroBrew09/xenon-lfp-analysis (2022).
- Codadu, N. K., et al. Divergent paths to seizure-like events. Physiol Rep. 7 (19), e14226(2019).
- Kirsch, G. E., Drewe, J. A. Gating-dependent mechanism of 4-aminopyridine block in two related potassium channels. J Gen Physiol. 102 (5), 797-816 (1993).
- Levesque, M., Salami, P., Behr, C., Avoli, M. Temporal lobe epileptiform activity following systemic administration of 4-aminopyridine in rats. Epilepsia. 54 (4), 596-604 (2013).
- Myers, T. L., Gonzalez, O. C., Stein, J. B., Bazhenov, M. Characterizing concentration-dependent neural dynamics of 4-Aminopyridine-induced epileptiform activity. Epilepsy J. 4 (2), 128(2018).
- Perreault, P., Avoli, M. Physiology and pharmacology of epileptiform activity induced by 4-aminopyridine in rat hippocampal slices. J Neurophysiol. 65 (4), 771-785 (1991).
- Rutecki, P. A., Lebeda, F. J., Johnston, D. 4-Aminopyridine produces epileptiform activity in hippocampus and enhances synaptic excitation and inhibition. J Neurophysiol. 57 (6), 1911-1924 (1987).
- Chen, Y., Chad, J. E., Cannon, R. C., Wheal, H. V. Reduced Mg2+ blockade of synaptically activated N-methyl-D-aspartate receptor-channels in CA1 pyramidal neurons in kainic acid-lesioned rat hippocampus. Neuroscience. 88 (3), 727-739 (1999).
- Fujiwara-Tsukamoto, Y., Isomura, Y., Takada, M. Comparable GABAergic mechanisms of hippocampal seizure-like activity in posttetanic and low-Mg2+ conditions. J Neurophysiol. 95 (3), 2013-2019 (2006).
- Swartzwelder, H. S., Anderson, W. W., Wilson, W. A. Mechanism of electrographic seizure generation in the hippocampal slice in Mg2+-free medium: the role of GABAa inhibition. Epilepsy Res. 2 (4), 239-245 (1988).
- Trevelyan, A. J., Graham, R. T., Parrish, R. R., Codadu, N. K. Synergistic positive feedback mechanisms underlying seizure initiation. Epilepsy Curr. 23 (1), 38-43 (2023).
- Croning, M. D., Haddad, G. G. Comparison of brain slice chamber designs for investigations of oxygen deprivation in vitro. J Neurosci Methods. 81 (1-2), 103-111 (1998).
- Hajos, N., Mody, I. Establishing a physiological environment for visualized in vitro brain slice recordings by increasing oxygen supply and modifying aCSF content. J Neurosci Methods. 183 (2), 107-113 (2009).
- Huang, Y., Williams, J. C., Johnson, S. M. Brain slice on a chip: opportunities and challenges of applying microfluidic technology to intact tissues. Lab Chip. 12 (12), 2103-2117 (2012).
- Andrew, R. D., et al. The critical role of spreading depolarizations in early brain injury: Consensus and contention. Neurocrit Care. 37 (Suppl 1), 83-101 (2022).
- Devonshire, I. M., Dommett, E. J., Grandy, T. H., Halliday, A. C., Greenfield, S. A. Environmental enrichment differentially modifies specific components of sensory-evoked activity in rat barrel cortex as revealed by simultaneous electrophysiological recordings and optical imaging in vivo. Neuroscience. 170 (2), 662-669 (2010).
- Parrish, R. R., Codadu, N. K., Mackenzie-Gray Scott, C., Trevelyan, A. J. Feedforward inhibition ahead of ictal wavefronts is provided by both parvalbumin- and somatostatin-expressing interneurons. J Physiol. 597 (8), 2297-2314 (2019).
- Wang, H., Jing, M., Li, Y. Lighting up the brain: genetically encoded fluorescent sensors for imaging neurotransmitters and neuromodulators. Curr Opin Neurobiol. 50, 171-178 (2018).
- Yaksi, E., Jamali, A., Diaz Verdugo, C., Jurisch-Yaksi, N. Past, present and future of zebrafish in epilepsy research. FEBS J. 288 (24), 7243-7255 (2021).
- He, M. F., et al. Ex vivo calcium imaging for drosophila model of epilepsy. J Vis Exp. 200, 65825(2023).
- Driscoll, N., et al. Multimodal in vivo recording using transparent graphene microelectrodes illuminates spatiotemporal seizure dynamics at the microscale. Commun Biol. 4 (1), 136(2021).
- Parrish, R. R., Grady, J., Codadu, N. K., Trevelyan, A. J., Racca, C. Simultaneous profiling of activity patterns in multiple neuronal subclasses. J Neurosci Methods. 303, 16-29 (2018).
- Valderhaug, V. D., et al. Criticality as a measure of developing proteinopathy in engineered human neural networks. bioRxiv. , (2020).
- Multi-electrode array (MEASs) to investigate pathogenetic disease mechanisms and pharmacological properties in iPSC-derived neurons modelling neuropsychiatric diseases. Carleo, G., Lee, Y. -S., Secondo, A., Miceli, F., Taglialatela, M. 2022 IEEE International Conference on Metrology for Extended Reality, Artificial Intelligence and Neural Engineering (MetroXRAINE), , Rome, Italy. 667-672 (2022).
- Ruz, I. D., Schultz, S. R. Localising and classifying neurons from high density MEA recordings. J Neurosci Methods. 233, 115-128 (2014).
- Franke, F., Natora, M., Boucsein, C., Munk, M. H. J., Obermayer, K. An online spike detection and spike classification algorithm capable of instantaneous resolution of overlapping spikes. J Comput Neurosci. 29 (1-2), 127-148 (2010).
- Vollgraf, R., Obermayer, K. Improved optimal linear filters for the discrimination of multichannel waveform templates for spike-sorting applications. IEEE Signal Processing Letters. 13 (3), 121-124 (2006).
- Muller, J., et al. High-resolution CMOS MEA platform to study neurons at subcellular, cellular, and network levels. Lab Chip. 15 (13), 2767-2780 (2015).
- Mapelli, L., et al. implementation, and functional validation of a new generation of microneedle 3D high-density CMOS multi-electrode array for brain tissue and spheroids. bioRxiv. , (2022).
- Reddy, D. S., Kuruba, R. Experimental models of status epilepticus and neuronal injury for evaluation of therapeutic interventions. Int J Mol Sci. 14 (9), 18284-18318 (2013).
- Parrish, R. R., Trevelyan, A. J. Stress-testing the brain to understand its breaking points. J Physiol. 596 (11), 2033-2034 (2018).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon