É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Atividade semelhante a convulsões de alta qualidade de fatias cerebrais agudas usando um sistema complementar de matriz de microeletrodos de alta densidade com semicondutor de óxido metálico
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Aqui, descrevemos um protocolo para o uso de sistemas complementares de matriz de microeletrodos de alta densidade de semicondutores de óxido metálico (CMOS-HD-MEAs) para registrar a atividade semelhante a convulsões de fatias cerebrais ex vivo .
Resumo
Os sistemas complementares de matriz de microeletrodos de alta densidade de óxido metálico-semicondutor (CMOS-HD-MEA) podem registrar a atividade neurofisiológica de culturas de células e fatias cerebrais ex vivo em detalhes eletrofisiológicos sem precedentes. Os CMOS-HD-MEAs foram otimizados pela primeira vez para registrar a atividade da unidade neuronal de alta qualidade a partir de culturas de células, mas também demonstraram produzir dados de qualidade a partir de cortes agudos de retina e cerebelar. Os pesquisadores usaram recentemente CMOS-HD-MEAs para registrar potenciais de campo local (LFPs) de fatias cerebrais agudas de roedores corticais. Um LFP de interesse é a atividade semelhante a convulsões. Embora muitos usuários tenham produzido descargas epileptiformes breves e espontâneas usando CMOS-HD-MEAs, poucos usuários produzem de forma confiável atividade semelhante a convulsões de qualidade. Muitos fatores podem contribuir para essa dificuldade, incluindo ruído elétrico, a natureza inconsistente de produzir atividade semelhante a convulsões ao usar câmaras de gravação submersas e a limitação de que os chips 2D CMOS-MEA registram apenas a partir da superfície da fatia do cérebro. As técnicas detalhadas neste protocolo devem permitir que os usuários induzam e registrem consistentemente atividades semelhantes a convulsões de alta qualidade de fatias cerebrais agudas com um sistema CMOS-HD-MEA. Além disso, este protocolo descreve o tratamento adequado dos chips CMOS-HD-MEA, o gerenciamento de soluções e fatias cerebrais durante a experimentação e a manutenção do equipamento.
Introdução
Os sistemas de matriz de microeletrodos de alta densidade (HD-MEA) disponíveis comercialmente, que incluem um chip MEA com milhares de pontos de gravação 1,2 e uma plataforma MEA para amplificar e digitalizar os dados, são uma ferramenta emergente para a pesquisa eletrofisiológica. Esses sistemas HD-MEA usam tecnologia de semicondutor de óxido metálico complementar (CMOS) para registrar dados eletrofisiológicos com alta sensibilidade de culturas de células e preparações de fatias cerebrais ex vivo. Esses sistemas MEA oferecem resolução espacial e temporal sem precedentes para pesquisas neurofisiológicas por meio de alta densidade de eletrodos e relações sinal-ruídode qualidade 3. Essa tecnologia tem sido usada principalmente para estudar potenciais de ação extracelulares, mas também pode capturar potenciais de campo local (LFPs) de alta qualidade de várias preparações neuronais de fatias cerebrais 4,5,6,7,8,9,10,11,12,13,14,15 . Devido à capacidade de gravação de alta resolução dos sistemas CMOS-HD-MEA mencionada acima, os usuários podem rastrear a atividade eletrofisiológica com grande precisão espacial 16,17,18. Esse recurso é particularmente relevante para rastrear padrões de propagação de LFPs de rede 5,12,15,19,20,21. Portanto, os sistemas CMOS-HD-MEA podem fornecer uma compreensão sem precedentes dos padrões de propagação da atividade fisiológica e patológica de várias culturas de células e preparações de fatias de cérebro. É importante notar que esses recursos dos sistemas CMOS-HD-MEA podem permitir que os pesquisadores contrastem os padrões de convulsões de diferentes regiões do cérebro simultaneamente e testem como vários compostos antiepilépticos afetam esses padrões. Ao fazer isso, fornece um método inovador para estudar a ictogênese e a propagação ictal e para entender como a farmacologia interrompe a atividade patológica da rede 7,10,14. Portanto, essas novas capacidades dos sistemas CMOS-HD-MEA podem contribuir significativamente para a pesquisa de distúrbios neurológicos, bem como auxiliar na pesquisa de descoberta de medicamentos 5,7,11,22. Nosso objetivo é fornecer detalhes sobre o uso de sistemas CMOS-HD-MEA para estudar a atividade semelhante a convulsões.
Ao usar sistemas CMOS-HD-MEA para estudar LFPs, como a atividade epileptiforme em fatias cerebrais agudas, os usuários podem enfrentar muitos desafios, incluindo ruído elétrico debilitante, manter a fatia saudável durante a experimentação e detectar um sinal de qualidade de um chip CMOS-MEA bidimensional (2D) que grava apenas da superfície da fatia cerebral. Este protocolo descreve as etapas básicas para o aterramento adequado da plataforma MEA e outros equipamentos usados na experimentação, uma etapa crucial que pode exigir personalização individual para cada configuração de laboratório. Além disso, discutimos as etapas para ajudar a manter o corte cerebral saudável durante longas gravações nas câmaras submersas usadas com os sistemas CMOS-HD-MEA 23,24,25. Além disso, em contraste com os métodos de gravação eletrofisiológica mais comuns, que registram profundamente a fatia do cérebro, a maioria dos sistemas CMOS-HD-MEA usa chips 2D que não penetram na fatia. Portanto, esses sistemas requerem uma camada externa neuronal saudável para produzir a maioria dos sinais LFP registrados. Outros desafios incluem a visualização da enorme quantidade de dados gerados por milhares de eletrodos. Para superar esses desafios, recomendamos um protocolo simples, mas eficaz, que aumente a probabilidade de alcançar uma atividade epileptiforme de rede de alta qualidade que se propaga pela fatia cerebral. Também incluímos uma breve descrição de uma interface gráfica do usuário (GUI) disponível publicamente que desenvolvemos com recursos associados para auxiliar na visualização de dados10.
Publicações anteriores forneceram protocolos relacionados para o uso de sistemas de registro MEA 26,27,28,29. No entanto, este trabalho visa auxiliar os experimentadores que usam sistemas CMOS-HD-MEA com chips 2D, especificamente aqueles que buscam estudar a atividade epileptiforme de alta qualidade a partir de fatias de cérebro. Além disso, comparamos duas das manipulações de solução mais comuns para indução de atividade semelhante a convulsões, ou seja, os paradigmas 0 Mg2+ e 4-AP, para ajudar os usuários a identificar os meios convulsivantes mais apropriados para sua aplicação específica. Embora o protocolo seja focado principalmente na geração de atividade semelhante a convulsões, ele pode ser modificado para explorar outros fenômenos eletrofisiológicos usando fatias de cérebro.
Protocolo
Os procedimentos envolvendo camundongos foram aprovados pelo Comitê Institucional de Cuidados e Uso de Animais (IACUC) da Universidade Brigham Young. Camundongos C57BL/6 machos e fêmeas (n = 8) com idade mínima de P21 foram utilizados nos experimentos a seguir.
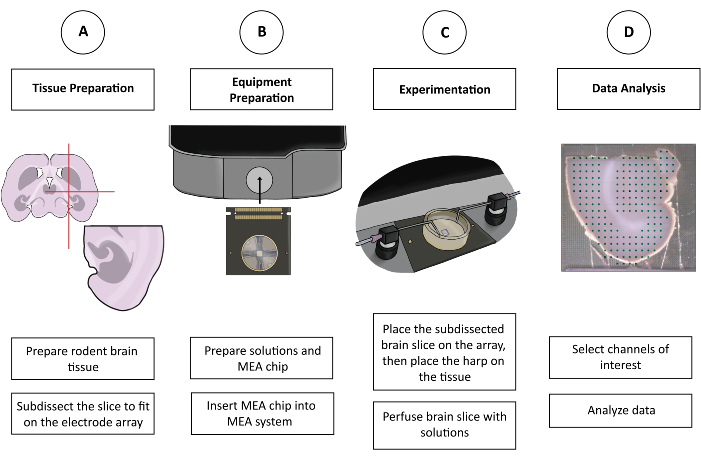
Figura 1: Figura esquemática da experimentação CMOS-HD-MEA. (A) A fatia do cérebro é preparada pelo método de corte preferido e subdissecada para caber no MEA. (B) Prepare as soluções e o chip CMOS-HD-MEA. (C) A fatia de cérebro subdissecada é colocada no feixe de eletrodos e banhada nas soluções apropriadas. (D) Os canais relevantes são selecionados a partir dos dados coletados. Os dados são então preparados para análise no programa preferido do usuário. Clique aqui para ver uma versão maior desta figura.
| Nome | Concentração (mM) | g/L | ||
| Cloreto de sódio (NaCl) | 126 | 7.36 | ||
| Cloreto de potássio (KCl) | 3.5 | 0.261 | ||
| Fosfato de di-hidrogênio sódico (NaH2PO4) | 1.26 | 0.151 | ||
| Bicarbonato de sódio (NaHCO3) | 26 | 2.18 | ||
| Glicose (C6H12O6) | 10 | 1.80 | ||
| Cloreto de magnésio (MgCl2) | 1 (de 1 M estoque) | 1 mL | ||
| Cloreto de cálcio (CaCl2) | 2 (a partir de 1 M de estoque) | 2 mL | ||
Tabela 1: Solução de aCSF.
1. Preparando soluções
- Prepare as soluções experimentais.
- Prepare 1 L de líquido cefalorraquidiano artificial (aCSF) (consulte a Tabela 1 para obter detalhes).
- Prepare 1 L de solução pró-convulsivante.
NOTA: As soluções usadas para gerar atividade semelhante a convulsões neste protocolo utilizaram aCSF com 100 μM de 4-aminopiridina (4-AP) ou aCSF livre de íons de magnésio. - Carbogenar todas as soluções por pelo menos 10 minutos antes de usar pedras porosas.
- Obter um copo para rejeitar as soluções.
- Colocar 1 L de aCSF, 1 L de solução pró-convulsivante e o copo de descarte numa superfície próxima do sistema de perfusão.
- Preparando as soluções de câmara de corte e retenção
- Prepare 0,5 L de aCSF, omitindo CaCl2 e adicionando 3 mM MgCl2 (em oposição a 1 mM) para criar uma solução de corte única. Guarde esta solução para uso na preparação de fatias de cérebro de roedores, colocando-a perto da área de preparação de fatias de cérebro agudas.
- Prepare 0,5 L de aCSF e coloque-o em uma câmara de retenção de tecido que será usada para reter fatias para uso em experimentação. Insira pedras porosas na câmara de retenção de tecido e carbogene a solução por pelo menos 10 minutos antes de colocar as fatias subdissecadas na câmara.
2. Preparando fatias de cérebro de roedor
- Prepare todas as soluções conforme descrito acima (ver secção 1). Certifique-se de que todas as soluções estejam devidamente carbogenadas durante o processo de preparação.
NOTA: A solução de corte (consulte a seção 1.2) é recomendada, mas outras soluções de corte podem ser usadas para obter fatias de cérebro de qualidade, como uma solução de corte desacarose 30. - Use um micrótomo vibratório para fazer fatias de cérebro de roedor de 350 μm. Os protocolos para uso desses instrumentos estão listados nas referências 30,31,32,33.
- Prepare as fatias de cérebro de roedor para caber na área do eletrodo de gravação do chip MEA (veja a Figura 2). Use uma lâmina cirúrgica tamanho 10 para subdissecar as fatias, conforme mostrado na Figura 1A e na Figura 2A , rolando suavemente a lâmina para frente e para trás na fatia do cérebro. Execute a subdissecção na câmara de corte do micrótomo vibratório. Tenha cuidado para não danificar a fatia cerebral subdissecada.
- Colocar as fatias subdissecadas numa câmara de fixação de tecido cheia de aCSF. Certifique-se de que a câmara foi carbogenada por pelo menos 10 minutos antes de adicionar as fatias subdissecadas.
3. Preparação do equipamento
- Preparando os chips e o sistema MEA (Figura 2 e Figura 3)
NOTA: Materiais duros como plásticos e metais podem danificar facilmente o chip MEA se entrarem em contato com força com a parte inferior do chip. Ao usar pipetas para transferir a solução para dentro ou para fora dos chips, não entre em contato com a parte inferior do poço do chip com a pipeta, especialmente os eletrodos de referência nas laterais do poço do chip e os eletrodos de registro no centro do poço do chip (Figura 2C). Em vez disso, adicione a solução passando o mouse sobre a parte inferior do poço de cavacos ou entrando em contato ou aproximando as bordas plásticas do poço de cavacos. Para retirar toda a solução facilmente, incline levemente o cavaco para permitir que a solução se acumule em um lado do poço e remova-o do topo da solução agrupada ou use um pano antiestático para enxugar levemente as áreas que ainda têm a solução. Não transporte o cavaco segurando o poço do cavaco ou os pinos do cavaco (Figura 2C). O poço de cavacos pode conter aproximadamente 4 mL de solução. Para as etapas subsequentes, encha bem o cavaco com aproximadamente 2 mL de solução, se não especificado de outra forma.- Designe pipetas de transferência para várias tarefas antes de iniciar a preparação do cavaco. Rotule uma pipeta de transferência para etanol, uma para resíduos, outra para aCSF e outras para quaisquer soluções restantes para evitar a mistura não intencional.
- Encha o poço do chip MEA com etanol à prova de 190 para que o fundo do poço do chip fique completamente coberto (Figura 3). Deixe o etanol descansar por 30-60 s e remova-o com uma pipeta de descarte.
- Encha o poço do chip MEA com aCSF e remova-o com uma pipeta de descarte para enxaguar o etanol residual do poço do chip. Adicione e remova o aCSF do poço de cavacos três vezes, usando as pipetas de resíduos e aCSF previamente designadas. Depois de lavar bem o chip três vezes, adicione aCSF e deixe repousar por pelo menos 30 s.
NOTA: Os eletrodos de registro exibem a menor quantidade de ruído de interações hidrofóbicas quando o aCSF permanece na câmara por pelo menos 45 min após o enxágue com etanol e aCSF. - Antes de encaixar o chip MEA, molhe um pano antiestático com etanol à prova de 190 e use-o para limpar os pinos do chip (Figura 2C).
- Deslize suavemente o chip MEA na plataforma MEA e engate o mecanismo de encaixe para travar o chip no lugar.
- Verifique se há bolhas nos eletrodos de registro e referência (Figura 2C). Se houver bolhas, pegue um pincel limpo e passe levemente sobre os eletrodos para removê-los.
- Verifique se há ruído no chip usando o software CMOS-HD-MEA34 e escaneie visualmente o mapa de cores falsas em busca de bolhas, oscilações não biológicas ou picos causados por interferência elétrica. Aterre o sistema MEA adequadamente para negar qualquer ruído encontrado.
NOTA: A configuração de aterramento dependerá do ambiente de gravação. Para os experimentos deste protocolo, a plataforma MEA e o sistema de perfusão foram aterrados.
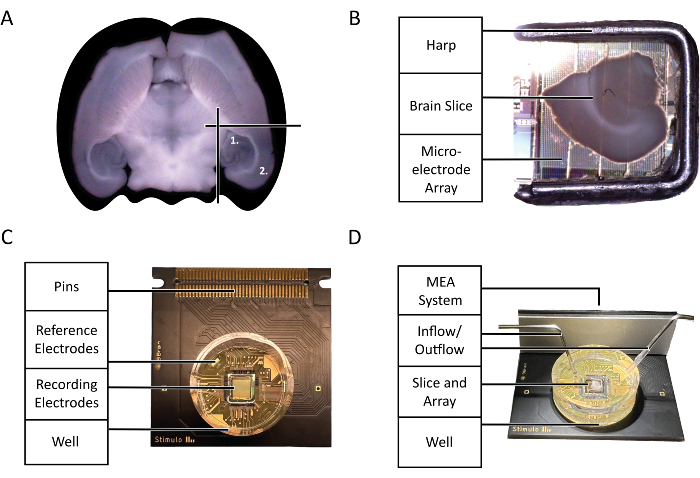
Figura 2: Diagramas de configuração e tecnologia. (A) Diagrama da seleção de fatias agudas de cérebro de camundongo usadas no experimento destacado por este protocolo. (1) Região do hipocampo (2) Região do neocórtex. (B) A colocação adequada de uma fatia aguda de cérebro de camundongo e harpa no microeletrodo Array (MEA). (C) A anatomia de um chip 3Brain Accura CMOS-HD-MEA. (D) A configuração adequada das entradas e saídas de perfusão. A entrada deve estar no fundo do poço do chip, enquanto a saída deve estar no lado oposto da entrada na parte superior do poço do chip para garantir um fluxo constante de aCSF fresco e oxigenado. Clique aqui para ver uma versão maior desta figura.
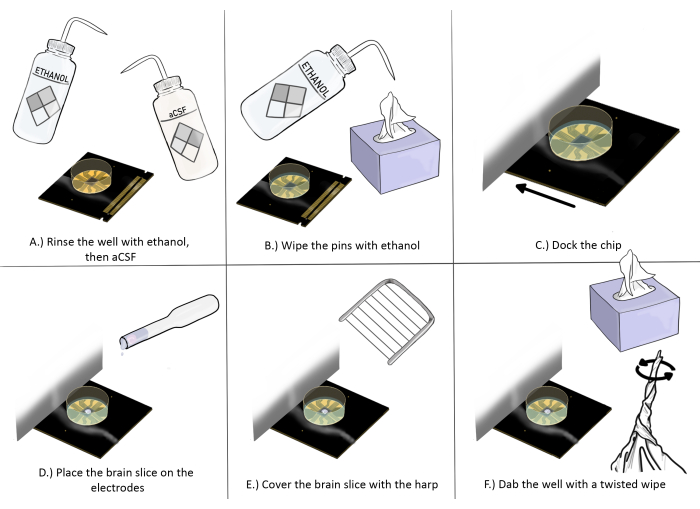
Figura 3: Diagrama esquemático de preparação e colocação de chips para experimentos de fatias de cérebro. (A) Enxágue bem o chip com etanol uma vez e depois com aCSF três vezes. (B) Limpe os pinos com etanol usando um pano antiestático. (C) Encaixe o chip. (D) Coloque a fatia do cérebro nos eletrodos. (E) Coloque a harpa na fatia do cérebro (consulte a Figura 2 para obter as diretrizes de colocação adequadas). (F) Enxugue o canto do poço dos eletrodos de gravação perto da fatia do cérebro com um pano antiestático torcido. Clique aqui para ver uma versão maior desta figura.
4. Experimentação
- Colocando a fatia do cérebro
- Coloque uma harpa de platina em um barco de pesagem (ou outra superfície limpa) perto da plataforma MEA. Cubra a harpa com cerca de 3mL de aCSF para reduzir suas tendências hidrofóbicas.
- Use uma tesoura para remover a parte fina da ponta de uma pipeta de transferência. Corte um terço, cerca de 1,5 pol., da ponta da pipeta.
NOTA: Esta pipeta modificada não contrai ou danifica a fatia do cérebro durante a coleta e o transporte. - Colete uma fatia de cérebro da câmara de retenção da fatia com a pipeta modificada. Dispense suavemente a fatia de cérebro e qualquer solução da pipeta no poço do cavaco. Para posicionar a fatia corretamente, libere suavemente mais aCSF de uma pipeta de transferência para manipular a posição da fatia do cérebro ou use um pincel macio para criar uma corrente na solução que empurra a fatia do cérebro para os eletrodos de gravação. Limite o contato com os eletrodos de gravação ou fatia do cérebro para minimizar os danos.
- Usando uma pinça, coloque suavemente a harpa sobre a fatia do cérebro com os fios para baixo para pressionar a fatia nos eletrodos de gravação. Evite entrar em contato com o feixe de eletrodos com a harpa. Oriente a harpa de forma que o lado sem armação fique voltado para a agulha de entrada e a armação da harpa não entre em contato com nenhum dos eletrodos de gravação (Figura 2C, D).
- Pegue uma pipeta de descarte e remova o excesso de aCSF. Pegue um pano antiestático, gire um canto para criar uma ponta e use-o para absorver o aCSF restante ao redor dos eletrodos de gravação sem tocar nos eletrodos de gravação, fatia de cérebro ou harpa (Figura 2).
- Usando uma pipeta de aCSF designada, adicione rapidamente aCSF carbogenado suficiente para cobrir a fatia do cérebro, cerca de 2 mL.
- Repita a etapa 4.1.5. e 4.1.6. mais duas vezes.
- Encha o poço com aCSF carbogenado até que o poço esteja aproximadamente 3/4 cheio, cerca de 3 mL.
- Tire uma foto da fatia do cérebro no chip MEA com um microscópio ou câmera. Certifique-se de que a foto tenha uma resolução alta o suficiente para ver as bordas do conjunto de eletrodos de gravação e a anatomia da fatia do cérebro.
- Executando o experimento
- Operando o sistema de perfusão
- Coloque os tubos de entrada e saída no béquer cheio de aCSF e as agulhas de entrada e saída no poço de cavacos. Coloque a agulha de entrada perto da parte inferior do chip do poço, do lado de fora dos eletrodos de gravação. Coloque a agulha de saída perto do topo do cavaco bem em direção à borda, de modo que o líquido suba quase até a borda do poço do cavaco, cerca de 4 mL, e a fatia fique submersa durante todo o experimento (Figura 2D).
- Defina o fluxo de entrada de perfusão para 5 mL / min e o fluxo de saída de perfusão para 7 mL / min.
NOTA: Recomenda-se que a taxa de saída exceda a taxa de entrada para evitar o transbordamento da solução para fora do poço do cavaco e para criar uma corrente de solução sobre a fatia do cérebro. - Ligue a entrada e a saída. Remova a agulha de entrada do poço do cavaco até que a agulha comece a produzir solução em vez de ar. Em seguida, coloque a agulha de volta em sua posição dentro do poço do chip, conforme descrito na etapa 4.2.1.1.
- Use um aquecedor de solução para manter a solução na temperatura fisiológica ou próxima dela, em torno de 34-37 °C.
- Deixe o aCSF perfundir sobre a fatia do cérebro por 10 min. Isso permitirá que a fatia se ajuste ao ambiente de gravação.
- Decorridos 10 minutos, desloque o tubo de saída para o copo de eliminação. Em seguida, desloque o tubo de entrada para o copo que contém a solução pró-convulsivante. Deixar que o líquido de aCSF não convulsivante seja eliminado do sistema de perfusão para o copo de descarte durante 10 min.
- Transferir o tubo de saída para o copo que contém a solução pró-convulsivante.
- Deixe a solução pró-convulsivante circular até que o experimento termine.
- Se a duração do registro do corte exceder 2 h, considere preparar outra solução pró-convulsivante para compensar o consumo de glicose do fatia cerebral ativa.
- Troca de fatias cerebrais
- Desligue o fluxo de entrada. Desligue a vazão 10-15 s depois.
- Use uma pinça para remover a harpa e coloque-a em um barco de pesagem ou outra superfície.
- Use uma pipeta modificada para extrair e descartar a fatia de cérebro. Não toque nos eletrodos de referência ou no conjunto de eletrodos de gravação (Figura 2C).
- Colocar o tubo de entrada no copo que contém a solução não convulsivante de aCSF. Colocar o tubo de saída no copo de eliminação. Deixe as agulhas de entrada e saída no poço de cavacos. Execute o sistema de perfusão por 10 min para remover toda a solução pró-convulsivante residual do poço de cavacos e do sistema de perfusão. Comece o processo de experimentação novamente com uma nova fatia de cérebro (começando na etapa 4.1).
- Operando o sistema de perfusão
- Terminando o experimento
- Limpando o equipamento
- Remova a harpa, a fatia de cérebro e a solução do poço, respectivamente.
- Desencaixe o chip MEA, coloque-o em uma superfície limpa e encha bem o chip com água deionizada.
- Descarte a água deionizada para limpar quaisquer sais deixados das soluções de aCSF.
- Usando uma pipeta de transferência, encha bem o chip com uma solução detergente. Passe a ponta da pipeta de transferência no poço do chip e aponte-a para os eletrodos de gravação. Aperte e solte o detergente rápida e repetidamente na pipeta de transferência para lavar vigorosamente o detergente sobre os eletrodos de gravação por 1 min. Deixe o detergente descansar por 5-10 min.
- Remova o detergente e enxágue bem o cavaco 4-6 vezes com água deionizada para remover a solução detergente e quaisquer solutos restantes.
- Remova toda a água do poço do cavaco, coloque um pano antiestático sobre o cavaco MEA e deixe durante a noite para permitir que o cavaco seque.
- Coloque um barco de pesagem ou outra placa estanque na plataforma de registro MEA onde o chip MEA estava anteriormente repousado. Coloque a harpa no centro da placa e coloque as agulhas de entrada e saída em ambos os lados da harpa.
- Limpe o sistema de perfusão e a harpa enviando 50 mL de água, 50 mL de etanol à prova de 140 e 200 mL de água, respectivamente, através do tubo para o copo de descarte.
NOTA: Neste momento, as velocidades de perfusão podem ser aumentadas para diminuir o tempo de limpeza, mas a velocidade de saída deve sempre exceder a velocidade de entrada para evitar inundações. - Limpe as pedras de carbogênio colocando-as em um copo de água destilada e enviando o carbogênio através do tubo por 5 min. Coloque-os para descansar em uma superfície seca e limpa durante a noite e cubra-os para evitar o acúmulo de poeira.
- Limpando o equipamento
5. Análise dos dados
NOTA: Há uma variedade de pacotes de análise usados para analisar dados eletrofisiológicos produzidos por CMOS-HD-MEAs, incluindo BrainWave da 3Brain, Yet Another Spike Sorter (YASS) e ferramentas Python personalizadas 34,35,36,37. Extraímos dados do formato de arquivo de dados BrainWave para uso na plataforma Xenon LFP Analysis para gerar os dados apresentados na Figura 4 e Figura 5. O código Matlab personalizado foi usado para analisar os dados na Figura 6. Os protocolos para a plataforma de análise de xenônio LFP estão disponíveis publicamente10. As etapas do protocolo a seguir são específicas para gravações feitas com Brainwave 438; Para outros sistemas, consulte a documentação de suporte relacionada a esses sistemas 34,35,36,37. Uma visão geral das etapas de análise tomadas para produzir os dados com este protocolo é fornecida abaixo. Para obter detalhes completos sobre como exportar, visualizar e analisar dados, incluindo vídeos tutoriais e todos os arquivos de código relevantes, consulte39.
- Exporte o arquivo de gravação para análise.
- Crie uma pasta para os arquivos de gravação de dados que serão reduzidos.
- Corte a imagem tirada durante a etapa 4.1.9 para incluir apenas a matriz de gravação com as fatias de cérebro.
- Abra o arquivo ExportToHDF5-ChannelSelection.py (este arquivo de código pode ser acessado no GitHub em xenon-lfp-analysis/code-files/3Brain-processing40) e copie e cole o URL listado na parte superior da janela em um navegador da Internet.
- Insira o caminho do arquivo para o arquivo de gravação de dados e carregue a imagem cortada da matriz associada à gravação.
- Em Selecionar canais para exportação, use a ferramenta de seleção de laço para selecionar as regiões hipocampal e neocortical mostradas na imagem recortada da matriz.
- Defina o número de linhas a serem ignoradas e o número de colunas a serem ignoradas para 2.
NOTA: Os arquivos de gravação geralmente são grandes; Se apropriado para a análise desejada, os dados exportados podem ser reduzidos. Existem opções para reduzir a amostragem espacialmente (pulando linhas e colunas de eletrodos de gravação) e temporalmente (reduzindo a amostragem para uma frequência de amostragem mais baixa). Na análise, cada 2 linhas e 2 colunas são ignoradas; Isso permite a redução da amostragem dos dados espacialmente sem ser tendencioso para determinados canais. Cada canal está distante o suficiente para que as diferenças de atividade possam ser significativas entre eles; portanto, nenhuma média de canal é executada. Ao pular linhas e colunas dos eletrodos, os dados de canais não selecionados não são incluídos quando o arquivo de saída é exportado. - Defina a frequência de redução da amostragem para 300 Hz.
- Clique no botão Exportar canais para gerar o arquivo de seleção de canal.
NOTA: O software salvará automaticamente o arquivo de seleção de canal na pasta onde o arquivo de gravação de dados foi salvo. - Abra o arquivo ExportToHDF5-ExtractDownsample.py (também encontrado no GitHub em xenon-lfp-analysis/code-files/3brain-processing40), cole o caminho do arquivo da pasta que contém o(s) arquivo(s) de seleção de canal e o(s) arquivo(s) de gravação de dados e pressione enter no teclado para gerar a versão reduzida do arquivo de gravação de dados.
- Usando a plataforma de análise
- No prompt de comando do computador, digite run_lfp_analysis e pressione Enter para abrir a plataforma de análise.
- Insira os caminhos da imagem e o arquivo de gravação com resolução reduzida e clique em enviar.
- Em Selecionar um intervalo de tempo para análise, selecione a gravação inteira.
- Em Selecionar canais para plotagens, destaque o hipocampo como Grupo-1 e o neocórtex como Grupo-2.
- Em Raster do canal, defina o Limite para 0,06 mV e a Duração do tempo para 0,02 s.
- Clique em Aplicar configurações e gerar raster.
- Use o gráfico raster para explorar diferentes canais que mostram padrões de atividade de interesse.
NOTA: Consulte a Figura 4 para exemplos de atividade semelhante a convulsões de alta qualidade (Figura 4A-D) versus atividade abaixo do ideal (Figura 4E-F).
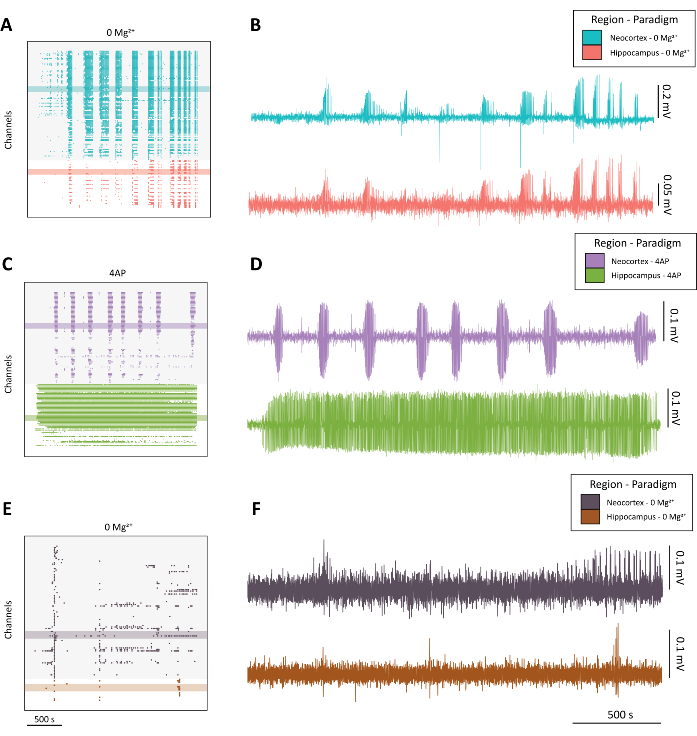
Figura 4: Exemplo de evolução da atividade epileptiforme dos paradigmas 0 Mg2+ e 4-AP. (A) Exemplo de gráfico raster da aplicação de aCSF com 0 Mg2+ durante aproximadamente 40 min. (B) Exemplo de traços eletrofisiológicos retirados do neocórtex (azul) e hipocampo (vermelho) demonstrando atividade epileptiforme do paradigma 0 Mg2+ . (C) Exemplo de gráfico raster da aplicação 100 mM 4-AP durante aproximadamente 40 min. (D) Exemplo de traços eletrofisiológicos retirados do neocórtex (roxo) e hipocampo (verde) demonstrando atividade epileptiforme da aplicação de 4-AP. (E) Exemplo de gráfico raster da aplicação de aCSF com 0 Mg2+ durante aproximadamente 40 minutos mostrando atividade de explosão em oposição à atividade semelhante a convulsões, conforme encontrado nos outros traços representativos. (F) Exemplo de traços eletrofisiológicos retirados do neocórtex (roxo escuro) e hipocampo (ferrugem) demonstrando atividade subótima do paradigma0 Mg 2+ destinado à comparação com a atividade semelhante a convulsões de qualidade encontrada em B e D. Clique aqui para ver uma versão maior desta figura.
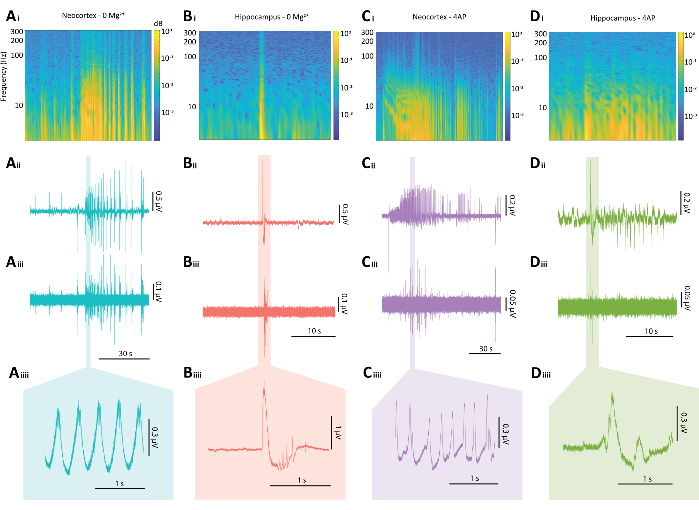
Figura 5: Resultados representativos de descargas epileptiformes dos paradigmas0 Mg 2+ e 4-AP. (A) Exemplos de gráficos de um evento típico semelhante a uma convulsão neocortical induzido pelo paradigma 0 Mg2+ , incluindo (Ai) um espectrograma de um evento semelhante a uma convulsão, (Aii) o traço eletrofisiológico associado, (Aiii) um filtro passa-alta de 80 Hz aplicado ao traço de Aii, (Aiiii) e uma seção ampliada do traço de Aii. (B) Gráficos de exemplo de uma explosão epileptiforme típica do hipocampo induzida pelo paradigma 0 Mg2+ , incluindo (Bi) um espectrograma da explosão epileptiforme, (Bii) o traço eletrofisiológico associado, (Biii) um filtro passa-alta de 80 Hz aplicado ao traço de Bii, (Biiii) e uma seção ampliada do traço de Bii (C) Gráficos de exemplo de um evento típico semelhante a uma convulsão neocortical induzido pelo paradigma 4-AP, incluindo (Ci) um espectrograma de atividade epileptiforme, (Cii) o traço eletrofisiológico associado, (Ciii) um filtro passa-alta de 80 Hz aplicado ao traço de Cii, (Ciiii) e uma seção ampliada do traço de Cii (D) Gráficos de exemplo de uma explosão epileptiforme do hipocampo sob o paradigma 4-AP, incluindo (Di) um espectrograma de atividade epileptiforme, (Dii) o traço eletrofisiológico associado, (Diii) um filtro passa-alta de 80 Hz aplicado ao traço de Dii, (Diiii) e uma seção ampliada do traço de Dii. Clique aqui para ver uma versão maior desta figura.
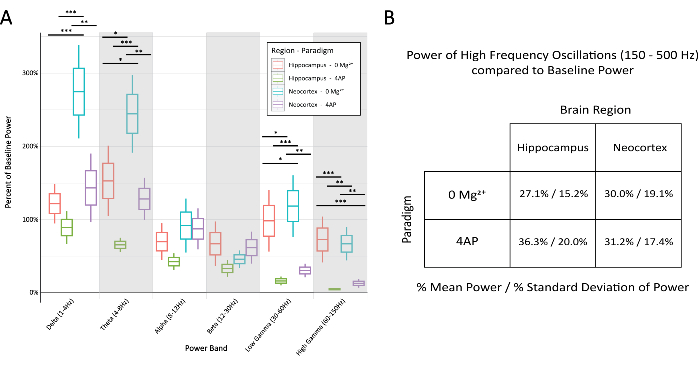
Figura 6: Comparação da porcentagem de potência basal nas várias bandas entre paradigma e região do cérebro durante descargas epileptiformes estereotipadas. (A) A potência durante as descargas epileptiformes foi significativamente diferente entre os paradigmas e as regiões cerebrais para a maioria das bandas de frequência (ANOVA de 2 vias com teste de Tukey, * P < 0,05, ** P < 0,001, * * P < 0,0001). A linha do meio para cada caixa representa a média, as bordas da caixa ±1 erro padrão da média (SEM) e as linhas mais externas ±2 SEM. (B) Tanto os paradigmas quanto as regiões cerebrais demonstraram poder limitado em bandas relacionadas à atividade de alta frequência acima de 150 Hz. Clique aqui para ver uma versão maior desta figura.
Resultados
Como é padrão ao visualizar a atividade de muitos canais 1,4,5,10, achamos benéfico primeiro gerar um gráfico raster dos dados que adquirimos com o CMOS-HD-MEA (Figura 4A,C,E). Esse gráfico pode criar uma visão panorâmica da atividade em todos os canais de gravação em cada fatia do cérebr...
Discussão
Este protocolo inclui diretrizes específicas relacionadas ao gerenciamento de fatias cerebrais agudas que abordam problemas comuns enfrentados pelos usuários de CMOS-HD-MEA, ou seja, desenvolvimento de ruído sob a fatia cerebral e manutenção de um ambiente saudável para a fatia cerebral. O desenvolvimento de ruído sob a fatia ocorre quando a fatia não está devidamente aderida à matriz; Se a fatia do cérebro não estiver adequadamente aderida, bolsas de ar podem se formar embai...
Divulgações
Os autores declaram que não há conflitos de interesse associados a esta pesquisa.
Agradecimentos
Os autores agradecem aos antigos e atuais membros do laboratório Parrish por suas edições neste manuscrito. Também gostaríamos de agradecer a Alessandro Maccione, da 3Brain, por seu feedback sobre este trabalho. Este trabalho foi financiado por um Prêmio de Investigador Júnior AES / EF e pelas Faculdades de Ciências da Vida e de Ciências Físicas e Matemáticas da Universidade Brigham Young.
Materiais
| Name | Company | Catalog Number | Comments |
| 2D Workbench | Cloudray | LM04CLLD26B | |
| 4-Aminopyridine | Sigma-Aldrich | 275875 | |
| Accura Chip | 3Brain | Accura HD-MEA | CMOS-HD-MEA chip |
| Agarose | Thermo Fisher Scientific | BP160-100 | |
| Vibration isolation table | Kinetic Systems | 91010124 | |
| Beaker for the slice holding chamber, 270 mL | VWR | 10754-772 | |
| BioCam | 3Brain | BioCAM DupleX | CMOS-HD-MEA platform |
| Brainwave Software | 3Brain | Version 4 | CMOS-HD-MEA software |
| Calcium Chloride | Thermo Fisher Scientific | BP510-500 | |
| Carbogen | Airgas | X02OX95C2003102 | |
| Carbogen | Airgas | 12005 | |
| Carbogen Stones | Supelco | 59277 | |
| Compresstome | Precissionary | VF-300-0Z | |
| Computer | Dell | Precission3650 | |
| Crocodile Clip Grounding Cables | JWQIDI | B06WGZG17W | |
| Detergent | Metrex | 10-4100-0000 | |
| D-Glucose | Macron Fine Chemicals | 4912-12 | |
| Dihydrogen Sodium Phosphate | Thermo Fisher Scientific | BP329-500 | |
| DinoCam | Dino-Lite | AM73915MZTL | |
| Ethanol | Thermo Fisher Scientific | A407P-4 | |
| Forceps | Fine Science Tools | 11980-13 | |
| Hot plate | Thermo Fisher Scientific | SP88857200 | |
| Ice Machine | Hoshizaki | F801MWH | |
| Inflow and outflow needles | Jensen Global | JG 18-3.0X | |
| Inline Solution Heater | Warner Instruments | SH-27B | |
| Isofluorine | Dechra | 08PB-STE22002-0122 | |
| Kim Wipes | Thermo Fisher Scientific | 06-666 | |
| Magnesium Chloride | Thermo Fisher Scientific | FLM33500 | |
| Micropipets | Gilson | F144069 | |
| Mili-Q Water Filter | Mili-Q | ZR0Q008WW | |
| Paintbrush | Daler Rowney | AF85 Round: 0 | |
| Paper Filter | Whatman | EW-06648-24 | |
| Parafilm | American National Can | PM996 | |
| Perfusion System | Multi Channel System | PPS2 | |
| Pipetor | Thermo Fisher Scientific | FB14955202 | |
| Platinum Harp | 3Brain | 3Brain | |
| Potassium Chloride | Thermo Fisher Scientific | P330-3 | |
| Razor blade | Personna | BP9020 | |
| Scale | Metter Toledo | AB204 | |
| Scissors | Solingen | 92008 | |
| Slice Holding Chamber | Custom | Custom | Custom 3D Printer Design, available upon request |
| Sodium Bicarbonate | Macron Fine Chemicals | 7412-06 | |
| Sodium Chloride | Thermo Fisher Scientific | S271-3 | |
| Temperature Control Box | Warner Instruments | TC344B | |
| Transfer Pipettes | Genesee Scientific | 30-200 | |
| Tubing | Tygon | B-44-3 TPE | |
| Vibratome VZ-300 | Precissionary | VF-00-VM-NC | |
| Weigh Boat | Electron Microscopy Sciences | 70040 |
Referências
- Obien, M. E. J., Frey, U. Large-scale, high-resolution microelectrode arrays for interrogation of neurons and networks. Adv Neurobiol. 22, 83-123 (2019).
- Schroter, M., et al. Functional imaging of brain organoids using high-density microelectrode arrays. MRS Bull. 47 (6), 530-544 (2022).
- Miccoli, B., et al. High-density electrical recording and impedance imaging with a multi-modal CMOS multi-electrode array chip. Front Neurosci. 13, 641 (2019).
- Emery, B. A., Hu, X., Khanzada, S., Kempermann, G., Amin, H. High-resolution CMOS-based biosensor for assessing hippocampal circuit dynamics in experience-dependent plasticity. Biosens Bioelectron. 237, 115471 (2023).
- Ferrea, E., et al. high-resolution electrophysiological imaging of field potentials in brain slices with microelectronic multielectrode arrays. Front Neural Circuits. 6, 80 (2012).
- Gagliano, G., et al. Non-linear frequency dependence of neurovascular coupling in the cerebellar cortex implies vasodilation-vasoconstriction competition. Cells. 11 (6), 1047 (2022).
- Goodchild, S. J., et al. Molecular pharmacology of selective Na(V)1.6 and dual Na(V)1.6/Na(V)1.2 channel inhibitors that suppress excitatory neuronal activity ex vivo. ACS Chem Neurosci. 15 (6), 1169-1184 (2024).
- Hu, X., Khanzada, S., Klutsch, D., Calegari, F., Amin, H. Implementation of biohybrid olfactory bulb on a high-density CMOS-chip to reveal large-scale spatiotemporal circuit information. Biosens Bioelectron. 198, 113834 (2022).
- Kim, S., et al. Alteration of neural network and hippocampal slice activation through exosomes derived from 5XFAD nasal lavage fluid. Int J Mol Sci. 24 (18), 14064 (2023).
- Mahadevan, A., Codadu, N. K., Parrish, R. R. Xenon LFP analysis platform is a novel graphical user interface for analysis of local field potential from large-scale MEA recordings. Front Neurosci. 16, 904931 (2022).
- Medrihan, L., Ferrea, E., Greco, B., Baldelli, P., Benfenati, F. Asynchronous GABA release is a key determinant of tonic inhibition and controls neuronal excitability: A study in the synapsin II-/- mouse. Cereb Cortex. 25 (10), 3356-3368 (2015).
- Monteverdi, A., Di Domenico, D., D'Angelo, E., Mapelli, L. Anisotropy and frequency dependence of signal propagation in the cerebellar circuit revealed by high-density multielectrode array recordings. Biomedicines. 11 (5), 1475 (2023).
- Obien, M. E. J., Hierlemann, A., Frey, U. Accurate signal-source localization in brain slices by means of high-density microelectrode arrays. Sci Rep. 9 (1), 788 (2019).
- Thouta, S., et al. Pharmacological determination of the fractional block of Nav channels required to impair neuronal excitability and ex vivo seizures. Front Cell Neurosci. 16, 964691 (2022).
- Tognolina, M., Monteverdi, A., D'Angelo, E. Discovering microcircuit secrets with multi-spot imaging and electrophysiological recordings: The example of cerebellar network dynamics. Front Cell Neurosci. 16, 805670 (2022).
- Hierlemann, A., Frey, U., Hafizovic, S., Heer, F. Growing cells atop microelectronic chips: Interfacing electrogenic cells in vitro with CMOS-based microelectrode arrays. Proceedings of the IEEE. 99 (2), 252-284 (2011).
- Maccione, A., et al. Experimental investigation on spontaneously active hippocampal cultures recorded by means of high-density MEAs: Analysis of the spatial resolution effects. Front Neuroeng. 3, 4 (2010).
- van Vliet, E., et al. Electrophysiological recording of re-aggregating brain cell cultures on multi-electrode arrays to detect acute neurotoxic effects. Neurotoxicology. 28 (6), 1136-1146 (2007).
- Emery, B. A., et al. Large-scale multimodal recordings on a high-density neurochip: Olfactory bulb and hippocampal networks. Annu Int Conf IEEE Eng Med Biol Soc. 2022, 3111-3114 (2022).
- Veleanu, M., et al. Modified climbing fiber/Purkinje cell synaptic connectivity in the cerebellum of the neonatal phencyclidine model of schizophrenia. Proc Natl Acad Sci U S A. 119 (21), e2122544119 (2022).
- Giansante, G., et al. Neuronal network activity and connectivity are impaired in a conditional knockout mouse model with PCDH19 mosaic expression. Mol Psychiatry. , (2023).
- Dossi, E., Blauwblomme, T., Nabbout, R., Huberfeld, G., Rouach, N. Multi-electrode array recordings of human epileptic postoperative cortical tissue. J Vis Exp. (92), e51870 (2014).
- Hajos, N., et al. Maintaining network activity in submerged hippocampal slices: importance of oxygen supply. Eur J Neurosci. 29 (2), 319-327 (2009).
- Hill, M. R., Greenfield, S. A. The membrane chamber: a new type of in vitro recording chamber. J Neurosci Methods. 195 (1), 15-23 (2011).
- Raimondo, J. V., et al. Methodological standards for in vitro models of epilepsy and epileptic seizures. A TASK1-WG4 report of the AES/ILAE Translational Task Force of the ILAE. Epilepsia. 58 (Suppl 4), 40-52 (2017).
- Hales, C. M., Rolston, J. D., Potter, S. M. How to culture, record and stimulate neuronal networks on micro-electrode arrays (MEAs). J Vis Exp. (39), 2056 (2010).
- Lin, C. H., Lee, J. K., LaBarge, M. A. Fabrication and use of microenvironment microarrays (MEArrays). J Vis Exp. (68), 4152 (2012).
- Panuccio, G., Colombi, I., Chiappalone, M. Recording and modulation of epileptiform activity in rodent brain slices coupled to microelectrode arrays. J Vis Exp. 135, 57548 (2018).
- Patel, C., Muthuswamy, J. High efficiency, site-specific transfection of adherent cells with siRNA using microelectrode arrays (MEA). J Vis Exp. 67, e4415 (2012).
- Ting, J. T., Daigle, T. L., Chen, Q., Feng, G. Acute brain slice methods for adult and aging animals: application of targeted patch clamp analysis and optogenetics. Methods Mol Biol. 1183, 221-242 (2014).
- Papouin, T., Haydon, P. G. Obtaining acute brain slices. Bio Protoc. 8 (2), e2699 (2018).
- Ting, J. T., et al. Preparation of acute brain slices using an optimized N-Methyl-D-glucamine protective recovery method. J Vis Exp. 132, 53825 (2018).
- Van Hoeymissen, E., Philippaert, K., Vennekens, R., Vriens, J., Held, K. Horizontal hippocampal slices of the mouse brain. J Vis Exp. (163), 61753 (2020).
- . 3Brain Available from: https://www.3brain.com/ (2022)
- Bridges, D. C., Tovar, K. R., Wu, B., Hansma, P. K., Kosik, K. S. MEA Viewer: A high-performance interactive application for visualizing electrophysiological data. PLoS One. 13 (2), e0192477 (2018).
- Hawrylycz, M., et al. Inferring cortical function in the mouse visual system through large-scale systems neuroscience. Proc Natl Acad Sci U S A. 113 (27), 7337-7344 (2016).
- Maccione, A., et al. Microelectronics, bioinformatics and neurocomputation for massive neuronal recordings in brain circuits with large scale multielectrode array probes. Brain Res Bull. 119 (Pt B), 118-126 (2015).
- . 3Brain Available from: https://www.3brain.com/products/software/brainwave4 (2022)
- Mahadevan, A. . Xenon LFP Analysis. , (2022).
- Mahadevan, A. . xenon-lfp-analysis github. , (2022).
- Codadu, N. K., et al. Divergent paths to seizure-like events. Physiol Rep. 7 (19), e14226 (2019).
- Kirsch, G. E., Drewe, J. A. Gating-dependent mechanism of 4-aminopyridine block in two related potassium channels. J Gen Physiol. 102 (5), 797-816 (1993).
- Levesque, M., Salami, P., Behr, C., Avoli, M. Temporal lobe epileptiform activity following systemic administration of 4-aminopyridine in rats. Epilepsia. 54 (4), 596-604 (2013).
- Myers, T. L., Gonzalez, O. C., Stein, J. B., Bazhenov, M. Characterizing concentration-dependent neural dynamics of 4-Aminopyridine-induced epileptiform activity. Epilepsy J. 4 (2), 128 (2018).
- Perreault, P., Avoli, M. Physiology and pharmacology of epileptiform activity induced by 4-aminopyridine in rat hippocampal slices. J Neurophysiol. 65 (4), 771-785 (1991).
- Rutecki, P. A., Lebeda, F. J., Johnston, D. 4-Aminopyridine produces epileptiform activity in hippocampus and enhances synaptic excitation and inhibition. J Neurophysiol. 57 (6), 1911-1924 (1987).
- Chen, Y., Chad, J. E., Cannon, R. C., Wheal, H. V. Reduced Mg2+ blockade of synaptically activated N-methyl-D-aspartate receptor-channels in CA1 pyramidal neurons in kainic acid-lesioned rat hippocampus. Neuroscience. 88 (3), 727-739 (1999).
- Fujiwara-Tsukamoto, Y., Isomura, Y., Takada, M. Comparable GABAergic mechanisms of hippocampal seizure-like activity in posttetanic and low-Mg2+ conditions. J Neurophysiol. 95 (3), 2013-2019 (2006).
- Swartzwelder, H. S., Anderson, W. W., Wilson, W. A. Mechanism of electrographic seizure generation in the hippocampal slice in Mg2+-free medium: the role of GABAa inhibition. Epilepsy Res. 2 (4), 239-245 (1988).
- Trevelyan, A. J., Graham, R. T., Parrish, R. R., Codadu, N. K. Synergistic positive feedback mechanisms underlying seizure initiation. Epilepsy Curr. 23 (1), 38-43 (2023).
- Croning, M. D., Haddad, G. G. Comparison of brain slice chamber designs for investigations of oxygen deprivation in vitro. J Neurosci Methods. 81 (1-2), 103-111 (1998).
- Hajos, N., Mody, I. Establishing a physiological environment for visualized in vitro brain slice recordings by increasing oxygen supply and modifying aCSF content. J Neurosci Methods. 183 (2), 107-113 (2009).
- Huang, Y., Williams, J. C., Johnson, S. M. Brain slice on a chip: opportunities and challenges of applying microfluidic technology to intact tissues. Lab Chip. 12 (12), 2103-2117 (2012).
- Andrew, R. D., et al. The critical role of spreading depolarizations in early brain injury: Consensus and contention. Neurocrit Care. 37 (Suppl 1), 83-101 (2022).
- Devonshire, I. M., Dommett, E. J., Grandy, T. H., Halliday, A. C., Greenfield, S. A. Environmental enrichment differentially modifies specific components of sensory-evoked activity in rat barrel cortex as revealed by simultaneous electrophysiological recordings and optical imaging in vivo. Neuroscience. 170 (2), 662-669 (2010).
- Parrish, R. R., Codadu, N. K., Mackenzie-Gray Scott, C., Trevelyan, A. J. Feedforward inhibition ahead of ictal wavefronts is provided by both parvalbumin- and somatostatin-expressing interneurons. J Physiol. 597 (8), 2297-2314 (2019).
- Wang, H., Jing, M., Li, Y. Lighting up the brain: genetically encoded fluorescent sensors for imaging neurotransmitters and neuromodulators. Curr Opin Neurobiol. 50, 171-178 (2018).
- Yaksi, E., Jamali, A., Diaz Verdugo, C., Jurisch-Yaksi, N. Past, present and future of zebrafish in epilepsy research. FEBS J. 288 (24), 7243-7255 (2021).
- He, M. F., et al. Ex vivo calcium imaging for drosophila model of epilepsy. J Vis Exp. 200, 65825 (2023).
- Driscoll, N., et al. Multimodal in vivo recording using transparent graphene microelectrodes illuminates spatiotemporal seizure dynamics at the microscale. Commun Biol. 4 (1), 136 (2021).
- Parrish, R. R., Grady, J., Codadu, N. K., Trevelyan, A. J., Racca, C. Simultaneous profiling of activity patterns in multiple neuronal subclasses. J Neurosci Methods. 303, 16-29 (2018).
- Valderhaug, V. D., et al. Criticality as a measure of developing proteinopathy in engineered human neural networks. bioRxiv. , (2020).
- Carleo, G., Lee, Y. -. S., Secondo, A., Miceli, F., Taglialatela, M. Multi-electrode array (MEASs) to investigate pathogenetic disease mechanisms and pharmacological properties in iPSC-derived neurons modelling neuropsychiatric diseases. , 667-672 (2022).
- Ruz, I. D., Schultz, S. R. Localising and classifying neurons from high density MEA recordings. J Neurosci Methods. 233, 115-128 (2014).
- Franke, F., Natora, M., Boucsein, C., Munk, M. H. J., Obermayer, K. An online spike detection and spike classification algorithm capable of instantaneous resolution of overlapping spikes. J Comput Neurosci. 29 (1-2), 127-148 (2010).
- Vollgraf, R., Obermayer, K. Improved optimal linear filters for the discrimination of multichannel waveform templates for spike-sorting applications. IEEE Signal Processing Letters. 13 (3), 121-124 (2006).
- Muller, J., et al. High-resolution CMOS MEA platform to study neurons at subcellular, cellular, and network levels. Lab Chip. 15 (13), 2767-2780 (2015).
- Mapelli, L., et al. implementation, and functional validation of a new generation of microneedle 3D high-density CMOS multi-electrode array for brain tissue and spheroids. bioRxiv. , (2022).
- Reddy, D. S., Kuruba, R. Experimental models of status epilepticus and neuronal injury for evaluation of therapeutic interventions. Int J Mol Sci. 14 (9), 18284-18318 (2013).
- Parrish, R. R., Trevelyan, A. J. Stress-testing the brain to understand its breaking points. J Physiol. 596 (11), 2033-2034 (2018).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados