Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Actividad similar a las convulsiones de alta calidad a partir de cortes agudos de cerebro utilizando un sistema complementario de matriz de microelectrodos de alta densidad de semiconductores de óxido metálico
* Estos autores han contribuido por igual
En este artículo
Resumen
Aquí, describimos un protocolo para el uso de sistemas complementarios de matriz de microelectrodos de alta densidad de semiconductores de óxido metálico (CMOS-HD-MEA) para registrar la actividad similar a las convulsiones de cortes de cerebro ex vivo .
Resumen
Los sistemas complementarios de microelectrodos de alta densidad de semiconductores de óxido metálico (CMOS-HD-MEA) pueden registrar la actividad neurofisiológica de cultivos celulares y cortes de cerebro ex vivo con un detalle electrofisiológico sin precedentes. Los CMOS-HD-MEA se optimizaron por primera vez para registrar la actividad de las unidades neuronales de alta calidad a partir de cultivos celulares, pero también se ha demostrado que producen datos de calidad a partir de cortes agudos de retina y cerebelos. Los investigadores han utilizado recientemente CMOS-HD-MEAs para registrar potenciales de campo local (LFP) a partir de cortes agudos de cerebro de roedores corticales. Un LFP de interés es la actividad similar a las convulsiones. Si bien muchos usuarios han producido descargas epileptiformes breves y espontáneas utilizando CMOS-HD-MEA, pocos usuarios producen de manera confiable una actividad similar a las convulsiones de calidad. Muchos factores pueden contribuir a esta dificultad, incluido el ruido eléctrico, la naturaleza inconsistente de producir actividad similar a las convulsiones cuando se usan cámaras de grabación sumergidas y la limitación de que los chips 2D CMOS-MEA solo graban desde la superficie del corte del cerebro. Las técnicas detalladas en este protocolo deberían permitir a los usuarios inducir y registrar de manera consistente una actividad similar a las convulsiones de alta calidad a partir de cortes agudos de cerebro con un sistema CMOS-HD-MEA. Además, este protocolo describe el tratamiento adecuado de los chips CMOS-HD-MEA, la gestión de soluciones y cortes de cerebro durante la experimentación y el mantenimiento del equipo.
Introducción
Los sistemas de matriz de microelectrodos de alta densidad (HD-MEA) disponibles en el mercado, que incluyen un chip MEA con miles de puntos de registro 1,2 y una plataforma MEA para amplificar y digitalizar los datos, son una herramienta emergente para la investigación electrofisiológica. Estos sistemas HD-MEA utilizan tecnología complementaria de semiconductores de óxido metálico (CMOS) para registrar datos electrofisiológicos con alta sensibilidad de cultivos celulares y preparaciones de cortes de cerebro ex vivo. Estos sistemas MEA ofrecen una resolución espacial y temporal sin precedentes para la investigación neurofisiológica a través de una alta densidad de electrodos y relaciones señal-ruido de calidad3. Esta tecnología se ha utilizado principalmente para estudiar los potenciales de acción extracelular, pero también puede capturar potenciales de campo local (LFP) de alta calidad de varias preparaciones de cortes de cerebro neuronales 4,5,6,7,8,9,10,11,12,13,14,15 . Debido a la capacidad de grabación de alta resolución mencionada anteriormente de los sistemas CMOS-HD-MEA, los usuarios pueden rastrear la actividad electrofisiológica con gran precisión espacial 16,17,18. Esta capacidad es particularmente relevante para el seguimiento de los patrones de propagación de los LFPde red 5,12,15,19,20,21. Por lo tanto, los sistemas CMOS-HD-MEA pueden proporcionar una comprensión sin precedentes de los patrones de propagación de la actividad fisiológica y patológica a partir de diversos cultivos celulares y preparaciones de cortes de cerebro. Cabe destacar que estas capacidades de los sistemas CMOS-HD-MEA pueden permitir a los investigadores contrastar los patrones de convulsiones de diferentes regiones del cerebro simultáneamente y analizar cómo varios compuestos antiepilépticos afectan estos patrones. De este modo, proporciona un método innovador para estudiar la ictogénesis y la propagación ictal y para comprender cómo la farmacología interrumpe la actividad patológica de la red 7,10,14. Por lo tanto, estas nuevas capacidades de los sistemas CMOS-HD-MEA pueden contribuir significativamente a la investigación de trastornos neurológicos, así como ayudar en la investigación de descubrimientos de fármacos 5,7,11,22. Nuestro objetivo es proporcionar detalles sobre el uso de los sistemas CMOS-HD-MEA para estudiar la actividad similar a las convulsiones.
Al utilizar sistemas CMOS-HD-MEA para estudiar los LFP, como la actividad epileptiforme en cortes agudos de cerebro, los usuarios pueden enfrentar muchos desafíos, incluido el ruido eléctrico debilitante, mantener el corte saludable durante la experimentación y detectar una señal de calidad de un chip CMOS-MEA BIDIMENSIONAL (2D) que registra solo desde la superficie del corte de cerebro. Este protocolo describe los pasos básicos para conectar a tierra correctamente la plataforma MEA y otros equipos utilizados en la experimentación, un paso crucial que puede requerir una personalización individual para cada configuración de laboratorio. Además, discutimos los pasos para ayudar a mantener el corte de cerebro saludable durante las grabaciones largas en las cámaras sumergidas utilizadas con los sistemas CMOS-HD-MEA 23,24,25. Además, a diferencia de los métodos de registro electrofisiológico más comunes, que registran desde lo profundo del corte cerebral, la mayoría de los sistemas CMOS-HD-MEA utilizan chips 2D que no penetran en el corte. Por lo tanto, estos sistemas requieren una capa externa neuronal saludable para producir la mayoría de las señales LFP registradas. Otros desafíos incluyen la visualización de la enorme cantidad de datos generados por miles de electrodos. Para superar estos desafíos, recomendamos un protocolo simple pero efectivo que aumente la probabilidad de lograr una actividad epileptiforme de red de alta calidad que se propague a través del corte del cerebro. También incluimos una breve descripción de una interfaz gráfica de usuario (GUI) disponible públicamente que desarrollamos con recursos asociados para ayudar en la visualización de datos10.
Publicaciones anteriores han proporcionado protocolos relacionados para el uso de sistemas de registro MEA 26,27,28,29. Sin embargo, este trabajo tiene como objetivo ayudar a los experimentadores que utilizan sistemas CMOS-HD-MEA con chips 2D, específicamente a aquellos que buscan estudiar la actividad epileptiforme de alta calidad a partir de cortes de cerebro. Además, comparamos dos de las manipulaciones de soluciones más comunes para la inducción de actividad similar a las convulsiones, a saber, los paradigmas 0 Mg2+ y 4-AP, para ayudar a los usuarios a identificar los medios convulsivos más apropiados para su aplicación específica. Aunque el protocolo se centra principalmente en la generación de actividad similar a las convulsiones, puede modificarse para explorar otros fenómenos electrofisiológicos utilizando cortes de cerebro.
Access restricted. Please log in or start a trial to view this content.
Protocolo
Los procedimientos con ratones fueron aprobados por el Comité Institucional de Cuidado y Uso de Animales (IACUC) de la Universidad Brigham Young. En los siguientes experimentos se utilizaron ratones machos y hembras (n = 8) C57BL/6 de al menos P21.
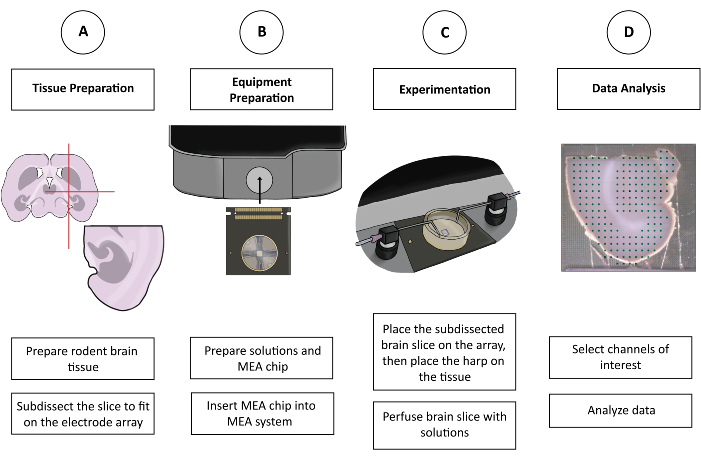
Figura 1: Figura esquemática de la experimentación CMOS-HD-MEA. (A) El corte de cerebro se prepara por el método de corte preferido de cada uno y se subdisecciona para que quepa en el MEA. (B) Prepare las soluciones y el chip CMOS-HD-MEA. (C) El corte de cerebro subdisecado se coloca en la guía de electrodos y se baña en las soluciones adecuadas. (D) Se seleccionan los canales relevantes de los datos recopilados. A continuación, los datos se preparan para su análisis en el programa preferido del usuario. Haga clic aquí para ver una versión más grande de esta figura.
| Nombre | Concentración (mM) | g/L | ||
| Cloruro de sodio (NaCl) | 126 | 7.36 | ||
| Cloruro de potasio (KCl) | 3.5 | 0.261 | ||
| Dihidrógeno Fosfato Sódico (NaH2PO4) | 1.26 | 0.151 | ||
| Bicarbonato de sodio (NaHCO3) | 26 | 2.18 | ||
| Glucosa (C6h12o6) | 10 | 1.80 | ||
| Cloruro de magnesio (MgCl2) | 1 (a partir de 1 M de culata) | 1 mL | ||
| Cloruro de calcio (CaCl2) | 2 (a partir de 1 m de culata) | 2 mL | ||
Tabla 1: Solución de aCSF.
1. Preparación de soluciones
- Preparar las soluciones experimentales.
- Prepare 1 L de líquido cefalorraquídeo artificial (aCSF, por sus siglas en inglés) (consulte la Tabla 1 para obtener más detalles).
- Prepare 1 litro de solución proconvulsiva.
NOTA: Las soluciones utilizadas para generar una actividad similar a las convulsiones en este protocolo utilizaron aCSF con 100 μM de 4-aminopiridina (4-AP) o aCSF libre de iones de magnesio. - Carbogenar todas las soluciones durante al menos 10 minutos antes de usar usando piedras porosas.
- Consigue un vaso de precipitados para desechar las soluciones.
- Coloque 1 L de aCSF, 1 L de solución proconvulsiva y el vaso de precipitados de desecho en una superficie cercana al sistema de perfusión.
- Preparación de las soluciones de la cámara de corte y retención
- Prepare 0,5 L de aCSF, omitiendo CaCl2 y agregando 3 mM de MgCl2 (en lugar de 1 mM) para crear una solución de corte única. Conserve esta solución para su uso en la preparación de cortes de cerebro de roedores colocándola cerca del área de preparación de cortes de cerebro agudos.
- Prepare 0,5 L de aCSF y colóquelo en una cámara de retención de tejidos que se utilizará para retener las rodajas para su uso en la experimentación. Inserte piedras porosas en la cámara de retención de tejido y carboreñe la solución durante al menos 10 minutos antes de colocar las rodajas subdisecadas en la cámara.
2. Preparación de rodajas de cerebro de roedor
- Prepare todas las soluciones como se ha descrito anteriormente (consulte la sección 1). Asegúrese de que todas las soluciones estén correctamente carbengenadas durante el proceso de preparación.
NOTA: Se recomienda la solución de corte (ver sección 1.2), pero se pueden utilizar otras soluciones de corte para obtener cortes de cerebro de calidad, como una solución de corte de sacarosa30. - Utilice un micrótomo vibratorio para hacer rodajas de cerebro de roedores de 350 μm. Los protocolos para el uso de estos instrumentos se enumeran en las referencias 30,31,32,33.
- Prepare los cortes de cerebro de roedor para que encajen en el área del electrodo de registro del chip MEA (consulte la Figura 2). Use una cuchilla quirúrgica de tamaño 10 para subdiseccionar las rebanadas, como se muestra en la Figura 1A y la Figura 2A , haciendo rodar suavemente la cuchilla hacia adelante y hacia atrás sobre la rebanada de cerebro. Realice la subdisección en la cámara de corte de micrótomo vibratorio. Tenga cuidado de no dañar el corte de cerebro subdisecado.
- Coloque las rodajas subdisecadas en una cámara de tejido llena de líquido cefalorraquídeo. Asegúrese de que la cámara haya sido carbogenada durante al menos 10 minutos antes de agregar las rodajas subdisecadas.
3. Preparación del equipo
- Preparación de los chips y el sistema MEA (Figura 2 y Figura 3)
NOTA: Los materiales duros como los plásticos y los metales pueden dañar fácilmente el chip MEA si entran en contacto con fuerza en la parte inferior del chip. Cuando utilice pipetas para transferir la solución dentro o fuera de las virutas, no entre en contacto con la pipeta la parte inferior del pocillo de la viruta, especialmente los electrodos de referencia en los lados del pocillo de la viruta y los electrodos de registro en el centro del pocillo de la viruta (Figura 2C). En su lugar, agregue la solución pasando el cursor sobre la parte inferior del pocillo del chip o contactando o aproximando los bordes de plástico del pocillo del chip. Para extraer toda la solución fácilmente, incline ligeramente la viruta para permitir que la solución se acumule a un lado del pozo de la viruta y retírela de la parte superior de la solución agrupada, o use una toallita antiestática para frotar ligeramente las áreas que aún tienen la solución. No transporte la viruta sujetándola bien o las clavijas de la viruta (Figura 2C). El pocillo de virutas puede contener aproximadamente 4 mL de solución. Para los pasos siguientes, llene bien el chip con aproximadamente 2 mL de solución si no se especifica lo contrario.- Designe pipetas de transferencia para diversas tareas antes de comenzar la preparación del chip. Etiquete una pipeta de transferencia para etanol, otra para residuos, otra para aCSF y otras para cualquier solución restante para evitar mezclas involuntarias.
- Llene el pocillo del chip MEA con etanol de 190 grados para que el fondo del pocillo del chip quede completamente cubierto (Figura 3). Deje reposar el etanol durante 30-60 s, luego retírelo con una pipeta de desecho.
- Llene el pocillo del chip MEA con aCSF y retírelo con una pipeta de desecho para enjuagar el etanol residual del pocillo del chip. Añada y extraiga aCSF del pocillo de virutas tres veces, utilizando las pipetas de residuos y aCSF previamente designadas. Después de lavar bien el chip tres veces, agregue aCSF y déjelo reposar durante al menos 30 s.
NOTA: Los electrodos de registro exhiben la menor cantidad de ruido de las interacciones hidrofóbicas cuando el aCSF permanece en la cámara durante al menos 45 minutos después del enjuague con etanol y aCSF. - Antes de acoplar el chip MEA, humedezca una toallita antiestática con etanol de 190 grados y utilícela para limpiar las clavijas del chip (Figura 2C).
- Deslice suavemente el chip MEA en la plataforma MEA y active el mecanismo de acoplamiento para bloquear el chip en su lugar.
- Revise los electrodos de registro y referencia para ver si hay burbujas (Figura 2C). Si hay burbujas, tome un pincel limpio y pase ligeramente por los electrodos para eliminarlas.
- Compruebe si hay ruido en el chip con el software CMOS-HD-MEA34 y escanee visualmente el mapa de colores falsos en busca de burbujas, oscilaciones no biológicas o picos causados por interferencias eléctricas. Conecte a tierra el sistema MEA adecuadamente para anular cualquier ruido encontrado.
NOTA: La configuración de la conexión a tierra dependerá del entorno de grabación. Para los experimentos de este protocolo, se conectó a tierra la plataforma MEA y el sistema de perfusión.
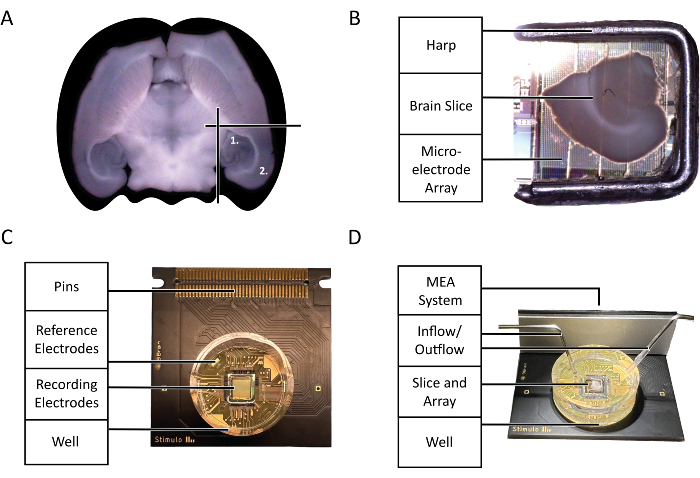
Figura 2: Diagramas de configuración y tecnología. (A) Diagrama de la selección de cortes agudos de cerebro de ratón utilizados en el experimento destacado por este protocolo. (1) Región del hipocampo (2) Región del neocórtex. (B) La colocación adecuada de un corte agudo de cerebro de ratón y un arpa en la matriz de microelectrodos (MEA). (C) La anatomía de un chip 3Brain Accura CMOS-HD-MEA. (D) La configuración adecuada de las entradas y salidas de perfusión. La entrada debe estar en lo profundo del pocillo del chip, mientras que la salida debe estar en el lado opuesto de la entrada en la parte superior del pocillo del chip para garantizar un flujo constante de aCSF fresco y oxigenado. Haga clic aquí para ver una versión más grande de esta figura.
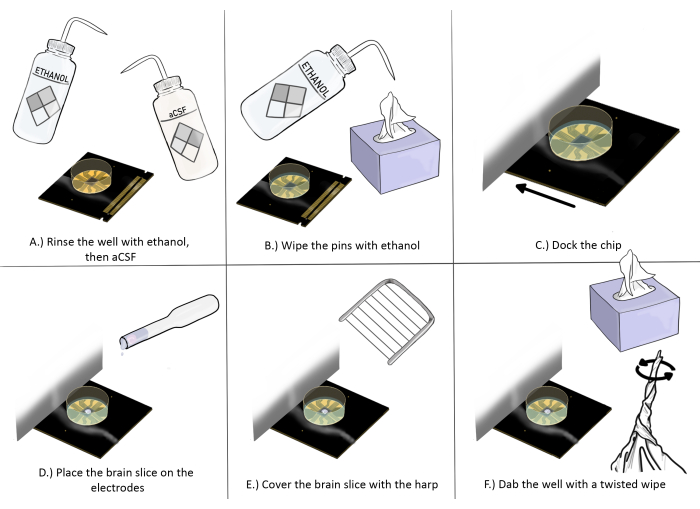
Figura 3: Diagrama esquemático de la preparación y colocación de las virutas para los experimentos de cortes de cerebro. (A) Enjuague bien la viruta con etanol una vez, luego aCSF tres veces. (B) Limpie los pines con etanol con una toallita antiestática. (C) Acople el chip. (D) Coloque el corte de cerebro en los electrodos. (E) Coloque el arpa en el corte de cerebro (consulte la Figura 2 para obtener las pautas de colocación adecuadas). (F) Frote la esquina del pocillo de los electrodos de registro cerca del corte de cerebro con una toallita antiestática retorcida. Haga clic aquí para ver una versión más grande de esta figura.
4. Experimentación
- Colocación de la rebanada de cerebro
- Coloque un arpa de platino en un bote de pesaje (u otra superficie limpia) cerca de la plataforma MEA. Cubra el arpa con aproximadamente 3 ml de aCSF para reducir sus tendencias hidrofóbicas.
- Utilice unas tijeras para extraer la parte de punta fina de una pipeta de transferencia. Corte un tercio, aproximadamente 1,5 pulgadas, de la punta de la pipeta.
NOTA: Esta pipeta modificada no constriñirá ni dañará el corte de cerebro durante la recolección y el transporte. - Recoja un trozo de cerebro de la cámara de retención de rodajas con la pipeta modificada. Dispense suavemente la rodaja de cerebro y cualquier solución de la pipeta en el pozo del chip. Para colocar el corte correctamente, libere suavemente más aCSF de una pipeta de transferencia para manipular la posición del corte de cerebro o use un pincel suave para crear una corriente en la solución que empuje el corte de cerebro a los electrodos de grabación. Limite el contacto con los electrodos de registro o el corte de cerebro para minimizar el daño.
- Con pinzas, coloque suavemente el arpa sobre el corte de cerebro con los hilos hacia abajo para presionar el corte en los electrodos de registro. Evite el contacto de la guía de electrodos con el arpa. Oriente el arpa de modo que el lado sin marco mire hacia la aguja de entrada y el marco del arpa no entre en contacto con ninguno de los electrodos de registro (Figura 2C, D).
- Tome una pipeta de desecho y retire el exceso de líquido líquido cefalorraquídeo. Tome una toallita antiestática, gire una esquina para crear una punta y utilícela para absorber el aCSF restante que rodea los electrodos de grabación sin tocar los electrodos de grabación, el corte de cerebro o el arpa (Figura 2).
- Usando una pipeta de aCSF designada, agregue rápidamente suficiente aCSF carbogenado para cubrir el corte de cerebro, aproximadamente 2 mL.
- Repita el paso 4.1.5. y 4.1.6. Dos veces más.
- Llene el pocillo con aCSF carbogenado hasta que el pocillo esté aproximadamente 3/4 lleno, aproximadamente 3 mL.
- Tome una imagen del corte de cerebro en el chip MEA con un microscopio o una cámara. Asegúrese de que la foto tenga una resolución lo suficientemente alta como para ver los bordes de la matriz de electrodos de grabación y la anatomía del corte de cerebro.
- Ejecución del experimento
- Funcionamiento del sistema de perfusión
- Coloque los tubos de entrada y salida en el vaso de precipitados lleno de aCSF y las agujas de entrada y salida en el compartimento de la viruta. Coloque la aguja de entrada cerca de la parte inferior del pozo del chip, justo fuera de los electrodos de registro. Coloque la aguja de salida cerca de la parte superior del pocillo de la viruta, hacia el borde, de modo que el líquido suba casi hasta el borde del pocillo de la viruta, aproximadamente 4 mL, y la rebanada se sumerja durante todo el experimento (Figura 2D).
- Ajuste el flujo de entrada de perfusión a 5 mL/min y el flujo de salida de perfusión a 7 mL/min.
NOTA: Se recomienda que la tasa de salida exceda la tasa de entrada para evitar el desbordamiento de la solución fuera del pocillo de chip y para crear una corriente de solución sobre la rebanada de cerebro. - Encienda el flujo de entrada y salida. Retire la aguja de entrada del pozo de viruta hasta que la aguja comience a emitir solución en lugar de aire. A continuación, vuelva a colocar la aguja en su posición dentro del pocillo del chip como se describe en el paso 4.2.1.1.
- Utilice un calentador de solución para mantener la solución a la temperatura fisiológica o cerca de ella, alrededor de 34-37 °C.
- Deje que el aCSF se fusione sobre la rebanada de cerebro durante 10 minutos. Esto permitirá que el corte se ajuste al entorno de grabación.
- Una vez transcurridos 10 minutos, mueva el tubo de salida al vaso de precipitados de desecho. A continuación, mueva el tubo de entrada al vaso de precipitados que contiene la solución proconvulsiva. Deje que el aCSF no convulsivo se elimine del sistema de perfusión y se introduzca en el vaso de precipitados de desecho durante 10 minutos.
- Transfiera el tubo de salida al vaso de precipitados que contiene la solución proconvulsiva.
- Deje que la solución proconvulsiva realice un ciclo hasta que termine el experimento.
- Si la duración del registro del corte supera las 2 horas, considere la posibilidad de preparar otra solución proconvulsiva para compensar el consumo de glucosa del corte de cerebro activo.
- Intercambio de rebanadas de cerebro
- Apague el flujo de entrada. Apague el flujo de salida 10-15 s después.
- Use pinzas para quitar el arpa y colóquela en un bote de pesaje u otra superficie.
- Utilice una pipeta modificada para extraer y desechar el trozo de cerebro. No toque los electrodos de referencia ni el conjunto de electrodos de registro (Figura 2C).
- Coloque el tubo de entrada en el vaso de precipitados que contiene la solución de aCSF no convulsiva. Coloque el tubo de salida en el vaso de precipitados de desecho. Deje las agujas de entrada y salida en el pozo de la viruta. Haga funcionar el sistema de perfusión durante 10 minutos para eliminar toda la solución proconvulsiva residual del pozo de viruta y del sistema de perfusión. Comience el proceso de experimentación de nuevo con un nuevo corte de cerebro (comenzando en el paso 4.1).
- Funcionamiento del sistema de perfusión
- Terminando el experimento
- Limpieza del equipo
- Retire el arpa, el trozo de cerebro y la solución del pozo, respectivamente.
- Desacople el chip MEA, colóquelo sobre una superficie limpia y llene bien el chip con agua desionizada.
- Deseche el agua desionizada para eliminar las sales que queden de las soluciones de aCSF.
- Con una pipeta de transferencia, llene bien la viruta con una solución de detergente. Coloque la punta de la pipeta de transferencia en el pocillo del chip y apúntela a los electrodos de registro. Exprima y suelte rápida y repetidamente el detergente en la pipeta de transferencia para lavar enérgicamente el detergente sobre los electrodos de registro durante 1 min. Deje reposar el detergente durante 5-10 min.
- Retire el detergente, luego enjuague bien el chip de 4 a 6 veces con agua desionizada para eliminar la solución de detergente y los solutos restantes.
- Retire toda el agua del pozo de la viruta, luego coloque una toallita antiestática sobre la viruta MEA y déjela toda la noche para permitir que la viruta se seque bien.
- Coloque un bote de pesaje u otra placa hermética en la plataforma de grabación MEA donde descansaba previamente el chip MEA. Coloque el arpa en el centro de la placa y coloque las agujas de entrada y salida a cada lado del arpa.
- Limpie el sistema de perfusión y el arpa enviando 50 mL de agua, 50 mL de etanol de 140 grados y 200 mL de agua, respectivamente, a través del tubo hacia el vaso de precipitados de desecho.
NOTA: En este momento, se pueden aumentar las velocidades de perfusión para disminuir el tiempo de limpieza, pero la velocidad de salida siempre debe exceder la velocidad de entrada para evitar inundaciones. - Limpie las piedras de carbógeno colocándolas en un vaso de precipitados con agua destilada y enviando el carbógeno a través del tubo durante 5 minutos. Colócalos sobre una superficie seca y limpia durante la noche y cúbrelos para evitar la acumulación de polvo.
- Limpieza del equipo
5. Análisis de datos
NOTA: Hay una variedad de paquetes de análisis utilizados para analizar datos electrofisiológicos producidos por CMOS-HD-MEA, incluido BrainWave de 3Brain, Yet Another Spike Sorter (YASS) y herramientas personalizadas de Python 34,35,36,37. Extrajimos datos del formato de archivo de datos BrainWave para su uso en la plataforma de análisis Xenon LFP para generar los datos presentados en la Figura 4 y la Figura 5. Se utilizó código personalizado de Matlab para analizar los datos de la Figura 6. Los protocolos para la plataforma de análisis de LFP de xenón están disponibles públicamente10. Los siguientes pasos del protocolo son específicos de las grabaciones realizadas con Brainwave 438; Para otros sistemas, consulte la documentación de respaldo relacionada con esos sistemas 34,35,36,37. A continuación se proporciona una descripción general de los pasos de análisis tomados para producir los datos con este protocolo. Para obtener detalles completos sobre cómo exportar, visualizar y analizar datos, incluidos los vídeos tutoriales y todos los archivos de código relevantes, consulte39.
- Exporte el archivo de grabación para su análisis.
- Cree una carpeta para los archivos de registro de datos que se reducirán de resolución.
- Recorte la imagen tomada durante el paso 4.1.9 para incluir solo la matriz de grabación con los cortes de cerebro.
- Abra el archivo ExportToHDF5-ChannelSelection.py (se puede acceder a este archivo de código en GitHub en xenon-lfp-analysis/code-files/3Brain-processing40) y copie y pegue la URL que aparece en la parte superior de la ventana en un navegador de Internet.
- Introduzca la ruta del archivo de grabación de datos y cargue la imagen recortada de la matriz asociada a la grabación.
- En Seleccionar canales para exportar, utilice la herramienta de selección de lazo para seleccionar las regiones del hipocampo y del neocortical que se muestran en la imagen recortada de la matriz.
- Establezca el número de filas que se van a omitir y el número de columnas que se van a omitir en 2.
NOTA: Los archivos de grabación suelen ser grandes; Si es apropiado para el análisis deseado, los datos exportados se pueden reducir de muestreo. Existen opciones para reducir la resolución espacial (saltando filas y columnas de electrodos de registro) y temporalmente (reduciendo la resolución a una frecuencia de muestreo más baja). En el análisis, se omiten cada 2 filas y 2 columnas; Esto permite reducir el muestreo de los datos espacialmente sin estar sesgado a ciertos canales. Cada canal está lo suficientemente alejado como para que las diferencias en la actividad puedan ser significativas entre ellos; por lo tanto, no se realiza ningún promedio de canal. Al saltar filas y columnas de los electrodos, los datos de los canales no seleccionados no se incluyen cuando se exporta el archivo de salida. - Ajuste la frecuencia de reducción de resolución a 300 Hz.
- Haga clic en el botón Exportar canales para generar el archivo de selección de canales.
NOTA: El software guardará automáticamente el archivo de selección de canal en la carpeta donde se guarda el archivo de grabación de datos. - Abra el archivo ExportToHDF5-ExtractDownsample.py (que también se encuentra en GitHub en xenon-lfp-analysis/code-files/3brain-processing40), pegue la ruta del archivo de la carpeta que contiene los archivos de selección de canal y los archivos de grabación de datos, y presione enter en el teclado para generar la versión reducida del archivo de grabación de datos.
- Uso de la plataforma de análisis
- En el símbolo del sistema de la computadora, escriba run_lfp_analysis y presione enter para abrir la plataforma de análisis.
- Introduzca las rutas de archivo de la imagen y el archivo de grabación con resolución reducida y haga clic en enviar.
- En Seleccionar un intervalo de tiempo para el análisis, seleccione toda la grabación.
- En Seleccionar canales para trazados, resalte el hipocampo como Grupo-1 y el neocórtex como Grupo-2.
- En Ráster de canal, establezca el Umbral en 0,06 mV y la Duración de tiempo en 0,02 s.
- Haga clic en Aplicar configuración y Generar ráster.
- Utilice el diagrama ráster para explorar diferentes canales que muestren patrones de actividad de interés.
NOTA: Consulte la Figura 4 para ver ejemplos de actividad similar a una convulsión de alta calidad (Figura 4A-D) en comparación con actividad subóptima (Figura 4E-F).
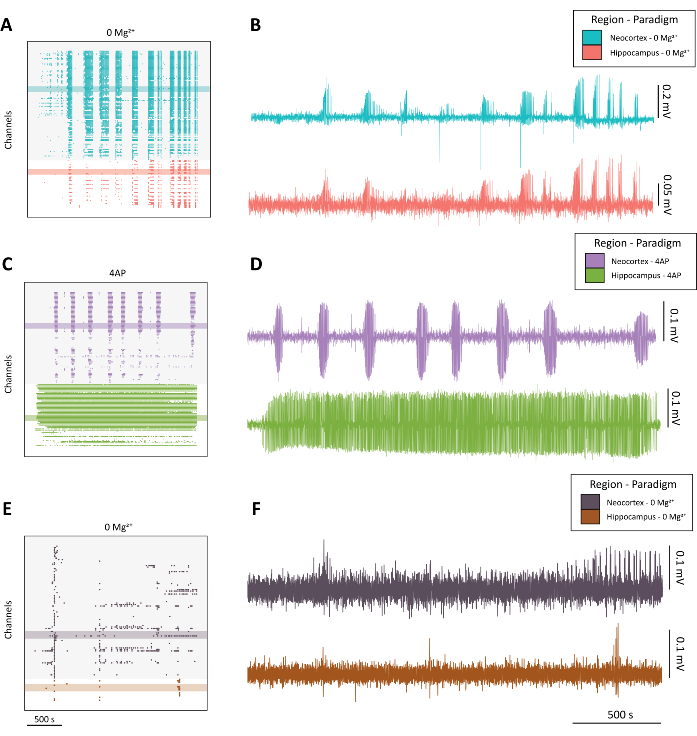
Figura 4: Ejemplo de evolución de la actividad epileptiforme a partir de los paradigmas 0 Mg2+ y 4-AP. (A) Ejemplo de diagrama ráster de la aplicación de aCSF con 0 Mg2+ durante aproximadamente 40 min. (B) Ejemplo de trazas de electrofisiología tomadas del neocórtex (azul) y el hipocampo (rojo) que demuestran la actividad epileptiforme del paradigma 0 Mg2+ . (C) Ejemplo de diagrama ráster de la aplicación 100 mM 4-AP durante aproximadamente 40 min. (D) Ejemplo de trazas electrofisiológicas tomadas del neocórtex (púrpura) y el hipocampo (verde) que demuestran actividad epileptiforme a partir de la aplicación de 4-AP. (E) Ejemplo de diagrama ráster de la aplicación de aCSF con 0 Mg2+ durante aproximadamente 40 minutos que muestra actividad de ráfaga en lugar de actividad similar a la convulsión como se encuentra en las otras trazas representativas. (F) Ejemplo: trazas de electrofisiología tomadas del neocórtex (púrpura oscuro) y el hipocampo (óxido) que demuestran una actividad subóptima del paradigma 0 Mg2+ destinado a la comparación con la actividad similar a las convulsiones de calidad encontrada en B y D. Haga clic aquí para ver una versión más grande de esta figura.
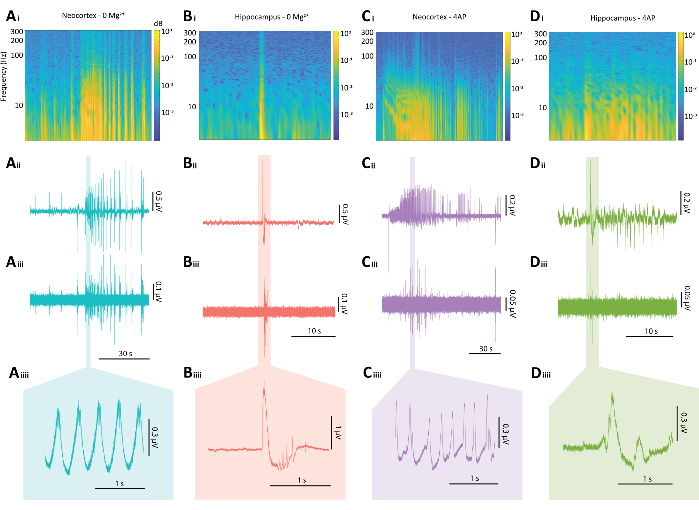
Figura 5: Resultados representativos de las descargas epileptiformes de los paradigmas 0 Mg2+ y 4-AP. (A) Ejemplos de gráficos de un evento típico similar a una convulsión neocortical inducido por el paradigma 0 Mg2+ que incluye (Ai) un espectrograma de un evento similar a una convulsión, (Aii) la traza electrofisiológica asociada, (Aiii) un filtro de paso alto de 80 Hz aplicado a la traza de Aii, (Aiiii) y una sección ampliada de la traza de Aii. (B) Ejemplos de gráficos de un estallido epileptiforme típico del hipocampo inducido por el paradigma de 0 Mg2+ que incluye (Bi) un espectrograma del estallido epileptiforme, (Bii) la traza electrofisiológica asociada, (Biii) un filtro de paso alto de 80 Hz aplicado a la traza de Bii, (Biiii) y una sección ampliada de la traza de Bii (C) Ejemplos de diagramas de un evento típico similar a una convulsión neocortical inducido por el paradigma 4-AP que incluye (Ci) un espectrograma de actividad epileptiforme, (Cii) la traza electrofisiológica asociada, (Ciii) un filtro de paso alto de 80 Hz aplicado a la traza de Cii, (Ciiii) y una sección ampliada de la traza de Cii (D) Ejemplos de gráficos de un estallido epileptiforme en el hipocampo bajo el paradigma 4-AP que incluye (Di) un espectrograma de actividad epileptiforme, (Dii) la traza electrofisiológica asociada, (Diii) un filtro de paso alto de 80 Hz aplicado a la traza de Dii, (Diiii) y una sección ampliada de la traza de Dii. Haga clic aquí para ver una versión más grande de esta figura.
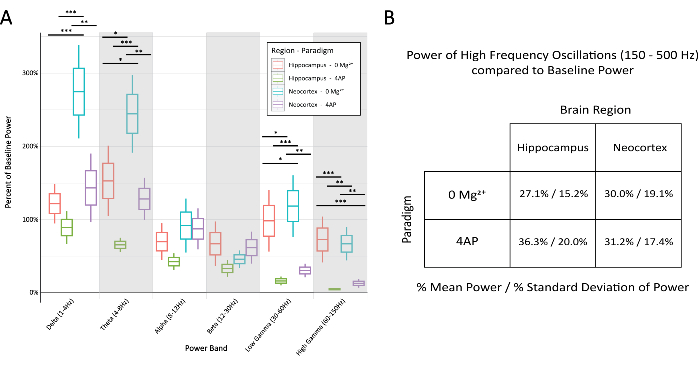
Figura 6: Comparación del porcentaje de potencia basal en las diversas bandas a través del paradigma y la región del cerebro durante las descargas epileptiformes estereotipadas. (A) La potencia durante las descargas epileptiformes fue significativamente diferente entre los paradigmas y las regiones cerebrales para la mayoría de las bandas de frecuencia (ANOVA de 2 vías con prueba de Tukey, *P < 0.05, **P < 0.001, ***P < 0.0001). La línea central de cada cuadro representa la media, los bordes del cuadro ±1 error estándar de la media (SEM) y las líneas más externas ±2 SEM. (B) Ambos paradigmas y regiones cerebrales demostraron una potencia limitada en las bandas relacionadas con la actividad de alta frecuencia por encima de 150 Hz. Haga clic aquí para ver una versión más grande de esta figura.
Access restricted. Please log in or start a trial to view this content.
Resultados
Como es habitual cuando se visualiza la actividad de muchos canales 1,4,5,10, nos parece beneficioso generar primero un gráfico ráster de los datos que adquirimos con el CMOS-HD-MEA (figura 4A,C,E). Este gráfico puede crear una vista panorámica de la actividad en todos los canales de grabación...
Access restricted. Please log in or start a trial to view this content.
Discusión
Este protocolo incluye pautas específicas relacionadas con el manejo agudo del corte de cerebro que abordan los problemas comunes que enfrentan los usuarios de CMOS-HD-MEA, a saber, el desarrollo de ruido debajo del corte de cerebro y el mantenimiento de un entorno saludable para el corte de cerebro. El desarrollo de ruido debajo del corte ocurre cuando el corte no se adhiere correctamente a la matriz; Si la rebanada de cerebro no se adhiere adecuadamente, se pueden formar bolsas de air...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores declaran que no existen conflictos de intereses asociados con este estudio de investigación.
Agradecimientos
Los autores agradecen a los miembros anteriores y actuales del laboratorio Parrish por sus ediciones en este manuscrito. También nos gustaría agradecer a Alessandro Maccione de 3Brain por sus comentarios sobre este trabajo. Este trabajo fue financiado por un premio AES/EF Junior Investigator Award y por los Colegios de Ciencias de la Vida y de Ciencias Físicas y Matemáticas de la Universidad Brigham Young.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| 2D Workbench | Cloudray | LM04CLLD26B | |
| 4-Aminopyridine | Sigma-Aldrich | 275875 | |
| Accura Chip | 3Brain | Accura HD-MEA | CMOS-HD-MEA chip |
| Agarose | Thermo Fisher Scientific | BP160-100 | |
| Vibration isolation table | Kinetic Systems | 91010124 | |
| Beaker for the slice holding chamber, 270 mL | VWR | 10754-772 | |
| BioCam | 3Brain | BioCAM DupleX | CMOS-HD-MEA platform |
| Brainwave Software | 3Brain | Version 4 | CMOS-HD-MEA software |
| Calcium Chloride | Thermo Fisher Scientific | BP510-500 | |
| Carbogen | Airgas | X02OX95C2003102 | |
| Carbogen | Airgas | 12005 | |
| Carbogen Stones | Supelco | 59277 | |
| Compresstome | Precissionary | VF-300-0Z | |
| Computer | Dell | Precission3650 | |
| Crocodile Clip Grounding Cables | JWQIDI | B06WGZG17W | |
| Detergent | Metrex | 10-4100-0000 | |
| D-Glucose | Macron Fine Chemicals | 4912-12 | |
| Dihydrogen Sodium Phosphate | Thermo Fisher Scientific | BP329-500 | |
| DinoCam | Dino-Lite | AM73915MZTL | |
| Ethanol | Thermo Fisher Scientific | A407P-4 | |
| Forceps | Fine Science Tools | 11980-13 | |
| Hot plate | Thermo Fisher Scientific | SP88857200 | |
| Ice Machine | Hoshizaki | F801MWH | |
| Inflow and outflow needles | Jensen Global | JG 18-3.0X | |
| Inline Solution Heater | Warner Instruments | SH-27B | |
| Isofluorine | Dechra | 08PB-STE22002-0122 | |
| Kim Wipes | Thermo Fisher Scientific | 06-666 | |
| Magnesium Chloride | Thermo Fisher Scientific | FLM33500 | |
| Micropipets | Gilson | F144069 | |
| Mili-Q Water Filter | Mili-Q | ZR0Q008WW | |
| Paintbrush | Daler Rowney | AF85 Round: 0 | |
| Paper Filter | Whatman | EW-06648-24 | |
| Parafilm | American National Can | PM996 | |
| Perfusion System | Multi Channel System | PPS2 | |
| Pipetor | Thermo Fisher Scientific | FB14955202 | |
| Platinum Harp | 3Brain | 3Brain | |
| Potassium Chloride | Thermo Fisher Scientific | P330-3 | |
| Razor blade | Personna | BP9020 | |
| Scale | Metter Toledo | AB204 | |
| Scissors | Solingen | 92008 | |
| Slice Holding Chamber | Custom | Custom | Custom 3D Printer Design, available upon request |
| Sodium Bicarbonate | Macron Fine Chemicals | 7412-06 | |
| Sodium Chloride | Thermo Fisher Scientific | S271-3 | |
| Temperature Control Box | Warner Instruments | TC344B | |
| Transfer Pipettes | Genesee Scientific | 30-200 | |
| Tubing | Tygon | B-44-3 TPE | |
| Vibratome VZ-300 | Precissionary | VF-00-VM-NC | |
| Weigh Boat | Electron Microscopy Sciences | 70040 |
Referencias
- Obien, M. E. J., Frey, U. Large-scale, high-resolution microelectrode arrays for interrogation of neurons and networks. Adv Neurobiol. 22, 83-123 (2019).
- Schroter, M., et al. Functional imaging of brain organoids using high-density microelectrode arrays. MRS Bull. 47 (6), 530-544 (2022).
- Miccoli, B., et al. High-density electrical recording and impedance imaging with a multi-modal CMOS multi-electrode array chip. Front Neurosci. 13, 641(2019).
- Emery, B. A., Hu, X., Khanzada, S., Kempermann, G., Amin, H. High-resolution CMOS-based biosensor for assessing hippocampal circuit dynamics in experience-dependent plasticity. Biosens Bioelectron. 237, 115471(2023).
- Ferrea, E., et al. high-resolution electrophysiological imaging of field potentials in brain slices with microelectronic multielectrode arrays. Front Neural Circuits. 6, 80(2012).
- Gagliano, G., et al. Non-linear frequency dependence of neurovascular coupling in the cerebellar cortex implies vasodilation-vasoconstriction competition. Cells. 11 (6), 1047(2022).
- Goodchild, S. J., et al. Molecular pharmacology of selective Na(V)1.6 and dual Na(V)1.6/Na(V)1.2 channel inhibitors that suppress excitatory neuronal activity ex vivo. ACS Chem Neurosci. 15 (6), 1169-1184 (2024).
- Hu, X., Khanzada, S., Klutsch, D., Calegari, F., Amin, H. Implementation of biohybrid olfactory bulb on a high-density CMOS-chip to reveal large-scale spatiotemporal circuit information. Biosens Bioelectron. 198, 113834(2022).
- Kim, S., et al. Alteration of neural network and hippocampal slice activation through exosomes derived from 5XFAD nasal lavage fluid. Int J Mol Sci. 24 (18), 14064(2023).
- Mahadevan, A., Codadu, N. K., Parrish, R. R. Xenon LFP analysis platform is a novel graphical user interface for analysis of local field potential from large-scale MEA recordings. Front Neurosci. 16, 904931(2022).
- Medrihan, L., Ferrea, E., Greco, B., Baldelli, P., Benfenati, F. Asynchronous GABA release is a key determinant of tonic inhibition and controls neuronal excitability: A study in the synapsin II-/- mouse. Cereb Cortex. 25 (10), 3356-3368 (2015).
- Monteverdi, A., Di Domenico, D., D'Angelo, E., Mapelli, L. Anisotropy and frequency dependence of signal propagation in the cerebellar circuit revealed by high-density multielectrode array recordings. Biomedicines. 11 (5), 1475(2023).
- Obien, M. E. J., Hierlemann, A., Frey, U. Accurate signal-source localization in brain slices by means of high-density microelectrode arrays. Sci Rep. 9 (1), 788(2019).
- Thouta, S., et al. Pharmacological determination of the fractional block of Nav channels required to impair neuronal excitability and ex vivo seizures. Front Cell Neurosci. 16, 964691(2022).
- Tognolina, M., Monteverdi, A., D'Angelo, E. Discovering microcircuit secrets with multi-spot imaging and electrophysiological recordings: The example of cerebellar network dynamics. Front Cell Neurosci. 16, 805670(2022).
- Hierlemann, A., Frey, U., Hafizovic, S., Heer, F. Growing cells atop microelectronic chips: Interfacing electrogenic cells in vitro with CMOS-based microelectrode arrays. Proceedings of the IEEE. 99 (2), 252-284 (2011).
- Maccione, A., et al. Experimental investigation on spontaneously active hippocampal cultures recorded by means of high-density MEAs: Analysis of the spatial resolution effects. Front Neuroeng. 3, 4(2010).
- van Vliet, E., et al. Electrophysiological recording of re-aggregating brain cell cultures on multi-electrode arrays to detect acute neurotoxic effects. Neurotoxicology. 28 (6), 1136-1146 (2007).
- Emery, B. A., et al. Large-scale multimodal recordings on a high-density neurochip: Olfactory bulb and hippocampal networks. Annu Int Conf IEEE Eng Med Biol Soc. 2022, 3111-3114 (2022).
- Veleanu, M., et al. Modified climbing fiber/Purkinje cell synaptic connectivity in the cerebellum of the neonatal phencyclidine model of schizophrenia. Proc Natl Acad Sci U S A. 119 (21), e2122544119(2022).
- Giansante, G., et al. Neuronal network activity and connectivity are impaired in a conditional knockout mouse model with PCDH19 mosaic expression. Mol Psychiatry. , (2023).
- Dossi, E., Blauwblomme, T., Nabbout, R., Huberfeld, G., Rouach, N. Multi-electrode array recordings of human epileptic postoperative cortical tissue. J Vis Exp. (92), e51870(2014).
- Hajos, N., et al. Maintaining network activity in submerged hippocampal slices: importance of oxygen supply. Eur J Neurosci. 29 (2), 319-327 (2009).
- Hill, M. R., Greenfield, S. A. The membrane chamber: a new type of in vitro recording chamber. J Neurosci Methods. 195 (1), 15-23 (2011).
- Raimondo, J. V., et al. Methodological standards for in vitro models of epilepsy and epileptic seizures. A TASK1-WG4 report of the AES/ILAE Translational Task Force of the ILAE. Epilepsia. 58 (Suppl 4), 40-52 (2017).
- Hales, C. M., Rolston, J. D., Potter, S. M. How to culture, record and stimulate neuronal networks on micro-electrode arrays (MEAs). J Vis Exp. (39), 2056(2010).
- Lin, C. H., Lee, J. K., LaBarge, M. A. Fabrication and use of microenvironment microarrays (MEArrays). J Vis Exp. (68), 4152(2012).
- Panuccio, G., Colombi, I., Chiappalone, M. Recording and modulation of epileptiform activity in rodent brain slices coupled to microelectrode arrays. J Vis Exp. 135, 57548(2018).
- Patel, C., Muthuswamy, J. High efficiency, site-specific transfection of adherent cells with siRNA using microelectrode arrays (MEA). J Vis Exp. 67, e4415(2012).
- Ting, J. T., Daigle, T. L., Chen, Q., Feng, G. Acute brain slice methods for adult and aging animals: application of targeted patch clamp analysis and optogenetics. Methods Mol Biol. 1183, 221-242 (2014).
- Papouin, T., Haydon, P. G. Obtaining acute brain slices. Bio Protoc. 8 (2), e2699(2018).
- Ting, J. T., et al. Preparation of acute brain slices using an optimized N-Methyl-D-glucamine protective recovery method. J Vis Exp. 132, 53825(2018).
- Van Hoeymissen, E., Philippaert, K., Vennekens, R., Vriens, J., Held, K. Horizontal hippocampal slices of the mouse brain. J Vis Exp. (163), 61753(2020).
- 3Brain. , Available from: https://www.3brain.com/ (2022).
- Bridges, D. C., Tovar, K. R., Wu, B., Hansma, P. K., Kosik, K. S. MEA Viewer: A high-performance interactive application for visualizing electrophysiological data. PLoS One. 13 (2), e0192477(2018).
- Hawrylycz, M., et al. Inferring cortical function in the mouse visual system through large-scale systems neuroscience. Proc Natl Acad Sci U S A. 113 (27), 7337-7344 (2016).
- Maccione, A., et al. Microelectronics, bioinformatics and neurocomputation for massive neuronal recordings in brain circuits with large scale multielectrode array probes. Brain Res Bull. 119 (Pt B), 118-126 (2015).
- 3Brain. , BrainWave 4 https://www.3brain.com/products/software/brainwave4 (2022).
- Mahadevan, A. Xenon LFP Analysis. , https://xenon-lfp-analysis.readthedocs.io/en/latest/Installation.html (2022).
- Mahadevan, A. xenon-lfp-analysis github. , https://github.com/MicroBrew09/xenon-lfp-analysis (2022).
- Codadu, N. K., et al. Divergent paths to seizure-like events. Physiol Rep. 7 (19), e14226(2019).
- Kirsch, G. E., Drewe, J. A. Gating-dependent mechanism of 4-aminopyridine block in two related potassium channels. J Gen Physiol. 102 (5), 797-816 (1993).
- Levesque, M., Salami, P., Behr, C., Avoli, M. Temporal lobe epileptiform activity following systemic administration of 4-aminopyridine in rats. Epilepsia. 54 (4), 596-604 (2013).
- Myers, T. L., Gonzalez, O. C., Stein, J. B., Bazhenov, M. Characterizing concentration-dependent neural dynamics of 4-Aminopyridine-induced epileptiform activity. Epilepsy J. 4 (2), 128(2018).
- Perreault, P., Avoli, M. Physiology and pharmacology of epileptiform activity induced by 4-aminopyridine in rat hippocampal slices. J Neurophysiol. 65 (4), 771-785 (1991).
- Rutecki, P. A., Lebeda, F. J., Johnston, D. 4-Aminopyridine produces epileptiform activity in hippocampus and enhances synaptic excitation and inhibition. J Neurophysiol. 57 (6), 1911-1924 (1987).
- Chen, Y., Chad, J. E., Cannon, R. C., Wheal, H. V. Reduced Mg2+ blockade of synaptically activated N-methyl-D-aspartate receptor-channels in CA1 pyramidal neurons in kainic acid-lesioned rat hippocampus. Neuroscience. 88 (3), 727-739 (1999).
- Fujiwara-Tsukamoto, Y., Isomura, Y., Takada, M. Comparable GABAergic mechanisms of hippocampal seizure-like activity in posttetanic and low-Mg2+ conditions. J Neurophysiol. 95 (3), 2013-2019 (2006).
- Swartzwelder, H. S., Anderson, W. W., Wilson, W. A. Mechanism of electrographic seizure generation in the hippocampal slice in Mg2+-free medium: the role of GABAa inhibition. Epilepsy Res. 2 (4), 239-245 (1988).
- Trevelyan, A. J., Graham, R. T., Parrish, R. R., Codadu, N. K. Synergistic positive feedback mechanisms underlying seizure initiation. Epilepsy Curr. 23 (1), 38-43 (2023).
- Croning, M. D., Haddad, G. G. Comparison of brain slice chamber designs for investigations of oxygen deprivation in vitro. J Neurosci Methods. 81 (1-2), 103-111 (1998).
- Hajos, N., Mody, I. Establishing a physiological environment for visualized in vitro brain slice recordings by increasing oxygen supply and modifying aCSF content. J Neurosci Methods. 183 (2), 107-113 (2009).
- Huang, Y., Williams, J. C., Johnson, S. M. Brain slice on a chip: opportunities and challenges of applying microfluidic technology to intact tissues. Lab Chip. 12 (12), 2103-2117 (2012).
- Andrew, R. D., et al. The critical role of spreading depolarizations in early brain injury: Consensus and contention. Neurocrit Care. 37 (Suppl 1), 83-101 (2022).
- Devonshire, I. M., Dommett, E. J., Grandy, T. H., Halliday, A. C., Greenfield, S. A. Environmental enrichment differentially modifies specific components of sensory-evoked activity in rat barrel cortex as revealed by simultaneous electrophysiological recordings and optical imaging in vivo. Neuroscience. 170 (2), 662-669 (2010).
- Parrish, R. R., Codadu, N. K., Mackenzie-Gray Scott, C., Trevelyan, A. J. Feedforward inhibition ahead of ictal wavefronts is provided by both parvalbumin- and somatostatin-expressing interneurons. J Physiol. 597 (8), 2297-2314 (2019).
- Wang, H., Jing, M., Li, Y. Lighting up the brain: genetically encoded fluorescent sensors for imaging neurotransmitters and neuromodulators. Curr Opin Neurobiol. 50, 171-178 (2018).
- Yaksi, E., Jamali, A., Diaz Verdugo, C., Jurisch-Yaksi, N. Past, present and future of zebrafish in epilepsy research. FEBS J. 288 (24), 7243-7255 (2021).
- He, M. F., et al. Ex vivo calcium imaging for drosophila model of epilepsy. J Vis Exp. 200, 65825(2023).
- Driscoll, N., et al. Multimodal in vivo recording using transparent graphene microelectrodes illuminates spatiotemporal seizure dynamics at the microscale. Commun Biol. 4 (1), 136(2021).
- Parrish, R. R., Grady, J., Codadu, N. K., Trevelyan, A. J., Racca, C. Simultaneous profiling of activity patterns in multiple neuronal subclasses. J Neurosci Methods. 303, 16-29 (2018).
- Valderhaug, V. D., et al. Criticality as a measure of developing proteinopathy in engineered human neural networks. bioRxiv. , (2020).
- Multi-electrode array (MEASs) to investigate pathogenetic disease mechanisms and pharmacological properties in iPSC-derived neurons modelling neuropsychiatric diseases. Carleo, G., Lee, Y. -S., Secondo, A., Miceli, F., Taglialatela, M. 2022 IEEE International Conference on Metrology for Extended Reality, Artificial Intelligence and Neural Engineering (MetroXRAINE), , Rome, Italy. 667-672 (2022).
- Ruz, I. D., Schultz, S. R. Localising and classifying neurons from high density MEA recordings. J Neurosci Methods. 233, 115-128 (2014).
- Franke, F., Natora, M., Boucsein, C., Munk, M. H. J., Obermayer, K. An online spike detection and spike classification algorithm capable of instantaneous resolution of overlapping spikes. J Comput Neurosci. 29 (1-2), 127-148 (2010).
- Vollgraf, R., Obermayer, K. Improved optimal linear filters for the discrimination of multichannel waveform templates for spike-sorting applications. IEEE Signal Processing Letters. 13 (3), 121-124 (2006).
- Muller, J., et al. High-resolution CMOS MEA platform to study neurons at subcellular, cellular, and network levels. Lab Chip. 15 (13), 2767-2780 (2015).
- Mapelli, L., et al. implementation, and functional validation of a new generation of microneedle 3D high-density CMOS multi-electrode array for brain tissue and spheroids. bioRxiv. , (2022).
- Reddy, D. S., Kuruba, R. Experimental models of status epilepticus and neuronal injury for evaluation of therapeutic interventions. Int J Mol Sci. 14 (9), 18284-18318 (2013).
- Parrish, R. R., Trevelyan, A. J. Stress-testing the brain to understand its breaking points. J Physiol. 596 (11), 2033-2034 (2018).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados