Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Activité épileptique de haute qualité à partir de coupes cérébrales aiguës à l’aide d’un système de réseau de microélectrodes haute densité à oxyde métallique, oxyde métallique et semi-conducteur complémentaire
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Ici, nous décrivons un protocole pour l’utilisation de systèmes complémentaires de réseaux de microélectrodes à haute densité d’oxydes métalliques et de semi-conducteurs (CMOS-HD-MEA) pour enregistrer une activité semblable à celle d’une crise à partir de coupes de cerveau ex vivo .
Résumé
Les systèmes CMOS-HD-MEA (complémentaires à micro-réseau de microélectrodes haute densité à oxyde métallique et semi-conducteurs) peuvent enregistrer l’activité neurophysiologique à partir de cultures cellulaires et de coupes de cerveau ex vivo avec des détails électrophysiologiques sans précédent. Les CMOS-HD-MEA ont d’abord été optimisés pour enregistrer l’activité des unités neuronales de haute qualité à partir de cultures cellulaires, mais il a également été démontré qu’ils produisaient des données de qualité à partir de coupes rétiniennes et cérébelleuses aiguës. Les chercheurs ont récemment utilisé CMOS-HD-MEA pour enregistrer les potentiels de champ locaux (LFP) à partir de coupes de cerveau de rongeurs corticaux aigus. L’une des LFP d’intérêt est l’activité semblable à une crise. Bien que de nombreux utilisateurs aient produit des décharges épileptiformes brèves et spontanées à l’aide de CMOS-HD-MEA, peu d’utilisateurs produisent de manière fiable une activité épileptique de qualité. De nombreux facteurs peuvent contribuer à cette difficulté, notamment le bruit électrique, la nature incohérente de la production d’une activité semblable à une crise lors de l’utilisation de chambres d’enregistrement immergées et la limitation du fait que les puces CMOS-MEA 2D n’enregistrent qu’à partir de la surface de la tranche de cerveau. Les techniques détaillées dans ce protocole devraient permettre aux utilisateurs d’induire et d’enregistrer de manière cohérente une activité convulsive de haute qualité à partir de coupes cérébrales aiguës avec un système CMOS-HD-MEA. De plus, ce protocole décrit le bon traitement des puces CMOS-HD-MEA, la gestion des solutions et des tranches de cerveau lors de l’expérimentation, et la maintenance des équipements.
Introduction
Les systèmes de microélectrodes à haute densité (HD-MEA) disponibles dans le commerce, qui comprennent une puce MEA avec des milliers de points d’enregistrement 1,2 et une plate-forme MEA pour amplifier et numériser les données, constituent un outil émergent pour la recherche électrophysiologique. Ces systèmes HD-MEA utilisent la technologie CMOS (Complementary metal-oxide-semiconductor) pour enregistrer des données électrophysiologiques avec une sensibilité élevée à partir de cultures cellulaires et de préparations de coupes de cerveau ex vivo. Ces systèmes MEA offrent une résolution spatiale et temporelle sans précédent à la recherche neurophysiologique grâce à une densité d’électrodes élevée et à des rapports signal/bruit de qualité3. Cette technologie a principalement été utilisée pour étudier les potentiels d’action extracellulaires, mais elle peut également capturer des potentiels de champ local (LFP) de haute qualité à partir de diverses préparations de coupes de cerveau neuronal 4,5,6,7,8,9,10,11,12,13,14,15 . En raison de la capacité d’enregistrement haute résolution mentionnée ci-dessus des systèmes CMOS-HD-MEA, les utilisateurs peuvent suivre l’activité électrophysiologique avec une grande précision spatiale 16,17,18. Cette capacité est particulièrement pertinente pour le suivi des modèles de propagation des LFPde réseau 5,12,15,19,20,21. Par conséquent, les systèmes CMOS-HD-MEA peuvent fournir une compréhension sans précédent des modèles de propagation de l’activité physiologique et pathologique à partir de diverses cultures cellulaires et préparations de coupes de cerveau. Il convient de noter en particulier que ces capacités des systèmes CMOS-HD-MEA peuvent permettre aux chercheurs de comparer simultanément les modèles de crises de différentes régions du cerveau et d’évaluer comment divers composés antiépileptiques affectent ces modèles. Ce faisant, il fournit une méthode innovante pour étudier l’ictogenèse et la propagation de l’ictal et pour comprendre comment la pharmacologie perturbe l’activité pathologique du réseau 7,10,14. Par conséquent, ces nouvelles capacités des systèmes CMOS-HD-MEA peuvent contribuer de manière significative à la recherche sur les troubles neurologiques, ainsi qu’à la recherche sur la découverte de médicaments 5,7,11,22. Notre objectif est de fournir des détails sur l’utilisation des systèmes CMOS-HD-MEA pour étudier l’activité convulsive.
Lors de l’utilisation de systèmes CMOS-HD-MEA pour étudier les LFP, telles que l’activité épileptiforme dans les tranches cérébrales aiguës, les utilisateurs peuvent être confrontés à de nombreux défis, notamment un bruit électrique débilitant, le maintien de la tranche en bonne santé pendant l’expérimentation et la détection d’un signal de qualité à partir d’une puce CMOS-MEA bidimensionnelle (2D) qui enregistre uniquement à partir de la surface de la tranche de cerveau. Ce protocole décrit les étapes de base pour mettre correctement à la terre la plate-forme MEA et d’autres équipements utilisés dans l’expérimentation, une étape cruciale qui peut nécessiter une personnalisation individuelle pour chaque configuration de laboratoire. De plus, nous discutons des étapes à suivre pour aider à maintenir la tranche de cerveau en bonne santé lors de longs enregistrements dans les chambres immergées utilisées avec les systèmes CMOS-HD-MEA 23,24,25. De plus, contrairement aux méthodes d’enregistrement électrophysiologique plus courantes, qui enregistrent au plus profond de la tranche de cerveau, la plupart des systèmes CMOS-HD-MEA utilisent des puces 2D qui ne pénètrent pas dans la tranche. Par conséquent, ces systèmes nécessitent une couche externe neuronale saine pour produire la majorité des signaux LFP enregistrés. Parmi les autres défis, citons la visualisation de la quantité massive de données générées par des milliers d’électrodes. Pour surmonter ces défis, nous recommandons un protocole simple mais efficace qui augmente la probabilité d’obtenir une activité épileptiforme de réseau de haute qualité qui se propage dans la tranche de cerveau. Nous incluons également une brève description d’une interface utilisateur graphique (GUI) accessible au public que nous avons développée avec les ressources associées pour faciliter la visualisation des données10.
Des publications antérieures ont fourni des protocoles connexes pour l’utilisation des systèmes d’enregistrement MEA 26,27,28,29. Cependant, ce travail vise à aider les expérimentateurs utilisant des systèmes CMOS-HD-MEA avec des puces 2D, en particulier ceux qui cherchent à étudier une activité épileptiforme de haute qualité à partir de coupes de cerveau. De plus, nous comparons deux des manipulations de solutions les plus courantes pour l’induction d’une activité convulsive, à savoir les paradigmes 0 Mg2+ et 4-AP, pour aider les utilisateurs à identifier les milieux convulsivants les plus appropriés pour leur application spécifique. Bien que le protocole soit principalement axé sur la génération d’une activité semblable à celle d’une crise, il peut être modifié pour explorer d’autres phénomènes électrophysiologiques à l’aide de tranches de cerveau.
Access restricted. Please log in or start a trial to view this content.
Protocole
Les procédures impliquant des souris ont été approuvées par le Comité institutionnel de soin et d’utilisation des animaux (IACUC) de l’Université Brigham Young. Des souris C57BL/6 mâles et femelles (n = 8) âgées d’au moins P21 ont été utilisées dans les expériences suivantes.
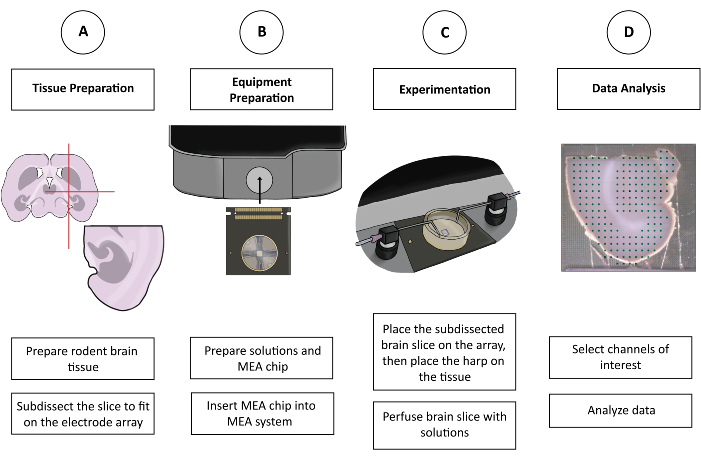
Figure 1 : Figure schématique de l’expérience CMOS-HD-MEA. (A) La tranche de cerveau est préparée par la méthode de coupe préférée et sous-disséquée pour s’adapter à la MEA. (B) Préparez les solutions et la puce CMOS-HD-MEA. (C) La tranche de cerveau sous-disséquée est placée sur le réseau d’électrodes et baignée dans les solutions appropriées. (D) Les canaux pertinents sont sélectionnés à partir des données collectées. Les données sont ensuite préparées pour l’analyse dans le programme préféré de l’utilisateur. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Nom | Concentration (mM) | g/L | ||
| Chlorure de sodium (NaCl) | 126 | 7.36 | ||
| Chlorure de potassium (KCl) | 3.5 | 0.261 | ||
| Phosphate de dihydrogène et de sodium (NaH2PO4) | 1.26 | 0.151 | ||
| Bicarbonate de sodium (NaHCO3) | 26 | 2.18 | ||
| Glucose (C6h12O6) | 10 | 1.80 | ||
| Chlorure de magnésium (MgCl2) | 1 (à partir de 1 M de stock) | 1 mL | ||
| Chlorure de calcium (CaCl2) | 2 (à partir de 1 M de stock) | 2 mL | ||
Tableau 1 : Solution de CSF.
1. Préparer des solutions
- Préparez les solutions expérimentales.
- Préparez 1 L de liquide céphalo-rachidien artificiel (LCR-A) (voir le tableau 1 pour plus de détails).
- Préparez 1 L de solution pro-convulsivante.
REMARQUE : Les solutions utilisées pour générer une activité convulsive dans ce protocole utilisaient soit un LCR avec 100 μM de 4-Aminopyridine (4-AP), soit un LCR sans ions magnésium. - Carbogéner toutes les solutions pendant au moins 10 min avant utilisation à l’aide de pierres poreuses.
- Procurez-vous un bécher pour jeter les solutions.
- Placez 1 L d’aCSF, 1 L de solution pro-convulsivante et le bécher jeté sur une surface proche du système de perfusion.
- Préparation des solutions de chambre de coupe et de maintien
- Préparez 0,5 L d’aCSF, en omettant CaCl2 et en ajoutant 3 mM de MgCl2 (au lieu de 1 mM) pour créer une solution de coupe unique. Conservez cette solution pour l’utiliser dans la préparation des tranches de cerveau de rongeur en la plaçant près de la zone de préparation des tranches de cerveau aiguës.
- Préparez 0,5 L de LCa et placez-le dans une chambre de rétention de tissus qui servira à retenir les tranches à des fins d’expérimentation. Insérez des pierres poreuses dans la chambre de rétention des tissus et carbogénez la solution pendant au moins 10 minutes avant de placer les tranches sous-disséquées dans la chambre.
2. Préparation des tranches de cerveau de rongeur
- Préparez toutes les solutions comme décrit ci-dessus (voir rubrique 1). Assurez-vous que toutes les solutions sont correctement carbogénées pendant le processus de préparation.
REMARQUE : La solution de coupe (voir rubrique 1.2) est recommandée, mais d’autres solutions de coupe peuvent être utilisées pour obtenir des tranches de cerveau de qualité, comme une solution de coupe de saccharose30. - À l’aide d’un microtome vibrant, faites des tranches de cerveau de rongeur de 350 μm. Les protocoles d’utilisation de ces instruments sont listés dans les références 30,31,32,33.
- Préparez les tranches de cerveau de rongeur pour qu’elles s’adaptent à la zone de l’électrode d’enregistrement de la puce MEA (voir Figure 2). Utilisez une lame chirurgicale de taille 10 pour sous-disséquer les tranches, comme illustré à la figure 1A et à la figure 2A , en faisant rouler doucement la lame d’avant en arrière sur la tranche de cerveau. Effectuez la sous-dissection dans la chambre de découpe du microtome vibrant. Faites attention de ne pas endommager la tranche de cerveau sous-disséquée.
- Placez les tranches sous-disséquées dans une chambre de rétention de tissu remplie d’aCSF. Assurez-vous que la chambre a été carbogénée pendant au moins 10 minutes avant d’ajouter les tranches sous-disséquées.
3. Préparation de l’équipement
- Préparation des puces et du système MEA (Figure 2 et Figure 3)
REMARQUE : Les matériaux durs comme les plastiques et les métaux peuvent facilement endommager la puce MEA s’ils entrent en contact avec force avec le bas de la puce. Lorsque vous utilisez des pipettes pour transférer la solution dans ou hors des puces, ne touchez pas le bas du puits de la puce avec la pipette, en particulier les électrodes de référence sur les côtés du puits de la puce et les électrodes d’enregistrement au centre du puits de la puce (Figure 2C). Au lieu de cela, ajoutez la solution en passant la souris sur le fond du puits de copeaux ou en contactant ou en approchant les bords en plastique du puits de copeaux. Pour retirer facilement toute la solution, inclinez légèrement la puce pour permettre à la solution de s’accumuler d’un côté du puits de la puce et retirez-la du haut de la solution accumulée, ou utilisez une lingette antistatique pour tamponner légèrement les zones qui ont encore la solution. Ne transportez pas la puce en la tenant bien ou sur les broches de la puce (Figure 2C). Le puits à copeaux peut contenir environ 4 ml de solution. Pour les étapes subséquentes, remplissez bien la copuce d’environ 2 mL de solution, sauf indication contraire.- Désignez les pipettes de transfert pour diverses tâches avant de commencer la préparation des puces. Étiquetez une pipette de transfert pour l’éthanol, une pour les déchets, une autre pour l’aCSF et d’autres pour toute solution restante afin d’éviter tout mélange involontaire.
- Remplissez le puits de la puce MEA avec de l’éthanol à 190 degrés de sorte que le fond du puits de la puce soit complètement recouvert (Figure 3). Laissez reposer l’éthanol pendant 30 à 60 s, puis retirez-le à l’aide d’une pipette de jet.
- Remplissez le puits de la puce MEA avec de l’aCSF et retirez-le avec une pipette de décharge pour rincer l’éthanol résiduel du puits de la puce. Ajoutez et retirez trois fois l’aCSF du puits à copeaux, à l’aide des pipettes de déchets et d’aCSF précédemment désignées. Après avoir bien lavé la puce trois fois, ajoutez aCSF et laissez-la reposer pendant au moins 30 s.
REMARQUE : Les électrodes d’enregistrement présentent le moins de bruit provenant d’interactions hydrophobes lorsque l’aLCR reste dans la chambre pendant au moins 45 minutes après le rinçage à l’éthanol et à l’aCSF. - Avant d’ancrer la puce MEA, mouillez une lingette antistatique avec de l’éthanol à 190 degrés et utilisez-la pour essuyer les broches de la puce (Figure 2C).
- Faites glisser doucement la puce MEA dans la plate-forme MEA et engagez le mécanisme d’amarrage pour verrouiller la puce en place.
- Vérifiez que les électrodes d’enregistrement et de référence ne contiennent pas de bulles (Figure 2C). Si des bulles sont présentes, prenez un pinceau propre et balayez légèrement les électrodes pour les enlever.
- Vérifiez que la puce n’est pas bruyante à l’aide du logiciel CMOS-HD-MEA34 et scannez visuellement la carte en fausses couleurs à la recherche de bulles, d’oscillations non biologiques ou de pointes causées par des interférences électriques. Mettez le système MEA à la terre de manière appropriée pour éliminer tout bruit rencontré.
REMARQUE : La configuration de la mise à la terre dépendra de l’environnement d’enregistrement. Pour les expériences de ce protocole, la plateforme MEA et le système de perfusion ont été mis à la terre.
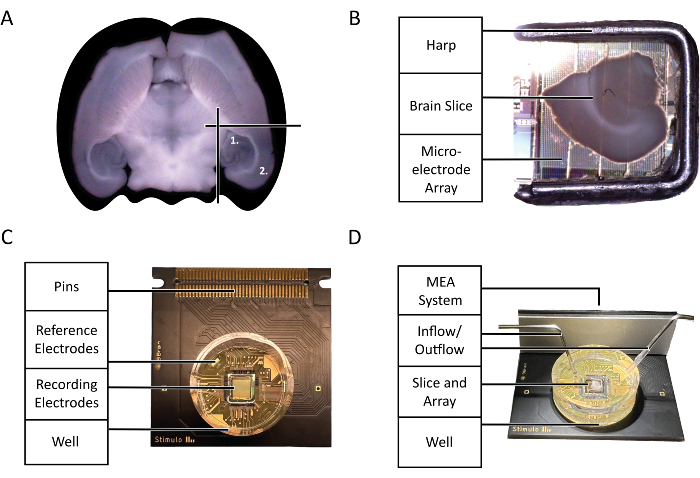
Figure 2 : Diagrammes de configuration et de technologie. (A) Schéma de la sélection des tranches de cerveau de souris aiguës utilisées dans l’expérience mise en évidence par ce protocole. (1) Région de l’hippocampe (2) Région du néocortex. (B) Le placement approprié d’une tranche de cerveau de souris aiguë et d’une harpe sur le réseau de microélectrodes (MEA). (C) L’anatomie d’une puce 3Brain Accura CMOS-HD-MEA. (D) La configuration appropriée des entrées et des sorties de perfusion. L’entrée doit se trouver profondément dans le puits de la puce, tandis que la sortie doit se trouver du côté opposé de l’entrée en haut du puits de la puce pour garantir un flux constant d’aCSF frais et oxygéné. Veuillez cliquer ici pour voir une version agrandie de cette figure.
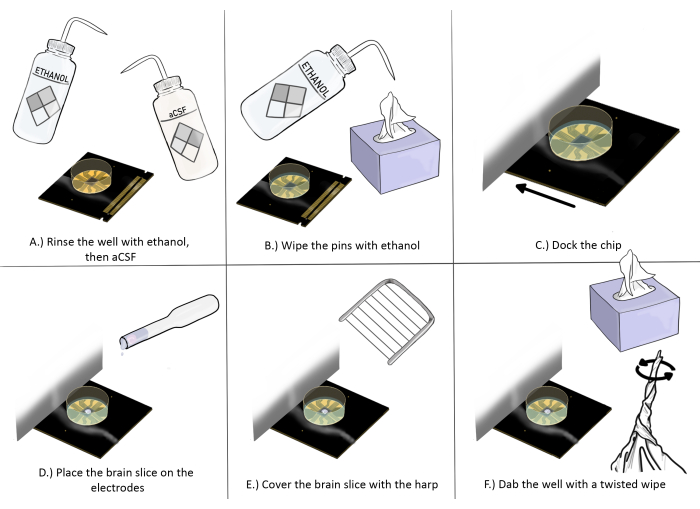
Figure 3 : Schéma de principe de la préparation et du placement des puces pour les expériences de coupe de cerveau. (A) Rincez bien la puce avec de l’éthanol une fois, puis trois fois du LCR. (B) Essuyez les broches avec de l’éthanol à l’aide d’une lingette antistatique. (C) Ancrez la puce. (D) Placez la tranche de cerveau sur les électrodes. (E) Placez la harpe sur la tranche de cerveau (voir la figure 2 pour les directives de placement approprié). (F) Tamponnez le coin du puits des électrodes d’enregistrement près de la tranche de cerveau avec une lingette antistatique tordue. Veuillez cliquer ici pour voir une version agrandie de cette figure.
4. Expérimentation
- Placer la tranche de cerveau
- Placez une harpe platine dans un bateau de pesée (ou une autre surface propre) près de la plate-forme MEA. Recouvrez la harpe d’environ 3 ml d’aCSF pour réduire ses tendances hydrophobes.
- À l’aide de ciseaux, retirez la partie à pointe mince d’une pipette de transfert. Coupez un tiers, environ 1,5 po, de la pointe de la pipette.
REMARQUE : Cette pipette modifiée ne resserrera pas et n’endommagera pas la tranche de cerveau pendant la collecte et le transport. - Prélevez une tranche de cerveau dans la chambre de maintien de la tranche à l’aide de la pipette modifiée. Versez doucement la tranche de cerveau et toute solution contenue dans la pipette dans le puits de la puce. Pour positionner correctement la tranche, libérez doucement une plus grande quantité d’aCSF à l’aide d’une pipette de transfert pour manipuler la position de la tranche de cerveau ou utilisez un pinceau doux pour créer un courant dans la solution qui pousse la tranche de cerveau sur les électrodes d’enregistrement. Limitez le contact avec les électrodes d’enregistrement ou la tranche de cerveau pour minimiser les dommages.
- À l’aide d’une pince, placez délicatement la harpe sur la tranche de cerveau avec les fils vers le bas pour appuyer la tranche sur les électrodes d’enregistrement. Évitez de mettre en contact le porte-électrodes avec la harpe. Orientez la harpe de manière à ce que le côté sans cadre soit orienté vers l’aiguille d’entrée et que le cadre de la harpe n’entre en contact avec aucune des électrodes d’enregistrement (Figure 2C, D).
- Prenez une pipette de rechange et retirez l’excès de LCR. Prenez une lingette antistatique, tournez un coin pour créer une pointe et utilisez-la pour absorber l’aCSF restant entourant les électrodes d’enregistrement sans toucher les électrodes d’enregistrement, la tranche de cerveau ou la harpe (Figure 2).
- À l’aide d’une pipette désignée pour l’aCSF, ajoutez rapidement une quantité suffisante de aCSF carbogéné pour couvrir la tranche de cerveau, soit environ 2 ml.
- Répétez l’étape 4.1.5. et 4.1.6. deux fois de plus.
- Remplissez le puits d’aCSF carbogéné jusqu’à ce que le puits soit rempli aux 3/4 environ, soit environ 3 ml.
- Prenez une photo de la tranche de cerveau sur la puce MEA avec un microscope ou une caméra. Assurez-vous que la photo a une résolution suffisamment élevée pour voir les bords du réseau d’électrodes d’enregistrement et l’anatomie de la tranche de cerveau.
- Déroulement de l’expérience
- Fonctionnement du système de perfusion
- Placez les tubes d’entrée et de sortie dans le bécher rempli d’aCSF et les aiguilles d’entrée et de sortie dans le puits à copeaux. Placez l’aiguille d’entrée près du bas du puits de la puce, juste à l’extérieur des électrodes d’enregistrement. Placez l’aiguille de sortie près du haut du puits de copeaux vers le bord de sorte que le liquide monte presque jusqu’au bord du puits de copeaux, soit environ 4 ml, et que la tranche soit immergée tout au long de l’expérience (figure 2D).
- Réglez l’entrée de perfusion à 5 mL/min et le débit de perfusion à 7 mL/min.
REMARQUE : Il est recommandé que le débit sortant dépasse le débit entrant pour éviter que la solution ne déborde du puits de la puce et pour créer un courant de solution sur la tranche de cerveau. - Activez l’entrée et la sortie. Retirez l’aiguille d’entrée du puits de copeaux jusqu’à ce que l’aiguille commence à sortir de la solution au lieu de l’air. Ensuite, remettez l’aiguille à sa position à l’intérieur du puits de puce comme décrit à l’étape 4.2.1.1.
- Utilisez un réchauffeur de solution pour maintenir la solution à une température physiologique ou proche, autour de 34-37 °C.
- Laissez l’aCSF perfuser sur la tranche de cerveau pendant 10 min. Cela permettra à la tranche de s’adapter à l’environnement d’enregistrement.
- Une fois que 10 minutes se sont écoulées, déplacez le tube de sortie vers le bécher de jet. Ensuite, déplacez le tube d’entrée dans le bécher contenant la solution pro-convulsivante. Laisser le LCRFa non convulsivant être évacué du système de perfusion dans le bécher pendant 10 minutes.
- Transférez le tube d’évacuation dans le bécher contenant la solution pro-convulsivante.
- Laissez la solution pro-convulsivante tourner jusqu’à la fin de l’expérience.
- Si la durée d’enregistrement de la tranche dépasse 2 h, envisagez de préparer une autre solution pro-convulsivante pour compenser la consommation de glucose de la tranche de cerveau active.
- Échange de tranches de cerveau
- Désactivez l’afflux entrant. Coupez le flux sortant 10-15 s après.
- À l’aide d’une pince, retirez la harpe et placez-la sur un bateau de pesée ou une autre surface.
- Utilisez une pipette modifiée pour extraire et jeter la tranche de cerveau. Ne touchez pas les électrodes de référence ou le réseau d’électrodes d’enregistrement (Figure 2C).
- Placez le tube d’entrée dans le bécher contenant la solution aCSF non convulsivante. Placez le tube de sortie dans le bécher de jet. Laissez bien les aiguilles d’entrée et de sortie dans la puce. Faites fonctionner le système de perfusion pendant 10 minutes pour éliminer toute solution pro-convulsivante résiduelle du puits de puce et du système de perfusion. Recommencez le processus d’expérimentation avec une nouvelle tranche de cerveau (à partir de l’étape 4.1).
- Fonctionnement du système de perfusion
- Terminer l’expérience
- Nettoyage de la plate-forme
- Retirez la harpe, la tranche de cerveau et la solution du puits, respectivement.
- Retirez la puce MEA, placez-la sur une surface propre et remplissez bien la puce avec de l’eau déminéralisée.
- Jetez l’eau déminéralisée pour éliminer les sels restants des solutions de CSF.
- À l’aide d’une pipette de transfert, remplissez bien la puce avec une solution détergente. Passez la pointe de la pipette de transfert dans le puits de la puce et pointez-la vers les électrodes d’enregistrement. Pressez et libérez rapidement et à plusieurs reprises le détergent dans la pipette de transfert pour laver vigoureusement le détergent sur les électrodes d’enregistrement pendant 1 min. Laissez reposer le détergent pendant 5 à 10 minutes.
- Retirez le détergent, puis rincez bien la copeau 4 à 6 fois avec de l’eau déminéralisée pour éliminer la solution détergente et tous les solutés restants.
- Retirez toute l’eau du puits de la puce, puis placez une lingette antistatique sur la puce MEA et laissez sécher toute la nuit pour permettre à la puce de bien sécher.
- Placez un bateau de pesée ou une autre plaque étanche sur la plate-forme d’enregistrement MEA où la puce MEA reposait précédemment. Placez la harpe au centre de la plaque et placez les aiguilles d’entrée et de sortie de chaque côté de la harpe.
- Nettoyez le système de perfusion et la harpe en envoyant 50 ml d’eau, 50 ml d’éthanol à 140 degrés et 200 ml d’eau, respectivement, à travers le tube dans le bécher de jet.
REMARQUE : À ce moment, les vitesses de perfusion peuvent être augmentées pour réduire le temps de nettoyage, mais la vitesse de sortie doit toujours dépasser la vitesse d’entrée pour éviter les inondations. - Nettoyez les pierres de carbogène en les plaçant dans un bécher d’eau distillée et en envoyant le carbogen à travers le tube pendant 5 min. Laissez-les reposer sur une surface sèche et propre pendant la nuit et couvrez-les pour éviter l’accumulation de poussière.
- Nettoyage de la plate-forme
5. Analyse des données
REMARQUE : Il existe une variété de progiciels d’analyse utilisés pour analyser les données électrophysiologiques produites par les CMOS-HD-MEA, y compris BrainWave de 3Brain, Yet Another Spike Sorter (YASS) et les outils Python personnalisés 34,35,36,37. Nous avons extrait des données du format de fichier de données BrainWave pour les utiliser dans la plate-forme d’analyse Xenon LFP afin de générer les données présentées dans les figures 4 et 5. Du code Matlab personnalisé a été utilisé pour analyser les données de la figure 6. Les protocoles de la plate-forme d’analyse Xenon LFP sont accessibles au public10. Les étapes de protocole suivantes sont spécifiques aux enregistrements réalisés avec Brainwave 438 ; Pour les autres systèmes, reportez-vous à la documentation de support relative à ces systèmes 34,35,36,37. Vous trouverez ci-dessous un aperçu des étapes d’analyse suivies pour produire les données à l’aide de ce protocole. Pour plus d’informations sur l’exportation, la visualisation et l’analyse des données, y compris des vidéos tutorielles et tous les fichiers de code pertinents, reportez-vous àla section 39.
- Exportez le fichier d’enregistrement pour analyse.
- Créez un dossier pour le(s) fichier(s) d’enregistrement de données qui seront sous-échantillonnés.
- Recadrez l’image prise à l’étape 4.1.9 pour n’inclure que le réseau d’enregistrement avec les tranches de cerveau.
- Ouvrez le fichier ExportToHDF5-ChannelSelection.py (ce fichier de code est accessible sur GitHub sous xenon-lfp-analysis/code-files/3Brain-processing40) et copiez et collez l’URL répertoriée en haut de la fenêtre dans un navigateur Internet.
- Entrez le chemin d’accès au fichier d’enregistrement de données et téléchargez l’image recadrée du tableau associé à l’enregistrement.
- Sous Sélectionner les canaux pour l’exportation, utilisez l’outil de sélection au lasso pour sélectionner les régions de l’hippocampe et du néocortical affichées sur l’image recadrée du réseau.
- Définissez à la fois le nombre de lignes à ignorer et le nombre de colonnes à ignorer sur 2.
REMARQUE : Les fichiers d’enregistrement sont souvent volumineux ; Si cela convient à l’analyse souhaitée, les données exportées peuvent être sous-échantillonnées. Il est possible de sous-échantillonner spatialement (en sautant les rangées et les colonnes d’électrodes d’enregistrement) et temporellement (en sous-échantillonnant à une fréquence d’échantillonnage inférieure). Dans l’analyse, toutes les 2 lignes et 2 colonnes sont ignorées ; Cela permet de sous-échantillonner les données dans l’espace sans être biaisé vers certains canaux. Chaque canal est suffisamment éloigné pour que les différences d’activité puissent être importantes entre eux ; Par conséquent, aucun calcul de moyenne de canal n’est effectué. Lorsque vous sautez des lignes et des colonnes des électrodes, les données des canaux non sélectionnés ne sont pas incluses lors de l’exportation du fichier de sortie. - Réglez la fréquence de sous-échantillonnage sur 300 Hz.
- Cliquez sur le bouton Exporter les chaînes pour générer le fichier de sélection de chaîne.
REMARQUE : Le logiciel enregistrera automatiquement le fichier de sélection de canal dans le dossier où le fichier d’enregistrement de données est enregistré. - Ouvrez le fichier ExportToHDF5-ExtractDownsample.py (également disponible sur GitHub sous xenon-lfp-analysis/code-files/3brain-processing40), collez le chemin d’accès du dossier contenant le(s) fichier(s) de sélection de canal et le(s) fichier(s) d’enregistrement de données, puis appuyez sur la touche Entrée du clavier pour générer la version sous-échantillonnée du fichier d’enregistrement de données.
- Utilisation de la plateforme d’analyse
- Dans l’invite de commande de l’ordinateur, tapez run_lfp_analysis et appuyez sur Entrée pour ouvrir la plate-forme d’analyse.
- Entrez les chemins d’accès aux fichiers de l’image et du fichier d’enregistrement sous-échantillonné, puis cliquez sur Envoyer.
- Sous Sélectionner une plage horaire pour l’analyse, sélectionnez l’enregistrement entier.
- Sous Select Channels for Plots (Sélectionner des canaux pour les tracés), mettez en surbrillance l’hippocampe en tant que groupe 1 et le néocortex en tant que groupe 2.
- Sous Raster de canal, définissez le seuil sur 0,06 mV et la durée sur 0,02 s.
- Cliquez sur Appliquer les paramètres et Générer un raster.
- Utilisez le tracé matriciel pour explorer différents canaux qui affichent des modèles d’activité d’intérêt.
REMARQUE : Voir la figure 4 pour des exemples d’activité de haute qualité semblable à une crise (figure 4A-D) par rapport à une activité sous-optimale (figure 4E-F).
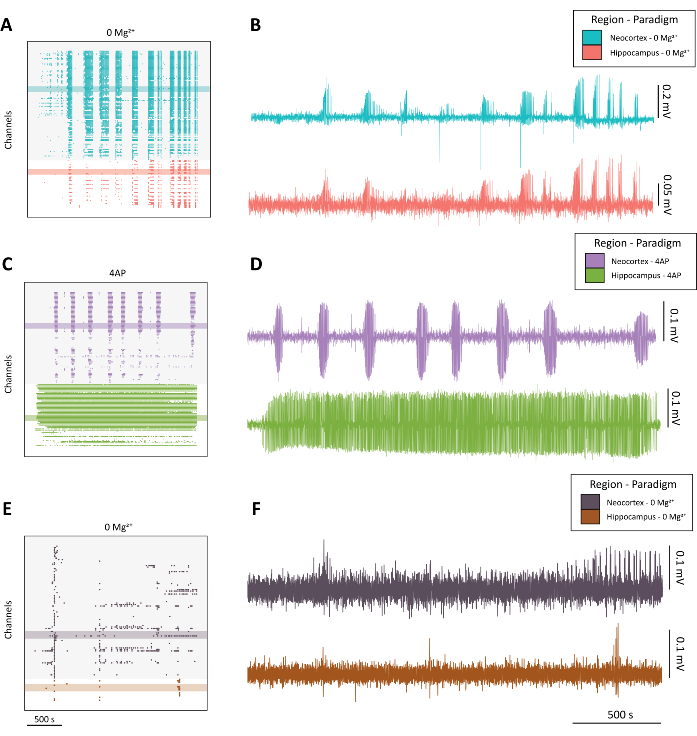
Figure 4 : Exemple d’évolution de l’activité épileptiforme à partir des paradigmes 0 Mg2+ et 4-AP. (A) Exemple de tracé matriciel de l’application d’un LCR avec 0 Mg2+ sur environ 40 min. (B) Exemple de traces électrophysiologiques prélevées sur le néocortex (bleu) et l’hippocampe (rouge) démontrant une activité épileptiforme selon le paradigme 0 Mg2+. (C) Exemple de tracé matriciel de l’application 100 mM 4-AP sur environ 40 min. (D) Exemple de traces électrophysiologiques prélevées sur le néocortex (violet) et l’hippocampe (vert) montrant une activité épileptiforme de l’application de 4-AP. (E) Exemple de graphique matriciel de l’application d’un LCR avec 0 Mg2+ sur environ 40 minutes montrant une activité d’éclatement par opposition à une activité semblable à une crise comme on le trouve dans les autres traces représentatives. (F) Exemple : traces électrophysiologiques prélevées dans le néocortex (violet foncé) et l’hippocampe (rouille) démontrant une activité sous-optimale du paradigme 0 Mg2+ destinée à la comparaison avec l’activité convulsive de qualité trouvée dans B et D. Veuillez cliquer ici pour voir une version plus grande de cette figure.
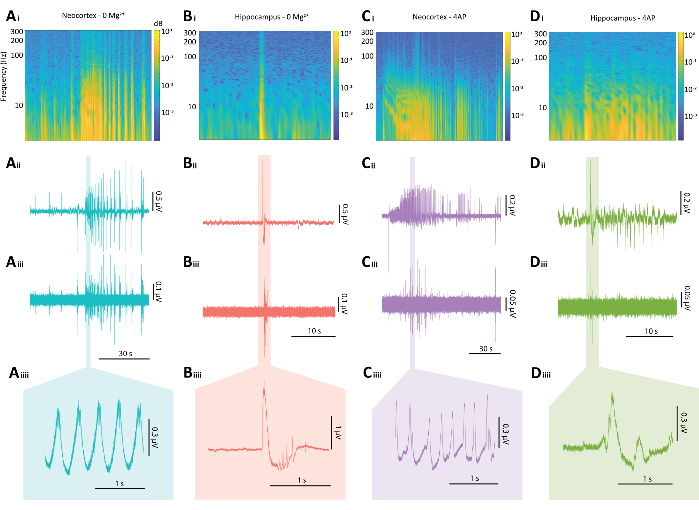
Figure 5 : Résultats représentatifs des décharges épileptiformes des paradigmes 0 Mg2+ et 4-AP. (A) Exemples de graphiques d’un événement typique de type épileptique néocortical induit par le paradigme 0 Mg2+ comprenant (Ai) un spectrogramme d’un événement convulsif, (Aii) la trace électrophysiologique associée, (Aiii) un filtre passe-haut de 80 Hz appliqué à la trace de Aii, (Aiiii) et une section agrandie de la trace de Aii. (B) Exemples de graphiques d’un sursaut épileptiforme hippocampique typique induit par le paradigme 0 Mg2+ comprenant (Bi) un spectrogramme du sursaut épileptiforme, (Bii) la trace électrophysiologique associée, (Biii) un filtre passe-haut de 80 Hz appliqué à la trace de Bii, (Biiii) et une section agrandie de la trace de Bii (C) Exemples de graphiques d’un événement typique de type convulsif néocortical induit par le paradigme 4-AP comprenant (Ci) un spectrogramme de l’activité épileptiforme, (Cii) la trace électrophysiologique associée, (Ciii) un filtre passe-haut de 80 Hz appliqué à la trace de Cii, (Ciiii) et une section agrandie de la trace de Cii (D) Exemples de tracés d’une explosion épileptiforme hippocampique sous le paradigme 4-AP comprenant (Di) un spectrogramme de l’activité épileptiforme, (Dii) la trace électrophysiologique associée, (Diii) un filtre passe-haut de 80 Hz appliqué à la trace de Dii, (Diiii) et une section agrandie de la trace de Dii. Veuillez cliquer ici pour voir une version agrandie de cette figure.
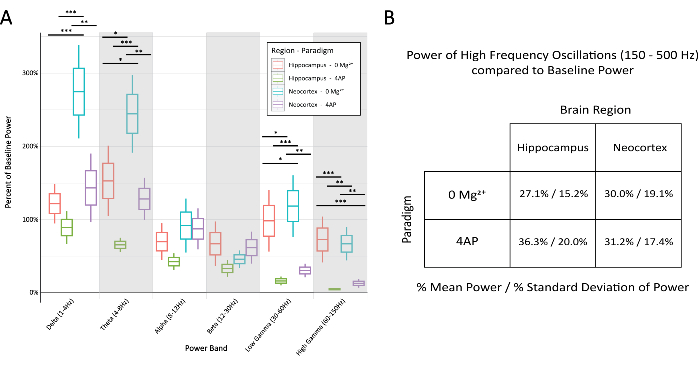
Figure 6 : Comparaison du pourcentage de la puissance de base dans les différentes bandes à travers le paradigme et la région du cerveau pendant les décharges épileptiformes stéréotypées. (A) La puissance pendant les décharges épileptiformes était significativement différente entre les paradigmes et les régions du cerveau pour la plupart des bandes de fréquences (ANOVA à 2 voies avec test de Tukey, *P < 0,05, P < 0,001, P < 0,0001). La ligne médiane de chaque case représente la moyenne, les limites de la case ±1 erreur type de la moyenne (SEM) et les lignes les plus externes ±2 SEM. (B) Les deux paradigmes et les régions du cerveau ont démontré une puissance limitée dans les bandes liées à l’activité à haute fréquence au-dessus de 150 Hz. Veuillez cliquer ici pour voir une version plus grande de cette figure.
Access restricted. Please log in or start a trial to view this content.
Résultats
Comme c’est la norme lorsque l’on visualise l’activité de plusieurs canaux 1,4,5,10, nous trouvons utile de générer d’abord un tracé raster des données que nous acquérons avec le CMOS-HD-MEA (figure 4A,C,E). Ce graphique peut créer une vue d’ensemble de l’activité dans tous les ...
Access restricted. Please log in or start a trial to view this content.
Discussion
Ce protocole comprend des lignes directrices spécifiques liées à la gestion des tranches de cerveau aiguës qui abordent les problèmes courants rencontrés par les utilisateurs de CMOS-HD-MEA, à savoir le développement du bruit sous la tranche de cerveau et le maintien d’un environnement sain pour la tranche de cerveau. Le développement de bruit sous la tranche se produit lorsque la tranche n’adhère pas correctement au réseau ; Si la tranche de cerveau n’adhère pas adéqu...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs déclarent qu’il n’y a pas de conflits d’intérêts associés à cette étude.
Remerciements
Les auteurs remercient les anciens et actuels membres du laboratoire Parrish pour leurs modifications sur ce manuscrit. Nous tenons également à remercier Alessandro Maccione de 3Brain pour ses commentaires sur ce travail. Ce travail a été financé par une bourse de chercheur junior AES/EF et par les collèges des sciences de la vie et des sciences physiques et mathématiques de l’Université Brigham Young.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| 2D Workbench | Cloudray | LM04CLLD26B | |
| 4-Aminopyridine | Sigma-Aldrich | 275875 | |
| Accura Chip | 3Brain | Accura HD-MEA | CMOS-HD-MEA chip |
| Agarose | Thermo Fisher Scientific | BP160-100 | |
| Vibration isolation table | Kinetic Systems | 91010124 | |
| Beaker for the slice holding chamber, 270 mL | VWR | 10754-772 | |
| BioCam | 3Brain | BioCAM DupleX | CMOS-HD-MEA platform |
| Brainwave Software | 3Brain | Version 4 | CMOS-HD-MEA software |
| Calcium Chloride | Thermo Fisher Scientific | BP510-500 | |
| Carbogen | Airgas | X02OX95C2003102 | |
| Carbogen | Airgas | 12005 | |
| Carbogen Stones | Supelco | 59277 | |
| Compresstome | Precissionary | VF-300-0Z | |
| Computer | Dell | Precission3650 | |
| Crocodile Clip Grounding Cables | JWQIDI | B06WGZG17W | |
| Detergent | Metrex | 10-4100-0000 | |
| D-Glucose | Macron Fine Chemicals | 4912-12 | |
| Dihydrogen Sodium Phosphate | Thermo Fisher Scientific | BP329-500 | |
| DinoCam | Dino-Lite | AM73915MZTL | |
| Ethanol | Thermo Fisher Scientific | A407P-4 | |
| Forceps | Fine Science Tools | 11980-13 | |
| Hot plate | Thermo Fisher Scientific | SP88857200 | |
| Ice Machine | Hoshizaki | F801MWH | |
| Inflow and outflow needles | Jensen Global | JG 18-3.0X | |
| Inline Solution Heater | Warner Instruments | SH-27B | |
| Isofluorine | Dechra | 08PB-STE22002-0122 | |
| Kim Wipes | Thermo Fisher Scientific | 06-666 | |
| Magnesium Chloride | Thermo Fisher Scientific | FLM33500 | |
| Micropipets | Gilson | F144069 | |
| Mili-Q Water Filter | Mili-Q | ZR0Q008WW | |
| Paintbrush | Daler Rowney | AF85 Round: 0 | |
| Paper Filter | Whatman | EW-06648-24 | |
| Parafilm | American National Can | PM996 | |
| Perfusion System | Multi Channel System | PPS2 | |
| Pipetor | Thermo Fisher Scientific | FB14955202 | |
| Platinum Harp | 3Brain | 3Brain | |
| Potassium Chloride | Thermo Fisher Scientific | P330-3 | |
| Razor blade | Personna | BP9020 | |
| Scale | Metter Toledo | AB204 | |
| Scissors | Solingen | 92008 | |
| Slice Holding Chamber | Custom | Custom | Custom 3D Printer Design, available upon request |
| Sodium Bicarbonate | Macron Fine Chemicals | 7412-06 | |
| Sodium Chloride | Thermo Fisher Scientific | S271-3 | |
| Temperature Control Box | Warner Instruments | TC344B | |
| Transfer Pipettes | Genesee Scientific | 30-200 | |
| Tubing | Tygon | B-44-3 TPE | |
| Vibratome VZ-300 | Precissionary | VF-00-VM-NC | |
| Weigh Boat | Electron Microscopy Sciences | 70040 |
Références
- Obien, M. E. J., Frey, U. Large-scale, high-resolution microelectrode arrays for interrogation of neurons and networks. Adv Neurobiol. 22, 83-123 (2019).
- Schroter, M., et al. Functional imaging of brain organoids using high-density microelectrode arrays. MRS Bull. 47 (6), 530-544 (2022).
- Miccoli, B., et al. High-density electrical recording and impedance imaging with a multi-modal CMOS multi-electrode array chip. Front Neurosci. 13, 641(2019).
- Emery, B. A., Hu, X., Khanzada, S., Kempermann, G., Amin, H. High-resolution CMOS-based biosensor for assessing hippocampal circuit dynamics in experience-dependent plasticity. Biosens Bioelectron. 237, 115471(2023).
- Ferrea, E., et al. high-resolution electrophysiological imaging of field potentials in brain slices with microelectronic multielectrode arrays. Front Neural Circuits. 6, 80(2012).
- Gagliano, G., et al. Non-linear frequency dependence of neurovascular coupling in the cerebellar cortex implies vasodilation-vasoconstriction competition. Cells. 11 (6), 1047(2022).
- Goodchild, S. J., et al. Molecular pharmacology of selective Na(V)1.6 and dual Na(V)1.6/Na(V)1.2 channel inhibitors that suppress excitatory neuronal activity ex vivo. ACS Chem Neurosci. 15 (6), 1169-1184 (2024).
- Hu, X., Khanzada, S., Klutsch, D., Calegari, F., Amin, H. Implementation of biohybrid olfactory bulb on a high-density CMOS-chip to reveal large-scale spatiotemporal circuit information. Biosens Bioelectron. 198, 113834(2022).
- Kim, S., et al. Alteration of neural network and hippocampal slice activation through exosomes derived from 5XFAD nasal lavage fluid. Int J Mol Sci. 24 (18), 14064(2023).
- Mahadevan, A., Codadu, N. K., Parrish, R. R. Xenon LFP analysis platform is a novel graphical user interface for analysis of local field potential from large-scale MEA recordings. Front Neurosci. 16, 904931(2022).
- Medrihan, L., Ferrea, E., Greco, B., Baldelli, P., Benfenati, F. Asynchronous GABA release is a key determinant of tonic inhibition and controls neuronal excitability: A study in the synapsin II-/- mouse. Cereb Cortex. 25 (10), 3356-3368 (2015).
- Monteverdi, A., Di Domenico, D., D'Angelo, E., Mapelli, L. Anisotropy and frequency dependence of signal propagation in the cerebellar circuit revealed by high-density multielectrode array recordings. Biomedicines. 11 (5), 1475(2023).
- Obien, M. E. J., Hierlemann, A., Frey, U. Accurate signal-source localization in brain slices by means of high-density microelectrode arrays. Sci Rep. 9 (1), 788(2019).
- Thouta, S., et al. Pharmacological determination of the fractional block of Nav channels required to impair neuronal excitability and ex vivo seizures. Front Cell Neurosci. 16, 964691(2022).
- Tognolina, M., Monteverdi, A., D'Angelo, E. Discovering microcircuit secrets with multi-spot imaging and electrophysiological recordings: The example of cerebellar network dynamics. Front Cell Neurosci. 16, 805670(2022).
- Hierlemann, A., Frey, U., Hafizovic, S., Heer, F. Growing cells atop microelectronic chips: Interfacing electrogenic cells in vitro with CMOS-based microelectrode arrays. Proceedings of the IEEE. 99 (2), 252-284 (2011).
- Maccione, A., et al. Experimental investigation on spontaneously active hippocampal cultures recorded by means of high-density MEAs: Analysis of the spatial resolution effects. Front Neuroeng. 3, 4(2010).
- van Vliet, E., et al. Electrophysiological recording of re-aggregating brain cell cultures on multi-electrode arrays to detect acute neurotoxic effects. Neurotoxicology. 28 (6), 1136-1146 (2007).
- Emery, B. A., et al. Large-scale multimodal recordings on a high-density neurochip: Olfactory bulb and hippocampal networks. Annu Int Conf IEEE Eng Med Biol Soc. 2022, 3111-3114 (2022).
- Veleanu, M., et al. Modified climbing fiber/Purkinje cell synaptic connectivity in the cerebellum of the neonatal phencyclidine model of schizophrenia. Proc Natl Acad Sci U S A. 119 (21), e2122544119(2022).
- Giansante, G., et al. Neuronal network activity and connectivity are impaired in a conditional knockout mouse model with PCDH19 mosaic expression. Mol Psychiatry. , (2023).
- Dossi, E., Blauwblomme, T., Nabbout, R., Huberfeld, G., Rouach, N. Multi-electrode array recordings of human epileptic postoperative cortical tissue. J Vis Exp. (92), e51870(2014).
- Hajos, N., et al. Maintaining network activity in submerged hippocampal slices: importance of oxygen supply. Eur J Neurosci. 29 (2), 319-327 (2009).
- Hill, M. R., Greenfield, S. A. The membrane chamber: a new type of in vitro recording chamber. J Neurosci Methods. 195 (1), 15-23 (2011).
- Raimondo, J. V., et al. Methodological standards for in vitro models of epilepsy and epileptic seizures. A TASK1-WG4 report of the AES/ILAE Translational Task Force of the ILAE. Epilepsia. 58 (Suppl 4), 40-52 (2017).
- Hales, C. M., Rolston, J. D., Potter, S. M. How to culture, record and stimulate neuronal networks on micro-electrode arrays (MEAs). J Vis Exp. (39), 2056(2010).
- Lin, C. H., Lee, J. K., LaBarge, M. A. Fabrication and use of microenvironment microarrays (MEArrays). J Vis Exp. (68), 4152(2012).
- Panuccio, G., Colombi, I., Chiappalone, M. Recording and modulation of epileptiform activity in rodent brain slices coupled to microelectrode arrays. J Vis Exp. 135, 57548(2018).
- Patel, C., Muthuswamy, J. High efficiency, site-specific transfection of adherent cells with siRNA using microelectrode arrays (MEA). J Vis Exp. 67, e4415(2012).
- Ting, J. T., Daigle, T. L., Chen, Q., Feng, G. Acute brain slice methods for adult and aging animals: application of targeted patch clamp analysis and optogenetics. Methods Mol Biol. 1183, 221-242 (2014).
- Papouin, T., Haydon, P. G. Obtaining acute brain slices. Bio Protoc. 8 (2), e2699(2018).
- Ting, J. T., et al. Preparation of acute brain slices using an optimized N-Methyl-D-glucamine protective recovery method. J Vis Exp. 132, 53825(2018).
- Van Hoeymissen, E., Philippaert, K., Vennekens, R., Vriens, J., Held, K. Horizontal hippocampal slices of the mouse brain. J Vis Exp. (163), 61753(2020).
- 3Brain. , Available from: https://www.3brain.com/ (2022).
- Bridges, D. C., Tovar, K. R., Wu, B., Hansma, P. K., Kosik, K. S. MEA Viewer: A high-performance interactive application for visualizing electrophysiological data. PLoS One. 13 (2), e0192477(2018).
- Hawrylycz, M., et al. Inferring cortical function in the mouse visual system through large-scale systems neuroscience. Proc Natl Acad Sci U S A. 113 (27), 7337-7344 (2016).
- Maccione, A., et al. Microelectronics, bioinformatics and neurocomputation for massive neuronal recordings in brain circuits with large scale multielectrode array probes. Brain Res Bull. 119 (Pt B), 118-126 (2015).
- 3Brain. , BrainWave 4 https://www.3brain.com/products/software/brainwave4 (2022).
- Mahadevan, A. Xenon LFP Analysis. , https://xenon-lfp-analysis.readthedocs.io/en/latest/Installation.html (2022).
- Mahadevan, A. xenon-lfp-analysis github. , https://github.com/MicroBrew09/xenon-lfp-analysis (2022).
- Codadu, N. K., et al. Divergent paths to seizure-like events. Physiol Rep. 7 (19), e14226(2019).
- Kirsch, G. E., Drewe, J. A. Gating-dependent mechanism of 4-aminopyridine block in two related potassium channels. J Gen Physiol. 102 (5), 797-816 (1993).
- Levesque, M., Salami, P., Behr, C., Avoli, M. Temporal lobe epileptiform activity following systemic administration of 4-aminopyridine in rats. Epilepsia. 54 (4), 596-604 (2013).
- Myers, T. L., Gonzalez, O. C., Stein, J. B., Bazhenov, M. Characterizing concentration-dependent neural dynamics of 4-Aminopyridine-induced epileptiform activity. Epilepsy J. 4 (2), 128(2018).
- Perreault, P., Avoli, M. Physiology and pharmacology of epileptiform activity induced by 4-aminopyridine in rat hippocampal slices. J Neurophysiol. 65 (4), 771-785 (1991).
- Rutecki, P. A., Lebeda, F. J., Johnston, D. 4-Aminopyridine produces epileptiform activity in hippocampus and enhances synaptic excitation and inhibition. J Neurophysiol. 57 (6), 1911-1924 (1987).
- Chen, Y., Chad, J. E., Cannon, R. C., Wheal, H. V. Reduced Mg2+ blockade of synaptically activated N-methyl-D-aspartate receptor-channels in CA1 pyramidal neurons in kainic acid-lesioned rat hippocampus. Neuroscience. 88 (3), 727-739 (1999).
- Fujiwara-Tsukamoto, Y., Isomura, Y., Takada, M. Comparable GABAergic mechanisms of hippocampal seizure-like activity in posttetanic and low-Mg2+ conditions. J Neurophysiol. 95 (3), 2013-2019 (2006).
- Swartzwelder, H. S., Anderson, W. W., Wilson, W. A. Mechanism of electrographic seizure generation in the hippocampal slice in Mg2+-free medium: the role of GABAa inhibition. Epilepsy Res. 2 (4), 239-245 (1988).
- Trevelyan, A. J., Graham, R. T., Parrish, R. R., Codadu, N. K. Synergistic positive feedback mechanisms underlying seizure initiation. Epilepsy Curr. 23 (1), 38-43 (2023).
- Croning, M. D., Haddad, G. G. Comparison of brain slice chamber designs for investigations of oxygen deprivation in vitro. J Neurosci Methods. 81 (1-2), 103-111 (1998).
- Hajos, N., Mody, I. Establishing a physiological environment for visualized in vitro brain slice recordings by increasing oxygen supply and modifying aCSF content. J Neurosci Methods. 183 (2), 107-113 (2009).
- Huang, Y., Williams, J. C., Johnson, S. M. Brain slice on a chip: opportunities and challenges of applying microfluidic technology to intact tissues. Lab Chip. 12 (12), 2103-2117 (2012).
- Andrew, R. D., et al. The critical role of spreading depolarizations in early brain injury: Consensus and contention. Neurocrit Care. 37 (Suppl 1), 83-101 (2022).
- Devonshire, I. M., Dommett, E. J., Grandy, T. H., Halliday, A. C., Greenfield, S. A. Environmental enrichment differentially modifies specific components of sensory-evoked activity in rat barrel cortex as revealed by simultaneous electrophysiological recordings and optical imaging in vivo. Neuroscience. 170 (2), 662-669 (2010).
- Parrish, R. R., Codadu, N. K., Mackenzie-Gray Scott, C., Trevelyan, A. J. Feedforward inhibition ahead of ictal wavefronts is provided by both parvalbumin- and somatostatin-expressing interneurons. J Physiol. 597 (8), 2297-2314 (2019).
- Wang, H., Jing, M., Li, Y. Lighting up the brain: genetically encoded fluorescent sensors for imaging neurotransmitters and neuromodulators. Curr Opin Neurobiol. 50, 171-178 (2018).
- Yaksi, E., Jamali, A., Diaz Verdugo, C., Jurisch-Yaksi, N. Past, present and future of zebrafish in epilepsy research. FEBS J. 288 (24), 7243-7255 (2021).
- He, M. F., et al. Ex vivo calcium imaging for drosophila model of epilepsy. J Vis Exp. 200, 65825(2023).
- Driscoll, N., et al. Multimodal in vivo recording using transparent graphene microelectrodes illuminates spatiotemporal seizure dynamics at the microscale. Commun Biol. 4 (1), 136(2021).
- Parrish, R. R., Grady, J., Codadu, N. K., Trevelyan, A. J., Racca, C. Simultaneous profiling of activity patterns in multiple neuronal subclasses. J Neurosci Methods. 303, 16-29 (2018).
- Valderhaug, V. D., et al. Criticality as a measure of developing proteinopathy in engineered human neural networks. bioRxiv. , (2020).
- Multi-electrode array (MEASs) to investigate pathogenetic disease mechanisms and pharmacological properties in iPSC-derived neurons modelling neuropsychiatric diseases. Carleo, G., Lee, Y. -S., Secondo, A., Miceli, F., Taglialatela, M. 2022 IEEE International Conference on Metrology for Extended Reality, Artificial Intelligence and Neural Engineering (MetroXRAINE), , Rome, Italy. 667-672 (2022).
- Ruz, I. D., Schultz, S. R. Localising and classifying neurons from high density MEA recordings. J Neurosci Methods. 233, 115-128 (2014).
- Franke, F., Natora, M., Boucsein, C., Munk, M. H. J., Obermayer, K. An online spike detection and spike classification algorithm capable of instantaneous resolution of overlapping spikes. J Comput Neurosci. 29 (1-2), 127-148 (2010).
- Vollgraf, R., Obermayer, K. Improved optimal linear filters for the discrimination of multichannel waveform templates for spike-sorting applications. IEEE Signal Processing Letters. 13 (3), 121-124 (2006).
- Muller, J., et al. High-resolution CMOS MEA platform to study neurons at subcellular, cellular, and network levels. Lab Chip. 15 (13), 2767-2780 (2015).
- Mapelli, L., et al. implementation, and functional validation of a new generation of microneedle 3D high-density CMOS multi-electrode array for brain tissue and spheroids. bioRxiv. , (2022).
- Reddy, D. S., Kuruba, R. Experimental models of status epilepticus and neuronal injury for evaluation of therapeutic interventions. Int J Mol Sci. 14 (9), 18284-18318 (2013).
- Parrish, R. R., Trevelyan, A. J. Stress-testing the brain to understand its breaking points. J Physiol. 596 (11), 2033-2034 (2018).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon