Method Article
通过 CMG 解旋酶对 DNA 解旋进行单分子实时可视化
摘要
该方案演示了执行单分子分析,以通过 CMG 解旋酶实时观察 DNA 解旋。它描述了 (1) 制备 DNA 底物,(2) 纯化荧光标记的 黑腹果蝇 CMG 解旋酶,(3) 制备用于全内反射荧光 (TIRF) 显微镜的微流体流动池,以及 (4) 单分子 DNA 展开测定。
摘要
忠实的基因组复制对于保持分裂细胞的遗传稳定性至关重要。DNA 复制在 S 期由称为复制体的动态蛋白质复合物进行。复制体的核心是 CDC45-MCM2-7-GINS (CMG) 解旋酶,它将 DNA 双螺旋的两条链分开,以便 DNA 聚合酶可以复制每条链。在基因组复制过程中,复制体必须克服大量的障碍和挑战。这些都威胁到基因组的稳定性,因为无法完全准确地复制 DNA 会导致突变、疾病或细胞死亡。因此,了解 CMG 在正常复制和复制应激期间如何在复制体中发挥作用是非常有趣的。在这里,我们描述了一种使用重组纯化蛋白的全内反射荧光 (TIRF) 显微镜测定,它允许通过单个 CMG 复合物实时可视化表面栓系的拉伸 DNA 分子。该分析提供了一个强大的平台,可以在单分子水平上研究 CMG 行为,从而可以直接观察解旋酶动力学,并实时控制反应条件。
引言
DNA 复制受到严格调节,因为细胞必须准确复制其基因组以防止突变、疾病或死亡。真核 DNA 复制由复制体复合物进行,该复合物解开亲本 DNA 并使用单链 DNA (ssDNA) 作为模板合成新的 DNA。在 G1 期,MCM2-7 的催化失活双六聚体在复制起点1 上加载到双链 DNA (dsDNA) 上。在 S 期,MCM2-7 复合物被 CDC45 和 GINS2 结合激活,形成 11 个亚基的 CMG 复合物(CDC45、MCM2-7、GINS)。每个 CMG 都以相反的方向启动 DNA 解旋,形成复制体围绕3 排列的核心单元。
二十年前,CMG 解旋酶首次被鉴定为 11 亚基复合体,对 DNA 复制至关重要4。从那时起,我们对 CMG 的理解有了很大的进步,从加载和激活 5,6 到 DNA 解旋和终止7。传统的生化和结构生物学技术对许多这些发现至关重要;然而,这些方法在研究 CMG 更动态方面的能力往往受到限制。单分子方法使用单个生物分子的物理操作来测量或可视化它们一次一个分子的活性。这可用于深入了解其他技术经常遗漏或无法检测到的蛋白质的实时动态 8,9。
在这里,我们描述了一种全内反射荧光 (TIRF) 显微镜测定法,以实时可视化 CMG 解旋酶的 DNA 解旋。将纯化的荧光标记 CMG 上样到含有预制 DNA 叉结构的长 DNA 的游离 3' 端。通过将 DNA 的每一端依次拴在表面,将线性 DNA 拉伸在微流体流动池中的生物素-PEG 盖玻片上。这种方法允许更均匀的 DNA 栓系,从而显著减少数据分析过程中必须考虑的变异。在 ATP-γ-s 存在下,CMG 被加载到叉子 3' 端的单链 DNA 上。ATP-γ-s 是一种缓慢水解的 ATP 类似物,允许 CMG 结合到 DNA 上,但不能解旋。随后添加 ATP 以及纯化的荧光标记 RPA,激活 CMG 并启动广泛的 DNA 解旋。从视觉上看,CMG 沿 DNA 易位,在其后面留下不断增长的 RPA 结合 ssDNA 片段。未栓系的 DNA 末端随 CMG 移动,由于 RPA 结合引起的压缩而形成一个"紧密球"。流通池设计允许在展开过程中随时更换缓冲液,从而在每次实验期间和实验期间提供很好的控制。
该协议分为四种方法,可以彼此独立执行。第 1 节描述了用于单分子检测的 20 kb 线性分叉 DNA 底物的制备。第 2 节概述了 黑腹果蝇 CMG (DmCMG) 的纯化和荧光标记。有关 DmCMG 表达的关键信息包含在注释部分。第 3 节介绍了可用于 TIRF 显微镜的微流体流通池的制备。第 4 节描述了如何进行单分子 DNA 解旋测定。
研究方案
1. 制备用于单分子检测的 20 kb 线性分叉 DNA(图 1)
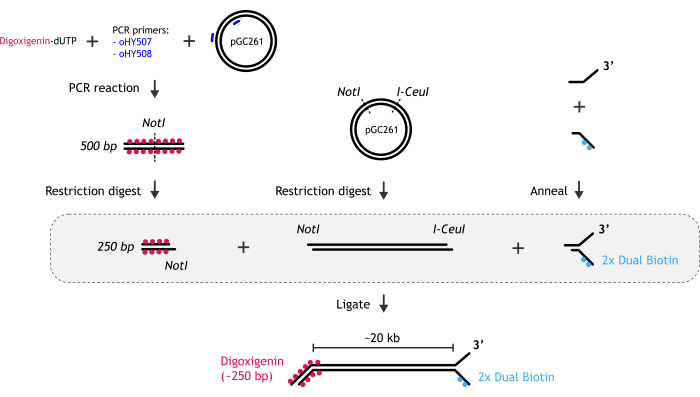
图 1:DNA 底物制备的图形表示。 (A) 生物素化的 DNA 叉端是通过对两个部分互补的寡核苷酸(生物素化和非生物素化寡核苷酸)进行退火而形成的。(B) pGC261 质粒与两种酶的限制性消化产生主要的 dsDNA 片段 (~20 kb),以产生两端具有不同突出端的线性 DNA。(C) 地高辛双链体 DNA 末端是在地高辛-dUTP 存在下进行 PCR 反应,然后进行限制性消化而获得的。 请单击此处查看此图的较大版本。
- 生成生物素化 DNA 叉端。
- 对叉子 DNA 进行退火。
- 将两种寡核苷酸(oHY502 和 oHYbio85)在 1x STE 缓冲液(100 mM NaCl、10 mM Tris,pH 8.0;1 mM 乙二胺四乙酸 [EDTA])中混合。最终寡核苷酸浓度为 10 μM,最终反应体积为 100 μL。
- 将反应物在 80 °C 下在加热块上孵育 5 分钟。关闭加热块,让反应逐渐冷却,将其留在加热块中,直到温度降至 30 °C 以下。
- 验证 Tris-硼酸盐-EDTA (TBE)-聚丙烯酰胺凝胶电泳 (PAGE) 凝胶上的退火效率。
- 制备 8% 丙烯酰胺 - TBE 凝胶(4 块凝胶:32.8 mL H2O;4.8 mL 10x TBE 缓冲液(1 M Tris、1 M 硼酸、20 mM EDTA)、9.6 mL 40% 丙烯酰胺/双溶液,37.5:1;0.8 mL 10% 过硫酸铵 [APS]、40 μL 四甲基乙二胺 [TEMED])。
注意:TBE、丙烯酰胺、APS、TEMED 有毒/有害。 - 在 RT 下,在 8% 丙烯酰胺 - TBE 凝胶上,在 80 V 下运行小体积 (1 μL) 退火寡核苷酸以及用 6x 上样染料稀释的单个寡核苷酸 1 小时。
- 将凝胶浸泡在补充有合适核酸染色剂的 1x TBE 中,在跷跷板摇床上浸泡 30 分钟,然后使用凝胶成像系统扫描凝胶。
- 制备 8% 丙烯酰胺 - TBE 凝胶(4 块凝胶:32.8 mL H2O;4.8 mL 10x TBE 缓冲液(1 M Tris、1 M 硼酸、20 mM EDTA)、9.6 mL 40% 丙烯酰胺/双溶液,37.5:1;0.8 mL 10% 过硫酸铵 [APS]、40 μL 四甲基乙二胺 [TEMED])。
- 在 TBE-PAGE 凝胶上分离退火的 DNA 以进行凝胶切除。
- 如果寡核苷酸正确退火,则在 BSA 存在下(~100 μL 退火样品、25 μL 6x 上样染料、2 μL 33 mg/mL BSA)分离样品的其余部分。要在凝胶上加载更大的体积,请将几个孔与无菌手术刀混合。
- 将凝胶置于补充有合适核酸染色剂的 1x TBE 中,在跷跷板摇床上放置 30 分钟。
- 将凝胶转移到玻璃托盘中,并在黑暗的房间中在蓝光下观察 DNA。使用蓝光时,请使用合适的防护屏或防护眼镜。
- 用无菌手术刀切出所需的带子。修剪掉多余的凝胶。
- 通过电洗脱纯化退火叉。
- 剪下一段足够长的透析管以插入凝胶块,在 1x TBE 中润湿,然后将一端夹住。
- 在 500 μL 1x TBE 中将 BSA 稀释至 0.3 mg/mL,然后将整个体积移液到管中。
- 使用金属刮刀将凝胶片滑入其中。将凝胶片移动到管的一侧,为 DNA 在电洗脱过程中迁移提供缓冲空间。
- 挤出多余的缓冲液并夹住管路的第二端。
注:剩余体积将决定最终体积和浓度。 - 将透析管放入溶于 1x TBE 缓冲液中的琼脂糖电泳槽中,确保管完全浸没。
注:确保将凝胶片放置在最靠近负极的一侧,以便 DNA 可以从凝胶中迁移到透析袋中。 - 在 80 V 下运行电洗脱 1-2 小时。
- 将电洗脱的 DNA 透析到 10 mM Tris 缓冲液 (pH 8) 中。
- 电洗脱后,从透析槽中取出透析袋,用纸巾擦干一端。避免用纸巾接触管子的中间部分,即凝胶和样品所在的位置。
- 从干燥端取下夹子,轻轻地在管内上下吹打样品,将 DNA 混合到缓冲液中。
- 将干净的刮刀浸入 1x TBE 缓冲液中,然后从管道中舀出凝胶片。
注:去除凝胶片时,请尽可能少地去除缓冲液。 - 再次将透析袋的开口端夹住,去除内部的任何空气。
- 将透析袋放入装有 1.5 L 10 mM Tris 缓冲液 (pH 8)、20 mM NaCl 和 2 mM MgCl2 的 2 L 烧杯中。用封口膜包裹夹子,将管路粘在烧杯的边缘,同时将管路完全浸入缓冲液中。
注:盐可稳定短寡核苷酸双链体。 - 使用磁棒轻轻搅拌缓冲液,并在室温 (RT) 下透析样品至少 3-4 小时或在 4 °C 下过夜。
- 透析后,用纸巾擦干透析袋的一端(不是为了稀释 DNA)并解开这一端。
- 在管内轻轻地上下移液样品,然后将其转移到干净的 1.5 mL 试管中。
- 使用超微量分光光度计测量 DNA 的浓度。
- 对叉子 DNA 进行退火。
- 生成 20 kb 片段
- 使用 NotI/I-CeuI 酶对 pGC261 质粒18 进行限制性消化。
- 在限制性内切酶生产商推荐的缓冲液中,轻轻混合 8 μL NotI-HF (20,000 U/mL)、8 μL I-CeuI (5,000 U/mL) 和 pGC261 质粒(200 μL 反应中的终质粒浓度 ~40 ng/μL)。
- 将反应物在 37 °C 下孵育过夜。
- 在琼脂糖凝胶上分离消化的 DNA,用于凝胶切除。
- 可选:在上样整个反应之前,先在 0.6% 琼脂糖凝胶上运行少量反应以测试消化效率。
- 按照步骤 1.2.2.3-1.2.2.4 制备 0.6% 琼脂糖凝胶。
- 将 0.48 g 琼脂糖粉末与 80 mL 1x TBE 缓冲液混合。将溶液微波加热至沸腾。涡旋以确保琼脂糖完全熔化。冷却几分钟,然后倒入指定的罐中。
- 要合并孔以容纳更大体积的样品,请事先在梳子的多个孔上贴上一条胶带。
- 凝胶凝固后,轻轻取下梳子,并在槽中加入 1x TBE 缓冲液。将样品与 DNA 上样染料轻轻混合,并在 120 V 下运行凝胶 1 小时。
- 将凝胶留在跷跷板摇床上,将凝胶置于补充有合适核酸染色剂的 1x TBE 中,直至染色。
- 将染色的凝胶转移到玻璃托盘上,并在黑暗的房间中在蓝光下观察 DNA。
- 用无菌手术刀切出所需的带子。修剪掉多余的凝胶。
- 可选:如步骤 1.1.4 和 1.1.5 中所述,通过电洗脱纯化所需的片段。此处省略透析缓冲液中的盐,因为这仅用于稳定短寡核苷酸双链体。
- 使用 NotI/I-CeuI 酶对 pGC261 质粒18 进行限制性消化。
- 地高辛双链 DNA 末端的生成
- 以 pGC261 DNA 为模板,用地高辛-dUTP (dig-dUTP) 进行 PCR 反应。
- 混合:400 μL 水、8 μL pGC261 (0.8 ng/μL)、3.5 μL 引物 oHY507 (100 μM)、3.5 μL 引物 oHY508 (100 μM)、8 μL dig-dUTP (1 mM)、400 μL 2x PCR 混合物(由 DNA 聚合酶 (20 μL) 制备)、10 mM dNTP (40 μL)、5x 高保真 (HF) 缓冲液 (400 μL) 和水 (540 μL)。
- 在热循环仪中运行以下反应:
98 °C - 1 分钟
30x: 98 °C - 20 s;65 °C - 20 秒;72 °C - 30 秒;
72 °C - 10 分钟
4 °C - 保持
- 消化和纯化地高辛标记的 PCR 产物
- 使用市售 DNA 纯化试剂盒纯化 PCR 产物。
- 将 10 μL NotI-HF (20,000 U/mL) 与 PCR 产物混合,加入酶供应商推荐的缓冲液(在 ~200 μL 反应中,最终 DNA 浓度为 ~50 ng/μL)。将反应物在 37 °C 下孵育过夜。
- 使用市售 DNA 纯化试剂盒纯化消化的 DNA。
- 以 pGC261 DNA 为模板,用地高辛-dUTP (dig-dUTP) 进行 PCR 反应。
- 组装组件以制备 DNA 底物。
- 在 200 μL 反应中,将生物素化叉端、20 kb DNA 片段和地高辛标记的 Not-I 处理的 PCR 片段与 5 μL T4 DNA 连接酶 (400,000 U/mL) 在酶供应商推荐的缓冲液中轻轻混合。然而,根据添加的 20 kb DNA 片段 (1-5 μg),添加大约 100 倍摩尔过量的生物素叉端和 PCR 片段。
- 将反应物分装到 PCR 管(每个 50 μL)中,并在 16 °C 下在热循环仪中孵育过夜。
- 在 0.6% 琼脂糖凝胶上分离连接的样品,并按照步骤 1.2.2 和 1.2.3 中的说明通过电洗脱纯化连接的 DNA。
- 将快速冷冻 DNA 储存在 -80 °C 下。
2. 黑腹果 蝇 CMG 的纯化(图 2)

图 2:从 4 L Hi Five 细胞中纯化 黑腹果蝇 CMG。 在 MOPS 缓冲液存在下,在 200 V 下,在 4%-12% Bis-Tris 聚丙烯酰胺凝胶上分离蛋白质。样品显示在纯化的每个阶段(细胞裂解物 - 2 μL,FLAG 洗脱 - 10 μL,第一个离子交换柱后 - 10 μL,标记后和第二个离子交换柱 - 1 μL。(A) 考马斯染色证实了荧光标记之前 (10 μL) 和 (1 μL) 荧光标记后 CMG 复合物的所有 11 个亚基的存在。(B) 通过使用长通红光 (LPR) 滤光片的荧光图像分析仪扫描 Cy5 来验证 MCM3 亚基的标记效率。 请单击此处查看此图的较大版本。
注:为了制备荧光标记 的黑腹果蝇 CMG,在 MCM3 亚基(在 pFastBac1 载体中)的 N 末端 FLAG 标签下游引入一个 TEV 切割位点 (ENLYFQG),后跟四个 Gly 残基10。为了表达复合物,使用了杆状病毒表达系统。对于初始转染,每个 CMG 亚基 (P1 病毒阶段) 分别使用 Sf21 细胞。为了进一步扩增病毒,使用了 Sf9 细胞 (P2 病毒阶段)。随后,用补充有 10% 胎牛血清的 0.5 mL P2 病毒(P3 病毒期)感染 Sf9 细胞培养物 (每个 CMG 亚基 100 mL;0.5 x 106 个细胞/mL)。为了在 4 L Hi Five 细胞 (1 x 106 个细胞/mL) 中表达整个 CMG 复合物,每个亚基使用 200 mL 的 P3 病毒。收获表达 CMG 复合物的 Hi Five 细胞后,可将细胞沉淀在液氮中快速冷冻并储存在 -80°C 下。 在冰上或 4 °C 下进行整个纯化。 缓冲液可以提前制备,前提是在使用前添加还原剂(DTT 或 2-巯基乙醇)和蛋白酶抑制剂 (CAUTION)。确保所有缓冲液都提前预冷、过滤和脱气。
- 准备以下缓冲液。
- 通过混合 25 mM 4-(2-羟乙基)-1-哌嗪乙磺酸 (HEPES) pH 7.5、0.02% 吐温-20、10% 甘油、15 mM KCl、2 mM MgCl2、2 mM 2-巯基乙醇、1 mM EDTA 和 1 mM 乙二醇-双(β-氨基乙基醚)-N、N、N'、N'-四乙酸 (EGTA) 来制备重悬缓冲液 A。
注意:2-巯基乙醇、DTT 和 EDTA 有毒/有害 - 制备 Tris 缓冲盐水(TBS;0.1 M 甘氨酸盐酸盐,pH 3.5)缓冲液,用于制备 ANTI-FLAG M2 亲和凝胶。
- 通过混合 25 mM HEPES pH 7.5、0.02% Tween-20、10% 甘油、100 mM KCl、1 mM DTT、1 mM EDTA 和 1 mM EGTA 来制备缓冲液 A-100。
- 通过添加 25 mM HEPES pH 7.5、50 mM 乙酸钠、10 mM 乙酸镁、10% 甘油和 1 mM DTT 来制备透析缓冲液。
- 通过混合 50 mM Tris-HCl、pH 7.5 和 150 mM NaCl 来制备 TBS 缓冲液。
- 通过混合 25 mM 4-(2-羟乙基)-1-哌嗪乙磺酸 (HEPES) pH 7.5、0.02% 吐温-20、10% 甘油、15 mM KCl、2 mM MgCl2、2 mM 2-巯基乙醇、1 mM EDTA 和 1 mM 乙二醇-双(β-氨基乙基醚)-N、N、N'、N'-四乙酸 (EGTA) 来制备重悬缓冲液 A。
- 将 4 L Hi Five 细胞培养物的沉淀重悬于 200 mL 补充有蛋白酶抑制剂混合物片剂的冷重悬缓冲液 A 中。倒置混合试管,直到整个沉淀完全重悬。
- 将重悬的细胞转移到预冷的 40 mL Dounce 匀浆器中。为了回收尽可能多的细胞,用相同类型的缓冲液洗涤用于冷冻细胞沉淀的试管,并添加到 Dounce 匀浆器中。确保均质器中的总体积不超过建议的限值。在冰上裂解细胞 60-70 次。
注意:确保柱塞到达管底部,但不要施加太大的压力,否则均质器可能会破裂。小心不要在液面以上取出柱塞,因为这会引入气泡。 - 重复上一步,直到整个样品均质化,然后将其混合在预冷的烧杯中。评估样品的最终体积。逐滴加入过滤后的 KCl 至终浓度为 100 mM,轻轻混合。
- 提前将离心机预冷至 4 °C。将样品倒入离心管中,确保达到生产商推荐的体积。在秤上平衡试管。
注意:体积过小的试管在离心过程中可能会破裂。 - 将裂解的细胞在 4 °C 下以 23,500 x g 离心 15-30 分钟。 同时,根据下一步准备 ANTI-FLAG M2 亲和凝胶。
- 轻轻混合含有 ANTI-FLAG M2 亲和凝胶(50% 悬浮液)的瓶子。切开 P-100 移液器吸头的末端(以加宽吸头),并立即将其将 4 mL 悬浮液(2 mL 珠子)转移到 20 mL 多萃制备色谱柱中。要恢复卡在吸头中的珠子,请用 TBS 清洗吸头并将其添加到柱中。让所有珠子沉淀在柱内,但要小心不要干燥它们。
- 用 3 倍珠子体积(共 6 mL)的 0.1 M 甘氨酸盐酸盐 pH 3.5 洗涤树脂。随后,用 3-5 个珠子体积的 TBS (6-10 mL) 洗涤珠子。接下来,用缓冲液 A-100 洗涤磁珠 3 次 (6 mL)。
注:请勿将微珠置于 0.1 M 盐酸甘氨酸 pH 3.5 溶液中超过 20 分钟。要提高流速压力和洗涤速度,请用盖子盖住色谱柱。任何时候都不要让树脂干燥。 - 制备填料后,在填料上方留下 2 mL 缓冲液 A-100,并关闭色谱柱出口。
- 离心后,将上清液轻轻倒入预冷的 50 mL 试管中。小心不要破坏颗粒。取一小份 (~10 μL) 稍后通过 SDS-PAGE 电泳进行验证。
- 将等量的 ANTI-FLAG M2 微珠(按照上一步制备)添加到每个 50 mL 试管中。尝试用几毫升额外的缓冲液 A-100 重悬于柱子中,并将重新悬浮的珠子转移到试管中,从而从柱子中回收所有珠子。
- 将样品(上清液)与 ANTI-FLAG M2 树脂一起孵育,在 4 °C 下旋转 2.5 小时。
- 孵育后,将样品在 4 °C 下以 1,000 x g 旋转 5 分钟。 使用移液管除去大部分上清液。使用切割尖端,将珠子重悬于几毫升剩余的上清液中,然后将它们转移到两个 15 mL 试管中。要回收粘附在 50 mL 试管壁上的任何微珠,请添加几毫升缓冲液 A-100 并将其也转移到 15 mL 试管中。
- 在 4 °C 下以 1,000 x g 的速度旋转 15 mL 试管 5 分钟。 去除上清液。
- 向每个试管中加入 ~14 mL 缓冲液 A-100 洗涤珠子,然后在 4 °C 下短暂、温和地旋转,然后在 4 °C 下以 1000 x g 旋转 5 分钟。 重复洗涤两次。每次洗涤液取小等分试样 (~10 μL),稍后通过 SDS-PAGE 电泳进行验证。
- 此步骤 (2.16) 是前面步骤 (2.13-2.15) 的替代过程。
- 与 ANTI-FLAG M2 填料孵育后,将样品倒入两个 20 mL 多余制备层析柱中。
- 如果选择这种方法,让磁珠沉淀在色谱柱底部,未结合的样品通过重力流通过色谱柱。随后,将缓冲液 A-100 直接添加到色谱柱中,让它通过重力流清洗样品。
- 将磁珠(重悬于缓冲液 A-100 中)转移到两个 10 mL 多余制备色谱柱中。
- 让缓冲液 A-100 穿过填料,当缓冲液水平达到填料顶部时关闭出口,然后加入补充有 200 μg/mL (DYKDDDDK) 肽(FLAG 洗脱缓冲液)的缓冲液 A-100 从磁珠中洗脱蛋白质。
- 对于第一次洗脱,加入 3 mL FLAG 洗脱缓冲液,并在室温下轻轻旋转关闭的色谱柱 15 分钟。打开出口以收集第一个洗脱馏分。
- 关闭出口,向每根色谱柱中加入 2 mL 的 FLAG 洗脱缓冲液。再旋转 10 分钟并收集第二个洗脱馏分。
- 将所有洗脱的馏分混合在一起 (~10 mL) 并保持在 4 °C。 取一小份洗脱的馏分 (~10 μL),稍后通过 SDS-PAGE 电泳进行验证。
- 使用 0.22 μm 针式过滤器过滤洗脱的样品。
- 根据制造商的方案,提前准备连接到蛋白质纯化系统的 CaptoHiRes Q (5/50) 色谱柱(高分辨率离子交换色谱柱)。随后,用缓冲液 A-100 平衡色谱柱,并将过滤后的样品上样到平衡的色谱柱上。
- 用 20 倍柱体积 (CV) 的缓冲液 A-100(总共 ~20 mL,因为柱的体积为 ~1 mL)洗涤柱。
- 要洗脱蛋白质,请提前准备两种缓冲液:缓冲液 A-100 和缓冲液 B,其成分与缓冲液 A-100 相同,但使用 550 mM KCl 而不是 100 mM KCl。将梯度洗脱设置为 20 CV (~20 mL),盐浓度从 100 mM 增加到 550 mM KCl。在馏分收集器中收集 0.3-0.5 mL 洗脱液。
注:CMG 应在缓冲液 B 浓度约为 70%-75% 的情况下洗脱。取小等分试样的洗涤液和洗脱级分进行 SDS-PAGE 电泳。 - 进行 SDS-PAGE 电泳以确认所选级分中存在 CMG。同时,按照制造商的方案清洁色谱柱和纯化系统。
- 通过每 1 mL 样品混合 50 μL TEV 蛋白酶 (1 mg/mL),将所选组分与 TEV 蛋白酶孵育过夜。
- 预湿透析管并确保膜未损坏。将 CMG/TEV 混合物加入透析管中,放入装有 1.5 L 预冷缓冲液 A-100 的 2 L 烧杯中,并放入内部磁棒中。在 4°C 下轻轻搅拌过夜。
- 在 5 mM CaCl2 存在下,将样品与 50 μM LD655 标记的肽和 10 μg/mL Sortase 酶混合。在 4 °C 下孵育反应,旋转 30 分钟,确保试管避光。在标记前后取一小份 SDS-PAGE 电泳。
- 在加载到纯化系统之前,使用 0.22 μm 离心过滤器过滤标记的样品,因为肽可能会沉淀。
- 如前所述(步骤 2.20),提前准备高分辨率离子交换色谱柱。这一次,用铝箔覆盖色谱柱和馏分收集系统,以保护样品避光。
- 要去除游离肽,请将过滤后的样品加载到高分辨率离子交换色谱柱上。以与之前描述相同的方式进行纯化(步骤 2.21-2.22)。确保标记的 CMG 以与之前相似的缓冲液-B 浓度洗脱。
- 进行 SDS-PAGE 电泳以验证所选组分的质量。要对荧光进行成像,上样前不要将样品煮沸,并确保电泳槽避光。首先使用凝胶成像系统观察蛋白质荧光,然后用考马斯染料对凝胶进行染色,并再次成像以可视化所有蛋白质。
- 在 4 °C 下,对照 1.5 L 透析缓冲液将所选组分透析过夜。如有必要,浓缩样品。
- 将蛋白质在液氮中快速冷冻并储存在 -80 °C 直至进一步使用。
3. 流通池的制备(图 3)
- 按照先前描述的方案11 准备生物素-PEG 盖玻片,省略了烘箱烘烤步骤。生物素-PEG 盖玻片在 RT 的真空下可稳定保存至少 1 个月。为了制备流通池,将生物素-PEG 盖玻片 (24 mm x 60 mm) 切成两半(约 24 mm x 30 mm)。
注意:注意不要触摸盖玻片的中心区域,那里是流路所在的位置。 - 通过将载玻片蚀刻并卡合成约 2.4 厘米 x 1 厘米的块来制备小玻璃片。
- 使用 1.4 毫米金刚石涂层钻头钻两个相距 0.8 毫米的孔,一个比另一个略宽(0.043 英寸 = 入口,0.048 英寸 = 出口)。尝试插入一段入口/出口管,使用相同的钻头加宽孔,直到管子适合,以测试孔的大小是否正确。确保管路配合足够紧密,不容易脱落。
注:使用较窄的入口管路以最大限度减少死体积。 - 将双面胶带剪成与玻璃片相同的形状。
- 对齐胶带上的载玻片,然后用针刺穿每个孔,以标记它们在胶带上的位置。使用剃须刀片,切出一个包含两个孔的通道。
注意:不要将通道切割得太长,因为当液体流入时,每个孔远侧的多余空间可能会保持干燥,并可能导致流动过程中出现气泡问题。切割时,尽量在一次干净的切割中切割长边,因为多次切割的磨损边缘会影响流道的质量。同样,请勿切入流道的边缘,因为切入可用部件会影响液体流动,甚至在组装好的流通池中泄漏。 - 用丙酮和纸巾清洁玻璃片直至干燥,然后放在干净的表面上。撕下胶带的一侧并将其粘在玻璃片上,使两个孔都完全位于通道内。
- 使用 p1000 移液器吸头将胶带密封在玻璃片上,以用力但适度的压力按压表面以消除气泡。检查整个表面以密封。
- 从两块玻璃片上撕下胶带的第二个边缘,然后将它们放在干净的表面上,粘性面朝上。将载玻片彼此靠近,但不要接触它们将连接到盖玻片的位置(在它们之间留出足够的空间,以便环氧树脂在每个玻璃片周围形成完全密封)。
- 用塑料镊子拿起半生物素-PEG 盖玻片,仅握住边缘(不要触摸将形成流道的区域),然后将 PEG 功能化的一面降低到粘合剂上。用手指向下按压以将其固定到位,然后用移液器吸头适度用力摩擦表面以去除气泡(不要在流道区域,否则可能会破裂),然后翻转以完成密封。
注意:小心处理盖玻片。 - 对于每个流通池,切割 ~10 cm 的每种聚乙烯管(PE20 和 PE60)。使用较窄的管子作为流通室的入口,以减少死体积。
- 用手将管子插入载玻片上的孔中。如果孔的直径正确,请确保管子在插入后自行在孔中竖立。以一个小角度 <45° 切割管子尖端,以使插入管子稍微容易一些,并防止管子在盖玻片上形成密封,如果末端是平的,可能会这样做(这可能会阻碍流动)。
注:将管路精确安装到玻璃件中,使流通池结构更加可靠,并有助于避免将气泡引入流通池。 - 将环氧树脂成分充分混合,然后使用 p200 尖端轻拍环氧树脂以密封管道并在每块玻璃片周围形成密封。添加足够的环氧树脂,使其上升到盖玻片圆的边缘,因为这也将加强精致的盖玻片。但是,请确保没有环氧树脂进入盖玻片的底部,因为它可能会阻止其平躺在载物台上。
注意: 如有必要,可以使用剃须刀片从底部刮掉少量多余的环氧树脂。 - 放置至少 30-60 分钟,让环氧树脂完全固化。
注:如果流通池不打算立即使用,请在完全固化后将其存放在室温下的真空下。 - 使用流通池后,回收玻璃片并无限期地重复使用它们。拉出管路,将流通池放入含有丙酮的载玻片罐中至少 24 小时,以软化环氧树脂和双面胶粘剂,从而轻松去除玻璃片。在重新用于制作另一个流通池之前,请用海绵和肥皂水擦洗玻璃片,擦干,然后用丙酮和薄纸擦洗。

图 3:流通池制备的图形表示。 (A) 切割双面胶带以匹配玻璃片的尺寸。将载玻片对准胶带顶部,并用针标记每个孔的位置。使用剃须刀片,在每个支架周围切开以创建一个通道。(B) 撕下胶带的一侧,将胶带粘在玻璃片上。确保两个孔都在通道内。撕下胶带的第二端,将生物素-PEG 盖玻片粘在上面。(C) 将聚乙烯管插入每个孔中,并用环氧树脂将管路密封到位,将每个玻璃片也密封到盖玻片上。(D) 使用两个通道后,拉出管路,将流通池放入装满丙酮的染色瓶中。大约 24 小时后,环氧树脂和胶带将软化,流通池的各层可以剥离。玻璃片可以回收并储存在丙酮中,以便无限期地重复使用,以制造下一个流通池。 请单击此处查看此图的较大版本。
4. 单分子 TIRF 检测,用于可视化 CMG 介导的 DNA 解旋
- 准备以下缓冲液。
- 制备封闭缓冲液(20 mM Tris,pH 8.0;50 mM NaCl;2 mM EDTA;0.2 mg/mL BSA)。使用 0.22 μm 注射器过滤器过滤缓冲液并将其储存在 4 °C。
- 制备 10x 反应缓冲液 1(250 mM HEPES pH 7.5;50 mM NaCl;100 mM 乙酸镁)。使用 0.22 μm 注射器过滤器过滤缓冲液并将其储存在 4 °C。
- 制备 10x 反应缓冲液 2(250 mM Tris,pH 7.5;100 mM 乙酸镁;1.25 M 谷氨酸钾;1 mM EDTA;0.025% 吐温-20)。使用 0.22 μm 注射器过滤器过滤缓冲液并将其储存在 4 °C。
- 将打开的试管放入干燥器中并在真空下放置 15 分钟,对大约 2.5 mL 的封闭缓冲液和 5 mL 的超纯水进行脱气。
注意:不要将试管装满到边缘,因为脱气过程中的气泡会导致液体飞溅。 - 将流通池放在显微镜载物台上,并在两端使用粘性腻子将其固定到位。请务必妥善固定,以防止流通池在采集过程中稍后移动。
- 将流通池出口管路连接到注射泵,注射泵的管路连接到注射器上,另一端有针头。将此针头插入出口管路以连接流通池。
- 将物镜加热器打开至 30 °C。
- 将管中的 1-2 mL 脱气水固定在流通池附近的单独胶粘剂腻子上,以便入口管可以插入并到达管底部。
- 让水流过通道,然后使用更快的水流去除进水管附近任何滞留的气泡,必要时添加更多水。否则,大气泡可能会在实验后期脱落并通过通道。静置几分钟,让液流完全停止,因为随着注射器内的压力稳定,快速液流会在停止后导致液体残留拉扯。
注:任何接触气泡的 DNA 都会变得无法使用,即使重新水化后也是如此。 - 将 100 μL 脱气封闭缓冲液添加到 20 μL 等分试样的 1 mg/mL 链霉亲和素中。将开口管连接到粘性腻子上,并将入口管从水中转移到链霉亲和素。以 40 μL/min 的速率流速流速 2 分钟(总共 80 μL)并孵育 5 分钟。
- 用封闭缓冲液洗去过量的链霉亲和素(100 μL 为 50 μL/min)。
- 流入生物素化 DNA,在含 25 nM SYTOX orange 的封闭缓冲液中稀释。使用 532 nm 激光器的实时取景进行成像,实时观察 DNA 拴系到表面。
- 当表面 DNA 达到近似密度时,流入同样含有 25 nM SYTOX(100 μL 为 50 μL/min)的封闭缓冲液中,以洗出游离 DNA。
- 流入在含有 25 nM SYTOX 橙(300 μL 为 100 μL/min)的封闭缓冲液中稀释的生物素化抗地高辛抗体 (~10 μg/mL)。
注:SYTOX 橙嵌入 DNA 的碱基对之间,延长了等值线长度。这意味着 SYTOX orange 中 DNA 的端到端长度增加,使第二个系绳能够从第一个系绳更远地附着在表面,从而在 SYTOX orange 稍后洗掉时获得更好的拉伸 DNA。在每个 DNA 末端使用不同的部分可以使 DNA 更均匀地拴系到盖玻片表面(图 4)。当两端都被生物素化时,它们在同一步骤中系绳,导致它们的附着位置发生显着变化。在一端使用地高辛可以在栓系第二端之前更好地控制 DNA 覆盖。这也允许通过孵育增加 DNA 覆盖率。 - 用封闭缓冲液(100 μL 为 50 μL/min)洗出生物素化的抗地高辛抗体和 SYTOX 橙。
- 配制 120 μL "ATP-g-s 混合物"(1x 反应缓冲液 1、0.75 mg/mL BSA、1.25 mg/mL 酪蛋白、8 mM DTT、0.33 mM ATP-G-S),向新试管中加入 30 μL,然后从原始试管中流出 50 μL(50 μL 为 50 μL/min)。这有助于最大限度地减少缓冲液通过出口管的回流或扩散混合的影响。
- 在 30 μL ATP-g-s 混合物中加入纯化的 CMG 至 ~100 nM(终浓度),然后以 20 μL/min 的速度流入 20 μL。孵育 15 分钟。
- 配制 120 μL "ATP/RPA 混合物"(1x 反应缓冲液 1、0.75 mg/mL BSA、8 mM DTT、3.3 mM ATP、20 nM EGFP-hRPA),并以 40 μL/min 流入流通池,浓度为 80 μL。
- 立即开始采集图像。每 30 秒为每帧获取 6 x 6 个视野(或实验的适当帧速率)。较低的帧速率可以减少激光曝光和荧光团的光漂白。用 488 nm 激光以 1% 的功率可视化 EGFP-hRPA。如果标记了 CMG,例如 LD655,则用 640 nm 激光(最大功率 = 30 mW)以 10% 的功率观察 CMG。使用 532 nm 激光以 0.5% 的功率观察 SYTOX 橙色染色的 DNA。使用每个激光器的曝光时间为 50-100 毫秒。
注:每次将流通池的入口管路从一根液体管转移到另一管液体时,在将其从液体中取出之前,先将管子的尖端轻拍管子底部的管子尖端数次。这有助于防止气泡在传输过程中进入。尽量减少入口管路尖端与液体接触的时间,不要将尖端指向上方或显著抬高尖端,因为这会导致液体流向通道并将空气吸入尖端。转移入口管路时,尽量不要对管路施加张力,因为这会将其从环氧树脂中拉出,将其固定到流通池上。对于 DNA 解旋成像,在 SYTOX orange 存在下以观察双链 DNA,将 10x 反应缓冲液 1 替换为 10x 反应缓冲液 2,用于 ATP/RPA 混合物。在这些情况下,SYTOX orange 与 DNA 的结合效果更好。每次实验结束时,请完全清空注射泵。偶尔加水清洗并排空几次,或拆卸并用肥皂水清洗。

图 4:DNA 拴系到表面。 (A) 当两端用生物素连接 DNA 底物时,两个栓系之间的距离可能会有所不同,具体取决于两端与表面的接触方式 (i)。通过在一端使用地高辛,可以暂时分离每一端的栓系,以获得更一致的栓系距离和更均匀的 DNA 拉伸 (ii)。(B) 显示两端栓系的 DNA(地高辛标记)并用荧光嵌入核酸染色剂染色的 DNA 示例视野。两端拴系的 DNA 显示为一条线,而仅由一端拴系的 DNA 显示为斑点。理想情况下,DNA 应尽可能密集地拴系,不要与其他 DNA 重叠。图像为 512 x 512 像素(像素大小 = 154.6 nm)。 请单击此处查看此图的较大版本。
结果
当 CMG 解旋 DNA 时,特征性的 RPA 束会随着时间的推移而生长(图 5)。解开的 DNA 的 5' 端被拴在表面;因此,它被视为 Tether 和 fork 之间的 RPA 信号的线性延伸。3' 端没有系留,因此随叉移动,并被观察为紧凑的 EGFP-RPA 信号。压实的未绕绕易位链的位置大致对应于复制叉的位置,该复制叉与 LD655-CMG 一起移动,通过 640 nm 激光可视化。
尽量减少对 DNA 底物的损伤很重要,因为单链 DNA 缺口等损伤会减少可观察到的解旋事件的数量,从而限制可以收集的数据量(图 6)。
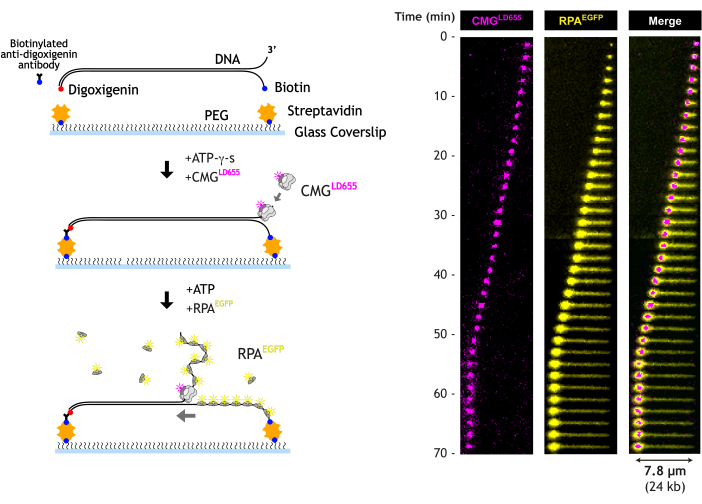
图 5:单分子 DNA 解旋测定。 DNA 底物被拴在盖玻片表面。将用 LD655 标记的纯化 CMG 与 DNA 在 ATP-g-s 中孵育 15 分钟。加入 ATP 和纯化的 EGFP 标记的 RPA,通过 CMG 启动广泛的 DNA 解旋。显示了代表性数据的卡通原理图(左)和运动图(右)。 请单击此处查看此图的较大版本。
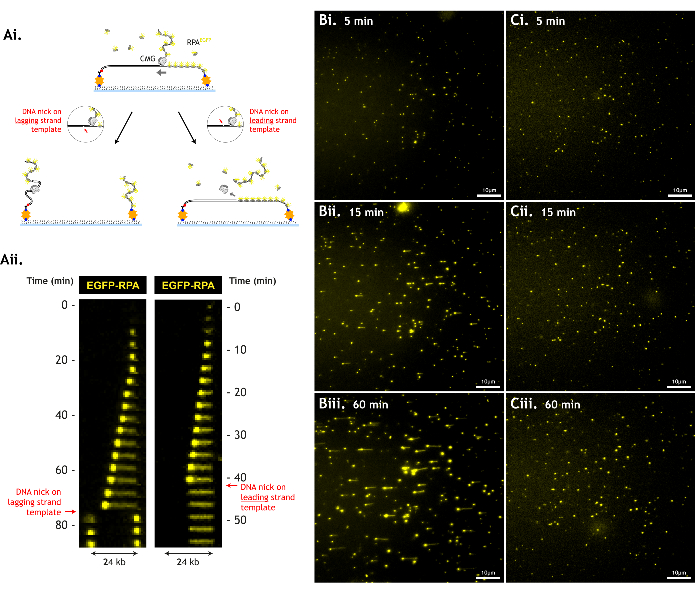
图 6:DNA 损伤会降低检测通量。 (A) CMG 无法在 DNA 骨架断裂(DNA 缺口)之后解旋 DNA。前导链模板上的缺口会导致 CMG 从 DNA 上滑落,并且 CMG 和前导链模板都丢失。滞后链模板上的缺口会导致滞后链模板与其余 DNA 分离,每个 DNA 片段缩回各自的系绳。这可以通过 (i) 卡通示意图和 (ii) 这些事件的运动图 (ii) 来说明。在单个视野中,(B) DNA 底物损伤轻微与 (C) DNA 底物损伤较重的代表性数据,分别是 (i) 5 min、(ii) 15 min 和 (iii) 60 min。受损较多的 DNA 底物不会产生长距离的展开,因为 CMG 更早地遇到缺口,尽管展开活性水平相似(5 分钟时生长的 RPA 斑点密度相似,表明相似的 CMG 加载/展开效率)。视野为 512 x 512 像素(像素大小 = 154.6 nm)。1% 激光功率 (488 nm) 成像 EGFP-RPA。比例尺显示 10 μm。 请单击此处查看此图的较大版本。
讨论
该测定提供了一个平台来观察和研究单个 CMG 的实时动态,既可以单独观察,也可以在所需附加因子的背景下进行。然而,与许多单分子荧光技术一样,有一些常见的挑战可能需要优化才能克服。这些通常与荧光团的长期成像(光漂白、亮度)、DNA 底物制备(DNA 损伤)、流通池表面的质量(背景噪声、非特异性相互作用)或纯化蛋白质制备的质量(核酸酶污染、标记效率)有关。
每个荧光团的光稳定性和亮度都不同,因此选择合适的分子很重要。当对荧光标记的寡聚蛋白(如 RPA)进行成像时,可以使用较低的激光功率,因为许多荧光团会在附近被激发,从而产生可见信号。对于单个荧光团的成像,例如在单个亚基上标记的 CMG,需要更高的激光功率才能清楚地观察荧光团。通过最大限度地减少激光曝光(例如降低图像拍摄频率),可以延长荧光团的使用寿命。此外,激发荧光团会产生活性氧 (ROS),这会导致光漂白。在成像缓冲液中加入除氧系统可以通过消除 ROS 来延长荧光团的使用寿命。但是,一些除氧系统会影响pH 值 12。
关于 DNA 底物制备,尽量减少 DNA 损伤(例如缺口或单链间隙)至关重要。过度损伤会阻止大量 DNA 解旋,从而限制可以收集的数据量。机械剪切、核酸酶污染导致的过度加热或成像过程中产生的 ROS 都可能造成损坏。使用大口径吸头进行移液,小心处理 DNA 样品,缓慢移液,避免轻弹样品,可以最大限度地减少剪切。通过减少激光曝光或在成像缓冲液中加入氧清除系统,可以最大限度地减少 ROS 的影响。制备 DNA 底物后,可以使用市售的 DNA 修复试剂盒在进行解旋反应之前修复损伤。
DNA 解旋的效率还取决于 CMG 的纯度和活性。最好在每个纯化步骤后通过 SDS-PAGE 电泳评估样品纯度,以确定需要优化的位置。如果在最后一步后观察到过多的污染物,则修改用于从 CaptoHiRes Q (5/50) 色谱柱洗脱的盐梯度体积可能会有所帮助。去除用于蛋白质标记的任何多余荧光肽也非常重要,因为它会在盖玻片表面产生不需要的背景。避免核酸酶污染也很重要,因为这会降解 DNA 底物。实验后,用 SYTOX orange 对剩余的 DNA 进行染色是检查 DNA 是否已显著降解的好方法。在实验过程中,一定程度的 DNA 损伤是不可避免的,但严重的损伤通常表明存在核酸酶污染问题。
该检测本身也受到衍射限制点分辨率的限制,需要荧光蛋白距离数百个碱基对(如果不是更多)才能将它们区分为单独的。这限制了可以观察到 CMG 进展和相互作用的细节。
我们为每个分析观察到的展开事件的数量各不相同。对于成功的实验,我们预计每个视场至少会看到几个足够长度的 RPA 区域,即 512x512 像素(像素大小 = 154.6 nm)。可以在同一实验中对多个视野进行成像,从而在必要时收集更多数据。这些束不需要具有相同的长度,也不需要到达 DNA 的末端即可使用。例如,可以通过在添加 CMG 之前测量 SYTOX 染色的 DNA 的长度来确定每个实验的平均系绳距离。这可用于通过将距离从"μm 行进"转换为"kb 展开"来估计任何 RPA 束的解开 DNA 量(只要解开的 DNA 足够多以明显移动叉子)。
CMG 在各种 DNA 底物上都表现出解旋活性,但必须在 polyT 瓣上提供至少 30 nt 的游离 3' DNA 末端,以适应 CMG10 的足迹。在叉子上包含多个生物素部分可确保强大的表面束缚。DNA 底物的其余部分可以通过多种方式重新设计,例如包括不同的 DNA 序列、长度和化学修饰。使用不同浓度的乙酸镁可以改变 DNA 的构象。在较高浓度 (≥10 mM) 的乙酸镁下,RPA 包被的 ssDNA 丝被压实,导致 DNA 在解旋过程中被 RPA 结合拉动。这可能很有用,因为它可以防止 DNA 过度移动,从而可以更准确地测量 CMG 的位置和展开进程。在低浓度 (~3 mM) 的乙酸镁下,RPA-ssDNA 在整个过程中保持松弛。
所描述的单分子检测代表了一个可以构建和修改以研究 DNA 复制的进一步方面的平台。在 DNA 复制过程中,CMG 充当复制体及其组件组装的核心。因此,可以将其他纯化的蛋白质添加到该测定中,包括 TIMELESS、TIPIN 和 CLASPIN 等辅助因子,以研究它们对 CMG 动力学的影响。这些蛋白质已被证明会影响复制分叉的速率13,但尚不清楚它们如何影响 CMG 展开的速率。因此,使用该测定法研究不同的复制酶体蛋白如何影响 CMG 会很有趣。如前所述,添加 DNA 聚合酶可以更好地了解 DNA 复制,而不仅仅是 DNA 解旋,如前面对酵母蛋白14 所述。此外,纯化修饰的 CMG 可能有助于更好地了解某些突变或翻译后修饰如何影响解旋酶活性15,16。此外,设计不同的 DNA 底物可以允许在模拟复制应激的各种条件下研究 CMG 的 DNA 解旋17。这些修饰包括 DNA 障碍 9,18、链间交联 19,20,2 1 和 DNA 链的不连续性22。
披露声明
作者没有相互竞争的经济利益或其他利益冲突。
致谢
我们感谢 Gheorghe Chistol 提供 pGC261 质粒和 Francis Crick 研究所化学生物学设施用于肽合成和标记。这项工作由弗朗西斯克里克研究所资助,该研究所获得了英国癌症研究中心、英国医学研究委员会和惠康信托基金 (CC2133) 的核心资助。
材料
| Name | Company | Catalog Number | Comments |
| 2-Mercaptoethanol | Sigma-Aldrich | M3148-25ML | |
| Acrylamide / Bis Solution 40% | BioRad | 1610148 | |
| Agarose UltraPure | Invitrogen | 16500-500 | Used to prepare Tris Buffer (pH 8) |
| ÄKTA Pure | GE Healthcare | - | Used with Fraction Collector F9-C; protein purification system |
| ANTI-FLAG M2 affinity gel | Sigma-Aldrich | A2220 | |
| APS 10% | Sigma-Aldrich | A3678-25G | |
| ATP (200 mM) | Sigma-Aldrich | A7699-5G | |
| ATP-g-s (100 mM) | Sigma-Aldrich | 11162306001 | |
| Biotin-PEG coverslip | N/A | N/A | Prepared as described: https://doi.org/10.1016/j.ymeth.2012.03.033 |
| Biotinylated anti-digoxigenin antibody | Perkin Elmer | N/A | |
| Blu Tack | Bostik | N/A | Adhesive putty |
| Boric acid | Thermo Fisher Scientific | B/3800/53 | |
| BSA; 33 mg/mL | SIGMA-ALDRICH | A3858-10G | Diluted from the stock |
| CaCl2 | Sigma-Aldrich | C7902 | |
| CaptoHiRes Q (5/50) | Cytiva | 29275878 | Connected to the AKTA protein purification system; high-resolution ion exchange chromatography column |
| Casein (5% in water, 50 mg/mL) | Sigma-Aldrich | C4765-10ML | |
| ChemiDoc system | Bio-Rad | N/A | |
| Clips | N/A | N/A | |
| Cutting board | N/A | N/A | |
| Dialysis tubing (3.5 kDa) | Fisher Scientific | 11425859 | |
| digoxigenin-11-dUTP | Roche | 11209256910 | |
| DNA loading dye 6x | NEB | B7024S | |
| dNTP 10 mM | NEB | N0447S | |
| Double-sided tape (0.14 mm thick) | N/A | N/A | |
| DTT | Fluorochem Limited | M02712-10G | |
| DYKDDDDK peptide | N/A | N/A | Synthetised by chemical biology STP of The Francis Crick Institute |
| EDTA | Thermo Fisher Scientific | D/0700/68 | |
| EGFP-RPA | N/A | N/A | Purified as desribed in ‘Single Molecule Analysis’, Series Methods in Molecular Biology, Vol. 783 (2011), Peterman, E. J. G. and Wuite G. J. L. editors, Humana Press. Protein information: 2 mM, human, expressed in E. coli. |
| EGTA | Sigma-Aldrich | 03779-10G | |
| Epoxy - 5 minute | Devcon | 20845 | |
| Fujifilm FLA-5000 | Fujifilm | FLA-5000 | Fluorescent image analyzer |
| GelRed Nucleic Acid Stain; 10000x | Cambridge Bioscience | 41003-BT | Example of nucleic acid stain which is a safer alternative to ethidium bromide. |
| Glass or quartz slide with holes for tubing | Dremel Model 395 | 1 mm thick slide, cut to 2.4 cm x 1cm, two holes drilled 1.4 mm apart. The holes should be drilled to different sizes to accommodate different tubing at each end. | |
| Glycerol | Fisher Scientific | G/0650/17 | |
| Glycine HCl, pH 3.5 | Sigma-Aldrich | G8898 | |
| Hamilton syringe, 1000 series GASTIGHT, PTFE luer lock 1010TLL, PTFE Luer lock (with slots), volume 10 mL | Merck | 26211-U | |
| HEPES pH 7.5 | Sigma-Aldrich | H4034 | |
| I-CeuI | NEB | R0699L | |
| KCl | Fisher Scientific | P/4280/53 | |
| Magnesium acetate | Sigma-Aldrich | M2545 | |
| Maxi GeBaFlex-tube Dialysis Kit; MWCO 14 kDa | Generon | D055 | |
| Metal spatula | N/A | N/A | |
| Metal tweezers | N/A | N/A | |
| MgCl2 | Sigma-Aldrich | M2393 | |
| Millex-GP (Syringe filters) 0.22 µm | Merck | SLGP033RS | |
| NaCl | Sigma-Aldrich | S7653 | |
| Needle | N/A | N/A | |
| NotI-HF | NEB | R3189L | |
| NuPAGE 4%–12% Bis-Tris Protein Gels | Thermo Fisher Scientific | 10247002; | |
| NuPAGE MOPS SDS Running Buffer | Thermo Fisher Scientific | NP0001 | |
| Objective heater | Okolab | N/A | |
| PCR mix - 2x | Prepared from Phusion DNA polymerase (20 µL), 10 mM dNTPs (40 µL), 5x Phusion HF buffer (400 µL) and water (540 µL). | ||
| Peptide NH2-CHHHHHHHHHHLPETGG-COOH, labelled with LD655-MAL | N/A | N/A | peptide NH2-CHHHHHHHHHHLPETGG-COOH, labeled with LD655-MAL (Lumidyne Technologies) on the cysteine residue, was synthetised and purified by the peptide chemistry STP of The Francis Crick Institute |
| pGC261 plasmid | N/A | N/A | https://doi.org/10.1016/j.cell.2018.10.053 |
| Phusion High-Fidelity DNA Polymerase | NEB | M0530L | |
| Plastic tweezers | N/A | N/A | |
| Polyethylene tubing PE20 (inner diameter 0.015”, outer diameter 0.043”) | Becton Dickinson | 427406 | |
| Polyethylene tubing PE60 (inner diameter 0.03”, outer diameter 0.048”) | Becton Dickinson | 427416 | |
| Poly-Prep Chromatography Columns; 10 ml and 20 ml | Bio-Rad | 7311550 | |
| Potassium Glutamate (L-glutamic acid potassium salt monohydrate) | Sigma | G1501-1KG | |
| Protease inhibitor cocktail (cOmplete, EDTA free) | Roche | 5056489001 | |
| Pump 11 Elite Infusion/Withdrawal Programmable Single Syringe | Harvard Apparatus | 70-4504 | |
| QIAquick PCR Purification Kit | QIAGEN | 28104 | |
| Razor blade | VWR International Ltd | 233-0156 | |
| rCutSmart Buffer; 10x | NEB | B6004S | |
| Sodium acetate | Thermo Fisher Scientific | S/2120/53 | |
| Sortase (pentamutant) | N/A | N/A | Purified based on: https://doi.org/10.1073/pnas.1101046108 |
| Spin-X Centrifuge Tube Filters; 0.22 µm cellulose acetate | Fisher Scientific | 10310361 | |
| Sterile scalpel | N/A | N/A | |
| Steritop Vacuum Driven Disposable Filtration System; 0.22 um; PES; | Millipore | S2GPT05RE | |
| Streptavidin (1 mg/mL) in 1x PBS buffer | Sigma | S4762-10MG | 20 µL aliquotes |
| SYBR Gold | Thermo Fisher Scientific | S11494 | Highly sensitive nucleic acid stain we used to visualise DNA fork substrate. |
| SYTOX orange; 5 µM in DMSO | Thermo Fisher Scientific | S11368 | |
| T4 DNA ligase | NEB | M0202M | |
| T4 DNA Ligase Reaction Buffer | NEB | B0202S | |
| TEMED | Sigma-Aldrich | T9281-25ML | |
| TEV protease (1 mg/mL); EZCut | Biovision | 7847-10000 | |
| Tissue grinders, Dounce type (40 mL, Wheaton) | DWK Life Sciences | 432-1273 | |
| Tris base | Sigma-Aldrich | T1503 | Used to prepare Tris Buffer (pH 8) |
| Tris HCl | Sigma-Aldrich | T3253 | |
| Tween-20 | Promega UK Ltd | H5152 |
参考文献
- Bell, S. P., Labib, K. Chromosome Duplication in Saccharomyces cerevisiae. Genetics. 203 (3), 1027-1067 (2016).
- Costa, A., et al. The structural basis for MCM2-7 helicase activation by GINS and Cdc45. Nat Struct Mol Biol. 18 (4), 471-477 (2011).
- Jones, M. L., Baris, Y., Taylor, M. R. G., Yeeles, J. T. P. Structure of a human replisome shows the organisation and interactions of a DNA replication machine. EMBO J. 40 (23), e108819(2021).
- Moyer, S. E., Lewis, P. W., Botchan, M. R. Isolation of the Cdc45/Mcm2-7/GINS (CMG) complex, a candidate for the eukaryotic DNA replication fork helicase. Proc Natl Acad Sci U S A. 103 (27), 10236-10241 (2006).
- Douglas, M. E., Ali, F. A., Costa, A., Diffley, J. F. X. The mechanism of eukaryotic CMG helicase activation. Nature. 555 (7695), 265-268 (2018).
- Ilves, I., Petojevic, T., Pesavento, J. J., Botchan, M. R. Activation of the MCM2-7 helicase by association with Cdc45 and GINS proteins. Mol Cell. 37 (2), 247-258 (2010).
- Dewar, J. M., Walter, J. C. Mechanisms of DNA replication termination. Nat Rev Mol Cell Biol. 18 (8), 507-516 (2017).
- Burnham, D. R., Kose, H. B., Hoyle, R. B., Yardimci, H. The mechanism of DNA unwinding by the eukaryotic replicative helicase. Nat Commun. 10 (1), 2159(2019).
- Kose, H. B., Larsen, N. B., Duxin, J. P., Yardimci, H. Dynamics of the eukaryotic replicative helicase at lagging-strand protein barriers support the steric exclusion model. Cell Rep. 26 (8), 2113-2125.e6 (2019).
- Kose, H. B., Xie, S., Cameron, G., Strycharska, M. S., Yardimci, H. Duplex DNA engagement and RPA oppositely regulate the DNA-unwinding rate of CMG helicase. Nat Commun. 11 (1), 3713(2020).
- Tanner, N. A., Van Oijen, A. M. Visualizing DNA replication at the single-molecule level. Methods Enzymol. 475, 259-278 (2010).
- Swoboda, M., et al. Enzymatic oxygen scavenging for photostability without ph drop in single-molecule experiments. ACS Nano. 6 (7), 6364-6369 (2012).
- Baris, Y., Taylor, M. R. G., Aria, V., Yeeles, J. T. P. Fast and efficient DNA replication with purified human proteins. Nature. 606, 204-210 (2022).
- Lewis, J. S., et al. Tunability of DNA polymerase stability during eukaryotic DNA replication. Mol Cell. 77 (1), 17-25.e5 (2020).
- Li, Z., Xu, X. Post-translational modifications of the mini-chromosome maintenance proteins in DNA replication. Genes. 10 (5), 331(2019).
- Lewis, J. S., et al. Mechanism of replication origin melting nucleated by CMG helicase assembly. Nature. 606 (7916), 1007-1014 (2022).
- Zeman, M. K., Cimprich, K. A. Causes and consequences of replication stress. Nat Cell Biol. 16 (1), 2-9 (2014).
- Sparks, J. L., et al. The CMG helicase bypasses DNA-protein cross-links to facilitate their repair. Cell. 176 (1-2), 167-181.e21 (2019).
- Taylor, M. R. G., Yeeles, J. T. P. The initial response of a eukaryotic replisome to DNA damage. Mol Cell. 70 (6), 1067-1080.e12 (2018).
- Amunugama, R., et al. Replication fork reversal during DNA interstrand crosslink repair requires CMG unloading. Cell Rep. 23 (12), 3419-3428 (2018).
- Wu, R. A., et al. TRAIP is a master regulator of DNA interstrand crosslink repair. Nature. 567 (7747), 267-272 (2019).
- Vrtis, K. B., Dewar, J. M., Chistol, G., Wu, R. A., Graham, T. G. W., Walter, J. C. Single-strand DNA breaks cause replisome disassembly. Mol Cell. 81 (6), 1309-1318.e6 (2021).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。