Method Article
CMGヘリカーゼによるDNA巻き戻しの1分子リアルタイム可視化
要約
このプロトコルは、CMGヘリカーゼによるDNA巻き戻しのライブ可視化のための単一分子アッセイの実施を示しています。(1)DNA基質の調製、(2)蛍光標識 されたDrosophila melanogaster CMGヘリカーゼの精製、(3)全反射蛍光(TIRF)顕微鏡用のマイクロ流体フローセルの調製、および(4)単一分子DNA巻き戻しアッセイについて説明します。
要約
忠実なゲノム複製は、分裂する細胞の遺伝的安定性を維持するために不可欠です。DNA複製は、S期にレプリソームと呼ばれるタンパク質の動的複合体によって行われます。レプリソームの中心にあるのはCDC45-MCM2-7-GINS(CMG)ヘリカーゼで、DNAポリメラーゼが各鎖をコピーできるように、DNA二重らせんの2本の鎖を分離します。ゲノム重複の間、レプリソームは多くの障害や課題を克服しなければなりません。これらはそれぞれ、DNAを完全かつ正確に複製できないと、突然変異、病気、または細胞死につながる可能性があるため、ゲノムの安定性を脅かします。したがって、CMGが通常の複製と複製ストレスの両方でレプリソームでどのように機能するかを理解することは非常に興味深いことです。ここでは、組換え精製タンパク質を用いた全反射蛍光(TIRF)顕微鏡アッセイについて述べます。これにより、個々のCMG複合体による表面につながれた伸長DNA分子のリアルタイム可視化が可能になります。このアッセイは、CMGの挙動を単一分子レベルで調査するための強力なプラットフォームを提供し、反応条件をリアルタイムで制御しながらヘリカーゼダイナミクスを直接観察することができます。
概要
DNA複製は厳密に制御されており、細胞は突然変異、病気、または死亡を防ぐためにそのゲノムを正確に複製する必要があります。真核生物のDNA複製は、親のDNAを巻き戻し、一本鎖DNA(ssDNA)をテンプレートとして新しいDNAを合成するレプリソーム複合体によって行われます。G1期では、MCM2-7の触媒的に不活性な二六量体が複製起点1の二本鎖DNA(dsDNA)にロードされます。S期では、CDC45とGINS2 が結合することでMCM2-7複合体が活性化され、11サブユニットのCMG複合体(CDC45、MCM2-7、GINS)が形成されます。各CMGは、DNAの巻き戻しを反対方向に開始し、レプリソームが3の周りで自分自身を配置するコアユニットを形成します。
20年前、CMGヘリカーゼは、DNA複製に必須の11サブユニット複合体として初めて同定された4。それ以来、CMGに対する私たちの理解は、ローディングと活性化5,6からDNAの巻き戻しと終了7まで、大幅に進歩しました。従来の生化学的および構造生物学的手法は、これらの発見の多くにとって重要でした。しかし、これらの方法は、CMGのよりダイナミックな側面を研究する能力が限られていることが多かった。単一分子法は、個々の生体分子を物理的に操作して、一度に1分子ずつその活性を測定または視覚化します。これを使用して、他の手法では見逃されたり検出できなかったりすることが多いタンパク質のリアルタイムのダイナミクスに関する洞察を得ることができます8,9。
ここでは、CMGヘリカーゼによるDNAの巻き戻しをリアルタイムで可視化するための全反射蛍光(TIRF)顕微鏡アッセイについて説明します。精製され、蛍光標識されたCMGは、既製のDNAフォーク構造を含む長いDNAの遊離3'末端にロードされます。直鎖状DNAは、DNAの両端を表面に順次テザリングすることにより、マイクロ流体フローセル内のビオチン-PEGカバースリップ上に引き伸ばされます。このアプローチにより、より均一なDNAテザリングが可能になり、データ解析中に考慮しなければならないばらつきが大幅に減少します。ATP-γ-sの存在下では、CMGはフォークの3'末端にある一本鎖DNAにロードされます。ATP-γ-sはゆっくりと加水分解可能なATPアナログであり、CMGがDNAに結合することはできますが、巻き戻すことはできません。その後、ATPを精製した蛍光タグ付きRPAとともに添加すると、CMGが活性化され、広範なDNAの巻き戻しが開始されます。視覚的には、CMGはDNAに沿って転座し、その背後にRPA結合ssDNAの成長管を残します。つながれていないDNA末端はCMGとともに移動し、RPA結合による圧縮により「タイトボール」を形成します。フローセルの設計により、巻き戻し中の任意のポイントでバッファーを交換できるため、各実験中および実験中に優れた制御が可能になります。
このプロトコルは4つの方法に分かれており、それぞれ独立して実行できます。セクション1では、1分子アッセイ用の20 kbの直鎖状フォークDNA基質の調製について説明します。セクション2では、 ショウジョウバエメラノガスターCMG (DmCMG)の精製と蛍光標識について概説します。 DmCMG の発現に関する重要な情報は、ノート セクションに含まれています。セクション3では、TIRF顕微鏡で使用できるマイクロ流体フローセルの調製について説明します。セクション4では、単一分子DNA巻き戻しアッセイの実施方法について説明します。
プロトコル
1. 1. 1分子アッセイに用いる20kb直鎖状フォークDNAの作製(図1)
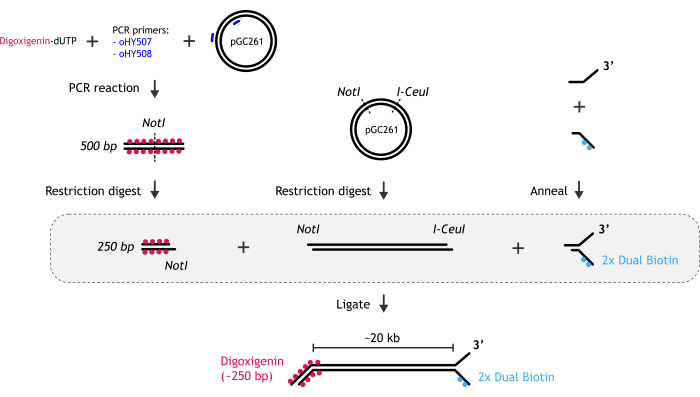
図1:DNA基質調製の図示: (A)ビオチン化DNAフォーク末端は、2つの部分的に相補的なオリゴヌクレオチド(ビオチン化オリゴヌクレオチドと非ビオチン化オリゴヌクレオチド)をアニーリングすることによって作成されます。(B)メインのdsDNA断片(~20 kb)は、pGC261プラスミドの制限酵素消化物と2つの酵素によって生成され、各末端に異なるオーバーハングを持つ直鎖DNAを作成します。(C)ジゴキシゲニン二本鎖DNA末端は、ジゴキシゲニン-dUTPの存在下で行われたPCR反応と、続いて制限消化物によって得られます。 この図の拡大版を表示するには、ここをクリックしてください。
- ビオチン化DNAフォークエンドの生成。
- フォークDNAをアニールします。
- 2つのオリゴヌクレオチド(oHY502およびoHYbio85)を1x STEバッファー(100 mM NaCl、10 mM Tris、pH 8.0、1 mMエチレンジアミン四酢酸[EDTA])に混合します。最終オリゴヌクレオチド濃度は10 μM、最終反応容量は100 μLです。
- 反応液を80°Cで5分間、ヒートブロック上でインキュベートします。ヒートブロックのスイッチを切り、反応を徐々に冷まし、温度が30°Cを下回るまでヒートブロックに残します。
- Tris-Borate-EDTA(TBE)-ポリアクリルアミドゲル電気泳動(PAGE)ゲルのアニーリング効率を検証します。
- 8%アクリルアミド-TBEゲル(4ゲル用:32.8 mLのH2O、4.8 mLの10x TBEバッファー(1 M Tris、1 Mホウ酸、20 mM EDTA)、9.6 mLの40%アクリルアミド/ビス溶液、37.5:1、0.8 mLの10%過硫酸アンモニウム[APS]、40 μLのテトラメチルエチレンジアミン[TEMED])を調製します。
注意:TBE、アクリルアミド、APS、TEMEDは有毒/有害です。 - 少量(1 μL)のアニーリングされたオリゴヌクレオチドと、6倍ローディング色素で希釈した個々のオリゴヌクレオチドを80 Vで80 Vで、室温で1時間分析します。
- シーソーロッキングシェーカーで、適切な核酸染色剤を添加した1x TBEにゲルを30分間浸し、ゲルイメージングシステムを使用してゲルをスキャンします。
- 8%アクリルアミド-TBEゲル(4ゲル用:32.8 mLのH2O、4.8 mLの10x TBEバッファー(1 M Tris、1 Mホウ酸、20 mM EDTA)、9.6 mLの40%アクリルアミド/ビス溶液、37.5:1、0.8 mLの10%過硫酸アンモニウム[APS]、40 μLのテトラメチルエチレンジアミン[TEMED])を調製します。
- アニーリングされたDNAをTBE-PAGEゲル上で分離し、ゲルを切除します。
- オリゴヌクレオチドが適切にアニーリングされている場合は、BSA(~100 μLのアニーリングサンプル、25 μLの6xローディング色素、2 μLの33 mg/mL BSA)の存在下で残りのサンプルを分離します。ゲルに大量の量をロードするには、いくつかのウェルを滅菌メスと組み合わせます。
- 適切な核酸染色剤を添加した1x TBEにゲルをシーソーロッキングシェーカーで30分間放置します。
- ゲルをガラストレイに移し、ブルーライトの下で暗い部屋でDNAを視覚化します。ブルーライトを使用する場合は、適切な保護スクリーンまたは保護メガネを使用してください。
- 滅菌メスで目的のバンドを切り取ります。余分なジェルを切り取ります。
- 焼きなましされたフォークを電気溶出によって精製します。
- ゲルピースを挿入するのに十分な長さの透析チューブをカットし、1x TBEで濡らし、片方の端をクリップで閉じます。
- BSAを1x TBEの500μLで0.3 mg/mLに希釈し、全容量をチューブにピペットで移します。
- 金属ヘラを使用して、ゲルピースを内側にスライドさせます。ゲル片をチューブの片側に移動させ、電気溶出中にDNAが移動するための緩衝スペースを確保します。
- 余分なバッファーを絞り出し、チューブのもう一方の端をクリップします。
注:残りの容量によって、最終的な容量と濃度が決まります。 - 透析チューブを1x TBEバッファーのアガロース電気泳動タンクに入れ、チューブが完全に水没していることを確認します。
注:DNAがゲルから透析バッグに移動できるように、ゲル片が負極に最も近い側に配置されていることを確認してください。 - 電気溶出を80Vで1〜2時間実行します。
- 電気溶出したDNAを10 mM Trisバッファー(pH 8)に透析します。
- 電気溶出後、透析バッグをタンクから取り出し、片方の端をティッシュで乾かします。チューブの中央部分、ゲルとサンプルが位置するティッシュに触れないでください。
- 乾燥させた端からクリップを取り外し、サンプルをチューブ内でゆっくりと上下にピペッティングして、DNAをバッファーに混合します。
- きれいなスパチュラを1x TBEバッファーに浸し、チューブからゲル片をすくい取ります。
注:ゲル片を取り外すときは、できるだけ緩衝液を取り除かないでください。 - 透析バッグの開放端を再びクリップで閉じ、内側の空気を取り除きます。
- 透析バッグを、1.5 Lの10 mM Tris緩衝液(pH 8)、20 mM NaCl、および2 mM MgCl2を充填した2 Lビーカーに入れます。クリップをパラフィルムで包み、チューブをビーカーの端に貼り付け、チューブをバッファーに完全に沈めます。
注:塩は短いオリゴ二本鎖を安定化させます。 - 磁気バーを使用してバッファーを穏やかに攪拌し、室温(RT)で少なくとも3〜4時間、または4°Cで一晩サンプルを透析します。
- 透析後、透析バッグの一方の端をティッシュで乾かし(DNAを希釈しないように)、この端のクリップを外します。
- サンプルをチューブ内で静かに上下に動かし、きれいな1.5 mLチューブに移します。
- 微量分光光度計を使用してDNAの濃度を測定します。
- フォークDNAをアニールします。
- 20 kb フラグメントの生成
- NotI/I-CeuI酵素を用いてpGC261プラスミド18 の制限消化を行います。
- 8 μL の NotI-HF (20,000 U/mL)、8 μL の I-CeuI (5,000 U/mL)、および pGC261 プラスミド (200 μL の反応での最終プラスミド濃度 ~40 ng/μL) を、制限酵素メーカーが推奨するバッファーで穏やかに混合します。
- 反応液を37°Cで一晩インキュベートします。
- 消化したDNAをアガロースゲル上で分離し、ゲルを切除します。
- オプション:まず0.6%アガロースゲルで少量の反応を行い、消化効率をテストしてから、反応全体をロードします。
- ステップ1.2.2.3-1.2.2.4に従って0.6%アガロースゲルを調製します。
- 0.48 gのアガロース粉末を80 mLの1x TBEバッファーと混合します。溶液が沸騰するまで電子レンジで加熱します。アガロースが完全に溶けていることを確認するために渦巻きます。数分間冷ましてから、指定されたタンクに注ぎます。
- 大量のサンプルに対応するためにウェルを組み合わせるには、事前にコームの複数のウェルにテープを貼り付けます。
- ゲルが固まったら、コームを静かに取り外し、タンクに1x TBEバッファーを補充します。サンプルをDNAローディング色素と穏やかに混合し、ゲルを120Vで1時間泳動します。
- ゲルを1x TBEに添加し、適切な核酸染色剤を添加し、シーソーロッキングシェーカーで染色するまで放置します。
- 染色したゲルをガラストレイに移し、ブルーライトの下で暗い部屋でDNAを視覚化します。
- 滅菌メスで目的のバンドを切り取ります。余分なジェルを切り取ります。
- オプション:ステップ1.1.4および1.1.5で説明されているように、電気溶出により目的のフラグメントを精製します。ここでは、透析バッファーからの塩を省略しますが、これは短いオリゴ二本鎖を安定させるためにのみ必要です。
- NotI/I-CeuI酵素を用いてpGC261プラスミド18 の制限消化を行います。
- ジゴキシゲニン二本鎖DNA末端の生成
- pGC261 DNAをテンプレートとして、ジゴキシゲニン-dUTP(dig-dUTP)でPCR反応を行います。
- 混合:400 μLの水、8 μLのpGC261(0.8 ng/μL)、3.5 μLのプライマーoHY507(100 μM)、3.5 μLのプライマーoHY508(100 μM)、8 μLのdig-dUTP(1 mM)、400 μLの2x PCRミックス(DNAポリメラーゼ(20 μL)、10 mM dNTP(40 μL)、5x High-Fidelity(HF)バッファー(400 μL)および水(540 μL)から調製)。
- サーマルサイクラーで以下の反応を実行します。
98 °C - 1 分
30倍: 98 °C - 20 秒;65 °C - 20 秒;72 °C - 30 秒;
72 °C - 10 分
4 °C - ホールド
- ジゴキシゲニン標識PCR産物の消化および精製
- 市販のDNA精製キットを使用してPCR産物を精製します。
- 10 μL NotI-HF (20,000 U/mL) を、酵素供給者が推奨するバッファー (~200 μL 反応での最終 DNA 濃度 ~50 ng/μL) で PCR 産物と混合します。反応液を37°Cで一晩インキュベートします。
- 市販のDNA精製キットを使用して、消化したDNAを精製します。
- pGC261 DNAをテンプレートとして、ジゴキシゲニン-dUTP(dig-dUTP)でPCR反応を行います。
- DNA基質を作るための部品を組み立てます。
- ビオチン化フォークエンド、20 kb DNAフラグメント、およびジゴキシゲニン標識Not-I処理PCRフラグメントを、酵素供給者が推奨するバッファー中の5 μLのT4 DNAリガーゼ(400,000 U/mL)と穏やかに混合し、200 μLの反応を行います。ただし、添加した20 kbのDNAフラグメント(1〜5 μg)に基づいて、ビオチン化フォーク末端とPCRフラグメントの両方を約100倍モル過剰に添加します。
- 反応液をPCRチューブ(各50 μL)に分注し、サーマルサイクラーで16°Cで一晩インキュベートします。
- ライゲーションしたサンプルを0.6%アガロースゲルで分離し、ステップ1.2.2および1.2.3で説明したように電気溶出によりライゲーションしたDNAを精製します。
- 瞬間凍結したDNAは-80°Cで保存してください。
2. Drosophila melanogaster CMGの精製(図2)

図2:4 LのHi Five細胞からのDrosophila melanogasterCMGの精製。 タンパク質は、MOPSバッファーの存在下で200V下で4%-12%Bis-Trisポリアクリルアミドゲルで分離されました。サンプルは、精製の各段階で示されています(細胞溶解物-2μL、FLAG溶出-10μL、第1のイオン交換カラム後-10μL、および標識後および第2のイオン交換カラム-1μL)。(B)MCM3サブユニットの標識効率は、ロングパスレッド(LPR)フィルターを使用した蛍光イメージアナライザーでCy5をスキャンすることにより検証されました。この図の拡大版を表示するには、ここをクリックしてください。
注:蛍光標識 されたショウジョウバエmelanogaster CMGを調製するために、TEV切断部位(ENLYFQG)とそれに続く4つのGly残基をMCM3サブユニット(pFastBac1ベクター)10上のN末端FLAGタグの下流に導入した。複合体を発現するために、バキュロウイルス発現系を用いた。最初のトランスフェクションでは、Sf21細胞を各CMGサブユニット(P1ウイルスステージ)ごとに別々に使用しました。ウイルスをさらに増幅するために、Sf9細胞を使用しました(P2ウイルスステージ)。続いて、Sf9細胞培養物(各CMGサブユニットにつき100mL;0.5×106 細胞/mL)に、10%ウシ胎児血清(P3ウイルスステージ)を添加した0.5mLのP2ウイルスを感染させた。4 L の Hi Five 細胞 (1 x 106 細胞/mL) で CMG 複合体全体を発現するために、各サブユニットに 200 mL の P3 ウイルスを使用しました。CMG複合体を発現するHi Five細胞を回収した後、細胞ペレットを液体窒素で急速凍結し、-80°Cで保存することができます。 氷上または4°Cで全体の精製を行います。 緩衝液は、還元剤(DTTまたは2-メルカプトエタノール)とプロテアーゼ阻害剤(CAUTION)を使用直前に添加することを条件に、事前に調製することができます。すべてのバッファーが事前に予冷され、ろ過され、脱気されていることを確認してください。
- 次のバッファーを準備します。
- 25 mM 4-(2-ヒドロキシエチル)-1-ピペラジンエタンスルホン酸(HEPES)pH 7.5、0.02% Tween-20、10%グリセロール、15 mM KCl、2 mM MgCl2、2 mM 2-メルカプトエタノール、1 mM EDTA、および1 mMエチレングリコール-ビス(β-アミノエチルエーテル)-N、N、N'、N'-四酢酸(EGTA)を混合して、再懸濁バッファーAを調製します。
注意:2-メルカプトエタノール、DTT、およびEDTAは有毒/有害です - ANTI-FLAG M2アフィニティーゲルを調製するためのトリス緩衝生理食塩水(TBS;0.1 M Glycine HCl、pH 3.5)緩衝液を調製します。
- 25 mM HEPES pH 7.5、0.02% Tween-20、10% グリセロール、100 mM KCl、1 mM DTT、1 mM EDTA、および 1 mM EGTA を混合して、バッファー A-100 を調製します。
- 25 mM HEPES pH 7.5、50 mM酢酸ナトリウム、10 mM酢酸マグネシウム、10%グリセロール、および1 mM DTTを添加して、透析バッファーを調製します。
- 50 mM Tris-HCl pH 7.5と150 mM NaClを混合してTBSバッファーを調製します。
- 25 mM 4-(2-ヒドロキシエチル)-1-ピペラジンエタンスルホン酸(HEPES)pH 7.5、0.02% Tween-20、10%グリセロール、15 mM KCl、2 mM MgCl2、2 mM 2-メルカプトエタノール、1 mM EDTA、および1 mMエチレングリコール-ビス(β-アミノエチルエーテル)-N、N、N'、N'-四酢酸(EGTA)を混合して、再懸濁バッファーAを調製します。
- Hi Five細胞の4 L培養液から、プロテアーゼ阻害剤カクテル錠剤を添加した200 mLの冷再懸濁緩衝液Aにペレットを再懸濁します。ペレット全体が完全に再懸濁されるまで、チューブを反転して混合します。
- 再懸濁した細胞を予冷した40 mL Dounceホモジナイザーに移します。できるだけ多くの細胞を回収するには、細胞ペレットの凍結に使用したチューブを同じタイプのバッファーで洗浄し、Dounceホモジナイザーに添加します。ホモゲナイザーの合計容量が推奨制限を超えないようにしてください。細胞を氷上で60〜70ストローク溶解します。
注意: プランジャーがチューブの底に達していることを確認しますが、ホモジナイザーが破損する可能性があるため、圧力をかけすぎないでください。液面より上でプランジャーを取り出さないように注意してください、気泡が発生するので注意してください。 - サンプル全体が均質化されるまで前のステップを繰り返し、予冷したビーカーで混合します。サンプルの最終容量を評価します。一滴ずつ、ろ過したKClを最終濃度100 mMまで加え、穏やかに混合します。
- 事前に遠心分離機を4°Cに予冷してください。サンプルを遠心分離チューブに注ぎ、生産者が推奨する容量に達することを確認します。はかりでチューブのバランスを取ります。
注:容量が小さすぎるチューブは、遠心分離中に破損する可能性があります。 - 溶解した細胞を23,500 x g で4°Cで15〜30分間遠心分離します。 それまでの間、次のステップに沿ってANTI-FLAG M2アフィニティーゲルを準備します。
- ANTI-FLAG M2アフィニティゲル(懸濁液50%)が入ったボトルを穏やかに混合します。P-100ピペットチップの端を切って(チップを広げるため)、それを使用してすぐに4 mLの懸濁液(2 mLのビーズ)を20 mLのポリプレップクロマトグラフィーカラムに移します。チップに付着したビーズを回収するには、チップをTBSで洗浄し、カラムに加えます。すべてのビーズが柱の内側に落ち着くまで待ちますが、乾燥させないように注意してください。
- 3倍ビーズ容量(合計6 mL)の0.1 M Glycine HCl pH 3.5でレジンを洗浄します。その後、ビーズを3〜5ビーズの量のTBS(6〜10 mL)で洗浄します。次に、ビーズをバッファーA-100で3回(6mL)洗浄します。
注:ビーズを0.1 M Glycine HCl pH 3.5溶液に20分以上放置しないでください。流量と洗浄速度を上げるには、カラムを蓋で覆います。レジンを乾燥させないでください。 - レジンを調製した後、レジンの上に2 mLのバッファーA-100を残し、カラムの出口を閉じます。
- 遠心分離後、上清を予冷した50mLチューブに静かに注ぎます。ペレットを乱さないように注意してください。少量のアリコート(~10 μL)を取って、後でSDS-PAGE電気泳動でバリデーションします。
- 同量のANTI-FLAG M2ビーズ(前のステップに沿って調製)を50 mLチューブのそれぞれに加えます。カラムからすべてのビーズを回収するには、バッファーA-100を数ミリリットル追加して再懸濁し、この再懸濁液をチューブに移します。
- サンプル(上清)をANTI-FLAG M2樹脂とインキュベートし、4°Cで2.5時間回転させます。
- インキュベーション後、サンプルを1,000 x g で4°Cで5分間遠心します。 ピペットを使用して、上澄みの大部分を取り除きます。カットチップを使用して、残りの上清の数ミリリットルにビーズを再懸濁し、それらを2本の15mLチューブに移します。50 mLチューブの壁に付着したビーズを回収するには、数ミリリットルのバッファーA-100を追加し、これを15 mLチューブにも移します。
- 15 mLチューブを1,000 x g で4°Cで5分間遠心します。 上清を取り除きます。
- 各チューブに~14 mLのバッファーA-100を加えてビーズを洗浄し、続いて4°Cで短く穏やかに回転させ、その後1000 x g で4°Cで5分間回転させます。 洗濯を2回繰り返します。各洗浄の少量アリコート(~10 μL)を取って、後でSDS-PAGE電気泳動でバリデーションします。
- この手順 (2.16) は、前の手順 (2.13-2.15) の代替手順です。
- ANTI-FLAG M2樹脂でインキュベートした後、サンプルを2つの20 mLポリプレップクロマトグラフィーカラムに注ぎます。
- この方法を選択する場合は、ビーズをカラムの底部に沈殿させ、結合していないサンプルを重力流によってカラムを通過します。その後、バッファーA-100をカラムに直接加え、重力流でサンプルを洗浄します。
- ビーズ(バッファーA-100に再懸濁)を2本の10 mLポリプレップクロマトグラフィーカラムに移します。
- バッファーA-100をレジンに通過させ、バッファーレベルがレジンの上部に達したら出口を閉じ、200 μg/mL(DYKDDDDK)ペプチド(FLAG溶出バッファー)を添加したバッファーA-100を添加してビーズからタンパク質を溶出します。
- 最初の溶出には、3 mLのFLAG溶出バッファーを加え、閉カラムを室温で15分間静かに回転させ、出口を開いて最初の溶出画分を収集します。
- コンセントを閉じ、2 mLのFLAG溶出バッファーを各カラムに加えます。さらに10分間回転させ、2回目の溶出画分を回収します。
- 溶出したすべての画分を混ぜ合わせ(~10 mL)、4°Cに保ちます。 溶出した画分(~10 μL)の少量アリコートを採取し、後でSDS-PAGE電気泳動でバリデーションします。
- 溶出したサンプルを0.22 μmシリンジフィルターでろ過します。
- 事前に、タンパク質精製システムに接続されたCaptoHiRes Q (5/50) カラム(高分解能イオン交換クロマトグラフィーカラム)を、メーカーのプロトコールに沿って調製してください。その後、バッファーA-100でカラムを平衡化し、ろ過したサンプルを平衡化したカラムにロードします。
- バッファー A-100 の 20 カラム容量 (CV) でカラムを洗浄します (カラムの容量が ~1 mL であるため、合計 ~20 mL)。
- タンパク質を溶出するには、あらかじめ2つのバッファーを準備します:バッファーA-100とバッファーBをバッファーA-100と同じ組成で、100 mM KClではなく550 mM KClにします。塩濃度を100 mMから550 mM KClに増やして、20 CV(~20 mL)のグラジエント溶出を設定します。フラクションコレクターで0.3〜0.5 mLの溶出を収集します。
注:CMGは、バッファーBの約70%〜75%の濃度で溶出する必要があります。洗浄液と溶出画分の少量のアリコートを取って、SDS-PAGE電気泳動を行います。 - SDS-PAGE電気泳動を行い、選択した画分にCMGが存在することを確認します。それまでの間、カラムと精製システムはメーカーのプロトコルに従って清掃してください。
- サンプル1 mLあたり50 μLのTEVプロテアーゼ(1 mg/mL)を混合することにより、選択した画分をTEVプロテアーゼと一晩インキュベートします。
- 透析チューブを予湿し、メンブレンが損傷していないことを確認します。CMG/TEVミックスを透析チューブに加え、1.5Lの予冷バッファーA-100と内部の磁気バーを入れた2Lビーカーに入れます。穏やかに攪拌しながら4°Cで一晩透析します。
- サンプルを50 μMのLD655標識ペプチドおよび10 μg/mLのSortase酵素と5 mM CaCl2の存在下で混合します。反応液を4°Cでインキュベートし、30分間回転させ、チューブが光から覆われていることを確認します。SDS-PAGE電気泳動のラベリングの前後に少量のアリコートを服用してください。
- ペプチドが沈殿する可能性があるため、精製システムにロードする前に、0.22 μmの遠心分離フィルターを使用して標識サンプルをろ過します。
- 前述したように、高分解能イオン交換クロマトグラフィーカラムを事前に調製します(ステップ2.20)。今回は、カラムとフラクションコレクターシステムをアルミホイルで覆い、サンプルを光から保護します。
- 遊離ペプチドを除去するには、ろ過したサンプルを高分解能イオン交換クロマトグラフィーカラムにロードします。前に説明したのと同じ方法で精製を実行します(ステップ2.21-2.22)。標識されたCMGが以前と同様の濃度のBuffer-Bで溶出することを確認してください。
- SDS-PAGE電気泳動を実施して、選択したフラクションの品質を検証します。蛍光をイメージングするには、ロードする前にサンプルを沸騰させず、電気泳動タンクが光から保護されていることを確認してください。まずゲルイメージングシステムを使用してタンパク質の蛍光を可視化し、次にCoomassie色素でゲルを染色し、再度イメージングしてすべてのタンパク質を可視化します。
- 選択した画分を4°Cで一晩、1.5 Lの透析バッファーに対して透析します。必要に応じて、サンプルを濃縮します。
- タンパク質を液体窒素で急速凍結し、さらに使用するまで-80°Cで保存します。
3. フローセルの準備(図3)
- ビオチン-PEGカバースリップは、オーブンベーキングステップを省略して、前述のプロトコル11に従って調製します。ビオチン-PEGカバースリップは、RTの真空下で少なくとも1ヶ月間安定です。フローセルを調製するには、ビオチン-PEGカバーガラス(24 mm x 60 mm)を半分(約24 mm x 30 mm)に切断します。
注意: フローチャネルがあるカバースリップの中央領域に触れないように注意してください。 - スライドガラスをエッチングして約2.4cm×1cmの小片にパチンと折って、小さなガラス片を準備します。
- 0.8 mmのダイヤモンドコーティングドリルビットを使用して、1.4 mm間隔で2つの穴を開けます(0.043インチ=入口、0.048インチ=出口)。穴が正しいサイズであることをテストするには、同じドリルビットを使用してチューブが収まるまで穴を広げるために、入口/出口チューブの一部を挿入してみてください。チューブが簡単に脱落しないように十分にしっかりとはまっていることを確認してください。
注意: デッドボリュームを最小限に抑えるために、より狭いインレットチューブが使用されます。 - ガラス片と同じ形の両面テープをカットします。
- スライドをテープに合わせ、各穴に針を刺してテープ上の位置に印を付けます。かみそりの刃を使用して、両方の穴を囲むチャネルを切り取ります。
注意: 液体が流入すると、各穴の向こう側の余分なスペースが乾燥したままになり、流れ中の気泡の問題を引き起こす可能性があるため、チャネルを長くカットしないでください。切断するときは、複数の切断による擦り切れたエッジが流路の品質に影響を与える可能性があるため、長いエッジを1回のきれいなカットで切断してみてください。同様に、使用可能な部品に切り込むと、液体の流れに影響を与えたり、組み立てられたフローセルに漏れたりする可能性があるため、流路の端に切り込みを入れないでください。 - ガラス片をアセトンとティッシュで乾くまできれいにしてから、きれいな面に置きます。粘着テープの片面をはがし、両方の穴がチャネル内に完全に入るようにガラス片に貼り付けます。
- p1000ピペットチップを使用してテープをガラス片に密封し、しっかりと適度な圧力で表面を押して気泡を取り除きます。全面を覆ってシールします。
- テープの2番目の端を両方のガラス片からはがし、粘着面を上にして清潔な面に置きます。スライドを互いに近づけて、カバーガラスに取り付けられる位置に触れないように配置します(エポキシが各ガラス片の周りに完全なシールを作成するのに十分なスペースをそれらの間に残します)。
- プラスチックピンセットでビオチン-PEGハーフカバースリップを持ち上げ、端だけを持って(流路を形成する部分には触れないように)、PEG機能面を接着剤の上に下げます。指で押し下げて固定し、ピペットチップで表面を適度にしっかりとこすって気泡(流路の部分では壊れる恐れがありますので除く)を取り除いてシールを完成させてから、裏返します。
注意: カバースリップの取り扱いには注意してください。 - 各フローセルについて、ポリエチレンチューブ(PE20およびPE60)の各タイプを~10cmカットします。細いチューブをフローチャンバーへの入口として使用して、デッドボリュームを減らします。
- スライドの穴にチューブを手で挿入します。穴の直径が正しい場合は、挿入後にチューブが穴に自立していることを確認してください。チューブの先端をわずかな角度<45°)でカットすると、チューブの挿入が少し容易になり、チューブがカバーガラスに対してシールを形成するのを防ぎます。これは、端が平らな場合に発生する可能性があります(これにより、流れが妨げられる可能性があります)。
注:チューブをガラス片に正確にはめ込むことで、フローセルの構造の信頼性が向上し、フローセルへの気泡の導入を回避できます。 - エポキシ成分をよく混合し、p200チップを使用してエポキシを軽くたたき、チューブをシールし、各ガラス片の周りにシールを作成します。カバーガラスの丸い端まで上がるように十分なエポキシ樹脂を追加すると、繊細なカバーガラスも補強されます。ただし、エポキシ樹脂がカバーガラスの下側に付着しないように注意してください。
注意: 必要に応じて、かみそりの刃を使用して少量の余分なエポキシを下側からこすり落とすことができます。 - エポキシが完全に硬化するまで、少なくとも30〜60分間放置します。
注意: フローセルをすぐに使用しない場合は、完全に硬化した後、RTで真空下で保管してください。 - フローセルを使用した後、ガラス片を回収し、無期限に再利用してください。チューブを引き出し、フローセルをアセトンの入ったスライドジャーに少なくとも24時間入れて、エポキシ樹脂と両面接着剤を軟化させ、ガラス片を簡単に取り除きます。再利用して別のフローセルを作る前に、ガラス片をスポンジと石鹸水でこすってきれいにし、乾かし、アセトンとティッシュペーパーでこすってください。

図3:フローセル調製の図表(A)ガラス片のサイズに合わせて両面テープをカットします。スライドをテープの上に合わせ、各穴の位置を針でマークします。カミソリの刃を使用して、各ホールドの周りをカットしてチャネルを作成します。(B)テープの片面をはがし、テープをガラス片に貼り付けます。両方の穴がチャネルの内側にあることを確認します。テープのもう一方の端をはがし、ビオチン-PEGカバースリップを上に貼り付けます。(C)ポリエチレンチューブを各穴に挿入し、チューブをエポキシで所定の位置に密封し、各ガラス片もカバーガラスに密封します。(D)両方のチャンネルを使用した後、チューブを引き出し、アセトンで満たされた染色ジャーにフローセルを入れます。約24時間後、エポキシとテープが軟化し、フローセルの層を剥がすことができます。ガラス片は回収してアセトンに保存でき、次のフローセルを作るために無期限に再利用できます。この図の拡大版を表示するには、ここをクリックしてください。
4. CMGを介したDNA巻き戻しを可視化するための単一分子TIRFアッセイ
- 次のバッファーを準備します。
- ブロッキングバッファー(20 mM Tris、pH 8.0、50 mM NaCl、2 mM EDTA、0.2 mg/mL BSA)を調製します。0.22 μmシリンジフィルターを使用してバッファーをろ過し、4°Cで保存します。
- 10x Reaction buffer 1(250 mM HEPES pH 7.5、50 mM NaCl、100 mM Magnesium Acetate)を調製します。0.22 μmシリンジフィルターを使用してバッファーをろ過し、4°Cで保存します。
- 10x Reaction buffer 2 (250 mM Tris, pH 7.5; 100 mM Magnesium Acetate; 1.25 M Potassium glutanate; 1 mM EDTA; 0.025% Tween-20) を調製します。0.22 μmシリンジフィルターを使用してバッファーをろ過し、4°Cで保存します。
- 約2.5 mLのブロッキングバッファーと5 mLの超純水を、開いたチューブをデシケーターに入れ、真空下で15分間放置して脱気します。
注意: 脱気中の気泡が液体の飛散を引き起こす可能性があるため、チューブを縁まで充填しないでください。 - フローセルを顕微鏡ステージに置き、両端に粘着パテを使用して所定の位置に固定します。取得中にフローセルが後で移動しないように、しっかりと固定することが重要です。
- フローセル出口チューブをシリンジポンプに接続し、シリンジにチューブが取り付けられ、もう一方の端に針が付いています。この針を出口チューブに挿入して、フローセルを接続します。
- 対物レンズヒーターを30°Cに切り替えます。
- チューブ内の1〜2 mLの脱気水をフローセルの近くにある別の接着剤パテに固定し、入口チューブを挿入してチューブの底に到達できるようにします。
- チャネルに水を流し、次に速い流れを使用してインレットチューブの近くに閉じ込められた気泡を取り除き、必要に応じて水を追加します。そうしないと、大きな気泡が実験の後半で外れてチャネルを通過する可能性があります。速い流れは、シリンジ内の圧力が安定するにつれて、液体が停止した後しばらくの間液体の残留引っ張りを引き起こす可能性があるため、流れが完全に停止するまで数分間放置します。
注:気泡に接触するDNAは、一度水和しても使用できなくなります。 - 100 μLの脱気ブロッキングバッファーを、1 mg/mLストレプトアビジンの20 μLアリコートに加えます。開いたチューブを接着剤パテに取り付け、インレットチューブを水からストレプトアビジンに移します。40 μL/minの速度で2分間(合計80 μL)流し、5分間インキュベートします。
- ブロッキングバッファー(100μLで50μL/min)で過剰なストレプトアビジンを洗い流します。
- ビオチン化DNAを25 nM SYTOXオレンジのブロッキングバッファーで希釈したフロー。532nmレーザーによるライブビューで画像化し、DNAが表面につながっている様子をリアルタイムで観察します。
- 表面上のDNAのおおよその密度が達成されたら、ブロッキングバッファーに25 nM SYTOX(100 μLで50 μL/min)を流し込み、遊離DNAを洗い流します。
- ビオチン化抗ジゴキシゲニン抗体(~10 μg/mL)を25 nM SYTOX orange(100 μL/min、300 μL)を含むブロッキングバッファーで希釈したフロー。
注:SYTOXオレンジはDNAの塩基対の間に挿入され、輪郭の長さを延長します。これは、SYTOXオレンジではDNAのエンドツーエンドの長さが長くなり、2番目のテザーが最初のテザーよりもさらに表面に付着するようになり、後でSYTOXオレンジが洗い流されたときにDNAがより引き伸ばされることを意味します。各DNA末端に異なる部分を使用することで、DNAをカバーガラス表面により均一にテザリングすることができます(図4)。両端がビオチン化されると、同じステップでつながれ、結合位置に大きなばらつきが生じます。一方の端にジゴキシゲニンを使用すると、もう一方の端をテザリングする前にDNAカバレッジをより細かく制御できます。これにより、インキュベーションによってDNAカバレッジを増やすこともできます。 - ビオチン化抗ジゴキシゲニン抗体とSYTOX Orangeをブロッキングバッファー(100 μLで50 μL/分)で洗い流します。
- 120 μLの「ATP-g-sミックス」(1x Reaction buffer 1、0.75 mg/mL BSA、1.25 mg/mL カゼイン、8 mM DTT、0.33 mM ATP-g-s)を調製し、新しいチューブに30 μLを加え、元のチューブから50 μLをフローセルに流します(50 μL/分で50 μL)。これにより、アウトレットチューブからの逆流または拡散によるバッファーの混合の影響を最小限に抑えることができます。
- 精製したCMGを30 μLのATP-g-sミックス中に~100 nM finalに添加し、20 μL/minで20 μLで流し込み、15分間インキュベートします。
- 120 μLの「ATP/RPAミックス」(1x Reaction buffer 1、0.75 mg/mL BSA、8 mM DTT、3.3 mM ATP、20 nM EGFP-hRPA)を調製し、40 μL/minで80 μLのフローセルに流します。
- すぐに画像の取得を開始します。30 秒ごとに各フレームの 6 x 6 の視野を取得します (または実験に適したフレーム レート)。フレームレートを低くすると、レーザーの露光と蛍光色素の光退色を減らすことができます。488 nm レーザーで 1% の出力で EGFP-hRPA を視覚化します。CMGがLD655などとラベル付けされている場合は、640 nmレーザー(最大出力= 30 mW)でCMGを10%の出力で視覚化します。SYTOXオレンジ染色DNAを532 nmレーザーで0.5%の出力で視覚化します。各レーザーは50〜100ミリ秒の露光で使用します。
注意: フローセルの入口チューブが液体のチューブから別のチューブに移されるたびに、チューブの底にチューブの先端を数回軽くたたいてから、液体から取り出します。これにより、移乗中に気泡が入り込むのを防ぐことができます。インレットチューブの先端が液体から出ないようにする時間を最小限に抑え、液体がチャネルに向かって排出され、先端に空気が引き込まれるため、先端を上に向けたり、先端を大幅に上げたりしないでください。インレットチューブを移設する際は、チューブに張力がかからないようにしてください。チューブをエポキシから引き抜いてフローセルに固定するおそれがあります。DNAアンワインディングをイメージングするには、SYTOX orangeの存在下で二本鎖DNAを可視化し、ATP/RPAミックス用の10x Reaction buffer 1を10x Reaction buffer 2に置き換えてください。SYTOX orangeは、これらの条件下でDNAによりよく結合します。各実験の終了時にシリンジポンプを完全に空にします。時々、水を入れて数回空にするか、石鹸水で分解して洗ってください。

図4:表面へのDNAテザリング。 (A)DNA基質の両端をビオチンでテザリングする場合、両端が表面にどのように接触するかによって、2つのテザー間の距離が変わることがあります(i)。一方の端にジゴキシゲニンを使用することで、各端のテザリングを時間的に分離して、より一貫したテザー距離とより均一に引き伸ばされたDNAを実現できます(ii)。(B)DNAが両端につながれ(ジゴキシゲニン標識)、蛍光インターカレート核酸染色で染色されたことを示す視野の例。両端でつながれたDNAは線として現れますが、一方の端だけにつながれたDNAは斑点として現れます。理想的には、DNAは他のDNAと重ならないようにできるだけ密集してつながれるべきです。画像は 512 x 512 ピクセル (ピクセル サイズ = 154.6 nm) です。 この図の拡大版を表示するには、ここをクリックしてください。
結果
CMGがDNAを巻き戻すと、特徴的なRPAトラクトが時間とともに成長します(図5)。巻き戻されたDNAの5'末端は表面につながれています。したがって、テザーとフォークの間のRPA信号の線形ストレッチと見なされます。3'の端はテザリングされていないため、フォークと一緒に移動し、コンパクトなEGFP-RPA信号として観察されます。圧縮された巻き戻された転座ストランドの位置は、レプリケーションフォークの位置とほぼ一致し、レプリケーションフォークは640nmレーザーで可視化されたLD655-CMGと一緒に移動します。
一本鎖DNAニックなどの損傷は、観察可能な巻き戻しイベントの数を減らし、収集できるデータ量を制限するため、DNA基質への損傷を最小限に抑えることが重要です(図6)。
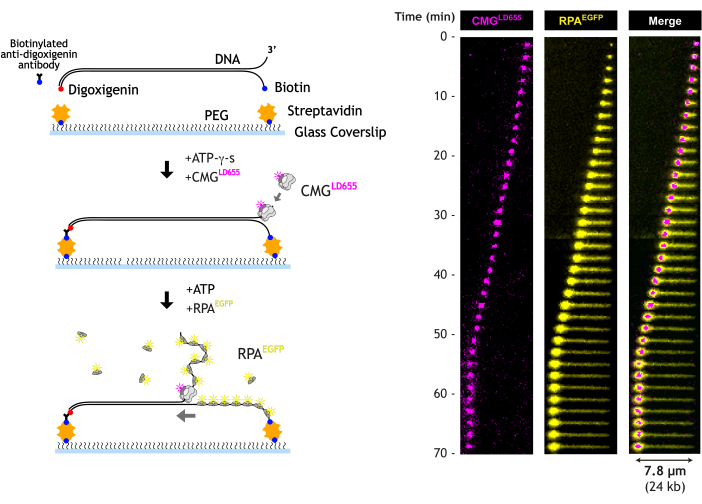
図5:1分子DNA巻き戻しアッセイ。 DNA基質はカバースリップ表面につながれています。LD655で標識した精製CMGをDNAとATP-g-s中で15分間インキュベートします。ATPと精製EGFP標識RPAを添加し、CMGによる広範なDNAアンワインドを開始します。漫画の概略図(左)と代表的なデータのキモグラフ(右)が示されています。 この図の拡大版を表示するには、ここをクリックしてください。
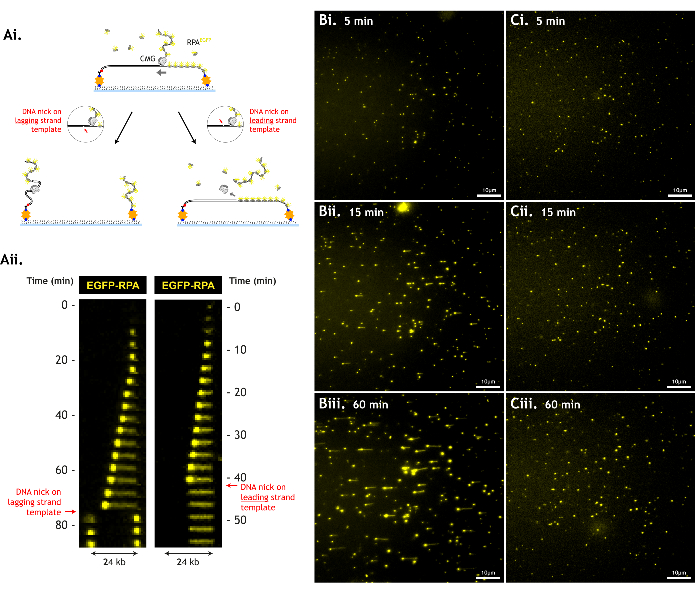
図6:DNA損傷はアッセイのスループットを低下させます。 (A)CMGは、DNAバックボーン(DNAニック)の切断を超えてDNAを巻き戻すことはできません。リーディングストランドテンプレートに傷が付くと、CMGがDNAから滑り落ち、CMGとリーディングストランドテンプレートの両方が失われます。ラギングストランドテンプレートにニックがあると、ラギングストランドテンプレートが他のDNAから分離し、各DNAピースがそれぞれのテザーに引っ込みます。これは、(i)漫画の回路図と(ii)これらのイベントのキモグラフ(ii)によって説明されています。(B)損傷が最小限に抑えられたDNA基質と(C)1つの視野で(i)5分、(ii)15分、(iii)60分で損傷が大きかったDNA基質の代表的なデータ。損傷したDNA基質は、同様のレベルの巻き戻し活動(5分で成長するRPAスポットの密度が同程度で、CMGのローディング/巻き戻し効率が類似していることを示す)にもかかわらず、CMGが早期に傷に遭遇するため、長い巻き戻しの経路を生成しません。視野は 512 x 512 ピクセル (ピクセル サイズ = 154.6 nm) です。レーザー出力1%(488nm)のイメージングEGFP-RPA。10 μmを示すスケールバー。 この図の拡大版を表示するには、ここをクリックしてください。
ディスカッション
このアッセイは、個々のCMGのリアルタイムダイナミクスを、単離および必要な追加因子のコンテキストの両方で観察および調査するためのプラットフォームを提供します。ただし、多くの単一分子蛍光技術と同様に、克服するために最適化が必要な一般的な課題がいくつかあります。これらは通常、長期間にわたる蛍光色素のイメージング(光退色、輝度)、DNA基質調製(DNA損傷)、フローセル表面の品質(バックグラウンドノイズ、非特異的相互作用)、または精製タンパク質調製物の品質(ヌクレアーゼ汚染、標識効率)に関連しています。
各蛍光色素は光安定性と輝度が異なるため、適切な分子を選択することが重要です。RPAのように蛍光標識されたオリゴマータンパク質をイメージングする場合、多くの蛍光色素が近接して励起され、可視信号を生成するため、より低いレーザー出力を使用できます。単一の蛍光色素をイメージングする場合、例えば、単一のサブユニットに標識されたCMGでは、蛍光色素をはっきりと観察するためには、より高いレーザー出力が必要です。蛍光色素の寿命は、画像が撮影される頻度を減らすなど、レーザーの曝露を最小限に抑えることで延長できます。さらに、蛍光色素を励起すると活性酸素種(ROS)が生成され、光退色の原因となる可能性があります。イメージングバッファーに酸素捕捉システムを含めると、ROSを除去して蛍光色素の寿命を延ばすことができます。ただし、一部の酸素捕捉システムはpH12に影響を与える可能性があります。
DNA基質の調製に関しては、傷や一本鎖のギャップなどのDNA損傷を最小限に抑えることが重要です。過度の損傷は、広範なDNAの巻き戻しを妨げ、収集できるデータの量を制限します。損傷は、機械的なせん断、ヌクレアーゼ汚染の結果としての過度の加熱、またはイメージング中に生成されるROSによって発生する可能性があります。DNAサンプルの取り扱いには、ピペッティング用の幅の広いチップを使用し、ゆっくりとピペッティングし、サンプルをフリックしないようにすることで、せん断を最小限に抑えることができます。ROSの影響は、レーザー曝露を減らすか、イメージングバッファーに酸素捕捉システムを含めることで最小限に抑えることができます。DNA基質の調製後、市販のDNA修復キットを使用して損傷を修復してから、巻き戻し反応を行うことが可能です。
DNA巻き戻しの効率は、CMGの純度と活性にも依存します。各精製ステップの後にSDS-PAGE電気泳動でサンプルの純度を評価し、最適化が必要な場所を特定することをお勧めします。最終ステップ後に観察される汚染物質が多すぎる場合は、CaptoHiRes Q (5/50) カラムからの溶出に使用される塩グラジエント量を変更することが役立つ場合があります。また、タンパク質の標識に使用される過剰な蛍光ペプチドを除去することも非常に重要です。これは、カバーガラスの表面に望ましくない背景を作り出す可能性があるためです。また、DNA基質を分解する可能性があるため、ヌクレアーゼ汚染を避けることも不可欠です。実験後、残りのDNAをSYTOXオレンジで染色することは、DNAが大幅に分解されているかどうかを確認する良い方法です。実験の過程である程度のDNA損傷は避けられませんが、重大な損傷はしばしば問題のあるヌクレアーゼ汚染を示しています。
また、このアッセイは、回折限界のあるスポットの分解能によって本質的に制限されるため、蛍光タンパク質を分離として区別するためには、蛍光タンパク質が数百塩基対(それ以上ではないにしても)離れている必要があります。これにより、CMG の進行と相互作用を観察できる詳細が制限されます。
各解析で観測されるアンワインド イベントの数は異なります。実験を成功させるには、視野あたり 512x512 ピクセル (ピクセル サイズ = 154.6 nm) の十分な長さの RPA トラクトが少なくとも 2 つ見られることが予想されます。同じ実験で複数の視野を画像化できるため、必要に応じてより多くのデータを収集できます。トラクトは、有用であるために同じ長さである必要はなく、DNAの末端に達する必要もありません。例えば、各実験の平均テザー距離は、CMGを添加する前にSYTOX染色DNAの長さを測定することで決定できます。これを使用して、距離を「μm移動」から「kb巻き戻し」に変換することにより、任意のRPAトラクトで巻き戻されたDNAの量を推定できます(フォークを目に見える形で動かすのに十分なDNAが巻き戻されている限り)。
CMGは、さまざまなDNA基質上で巻き戻し活性を示しますが、CMG10のフットプリントに対応するためには、少なくとも30 ntのpolyTフラップ上に遊離3' DNA末端を提供することが不可欠です。フォークに複数のビオチン部分を含めることで、堅牢な表面テザリングが保証されます。残りのDNA基質は、さまざまなDNA配列、長さ、化学修飾を含めるなど、さまざまな方法で再設計できます。DNAのコンフォメーションは、異なる濃度の酢酸マグネシウムを使用することで変更することができます。高濃度(≥10 mM)の酢酸マグネシウムでは、RPAコーティングされたssDNAフィラメントが圧縮され、巻き戻し中にRPA結合によってDNAが引き出されます。これは、DNAが過度に移動するのを防ぎ、CMGの位置と巻き戻し進行の位置をより正確に測定できるため、有用です。低濃度(~3 mM)の酢酸マグネシウムでは、RPA-ssDNAは全体を通してリラックスしたままです。
記載されている1分子アッセイは、DNA複製のさらなる側面を調査するために構築および変更できるプラットフォームを表しています。DNA複製中、CMGはレプリソームとその構成要素が組み立てるコアとして機能します。したがって、TIMELESS、TIPIN、CLASPINなどのアクセサリーファクターを含む精製タンパク質をこのアッセイに追加して、CMGダイナミクスへの影響を研究することができます。これらのタンパク質は、複製フォーク13の速度に影響を与えることが示されているが、それらがCMGの巻き戻し速度にどのように影響するかは明らかではない。したがって、このアッセイを使用して、さまざまなレプリソームタンパク質がCMGにどのように影響するかを調査することは興味深いでしょう。DNAポリメラーゼの添加は、酵母タンパク質14で前に説明したように、DNAの巻き戻しだけでは行わなかったDNA複製についてのより良い洞察を得るかもしれない。さらに、修飾されたCMGの精製は、特定の突然変異または翻訳後修飾がヘリカーゼ活性にどのように影響するかについてのより良い理解を提供するかもしれない15,16。さらに、異なるDNA基質を設計することにより、CMGによるDNA巻き戻しを、複製ストレス17を模倣する様々な条件下で研究することができる。これらの修飾は、DNA障害物9、18、鎖間架橋19、20、21、およびDNA鎖22の不連続性を含む。
開示事項
著者は、競合する金銭的利益またはその他の利益相反を持っていません。
謝辞
pGC261プラスミドを提供してくださったGheorghe Chistol氏と、ペプチド合成と標識のためのFrancis Crick Institute Chemical Biology Facilityに感謝します。この研究は、Cancer Research UK、UK Medical Research Council、およびThe Wellcome Trust(CC2133)から主要な資金提供を受けているFrancis Crick Instituteによって資金提供されました。
資料
| Name | Company | Catalog Number | Comments |
| 2-Mercaptoethanol | Sigma-Aldrich | M3148-25ML | |
| Acrylamide / Bis Solution 40% | BioRad | 1610148 | |
| Agarose UltraPure | Invitrogen | 16500-500 | Used to prepare Tris Buffer (pH 8) |
| ÄKTA Pure | GE Healthcare | - | Used with Fraction Collector F9-C; protein purification system |
| ANTI-FLAG M2 affinity gel | Sigma-Aldrich | A2220 | |
| APS 10% | Sigma-Aldrich | A3678-25G | |
| ATP (200 mM) | Sigma-Aldrich | A7699-5G | |
| ATP-g-s (100 mM) | Sigma-Aldrich | 11162306001 | |
| Biotin-PEG coverslip | N/A | N/A | Prepared as described: https://doi.org/10.1016/j.ymeth.2012.03.033 |
| Biotinylated anti-digoxigenin antibody | Perkin Elmer | N/A | |
| Blu Tack | Bostik | N/A | Adhesive putty |
| Boric acid | Thermo Fisher Scientific | B/3800/53 | |
| BSA; 33 mg/mL | SIGMA-ALDRICH | A3858-10G | Diluted from the stock |
| CaCl2 | Sigma-Aldrich | C7902 | |
| CaptoHiRes Q (5/50) | Cytiva | 29275878 | Connected to the AKTA protein purification system; high-resolution ion exchange chromatography column |
| Casein (5% in water, 50 mg/mL) | Sigma-Aldrich | C4765-10ML | |
| ChemiDoc system | Bio-Rad | N/A | |
| Clips | N/A | N/A | |
| Cutting board | N/A | N/A | |
| Dialysis tubing (3.5 kDa) | Fisher Scientific | 11425859 | |
| digoxigenin-11-dUTP | Roche | 11209256910 | |
| DNA loading dye 6x | NEB | B7024S | |
| dNTP 10 mM | NEB | N0447S | |
| Double-sided tape (0.14 mm thick) | N/A | N/A | |
| DTT | Fluorochem Limited | M02712-10G | |
| DYKDDDDK peptide | N/A | N/A | Synthetised by chemical biology STP of The Francis Crick Institute |
| EDTA | Thermo Fisher Scientific | D/0700/68 | |
| EGFP-RPA | N/A | N/A | Purified as desribed in ‘Single Molecule Analysis’, Series Methods in Molecular Biology, Vol. 783 (2011), Peterman, E. J. G. and Wuite G. J. L. editors, Humana Press. Protein information: 2 mM, human, expressed in E. coli. |
| EGTA | Sigma-Aldrich | 03779-10G | |
| Epoxy - 5 minute | Devcon | 20845 | |
| Fujifilm FLA-5000 | Fujifilm | FLA-5000 | Fluorescent image analyzer |
| GelRed Nucleic Acid Stain; 10000x | Cambridge Bioscience | 41003-BT | Example of nucleic acid stain which is a safer alternative to ethidium bromide. |
| Glass or quartz slide with holes for tubing | Dremel Model 395 | 1 mm thick slide, cut to 2.4 cm x 1cm, two holes drilled 1.4 mm apart. The holes should be drilled to different sizes to accommodate different tubing at each end. | |
| Glycerol | Fisher Scientific | G/0650/17 | |
| Glycine HCl, pH 3.5 | Sigma-Aldrich | G8898 | |
| Hamilton syringe, 1000 series GASTIGHT, PTFE luer lock 1010TLL, PTFE Luer lock (with slots), volume 10 mL | Merck | 26211-U | |
| HEPES pH 7.5 | Sigma-Aldrich | H4034 | |
| I-CeuI | NEB | R0699L | |
| KCl | Fisher Scientific | P/4280/53 | |
| Magnesium acetate | Sigma-Aldrich | M2545 | |
| Maxi GeBaFlex-tube Dialysis Kit; MWCO 14 kDa | Generon | D055 | |
| Metal spatula | N/A | N/A | |
| Metal tweezers | N/A | N/A | |
| MgCl2 | Sigma-Aldrich | M2393 | |
| Millex-GP (Syringe filters) 0.22 µm | Merck | SLGP033RS | |
| NaCl | Sigma-Aldrich | S7653 | |
| Needle | N/A | N/A | |
| NotI-HF | NEB | R3189L | |
| NuPAGE 4%–12% Bis-Tris Protein Gels | Thermo Fisher Scientific | 10247002; | |
| NuPAGE MOPS SDS Running Buffer | Thermo Fisher Scientific | NP0001 | |
| Objective heater | Okolab | N/A | |
| PCR mix - 2x | Prepared from Phusion DNA polymerase (20 µL), 10 mM dNTPs (40 µL), 5x Phusion HF buffer (400 µL) and water (540 µL). | ||
| Peptide NH2-CHHHHHHHHHHLPETGG-COOH, labelled with LD655-MAL | N/A | N/A | peptide NH2-CHHHHHHHHHHLPETGG-COOH, labeled with LD655-MAL (Lumidyne Technologies) on the cysteine residue, was synthetised and purified by the peptide chemistry STP of The Francis Crick Institute |
| pGC261 plasmid | N/A | N/A | https://doi.org/10.1016/j.cell.2018.10.053 |
| Phusion High-Fidelity DNA Polymerase | NEB | M0530L | |
| Plastic tweezers | N/A | N/A | |
| Polyethylene tubing PE20 (inner diameter 0.015”, outer diameter 0.043”) | Becton Dickinson | 427406 | |
| Polyethylene tubing PE60 (inner diameter 0.03”, outer diameter 0.048”) | Becton Dickinson | 427416 | |
| Poly-Prep Chromatography Columns; 10 ml and 20 ml | Bio-Rad | 7311550 | |
| Potassium Glutamate (L-glutamic acid potassium salt monohydrate) | Sigma | G1501-1KG | |
| Protease inhibitor cocktail (cOmplete, EDTA free) | Roche | 5056489001 | |
| Pump 11 Elite Infusion/Withdrawal Programmable Single Syringe | Harvard Apparatus | 70-4504 | |
| QIAquick PCR Purification Kit | QIAGEN | 28104 | |
| Razor blade | VWR International Ltd | 233-0156 | |
| rCutSmart Buffer; 10x | NEB | B6004S | |
| Sodium acetate | Thermo Fisher Scientific | S/2120/53 | |
| Sortase (pentamutant) | N/A | N/A | Purified based on: https://doi.org/10.1073/pnas.1101046108 |
| Spin-X Centrifuge Tube Filters; 0.22 µm cellulose acetate | Fisher Scientific | 10310361 | |
| Sterile scalpel | N/A | N/A | |
| Steritop Vacuum Driven Disposable Filtration System; 0.22 um; PES; | Millipore | S2GPT05RE | |
| Streptavidin (1 mg/mL) in 1x PBS buffer | Sigma | S4762-10MG | 20 µL aliquotes |
| SYBR Gold | Thermo Fisher Scientific | S11494 | Highly sensitive nucleic acid stain we used to visualise DNA fork substrate. |
| SYTOX orange; 5 µM in DMSO | Thermo Fisher Scientific | S11368 | |
| T4 DNA ligase | NEB | M0202M | |
| T4 DNA Ligase Reaction Buffer | NEB | B0202S | |
| TEMED | Sigma-Aldrich | T9281-25ML | |
| TEV protease (1 mg/mL); EZCut | Biovision | 7847-10000 | |
| Tissue grinders, Dounce type (40 mL, Wheaton) | DWK Life Sciences | 432-1273 | |
| Tris base | Sigma-Aldrich | T1503 | Used to prepare Tris Buffer (pH 8) |
| Tris HCl | Sigma-Aldrich | T3253 | |
| Tween-20 | Promega UK Ltd | H5152 |
参考文献
- Bell, S. P., Labib, K. Chromosome Duplication in Saccharomyces cerevisiae. Genetics. 203 (3), 1027-1067 (2016).
- Costa, A., et al. The structural basis for MCM2-7 helicase activation by GINS and Cdc45. Nat Struct Mol Biol. 18 (4), 471-477 (2011).
- Jones, M. L., Baris, Y., Taylor, M. R. G., Yeeles, J. T. P. Structure of a human replisome shows the organisation and interactions of a DNA replication machine. EMBO J. 40 (23), e108819(2021).
- Moyer, S. E., Lewis, P. W., Botchan, M. R. Isolation of the Cdc45/Mcm2-7/GINS (CMG) complex, a candidate for the eukaryotic DNA replication fork helicase. Proc Natl Acad Sci U S A. 103 (27), 10236-10241 (2006).
- Douglas, M. E., Ali, F. A., Costa, A., Diffley, J. F. X. The mechanism of eukaryotic CMG helicase activation. Nature. 555 (7695), 265-268 (2018).
- Ilves, I., Petojevic, T., Pesavento, J. J., Botchan, M. R. Activation of the MCM2-7 helicase by association with Cdc45 and GINS proteins. Mol Cell. 37 (2), 247-258 (2010).
- Dewar, J. M., Walter, J. C. Mechanisms of DNA replication termination. Nat Rev Mol Cell Biol. 18 (8), 507-516 (2017).
- Burnham, D. R., Kose, H. B., Hoyle, R. B., Yardimci, H. The mechanism of DNA unwinding by the eukaryotic replicative helicase. Nat Commun. 10 (1), 2159(2019).
- Kose, H. B., Larsen, N. B., Duxin, J. P., Yardimci, H. Dynamics of the eukaryotic replicative helicase at lagging-strand protein barriers support the steric exclusion model. Cell Rep. 26 (8), 2113-2125.e6 (2019).
- Kose, H. B., Xie, S., Cameron, G., Strycharska, M. S., Yardimci, H. Duplex DNA engagement and RPA oppositely regulate the DNA-unwinding rate of CMG helicase. Nat Commun. 11 (1), 3713(2020).
- Tanner, N. A., Van Oijen, A. M. Visualizing DNA replication at the single-molecule level. Methods Enzymol. 475, 259-278 (2010).
- Swoboda, M., et al. Enzymatic oxygen scavenging for photostability without ph drop in single-molecule experiments. ACS Nano. 6 (7), 6364-6369 (2012).
- Baris, Y., Taylor, M. R. G., Aria, V., Yeeles, J. T. P. Fast and efficient DNA replication with purified human proteins. Nature. 606, 204-210 (2022).
- Lewis, J. S., et al. Tunability of DNA polymerase stability during eukaryotic DNA replication. Mol Cell. 77 (1), 17-25.e5 (2020).
- Li, Z., Xu, X. Post-translational modifications of the mini-chromosome maintenance proteins in DNA replication. Genes. 10 (5), 331(2019).
- Lewis, J. S., et al. Mechanism of replication origin melting nucleated by CMG helicase assembly. Nature. 606 (7916), 1007-1014 (2022).
- Zeman, M. K., Cimprich, K. A. Causes and consequences of replication stress. Nat Cell Biol. 16 (1), 2-9 (2014).
- Sparks, J. L., et al. The CMG helicase bypasses DNA-protein cross-links to facilitate their repair. Cell. 176 (1-2), 167-181.e21 (2019).
- Taylor, M. R. G., Yeeles, J. T. P. The initial response of a eukaryotic replisome to DNA damage. Mol Cell. 70 (6), 1067-1080.e12 (2018).
- Amunugama, R., et al. Replication fork reversal during DNA interstrand crosslink repair requires CMG unloading. Cell Rep. 23 (12), 3419-3428 (2018).
- Wu, R. A., et al. TRAIP is a master regulator of DNA interstrand crosslink repair. Nature. 567 (7747), 267-272 (2019).
- Vrtis, K. B., Dewar, J. M., Chistol, G., Wu, R. A., Graham, T. G. W., Walter, J. C. Single-strand DNA breaks cause replisome disassembly. Mol Cell. 81 (6), 1309-1318.e6 (2021).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved