Method Article
Visualização em tempo real de molécula única do desenrolamento do DNA por CMG helicase
Neste Artigo
Resumo
Este protocolo demonstra a realização de um ensaio de molécula única para visualização ao vivo do desenrolamento do DNA por CMG helicase. Ele descreve (1) a preparação de um substrato de DNA, (2) a purificação de Drosophila melanogaster CMG helicase marcada com fluorescência, (3) a preparação de uma célula de fluxo microfluídico para microscopia de fluorescência de reflexão interna total (TIRF) e (4) o ensaio de desenrolamento de DNA de molécula única.
Resumo
A duplicação fiel do genoma é essencial para preservar a estabilidade genética das células em divisão. A replicação do DNA é realizada durante a fase S por um complexo dinâmico de proteínas denominado replissoma. No coração do replissoma está a helicase CDC45-MCM2-7-GINS (CMG), que separa as duas fitas da dupla hélice do DNA de modo que as DNA polimerases possam copiar cada fita. Durante a duplicação do genoma, os replossomos devem superar uma infinidade de obstáculos e desafios. Cada um deles ameaça a estabilidade do genoma, pois a falha em replicar o DNA de forma completa e precisa pode levar a mutações, doenças ou morte celular. Portanto, é de grande interesse entender como o CMG funciona no replissoma durante a replicação normal e o estresse de replicação. Aqui, descrevemos um ensaio de microscopia de fluorescência de reflexão interna total (TIRF) usando proteínas purificadas recombinantes, que permite a visualização em tempo real de moléculas de DNA esticadas amarradas à superfície por complexos CMG individuais. Este ensaio fornece uma plataforma poderosa para investigar o comportamento do CMG no nível de molécula única, permitindo que a dinâmica da helicase seja observada diretamente com controle em tempo real sobre as condições de reação.
Introdução
A replicação do DNA é rigidamente regulada, pois uma célula deve duplicar seu genoma com precisão para evitar mutações, doenças ou morte. A replicação do DNA eucariótico é realizada pelo complexo replissoma, que desenrola o DNA parental e usa o DNA de fita simples (ssDNA) como modelo para sintetizar novo DNA. Na fase G1, hexâmeros duplos cataliticamente inativos de MCM2-7 são carregados no DNA de fita dupla (dsDNA) nas origens de replicação1. Na fase S, os complexos MCM2-7 são ativados pela ligação de CDC45 e GINS2 para formar complexos CMG de 11 subunidades (CDC45, MCM2-7, GINS). Cada CMG inicia o desenrolamento do DNA em direções opostas, formando a unidade central que o replissoma se organiza em torno de3.
Duas décadas atrás, a helicase CMG foi identificada pela primeira vez como um complexo de 11 subunidades, essencial para a replicação do DNA4. Desde então, nossa compreensão do CMG avançou consideravelmente, desde o carregamento e ativação 5,6, até o desenrolamento e terminação do DNA7. As técnicas tradicionais de biologia bioquímica e estrutural têm sido críticas para muitas dessas descobertas; no entanto, esses métodos eram frequentemente limitados em sua capacidade de estudar os aspectos mais dinâmicos do CMG. Os métodos de molécula única usam a manipulação física de biomoléculas individuais para medir ou visualizar sua atividade, uma molécula de cada vez. Isso pode ser usado para fornecer informações sobre a dinâmica em tempo real de proteínas que muitas vezes são perdidas ou indetectáveis por outras técnicas 8,9.
Aqui, descrevemos um ensaio de microscopia de fluorescência de reflexão interna total (TIRF) para visualizar o desenrolamento do DNA por CMG helicase em tempo real. O CMG purificado e marcado com fluorescência é carregado na extremidade livre de 3 'de DNA longo contendo uma estrutura de garfo de DNA pré-fabricada. O DNA linear é esticado em uma lamínula de biotina-PEG em uma célula de fluxo microfluídico, amarrando cada extremidade do DNA sequencialmente à superfície. Essa abordagem permite uma amarração de DNA mais uniforme, o que reduz significativamente a variação que deve ser contabilizada durante a análise de dados. Na presença de ATP-γ-s, o CMG é carregado no DNA de fita simples na extremidade 3' do garfo. O ATP-γ-s é um análogo de ATP lentamente hidrolisável, que permite a ligação do CMG ao DNA, mas não o desenrolamento. A adição subsequente de ATP, juntamente com RPA purificado e marcado com fluorescência, ativa o CMG e inicia o extenso desenrolamento do DNA. Visualmente, o CMG se transloca ao longo do DNA, deixando um trato crescente de ssDNA ligado a RPA atrás dele. A extremidade de DNA não amarrada viaja com o CMG, formando uma "bola apertada" devido à compactação causada pela ligação do RPA. O design da célula de fluxo permite que o buffer seja trocado a qualquer momento durante o desenrolamento, proporcionando grande controle durante e sobre cada experimento.
Este protocolo é dividido em quatro métodos, que podem ser realizados independentemente um do outro. A seção 1 descreve a preparação de um substrato de DNA bifurcado linear de 20 kb para ensaios de molécula única. A Seção 2 descreve a purificação e a marcação fluorescente de Drosophila melanogaster CMG (DmCMG). As principais informações sobre a expressão de DmCMG estão incluídas na seção de notas. A seção 3 cobre a preparação de uma célula de fluxo microfluídico que pode ser usada em um microscópio TIRF. A seção 4 descreve como realizar o ensaio de desenrolamento de DNA de molécula única.
Protocolo
1. Preparação de DNA bifurcado linear de 20 kb usado em ensaios de molécula única (Figura 1)
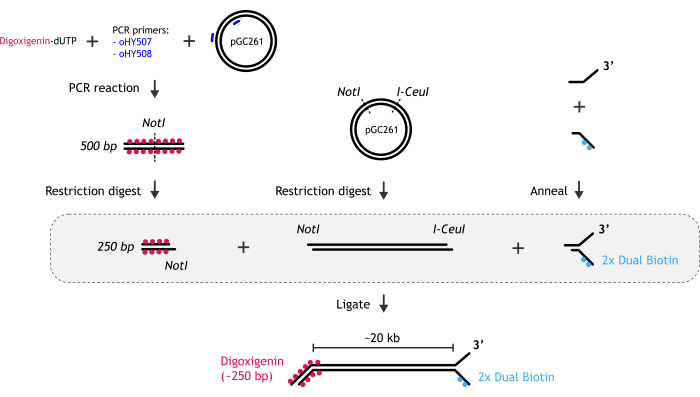
Figura 1: Representação gráfica da preparação do substrato de DNA. (A) A extremidade do garfo de DNA biotinilado é criada pelo recozimento de dois oligonucleotídeos parcialmente complementares: biotinilado e não biotinilado. (B) O fragmento principal de dsDNA (~ 20 kb) é gerado por digestão de restrição do plasmídeo pGC261 com duas enzimas para criar um DNA linear com diferentes saliências em cada extremidade. (C) A extremidade do DNA duplex da digoxigenina é obtida por uma reação de PCR realizada na presença de digoxigenina-dUTP, seguida de digestão de restrição. Clique aqui para ver uma versão maior desta figura.
- Geração de extremidade de garfo de DNA biotinilado.
- Recozer o DNA do garfo.
- Misture dois oligonucleotídeos (oHY502 e oHYbio85) em 1x tampão STE (100 mM NaCl, 10 mM Tris, pH 8,0; 1 mM de ácido etilenodiaminotetracético [EDTA]). A concentração final de oligonucleotídeos é de 10 μM e o volume final da reação é de 100 μL.
- Incubar a reação a 80 °C durante 5 min num bloco de calor. Desligue o bloco de calor e deixe a reação esfriar gradualmente, deixando-o no bloco de calor até que a temperatura caia abaixo de 30 °C.
- Valide a eficiência de recozimento em um gel de eletroforese em gel de tris-borato-EDTA (TBE)-poliacrilamida (PAGE).
- Prepare 8% de acrilamida - gel TBE (para 4 géis: 32,8 mL de H2O; 4,8 mL de tampão 10x TBE (1 M Tris, 1 M ácido bórico, 20 mM EDTA), 9,6 mL de solução de acrilamida/bis a 40%, 37,5:1; 0,8 mL de persulfato de amônio a 10% [APS], 40 μL de tetrametiletilenodiamina [TEMED]).
CUIDADO: TBE, acrilamida, APS, TEMED são tóxicos/prejudiciais. - Execute um pequeno volume (1 μL) de oligonucleotídeos recozidos ao lado dos oligonucleotídeos individuais diluídos com corante de carga 6x no gel de acrilamida - TBE a 8% a 80 V, por 1 h em RT.
- Mergulhe o gel em 1x TBE suplementado com uma coloração de ácido nucleico adequada por 30 min em um agitador de balanço de gangorra e, em seguida, escaneie o gel usando um sistema de imagem de gel.
- Prepare 8% de acrilamida - gel TBE (para 4 géis: 32,8 mL de H2O; 4,8 mL de tampão 10x TBE (1 M Tris, 1 M ácido bórico, 20 mM EDTA), 9,6 mL de solução de acrilamida/bis a 40%, 37,5:1; 0,8 mL de persulfato de amônio a 10% [APS], 40 μL de tetrametiletilenodiamina [TEMED]).
- Resolva o DNA recozido no gel TBE-PAGE para excisão do gel.
- Se os oligonucleotídeos forem recozidos corretamente, resolva o restante da amostra na presença de BSA (~ 100 μL de amostra recozida, 25 μL de corante de carga 6x, 2 μL de 33 mg / mL de BSA). Para carregar o volume maior no gel, combine vários poços com um bisturi estéril.
- Deixe o gel em 1x TBE suplementado com coloração de ácido nucleico adequada por 30 min em um agitador de balanço de gangorra.
- Transfira o gel para uma bandeja de vidro e visualize o DNA em uma sala escura sob luz azul. Use uma tela de proteção adequada ou óculos de proteção ao usar luz azul.
- Corte a faixa desejada com um bisturi estéril. Corte o excesso de gel.
- Purificar o garfo recozido por eletroeluição.
- Corte um pedaço de tubo de diálise longo o suficiente para inserir o pedaço de gel, molhe-o em 1x TBE e feche uma extremidade.
- Diluir a BSA a 0,3 mg/mL em 500 μL de 1x TBE e pipetar todo o volume para o tubo.
- Use a espátula de metal para deslizar a peça de gel para dentro. Mova a peça de gel para um lado do tubo para dar espaço tampão para o DNA migrar durante a eletroeluição.
- Esprema o excesso de tampão e prenda a segunda extremidade do tubo.
NOTA: O volume restante determinará o volume e a concentração finais. - Coloque o tubo de diálise no tanque de eletroforese de agarose em 1x tampão TBE, garantindo que o tubo esteja totalmente submerso.
NOTA: Certifique-se de que a peça de gel seja colocada no lado mais próximo do eletrodo negativo para que o DNA possa migrar do gel para a bolsa de diálise. - Execute a eletroeluição a 80 V por 1-2 h.
- Dialisar o ADN electroeluído para um tampão Tris 10 mM (pH 8).
- Após a eletroeluição, retire a bolsa de diálise do tanque e seque uma extremidade com lenço de papel. Evite tocar na parte central do tubo com tecido, onde o gel e a amostra estão localizados.
- Remova o clipe da extremidade seca e pipete suavemente a amostra para cima e para baixo dentro do tubo para misturar o DNA no tampão.
- Mergulhe uma espátula limpa em 1x tampão TBE e retire o pedaço de gel do tubo.
NOTA: Ao remover o pedaço de gel, remova o mínimo de tampão possível. - Prenda a extremidade aberta da bolsa de diálise novamente, removendo o ar de dentro.
- Coloque a bolsa de diálise em um béquer de 2 L cheio de 1,5 L de tampão Tris 10 mM (pH 8), NaCl 20 mM e MgCl 2 mM2. Enrole os clipes com parafilme para colar o tubo na borda do béquer enquanto submerge totalmente o tubo no tampão.
NOTA: O sal estabiliza oligoduplexes curtos. - Use uma barra magnética para agitar suavemente o tampão e dialisar a amostra à temperatura ambiente (RT) por pelo menos 3-4 h ou a 4 ° C durante a noite.
- Após a diálise, seque uma extremidade da bolsa de diálise com um lenço de papel (para não diluir o DNA) e solte essa extremidade.
- Pipete suavemente a amostra para cima e para baixo dentro do tubo e transfira-a para um tubo limpo de 1,5 mL.
- Medir a concentração do ADN utilizando um espectrofotómetro de microvolume.
- Recozer o DNA do garfo.
- Geração de fragmento de 20 kb
- Realize a digestão de restrição do plasmídeo pGC26118 usando enzimas NotI / I-CeuI.
- Misture suavemente 8 μL de NotI-HF (20.000 U/mL), 8 μL de I-CeuI (5.000 U/mL) e o plasmídeo pGC261 (concentração final de plasmídeo ~40 ng/μL em uma reação de 200 μL) em um tampão recomendado pelos produtores de enzimas de restrição.
- Incubar a reação a 37 °C durante a noite.
- Resolva o DNA digerido no gel de agarose para excisão em gel.
- OPCIONAL: Execute uma pequena quantidade da reação em um gel de agarose a 0,6% primeiro para testar a eficiência da digestão antes de carregar toda a reação.
- Prepare o gel de agarose a 0,6% seguindo as etapas 1.2.2.3-1.2.2.4.
- Misture 0,48 g de pó de agarose com 80 mL de tampão 1x TBE. Microondas a solução até que esteja fervendo. Agite para garantir que a agarose esteja totalmente derretida. Deixe esfriar por alguns minutos e despeje no tanque designado.
- Para combinar poços para acomodar o maior volume da amostra, cole um pedaço de fita adesiva em vários poços do pente com antecedência.
- Assim que o gel solidificar, remova suavemente o pente e encha o tanque com 1x tampão TBE. Misture suavemente a amostra com corante de carga de DNA e execute o gel a 120 V por 1 h.
- Deixe o gel em 1x TBE suplementado com uma coloração de ácido nucleico adequada em um agitador de balanço de gangorra até ficar manchado.
- Transfira o gel corado para uma bandeja de vidro e visualize o DNA em uma sala escura sob luz azul.
- Corte a faixa desejada com um bisturi estéril. Corte o excesso de gel.
- Opcional: Purifique o fragmento desejado por eletroeluição, conforme descrito nas etapas 1.1.4 e 1.1.5. Omita o sal do tampão de diálise aqui, pois isso só é necessário para estabilizar o oligoduplex curto.
- Realize a digestão de restrição do plasmídeo pGC26118 usando enzimas NotI / I-CeuI.
- Geração de extremidade de DNA duplex de digoxigenina
- Realize a reação de PCR com digoxigenina-dUTP (dig-dUTP) usando o DNA pGC261 como modelo.
- Mistura: 400 μL de água, 8 μL de pGC261 (0,8 ng/μL), 3,5 μL de primer oHY507 (100 μM), 3,5 μL de primer oHY508 (100 μM), 8 μL de dig-dUTP (1 mM), 400 μL de mistura de PCR 2x (preparada a partir de DNA polimerase (20 μL), 10 mM dNTPs (40 μL), 5x tampão de alta fidelidade (HF) (400 μL) e água (540 μL).
- Execute a seguinte reação em um termociclador:
98 °C - 1 min
30x: 98 °C - 20 s; 65 °C - 20 s; 72 °C - 30 s;
72 °C - 10 min
4 °C - Segurar
- Digerir e purificar o produto de PCR marcado com digoxigenina
- Purifique o produto de PCR usando um kit comercial de purificação de DNA.
- Misture 10 μL de NotI-HF (20.000 U/mL) com o produto de PCR em um tampão recomendado pelo fornecedor da enzima (concentração final de DNA de ~50 ng/μL em uma reação de ~200 μL). Incubar a reação a 37 °C durante a noite.
- Purifique o DNA digerido usando um kit comercial de purificação de DNA.
- Realize a reação de PCR com digoxigenina-dUTP (dig-dUTP) usando o DNA pGC261 como modelo.
- Monte os componentes para fazer o substrato de DNA.
- Misture delicadamente a extremidade do garfo biotinilada, o fragmento de DNA de 20 kb e o fragmento de PCR tratado com Not-I marcado com digoxigenina com 5 μL de T4 DNA ligase (400.000 U/mL) em um tampão recomendado pelo fornecedor da enzima em uma reação de 200 μL. No entanto, com base no fragmento de DNA de 20 kb adicionado (1-5 μg), adicione aproximadamente um excesso molar de 100 vezes a extremidade do garfo biotinilada e o fragmento de PCR.
- Alicitar a reação em tubos de PCR (50 μL cada) e incubar a 16 °C em um termociclador durante a noite.
- Resolva a amostra ligada em um gel de agarose a 0,6% e purifique o DNA ligado por eletroeluição, conforme descrito nas etapas 1.2.2 e 1.2.3.
- Armazenar o ADN congelado a -80 °C.
2. Purificação de Drosophila melanogaster CMG (Figura 2)

Figura 2: Purificação de Drosophila melanogaster CMG de 4 L de células Hi Five. As proteínas foram resolvidas em gel de poliacrilamida Bis-Tris 4%-12% sob 200 V na presença de tampão MOPS. A amostra é mostrada em cada estágio da purificação (lisado celular - 2 μL, eluição FLAG - 10 μL, após a primeira coluna de troca iônica - 10 μL, e após a marcação e a segunda coluna de troca iônica - 1 μL. (A) A coloração de Coomassie confirma a presença de todas as 11 subunidades do complexo CMG antes (10 μL) e depois (1 μL) da marcação fluorescente. (B) A eficiência de marcação da subunidade MCM3 foi validada por varredura para Cy5 com um analisador de imagem fluorescente usando um filtro vermelho de passagem longa (LPR). Clique aqui para ver uma versão maior desta figura.
NOTA: Para preparar Drosophila melanogaster CMG marcada com fluorescência, um local de clivagem TEV (ENLYFQG) seguido por quatro resíduos Gly foi introduzido a jusante da etiqueta FLAG N-terminal na subunidade MCM3 (no vetor pFastBac1) 10 . Para expressar o complexo, foi utilizado o sistema de expressão de baculovírus. Para a transfecção inicial, as células Sf21 foram usadas separadamente para cada subunidade CMG (estágio do vírus P1). Para amplificar ainda mais os vírus, foram utilizadas células Sf9 (estágio do vírus P2). Posteriormente, as culturas de células Sf9 (100 mL para cada subunidade CMG; 0,5 x 106 células/mL) foram infectadas com 0,5 mL do vírus P2 suplementado com 10% de soro fetal de bezerro (estágio do vírus P3). Para expressar todo o complexo CMG em 4 L de células Hi Five (1 x 106 células/mL), foram utilizados 200 mL de vírus P3 para cada uma das subunidades. Após a colheita das células Hi Five que expressam o complexo CMG, o pellet celular pode ser congelado rapidamente em nitrogênio líquido e armazenado a -80 ° C. Efectuar toda a purificação com gelo ou a 4 °C. Os tampões podem ser preparados com antecedência, desde que os agentes redutores (DTT ou 2-mercaptoetanol) e inibidores de protease (CUIDADO) sejam adicionados imediatamente antes do uso. Certifique-se de que todos os buffers sejam pré-resfriados com antecedência, filtrados e desgaseificados.
- Prepare os buffers a seguir.
- Prepare o tampão de ressuspensão A misturando 25 mM de ácido 4-(2-hidroxietil)-1-piperazineetanossulfônico (HEPES) pH 7,5, 0,02% de Tween-20, 10% de glicerol, 15 mM de KCl, 2 mM de MgCl2, 2 mM de 2-mercaptoetanol, 1 mM de EDTA e 1 mM de etilenoglicol-bis (éter β-aminoetílico)-N,N,N',N'-ácido tetracético (EGTA).
CUIDADO: 2-mercaptoetanol, TDT e EDTA são tóxicos/prejudiciais - Prepare o tampão salino tamponada com Tris (TBS; 0,1 M Glycine HCl, pH 3,5) para preparar o gel de afinidade ANTI-FLAG M2.
- Prepare o tampão A-100 misturando 25 mM HEPES pH 7.5, 0.02% Tween-20, 10% de glicerol, 100 mM KCl, 1 mM DTT, 1 mM EDTA e 1 mM EGTA.
- Prepare o tampão de diálise adicionando HEPES 25 mM pH 7.5, acetato de sódio 50 mM, acetato de magnésio 10 mM, glicerol 10% e DTT 1 mM.
- Prepare o tampão TBS misturando 50 mM Tris-HCl pH 7,5 e 150 mM NaCl.
- Prepare o tampão de ressuspensão A misturando 25 mM de ácido 4-(2-hidroxietil)-1-piperazineetanossulfônico (HEPES) pH 7,5, 0,02% de Tween-20, 10% de glicerol, 15 mM de KCl, 2 mM de MgCl2, 2 mM de 2-mercaptoetanol, 1 mM de EDTA e 1 mM de etilenoglicol-bis (éter β-aminoetílico)-N,N,N',N'-ácido tetracético (EGTA).
- Ressuspenda o pellet de 4 L de cultura de células Hi Five em 200 mL de tampão de ressuspensão a frio A suplementado com comprimidos de coquetel de inibidores de protease. Misture os tubos por inversão até que todo o pellet esteja totalmente ressuspenso.
- Transfira as células ressuspensas para o homogeneizador Dounce pré-resfriado de 40 mL. Para recuperar o maior número possível de células, lave os tubos usados para o congelamento de pellets de células com o mesmo tipo de tampão e adicione ao homogeneizador Dounce. Certifique-se de que o volume total no homogeneizador não exceda o limite recomendado. Lise as células no gelo em 60-70 golpes.
NOTA: Certifique-se de que o êmbolo atinja o fundo do tubo, mas não coloque muita pressão, pois o homogeneizador pode quebrar. Tenha cuidado para não retirar o êmbolo acima do nível do líquido, pois isso introduzirá bolhas. - Repetir o passo anterior até que toda a amostra esteja homogeneizada e combiná-la no copo pré-arrefecido. Avalie o volume final da amostra. Gota a gota, adicione KCl filtrado à concentração final de 100 mM, misturando suavemente.
- Pré-arrefecer a centrífuga a 4 °C com antecedência. Despeje a amostra em tubos de centrifugação, garantindo que o volume recomendado pelo produtor seja atingido. Equilibre os tubos em uma balança.
NOTA: Tubos com volume muito pequeno podem quebrar durante a centrifugação. - Centrifugar as células lisadas a 23.500 x g durante 15-30 min a 4 °C. Enquanto isso, prepare o gel de afinidade ANTI-FLAG M2 de acordo com a próxima etapa.
- Misture delicadamente o frasco contendo o gel de afinidade ANTI-FLAG M2 (50% de suspensão). Corte a extremidade de uma ponta de pipeta P-100 (para alargar a ponta) e use-a para transferir imediatamente 4 mL de suspensão (2 mL de contas) para uma coluna de cromatografia de polipreparação de 20 mL. Para recuperar as contas presas na ponta, lave a ponta com TBS e adicione-a à coluna. Deixe todas as contas assentarem dentro da coluna, mas tome cuidado para não secá-las.
- Lave a resina com 3 grânulos de volume (6 mL no total) de 0,1 M Glicina HCl pH 3,5. Em seguida, lave as esferas com 3-5 esferas de volume de TBS (6-10 mL). Em seguida, lave as esferas 3 vezes (6 mL) com tampão A-100.
NOTA: Não deixe os grânulos na solução de Glycine HCl pH 3,5 0,1 M por mais de 20 min. Para aumentar a pressão do fluxo e a velocidade de lavagem, cubra a coluna com uma tampa. Não deixe a resina secar em nenhum momento. - Depois de preparar a resina, deixe 2 mL de tampão A-100 acima da resina e feche a saída da coluna.
- Após a centrifugação, despeje suavemente o sobrenadante em tubos pré-resfriados de 50 mL. Tenha cuidado para não interromper o pellet. Pegue uma pequena alíquota (~ 10 μL) para validar por eletroforese SDS-PAGE posteriormente.
- Adicione uma quantidade igual de esferas ANTI-FLAG M2 (preparadas de acordo com a etapa anterior) em cada um dos tubos de 50 mL. Tente recuperar todos os grânulos da coluna ressuspendendo-os com alguns mililitros adicionais de Buffer A-100 e transferindo essa ressuspensão para os tubos.
- Incubar a amostra (sobrenadante) com resina ANTI-FLAG M2, girando por 2,5 h a 4 °C.
- Após a incubação, centrifugar a amostra a 1.000 x g durante 5 min a 4 °C. Usando uma pipeta, remova a maior parte do sobrenadante. Usando uma ponta cortada, ressuspenda as contas em alguns mililitros do sobrenadante restante e transfira-as para dois tubos de 15 mL. Para recuperar quaisquer grânulos presos nas paredes dos tubos de 50 mL, adicione alguns mililitros de Buffer A-100 e transfira-o para os tubos de 15 mL também.
- Gire os tubos de 15 mL a 1.000 x g por 5 min a 4 ° C. Remova o sobrenadante.
- Lave os grânulos adicionando ~ 14 mL de tampão A-100 a cada tubo, seguido de rotação curta e suave a 4 ° C e subsequente centrifugação a 1000 x g por 5 min a 4 ° C. Repita as lavagens duas vezes. Pegue pequenas alíquotas de cada lavagem (~ 10 μL) para validar por eletroforese SDS-PAGE posteriormente.
- Esta etapa (2.16) é um procedimento alternativo às etapas anteriores (2.13-2.15).
- Após a incubação com a resina ANTI-FLAG M2, despeje a amostra em duas colunas de cromatografia de polipreparação de 20 mL.
- Se escolher este método, deixe os grânulos assentarem na parte inferior da coluna e a amostra não ligada passar pela coluna pelo fluxo de gravidade. Em seguida, adicione o tampão A-100 diretamente à coluna e deixe-o lavar a amostra por fluxo por gravidade.
- Transfira os grânulos (ressuspensos no tampão A-100) para duas colunas de cromatografia de polipreparação de 10 mL.
- Deixe o tampão A-100 passar pela resina, feche a saída quando o nível do tampão atingir o topo da resina e elua a proteína dos grânulos adicionando o tampão A-100 suplementado com peptídeo de 200 μg / mL (DYKDDDDK) (tampão de eluição FLAG).
- Para a primeira eluição, adicione 3 mL do tampão de eluição FLAG e gire suavemente a coluna fechada por 15 min em RT. Abra a saída para coletar a primeira fração de eluição.
- Feche a saída e adicione 2 mL do tampão de eluição FLAG a cada coluna. Girar por mais 10 min e coletar a segunda fração de eluição.
- Combinar todas as fracções eluídas (~10 ml) e conservar a 4 °C. Pegue uma pequena alíquota da fração eluída (~ 10 μL) para validar por eletroforese SDS-PAGE posteriormente.
- Filtrar a amostra eluída utilizando filtros de seringa de 0,22 μm.
- Prepare previamente a coluna CaptoHiRes Q (5/50) (coluna de cromatografia de troca iônica de alta resolução) conectada a um sistema de purificação de proteínas, de acordo com o protocolo do fabricante. Em seguida, equilibre a coluna com o tampão A-100 e carregue a amostra filtrada na coluna equilibrada.
- Lave a coluna com 20 volumes de coluna (CV) de tampão A-100 (~ 20 mL no total, pois o volume da coluna é ~ 1 mL).
- Para eluir a proteína, prepare dois tampões com antecedência: Tampão A-100 e Tampão B com a mesma composição do Tampão A-100, mas com 550 mM KCl em vez de 100 mM KCl. Configure a eluição gradiente para 20 CV (~ 20 mL) com concentração crescente de sal de 100 mM para 550 mM KCl. Colete eluições de 0,3-0,5 mL em um coletor de frações.
NOTA: O CMG deve eluir em torno de 70%-75% de concentração de tampão B. Pegue pequenas alíquotas das frações de lavagem e eluídas para eletroforese SDS-PAGE. - Realize eletroforese SDS-PAGE para confirmar que o CMG está presente nas frações escolhidas. Enquanto isso, limpe a coluna e o sistema de purificação de acordo com o protocolo do fabricante.
- Incubar as frações escolhidas com protease TEV durante a noite, misturando 50 μL de protease TEV (1 mg/mL) por 1 mL de amostra.
- Umedeça previamente um tubo de diálise e certifique-se de que a membrana não esteja danificada. Adicione a mistura CMG/TEV ao tubo de diálise e coloque em um béquer de 2 L com 1,5 L de tampão pré-resfriado A-100 e a barra magnética dentro. Dialisar com agitação suave durante a noite a 4°C.
- Misture a amostra com 50 μM de peptídeo marcado com LD655 e 10 μg/mL de enzima Sortase na presença de 5 mM de CaCl2. Incubar a reação a 4 °C, girando por 30 min, garantindo que o tubo esteja coberto de luz. Pegue uma pequena alíquota antes e depois da rotulagem para eletroforese SDS-PAGE.
- Filtre a amostra rotulada usando filtros de centrífuga de 0,22 μm antes de carregá-la no sistema de purificação, pois o peptídeo pode precipitar.
- Preparar previamente a coluna de cromatografia de troca iónica de alta resolução, conforme descrito anteriormente (passo 2.20). Desta vez, cubra a coluna e o sistema coletor de frações com papel alumínio para proteger a amostra da luz.
- Para remover o peptídeo livre, carregue a amostra filtrada na coluna de cromatografia de troca iônica de alta resolução. Execute a purificação da mesma forma descrita anteriormente (etapas 2.21-2.22). Certifique-se de que o CMG rotulado elui em uma concentração de Buffer-B semelhante à anterior.
- Realize eletroforese SDS-PAGE para validar a qualidade das frações escolhidas. Para obter imagens da fluorescência, não ferva as amostras antes do carregamento e certifique-se de que o tanque de eletroforese esteja protegido da luz. Visualize a fluorescência da proteína usando um sistema de imagem de gel primeiro, depois pinte o gel com o corante Coomassie e visualize-o novamente para visualizar todas as proteínas.
- Dialisar as frações escolhidas durante a noite a 4 °C, contra 1,5 L do tampão de diálise. Se necessário, concentrar a amostra.
- Congelar rapidamente a proteína em nitrogênio líquido e armazená-la a -80 °C até uso posterior.
3. Preparação da célula de fluxo (Figura 3)
- Prepare lamínulas de biotina-PEG seguindo um protocolodescrito anteriormente 11, omitindo a etapa de cozimento do forno. As lamínulas de biotina-PEG são estáveis por pelo menos 1 mês sob vácuo em RT. Para a preparação da célula de fluxo, corte a lamínula de biotina-PEG (24 mm x 60 mm) ao meio (aproximadamente 24 mm x 30 mm).
NOTA: Tome cuidado para não tocar na área central da lamínula onde estarão os canais de fluxo. - Prepare pequenos pedaços de vidro gravando e encaixando uma lâmina de vidro em pedaços de aproximadamente 2,4 cm x 1 cm.
- Faça dois furos separados por 1,4 mm usando uma broca revestida de diamante de 0,8 mm, um ligeiramente mais largo que o outro (0,043" = entrada, 0,048" = saída). Teste se os furos são do tamanho correto tentando inserir um pedaço da tubulação de entrada/saída, usando a mesma broca para alargar o furo até que a tubulação se encaixe. Certifique-se de que o tubo se encaixe bem o suficiente para não cair facilmente.
NOTA: Tubulação de entrada mais estreita é usada para minimizar o volume morto. - Corte a fita dupla-face no mesmo formato dos pedaços de vidro.
- Alinhe o slide na fita e enfie uma agulha em cada orifício para marcar suas posições na fita. Usando uma lâmina de barbear, corte um canal que englobe os dois orifícios.
NOTA: Não corte o canal por muito tempo, pois o excesso de espaço no lado mais distante de cada orifício pode permanecer seco quando o líquido flui e pode causar problemas com bolhas de ar durante o fluxo. Ao cortar, tente cortar as bordas longas em um único corte limpo, pois as bordas desgastadas de vários cortes podem afetar a qualidade do canal de fluxo. Para um fim semelhante, não corte as bordas do canal de fluxo, pois cortes na peça utilizável podem afetar o fluxo de líquido ou até mesmo vazar na célula de fluxo montada. - Limpe a peça de vidro com acetona e lenço de papel até secar e coloque sobre uma superfície limpa. Retire um lado da fita adesiva e cole-a no pedaço de vidro de forma que ambos os orifícios fiquem totalmente dentro do canal.
- Sele a fita na peça de vidro usando uma ponta de pipeta p1000 para eliminar bolhas pressionando a superfície com pressão firme, mas moderada. Percorra toda a superfície para selar.
- Retire a segunda borda da fita de ambos os pedaços de vidro e coloque-os em uma superfície limpa com o lado adesivo para cima. Coloque as lâminas próximas umas das outras, mas sem se tocarem na posição em que serão fixadas na lamínula (deixe espaço suficiente entre elas para que o epóxi crie uma vedação completa ao redor de cada peça de vidro).
- Pegue a meia lamínula de biotina-PEG usando uma pinça de plástico, segurando apenas pela borda (não toque na área que formará o canal de fluxo) e abaixe o lado funcionalizado com PEG sobre o adesivo. Prenda pressionando com um dedo para fixá-lo no lugar e, em seguida, complete a vedação esfregando a superfície moderadamente firmemente com uma ponta de pipeta para remover as bolhas de ar (não na área do canal de fluxo, pois isso pode quebrar) e, em seguida, vire.
NOTA: Manuseie a lamínula com cuidado. - Para cada célula de fluxo, corte ~10 cm de cada tipo de tubo de polietileno (PE20 e PE60). Use o tubo mais estreito como entrada para a câmara de fluxo para reduzir o volume morto.
- Insira a tubulação nos orifícios da corrediça com a mão. Se o diâmetro dos orifícios estiver correto, certifique-se de que a tubulação fique em pé no orifício sozinha depois de inserida. Corte a ponta do tubo em um pequeno ângulo <45° para facilitar um pouco a inserção do tubo e evitar que o tubo forme uma vedação contra a lamínula, o que pode acontecer se a extremidade for plana (isso pode obstruir o fluxo).
NOTA: O encaixe preciso da tubulação na peça de vidro torna a construção da célula de fluxo mais confiável e ajuda a evitar a introdução de bolhas na célula de fluxo. - Misture bem os componentes epóxi e, em seguida, use uma ponta p200 para aplicar epóxi para selar a tubulação e criar uma vedação ao redor de cada peça de vidro. Adicione epóxi suficiente para que suba até a borda da lamínula, pois isso também reforçará a lamínula delicada. Certifique-se, no entanto, de que nenhum epóxi entre na parte inferior da lamínula, pois isso pode impedir que ela fique plana no palco.
NOTA: Se necessário, pequenas quantidades de excesso de epóxi podem ser raspadas da parte inferior usando uma lâmina de barbear. - Deixe por pelo menos 30-60 min para que o epóxi cure completamente.
NOTA: Se a célula de fluxo não for usada imediatamente, armazene-a sob vácuo em RT após a cura completa. - Depois de usar a célula de fluxo, recupere as peças de vidro e reutilize-as indefinidamente. Puxe o tubo para fora e coloque a célula de fluxo em um frasco deslizante contendo acetona por pelo menos 24 h para amolecer o epóxi e o adesivo de dupla face, permitindo que os pedaços de vidro sejam facilmente removidos. Antes de reutilizar para fazer outra célula de fluxo, limpe os pedaços de vidro esfregando-os com uma esponja e água e sabão, seque-os e esfregue-os com acetona e papel de seda.

Figura 3: Representação gráfica da preparação da célula de fluxo. (A) Corte fita dupla-face para corresponder ao tamanho do pedaço de vidro. Alinhe a lâmina na parte superior da fita e marque a posição de cada orifício com uma agulha. Usando uma lâmina de barbear, corte ao redor de cada porão para criar um canal. (B) Descasque um lado da fita e cole-a no pedaço de vidro. Certifique-se de que ambos os orifícios estejam dentro do canal. Descasque a segunda extremidade da fita e cole a lamínula de biotina-PEG por cima. (C) Insira o tubo de polietileno em cada orifício e sele o tubo no lugar com epóxi, selando cada peça de vidro na lamínula também. (D) Depois de usar os dois canais, retire a tubulação e coloque a célula de fluxo em um frasco de coloração cheio de acetona. Após aproximadamente 24 h, o epóxi e a fita terão amolecido e as camadas da célula de fluxo podem ser separadas. As peças de vidro podem ser recuperadas e armazenadas em acetona para serem reutilizadas indefinidamente para fazer a próxima célula de fluxo. Clique aqui para ver uma versão maior desta figura.
4. Ensaio TIRF de molécula única para visualizar o desenrolamento do DNA mediado por CMG
- Prepare os buffers a seguir.
- Prepare o tampão de bloqueio (20 mM Tris, pH 8,0; 50 mM NaCl; 2 mM EDTA; 0,2 mg / mL BSA). Filtrar o tampão com um filtro de seringa de 0,22 μm e conservá-lo a 4 °C.
- Prepare 10x tampão de reação 1 (250 mM HEPES pH 7,5; 50 mM NaCl; 100 mM Acetato de magnésio). Filtrar o tampão com um filtro de seringa de 0,22 μm e conservá-lo a 4 °C.
- Prepare 10x tampão de reação 2 (250 mM Tris, pH 7,5; 100 mM de acetato de magnésio; 1,25 M de glutamato de potássio; 1 mM de EDTA; 0,025% Tween-20). Filtrar o tampão com um filtro de seringa de 0,22 μm e conservá-lo a 4 °C.
- Desgaseifique aproximadamente 2,5 mL de tampão de bloqueio e 5 mL de água ultrapura, colocando os tubos abertos em um dessecador e deixando-os sob vácuo por 15 min.
NOTA: Não encha os tubos até a borda, pois as bolhas durante a desgaseificação podem causar respingos de líquido. - Coloque a célula de fluxo em um microscópio stage e prenda-a no lugar usando uma massa adesiva em cada extremidade. É importante proteger bem para evitar que a célula de fluxo se mova mais tarde durante a aquisição.
- Conecte a tubulação de saída da célula de fluxo à bomba da seringa, que possui a tubulação conectada à seringa com uma agulha na outra extremidade. Insira esta agulha na tubulação de saída para conectar a célula de fluxo.
- Ligue o aquecedor da objetiva a 30 °C.
- Prenda 1-2 mL de água desgaseificada em um tubo a um pedaço separado de massa adesiva próximo à célula de fluxo, de modo que o tubo de entrada possa ser inserido e atingir o fundo do tubo.
- Flua a água pelo canal e, em seguida, use um fluxo mais rápido para remover quaisquer bolhas presas perto da tubulação de entrada, adicionando mais água, se necessário. Caso contrário, grandes bolhas podem ser desalojadas mais tarde nos experimentos e passar pelo canal. Deixe por alguns minutos para que o fluxo pare totalmente, pois o fluxo rápido pode causar a extração residual do líquido por um tempo depois de parado, à medida que a pressão dentro da seringa se estabiliza.
NOTA: Qualquer DNA que entre em contato com uma bolha torna-se inutilizável, mesmo depois de reidratado. - Adicionar 100 μL de tampão de bloqueio desgaseificado a uma alíquota de 20 μL de estreptavidina a 1 mg/ml. Fixe o tubo aberto à massa adesiva e transfira o tubo de entrada da água para a estreptavidina. Flua a uma taxa de 40 μL/min por 2 min (80 μL no total) e incube por 5 min.
- Lave o excesso de estreptavidina com o tampão de bloqueio (50 μL / min para 100 μL).
- Fluxo em DNA biotinilado, diluído em tampão de bloqueio com laranja SYTOX 25 nM. Imagem usando visualização ao vivo com o laser de 532 nm para observar o DNA se ligando à superfície em tempo real.
- Quando a densidade aproximada de DNA na superfície for alcançada, flua no tampão de bloqueio, também com 25 nM SYTOX (50 μL / min para 100 μL), para lavar o DNA livre.
- Fluxo em anticorpo antidigoxigenina biotinilado (~ 10 μg / mL) diluído em tampão de bloqueio contendo 25 nM de laranja SYTOX (100 μL / min para 300 μL).
NOTA: O laranja SYTOX intercala entre os pares de bases do DNA, estendendo o comprimento do contorno. Isso significa que o comprimento de ponta a ponta do DNA aumenta na laranja SYTOX, permitindo que a segunda corda se prenda à superfície mais longe da primeira, dando um DNA melhor esticado quando a laranja SYTOX é lavada mais tarde. O uso de uma porção diferente em cada extremidade do DNA permite uma amarração mais uniforme do DNA à superfície da lamínula (Figura 4). Quando ambas as extremidades são biotiniladas, elas se amarram na mesma etapa, resultando em uma variação significativa em suas posições de fixação. O uso de digoxigenina em uma extremidade dá mais controle sobre a cobertura do DNA antes de amarrar a segunda extremidade. Isso também permite que a cobertura de DNA seja aumentada por incubação. - Lave o anticorpo anti-digoxigenina biotinilado e o laranja SYTOX com tampão de bloqueio (50 μL / min para 100 μL).
- Completar 120 μL de 'mistura de ATP-g-s' (1x tampão de reação 1, 0,75 mg/mL BSA, 1,25 mg/mL de caseína, 8 mM DTT, 0,33 mM ATP-g-s), adicionar 30 μL a um novo tubo e, em seguida, fluir 50 μL do tubo original para a célula de fluxo (50 μL/min para 50 μL). Isso ajuda a minimizar o efeito da mistura de tampão por meio de refluxo ou difusão da tubulação de saída.
- Adicione CMG purificado a ~ 100 nM final em 30 μL de mistura de ATP-g-s e, em seguida, flua a 20 μL / min por 20 μL. Incube por 15 min.
- Compõem 120 μL de 'mistura ATP/RPA' (1x tampão de reação 1, 0,75 mg/mL BSA, 8 mM DTT, 3,3 mM ATP, 20 nM EGFP-hRPA) e fluxo para a célula de fluxo a 40 μL/min para 80 μL.
- Comece imediatamente a adquirir imagens. Adquira 6 x 6 campos de visão para cada quadro a cada 30 s (ou uma taxa de quadros apropriada para o experimento). Taxas de quadros mais baixas podem reduzir a exposição ao laser e o fotobranqueamento com fluoróforo. Visualize EGFP-hRPA com um laser de 488 nm a 1% de potência. Se o CMG estiver rotulado, por exemplo, LD655, visualize o CMG com laser de 640 nm (potência máxima = 30 mW) a 10% de potência. Visualize o DNA corado de laranja SYTOX com um laser de 532 nm a 0,5% de potência. Use cada laser com exposição de 50-100 ms.
NOTA: Cada vez que a tubulação de entrada da célula de fluxo for transferida de um tubo de líquido para outro, passe a ponta da tubulação na parte inferior do tubo várias vezes antes de removê-la do líquido. Isso ajuda a evitar que bolhas entrem durante a transferência. Minimize o tempo em que a ponta da tubulação de entrada fica fora do líquido e não aponte a ponta para cima ou levante a ponta significativamente, pois isso faz com que o líquido escorra em direção ao canal e puxe o ar para dentro da ponta. Ao transferir a tubulação de entrada, tente não colocar tensão na tubulação, pois isso pode puxá-la para fora do epóxi, prendendo-a à célula de fluxo. Para o desenrolamento do DNA de imagem, na presença de laranja SYTOX para visualizar o DNA de fita dupla, substitua o tampão de reação 10x 1 pelo tampão de reação 10x 2 para a mistura ATP/RPA. A laranja SYTOX liga-se melhor ao ADN nestas condições. Esvazie totalmente a bomba de seringa no final de cada experimento. Lave ocasionalmente enchendo com água e esvaziando várias vezes ou desmontando e lavando com água e sabão.

Figura 4: Amarração de DNA à superfície. (A) Ao amarrar substratos de DNA com biotina em ambas as extremidades, a distância entre as duas amarras pode variar dependendo de como as extremidades entram em contato com a superfície (i). Usando digoxigenina em uma extremidade, a amarração de cada extremidade pode ser separada temporariamente para distâncias de amarração mais consistentes e DNA esticado de maneira mais uniforme (ii). (B) Exemplo de campo de visão mostrando DNA amarrado por ambas as extremidades (marcado com digoxigenina) e corado com coloração de ácido nucleico intercalante fluorescente. O DNA, que é amarrado por ambas as extremidades, aparece como uma linha, enquanto o DNA amarrado por apenas uma extremidade aparece como manchas. Idealmente, o DNA deve ser amarrado o mais densamente possível, sem sobrepor outro DNA. A imagem tem 512 x 512 pixels (tamanho do pixel = 154,6 nm). Clique aqui para ver uma versão maior desta figura.
Resultados
Quando o CMG desenrola o DNA, um trato característico de RPA cresce com o tempo (Figura 5). A extremidade 5 'do DNA desenrolado está amarrada à superfície; portanto, é visto como um trecho linear do sinal RPA entre a corda e o garfo. A extremidade 3' não está amarrada e, portanto, se move com o garfo e é observada como um sinal EGFP-RPA compacto. A posição da fita de translocação desenrolada compactada corresponde aproximadamente à posição do garfo de replicação, que se move junto com o LD655-CMG visualizado por meio de um laser de 640 nm.
É importante minimizar os danos ao substrato do DNA, pois danos como cortes de DNA de fita simples reduzem o número de eventos de desenrolamento observáveis, limitando a quantidade de dados que podem ser coletados (Figura 6).
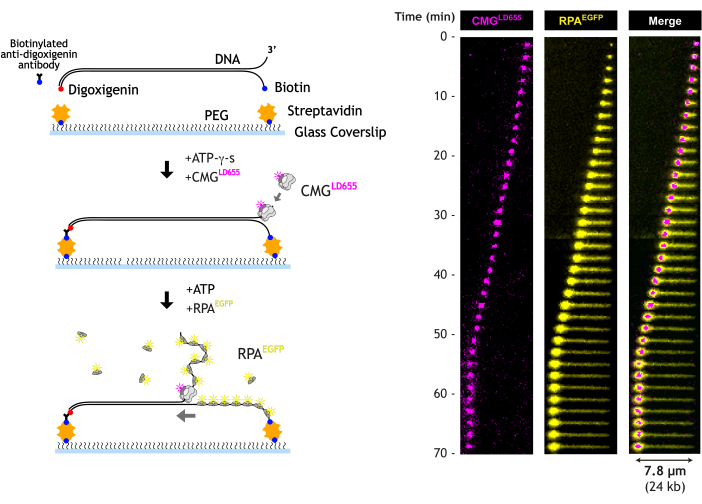
Figura 5: Ensaio de desenrolamento de DNA de molécula única. O substrato de DNA é amarrado a uma superfície de lamínula. O CMG purificado marcado com LD655 é incubado com o DNA por 15 min em ATP-g-s. ATP e RPA purificado marcado com EGFP são adicionados, iniciando um extenso desenrolamento de DNA por CMG. Um esquema de desenho animado (à esquerda) e um quimógrafo de dados representativos (à direita) são mostrados. Clique aqui para ver uma versão maior desta figura.
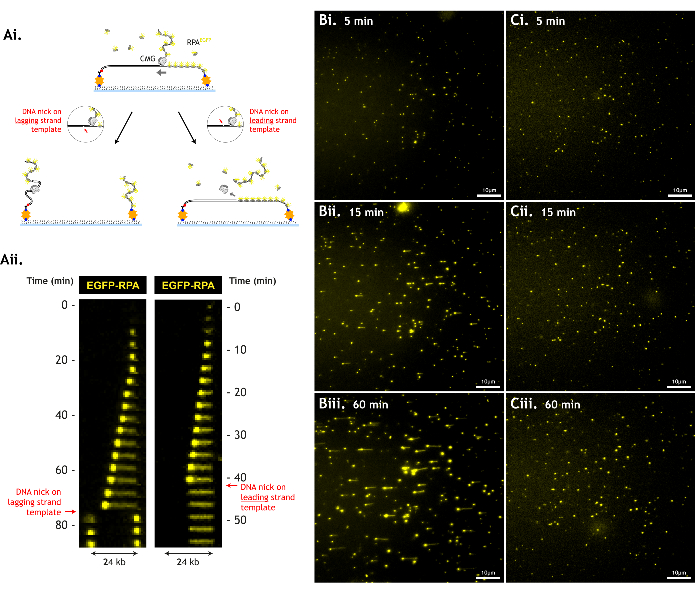
Figura 6: Danos ao DNA reduzem o rendimento do ensaio. (A) O CMG não pode desenrolar o DNA após uma quebra na estrutura do DNA (corte de DNA). Um corte no modelo de fita principal faz com que o CMG deslize para fora do DNA, e tanto o CMG quanto o modelo de fita principal são perdidos. Um corte no modelo de fita atrasada faz com que o modelo de fita atrasada se separe do resto do DNA, e cada pedaço de DNA se retrai para sua respectiva amarra. Isso é ilustrado por (i) esquemas de desenhos animados e (ii) quimógrafos desses eventos (ii). Dados representativos com (B) um substrato de DNA minimamente danificado versus (C) um substrato de DNA mais danificado em (i) 5 min, (ii) 15 min e (iii) 60 min em um único campo de visão. O substrato de DNA mais danificado não gera longos trechos de desenrolamento, pois os CMGs encontram cortes mais cedo, apesar dos níveis semelhantes de atividade de desenrolamento (densidade semelhante de pontos de RPA em crescimento em 5 min, indicando eficiência de carregamento/desenrolamento de CMG semelhante). O campo de visão é de 512 x 512 pixels (tamanho do pixel = 154,6 nm). 1% de potência do laser (488 nm) de geração de imagens EGFP-RPA. Barra de escala mostrando 10 μm. Clique aqui para ver uma versão maior desta figura.
Discussão
Este ensaio fornece uma plataforma para observar e investigar a dinâmica em tempo real de CMGs individuais, tanto isoladamente quanto no contexto dos fatores adicionais desejados. No entanto, como acontece com muitas técnicas de fluorescência de molécula única, existem alguns desafios comuns que podem exigir otimização para serem superados. Estes geralmente estão relacionados a fluoróforos de imagem por longos períodos de tempo (fotobranqueamento, brilho), preparação de substrato de DNA (danos ao DNA), a qualidade da superfície da célula de fluxo (ruído de fundo, interações não específicas) ou a qualidade da preparação de proteína purificada (contaminação de nuclease, eficiência de marcação).
Cada fluoróforo varia em fotoestabilidade e brilho, por isso é importante escolher uma molécula apropriada. Ao obter imagens de proteínas oligoméricas marcadas com fluorescência, como RPA, uma potência de laser mais baixa pode ser usada, pois muitos fluoróforos serão excitados nas proximidades, gerando um sinal visível. Para imagens de fluoróforos únicos, por exemplo, CMG marcado em uma única subunidade, é necessária uma potência de laser mais alta para observar o fluoróforo claramente. A vida útil do fluoróforo pode ser estendida minimizando a exposição ao laser, como reduzindo a frequência com que as imagens são tiradas. Além disso, excitar um fluoróforo gera espécies reativas de oxigênio (ROS), que podem contribuir para o fotobranqueamento. A inclusão de um sistema de eliminação de oxigênio no tampão de imagem pode prolongar a vida útil dos fluoróforos, eliminando as ROS. No entanto, alguns sistemas de sequestro de oxigênio podem afetar o pH12.
Em relação à preparação do substrato de DNA, é crucial minimizar os danos ao DNA, como cortes ou lacunas de fita simples. Danos excessivos impedem o desenrolamento extensivo do DNA, limitando a quantidade de dados que podem ser coletados. Os danos podem surgir de cisalhamento mecânico, aquecimento excessivo, como resultado da contaminação da nuclease ou ROS geradas durante a imagem. O cisalhamento pode ser minimizado manuseando a amostra de DNA com cuidado, usando pontas de furo largo para pipetagem, pipetando lentamente e evitando sacudir a amostra. O efeito das ROS pode ser minimizado reduzindo a exposição ao laser ou incluindo um sistema de eliminação de oxigênio no tampão de imagem. Após a preparação do substrato de DNA, é possível usar kits comerciais de reparo de DNA para reparar o dano antes de realizar uma reação de desenrolamento.
A eficiência do desenrolamento do DNA também depende da pureza e atividade do CMG. É uma boa prática avaliar a pureza da amostra após cada etapa de purificação por eletroforese SDS-PAGE para determinar onde a otimização é necessária. Se muitos contaminantes forem observados após a etapa final, pode ser útil modificar os volumes de gradiente de sal usados para a eluição da coluna CaptoHiRes Q (5/50). Também é muito importante remover qualquer excesso de peptídeo fluorescente usado para a rotulagem da proteína, pois pode criar um fundo indesejável na superfície da lamínula. Também é essencial evitar a contaminação por nucleases, pois isso pode degradar o substrato do DNA. Após um experimento, corar o DNA restante com laranja SYTOX pode ser uma boa maneira de verificar se o DNA foi degradado significativamente ou não. Um certo nível de dano ao DNA é inevitável ao longo de um experimento, mas danos significativos geralmente indicam contaminação problemática da nuclease.
O ensaio também é inerentemente limitado pela resolução de pontos limitados por difração, exigindo que as proteínas fluorescentes estejam a centenas de pares de bases de distância (se não mais) para distingui-las como separadas. Isso limita os detalhes em que a progressão e as interações do CMG podem ser observadas.
O número de eventos de desenrolamento que observamos para cada análise varia. Para um experimento bem-sucedido, esperamos ver pelo menos vários trechos de RPA de comprimento suficiente por campo de visão de 512x512 pixels (tamanho do pixel = 154,6 nm). Vários campos de visão podem ser visualizados no mesmo experimento, permitindo mais coleta de dados quando necessário. Os tratos não precisam ter o mesmo comprimento nem chegar ao final do DNA para serem úteis. Por exemplo, a distância média do cabo para cada experimento pode ser determinada medindo o comprimento do DNA corado com SYTOX antes de adicionar CMG. Isso pode ser usado para estimar quanto DNA foi desenrolado para qualquer trato de RPA (desde que DNA suficiente seja desenrolado para mover visivelmente o garfo), convertendo a distância de 'μm percorrido' para 'kb desenrolado'.
O CMG exibe atividade de desenrolamento em uma variedade de substratos de DNA, mas é essencial fornecer uma extremidade de DNA 3 'livre em um retalho poliT de pelo menos 30 nt para acomodar a pegada do CMG10. A inclusão de várias porções de biotina no garfo garante uma amarração robusta da superfície. O resto do substrato de DNA pode ser redesenhado de várias maneiras, como incluir diferentes sequências de DNA, comprimentos e modificações químicas. A conformação do DNA pode ser alterada usando diferentes concentrações de acetato de magnésio. Em concentrações mais altas (≥10 mM) de acetato de magnésio, o filamento de ssDNA revestido com RPA é compactado, levando o DNA a ser puxado pela ligação de RPA durante o desenrolamento. Isso pode ser útil, pois evita que o DNA se mova excessivamente, permitindo que a posição do CMG e da progressão do desenrolamento sejam medidas com mais precisão. Em baixas concentrações (~ 3 mM) de acetato de magnésio, o RPA-ssDNA permanece relaxado por toda parte.
O ensaio de molécula única descrito representa uma plataforma que pode ser construída e modificada para investigar outros aspectos da replicação do DNA. Durante a replicação do DNA, o CMG atua como um núcleo em torno do qual o replissoma e seus componentes se reúnem. Portanto, proteínas purificadas adicionais podem ser adicionadas a este ensaio, incluindo fatores acessórios como TIMELESS, TIPIN e CLASPIN, para estudar seu efeito na dinâmica do CMG. Essas proteínas demonstraram afetar a taxa de garfos de replicação13, mas não está claro como elas afetam a taxa de desenrolamento do CMG. Portanto, seria interessante investigar como diferentes proteínas do replissoma afetam o CMG usando este ensaio. A adição de DNA polimerases pode fornecer uma melhor visão da replicação do DNA além do desenrolamento do DNA sozinho, conforme descrito anteriormente com proteínas de levedura14. Além disso, a purificação do CMG modificado pode fornecer uma melhor compreensão de como certas mutações ou modificações pós-traducionais afetam a atividade da helicase15,16. Além disso, o design de diferentes substratos de DNA pode permitir que o desenrolamento do DNA por CMG seja estudado sob uma variedade de condições que imitam o estresse de replicação17. Essas modificações incluem obstáculos de DNA 9,18, ligações cruzadas entre fitas 19,20,2 1 e descontinuidades nas fitas de DNA22.
Divulgações
Os autores não têm interesses financeiros concorrentes ou outros conflitos de interesse.
Agradecimentos
Agradecemos a Gheorghe Chistol por fornecer o plasmídeo pGC261 e ao Francis Crick Institute Chemical Biology Facility para síntese e marcação de peptídeos. Este trabalho foi financiado pelo Instituto Francis Crick, que recebe financiamento básico da Cancer Research UK, do UK Medical Research Council e do The Wellcome Trust (CC2133).
Materiais
| Name | Company | Catalog Number | Comments |
| 2-Mercaptoethanol | Sigma-Aldrich | M3148-25ML | |
| Acrylamide / Bis Solution 40% | BioRad | 1610148 | |
| Agarose UltraPure | Invitrogen | 16500-500 | Used to prepare Tris Buffer (pH 8) |
| ÄKTA Pure | GE Healthcare | - | Used with Fraction Collector F9-C; protein purification system |
| ANTI-FLAG M2 affinity gel | Sigma-Aldrich | A2220 | |
| APS 10% | Sigma-Aldrich | A3678-25G | |
| ATP (200 mM) | Sigma-Aldrich | A7699-5G | |
| ATP-g-s (100 mM) | Sigma-Aldrich | 11162306001 | |
| Biotin-PEG coverslip | N/A | N/A | Prepared as described: https://doi.org/10.1016/j.ymeth.2012.03.033 |
| Biotinylated anti-digoxigenin antibody | Perkin Elmer | N/A | |
| Blu Tack | Bostik | N/A | Adhesive putty |
| Boric acid | Thermo Fisher Scientific | B/3800/53 | |
| BSA; 33 mg/mL | SIGMA-ALDRICH | A3858-10G | Diluted from the stock |
| CaCl2 | Sigma-Aldrich | C7902 | |
| CaptoHiRes Q (5/50) | Cytiva | 29275878 | Connected to the AKTA protein purification system; high-resolution ion exchange chromatography column |
| Casein (5% in water, 50 mg/mL) | Sigma-Aldrich | C4765-10ML | |
| ChemiDoc system | Bio-Rad | N/A | |
| Clips | N/A | N/A | |
| Cutting board | N/A | N/A | |
| Dialysis tubing (3.5 kDa) | Fisher Scientific | 11425859 | |
| digoxigenin-11-dUTP | Roche | 11209256910 | |
| DNA loading dye 6x | NEB | B7024S | |
| dNTP 10 mM | NEB | N0447S | |
| Double-sided tape (0.14 mm thick) | N/A | N/A | |
| DTT | Fluorochem Limited | M02712-10G | |
| DYKDDDDK peptide | N/A | N/A | Synthetised by chemical biology STP of The Francis Crick Institute |
| EDTA | Thermo Fisher Scientific | D/0700/68 | |
| EGFP-RPA | N/A | N/A | Purified as desribed in ‘Single Molecule Analysis’, Series Methods in Molecular Biology, Vol. 783 (2011), Peterman, E. J. G. and Wuite G. J. L. editors, Humana Press. Protein information: 2 mM, human, expressed in E. coli. |
| EGTA | Sigma-Aldrich | 03779-10G | |
| Epoxy - 5 minute | Devcon | 20845 | |
| Fujifilm FLA-5000 | Fujifilm | FLA-5000 | Fluorescent image analyzer |
| GelRed Nucleic Acid Stain; 10000x | Cambridge Bioscience | 41003-BT | Example of nucleic acid stain which is a safer alternative to ethidium bromide. |
| Glass or quartz slide with holes for tubing | Dremel Model 395 | 1 mm thick slide, cut to 2.4 cm x 1cm, two holes drilled 1.4 mm apart. The holes should be drilled to different sizes to accommodate different tubing at each end. | |
| Glycerol | Fisher Scientific | G/0650/17 | |
| Glycine HCl, pH 3.5 | Sigma-Aldrich | G8898 | |
| Hamilton syringe, 1000 series GASTIGHT, PTFE luer lock 1010TLL, PTFE Luer lock (with slots), volume 10 mL | Merck | 26211-U | |
| HEPES pH 7.5 | Sigma-Aldrich | H4034 | |
| I-CeuI | NEB | R0699L | |
| KCl | Fisher Scientific | P/4280/53 | |
| Magnesium acetate | Sigma-Aldrich | M2545 | |
| Maxi GeBaFlex-tube Dialysis Kit; MWCO 14 kDa | Generon | D055 | |
| Metal spatula | N/A | N/A | |
| Metal tweezers | N/A | N/A | |
| MgCl2 | Sigma-Aldrich | M2393 | |
| Millex-GP (Syringe filters) 0.22 µm | Merck | SLGP033RS | |
| NaCl | Sigma-Aldrich | S7653 | |
| Needle | N/A | N/A | |
| NotI-HF | NEB | R3189L | |
| NuPAGE 4%–12% Bis-Tris Protein Gels | Thermo Fisher Scientific | 10247002; | |
| NuPAGE MOPS SDS Running Buffer | Thermo Fisher Scientific | NP0001 | |
| Objective heater | Okolab | N/A | |
| PCR mix - 2x | Prepared from Phusion DNA polymerase (20 µL), 10 mM dNTPs (40 µL), 5x Phusion HF buffer (400 µL) and water (540 µL). | ||
| Peptide NH2-CHHHHHHHHHHLPETGG-COOH, labelled with LD655-MAL | N/A | N/A | peptide NH2-CHHHHHHHHHHLPETGG-COOH, labeled with LD655-MAL (Lumidyne Technologies) on the cysteine residue, was synthetised and purified by the peptide chemistry STP of The Francis Crick Institute |
| pGC261 plasmid | N/A | N/A | https://doi.org/10.1016/j.cell.2018.10.053 |
| Phusion High-Fidelity DNA Polymerase | NEB | M0530L | |
| Plastic tweezers | N/A | N/A | |
| Polyethylene tubing PE20 (inner diameter 0.015”, outer diameter 0.043”) | Becton Dickinson | 427406 | |
| Polyethylene tubing PE60 (inner diameter 0.03”, outer diameter 0.048”) | Becton Dickinson | 427416 | |
| Poly-Prep Chromatography Columns; 10 ml and 20 ml | Bio-Rad | 7311550 | |
| Potassium Glutamate (L-glutamic acid potassium salt monohydrate) | Sigma | G1501-1KG | |
| Protease inhibitor cocktail (cOmplete, EDTA free) | Roche | 5056489001 | |
| Pump 11 Elite Infusion/Withdrawal Programmable Single Syringe | Harvard Apparatus | 70-4504 | |
| QIAquick PCR Purification Kit | QIAGEN | 28104 | |
| Razor blade | VWR International Ltd | 233-0156 | |
| rCutSmart Buffer; 10x | NEB | B6004S | |
| Sodium acetate | Thermo Fisher Scientific | S/2120/53 | |
| Sortase (pentamutant) | N/A | N/A | Purified based on: https://doi.org/10.1073/pnas.1101046108 |
| Spin-X Centrifuge Tube Filters; 0.22 µm cellulose acetate | Fisher Scientific | 10310361 | |
| Sterile scalpel | N/A | N/A | |
| Steritop Vacuum Driven Disposable Filtration System; 0.22 um; PES; | Millipore | S2GPT05RE | |
| Streptavidin (1 mg/mL) in 1x PBS buffer | Sigma | S4762-10MG | 20 µL aliquotes |
| SYBR Gold | Thermo Fisher Scientific | S11494 | Highly sensitive nucleic acid stain we used to visualise DNA fork substrate. |
| SYTOX orange; 5 µM in DMSO | Thermo Fisher Scientific | S11368 | |
| T4 DNA ligase | NEB | M0202M | |
| T4 DNA Ligase Reaction Buffer | NEB | B0202S | |
| TEMED | Sigma-Aldrich | T9281-25ML | |
| TEV protease (1 mg/mL); EZCut | Biovision | 7847-10000 | |
| Tissue grinders, Dounce type (40 mL, Wheaton) | DWK Life Sciences | 432-1273 | |
| Tris base | Sigma-Aldrich | T1503 | Used to prepare Tris Buffer (pH 8) |
| Tris HCl | Sigma-Aldrich | T3253 | |
| Tween-20 | Promega UK Ltd | H5152 |
Referências
- Bell, S. P., Labib, K. Chromosome Duplication in Saccharomyces cerevisiae. Genetics. 203 (3), 1027-1067 (2016).
- Costa, A., et al. The structural basis for MCM2-7 helicase activation by GINS and Cdc45. Nat Struct Mol Biol. 18 (4), 471-477 (2011).
- Jones, M. L., Baris, Y., Taylor, M. R. G., Yeeles, J. T. P. Structure of a human replisome shows the organisation and interactions of a DNA replication machine. EMBO J. 40 (23), e108819(2021).
- Moyer, S. E., Lewis, P. W., Botchan, M. R. Isolation of the Cdc45/Mcm2-7/GINS (CMG) complex, a candidate for the eukaryotic DNA replication fork helicase. Proc Natl Acad Sci U S A. 103 (27), 10236-10241 (2006).
- Douglas, M. E., Ali, F. A., Costa, A., Diffley, J. F. X. The mechanism of eukaryotic CMG helicase activation. Nature. 555 (7695), 265-268 (2018).
- Ilves, I., Petojevic, T., Pesavento, J. J., Botchan, M. R. Activation of the MCM2-7 helicase by association with Cdc45 and GINS proteins. Mol Cell. 37 (2), 247-258 (2010).
- Dewar, J. M., Walter, J. C. Mechanisms of DNA replication termination. Nat Rev Mol Cell Biol. 18 (8), 507-516 (2017).
- Burnham, D. R., Kose, H. B., Hoyle, R. B., Yardimci, H. The mechanism of DNA unwinding by the eukaryotic replicative helicase. Nat Commun. 10 (1), 2159(2019).
- Kose, H. B., Larsen, N. B., Duxin, J. P., Yardimci, H. Dynamics of the eukaryotic replicative helicase at lagging-strand protein barriers support the steric exclusion model. Cell Rep. 26 (8), 2113-2125.e6 (2019).
- Kose, H. B., Xie, S., Cameron, G., Strycharska, M. S., Yardimci, H. Duplex DNA engagement and RPA oppositely regulate the DNA-unwinding rate of CMG helicase. Nat Commun. 11 (1), 3713(2020).
- Tanner, N. A., Van Oijen, A. M. Visualizing DNA replication at the single-molecule level. Methods Enzymol. 475, 259-278 (2010).
- Swoboda, M., et al. Enzymatic oxygen scavenging for photostability without ph drop in single-molecule experiments. ACS Nano. 6 (7), 6364-6369 (2012).
- Baris, Y., Taylor, M. R. G., Aria, V., Yeeles, J. T. P. Fast and efficient DNA replication with purified human proteins. Nature. 606, 204-210 (2022).
- Lewis, J. S., et al. Tunability of DNA polymerase stability during eukaryotic DNA replication. Mol Cell. 77 (1), 17-25.e5 (2020).
- Li, Z., Xu, X. Post-translational modifications of the mini-chromosome maintenance proteins in DNA replication. Genes. 10 (5), 331(2019).
- Lewis, J. S., et al. Mechanism of replication origin melting nucleated by CMG helicase assembly. Nature. 606 (7916), 1007-1014 (2022).
- Zeman, M. K., Cimprich, K. A. Causes and consequences of replication stress. Nat Cell Biol. 16 (1), 2-9 (2014).
- Sparks, J. L., et al. The CMG helicase bypasses DNA-protein cross-links to facilitate their repair. Cell. 176 (1-2), 167-181.e21 (2019).
- Taylor, M. R. G., Yeeles, J. T. P. The initial response of a eukaryotic replisome to DNA damage. Mol Cell. 70 (6), 1067-1080.e12 (2018).
- Amunugama, R., et al. Replication fork reversal during DNA interstrand crosslink repair requires CMG unloading. Cell Rep. 23 (12), 3419-3428 (2018).
- Wu, R. A., et al. TRAIP is a master regulator of DNA interstrand crosslink repair. Nature. 567 (7747), 267-272 (2019).
- Vrtis, K. B., Dewar, J. M., Chistol, G., Wu, R. A., Graham, T. G. W., Walter, J. C. Single-strand DNA breaks cause replisome disassembly. Mol Cell. 81 (6), 1309-1318.e6 (2021).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados