Method Article
Одномолекулярная визуализация раскручивания ДНК в реальном времени с помощью хеликазы CMG
В этой статье
Резюме
Этот протокол демонстрирует выполнение анализа одной молекулы для визуализации раскручивания ДНК с помощью хеликазы CMG в реальном времени. В ней описывается (1) получение субстрата ДНК, (2) очистка флуоресцентно меченой геликазы Drosophila melanogaster CMG, (3) подготовка микрофлюидной проточной ячейки для микроскопии полного внутреннего отражения (TIRF) и (4) анализ раскручивания одномолекулярной ДНК.
Аннотация
Точная дупликация генома имеет важное значение для сохранения генетической стабильности делящихся клеток. Репликация ДНК осуществляется во время S-фазы динамическим комплексом белков, называемым реплисомой. В основе репликазы лежит геликаза CDC45-MCM2-7-GINS (CMG), которая разделяет две нити двойной спирали ДНК таким образом, что ДНК-полимеразы могут копировать каждую цепь. Во время дупликации генома реплисомы должны преодолеть множество препятствий и проблем. Каждый из этих факторов угрожает стабильности генома, так как неспособность полностью и точно воспроизвести ДНК может привести к мутациям, заболеваниям или гибели клеток. Поэтому очень интересно понять, как CMG функционирует в репликсоме как при нормальной репликации, так и при репликационном стрессе. В данной работе мы описываем микроскопию полного внутреннего отражения (TIRF) с использованием рекомбинантных очищенных белков, которая позволяет в режиме реального времени визуализировать поверхностно привязанные растянутые молекулы ДНК отдельными комплексами CMG. Этот анализ обеспечивает мощную платформу для исследования поведения CMG на уровне отдельных молекул, позволяя напрямую наблюдать динамику геликаза с контролем условий реакции в режиме реального времени.
Введение
Репликация ДНК строго регулируется, так как клетка должна точно дублировать свой геном, чтобы предотвратить мутации, болезни или смерть. Репликация эукариотической ДНК осуществляется с помощью комплекса репликосом, который раскручивает родительскую ДНК и использует одноцепочечную ДНК (одноцепочечную ДНК) в качестве матрицы для синтеза новой ДНК. В фазе G1 каталитически неактивные двойные гексамеры MCM2-7 загружаются на двухцепочечную ДНК (дцДНК) в начале репликации1. В S-фазе комплексы MCM2-7 активируются путем связывания CDC45 иGINS2 с образованием 11-субъединичных комплексов CMG (CDC45, MCM2-7, GINS). Каждый CMG инициирует раскручивание ДНК в противоположных направлениях, образуя ядро, вокруг которого репсисома располагается вокруг3.
Два десятилетия назад геликаза CMG была впервые идентифицирована как комплекс из 11 субъединиц, необходимый для репликации ДНК4. С тех пор наше понимание CMG значительно продвинулось вперед, от загрузки и активации 5,6 до раскручивания и завершения ДНК7. Традиционные методы биохимической и структурной биологии сыграли решающую роль во многих из этих открытий; однако эти методы часто были ограничены в своих возможностях изучения более динамичных аспектов CMG. Методы с использованием одной молекулы используют физические манипуляции с отдельными биомолекулами для измерения или визуализации их активности по одной молекуле за раз. Это может быть использовано для получения представления о динамике белков в реальном времени, которые часто пропускаются или не обнаруживаются с помощью других методов 8,9.
В этой статье мы описываем микроскопию полного внутреннего отражения (TIRF) для визуализации раскручивания ДНК хеликазой CMG в режиме реального времени. Очищенный, флуоресцентно меченный CMG загружается на свободный 3'-конец длинной ДНК, содержащей предварительно созданную структуру ДНК-вилки. Линейная ДНК растягивается на покровном стекле биотин-ПЭГ в микрофлюидной проточной ячейке путем последовательного привязывания каждого конца ДНК к поверхности. Такой подход позволяет обеспечить более равномерное связывание ДНК, что значительно снижает вариации, которые необходимо учитывать при анализе данных. В присутствии АТФ-γ-ов, CMG загружается на одноцепочечную ДНК на 3'-конце вилки. АТФ-γ-s является медленно гидролизуемым аналогом АТФ, который позволяет ЦМГ связываться с ДНК, но не раскручиваться. Последующее добавление АТФ, наряду с очищенной, флуоресцентно меченной РПА, активирует CMG и инициирует обширное раскручивание ДНК. Визуально CMG перемещается вдоль ДНК, оставляя за собой растущий участок RPA-связанной одноцепочной ДНК. Несвязанный конец ДНК перемещается с CMG, образуя «плотный шар» из-за уплотнения, вызванного связыванием RPA. Конструкция проточной ячейки позволяет заменять буфер в любой момент во время размотки, обеспечивая отличный контроль во время и во время каждого эксперимента.
Этот протокол разделен на четыре метода, которые могут выполняться независимо друг от друга. В разделе 1 описывается приготовление 20-килобайтного линейного разветвленного субстрата ДНК для одномолекулярных анализов. В разделе 2 описывается очистка и флуоресцентное мечение Drosophila melanogaster CMG (DmCMG). Ключевая информация о выражении DmCMG включена в раздел примечаний. Раздел 3 посвящен подготовке микрофлюидной проточной ячейки, которую можно использовать на микроскопе TIRF. В разделе 4 описано, как провести анализ раскручивания одномолекулярной ДНК.
протокол
1. Получение линейной разветвленной ДНК размером 20 кб, используемой в одномолекулярных анализах (Рисунок 1)
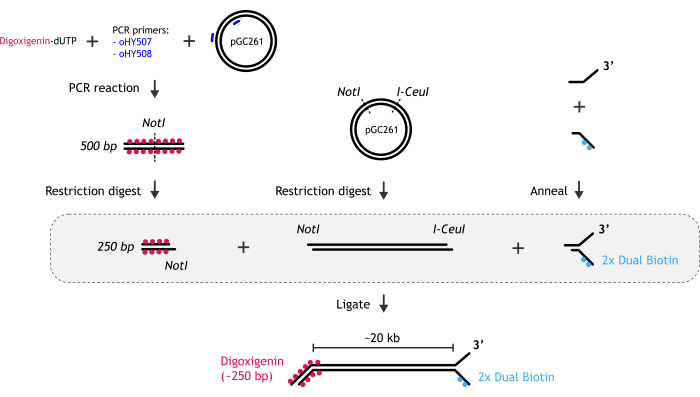
Рисунок 1: Графическое представление подготовки субстрата ДНК. (A) Биотинилированный конец вилки ДНК создается путем отжига двух частично комплементарных олигонуклеотидов: биотинилированного и небиотинилированного. (B) Основной фрагмент дцДНК (~20 kb) генерируется путем рестрикционного расщепления плазмиды pGC261 двумя ферментами для создания линейной ДНК с различными выступами на каждом конце. (C) Конец дуплексной ДНК дигоксигенина получают путем ПЦР-реакции, проводимой в присутствии дигоксигенина-dUTP, с последующим рестрикционным расщеплением. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
- Генерация биотинилированной ДНК на конце вилки.
- Отжигаем вилку ДНК.
- Смешайте два олигонуклеотида (oHY502 и oHYbio85) в 1x STE-буфере (100 мМ NaCl, 10 мМ Tris, pH 8,0; 1 мМ этилендиаминтетрауксусная кислота [ЭДТА]). Конечная концентрация олигонуклеотида составляет 10 мкМ, а конечный объем реакции — 100 мкл.
- Инкубируйте реакцию при 80 °C в течение 5 минут на тепловом блоке. Выключите нагревательный блок и дайте реакции постепенно остыть, оставляя его в тепловом блоке до тех пор, пока температура не упадет ниже 30 °C.
- Проверьте эффективность отжига геля для электрофореза (PAGE) в виде полиакриламидного геля Tris-Borate-EDTA (TBE).
- Приготовьте 8% акриламид - гель TBE (для 4 гелей: 32,8 мл H2O; 4,8 мл 10x буфера TBE (1 М Трис, 1 М борная кислота, 20 мМ ЭДТА), 9,6 мл 40% раствора акриламида/Бис, 37,5:1; 0,8 мл 10% персульфата аммония [APS], 40 μл тетраметилэтилендиамина [TEMED]).
ВНИМАНИЕ: КЭ, акриламид, APS, TEMED токсичны/вредны. - Пропустите небольшой объем (1 мкл) отожженных олигонуклеотидов вместе с отдельными олигонуклеотидами, разбавленными 6-кратным загрузочным красителем на 8% акриламид - геле TBE при 80 В, в течение 1 ч в режиме РТ.
- Замочите гель в 1x TBE, дополненном подходящим пятном для нуклеиновых кислот, на 30 минут на качающемся качателе, затем отсканируйте гель с помощью системы визуализации геля.
- Приготовьте 8% акриламид - гель TBE (для 4 гелей: 32,8 мл H2O; 4,8 мл 10x буфера TBE (1 М Трис, 1 М борная кислота, 20 мМ ЭДТА), 9,6 мл 40% раствора акриламида/Бис, 37,5:1; 0,8 мл 10% персульфата аммония [APS], 40 μл тетраметилэтилендиамина [TEMED]).
- Разрешите отожженную ДНК на геле TBE-PAGE для иссечения геля.
- Если олигонуклеотиды отжигаются правильно, растворите остальную часть образца в присутствии БСА (~100 мкл отожженного образца, 25 мкл 6-кратного загрузочного красителя, 2 мкл 33 мг/мл БСА). Чтобы нагрузить на гель больший объем, соедините несколько лунок стерильным скальпелем.
- Оставьте гель в 1x TBE, дополненный подходящим пятном для нуклеиновых кислот, на 30 минут на качающемся качающемся шейкере.
- Переложите гель в стеклянный лоток и визуализируйте ДНК в темной комнате под синим светом. При использовании синего света используйте подходящий защитный экран или защитные очки.
- Вырежьте нужную полосу стерильным скальпелем. Срежьте излишки геля.
- Очистите отожженную вилку электроэлюированием.
- Отрежьте кусок диализной трубки достаточно длинно, чтобы вставить кусочек геля, намочите его в 1x TBE и зафиксируйте один конец.
- Разведите БСА до 0,3 мг/мл в 500 мкл 1x TBE и нанесите пипеткой весь объем в трубку.
- С помощью металлического шпателя вставьте внутрь гелевый кусочек. Переместите кусочек геля на одну сторону трубки, чтобы освободить буферное пространство для миграции ДНК во время электроэлюирования.
- Отожмите лишний буфер и зафиксируйте второй конец трубки.
ПРИМЕЧАНИЕ: Оставшийся объем определяет окончательный объем и концентрацию. - Поместите диализную трубку в резервуар для агарозного электрофореза в буфере 1x TBE, убедившись, что трубка полностью погружена в воду.
ПРИМЕЧАНИЕ: Убедитесь, что кусочек геля расположен на стороне, ближайшей к отрицательному электроду, чтобы ДНК могла мигрировать из геля в мешок для диализа. - Запустите электроэлюирование при напряжении 80 В в течение 1-2 часов.
- Диализируйте электроэлюированную ДНК в 10 мМ трис-буфер (pH 8).
- После электроэлюирования достаньте диализный мешок из бака и высушите один конец салфеткой. Избегайте соприкосновения с тканью средней части трубки, где находится гель и образец.
- Снимите зажим с высушенного конца и осторожно проведите образец вверх и вниз внутри трубки, чтобы смешать ДНК с буфером.
- Опустите чистый шпатель в буфер 1x TBE и выньте гелевый кусочек из трубки.
ПРИМЕЧАНИЕ: Снимая гелевый кусочек, удалите как можно меньше буфера. - Снова зажмите открытый конец диализного пакета, удаляя воздух изнутри.
- Поместите диализный мешок в стакан объемом 2 л, наполненный 1,5 л 10 мМ трис-буфера (pH 8), 20 мМ NaCl и 2 мМ MgCl2. Оберните зажимы парапленкой, чтобы приклеить трубку к краю стакана, полностью погрузив трубку в буфер.
ПРИМЕЧАНИЕ: Соль стабилизирует короткие олигодуплексы. - С помощью магнитного стержня осторожно перемешайте буфер и диализируйте образец при комнатной температуре (RT) в течение не менее 3-4 часов или при 4 °C в течение ночи.
- После диализа высушите один конец диализного мешка салфеткой (чтобы не разбавить ДНК) и отсоедините этот конец.
- Аккуратно проводите пипеткой образец вверх и вниз внутри трубки и переложите его в чистую пробирку объемом 1,5 мл.
- Измерьте концентрацию ДНК с помощью микрообъемного спектрофотометра.
- Отжигаем вилку ДНК.
- Генерация фрагмента размером 20 КБ
- Провести рестрикционный расщепление pGC261 плазмиды18 с использованием ферментов NotI/I-CeuI.
- Аккуратно смешайте 8 мкл NotI-HF (20 000 ЕД/мл), 8 мкл I-CeuI (5 000 ЕД/мл) и плазмиду pGC261 (конечная концентрация плазмиды ~40 нг/мкл в реакции 200 мкл) в буфере, рекомендованном производителями ферментов рестрикции.
- Инкубируйте реакцию при 37 °C в течение ночи.
- Растворите расщепленную ДНК на агарозном геле для иссечения геля.
- ДОПОЛНИТЕЛЬНО: Сначала запустите небольшое количество реакции на 0,6% агарозном геле, чтобы проверить эффективность пищеварения, прежде чем загружать всю реакцию.
- Приготовьте 0,6% агарозный гель, следуя шагам 1.2.2.3-1.2.2.4.
- Смешайте 0,48 г порошка агарозы с 80 мл 1x TBE буфера. Разогрейте раствор в микроволновой печи, пока он не закипит. Перемешайте, чтобы агароза полностью расплавилась. Дайте остыть в течение нескольких минут, затем перелейте в предназначенную для этого емкость.
- Чтобы объединить лунки для размещения большего объема образца, заранее наклейте кусок ленты на несколько лунок соты.
- Как только гель застынет, аккуратно снимите расческу и долейте в бак 1x TBE буфер. Аккуратно перемешайте образец с ДНК-загружающим красителем и запустите гель при напряжении 120 В в течение 1 часа.
- Оставьте гель в 1x TBE, дополненный подходящим пятном для нуклеиновых кислот, на качающемся шейкере-качалке до тех пор, пока он не окрасится.
- Переложите окрашенный гель в стеклянный лоток и визуализируйте ДНК в темной комнате под синим светом.
- Вырежьте нужную полосу стерильным скальпелем. Срежьте излишки геля.
- Необязательно: Очистите нужный фрагмент с помощью электроэлюирования, как описано в шагах 1.1.4 и 1.1.5. Здесь исключите соль из диализного буфера, так как она нужна только для стабилизации короткого олигодуплекса.
- Провести рестрикционный расщепление pGC261 плазмиды18 с использованием ферментов NotI/I-CeuI.
- Генерация конца дуплексной ДНК дигоксигенина
- Проведите ПЦР-реакцию с дигоксигенином-dUTP (dig-dUTP) с использованием ДНК pGC261 в качестве матрицы.
- Смесь: 400 мкл воды, 8 мкл pGC261 (0,8 нг/мкл), 3,5 мкл праймера oHY507 (100 мкМ), 3,5 мкл праймера oHY508 (100 мкМ), 8 мкл dig-dUTP (1 мМ), 400 мкл 2x ПЦР-смеси (приготовленной из ДНК-полимеразы (20 μл), 10 мМ dNTP (40 μл), 5x высокоточного (HF) буфера (400 μл) и воды (540 μл).
- Запустите следующую реакцию в термоамплификаторе:
98 °C - 1 мин
30x: 98 °C - 20 с; 65 °C - 20 с; 72 °C - 30 с;
72 °C - 10 мин
4 °C - Удерживать
- Переваривание и очистка ПЦР-продукта, меченного дигоксигенином
- Очистите продукт ПЦР с помощью коммерческого набора для очистки ДНК.
- Смешайте 10 мкл NotI-HF (20 000 ЕД/мл) с продуктом ПЦР в буфере, рекомендованном поставщиком фермента (конечная концентрация ДНК ~50 нг/мкл в реакции ~200 μл). Инкубируйте реакцию при 37 °C в течение ночи.
- Очистите переваренную ДНК с помощью коммерческого набора для очистки ДНК.
- Проведите ПЦР-реакцию с дигоксигенином-dUTP (dig-dUTP) с использованием ДНК pGC261 в качестве матрицы.
- Соберите компоненты для создания субстрата ДНК.
- Аккуратно смешайте биотинилированный конец вилки, фрагмент ДНК объемом 20 кб и меченный дигоксигенином фрагмент ПЦР, обработанный Not-I, с 5 мкл ДНК-лигазы Т4 (400 000 ЕД/мл) в буфере, рекомендованном поставщиком фермента в реакции объемом 200 мкл. Однако, основываясь на добавленном фрагменте ДНК размером 20 кб (1-5 мкг), добавьте примерно в 100 раз больше молярного избытка как биотинилированного конца вилки, так и фрагмента ПЦР.
- Распределите реакцию в ПЦР-пробирки (по 50 мкл каждая) и инкубируйте при 16 °C в термоамплификаторе на ночь.
- Разрешите лигированный образец на 0,6% агарозном геле и очистите лигированную ДНК с помощью электроэлюирования, как описано в шагах 1.2.2 и 1.2.3.
- Храните мгновенно замороженную ДНК при температуре -80 °C.
2. Очистка Drosophila melanogaster CMG (Рисунок 2)

Рисунок 2: Очистка Drosophila melanogaster CMG из 4 л клеток Hi Five. Белки растворяли на 4%-12% полиакриламидном геле Bis-Tris при 200 В в присутствии буфера MOPS. Образец показывают на каждой стадии очистки (лизат клеток - 2 μл, элюирование FLAG - 10 μл, после первой ионообменной колонки - 10 μл, а после мечения и второй ионообменной колонки - 1 μл. (А) окрашивание Кумасси подтверждает наличие всех 11 субъединиц комплекса CMG до (10 μл), и после (1 μл) флуоресцентного мечения. (B) Эффективность мечения субъединицы MCM3 была проверена путем сканирования Cy5 с помощью флуоресцентного анализатора изображений с использованием длинночастотного красного фильтра (LPR). Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
Примечание: Для получения флуоресцентно меченого Drosophila melanogaster CMG сайт расщепления TEV (ENLYFQG), за которым следуют четыре остатка Gly, вводили после N-концевой метки FLAG на субъединице MCM3 (в векторе pFastBac1)10. Для экспрессии комплекса использовалась система экспрессии бакуловируса. Для первичной трансфекции клетки Sf21 использовали отдельно для каждой субъединицы CMG (стадия вируса P1). Для дальнейшей амплификации вирусов использовали клетки Sf9 (стадия вируса P2). Впоследствии клеточные культуры Sf9 (100 мл для каждой субъединицы CMG; 0,5 x 106 клеток/мл) инфицировали 0,5 мл вируса P2 с добавлением 10% сыворотки эмбрионального теленка (стадия вируса P3). Для экспрессии всего комплекса CMG в 4 л клеток Hi Five (1 x 106 клеток/мл) использовали по 200 мл вирусов P3 для каждой из субъединиц. После сбора клеток Hi Five, экспрессирующих комплекс CMG, гранулу клетки можно мгновенно заморозить в жидком азоте и хранить при температуре -80°C. Проведите всю очистку на льду или при температуре 4 °C. Буферы могут быть приготовлены заранее, при условии, что восстановители (DTT или 2-меркаптоэтанол) и ингибиторы протеазы (CAUTION) добавляются непосредственно перед использованием. Убедитесь, что все буферы предварительно охлаждены, отфильтрованы и дегазированы.
- Подготовьте следующие буферы.
- Приготовьте ресуспензионный буфер А, смешав 25 мМ 4-(2-гидроксиэтил)-1-пиперазинэтансульфоновую кислоту (HEPES) pH 7,5, 0,02% твин-20, 10% глицерин, 15 мМ KCl, 2 мМ MgCl2, 2 мМ 2-меркаптоэтанол, 1 мМ ЭДТА и 1 мМ этиленгликол-бис (β-аминоэтиловый эфир)-N,N,N',N'-тетрауксусную кислоту (EGTA).
ВНИМАНИЕ: 2-меркаптоэтанол, DTT и ЭДТА токсичны/вредны - Приготовьте трис-буферный физиологический раствор (TBS; 0,1 M Glycine HCl, pH 3,5) буфер для приготовления аффинного геля ANTI-FLAG M2.
- Приготовьте буфер A-100, смешав 25 мМ HEPES pH 7,5, 0,02% Tween-20, 10% глицерина, 100 мМ KCl, 1 мМ DTT, 1 мМ ЭДТА и 1 мМ EGTA.
- Приготовьте диализный буфер, добавив 25 мМ HEPES pH 7,5, 50 мМ ацетата натрия, 10 мМ ацетата магния, 10% глицерина и 1 мМ DTT.
- Приготовьте буфер TBS, смешав 50 мМ Tris-HCl pH 7,5 и 150 мМ NaCl.
- Приготовьте ресуспензионный буфер А, смешав 25 мМ 4-(2-гидроксиэтил)-1-пиперазинэтансульфоновую кислоту (HEPES) pH 7,5, 0,02% твин-20, 10% глицерин, 15 мМ KCl, 2 мМ MgCl2, 2 мМ 2-меркаптоэтанол, 1 мМ ЭДТА и 1 мМ этиленгликол-бис (β-аминоэтиловый эфир)-N,N,N',N'-тетрауксусную кислоту (EGTA).
- Ресуспендируйте пеллету из 4 л культуры клеток Hi Five в 200 мл холодного ресуспензионного буфера А с добавлением коктейльных таблеток ингибитора протеазы. Перемешайте трубки методом инверсии до тех пор, пока вся гранула не будет полностью суспендирована.
- Перенесите ресуспендированные клетки в предварительно охлажденный гомогенизатор Dounce объемом 40 мл. Чтобы восстановить как можно больше клеток, промойте пробирки, используемые для заморозки клеточных гранул, тем же типом буфера и добавьте в гомогенизатор Dounce. Следите за тем, чтобы общий объем в гомогенизаторе не превышал рекомендуемого предела. Лизируйте клетки на льду на 60-70 ударов.
ПРИМЕЧАНИЕ: Убедитесь, что поршень достигает дна трубки, но не оказывайте слишком большого давления, так как гомогенизатор может сломаться. Будьте осторожны и не вынимайте поршень выше уровня жидкости, так как это приведет к появлению пузырьков. - Повторяйте предыдущий шаг до тех пор, пока весь образец не будет гомогенизирован, и смешайте его в предварительно охлажденном стакане. Оцените окончательный объем пробы. По каплям добавляем отфильтрованный KCl до конечной концентрации 100 мМ, аккуратно перемешивая.
- Предварительно охладите центрифугу до 4 °C. Налейте образец в центрифужные пробирки, убедившись, что достигнут объем, рекомендованный производителем. Сбалансируйте трубки на весах.
ПРИМЕЧАНИЕ: Пробирки со слишком маленьким объемом могут сломаться во время центрифугирования. - Центрифугируйте лизированные клетки при давлении 23 500 x g в течение 15-30 мин при 4 °C. Тем временем приготовьте аффинитивный гель ANTI-FLAG M2 в соответствии со следующим шагом.
- Аккуратно перемешайте флакон, содержащий аффинный гель ANTI-FLAG M2 (50% суспензия). Отрежьте конец наконечника для пипетки P-100 (чтобы расширить кончик) и с его помощью немедленно перенесите 4 мл суспензии (2 мл гранул) в колонку для поли-препа объемом 20 мл. Чтобы восстановить застрявшие в наконечнике бусины, промойте наконечник с помощью TBS и добавьте его в столбик. Дайте всем бусинам осесть внутри колонны, но будьте осторожны, чтобы не высушить их.
- Промойте смолу 3x шариками объемом (всего 6 мл) 0,1 М Глицин HCl pH 3,5. Впоследствии промойте бусины объемом 3-5 бусин TBS (6-10 мл). Далее промойте бусины 3 раза (6 мл) Буфером А-100.
ПРИМЕЧАНИЕ: Не оставляйте шарики в 0,1 М растворе Glycine HCl pH 3,5 дольше 20 минут. Чтобы увеличить давление потока и скорость мойки, накройте колонку крышкой. Ни в коем случае не допускайте высыхания смолы. - После приготовления смолы оставьте 2 мл буфера А-100 над смолой и закройте выходное отверстие колонны.
- После центрифугирования аккуратно влейте надосадочную жидкость в предварительно охлажденные пробирки объемом 50 мл. Будьте осторожны, чтобы не нарушить гранулу. Возьмите небольшую аликвоту (~10 мкл) для последующей валидации с помощью электрофореза SDS-PAGE.
- Добавьте равное количество шариков ANTI-FLAG M2 (приготовленных в соответствии с предыдущим шагом) в каждую из пробирок объемом 50 мл. Попробуйте восстановить все бусины из колонки, повторно взвесив их парой дополнительных миллилитров Buffer A-100 и перенеся эту ресуспензию на трубки.
- Инкубировать образец (надосадочную жидкость) со смолой ANTI-FLAG M2, вращая в течение 2,5 ч при температуре 4 °C.
- После инкубации образец вращают при температуре 1 000 x g в течение 5 минут при 4 °C. С помощью пипетки удалите большую часть надосадочной жидкости. С помощью срезанного кончика суспендируйте бусины в нескольких миллилитрах оставшейся надосадочной жидкости и переложите их в две пробирки по 15 мл. Чтобы удалить шарики, прилипшие к стенкам пробирок объемом 50 мл, добавьте несколько миллилитров буфера А-100 и перенесите его в пробирки объемом 15 мл.
- Вращайте пробирки объемом 15 мл при температуре 1 000 x g в течение 5 минут при температуре 4 °C. Удалите надосадочную жидкость.
- Промойте шарики, добавив ~14 мл буфера A-100 в каждую пробирку, затем короткое, осторожное вращение при 4 °C и последующее вращение при 1000 x g в течение 5 минут при 4 °C. Повторите промывки дважды. Возьмите небольшие аликвоты для каждой промывки (~10 μл) для последующей валидации с помощью электрофореза SDS-PAGE.
- Этот шаг (2.16) является процедурой, альтернативной предыдущим шагам (2.13-2.15).
- После инкубации со смолой ANTI-FLAG M2 вылейте образец в две поли-преп-хроматографические колонки объемом 20 мл.
- Если вы выбираете этот метод, пусть бусины осядут на дне колонны, а несвязанный образец пройдет через колонну под действием силы тяжести. Затем добавьте буфер А-100 непосредственно в колонку и дайте ему промыть образец под действием силы тяжести.
- Перенесите шарики (ресуспендированные в буфере А-100) в две 10-мл поли-подготовительные хроматографические колонки.
- Дайте буферу A-100 пройти через смолу, закройте выпускное отверстие, когда уровень буфера достигнет верхней части смолы, и вымывайте белок из гранул, добавив буфер A-100 с добавлением пептида 200 мкг/мл (DYKDDDDK) (буфер для элюирования FLAG).
- Для первого элюирования добавьте 3 мл элюирующего буфера FLAG и осторожно вращайте закрытую колонку в течение 15 минут при температуре RT. Откройте выпускное отверстие, чтобы собрать первую фракцию элюирования.
- Закройте розетку и добавьте по 2 мл элюирующего буфера FLAG в каждую колонку. Вращаем еще 10 минут и собираем вторую фракцию элюирования.
- Соедините все элюированные фракции вместе (~10 мл) и держите при температуре 4 °C. Возьмите небольшую аликвоту элюированной фракции (~10 μл) для последующей валидации с помощью электрофореза SDS-PAGE.
- Отфильтруйте элюированный образец с помощью шприцевых фильтров 0,22 мкм.
- Заранее подготовьте колонку CaptoHiRes Q (5/50) (колонку ионообменной хроматографии высокого разрешения), подключенную к системе очистки белка, в соответствии с протоколом производителя. Затем уравновесьте колонку с помощью буфера А-100 и загрузите отфильтрованную пробу в уравновешенную колонку.
- Промойте колонку 20 объемами колонки (CV) Buffer A-100 (всего ~20 мл, так как объем колонки составляет ~1 мл).
- Для элюирования белка заранее приготовьте два буфера: Буфер А-100 и Буфер В с тем же составом, что и Буфер А-100, но с 550 мМ KCl вместо 100 мМ KCl. Настройте градиентное элюирование на 20 CV (~20 мл) с возрастающей концентрацией соли со 100 мМ до 550 мМ KCl. Соберите 0,3-0,5 мл элюирования в сборнике фракций.
ПРИМЕЧАНИЕ: CMG должен разбавляться при концентрации около 70%-75% буфера B. Возьмите небольшие аликвоты промывочной и элюированной фракций для электрофореза SDS-PAGE. - Проведите электрофорез SDS-PAGE, чтобы подтвердить, что CMG присутствует в выбранных фракциях. Тем временем очистите колонну и систему очистки в соответствии с протоколом производителя.
- Инкубируйте выбранные фракции с протеазой TEV в течение ночи, смешав 50 мкл протеазы TEV (1 мг/мл) на 1 мл образца.
- Предварительно смочите диализную трубку и убедитесь, что мембрана не повреждена. Добавьте смесь CMG/TEV в диализную трубку и поместите в стакан объемом 2 л с 1,5 л предварительно охлажденного буфера A-100 и магнитным стержнем внутри. Диализируйте при слабом помешивании в течение ночи при температуре 4°C.
- Смешайте образец с 50 мкМ пептида, меченного LD655, и 10 мкг/мл фермента сортазы в присутствии 5 мМ CaCl2. Инкубируйте реакцию при температуре 4 °C, вращая в течение 30 минут, следя за тем, чтобы пробирка была закрыта от света. Возьмите небольшую аликвоту до и после маркировки для электрофореза SDS-PAGE.
- Перед загрузкой в систему очистки отфильтруйте маркированный образец с помощью центрифужных фильтров 0,22 мкм, так как пептид может выпасть в осадок.
- Заранее подготовьте колонку ионообменной хроматографии с высоким разрешением, как описано ранее (шаг 2.20). На этот раз накройте колонну и систему сбора фракций алюминиевой фольгой, чтобы защитить пробу от света.
- Чтобы удалить свободный пептид, загрузите отфильтрованный образец в колонку ионообменной хроматографии высокого разрешения. Проведите очищение так же, как описано ранее (шаги 2.21-2.22). Убедитесь, что меченый CMG элюируется при концентрации буфера-B, аналогичной той, которая была ранее.
- Выполните электрофорез SDS-PAGE для проверки качества выбранных фракций. Чтобы получить изображение флуоресценции, не кипятите образцы перед загрузкой и убедитесь, что резервуар для электрофореза защищен от света. Сначала визуализируйте флуоресценцию белка с помощью системы визуализации геля, затем окрасьте гель красителем Кумасси и снова визуализируйте его, чтобы визуализировать все белки.
- Выбранные фракции диализируют в течение ночи при температуре 4 °C, против 1,5 л диализного буфера. При необходимости сконцентрируйте образец.
- Мгновенно заморозьте белок в жидком азоте и храните его при температуре -80 °C до дальнейшего использования.
3. Подготовка проточной ячейки (Рисунок 3)
- Приготовьте покровные листы биотин-ПЭГ в соответствии с ранее описанным протоколом11, опуская этап выпечки в духовке. Покровные стекла Biotin-PEG стабильны не менее 1 месяца под вакуумом при RT. Для приготовления проточной ячейки разрежьте покровное стекло биотин-ПЭГ (24 мм x 60 мм) пополам (примерно 24 мм x 30 мм).
ПРИМЕЧАНИЕ: Будьте осторожны, чтобы не касаться центральной части покровного стекла, где будут находиться проточные каналы. - Подготовьте небольшие стеклянные кусочки путем травления и защелкивания предметного стекла на кусочки размером примерно 2,4 см x 1 см.
- Просверлите два отверстия на расстоянии 1,4 мм друг от друга с помощью алмазного сверла диаметром 0,8 мм, одно немного шире другого (0,043 дюйма = вход, 0,048 дюйма = выход). Убедитесь, что отверстия имеют правильный размер, попытавшись вставить кусок входной/выходной трубки, используя то же сверло, чтобы расширить отверстие до тех пор, пока трубка не войдет. Убедитесь, что трубка плотно прилегает достаточно плотно, чтобы не выпасть легко.
ПРИМЕЧАНИЕ: Более узкая впускная трубка используется для минимизации мертвого объема. - Нарежьте двухсторонним скотчем той же формы, что и стеклянные кусочки.
- Выровняйте предметное стекло на ленте и проколите иглой каждое отверстие, чтобы отметить их положение на ленте. С помощью лезвия бритвы вырежьте канал, охватывающий оба отверстия.
ПРИМЕЧАНИЕ: Не режьте канал слишком длинным, так как избыточное пространство на дальней стороне каждого отверстия может оставаться сухим при попадании жидкости и может привести к проблемам с пузырьками воздуха во время потока. При резке старайтесь обрезать длинные края за один чистый разрез, так как изношенные края от нескольких разрезов могут повлиять на качество проточного канала. С аналогичной целью не врезайтесь в края проточного канала, так как разрезы в пригодной для использования детали могут повлиять на поток жидкости или даже привести к утечке в собранной проточной ячейке. - Очистите стеклянный кусок ацетоном и салфеткой до полного высыхания, затем положите на чистую поверхность. Отклейте одну сторону клейкой ленты и приклейте ее к стеклянному кусочку таким образом, чтобы оба отверстия полностью находились внутри канала.
- Приклейте ленту к стеклянному изделию с помощью наконечника для пипетки p1000 для устранения пузырьков, прижимая поверхность с сильным, но умеренным давлением. Пройдитесь по всей поверхности для герметизации.
- Снимите второй край ленты с обоих кусочков стекла и положите их на чистую поверхность липкой стороной вверх. Расположите предметные стекла близко друг к другу, но не соприкасаясь в том месте, где они будут прикреплены к покровному стеклу (оставьте между ними достаточно места, чтобы эпоксидная смола могла создать полное уплотнение вокруг каждого стеклянного элемента).
- Возьмите половинку покровного стекла биотин-ПЭГ с помощью пластикового пинцета, придерживаясь только за край (не касайтесь участка, который будет формировать канал потока), и опустите функционализированную сторону ПЭГ на клей. Закрепите, надавливая пальцем, чтобы зафиксировать его на месте, затем завершите уплотнение, умеренно сильно потерев поверхность наконечником пипетки, чтобы удалить пузырьки воздуха (не в области проточного канала, так как это может сломаться), затем переверните.
ПРИМЕЧАНИЕ: Обращайтесь с чехлом осторожно. - Для каждой проточной ячейки отрежьте ~10 см каждого типа полиэтиленовых трубок (PE20 и PE60). Используйте более узкую трубку в качестве входного отверстия в проточную камеру, чтобы уменьшить мертвый объем.
- Вставьте трубку в отверстия в предметном стекле вручную. Если диаметр отверстий правильный, убедитесь, что трубка стоит в отверстии самостоятельно после вставки. Разрежьте кончик трубки под небольшим углом <45°, чтобы немного облегчить вставку трубки и предотвратить образование уплотнения трубки против покровного стекла, что может произойти, если конец плоский (это может затруднить поток).
ПРИМЕЧАНИЕ: Точная посадка трубки в стеклянную деталь делает конструкцию проточной ячейки более надежной и помогает избежать попадания пузырьков в проточную ячейку. - Хорошо перемешайте компоненты эпоксидной смолы, затем с помощью наконечника p200 промокните эпоксидную смолу, чтобы герметизировать трубку и создать уплотнение вокруг каждого стеклянного элемента. Добавьте достаточное количество эпоксидной смолы, чтобы она доходила до края покровного стекла круглая, так как это также усилит деликатный покровный лист. Однако убедитесь, что эпоксидная смола не попала на нижнюю сторону чехла, так как это может помешать ему лежать на сцене.
ПРИМЕЧАНИЕ: При необходимости небольшое количество излишков эпоксидной смолы можно соскоблить с нижней стороны с помощью бритвенного лезвия. - Оставьте минимум на 30-60 минут, чтобы эпоксидная смола полностью застыла.
ПРИМЕЧАНИЕ: Если проточную ячейку нельзя использовать сразу, храните ее в вакууме при RT после полного отверждения. - После использования проточной ячейки соберите осколки стекла и используйте их повторно в течение неограниченного времени. Вытащите трубку и поместите проточную ячейку в банку с ацетоном не менее чем на 24 часа, чтобы размягчить эпоксидную смолу и двусторонний клей, что позволит легко удалить стеклянные осколки. Перед повторным использованием для изготовления еще одной проточной ячейки очистите стеклянные элементы, протерев их губкой и мыльной водой, высушите их и потрите ацетоном и папиросной бумагой.

Иллюстрация 3: Графическое изображение подготовки проточной ячейки. (A) Отрежьте двустороннюю ленту в соответствии с размером стеклянной детали. Выровняйте горку поверх ленты и отметьте иголкой положение каждого отверстия. С помощью лезвия бритвы разрежьте вокруг каждого зацепа, чтобы создать канал. (B) Отклейте одну сторону ленты и приклейте ленту к стеклянной части. Убедитесь, что оба отверстия находятся внутри канала. Снимите второй конец ленты и приклейте сверху покровное стекло биотин-ПЭГ. (C) Вставьте полиэтиленовую трубку в каждое отверстие и запечатайте трубку на месте эпоксидной смолой, приклеив каждую стеклянную деталь к покровному стеклу. (D) После использования обоих каналов вытащите трубку и поместите проточную ячейку в банку для окрашивания, наполненную ацетоном. Примерно через 24 часа эпоксидная смола и лента станут мягкими, и слои проточной ячейки можно будет отделить. Кусочки стекла могут быть восстановлены и храниться в ацетоне для повторного использования в течение неограниченного срока для изготовления следующей проточной ячейки. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
4. Анализ одной молекулы TIRF для визуализации CMG-опосредованного раскручивания ДНК
- Подготовьте следующие буферы.
- Приготовьте блокирующий буфер (20 мМ Трис, pH 8,0; 50 мМ NaCl; 2 мМ ЭДТА; 0,2 мг/мл BSA). Отфильтруйте буфер с помощью шприцевого фильтра 0,22 мкм и храните его при температуре 4 °C.
- Приготовьте 10x Реакционный буфер 1 (250 мМ HEPES pH 7,5; 50 мМ NaCl; 100 мМ ацетата магния). Отфильтруйте буфер с помощью шприцевого фильтра 0,22 мкм и храните его при температуре 4 °C.
- Приготовьте 10x Реакционный буфер 2 (250 мМ Трис, pH 7,5; 100 мМ ацетата магния; 1,25 М глутамата калия; 1 мМ ЭДТА; 0,025% Tween-20). Отфильтруйте буфер с помощью шприцевого фильтра 0,22 мкм и храните его при температуре 4 °C.
- Дегазируйте примерно 2,5 мл блокирующего буфера и 5 мл сверхчистой воды, поместив открытые пробирки в эксикатор и оставив их под вакуумом на 15 минут.
ПРИМЕЧАНИЕ: Не заполняйте трубки до краев, так как пузырьки во время дегазации могут привести к разбрызгиванию жидкости. - Поместите проточную ячейку на предметный столик для микроскопа и закрепите ее на месте с помощью клеевой шпаклевки с каждого конца. Важно хорошо закрепить, чтобы предотвратить смещение проточной ячейки позже во время захвата.
- Подсоедините выходную трубку проточной ячейки к шприцевому насосу, трубка которого прикреплена к шприцу с помощью иглы на другом конце. Вставьте эту иглу в выпускную трубку, чтобы подсоединить проточную ячейку.
- Включите обогреватель объектива на 30 °C.
- Закрепите 1-2 мл дегазированной воды в трубке на отдельном куске клеевой шпаклевки рядом с проточной ячейкой таким образом, чтобы входная трубка могла быть вставлена и достигать дна трубки.
- Пропустите воду через канал, затем используйте более быстрый поток, чтобы удалить все застрявшие пузырьки возле входной трубки, при необходимости добавив больше воды. В противном случае крупные пузырьки могут сместиться позже в экспериментах и пройти через канал. Оставьте на несколько минут, чтобы поток полностью прекратился, так как быстрый поток может вызвать остаточное вытягивание жидкости в течение некоторого времени после его остановки, так как давление внутри шприца стабилизируется.
ПРИМЕЧАНИЕ: Любая ДНК, которая контактирует с пузырем, становится непригодной для использования, даже после регидратации. - Добавьте 100 мкл дегазированного блокирующего буфера к 20 мкл аликвоты 1 мг/мл стрептавидина. Приложите открытую трубку к клеевой замазке и перенесите входную трубку из воды в стрептавидин. Расходуйте со скоростью 40 мкл/мин в течение 2 мин (всего 80 мкл) и инкубируйте в течение 5 мин.
- Промойте избыток стрептавидина с помощью блокирующего буфера (50 мкл/мин для 100 мкл).
- Поток в биотинилированной ДНК, разведенной в блокирующем буфере с 25 нМ оранжевым SYTOX. Изображение с помощью лазера с длиной волны 532 нм позволяет наблюдать за тем, как ДНК привязывается к поверхности в режиме реального времени.
- Когда приблизительная плотность ДНК на поверхности достигнута, влейте в блокирующий буфер, также с 25 нМ SYTOX (50 мкл/мин для 100 мкл), чтобы вымыть свободную ДНК.
- Поток биотинилированного антидигоксигенинового антитела (~10 мкг/мл), разведенного в блокирующем буфере, содержащем 25 нМ оранжевого SYTOX (100 мкл/мин для 300 мкл).
Примечание: Оранжевый цвет SYTOX интеркалируется между парами оснований ДНК, увеличивая длину контура. Это означает, что длина ДНК SYTOX оранжевого цвета увеличивается от конца до конца, что позволяет второму тросу прикрепляться к поверхности дальше от первого, обеспечивая лучшую растянутость ДНК, когда оранжевый SYTOX вымывается позже. Использование разной части на каждом конце ДНК обеспечивает более равномерное связывание ДНК с поверхностью покровного стекла (Рисунок 4). Когда оба конца биотинилированы, они привязываются на одном и том же этапе, что приводит к значительным изменениям в положениях их крепления. Использование дигоксигенина на одном конце дает больший контроль над покрытием ДНК перед связыванием второго конца. Это также позволяет увеличить покрытие ДНК за счет инкубации. - Промойте биотинилированное антитело к дигоксигенину и оранжевый SYTOX с блокирующим буфером (50 мкл/мин для 100 мкл).
- Приготовьте 120 мкл «смеси АТФ-г-с» (1x Реакционный буфер 1, 0,75 мг/мл БСА, 1,25 мг/мл казеина, 8 мМ DTT, 0,33 мМ АТФ-г-с), добавьте 30 мкл в новую пробирку, затем слейте 50 мкл из исходной пробирки в проточную ячейку (50 мкл/мин для 50 мкл). Это помогает свести к минимуму эффект буферного смешивания за счет обратного потока или диффузии из выпускной трубки.
- Добавьте очищенный CMG до ~100 нМ в 30 мкл смеси АТФ-г-с, затем влейте со скоростью 20 мкл/мин в течение 20 мкл. Инкубируйте в течение 15 мин.
- Приготовьте 120 мкл «смеси АТФ/РПА» (1x Реакционный буфер 1, 0,75 мг/мл БСА, 8 мМ DTT, 3,3 мМ АТФ, 20 нМ EGFP-hRPA) и влейте в проточную ячейку со скоростью 40 мкл/мин для 80 мкл.
- Немедленно приступайте к получению изображений. Получайте поля зрения 6 x 6 для каждого кадра каждые 30 с (или подходящую частоту кадров для эксперимента). Более низкая частота кадров может уменьшить воздействие лазера и фотообесцвечивание флуорофора. Визуализируйте EGFP-hRPA с помощью лазера с длиной волны 488 нм при мощности 1%. Если CMG помечена, например, LD655, визуализируйте CMG с помощью лазера с длиной волны 640 нм (максимальная мощность = 30 мВт) при мощности 10%. Визуализируйте окрашенную в оранжевый цвет ДНК SYTOX с помощью лазера с длиной волны 532 нм при мощности 0,5%. Используйте каждый лазер с экспозицией 50-100 мс.
ПРИМЕЧАНИЕ: Каждый раз, когда входная трубка проточной ячейки переносится из одной пробирки с жидкостью в другую, промокните кончик трубки на дне пробирки несколько раз, прежде чем извлечь его из жидкости. Это помогает предотвратить попадание пузырьков во время перемещения. Сведите к минимуму время, в течение которого наконечник входной трубки удерживается жидкостью, и не направляйте наконечник вверх и не поднимайте наконечник значительно, так как это приводит к стеканию жидкости в сторону канала и втягиванию воздуха в наконечник. При переносе входной трубки старайтесь не натягивать трубку, так как это может вытянуть ее из эпоксидной смолы, закрепив ее на проточной ячейке. Для визуализации раскручивания ДНК в присутствии оранжевого SYTOX для визуализации двухцепочечной ДНК замените 10x Реакционный буфер 1 на 10x Реакционный буфер 2 для смеси АТФ/РПА. В этих условиях оранжевый SYTOX лучше связывается с ДНК. Полностью опорожняйте шприцевую помпу в конце каждого эксперимента. Время от времени мойтесь, наполняя водой и опорожняя несколько раз или разбирая и стирая мыльным раствором.

Рисунок 4: Привязка ДНК к поверхности. (А) При связывании субстратов ДНК с биотином на обоих концах расстояние между двумя тросами может варьироваться в зависимости от того, как концы соприкасаются с поверхностью (i). Используя дигоксигенин на одном конце, привязка каждого конца может быть временно разделена для получения более постоянных расстояний между связями и более равномерно растянутой ДНК (ii). (B) Пример поля зрения, показывающий ДНК, привязанную с обоих концов (меченую дигоксигенином) и окрашенную флуоресцентным интеркалирующим окрашиванием нуклеиновой кислоты. ДНК, которая связана обоими концами, выглядит как линия, в то время как ДНК, связанная только одним концом, выглядит как пятна. В идеале ДНК должна быть связана как можно плотнее, не перекрывая другую ДНК. Изображение имеет размер 512 x 512 пикселей (размер пикселя = 154,6 нм). Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
Результаты
Когда CMG раскручивает ДНК, характерный тракт RPA со временем будет расти (рис. 5). 5'-конец раскрученной ДНК привязан к поверхности; следовательно, он рассматривается как линейный участок сигнала RPA между тросом и вилкой. 3'-конец не привязан и, следовательно, движется вместе с вилкой и наблюдается как компактный сигнал EGFP-RPA. Положение уплотненной размотанной транслокационной нити примерно соответствует положению репликационной вилки, которая перемещается вместе с LD655-CMG, визуализируемым с помощью лазера с длиной волны 640 нм.
Важно свести к минимуму повреждение субстрата ДНК, так как такие повреждения, как одноцепочечные дефекты ДНК, уменьшают количество наблюдаемых событий раскручивания, ограничивая объем данных, которые могут быть собраны (рис. 6).
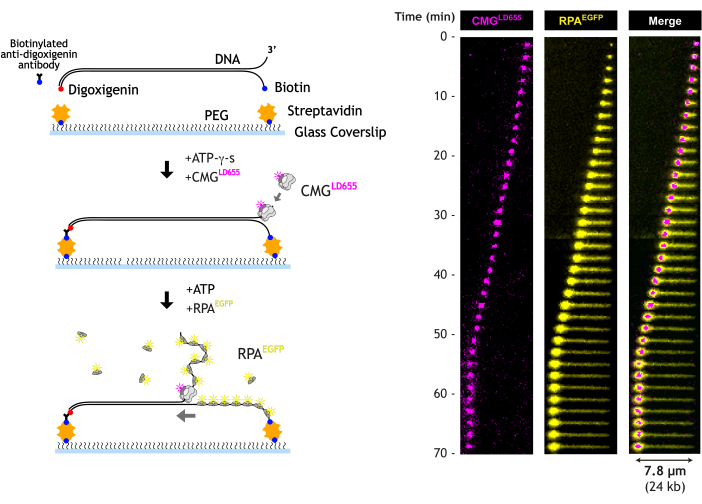
Рисунок 5: Анализ раскручивания одномолекулярной ДНК. Субстрат ДНК привязан к покровной поверхности. Очищенный CMG, меченный LD655, инкубируют с ДНК в течение 15 мин в АТФ-g-s. Добавляются АТФ и очищенная EGFP-меченая RPA, что инициирует обширное раскручивание ДНК CMG. Показана мультяшная схема (слева) и кимография репрезентативных данных (справа). Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
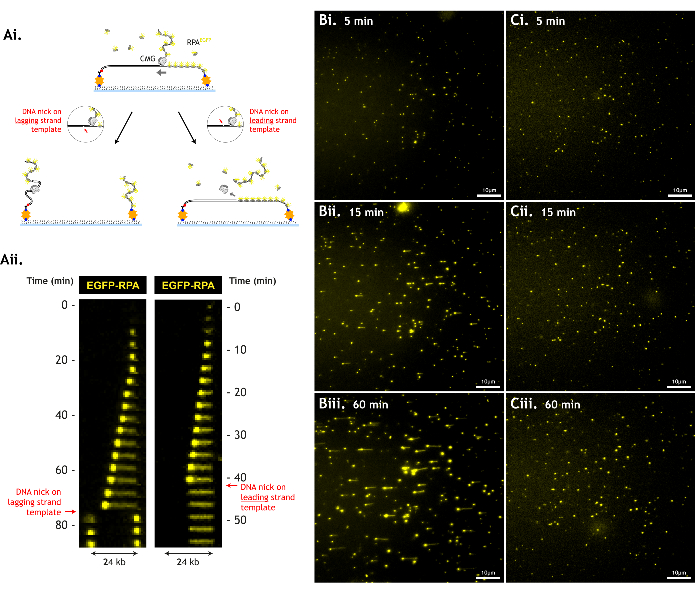
Рисунок 6: Повреждение ДНК снижает производительность анализа. (A) CMG не может раскручивать ДНК после разрыва в костяке ДНК (DNA nick). Зазубрины на матрице ведущей цепи приводят к соскальзыванию CMG с ДНК, и обе матрицы CMG и ведущей цепи теряются. Зазубрин на шаблоне отстающей цепи приводит к тому, что шаблон отстающей цепи отделяется от остальной части ДНК, и каждый фрагмент ДНК втягивается к соответствующему тросу. Это иллюстрируется (i) мультяшными схемами и (ii) кимографиями этих событий; (ii). Репрезентативные данные с (В) минимально поврежденным субстратом ДНК по сравнению с (В) более поврежденным субстратом ДНК через (i) 5 мин, (ii) 15 мин и (iii) 60 мин в одном поле зрения. Более поврежденный субстрат ДНК не генерирует длинных отрезков разматывания, как СМГ сталкиваются с зазубринами ранее, несмотря на схожие уровни активности разматывания (одинаковая плотность растущих пятен RPA через 5 минут, что указывает на аналогичную эффективность загрузки/размотывания КМГ). Поле зрения составляет 512 x 512 пикселей (размер пикселя = 154,6 нм). Мощность лазера 1% (488 нм) для визуализации EGFP-RPA. Масштабная линейка 10 мкм. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Обсуждение
Этот анализ обеспечивает платформу для наблюдения и исследования динамики отдельных CMG в режиме реального времени, как изолированно, так и в контексте желаемых дополнительных факторов. Однако, как и во многих методах флуоресценции с использованием одиночных молекул, существуют некоторые общие проблемы, для преодоления которых может потребоваться оптимизация. Обычно они связаны с визуализацией флуорофоров в течение длительных периодов времени (фотообесцвечивание, яркость), подготовкой субстрата ДНК (повреждение ДНК), качеством поверхности проточной ячейки (фоновый шум, неспецифические взаимодействия) или качеством очищенного белкового препарата (загрязнение нуклеазами, эффективность мечения).
Каждый флуорофор различается по светостабильности и яркости, поэтому важно выбрать подходящую молекулу. При визуализации флуоресцентно меченных олигомерных белков, таких как RPA, можно использовать более низкую мощность лазера, так как многие флуорофоры будут возбуждаться в непосредственной близости, генерируя видимый сигнал. Для визуализации отдельных флуорофоров, например, CMG, меченных на одной субъединице, требуется более высокая мощность лазера, чтобы четко наблюдать флуорофор. Срок службы флуорофора может быть увеличен за счет минимизации лазерного воздействия, например, за счет уменьшения частоты получения изображений. Кроме того, возбуждение флуорофора приводит к образованию активных форм кислорода (АФК), которые могут способствовать фотообесцвечиванию. Включение системы очистки кислорода в буфер визуализации может продлить срок службы флуорофоров за счет устранения АФК. Тем не менее, некоторые системы поглощения кислорода могут влиять на pH12.
Что касается подготовки субстрата ДНК, крайне важно свести к минимуму повреждения ДНК, такие как зазубрины или одноцепочечные промежутки. Чрезмерное повреждение препятствует обширному раскручиванию ДНК, ограничивая объем данных, которые могут быть собраны. Повреждение может возникнуть в результате механического сдвига, чрезмерного нагрева, в результате загрязнения нуклеазами или АФК, образующихся во время визуализации. Сдвиг можно свести к минимуму, если бережно обращаться с образцом ДНК с помощью широкопроходных наконечников для пипетирования, медленного пипетирования и избегания перелистывания образца. Эффект АФК может быть сведен к минимуму либо за счет уменьшения лазерной нагрузки, либо за счет включения системы очистки кислорода в буфер изображения. После подготовки субстрата ДНК можно использовать коммерческие наборы для восстановления ДНК для восстановления повреждения перед проведением реакции раскручивания.
Эффективность раскручивания ДНК также зависит от чистоты и активности КМГ. Хорошей практикой является оценка чистоты образца после каждого этапа очистки с помощью электрофореза SDS-PAGE, чтобы определить, где необходима оптимизация. Если после заключительного этапа наблюдается слишком большое количество загрязняющих веществ, может быть полезно изменить объемы градиента соли, используемые для элюирования из колонки CaptoHiRes Q (5/50). Также очень важно удалить избыток флуоресцентного пептида, используемого для мечения белка, так как он может создать нежелательный фон на поверхности покровного стекла. Также важно избегать загрязнения нуклеазами, так как это может привести к разрушению субстрата ДНК. После эксперимента окрашивание оставшейся ДНК в оранжевый цвет SYTOX может быть хорошим способом проверить, значительно ли деградировала ДНК или нет. Определенный уровень повреждения ДНК неизбежен в ходе эксперимента, но значительные повреждения часто указывают на проблемное загрязнение нуклеазами.
Анализ также по своей сути ограничен разрешением дифракционно-ограниченных пятен, что требует, чтобы флуоресцентные белки находились на расстоянии сотен пар оснований (если не больше), чтобы различать их как отдельные. Это ограничивает детализацию, с которой можно наблюдать прогрессирование КМГ и взаимодействия.
Количество событий раскручивания, которые мы наблюдаем для каждого анализа, варьируется. Для успешного эксперимента мы ожидаем увидеть как минимум несколько RPA-трактов достаточной длины на поле зрения 512x512 пикселей (размер пикселя = 154,6 нм). В одном эксперименте можно визуализировать несколько полей зрения, что позволяет собирать больше данных при необходимости. Тракты не обязательно должны быть одинаковой длины или доходить до конца ДНК, чтобы быть полезными. Например, среднее расстояние до троса для каждого эксперимента можно определить, измерив длину окрашенной SYTOX ДНК перед добавлением CMG. Это может быть использовано для оценки того, сколько ДНК было раскручено для любого RPA-тракта (при условии, что ДНК достаточно для видимого перемещения вилки) путем преобразования расстояния от «пройдено μm» до «kb раскручено».
CMG проявляет разматывающую активность на различных субстратах ДНК, но важно обеспечить свободный 3'-конец ДНК на лоскуте polyT с углом не менее 30 нт, чтобы обеспечить посадочное место CMG10. Включение нескольких биотиновых фрагментов на вилке обеспечивает надежную поверхностную привязку. Остальная часть субстрата ДНК может быть перепроектирована множеством способов, например, с включением различных последовательностей ДНК, длины и химических модификаций. Конформация ДНК может быть изменена при использовании различных концентраций ацетата магния. При более высоких концентрациях (≥10 мМ) ацетата магния нить одноцеклеточной ДНК, покрытая RPA, уплотняется, что приводит к тому, что ДНК вытягивается за счет связывания RPA во время раскручивания. Это может быть полезно, так как предотвращает чрезмерное перемещение ДНК, позволяя более точно измерить положение CMG и прогрессию раскручивания. При низких концентрациях (~3 мМ) ацетата магния RPA-ssDNA остается расслабленной на протяжении всего процесса.
Описанный анализ на основе одной молекулы представляет собой платформу, которая может быть построена и модифицирована для исследования дальнейших аспектов репликации ДНК. Во время репликации ДНК CMG выступает в качестве ядра, вокруг которого собирается репсисома и ее компоненты. Таким образом, в этот анализ могут быть добавлены дополнительные очищенные белки, включая вспомогательные факторы, такие как TIMELESS, TIPIN и CLASPIN, для изучения их влияния на динамику CMG. Было показано, что эти белки влияют на скорость репликациивилок 13, но неясно, как они влияют на скорость раскручивания CMG. Поэтому было бы интересно исследовать, как различные реплисомные белки влияют на CMG с помощью этого анализа. Добавление ДНК-полимераз может дать лучшее представление о репликации ДНК, выходящей за рамки только раскручивания ДНК, как описано ранее на примере белков дрожжей14. Кроме того, очистка модифицированного CMG может обеспечить лучшее понимание того, как определенные мутации или посттрансляционные модификации влияют на активность геликазы15,16. Кроме того, проектирование различных субстратов ДНК может позволить изучать раскручивание ДНК методом CMG в различных условиях, имитирующих репликационный стресс17. Эти модификации включают препятствия 9,18 ДНК, межцепочечные сшивки 19,20,21 и разрывы в цепяхДНК 22.
Раскрытие информации
Авторы не имеют конкурирующих финансовых интересов или других конфликтов интересов.
Благодарности
Мы благодарим Георге Кистола за предоставление плазмиды pGC261 и Центр химической биологии Института Фрэнсиса Крика за синтез и мечение пептидов. Эта работа финансировалась Институтом Фрэнсиса Крика, который получает основное финансирование от Cancer Research UK, UK Medical Research Council и The Wellcome Trust (CC2133).
Материалы
| Name | Company | Catalog Number | Comments |
| 2-Mercaptoethanol | Sigma-Aldrich | M3148-25ML | |
| Acrylamide / Bis Solution 40% | BioRad | 1610148 | |
| Agarose UltraPure | Invitrogen | 16500-500 | Used to prepare Tris Buffer (pH 8) |
| ÄKTA Pure | GE Healthcare | - | Used with Fraction Collector F9-C; protein purification system |
| ANTI-FLAG M2 affinity gel | Sigma-Aldrich | A2220 | |
| APS 10% | Sigma-Aldrich | A3678-25G | |
| ATP (200 mM) | Sigma-Aldrich | A7699-5G | |
| ATP-g-s (100 mM) | Sigma-Aldrich | 11162306001 | |
| Biotin-PEG coverslip | N/A | N/A | Prepared as described: https://doi.org/10.1016/j.ymeth.2012.03.033 |
| Biotinylated anti-digoxigenin antibody | Perkin Elmer | N/A | |
| Blu Tack | Bostik | N/A | Adhesive putty |
| Boric acid | Thermo Fisher Scientific | B/3800/53 | |
| BSA; 33 mg/mL | SIGMA-ALDRICH | A3858-10G | Diluted from the stock |
| CaCl2 | Sigma-Aldrich | C7902 | |
| CaptoHiRes Q (5/50) | Cytiva | 29275878 | Connected to the AKTA protein purification system; high-resolution ion exchange chromatography column |
| Casein (5% in water, 50 mg/mL) | Sigma-Aldrich | C4765-10ML | |
| ChemiDoc system | Bio-Rad | N/A | |
| Clips | N/A | N/A | |
| Cutting board | N/A | N/A | |
| Dialysis tubing (3.5 kDa) | Fisher Scientific | 11425859 | |
| digoxigenin-11-dUTP | Roche | 11209256910 | |
| DNA loading dye 6x | NEB | B7024S | |
| dNTP 10 mM | NEB | N0447S | |
| Double-sided tape (0.14 mm thick) | N/A | N/A | |
| DTT | Fluorochem Limited | M02712-10G | |
| DYKDDDDK peptide | N/A | N/A | Synthetised by chemical biology STP of The Francis Crick Institute |
| EDTA | Thermo Fisher Scientific | D/0700/68 | |
| EGFP-RPA | N/A | N/A | Purified as desribed in ‘Single Molecule Analysis’, Series Methods in Molecular Biology, Vol. 783 (2011), Peterman, E. J. G. and Wuite G. J. L. editors, Humana Press. Protein information: 2 mM, human, expressed in E. coli. |
| EGTA | Sigma-Aldrich | 03779-10G | |
| Epoxy - 5 minute | Devcon | 20845 | |
| Fujifilm FLA-5000 | Fujifilm | FLA-5000 | Fluorescent image analyzer |
| GelRed Nucleic Acid Stain; 10000x | Cambridge Bioscience | 41003-BT | Example of nucleic acid stain which is a safer alternative to ethidium bromide. |
| Glass or quartz slide with holes for tubing | Dremel Model 395 | 1 mm thick slide, cut to 2.4 cm x 1cm, two holes drilled 1.4 mm apart. The holes should be drilled to different sizes to accommodate different tubing at each end. | |
| Glycerol | Fisher Scientific | G/0650/17 | |
| Glycine HCl, pH 3.5 | Sigma-Aldrich | G8898 | |
| Hamilton syringe, 1000 series GASTIGHT, PTFE luer lock 1010TLL, PTFE Luer lock (with slots), volume 10 mL | Merck | 26211-U | |
| HEPES pH 7.5 | Sigma-Aldrich | H4034 | |
| I-CeuI | NEB | R0699L | |
| KCl | Fisher Scientific | P/4280/53 | |
| Magnesium acetate | Sigma-Aldrich | M2545 | |
| Maxi GeBaFlex-tube Dialysis Kit; MWCO 14 kDa | Generon | D055 | |
| Metal spatula | N/A | N/A | |
| Metal tweezers | N/A | N/A | |
| MgCl2 | Sigma-Aldrich | M2393 | |
| Millex-GP (Syringe filters) 0.22 µm | Merck | SLGP033RS | |
| NaCl | Sigma-Aldrich | S7653 | |
| Needle | N/A | N/A | |
| NotI-HF | NEB | R3189L | |
| NuPAGE 4%–12% Bis-Tris Protein Gels | Thermo Fisher Scientific | 10247002; | |
| NuPAGE MOPS SDS Running Buffer | Thermo Fisher Scientific | NP0001 | |
| Objective heater | Okolab | N/A | |
| PCR mix - 2x | Prepared from Phusion DNA polymerase (20 µL), 10 mM dNTPs (40 µL), 5x Phusion HF buffer (400 µL) and water (540 µL). | ||
| Peptide NH2-CHHHHHHHHHHLPETGG-COOH, labelled with LD655-MAL | N/A | N/A | peptide NH2-CHHHHHHHHHHLPETGG-COOH, labeled with LD655-MAL (Lumidyne Technologies) on the cysteine residue, was synthetised and purified by the peptide chemistry STP of The Francis Crick Institute |
| pGC261 plasmid | N/A | N/A | https://doi.org/10.1016/j.cell.2018.10.053 |
| Phusion High-Fidelity DNA Polymerase | NEB | M0530L | |
| Plastic tweezers | N/A | N/A | |
| Polyethylene tubing PE20 (inner diameter 0.015”, outer diameter 0.043”) | Becton Dickinson | 427406 | |
| Polyethylene tubing PE60 (inner diameter 0.03”, outer diameter 0.048”) | Becton Dickinson | 427416 | |
| Poly-Prep Chromatography Columns; 10 ml and 20 ml | Bio-Rad | 7311550 | |
| Potassium Glutamate (L-glutamic acid potassium salt monohydrate) | Sigma | G1501-1KG | |
| Protease inhibitor cocktail (cOmplete, EDTA free) | Roche | 5056489001 | |
| Pump 11 Elite Infusion/Withdrawal Programmable Single Syringe | Harvard Apparatus | 70-4504 | |
| QIAquick PCR Purification Kit | QIAGEN | 28104 | |
| Razor blade | VWR International Ltd | 233-0156 | |
| rCutSmart Buffer; 10x | NEB | B6004S | |
| Sodium acetate | Thermo Fisher Scientific | S/2120/53 | |
| Sortase (pentamutant) | N/A | N/A | Purified based on: https://doi.org/10.1073/pnas.1101046108 |
| Spin-X Centrifuge Tube Filters; 0.22 µm cellulose acetate | Fisher Scientific | 10310361 | |
| Sterile scalpel | N/A | N/A | |
| Steritop Vacuum Driven Disposable Filtration System; 0.22 um; PES; | Millipore | S2GPT05RE | |
| Streptavidin (1 mg/mL) in 1x PBS buffer | Sigma | S4762-10MG | 20 µL aliquotes |
| SYBR Gold | Thermo Fisher Scientific | S11494 | Highly sensitive nucleic acid stain we used to visualise DNA fork substrate. |
| SYTOX orange; 5 µM in DMSO | Thermo Fisher Scientific | S11368 | |
| T4 DNA ligase | NEB | M0202M | |
| T4 DNA Ligase Reaction Buffer | NEB | B0202S | |
| TEMED | Sigma-Aldrich | T9281-25ML | |
| TEV protease (1 mg/mL); EZCut | Biovision | 7847-10000 | |
| Tissue grinders, Dounce type (40 mL, Wheaton) | DWK Life Sciences | 432-1273 | |
| Tris base | Sigma-Aldrich | T1503 | Used to prepare Tris Buffer (pH 8) |
| Tris HCl | Sigma-Aldrich | T3253 | |
| Tween-20 | Promega UK Ltd | H5152 |
Ссылки
- Bell, S. P., Labib, K. Chromosome Duplication in Saccharomyces cerevisiae. Genetics. 203 (3), 1027-1067 (2016).
- Costa, A., et al. The structural basis for MCM2-7 helicase activation by GINS and Cdc45. Nat Struct Mol Biol. 18 (4), 471-477 (2011).
- Jones, M. L., Baris, Y., Taylor, M. R. G., Yeeles, J. T. P. Structure of a human replisome shows the organisation and interactions of a DNA replication machine. EMBO J. 40 (23), e108819(2021).
- Moyer, S. E., Lewis, P. W., Botchan, M. R. Isolation of the Cdc45/Mcm2-7/GINS (CMG) complex, a candidate for the eukaryotic DNA replication fork helicase. Proc Natl Acad Sci U S A. 103 (27), 10236-10241 (2006).
- Douglas, M. E., Ali, F. A., Costa, A., Diffley, J. F. X. The mechanism of eukaryotic CMG helicase activation. Nature. 555 (7695), 265-268 (2018).
- Ilves, I., Petojevic, T., Pesavento, J. J., Botchan, M. R. Activation of the MCM2-7 helicase by association with Cdc45 and GINS proteins. Mol Cell. 37 (2), 247-258 (2010).
- Dewar, J. M., Walter, J. C. Mechanisms of DNA replication termination. Nat Rev Mol Cell Biol. 18 (8), 507-516 (2017).
- Burnham, D. R., Kose, H. B., Hoyle, R. B., Yardimci, H. The mechanism of DNA unwinding by the eukaryotic replicative helicase. Nat Commun. 10 (1), 2159(2019).
- Kose, H. B., Larsen, N. B., Duxin, J. P., Yardimci, H. Dynamics of the eukaryotic replicative helicase at lagging-strand protein barriers support the steric exclusion model. Cell Rep. 26 (8), 2113-2125.e6 (2019).
- Kose, H. B., Xie, S., Cameron, G., Strycharska, M. S., Yardimci, H. Duplex DNA engagement and RPA oppositely regulate the DNA-unwinding rate of CMG helicase. Nat Commun. 11 (1), 3713(2020).
- Tanner, N. A., Van Oijen, A. M. Visualizing DNA replication at the single-molecule level. Methods Enzymol. 475, 259-278 (2010).
- Swoboda, M., et al. Enzymatic oxygen scavenging for photostability without ph drop in single-molecule experiments. ACS Nano. 6 (7), 6364-6369 (2012).
- Baris, Y., Taylor, M. R. G., Aria, V., Yeeles, J. T. P. Fast and efficient DNA replication with purified human proteins. Nature. 606, 204-210 (2022).
- Lewis, J. S., et al. Tunability of DNA polymerase stability during eukaryotic DNA replication. Mol Cell. 77 (1), 17-25.e5 (2020).
- Li, Z., Xu, X. Post-translational modifications of the mini-chromosome maintenance proteins in DNA replication. Genes. 10 (5), 331(2019).
- Lewis, J. S., et al. Mechanism of replication origin melting nucleated by CMG helicase assembly. Nature. 606 (7916), 1007-1014 (2022).
- Zeman, M. K., Cimprich, K. A. Causes and consequences of replication stress. Nat Cell Biol. 16 (1), 2-9 (2014).
- Sparks, J. L., et al. The CMG helicase bypasses DNA-protein cross-links to facilitate their repair. Cell. 176 (1-2), 167-181.e21 (2019).
- Taylor, M. R. G., Yeeles, J. T. P. The initial response of a eukaryotic replisome to DNA damage. Mol Cell. 70 (6), 1067-1080.e12 (2018).
- Amunugama, R., et al. Replication fork reversal during DNA interstrand crosslink repair requires CMG unloading. Cell Rep. 23 (12), 3419-3428 (2018).
- Wu, R. A., et al. TRAIP is a master regulator of DNA interstrand crosslink repair. Nature. 567 (7747), 267-272 (2019).
- Vrtis, K. B., Dewar, J. M., Chistol, G., Wu, R. A., Graham, T. G. W., Walter, J. C. Single-strand DNA breaks cause replisome disassembly. Mol Cell. 81 (6), 1309-1318.e6 (2021).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены