Method Article
הדמיה בזמן אמת של מולקולה בודדת של שחרור DNA על ידי מנחת מסוקים CMG
In This Article
Summary
פרוטוקול זה מדגים ביצוע בדיקת מולקולה בודדת להדמיה חיה של שחרור DNA על ידי מנחת מסוקים CMG. הוא מתאר (1) הכנת מצע דנ"א, (2) טיהור פלואורסצנטי המסומן כמנחת מסוקים Drosophila melanogaster CMG, (3) הכנת תא זרימה מיקרופלואידי למיקרוסקופ פלואורסצנטי של השתקפות פנימית כוללת (TIRF), ו-(4) בדיקת שחרור DNA של מולקולה יחידה.
Abstract
שכפול גנום נאמן חיוני לשימור היציבות הגנטית של תאים מתחלקים. שכפול DNA מתבצע בשלב S על ידי קומפלקס דינמי של חלבונים המכונה רפליזום. בלב הרפליזום נמצא הליקז CDC45-MCM2-7-GINS (CMG), המפריד בין שני הגדילים של הסליל הכפול של הדנ"א כך שפולימראז DNA יכול להעתיק כל גדיל. במהלך שכפול הגנום, על הרפליזומים להתגבר על שפע של מכשולים ואתגרים. כל אחד מאלה מאיים על יציבות הגנום, שכן כישלון לשכפל דנ"א באופן מלא ומדויק עלול להוביל למוטציות, מחלות או מוות תאי. לכן, יש עניין רב להבין כיצד CMG מתפקד ברפליזום הן במהלך שכפול נורמלי והן במהלך לחץ שכפול. במאמר זה אנו מתארים בדיקת מיקרוסקופ פלואורסצנטי של השתקפות פנימית כוללת (TIRF) באמצעות חלבונים מטוהרים רקומביננטיים, המאפשרת הדמיה בזמן אמת של מולקולות DNA מתוחות הקשורות לפני השטח על ידי קומפלקסים CMG בודדים. בדיקה זו מספקת פלטפורמה רבת עוצמה לחקר התנהגות CMG ברמת המולקולה היחידה, ומאפשרת לצפות ישירות בדינמיקת מנחת המסוקים עם שליטה בזמן אמת על תנאי התגובה.
Introduction
שכפול הדנ"א מוסדר היטב, מכיוון שתא חייב לשכפל את הגנום שלו באופן מדויק כדי למנוע מוטציות, מחלות או מוות. שכפול הדנ"א האיקריוטי מתבצע על ידי קומפלקס הרפליזום, אשר משחרר את הדנ"א ההורי ומשתמש בדנ"א חד-גדילי (ssDNA) כתבנית לסנתז דנ"א חדש אליו. בשלב G1, הקסמרים כפולים לא פעילים קטליטית של MCM2-7 נטענים על דנ"א דו-גדילי (dsDNA) במקורות שכפול1. בשלב S, קומפלקסים MCM2-7 מופעלים על ידי קשירה של CDC45 ו- GINS2 ליצירת קומפלקסים CMG של 11 תת-יחידות (CDC45, MCM2-7, GINS). כל CMG יוזם שחרור DNA בכיוונים מנוגדים, ויוצר את יחידת הליבה שהרפליזום מסדר את עצמו סביב3.
לפני שני עשורים זוהה לראשונה מנחת המסוקים CMG כקומפלקס בן 11 תת-יחידות, החיוני לשכפול DNA4. מאז, ההבנה שלנו של CMG התקדמה במידה ניכרת, מטעינה והפעלה 5,6, לשחרור DNA וסיום7. טכניקות ביוכימיות וביולוגיות מבניות מסורתיות היו קריטיות לרבות מהתגליות הללו; עם זאת, שיטות אלה היו לעתים קרובות מוגבלות ביכולתן לחקור את ההיבטים הדינמיים יותר של CMG. שיטות של מולקולה בודדת משתמשות במניפולציה פיזיקלית של ביומולקולות בודדות כדי למדוד או לדמיין את פעילותן מולקולה אחת בכל פעם. זה יכול לשמש כדי לספק תובנה לגבי הדינמיקה בזמן אמת של חלבונים כי הם לעתים קרובות החמיץ או בלתי ניתן לגילוי על ידי טכניקות אחרות 8,9.
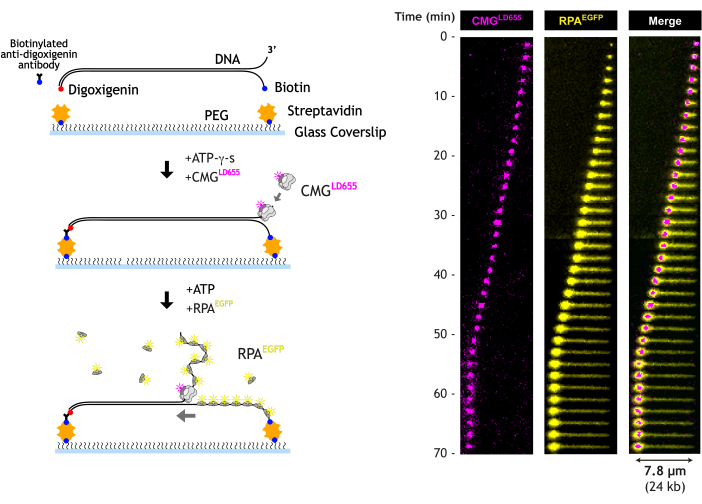
במאמר זה אנו מתארים בדיקה כוללת של מיקרוסקופ פלואורסצנטי של השתקפות פנימית (TIRF) כדי להמחיש את שחרור הדנ"א על ידי מנחת מסוקים CMG בזמן אמת. CMG מטוהר ומסומן באופן פלואורסצנטי נטען על הקצה החופשי של 3 אינץ' של DNA ארוך המכיל מבנה מזלג DNA מוכן מראש. דנ"א ליניארי נמתח על גבי כיסוי ביוטין-PEG בתא זרימה מיקרופלואידי על ידי קשירת כל קצה של הדנ"א ברצף לפני השטח. גישה זו מאפשרת קשירת DNA אחידה יותר, אשר מפחיתה באופן משמעותי את השונות שיש לקחת בחשבון במהלך ניתוח הנתונים. בנוכחות ATP-γ-s, CMG נטען על הדנ"א החד-גדילי בקצה 3' של המזלג. ATP-γ-s הוא אנלוגי ATP הניתן להידרוליזה איטית, המאפשר ל-CMG להיקשר לדנ"א אך לא להשתחרר. הוספה מאוחרת יותר של ATP, יחד עם RPA מטוהר ומתויג פלואורסצנטית, מפעילה CMG ומתחילה שחרור DNA נרחב. מבחינה ויזואלית, CMG עובר טרנסלוקציה לאורך הדנ"א, ומשאיר אחריו מערכת הולכת וגדלה של ssDNA הקשור ל-RPA. קצה הדנ"א הבלתי קשור נע עם CMG, ויוצר "כדור הדוק" עקב דחיסה הנגרמת על ידי קשירת RPA. תכנון תא הזרימה מאפשר להחליף את החיץ בכל נקודה במהלך השחרור, מה שמעניק שליטה רבה במהלך כל ניסוי ומעליו.
פרוטוקול זה מחולק לארבע שיטות, אשר ניתן לבצע בנפרד אחד מהשני. סעיף 1 מתאר את הכנת מצע DNA ליניארי מפוצל של 20 קילו-בתים לבדיקות של מולקולה בודדת. סעיף 2 מתאר את הטיהור והתיוג הפלואורסצנטי של Drosophila melanogaster CMG (DmCMG). מידע מרכזי על הביטוי של DmCMG כלול בסעיף ההערות. סעיף 3 מכסה את הכנת תא זרימה מיקרופלואידי שניתן להשתמש בו במיקרוסקופ TIRF. סעיף 4 מתאר כיצד לבצע את בדיקת שחרור הדנ"א של מולקולה בודדת.
Protocol
1. הכנת דנ"א ליניארי מפוצל של 20 קילובייט המשמש בבדיקות של מולקולות בודדות (איור 1)
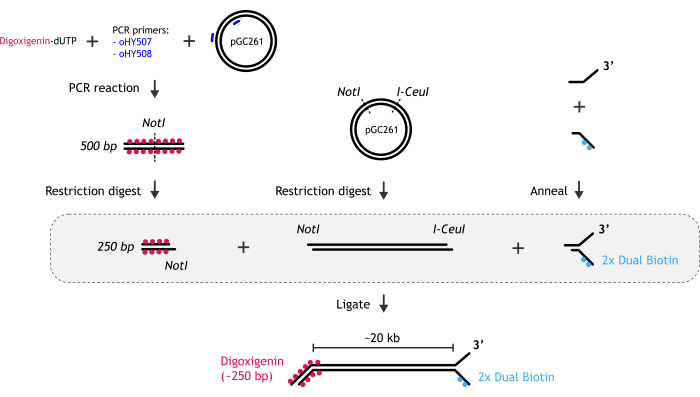
איור 1: ייצוג גרפי של הכנת מצע דנ"א. (A) קצה מזלג הדנ"א הביוטינילי נוצר על-ידי חישול שני אוליגונוקלאוטידים משלימים חלקית: ביוטינילציה ולא ביוטינילציה. (B) מקטע dsDNA הראשי (~20 kb) נוצר על ידי תקציר הגבלה של פלסמיד pGC261 עם שני אנזימים ליצירת דנ"א ליניארי עם שלוחות שונות בכל קצה. (C) קצה הדנ"א הדו-צדדי של דיגוקסיגנין מתקבל על ידי תגובת PCR המבוצעת בנוכחות digoxigenin-dUTP, ואחריה תקציר הגבלה. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
- יצירת מזלג DNA ביוטינילציה.
- אנאל את הדנ"א של המזלג.
- יש לערבב שני אוליגונוקלאוטידים (oHY502 ו-oHYbio85) במאגר STE אחד (100 mM NaCl, 10 mM Tris, pH 8.0; 1 mM ethylenediaminetetraacetic acid [EDTA]). ריכוז האוליגונוקלאוטיד הסופי הוא 10 מיקרומטר, ונפח התגובה הסופי הוא 100 מיקרוליטר.
- לדגור על התגובה ב 80 ° C במשך 5 דקות על בלוק חום. כבו את בלוק החום ואפשרו לתגובה להתקרר בהדרגה, והשאירו אותה בבלוק החום עד שהטמפרטורה יורדת מתחת ל -30 מעלות צלזיוס.
- אמת את יעילות החישול בג'ל Tris-Borate-EDTA (TBE)- Polyacrylamide gel electrophoresis (PAGE).
- הכן 8% אקרילאמיד - ג'ל TBE (עבור 4 ג'לים: 32.8 מ"ל של H2O; 4.8 מ"ל של 10x TBE buffer (1 M Tris, 1 M חומצה בורית, 20 mM EDTA), 9.6 מ"ל של 40% Acrylamide/Bis Solution, 37.5: 1; 0.8 מ"ל של 10% אמוניום פרסולפט [APS], 40 מיקרוליטר של טטרמתילאתילאנדיאמין [TEMED]).
אזהרה: TBE, אקרילאמיד, APS, TEMED הם רעילים/מזיקים. - הפעל נפח קטן (1 μL) של אוליגונוקלאוטידים מחושלים לצד אוליגונוקלאוטידים בודדים מדוללים בצבע העמסה פי 6 על ג'ל אקרילאמיד - TBE 8% ב- 80 V, למשך שעה אחת ב- RT.
- השרו את הג'ל ב-1x TBE בתוספת כתם חומצת גרעין מתאים למשך 30 דקות על שייקר נדנדה נדנדה, ולאחר מכן סרקו את הג'ל באמצעות מערכת הדמיה בג'ל.
- הכן 8% אקרילאמיד - ג'ל TBE (עבור 4 ג'לים: 32.8 מ"ל של H2O; 4.8 מ"ל של 10x TBE buffer (1 M Tris, 1 M חומצה בורית, 20 mM EDTA), 9.6 מ"ל של 40% Acrylamide/Bis Solution, 37.5: 1; 0.8 מ"ל של 10% אמוניום פרסולפט [APS], 40 מיקרוליטר של טטרמתילאתילאנדיאמין [TEMED]).
- יש לפתור DNA מחושל בג'ל TBE-PAGE לכריתת ג'ל.
- אם האוליגונוקלאוטידים מחושלים כראוי, פתרו את שאר הדגימה בנוכחות BSA (~ 100 μL של דגימה מחושלת, 25 μL של 6x טעינת צבע, 2 μL של 33 מ"ג / מ"ל BSA). כדי לטעון את נפח גדול יותר על ג'ל, לשלב כמה בארות עם אזמל סטרילי.
- השאירו את הג'ל ב-1x TBE בתוספת כתם חומצת גרעין מתאים למשך 30 דקות על שייקר נדנדה.
- העבירו את הג'ל למגש זכוכית ודמיינו את הדנ"א בחדר חשוך תחת אור כחול. השתמש במסך מגן מתאים או משקפי מגן בעת שימוש באור כחול.
- חותכים את הרצועה הרצויה עם אזמל סטרילי. יש לקצץ את עודפי הג'ל.
- לטהר את המזלג מחושל על ידי electroelution.
- חותכים חתיכת צינור דיאליזה מספיק זמן כדי להכניס את חתיכת הג'ל, מרטיבים אותה ב-1x TBE וסוגרים קצה אחד.
- לדלל BSA ל 0.3 מ"ג / מ"ל ב 500 μL של 1x TBE ו pipete את כל נפח לתוך הצינור.
- השתמשו במרית המתכת כדי להחליק את חתיכת הג'ל פנימה. הזיזו את חתיכת הג'ל לצד אחד של הצינורית כדי לתת מקום חיץ לדנ"א לנדוד לתוכו במהלך האלקטרואלוציה.
- סחטו החוצה את החיץ העודף וחתכו את הקצה השני של הצינור.
הערה: אמצעי האחסון הנותר יקבע את הנפח והריכוז הסופיים. - הכניסו את צינור הדיאליזה למיכל האלקטרופורזה של האגרוז במאגר TBE אחד, וודאו שהצינורית שקועה במלואה.
הערה: ודא שחתיכת הג'ל ממוקמת בצד הקרוב ביותר לאלקטרודה השלילית כדי שהדנ"א יוכל לנדוד החוצה מהג'ל לתוך שקית הדיאליזה. - הפעל את האלקטרואלוציה ב 80 V למשך 1-2 שעות.
- Dialyze את ה- DNA electroeluted לתוך 10 mM Tris buffer (pH 8).
- לאחר אלקטרואלוציה, להוציא את שקית הדיאליזה מהמיכל ולייבש קצה אחד עם רקמות. הימנע מלגעת בחלק האמצעי של הצינור עם רקמות, שם הג'ל ואת הדגימה נמצאים.
- הסר את האטב מהקצה המיובש ומזלף בעדינות את הדגימה למעלה ולמטה בתוך הצינורית כדי לערבב את הדנ"א לתוך החיץ.
- טבלו מרית נקייה במאגר TBE אחד וגרפו את חתיכת הג'ל מהצינור.
הערה: בעת הסרת חתיכת הג'ל, הסר מאגר קטן ככל האפשר. - סגרו שוב את הקצה הפתוח של שקית הדיאליזה, והסירו אוויר מבפנים.
- הכניסו את שקית הדיאליזה לכוס 2 ליטר מלאה ב-1.5 ליטר של 10 mM Tris buffer (pH 8), 20 mM NaCl ו-2 mM MgCl2. עטפו את האטבים בפרפילם כדי להדביק את הצינור לקצה הכד תוך כדי טבילה מלאה של הצינור בחיץ.
הערה: מלח מייצב דופלקסים קצרים של אוליגו. - השתמש בפס מגנטי כדי לערבב בעדינות את החיץ ולנטרל את הדגימה בטמפרטורת החדר (RT) למשך 3-4 שעות לפחות או ב -4 ° C למשך הלילה.
- לאחר הדיאליזה, יבשו קצה אחד של שקית הדיאליזה עם טישו (לא כדי לדלל את הדנ"א) ושחררו את הקצה הזה.
- מחזירים בעדינות את הדגימה למעלה ולמטה בתוך הצינור ומעבירים אותה לצינור נקי של 1.5 מ"ל.
- למדוד את ריכוז הדנ"א באמצעות ספקטרופוטומטר מיקרו-נפח.
- אנאל את הדנ"א של המזלג.
- דור של שבר 20-kb
- בצע עיכול הגבלה של פלסמיד18 pGC261 באמצעות אנזימי NotI/I-CeuI.
- ערבבו בעדינות 8 μL של NotI-HF (20,000 U/mL), 8 μL של I-CeuI (5,000 U/mL), ואת פלסמיד pGC261 (ריכוז פלסמיד סופי ~40 ng/μL בתגובה של 200 μL) במאגר המומלץ על ידי יצרני אנזימי הגבלה.
- לדגור את התגובה ב 37 ° C במשך הלילה.
- לפתור DNA מעוכל על ג'ל agarose לכריתת ג'ל.
- אופציונלי: יש להריץ כמות קטנה של התגובה על ג'ל אגרוז 0.6% תחילה כדי לבדוק את יעילות העיכול לפני טעינת התגובה כולה.
- הכינו 0.6% ג'ל אגרוז לפי השלבים 1.2.2.3-1.2.2.4.
- מערבבים 0.48 גרם אבקת אגרוז עם 80 מ"ל של 1x TBE buffer. חממו את התמיסה במיקרוגל עד לרתיחה. מערבלים כדי להבטיח שהאגרוז נמס במלואו. מניחים להתקרר במשך מספר דקות, ולאחר מכן יוצקים לתוך המיכל המיועד.
- כדי לשלב בארות כדי להכיל את הנפח הגדול יותר של הדגימה, הדביקו פיסת סרט על פני בארות מרובות של המסרק מראש.
- לאחר שהג'ל התמצק, הסר בעדינות את המסרק ומלא את המיכל במאגר TBE 1x. ערבבו בעדינות את הדגימה עם צבע טעינת DNA והפעילו את הג'ל במתח של 120 וולט למשך שעה אחת.
- השאירו את הג'ל ב-1x TBE בתוספת כתם חומצת גרעין מתאים על שייקר נדנדה נדנדה עד להכתמה.
- העבירו את הג'ל המוכתם למגש זכוכית ודמיינו את הדנ"א בחדר חשוך תחת אור כחול.
- חותכים את הרצועה הרצויה עם אזמל סטרילי. יש לקצץ את עודפי הג'ל.
- אופציונלי: לטהר את המקטע הרצוי באמצעות אלקטרואלוציה כמתואר בשלבים 1.1.4 ו-1.1.5. השמיט את המלח ממאגר הדיאליזה כאן, מכיוון שזה נחוץ רק כדי לייצב את דופלקס האוליגו הקצר.
- בצע עיכול הגבלה של פלסמיד18 pGC261 באמצעות אנזימי NotI/I-CeuI.
- דור של digoxigenin דופלקס DNA סוף
- בצע תגובת PCR עם digoxigenin-dUTP (dig-dUTP) באמצעות pGC261 DNA כתבנית.
- ערבוב: 400 μL מים, 8 μL של pGC261 (0.8 ng/μL), 3.5 μL של פריימר oHY507 (100 μM), 3.5 μL של פריימר oHY508 (100 μM), 8 μL של dig-dUTP (1 mM), 400 μL של 2x PCR תערובת (מוכן DNA פולימראז (20 μL), 10 mM dNTPs (40 μL), 5x נאמנות גבוהה (HF) חיץ (400 μL) ומים (540 μL).
- הפעל את התגובה הבאה במחזור תרמי:
98 °C - 1 דקות
30x: 98 °C - 20 שניות; 65 °C - 20 שניות; 72 °C - 30 שניות;
72 °C - 10 דקות
4 °C - החזק
- לעכל ולטהר מוצר PCR עם תווית digoxigenin
- לטהר מוצר PCR באמצעות ערכת טיהור DNA מסחרית.
- יש לערבב 10 μL NotI-HF (20,000 U/mL) עם מוצר ה-PCR במאגר המומלץ על ידי ספק האנזים (ריכוז DNA סופי של ~50 ng/μL בתגובה ~200 μL). לדגור את התגובה ב 37 ° C במשך הלילה.
- לטהר את הדנ"א המעוכל באמצעות ערכת טיהור DNA מסחרית.
- בצע תגובת PCR עם digoxigenin-dUTP (dig-dUTP) באמצעות pGC261 DNA כתבנית.
- להרכיב רכיבים כדי להפוך את מצע ה- DNA.
- ערבבו בעדינות את קצה המזלג הביוטינילציה, מקטע DNA של 20 קילו-בתים, ומקטע PCR שכותרתו דיגוקסיגנין Not-I עם 5 μL של ליגאז DNA T4 (400,000 U/mL) במאגר המומלץ על ידי ספק האנזים בתגובה של 200 μL. עם זאת, בהתבסס על מקטע DNA של 20 קילו-בתים שנוסף (1-5 מיקרוגרם), הוסף עודף מולארי של בערך פי 100 הן של קצה המזלג הביוטינילציה והן של מקטע ה-PCR.
- Aliquot את התגובה לתוך צינורות PCR (50 μL כל אחד) לדגור ב 16 ° C במחזור תרמי לילה.
- יש לפתור את הדגימה הקשורה על ג'ל אגרוז 0.6% ולטהר DNA קשור על ידי אלקטרואלוציה כמתואר בשלבים 1.2.2 ו-1.2.3.
- אחסן DNA קפוא פלאש ב -80 ° C.
2. טיהור של Drosophila melanogaster CMG (איור 2)

איור 2: טיהור של Drosophila melanogaster CMG מ-4 ליטר של תאי Hi Five. החלבונים נפתרו על 4%-12% ג'ל פוליאקרילאמיד Bis-Tris מתחת ל-200 וולט בנוכחות חיץ MOPS. הדגימה מוצגת בכל שלב של הטיהור (תא ליזט - 2 μL, FLAG elution - 10 μL, לאחר עמודת חילופי היונים הראשונה - 10 μL, ולאחר התיוג ועמודת חילופי היונים השנייה - 1 μL. (A) צביעת קומאסי מאשרת את נוכחותן של כל 11 יחידות המשנה של קומפלקס CMG לפני (10 μL), ואחרי (1 μL) תיוג פלואורסצנטי. (B) יעילות ההתוויה של תת-היחידה MCM3 אומתה על-ידי סריקה עבור Cy5 עם מנתח תמונות פלואורסצנטי באמצעות מסנן אדום בעל מעבר ארוך (LPR). אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
הערה: כדי להכין את Drosophila melanogaster CMG המסומן באופן פלואורסצנטי, הוכנס אתר מחשוף TEV (ENLYFQG) ואחריו ארבע שאריות Gly במורד הזרם של תג FLAG N-terminal בתת-היחידה MCM3 (בווקטור pFastBac1)10. כדי לבטא את המתחם, נעשה שימוש במערכת הביטוי baculovirus. עבור טרנספקציה ראשונית, תאי Sf21 שימשו בנפרד עבור כל תת-יחידה CMG (שלב וירוס P1). כדי להגביר עוד יותר את הנגיפים, נעשה שימוש בתאי Sf9 (שלב וירוס P2). לאחר מכן, תרביות תאי Sf9 (100 מ"ל לכל תת-יחידה CMG; 0.5 x 106 תאים / מ"ל) היו נגועות בנגיף P2 0.5 מ"ל בתוספת 10% נסיוב עגל עובר (שלב וירוס P3). כדי לבטא את כל קומפלקס CMG ב 4 L של Hi חמישה תאים (1 x 106 תאים / מ"ל), 200 מ"ל של וירוסים P3 שימשו עבור כל אחת מתתי היחידות. לאחר קצירת תאי Hi Five המבטאים את קומפלקס CMG, ניתן להקפיא את גלולת התא בחנקן נוזלי ולאחסן אותה בטמפרטורה של -80°C. בצע את הטיהור כולו על קרח או ב 4 °C (75 °F). ניתן להכין את המאגרים מראש, בתנאי שמוסיפים את החומרים המחזרים (DTT או 2-Mercaptoethanol) ומעכבי פרוטאז (CAUTION) ממש לפני השימוש. ודא שכל המאגרים מקוררים מראש, מסוננים ומנוטרלים גזים.
- הכינו את המאגרים הבאים.
- הכן את חיץ ההשעיה A על-ידי ערבוב 25 mM 4-(2-hydroxyethyl)-1-piperazineethanesulfonic acid (HEPES) pH 7.5, 0.02% Tween-20, 10% glycerol, 15 mM KCl, 2 mM MgCl2, 2 mM 2-mercaptoethanol, 1 mM EDTA ו-1 mM אתילן גליקול-ביס (β-אתר אמינואתיל)-N,N,N′,N′-חומצה טטראצטית (EGTA).
אזהרה: 2-מרקפטואתנול, DTT ו-EDTA רעילים/מזיקים - הכינו חיץ מלח חוצץ Tris-buffered (TBS; 0.1 M Glycine HCl, pH 3.5) להכנת ג'ל זיקה ANTI-FLAG M2.
- הכינו את Buffer A-100 על ידי ערבוב 25 mM HEPES pH 7.5, 0.02% Tween-20, 10% גליצרול, 100 mM KCl, 1 mM DTT, 1 mM EDTA ו-1 mM EGTA.
- הכינו את מאגר הדיאליזה על ידי הוספת 25 mM HEPES pH 7.5, 50 mM נתרן אצטט, 10 mM מגנזיום אצטט, 10% גליצרול ו-1 mM DTT.
- הכן מאגר TBS על ידי ערבוב 50 mM Tris-HCl pH 7.5 ו- 150 mM NaCl.
- הכן את חיץ ההשעיה A על-ידי ערבוב 25 mM 4-(2-hydroxyethyl)-1-piperazineethanesulfonic acid (HEPES) pH 7.5, 0.02% Tween-20, 10% glycerol, 15 mM KCl, 2 mM MgCl2, 2 mM 2-mercaptoethanol, 1 mM EDTA ו-1 mM אתילן גליקול-ביס (β-אתר אמינואתיל)-N,N,N′,N′-חומצה טטראצטית (EGTA).
- גלולת השהיה מתרבית 4 ליטר של Hi Five תאים ב-200 מ"ל של חיץ מתלה קר A בתוספת טבליות קוקטייל מעכבות פרוטאז. מערבבים את הצינורות בהיפוך עד שכל הגלולה מושהית מחדש.
- להעביר את התאים resuspended לתוך precooled 40 מ"ל Dounce homogenizer. כדי לשחזר תאים רבים ככל האפשר, לשטוף את הצינורות המשמשים להקפאת גלולת התא עם אותו סוג של חיץ ולהוסיף הומוגנייזר Dounce. ודא שהנפח הכולל בהומוגנייזר אינו חורג מהגבול המומלץ. לשכב את התאים על קרח על ידי 60-70 שבץ.
הערה: ודא שהבוכנה מגיעה לתחתית הצינור, אך אל תפעיל לחץ רב מדי מכיוון שההומוגנייזר עלול להישבר. היזהר לא להוציא את הבוכנה מעל רמת הנוזל, כמו זה יציג בועות. - חזור על השלב הקודם עד שכל הדגימה הומוגנית ושלב אותה בכד המקורר מראש. הערך את הנפח הסופי של המדגם. טיפה אחר טיפה, מוסיפים KCl מסונן לריכוז הסופי של 100 mM, תוך ערבוב עדין.
- מצננים מראש את הצנטריפוגה ל-4°C. יוצקים את הדגימה לתוך צינורות צנטריפוגה, להבטיח את נפח מומלץ על ידי היצרן הוא הגיע. אזנו את הצינורות על סולם.
הערה: צינורות בעלי נפח קטן מדי עלולים להישבר במהלך צנטריפוגה. - צנטריפוגה את התאים lysed ב 23,500 x גרם במשך 15-30 דקות ב 4 ° C. בינתיים, הכינו ג'ל זיקה ANTI-FLAG M2 בהתאם לשלב הבא.
- יש לערבב בעדינות את הבקבוק המכיל ג'ל זיקה ANTI-FLAG M2 (50% תרחיף). חתכו את קצהו של קצה פיפטה P-100 (כדי להרחיב את הקצה) והשתמשו בו כדי להעביר מיד 4 מ"ל של מתלה (2 מ"ל חרוזים) לתוך עמודת כרומטוגרפיה פולי-הכנה של 20 מ"ל. כדי לשחזר את החרוזים שנתקעו בקצה, שטפו את הקצה עם TBS והוסיפו אותו לעמודה. הניחו לכל החרוזים להתיישב בתוך העמודה, אך היזהרו שלא לייבש אותם.
- שטפו את השרף בנפח 3x חרוזים (6 מ"ל בסך הכל) של 0.1 M גליצין הידרוכלוריד pH 3.5. לאחר מכן, לשטוף את החרוזים עם נפח 3-5 חרוזים של TBS (6-10 מ"ל). לאחר מכן, לשטוף את החרוזים 3 פעמים (6 מ"ל) עם Buffer A-100.
הערה: אין להשאיר את החרוזים בתמיסת 0.1 M גליצין HCl pH 3.5 למשך יותר מ-20 דקות. כדי להגביר את לחץ הזרימה ואת מהירות הכביסה, כסו את העמוד במכסה. אין לאפשר לשרף להתייבש בשום שלב. - לאחר הכנת השרף, להשאיר 2 מ"ל של חיץ A-100 מעל השרף ולסגור את היציאה של העמודה.
- לאחר הצנטריפוגה, יוצקים בעדינות את supernatant לתוך צינורות 50 מ"ל מקורר מראש. היזהרו לא להפריע לכדור. קח aliquot קטן (~ 10 μL) כדי לאמת על ידי אלקטרופורזה SDS-PAGE מאוחר יותר.
- הוסף כמות שווה של חרוזי ANTI-FLAG M2 (שהוכנו בהתאם לשלב הקודם) לכל אחד מהצינורות של 50 מ"ל. נסה לשחזר את כל החרוזים מהטור על ידי השעייתם מחדש עם כמה מיליליטרים נוספים של חיץ A-100 והעברת השעיה זו לצינורות.
- לדגור את הדגימה (supernatant) עם שרף M2 ANTI-FLAG, מסתובב במשך 2.5 שעות ב 4 ° C.
- לאחר הדגירה, סובבו את הדגימה בטמפרטורה של 1,000 x גרם למשך 5 דקות בטמפרטורה של 4°C. בעזרת פיפטה, להסיר את רוב supernatant. בעזרת קצה חתוך, מרחפים מחדש את החרוזים בכמה מיליליטרים של הסופרנאטנט הנותר ומעבירים אותם לשני צינורות של 15 מ"ל. כדי לשחזר את כל החרוזים הדבוקים לדפנות צינורות 50 מ"ל, הוסף כמה מיליליטרים של חיץ A-100 והעבר את זה גם לצינורות 15 מ"ל.
- סובב את צינורות 15 מ"ל ב 1,000 x גרם במשך 5 דקות ב 4 ° C. הסר את supernatant.
- שטפו את החרוזים על ידי הוספת ~ 14 מ"ל של Buffer A-100 לכל צינור, ולאחר מכן סיבוב קצר ועדין ב 4 °C ולאחר מכן מסתובב ב 1000 x גרם במשך 5 דקות ב 4 °C. חזרו על השטיפות פעמיים. קח aliquots קטנים של כל כביסה (~ 10 μL) כדי לאמת על ידי אלקטרופורזה SDS-PAGE מאוחר יותר.
- שלב זה (2.16) מהווה הליך חלופי לשלבים הקודמים (2.13-2.15).
- לאחר הדגירה עם שרף ANTI-FLAG M2, יוצקים את הדגימה לשתי עמודות כרומטוגרפיה פולי-פרפ 20 מ"ל.
- אם בוחרים בשיטה זו, תנו לחרוזים להתיישב בתחתית הטור ולדגימה הלא מאוגדת לעבור דרך הטור לפי זרימת הכבידה. לאחר מכן, הוסף חיץ A-100 ישירות לעמודה ותן לו לשטוף את הדגימה באמצעות זרימת הכבידה.
- העבר את החרוזים (התלויים מחדש במאגר A-100) לשתי עמודות כרומטוגרפיה פולי-מוכנות של 10 מ"ל.
- אפשר ל-Buffer A-100 לעבור דרך השרף, סגור את השקע כאשר רמת החיץ מגיעה לחלק העליון של השרף, והסר את החלבון מהחרוזים על ידי הוספת Buffer A-100 בתוספת פפטיד 200 מיקרוגרם/מ"ל (DYKDDDDK) (מאגר אלוציה FLAG).
- עבור ההדבקה הראשונה, הוסף 3 מ"ל של מאגר ה- elution של FLAG וסובב בעדינות את העמודה הסגורה למשך 15 דקות ב- RT. פתח את השקע כדי לאסוף את שבר ה- elution הראשון.
- סגור את השקע והוסף 2 מ"ל של מאגר FLAG elution לכל עמודה. סובבו במשך 10 דקות נוספות ואספו את שבר האלוציה השני.
- ערבבו את כל השברים המדוללים יחד (~10 מ"ל) ושמרו על טמפרטורה של 4°C. קח aliquot קטן של החלק המדולל (~ 10 μL) כדי לאמת על ידי אלקטרופורזה SDS-PAGE מאוחר יותר.
- סנן את הדגימה המדוללת באמצעות מסנני מזרק 0.22 מיקרומטר.
- מראש, הכינו את עמודת CaptoHiRes Q (5/50) (עמודת כרומטוגרפיה של חילופי יונים ברזולוציה גבוהה) המחוברת למערכת טיהור חלבונים, בהתאם לפרוטוקול היצרן. לאחר מכן, אזן את העמודה עם Buffer A-100 וטען את הדגימה המסוננת לעמודה המאוזנת.
- שטוף את העמודה עם 20 כרכי עמודות (CV) של Buffer A-100 (~ 20 מ"ל סה"כ מכיוון שנפח העמודה הוא ~ 1 מ"ל).
- כדי לנטרל את החלבון, הכינו מראש שני חוצצים: Buffer A-100 ו-Buffer B עם הרכב זהה לזה של Buffer A-100, אך עם 550 mM KCl במקום 100 mM KCl. הגדר את ה-gradient elution עבור 20 CV (~20 mL) עם ריכוז מלח עולה מ-100 mM ל-550 mM KCl. אסוף 0.3-0.5 מ"ל elutions באספן שברים.
הערה: CMG צריך ללוט בסביבות 70%-75% ריכוז של Buffer B. קח aliquots קטנים של לשטוף שברים מדוללים עבור אלקטרופורזה SDS-PAGE. - בצע אלקטרופורזה SDS-PAGE כדי לאשר כי CMG קיים בשברים שנבחרו. בינתיים, נקו את העמודה ואת מערכת הטיהור לפי פרוטוקול היצרן.
- דגרו על השברים שנבחרו עם פרוטאז TEV למשך הלילה על ידי ערבוב 50 מיקרוליטר של פרוטאז TEV (1 מ"ג/מ"ל) לכל 1 מ"ל דגימה.
- הרטיבו מראש צינור דיאליזה וודאו שהקרום אינו ניזוק. הוסיפו את תערובת CMG/TEV לצינורות הדיאליזה והכניסו לכוס 2 ליטר עם 1.5 ליטר של Buffer A-100 מקורר מראש והמוט המגנטי בפנים. יש לטשטש עם ערבוב עדין למשך הלילה ב-4°C.
- ערבבו את הדגימה עם 50 מיקרומטר של פפטיד המסומן בתווית LD655 ואנזים Sortase 10 מיקרוגרם/מ"ל בנוכחות 5 mM CaCl2. לדגור את התגובה ב 4 ° C, מסתובב במשך 30 דקות, להבטיח את הצינור מכוסה מאור. קח aliquot קטן לפני ואחרי התיוג עבור אלקטרופורזה SDS-PAGE.
- סנן את הדגימה המסומנת באמצעות מסנני צנטריפוגות של 0.22 מיקרומטר לפני הטעינה למערכת הטיהור, מכיוון שהפפטיד עלול לזרז.
- הכן מראש את עמודת הכרומטוגרפיה של חילופי יונים ברזולוציה גבוהה, כמתואר קודם לכן (שלב 2.20). הפעם, כסו את העמוד ואת מערכת אספן השברים ברדיד אלומיניום כדי להגן על הדגימה מפני אור.
- כדי להסיר את הפפטיד החופשי, טען את הדגימה המסוננת בעמודת הכרומטוגרפיה של חילופי יונים ברזולוציה גבוהה. בצע את הטיהור באותו אופן שתואר קודם לכן (שלבים 2.21-2.22). ודא שה-CMG המסומן פולט בריכוז של Buffer-B בדומה לזה שהיה קודם.
- בצע אלקטרופורזה SDS-PAGE כדי לאמת את איכות השברים שנבחרו. כדי לצלם את הפלואורסצנטיות, אין להרתיח את הדגימות לפני ההעמסה, ולוודא שמיכל האלקטרופורזה מוגן מפני אור. דמיינו תחילה פלואורסצנטיות של חלבונים באמצעות מערכת הדמיית ג'ל, ולאחר מכן צבעו את הג'ל בצבע קומאסי ודמיינו אותו שוב כדי להמחיש את כל החלבונים.
- דיאליזה בחרה שברים למשך הלילה בטמפרטורה של 4°C, כנגד 1.5 ליטר של חיץ הדיאליזה. במידת הצורך, רכז את המדגם.
- יש להקפיא את החלבון בחנקן נוזלי ולאחסן אותו בטמפרטורה של -80°C עד לשימוש נוסף.
3. הכנת תא הזרימה (איור 3)
- הכינו כיסויי ביוטין-PEG לפי פרוטוקול11 שתואר קודם לכן, תוך השמטת שלב האפייה בתנור. כיסויי ביוטין-PEG יציבים למשך חודש אחד לפחות תחת ואקום ב-RT. להכנת תא הזרימה, חתכו את כיסוי הביוטין-PEG (24 מ"מ x 60 מ"מ) לשניים (כ-24 מ"מ x 30 מ"מ).
הערה: יש להקפיד לא לגעת באזור המרכזי של הכיסוי שבו יהיו ערוצי הזרימה. - הכינו חתיכות זכוכית קטנות על ידי חריטה והצמדה של מגלשת זכוכית לחתיכות בגודל של כ-2.4X1 ס"מ.
- קדח שני חורים במרחק של 1.4 מ"מ זה מזה באמצעות מקדח מצופה יהלום בקוטר 0.8 מ"מ, אחד מעט רחב יותר מהשני (0.043 אינץ' = כניסה, 0.048 אינץ' = שקע). בדוק שהחורים בגודל הנכון על ידי ניסיון להכניס חתיכה מצינור הכניסה / יציאה, באמצעות אותו מקדח כדי להרחיב את החור עד שהצינור מתאים. ודא כי הצינור מתאים מספיק חזק כדי לא ליפול בקלות.
הערה: צינורות כניסה צרים יותר משמשים למזעור הנפח המת. - חותכים סרט דו-צדדי באותה צורה כמו חתיכות הזכוכית.
- ישר את השקופית על הקלטת ודקר מחט דרך כל חור כדי לסמן את מיקומם על הקלטת. בעזרת סכין גילוח, חותכים תעלה המקיפה את שני החורים.
הערה: אין לחתוך את התעלה זמן רב מדי, מכיוון ששטח עודף בצד הרחוק של כל חור עלול להישאר יבש כאשר נוזלים מוזרמים פנימה ויכול להוביל לבעיות עם בועות אוויר במהלך הזרימה. בשעת החיתוך, נסו לחתוך את הקצוות הארוכים בחיתוך נקי אחד, שכן קצוות מרופטים מחתכים מרובים עלולים להשפיע על איכות ערוץ הזרימה. למטרה דומה, אין לחתוך בשולי ערוץ הזרימה, שכן חתכים בחתיכה השמישה יכולים להשפיע על זרימת הנוזל או אפילו לדלוף בתא הזרימה המורכב. - נקו את חתיכת הזכוכית עם אצטון ורקמות עד לייבוש, ולאחר מכן הניחו על משטח נקי. מקלפים צד אחד של סרט ההדבקה ומדביקים אותו על חתיכת הזכוכית כך ששני החורים יהיו לגמרי בתוך התעלה.
- אטמו את סרט הדבק לחתיכת הזכוכית באמצעות קצה פיפטה p1000 כדי לסלק בועות על ידי לחיצה על המשטח בלחץ יציב אך מתון. יש לעבור על כל המשטח כדי לאטום.
- מקלפים את הקצה השני של הסרט משני חלקי הזכוכית ומניחים אותם על משטח נקי כשהצד הדביק כלפי מעלה. הניחו את המגלשות צמודות זו לזו אך לא נוגעות בתנוחה בה הן יהיו מחוברות לכיסוי (השאירו מספיק מקום ביניהן כדי שהאפוקסי ייצור אטימה מלאה סביב כל חתיכת זכוכית).
- הרימו את כיסוי חצי ביוטין-PEG באמצעות פינצטה מפלסטיק, המחזיקים רק בקצה (אל תיגעו באזור שייצור את תעלת הזרימה), והורידו את הצד המתפקד ל-PEG אל הדבק. יש לאבטח על ידי לחיצה עם אצבע כלפי מטה כדי לקבע אותו במקומו, ולאחר מכן להשלים את האטימה על ידי שפשוף המשטח בחוזקה מתונה עם קצה פיפטה כדי להסיר בועות אוויר (לא על האזור של תעלת הזרימה מכיוון שזה עלול להישבר), ואז להתהפך.
הערה: טפל בכיסוי בזהירות. - עבור כל תא זרימה, לחתוך ~ 10 ס"מ מכל סוג של צינורות פוליאתילן (PE20 ו PE60). השתמש בצינור הצר יותר ככניסה לתא הזרימה כדי להפחית נפח מת.
- הכנס צינורות לחורים בשקופית ביד. אם קוטר החורים נכון, יש לוודא שהצינור עומד בתוך החור בכוחות עצמו לאחר הכנסתו. חתכו את קצה הצינור בזווית קלה <45° כדי להקל מעט על החדרת הצינור ולמנוע מהצינור ליצור אטם כנגד הכיסוי, מה שהוא עלול לעשות אם הקצה שטוח (זה עלול לחסום את הזרימה).
הערה: ההתאמה המדויקת של הצינור לחתיכת הזכוכית הופכת את בניית תאי הזרימה לאמינה יותר ומסייעת למנוע הכנסת בועות לתא הזרימה. - ערבבו היטב את רכיבי האפוקסי ולאחר מכן השתמשו בקצה p200 כדי לטפוח אפוקסי כדי לאטום את הצינורית וליצור איטום סביב כל חתיכת זכוכית. הוסיפו מספיק אפוקסי כך שיעלה לקצה הכיסוי העגול, שכן זה יחזק גם את הכיסוי העדין. עם זאת, יש לוודא שאף אפוקסי לא יעלה על החלק התחתון של הכיסוי, מכיוון שהוא עלול למנוע ממנו לשכב שטוח על הבמה.
הערה: במידת הצורך, ניתן לגרד כמויות קטנות של עודפי אפוקסי מהחלק התחתון באמצעות סכין גילוח. - השאירו לפחות 30-60 דקות כדי שהאפוקסי יחלים לחלוטין.
הערה: אם אין להשתמש בתא הזרימה מיד, אחסן אותו תחת ואקום ב-RT לאחר ריפוי מלא. - לאחר השימוש בתא הזרימה, לשחזר את חתיכות זכוכית ולעשות בהם שימוש חוזר ללא הגבלת זמן. משכו את הצינור החוצה והכניסו את תא הזרימה לצנצנת שקופיות המכילה אצטון למשך 24 שעות לפחות כדי לרכך את האפוקסי והדבק הדו-צדדי, מה שמאפשר להסיר את חתיכות הזכוכית בקלות. לפני שימוש חוזר ליצירת תא זרימה נוסף, נקו את חתיכות הזכוכית על ידי קרצוף שלהן עם ספוג ומי סבון, ייבשו אותן וקרצפו אותן עם אצטון ונייר טישו.

איור 3: ייצוג גרפי של הכנת תא הזרימה. (A) גזרו סרט דו-צדדי כך שיתאים לגודל חתיכת הזכוכית. ישר את השקופית על גבי הסרט וסמן את המיקום של כל חור עם מחט. בעזרת סכין גילוח, חתכו סביב כל אחיזה ליצירת ערוץ. (B) מקלפים צד אחד של הסרט ומדביקים את הסרט על חתיכת הזכוכית. ודא ששני החורים נמצאים בתוך התעלה. מקלפים את הקצה השני של הסרט ומדביקים את כיסוי הביוטין-PEG מלמעלה. (C) הכניסו את צינור הפוליאתילן לכל חור ואטמו את הצינורית במקומה עם אפוקסי, ואטמו כל פיסת זכוכית גם לכיסוי. (D) לאחר השימוש בשני הערוצים, משכו את הצינור והכניסו את תא הזרימה לצנצנת מכתימה מלאה באצטון. לאחר כ-24 שעות, האפוקסי והסרט יתרככו, וניתן יהיה לקלף את שכבות תא הזרימה. ניתן לשחזר את חתיכות הזכוכית ולאחסן אותן באצטון לשימוש חוזר ללא הגבלת זמן ליצירת תא הזרימה הבא. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
4. בדיקת TIRF של מולקולה בודדת כדי להמחיש שחרור DNA בתיווך CMG
- הכינו את המאגרים הבאים.
- הכן מאגר חוסם (20 mM Tris, pH 8.0; 50 mM NaCl; 2 mM EDTA; 0.2 mg/mL BSA). סנן את המאגר באמצעות מסנן מזרק 0.22 מיקרומטר ואחסן אותו בטמפרטורה של 4°C.
- הכן 10x חיץ תגובה 1 (250 mM HEPES pH 7.5; 50 mM NaCl; 100 mM מגנזיום אצטט). סנן את המאגר באמצעות מסנן מזרק 0.22 מיקרומטר ואחסן אותו בטמפרטורה של 4°C.
- הכינו 10x חיץ תגובה 2 (250 mM Tris, pH 7.5; 100 mM מגנזיום אצטט; 1.25 M אשלגן גלוטמט; 1 mM EDTA; 0.025% Tween-20). סנן את המאגר באמצעות מסנן מזרק 0.22 מיקרומטר ואחסן אותו בטמפרטורה של 4°C.
- נטרל כ -2.5 מ"ל של חיץ חוסם ו -5 מ"ל של מים טהורים במיוחד על ידי הנחת הצינורות הפתוחים במייבש והשארתם תחת ואקום למשך 15 דקות.
הערה: אין למלא צינורות עד הקצה, מכיוון שבועות במהלך פירוק גזים עלולות לגרום להתזת נוזלים. - הניחו את תא הזרימה על במת מיקרוסקופ ואבטחו אותו במקומו באמצעות מרק דבק בכל קצה. חשוב לאבטח היטב כדי למנוע מתא הזרימה לנוע מאוחר יותר במהלך הרכישה.
- חבר את צינור יציאת תא הזרימה למשאבת המזרק, אשר יש צינור מחובר מזרק עם מחט בקצה השני. הכנס מחט זו לצינור היציאה כדי לחבר את תא הזרימה.
- הפעילו את תנור החימום האובייקטיבי ל-30°C.
- אחסנו 1-2 מ"ל מים בצינור לחתיכה נפרדת של מרק דבק בסמוך לתא הזרימה, כך שניתן יהיה להחדיר את צינור הכניסה ולהגיע לתחתית הצינור.
- הזרימו מים דרך התעלה, ולאחר מכן השתמשו בזרימה מהירה יותר כדי להסיר בועות לכודות ליד צינור הכניסה, והוסיפו עוד מים במידת הצורך. אחרת, בועות גדולות עלולות להשתחרר מאוחר יותר בניסויים ולעבור דרך התעלה. השאירו למשך מספר דקות עד שהזרימה תיפסק במלואה, שכן הזרימה המהירה עלולה לגרום למשיכה שיורית של נוזל למשך זמן מה לאחר הפסקתה, שכן הלחץ בתוך המזרק מתייצב.
הערה: כל דנ"א שבא במגע עם בועה הופך לבלתי שמיש, אפילו לאחר התייבשות. - הוסף 100 μL של חיץ חוסם degassed ל 20 μL aliquot של 1 מ"ג / מ"ל streptavidin. חברו את הצינור הפתוח למרק הדבק והעבירו את צינור הכניסה מהמים לסטרפטווידין. זרימה בקצב של 40 מיקרוליטר/דקה למשך 2 דקות (סה"כ 80 מיקרוליטר) ודגרה במשך 5 דקות.
- שטפו את עודפי הסטרפטאווידין עם המאגר החוסם (50 מיקרוליטר/דקה עבור 100 מיקרוליטר).
- זרימה בדנ"א ביוטינילציה, מדולל בחיץ חוסם עם תפוז SYTOX 25 ננומטר. תמונה באמצעות תצוגה חיה עם לייזר 532 ננומטר כדי לצפות בדנ"א נקשר לפני השטח בזמן אמת.
- כאשר מושגת הצפיפות המשוערת של דנ"א על פני השטח, זרימה במאגר חוסם, גם כן עם 25 ננומטר SYTOX (50 μL/min עבור 100 μL), כדי לשטוף את הדנ"א החופשי.
- זרימה בנוגדן אנטי-דיגוקסיגנין ביוטינילציה (~10 מיקרוגרם/מ"ל) מדולל במאגר חוסם המכיל 25 ננומטר תפוז SYTOX (100 מיקרוליטר/דקה עבור 300 מיקרוליטר).
הערה: תפוז SYTOX עובר אינטרקלציה בין זוגות הבסיס של הדנ"א, ומאריך את אורך קווי המתאר. משמעות הדבר היא שהאורך מקצה לקצה של הדנ"א גדל בכתום SYTOX, מה שמאפשר לקשירה השנייה להיצמד לפני השטח רחוק יותר מהראשונה, מה שנותן דנ"א מתוח טוב יותר כאשר תפוז SYTOX נשטף מאוחר יותר. שימוש במויטי שונה בכל קצה דנ"א מאפשר קשירה אחידה יותר של דנ"א למשטח הכיסוי (איור 4). כאשר שני הקצוות עוברים ביוטינילציה, הם נקשרים באותו שלב, וכתוצאה מכך נוצרת שונות משמעותית במיקומי ההתקשרות שלהם. שימוש בדיגוקסיגנין בקצה אחד נותן שליטה רבה יותר על כיסוי הדנ"א לפני קשירת הקצה השני. זה גם מאפשר להגדיל את כיסוי הדנ"א על ידי הדגירה. - שטפו את נוגדן האנטי-דיגוקסיגנין הביוטינילציה ואת תפוז SYTOX עם חיץ חוסם (50 μL/min עבור 100 μL).
- לפצות 120 μL של 'תערובת ATP-g-s' (1x חיץ תגובה 1, 0.75 מ"ג / מ"ל BSA, 1.25 מ"ג / מ"ל קזאין, 8 mM DTT, 0.33 mM ATP-g-s), להוסיף 30 μL לצינור חדש, ולאחר מכן להזרים 50 μL מן הצינור המקורי לתוך תא הזרימה (50 μL / min עבור 50 μL). זה עוזר למזער את ההשפעה של ערבוב חיץ באמצעות זרימה חוזרת או דיפוזיה מצינור היציאה.
- הוסף CMG מטוהר ל~100 ננומטר סופי בתערובת ATP-g-s של 30 מיקרוליטר, ולאחר מכן זרם פנימה במהירות של 20 μL/min למשך 20 μL. דגירה במשך 15 דקות.
- מהווים 120 μL של 'תערובת ATP/RPA' (1x Reaction buffer 1, 0.75 mg/mL BSA, 8 mM DTT, 3.3 mM ATP, 20 nM EGFP-hRPA), וזורמים לתא זרימה ב-40 μL/min עבור 80 μL.
- מיד להתחיל לרכוש תמונות. רכוש 6 x 6 שדות ראייה לכל מסגרת כל 30 שניות (או קצב מסגרות מתאים לניסוי). קצבי פריימים נמוכים יותר יכולים להפחית את החשיפה ללייזר ואת ההלבנה הפלואורופורית. הצג באופן חזותי את EGFP-hRPA עם לייזר של 488 ננומטר בעוצמה של 1%. אם CMG מסומן, למשל, LD655, דמיינו CMG עם לייזר של 640 ננומטר (הספק מרבי = 30 mW) בהספק של 10%. דמיינו DNA מוכתם בכתום של SYTOX באמצעות לייזר של 532 ננומטר בעוצמה של 0.5%. השתמש בכל לייזר בחשיפה של 50-100 אלפיות השנייה.
הערה: בכל פעם שצינור הכניסה של תא הזרימה מועבר מצינור נוזל אחד למשנהו, טפחו את קצה הצינור בתחתית הצינור מספר פעמים לפני הוצאתו מהנוזל. זה עוזר למנוע מבועות להיכנס במהלך ההעברה. צמצמו את הזמן שבו קצה צינור הכניסה מוחזק מחוץ לנוזל, ואל תכוונו את הקצה כלפי מעלה ואל תרימו את הקצה באופן משמעותי, שכן הדבר גורם לנוזל להתנקז לכיוון התעלה ולמשוך אוויר לקצה. בעת העברת צינור הכניסה, נסו לא להפעיל מתח על הצינור מכיוון שזה יכול למשוך אותו החוצה מהאפוקסי, ולאבטח אותו לתא הזרימה. לשחרור DNA בהדמיה, בנוכחות תפוז SYTOX כדי להמחיש DNA דו-גדילי, החלף את 10x Reaction buffer 1 ב-10x Reaction buffer 2 עבור תערובת ATP/RPA. תפוז SYTOX נקשר לדנ"א טוב יותר בתנאים אלה. רוקנו לחלוטין את משאבת המזרק בסוף כל ניסוי. יש לשטוף מדי פעם על ידי מילוי במים וריקון מספר פעמים או פירוק ושטיפה במי סבון.

איור 4: דנ"א שנקשר לפני השטח. (A) כאשר קושרים מצעי דנ"א עם ביוטין בשני הקצוות, המרחק בין שני הקשרים יכול להשתנות בהתאם לאופן שבו הקצוות באים במגע עם פני השטח (i). על ידי שימוש בדיגוקסיגנין בקצה אחד, ניתן להפריד באופן זמני את הקשירה של כל קצה לקבלת מרחקי קשירה עקביים יותר ודנ"א נמתח באופן אחיד יותר (ii). (B) שדה ראייה לדוגמה המראה דנ"א קשור בשני קצותיו (מסומן בתווית דיגוקסיגנין) ומוכתם בכתם פלואורסצנטי של חומצת גרעין. דנ"א, הקשור בשני קצותיו, מופיע כקו, ואילו דנ"א הקשור בקצה אחד בלבד מופיע ככתמים. באופן אידיאלי, הדנ"א צריך להיות קשור בצפיפות ככל האפשר מבלי לחפוף דנ"א אחר. התמונה היא 512 x 512 פיקסלים (גודל פיקסל = 154.6 nm). אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
תוצאות
כאשר CMG משחרר דנ"א, מערכת RPA אופיינית תגדל עם הזמן (איור 5). קצה ה-5' של הדנ"א הלא פצוע קשור לפני השטח; לפיכך, הוא נראה כקטע ליניארי של אות RPA בין הקשירה למזלג. קצה 3' אינו קשור, ולכן נע עם המזלג ונצפה כאות EGFP-RPA קומפקטי. המיקום של גדיל טרנסלוקציה דחוס unwound מתאים בערך למיקום של מזלג השכפול, אשר נע יחד עם LD655-CMG דמיינו באמצעות לייזר 640 ננומטר.
חשוב למזער את הנזק למצע הדנ"א, מאחר שנזק כמו ניקים חד-גדיליים של דנ"א מפחית את מספר האירועים הנצפים, ומגביל את כמות הנתונים שניתן לאסוף (איור 6).
איור 5: בדיקת שחרור דנ"א של מולקולה יחידה. מצע הדנ"א קשור למשטח כיסוי. CMG מטוהר המסומן ב-LD655 מודגר עם הדנ"א במשך 15 דקות ב-ATP-g-s. ATP ו-RPA מטוהר המסומן בתווית EGFP מתווספים, ומתחילים שחרור DNA נרחב על ידי CMG. סכימת קריקטורה (משמאל) וקימוגרף של נתונים מייצגים (מימין) מוצגים. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
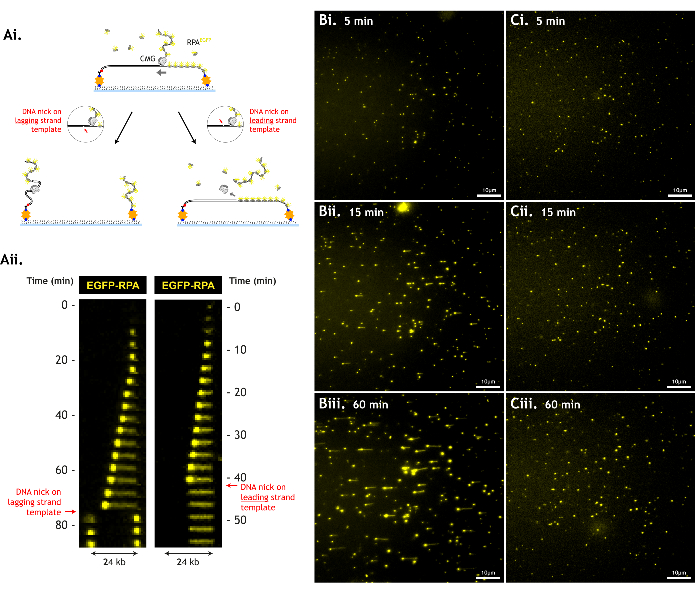
איור 6: נזק לדנ"א מפחית את תפוקת הבדיקה. (A) CMG לא יכול לשחרר דנ"א מעבר להפסקה בעמוד השדרה של הדנ"א (DNA nick). ניק בתבנית הגדיל המוביל גורם ל-CMG להחליק מהדנ"א, וגם תבנית CMG וגם תבנית הגדיל המוביל אובדות. ניק בתבנית הגדיל המפגר גורם לתבנית הגדיל המפגר להיפרד משאר הדנ"א, וכל פיסת דנ"א נסוגה לקשירה המתאימה לה. הדבר מומחש על-ידי (i) סכמות מצוירות ו-(ii) קימוגרפיות של אירועים אלה (ii). נתונים מייצגים עם (B) מצע DNA פגום באופן מינימלי לעומת (C) מצע DNA פגום יותר ב- (i) 5 דקות, (ii) 15 דקות ו- (iii) 60 דקות בשדה ראייה יחיד. מצע הדנ"א הפגוע יותר אינו מייצר רצועות ארוכות של השתחררות, מכיוון שה-CMGs נתקלים בניקים בשלב מוקדם יותר, למרות רמות דומות של פעילות שחרור (צפיפות דומה של כתמי RPA גדלים ב-5 דקות, מה שמצביע על יעילות העמסה/שחרור CMG דומה). שדה הראייה הוא 512 x 512 פיקסלים (גודל פיקסל = 154.6 nm). 1% עוצמת לייזר (488 ננומטר) הדמיה EGFP-RPA. סרגל קנה מידה המציג 10 מיקרומטר. לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
Discussion
בדיקה זו מספקת פלטפורמה להתבונן ולחקור את הדינמיקה בזמן אמת של CMGs בודדים, הן בבידוד והן בהקשר של הגורמים הנוספים הרצויים. עם זאת, כמו בטכניקות פלואורסצנטיות רבות של מולקולה בודדת, ישנם כמה אתגרים נפוצים שיכולים לדרוש אופטימיזציה כדי להתגבר עליהם. אלה מתייחסים בדרך כלל לדימות פלואורופורים לאורך תקופות זמן ארוכות (הלבנה, בהירות), הכנת מצע DNA (נזק לדנ"א), איכות פני התא הזורמים (רעשי רקע, אינטראקציות לא ספציפיות), או איכות הכנת החלבון המטוהר (זיהום נוקלאז, יעילות התוויה).
כל פלואורופור משתנה ביציבות האור ובבהירותו, ולכן חשוב לבחור מולקולה מתאימה. כאשר הדמיה של חלבונים אוליגומריים המסומנים באופן פלואורסצנטי, כמו RPA, ניתן להשתמש בעוצמת לייזר נמוכה יותר מכיוון שפלואורופורים רבים יתעוררו בסמיכות, וייצרו אות נראה. עבור הדמיה של פלואורופורים בודדים, לדוגמה, CMG המסומן על תת-יחידה יחידה, יש צורך בעוצמת לייזר גבוהה יותר כדי לצפות בפלואורופור בבירור. ניתן להאריך את חיי הפלואורופור על ידי מזעור החשיפה ללייזר, למשל על ידי הפחתת התדירות שבה התמונות מצולמות. נוסף על כך, פלואורופור מעורר מייצר מיני חמצן תגובתי (ROS), אשר יכול לתרום להלבנה פוטו. הכללת מערכת חיטוי חמצן במאגר ההדמיה יכולה להאריך את חיי הפלואורופורים על ידי ביטול ROS. עם זאת, כמה מערכות חיטוי חמצן יכול להשפיע על pH12.
לגבי הכנת מצע DNA, חיוני למזער את הנזק לדנ"א, כגון ניקים או רווחים חד-גדיליים. נזק מוגזם מונע שחרור נרחב של הדנ"א, ומגביל את כמות הנתונים שניתן לאסוף. נזק יכול לנבוע מגזירה מכנית, חימום מוגזם, כתוצאה מזיהום נוקלאז, או ROS שנוצר במהלך ההדמיה. ניתן למזער את הגזירה על ידי טיפול זהיר בדגימת הדנ"א על ידי שימוש בקצוות רחבים לפיפט, פיפטינג איטי והימנעות מהזזת הדגימה. ניתן למזער את ההשפעה של ROS על ידי הפחתת החשיפה ללייזר או הכללת מערכת ניקוי חמצן במאגר ההדמיה. לאחר הכנת מצע הדנ"א, ניתן להשתמש בערכות תיקון DNA מסחריות כדי לתקן את הנזק לפני ביצוע תגובה משחררת.
יעילות שחרור הדנ"א תלויה גם בטוהר ובפעילות של CMG. מומלץ להעריך את טוהר הדגימה לאחר כל שלב טיהור על ידי אלקטרופורזה SDS-PAGE כדי לקבוע היכן יש צורך באופטימיזציה. אם נצפים יותר מדי מזהמים לאחר השלב הסופי, זה עשוי לעזור לשנות את נפחי שיפוע המלח המשמשים לפליטה מעמודת CaptoHiRes Q (5/50). כמו כן, חשוב מאוד להסיר כל עודף פפטיד פלואורסצנטי המשמש לסימון החלבון, מכיוון שהוא יכול ליצור רקע לא רצוי על משטח הכיסוי. זה גם חיוני כדי למנוע זיהום נוקלז, כמו זה יכול להשפיל את מצע ה- DNA. לאחר ניסוי, צביעת הדנ"א שנותר בתפוז SYTOX יכולה להיות דרך טובה לבדוק אם הדנ"א פורק באופן משמעותי או לא. רמה מסוימת של נזק לדנ"א היא בלתי נמנעת במהלך ניסוי, אך נזק משמעותי מצביע לעתים קרובות על זיהום נוקלאז בעייתי.
הבדיקה מוגבלת מטבעה גם על ידי הרזולוציה של כתמים מוגבלים בעקיפה, מה שמחייב חלבונים פלואורסצנטיים להיות במרחק של מאות זוגות בסיסים (אם לא יותר) כדי להבדיל ביניהם כנפרדים. זה מגביל את הפרטים שבהם התקדמות CMG ואינטראקציות ניתן לצפות.
מספר האירועים שאנו צופים בהם עבור כל ניתוח משתנה. עבור ניסוי מוצלח, אנו מצפים לראות לפחות כמה קטעי RPA באורך מספיק לכל שדה ראייה 512x512 פיקסלים (גודל פיקסל = 154.6 ננומטר). ניתן לצלם שדות ראייה מרובים באותו ניסוי, מה שמאפשר איסוף נתונים נוסף בעת הצורך. האסופות לא צריכות להיות באותו אורך ולא להגיע לסוף הדנ"א כדי להיות שימושיות. לדוגמה, ניתן לקבוע את מרחק הקשירה הממוצע עבור כל ניסוי על ידי מדידת אורך הדנ"א המוכתם ב-SYTOX לפני הוספת CMG. זה יכול לשמש כדי להעריך כמה דנ"א כבר unwound עבור כל מערכת RPA (כל עוד מספיק DNA הוא unwound כדי להזיז את המזלג באופן גלוי) על ידי המרת המרחק מ "μm נסע" ל "kb unwound".
CMG מפגין פעילות משחררת על מגוון מצעי DNA, אך חיוני לספק קצה DNA חופשי של 3' על דש polyT של לפחות 30 nt כדי להכיל את טביעת הרגל של CMG10. הכללת מוטות ביוטין מרובים במזלג מבטיחה קשירה חזקה של פני השטח. שאר מצע הדנ"א יכול להיות מתוכנן מחדש במגוון דרכים, כגון לכלול רצפי דנ"א שונים, אורכים ושינויים כימיים. הקונפורמציה של ה- DNA יכולה להשתנות באמצעות ריכוזים שונים של מגנזיום אצטט. בריכוזים גבוהים יותר (≥10 מילימול) של מגנזיום אצטט, חוט הלהט ssDNA מצופה RPA נדחס, מה שמוביל למשיכת הדנ"א הנלמדת על ידי קשירת RPA במהלך הרפיה. זה יכול להיות שימושי מכיוון שהוא מונע מהדנ"א לנוע יתר על המידה, ומאפשר למדוד בצורה מדויקת יותר את המיקום של CMG ושל התקדמות משחררת. בריכוזים נמוכים (~ 3 mM) של מגנזיום אצטט, RPA-ssDNA נשאר רגוע לאורך כל הדרך.
הבדיקה המתוארת של מולקולה בודדת מייצגת פלטפורמה שניתן לבנות עליה ולשנות אותה כדי לחקור היבטים נוספים של שכפול DNA. במהלך שכפול הדנ"א, CMG פועל כליבה שהרפליזום ומרכיביו מתאספים סביבה. לכן, ניתן להוסיף חלבונים מטוהרים נוספים לבדיקה זו, כולל גורמים נלווים כמו TIMELESS, TIPIN, ו- CLASPIN, כדי לחקור את השפעתם על דינמיקת CMG. חלבונים אלה הוכחו כמשפיעים על קצב השכפול של מזלגות13, אך לא ברור כיצד הם משפיעים על קצב שחרור CMG. לכן, יהיה מעניין לחקור כיצד חלבונים שונים משפיעים על CMG באמצעות בדיקה זו. תוספת של DNA פולימראז עשויה לתת תובנה טובה יותר לגבי שכפול דנ"א מעבר לשחרור הדנ"א בלבד, כפי שתואר קודם לכן עם חלבוני שמרים14. יתר על כן, טיהור של CMG שונה עשוי לספק הבנה טובה יותר של האופן שבו מוטציות מסוימות או שינויים לאחר תרגום משפיעים על פעילות הליקז15,16. בנוסף, תכנון מצעי דנ"א שונים יכול לאפשר לדנ"א להתפרק על ידי CMG במגוון תנאים המחקים את עקה השכפול17. שינויים אלה כוללים מכשולי דנ"א 9,18, קישורים צולבים בין-גדיליים 19,20,2 1, ואי-רציפות בגדילי הדנ"א22.
Disclosures
למחברים אין אינטרסים כלכליים מתחרים או ניגודי אינטרסים אחרים.
Acknowledgements
אנו מודים לגאורגה צ'יסטול על אספקת פלסמיד pGC261 ולמתקן לביולוגיה כימית של מכון פרנסיס קריק לסינתזה ותיוג פפטידים. עבודה זו מומנה על ידי מכון פרנסיס קריק, המקבל מימון ליבה מ- Cancer Research UK, המועצה הבריטית למחקר רפואי ו- Wellcome Trust (CC2133).
Materials
| Name | Company | Catalog Number | Comments |
| 2-Mercaptoethanol | Sigma-Aldrich | M3148-25ML | |
| Acrylamide / Bis Solution 40% | BioRad | 1610148 | |
| Agarose UltraPure | Invitrogen | 16500-500 | Used to prepare Tris Buffer (pH 8) |
| ÄKTA Pure | GE Healthcare | - | Used with Fraction Collector F9-C; protein purification system |
| ANTI-FLAG M2 affinity gel | Sigma-Aldrich | A2220 | |
| APS 10% | Sigma-Aldrich | A3678-25G | |
| ATP (200 mM) | Sigma-Aldrich | A7699-5G | |
| ATP-g-s (100 mM) | Sigma-Aldrich | 11162306001 | |
| Biotin-PEG coverslip | N/A | N/A | Prepared as described: https://doi.org/10.1016/j.ymeth.2012.03.033 |
| Biotinylated anti-digoxigenin antibody | Perkin Elmer | N/A | |
| Blu Tack | Bostik | N/A | Adhesive putty |
| Boric acid | Thermo Fisher Scientific | B/3800/53 | |
| BSA; 33 mg/mL | SIGMA-ALDRICH | A3858-10G | Diluted from the stock |
| CaCl2 | Sigma-Aldrich | C7902 | |
| CaptoHiRes Q (5/50) | Cytiva | 29275878 | Connected to the AKTA protein purification system; high-resolution ion exchange chromatography column |
| Casein (5% in water, 50 mg/mL) | Sigma-Aldrich | C4765-10ML | |
| ChemiDoc system | Bio-Rad | N/A | |
| Clips | N/A | N/A | |
| Cutting board | N/A | N/A | |
| Dialysis tubing (3.5 kDa) | Fisher Scientific | 11425859 | |
| digoxigenin-11-dUTP | Roche | 11209256910 | |
| DNA loading dye 6x | NEB | B7024S | |
| dNTP 10 mM | NEB | N0447S | |
| Double-sided tape (0.14 mm thick) | N/A | N/A | |
| DTT | Fluorochem Limited | M02712-10G | |
| DYKDDDDK peptide | N/A | N/A | Synthetised by chemical biology STP of The Francis Crick Institute |
| EDTA | Thermo Fisher Scientific | D/0700/68 | |
| EGFP-RPA | N/A | N/A | Purified as desribed in ‘Single Molecule Analysis’, Series Methods in Molecular Biology, Vol. 783 (2011), Peterman, E. J. G. and Wuite G. J. L. editors, Humana Press. Protein information: 2 mM, human, expressed in E. coli. |
| EGTA | Sigma-Aldrich | 03779-10G | |
| Epoxy - 5 minute | Devcon | 20845 | |
| Fujifilm FLA-5000 | Fujifilm | FLA-5000 | Fluorescent image analyzer |
| GelRed Nucleic Acid Stain; 10000x | Cambridge Bioscience | 41003-BT | Example of nucleic acid stain which is a safer alternative to ethidium bromide. |
| Glass or quartz slide with holes for tubing | Dremel Model 395 | 1 mm thick slide, cut to 2.4 cm x 1cm, two holes drilled 1.4 mm apart. The holes should be drilled to different sizes to accommodate different tubing at each end. | |
| Glycerol | Fisher Scientific | G/0650/17 | |
| Glycine HCl, pH 3.5 | Sigma-Aldrich | G8898 | |
| Hamilton syringe, 1000 series GASTIGHT, PTFE luer lock 1010TLL, PTFE Luer lock (with slots), volume 10 mL | Merck | 26211-U | |
| HEPES pH 7.5 | Sigma-Aldrich | H4034 | |
| I-CeuI | NEB | R0699L | |
| KCl | Fisher Scientific | P/4280/53 | |
| Magnesium acetate | Sigma-Aldrich | M2545 | |
| Maxi GeBaFlex-tube Dialysis Kit; MWCO 14 kDa | Generon | D055 | |
| Metal spatula | N/A | N/A | |
| Metal tweezers | N/A | N/A | |
| MgCl2 | Sigma-Aldrich | M2393 | |
| Millex-GP (Syringe filters) 0.22 µm | Merck | SLGP033RS | |
| NaCl | Sigma-Aldrich | S7653 | |
| Needle | N/A | N/A | |
| NotI-HF | NEB | R3189L | |
| NuPAGE 4%–12% Bis-Tris Protein Gels | Thermo Fisher Scientific | 10247002; | |
| NuPAGE MOPS SDS Running Buffer | Thermo Fisher Scientific | NP0001 | |
| Objective heater | Okolab | N/A | |
| PCR mix - 2x | Prepared from Phusion DNA polymerase (20 µL), 10 mM dNTPs (40 µL), 5x Phusion HF buffer (400 µL) and water (540 µL). | ||
| Peptide NH2-CHHHHHHHHHHLPETGG-COOH, labelled with LD655-MAL | N/A | N/A | peptide NH2-CHHHHHHHHHHLPETGG-COOH, labeled with LD655-MAL (Lumidyne Technologies) on the cysteine residue, was synthetised and purified by the peptide chemistry STP of The Francis Crick Institute |
| pGC261 plasmid | N/A | N/A | https://doi.org/10.1016/j.cell.2018.10.053 |
| Phusion High-Fidelity DNA Polymerase | NEB | M0530L | |
| Plastic tweezers | N/A | N/A | |
| Polyethylene tubing PE20 (inner diameter 0.015”, outer diameter 0.043”) | Becton Dickinson | 427406 | |
| Polyethylene tubing PE60 (inner diameter 0.03”, outer diameter 0.048”) | Becton Dickinson | 427416 | |
| Poly-Prep Chromatography Columns; 10 ml and 20 ml | Bio-Rad | 7311550 | |
| Potassium Glutamate (L-glutamic acid potassium salt monohydrate) | Sigma | G1501-1KG | |
| Protease inhibitor cocktail (cOmplete, EDTA free) | Roche | 5056489001 | |
| Pump 11 Elite Infusion/Withdrawal Programmable Single Syringe | Harvard Apparatus | 70-4504 | |
| QIAquick PCR Purification Kit | QIAGEN | 28104 | |
| Razor blade | VWR International Ltd | 233-0156 | |
| rCutSmart Buffer; 10x | NEB | B6004S | |
| Sodium acetate | Thermo Fisher Scientific | S/2120/53 | |
| Sortase (pentamutant) | N/A | N/A | Purified based on: https://doi.org/10.1073/pnas.1101046108 |
| Spin-X Centrifuge Tube Filters; 0.22 µm cellulose acetate | Fisher Scientific | 10310361 | |
| Sterile scalpel | N/A | N/A | |
| Steritop Vacuum Driven Disposable Filtration System; 0.22 um; PES; | Millipore | S2GPT05RE | |
| Streptavidin (1 mg/mL) in 1x PBS buffer | Sigma | S4762-10MG | 20 µL aliquotes |
| SYBR Gold | Thermo Fisher Scientific | S11494 | Highly sensitive nucleic acid stain we used to visualise DNA fork substrate. |
| SYTOX orange; 5 µM in DMSO | Thermo Fisher Scientific | S11368 | |
| T4 DNA ligase | NEB | M0202M | |
| T4 DNA Ligase Reaction Buffer | NEB | B0202S | |
| TEMED | Sigma-Aldrich | T9281-25ML | |
| TEV protease (1 mg/mL); EZCut | Biovision | 7847-10000 | |
| Tissue grinders, Dounce type (40 mL, Wheaton) | DWK Life Sciences | 432-1273 | |
| Tris base | Sigma-Aldrich | T1503 | Used to prepare Tris Buffer (pH 8) |
| Tris HCl | Sigma-Aldrich | T3253 | |
| Tween-20 | Promega UK Ltd | H5152 |
References
- Bell, S. P., Labib, K. Chromosome Duplication in Saccharomyces cerevisiae. Genetics. 203 (3), 1027-1067 (2016).
- Costa, A., et al. The structural basis for MCM2-7 helicase activation by GINS and Cdc45. Nat Struct Mol Biol. 18 (4), 471-477 (2011).
- Jones, M. L., Baris, Y., Taylor, M. R. G., Yeeles, J. T. P. Structure of a human replisome shows the organisation and interactions of a DNA replication machine. EMBO J. 40 (23), e108819(2021).
- Moyer, S. E., Lewis, P. W., Botchan, M. R. Isolation of the Cdc45/Mcm2-7/GINS (CMG) complex, a candidate for the eukaryotic DNA replication fork helicase. Proc Natl Acad Sci U S A. 103 (27), 10236-10241 (2006).
- Douglas, M. E., Ali, F. A., Costa, A., Diffley, J. F. X. The mechanism of eukaryotic CMG helicase activation. Nature. 555 (7695), 265-268 (2018).
- Ilves, I., Petojevic, T., Pesavento, J. J., Botchan, M. R. Activation of the MCM2-7 helicase by association with Cdc45 and GINS proteins. Mol Cell. 37 (2), 247-258 (2010).
- Dewar, J. M., Walter, J. C. Mechanisms of DNA replication termination. Nat Rev Mol Cell Biol. 18 (8), 507-516 (2017).
- Burnham, D. R., Kose, H. B., Hoyle, R. B., Yardimci, H. The mechanism of DNA unwinding by the eukaryotic replicative helicase. Nat Commun. 10 (1), 2159(2019).
- Kose, H. B., Larsen, N. B., Duxin, J. P., Yardimci, H. Dynamics of the eukaryotic replicative helicase at lagging-strand protein barriers support the steric exclusion model. Cell Rep. 26 (8), 2113-2125.e6 (2019).
- Kose, H. B., Xie, S., Cameron, G., Strycharska, M. S., Yardimci, H. Duplex DNA engagement and RPA oppositely regulate the DNA-unwinding rate of CMG helicase. Nat Commun. 11 (1), 3713(2020).
- Tanner, N. A., Van Oijen, A. M. Visualizing DNA replication at the single-molecule level. Methods Enzymol. 475, 259-278 (2010).
- Swoboda, M., et al. Enzymatic oxygen scavenging for photostability without ph drop in single-molecule experiments. ACS Nano. 6 (7), 6364-6369 (2012).
- Baris, Y., Taylor, M. R. G., Aria, V., Yeeles, J. T. P. Fast and efficient DNA replication with purified human proteins. Nature. 606, 204-210 (2022).
- Lewis, J. S., et al. Tunability of DNA polymerase stability during eukaryotic DNA replication. Mol Cell. 77 (1), 17-25.e5 (2020).
- Li, Z., Xu, X. Post-translational modifications of the mini-chromosome maintenance proteins in DNA replication. Genes. 10 (5), 331(2019).
- Lewis, J. S., et al. Mechanism of replication origin melting nucleated by CMG helicase assembly. Nature. 606 (7916), 1007-1014 (2022).
- Zeman, M. K., Cimprich, K. A. Causes and consequences of replication stress. Nat Cell Biol. 16 (1), 2-9 (2014).
- Sparks, J. L., et al. The CMG helicase bypasses DNA-protein cross-links to facilitate their repair. Cell. 176 (1-2), 167-181.e21 (2019).
- Taylor, M. R. G., Yeeles, J. T. P. The initial response of a eukaryotic replisome to DNA damage. Mol Cell. 70 (6), 1067-1080.e12 (2018).
- Amunugama, R., et al. Replication fork reversal during DNA interstrand crosslink repair requires CMG unloading. Cell Rep. 23 (12), 3419-3428 (2018).
- Wu, R. A., et al. TRAIP is a master regulator of DNA interstrand crosslink repair. Nature. 567 (7747), 267-272 (2019).
- Vrtis, K. B., Dewar, J. M., Chistol, G., Wu, R. A., Graham, T. G. W., Walter, J. C. Single-strand DNA breaks cause replisome disassembly. Mol Cell. 81 (6), 1309-1318.e6 (2021).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved