Method Article
Visualizzazione in tempo reale a singola molecola dello svolgimento del DNA mediante elicasi CMG
In questo articolo
Riepilogo
Questo protocollo dimostra l'esecuzione di un saggio a singola molecola per la visualizzazione dal vivo dello svolgimento del DNA mediante elicasi CMG. Descrive (1) la preparazione di un substrato di DNA, (2) la purificazione dell'elicasi CMG di Drosophila melanogaster marcata in fluorescenza, (3) la preparazione di una cella a flusso microfluidico per la microscopia a fluorescenza a riflessione interna totale (TIRF) e (4) il saggio di svolgimento del DNA a singola molecola.
Abstract
La duplicazione fedele del genoma è essenziale per preservare la stabilità genetica delle cellule in divisione. La replicazione del DNA viene effettuata durante la fase S da un complesso dinamico di proteine chiamato replosoma. Al centro del replisoma c'è l'elicasi CDC45-MCM2-7-GINS (CMG), che separa i due filamenti della doppia elica del DNA in modo tale che le DNA polimerasi possano copiare ciascun filamento. Durante la duplicazione del genoma, i replisomi devono superare una pletora di ostacoli e sfide. Ognuno di questi minaccia la stabilità del genoma, poiché l'incapacità di replicare il DNA in modo completo e accurato può portare a mutazioni, malattie o morte cellulare. Pertanto, è di grande interesse capire come funziona il CMG nel reposoma sia durante la normale replicazione che durante lo stress di replicazione. Qui, descriviamo un saggio di microscopia a fluorescenza a riflessione interna totale (TIRF) utilizzando proteine purificate ricombinanti, che consente la visualizzazione in tempo reale di molecole di DNA allungate legate alla superficie da singoli complessi CMG. Questo test fornisce una potente piattaforma per studiare il comportamento del CMG a livello di singola molecola, consentendo di osservare direttamente la dinamica dell'elicasi con un controllo in tempo reale sulle condizioni di reazione.
Introduzione
La replicazione del DNA è strettamente regolata, poiché una cellula deve duplicare accuratamente il proprio genoma per prevenire mutazioni, malattie o morte. La replicazione del DNA eucariotico viene effettuata dal complesso replosomico, che svolge il DNA parentale e utilizza il DNA a filamento singolo (ssDNA) come modello per sintetizzare nuovo DNA. Nella fase G1, i doppi esameri cataliticamente inattivi di MCM2-7 vengono caricati su DNA a doppio filamento (dsDNA) alle origini di replicazione1. Nella fase S, i complessi MCM2-7 sono attivati dal legame di CDC45 e GINS2 per formare complessi CMG a 11 subunità (CDC45, MCM2-7, GINS). Ogni CMG avvia il srotolamento del DNA in direzioni opposte, formando l'unità centrale attorno alla quale il replisoma si dispone attorno a3.
Due decenni fa l'elicasi CMG è stata identificata per la prima volta come un complesso di 11 subunità, essenziale per la replicazione del DNA4. Da allora, la nostra comprensione del CMG è progredita considerevolmente, dal caricamento e attivazione 5,6, allo svolgimento e alla terminazione del DNA7. Le tecniche tradizionali di biochimica e biologia strutturale sono state fondamentali per molte di queste scoperte; tuttavia, questi metodi erano spesso limitati nella loro capacità di studiare gli aspetti più dinamici della CMG. I metodi a singola molecola utilizzano la manipolazione fisica di singole biomolecole per misurare o visualizzare la loro attività una molecola alla volta. Questo può essere utilizzato per fornire informazioni sulla dinamica in tempo reale di proteine che spesso non vengono rilevate o non rilevate da altre tecniche 8,9.
Qui, descriviamo un saggio di microscopia a fluorescenza a riflessione interna totale (TIRF) per visualizzare lo svolgimento del DNA mediante elicasi CMG in tempo reale. Il CMG purificato e marcato in fluorescenza viene caricato sull'estremità libera 3' del DNA lungo contenente una struttura a forcella di DNA prefabbricata. Il DNA lineare viene allungato su un vetrino coprioggetti biotina-PEG in una cella a flusso microfluidico legando ciascuna estremità del DNA in sequenza alla superficie. Questo approccio consente un tethering del DNA più uniforme, che riduce significativamente la variazione di cui è necessario tenere conto durante l'analisi dei dati. In presenza di ATP-γ-s, il CMG viene caricato sul DNA a singolo filamento all'estremità 3' della forcella. L'ATP-γ-s è un analogo dell'ATP lentamente idrolizzabile, che consente al CMG di legarsi al DNA ma non di srotolarsi. La successiva aggiunta di ATP, insieme all'RPA purificato e marcato in fluorescenza, attiva il CMG e avvia un'ampia svolgimento del DNA. Visivamente, il CMG trasloca lungo il DNA, lasciando dietro di sé un tratto crescente di ssDNA legato all'RPA. L'estremità del DNA libera viaggia con il CMG, formando una "palla stretta" a causa della compattazione causata dal legame RPA. Il design della cella di flusso consente di sostituire il tampone in qualsiasi momento durante lo svolgimento, offrendo un ottimo controllo durante e durante ogni esperimento.
Questo protocollo è suddiviso in quattro metodi, che possono essere eseguiti indipendentemente l'uno dall'altro. La sezione 1 descrive la preparazione di un substrato di DNA biforcato lineare da 20 kb per saggi su singola molecola. La sezione 2 delinea la purificazione e la marcatura fluorescente di Drosophila melanogaster CMG (DmCMG). Le informazioni chiave sull'espressione di DmCMG sono incluse nella sezione note. La sezione 3 riguarda la preparazione di una cella a flusso microfluidica che può essere utilizzata su un microscopio TIRF. La sezione 4 descrive come eseguire il saggio di svolgimento del DNA a singola molecola.
Protocollo
1. Preparazione di DNA biforcato lineare da 20 kb utilizzato in saggi a singola molecola (Figura 1)
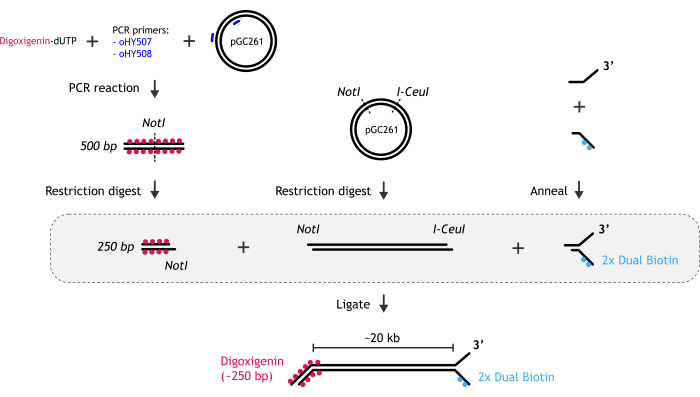
Figura 1: Rappresentazione grafica della preparazione del substrato di DNA. (A) L'estremità della forcella del DNA biotinilato viene creata mediante ricottura di due oligonucleotidi parzialmente complementari: biotinilato e non biotinilato. (B) Il frammento principale di dsDNA (~20 kb) è generato dalla restrizione del plasmide pGC261 con due enzimi per creare un DNA lineare con diverse sporgenze a ciascuna estremità. (C) L'estremità duplex DNA della digossigenina è ottenuta mediante una reazione PCR eseguita in presenza di digossigenina-dUTP, seguita da digest di restrizione. Clicca qui per visualizzare una versione più grande di questa figura.
- Generazione di estremità della forcella di DNA biotinilato.
- Ricottura del DNA della forchetta.
- Miscelare due oligonucleotidi (oHY502 e oHYbio85) in 1x tampone STE (100 mM NaCl, 10 mM Tris, pH 8,0; 1 mM di acido etilendiamminotetraacetico [EDTA]). La concentrazione finale di oligonucleotidi è di 10 μM e il volume di reazione finale è di 100 μL.
- Incubare la reazione a 80 °C per 5 minuti su un blocco termico. Spegnere il blocco termico e lasciare raffreddare gradualmente la reazione, lasciandolo nel blocco termico fino a quando la temperatura non scende sotto i 30 °C.
- Convalidare l'efficienza di ricottura su un gel per elettroforesi su gel di tris-borato-EDTA (TBE)-poliacrilammide (PAGE).
- Preparare il gel TBE all'8% di acrilammide (per 4 gel: 32,8 mL di H2O; 4,8 mL di tampone 10x TBE (1 M Tris, 1 M acido borico, 20 mM EDTA), 9,6 mL di soluzione di acrilammide/bis al 40%, 37,5:1; 0,8 mL di persolfato di ammonio al 10% [APS], 40 μL di tetrametiletilendiammina [TEMED]).
ATTENZIONE: TBE, acrilammide, APS, TEMED sono tossici/nocivi. - Far scorrere un piccolo volume (1 μL) di oligonucleotidi ricotti insieme ai singoli oligonucleotidi diluiti con colorante di carico 6x sul gel di acrilammide - TBE all'8% a 80 V, per 1 ora a RT.
- Immergere il gel in 1x TBE integrato con un colorante di acido nucleico adatto per 30 minuti su uno shaker oscillante, quindi scansionare il gel utilizzando un sistema di imaging su gel.
- Preparare il gel TBE all'8% di acrilammide (per 4 gel: 32,8 mL di H2O; 4,8 mL di tampone 10x TBE (1 M Tris, 1 M acido borico, 20 mM EDTA), 9,6 mL di soluzione di acrilammide/bis al 40%, 37,5:1; 0,8 mL di persolfato di ammonio al 10% [APS], 40 μL di tetrametiletilendiammina [TEMED]).
- Risolvere il DNA ricotto sul gel TBE-PAGE per l'escissione del gel.
- Se gli oligonucleotidi vengono ricotti correttamente, risolvere il resto del campione in presenza di BSA (~100 μL di campione ricotto, 25 μL di colorante di caricamento 6x, 2 μL di 33 mg/mL di BSA). Per caricare il volume maggiore sul gel, combinare diversi pozzetti con un bisturi sterile.
- Lasciare il gel in 1x TBE integrato con un colorante di acido nucleico adatto per 30 minuti su uno shaker oscillante.
- Trasferisci il gel in un vassoio di vetro e visualizza il DNA in una stanza buia sotto la luce blu. Utilizzare uno schermo protettivo adatto o occhiali protettivi quando si utilizza la luce blu.
- Ritaglia la fascia desiderata con un bisturi sterile. Elimina il gel in eccesso.
- Purificare la forca ricottura mediante elettroeluizione.
- Tagliare un pezzo di tubo per dialisi abbastanza a lungo da inserire il pezzo di gel, bagnarlo in 1x TBE e chiudere un'estremità.
- Diluire BSA a 0,3 mg/mL in 500 μL di 1x TBE e pipettare l'intero volume nel tubo.
- Usa la spatola di metallo per far scorrere il pezzo di gel all'interno. Spostare il pezzo di gel su un lato del tubo per dare spazio tampone in cui il DNA possa migrare durante l'elettroeluizione.
- Spremere il tampone in eccesso e agganciare la seconda estremità del tubo.
NOTA: Il volume rimanente determinerà il volume e la concentrazione finali. - Posizionare il tubo per dialisi nel serbatoio per elettroforesi dell'agarosio in 1 tampone TBE, assicurandosi che il tubo sia completamente sommerso.
NOTA: Assicurarsi che il pezzo di gel sia posizionato sul lato più vicino all'elettrodo negativo in modo che il DNA possa migrare fuori dal gel nella sacca per dialisi. - Far funzionare l'elettroeluizione a 80 V per 1-2 ore.
- Dializzare il DNA elettroeluito in 10 mM di tampone Tris (pH 8).
- Dopo l'elettroeluizione, estrarre la sacca per dialisi dal serbatoio e asciugare un'estremità con un fazzoletto. Evitare di toccare la parte centrale del tubo con il fazzoletto, dove si trovano il gel e il campione.
- Rimuovere la clip dall'estremità essiccata e pipettare delicatamente il campione su e giù all'interno del tubo per mescolare il DNA nel tampone.
- Immergi una spatola pulita in 1x tampone TBE e raccogli il pezzo di gel dal tubo.
NOTA: Durante la rimozione del pezzo di gel, rimuovere il meno tampone possibile. - Riagganciare l'estremità aperta della sacca per dialisi, rimuovendo l'aria dall'interno.
- Posizionare la sacca per dialisi in un becher da 2 L riempito con 1,5 L di tampone Tris 10 mM (pH 8), 20 mM di NaCl e 2 mM di MgCl2. Avvolgere le clip con parafilm per attaccare il tubo al bordo del becher mentre si immerge completamente il tubo nel tampone.
NOTA: Il sale stabilizza i duplex oligo corti. - Utilizzare una barra magnetica per mescolare delicatamente il tampone e dializzare il campione a temperatura ambiente (RT) per almeno 3-4 ore o a 4 °C durante la notte.
- Dopo la dialisi, asciugare un'estremità della sacca per dialisi con un fazzoletto (per non diluire il DNA) e sganciare questa estremità.
- Pipettare delicatamente il campione su e giù all'interno del tubo e trasferirlo in una provetta pulita da 1,5 ml.
- Misurare la concentrazione del DNA utilizzando uno spettrofotometro a microvolume.
- Ricottura del DNA della forchetta.
- Generazione di un frammento da 20 kb
- Eseguire il digest di restrizione del plasmide18 pGC261 utilizzando gli enzimi NotI/I-CeuI.
- Miscelare delicatamente 8 μL di NotI-HF (20.000 U/mL), 8 μL di I-CeuI (5.000 U/mL) e il plasmide pGC261 (concentrazione finale del plasmide ~40 ng/μL in una reazione di 200 μL) in un tampone raccomandato dai produttori di enzimi di restrizione.
- Incubare la reazione a 37 °C per una notte.
- Risolvi il DNA digerito su gel di agarosio per l'escissione del gel.
- FACOLTATIVO: Eseguire una piccola quantità della reazione su un gel di agarosio allo 0,6% per testare l'efficienza della digestione prima di caricare l'intera reazione.
- Preparare il gel di agarosio allo 0,6% seguendo i passaggi 1.2.2.3-1.2.2.4.
- Mescolare 0,48 g di polvere di agarosio con 80 ml di tampone 1x TBE. Cuocere la soluzione nel microonde fino a quando non bolle. Agitare per assicurarsi che l'agarosio sia completamente sciolto. Lasciar raffreddare per qualche minuto, quindi versare nell'apposito serbatoio.
- Per combinare i pozzetti in modo da accogliere il volume maggiore del campione, attaccare prima un pezzo di nastro adesivo su più pozzetti del pettine.
- Una volta che il gel si è solidificato, rimuovere delicatamente il pettine e riempire il serbatoio con 1x tampone TBE. Mescolare delicatamente il campione con il colorante che carica DNA e far scorrere il gel a 120 V per 1 ora.
- Lasciare il gel in 1x TBE integrato con un colorante di acido nucleico adatto su uno shaker oscillante a dondolo fino a quando non si macchia.
- Trasferisci il gel colorato in un vassoio di vetro e visualizza il DNA in una stanza buia sotto la luce blu.
- Ritaglia la fascia desiderata con un bisturi sterile. Elimina il gel in eccesso.
- Opzionale: Purificare il frammento desiderato mediante elettroeluizione come descritto nei passaggi 1.1.4 e 1.1.5. Omettere qui il sale dal tampone per dialisi, poiché è necessario solo per stabilizzare l'oligo duplex corto.
- Eseguire il digest di restrizione del plasmide18 pGC261 utilizzando gli enzimi NotI/I-CeuI.
- Generazione di digossigenina duplex DNA end
- Eseguire la reazione PCR con digossigenina-dUTP (dig-dUTP) utilizzando il DNA pGC261 come stampo.
- Miscela: 400 μL di acqua, 8 μL di pGC261 (0,8 ng/μL), 3,5 μL di primer oHY507 (100 μM), 3,5 μL di primer oHY508 (100 μM), 8 μL di dig-dUTP (1 mM), 400 μL di 2 miscele PCR (preparate da DNA polimerasi (20 μL), 10 mM di dNTP (40 μL), 5 tamponi ad alta fedeltà (HF) (400 μL) e acqua (540 μL).
- Eseguire la seguente reazione in un termociclatore:
98 °C - 1 min
30x: 98 °C - 20 s; 65 °C - 20 s; 72 °C - 30 s;
72 °C - 10 min
4 °C - Mantenimento
- Digerire e purificare il prodotto PCR marcato con digossigenina
- Purificare il prodotto PCR utilizzando un kit di purificazione del DNA commerciale.
- Miscelare 10 μL di NotI-HF (20.000 U/mL) con il prodotto della PCR in un tampone raccomandato dal fornitore dell'enzima (concentrazione finale di DNA di ~50 ng/μL in una reazione di ~200 μL). Incubare la reazione a 37 °C per una notte.
- Purifica il DNA digerito utilizzando un kit di purificazione del DNA commerciale.
- Eseguire la reazione PCR con digossigenina-dUTP (dig-dUTP) utilizzando il DNA pGC261 come stampo.
- Assembla i componenti per creare il substrato del DNA.
- Mescolare delicatamente l'estremità della forchetta biotinilata, il frammento di DNA da 20 kb e il frammento di PCR trattato con Not-I marcato con digossigenina con 5 μL di DNA ligasi T4 (400.000 U/mL) in un tampone raccomandato dal fornitore dell'enzima in una reazione da 200 μL. Tuttavia, in base al frammento di DNA di 20 kb aggiunto (1-5 μg), aggiungere un eccesso molare di circa 100 volte sia dell'estremità della forcella biotinilata che del frammento di PCR.
- Aliquotare la reazione in provette per PCR (50 μl ciascuna) e incubare a 16 °C in un termociclatore per una notte.
- Risolvere il campione legato su un gel di agarosio allo 0,6% e purificare il DNA legato mediante elettroeluizione come descritto nei passaggi 1.2.2 e 1.2.3.
- Conservare il DNA congelato a -80 °C.
2. Purificazione di Drosophila melanogaster CMG (Figura 2)

Figura 2: Purificazione di Drosophila melanogaster CMG da 4 L di cellule Hi Five. Le proteine sono state risolte su gel di poliacrilammide Bis-Tris al 4%-12% sotto i 200 V in presenza di tampone MOPS. Il campione viene mostrato in ogni fase della purificazione (lisato cellulare - 2 μL, eluizione FLAG - 10 μL, dopo la prima colonna a scambio ionico - 10 μL e dopo la marcatura e la seconda colonna a scambio ionico - 1 μL). (A) La colorazione di Coomassie conferma la presenza di tutte le 11 subunità del complesso CMG prima (10 μL) e dopo (1 μL) la marcatura fluorescente. (B) L'efficienza di marcatura della subunità MCM3 è stata convalidata mediante scansione di Cy5 con un analizzatore di immagini fluorescenti utilizzando un filtro rosso passa-lungo (LPR). Clicca qui per visualizzare una versione più grande di questa figura.
NOTA: Per preparare un CMG marcato con Drosophila melanogaster in fluorescenza, è stato introdotto un sito di scissione TEV (ENLYFQG) seguito da quattro residui di Gly a valle del tag FLAG N-terminale sulla subunità MCM3 (nel vettore pFastBac1)10. Per esprimere il complesso, è stato utilizzato il sistema di espressione del baculovirus. Per la trasfezione iniziale, le cellule Sf21 sono state utilizzate separatamente per ciascuna subunità CMG (stadio del virus P1). Per amplificare ulteriormente i virus, sono state utilizzate cellule Sf9 (stadio virale P2). Successivamente, le colture cellulari Sf9 (100 mL per ciascuna subunità CMG; 0,5 x 106 cellule/mL) sono state infettate con 0,5 mL di virus P2 integrato con il 10% di siero fetale di vitello (stadio del virus P3). Per esprimere l'intero complesso CMG in 4 L di cellule Hi Five (1 x 106 cellule/mL), sono stati utilizzati 200 mL di virus P3 per ciascuna delle subunità. Dopo aver raccolto le cellule Hi Five che esprimono il complesso CMG, il pellet cellulare può essere congelato in azoto liquido e conservato a -80°C. Eseguire l'intera purificazione su ghiaccio o a 4 °C. I tamponi possono essere preparati in anticipo, a condizione che gli agenti riducenti (DTT o 2-Mercaptoetanolo) e gli inibitori della proteasi (ATTENZIONE) vengano aggiunti appena prima dell'uso. Assicurarsi che tutti i tamponi siano preventivamente preraffreddati, filtrati e degassati.
- Preparare i seguenti buffer.
- Preparare il tampone di risospensione A miscelando 25 mM di acido 4-(2-idrossietil)-1-piperazineetanesolfonico (HEPES) pH 7,5, 0,02% Tween-20, 10% glicerolo, 15 mM KCl, 2 mM MgCl2, 2 mM 2-mercaptoetanolo, 1 mM EDTA e 1 mM di glicole etilenico-bis (β-amminoetil etere)-N,N,N′,N′-acido tetraacetico (EGTA).
ATTENZIONE: il 2-mercaptoetanolo, il DTT e l'EDTA sono tossici/nocivi - Preparare il tampone salino tamponato Tris (TBS; 0,1 M Glycine HCl, pH 3,5) per la preparazione del gel di affinità ANTI-FLAG M2.
- Preparare il tampone A-100 miscelando 25 mM HEPES pH 7,5, 0,02% Tween-20, 10% glicerolo, 100 mM KCl, 1 mM DTT, 1 mM EDTA e 1 mM EGTA.
- Preparare il tampone per dialisi aggiungendo 25 mM di HEPES pH 7,5, 50 mM di acetato di sodio, 10 mM di acetato di magnesio, 10% di glicerolo e 1 mM di DTT.
- Preparare il tampone TBS miscelando 50 mM di Tris-HCl pH 7,5 e 150 mM di NaCl.
- Preparare il tampone di risospensione A miscelando 25 mM di acido 4-(2-idrossietil)-1-piperazineetanesolfonico (HEPES) pH 7,5, 0,02% Tween-20, 10% glicerolo, 15 mM KCl, 2 mM MgCl2, 2 mM 2-mercaptoetanolo, 1 mM EDTA e 1 mM di glicole etilenico-bis (β-amminoetil etere)-N,N,N′,N′-acido tetraacetico (EGTA).
- Pellet di risospensione da 4 L di colture Hi Five in 200 mL di tampone di risospensione fredda A integrato con compresse cocktail di inibitori della proteasi. Miscelare i tubi per inversione fino a quando l'intero pellet è completamente risospeso.
- Trasferire le cellule risospese in un omogeneizzatore Dounce da 40 mL preraffreddato. Per recuperare il maggior numero possibile di cellule, lavare le provette utilizzate per il congelamento dei pellet cellulari con lo stesso tipo di tampone e aggiungerle all'omogeneizzatore Dounce. Assicurarsi che il volume totale nell'omogeneizzatore non superi il limite consigliato. Lisare le cellule sul ghiaccio con 60-70 colpi.
NOTA: Assicurarsi che lo stantuffo raggiunga il fondo del tubo, ma non esercitare troppa pressione poiché l'omogeneizzatore potrebbe rompersi. Fare attenzione a non estrarre lo stantuffo oltre il livello del liquido, poiché ciò introdurrà bolle. - Ripetere il passaggio precedente fino a quando l'intero campione non è omogeneizzato e unirlo nel becher preraffreddato. Valutare il volume finale del campione. Goccia a goccia, aggiungere KCl filtrato alla concentrazione finale di 100 mM, mescolando delicatamente.
- Preraffreddare la centrifuga a 4 °C in anticipo. Versare il campione nelle provette da centrifugazione, assicurandosi che venga raggiunto il volume raccomandato dal produttore. Bilanciare i tubi su una bilancia.
NOTA: Le provette con un volume troppo piccolo possono rompersi durante la centrifugazione. - Centrifugare le celle lisate a 23.500 x g per 15-30 minuti a 4 °C. Nel frattempo, preparare il gel di affinità ANTI-FLAG M2 in linea con il passaggio successivo.
- Mescolare delicatamente il flacone contenente il gel di affinità ANTI-FLAG M2 (50% sospensione). Tagliare l'estremità di un puntale per pipetta P-100 (per allargare il puntale) e utilizzarlo per trasferire immediatamente 4 mL di sospensione (2 mL di perline) in una colonna cromatografica Poly-Prep da 20 mL. Per recuperare le perline incastrate nella punta, lavare la punta con TBS e aggiungerla alla colonna. Lascia che tutte le perline si depositino all'interno della colonna, ma fai attenzione a non asciugarle.
- Lavare la resina con 3 perle di volume (6 ml totali) di 0,1 M di glicina HCl pH 3,5. Successivamente, lavare le perle con 3-5 perle volume di TBS (6-10 mL). Quindi, lavare le perline 3 volte (6 ml) con il tampone A-100.
NOTA: Non lasciare le perle nella soluzione 0.1 M Glycine HCl pH 3.5 per più di 20 minuti. Per aumentare la pressione del flusso e la velocità di lavaggio, coprire la colonna con un coperchio. Non lasciare che la resina si asciughi in nessun punto. - Dopo aver preparato la resina, lasciare 2 mL di Buffer A-100 sopra la resina e chiudere l'uscita della colonna.
- Dopo la centrifugazione, versare delicatamente il surnatante in provette preraffreddate da 50 mL. Fare attenzione a non rompere il pellet. Prelevare una piccola aliquota (~10 μL) per convalidarla mediante elettroforesi SDS-PAGE in un secondo momento.
- Aggiungere una quantità uguale di perle ANTI-FLAG M2 (preparate in linea con il passaggio precedente) in ciascuna delle provette da 50 mL. Cercate di recuperare tutte le perle dalla colonna risospendendole con un paio di millilitri aggiuntivi di Buffer A-100 e trasferendo questa risospensione nei tubi.
- Incubare il campione (surnatante) con la resina ANTI-FLAG M2, ruotando per 2,5 ore a 4 °C.
- Dopo l'incubazione, centrifugare il campione a 1.000 x g per 5 minuti a 4 °C. Utilizzando una pipetta, rimuovere la maggior parte del surnatante. Utilizzando una punta tagliata, risospendere le perle in pochi millilitri del surnatante rimanente e trasferirle in due provette da 15 ml. Per recuperare eventuali perle attaccate alle pareti delle provette da 50 mL, aggiungere alcuni millilitri di Buffer A-100 e trasferirlo anche nelle provette da 15 mL.
- Centrifugare le provette da 15 mL a 1.000 x g per 5 minuti a 4 °C. Rimuovere il surnatante.
- Lavare le perle aggiungendo ~14 mL di Buffer A-100 a ciascuna provetta, seguita da una rotazione breve e delicata a 4 °C e successiva centrifuga a 1000 x g per 5 minuti a 4 °C. Ripetere i lavaggi due volte. Prelevare piccole aliquote di ogni lavaggio (~10 μL) per convalidarle successivamente mediante elettroforesi SDS-PAGE.
- Questo passaggio (2.16) è una procedura alternativa ai passaggi precedenti (2.13-2.15).
- Dopo l'incubazione con la resina ANTI-FLAG M2, versare il campione in due colonne cromatografiche poly-prep da 20 mL.
- Se si sceglie questo metodo, lasciare che le perle si depositino sul fondo della colonna e che il campione non legato attraversi la colonna per gravità. Successivamente, aggiungere il tampone A-100 direttamente alla colonna e lasciare che lavi il campione tramite flusso per gravità.
- Trasferire le microsfere (risospese nel tampone A-100) in due colonne per cromatografia poli-prep da 10 mL.
- Lasciare che il tampone A-100 passi attraverso la resina, chiudere l'uscita quando il livello del tampone raggiunge la parte superiore della resina ed eluire la proteina dalle perle aggiungendo il tampone A-100 integrato con 200 μg/mL (DYKDDDDK) peptide (tampone di eluizione FLAG).
- Per la prima eluizione, aggiungere 3 mL del tampone di eluizione FLAG e ruotare delicatamente la colonna chiusa per 15 minuti a RT. Aprire l'uscita per raccogliere la prima frazione di eluizione.
- Chiudere l'uscita e aggiungere 2 mL del tampone di eluizione FLAG a ciascuna colonna. Ruotare per altri 10 minuti e raccogliere la seconda frazione di eluizione.
- Combinare tutte le frazioni eluite insieme (~10 mL) e mantenere a 4 °C. Prelevare una piccola aliquota della frazione eluita (~10 μL) per convalidarla mediante elettroforesi SDS-PAGE in un secondo momento.
- Filtrare il campione eluito utilizzando filtri per siringa da 0,22 μm.
- In anticipo, preparare la colonna CaptoHiRes Q (5/50) (colonna per cromatografia a scambio ionico ad alta risoluzione) collegata a un sistema di purificazione delle proteine, in linea con il protocollo del produttore. Successivamente, equilibrare la colonna con il tampone A-100 e caricare il campione filtrato sulla colonna bilanciata.
- Lavare la colonna con 20 volumi di colonna (CV) di tampone A-100 (~20 mL in totale, poiché il volume della colonna è ~1 mL).
- Per eluire la proteina, preparare in anticipo due tamponi: il tampone A-100 e il tampone B con la stessa composizione del tampone A-100, ma con 550 mM di KCl invece di 100 mM di KCl. Impostare l'eluizione a gradiente per 20 CV (~20 mL) con una concentrazione salina crescente da 100 mM a 550 mM KCl. Raccogliere 0,3-0,5 mL di eluizioni in un collettore di frazioni.
NOTA: Il CMG dovrebbe eluire a circa il 70%-75% di concentrazione del tampone B. Prelevare piccole aliquote delle frazioni di lavaggio e eluite per l'elettroforesi SDS-PAGE. - Eseguire l'elettroforesi SDS-PAGE per confermare che il CMG è presente nelle frazioni scelte. Nel frattempo, pulire la colonna e il sistema di purificazione secondo il protocollo del produttore.
- Incubare le frazioni scelte con proteasi TEV durante la notte mescolando 50 μL di proteasi TEV (1 mg/mL) per 1 mL di campione.
- Prebagnare una provetta per dialisi e assicurarsi che la membrana non sia danneggiata. Aggiungere la miscela CMG/TEV nel tubo per dialisi e metterla in un becher da 2 L con 1,5 L di tampone A-100 preraffreddato e la barra magnetica all'interno. Dializzare mescolando delicatamente per una notte a 4°C.
- Miscelare il campione con 50 μM di peptide marcato con LD655 e 10 μg/mL di enzima sortasi in presenza di 5 mM di CaCl2. Incubare la reazione a 4 °C, ruotando per 30 minuti, assicurandosi che la provetta sia coperta dalla luce. Prelevare una piccola aliquota prima e dopo la marcatura per l'elettroforesi SDS-PAGE.
- Filtrare il campione marcato utilizzando filtri centrifughi da 0,22 μm prima di caricarlo sul sistema di purificazione, poiché il peptide potrebbe precipitare.
- Preparare in anticipo la colonna per cromatografia a scambio ionico ad alta risoluzione, come descritto in precedenza (passaggio 2.20). Questa volta, coprire la colonna e il sistema di raccolta delle frazioni con un foglio di alluminio per proteggere il campione dalla luce.
- Per rimuovere il peptide libero, caricare il campione filtrato sulla colonna cromatografica a scambio ionico ad alta risoluzione. Eseguire la purificazione nello stesso modo descritto in precedenza (passaggi 2.21-2.22). Assicurarsi che il CMG etichettato eluisca a una concentrazione di Buffer-B simile a quella precedente.
- Eseguire l'elettroforesi SDS-PAGE per convalidare la qualità delle frazioni scelte. Per visualizzare la fluorescenza, non far bollire i campioni prima di caricarli e assicurarsi che il serbatoio dell'elettroforesi sia protetto dalla luce. Visualizza prima la fluorescenza delle proteine utilizzando un sistema di imaging su gel, quindi colora il gel con il colorante Coomassie e riproduci un'immagine per visualizzare tutte le proteine.
- Dializzare le frazioni scelte per una notte a 4 °C, contro 1,5 L del tampone di dialisi. Se necessario, concentrare il campione.
- Congelare la proteina in azoto liquido e conservarla a -80 °C fino a nuovo utilizzo.
3. Preparazione della cella a flusso (Figura 3)
- Preparare i vetrini coprioggetti biotina-PEG seguendo un protocollo11 precedentemente descritto, omettendo la fase di cottura in forno. I vetrini coprioggetti Biotina-PEG sono stabili per almeno 1 mese sotto vuoto a RT. Per la preparazione della cella a flusso, tagliare a metà il vetrino coprioggetti in biotina-PEG (24 mm x 60 mm) (circa 24 mm x 30 mm).
NOTA: Fare attenzione a non toccare la zona centrale del vetrino coprioggetti dove si troveranno i canali di flusso. - Preparare piccoli pezzi di vetro incidendo e spezzando un vetrino in pezzi di circa 2,4 cm x 1 cm.
- Praticare due fori a 1.4 mm di distanza utilizzando una punta diamantata da 0.8 mm, uno leggermente più largo dell'altro (0.043" = ingresso, 0.048" = uscita). Verificare che i fori siano della dimensione corretta provando a inserire un pezzo del tubo di ingresso/uscita, utilizzando la stessa punta da trapano per allargare il foro fino a quando il tubo non si adatta. Assicurarsi che il tubo sia sufficientemente aderente da non cadere facilmente.
NOTA: Il tubo di ingresso più stretto viene utilizzato per ridurre al minimo il volume morto. - Tagliare del nastro biadesivo della stessa forma dei pezzi di vetro.
- Allineare il vetrino sul nastro e infilzare un ago attraverso ogni foro per segnare le loro posizioni sul nastro. Usando una lama di rasoio, taglia un canale che includa entrambi i fori.
NOTA: Non tagliare il canale troppo a lungo, poiché lo spazio in eccesso sul lato opposto di ciascun foro può rimanere asciutto quando il liquido viene versato e può causare problemi con bolle d'aria durante il flusso. Durante il taglio, provare a tagliare i bordi lunghi in un unico taglio netto, poiché i bordi sfilacciati da più tagli possono influire sulla qualità del canale di flusso. Allo stesso modo, non tagliare i bordi del canale di flusso, poiché i tagli nel pezzo utilizzabile possono influenzare il flusso di liquido o addirittura perdere nella cella di flusso assemblata. - Pulire il pezzo di vetro con acetone e fazzoletto fino a quando non si asciuga, quindi posizionarlo su una superficie pulita. Staccare un lato del nastro adesivo e incollarlo sul pezzo di vetro in modo che entrambi i fori siano completamente all'interno del canale.
- Sigillare il nastro adesivo sul pezzo di vetro utilizzando una punta per pipetta p1000 per eliminare le bolle premendo la superficie con una pressione decisa ma moderata. Passare su tutta la superficie per sigillare.
- Stacca il secondo bordo del nastro da entrambi i pezzi di vetro e posizionali su una superficie pulita con il lato adesivo rivolto verso l'alto. Posizionare i vetrini uno vicino all'altro ma senza toccarli nella posizione in cui saranno attaccati al vetrino coprioggetti (lasciare abbastanza spazio tra di loro affinché la resina epossidica crei una tenuta completa attorno a ciascun pezzo di vetro).
- Prelevare il vetrino coprioggetti mezzo biotina-PEG utilizzando una pinzetta di plastica, tenendolo solo per il bordo (non toccare l'area che formerà il canale di flusso), e abbassare il lato funzionalizzato PEG sull'adesivo. Fissare premendo con un dito per fissarlo in posizione, quindi completare la sigillatura strofinando la superficie con moderatamente decisione con la punta di una pipetta per rimuovere le bolle d'aria (non sull'area del canale di flusso in quanto potrebbe rompersi), quindi capovolgere.
NOTA: Maneggiare il vetrino coprioggetti con cura. - Per ogni cella di flusso, tagliare ~10 cm di ogni tipo di tubo in polietilene (PE20 e PE60). Utilizzare il tubo più stretto come ingresso alla camera di flusso per ridurre il volume morto.
- Inserire manualmente il tubo nei fori della slitta. Se il diametro dei fori è corretto, assicurarsi che il tubo si trovi da solo nel foro una volta inserito. Tagliare la punta del tubo con una leggera angolazione di <45° per facilitare leggermente l'inserimento del tubo ed evitare che il tubo formi una tenuta contro il vetrino coprioggetti, cosa che potrebbe fare se l'estremità è piatta (questo potrebbe ostruire il flusso).
NOTA: L'accurato montaggio del tubo nel pezzo di vetro rende la costruzione della cella di flusso più affidabile e aiuta a evitare l'introduzione di bolle nella cella di flusso. - Mescola bene i componenti epossidici, quindi usa una punta p200 per tamponare la resina epossidica per sigillare il tubo e creare una tenuta attorno a ciascun pezzo di vetro. Aggiungi abbastanza resina epossidica in modo che arrivi fino al bordo del coprioggetti rotondo, in quanto ciò rafforzerà anche il delicato vetrino coprioggetti. Assicurarsi, tuttavia, che la resina epossidica non penetri sul lato inferiore del vetrino coprioggetti, in quanto potrebbe impedirne l'appoggio sul palco.
NOTA: Se necessario, piccole quantità di resina epossidica in eccesso possono essere raschiate via dalla parte inferiore utilizzando una lama di rasoio. - Lasciare agire per almeno 30-60 minuti affinché la resina epossidica si indurisca completamente.
NOTA: Se la cella di flusso non deve essere utilizzata immediatamente, conservarla sotto vuoto a RT dopo la completa polimerizzazione. - Dopo aver utilizzato la cella a flusso, recuperare i pezzi di vetro e riutilizzarli a tempo indeterminato. Estrarre il tubo e posizionare la cella di flusso in un barattolo contenente acetone per almeno 24 ore per ammorbidire la resina epossidica e il biadesivo, consentendo una facile rimozione dei pezzi di vetro. Prima di riutilizzarli per creare un'altra cella a flusso, pulire i pezzi di vetro strofinandoli con una spugna e acqua saponata, asciugarli e strofinarli con acetone e carta velina.

Figura 3: Rappresentazione grafica della preparazione della cella di flusso. (A) Tagliare il nastro biadesivo in modo che corrisponda alle dimensioni del pezzo di vetro. Allineare la diapositiva sulla parte superiore del nastro e segnare la posizione di ciascun foro con un ago. Usando una lama di rasoio, taglia intorno a ogni presa per creare un canale. (B) Staccare un lato del nastro e incollare il nastro sul pezzo di vetro. Assicurarsi che entrambi i fori siano all'interno del canale. Staccare la seconda estremità del nastro e incollare sopra il vetrino coprioggetti in biotina-PEG. (C) Inserire il tubo in polietilene in ciascun foro e sigillare il tubo in posizione con resina epossidica, sigillando anche ogni pezzo di vetro al vetrino coprioggetti. (D) Dopo aver utilizzato entrambi i canali, estrarre il tubo e posizionare la cella di flusso in un barattolo di colorazione riempito di acetone. Dopo circa 24 ore, la resina epossidica e il nastro si saranno ammorbiditi e gli strati della cella di flusso potranno essere staccati. I pezzi di vetro possono essere recuperati e conservati in acetone per essere riutilizzati all'infinito per realizzare la successiva cella di flusso. Clicca qui per visualizzare una versione più grande di questa figura.
4. Saggio TIRF a singola molecola per visualizzare lo svolgimento del DNA mediato da CMG
- Preparare i seguenti buffer.
- Preparare il tampone bloccante (20 mM Tris, pH 8,0; 50 mM NaCl; 2 mM EDTA; 0,2 mg/mL BSA). Filtrare il tampone con un filtro per siringa da 0,22 μm e conservarlo a 4 °C.
- Preparare 10 tamponi di reazione 1 (250 mM HEPES pH 7,5; 50 mM NaCl; 100 mM di acetato di magnesio). Filtrare il tampone con un filtro per siringa da 0,22 μm e conservarlo a 4 °C.
- Preparare 10 tamponi di reazione 2 (250 mM Tris, pH 7,5; 100 mM di acetato di magnesio; 1,25 M di glutammato di potassio; 1 mM EDTA; 0,025% Tween-20). Filtrare il tampone con un filtro per siringa da 0,22 μm e conservarlo a 4 °C.
- Degassare circa 2,5 mL di tampone bloccante e 5 mL di acqua ultrapura mettendo le provette aperte in un essiccatore e lasciandole sotto vuoto per 15 minuti.
NOTA: Non riempire i tubi fino all'orlo, poiché le bolle durante il degasaggio possono causare schizzi di liquido. - Posizionare la cella di flusso su un tavolino da microscopio e fissarla in posizione utilizzando uno stucco adesivo a ciascuna estremità. È importante fissarlo bene per evitare che la cella a flusso si muova più tardi durante l'acquisizione.
- Collegare il tubo di uscita della cella a flusso alla pompa a siringa, che ha un tubo collegato alla siringa con un ago all'altra estremità. Inserire questo ago nel tubo di uscita per collegare la cella di flusso.
- Accendere il riscaldatore dell'obiettivo a 30 °C.
- Fissare 1-2 mL di acqua degassata in una provetta in un pezzo separato di stucco adesivo vicino alla cella di flusso in modo che il tubo di ingresso possa essere inserito e raggiungere il fondo della provetta.
- Far scorrere l'acqua attraverso il canale, quindi utilizzare un flusso più veloce per rimuovere eventuali bolle intrappolate vicino al tubo di ingresso, aggiungendo altra acqua se necessario. In caso contrario, le bolle di grandi dimensioni potrebbero staccarsi più avanti negli esperimenti e passare attraverso il canale. Lasciare agire per alcuni minuti affinché il flusso si fermi completamente, poiché il flusso veloce può causare l'estrazione residua del liquido per un po' di tempo dopo l'arresto, poiché la pressione all'interno della siringa si stabilizza.
NOTA: Qualsiasi DNA che entra in contatto con una bolla diventa inutilizzabile, anche una volta reidratato. - Aggiungere 100 μL di tampone bloccante degassato a un'aliquota di 20 μL di 1 mg/mL di streptavidina. Attacca il tubo aperto allo stucco adesivo e trasferisci il tubo di ingresso dall'acqua alla streptavidina. Flusso a una velocità di 40 μl/min per 2 min (80 μl in totale) e incubazione per 5 min.
- Lavare via la streptavidina in eccesso con il tampone bloccante (50 μL/min per 100 μL).
- Flusso in DNA biotinilato, diluito in tampone bloccante con 25 nM di SYTOX orange. Immagine utilizzando la visualizzazione dal vivo con il laser a 532 nm per osservare il DNA che si lega alla superficie in tempo reale.
- Quando si raggiunge la densità approssimativa del DNA sulla superficie, far scorrere il tampone bloccante, anche con 25 nM di SYTOX (50 μL/min per 100 μL), per lavare via il DNA libero.
- Flusso in anticorpo biotinilato anti-digossigenina (~10 μg/mL) diluito in tampone bloccante contenente 25 nM di arancio SYTOX (100 μL/min per 300 μL).
NOTA: L'arancia SYTOX si intercala tra le coppie di basi del DNA, estendendo la lunghezza del contorno. Ciò significa che la lunghezza end-to-end del DNA aumenta in arancione SYTOX, consentendo al secondo cavo di attaccarsi alla superficie più lontano dal primo, fornendo un DNA meglio allungato quando l'arancio SYTOX viene lavato via in seguito. L'uso di una parte diversa su ciascuna estremità del DNA consente un ancoraggio più uniforme del DNA alla superficie del vetrino coprioggetti (Figura 4). Quando entrambe le estremità sono biotinilate, si legano nello stesso passaggio, con conseguente variazione significativa nelle loro posizioni di attacco. L'uso della digossigenina a un'estremità offre un maggiore controllo sulla copertura del DNA prima di legare la seconda estremità. Ciò consente anche di aumentare la copertura del DNA mediante incubazione. - Lavare via l'anticorpo biotinilato anti-digossigenina e SYTOX orange con tampone bloccante (50 μL/min per 100 μL).
- Preparare 120 μl di "miscela ATP-g-s" (1x tampone di reazione 1, 0,75 mg/mL BSA, 1,25 mg/mL caseina, 8 mM DTT, 0,33 mM ATP-g-s), aggiungere 30 μl a una nuova provetta, quindi far fluire 50 μl dalla provetta originale nella cella di flusso (50 μl/min per 50 μl). Questo aiuta a ridurre al minimo l'effetto della miscelazione del tampone attraverso il riflusso o la diffusione dal tubo di uscita.
- Aggiungere CMG purificato a ~100 nM finale in 30 μL di miscela ATP-g-s, quindi far fluire a 20 μL/min per 20 μL. Incubare per 15 min.
- Preparare 120 μl di "miscela ATP/RPA" (1x tampone di reazione 1, 0,75 mg/mL BSA, 8 mM DTT, 3,3 mM ATP, 20 nM EGFP-hRPA) e fluire nella cella a flusso a 40 μl/min per 80 μl.
- Inizia immediatamente ad acquisire le immagini. Acquisisci 6 x 6 campi visivi per ogni fotogramma ogni 30 s (o un frame rate appropriato per l'esperimento). Frame rate più bassi possono ridurre l'esposizione al laser e il fotosbiancamento del fluoroforo. Visualizza EGFP-hRPA con un laser a 488 nm all'1% di potenza. Se CMG è etichettato, ad esempio, LD655, visualizzare CMG con laser a 640 nm (potenza massima = 30 mW) al 10% di potenza. Visualizza il DNA colorato di arancione SYTOX con un laser a 532 nm allo 0,5% di potenza. Utilizzare ciascun laser con un'esposizione di 50-100 ms.
NOTA: Ogni volta che il tubo di ingresso della cella di flusso viene trasferito da un tubo di liquido all'altro, tamponare più volte la punta del tubo sul fondo del tubo prima di rimuoverlo dal liquido. Questo aiuta a prevenire l'ingresso di bolle durante il trasferimento. Ridurre al minimo il tempo in cui la punta del tubo di ingresso viene tenuta fuori dal liquido e non puntare la punta verso l'alto o sollevare la punta in modo significativo, poiché ciò provoca il drenaggio del liquido verso il canale e l'aria nell'aspirazione. Durante il trasferimento del tubo di ingresso, cercare di non mettere in tensione il tubo in quanto ciò potrebbe estrarlo dalla resina epossidica, fissandolo alla cella di flusso. Per l'imaging dello svolgimento del DNA, in presenza di SYTOX orange per visualizzare il DNA a doppio filamento, sostituire 10x il tampone di reazione 1 con 10x il tampone di reazione 2 per la miscela ATP/RPA. L'arancione SYTOX si lega meglio al DNA in queste condizioni. Svuotare completamente la pompa a siringa al termine di ogni esperimento. Lavare di tanto in tanto riempiendo con acqua e svuotando più volte o smontando e lavando con acqua e sapone.

Figura 4: Legame del DNA alla superficie. (A) Quando si legano substrati di DNA con biotina ad entrambe le estremità, la distanza tra i due lacci può variare a seconda di come le estremità entrano in contatto con la superficie (i). Utilizzando la digossigenina a un'estremità, il legame di ciascuna estremità può essere separato temporalmente per distanze di legame più coerenti e DNA allungato in modo più uniforme (ii). (B) Esempio di campo visivo che mostra il DNA legato da entrambe le estremità (marcato con digossigenina) e colorato con una colorazione fluorescente di acido nucleico intercalante. Il DNA, che è legato da entrambe le estremità, appare come una linea, mentre il DNA legato da una sola estremità appare come macchie. Idealmente, il DNA dovrebbe essere legato il più densamente possibile senza sovrapporsi ad altro DNA. L'immagine è di 512 x 512 pixel (dimensione pixel = 154,6 nm). Clicca qui per visualizzare una versione più grande di questa figura.
Risultati
Quando il CMG svolge il DNA, un caratteristico tratto di RPA crescerà nel tempo (Figura 5). L'estremità 5' del DNA srotolato è legata alla superficie; quindi, è visto come un tratto lineare del segnale RPA tra il cavo e la forcella. L'estremità 3' non è legata e, quindi, si muove con la forcella e viene osservata come un segnale EGFP-RPA compatto. La posizione del filamento di traslocazione compattato non svolto corrisponde approssimativamente alla posizione della forcella di replicazione, che si muove insieme a LD655-CMG visualizzata tramite un laser a 640 nm.
È importante ridurre al minimo i danni al substrato di DNA, poiché danni come le tacche di DNA a singolo filamento riducono il numero di eventi di svolgimento osservabili, limitando la quantità di dati che possono essere raccolti (Figura 6).
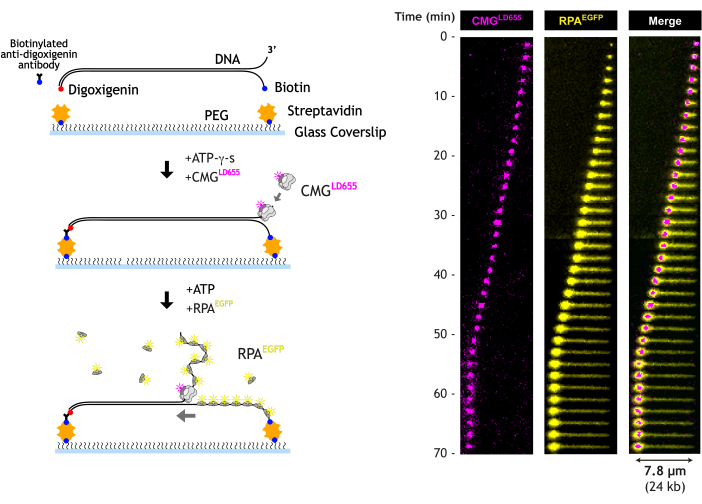
Figura 5: Saggio di svolgimento del DNA a singola molecola. Il substrato di DNA è legato a una superficie del vetrino coprioggetti. Il CMG purificato marcato con LD655 viene incubato con il DNA per 15 minuti in ATP-g-s. Vengono aggiunti ATP e RPA purificato marcato con EGFP, avviando un'ampia svolgimento del DNA da parte del CMG. Vengono mostrati uno schema a fumetti (a sinistra) e un chimografo di dati rappresentativi (a destra). Clicca qui per visualizzare una versione più grande di questa figura.
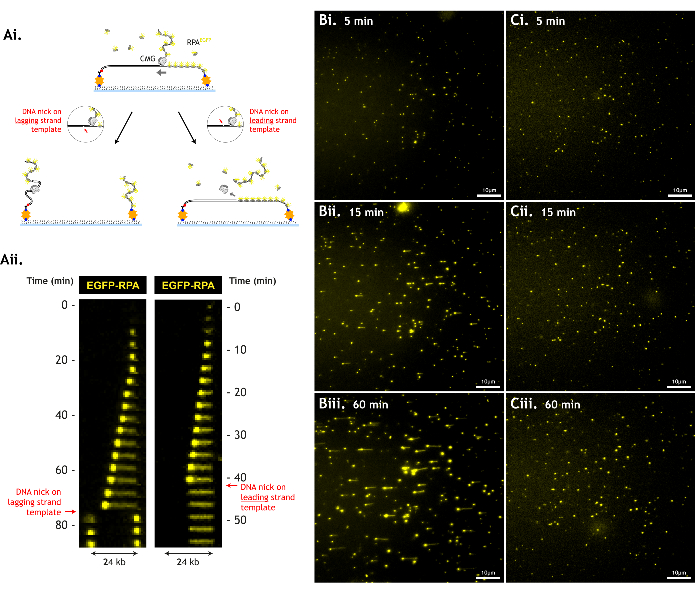
Figura 6: Il danno al DNA riduce la produttività del saggio. (A) Il CMG non è in grado di svolgere il DNA oltre una rottura nella spina dorsale del DNA (intaccatura del DNA). Un'intaccatura sul modello del filamento principale provoca lo scivolamento del CMG dal DNA e sia il CMG che il modello del filamento principale vengono persi. Un'intaccatura sul modello del filamento in ritardo fa sì che il modello del filamento in ritardo si separi dal resto del DNA e ogni pezzo di DNA si ritrae nel rispettivo legame. Ciò è illustrato da (i) schemi di cartoni animati e (ii) chimografie di questi eventi (ii). Dati rappresentativi con (B) un substrato di DNA minimamente danneggiato rispetto a (C) un substrato di DNA più danneggiato a (i) 5 minuti, (ii) 15 minuti e (iii) 60 minuti in un singolo campo visivo. Il substrato di DNA più danneggiato non genera lunghi tratti di svolgimento, poiché i CMG incontrano intaccature prima, nonostante livelli simili di attività di svolgimento (densità simile di punti RPA in crescita a 5 minuti, indicando un'efficienza di carico/svolgimento CMG simile). Il campo visivo è di 512 x 512 pixel (dimensione dei pixel = 154,6 nm). 1% di potenza laser (488 nm) imaging EGFP-RPA. Barra della scala che mostra 10 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
Questo test fornisce una piattaforma per osservare e studiare le dinamiche in tempo reale dei singoli CMG, sia in isolamento che nel contesto dei fattori aggiuntivi desiderati. Tuttavia, come per molte tecniche di fluorescenza a singola molecola, ci sono alcune sfide comuni che possono richiedere un'ottimizzazione per essere superate. Questi di solito si riferiscono all'imaging di fluorofori per lunghi periodi di tempo (fotosbiancamento, luminosità), alla preparazione del substrato di DNA (danno al DNA), alla qualità della superficie della cella di flusso (rumore di fondo, interazioni non specifiche) o alla qualità della preparazione proteica purificata (contaminazione da nucleasi, efficienza di marcatura).
Ogni fluoroforo varia in fotostabilità e luminosità, quindi è importante scegliere una molecola appropriata. Quando si esegue l'imaging di proteine oligomeriche marcate in fluorescenza, come l'RPA, è possibile utilizzare una potenza laser inferiore poiché molti fluorofori verranno eccitati nelle immediate vicinanze, generando un segnale visibile. Per l'imaging di singoli fluorofori, ad esempio CMG marcati su una singola subunità, è necessaria una maggiore potenza laser per osservare chiaramente il fluoroforo. La durata del fluoroforo può essere prolungata riducendo al minimo l'esposizione al laser, ad esempio riducendo la frequenza con cui vengono scattate le immagini. Inoltre, l'eccitazione di un fluoroforo genera specie reattive dell'ossigeno (ROS), che possono contribuire al fotosbiancamento. L'inclusione di un sistema di evacuazione dell'ossigeno nel tampone di imaging può prolungare la durata dei fluorofori eliminando i ROS. Tuttavia, alcuni sistemi di evacuazione dell'ossigeno possono influenzare il pH12.
Per quanto riguarda la preparazione del substrato di DNA, è fondamentale ridurre al minimo i danni al DNA, come intaccature o spazi vuoti a singolo filamento. Un danno eccessivo impedisce lo svolgimento esteso del DNA, limitando la quantità di dati che possono essere raccolti. I danni possono derivare da taglio meccanico, riscaldamento eccessivo, a causa della contaminazione da nucleasi o ROS generati durante l'imaging. Il taglio può essere ridotto al minimo maneggiando il campione di DNA con cura, utilizzando punte a foro largo per il pipettaggio, il pipettaggio lento ed evitando di scuotere il campione. L'effetto dei ROS può essere minimizzato riducendo l'esposizione al laser o includendo un sistema di evacuazione dell'ossigeno nel tampone di imaging. Dopo la preparazione del substrato di DNA, è possibile utilizzare kit di riparazione del DNA commerciali per riparare il danno prima di eseguire una reazione di svolgimento.
L'efficienza dello svolgimento del DNA dipende anche dalla purezza e dall'attività del CMG. È buona norma valutare la purezza del campione dopo ogni fase di purificazione mediante elettroforesi SDS-PAGE per determinare dove è necessaria l'ottimizzazione. Se si osservano troppi contaminanti dopo la fase finale, può essere utile modificare i volumi del gradiente salino utilizzati per l'eluizione dalla colonna CaptoHiRes Q (5/50). È anche molto importante rimuovere l'eccesso di peptide fluorescente utilizzato per la marcatura delle proteine, in quanto può creare uno sfondo indesiderato sulla superficie del vetrino. È inoltre essenziale evitare la contaminazione da nucleasi, in quanto ciò può degradare il substrato del DNA. Dopo un esperimento, colorare il DNA rimanente con l'arancione SYTOX può essere un buon modo per verificare se il DNA è stato degradato in modo significativo o meno. Un certo livello di danno al DNA è inevitabile nel corso di un esperimento, ma un danno significativo spesso indica una contaminazione problematica della nucleasi.
Il test è anche intrinsecamente limitato dalla risoluzione dei punti limitati dalla diffrazione, che richiede che le proteine fluorescenti siano distanti centinaia di paia di basi (se non di più) per distinguerle come separate. Ciò limita il dettaglio in cui è possibile osservare la progressione e le interazioni del CMG.
Il numero di eventi di svolgimento osservati per ogni analisi varia. Per un esperimento di successo, ci aspettiamo di vedere almeno diversi tratti RPA di lunghezza sufficiente per campo visivo 512x512 pixel (dimensione dei pixel = 154,6 nm). È possibile visualizzare più campi visivi nello stesso esperimento, consentendo una maggiore raccolta di dati quando necessario. I tratti non devono essere della stessa lunghezza né raggiungere la fine del DNA per essere utili. Ad esempio, la distanza media del cavo per ogni esperimento può essere determinata misurando la lunghezza del DNA colorato con SYTOX prima di aggiungere CMG. Questo può essere utilizzato per stimare la quantità di DNA che è stata srotolata per qualsiasi tratto di RPA (a condizione che sia srotolato abbastanza DNA da muovere visibilmente la forcella) convertendo la distanza da "μm percorso" a "kb svolto".
Il CMG mostra attività di svolgimento su una varietà di substrati di DNA, ma è essenziale fornire un'estremità libera di DNA 3' su un lembo di polyT di almeno 30 nt per accogliere l'impronta di CMG10. L'inclusione di più frazioni di biotina sulla forcella garantisce un robusto ancoraggio superficiale. Il resto del substrato di DNA può essere riprogettato in una moltitudine di modi, ad esempio per includere diverse sequenze di DNA, lunghezze e modifiche chimiche. La conformazione del DNA può essere alterata utilizzando diverse concentrazioni di acetato di magnesio. A concentrazioni più elevate (≥10 mM) di acetato di magnesio, il filamento di ssDNA rivestito di RPA viene compattato, portando al DNA estratto dal legame RPA durante lo svolgimento. Questo può essere utile in quanto impedisce al DNA di muoversi eccessivamente, consentendo di misurare in modo più accurato la posizione del CMG e la progressione di svolgimento. A basse concentrazioni (~3 mM) di acetato di magnesio, l'RPA-ssDNA rimane rilassato per tutto il tempo.
Il saggio a singola molecola descritto rappresenta una piattaforma che può essere costruita e modificata per studiare ulteriori aspetti della replicazione del DNA. Durante la replicazione del DNA, il CMG agisce come un nucleo attorno al quale si assemblano il replisoma e i suoi componenti. Pertanto, è possibile aggiungere ulteriori proteine purificate a questo test, inclusi fattori accessori come TIMELESS, TIPIN e CLASPIN, per studiare il loro effetto sulla dinamica del CMG. È stato dimostrato che queste proteine influenzano la velocità di replicazione delle forcelle13, ma non è chiaro come influenzino la velocità di svolgimento della CMG. Pertanto, sarebbe interessante studiare come le diverse proteine replisomiche influenzano il CMG utilizzando questo test. L'aggiunta di DNA polimerasi può fornire una migliore comprensione della replicazione del DNA oltre al solo svolgimento del DNA, come descritto in precedenza con le proteine del lievito14. Inoltre, la purificazione del CMG modificato può fornire una migliore comprensione di come alcune mutazioni o modificazioni post-traduzionali influenzano l'attività dell'elicasi15,16. Inoltre, la progettazione di diversi substrati di DNA può consentire di studiare lo svolgimento del DNA mediante CMG in una varietà di condizioni che imitano lo stress di replicazione17. Queste modifiche includono ostacoli al DNA 9,18, legami incrociati tra filamenti 19,20,2 1 e discontinuità nei filamenti di DNA22.
Divulgazioni
Gli autori non hanno interessi finanziari concorrenti o altri conflitti di interesse.
Riconoscimenti
Ringraziamo Gheorghe Chistol per aver fornito il plasmide pGC261 e la Francis Crick Institute Chemical Biology Facility per la sintesi e l'etichettatura dei peptidi. Questo lavoro è stato finanziato dal Francis Crick Institute, che riceve finanziamenti di base dal Cancer Research UK, dal Medical Research Council del Regno Unito e dal Wellcome Trust (CC2133).
Materiali
| Name | Company | Catalog Number | Comments |
| 2-Mercaptoethanol | Sigma-Aldrich | M3148-25ML | |
| Acrylamide / Bis Solution 40% | BioRad | 1610148 | |
| Agarose UltraPure | Invitrogen | 16500-500 | Used to prepare Tris Buffer (pH 8) |
| ÄKTA Pure | GE Healthcare | - | Used with Fraction Collector F9-C; protein purification system |
| ANTI-FLAG M2 affinity gel | Sigma-Aldrich | A2220 | |
| APS 10% | Sigma-Aldrich | A3678-25G | |
| ATP (200 mM) | Sigma-Aldrich | A7699-5G | |
| ATP-g-s (100 mM) | Sigma-Aldrich | 11162306001 | |
| Biotin-PEG coverslip | N/A | N/A | Prepared as described: https://doi.org/10.1016/j.ymeth.2012.03.033 |
| Biotinylated anti-digoxigenin antibody | Perkin Elmer | N/A | |
| Blu Tack | Bostik | N/A | Adhesive putty |
| Boric acid | Thermo Fisher Scientific | B/3800/53 | |
| BSA; 33 mg/mL | SIGMA-ALDRICH | A3858-10G | Diluted from the stock |
| CaCl2 | Sigma-Aldrich | C7902 | |
| CaptoHiRes Q (5/50) | Cytiva | 29275878 | Connected to the AKTA protein purification system; high-resolution ion exchange chromatography column |
| Casein (5% in water, 50 mg/mL) | Sigma-Aldrich | C4765-10ML | |
| ChemiDoc system | Bio-Rad | N/A | |
| Clips | N/A | N/A | |
| Cutting board | N/A | N/A | |
| Dialysis tubing (3.5 kDa) | Fisher Scientific | 11425859 | |
| digoxigenin-11-dUTP | Roche | 11209256910 | |
| DNA loading dye 6x | NEB | B7024S | |
| dNTP 10 mM | NEB | N0447S | |
| Double-sided tape (0.14 mm thick) | N/A | N/A | |
| DTT | Fluorochem Limited | M02712-10G | |
| DYKDDDDK peptide | N/A | N/A | Synthetised by chemical biology STP of The Francis Crick Institute |
| EDTA | Thermo Fisher Scientific | D/0700/68 | |
| EGFP-RPA | N/A | N/A | Purified as desribed in ‘Single Molecule Analysis’, Series Methods in Molecular Biology, Vol. 783 (2011), Peterman, E. J. G. and Wuite G. J. L. editors, Humana Press. Protein information: 2 mM, human, expressed in E. coli. |
| EGTA | Sigma-Aldrich | 03779-10G | |
| Epoxy - 5 minute | Devcon | 20845 | |
| Fujifilm FLA-5000 | Fujifilm | FLA-5000 | Fluorescent image analyzer |
| GelRed Nucleic Acid Stain; 10000x | Cambridge Bioscience | 41003-BT | Example of nucleic acid stain which is a safer alternative to ethidium bromide. |
| Glass or quartz slide with holes for tubing | Dremel Model 395 | 1 mm thick slide, cut to 2.4 cm x 1cm, two holes drilled 1.4 mm apart. The holes should be drilled to different sizes to accommodate different tubing at each end. | |
| Glycerol | Fisher Scientific | G/0650/17 | |
| Glycine HCl, pH 3.5 | Sigma-Aldrich | G8898 | |
| Hamilton syringe, 1000 series GASTIGHT, PTFE luer lock 1010TLL, PTFE Luer lock (with slots), volume 10 mL | Merck | 26211-U | |
| HEPES pH 7.5 | Sigma-Aldrich | H4034 | |
| I-CeuI | NEB | R0699L | |
| KCl | Fisher Scientific | P/4280/53 | |
| Magnesium acetate | Sigma-Aldrich | M2545 | |
| Maxi GeBaFlex-tube Dialysis Kit; MWCO 14 kDa | Generon | D055 | |
| Metal spatula | N/A | N/A | |
| Metal tweezers | N/A | N/A | |
| MgCl2 | Sigma-Aldrich | M2393 | |
| Millex-GP (Syringe filters) 0.22 µm | Merck | SLGP033RS | |
| NaCl | Sigma-Aldrich | S7653 | |
| Needle | N/A | N/A | |
| NotI-HF | NEB | R3189L | |
| NuPAGE 4%–12% Bis-Tris Protein Gels | Thermo Fisher Scientific | 10247002; | |
| NuPAGE MOPS SDS Running Buffer | Thermo Fisher Scientific | NP0001 | |
| Objective heater | Okolab | N/A | |
| PCR mix - 2x | Prepared from Phusion DNA polymerase (20 µL), 10 mM dNTPs (40 µL), 5x Phusion HF buffer (400 µL) and water (540 µL). | ||
| Peptide NH2-CHHHHHHHHHHLPETGG-COOH, labelled with LD655-MAL | N/A | N/A | peptide NH2-CHHHHHHHHHHLPETGG-COOH, labeled with LD655-MAL (Lumidyne Technologies) on the cysteine residue, was synthetised and purified by the peptide chemistry STP of The Francis Crick Institute |
| pGC261 plasmid | N/A | N/A | https://doi.org/10.1016/j.cell.2018.10.053 |
| Phusion High-Fidelity DNA Polymerase | NEB | M0530L | |
| Plastic tweezers | N/A | N/A | |
| Polyethylene tubing PE20 (inner diameter 0.015”, outer diameter 0.043”) | Becton Dickinson | 427406 | |
| Polyethylene tubing PE60 (inner diameter 0.03”, outer diameter 0.048”) | Becton Dickinson | 427416 | |
| Poly-Prep Chromatography Columns; 10 ml and 20 ml | Bio-Rad | 7311550 | |
| Potassium Glutamate (L-glutamic acid potassium salt monohydrate) | Sigma | G1501-1KG | |
| Protease inhibitor cocktail (cOmplete, EDTA free) | Roche | 5056489001 | |
| Pump 11 Elite Infusion/Withdrawal Programmable Single Syringe | Harvard Apparatus | 70-4504 | |
| QIAquick PCR Purification Kit | QIAGEN | 28104 | |
| Razor blade | VWR International Ltd | 233-0156 | |
| rCutSmart Buffer; 10x | NEB | B6004S | |
| Sodium acetate | Thermo Fisher Scientific | S/2120/53 | |
| Sortase (pentamutant) | N/A | N/A | Purified based on: https://doi.org/10.1073/pnas.1101046108 |
| Spin-X Centrifuge Tube Filters; 0.22 µm cellulose acetate | Fisher Scientific | 10310361 | |
| Sterile scalpel | N/A | N/A | |
| Steritop Vacuum Driven Disposable Filtration System; 0.22 um; PES; | Millipore | S2GPT05RE | |
| Streptavidin (1 mg/mL) in 1x PBS buffer | Sigma | S4762-10MG | 20 µL aliquotes |
| SYBR Gold | Thermo Fisher Scientific | S11494 | Highly sensitive nucleic acid stain we used to visualise DNA fork substrate. |
| SYTOX orange; 5 µM in DMSO | Thermo Fisher Scientific | S11368 | |
| T4 DNA ligase | NEB | M0202M | |
| T4 DNA Ligase Reaction Buffer | NEB | B0202S | |
| TEMED | Sigma-Aldrich | T9281-25ML | |
| TEV protease (1 mg/mL); EZCut | Biovision | 7847-10000 | |
| Tissue grinders, Dounce type (40 mL, Wheaton) | DWK Life Sciences | 432-1273 | |
| Tris base | Sigma-Aldrich | T1503 | Used to prepare Tris Buffer (pH 8) |
| Tris HCl | Sigma-Aldrich | T3253 | |
| Tween-20 | Promega UK Ltd | H5152 |
Riferimenti
- Bell, S. P., Labib, K. Chromosome Duplication in Saccharomyces cerevisiae. Genetics. 203 (3), 1027-1067 (2016).
- Costa, A., et al. The structural basis for MCM2-7 helicase activation by GINS and Cdc45. Nat Struct Mol Biol. 18 (4), 471-477 (2011).
- Jones, M. L., Baris, Y., Taylor, M. R. G., Yeeles, J. T. P. Structure of a human replisome shows the organisation and interactions of a DNA replication machine. EMBO J. 40 (23), e108819 (2021).
- Moyer, S. E., Lewis, P. W., Botchan, M. R. Isolation of the Cdc45/Mcm2-7/GINS (CMG) complex, a candidate for the eukaryotic DNA replication fork helicase. Proc Natl Acad Sci U S A. 103 (27), 10236-10241 (2006).
- Douglas, M. E., Ali, F. A., Costa, A., Diffley, J. F. X. The mechanism of eukaryotic CMG helicase activation. Nature. 555 (7695), 265-268 (2018).
- Ilves, I., Petojevic, T., Pesavento, J. J., Botchan, M. R. Activation of the MCM2-7 helicase by association with Cdc45 and GINS proteins. Mol Cell. 37 (2), 247-258 (2010).
- Dewar, J. M., Walter, J. C. Mechanisms of DNA replication termination. Nat Rev Mol Cell Biol. 18 (8), 507-516 (2017).
- Burnham, D. R., Kose, H. B., Hoyle, R. B., Yardimci, H. The mechanism of DNA unwinding by the eukaryotic replicative helicase. Nat Commun. 10 (1), 2159 (2019).
- Kose, H. B., Larsen, N. B., Duxin, J. P., Yardimci, H. Dynamics of the eukaryotic replicative helicase at lagging-strand protein barriers support the steric exclusion model. Cell Rep. 26 (8), 2113-2125.e6 (2019).
- Kose, H. B., Xie, S., Cameron, G., Strycharska, M. S., Yardimci, H. Duplex DNA engagement and RPA oppositely regulate the DNA-unwinding rate of CMG helicase. Nat Commun. 11 (1), 3713 (2020).
- Tanner, N. A., Van Oijen, A. M. Visualizing DNA replication at the single-molecule level. Methods Enzymol. 475, 259-278 (2010).
- Swoboda, M., et al. Enzymatic oxygen scavenging for photostability without ph drop in single-molecule experiments. ACS Nano. 6 (7), 6364-6369 (2012).
- Baris, Y., Taylor, M. R. G., Aria, V., Yeeles, J. T. P. Fast and efficient DNA replication with purified human proteins. Nature. 606, 204-210 (2022).
- Lewis, J. S., et al. Tunability of DNA polymerase stability during eukaryotic DNA replication. Mol Cell. 77 (1), 17-25.e5 (2020).
- Li, Z., Xu, X. Post-translational modifications of the mini-chromosome maintenance proteins in DNA replication. Genes. 10 (5), 331 (2019).
- Lewis, J. S., et al. Mechanism of replication origin melting nucleated by CMG helicase assembly. Nature. 606 (7916), 1007-1014 (2022).
- Zeman, M. K., Cimprich, K. A. Causes and consequences of replication stress. Nat Cell Biol. 16 (1), 2-9 (2014).
- Sparks, J. L., et al. The CMG helicase bypasses DNA-protein cross-links to facilitate their repair. Cell. 176 (1-2), 167-181.e21 (2019).
- Taylor, M. R. G., Yeeles, J. T. P. The initial response of a eukaryotic replisome to DNA damage. Mol Cell. 70 (6), 1067-1080.e12 (2018).
- Amunugama, R., et al. Replication fork reversal during DNA interstrand crosslink repair requires CMG unloading. Cell Rep. 23 (12), 3419-3428 (2018).
- Wu, R. A., et al. TRAIP is a master regulator of DNA interstrand crosslink repair. Nature. 567 (7747), 267-272 (2019).
- Vrtis, K. B., Dewar, J. M., Chistol, G., Wu, R. A., Graham, T. G. W., Walter, J. C. Single-strand DNA breaks cause replisome disassembly. Mol Cell. 81 (6), 1309-1318.e6 (2021).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon