Method Article
CMG Helicase를 이용한 DNA 풀림의 단일 분자 실시간 시각화
요약
이 프로토콜은 CMG helicase에 의한 DNA 풀림의 실시간 시각화를 위한 단일 분자 분석을 수행하는 방법을 보여줍니다. (1) DNA 기질 준비, (2) 형광 표지된 Drosophila melanogaster CMG helicase를 정제하는 방법, (3) 전반사 형광(TIRF) 현미경 검사를 위한 미세유체 플로우 셀을 준비하는 방법, (4) 단일 분자 DNA 풀기 분석에 대해 설명합니다.
초록
충실한 게놈 복제는 분열하는 세포의 유전적 안정성을 보존하는 데 필수적입니다. DNA 복제는 레플리솜(replisome)이라고 하는 단백질의 동적 복합체에 의해 S 단계에서 수행됩니다. 레플리솜의 중심에는 CDC45-MCM2-7-GINS(CMG) 헬리카제가 있으며, 이는 DNA 이중 나선의 두 가닥을 분리하여 DNA 중합효소가 각 가닥을 복제할 수 있도록 합니다. 게놈 복제 과정에서 replisomes는 수많은 장애물과 도전을 극복해야 합니다. 이들 각각은 DNA를 완전하고 정확하게 복제하지 못하면 돌연변이, 질병 또는 세포 사멸로 이어질 수 있기 때문에 게놈 안정성을 위협합니다. 따라서 CMG가 일반 복제와 복제 스트레스 동안 replisome에서 어떻게 작동하는지 이해하는 것이 매우 중요합니다. 여기에서는 개별 CMG 복합체에 의한 표면 테더링 스트레치 DNA 분자를 실시간으로 시각화할 수 있는 재조합 정제 단백질을 사용한 전반사 형광(TIRF) 현미경 분석에 대해 설명합니다. 이 분석은 단일 분자 수준에서 CMG 거동을 조사할 수 있는 강력한 플랫폼을 제공하여 반응 조건에 대한 실시간 제어를 통해 나선형 역학을 직접 관찰할 수 있습니다.
서문
DNA 복제는 세포가 돌연변이, 질병 또는 죽음을 방지하기 위해 게놈을 정확하게 복제해야 하기 때문에 엄격하게 조절됩니다. 진핵생물 DNA 복제는 부모의 DNA를 풀고 단일 가닥 DNA(ssDNA)를 새로운 DNA를 합성하기 위한 템플릿으로 사용하는 replisome complex에 의해 수행됩니다. G1 단계에서 MCM2-7의 촉매 비활성 이중 헥사머는 복제 기원1에서 이중 가닥 DNA(dsDNA)에 로드됩니다. S 단계에서 MCM2-7 복합체는 CDC45 및 GINS2 의 결합에 의해 활성화되어 11-서브유닛 CMG 복합체(CDC45, MCM2-7, GINS)를 형성합니다. 각 CMG는 반대 방향으로 DNA 풀기를 시작하여 replisome이3 주위에 배열하는 핵심 단위를 형성합니다.
20년 전 CMG 헬리카제는 DNA 복제에 필수적인 11-서브유닛 복합체로 처음 확인되었다4. 그 이후로 CMG에 대한 우리의 이해는 로딩 및 활성화 5,6에서 DNA 풀기 및 종료7에 이르기까지 상당히 발전했습니다. 전통적인 생화학 및 구조 생물학 기술은 이러한 발견 중 많은 부분에 중요했습니다. 그러나 이러한 방법은 CMG의 보다 동적인 측면을 연구하는 능력이 제한되는 경우가 많았습니다. 단일 분자 방법은 개별 생체 분자의 물리적 조작을 사용하여 한 번에 한 분자씩 생체 분자의 활동을 측정하거나 시각화합니다. 이는 다른 기술에 의해 종종 누락되거나 검출할 수 없는 단백질의 실시간 역학에 대한 통찰력을 제공하는 데 사용할 수 있습니다 8,9.
여기에서는 CMG helicase에 의한 DNA 풀림을 실시간으로 시각화하기 위한 전반사 형광(TIRF) 현미경 분석에 대해 설명합니다. 정제되고 형광으로 표지된 CMG는 사전 만들어진 DNA 포크 구조를 포함하는 긴 DNA의 유리 3' 말단에 로드됩니다. 선형 DNA는 DNA의 각 말단을 표면에 순차적으로 연결하여 미세유체 플로우 셀의 비오틴-PEG 커버슬립에 늘어납니다. 이 접근 방식을 사용하면 보다 균일한 DNA 테더링이 가능하여 데이터 분석 중에 고려해야 하는 변동을 크게 줄일 수 있습니다. ATP-γ-s가 있는 경우 CMG는 포크의 3' 말단에 있는 단일 가닥 DNA에 로드됩니다. ATP-γ-s는 천천히 가수분해가 가능한 ATP 아날로그로, CMG가 DNA에 결합할 수 있지만 풀릴 수는 없습니다. 이후에 정제된 형광 태깅 RPA와 함께 ATP를 추가하면 CMG가 활성화되고 광범위한 DNA 풀기가 시작됩니다. 시각적으로 CMG는 DNA를 따라 전좌되어 RPA 결합 ssDNA의 증가 영역을 남깁니다. 테더링되지 않은 DNA 말단은 CMG와 함께 이동하여 RPA 바인딩으로 인한 압축으로 인해 "타이트 볼"을 형성합니다. 플로우 셀 설계는 언와인딩 중 언제든지 버퍼를 교체할 수 있도록 하여 각 실험 중 및 각 실험에 대한 뛰어난 제어를 제공합니다.
이 프로토콜은 서로 독립적으로 수행할 수 있는 네 가지 방법으로 나뉩니다. 섹션 1에서는 단일 분자 분석을 위한 20kb 선형 분기형 DNA 기질의 준비에 대해 설명합니다. 섹션 2는 Drosophila melanogaster CMG(DmCMG)의 정제 및 형광 라벨링에 대해 간략하게 설명합니다. DmCMG 표현에 대한 주요 정보는 참고 섹션에 포함되어 있습니다. 섹션 3에서는 TIRF 현미경에 사용할 수 있는 미세유체 플로우 셀의 준비에 대해 다룹니다. 섹션 4에서는 단일 분자 DNA 풀기 분석을 수행하는 방법을 설명합니다.
프로토콜
1. 단일 분자 분석에 사용되는 20kb 선형 분기형 DNA의 준비(그림 1)
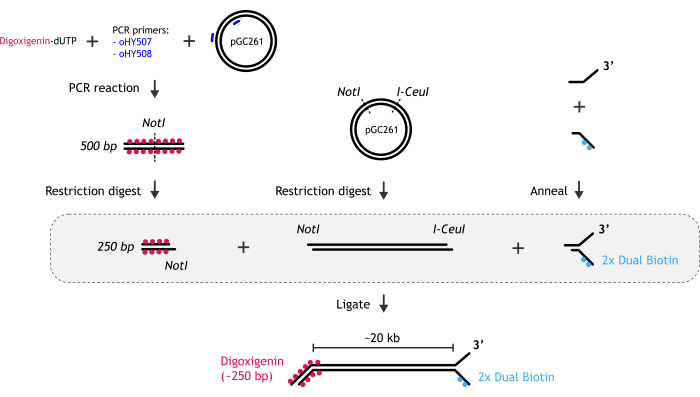
그림 1: DNA 기질 준비의 그래픽 표현. (A) 비오틴화된 DNA 포크 말단은 부분적으로 상호 보완적인 두 개의 올리고뉴클레오티드(비오틴화 및 비오틴화 올리고뉴클레오티드)를 어닐링하여 생성됩니다. (B) 주요 dsDNA 단편(~20kb)은 두 개의 효소가 있는 pGC261 플라스미드의 제한 분해에 의해 생성되어 각 말단에 다른 돌출부를 가진 선형 DNA를 생성합니다. (C) 디곡시제닌 듀플렉스 DNA 말단은 디곡시제닌-dUTP의 존재 하에 수행된 PCR 반응에 의해 얻어진 후 제한 분해에 의해 얻어진다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
- 비오틴화된 DNA 포크 말단의 생성.
- 포크 DNA를 어닐링합니다.
- 두 개의 올리고뉴클레오티드(oHY502 및 oHYbio85)를 1x STE 완충액(100mM NaCl, 10mM Tris, pH 8.0, 1mM 에틸렌디아민테트라아세트산[EDTA])에 혼합합니다. 최종 올리고뉴클레오티드 농도는 10μM이고 최종 반응 부피는 100μL입니다.
- 80°C에서 히트 블록에서 5분 동안 반응을 배양합니다. 히트 블록을 끄고 반응을 서서히 냉각시켜 온도가 30°C 이하로 떨어질 때까지 히트 블록에 그대로 둡니다.
- Tris-Borate-EDTA(TBE)-Polyacrylamide 겔 전기영동(PAGE) 겔에서 어닐링 효율을 검증합니다.
- 8% 아크릴아마이드 - TBE 겔(4개 겔용: H2O32.8mL, 10x TBE 완충액 4.8mL(1M Tris, 1M 붕산, 20mM EDTA), 40% 아크릴아미드/Bis 용액 9.6mL, 37.5:1, 10% 과황산암모늄[APS] 0.8mL, 테트라메틸에틸렌디아민[TEMED] 40μL).
주의: TBE, 아크릴아마이드, APS, TEMED는 독성/유해합니다. - 6배 로딩 염료로 희석한 개별 올리고뉴클레오티드와 함께 소량(1μL)의 어닐링된 올리고뉴클레오티드를 80V에서 80V에서 1시간 동안 실행합니다.
- 적절한 핵산 염색제가 첨가된 1x TBE에 겔을 시소 로킹 셰이커에서 30분 동안 담근 다음 겔 이미징 시스템을 사용하여 겔을 스캔합니다.
- 8% 아크릴아마이드 - TBE 겔(4개 겔용: H2O32.8mL, 10x TBE 완충액 4.8mL(1M Tris, 1M 붕산, 20mM EDTA), 40% 아크릴아미드/Bis 용액 9.6mL, 37.5:1, 10% 과황산암모늄[APS] 0.8mL, 테트라메틸에틸렌디아민[TEMED] 40μL).
- 겔 절제를 위해 TBE-PAGE 겔에서 어닐링된 DNA를 분해합니다.
- 올리고뉴클레오티드가 제대로 어닐링된 경우 BSA가 있는 상태에서 샘플의 나머지 부분을 분리합니다(어닐링된 샘플 ~100μL, 6x 로딩 염료 25μL, 33mg/mL BSA 2μL). 겔에 더 많은 양을 로드하려면 여러 웰을 멸균 메스와 결합하십시오.
- 적절한 핵산 염색이 보충된 1x TBE에 겔을 넣고 시소 로킹 셰이커에서 30분 동안 그대로 둡니다.
- 겔을 유리 트레이에 옮기고 청색광 아래의 어두운 방에서 DNA를 시각화합니다. 블루라이트를 사용할 때는 적절한 보호 스크린이나 보호 안경을 사용하십시오.
- 멸균 메스로 원하는 밴드를 잘라냅니다. 여분의 젤을 잘라냅니다.
- 전기 용리로 어닐링된 포크를 정제합니다.
- 젤 조각을 삽입할 수 있을 만큼 충분히 길게 투석 튜브 조각을 자르고 1x TBE에 적신 다음 한쪽 끝을 닫습니다.
- 500 μL의 1x TBE에서 BSA를 0.3 mg/mL로 희석하고 전체 부피를 튜브에 피펫팅합니다.
- 금속 주걱을 사용하여 젤 조각을 안으로 밀어 넣습니다. 겔 조각을 튜브의 한쪽으로 이동하여 전기 용출 중에 DNA가 이동할 수 있는 완충 공간을 제공합니다.
- 여분의 버퍼를 짜내고 튜브의 두 번째 끝을 자릅니다.
알림: 남은 부피에 따라 최종 부피와 농도가 결정됩니다. - 투석 튜브를 1x TBE 버퍼의 아가로스 전기영동 탱크에 넣고 튜브가 완전히 잠기도록 합니다.
알림: DNA가 겔에서 투석 백으로 이동할 수 있도록 겔 조각이 음극에 가장 가까운 쪽에 배치되었는지 확인하십시오. - 80-1시간 동안 2V에서 전기 용리를 실행합니다.
- 전기용출된 DNA를 10mM Tris buffer(pH 8)로 투석합니다.
- 전기 용출 후 탱크에서 투석 백을 꺼내 한쪽 끝을 티슈로 건조시킵니다. 겔과 샘플이 있는 티슈로 튜브의 중간 부분을 만지지 마십시오.
- 건조된 끝에서 클립을 제거하고 샘플을 튜브 내부에서 위아래로 부드럽게 파이프하여 DNA를 완충액에 혼합합니다.
- 깨끗한 주걱을 1x TBE 버퍼에 담그고 튜브에서 젤 조각을 퍼냅니다.
알림: 젤 조각을 제거하는 동안 가능한 한 적은 버퍼를 제거하십시오. - 투석 백의 열린 끝을 다시 끼워 내부의 공기를 제거합니다.
- 투석 백을 1.5L의 10mM Tris buffer(pH 8), 20mM NaCl 및 2mM MgCl2로 채워진 2L 비커에 넣습니다. 클립을 파라필름으로 감싸 튜브를 비커 가장자리에 붙이고 튜브를 버퍼에 완전히 담그십시오.
알림: 소금은 짧은 올리고 듀플렉스를 안정화합니다. - 마그네틱 바를 사용하여 완충액을 부드럽게 저어주고 샘플을 실온(RT)에서 최소 3-4시간 동안 또는 밤새 4°C에서 투석합니다.
- 투석 후에는 투석 백의 한쪽 끝을 티슈로 말리고(DNA를 희석하지 않기 위해) 이 끝 부분을 풉니다.
- 튜브 내부에서 샘플을 위아래로 부드럽게 피펫팅하고 깨끗한 1.5mL 튜브에 옮깁니다.
- 마이크로볼륨 분광 광도계를 사용하여 DNA의 농도를 측정합니다.
- 포크 DNA를 어닐링합니다.
- 20kb 단편 생성
- NotI/I-CeuI 효소를 사용하여 pGC261 plasmid18 의 restriction digest를 수행합니다.
- 제한효소 생산자가 권장하는 완충액에 8 μL의 NotI-HF(20,000 U/mL), 8 μL의 I-CeuI(5,000 U/mL) 및 pGC261 플라스미드(200 μL 반응에서 최종 플라스미드 농도 ~40 ng/μL)를 부드럽게 혼합합니다.
- 37°C에서 하룻밤 동안 반응을 배양합니다.
- 겔 절제를 위해 아가로스 겔에서 분해된 DNA를 분해합니다.
- 선택 사항: 전체 반응을 로드하기 전에 먼저 0.6% 아가로스 겔에 소량의 반응을 실행하여 분해 효율을 테스트합니다.
- 0.6단계 1.2.2.3-1.2.2.4에 따라 1% 아가로스 겔을 준비합니다.
- 아가로스 분말 0.48g을 1x TBE 완충액 80mL와 혼합합니다. 끓을 때까지 용액을 전자레인지에 돌립니다. 아가로스가 완전히 녹을 때까지 휘젓습니다. 몇 분 동안 식힌 다음 지정된 탱크에 붓습니다.
- 더 많은 양의 샘플을 수용하기 위해 웰을 결합하려면 미리 빗의 여러 웰에 테이프 조각을 붙입니다.
- 겔이 응고되면 빗을 부드럽게 제거하고 탱크에 1x TBE 버퍼를 채웁니다. 샘플을 DNA 로딩 염료와 부드럽게 혼합하고 겔을 120V에서 1시간 동안 실행합니다.
- 염색될 때까지 적절한 핵산 염색이 보충된 1x TBE에 겔을 시소 로킹 셰이커에 그대로 둡니다.
- 염색된 겔을 유리 트레이에 옮기고 청색광 아래의 어두운 방에서 DNA를 시각화합니다.
- 멸균 메스로 원하는 밴드를 잘라냅니다. 여분의 젤을 잘라냅니다.
- 선택 사항: 1.1.4 및 1.1.5 단계에 설명된 대로 전기 용리로 원하는 단편을 정제합니다. 여기서 투석 완충액의 소금은 생략하는데, 이는 짧은 oligo duplex를 안정화하는 데만 필요하기 때문입니다.
- NotI/I-CeuI 효소를 사용하여 pGC261 plasmid18 의 restriction digest를 수행합니다.
- digoxigenin duplex DNA 말단의 생성
- pGC261 DNA를 템플릿으로 사용하여 digoxigenin-dUTP(dig-dUTP)로 PCR 반응을 수행합니다.
- 혼합물: 물 400 μL, pGC261 8 μL(0.8 ng/μL), 3.5 μL oHY507 프라이머(100 μM), 3.5 μL 프라이머 oHY508 (100 μM), 8 μL dig-dUTP (1 mM), 400 μL의 2x PCR 믹스(DNA 중합효소(20 μL), 10 mM dNTPs(40 μL), 5x 고충실도(HF) 완충액(400 μL) 및 물(540 μL)로 제조.
- 열 순환기에서 다음 반응을 실행합니다.
98 °C - 1 분
30x: 98°C - 20초; 65 °C - 20 초; 72 °C - 30 초;
72 °C - 10 분
4 °C - 보류
- digoxigenin-labeled PCR 산물의 분해 및 정제
- 상용 DNA 정제 키트를 사용하여 PCR 산물을 정제합니다.
- 효소 공급업체에서 권장하는 완충액에 10μL NotI-HF(20,000U/mL)를 PCR 산물과 혼합합니다(~200μL 반응에서 최종 DNA 농도는 ~50ng/μL). 37°C에서 하룻밤 동안 반응을 배양합니다.
- 상용 DNA 정제 키트를 사용하여 분해된 DNA를 정제합니다.
- pGC261 DNA를 템플릿으로 사용하여 digoxigenin-dUTP(dig-dUTP)로 PCR 반응을 수행합니다.
- DNA 기질을 만들기 위해 구성 요소를 조립합니다.
- 200 μL 반응에서 효소 공급업체가 권장하는 완충액에 비오틴화된 포크 말단, 20-kb DNA 단편 및 디곡시제닌 표지된 Not-I 처리된 PCR 단편을 5 μL의 T4 DNA 리가아제(400,000 U/mL)와 부드럽게 혼합합니다. 그러나, 첨가된 20-kb DNA 단편(1-5 μg)에 기초하여, 비오틴화된 포크 말단과 PCR 단편 모두의 대략 100배 몰 초과분을 첨가한다.
- 반응을 PCR 튜브(각 50μL)에 분취하고 16°C에서 열 순환기에서 밤새 배양합니다.
- 0.6% 아가로스 겔에서 결찰된 샘플을 분리하고 1.2.2 및 1.2.3 단계에 설명된 대로 전기 용출로 결찰된 DNA를 정제합니다.
- 급속 동결 DNA는 -80°C에서 보관합니다.
2. Drosophila melanogaster CMG의 정제 (그림 2)

그림 2: Hi Five 세포 4L에서 Drosophila melanogaster CMG의 정제. 단백질은 MOPS 완충액의 존재 하에 200V 미만의 4%-12% Bis-Tris 폴리아크릴아미드 겔에서 분해되었습니다. 샘플은 정제의 각 단계(세포 용해물 - 2 μL, FLAG 용리 - 10 μL, 첫 번째 이온 교환 컬럼 후 - 10 μL, 라벨링 후 및 두 번째 이온 교환 컬럼 - 1 μL)에서 표시됩니다. (A) Coomassie 염색은 형광 라벨링 전(10 μL) 및 후(1 μL) CMG 복합체의 11개 소단위체의 존재를 모두 확인합니다. (B) MCM3 서브유닛의 라벨링 효율성은 LPR(Long Pass Red) 필터를 사용하여 형광 이미지 분석기로 Cy5를 스캔하여 검증되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
참고: 형광 표지된 Drosophila melanogaster CMG를 제조하기 위해 TEV 절단 부위(ENLYFQG)와 4개의 Gly 잔기를 MCM3 서브유닛(pFastBac1 벡터)의 N-말단 FLAG 태그 다운스트림에 도입했습니다(pFastBac1 벡터)10. 복합체를 발현하기 위해 바큘로바이러스 발현 시스템을 사용하였다. 초기 transfection을 위해 Sf21 세포를 각 CMG subunit(P1 바이러스 단계)에 대해 별도로 사용했습니다. 바이러스를 더욱 증폭시키기 위해 Sf9 세포를 사용했습니다(P2 바이러스 단계). 이어서, Sf9 세포 배양(각 CMG 소단위당 100mL, 0.5 x 106 cells/mL)을 10% 태아 송아지 혈청(P3 바이러스 단계)이 보충된 0.5mL P2 바이러스로 감염시켰다. 전체 CMG 복합체를 4L의 Hi Five 세포(1 x 106 cells/mL)로 발현하기 위해 각 소단위체에 대해 200mL의 P3 바이러스를 사용했습니다. CMG 복합체를 발현하는 Hi Five 세포를 수확한 후 세포 펠릿을 액체 질소에 급속 냉동하고 -80°C에서 보관할 수 있습니다. 얼음 또는 4 °C에서 전체 정제를 수행하십시오. 완충액은 환원제(DTT 또는 2-메르캅토에탄올) 및 프로테아제 억제제(주의)를 사용 직전에 첨가하는 조건으로 미리 준비할 수 있습니다. 모든 버퍼가 미리 사전 냉각되고 여과 및 가스가 제거되었는지 확인하십시오.
- 다음 버퍼를 준비합니다.
- 25 mM 4-(2-하이드록시에틸)-1-피페라진에탄술폰산(HEPES) pH 7.5, 0.02% 트윈-20, 10% 글리세롤, 15 mM KCl, 2 mMMgCl2, 2 mM 2-메르캅토에탄올, 1 mM EDTA, 및 1 mM 에틸렌 글리콜-비스(β-아미노에틸 에테르)-N,N,N′,N′-테트라아세트산(EGTA)을 혼합하여 재현탁액 A를 제조한다.
주의: 2-메르캅토에탄올, DTT 및 EDTA는 독성/유해합니다. - ANTI-FLAG M2 친화성 겔을 제조하기 위해 트리스 완충 식염수(TBS; 0.1M Glycine HCl, pH 3.5) 완충액을 준비합니다.
- 25 mM HEPES pH 7.5, 0.02% 트윈-20, 10% 글리세롤, 100 mM KCl, 1 mM DTT, 1 mM EDTA 및 1 mM EGTA를 혼합하여 완충액 A-100을 제조합니다.
- 25mM HEPES pH 7.5, 50mM 아세트산나트륨, 10mM 마그네슘 아세테이트, 10% 글리세롤 및 1mM DTT를 첨가하여 투석 완충액을 준비합니다.
- 50mM Tris-HCl pH 7.5 및 150mM NaCl을 혼합하여 TBS 완충액을 제조합니다.
- 25 mM 4-(2-하이드록시에틸)-1-피페라진에탄술폰산(HEPES) pH 7.5, 0.02% 트윈-20, 10% 글리세롤, 15 mM KCl, 2 mMMgCl2, 2 mM 2-메르캅토에탄올, 1 mM EDTA, 및 1 mM 에틸렌 글리콜-비스(β-아미노에틸 에테르)-N,N,N′,N′-테트라아세트산(EGTA)을 혼합하여 재현탁액 A를 제조한다.
- 프로테아제 억제제 칵테일 정제를 보충한 200mL의 Cold Resuspension Buffer A 내 Hi Five 세포의 4L 배양에서 얻은 재현탁 펠릿. 전체 펠릿이 완전히 재현탁될 때까지 튜브를 반전으로 혼합합니다.
- 재현탁 세포를 사전 냉각된 40mL Dounce 균질화기로 옮깁니다. 가능한 한 많은 세포를 회수하려면 세포 펠릿 동결에 사용되는 튜브를 동일한 유형의 완충액으로 세척하고 Dounce 균질화기에 첨가하십시오. 균질화기의 총 부피가 권장 한계를 초과하지 않는지 확인하십시오. 얼음 위의 세포를 60-70 스트로크로 용해시킵니다.
알림: 플런저가 튜브 바닥에 닿았는지 확인하되 균질기가 파손될 수 있으므로 너무 많은 압력을 가하지 마십시오. 플런저를 액체 높이 위로 빼면 기포가 생길 수 있으므로 주의하십시오. - 전체 샘플이 균질화될 때까지 이전 단계를 반복하고 예냉된 비커에 결합합니다. 샘플의 최종 부피를 평가합니다. 한 방울 한 방울 여과된 KCl을 최종 농도 100mM에 넣고 부드럽게 섞습니다.
- 원심분리기를 미리 4°C로 사전 냉각합니다. 샘플을 원심분리 튜브에 붓고 생산자가 권장하는 양에 도달했는지 확인합니다. 저울에서 튜브의 균형을 맞춥니다.
알림: 부피가 너무 작은 튜브는 원심분리 중에 파손될 수 있습니다. - 용해된 세포를 23,500 x g 에서 4°C에서 15-30분 동안 원심분리합니다. 그 동안 다음 단계에 따라 ANTI-FLAG M2 친화 젤을 준비하십시오.
- ANTI-FLAG M2 친화성 젤(현탁액 50%)이 들어있는 병을 부드럽게 섞습니다. P-100 피펫 팁의 끝을 자르고(팁을 넓히기 위해) 이를 사용하여 4mL의 현탁액(2mL의 비드)을 20mL 폴리-프렙 크로마토그래피 컬럼에 즉시 전사합니다. 팁에 붙어있는 비드를 복구하려면 TBS로 팁을 세척하고 컬럼에 추가하십시오. 모든 구슬이 기둥 내부에 가라앉도록 하되 건조시키지 않도록 주의하십시오.
- 0.1M Glycine HCl, pH 3.5의 3x 비드 부피(총 6mL)로 수지를 세척합니다. 그 후, 3-5 비드 부피의 TBS (6-10 mL)로 비드를 세척합니다. 다음으로, Buffer A-100으로 비드를 3회(6mL) 세척합니다.
알림: 비드를 0.1M Glycine HCl pH 3.5 용액에 20분 이상 두지 마십시오. 흐름 압력과 세척 속도를 높이려면 컬럼을 뚜껑으로 덮으십시오. 수지가 어느 시점에서도 건조되지 않도록 하십시오. - 레진을 준비한 후 레진 위에 Buffer A-100 2mL를 두고 컬럼의 배출구를 닫습니다.
- 원심분리 후 상층액을 사전 냉각된 50mL 튜브에 부드럽게 붓습니다. 펠릿을 방해하지 않도록 주의하십시오. 나중에 SDS-PAGE 전기 영동으로 검증하기 위해 작은 부분 표본(~10μL)을 사용합니다.
- 동일한 양의 ANTI-FLAG M2 비드(이전 단계에 따라 준비)를 각 50mL 튜브에 추가합니다. Buffer A-100 몇 밀리리터를 추가로 현탁시키고 이 재현탁액을 튜브로 옮겨 컬럼에서 모든 비드를 회수하십시오.
- 샘플(상층액)을 ANTI-FLAG M2 수지로 4°C에서 2.5시간 동안 회전시켜 배양합니다.
- 배양 후 샘플을 1,000 x g 에서 4°C에서 5분 동안 회전시킵니다. 피펫을 사용하여 대부분의 상층액을 제거합니다. 절단된 팁을 사용하여 비드를 남아 있는 상층액 몇 밀리리터에 재현탁하고 두 개의 15mL 튜브에 옮깁니다. 50-mL 튜브의 벽에 붙어있는 비드를 회수하려면 Buffer A-100 몇 밀리리터를 추가하고 이를 15-mL 튜브에도 옮깁니다.
- 15mL 튜브를 1,000 x g 에서 4°C에서 5분 동안 회전합니다. 상층액을 제거합니다.
- 각 튜브에 ~14mL의 Buffer A-100을 첨가하여 비드를 세척한 다음 4°C에서 짧고 부드럽게 회전한 다음 4°C에서 5분 동안 1000 x g 으로 회전시킵니다. 세탁을 두 번 반복하십시오. 각 세척의 소량(~10μL)을 취하여 나중에 SDS-PAGE 전기영동으로 검증합니다.
- 이 단계(2.16)는 이전 단계(2.13-2.15)의 대체 절차입니다.
- ANTI-FLAG M2 수지로 배양한 후 샘플을 2개의 20mL 폴리 분취 크로마토그래피 컬럼에 붓습니다.
- 이 방법을 선택하는 경우 비드가 컬럼 바닥에 가라앉도록 하고 결합되지 않은 샘플이 중력 흐름에 의해 컬럼을 통과하도록 합니다. 그런 다음 Buffer A-100을 컬럼에 직접 추가하고 중력 흐름을 통해 샘플을 세척하도록 합니다.
- 비드(Buffer A-100에 재현탁)를 2개의 10mL poly-prep 크로마토그래피 컬럼으로 옮깁니다.
- Buffer A-100이 레진을 통과하도록 하고, 버퍼 레벨이 레진 상단에 도달하면 배출구를 닫고, 200μg/mL(DYKDDDDK) 펩타이드(FLAG 용출 버퍼)가 보충된 Buffer A-100을 첨가하여 비드에서 단백질을 용리합니다.
- 첫 번째 용출의 경우 FLAG 용출 완충액 3mL를 추가하고 닫힌 컬럼을 RT에서 15분 동안 부드럽게 회전시킵니다. 배출구를 열어 첫 번째 용리 분획을 수집합니다.
- 배출구를 닫고 각 컬럼에 FLAG 용출 완충액 2mL를 추가합니다. 10분 더 회전하여 두 번째 용리 분획을 수집합니다.
- 모든 용리된 분획을 함께 결합하고(~10mL) 4°C에서 보관합니다. 나중에 SDS-PAGE 전기영동으로 검증하기 위해 용리된 분획(~10μL)의 작은 부분 표본을 취하십시오.
- 0.22μm 주사기 필터를 사용하여 용리된 샘플을 필터링합니다.
- 제조업체의 프로토콜에 따라 단백질 정제 시스템에 연결된 CaptoHiRes Q(5/50) 컬럼(고분해능 이온 교환 크로마토그래피 컬럼)을 미리 준비합니다. 그 후, Buffer A-100으로 컬럼을 평형화하고 여과된 샘플을 평형화된 컬럼에 로드합니다.
- Buffer A-100의 20 컬럼 부피(CV)로 컬럼을 세척합니다(컬럼 부피가 ~1mL이므로 총 ~20mL).
- 단백질을 용리하려면 Buffer A-100 및 Buffer B의 두 가지 완충액을 미리 준비합니다. Buffer A-100 및 Buffer B는 Buffer A-100과 조성이 동일하지만 100 mM KCl 대신 550 mM KCl을 사용합니다. 염 농도를 100 mM에서 550 mM KCl로 증가시켜 20 CV(~20 mL)에 대한 그래디언트 용리를 설정합니다. 분획 분취기에서 0.3-0.5 mL 용리액을 수집합니다.
참고: CMG는 버퍼 B의 약 70%-75% 농도에서 용리되어야 합니다. SDS-PAGE 전기영동을 위해 세척 및 용출된 분획의 소량 분취액을 취하십시오. - SDS-PAGE 전기영동을 수행하여 선택한 분획에 CMG가 존재하는지 확인합니다. 그 동안 제조업체의 프로토콜에 따라 컬럼과 정제 시스템을 청소하십시오.
- 샘플 1mL당 50μL의 TEV 프로테아제(1mg/mL)를 혼합하여 선택한 분획을 TEV 프로테아제로 밤새 배양합니다.
- 투석 튜브를 미리 적시고 멤브레인이 손상되지 않았는지 확인합니다. CMG/TEV 혼합물을 투석 튜브에 넣고 1.5 L의 사전 냉각된 Buffer A-100 과 내부의 마그네틱 바가 있는 2 L 비커에 넣습니다. 4°C에서 하룻밤 동안 부드럽게 저어주어 투석합니다.
- 5 mM CaCl2 가 있는 상태에서 샘플을 50 μM의 LD655 표지 펩타이드 및 10 μg/mL Sortase 효소와 혼합합니다. 4°C에서 30분 동안 회전하여 튜브가 빛으로부터 덮여 있는지 확인합니다. SDS-PAGE 전기영동을 위한 라벨링 전후에 소량의 부분 표본을 채취하십시오.
- 펩타이드가 침전될 수 있으므로 정제 시스템에 로드하기 전에 0.22μm 원심분리기 필터를 사용하여 라벨링된 샘플을 여과하십시오.
- 앞에서 설명한 대로 고분해능 이온 교환 크로마토그래피 컬럼을 미리 준비합니다(단계 2.20). 이번에는 컬럼과 분획 분취기 시스템을 알루미늄 호일로 덮어 샘플을 빛으로부터 보호합니다.
- 유리 펩타이드를 제거하려면 여과된 샘플을 고분해능 이온 교환 크로마토그래피 컬럼에 로드합니다. 앞에서 설명한 것과 동일한 방법으로 정제를 수행합니다(2.21-2.22단계). 레이블이 지정된 CMG가 이전과 유사한 Buffer-B 농도로 용리되는지 확인합니다.
- SDS-PAGE 전기영동을 수행하여 선택한 분획의 품질을 검증합니다. 형광을 이미지화하려면 로딩하기 전에 샘플을 끓이지 말고 전기영동 탱크가 빛으로부터 보호되는지 확인하십시오. 먼저 겔 이미징 시스템을 사용하여 단백질 형광을 시각화한 다음 Coomassie 염료로 겔을 염색하고 다시 이미징하여 모든 단백질을 시각화합니다.
- 선택한 분획을 4°C에서 1.5L의 투석 완충액에 대해 밤새 투석합니다. 필요한 경우 샘플을 농축합니다.
- 단백질을 액체 질소에 급속 냉동하고 나중에 사용할 때까지 -80 °C에서 보관하십시오.
3. 플로우 셀(flow cell) 준비(그림 3)
- 오븐 베이킹 단계를 생략하고 이전에 설명한 프로토콜11에 따라 비오틴-PEG 커버슬립을 준비합니다. 비오틴-PEG 커버슬립은 RT의 진공 상태에서 최소 1개월 동안 안정적입니다. 플로우 셀을 준비하기 위해 비오틴-PEG 커버슬립(24mm x 60mm)을 반(약 24mm x 30mm)으로 자릅니다.
알림: 흐름 채널이 있을 커버슬립의 중앙 영역을 만지지 않도록 주의하십시오. - 유리 슬라이드를 약 2.4cm x 1cm 조각으로 에칭하고 스냅하여 작은 유리 조각을 준비합니다.
- 하나는 다른 하나보다 약간 넓은 0.8mm 다이아몬드 코팅 드릴 비트를 사용하여 1.4mm 간격으로 두 개의 구멍을 뚫습니다(0.043" = 입구, 0.048" = 출구). 동일한 드릴 비트를 사용하여 튜브가 맞을 때까지 구멍을 넓히고 입구/출구 튜브의 일부를 삽입하여 구멍의 크기가 올바른지 테스트합니다. 튜브가 쉽게 떨어지지 않을 만큼 단단히 끼워졌는지 확인하십시오.
알림: 데드 볼륨을 최소화하기 위해 더 좁은 입구 튜브가 사용됩니다. - 유리 조각과 같은 모양으로 양면 테이프를 자릅니다.
- 슬라이드를 테이프에 맞추고 각 구멍을 통해 바늘을 찔러 테이프의 위치를 표시합니다. 면도날을 사용하여 두 구멍을 둘러싸는 채널을 자릅니다.
알림: 액체가 유입될 때 각 구멍의 반대쪽에 있는 과도한 공간이 건조한 상태로 유지될 수 있고 흐름 중 기포 문제가 발생할 수 있으므로 채널을 너무 오래 자르지 마십시오. 절단할 때 여러 절단으로 인해 닳은 가장자리가 흐름 채널의 품질에 영향을 줄 수 있으므로 긴 가장자리를 한 번에 깨끗하게 절단하십시오. 유사한 목적으로, 사용 가능한 부분을 절단하면 액체의 흐름에 영향을 미치거나 조립된 플로우 셀의 누출에 영향을 줄 수 있으므로 흐름 채널의 가장자리를 절단하지 마십시오. - 아세톤과 티슈로 유리 조각을 마를 때까지 청소한 다음 깨끗한 표면에 놓습니다. 접착 테이프의 한쪽 면을 벗겨내고 두 구멍이 채널 내부에 완전히 오도록 유리 조각에 붙입니다.
- p1000 피펫 팁을 사용하여 테이프를 유리 조각에 밀봉하여 단단하지만 적당한 압력으로 표면을 눌러 기포를 제거합니다. 밀봉하기 위해 전체 표면을 살펴보십시오.
- 두 유리 조각에서 테이프의 두 번째 가장자리를 떼어내고 끈적한 면이 위로 향하게 하여 깨끗한 표면에 놓습니다. 슬라이드를 서로 가깝게 놓되 커버슬립에 부착될 위치에 닿지 않도록 합니다(에폭시가 각 유리 조각 주위를 완전히 밀봉할 수 있도록 슬라이드 사이에 충분한 공간을 두십시오).
- 플라스틱 핀셋을 사용하여 가장자리만 잡고 (흐름 채널을 형성할 영역을 만지지 마십시오) PEG 기능성 면을 접착제 위로 내립니다. 손가락으로 눌러 제자리에 고정한 다음 피펫 팁으로 표면을 적당히 단단히 문질러 기포를 제거한 다음(파손될 수 있으므로 흐름 채널 영역이 아님) 뒤집어 밀봉을 완료합니다.
알림: 커버슬립을 주의해서 다루십시오. - 각 플로우 셀에 대해 각 유형의 폴리에틸렌 튜브(PE20 및 PE60)를 ~10cm 자릅니다. 더 좁은 튜브를 플로우 챔버의 입구로 사용하여 데드 볼륨을 줄입니다.
- 슬라이드의 구멍에 튜브를 손으로 삽입합니다. 구멍의 직경이 올바르면 튜브가 삽입된 후 자체적으로 구멍에 서 있는지 확인하십시오. 튜빙 팁을 약간 <45°로 절단하여 튜빙을 약간 쉽게 삽입하고 튜빙이 커버슬립에 대해 밀봉을 형성하는 것을 방지합니다. 이는 끝이 평평한 경우(흐름을 방해할 수 있음).
참고: 튜브를 유리 조각에 정확하게 장착하면 플로우 셀 구성이 더욱 안정적이며 플로우 셀에 기포가 유입되는 것을 방지할 수 있습니다. - 에폭시 구성 요소를 잘 섞은 다음 p200 팁을 사용하여 에폭시를 두드려 튜브를 밀봉하고 각 유리 조각 주위를 밀봉합니다. 커버슬립 라운드의 가장자리까지 올라갈 수 있도록 충분한 에폭시를 추가하면 섬세한 커버슬립도 강화됩니다. 그러나 커버슬립의 아래쪽에 에폭시가 묻지 않도록 하십시오.tage.
알림: 필요한 경우 면도날을 사용하여 밑면의 소량의 과도한 에폭시를 긁어낼 수 있습니다. - 에폭시가 완전히 경화될 때까지 최소 30-60분 동안 그대로 두십시오.
참고: 플로우 셀을 즉시 사용하지 않을 경우 완전히 경화된 후 RT에서 진공 상태로 보관하십시오. - 플로우 셀을 사용한 후에는 유리 조각을 회수하여 무기한 재사용하십시오. 튜브를 당겨 빼내고 플로우 셀을 아세톤이 들어 있는 슬라이드 용기에 24시간 이상 넣어 에폭시 및 양면 접착제를 부드럽게 하여 유리 조각을 쉽게 제거할 수 있도록 합니다. 다른 플로우 셀을 만들기 위해 재사용하기 전에 유리 조각을 스폰지와 비눗물로 문질러 닦고 건조시킨 다음 아세톤과 티슈 페이퍼로 문지릅니다.

그림 3: 플로우 셀 준비의 그래픽 표현. (A) 유리 조각의 크기에 맞게 양면 테이프를 자릅니다. 테이프 위에 슬라이드를 맞추고 바늘로 각 구멍의 위치를 표시합니다. 면도날을 사용하여 각 홀드 주위를 잘라 채널을 만듭니다. (B) 테이프의 한쪽 면을 벗겨내고 테이프를 유리 조각에 붙입니다. 두 구멍이 모두 채널 내부에 있는지 확인하십시오. 테이프의 두 번째 끝을 벗기고 그 위에 비오틴-PEG 커버슬립을 붙입니다. (C) 폴리에틸렌 튜브를 각 구멍에 삽입하고 튜브를 에폭시로 제자리에 밀봉하고 각 유리 조각도 커버슬립에 밀봉합니다. (D) 두 채널을 모두 사용한 후 튜브를 당겨 빼내고 플로우 셀을 아세톤으로 채워진 염색 용기에 넣습니다. 약 24시간이 지나면 에폭시와 테이프가 부드러워지고 플로우 셀의 층이 벗겨질 수 있습니다. 유리 조각은 회수하여 아세톤에 보관하여 다음 플로우 셀을 만드는 데 무기한 재사용할 수 있습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
4. CMG 매개 DNA 풀림을 시각화하기 위한 단일 분자 TIRF 분석
- 다음 버퍼를 준비합니다.
- 차단 완충액(20mM Tris, pH 8.0, 50mM NaCl, 2mM EDTA, 0.2mg/mL BSA)을 준비합니다. 0.22μm 주사기 필터를 사용하여 버퍼를 여과하고 4°C에서 보관합니다.
- 10x 반응 완충액 1(250mM HEPES pH 7.5, 50mM NaCl, 100mM 마그네슘 아세테이트)을 준비합니다. 0.22μm 주사기 필터를 사용하여 버퍼를 여과하고 4°C에서 보관합니다.
- 10x 반응 완충액 2 (250 mM Tris, pH 7.5; 100 mM 마그네슘 아세테이트; 1.25 M 글루타민산 칼륨; 1 mM EDTA; 0.025 % 트윈 -20)를 준비합니다. 0.22μm 주사기 필터를 사용하여 버퍼를 여과하고 4°C에서 보관합니다.
- 약 2.5mL의 차단 완충액과 5mL의 초순수를 탈가스화하여 열린 튜브를 데시케이터에 넣고 15분 동안 진공 상태로 둡니다.
알림: 가스 제거 중 기포로 인해 액체가 튀게 될 수 있으므로 튜브를 가장자리까지 채우지 마십시오. - 플로우 셀을 현미경 s에 놓습니다.tage 양쪽 끝에 접착 퍼티를 사용하여 제자리에 고정합니다. 나중에 획득 중에 플로우 셀이 움직이지 않도록 잘 고정하는 것이 중요합니다.
- 플로우 셀 출구 튜브를 주사기 펌프에 연결하고, 주사기 펌프에는 튜브가 다른 쪽 끝에 바늘이 있습니다. 이 바늘을 배출 튜브에 삽입하여 플로우 셀을 연결합니다.
- 대물 히터를 30°C로 켭니다.
- 튜브에 있는 1-2mL의 탈기된 물을 플로우 셀 근처의 별도의 접착 퍼티 조각에 고정하여 입구 튜브가 삽입되어 튜브 바닥에 닿을 수 있도록 합니다.
- 채널을 통해 물을 흘린 다음 더 빠른 흐름을 사용하여 입구 튜브 근처에 갇힌 기포를 제거하고 필요한 경우 더 많은 물을 추가합니다. 그렇지 않으면 실험 후반부에 큰 기포가 제거되어 채널을 통과할 수 있습니다. 주사기 내부의 압력이 안정화됨에 따라 빠른 흐름으로 인해 정지된 후 잠시 동안 액체가 잔류 당겨질 수 있으므로 흐름이 완전히 멈출 때까지 몇 분 동안 그대로 두십시오.
참고: 거품과 접촉하는 모든 DNA는 한 번 재수화되더라도 사용할 수 없게 됩니다. - 1mg/mL 스트렙타비딘 20μL 부분 표본에 100μL의 탈가스 차단 완충액을 추가합니다. 열린 튜브를 접착 퍼티에 부착하고 물에서 streptavidin으로 입구 튜브를 옮깁니다. 2분(총 80μL) 동안 40μL/분의 속도로 흐르고 5분 동안 배양합니다.
- 차단 완충액(100μL의 경우 50μL/분)으로 과도한 스트렙타비딘을 씻어냅니다.
- 25nM SYTOX 오렌지색으로 차단 완충액에 희석된 비오틴화된 DNA의 흐름. 532nm 레이저로 라이브 뷰를 사용하여 DNA가 표면에 붙어있는 것을 실시간으로 관찰하는 이미지.
- 표면에 있는 DNA의 대략적인 밀도에 도달하면 25nM SYTOX(100μL의 경우 50μL/min)를 사용하여 차단 완충액으로 흐름시켜 자유 DNA를 씻어냅니다.
- 25 nM SYTOX 오렌지(300 μL의 경우 100 μL/min)를 함유하는 차단 완충액에 희석된 비오틴화된 항-디곡시제닌 항체(~10 μg/mL)의 흐름.
참고: SYTOX 오렌지는 DNA의 염기쌍 사이를 삽입하여 윤곽 길이를 확장합니다. 이는 SYTOX 오렌지에서 DNA의 종단 간 길이가 증가하여 두 번째 테더가 첫 번째 테더에서 더 멀리 표면에 부착될 수 있도록 하여 나중에 SYTOX 오렌지가 씻겨 나갈 때 DNA가 더 잘 늘어난다는 것을 의미합니다. 각 DNA 말단에 다른 부분을 사용하면 DNA를 커버슬립 표면에 보다 균일하게 테더링할 수 있습니다(그림 4). 양쪽 끝이 비오틴화되면 동일한 단계에서 테더링되어 부착 위치에 상당한 변화가 발생합니다. 한쪽 말단에서 디곡시제닌을 사용하면 두 번째 말단을 테더링하기 전에 DNA 범위를 더 잘 제어할 수 있습니다. 이것은 또한 배양을 통해 DNA 적용 범위를 증가시킬 수 있습니다. - 차단 완충액(100μL의 경우 50μL/min)으로 비오틴화된 항-디곡시제닌 항체와 SYTOX 오렌지를 씻어냅니다.
- 120 μL의 'ATP-g-s mix'(1x 반응 완충액 1, 0.75 mg/mL BSA, 1.25 mg/mL 카제인, 8 mM DTT, 0.33 mM ATP-g-s)를 구성하고, 새 튜브에 30 μL를 추가한 다음 원래 튜브에서 50 μL를 플로우 셀로 흘립니다(50 μL의 경우 50 μL/min). 이는 출구 튜브로부터의 역류 또는 확산을 통한 완충액 혼합의 영향을 최소화하는 데 도움이 됩니다.
- 30 μL의 ATP-g-s 혼합물에 정제된 CMG를 ~100 nM final에 첨가한 다음 20 μL/min으로 20 μL로 유입시킵니다. 15분 동안 배양합니다.
- 120μL의 'ATP/RPA 혼합물'(반응 완충액 1개, 0.75mg/mL BSA, 8mM DTT, 3.3mM ATP, 20nM EGFP-hRPA)을 구성하고 80μL에 대해 40μL/min의 속도로 플로우 셀로 유동합니다.
- 즉시 이미지 획득을 시작합니다. 30초마다 각 프레임에 대해 6 x 6 시야각을 획득합니다(또는 실험에 적합한 프레임 속도). 프레임 속도를 낮추면 레이저 노출과 형광단 광표백을 줄일 수 있습니다. 1% 출력에서 488nm 레이저로 EGFP-hRPA를 시각화합니다. CMG가 레이블이 지정된 경우(예: LD655) 10% 전력에서 640nm 레이저(최대 출력 = 30mW)로 CMG를 시각화합니다. 0.5% 출력으로 532nm 레이저로 SYTOX 오렌지 염색 DNA를 시각화합니다. 각 레이저는 50-100ms 노출로 사용하십시오.
참고: 플로우 셀의 입구 튜브가 한 액체 튜브에서 다른 튜브로 옮겨질 때마다 튜브 바닥에 있는 튜브 끝을 여러 번 두드린 후 액체에서 제거하십시오. 이렇게 하면 전송 중에 기포가 들어가는 것을 방지할 수 있습니다. 입구 튜브의 팁에 액체가 닿지 않는 시간을 최소화하고 액체가 채널 쪽으로 배출되어 팁으로 공기를 끌어당기므로 팁을 위쪽으로 향하게 하거나 팁을 크게 올리지 마십시오. 입구 튜브를 이송하는 동안 튜브에 장력을 가하면 튜브가 에폭시에서 당겨져 플로우 셀에 고정될 수 있으므로 튜브에 장력을 가하지 마십시오. 이중 가닥 DNA를 시각화하기 위해 SYTOX 오렌지가 있는 상태에서 DNA 풀기를 이미징하기 위해 ATP/RPA 혼합물에 대해 10x 반응 버퍼 1을 10x 반응 버퍼 2로 교체합니다. SYTOX 오렌지는 이러한 조건에서 DNA에 더 잘 결합합니다. 각 실험이 끝날 때 주사기 펌프를 완전히 비우십시오. 물을 채우고 여러 번 비우거나 비눗물로 분해하여 세척하여 때때로 씻으십시오.

그림 4: DNA가 표면에 묶여 있습니다. (A) 양쪽 끝에서 비오틴으로 DNA 기질을 테더링할 때 두 테더 사이의 거리는 끝단이 표면 (i)과 접촉하는 방식에 따라 달라질 수 있습니다. 한쪽 말단에 디곡시게닌을 사용하면 각 말단의 테더링을 시간적으로 분리하여 보다 일관된 테더 거리와 더 균일하게 늘어난 DNA(ii)를 얻을 수 있습니다. (B) 양쪽 끝이 연결되어 있고(디곡시게닌 라벨링) 형광 삽입 핵산 염색으로 염색된 DNA를 보여주는 시야의 예. 양쪽 끝으로 묶여 있는 DNA는 선으로 나타나고, 한쪽 끝으로만 묶여 있는 DNA는 점으로 나타납니다. 이상적으로는 DNA가 다른 DNA와 겹치지 않도록 가능한 한 조밀하게 묶여 있어야 합니다. 이미지는 512 x 512 픽셀(픽셀 크기 = 154.6nm)입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
결과
CMG가 DNA를 풀면 시간이 지남에 따라 특징적인 RPA 영역이 커집니다(그림 5). 풀린 DNA의 5' 말단은 표면에 묶여 있습니다. 따라서 테더와 포크 사이의 RPA 신호의 선형 스트레치로 간주됩니다. 3' 말단은 테더링되지 않으므로 포크와 함께 이동하며 컴팩트 EGFP-RPA 신호로 관찰됩니다. 압축된 풀림 전위 가닥의 위치는 640nm 레이저를 통해 시각화된 LD655-CMG와 함께 이동하는 복제 포크의 위치와 거의 일치합니다.
단일 가닥 DNA 흠집과 같은 손상은 관찰 가능한 풀림 이벤트의 수를 줄여 수집할 수 있는 데이터의 양을 제한하기 때문에 DNA 기질의 손상을 최소화하는 것이 중요합니다(그림 6).
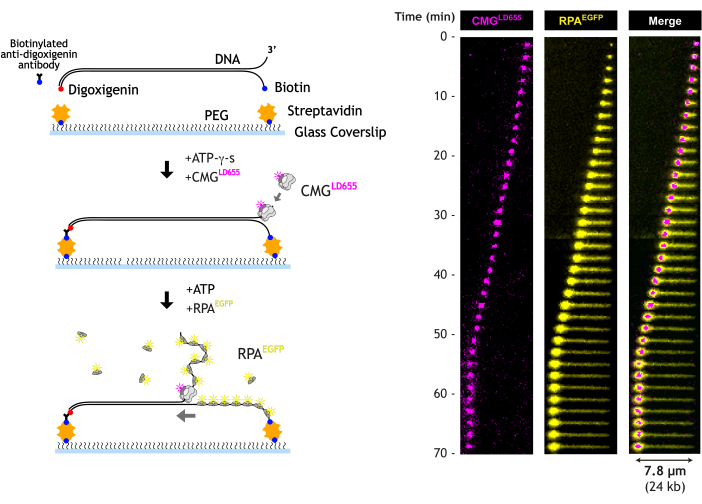
그림 5: 단일 분자 DNA 풀기 분석. DNA 기질은 커버슬립 표면에 묶여 있습니다. LD655로 표지된 정제된 CMG를 ATP-g-s에서 15분 동안 DNA와 함께 배양합니다. ATP 및 정제된 EGFP 표지 RPA가 추가되어 CMG에 의한 광범위한 DNA 풀림이 시작됩니다. 만화 회로도(왼쪽)와 대표 데이터의 카이모그래프(오른쪽)가 표시됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
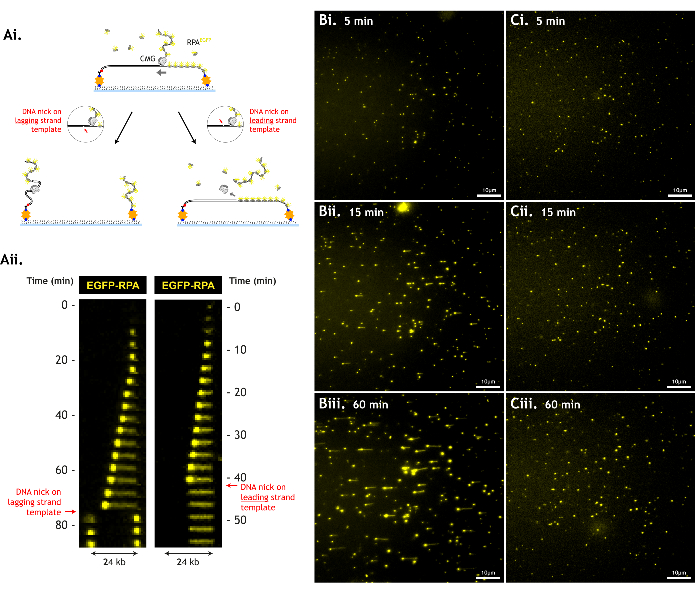
그림 6: DNA 손상으로 인해 분석 처리량이 감소합니다. (A) CMG는 DNA 백본의 절단(DNA 닉)을 지나 DNA를 풀 수 없습니다. 선행 가닥 템플릿에 흠집이 생기면 CMG가 DNA에서 미끄러져 나가고 CMG와 선행 가닥 템플릿이 모두 손실됩니다. 래깅 스트랜드 템플릿에 흠집이 생기면 래깅 스트랜드 템플릿이 DNA의 나머지 부분과 분리되고 각 DNA 조각은 각각의 테더로 수축됩니다. 이것은 (i) 만화 설계도와 (ii) 이러한 사건의 키모그래프 (ii)에 의해 설명됩니다. 단일 시야에서 (i) 5분, (ii) 15분, (iii) 60분에서 (B) 최소한으로 손상된 DNA 기질 대 (C) 더 손상된 DNA 기질이 있는 대표 데이터. 더 많이 손상된 DNA 기질은 유사한 수준의 풀림 활동(5분에서 성장하는 RPA 스폿의 유사한 밀도, 유사한 CMG 로딩/풀기 효율성을 나타냄)에도 불구하고 CMG가 초기에 흠집을 만나기 때문에 긴 풀림 경로를 생성하지 않습니다. 시야각은 512 x 512 픽셀(픽셀 크기 = 154.6nm)입니다. 1% 레이저 출력(488nm) 이미징 EGFP-RPA. 10 μm를 보여주는 스케일 바. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
토론
이 분석은 개별 CMG의 실시간 역학을 단독으로 그리고 원하는 추가 요인의 맥락에서 관찰하고 조사할 수 있는 플랫폼을 제공합니다. 그러나 많은 단일 분자 형광 기술과 마찬가지로 극복하기 위해 최적화가 필요할 수 있는 몇 가지 일반적인 문제가 있습니다. 이는 일반적으로 장기간에 걸친 형광단 이미징(광표백, 밝기), DNA 기판 준비(DNA 손상), 플로우 셀 표면의 품질(배경 노이즈, 비특이적 상호 작용) 또는 정제된 단백질 준비의 품질(뉴클레아제 오염, 라벨링 효율성)과 관련이 있습니다.
각 형광단은 광안정성과 밝기가 다르므로 적절한 분자를 선택하는 것이 중요합니다. RPA와 같이 형광 표지된 올리고머 단백질을 이미징할 때 많은 형광단이 가까운 거리에서 여기되어 가시 신호를 생성하므로 더 낮은 레이저 출력을 사용할 수 있습니다. 예를 들어 단일 subunit에 라벨링된 CMG와 같은 단일 형광단을 이미징하려면 형광단을 명확하게 관찰하기 위해 더 높은 레이저 출력이 필요합니다. Fluorophore 수명은 이미지 촬영 빈도를 줄이는 것과 같이 레이저 노출을 최소화함으로써 연장될 수 있습니다. 또한 형광단을 여기시키면 활성산소종(ROS)이 생성되어 광표백에 기여할 수 있습니다. 이미징 버퍼에 산소 제거 시스템을 포함하면 ROS를 제거하여 형광단의 수명을 연장할 수 있습니다. 그러나 일부 산소 제거 시스템은 pH12에 영향을 줄 수 있습니다.
DNA 기질 준비와 관련하여 흠집이나 단일 가닥 틈과 같은 DNA 손상을 최소화하는 것이 중요합니다. 과도한 손상은 광범위한 DNA 풀림을 방해하여 수집할 수 있는 데이터의 양을 제한합니다. 손상은 기계적 전단, 과도한 가열, 뉴클레아제 오염 또는 이미징 중 생성된 ROS로 인해 발생할 수 있습니다. DNA 시료를 조심스럽게 다루면 피펫팅을 위한 넓은 구경 팁을 사용하고, 천천히 피펫팅하고, 시료를 튕기지 않음으로써 전단을 최소화할 수 있습니다. ROS의 영향은 레이저 노출을 줄이거나 이미징 버퍼에 산소 제거 시스템을 포함하여 최소화할 수 있습니다. DNA 기질의 준비 후, 풀림 반응을 수행하기 전에 손상을 복구하기 위해 상용 DNA 복구 키트를 사용할 수 있습니다.
DNA 풀림의 효율성은 CMG의 순도와 활성에 따라 달라집니다. 각 정제 단계 후에 SDS-PAGE 전기영동으로 시료 순도를 평가하여 최적화가 필요한 부분을 결정하는 것이 좋습니다. 최종 단계 후에 너무 많은 오염 물질이 관찰되면 CaptoHiRes Q(5/50) 컬럼에서 용리에 사용되는 염 그래디언트 부피를 수정하는 데 도움이 될 수 있습니다. 또한 단백질 라벨링에 사용되는 과도한 형광 펩타이드를 제거하는 것이 매우 중요한데, 이는 커버슬립 표면에 원치 않는 배경을 생성할 수 있기 때문입니다. 또한 DNA 기질을 저하시킬 수 있는 뉴클레아제 오염을 피하는 것이 중요합니다. 실험 후 남은 DNA를 SYTOX 오렌지색으로 염색하는 것은 DNA가 크게 저하되었는지 여부를 확인하는 좋은 방법이 될 수 있습니다. 실험 과정에서 특정 수준의 DNA 손상은 피할 수 없지만, 심각한 손상은 종종 문제가 있는 뉴클레아제 오염을 나타냅니다.
또한 이 분석은 회절 제한 스폿의 분리능에 의해 본질적으로 제한되며, 형광 단백질을 별개의 것으로 구별하기 위해 수백 개의 염기쌍(그 이상은 아니더라도) 떨어져 있어야 합니다. 이로 인해 CMG 진행 및 상호 작용을 관찰할 수 있는 세부 정보가 제한됩니다.
각 분석에 대해 관찰되는 해제 이벤트의 수는 다양합니다. 성공적인 실험을 위해서는 시야각 512x512 픽셀(픽셀 크기 = 154.6nm)당 충분한 길이의 RPA 트랙이 최소 여러 개 있을 것으로 예상됩니다. 동일한 실험에서 여러 시야를 이미지화할 수 있으므로 필요한 경우 더 많은 데이터를 수집할 수 있습니다. 관은 길이가 같을 필요도 아니고, DNA의 끝부분까지 닿을 필요도 없다. 예를 들어, CMG를 추가하기 전에 SYTOX 염색된 DNA의 길이를 측정하여 각 실험의 평균 테더 거리를 결정할 수 있습니다. 이는 'μm traveled'에서 'kb unwound'로 거리를 변환하여 RPA tract(포크를 눈에 띄게 움직일 수 있을 만큼 충분한 DNA가 풀리는 한)에 대해 얼마나 많은 DNA가 풀렸는지 추정하는 데 사용할 수 있습니다.
CMG는 다양한 DNA 기질에서 풀림 활성을 나타내지만, CMG10의 발자국을 수용하기 위해 최소 30nt의 polyT 플랩에 무료 3' DNA 말단을 제공하는 것이 필수적입니다. 포크에 여러 비오틴 부분을 포함하면 강력한 표면 테더링이 보장됩니다. DNA 기질의 나머지 부분은 다양한 DNA 염기서열, 길이 및 화학적 변형을 포함하는 등 다양한 방법으로 재설계할 수 있습니다. DNA의 형태는 다양한 농도의 마그네슘 아세테이트를 사용하여 변경할 수 있습니다. 더 높은 농도(≥10mM)의 마그네슘 아세테이트에서 RPA 코팅된 ssDNA 필라멘트가 압축되어 풀림 중에 RPA 결합에 의해 DNA가 당겨집니다. 이는 DNA가 과도하게 움직이는 것을 방지하여 CMG의 위치와 풀림 진행을 보다 정확하게 측정할 수 있기 때문에 유용할 수 있습니다. 낮은 농도(~3mM)의 마그네슘 아세테이트에서 RPA-ssDNA는 전체적으로 편안한 상태를 유지합니다.
설명된 단일 분자 분석은 DNA 복제의 추가 측면을 조사하기 위해 구축되고 수정될 수 있는 플랫폼을 나타냅니다. DNA 복제 중에 CMG는 리플리솜과 그 구성 요소가 조립되는 코어 역할을 합니다. 따라서 CMG 역학에 미치는 영향을 연구하기 위해 TIMELESS, TIPIN 및 CLASPIN과 같은 보조 인자를 포함하여 이 분석에 추가 정제된 단백질을 추가할 수 있습니다. 이러한 단백질은 복제 포크(replication forks)13의 속도에 영향을 미치는 것으로 나타났지만, CMG 풀림 속도에 어떤 영향을 미치는지는 명확하지 않다. 따라서 이 분석을 사용하여 다양한 replisome 단백질이 CMG에 어떻게 영향을 미치는지 조사하는 것이 흥미로울 것입니다. DNA 중합효소의 추가는 이전에 효모 단백질에 대해 설명한 바와 같이 DNA 풀림만을 넘어 DNA 복제에 대한 더 나은 통찰력을 제공할 수 있습니다14. 또한, 변형된 CMG의 정제는 특정 돌연변이 또는 번역 후 변형이 헬리카제 활성에 어떻게 영향을 미치는지에 대한 더 나은 이해를 제공할 수있다 15,16. 또한, 상이한 DNA 기질을 설계하면 복제 스트레스를 모방하는 다양한 조건에서 CMG에 의한 DNA 풀림을 연구할 수 있다17. 이러한 변형에는 DNA 장애물(9,18), 가닥간 가교(inter-strand crosslinks)(19,20,2,1) 및 DNA 가닥(22)의 불연속성(discontinuities)이 포함된다.
공개
저자는 상충되는 재정적 이익 또는 기타 이해 상충이 없습니다.
감사의 말
pGC261 플라스미드를 제공한 Gheorghe Chistol과 펩타이드 합성 및 라벨링을 위한 Francis Crick Institute Chemical Biology Facility에 감사드립니다. 이 연구는 Francis Crick Institute의 자금 지원을 받았으며, Francis Crick Institute는 Cancer Research UK, UK Medical Research Council 및 The Wellcome Trust(CC2133)로부터 핵심 자금을 지원받았습니다.
자료
| Name | Company | Catalog Number | Comments |
| 2-Mercaptoethanol | Sigma-Aldrich | M3148-25ML | |
| Acrylamide / Bis Solution 40% | BioRad | 1610148 | |
| Agarose UltraPure | Invitrogen | 16500-500 | Used to prepare Tris Buffer (pH 8) |
| ÄKTA Pure | GE Healthcare | - | Used with Fraction Collector F9-C; protein purification system |
| ANTI-FLAG M2 affinity gel | Sigma-Aldrich | A2220 | |
| APS 10% | Sigma-Aldrich | A3678-25G | |
| ATP (200 mM) | Sigma-Aldrich | A7699-5G | |
| ATP-g-s (100 mM) | Sigma-Aldrich | 11162306001 | |
| Biotin-PEG coverslip | N/A | N/A | Prepared as described: https://doi.org/10.1016/j.ymeth.2012.03.033 |
| Biotinylated anti-digoxigenin antibody | Perkin Elmer | N/A | |
| Blu Tack | Bostik | N/A | Adhesive putty |
| Boric acid | Thermo Fisher Scientific | B/3800/53 | |
| BSA; 33 mg/mL | SIGMA-ALDRICH | A3858-10G | Diluted from the stock |
| CaCl2 | Sigma-Aldrich | C7902 | |
| CaptoHiRes Q (5/50) | Cytiva | 29275878 | Connected to the AKTA protein purification system; high-resolution ion exchange chromatography column |
| Casein (5% in water, 50 mg/mL) | Sigma-Aldrich | C4765-10ML | |
| ChemiDoc system | Bio-Rad | N/A | |
| Clips | N/A | N/A | |
| Cutting board | N/A | N/A | |
| Dialysis tubing (3.5 kDa) | Fisher Scientific | 11425859 | |
| digoxigenin-11-dUTP | Roche | 11209256910 | |
| DNA loading dye 6x | NEB | B7024S | |
| dNTP 10 mM | NEB | N0447S | |
| Double-sided tape (0.14 mm thick) | N/A | N/A | |
| DTT | Fluorochem Limited | M02712-10G | |
| DYKDDDDK peptide | N/A | N/A | Synthetised by chemical biology STP of The Francis Crick Institute |
| EDTA | Thermo Fisher Scientific | D/0700/68 | |
| EGFP-RPA | N/A | N/A | Purified as desribed in ‘Single Molecule Analysis’, Series Methods in Molecular Biology, Vol. 783 (2011), Peterman, E. J. G. and Wuite G. J. L. editors, Humana Press. Protein information: 2 mM, human, expressed in E. coli. |
| EGTA | Sigma-Aldrich | 03779-10G | |
| Epoxy - 5 minute | Devcon | 20845 | |
| Fujifilm FLA-5000 | Fujifilm | FLA-5000 | Fluorescent image analyzer |
| GelRed Nucleic Acid Stain; 10000x | Cambridge Bioscience | 41003-BT | Example of nucleic acid stain which is a safer alternative to ethidium bromide. |
| Glass or quartz slide with holes for tubing | Dremel Model 395 | 1 mm thick slide, cut to 2.4 cm x 1cm, two holes drilled 1.4 mm apart. The holes should be drilled to different sizes to accommodate different tubing at each end. | |
| Glycerol | Fisher Scientific | G/0650/17 | |
| Glycine HCl, pH 3.5 | Sigma-Aldrich | G8898 | |
| Hamilton syringe, 1000 series GASTIGHT, PTFE luer lock 1010TLL, PTFE Luer lock (with slots), volume 10 mL | Merck | 26211-U | |
| HEPES pH 7.5 | Sigma-Aldrich | H4034 | |
| I-CeuI | NEB | R0699L | |
| KCl | Fisher Scientific | P/4280/53 | |
| Magnesium acetate | Sigma-Aldrich | M2545 | |
| Maxi GeBaFlex-tube Dialysis Kit; MWCO 14 kDa | Generon | D055 | |
| Metal spatula | N/A | N/A | |
| Metal tweezers | N/A | N/A | |
| MgCl2 | Sigma-Aldrich | M2393 | |
| Millex-GP (Syringe filters) 0.22 µm | Merck | SLGP033RS | |
| NaCl | Sigma-Aldrich | S7653 | |
| Needle | N/A | N/A | |
| NotI-HF | NEB | R3189L | |
| NuPAGE 4%–12% Bis-Tris Protein Gels | Thermo Fisher Scientific | 10247002; | |
| NuPAGE MOPS SDS Running Buffer | Thermo Fisher Scientific | NP0001 | |
| Objective heater | Okolab | N/A | |
| PCR mix - 2x | Prepared from Phusion DNA polymerase (20 µL), 10 mM dNTPs (40 µL), 5x Phusion HF buffer (400 µL) and water (540 µL). | ||
| Peptide NH2-CHHHHHHHHHHLPETGG-COOH, labelled with LD655-MAL | N/A | N/A | peptide NH2-CHHHHHHHHHHLPETGG-COOH, labeled with LD655-MAL (Lumidyne Technologies) on the cysteine residue, was synthetised and purified by the peptide chemistry STP of The Francis Crick Institute |
| pGC261 plasmid | N/A | N/A | https://doi.org/10.1016/j.cell.2018.10.053 |
| Phusion High-Fidelity DNA Polymerase | NEB | M0530L | |
| Plastic tweezers | N/A | N/A | |
| Polyethylene tubing PE20 (inner diameter 0.015”, outer diameter 0.043”) | Becton Dickinson | 427406 | |
| Polyethylene tubing PE60 (inner diameter 0.03”, outer diameter 0.048”) | Becton Dickinson | 427416 | |
| Poly-Prep Chromatography Columns; 10 ml and 20 ml | Bio-Rad | 7311550 | |
| Potassium Glutamate (L-glutamic acid potassium salt monohydrate) | Sigma | G1501-1KG | |
| Protease inhibitor cocktail (cOmplete, EDTA free) | Roche | 5056489001 | |
| Pump 11 Elite Infusion/Withdrawal Programmable Single Syringe | Harvard Apparatus | 70-4504 | |
| QIAquick PCR Purification Kit | QIAGEN | 28104 | |
| Razor blade | VWR International Ltd | 233-0156 | |
| rCutSmart Buffer; 10x | NEB | B6004S | |
| Sodium acetate | Thermo Fisher Scientific | S/2120/53 | |
| Sortase (pentamutant) | N/A | N/A | Purified based on: https://doi.org/10.1073/pnas.1101046108 |
| Spin-X Centrifuge Tube Filters; 0.22 µm cellulose acetate | Fisher Scientific | 10310361 | |
| Sterile scalpel | N/A | N/A | |
| Steritop Vacuum Driven Disposable Filtration System; 0.22 um; PES; | Millipore | S2GPT05RE | |
| Streptavidin (1 mg/mL) in 1x PBS buffer | Sigma | S4762-10MG | 20 µL aliquotes |
| SYBR Gold | Thermo Fisher Scientific | S11494 | Highly sensitive nucleic acid stain we used to visualise DNA fork substrate. |
| SYTOX orange; 5 µM in DMSO | Thermo Fisher Scientific | S11368 | |
| T4 DNA ligase | NEB | M0202M | |
| T4 DNA Ligase Reaction Buffer | NEB | B0202S | |
| TEMED | Sigma-Aldrich | T9281-25ML | |
| TEV protease (1 mg/mL); EZCut | Biovision | 7847-10000 | |
| Tissue grinders, Dounce type (40 mL, Wheaton) | DWK Life Sciences | 432-1273 | |
| Tris base | Sigma-Aldrich | T1503 | Used to prepare Tris Buffer (pH 8) |
| Tris HCl | Sigma-Aldrich | T3253 | |
| Tween-20 | Promega UK Ltd | H5152 |
참고문헌
- Bell, S. P., Labib, K. Chromosome Duplication in Saccharomyces cerevisiae. Genetics. 203 (3), 1027-1067 (2016).
- Costa, A., et al. The structural basis for MCM2-7 helicase activation by GINS and Cdc45. Nat Struct Mol Biol. 18 (4), 471-477 (2011).
- Jones, M. L., Baris, Y., Taylor, M. R. G., Yeeles, J. T. P. Structure of a human replisome shows the organisation and interactions of a DNA replication machine. EMBO J. 40 (23), e108819 (2021).
- Moyer, S. E., Lewis, P. W., Botchan, M. R. Isolation of the Cdc45/Mcm2-7/GINS (CMG) complex, a candidate for the eukaryotic DNA replication fork helicase. Proc Natl Acad Sci U S A. 103 (27), 10236-10241 (2006).
- Douglas, M. E., Ali, F. A., Costa, A., Diffley, J. F. X. The mechanism of eukaryotic CMG helicase activation. Nature. 555 (7695), 265-268 (2018).
- Ilves, I., Petojevic, T., Pesavento, J. J., Botchan, M. R. Activation of the MCM2-7 helicase by association with Cdc45 and GINS proteins. Mol Cell. 37 (2), 247-258 (2010).
- Dewar, J. M., Walter, J. C. Mechanisms of DNA replication termination. Nat Rev Mol Cell Biol. 18 (8), 507-516 (2017).
- Burnham, D. R., Kose, H. B., Hoyle, R. B., Yardimci, H. The mechanism of DNA unwinding by the eukaryotic replicative helicase. Nat Commun. 10 (1), 2159 (2019).
- Kose, H. B., Larsen, N. B., Duxin, J. P., Yardimci, H. Dynamics of the eukaryotic replicative helicase at lagging-strand protein barriers support the steric exclusion model. Cell Rep. 26 (8), 2113-2125.e6 (2019).
- Kose, H. B., Xie, S., Cameron, G., Strycharska, M. S., Yardimci, H. Duplex DNA engagement and RPA oppositely regulate the DNA-unwinding rate of CMG helicase. Nat Commun. 11 (1), 3713 (2020).
- Tanner, N. A., Van Oijen, A. M. Visualizing DNA replication at the single-molecule level. Methods Enzymol. 475, 259-278 (2010).
- Swoboda, M., et al. Enzymatic oxygen scavenging for photostability without ph drop in single-molecule experiments. ACS Nano. 6 (7), 6364-6369 (2012).
- Baris, Y., Taylor, M. R. G., Aria, V., Yeeles, J. T. P. Fast and efficient DNA replication with purified human proteins. Nature. 606, 204-210 (2022).
- Lewis, J. S., et al. Tunability of DNA polymerase stability during eukaryotic DNA replication. Mol Cell. 77 (1), 17-25.e5 (2020).
- Li, Z., Xu, X. Post-translational modifications of the mini-chromosome maintenance proteins in DNA replication. Genes. 10 (5), 331 (2019).
- Lewis, J. S., et al. Mechanism of replication origin melting nucleated by CMG helicase assembly. Nature. 606 (7916), 1007-1014 (2022).
- Zeman, M. K., Cimprich, K. A. Causes and consequences of replication stress. Nat Cell Biol. 16 (1), 2-9 (2014).
- Sparks, J. L., et al. The CMG helicase bypasses DNA-protein cross-links to facilitate their repair. Cell. 176 (1-2), 167-181.e21 (2019).
- Taylor, M. R. G., Yeeles, J. T. P. The initial response of a eukaryotic replisome to DNA damage. Mol Cell. 70 (6), 1067-1080.e12 (2018).
- Amunugama, R., et al. Replication fork reversal during DNA interstrand crosslink repair requires CMG unloading. Cell Rep. 23 (12), 3419-3428 (2018).
- Wu, R. A., et al. TRAIP is a master regulator of DNA interstrand crosslink repair. Nature. 567 (7747), 267-272 (2019).
- Vrtis, K. B., Dewar, J. M., Chistol, G., Wu, R. A., Graham, T. G. W., Walter, J. C. Single-strand DNA breaks cause replisome disassembly. Mol Cell. 81 (6), 1309-1318.e6 (2021).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유