Method Article
Visualisation en temps réel d’une molécule unique du déroulement de l’ADN par CMG Helicase
Dans cet article
Résumé
Ce protocole démontre la réalisation d’un test sur une seule molécule pour la visualisation en direct du déroulement de l’ADN par l’hélicase CMG. Il décrit (1) la préparation d’un substrat d’ADN, (2) la purification de l’hélicase CMG de Drosophila melanogaster marquée par fluorescence, (3) la préparation d’une cellule d’écoulement microfluidique pour la microscopie à fluorescence par réflexion interne totale (TIRF) et (4) le test de déroulement de l’ADN à molécule unique.
Résumé
La duplication fidèle du génome est essentielle pour préserver la stabilité génétique des cellules en division. La réplication de l’ADN est effectuée pendant la phase S par un complexe dynamique de protéines appelé réplisome. Au cœur du réplisome se trouve l’hélicase CDC45-MCM2-7-GINS (CMG), qui sépare les deux brins de la double hélice de l’ADN de sorte que les ADN polymérases peuvent copier chaque brin. Lors de la duplication du génome, les réplisomes doivent surmonter une pléthore d’obstacles et de défis. Chacun d’entre eux menace la stabilité du génome, car l’incapacité à répliquer l’ADN complètement et avec précision peut entraîner des mutations, des maladies ou la mort cellulaire. Par conséquent, il est très intéressant de comprendre comment CMG fonctionne dans le réplisome lors d’une réplication normale et d’un stress de réplication. Ici, nous décrivons un test de microscopie à fluorescence par réflexion interne totale (TIRF) utilisant des protéines purifiées recombinantes, qui permet de visualiser en temps réel des molécules d’ADN étirées attachées à la surface par des complexes CMG individuels. Ce test fournit une plate-forme puissante pour étudier le comportement de la CMG au niveau de la molécule unique, permettant d’observer directement la dynamique des hélicases avec un contrôle en temps réel des conditions de réaction.
Introduction
La réplication de l’ADN est étroitement régulée, car une cellule doit dupliquer son génome avec précision pour éviter les mutations, la maladie ou la mort. La réplication de l’ADN eucaryote est effectuée par le complexe réplisome, qui déroule l’ADN parental et utilise l’ADN simple brin (ADNsb) comme modèle pour synthétiser un nouvel ADN. Dans la phase G1, les doubles hexamères catalytiquement inactifs de MCM2-7 sont chargés sur de l’ADN double brin (ADNdb) aux origines de réplication1. Dans la phase S, les complexes MCM2-7 sont activés par la liaison de CDC45 et GINS2 pour former des complexes CMG à 11 sous-unités (CDC45, MCM2-7, GINS). Chaque CMG initie le déroulement de l’ADN dans des directions opposées, formant l’unité centrale autour de laquelle le réplisome s’organise autour de3.
Il y a deux décennies, l’hélicase CMG a été identifiée pour la première fois comme un complexe de 11 sous-unités, essentiel à la réplication de l’ADN4. Depuis lors, notre compréhension de la CMG a considérablement progressé, de la charge et de l’activation 5,6 au déroulement et à la terminaison de l’ADN7. Les techniques traditionnelles de biochimie et de biologie structurale ont joué un rôle essentiel dans bon nombre de ces découvertes. cependant, ces méthodes étaient souvent limitées dans leur capacité à étudier les aspects les plus dynamiques de la CMG. Les méthodes à molécule unique utilisent la manipulation physique de biomolécules individuelles pour mesurer ou visualiser leur activité, une molécule à la fois. Cela peut être utilisé pour donner un aperçu de la dynamique en temps réel de protéines qui sont souvent manquées ou indétectables par d’autres techniques 8,9.
Ici, nous décrivons un test de microscopie à fluorescence par réflexion interne totale (TIRF) pour visualiser le déroulement de l’ADN par hélicase CMG en temps réel. Le CMG purifié et marqué par fluorescence est chargé sur l’extrémité libre de 3 pieds d’ADN long contenant une structure de fourche d’ADN préfabriquée. L’ADN linéaire est étiré sur une lamelle de biotine-PEG dans une cellule d’écoulement microfluidique en attachant chaque extrémité de l’ADN séquentiellement à la surface. Cette approche permet une connexion plus uniforme de l’ADN, ce qui réduit considérablement la variation qui doit être prise en compte lors de l’analyse des données. En présence d’ATP-γ-s, CMG est chargé sur l’ADN simple brin à l’extrémité 3' de la fourche. L’ATP-γ-s est un analogue de l’ATP lentement hydrolysable, qui permet à la CMG de se lier à l’ADN sans se dérouler. L’ajout ultérieur d’ATP, ainsi que de RPA purifié et marqué par fluorescence, active la CMG et déclenche un déroulement étendu de l’ADN. Visuellement, CMG se déplace le long de l’ADN, laissant derrière lui un volume croissant d’ADNsb lié à la RPA. L’extrémité de l’ADN non attachée se déplace avec la CMG, formant une « boule serrée » en raison du compactage causé par la liaison RPA. La conception de la cellule d’écoulement permet de remplacer le tampon à tout moment pendant le déroulement, ce qui donne un grand contrôle pendant et sur chaque expérience.
Ce protocole est divisé en quatre méthodes, qui peuvent être exécutées indépendamment les unes des autres. La section 1 décrit la préparation d’un substrat d’ADN linéaire fourchu de 20 kb pour des essais de molécules uniques. La section 2 décrit la purification et le marquage fluorescent de Drosophila melanogaster CMG (DmCMG). Les informations clés sur l’expression de DmCMG sont incluses dans la section des notes. La section 3 traite de la préparation d’une cellule d’écoulement microfluidique qui peut être utilisée sur un microscope TIRF. La section 4 décrit comment effectuer le test de déroulement de l’ADN à molécule unique.
Protocole
1. Préparation d’ADN linéaire fourchu de 20 kb utilisé dans des essais de molécules uniques (Figure 1)
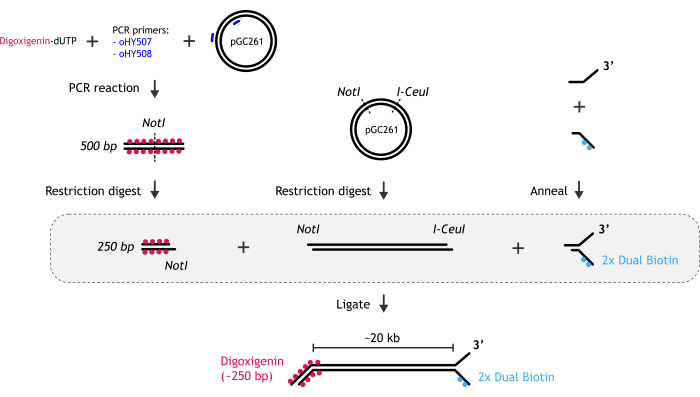
Figure 1 : Représentation graphique de la préparation du substrat d’ADN. (A) L’extrémité de la fourche de l’ADN biotinylé est créée par le recuit de deux oligonucléotides partiellement complémentaires : l’oligonucléotide biotinylé et l’autre non biotinylé. (B) Le fragment principal d’ADNdb (~20 kb) est généré par digestion de restriction du plasmide pGC261 avec deux enzymes pour créer un ADN linéaire avec différents surplombs à chaque extrémité. (C) L’extrémité de l’ADN duplex de la digoxigénine est obtenue par une réaction PCR réalisée en présence de digoxigénine-dUTP, suivie d’une digestion de restriction. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Génération de l’extrémité de la fourche de l’ADN biotinylé.
- Recuit l’ADN de la fourche.
- Mélanger deux oligonucléotides (oHY502 et oHYbio85) dans 1 tampon STE (100 mM de NaCl, 10 mM de Tris, pH 8,0 ; 1 mM d’acide éthylènediaminetétraacétique [EDTA]). La concentration finale en oligonucléotides est de 10 μM et le volume final de la réaction est de 100 μL.
- Incuber la réaction à 80 °C pendant 5 min sur un bloc thermique. Éteignez le bloc chauffant et laissez la réaction refroidir progressivement, en le laissant dans le bloc chauffant jusqu’à ce que la température descende en dessous de 30 °C.
- Validez l’efficacité du recuit sur un gel d’électrophorèse sur gel Tris-Borate-EDTA (TBE)- Polyacrylamide (PAGE).
- Préparez un gel d’acrylamide à 8 % et de TBE (pour 4 gels : 32,8 mL de H2O ; 4,8 mL de 10 tampons TBE (1 M de Tris, 1 M d’acide borique, 20 mM d’EDTA), 9,6 mL de solution d’acrylamide/Bis à 40 %, 37,5:1 ; 0,8 mL de persulfate d’ammonium à 10 % [APS], 40 μL de tétraméthyléthylènediamine [TEMED]).
ATTENTION : L’ETB, l’acrylamide, l’APS, le TEMED sont toxiques/nocifs. - Faites fonctionner un petit volume (1 μL) d’oligonucléotides recuits à côté des oligonucléotides individuels dilués avec un colorant de charge 6x sur le gel acrylamide - TBE à 8 % à 80 V, pendant 1 h à RT.
- Faites tremper le gel dans 1x TBE complété par un colorant d’acide nucléique approprié pendant 30 min sur un agitateur à bascule, puis scannez le gel à l’aide d’un système d’imagerie sur gel.
- Préparez un gel d’acrylamide à 8 % et de TBE (pour 4 gels : 32,8 mL de H2O ; 4,8 mL de 10 tampons TBE (1 M de Tris, 1 M d’acide borique, 20 mM d’EDTA), 9,6 mL de solution d’acrylamide/Bis à 40 %, 37,5:1 ; 0,8 mL de persulfate d’ammonium à 10 % [APS], 40 μL de tétraméthyléthylènediamine [TEMED]).
- Résolution de l’ADN recuit sur gel TBE-PAGE pour l’excision de gel.
- Si les oligonucléotides sont correctement recuits, résoudre le reste de l’échantillon en présence de BSA (~100 μL d’échantillon recuit, 25 μL de colorant de charge 6x, 2 μL de BSA 33 mg/mL). Pour charger le plus grand volume sur le gel, combinez plusieurs puits avec un scalpel stérile.
- Laissez le gel dans 1x TBE complété par une coloration d’acide nucléique appropriée pendant 30 min sur un agitateur à bascule.
- Transférez le gel dans un plateau en verre et visualisez l’ADN dans une pièce sombre sous lumière bleue. Utilisez un écran de protection approprié ou des lunettes de protection lorsque vous utilisez de la lumière bleue.
- Découpez la bande souhaitée à l’aide d’un scalpel stérile. Coupez l’excès de gel.
- Purifier la fourchette recuite par électroélution.
- Coupez un morceau de tube de dialyse suffisamment long pour insérer le morceau de gel, mouillez-le dans 1x TBE, et coupez une extrémité.
- Diluer la BSA à 0,3 mg/mL dans 500 μL de 1x TBE et pipeter tout le volume dans la tubulure.
- Utilisez la spatule en métal pour glisser le morceau de gel à l’intérieur. Déplacez le morceau de gel d’un côté du tube pour donner de l’espace tampon dans lequel l’ADN peut migrer pendant l’électroélution.
- Essorez l’excédent de tampon et coupez la deuxième extrémité du tube.
REMARQUE : Le volume restant déterminera le volume final et la concentration. - Placez la tubulure de dialyse dans le réservoir d’électrophorèse à agarose dans 1 tampon TBE, en vous assurant que la tubulure est complètement immergée.
REMARQUE : Assurez-vous que le morceau de gel est placé du côté le plus proche de l’électrode négative afin que l’ADN puisse migrer hors du gel dans le sac de dialyse. - Faites fonctionner l’électroélution à 80 V pendant 1 à 2 h.
- Dialysez l’ADN électroélué en tampon Tris de 10 mM (pH 8).
- Après l’électroélution, sortez le sac de dialyse du réservoir et séchez une extrémité avec du mouchoir. Évitez de toucher la partie centrale du tube avec du tissu, où se trouvent le gel et l’échantillon.
- Retirez le clip de l’extrémité séchée et faites doucement piper l’échantillon de haut en bas à l’intérieur du tube pour mélanger l’ADN dans le tampon.
- Trempez une spatule propre dans 1x tampon TBE et retirez le morceau de gel du tube.
REMARQUE : Lors du retrait du morceau de gel, retirez le moins de tampon possible. - Clipsez à nouveau l’extrémité ouverte du sac de dialyse, en éliminant tout air de l’intérieur.
- Placez le sac de dialyse dans un bécher de 2 L rempli de 1,5 L de tampon Tris 10 mM (pH 8), 20 mM de NaCl et 2 mM de MgCl2. Enveloppez les clips avec du parafilm pour coller le tube sur le bord du bécher tout en immergeant complètement le tube dans le tampon.
REMARQUE : Le sel stabilise les oligo-duplex courts. - À l’aide d’une barre magnétique, remuez doucement le tampon et dialysez l’échantillon à température ambiante (RT) pendant au moins 3 à 4 h ou à 4 °C pendant la nuit.
- Après la dialyse, séchez une extrémité du sac de dialyse avec un mouchoir (pour ne pas diluer l’ADN) et déclipsez cette extrémité.
- Pipetez doucement l’échantillon de haut en bas à l’intérieur de la tubulure et transférez-le dans un tube propre de 1,5 ml.
- Mesurez la concentration de l’ADN à l’aide d’un spectrophotomètre à microvolume.
- Recuit l’ADN de la fourche.
- Génération d’un fragment de 20 ko
- Effectuer une digestion de restriction du plasmide18 pGC261 à l’aide d’enzymes NotI/I-CeuI.
- Mélangez délicatement 8 μL de NotI-HF (20 000 U/mL), 8 μL d’I-CeuI (5 000 U/mL) et le plasmide pGC261 (concentration finale du plasmide ~40 ng/μL dans une réaction de 200 μL) dans un tampon recommandé par les fabricants d’enzymes de restriction.
- Incuber la réaction à 37 °C pendant la nuit.
- Résolution de l’ADN digéré sur gel d’agarose pour l’excision du gel.
- FACULTATIF : Exécutez d’abord une petite quantité de la réaction sur un gel d’agarose à 0,6 % pour tester l’efficacité de la digestion avant de charger l’ensemble de la réaction.
- Préparez un gel d’agarose à 0,6 % en suivant les étapes 1.2.2.3-1.2.2.4.
- Mélanger 0,48 g de poudre d’agarose avec 80 ml de 1 tampon TBE. Passez la solution au micro-ondes jusqu’à ébullition. Remuez pour vous assurer que l’agarose est complètement fondue. Laisser refroidir quelques minutes, puis verser dans le réservoir désigné.
- Pour combiner les puits afin d’accueillir le plus grand volume de l’échantillon, collez au préalable un morceau de ruban adhésif sur plusieurs puits du peigne.
- Une fois que le gel s’est solidifié, retirez délicatement le peigne et remplissez le réservoir avec 1x tampon TBE. Mélangez délicatement l’échantillon avec le colorant de charge d’ADN et faites fonctionner le gel à 120 V pendant 1 h.
- Laissez le gel dans 1x TBE complété par une coloration d’acide nucléique appropriée sur un agitateur à bascule jusqu’à ce qu’il soit coloré.
- Transférez le gel coloré dans un plateau en verre et visualisez l’ADN dans une pièce sombre sous une lumière bleue.
- Découpez la bande souhaitée à l’aide d’un scalpel stérile. Coupez l’excès de gel.
- Facultatif : Purifiez le fragment souhaité par électroélution comme décrit aux étapes 1.1.4 et 1.1.5. Omettez ici le sel du tampon de dialyse, car il n’est nécessaire que pour stabiliser l’oligo-duplex court.
- Effectuer une digestion de restriction du plasmide18 pGC261 à l’aide d’enzymes NotI/I-CeuI.
- Génération de l’extrémité de l’ADN duplex de la digoxigénine
- Effectuez une réaction PCR avec digoxigénine-dUTP (dig-dUTP) en utilisant l’ADN pGC261 comme matrice.
- Mélange : 400 μL d’eau, 8 μL de pGC261 (0,8 ng/μL), 3,5 μL d’amorce oHY507 (100 μM), 3,5 μL d’amorce oHY508 (100 μM), 8 μL de dig-dUTP (1 mM), 400 μL de 2 mélanges PCR (préparés à partir d’ADN polymérase (20 μL), 10 mM dNTPs (40 μL), 5 tampons haute fidélité (HF) (400 μL) et de l’eau (540 μL).
- Exécutez la réaction suivante dans un thermocycleur :
98 °C - 1 min
30x : 98 °C - 20 s ; 65 °C - 20 s ; 72 °C - 30 s ;
72 °C - 10 min
4 °C - Maintien
- Digérer et purifier un produit PCR marqué à la digoxigénine
- Purifiez le produit PCR à l’aide d’un kit de purification d’ADN commercial.
- Mélanger 10 μL de NotI-HF (20 000 U/mL) avec le produit PCR dans un tampon recommandé par le fournisseur de l’enzyme (concentration finale d’ADN de ~50 ng/μL dans une réaction de ~200 μL). Incuber la réaction à 37 °C pendant la nuit.
- Purifiez l’ADN digéré à l’aide d’un kit de purification d’ADN commercial.
- Effectuez une réaction PCR avec digoxigénine-dUTP (dig-dUTP) en utilisant l’ADN pGC261 comme matrice.
- Assemblez les composants pour fabriquer le substrat d’ADN.
- Mélangez délicatement l’extrémité de la fourche biotinylée, le fragment d’ADN de 20 kb et le fragment de PCR Not-I marqué à la digoxigénine avec 5 μL d’ADN ligase T4 (400 000 U/mL) dans un tampon recommandé par le fournisseur d’enzyme dans une réaction de 200 μL. Cependant, sur la base du fragment d’ADN de 20 kb ajouté (1-5 μg), ajoutez environ 100 fois l’excès molaire de l’extrémité de la fourche biotinylée et du fragment de PCR.
- Aliquote la réaction dans des tubes PCR (50 μL chacun) et incubation à 16 °C dans un thermocycleur pendant la nuit.
- Résolvez l’échantillon ligaturé sur un gel d’agarose à 0,6 % et purifiez l’ADN ligaturé par électroélution comme décrit aux étapes 1.2.2 et 1.2.3.
- Conservez l’ADN congelé instantané à -80 °C.
2. Purification de la CMG de Drosophila melanogaster (Figure 2)

Figure 2 : Purification de la CMG de Drosophila melanogaster à partir de 4 L de cellules Hi Five. Les protéines ont été résolues sur un gel de polyacrylamide Bis-Tris à 4 % à 12 % sous 200 V en présence d’un tampon MOPS. L’échantillon est montré à chaque étape de la purification (lysat cellulaire - 2 μL, élution FLAG - 10 μL, après la première colonne d’échange d’ions - 10 μL, et après le marquage et la deuxième colonne d’échange d’ions - 1 μL. (A) La coloration de Coomassie confirme la présence des 11 sous-unités du complexe CMG avant (10 μL), et après (1 μL) marquage fluorescent. (B) L’efficacité de marquage de la sous-unité MCM3 a été validée par balayage de Cy5 avec un analyseur d’images fluorescentes utilisant un filtre rouge passe-long (LPR). Veuillez cliquer ici pour voir une version agrandie de cette figure.
REMARQUE : Pour préparer la CMG de Drosophila melanogaster marquée par fluorescence, un site de clivage TEV (ENLYFQG) suivi de quatre résidus Gly a été introduit en aval de l’étiquette N-terminale FLAG sur la sous-unité MCM3 (dans le vecteur pFastBac1)10. Pour exprimer le complexe, le système d’expression du baculovirus a été utilisé. Pour la transfection initiale, les cellules Sf21 ont été utilisées séparément pour chaque sous-unité CMG (stade du virus P1). Pour amplifier davantage les virus, des cellules Sf9 ont été utilisées (stade viral P2). Par la suite, des cultures de cellules Sf9 (100 mL pour chaque sous-unité de CMG ; 0,5 x 106 cellules/mL) ont été infectées par 0,5 mL de virus P2 complété par 10 % de sérum de veau fœtal (stade du virus P3). Pour exprimer l’ensemble du complexe CMG dans 4 L de cellules Hi Five (1 x 106 cellules/mL), 200 mL de virus P3 ont été utilisés pour chacune des sous-unités. Après la récolte des cellules Hi Five exprimant le complexe CMG, la pastille de cellules peut être congelée dans de l’azote liquide et stockée à -80°C. Effectuez l’ensemble de la purification sur glace ou à 4 °C. Les tampons peuvent être préparés à l’avance, à condition d’ajouter les agents réducteurs (DTT ou 2-Mercaptoéthanol) et les inhibiteurs de protéase (ATTENTION) juste avant l’utilisation. Assurez-vous que tous les tampons sont prérefroidis à l’avance, filtrés et dégazés.
- Préparez les tampons suivants.
- Préparez le tampon de remise en suspension A en mélangeant 25 mM d’acide 4-(2-hydroxyéthyl)-1-pipérazine-éthananesulfonique (HEPES) pH 7,5, 0,02 % de Tween-20, 10 % de glycérol, 15 mM de KCl, 2 mM de MgCl2, 2 mM de 2-mercaptoéthanol, 1 mM d’EDTA et 1 mM d’éthylène glycol-bis (éther β-aminoéthylique)-N,N,N′,N′-acide tétraacétique (EGTA).
ATTENTION : Le 2-mercaptoéthanol, le DTT et l’EDTA sont toxiques/nocifs - Préparez un tampon salin tamponné au tris (TBS ; HCl de glycine 0,1 M, pH 3,5) pour la préparation du gel d’affinité ANTI-FLAG M2.
- Préparez le tampon A-100 en mélangeant 25 mM HEPES pH 7,5, 0,02 % Tween-20, 10 % de glycérol, 100 mM de KCl, 1 mM de DTT, 1 mM d’EDTA et 1 mM d’EGTA.
- Préparez le tampon de dialyse en ajoutant 25 mM de HEPES pH 7,5, 50 mM d’acétate de sodium, 10 mM d’acétate de magnésium, 10 % de glycérol et 1 mM de DTT.
- Préparez le tampon TBS en mélangeant 50 mM de Tris-HCl pH 7,5 et 150 mM de NaCl.
- Préparez le tampon de remise en suspension A en mélangeant 25 mM d’acide 4-(2-hydroxyéthyl)-1-pipérazine-éthananesulfonique (HEPES) pH 7,5, 0,02 % de Tween-20, 10 % de glycérol, 15 mM de KCl, 2 mM de MgCl2, 2 mM de 2-mercaptoéthanol, 1 mM d’EDTA et 1 mM d’éthylène glycol-bis (éther β-aminoéthylique)-N,N,N′,N′-acide tétraacétique (EGTA).
- Remettre en suspension la pastille à partir d’une culture de 4 L de cellules Hi Five dans 200 mL de tampon de remise en suspension à froid A complété par des comprimés cocktail inhibiteurs de protéase. Mélangez les tubes par inversion jusqu’à ce que la totalité de la pastille soit complètement remise en suspension.
- Transférez les cellules remises en suspension dans un homogénéisateur Dounce de 40 mL prérefroidi. Pour récupérer le plus grand nombre possible de cellules, lavez les tubes utilisés pour la congélation des granulés de cellules avec le même type de tampon et ajoutez-les à l’homogénéisateur Dounce. Assurez-vous que le volume total dans l’homogénéisateur ne dépasse pas la limite recommandée. Lyser les cellules sur la glace par 60 à 70 coups.
REMARQUE : Assurez-vous que le piston atteint le bas du tube, mais n’exercez pas trop de pression car l’homogénéisateur pourrait se briser. Attention à ne pas sortir le piston au-dessus du niveau du liquide, car cela introduirait des bulles. - Répétez l’étape précédente jusqu’à ce que l’échantillon entier soit homogénéisé et combinez-le dans le bécher prérefroidi. Évaluez le volume final de l’échantillon. Goutte à goutte, ajoutez du KCl filtré à la concentration finale de 100 mM, en mélangeant délicatement.
- Prérefroidissez la centrifugeuse à 4 °C à l’avance. Versez l’échantillon dans des tubes de centrifugation, en veillant à ce que le volume recommandé par le producteur soit atteint. Équilibrez les tubes sur une balance.
REMARQUE : Les tubes d’un volume trop petit peuvent se briser lors de la centrifugation. - Centrifuger les cellules lysées à 23 500 x g pendant 15 à 30 min à 4 °C. Pendant ce temps, préparez le gel d’affinité ANTI-FLAG M2 en ligne avec l’étape suivante.
- Mélangez délicatement le flacon contenant le gel affinitaire ANTI-FLAG M2 (suspension à 50%). Coupez l’extrémité d’une pointe de pipette P-100 (pour élargir l’pointe) et utilisez-la pour transférer immédiatement 4 ml de suspension (2 ml de billes) dans une colonne de chromatographie poly-prep de 20 ml. Pour récupérer les billes coincées dans la pointe, lavez la pointe avec du TBS et ajoutez-la à la colonne. Laissez toutes les perles se déposer à l’intérieur de la colonne, mais attention à ne pas les sécher.
- Lavez la résine avec 3 fois le volume de billes (6 mL au total) de 0,1 M de chlorhydrate de glycine pH 3,5. Par la suite, lavez les perles avec 3 à 5 billes de volume de TBS (6 à 10 ml). Ensuite, lavez les perles 3 fois (6 ml) avec le tampon A-100.
REMARQUE : Ne laissez pas les billes dans la solution de 0,1 M de chlorhydrate de glycine pH 3,5 pendant plus de 20 minutes. Pour augmenter la pression d’écoulement et la vitesse de lavage, couvrez la colonne avec un couvercle. Ne laissez pas la résine sécher à aucun moment. - Après avoir préparé la résine, laissez 2 mL de tampon A-100 au-dessus de la résine et fermez la sortie de la colonne.
- Après la centrifugation, verser délicatement le surnageant dans des tubes prérefroidis de 50 ml. Attention à ne pas perturber la pastille. Prélever une petite aliquote (~10 μL) pour valider par électrophorèse SDS-PAGE plus tard.
- Ajoutez une quantité égale de billes ANTI-FLAG M2 (préparées conformément à l’étape précédente) dans chacun des tubes de 50 ml. Essayez de récupérer toutes les billes de la colonne en les remettant en suspension avec quelques millilitres supplémentaires de Buffer A-100 et en transférant cette remise en suspension dans les tubes.
- Incuber l’échantillon (surnageant) avec la résine ANTI-FLAG M2, en tournant pendant 2,5 h à 4 °C.
- Après l’incubation, faire tourner l’échantillon à 1 000 x g pendant 5 min à 4 °C. À l’aide d’une pipette, prélever la majorité du surnageant. À l’aide d’une pointe coupée, remettre les billes en suspension dans quelques millilitres du surnageant restant et les transférer dans deux tubes de 15 ml. Pour récupérer les billes collées aux parois des tubes de 50 ml, ajoutez quelques millilitres de tampon A-100 et transférez-le également dans les tubes de 15 ml.
- Faites tourner les tubes de 15 mL à 1 000 x g pendant 5 min à 4 °C. Retirez le surnageant.
- Lavez les billes en ajoutant ~14 ml de tampon A-100 dans chaque tube, puis faites une rotation courte et douce à 4 °C, puis essorez à 1000 x g pendant 5 min à 4 °C. Répétez les lavages deux fois. Prélever de petites aliquotes de chaque lavage (~10 μL) pour valider par électrophorèse SDS-PAGE plus tard.
- Cette étape (2.16) est une procédure alternative aux étapes précédentes (2.13-2.15).
- Après l’incubation avec la résine ANTI-FLAG M2, verser l’échantillon dans deux colonnes de chromatographie poly-prep de 20 mL.
- Si vous choisissez cette méthode, laissez les billes se déposer au fond de la colonne et l’échantillon non lié traverser la colonne par l’écoulement gravitaire. Ensuite, ajoutez le tampon A-100 directement dans la colonne et laissez-le laver l’échantillon par gravité.
- Transférez les billes (remises en suspension dans le tampon A-100) dans deux colonnes de chromatographie poly-prep de 10 ml.
- Laissez le tampon A-100 passer à travers la résine, fermez la sortie lorsque le niveau du tampon atteint le haut de la résine et éluez la protéine des billes en ajoutant le tampon A-100 complété par un peptide de 200 μg/mL (DYKDDDDK) (tampon d’élution FLAG).
- Pour la première élution, ajoutez 3 ml du tampon d’élution FLAG et tournez doucement la colonne fermée pendant 15 minutes à RT. Ouvrez la sortie pour recueillir la première fraction d’élution.
- Fermez la sortie et ajoutez 2 ml du tampon d’élution FLAG dans chaque colonne. Tournez encore 10 minutes et récupérez la deuxième fraction d’élution.
- Mélanger toutes les fractions éluées ensemble (~10 mL) et conserver à 4 °C. Prélever une petite aliquote de la fraction éluée (~10 μL) pour valider par électrophorèse SDS-PAGE plus tard.
- Filtrez l’échantillon élué à l’aide de filtres à seringue de 0,22 μm.
- Préparez à l’avance la colonne CaptoHiRes Q (5/50) (colonne de chromatographie échangeuse d’ions à haute résolution) connectée à un système de purification de protéines, conformément au protocole du fabricant. Ensuite, équilibrez la colonne avec le tampon A-100 et chargez l’échantillon filtré sur la colonne équilibrée.
- Laver la colonne avec 20 volumes de colonne (CV) de tampon A-100 (~20 mL au total car le volume de la colonne est de ~1 mL).
- Pour éluer la protéine, préparez deux tampons à l’avance : le tampon A-100 et le tampon B avec la même composition que le tampon A-100, mais avec 550 mM de KCl au lieu de 100 mM de KCl. Réglez l’élution de gradient pour 20 CV (~20 mL) avec une concentration de sel croissante de 100 mM à 550 mM de KCl. Collectez des élutions de 0,3 à 0,5 mL dans un collecteur de fractions.
REMARQUE : La CMG doit éluer à une concentration d’environ 70 % à 75 % du tampon B. Prélever de petites aliquotes des fractions de lavage et éluées pour l’électrophorèse SDS-PAGE. - Effectuez une électrophorèse SDS-PAGE pour confirmer que la CMG est présente dans les fractions choisies. Pendant ce temps, nettoyez la colonne et le système de purification selon le protocole du fabricant.
- Incuber les fractions choisies avec la protéase TEV pendant la nuit en mélangeant 50 μL de protéase TEV (1 mg/mL) par 1 mL d’échantillon.
- Prémouillez un tube de dialyse et assurez-vous que la membrane n’est pas endommagée. Ajoutez le mélange CMG/TEV dans la tubulure de dialyse et placez-le dans un bécher de 2 L avec 1,5 L de tampon A-100 prérefroidi et la barre magnétique à l’intérieur. Dialyser en remuant doucement pendant une nuit à 4°C.
- Mélanger l’échantillon avec 50 μM de peptide marqué LD655 et 10 μg/mL d’enzyme Sortase en présence de 5 mM de CaCl2. Incuber la réaction à 4 °C, en tournant pendant 30 min, en veillant à ce que le tube soit protégé de la lumière. Prélever une petite aliquote avant et après l’étiquetage pour l’électrophorèse SDS-PAGE.
- Filtrez l’échantillon étiqueté à l’aide de filtres centrifuges de 0,22 μm avant de le charger dans le système de purification, car le peptide peut précipiter.
- Préparez à l’avance la colonne de chromatographie par échange d’ions à haute résolution, comme décrit précédemment (étape 2.20). Cette fois, couvrez la colonne et le système de collecte de fractions avec une feuille d’aluminium pour protéger l’échantillon de la lumière.
- Pour retirer le peptide libre, chargez l’échantillon filtré sur la colonne de chromatographie par échange d’ions à haute résolution. Effectuez la purification de la même manière que celle décrite précédemment (étapes 2.21-2.22). Assurez-vous que la CMG étiquetée élue à une concentration de tampon-B similaire à celle précédente.
- Effectuer l’électrophorèse SDS-PAGE pour valider la qualité des fractions choisies. Pour obtenir une image de fluorescence, ne faites pas bouillir les échantillons avant le chargement et assurez-vous que le réservoir d’électrophorèse est protégé de la lumière. Visualisez d’abord la fluorescence des protéines à l’aide d’un système d’imagerie sur gel, puis colorez le gel avec le colorant Coomassie et imagez-le à nouveau pour visualiser toutes les protéines.
- Dialysez les fractions choisies pendant la nuit à 4 °C, contre 1,5 L de tampon de dialyse. Si nécessaire, concentrez l’échantillon.
- Congelez rapidement la protéine dans de l’azote liquide et stockez-la à -80 °C jusqu’à une nouvelle utilisation.
3. Préparation de la cellule d’écoulement (Figure 3)
- Préparez les lamelles de protection à la biotine-PEG en suivant le protocole11 décrit précédemment, en omettant l’étape de cuisson au four. Les lamelles de recouvrement Biotin-PEG sont stables pendant au moins 1 mois sous vide à RT. Pour la préparation de la cellule d’écoulement, coupez la lamelle de biotine-PEG (24 mm x 60 mm) en deux (environ 24 mm x 30 mm).
REMARQUE : Veillez à ne pas toucher la zone centrale de la lamelle où se trouveront les canaux d’écoulement. - Préparez de petits morceaux de verre en gravant et en cassant une lame de verre en morceaux d’environ 2,4 cm x 1 cm.
- Percez deux trous espacés de 1,4 mm à l’aide d’un foret diamanté de 0,8 mm, l’un légèrement plus large que l’autre (0,043 » = entrée, 0,048 » = sortie). Vérifiez que les trous sont de la bonne taille en essayant d’insérer un morceau du tube d’entrée/sortie, en utilisant le même foret pour élargir le trou jusqu’à ce que le tube s’adapte. Assurez-vous que le tube est suffisamment bien ajusté pour ne pas tomber facilement.
REMARQUE : Un tube d’entrée plus étroit est utilisé pour minimiser le volume mort. - Coupez du ruban adhésif double face de la même forme que les morceaux de verre.
- Alignez la glissière sur le ruban et enfoncez une aiguille dans chaque trou pour marquer leurs positions sur le ruban. À l’aide d’une lame de rasoir, découpez un canal qui englobe les deux trous.
REMARQUE : Ne coupez pas le canal trop long, car l’espace excessif de l’autre côté de chaque trou peut rester sec lorsque le liquide s’écoule et peut entraîner des problèmes de bulles d’air pendant l’écoulement. Lors de la coupe, essayez de couper les bords longs en une seule coupe nette, car les bords effilochés de plusieurs coupes peuvent affecter la qualité du canal d’écoulement. Dans le même but, ne coupez pas les bords du canal d’écoulement, car les coupes dans la pièce utilisable peuvent affecter l’écoulement du liquide ou même une fuite dans la cellule d’écoulement assemblée. - Nettoyez le morceau de verre avec de l’acétone et un mouchoir en papier jusqu’à ce qu’il soit sec, puis placez-le sur une surface propre. Décollez un côté du ruban adhésif et collez-le sur le morceau de verre de sorte que les deux trous soient complètement à l’intérieur du canal.
- Scellez le ruban sur le morceau de verre à l’aide d’une pointe de pipette p1000 pour éliminer les bulles en appuyant sur la surface avec une pression ferme mais modérée. Passez sur toute la surface pour sceller.
- Décollez le deuxième bord du ruban adhésif des deux morceaux de verre et placez-les sur une surface propre avec le côté collant vers le haut. Placez les lames à proximité les unes des autres mais sans les toucher dans la position où elles seront attachées à la lamelle (laissez suffisamment d’espace entre elles pour que l’époxy crée un joint complet autour de chaque pièce de verre).
- Prenez la demi-lamelle de protection en biotine-PEG à l’aide d’une pince à épiler en plastique, en la tenant uniquement par le bord (ne touchez pas la zone qui formera le canal d’écoulement), et abaissez le côté fonctionnalisé en PEG sur l’adhésif. Fixez-le en appuyant avec un doigt pour le fixer en place, puis complétez l’étanchéité en frottant modérément fermement la surface avec une pointe de pipette pour éliminer les bulles d’air (pas sur la zone du canal d’écoulement car cela pourrait se briser), puis retournez-le.
REMARQUE : Manipulez la lamelle avec précaution. - Pour chaque cellule d’écoulement, coupez ~10 cm de chaque type de tube en polyéthylène (PE20 et PE60). Utilisez le tube plus étroit comme entrée dans la chambre d’écoulement pour réduire le volume mort.
- Insérez le tube dans les trous de la glissière à la main. Si le diamètre des trous est correct, assurez-vous que le tube se dresse tout seul dans le trou une fois inséré. Coupez l’embout du tube à un léger angle <45° pour faciliter légèrement l’insertion du tube et empêcher le tube de former un joint contre la lamelle, ce qui peut être le cas si l’extrémité est plate (cela peut obstruer l’écoulement).
REMARQUE : L’ajustement précis du tube dans la pièce de verre rend la construction de la cellule d’écoulement plus fiable et permet d’éviter l’introduction de bulles dans la cellule d’écoulement. - Mélangez bien les composants époxy, puis utilisez une pointe p200 pour tamponner l’époxy afin de sceller le tube et de créer un joint autour de chaque morceau de verre. Ajoutez suffisamment d’époxy pour qu’il remonte jusqu’au bord du rond de lamelle, car cela renforcera également la lamelle délicate. Assurez-vous toutefois qu’aucun époxy ne pénètre sur la face inférieure de la lamelle, car cela pourrait l’empêcher de reposer à plat sur la scène.
REMARQUE : Si nécessaire, de petites quantités d’époxy en excès peuvent être grattées sur la face inférieure à l’aide d’une lame de rasoir. - Laissez agir au moins 30 à 60 minutes pour que l’époxy durcisse complètement.
REMARQUE : Si la cellule d’écoulement ne doit pas être utilisée immédiatement, stockez-la sous vide à RT après durcissement complet. - Après avoir utilisé la cellule d’écoulement, récupérez les morceaux de verre et réutilisez-les indéfiniment. Retirez le tube et placez la cellule d’écoulement dans un bocal contenant de l’acétone pendant au moins 24 h pour ramollir l’époxy et l’adhésif double face, ce qui permet de retirer facilement les morceaux de verre. Avant de les réutiliser pour fabriquer une autre cellule d’écoulement, nettoyez les morceaux de verre en les frottant avec une éponge et de l’eau savonneuse, séchez-les et frottez-les avec de l’acétone et du papier de soie.

Figure 3 : Représentation graphique de la préparation de la cellule d’écoulement. (A) Coupez du ruban adhésif double face pour qu’il corresponde à la taille de la pièce de verre. Alignez la glissière sur le dessus du ruban adhésif et marquez la position de chaque trou avec une aiguille. À l’aide d’une lame de rasoir, coupez autour de chaque prise pour créer un canal. (B) Décollez un côté du ruban et collez-le sur le morceau de verre. Assurez-vous que les deux trous sont à l’intérieur du canal. Décollez la deuxième extrémité du ruban adhésif et collez la lamelle de protection biotine-PEG sur le dessus. (C) Insérez le tube en polyéthylène dans chaque trou et scellez le tube en place avec de l’époxy, en scellant également chaque morceau de verre à la lamelle. (D) Après avoir utilisé les deux canaux, retirez le tube et placez la cellule d’écoulement dans un bocal de coloration rempli d’acétone. Après environ 24 h, l’époxy et le ruban se seront ramollis et les couches de la cellule d’écoulement pourront être décollées. Les morceaux de verre peuvent être récupérés et stockés dans de l’acétone pour être réutilisés indéfiniment pour la fabrication de la prochaine cellule d’écoulement. Veuillez cliquer ici pour voir une version agrandie de cette figure.
4. Test TIRF à molécule unique pour visualiser le déroulement de l’ADN médié par CMG
- Préparez les tampons suivants.
- Préparez le tampon de blocage (20 mM de Tris, pH 8,0 ; 50 mM de NaCl ; 2 mM d’EDTA ; 0,2 mg/mL de BSA). Filtrez le tampon à l’aide d’un filtre à seringue de 0,22 μm et stockez-le à 4 °C.
- Préparez 10x tampon de réaction 1 (250 mM HEPES pH 7,5 ; 50 mM NaCl ; 100 mM acétate de magnésium). Filtrez le tampon à l’aide d’un filtre à seringue de 0,22 μm et stockez-le à 4 °C.
- Préparez 10x tampon de réaction 2 (250 mM Tris, pH 7,5 ; 100 mM d’acétate de magnésium ; 1,25 M de glutamate de potassium ; 1 mM d’EDTA ; 0,025 % de Tween-20). Filtrez le tampon à l’aide d’un filtre à seringue de 0,22 μm et stockez-le à 4 °C.
- Dégazez environ 2,5 mL de tampon de blocage et 5 mL d’eau ultrapure en plaçant les tubes ouverts dans un dessiccateur et en les laissant sous vide pendant 15 min.
REMARQUE : Ne remplissez pas les tubes à ras bord, car les bulles pendant le dégazage peuvent provoquer des éclaboussures de liquide. - Placez la cellule d’écoulement sur une platine de microscope et fixez-la en place à l’aide d’un mastic adhésif à chaque extrémité. Il est important de bien sécuriser pour éviter que la cellule d’écoulement ne se déplace plus tard lors de l’acquisition.
- Connectez le tube de sortie de la cellule d’écoulement à la pompe à seringue, qui a un tube fixé à la seringue avec une aiguille à l’autre extrémité. Insérez cette aiguille dans le tube de sortie pour connecter la cellule d’écoulement.
- Allumez le chauffage de l’objectif à 30 °C.
- Fixez 1 à 2 ml d’eau dégazée dans un tube sur un morceau de mastic adhésif séparé près de la cellule d’écoulement afin que le tube d’entrée puisse être inséré et atteindre le fond du tube.
- Faites couler de l’eau dans le canal, puis utilisez un débit plus rapide pour éliminer les bulles piégées près du tube d’entrée, en ajoutant plus d’eau si nécessaire. Sinon, de grosses bulles risquent de se déloger plus tard dans les expériences et de passer dans le canal. Laissez agir quelques minutes pour que le débit s’arrête complètement, car le débit rapide peut provoquer une traction résiduelle du liquide pendant un certain temps après son arrêt, à mesure que la pression à l’intérieur de la seringue se stabilise.
REMARQUE : Tout ADN qui entre en contact avec une bulle devient inutilisable, même une fois réhydraté. - Ajouter 100 μL de tampon de blocage dégazé à une aliquote de 20 μL de 1 mg/mL de streptavidine. Fixez le tube ouvert au mastic adhésif et transférez le tube d’entrée de l’eau à la streptavidine. Débit à un débit de 40 μL/min pendant 2 min (80 μL au total) et incubation pendant 5 min.
- Éliminer l’excès de streptavidine à l’aide du tampon de blocage (50 μL/min pour 100 μL).
- Écoulement dans l’ADN biotinylé, dilué dans un tampon bloquant avec 25 nM SYTOX orange. Image en direct avec le laser de 532 nm pour observer l’ADN se connecter à la surface en temps réel.
- Lorsque la densité approximative de l’ADN à la surface est atteinte, écoulez dans un tampon de blocage, également avec 25 nM de SYTOX (50 μL/min pour 100 μL), pour éliminer l’ADN libre.
- Écoulement dans l’anticorps anti-digoxigénine biotinylé (~10 μg/mL) dilué dans un tampon bloquant contenant 25 nM d’orange SYTOX (100 μL/min pour 300 μL).
REMARQUE : L’orange SYTOX s’intercale entre les paires de bases de l’ADN, allongeant ainsi la longueur du contour. Cela signifie que la longueur de bout en bout de l’ADN augmente dans SYTOX orange, ce qui permet à la deuxième attache de se fixer à la surface plus loin de la première, ce qui donne un ADN mieux étiré lorsque l’orange SYTOX est lavé plus tard. L’utilisation d’une fraction différente à chaque extrémité de l’ADN permet une liaison plus uniforme de l’ADN à la surface de la lamelle (figure 4). Lorsque les deux extrémités sont biotinylées, elles s’attachent dans la même étape, ce qui entraîne une variation significative de leurs positions d’attache. L’utilisation de la digoxigénine à une extrémité donne plus de contrôle sur la couverture de l’ADN avant d’attacher la deuxième extrémité. Cela permet également d’augmenter la couverture de l’ADN par l’incubation. - Laver l’anticorps anti-digoxigénine biotinylé et SYTOX orange avec tampon bloquant (50 μL/min pour 100 μL).
- Préparer 120 μL de « mélange ATP-g-s » (1x tampon de réaction 1, 0,75 mg/mL BSA, 1,25 mg/mL de caséine, 8 mM DTT, 0,33 mM d’ATP-g-s), ajouter 30 μL dans un nouveau tube, puis faire couler 50 μL du tube d’origine dans la cellule d’écoulement (50 μL/min pour 50 μL). Cela permet de minimiser l’effet du mélange tampon par refoulement ou diffusion à partir du tube de sortie.
- Ajouter la CMG purifiée à ~100 nM final dans 30 μL de mélange ATP-g-s, puis laisser couler à 20 μL/min pendant 20 μL. Incuber pendant 15 min.
- Préparer 120 μL de « mélange ATP/RPA » (1x tampon de réaction 1, 0,75 mg/mL BSA, 8 mM DTT, 3,3 mM d’ATP, 20 nM EGFP-hRPA), et s’écouler dans la cellule d’écoulement à 40 μL/min pendant 80 μL.
- Commencez immédiatement à acquérir des images. Acquérez des champs de vision de 6 x 6 pour chaque image toutes les 30 s (ou une fréquence d’images appropriée pour l’expérience). Des fréquences d’images plus faibles peuvent réduire l’exposition au laser et le photoblanchiment au fluorophore. Visualisez EGFP-hRPA avec un laser de 488 nm à 1 % de puissance. Si CMG est étiqueté, par exemple LD655, visualisez CMG avec un laser de 640 nm (puissance maximale = 30 mW) à 10 % de puissance. Visualisez l’ADN coloré en orange de SYTOX avec un laser de 532 nm à 0,5% de puissance. Utilisez chaque laser avec une exposition de 50 à 100 ms.
REMARQUE : Chaque fois que le tube d’entrée de la cellule d’écoulement est transféré d’un tube de liquide à un autre, tamponnez plusieurs fois l’extrémité du tube au fond du tube avant de le retirer du liquide. Cela permet d’éviter que des bulles ne pénètrent pendant le transfert. Minimisez le temps pendant lequel l’extrémité du tube d’entrée est maintenue hors du liquide, et ne pointez pas l’extrémité vers le haut ou ne soulevez pas l’extrémité de manière significative, car cela provoque l’écoulement du liquide vers le canal et aspire de l’air dans l’embout. Lors du transfert du tube d’entrée, essayez de ne pas mettre de tension sur le tube car cela peut le faire sortir de l’époxy, le fixant à la cellule d’écoulement. Pour le déroulement de l’ADN d’imagerie, en présence de SYTOX orange pour visualiser l’ADN double brin, remplacez 10x tampon de réaction 1 par 10x tampon de réaction 2 pour le mélange ATP/RPA. L’orange SYTOX se lie mieux à l’ADN dans ces conditions. Videz complètement le pousse-seringue à la fin de chaque expérience. Lavez de temps en temps en remplissant d’eau et en vidant plusieurs fois ou en démontant et en lavant à l’eau savonneuse.

Figure 4 : L’ADN se rattache à la surface. (A) Lors de l’attache de substrats d’ADN avec de la biotine aux deux extrémités, la distance entre les deux attaches peut varier en fonction de la façon dont les extrémités entrent en contact avec la surface (i). En utilisant la digoxigénine à une extrémité, l’attache de chaque extrémité peut être séparée temporellement pour des distances d’attache plus cohérentes et un ADN plus uniformément étiré (ii). (B) Exemple de champ de vision montrant de l’ADN attaché aux deux extrémités (marqué à la digoxigénine) et coloré par une coloration intercalaire fluorescente d’acide nucléique. L’ADN, qui est attaché aux deux extrémités, apparaît sous la forme d’une ligne, tandis que l’ADN attaché à une seule extrémité apparaît sous forme de taches. Idéalement, l’ADN devrait être attaché aussi densément que possible sans chevaucher d’autres ADN. L’image fait 512 x 512 pixels (taille de pixel = 154,6 nm). Veuillez cliquer ici pour voir une version agrandie de cette figure.
Résultats
Lorsque CMG déroule l’ADN, un tractus RPA caractéristique se développe au fil du temps (Figure 5). L’extrémité 5' de l’ADN déroulé est attachée à la surface ; par conséquent, il est considéré comme un étirement linéaire du signal RPA entre la sangle et la fourche. L’extrémité 3' n’est pas attachée et, par conséquent, se déplace avec la fourche et est observée comme un signal EGFP-RPA compact. La position du brin de translocation compacté et déroulé correspond approximativement à la position de la fourche de réplication, qui se déplace avec la LD655-CMG visualisée via un laser de 640 nm.
Il est important de minimiser les dommages au substrat de l’ADN, car les dommages tels que les entailles d’ADN simple brin réduisent le nombre d’événements de déroulement observables, limitant ainsi la quantité de données pouvant être collectées (Figure 6).
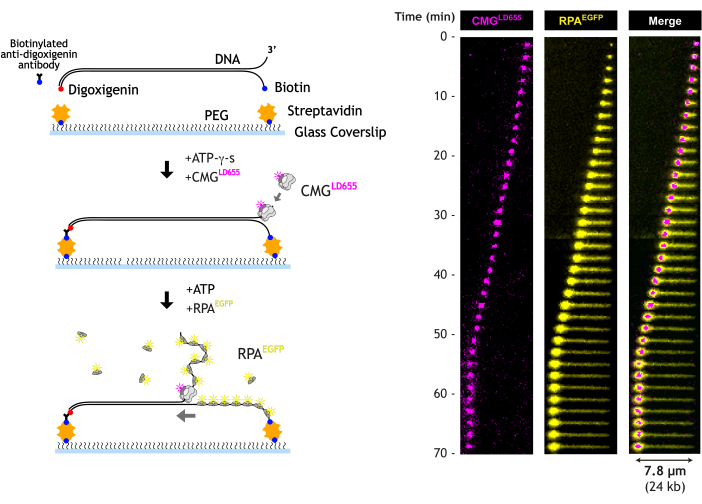
Figure 5 : Essai de déroulement de l’ADN d’une seule molécule. Le substrat d’ADN est attaché à une surface de lamelle. La CMG purifiée marquée au LD655 est incubée avec l’ADN pendant 15 min dans de l’ATP-g-s. De l’ATP et du RPA purifié marqué à l’EGFP sont ajoutés, initiant un déroulement étendu de l’ADN par CMG. Un schéma de dessin animé (à gauche) et un kymographe de données représentatives (à droite) sont montrés. Veuillez cliquer ici pour voir une version agrandie de cette figure.
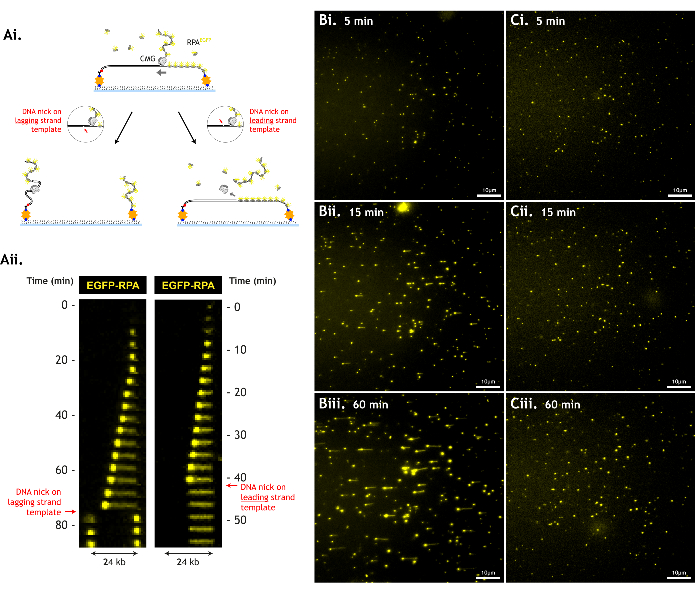
Figure 6 : Les dommages à l’ADN réduisent le débit du test. (A) CMG ne peut pas dérouler l’ADN au-delà d’une rupture dans le squelette de l’ADN (entaille d’ADN). Une entaille sur le modèle du brin principal fait glisser la passerelle de gestion de domaine hors de l’ADN, et la matrice de la passerelle de gestion de la chaîne et la matrice de brin principal sont perdues. Une entaille sur le modèle du brin retardé provoque la séparation du modèle du reste de l’ADN, et chaque morceau d’ADN se rétracte vers son attache respective. Ceci est illustré par (i) des schémas de dessins animés et (ii) des kymographes de ces événements (ii). Données représentatives avec (B) un substrat d’ADN peu endommagé par rapport à (C) un substrat d’ADN plus endommagé à (i) 5 min, (ii) 15 min et (iii) 60 min dans un seul champ de vision. Le substrat d’ADN le plus endommagé ne génère pas de longues périodes de déroulement, car les CMG rencontrent des entailles plus tôt, malgré des niveaux similaires d’activité de déroulement (densité similaire de points de croissance RPA à 5 min, indiquant une efficacité de chargement/déroulement CMG similaire). Le champ de vision est de 512 x 512 pixels (taille de pixel = 154,6 nm). Imagerie EGFP-RPA de puissance laser de 1 % (488 nm). Barre d’échelle montrant 10 μm. Veuillez cliquer ici pour voir une version plus grande de cette figure.
Discussion
Ce test fournit une plate-forme pour observer et étudier la dynamique en temps réel des CMG individuels, à la fois de manière isolée et dans le contexte des facteurs supplémentaires souhaités. Cependant, comme pour de nombreuses techniques de fluorescence à molécule unique, il existe des défis courants qui peuvent nécessiter une optimisation. Ceux-ci concernent généralement l’imagerie des fluorophores sur de longues périodes (photoblanchiment, luminosité), la préparation du substrat de l’ADN (dommages à l’ADN), la qualité de la surface de la cellule d’écoulement (bruit de fond, interactions non spécifiques) ou la qualité de la préparation des protéines purifiées (contamination par les nucléases, efficacité du marquage).
Chaque fluorophore varie en termes de photostabilité et de luminosité, il est donc important de choisir une molécule appropriée. Lors de l’imagerie de protéines oligomères marquées par fluorescence, comme la RPA, une puissance laser plus faible peut être utilisée car de nombreux fluorophores seront excités à proximité, générant un signal visible. Pour l’imagerie de fluorophores uniques, par exemple, CMG marqués sur une seule sous-unité, une puissance laser plus élevée est nécessaire pour observer clairement le fluorophore. La durée de vie des fluorophores peut être prolongée en minimisant l’exposition au laser, par exemple en réduisant la fréquence à laquelle les images sont prises. De plus, l’excitation d’un fluorophore génère des espèces réactives de l’oxygène (ROS), qui peuvent contribuer au photoblanchiment. L’inclusion d’un système de piégeage de l’oxygène dans le tampon d’imagerie peut prolonger la durée de vie des fluorophores en éliminant les ROS. Cependant, certains systèmes de piégeage de l’oxygène peuvent affecter le pH12.
En ce qui concerne la préparation du substrat d’ADN, il est crucial de minimiser les dommages à l’ADN, tels que les entailles ou les espaces monocaténaires. Des dommages excessifs empêchent un déroulement important de l’ADN, ce qui limite la quantité de données pouvant être collectées. Les dommages peuvent résulter d’un cisaillement mécanique, d’un échauffement excessif, d’une contamination par des nucléases ou de ROS générés pendant l’imagerie. Le cisaillement peut être minimisé en manipulant l’échantillon d’ADN avec soin en utilisant des pointes à large alésage pour le pipetage, en pipetant lentement et en évitant de tordre l’échantillon. L’effet des ROS peut être minimisé en réduisant l’exposition au laser ou en incluant un système de récupération de l’oxygène dans le tampon d’imagerie. Après la préparation du substrat d’ADN, il est possible d’utiliser des kits de réparation d’ADN commerciaux pour réparer les dommages avant d’effectuer une réaction de déroulement.
L’efficacité du déroulement de l’ADN dépend également de la pureté et de l’activité de la CMG. Il est recommandé d’évaluer la pureté de l’échantillon après chaque étape de purification par électrophorèse SDS-PAGE afin de déterminer où l’optimisation est nécessaire. Si trop de contaminants sont observés après l’étape finale, il peut être utile de modifier les volumes de gradient de sel utilisés pour l’élution de la colonne CaptoHiRes Q (5/50). Il est également très important d’éliminer tout excès de peptide fluorescent utilisé pour le marquage des protéines, car il peut créer un arrière-plan indésirable sur la surface de la lamelle. Il est également essentiel d’éviter la contamination par les nucléases, car cela peut dégrader le substrat de l’ADN. Après une expérience, la coloration de l’ADN restant avec de l’orange SYTOX peut être un bon moyen de vérifier si l’ADN a été dégradé de manière significative ou non. Un certain niveau de dommages à l’ADN est inévitable au cours d’une expérience, mais des dommages importants indiquent souvent une contamination problématique par les nucléases.
Le test est également intrinsèquement limité par la résolution des taches limitées par la diffraction, ce qui nécessite que les protéines fluorescentes soient à des centaines de paires de bases (sinon plus) pour les distinguer comme séparées. Cela limite les détails dans lesquels la progression et les interactions de la CMG peuvent être observées.
Le nombre d’événements de dénouement que nous observons pour chaque analyse varie. Pour une expérience réussie, nous nous attendons à voir au moins plusieurs faisceaux RPA de longueur suffisante par champ de vision de 512x512 pixels (taille de pixel = 154,6 nm). Plusieurs champs de vision peuvent être imagés dans la même expérience, ce qui permet de collecter davantage de données si nécessaire. Les tracts n’ont pas besoin d’être de la même longueur ni d’atteindre la fin de l’ADN pour être utiles. Par exemple, la distance moyenne d’attache pour chaque expérience peut être déterminée en mesurant la longueur de l’ADN coloré par SYTOX avant d’ajouter la CMG. Cela peut être utilisé pour estimer la quantité d’ADN qui a été déroulée pour n’importe quel tractus RPA (tant qu’il y a suffisamment d’ADN déroulé pour déplacer visiblement la fourche) en convertissant la distance de « μm parcouru » en « kb déroulé ».
CMG présente une activité de déroulement sur une variété de substrats d’ADN, mais il est essentiel de fournir une extrémité d’ADN 3' libre sur un lambeau polyT d’au moins 30 nt pour s’adapter à l’empreinte de CMG10. L’inclusion de plusieurs fractions de biotine à la fourche assure une attache de surface robuste. Le reste du substrat d’ADN peut être redessiné de multiples façons, par exemple pour inclure différentes séquences d’ADN, longueurs et modifications chimiques. La conformation de l’ADN peut être modifiée en utilisant différentes concentrations d’acétate de magnésium. À des concentrations plus élevées (≥10 mM) d’acétate de magnésium, le filament d’ADNsb enrobé de RPA est compacté, ce qui permet à l’ADN d’être tiré par la liaison RPA pendant le déroulement. Cela peut être utile car cela empêche l’ADN de bouger excessivement, ce qui permet de mesurer plus précisément la position de la CMG et la progression du déroulement. À de faibles concentrations (~3 mM) d’acétate de magnésium, l’ADNs-RPA reste détendu tout au long de la période.
Le test à molécule unique décrit représente une plate-forme qui peut être construite et modifiée pour étudier d’autres aspects de la réplication de l’ADN. Lors de la réplication de l’ADN, la CMG agit comme un noyau autour duquel le réplisome et ses composants s’assemblent. Par conséquent, des protéines purifiées supplémentaires peuvent être ajoutées à ce test, y compris des facteurs accessoires tels que TIMELESS, TIPIN et CLASPIN, pour étudier leur effet sur la dynamique de la CMG. Il a été démontré que ces protéines affectent le taux de fourchesde réplication 13, mais il n’est pas clair comment elles affectent le taux de déroulement de la CMG. Par conséquent, il serait intéressant d’étudier comment différentes protéines réplisomes affectent la CMG à l’aide de ce test. L’ajout d’ADN polymérases peut donner un meilleur aperçu de la réplication de l’ADN au-delà du seul déroulement de l’ADN, comme décrit précédemment avec les protéines de levure14. De plus, la purification de CMG modifiés peut permettre de mieux comprendre comment certaines mutations ou modifications post-traductionnelles affectent l’activité de l’hélicase15,16. De plus, la conception de différents substrats d’ADN peut permettre d’étudier le déroulement de l’ADN par CMG dans diverses conditions imitant le stress de réplication17. Ces modifications comprennent les obstaclesde l’ADN 9,18, les réticulations interbrins 19,20,2 1 et les discontinuités dans les brins d’ADN22.
Déclarations de divulgation
Les auteurs n’ont pas d’intérêts financiers concurrents ou d’autres conflits d’intérêts.
Remerciements
Nous remercions Gheorghe Chistol pour la fourniture du plasmide pGC261 et l’installation de biologie chimique de l’Institut Francis Crick pour la synthèse et le marquage des peptides. Ce travail a été financé par l’Institut Francis Crick, qui reçoit un financement de base de Cancer Research UK, du Conseil de la recherche médicale du Royaume-Uni et du Wellcome Trust (CC2133).
matériels
| Name | Company | Catalog Number | Comments |
| 2-Mercaptoethanol | Sigma-Aldrich | M3148-25ML | |
| Acrylamide / Bis Solution 40% | BioRad | 1610148 | |
| Agarose UltraPure | Invitrogen | 16500-500 | Used to prepare Tris Buffer (pH 8) |
| ÄKTA Pure | GE Healthcare | - | Used with Fraction Collector F9-C; protein purification system |
| ANTI-FLAG M2 affinity gel | Sigma-Aldrich | A2220 | |
| APS 10% | Sigma-Aldrich | A3678-25G | |
| ATP (200 mM) | Sigma-Aldrich | A7699-5G | |
| ATP-g-s (100 mM) | Sigma-Aldrich | 11162306001 | |
| Biotin-PEG coverslip | N/A | N/A | Prepared as described: https://doi.org/10.1016/j.ymeth.2012.03.033 |
| Biotinylated anti-digoxigenin antibody | Perkin Elmer | N/A | |
| Blu Tack | Bostik | N/A | Adhesive putty |
| Boric acid | Thermo Fisher Scientific | B/3800/53 | |
| BSA; 33 mg/mL | SIGMA-ALDRICH | A3858-10G | Diluted from the stock |
| CaCl2 | Sigma-Aldrich | C7902 | |
| CaptoHiRes Q (5/50) | Cytiva | 29275878 | Connected to the AKTA protein purification system; high-resolution ion exchange chromatography column |
| Casein (5% in water, 50 mg/mL) | Sigma-Aldrich | C4765-10ML | |
| ChemiDoc system | Bio-Rad | N/A | |
| Clips | N/A | N/A | |
| Cutting board | N/A | N/A | |
| Dialysis tubing (3.5 kDa) | Fisher Scientific | 11425859 | |
| digoxigenin-11-dUTP | Roche | 11209256910 | |
| DNA loading dye 6x | NEB | B7024S | |
| dNTP 10 mM | NEB | N0447S | |
| Double-sided tape (0.14 mm thick) | N/A | N/A | |
| DTT | Fluorochem Limited | M02712-10G | |
| DYKDDDDK peptide | N/A | N/A | Synthetised by chemical biology STP of The Francis Crick Institute |
| EDTA | Thermo Fisher Scientific | D/0700/68 | |
| EGFP-RPA | N/A | N/A | Purified as desribed in ‘Single Molecule Analysis’, Series Methods in Molecular Biology, Vol. 783 (2011), Peterman, E. J. G. and Wuite G. J. L. editors, Humana Press. Protein information: 2 mM, human, expressed in E. coli. |
| EGTA | Sigma-Aldrich | 03779-10G | |
| Epoxy - 5 minute | Devcon | 20845 | |
| Fujifilm FLA-5000 | Fujifilm | FLA-5000 | Fluorescent image analyzer |
| GelRed Nucleic Acid Stain; 10000x | Cambridge Bioscience | 41003-BT | Example of nucleic acid stain which is a safer alternative to ethidium bromide. |
| Glass or quartz slide with holes for tubing | Dremel Model 395 | 1 mm thick slide, cut to 2.4 cm x 1cm, two holes drilled 1.4 mm apart. The holes should be drilled to different sizes to accommodate different tubing at each end. | |
| Glycerol | Fisher Scientific | G/0650/17 | |
| Glycine HCl, pH 3.5 | Sigma-Aldrich | G8898 | |
| Hamilton syringe, 1000 series GASTIGHT, PTFE luer lock 1010TLL, PTFE Luer lock (with slots), volume 10 mL | Merck | 26211-U | |
| HEPES pH 7.5 | Sigma-Aldrich | H4034 | |
| I-CeuI | NEB | R0699L | |
| KCl | Fisher Scientific | P/4280/53 | |
| Magnesium acetate | Sigma-Aldrich | M2545 | |
| Maxi GeBaFlex-tube Dialysis Kit; MWCO 14 kDa | Generon | D055 | |
| Metal spatula | N/A | N/A | |
| Metal tweezers | N/A | N/A | |
| MgCl2 | Sigma-Aldrich | M2393 | |
| Millex-GP (Syringe filters) 0.22 µm | Merck | SLGP033RS | |
| NaCl | Sigma-Aldrich | S7653 | |
| Needle | N/A | N/A | |
| NotI-HF | NEB | R3189L | |
| NuPAGE 4%–12% Bis-Tris Protein Gels | Thermo Fisher Scientific | 10247002; | |
| NuPAGE MOPS SDS Running Buffer | Thermo Fisher Scientific | NP0001 | |
| Objective heater | Okolab | N/A | |
| PCR mix - 2x | Prepared from Phusion DNA polymerase (20 µL), 10 mM dNTPs (40 µL), 5x Phusion HF buffer (400 µL) and water (540 µL). | ||
| Peptide NH2-CHHHHHHHHHHLPETGG-COOH, labelled with LD655-MAL | N/A | N/A | peptide NH2-CHHHHHHHHHHLPETGG-COOH, labeled with LD655-MAL (Lumidyne Technologies) on the cysteine residue, was synthetised and purified by the peptide chemistry STP of The Francis Crick Institute |
| pGC261 plasmid | N/A | N/A | https://doi.org/10.1016/j.cell.2018.10.053 |
| Phusion High-Fidelity DNA Polymerase | NEB | M0530L | |
| Plastic tweezers | N/A | N/A | |
| Polyethylene tubing PE20 (inner diameter 0.015”, outer diameter 0.043”) | Becton Dickinson | 427406 | |
| Polyethylene tubing PE60 (inner diameter 0.03”, outer diameter 0.048”) | Becton Dickinson | 427416 | |
| Poly-Prep Chromatography Columns; 10 ml and 20 ml | Bio-Rad | 7311550 | |
| Potassium Glutamate (L-glutamic acid potassium salt monohydrate) | Sigma | G1501-1KG | |
| Protease inhibitor cocktail (cOmplete, EDTA free) | Roche | 5056489001 | |
| Pump 11 Elite Infusion/Withdrawal Programmable Single Syringe | Harvard Apparatus | 70-4504 | |
| QIAquick PCR Purification Kit | QIAGEN | 28104 | |
| Razor blade | VWR International Ltd | 233-0156 | |
| rCutSmart Buffer; 10x | NEB | B6004S | |
| Sodium acetate | Thermo Fisher Scientific | S/2120/53 | |
| Sortase (pentamutant) | N/A | N/A | Purified based on: https://doi.org/10.1073/pnas.1101046108 |
| Spin-X Centrifuge Tube Filters; 0.22 µm cellulose acetate | Fisher Scientific | 10310361 | |
| Sterile scalpel | N/A | N/A | |
| Steritop Vacuum Driven Disposable Filtration System; 0.22 um; PES; | Millipore | S2GPT05RE | |
| Streptavidin (1 mg/mL) in 1x PBS buffer | Sigma | S4762-10MG | 20 µL aliquotes |
| SYBR Gold | Thermo Fisher Scientific | S11494 | Highly sensitive nucleic acid stain we used to visualise DNA fork substrate. |
| SYTOX orange; 5 µM in DMSO | Thermo Fisher Scientific | S11368 | |
| T4 DNA ligase | NEB | M0202M | |
| T4 DNA Ligase Reaction Buffer | NEB | B0202S | |
| TEMED | Sigma-Aldrich | T9281-25ML | |
| TEV protease (1 mg/mL); EZCut | Biovision | 7847-10000 | |
| Tissue grinders, Dounce type (40 mL, Wheaton) | DWK Life Sciences | 432-1273 | |
| Tris base | Sigma-Aldrich | T1503 | Used to prepare Tris Buffer (pH 8) |
| Tris HCl | Sigma-Aldrich | T3253 | |
| Tween-20 | Promega UK Ltd | H5152 |
Références
- Bell, S. P., Labib, K. Chromosome Duplication in Saccharomyces cerevisiae. Genetics. 203 (3), 1027-1067 (2016).
- Costa, A., et al. The structural basis for MCM2-7 helicase activation by GINS and Cdc45. Nat Struct Mol Biol. 18 (4), 471-477 (2011).
- Jones, M. L., Baris, Y., Taylor, M. R. G., Yeeles, J. T. P. Structure of a human replisome shows the organisation and interactions of a DNA replication machine. EMBO J. 40 (23), e108819(2021).
- Moyer, S. E., Lewis, P. W., Botchan, M. R. Isolation of the Cdc45/Mcm2-7/GINS (CMG) complex, a candidate for the eukaryotic DNA replication fork helicase. Proc Natl Acad Sci U S A. 103 (27), 10236-10241 (2006).
- Douglas, M. E., Ali, F. A., Costa, A., Diffley, J. F. X. The mechanism of eukaryotic CMG helicase activation. Nature. 555 (7695), 265-268 (2018).
- Ilves, I., Petojevic, T., Pesavento, J. J., Botchan, M. R. Activation of the MCM2-7 helicase by association with Cdc45 and GINS proteins. Mol Cell. 37 (2), 247-258 (2010).
- Dewar, J. M., Walter, J. C. Mechanisms of DNA replication termination. Nat Rev Mol Cell Biol. 18 (8), 507-516 (2017).
- Burnham, D. R., Kose, H. B., Hoyle, R. B., Yardimci, H. The mechanism of DNA unwinding by the eukaryotic replicative helicase. Nat Commun. 10 (1), 2159(2019).
- Kose, H. B., Larsen, N. B., Duxin, J. P., Yardimci, H. Dynamics of the eukaryotic replicative helicase at lagging-strand protein barriers support the steric exclusion model. Cell Rep. 26 (8), 2113-2125.e6 (2019).
- Kose, H. B., Xie, S., Cameron, G., Strycharska, M. S., Yardimci, H. Duplex DNA engagement and RPA oppositely regulate the DNA-unwinding rate of CMG helicase. Nat Commun. 11 (1), 3713(2020).
- Tanner, N. A., Van Oijen, A. M. Visualizing DNA replication at the single-molecule level. Methods Enzymol. 475, 259-278 (2010).
- Swoboda, M., et al. Enzymatic oxygen scavenging for photostability without ph drop in single-molecule experiments. ACS Nano. 6 (7), 6364-6369 (2012).
- Baris, Y., Taylor, M. R. G., Aria, V., Yeeles, J. T. P. Fast and efficient DNA replication with purified human proteins. Nature. 606, 204-210 (2022).
- Lewis, J. S., et al. Tunability of DNA polymerase stability during eukaryotic DNA replication. Mol Cell. 77 (1), 17-25.e5 (2020).
- Li, Z., Xu, X. Post-translational modifications of the mini-chromosome maintenance proteins in DNA replication. Genes. 10 (5), 331(2019).
- Lewis, J. S., et al. Mechanism of replication origin melting nucleated by CMG helicase assembly. Nature. 606 (7916), 1007-1014 (2022).
- Zeman, M. K., Cimprich, K. A. Causes and consequences of replication stress. Nat Cell Biol. 16 (1), 2-9 (2014).
- Sparks, J. L., et al. The CMG helicase bypasses DNA-protein cross-links to facilitate their repair. Cell. 176 (1-2), 167-181.e21 (2019).
- Taylor, M. R. G., Yeeles, J. T. P. The initial response of a eukaryotic replisome to DNA damage. Mol Cell. 70 (6), 1067-1080.e12 (2018).
- Amunugama, R., et al. Replication fork reversal during DNA interstrand crosslink repair requires CMG unloading. Cell Rep. 23 (12), 3419-3428 (2018).
- Wu, R. A., et al. TRAIP is a master regulator of DNA interstrand crosslink repair. Nature. 567 (7747), 267-272 (2019).
- Vrtis, K. B., Dewar, J. M., Chistol, G., Wu, R. A., Graham, T. G. W., Walter, J. C. Single-strand DNA breaks cause replisome disassembly. Mol Cell. 81 (6), 1309-1318.e6 (2021).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon