Method Article
用于神经解剖学分析的全 Marmoset 大脑的连续双光子断层扫描
摘要
连续双光子断层扫描 (STPT) 成像是一种通过将双光子成像与自动载物台控制和切片机切片相结合,以三维形状对组织块进行成像的技术。在这里,我们描述了一种为狨猴大脑实施它的协议,以更好地了解其结构特征。
摘要
连续双光子断层扫描 (STPT) 是一种通过将双光子成像与自动载物台控制和切片机切片相结合,以三维形状对组织块进行成像的技术。我们成功地实施了这种技术来追踪狨猴大脑中的轴突投射。在这里,描述了导致整个狨猴大脑可靠体积成像的详细实验程序。成功成像的一个关键过程是去除大脑周围的脑膜,这会干扰切片。这种方法的一大优点是切片切片可用于额外的染色。在原始设置中,切片部分在水浴中打乱。这些部分可以根据皮层中的血管模式按其原始顺序正确对齐。有效组织学的一个例子是通过简单的光反射来可视化髓鞘结构,它可以与尼氏染色相结合来定义解剖边界。这些切片还可用于非荧光顺行和逆行示踪剂的免疫学检测,这些示踪剂可以注册到 STPT 数据中,以便对多个数据进行分层。
引言
在神经解剖学研究中,研究人员需要在整个大脑的背景下观察微米级结构(例如,轴突和钮扣)。这项艰巨的任务通常是通过目视检查连续切片并寻找感兴趣区域以进行详细成像、分析和照片记录来完成的。然而,随着技术的进步,以高分辨率对整个大脑进行成像以进行全脑分析成为可能。在 Oh 等人的一项开创性研究中1,数百只小鼠大脑接受了示踪剂注射用于连接组学分析,这些显踪剂通过连续双光子断层扫描 (STPT) 成像技术进行处理2。这项研究的特点是他们选择了顺行示踪剂来量化 "连通性"。虽然顺行示踪剂提供了关于轴突分布的非常详细的空间信息,但神经解剖学家一直依赖费力的手动分割进行分析。通过自动化各种分析程序,包括这个分割步骤,他们成功地"大规模生产"了用于多用途的即用型高分辨率示踪剂数据。鉴于使用他们的示踪数据 3,4,5 和他们的标准大脑6 的各种研究,他们的方法的有效性是显而易见的。
从历史上看,自 1950 年代退化方法发展以来,非人灵长类动物大脑的神经连接引起了许多神经解剖学家的注意,到 1970 年代的顺行/逆行物质运输方法,再到现在的病毒策略 7,8。因此,存在大量研究灵长类动物神经连接的文献。特别是,许多研究人员研究了猕猴大脑复杂的皮质连接,他们的结果已被整理成表格(例如,CoCoMac9)。尽管这些经典研究有用,但存在一些局限性。首先,由于每项研究只关注有限的大脑区域,因此获得的信息不可避免地变得零碎。其次,每项研究使用不同的方法和条件。因此,跨研究的定量评估变得复杂。第三,连接性通常显示为代表性切片的相机清晰度或作者定义的大脑区域的半定量表格/图表。换句话说,从复杂的大脑结构中只提取非常有限的信息,以便在已发表的文献中呈现。随着磁共振成像 (MRI) 技术的发展,低分辨率的全脑研究变得突出。然而,我们对小鼠神经连接和灵长类动物神经连接的了解之间存在很大的细节水平差距。
考虑到这样的背景,我们着手对常见的狨猴大脑进行全面的顺行追踪10,11。虽然狨猴的大脑比猕猴的大脑小得多、光滑得多,但它表现出灵长类动物的明显迹象,例如存在区域 MT 和颗粒状前额叶区域,这在啮齿动物中都没有明确定义12,13。在这里,小尺寸是一个很大的优势,因为即使是狨猴的大脑也比老鼠的大脑重十倍。幸运的是,我们可以通过对原始版本的 TissueCyte1000(以下简称全组织成像系统)作软件(用于 STPT 成像2 的市售显微镜)进行最少的更新来对整个狨猴大脑进行成像。此更新是为了在切片之前允许舞台的额外移动。当前版本现在足以处理狨猴的大脑。本文分享了处理狨猴大脑进行 STPT 成像的协议。还提供了进一步增强该方法实用性的成像后协议。
研究方案
所有实验程序均按照 1996 年修订的美国国立卫生研究院实验动物护理和使用指南(NIH 出版物第 80-23 号)和日本生理学会的"生理科学领域动物护理和使用指导原则"进行,并得到理化学研究所实验动物委员会的批准 (W2020-2-009(2))。
1. 示踪剂注射
- 根据先前报告的程序向狨猴大脑注射荧光和非荧光示踪剂14.
- 关于与 STPT 方法的组合,确保示踪剂的荧光足够强,无需增强即可检测到。采用双载体 TET-Off 系统10 以增强表达。
注:在完成 STPT 后,也可以使用产生的切片注入和检测非荧光示踪剂。非荧光示踪剂的实例包括生物素化葡聚糖胺 (BDA)、在 AAV2 retro 中表达 cre 的 AAV,用于逆行检测输入细胞核10,以及基于 GFP 的 smFP 标签 AAV(见下文)。
- 关于与 STPT 方法的组合,确保示踪剂的荧光足够强,无需增强即可检测到。采用双载体 TET-Off 系统10 以增强表达。
- 灌注固定并在 4 周后获得狨猴脑。
- 为了麻醉狨猴,肌肉注射美托咪定 (0.04 mg/kg)、咪达唑仑 (0.4 mg/kg) 和布托啡诺 (0.4 mg/kg),称为 MMB,然后腹膜内注射硫喷妥钠 (100 mg/kg)。
- 确认痛反射消失后,切开胸腔露出心脏,切开右心房,让血液和固定液流出。
- 然后切开左心室以插入灌注针,首先用前缀溶液(250 mM 蔗糖、5 mM MgCl2 在 0.02 M 磷酸盐缓冲液 [PB,pH 7.4] 中)冲洗血液几分钟(~50-100 mL),然后用 4% 多聚甲醛/0.1 M 磷酸盐缓冲液(PB;在 ~20 分钟内为动物体重的 2-3 倍)固定。
- 良好的灌注对于获得良好的结果至关重要。插入灌注针时,将其沿隔膜推向主动脉,以确保固定剂的良好流动。当针尖到达主动脉时变得可见。缩回直到尖端几乎不可见,这样它就不会绕过颈动脉。
- 将大脑在 4% 多聚甲醛/0.1 M PB 中在 4 °C 下保持 48 小时,然后转移至 50 mM PB。如果不立即使用,请将大脑储存在 0.75% 甘氨酸/0.1 M PB 中,以防止由于过度固定而引起的自发荧光。
2. 样品制备(图 1)
- 将固定的大脑与胶原酶(1 mg/mL在3 mM CaCl 2中,2 在10 mL tris缓冲盐水[TBS]中)在37°C下孵育1小时。
注意:孵育前将大脑在 37 °C 下预热 5 分钟。 - 用棉签小心地摩擦大脑表面,以剥落软脑膜和其他脑膜(图 1A, 补充视频)。用细镊子剥开脱落的脑膜。
注意:去除中脑周围的脑膜很重要,包括上丘或丘脑顶部的脑膜。很难揭露这些隐藏在内心深处的结构。但是,请尽可能去除脑膜。否则,它们通常保持未切割、漂浮并干扰成像和切片(图 2A)。 - 在嵌入前对大脑进行 离体 MRI 时要格外小心,在此期间大脑浸入氟溶液中 15。如果将剩余的氟嵌入在一起,则在成像时可能会在物镜下方形成微小的气泡。为避免这种情况,在嵌入前将大脑保持在 PB 中 1 周。
注意:这个等待步骤通常不是必需的,但在与 离体 MRI 结合使用时至关重要。 - 通过将 0.2 g NaBH4 溶解在 100 mL 加热至 40°C 的 50 mM 硼酸盐缓冲液 (pH 9.2) 中来制备 NaBH4 缓冲液。 松开盖子,让 CO2 气体排出。将溶液放松盖子过夜。用铝箔包裹瓶子以保护其避光,直到使用。
- 通过在 100 mL PB 中搅拌 2.25 g 琼脂糖和 0.21 g NaIO4 制备氧化琼脂糖 2-3 小时。用真空抽吸过滤溶液,并用 PB 的 3 次更换洗涤。将琼脂糖重悬于 50 mL PB 中。
- 在微波炉中完全熔化琼脂糖并冷却至 60-65 °C。
- 将大脑放入定制的腔室中,并将大脑嵌入琼脂糖中。小心将琼脂糖引入胼胝体下方的空腔中(图 1B)。
注意:首先在室温下预热大脑,然后在 65 °C 下预热 5 分钟,以使琼脂糖沉淀在大脑表面而不会凝固。 - 拆卸腔室,将琼脂糖块浸入 NaBH4 缓冲液中,在 4 °C 下过夜。
- 在 4 °C 下 1-2 周内将缓冲液多次更改为 PB。
注意:如果缓冲液交换不完全,组织自发荧光会变得非常微弱。 - 通过连接四个带有环氧树脂即时混合的钕磁铁来制作载物台。
- 用强力粘合剂(例如强力胶)将琼脂糖块安装到载物台上(图 1C)。
3. 组织处理
- 根据制造商的说明作整个组织成像系统。还公布了详细的协议16.目前的程序主要用于 TissueCyte1000,但也可以应用于其他型号,例如 TissueCyte 1600FC。
- 模型之间的作略有不同,但请遵循以下通常很重要的要点。
- 整个狨猴大脑可以从前端到后端进行冠状处理,而无需对硬件进行任何更改。确保在放置大脑时不超过载物台移动限制。
- 微调刀片的角度,确保刀片两端的表面深度在 10 μm 以内。由于髓鞘形成致密,即使深度差异为 10 μm,也可能导致不同的激光穿透成年狨猴白质。出于同样的原因,将成像平面设置为距表面约 25-35 μm。
- 使用陶瓷刀片(推荐)以 50 μm 的间隔(~超过 650 片)切割整个大脑。
- 脑膜保持未切割状态,如果未正确去除,则会干扰切片(图 2)。一些脑膜(例如,海马体或丘脑膜周围的脑膜)难以去除。注意到时,用细镊子手动提取这些脑膜。
4. 辅助组织学技术
- 章节检索
- 回收组织切片以进行各种组织学染色。按切片顺序准确对齐这些部分。追踪皮质层的血管以进行对齐(图 3)。
- 从每个切片上切掉多余的琼脂糖,以便更好地处理。
注意:此过程不必过度。琼脂糖用于将组织片段固定到位,而不会干扰组织学加工。 - 背光成像
- 将切片安装到载玻片上并干燥。
- 用 PBS 重新水化切片并放置盖玻片进行成像。
- 当荧光显微镜(图 4)在明场模式下对切片成像时,观察无染色的髓鞘形成模式。调整曝光时间,以便可以看到光反射。
注意:任何显微镜只要可以使用暗场照明,就可以使用。 - 取下盖玻片,进行尼氏染色或任何其他染色。任何标准染色程序都足够了。
- 使用这种方法直接比较不同的染色模式与髓鞘形成模式(图 4)。
- BDA 成像
- 有关详细过程,请参阅 表 1 。
- 首先,用 Dent 溶液(20% DMSO、80% 甲醇)处理切片。Dent 溶液中包含的甲醇显着增强了轴突中的 BDA 信号。
- 使用酪胺信号放大 (TSA) 方法增强要进行荧光检测的 BDA 信号17。
注意:TSA 生物素溶液可以从供应商处购买,但内部试剂更便宜、更有效。 - 要注册到 STPT 图像,请在使用安装介质将切片安装到载玻片玻璃上后拍摄背光图像。
- 意大利面怪兽荧光蛋白 (smFP) 染色
注:"意大利面怪物"荧光蛋白 (smFPs) 是具有多个表位标签18 的非荧光 GFP 变体家族。它们在免疫检测时提供出色的荧光信号,适合用作荧光示踪剂的配套示踪剂。有关 Addgene 提供的 AAV 构建体,请参见 表 2 。- 按照 表 1 中提供的详细过程进行作。
5. 成像后数据处理
- 数据完整性测试
注意:STPT 会生成大量图像文件。当成年狨猴的大脑以 50 μm 的间隔进行冠状切片时,通常需要 650 多个切片才能覆盖整个大脑。每个部分的数据都由在三个通道中捕获的一系列平铺图像组成。这些图像文件与记录每个切片位置的元数据文件一起存储在单个文件夹中。为了最大限度地减少数据总量和处理时间,成像以运行块的形式进行,在此期间,平铺的 X 和 Y 步数逐渐变化。通常,20-30 个运行块,每个运行块包括 30-50 个切片的成像,构成了单个大脑的完整数据集。为了利用这些数据,需要几个图像处理步骤,包括拼接平铺图像、分割荧光信号以及将体积数据配准到标准模板等。- 使用各种软件工具或通过根据研究人员的特定需求量身定制的专用管道单独执行图像处理步骤。图像处理的细节超出了本文的范围,可以在其他出版物11,19 中找到。
- 无论使用何种过程,都要确保数据的组织一致。
注意:由于整个映像过程涉及许多运行块并跨越数天,因此中途取消运行并重新重新启动的情况并不少见。在这种情况下,数据结构可能会中断,从而阻碍图像处理管道的正常执行。 - 为了最大限度地降低数据结构中断的风险,请运行 Python 脚本来检查 GitHub (github.com/watkarbey/STPT_depo) 上提供的数据结构 (kn_pipeline_check_mosaic.py) 的完整性。
- 将染色图像配准到 STPT 图像
注意:可以使用 ImageJ 的 bUnwarpJ 插件执行染色图像到 STPT 图像的非线性转换。- 调整目标 (STPT) 和源(染色)图像的图像大小,使它们大致相等。
- 对每个图像进行二值化以可视化各部分的形状。
- 运行 bUnwarpJ。 请记住检查 Save Transformations 选项。
- 再次启动 bUnwarpJ 。选择二值化前的原始图像作为源图像。然后单击 Load Elastic Transformation 并选择保存的转换。
结果
在这里使用的典型设置中,成年狨猴的整个大脑可以在大约 1 周内以 ~1.3 x 1.3 μm/像素的分辨率和 50 μm 的切片间隔成像(图 1)。这相当于图像拼接后三个通道中的 ~650 张冠状图像。配备 16 倍物镜(尼康 16xW CFI75 LWD;NA = 0.80),单次拍摄的视场约为 1 x 1 毫米。通过拼接这些照片获得整个冠状表面的图像(图 5A)。Z 方向的对齐非常好,通过简单地堆叠冠状图像数据即可获得良好的 3D 图像(图 5B,E)。为了标准化,marmoset brain11 的 STPT 模板可用于 3D-3D 配准(图 5F)。这种数据转换是全脑神经解剖学的关键方面之一,其中感兴趣区域被分配一个独立于解剖注释的绝对空间坐标。一旦样本大脑被注册到标准空间,就可以很容易地取出感兴趣的子区域进行进一步分析(图 5F,G)。特别是,皮质区域可以使用预定参数转换为平面图(图 5H)。
成像过程中生成的切片可用于各种组织学目的。如图 4A、B 所示,无染色的背光成像提供了与真实髓鞘染色非常相似的模式。这可能是髓鞘染色的绝佳替代方案。此外,如果在尼氏染色之前获得背光图像,则可以使用同一切片来获得髓鞘和尼氏染色的模式,从而为识别区域和层提供有用的信息(图 5C-E)。这些切片也可用于免疫学染色。在图 5J 中,用 NeuN 抗体对注射中心周围的切片进行复染,以估计 AAV 病毒的转导效率。除了荧光示踪剂外,还注射非荧光示踪剂并在切片检索后进行组织学检测也是一种很好的策略。在我们之前的研究中,我们将顺行绿色示踪剂与逆行"cre"载体相结合,后来通过抗 cre 抗体10 检测到。在图 6A-E 的示例中,将 BDA 注射到绿色示踪剂(三叶草)的对侧并对其进行荧光检测。请注意,红色 BDA 信号可以注册到 TissueCyte 图像中,以定位在整个大脑坐标中。在图 6F-I 的另一个示例中,smFP-myc 被注射到顶叶区域(图 6G),而绿色示踪剂被注射到额叶区域。这样,可以将多个示踪剂注射到同一只动物中,而不会干扰成像。使用 STPT 切片进行额外染色的一大优点是可以确定同一大脑的荧光和非荧光示踪剂之间的关系。因此,我们能够确定高精度10 的皮质皮质投射的互易性。另一个优点是,染色切片的 3D 坐标可以映射回 STPT 数据,然后映射到标准模板。因此,可能没有必要使用所有检索到的切片进行染色。为了更好地解释,可以选择切片进行染色,以向 STPT 数据添加更多上下文。
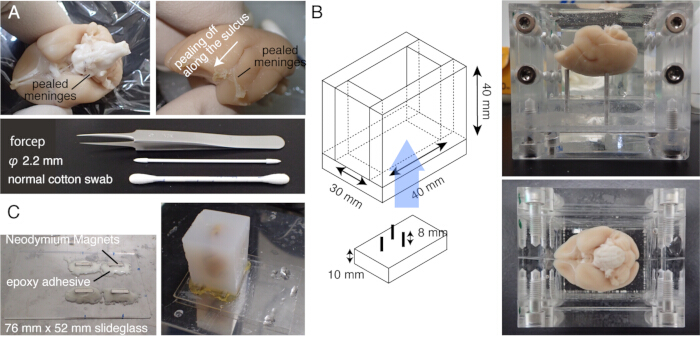
图 1:STPT 的样品制备。 (A) 使用棉签去除脑膜。脑干周围的脑膜可以用细尖镊子去除。用棉签摩擦手动去除狨猴脑周围的脑膜。照片显示了从外侧沟内剥落的脑膜。(B) 用于琼脂糖包埋的亚克力盒。针脚是可移动的,用于调整大脑的角度以接近立体定位固定位置20。(C) 由 76 mm x 52 mm 载玻片和四个钕磁铁制成的磁性载玻片,由环氧树脂粘合剂连接。琼脂糖块用强力胶连接到磁性载物台上。 请单击此处查看此图的较大版本。
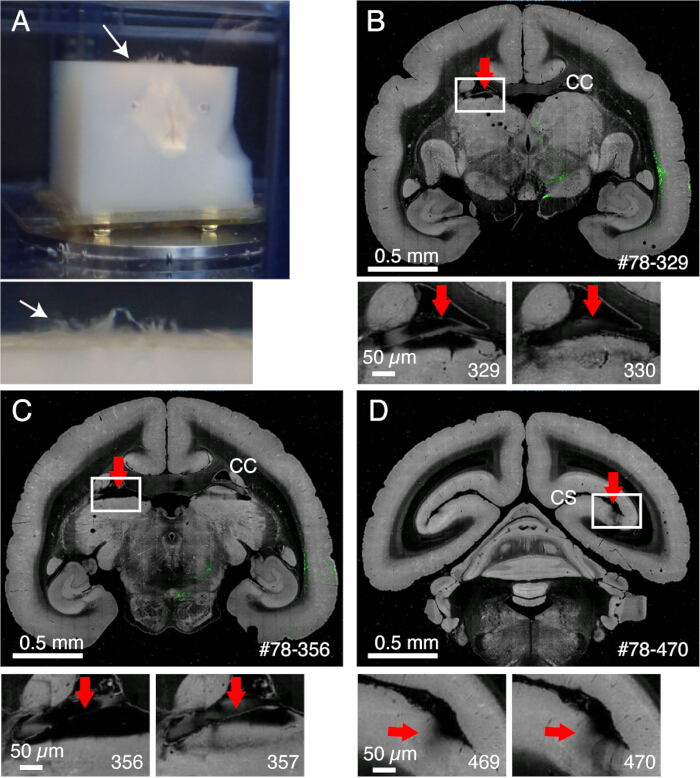
图 2:脑膜对成像的影响。 (A) 未切割的脑膜会向上漂浮,如白色箭头所示。在这个例子中,可以看到脑膜的广泛突出,因为它们在琼脂糖包埋之前没有被去除。通常,脑膜仅在几个困难的区域保持未切割状态。(B) 由于胼胝体和丘脑上部之间存在脑膜而导致的不良切片示例。在这种情况下,不良切片会导致深切片 (329) 和相对正常的切片 (330) 交替出现。#78-329 代表 Brain/MINDS 数据门户中的样本编号 78 第 329 节 (C) 成像不良的另一个例子。在更糟糕的情况下,红色箭头所示的丘脑核可能会完全脱落。比例尺:0.5 mm(上面板),0.5 μm(下面板)。(D) 成像不良的另一个例子。钙骨沟深处的脑膜很难去除。在第 469 节和第 470 节中看到的阴影是由目标下方的浮动脑膜引起的。比例尺:0.5 mm(上面板),0.5 μm(下面板)。 请单击此处查看此图的较大版本。
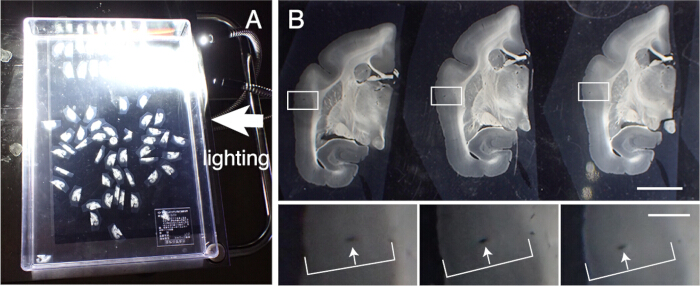
图 3:按顺序排列组织切片。 (A) 在塑料容器 (31 cm x 22.5 cm) 中,最多可以按顺序正确对齐 50 个狨猴冠状切片。为了可视化详细的结构,将容器放在黑纸上并从侧面照明。这些部分首先按顺序大致对齐,然后进行精确对齐。(B) 精确对位使用血管作为标志物。大脑皮层包含许多垂直穿过皮质层的血管。它们被识别为细长的孔,系统地改变它们在皮质层内的位置(白色箭头)。使用这种方法,即使是间隔为 50 μm 的切片也可以精确对齐。这些血管孔可以在 SPTP 切片图像中识别出来进行确认。比例尺:5 毫米(上面板),1 毫米(下面板)。 请单击此处查看此图的较大版本。
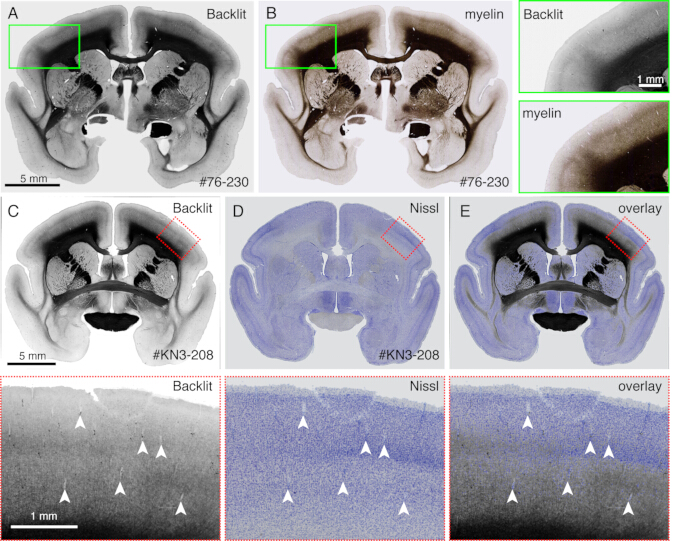
图 4:替代髓鞘染色的背光图像。(A,B) 相同的切片用于背光成像和髓鞘染色。首先,将切片安装在载玻片上,干燥,用 PBS 再水化,盖玻片,并使用光学显微镜成像(材料表)。取下盖玻片后,将同一切片用于髓鞘染色21 并使用荧光显微镜成像。使用 ImageJ 的 bUnwarpJ 插件将背光图像注册到髓鞘图像。绿色框显示每个图像的放大视图。请注意,这些图像显示几乎相同的模式,除了髓鞘染色更好地可视化纤维结构。(C-E)相同的切片用于背光成像和 Nissl 染色。首先使用 bUnwarpJ 插件将背光图像的低阈值蒙版注册到 Nissl 图像的低阈值蒙版,并使用相同的参数转换原始图像。请注意,血管(白色箭头)在两个图像之间匹配良好。因为这两张图片是针对同一切片的,所以匹配几乎是完美的,可以直接比较髓鞘和尼氏模式来识别皮层。比例尺:5 毫米(面板 A-E)。请单击此处查看此图的较大版本。
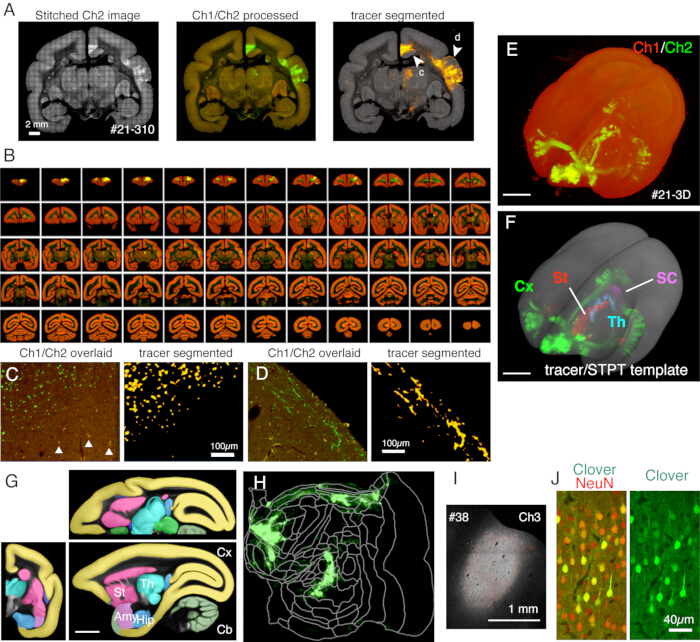
图 5:STPT 成像的典型结果。 (A) 示例切片(样品 #21 的第 310 节)的 Ch2(绿色)图像的简单平铺。如果不进行背景校正,则每个图块的边框都是可见的。在中间列中,图块分别对 Ch1(红色)和 Ch2(绿色)进行了背景校正。为了减少脂褐素的信号(参见图 C),从 Ch2 中减去 Ch1 信号并显示绿色。为了减少 Ch1 的脂褐素信号,ImageJ 在拼接之前使用了"Remove Outilers.." 命令。在右列中,示踪信号由图像处理管道11 分割。箭头 (c, d) 显示了放大视图在面板 C 和 D 中的显示位置。比例尺:2 mm。(B) 样品 #21 的序列截面概述。STPT 为该样本生成了 635 张高分辨率冠状图像。(C) 图 A 中箭头 c 显示的高放大倍率视图。这是 Ch1(红色)和 Ch2(绿色)的简单叠加,无需进一步处理。三角形显示脂褐素荧光信号,显示出广泛的光谱。示踪剂分割算法准确地将示踪信号与脂褐素背景区分开来,尽管它们的形状非常相似(右图)。比例尺:100 μm。(D) 高放大倍率视图的另一个示例。请注意,第 1 层中的细轴突纤维可以清晰可见。比例尺:100 μm。(E) 原始 STPT 图像的 3D 重建。图 B 所示的 635 张低分辨率冠状图像用作使用 fluorender22 进行 3D 可视化的 tiff 堆栈。比例尺:5 毫米。(F) 注册到 STPT 模板(灰色)的分段示踪信号的 3D 重建。不同脑区的示踪信号用不同的颜色表示。这些区域是通过使用面板 G 中显示的注释剪下来的。比例尺:5 毫米。(G) STPT 模板覆盖不同大脑区域的注释。比例尺:5 毫米。(H) 图 F 中显示的皮质示踪信号以平面图的形式显示。(I) 通过 Ch3 荧光鉴定注射部位,Ch3 荧光对示踪剂荧光不太敏感并且保持不饱和。比例尺:1 mm。(J) 用 NeuN 抗体对注射中心周围的切片进行染色,显示大约 30% 的神经元显示出三叶草绿色荧光的强烈表达。比例尺:40 μm。缩写:Cx;皮层,St;纹状体,Th;丘脑,SC;上丘。艾米;杏仁核,臀部;海马体,Cb;小脑。 请单击此处查看此图的较大版本。
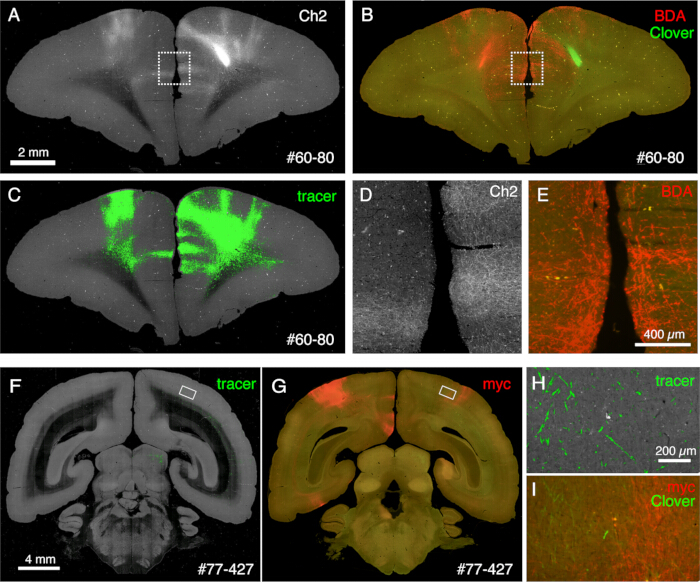
图 6:STPT 后组织学显示多个非荧光示踪剂。 (A-C)STPT 图像与 BDA 染色图像的比较。在这个样本中,BDA 被注射到 Clover 注射液的对侧。图 A 和 C 显示了 STPT 数据的 Ch2 图像和示踪剂分割。图 B 显示了 BDA 的 STPT 后染色。由于切片的甲醇处理,三叶草荧光减弱。比例尺:2 毫米。(D,E) 面板 A 和 B 中的虚线框被放大。比例尺:100 μm。(F,G) STPT 图像与抗 myc 标签抗体染色的比较。在这个样本中,三叶草注射是针对 PFC,而 AAV-smFP-myc 是注射到对侧顶叶皮层。白色矩形在面板 H 和 I 中被放大。比例尺:4 毫米。(H) 示踪剂分割以绿色显示。比例尺:200 μm。(I) myc 染色显示为红色。三叶草荧光显示为绿色。比例尺:200 μm。图 H 和图 I 中的绿色信号位于相似的位置,但不相同,因为 STPT 仅检索 ~10 μm 的光学切片。请单击此处查看此图的较大版本。
| BDA 荧光染色实验方案 | |
| 去除琼脂糖 | |
| TBS 清洗 | 10 分钟 (2x) |
| 1% H2O2 的 Dent 溶液 | 10 分钟 |
| TBS 清洗 | 短 |
| 0.5% TNB 阻塞 | 1 小时 |
| TNB 中的 StAvHRP (1:4000) | 2 过夜 |
| TNT 清洗 | 10 分钟 (3x) |
| TSA 生物素 (1:4000) 在 0.1 M 硼酸盐 (pH8.5) + 0.003% H2O2 中 | 2 小时 |
| TNT 清洗 | 10 分钟 (3x) |
| TNT 中的 Cy3-链霉亲和素 (1:1000) | 3 小时 |
| TNT 清洗 | 10 分钟 (2x) |
| TBS 清洗 | 保留该截面直到安装 |
| 使用抗淬灭封固剂将切片安装到载玻片上 | |
| 抗 MyC 荧光染色方案 | |
| 去除琼脂糖 | |
| TBS 清洗 | 10 分钟 (2x) |
| 在 IB 中阻止 | 1 小时 |
| IB 中的抗 Myc (1:4000) 抗体 | 2 个过夜 |
| TNT 清洗 | 10 分钟 (3x) |
| TNT 中的抗小鼠 Cy3 (1:1000) | 3 小时 |
| TNT 清洗 | 10 分钟 (2x) |
| TBS 清洗 | 保留该截面直到安装 |
| 使用抗淬灭封固剂将切片安装到载玻片上 | |
| 缓冲液/溶液 | 组成 |
| 0.5% 国能 | 0.5% TSA 封闭试剂,溶于 TS7.5 中 |
| Dent 的解决方案 | 20% DMSO、80% 甲醇 |
| 浸没缓冲液 (IB) | 10% FBS、2% BSA、0.5% TritonX100 的 TBS 溶液 |
| TBS(Tris 缓冲盐水) | 25 mM Tris、137 mM NaCl、2.7 mM KCl (pH 7.4) |
| 三硝基甲苯 | 0.05 % TS7.5 中的 Tween20 |
| TS7.5 系列 | 0.1 M TRIS-HCl,pH 7.5,0.15 M 氯化钠 |
表 1:BDA 荧光染色和抗 myc 荧光染色方案
| 用于 AAV 示踪剂的质粒 | Addgene 编号 | 推荐抗体 | 建议的稀释度 | 预期结果 |
| pAAV-EF1_Cre | 201198 | Millipore 克隆 2D8 | 1:1000 | 对细胞有益 |
| pAAVCam1.3_smFP_Myc | 201205 | MBL M192 My3 (鼠标) | 1:4000 | 非常好 |
| pAAVCam1.3_smFP_HA | 201206 | CST C29F4 (兔) | 1:1000 | 对细胞有益 |
| pAAVCam1.3_smFP_FLAG | 201207 | MBL PM020B(兔子) | 1:1000 | 还行 |
| AAVTRE3_smFP_Myc | 201208 | |||
| AAVTRE3_smFP_HA | 201209 | |||
| AAVTRE3_smFP_FLAG | 201210 |
表 2:可用于生产非荧光示踪剂的 Addgene 质粒列表。cre 构建体靶向细胞核,适用于被 AAV2 retro 包裹的逆行追踪。smFP_HA构建体都适用于细胞体检测(和逆行)。
补充视频:脑膜去除的显微镜视图。请点击此处下载此视频。
讨论
本文解释了处理狨猴大脑以进行全脑处理的实用解决方案,以及增强 STPT 技术实用性的辅助组织学技术。使用 STPT 的"全脑神经解剖学"的优势在于,您可以获得任何感兴趣区域的 3D 坐标,无论它是否经过解剖学注释。通过高精度 3D 到 3D 配准,可以将这些坐标转换为用于叠加多个数据集的标准模板。这样,标准模板就可以用作数据集成的媒介。这是我们前额叶皮层 (PFC) 映射项目10 的一个重要方面,其中分析了从许多个体获得的数据。此外,映射到标准模板的信息可以与已经映射的各种数据进行比较,无论是尼氏菌、髓鞘模式、示踪数据、MRI(包括弥散 MRI)数据还是解剖注释11。重要的是,它还可以与尚未出现的技术将获得的未来数据进行比较。目前存在多个狨猴大脑模板,它们基于尼氏染色23、MRI23、24、25、26、27、28 和 STPT11。但它们可以根据图像对比度和预先计算的参数转换为彼此的坐标11。跨研究的全脑规模的数据整合有助于更好地理解大脑作为一个系统。这种策略起作用的先决条件是在整个大脑中可靠地采集数据。下面讨论了与当前协议相关的关键步骤和潜在问题。
STPT 成像最脆弱的过程之一是组织切片。如上所述,脑膜通常保持未切割状态,可能会干扰切片。特别是,丘脑核和上丘是两个受影响最大的大脑区域:根据脑膜去除和嵌入琼脂糖的彻底程度,它们可以在切片过程中从组织块中剥离出来。这些深部区域很难从外部接近,并且在去除脑膜时很容易破裂。仔细但彻底的脑膜切除对于成功的成像至关重要。另一个问题是切片切片向后翻转到组织块上,有时即使切片后切片仍保持连接时,就会发生这种情况。可以通过塑造块状物来最小化它,使刀片在最末端倾斜切割。
通过深入组织块成像,STPT 避免了其表面的凹凸不平。虽然荧光信号可以很容易地穿过皮质区域,但它们在有髓区域高度减弱。因此,需要仔细确定成像深度,以平衡整个块表面的一致成像与富含髓鞘区域(如白质)中信号的亮度。在这里使用的设置中,我们通常瞄准距表面 25-35 μm。还需要注意的是,长时间存放后,切割表面可能会变得不均匀收缩。为了最大限度地减少成像区域,我们将成像会话分成 20-30 次运行,具有不同的阶段设置,持续 5-6 天。我们要么使运行之间的间隔小于 2 小时,要么在每次运行前确认表面深度并调整载物台高度。
在该协议中,BDA 信号通过 TSA 方法放大。这种方法非常有效,即使在相对较低的分辨率下也可以检测顺行传输的 BDA 信号(例如, 图 6B)。TSA 生物素可从 Akoya Biosciences 市售,但自制解决方案显示出更好的增强效果。另一方面,抗体和溶液的稀释度需要仔细调整以获得最佳结果。用甲醇溶液对切片进行预处理至关重要。如果不进行预处理,BDA 信号几乎无法在有髓轴突中检测到。
当使用顺行示踪剂时,由于荧光信号的饱和,通常很难确定确切的注射部位。在本研究中使用的整个组织成像系统设置中,我们使用蓝色通道来识别感染细胞(图 5I)。即使红色和绿色通道饱和,通常也可以在蓝色通道中检测到每个受感染的神经元。Allen Mouse Brain Connectivity Atlas1 也是如此。检查来源细胞很重要,因为感染有时只涉及特定层。我们在小鼠脑连接图谱中相当频繁地遇到这种部分感染,可能是因为使用了离子电渗疗法29。病毒示踪剂的横向传播可能或多或少地可变,具体取决于注射。这种可变性可能会影响跟踪的结果,需要仔细规范化。
将获得的 3D 图像成功配准到标准模板上是全脑神经解剖学的关键过程。将 STPT 图像配准到 STPT 模板可能非常准确,我们只观察到具有高图像对比度的边界的几个体素(50 μm 等立方)的偏差10。尽管如此,注册的作用是有限的。由于脑膜去除过程,STPT 样本通常在半球之间以及皮层和中脑/后脑之间有间隙,而脑组织在 体内 MRI 图像中紧密堆积。这种差异很难通过注册来调整。狨猴皮层大部分没有脑沟,但有些个体的顶内沟非常深。这样的沟在注册时将丢失(发生自上而下的融合)。尽管配准是一项强大的技术,但有必要返回原始数据以确认获得的结果。
海量图像数据的生成既是这种技术的优点,也是局限性。虽然它提高了数据集的完整性,但它需要仔细管理获取的数据并开发自动化图像处理管道以实现高效的数据解释。未来,生成式人工智能 (AI) 在构建图像处理管道中的应用可能会显着简化这一过程。系统全脑成像也使用基于载玻片扫描仪的方法进行21,30。与这些方法相比,STPT 不需要额外的 3D 重建计算。结合光学切片,我们已经证明 STPT 有可能在第31 节中重建甚至轴突节段。随着组织清除技术的进一步结合,Economo 等人开发了一种对整个稀疏标记的神经元进行成像的方法32,33。最新版本的 TissueCyte 现在提供附加激光器以增强红色荧光蛋白的激发或切片捕获单元以自动恢复切片的选项。随着这些进步,全脑方法将变得更加高效,为全面了解灵长类动物的大脑(包括人类的大脑)奠定基础。
披露声明
作者没有什么可披露的。
致谢
我们非常感谢 Yamamori 实验室和动物设施 (RRD) 的技术人员的帮助。我们感谢 RIKEN CBS-Olympus 协作中心在共聚焦图像采集方面提供的技术援助。这项工作得到了日本文部科学省创新领域科学研究计划(资助号 22123009)、日本 AMED 的 Brain/MINDS 和 Brain/MINDS2.0(JP15dm0207001、JP23wm0625001 和 JP24wm0625218)和 JSPS KAKENHI 资助号 24K09678 对 AW 的支持。
材料
| Name | Company | Catalog Number | Comments |
| Agarose type I | Sigma | A6013 | |
| All-in-one fluorescence microscope | Keyence | BZ-X series | This study used BZ-X710 |
| anti-mouse Cy3 | Jackson Immuno Research Laboratories, Inc. | 115-165-003 | |
| anti-myc antibody | MBL | M192 | mouse monoclonal |
| Butorphanol | Meiji Animal Health | Vetorphale | |
| Ceramic blade | TissueVision | N/A | |
| Collagenase type I | Wako Chemical | #031-17601 | Aliquot into 100 µL x 10 of 100 mg/mL in PBS |
| Cotton swab (HUBY-340) | HUBY | BB-013SP | micro cotton swabs |
| Custom-made chamber | N/A | N/A | The chamber consist of four side plates and one bottom plates, all made of 1 cm thick acrylic plates, which can be securely fastened together using screw bolts. An additional acrylic plate is placed inside the chamber, with three metal poles inserted through it to hold the marmoset brain in an inverted position. |
| Cy3-Streptavidin | Jackson Immuno Research Laboratories, Inc. | 016-160-084 | |
| Dumont #5 forceps, Inox | N/A | N/A | |
| Epoxy instant mix | Locktite | N/A | |
| FIJI | NIH | https://imagej.net/software/fiji/downloads | |
| kn_pipeline_check_mosaic.py | N/A | N/A | github.com/watkarbey/STPT_depo; This is a checking script for TissueCyte data. |
| Medetomidine | Zenoaq | Domitor | |
| Midazolam | Sandoz | N/A | |
| NaBH4 | Sigma | #452882 | |
| NaIO4 | Sigma | S1878 | |
| Perfusion needle | Natsume Seisakusho Co., Ltd | KN-348, 20G-50 | |
| Slideglass (76 mm x 52 mm) | Matsunami | S9111 | |
| StAvHRP | Jackson Immuno Research Laboratories, Inc. | 016-030-084 | HRP-streptavidin (titration needs to be carefully determined) |
| TissueCyte1000 | TissueVision | https://www.tissuevision.com/tissuecyte | |
| TSA blocking reagent | Kiko Tech | FP1012 | Refer to TSA-biotin kit (Akoya Biosciences) |
| TSA-biotin | House-made | N/A | See Okamoto et al (ref. 16) |
| VECTASHIELD HardSet Mounting Medium | Vector laboratories Inc | N/A | Antifade mounting medium |
参考文献
- Oh, S. W., Harris, J. A., Ng, L., Winslow, B., Cain, N., et al. A mesoscale connectome of the mouse brain. Nature. 508 (7495), 207-214 (2014).
- Ragan, T., et al. Serial two-photon tomography for automated ex vivo mouse brain imaging. Nat Methods. 9 (3), 255-258 (2012).
- Liang, Z., Arefin, T. M., Lee, C. H., Zhang, J. Using mesoscopic tract-tracing data to guide the estimation of fiber orientation distributions in the mouse brain from diffusion MRI. Neuroimage. 270, 119999(2023).
- Watakabe, A., Hirokawa, J. Cortical networks of the mouse brain elaborate within the gray matter. Brain Struct Funct. 223 (8), 3633-3652 (2018).
- Ypma, R. J. F., Bullmore, E. T. Statistical analysis of tract-tracing experiments demonstrates a dense, complex cortical network in the mouse. PLoS Comput Biol. 12 (9), e1005104(2016).
- Wang, Q., et al. The Allen mouse brain common coordinate framework: A 3D reference atlas. Cell. 181 (4), 936-953.e20 (2020).
- Köbbert, C., Apps, R., Bechmann, I., Lanciego, J. L., Mey, J., Thanos, S. Current concepts in neuroanatomical tracing. Prog Neurobiol. 62 (4), 327-351 (2000).
- Lanciego, J. L., Wouterlood, F. G. Neuroanatomical tract-tracing techniques that did go viral. Brain Struct Funct. 225 (4), 1193-1224 (2020).
- Bakker, R., Wachtler, T., Diesmann, M. CoCoMac 2.0 and the future of tract-tracing databases. Front Neuroinform. 6, (2012).
- Watakabe, A., et al. Local and long-distance organization of prefrontal cortex circuits in the marmoset brain. Neuron. 111 (14), 2258-2273.e10 (2023).
- Skibbe, H., et al. The Brain/MINDS marmoset connectivity resource: An open-access platform for cellular-level tracing and tractography in the primate brain. PLoS Biol. 21 (6), e3002158(2023).
- Mitchell, J. F., Leopold, D. A. The marmoset monkey as a model for visual neuroscience. Neurosci Res. 93, 20-46 (2015).
- Okano, H. Current status of and perspectives on the application of marmosets in neurobiology. Annu Rev Neurosci. 44, 27-48 (2021).
- Watakabe, A., et al. Comparative analyses of adeno-associated viral vector serotypes 1, 2, 5, 8 and 9 in marmoset, mouse and macaque cerebral cortex. Neurosci Res. 93, 144-157 (2015).
- Hata, J., et al. Multi-modal brain magnetic resonance imaging database covering marmosets with a wide age range. Sci Data. 10, 221(2023).
- Liwang, J. K., Bennett, H. C., Pi, H. -J., Kim, Y. Protocol for using serial two-photon tomography to map cell types and cerebrovasculature at single-cell resolution in the whole adult mouse brain. STAR Protoc. 4 (1), 102048(2023).
- Okamoto, S., et al. Exclusive labeling of direct and indirect pathway neurons in the mouse neostriatum by an adeno-associated virus vector with Cre/lox system. STAR Protoc. 2 (1), 100230(2021).
- Viswanathan, S., et al. High-performance probes for light and electron microscopy. Nat Methods. 12 (6), 568-576 (2015).
- Kuan, L., et al. Neuroinformatics of the Allen mouse brain connectivity atlas. Methods. 73, 4-17 (2015).
- Paxinos, G., Watson, C., Petrides, M., Rosa, M., Tokuno, H. The Marmoset Brain in Stereotaxic Coordinates. , Academic Press. London, Waltham, MA. (2012).
- Abe, H., et al. 3D reconstruction of brain section images for creating axonal projection maps in marmosets. J Neurosci Methods. 286, 102-113 (2017).
- Wan, Y., et al. FluoRender: Joint freehand segmentation and visualization for many-channel fluorescence data analysis. BMC Bioinformatics. 18 (1), 280(2017).
- Majka, P., et al. Towards a comprehensive atlas of cortical connections in a primate brain: Mapping tracer injection studies of the common marmoset into a reference digital template. J Comp Neurol. 524 (11), 2161-2181 (2016).
- Liu, C., Yen, C. C. -C., Szczupak, D., Tian, X., Glen, D., Silva, A. C. Marmoset brain mapping V3: Population multi-modal standard volumetric and surface-based templates. Neuroimage. 226, 117620(2021).
- Liu, C., et al. A digital 3D atlas of the marmoset brain based on multi-modal MRI. Neuroimage. 169, 106-116 (2018).
- Liu, C., et al. A resource for the detailed 3D mapping of white matter pathways in the marmoset brain. Nat Neurosci. 23 (2), 271-280 (2020).
- Woodward, A., et al. The Brain/MINDS 3D digital marmoset brain atlas. Sci Data. 5, 180009(2018).
- Saleem, K. S., Avram, A. V., Glen, D., Schram, V., Basser, P. J. The subcortical atlas of the marmoset ("SAM") monkey based on high-resolution MRI and histology. Cereb Cortex. 34 (4), bhae120(2024).
- Watakabe, A., Hirokawa, J. Cortical networks of the mouse brain elaborate within the gray matter. Brain Struct Funct. 223 (8), 3633-3652 (2018).
- Lin, M. K., et al. A high-throughput neurohistological pipeline for brain-wide mesoscale connectivity mapping of the common marmoset. eLife. 8, e40042(2019).
- Skibbe, H., et al. PAT-probabilistic axon tracking for densely labeled neurons in large 3-D micrographs. IEEE Trans Med Imaging. 38 (1), 69-78 (2019).
- Economo, M. N., et al. A platform for brain-wide imaging and reconstruction of individual neurons. eLife. 5, e10566(2016).
- Winnubst, J., et al. Reconstruction of 1,000 projection neurons reveals new cell types and organization of long-range connectivity in the mouse brain. Cell. 179 (1), 268-281.e13 (2019).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。