Method Article
Serielle Zwei-Photonen-Tomographie des gesamten Weißbüschelaffen-Gehirns für neuroanatomische Analysen
In diesem Artikel
Zusammenfassung
Die serielle Zwei-Photonen-Tomographie (STPT) ist eine Technik, bei der eine Gewebemasse in ihrer dreidimensionalen Form abgebildet wird, indem die Zwei-Photonen-Bildgebung mit der automatischen Tischsteuerung und dem Mikrotom-Schneiden kombiniert wird. Hier beschreiben wir ein Protokoll zur Implementierung für Weißbüschelaffengehirne, um ihre strukturellen Merkmale besser zu verstehen.
Zusammenfassung
Die serielle Zwei-Photonen-Tomographie (STPT) ist eine Technik, mit der eine Gewebemasse in ihrer dreidimensionalen Form abgebildet wird, indem die Zwei-Photonen-Bildgebung mit der automatischen Tischsteuerung und dem Mikrotom-Schneiden kombiniert wird. Wir haben diese Technik erfolgreich zur Verfolgung der axonalen Projektionen im Weißbüschelaffengehirn eingesetzt. Hier werden die detaillierten experimentellen Verfahren beschrieben, die zu einer zuverlässigen volumetrischen Bildgebung des gesamten Weißbüschelaffengehirns führten. Ein Schlüsselverfahren für eine erfolgreiche Bildgebung war die Entfernung von Hirnhäuten, die das Gehirn umgeben, was das Schneiden beeinträchtigt. Ein großer Vorteil dieser Methode besteht darin, dass die geschnittenen Abschnitte für zusätzliche Färbungen verwendet werden können. Im ursprünglichen Aufbau werden die geschnittenen Abschnitte im Wasserbad verrührt. Diese Abschnitte können in ihrer ursprünglichen Reihenfolge entsprechend den Blutgefäßmustern in der Hirnrinde korrekt ausgerichtet werden. Ein Beispiel für eine effektive Histologie ist die Visualisierung der Myelinstruktur durch einfache Lichtreflexion, die mit der Nissl-Färbung kombiniert werden kann, um anatomische Grenzen zu definieren. Diese Schnitte können auch für den immunologischen Nachweis von nicht-fluoreszierenden anterograden und retrograden Tracern verwendet werden, die in den STPT-Daten registriert werden können, um mehrere Daten zu schichten.
Einleitung
In neuroanatomischen Studien stehen Forscher vor der Notwendigkeit, Strukturen im Mikrometerbereich (z.B. Axone und Boutons) im Kontext des gesamten Gehirns zu beobachten. Diese schwierige Aufgabe wurde in der Regel durch die visuelle Inspektion der Serienabschnitte und die Suche nach dem Bereich von Interesse für detaillierte Bildgebung, Analysen und Fotoaufnahmen angegangen. Mit dem technologischen Fortschritt wird es jedoch möglich, das gesamte Gehirn mit hoher Auflösung für die Ganzhirnanalyse abzubilden. In einer bahnbrechenden Studie von Oh et al.1 erhielten Hunderte von Mäusegehirnen Tracer-Injektionen für die Konnektomanalyse, die mit Hilfe der seriellen Zwei-Photonen-Tomographie (STPT) Bildgebungstechnikverarbeitet wurden 2. Das Merkmal dieser Studie war, dass sie anterograde Tracer auswählten, um die "Konnektivität" zu quantifizieren. Während anterograde Tracer sehr detaillierte räumliche Informationen über die axonale Verteilung liefern, haben sich Neuroanatomen für ihre Analyse auf eine aufwendige manuelle Segmentierung verlassen. Durch die Automatisierung verschiedener Analyseverfahren, einschließlich dieses Segmentierungsschritts, gelang es ihnen, die "Massenproduktion" von gebrauchsfertigen, hochauflösenden Tracer-Daten für den Mehrzweckgebrauch zu erreichen. Die Wirksamkeit ihres Ansatzes liegt auf der Hand, wenn man bedenkt, dass eine Vielzahl von Studien ihre Tracerdaten 3,4,5 und ihr Standardgehirn6 verwendet haben.
Historisch gesehen hat die neuronale Konnektivität von nicht-menschlichen Primatengehirnen die Aufmerksamkeit vieler Neuroanatomen auf sich gezogen, seit der Entwicklung der Degenerationsmethode in den 1950er Jahren, über anterograde/retrograde Substanztransportmethoden in den 1970er Jahren bis hin zur heutigen viralen Strategie 7,8. Daher gibt es eine Vielzahl von Literaturstücken, die die neuronalen Verbindungen von Primaten untersuchen. Insbesondere haben viele Forscher die komplexe kortikokortikale Konnektivität des Makakengehirns untersucht, und ihre Ergebnisse wurden für die Tabellierung kuratiert (z. B. CoCoMac9). Obwohl diese klassischen Studien nützlich waren, hatten sie mehrere Einschränkungen. Erstens, da sich jede Studie nur auf begrenzte Hirnregionen konzentriert, werden die gewonnenen Informationen unweigerlich fragmentarisch. Zweitens verwendet jede Studie unterschiedliche Methoden und Bedingungen. Damit wird die quantitative Auswertung über Studien hinweg kompliziert. Drittens wird die Konnektivität in der Regel entweder als Camera Lucida repräsentativer Schnitte oder als semiquantitative Tabelle/Grafik für vom Autor definierte Hirnregionen dargestellt. Mit anderen Worten, es werden nur sehr begrenzte Informationen aus einer komplexen Gehirnstruktur für die Präsentation in der veröffentlichten Literatur extrahiert. Mit der Entwicklung der Magnetresonanztomographie (MRT) gewannen Ganzhirnuntersuchungen mit niedriger Auflösung an Bedeutung. Es gibt jedoch eine große Lücke zwischen dem Detaillierungsgrad dessen, was wir über neuronale Verbindungen bei Mäusen und denen bei Primaten wissen.
Vor diesem Hintergrund machten wir uns daran, eine umfassende anterograde Nachverfolgung der Gehirne von Weißbüschelaffen durchzuführen10,11. Obwohl das Weißbüschelaffen-Pendant viel kleiner und glatter ist als das Gehirn der Makaken, zeigt es deutliche Anzeichen von Primaten, wie z.B. das Vorhandensein des Bereichs MT und der körnigen präfrontalen Bereiche, von denen keiner bei Nagetieren klar definiert ist12,13. Hier war die geringe Größe ein großer Vorteil, denn selbst das Weißbüschelaffengehirn wiegt zehnmal mehr als das Mäusegehirn. Glücklicherweise konnten wir das gesamte Weißbüschelaffen-Gehirn mit minimalen Updates der Originalversion der Betriebssoftware TissueCyte1000 (im Folgenden als Ganzgewebe-Bildgebungssystem bezeichnet), dem kommerziell erhältlichen Mikroskop für die STPT-Bildgebung2, abbilden. Dieses Update sollte eine zusätzliche Bewegung der Bühne vor dem Schneiden ermöglichen. Die aktuelle Version reicht nun für die Verarbeitung des Weißbüschelaffengehirns aus. In diesem Artikel wird ein Protokoll für den Umgang mit dem Weißbüschelaffengehirn für die STPT-Bildgebung vorgestellt. Das Post-Imaging-Protokoll, das den Nutzen dieser Methode weiter erhöht, wird ebenfalls bereitgestellt.
Protokoll
Alle experimentellen Verfahren wurden gemäß dem 1996 überarbeiteten Leitfaden des National Institute of Health für die Pflege und Verwendung von Labortieren (NIH-Veröffentlichungen Nr. 80-23) und den "Leitprinzipien für die Pflege und Verwendung von Tieren auf dem Gebiet der physiologischen Wissenschaft" der Japanischen Physiologischen Gesellschaft durchgeführt und vom Experimental Animal Committee des RIKEN genehmigt (W2020-2-009(2)).
1. Tracer-Injektion
- Injektionen von fluoreszierenden und nicht-fluoreszierenden Tracern in das Weißbüschelaffengehirn gemäß den zuvor beschriebenen Verfahrendurchführen 14.
- Bei der Kombination mit der STPT-Methode ist darauf zu achten, dass die Fluoreszenz der Tracer stark genug ist, um ohne Verstärkung nachgewiesen werden zu können. Verwenden Sie ein Doppelvektor-TET-Off-System10 für einen verbesserten Ausdruck.
HINWEIS: Nicht fluoreszierende Tracer können auch nach der STPT mit den hergestellten Scheiben injiziert und nachgewiesen werden. Beispiele für nicht-fluoreszierende Tracer sind biotinyliertes Dextran-Amin (BDA), cre-exprimierendes AAV in AAV2 retro für den retrograden Nachweis der Eingangszellkerne10 sowie GFP-basierte smFP-tag-AAVs (siehe unten).
- Bei der Kombination mit der STPT-Methode ist darauf zu achten, dass die Fluoreszenz der Tracer stark genug ist, um ohne Verstärkung nachgewiesen werden zu können. Verwenden Sie ein Doppelvektor-TET-Off-System10 für einen verbesserten Ausdruck.
- Perfusionsfixierung und Erhalt des Weißbüschelaffengehirns nach 4 Wochen.
- Um den Weißbüschelaffen zu betäuben, verabreichen Sie Medetomidin (0,04 mg/kg), Midazolam (0,4 mg/kg) und Butorphanol (0,4 mg/kg), MMB genannt, intramuskulär, gefolgt von einer intraperitonealen Injektion von Thiopental-Natrium (100 mg/kg).
- Nachdem Sie bestätigt haben, dass der Schmerzreflex verloren gegangen ist, schneiden Sie die Brusthöhle auf, um das Herz freizulegen, und schneiden Sie den rechten Vorhof, damit das Blut und das Fixiermittel austreten können.
- Anschließend wird der linke Ventrikel durchtrennt, um die Perfusionsnadel einzuführen, um das Blut zunächst mit der Präfixlösung (250 mM Saccharose, 5 mM MgCl2 in 0,02 M Phosphatpuffer [PB, pH 7,4]) für einige Minuten (~50-100 mL) zu spülen, gefolgt von der Fixierung mit 4% Paraformaldehyd/0,1 M Phosphatpuffer (PB; 2-3 mal das Gewicht des Tieres über ~20 min).
- Eine gute Durchblutung ist entscheidend für ein gutes Ergebnis. Wenn Sie die Perfusionsnadel einführen, schieben Sie sie entlang des Septums in Richtung Aorta, um einen guten Fluss des Fixiermittels zu gewährleisten. Die Spitze der Nadel wird sichtbar, wenn sie die Aorta erreicht. Ziehen Sie sich zurück, bis die Spitze kaum sichtbar ist, damit sie die Halsschlagader nicht umgeht.
- Halten Sie das Gehirn 48 h lang bei 4 μm PB in 4 % Paraformaldehyd/0,1 M PB und übertragen Sie es auf 50 mM PB. Wenn es nicht sofort verwendet wird, lagern Sie das Gehirn in 0,75 % Glycin/0,1 M PB, um Autofluoreszenz aufgrund einer Überfixierung zu verhindern.
2. Probenvorbereitung (Abbildung 1)
- Inkubieren Sie das fixierte Gehirn 1 h lang mit Kollagenase (1 mg/ml in 3 mM CaCl,2 in 10 ml tris-gepufferter Kochsalzlösung [TBS]) bei 37 °C.
HINWEIS: Das Gehirn vor der Inkubation 5 Minuten lang bei 37 °C vorwärmen. - Reiben Sie die Gehirnoberfläche vorsichtig mit einem Wattestäbchen ab, um die Pia-Materie und andere Hirnhäute abzuziehen (Abbildung 1A, Ergänzendes Video). Schälen Sie die abgelösten Hirnhäute mit einer feinen Pinzette.
HINWEIS: Es ist wichtig, die Hirnhäute um das Mittelhirn herum zu entfernen, einschließlich des Colliculus superior oder der oberen Thalamus. Es ist schwierig, diese Strukturen, die tief im Inneren verborgen sind, freizulegen. Entfernen Sie jedoch die Hirnhäute so weit wie möglich. Andernfalls bleiben sie oft ungeschnitten, schwimmen nach oben und stören die Bildgebung und das Schneiden (Abbildung 2A). - Seien Sie besonders vorsichtig, wenn das Gehirn vor der Einbettung einer Ex-vivo-MRT unterzogen wird, bei der das Gehirn in Fluorlösung15 getaucht wird. Das übrig gebliebene Fluor könnte, wenn es zusammen eingebettet ist, während der Bildgebung winzige Luftblasen unter dem Objektiv bilden. Um dies zu vermeiden, halten Sie das Gehirn vor dem Einbetten 1 Woche lang in PB.
HINWEIS: Dieser Warteschritt ist in der Regel nicht notwendig, aber in Kombination mit einer ex vivo MRT kritisch. - Bereiten Sie den NaBH4-Puffer vor, indem Sie 0,2 g NaBH4 in 100 mL 50 mM Boratpuffer (pH 9,2) lösen, der auf 40 °C erwärmt wurde. Lassen Sie die Kappe locker, um das CO2 - Gas abzulassen. Bewahren Sie die Lösung über Nacht mit gelöster Kappe auf. Wickeln Sie die Flasche in Alufolie ein, um sie bis zum Gebrauch vor Licht zu schützen.
- Stellen Sie oxidierte Agarose her, indem Sie 2,25 g Agarose und 0,21 g NaIO4 in 100 mL PB für 2-3 h rühren. Filtern Sie die Lösung mit Vakuumsaugen und waschen Sie sie mit drei Änderungen des PB. Resuspendieren Sie die Agarose in 50 ml PB.
- Die Agarose in der Mikrowelle vollständig schmelzen und auf 60-65 °C abkühlen lassen.
- Platzieren Sie das Gehirn in einer speziell angefertigten Kammer und betten Sie das Gehirn in Agarose ein. Achten Sie darauf, dass die Agarose in den Hohlraum unterhalb des Corpus callosum eingeführt wird (Abbildung 1B).
HINWEIS: Das Gehirn zuerst bei Raumtemperatur und dann 5 Minuten lang bei 65 °C vorwärmen, damit sich die Agarose auf der Gehirnoberfläche absetzen kann, ohne sich zu verfestigen. - Die Kammer wird zerlegt und der Agaroseblock über Nacht bei 4 °C in den NaBH4-Puffer getaucht.
- Wechseln Sie den Puffer mehrmals über 1-2 Wochen bei 4 °C auf PB.
HINWEIS: Die Autofluoreszenz des Gewebes wird sehr schwach, wenn der Pufferaustausch unvollständig ist. - Stellen Sie einen Dia-Glastisch her, indem Sie vier Neodym-Magnete mit Epoxid-Instant-Mix anbringen.
- Montieren Sie den Agaroseblock mit einem starken Kleber (z. B. Sekundenkleber) auf der Bühne (Abbildung 1C).
3. Gewebeaufbereitung
- Betreiben Sie das gesamte Gewebebildgebungssystem gemäß den Anweisungen des Herstellers. Ein ausführliches Protokoll wird ebenfalls veröffentlicht16. Das derzeitige Verfahren wird hauptsächlich für TissueCyte1000 verwendet, kann aber auch auf andere Modelle, wie z. B. TissueCyte 1600FC, angewendet werden.
- Die Bedienung unterscheidet sich geringfügig zwischen den Modellen, aber befolgen Sie die folgenden Punkte, die allgemein wichtig sind.
- Das gesamte Weißbüschelaffen-Gehirn kann koronal von den vorderen bis zu den hinteren Enden bearbeitet werden, ohne dass sich die Hardware verändern muss. Achten Sie darauf, dass die Grenzen der Tischbewegung beim Platzieren des Gehirns nicht überschritten werden.
- Passen Sie den Winkel der Klingen fein ab, um sicherzustellen, dass die Oberflächentiefen an beiden Enden der Klinge innerhalb von 10 μm liegen. Aufgrund der dichten Myelinisierung kann selbst ein Tiefenunterschied von 10 μm zu unterschiedlichen Laserdurchdringungen durch die weiße Substanz adulter Weißbüschelaffen führen. Aus dem gleichen Grund stellen Sie die Bildebene auf etwa 25-35 μm über der Oberfläche ein.
- Verwenden Sie eine Keramikklinge (empfohlen), um das gesamte Gehirn in Abständen von 50 μm (~über 650 Scheiben) zu schneiden.
- Die Hirnhäute bleiben ungeschnitten und stören das Schneiden, wenn sie nicht ordnungsgemäß entfernt werden (Abbildung 2). Einige Hirnhäute (z. B. solche, die den Hippocampus oder den Pulvinar umgeben) sind schwer zu entfernen. Ziehen Sie diese Hirnhäute manuell mit einer feinen Zange heraus, wenn sie bemerkt werden.
4. Histologische Hilfstechniken
- Abrufen von Abschnitten
- Entnehmen Sie die Gewebeschnitte, um verschiedene histologische Färbungen durchzuführen. Richten Sie diese Abschnitte in der Reihenfolge des Schneidens genau aus. Verfolgen Sie die Blutgefäße zur Ausrichtung durch die kortikalen Schichten (Abbildung 3).
- Schneiden Sie überschüssige Agarose von jedem Abschnitt ab, um die Handhabung zu verbessern.
HINWEIS: Dieser Vorgang muss nicht übermäßig sein. Die Agarose dient dazu, Gewebesegmente an Ort und Stelle zu halten, ohne die histologische Verarbeitung zu beeinträchtigen. - Bildgebung mit Gegenlicht
- Montieren Sie die Abschnitte auf ein Diaglas und trocknen Sie sie.
- Rehydrieren Sie den Abschnitt mit PBS und legen Sie das Deckglas für die Bildgebung auf.
- Beobachten Sie ein Myelinisierungsmuster ohne Färbung, wenn der Schnitt mit einem Fluoreszenzmikroskop (Abbildung 4) im Hellfeldmodus abgebildet wird. Stellen Sie die Belichtungszeit so ein, dass die Lichtreflexion sichtbar ist.
HINWEIS: Jedes Mikroskop kann verwendet werden, wenn es Dunkelfeldbeleuchtung verwenden kann. - Entfernen Sie das Deckglas und fahren Sie mit der Nissl-Färbung oder einer anderen Färbung fort. Jedes Standard-Färbeverfahren ist ausreichend.
- Verwenden Sie diese Methode, um verschiedene Färbemuster direkt mit den Myelinisierungsmustern zu vergleichen (Abbildung 4).
- BDA-Bildgebung
- In Tabelle 1 finden Sie ein detailliertes Vorgehen.
- Behandeln Sie zunächst die Abschnitte mit Dent's Solution (20% DMSO, 80% Methanol). Methanol, das in Dents Lösung enthalten ist, verstärkt die BDA-Signale in den Axonen dramatisch.
- Verwenden Sie die Tyramid-Signalverstärkungsmethode (TSA), um das BDA-Signal zu verstärken, das fluoreszierend detektiert werdensoll 17.
HINWEIS: TSA-Biotin-Lösung kann vom Verkäufer gekauft werden, aber das hauseigene Reagenz ist viel billiger und wirksamer. - Um sich für das STPT-Image zu registrieren, nehmen Sie ein hintergrundbeleuchtetes Image auf, nachdem Sie den Abschnitt mit dem Montagemedium auf ein Diaglas montiert haben.
- Färbung von Spaghettimonster-Fluoreszenzproteinen (smFP)
HINWEIS: Die fluoreszierenden Proteine (smFPs) des "Spaghettimonsters" sind eine Familie von nicht fluoreszierenden GFP-Varianten mit mehreren Epitop-Tags18. Sie liefern hervorragende Fluoreszenzsignale bei der Immundetektion und eignen sich als Begleittracer für fluoreszierende Tracer. In Tabelle 2 finden Sie die AAV-Konstrukte, die von Addgene verfügbar sind.- Befolgen Sie das detaillierte Verfahren in Tabelle 1.
5. Datenverarbeitung nach der Bildgebung
- Test der Datenintegrität
HINWEIS: STPT generiert eine große Anzahl von Bilddateien. Wenn das Gehirn eines erwachsenen Weißbüschelaffen in Abständen von 50 μm koronal geschnitten wird, sind in der Regel mehr als 650 Schnitte erforderlich, um das gesamte Gehirn abzudecken. Die Daten jedes Abschnitts bestehen aus einer Reihe von Kachelbildern, die in drei Kanälen erfasst wurden. Diese Bilddateien werden in einem einzigen Ordner zusammen mit einer Metadatendatei gespeichert, in der die Position der einzelnen Kacheln aufgezeichnet wird. Um die Gesamtdatenmenge und die Verarbeitungszeit zu minimieren, wird die Bildverarbeitung in Blöcken von Durchläufen durchgeführt, in denen sich die Anzahl der X- und Y-Schritte für die Kachelung allmählich ändert. In der Regel bilden 20-30 Blöcke von Durchläufen, die jeweils die Bildgebung von 30-50 Schnitten umfassen, den vollständigen Datensatz für ein einzelnes Gehirn. Um diese Daten zu nutzen, sind mehrere Bildverarbeitungsschritte erforderlich, darunter das Zusammenfügen der gekachelten Bilder, die Segmentierung von Fluoreszenzsignalen und die Registrierung der volumetrischen Daten in einer Standardvorlage.- Führen Sie die Bildverarbeitungsschritte individuell mit verschiedenen Software-Tools oder über dedizierte Pipelines durch, die auf die spezifischen Bedürfnisse der Forscher zugeschnitten sind. Einzelheiten zur Bildbearbeitung gehen über den Rahmen dieses Beitrags hinaus und finden sich in anderen Publikationen11,19.
- Unabhängig vom verwendeten Verfahren stellen Sie sicher, dass die Daten konsistent organisiert sind.
HINWEIS: Da der gesamte Imaging-Prozess viele Blöcke von Durchläufen umfasst und sich über mehrere Tage erstreckt, ist es nicht ungewöhnlich, dass ein Lauf auf halbem Weg abgebrochen und neu gestartet wird. In solchen Fällen kann die Datenstruktur gestört werden, was die ordnungsgemäße Ausführung der Bildverarbeitungspipeline behindert. - Um das Risiko einer Unterbrechung der Datenstruktur zu minimieren, führen Sie ein Python-Skript aus, um die Integrität der Datenstruktur (kn_pipeline_check_mosaic.py) zu überprüfen, die auf GitHub (github.com/watkarbey/STPT_depo) verfügbar ist.
- Registrierung des gefärbten Bildes auf dem STPT-Bild
HINWEIS: Die nichtlineare Transformation des gefärbten Bildes in das STPT-Bild kann mit dem bUnwarpJ-Plugin von ImageJ durchgeführt werden.- Passen Sie die Größe der Bilder des Zielbildes (STPT) und des Quellbildes (gefärbt) so an, dass sie ungefähr gleich sind.
- Binarisieren Sie jedes Bild, um die Formen der Abschnitte zu visualisieren.
- Führen Sie bUnwarpJ aus. Denken Sie daran, die Option Transformationen speichern zu aktivieren.
- Starten Sie bUnwarpJ erneut. Wählen Sie das Originalbild vor der Binarisierung als Quellbild aus. Klicken Sie dann auf Load Elastic Transformation und wählen Sie die gespeicherte Transformation aus.
Ergebnisse
In dem hier verwendeten typischen Aufbau kann das gesamte Gehirn eines erwachsenen Weißbüschelaffen mit einer Auflösung von ~1,3 x 1,3 μm/Pixel mit einem Schnittintervall von 50 μm in etwa 1 Woche abgebildet werden (Abbildung 5). Dies entspricht ~650 koronalen Bildern in drei Kanälen nach dem Bild-Stitching. Mit einem 16-fach Objektiv (Nikon 16xW CFI75 LWD; NA = 0,80), beträgt das Gesichtsfeld einer Einzelaufnahme etwa 1 x 1 mm. Das Bild für die gesamte koronale Oberfläche erhält man durch Zusammenfügen dieser Aufnahmen (Abbildung 5A). Die Ausrichtung in Z-Richtung ist ausgezeichnet und ein gutes 3D-Bild wird durch einfaches Stapeln der koronalen Bilddaten erhalten (Abbildung 5B,E). Zur Standardisierung kann das STPT-Template für das Weißbüschelaffen-Gehirn11 für die 3D-3D-Registrierung verwendet werden (Abbildung 5F). Diese Datentransformation ist einer der Schlüsselaspekte der gesamten Neuroanatomie des Gehirns, in der einer interessierenden Region eine absolute Raumkoordinate zugewiesen wird, die unabhängig von der anatomischen Annotation ist. Sobald das Probengehirn im Standardraum registriert ist, kann man die interessierenden Subregionen für die weitere Analyse leicht herausnehmen (Abbildung 5F,G). Insbesondere können die kortikalen Regionen mit Hilfe eines vorgegebenen Parameters in eine flache Karte transformiert werden (Abbildung 5H).
Die bei der Bildgebung erzeugten Schnitte können für verschiedene histologische Zwecke verwendet werden. Wie in Abbildung 4A,B gezeigt, ergibt die Hinterlicht-Bildgebung ohne Färbung ein sehr ähnliches Muster wie die authentische Myelinfärbung. Dies kann eine hervorragende Alternative zur Myelinfärbung sein. Wenn das Gegenlichtbild vor der Nissl-Färbung aufgenommen wurde, kann derselbe Schnitt verwendet werden, um die Muster sowohl der Myelin- als auch der Nissl-Färbung zu erhalten, was nützliche Informationen zur Identifizierung von Bereichen und Schichten liefert (Abbildung 5C-E). Diese Schnitte können auch für immunologische Färbungen verwendet werden. In Abbildung 5J wurde der Abschnitt um das Injektionszentrum mit NeuN-Antikörpern gegengefärbt, um die Transduktionseffizienz des AAV-Virus abzuschätzen. Es ist auch eine gute Strategie, zusätzlich zu den fluoreszierenden Tracern nicht-fluoreszierende Tracer zu injizieren und diese nach der Schnittentnahme histologisch nachzuweisen. In unserer vorherigen Studie kombinierten wir anterograde grüne Tracer mit einem retrograden "cre"-Vektor, der später mit dem Anti-Cre-Antikörper10 nachgewiesen wurde. In einem Beispiel von Abbildung 6A-E wurde BDA in die kontralaterale Seite des grünen Tracers (Kleeblatt) injiziert und fluoreszierend detektiert. Beachten Sie, dass die roten BDA-Signale im TissueCyte-Bild registriert werden können, um in der gesamten Gehirnkoordinate lokalisiert zu werden. In einem anderen Beispiel von Abbildung 6F-I wurde smFP-myc in den parietalen Bereich injiziert (Abbildung 6G), während der grüne Tracer in den frontalen Bereich injiziert wurde. Auf diese Weise können mehrere Tracer in dasselbe Tier injiziert werden, ohne die Bildgebung zu beeinträchtigen. Ein großer Vorteil der Verwendung der STPT-Schnitte für zusätzliche Färbungen besteht darin, dass die Beziehung zwischen den fluoreszierenden und den nicht-fluoreszierenden Tracern für dasselbe Gehirn bestimmt werden kann. Auf diese Weise konnten wir die Reziprozität der kortikokortikalen Projektionen mit hoher Genauigkeitvon 10 bestimmen. Ein weiterer Vorteil ist, dass die 3D-Koordinaten der gefärbten Abschnitte auf die STPT-Daten und dann auf die Standardvorlage zurückgeführt werden können. Daher ist es möglicherweise nicht erforderlich, alle entnommenen Abschnitte für die Färbung zu verwenden. Zur besseren Interpretation können Abschnitte für die Färbung ausgewählt werden, um den STPT-Daten weiteren Kontext hinzuzufügen.
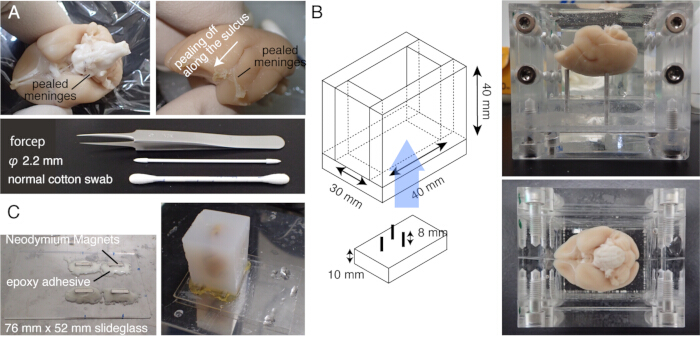
Abbildung 1: Probenvorbereitung für STPT. (A) Meninge-Entnahme mit Wattestäbchen. Die Hirnhäute, die den Hirnstamm umgeben, können mit einer feinen Pinzette entfernt werden. Die Hirnhäute, die das Weißbüschelaffengehirn umgeben, werden manuell durch Reiben mit Wattestäbchen entfernt. Das Foto zeigt die Hirnhäute, die aus dem Sulcus lateralis heraus geschält sind. (B) Die Acrylbox, die zum Einbetten von Agarose verwendet wird. Die Stifte sind beweglich und werden verwendet, um den Winkel des Gehirns so einzustellen, dass er nahe an der stereotaktisch fixierten Position20 liegt. (C) Ein magnetischer Objektträger aus 76 mm x 52 mm großem Gleitglas und vier Neodym-Magneten, die mit Epoxidkleber befestigt sind. Der Agaroseblock wird mit Sekundenkleber auf dem Magnettisch befestigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
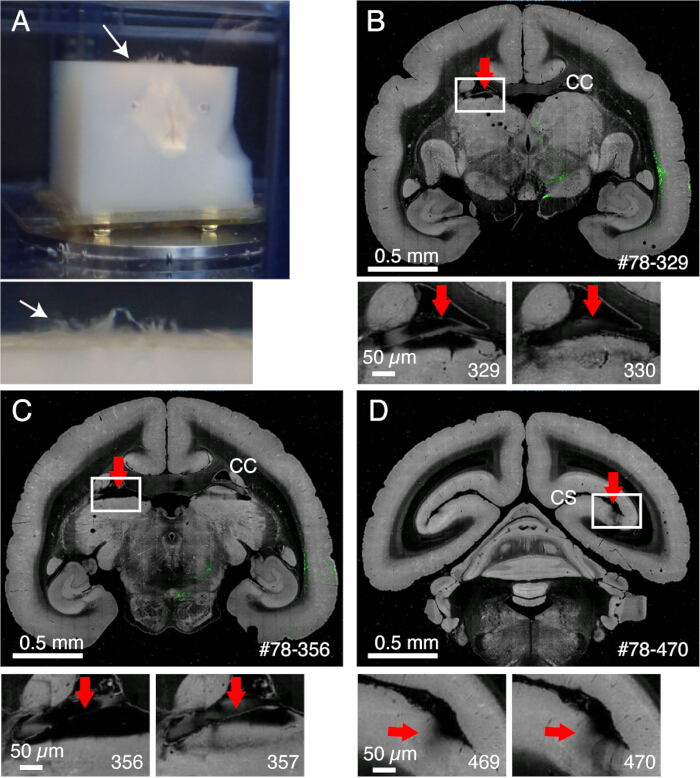
Abbildung 2: Der Einfluss von Hirnhäuten auf die Bildgebung. (A) Hirnhäute, die ungeschnitten bleiben, schwimmen nach oben, wie durch den weißen Pfeil dargestellt. In diesem Beispiel ist eine ausgedehnte Vorwölbung der Hirnhäute zu sehen, weil sie vor der Agarose-Einbettung nicht entfernt wurden. In der Regel bleiben Hirnhäute nur in einigen schwierigen Regionen ungeschnitten. (B) Ein Beispiel für schlechtes Schneiden aufgrund des Vorhandenseins von Hirnhäuten zwischen dem Corpus callosum und dem oberen Teil des Thalamus. In diesem Fall führte das schlechte Schneiden zum Wechsel eines tiefen Schnitts (329) und eines relativ normalen Schnitts (330). #78-329 steht für Probe Nr. 78 Abschnitt Nr. 329 im Brain/MINDS Datenportal (C) Ein weiteres Beispiel für schlechte Bildgebung. Im schlimmsten Fall kann sich der Nucleus pulvinaris, der durch den roten Pfeil gekennzeichnet ist, vollständig lösen. Maßstabsleisten: 0,5 mm (obere Platte), 0,5 μm (untere Platte). (D) Ein weiteres Beispiel für schlechte Bildgebung. Die Hirnhäute tief im Sulcus calcarine sind schwer zu entfernen. Die Schatten, die in den Abschnitten 469 und 470 zu sehen sind, werden durch die schwimmenden Hirnhäute verursacht, die unter das Ziel kamen. Maßstabsleisten: 0,5 mm (obere Platte), 0,5 μm (untere Platte). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
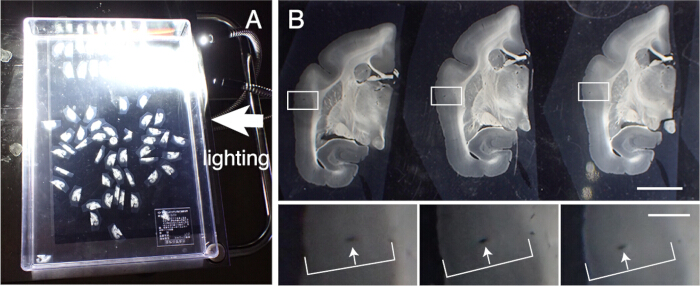
Abbildung 3: Ausrichtung der Gewebeschnitte in der richtigen Reihenfolge. (A) Bis zu 50 Weißbüschelaffen-Koronalabschnitte können in einem Plastikbehälter (31 cm x 22,5 cm) korrekt ausgerichtet werden. Um die detaillierten Strukturen zu visualisieren, wird der Container auf schwarzes Papier gelegt und von der Seite beleuchtet. Diese Abschnitte werden zunächst grob in der Reihenfolge ausgerichtet und dann einer genauen Ausrichtung unterzogen. (B) Bei der präzisen Ausrichtung wird das Blutgefäß als Marker verwendet. Die Großhirnrinde enthält viele Blutgefäße, die vertikal durch die kortikalen Schichten verlaufen. Sie sind als längliche Löcher gekennzeichnet, die ihre Position innerhalb der kortikalen Schichten systematisch ändern (weiße Pfeile). Mit dieser Methode können auch Abschnitte mit 50 μm Abstand exakt ausgerichtet werden. Diese Blutgefäßlöcher können zur Bestätigung in den Bildern des SPTP-Schnitts identifiziert werden. Maßstabsleiste: 5 mm (obere Platte), 1 mm (untere Platte). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
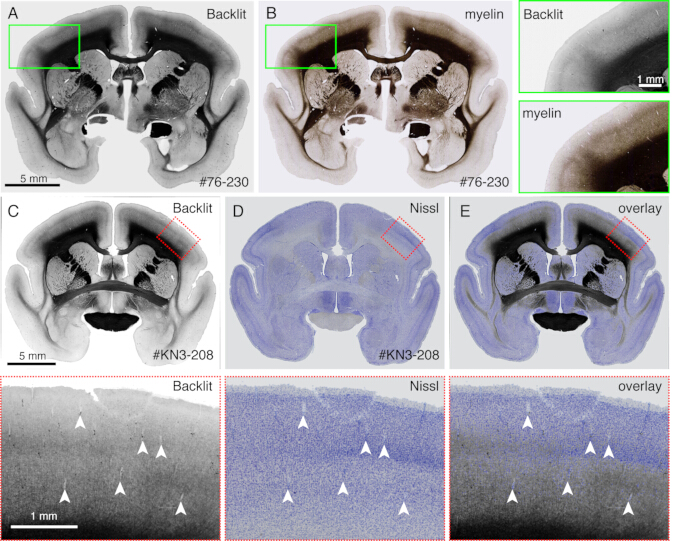
Abbildung 4: Gegenlichtbild als Ersatz für die Myelinfärbung. (A,B) Der identische Ausschnitt wurde für die Hinterlichtbildgebung und die Myelinfärbung verwendet. Zunächst wurde der Schnitt auf das Objektträgerglas aufgebracht, getrocknet, mit PBS rehydriert, mit Deckglas überzogen und mit einem Lichtmikroskop abgebildet (Table of Materials). Nach dem Entfernen des Deckglases wurde derselbe Schnitt für die Myelinfärbung21 verwendet und mit einem Fluoreszenzmikroskop abgebildet. Das Backlit-Image wurde mit dem bUnwarpJ-Plugin von ImageJ auf dem Myelin-Image registriert. Die grünen Felder zeigen die vergrößerten Ansichten der einzelnen Bilder. Beachten Sie, dass diese Bilder fast identische Muster zeigen, mit der Ausnahme, dass die Myelinfärbung die fibrösen Strukturen besser sichtbar macht. (C-E) Der identische Ausschnitt wurde für die Hinterlichtbildgebung und die Nissl-Färbung verwendet. Die niedrigschwellige Maske für das hinterleuchtete Bild wurde zuerst mit der niedrigschwelligen Maske für das Nissl-Bild mit dem bUnwarpJ-Plugin registriert, und das Originalbild wurde mit demselben Parameter transformiert. Beachten Sie, dass die Blutgefäße (weiße Pfeilspitzen) zwischen den beiden Bildern gut aufeinander abgestimmt sind. Da diese beiden Bilder für denselben Ausschnitt sind, ist die Übereinstimmung nahezu perfekt, und man kann die Myelin- und Nissl-Muster direkt vergleichen, um die kortikalen Schichten zu identifizieren. Maßstabsleisten: 5 mm (Paneele A-E). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
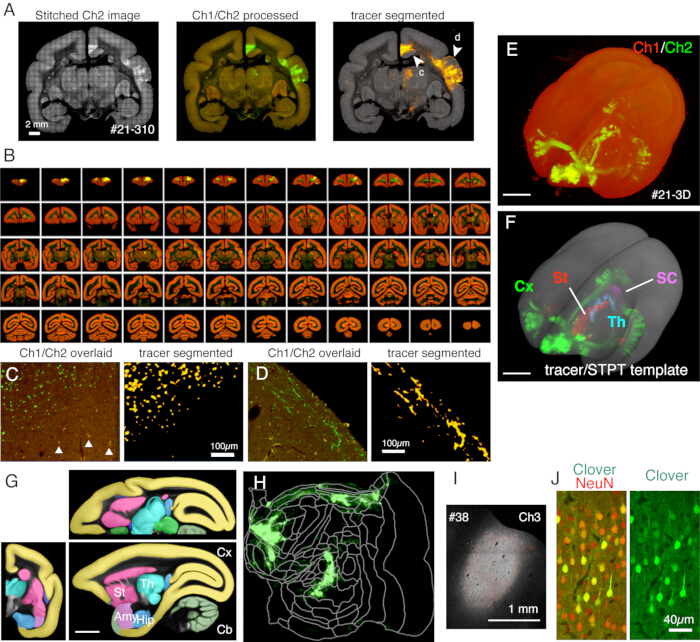
Abbildung 5: Typisches Ergebnis der STPT-Bildgebung. (A) Eine einfache Kachelung von Ch2 (grün) Bildern eines Beispielschnitts (Abschnitt 310 von Beispiel #21). Ohne Hintergrundkorrektur sind die Ränder für jede Kachel sichtbar. In der mittleren Spalte wurden die Kacheln mit Hintergrundkorrektur für Ch1 (rot) bzw. Ch2 (grün) zusammengefügt. Um die Signale von Lipofuszin zu reduzieren (siehe Panel C), wurden die Ch1-Signale von Ch2 subtrahiert und grün dargestellt. Um die Lipofuszinsignale von Ch1 zu reduzieren, wurde der Befehl "Outiler entfernen..." vor dem Stitching durch ImageJ verwendet. In der rechten Spalte wurden die Tracersignale nach Bildverarbeitungspipeline11 segmentiert. Die Pfeilspitzen (c, d) zeigen die Positionen, an denen die vergrößerten Ansichten in den Feldern C und D angezeigt werden. Maßstabsleiste: 2 mm. (B) Übersicht der Serienschnitte für Beispiel #21. STPT generierte für diese Probe 635 hochauflösende koronale Bilder. (C) Eine Ansicht mit hoher Vergrößerung, die durch die Pfeilspitze c in Feld A dargestellt ist. Dabei handelt es sich um eine einfache Überlagerung von Ch1 (rot) und Ch2 (grün) ohne weitere Verarbeitung. Die Dreiecke zeigen Lipofuszin-Fluoreszenzsignale, die ein weit verbreitetes Spektrum aufweisen. Der Tracer-Segmentierungsalgorithmus unterscheidet die Tracer-Signale trotz einer sehr ähnlichen Form genau vom Lipofuszin-Hintergrund (rechtes Bild). Maßstabsleiste: 100 μm. (D) Ein weiteres Beispiel für eine Ansicht mit hoher Vergrößerung. Beachten Sie, dass feine Axonfasern in Schicht 1 gut sichtbar sein können. Maßstabsleiste: 100 μm. (E) Die 3D-Rekonstruktion von Original-STPT-Bildern. Die 635 niedrig aufgelösten koronalen Bilder, wie sie in Panel B gezeigt werden, wurden als tiff-Stack für die 3D-Visualisierung mit Fluorender22 verwendet. Maßstabsbalken: 5 mm. (F) Die 3D-Rekonstruktion der segmentierten Tracersignale, die in der STPT-Vorlage (grau) registriert sind. Die Tracer-Signale in verschiedenen Hirnregionen wurden durch unterschiedliche Farben dargestellt. Diese Regionen wurden unter Verwendung der in Feld G gezeigten Annotation ausgeschnitten. Maßstabsleiste: 5 mm. (G) STPT-Schablone, die mit der Annotation verschiedener Hirnregionen überlagert ist. Maßstabsbalken: 5 mm. (H) Das in Feld F gezeigte kortikale Tracersignal wurde in Form einer Flatmap dargestellt. (I) Identifizierung der Injektionsstelle durch die Ch3-Fluoreszenz, die weniger empfindlich auf die Tracerfluoreszenz reagiert und ungesättigt bleibt. Maßstab: 1 mm. (J) Die Färbung des Abschnitts um das Injektionszentrum mit NeuN-Antikörpern zeigte, dass etwa 30 % der Neuronen eine starke Expression von Kleegrünfluoreszenz zeigten. Maßstab: 40 μm. Abkürzungen: Cx; Kortex, St; striatum, Th; Thalamus, SC; Colliculus superior. Amy; amygdala, Hüfte; Hippocampus, Cb; Kleinhirn. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
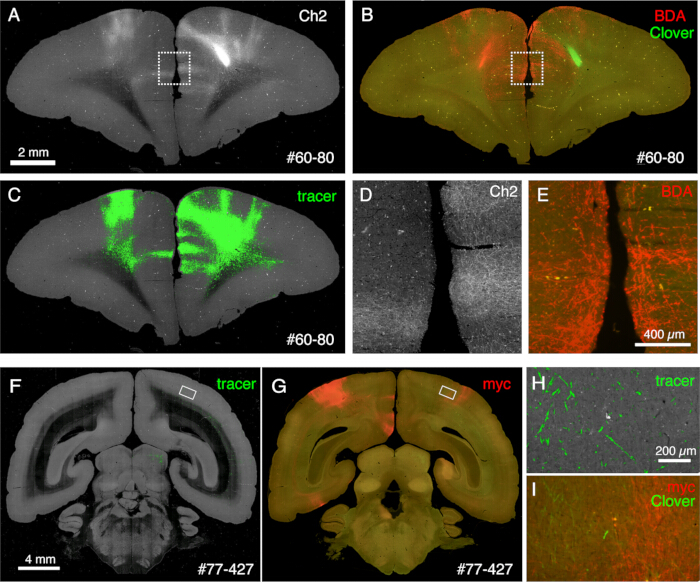
Abbildung 6: Post-STPT-Histologie mit mehreren nicht-fluoreszierenden Tracern. (A-C) Vergleich des STPT-Bildes mit dem BDA-gefärbten Bild. In dieser Probe wird BDA in die kontralaterale Seite der Clover-Injektion injiziert. Die Felder A und C zeigen das Ch2-Bild und die Tracer-Segmentierung der STPT-Daten. Panel B zeigt die Post-STPT-Färbung für BDA. Die Kleefluoreszenz wird durch die Methanolbehandlung des Schnitts verringert. Maßstabsleiste: 2 mm. (D,E) Die gestrichelten Kästchen in den Feldern A und B sind vergrößert. Maßstabsbalken: 100 μm. (F,G) Vergleich des STPT-Bildes mit der Färbung von Anti-Myc-Tag-Antikörpern. In dieser Probe erfolgt die Clover-Injektion in den PFC, während das AAV-smFP-myc in den kontralateralen parietalen Kortex injiziert wird. Die weißen Rechtecke sind in den Feldern H und I vergrößert. Maßstabsbalken: 4 mm. (H) Die Tracer-Segmentierung ist grün dargestellt. Maßstab: 200 μm. (I) Die Myc-Färbung ist rot dargestellt. Die Kleefluoreszenz ist grün dargestellt. Maßstab: 200 μm. Die grünen Signale in den Feldern H und I sind in einer ähnlichen Position, aber nicht identisch, da STPT nur ~10 μm optischen Abschnitt abruft. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| BDA-Protokoll für die Fluoreszenzfärbung | |
| Agarose entfernen | |
| TBS-Wäsche | ca. 10 min (2x) |
| 1% H2O2 in der Dent-Lösung | ca. 10 Minuten |
| TBS-Wäsche | Kurz |
| 0,5 % TNB-Blockierung | 1 h |
| StAvHRP (1:4000) in TNB | 2 über Nacht |
| TNT-Wäsche | ca. 10 min (3x) |
| TSA Biotin (1:4000) in 0,1 M Borat (pH8,5) + 0,003 % H2O2 | ca. 2 h |
| TNT-Wäsche | ca. 10 min (3x) |
| Cy3-streptavidin (1:1000) in TNT | 3 h |
| TNT-Wäsche | ca. 10 min (2x) |
| TBS-Wäsche | Bewahren Sie den Abschnitt bis zur Montage auf |
| Sektion mit einem lichtbeständigen Einbettmedium auf Diaglas montieren | |
| Anti-Myc-Fluoreszenz-Färbeprotokoll | |
| Agarose entfernen | |
| TBS-Wäsche | ca. 10 min (2x) |
| Sperren im IB | 1 h |
| Anti-Myc (1:4000) in IB | 2 über Nacht |
| TNT-Wäsche | ca. 10 min (3x) |
| Anti-Maus Cy3 (1:1000) in TNT | 3 h |
| TNT-Wäsche | ca. 10 min (2x) |
| TBS-Wäsche | Bewahren Sie den Abschnitt bis zur Montage auf |
| Sektion mit einem lichtbeständigen Einbettmedium auf Diaglas montieren | |
| Puffer/Lösungen | Zusammensetzung |
| 0,5 % TNB | 0,5 % TSA-Blockierungsreagenz in TS7.5 |
| Die Lösung von Dent | 20 % DMSO, 80 % Methanol |
| Immersionspuffer (IB) | 10 % FBS, 2 % BSA, 0,5 % TritonX100 in TBS |
| TBS (Tris-gepufferte Kochsalzlösung) | 25 mM Tris, 137 mM NaCl, 2,7 mM KCl (pH 7,4) |
| TNT | 0,05 % Tween20 in TS7,5 |
| TS7.5 | 0,1 M TRIS-HCl, pH 7,5, 0,15 M NaCl |
Tabelle 1: BDA-Fluoreszenzfärbung und Anti-Myc-Fluoreszenzfärbeprotokoll
| Plasmid für AAV-Tracer | Addgene Nein. | Empfohlener Antikörper | Empfohlene Verdünnung | Erwartetes Ergebnis |
| pAAV-EF1_Cre | 201198 | Millipore-Klon 2D8 | 1:1000 | gut für die Zellen |
| pAAVCam1.3_smFP_Myc | 201205 | MBL M192 My3 (Maus) | 1:4000 | Ausgezeichnet |
| pAAVCam1.3_smFP_HA | 201206 | CST C29F4 (Kaninchen) | 1:1000 | gut für die Zellen |
| pAAVCam1.3_smFP_FLAG | 201207 | MBL PM020B (Kaninchen) | 1:1000 | OKAY |
| AAVTRE3_smFP_Myc | 201208 | |||
| AAVTRE3_smFP_HA | 201209 | |||
| AAVTRE3_smFP_FLAG | 201210 |
Tabelle 2: Liste der Addgene-Plasmide, die für die Herstellung von nicht-fluoreszierenden Tracern zur Verfügung stehen. Das cre-Konstrukt zielt auf den Zellkern ab und eignet sich für retrograde Abtastung, die von AAV2-Retro umhüllt ist. smFP_HA Konstrukt ist gut für die Erkennung von Zellkörpern (und retrograd).
Ergänzendes Video: Mikroskopische Ansicht der Entfernung von Hirnhäuten. Bitte klicken Sie hier, um dieses Video herunterzuladen.
Diskussion
In diesem Artikel wurden die praktischen Lösungen für den Umgang mit Weißbüschelaffengehirnen für die Ganzhirnverarbeitung sowie zusätzliche histologische Techniken erläutert, die den Nutzen der STPT-Technik erhöhen. Die Stärke der "Neuroanatomie des gesamten Gehirns" mit STPT besteht darin, dass Sie die 3D-Koordinaten jeder interessierenden Region erhalten können, unabhängig davon, ob sie anatomisch annotiert ist oder nicht. Durch die hochpräzise 3D-zu-3D-Registrierung ist es möglich, diese Koordinaten in eine Standardvorlage für die Überlagerung mehrerer Datensätze umzuwandeln. Auf diese Weise dient das Standard-Template als Medium der Datenintegration. Dies war ein wesentlicher Aspekt unseres Projekts zur Kartierung des präfrontalen Kortex (PFC)10, bei dem Daten von vielen Personen analysiert wurden. Darüber hinaus können die Informationen, die dem Standard-Template zugeordnet werden, mit verschiedenen Daten verglichen werden, die bereits kartiert wurden, sei es Nissl, Myelinmuster, Tracerdaten, MRT-Daten (einschließlich Diffusions-MRT) oder anatomische Annotationen11. Wichtig ist, dass sie auch mit zukünftigen Daten verglichen werden können, die durch noch nicht entstehende Technologien gewonnen werden. Derzeit gibt es mehrere Templates für das Weißbüschelaffengehirn, die auf der Nissl-Färbung23, MRT 23,24,25,26,27,28 und STPT11 basieren. Sie können jedoch auf der Grundlage von Bildkontrasten und vorberechneten Parametern in die Koordinaten der jeweils anderen transformiert werden11. Die Datenintegration auf der gesamten Gehirnskala über Studien hinweg trägt zu einem besseren Verständnis des Gehirns als System bei. Voraussetzung dafür, dass diese Strategie funktioniert, ist eine zuverlässige Datenerfassung im gesamten Gehirn. Im Folgenden werden kritische Schritte und mögliche Probleme im Zusammenhang mit dem aktuellen Protokoll erläutert.
Eines der anfälligsten Verfahren der STPT-Bildgebung ist das Tissue Slicing. Wie oben erwähnt, bleiben die Hirnhäute oft ungeschnitten und können das Schneiden stören. Insbesondere der Nucleus pulvinaris und der Colliculus superior sind die beiden am stärksten betroffenen Hirnregionen: Je nach Gründlichkeit der Meningeentfernung und Einbettung in Agarose können sie beim Schneiden aus dem Gewebeblock entfernt werden. Diese tiefen Regionen sind von außen schwer zugänglich und können bei der Hirnhautentfernung leicht brechen. Eine sorgfältige, aber gründliche Entfernung der Hirnhaut ist entscheidend für eine erfolgreiche Bildgebung. Ein weiteres Problem ist das Zurückklappen von geschnittenen Schnitten auf den Gewebeblock, was manchmal auftritt, wenn die Schnitte auch nach dem Schneiden noch haften. Sie kann minimiert werden, indem der Block so geformt wird, dass die Klinge ganz am Ende schräg schneidet.
Durch die Bildgebung tief in den Gewebeblock hinein vermeidet STPT die Unebenheit seiner Oberfläche. Während die Fluoreszenzsignale die kortikale Region recht leicht passieren können, sind sie in den myelinisierten Regionen stark vermindert. Daher muss die Abbildungstiefe sorgfältig bestimmt werden, um ein Gleichgewicht zwischen der konsistenten Abbildung über die gesamte Blockoberfläche und der Helligkeit der Signale in der myelinreichen Region, wie z. B. der weißen Substanz, herzustellen. In dem hier verwendeten Aufbau zielen wir in der Regel auf 25-35 μm von der Oberfläche ab. Es muss auch darauf geachtet werden, dass die Schnittfläche nach langer Lagerung ungleichmäßig schrumpfen kann. Um den Bildgebungsbereich zu minimieren, teilen wir die Bildgebungssitzung in 20-30 Läufe mit unterschiedlichen Stufeneinstellungen für 5-6 Tage auf. Wir machen entweder das Intervall zwischen den Läufen weniger als 2 h oder bestätigen die Oberflächentiefe und passen die Bühnenhöhe vor jedem Lauf an.
In diesem Protokoll wurde das BDA-Signal durch die TSA-Methode verstärkt. Diese Methode ist hocheffektiv und kann anterograd transportierte BDA-Signale auch bei relativ geringer Auflösung detektieren (z.B. Abbildung 6B). TSA-Biotin ist von Akoya Biosciences im Handel erhältlich, aber die hausgemachte Lösung zeigt eine viel bessere Verbesserung. Auf der anderen Seite erfordert die Verdünnung des Antikörpers und der Lösung eine sorgfältige Anpassung, um das optimale Ergebnis zu erzielen. Die Vorbehandlung des Abschnitts mit einer Methanollösung ist entscheidend. Ohne Vorbehandlung werden die BDA-Signale in den myelinisierten Axonen kaum nachgewiesen.
Wenn ein anterograder Tracer verwendet wird, ist es aufgrund der Sättigung der Fluoreszenzsignale oft schwierig, die genaue Injektionsstelle zu identifizieren. In dem gesamten Gewebebildgebungssystem, das in dieser Studie verwendet wurde, verwenden wir den blauen Kanal, um die infizierten Zellen zu identifizieren (Abbildung 5I). Selbst wenn der rote und der grüne Kanal gesättigt sind, ist in der Regel jedes einzelne infizierte Neuron im blauen Kanal nachweisbar. Das gilt auch für den Allen Mouse Brain Connectivity Atlas1. Die Untersuchung der Ursprungszellen ist wichtig, da die Infektion manchmal nur bestimmte Schichten betrifft. Für den Mouse Brain Connectivity Atlas sind solche Teilinfektionen recht häufig angetroffen, vielleicht aufgrund der Anwendung der Iontophorese-Methode29. Die laterale Ausbreitung der viralen Tracer kann je nach Injektion mehr oder weniger variabel sein. Diese Variabilität kann sich möglicherweise auf das Ergebnis der Ablaufverfolgung auswirken und erfordert eine sorgfältige Normalisierung.
Die erfolgreiche Registrierung des erhaltenen 3D-Bildes in der Standardvorlage ist ein Schlüsselprozess der gesamten Neuroanatomie des Gehirns. Die Registrierung des STPT-Bildes in der STPT-Vorlage kann ziemlich genau sein, und wir haben nur Abweichungen von wenigen Voxeln (50 μm isokubisch) für die Ränder mit einem hohen Bildkontrastbeobachtet 10. Dennoch gibt es eine Grenze für die Möglichkeiten der Registrierung. Aufgrund des Prozesses der Meningeentfernung weisen die STPT-Proben in der Regel Lücken zwischen den Hemisphären und zwischen der Hirnrinde und dem Mittelhirn/Hinterhirn auf, während das Hirngewebe in den in vivo MRT-Bildern dicht gepackt ist. Solche Unterschiede lassen sich nur schwer durch Registrierung ausgleichen. Die Weißbüschelaffenrinde ist meist frei von Sulki, aber der intraparietale Sulcus ist bei einigen Individuen sehr tief. Ein solcher Sulci geht bei der Registrierung verloren (es kommt zu einer Top-to-Top-Verschmelzung). Obwohl die Registrierung eine leistungsstarke Technik ist, ist es notwendig, auf die Rohdaten zurückzugreifen, um das erhaltene Ergebnis zu bestätigen.
Die Erzeugung massiver Bilddaten ist sowohl eine Stärke als auch eine Einschränkung dieser Technik. Dies erhöht zwar die Vollständigkeit des Datensatzes, erfordert jedoch eine sorgfältige Verwaltung der erfassten Daten und die Entwicklung einer automatisierten Bildverarbeitungspipeline für eine effiziente Dateninterpretation. In Zukunft kann der Einsatz von generativer Künstlicher Intelligenz (KI) beim Aufbau von Bildverarbeitungspipelines diesen Prozess deutlich vereinfachen. Eine systematische Bildgebung des gesamten Gehirns wurde auch mit auf Objektträgerscannern basierenden Methoden durchgeführt21,30. Im Vergleich zu solchen Methoden erfordert STPT keine zusätzlichen Berechnungen für die 3D-Rekonstruktion. In Kombination mit optischen Schnitten haben wir gezeigt, dass STPT das Potenzial hat, sogar Axonsegmente über die Schnitte31 hinweg zu rekonstruieren. Mit der weiteren Kombination von Gewebe-Clearance-Techniken entwickelten Economo et al. eine Methode, um die Gesamtheit spärlich markierter Neuronen abzubilden32,33. Die neuesten Versionen von TissueCyte bieten nun Optionen für einen zusätzlichen Laser zur Verbesserung der Anregung von rot fluoreszierenden Proteinen oder eine Schnitterfassungseinheit für die automatische Wiederherstellung von Schnitten. Mit diesen Fortschritten wird der Ganzhirnansatz effizienter und bietet eine Grundlage für ein umfassendes Verständnis des Gehirns von Primaten, einschließlich des menschlichen Gehirns.
Offenlegungen
Die Autoren haben nichts offenzulegen.
Danksagungen
Wir danken dem technischen Personal in den Labor- und Tiereinrichtungen (RRD) von Yamamori für ihre Hilfe. Wir danken dem RIKEN CBS-Olympus Collaboration Center für die technische Unterstützung bei der konfokalen Bildaufnahme. Diese Arbeit wurde unterstützt durch das Program for Scientific Research on Innovative Areas (Fördernummer 22123009) von MEXT, Japan, durch Brain/MINDS und Brain/MINDS2.0 von AMED, Japan (JP15dm0207001, JP23wm0625001 und JP24wm0625218) und durch JSPS KAKENHI Fördernummer 24K09678 an A.W.
Materialien
| Name | Company | Catalog Number | Comments |
| Agarose type I | Sigma | A6013 | |
| All-in-one fluorescence microscope | Keyence | BZ-X series | This study used BZ-X710 |
| anti-mouse Cy3 | Jackson Immuno Research Laboratories, Inc. | 115-165-003 | |
| anti-myc antibody | MBL | M192 | mouse monoclonal |
| Butorphanol | Meiji Animal Health | Vetorphale | |
| Ceramic blade | TissueVision | N/A | |
| Collagenase type I | Wako Chemical | #031-17601 | Aliquot into 100 µL x 10 of 100 mg/mL in PBS |
| Cotton swab (HUBY-340) | HUBY | BB-013SP | micro cotton swabs |
| Custom-made chamber | N/A | N/A | The chamber consist of four side plates and one bottom plates, all made of 1 cm thick acrylic plates, which can be securely fastened together using screw bolts. An additional acrylic plate is placed inside the chamber, with three metal poles inserted through it to hold the marmoset brain in an inverted position. |
| Cy3-Streptavidin | Jackson Immuno Research Laboratories, Inc. | 016-160-084 | |
| Dumont #5 forceps, Inox | N/A | N/A | |
| Epoxy instant mix | Locktite | N/A | |
| FIJI | NIH | https://imagej.net/software/fiji/downloads | |
| kn_pipeline_check_mosaic.py | N/A | N/A | github.com/watkarbey/STPT_depo; This is a checking script for TissueCyte data. |
| Medetomidine | Zenoaq | Domitor | |
| Midazolam | Sandoz | N/A | |
| NaBH4 | Sigma | #452882 | |
| NaIO4 | Sigma | S1878 | |
| Perfusion needle | Natsume Seisakusho Co., Ltd | KN-348, 20G-50 | |
| Slideglass (76 mm x 52 mm) | Matsunami | S9111 | |
| StAvHRP | Jackson Immuno Research Laboratories, Inc. | 016-030-084 | HRP-streptavidin (titration needs to be carefully determined) |
| TissueCyte1000 | TissueVision | https://www.tissuevision.com/tissuecyte | |
| TSA blocking reagent | Kiko Tech | FP1012 | Refer to TSA-biotin kit (Akoya Biosciences) |
| TSA-biotin | House-made | N/A | See Okamoto et al (ref. 16) |
| VECTASHIELD HardSet Mounting Medium | Vector laboratories Inc | N/A | Antifade mounting medium |
Referenzen
- Oh, S. W., Harris, J. A., Ng, L., Winslow, B., Cain, N., et al. A mesoscale connectome of the mouse brain. Nature. 508 (7495), 207-214 (2014).
- Ragan, T., et al. Serial two-photon tomography for automated ex vivo mouse brain imaging. Nat Methods. 9 (3), 255-258 (2012).
- Liang, Z., Arefin, T. M., Lee, C. H., Zhang, J. Using mesoscopic tract-tracing data to guide the estimation of fiber orientation distributions in the mouse brain from diffusion MRI. Neuroimage. 270, 119999(2023).
- Watakabe, A., Hirokawa, J. Cortical networks of the mouse brain elaborate within the gray matter. Brain Struct Funct. 223 (8), 3633-3652 (2018).
- Ypma, R. J. F., Bullmore, E. T. Statistical analysis of tract-tracing experiments demonstrates a dense, complex cortical network in the mouse. PLoS Comput Biol. 12 (9), e1005104(2016).
- Wang, Q., et al. The Allen mouse brain common coordinate framework: A 3D reference atlas. Cell. 181 (4), 936-953.e20 (2020).
- Köbbert, C., Apps, R., Bechmann, I., Lanciego, J. L., Mey, J., Thanos, S. Current concepts in neuroanatomical tracing. Prog Neurobiol. 62 (4), 327-351 (2000).
- Lanciego, J. L., Wouterlood, F. G. Neuroanatomical tract-tracing techniques that did go viral. Brain Struct Funct. 225 (4), 1193-1224 (2020).
- Bakker, R., Wachtler, T., Diesmann, M. CoCoMac 2.0 and the future of tract-tracing databases. Front Neuroinform. 6, (2012).
- Watakabe, A., et al. Local and long-distance organization of prefrontal cortex circuits in the marmoset brain. Neuron. 111 (14), 2258-2273.e10 (2023).
- Skibbe, H., et al. The Brain/MINDS marmoset connectivity resource: An open-access platform for cellular-level tracing and tractography in the primate brain. PLoS Biol. 21 (6), e3002158(2023).
- Mitchell, J. F., Leopold, D. A. The marmoset monkey as a model for visual neuroscience. Neurosci Res. 93, 20-46 (2015).
- Okano, H. Current status of and perspectives on the application of marmosets in neurobiology. Annu Rev Neurosci. 44, 27-48 (2021).
- Watakabe, A., et al. Comparative analyses of adeno-associated viral vector serotypes 1, 2, 5, 8 and 9 in marmoset, mouse and macaque cerebral cortex. Neurosci Res. 93, 144-157 (2015).
- Hata, J., et al. Multi-modal brain magnetic resonance imaging database covering marmosets with a wide age range. Sci Data. 10, 221(2023).
- Liwang, J. K., Bennett, H. C., Pi, H. -J., Kim, Y. Protocol for using serial two-photon tomography to map cell types and cerebrovasculature at single-cell resolution in the whole adult mouse brain. STAR Protoc. 4 (1), 102048(2023).
- Okamoto, S., et al. Exclusive labeling of direct and indirect pathway neurons in the mouse neostriatum by an adeno-associated virus vector with Cre/lox system. STAR Protoc. 2 (1), 100230(2021).
- Viswanathan, S., et al. High-performance probes for light and electron microscopy. Nat Methods. 12 (6), 568-576 (2015).
- Kuan, L., et al. Neuroinformatics of the Allen mouse brain connectivity atlas. Methods. 73, 4-17 (2015).
- Paxinos, G., Watson, C., Petrides, M., Rosa, M., Tokuno, H. The Marmoset Brain in Stereotaxic Coordinates. , Academic Press. London, Waltham, MA. (2012).
- Abe, H., et al. 3D reconstruction of brain section images for creating axonal projection maps in marmosets. J Neurosci Methods. 286, 102-113 (2017).
- Wan, Y., et al. FluoRender: Joint freehand segmentation and visualization for many-channel fluorescence data analysis. BMC Bioinformatics. 18 (1), 280(2017).
- Majka, P., et al. Towards a comprehensive atlas of cortical connections in a primate brain: Mapping tracer injection studies of the common marmoset into a reference digital template. J Comp Neurol. 524 (11), 2161-2181 (2016).
- Liu, C., Yen, C. C. -C., Szczupak, D., Tian, X., Glen, D., Silva, A. C. Marmoset brain mapping V3: Population multi-modal standard volumetric and surface-based templates. Neuroimage. 226, 117620(2021).
- Liu, C., et al. A digital 3D atlas of the marmoset brain based on multi-modal MRI. Neuroimage. 169, 106-116 (2018).
- Liu, C., et al. A resource for the detailed 3D mapping of white matter pathways in the marmoset brain. Nat Neurosci. 23 (2), 271-280 (2020).
- Woodward, A., et al. The Brain/MINDS 3D digital marmoset brain atlas. Sci Data. 5, 180009(2018).
- Saleem, K. S., Avram, A. V., Glen, D., Schram, V., Basser, P. J. The subcortical atlas of the marmoset ("SAM") monkey based on high-resolution MRI and histology. Cereb Cortex. 34 (4), bhae120(2024).
- Watakabe, A., Hirokawa, J. Cortical networks of the mouse brain elaborate within the gray matter. Brain Struct Funct. 223 (8), 3633-3652 (2018).
- Lin, M. K., et al. A high-throughput neurohistological pipeline for brain-wide mesoscale connectivity mapping of the common marmoset. eLife. 8, e40042(2019).
- Skibbe, H., et al. PAT-probabilistic axon tracking for densely labeled neurons in large 3-D micrographs. IEEE Trans Med Imaging. 38 (1), 69-78 (2019).
- Economo, M. N., et al. A platform for brain-wide imaging and reconstruction of individual neurons. eLife. 5, e10566(2016).
- Winnubst, J., et al. Reconstruction of 1,000 projection neurons reveals new cell types and organization of long-range connectivity in the mouse brain. Cell. 179 (1), 268-281.e13 (2019).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten