Method Article
Tomografia seriale a due fotoni dell'intero cervello dell'uistitì per analisi neuroanatomiche
In questo articolo
Riepilogo
L'imaging con tomografia seriale a due fotoni (STPT) è una tecnica per visualizzare una massa di tessuto nella sua forma tridimensionale combinando l'imaging a due fotoni con il controllo automatico dello stadio e lo slicing del microtomo. Qui descriviamo un protocollo per implementarlo per i cervelli degli uistitì per comprendere meglio le loro caratteristiche strutturali.
Abstract
La tomografia seriale a due fotoni (STPT) è una tecnica per visualizzare una massa di tessuto nella sua forma tridimensionale combinando l'imaging a due fotoni con il controllo automatico dello stadio e il taglio del microtomo. Abbiamo implementato con successo questa tecnica per tracciare le proiezioni assonali nel cervello dell'uistitì. Qui vengono descritte le dettagliate procedure sperimentali che hanno portato a un imaging volumetrico affidabile dell'intero cervello dell'uistitì. Un processo chiave per il successo dell'imaging è stata la rimozione delle meningi che circondano il cervello, che interferisce con l'affettatura. Un grande vantaggio di questa metodologia è che le sezioni affettate possono essere utilizzate per ulteriori colorazioni. Nella configurazione originale, le sezioni affettate vengono strapazzate nel bagno d'acqua. Queste sezioni possono essere correttamente allineate nel loro ordine originale in base ai modelli dei vasi sanguigni nella corteccia. Un esempio di istologia efficace è la visualizzazione della struttura della mielina mediante semplice riflessione della luce, che può essere combinata con la colorazione Nissl per definire i bordi anatomici. Queste sezioni possono essere utilizzate anche per la rilevazione immunologica di traccianti anterogradi e retrogradi non fluorescenti, che possono essere registrati nei dati STPT per la stratificazione di più dati.
Introduzione
Negli studi neuroanatomici, i ricercatori si trovano di fronte alla necessità di osservare strutture di ordine micrometrico (ad esempio, assoni e bottoni) nel contesto dell'intero cervello. Questo difficile compito è stato tipicamente affrontato ispezionando visivamente le sezioni seriali e cercando la regione di interesse per l'imaging dettagliato, le analisi e la registrazione fotografica. Con il progresso tecnologico, tuttavia, sta diventando possibile visualizzare l'intero cervello ad alta risoluzione per l'analisi dell'intero cervello. In uno studio innovativo di Oh et al.1, centinaia di cervelli di topo hanno ricevuto iniezioni di traccianti per l'analisi connettomica, che sono state elaborate mediante la tecnica di imaging della tomografia seriale a due fotoni (STPT)2. La caratteristica di questo studio è stata quella di aver selezionato traccianti anterogradi per quantificare la "connettività". Mentre i traccianti anterogradi forniscono informazioni spaziali molto dettagliate sulla distribuzione assonale, i neuroanatomisti si sono affidati a una laboriosa segmentazione manuale per la sua analisi. Automatizzando varie procedure analitiche, tra cui questa fase di segmentazione, sono riusciti nella "produzione di massa" di dati traccianti ad alta risoluzione pronti all'uso per usi multiuso. L'efficacia del loro approccio è evidente, data una varietà di studi che hanno utilizzato i loro dati traccianti 3,4,5 e il loro cervello standard6.
Storicamente, la connettività neurale dei cervelli di primati non umani ha attirato l'attenzione di molti neuroanatomisti dallo sviluppo del metodo di degenerazione negli anni '50, ai metodi di trasporto di sostanze anterogrado/retrogrado negli anni '70 fino all'attuale strategia virale 7,8. Pertanto, esiste una vasta letteratura che indaga le connessioni neurali dei primati. In particolare, molti ricercatori hanno studiato la complessa connettività corticocorticale del cervello del macaco e i loro risultati sono stati curati per la tabulazione (ad esempio, CoCoMac9). Sebbene utili, questi studi classici presentavano diverse limitazioni. In primo luogo, poiché ogni studio si concentra solo su regioni cerebrali limitate, le informazioni ottenute diventano inevitabilmente frammentarie. In secondo luogo, ogni studio utilizza metodi e condizioni diverse. Pertanto, la valutazione quantitativa tra gli studi diventa complicata. In terzo luogo, la connettività è solitamente mostrata come una camera lucida di sezioni rappresentative o come una tabella/grafico semi-quantitativo per le regioni cerebrali definite dall'autore. In altre parole, solo informazioni molto limitate da una struttura cerebrale complessa vengono estratte per la presentazione nella letteratura pubblicata. Con lo sviluppo delle tecniche di risonanza magnetica (MRI), gli studi sull'intero cervello a bassa risoluzione sono diventati importanti. Tuttavia, c'è un grande divario tra i livelli di dettaglio tra ciò che sappiamo sulle connessioni neurali nei topi e quelle nei primati.
Con questo background in mente, abbiamo deciso di eseguire un tracciamento anterogrado completo del cervello dell'uistitì comune10,11. Sebbene molto più piccolo e liscio del cervello del macaco, la controparte dell'uistitì mostra chiari segni di primati, come la presenza dell'area MT e delle aree prefrontali granulari, nessuna delle quali è chiaramente definita nei roditori12,13. In questo caso, le dimensioni ridotte sono state un grande vantaggio perché anche il cervello dell'uistitì pesa dieci volte di più del cervello del topo. Fortunatamente, abbiamo potuto visualizzare l'intero cervello dell'uistitì con aggiornamenti minimi della versione originale del software operativo TissueCyte1000 (d'ora in poi indicato come sistema di imaging dell'intero tessuto), il microscopio disponibile in commercio per l'imaging STPT2. Questo aggiornamento è stato per consentire un movimento extra del livello prima dell'affettatura. La versione attuale è ora sufficiente per l'elaborazione del cervello dell'uistitì. Questo articolo condivide un protocollo per gestire il cervello dell'uistitì per l'imaging STPT. Viene fornito anche il protocollo post-imaging che migliora ulteriormente l'utilità di questo metodo.
Protocollo
Tutte le procedure sperimentali sono state eseguite seguendo la Guida dell'Istituto Nazionale di Salute per la cura e l'uso degli animali da laboratorio (Pubblicazioni NIH n. 80-23) rivista nel 1996 e i "Principi guida per la cura e l'uso degli animali nel campo della scienza fisiologica" della Società fisiologica giapponese e sono state approvate dal Comitato per gli animali sperimentali di RIKEN (W2020-2-009(2)).
1. Iniezione di tracciante
- Eseguire iniezioni di traccianti fluorescenti e non fluorescenti al cervello dell'uistitì secondo le procedure precedentemente riportate14.
- Per quanto riguarda la combinazione con il metodo STPT, assicurarsi che la fluorescenza dei traccianti sia sufficientemente forte da essere rilevata senza miglioramenti. Adotta un sistema TET-Off a doppio vettore10 per una migliore espressione.
NOTA: I traccianti non fluorescenti possono anche essere iniettati e rilevati dopo che l'STPT è stato eseguito utilizzando le fette prodotte. Esempi di traccianti non fluorescenti includono la destrano ammina biotinilata (BDA), l'AAV che esprime cre in AAV2 retrogrado per la rilevazione retrograda dei nuclei delle cellule di input10, nonché gli AAV smFP-tag basati su GFP (vedi sotto).
- Per quanto riguarda la combinazione con il metodo STPT, assicurarsi che la fluorescenza dei traccianti sia sufficientemente forte da essere rilevata senza miglioramenti. Adotta un sistema TET-Off a doppio vettore10 per una migliore espressione.
- Fissare la perfusione e ottenere il cervello dell'uistitì dopo 4 settimane.
- Per anestetizzare l'uistitì, somministrare medetomidina (0,04 mg/kg), midazolam (0,4 mg/kg) e butorfanolo (0,4 mg/kg), chiamato MMB, per via intramuscolare, seguita da iniezione intraperitoneale di tiopentale sodico (100 mg/kg).
- Dopo aver confermato che il riflesso del dolore è perso, aprire la cavità toracica per esporre il cuore e tagliare l'atrio destro per consentire l'uscita del sangue e del fissativo.
- Quindi tagliare il ventricolo sinistro per inserire l'ago di perfusione per il primo lavaggio del sangue con la soluzione di prefisso (250 mM di saccarosio, 5 mM di MgCl2 in tampone fosfato 0,02 M [PB, pH 7,4]) per alcuni minuti (~50-100 mL), seguita dalla fissazione con paraformaldeide al 4% / tampone fosfato 0,1 M (PB; 2-3 volte il peso dell'animale in ~20 minuti).
- Una buona perfusione è fondamentale per un buon risultato. Quando si inserisce l'ago per perfusione, farlo avanzare lungo il setto verso l'aorta per garantire un buon flusso del fissativo. La punta dell'ago diventa visibile quando raggiunge l'aorta. Ritrarre fino a quando la punta è appena visibile in modo da non bypassare l'arteria carotide.
- Mantenere il cervello in paraformaldeide al 4%/0,1 M PB per 48 ore a 4 °C e trasferire a 50 mM PB. Se non viene utilizzato immediatamente, conservare il cervello in glicina allo 0,75% / 0,1 M PB per prevenire l'autofluorescenza dovuta all'iperfissazione.
2. Preparazione del campione (Figura 1)
- Incubare il cervello fisso con collagenasi (1 mg/mL in 3 mM CaCl,2 in 10 mL di soluzione salina tamponata tris [TBS]) a 37 °C per 1 ora.
NOTA: Preriscaldare il cervello a 37 °C per 5 minuti prima dell'incubazione. - Strofinare accuratamente la superficie del cervello con un batuffolo di cotone per rimuovere la materia pia e altre meningi (Figura 1A, video supplementare). Usa una pinza fine per sbucciare le meningi staccate.
NOTA: È importante rimuovere le meningi che circondano il mesencefalo, compreso il collicolo superiore o quelle nella parte superiore del talamo. È difficile smascherare queste strutture che si nascondono nel profondo. Tuttavia, rimuovere le meningi il più possibile. In caso contrario, spesso rimangono non tagliati, galleggiano verso l'alto e interferiscono con l'imaging e lo slicing (Figura 2A). - Prestare particolare attenzione quando il cervello viene sottoposto a risonanza magnetica ex vivo prima dell'inclusione, durante la quale il cervello viene immerso nella soluzione di fluoro15. Il fluoro rimanente, se incorporato insieme, potrebbe formare minuscole bolle d'aria sotto l'obiettivo durante l'imaging. Per evitare ciò, mantieni il cervello in PB per 1 settimana prima dell'incorporamento.
NOTA: Questa fase di attesa di solito non è necessaria, ma è fondamentale se combinata con la risonanza magnetica ex vivo . - Preparare il tampone NaBH4 sciogliendo 0,2 g di NaBH4 in 100 mL di tampone borato da 50 mM (pH 9,2) riscaldato a 40°C. Lasciare il tappo allentato per far uscire il gas CO2 . Conservare la soluzione per una notte con il tappo allentato. Avvolgere la bottiglia in un foglio di alluminio per proteggerla dalla luce fino all'uso.
- Preparare l'agarosio ossidato mescolando 2,25 g di agarosio e 0,21 g di NaIO4 in 100 mL di PB per 2-3 ore. Filtrare la soluzione con aspirazione sottovuoto e lavarla con tre cambi di PB. Risospendere l'agarosio in 50 mL di PB.
- Sciogliere completamente l'agarosio in un forno a microonde e raffreddare a 60-65 °C.
- Metti il cervello in una camera su misura e incorpora il cervello nell'agarosio. Fare attenzione a introdurre l'agarosio nella cavità sottostante il corpo calloso (Figura 1B).
NOTA: Preriscaldare il cervello prima a temperatura ambiente e poi a 65 °C per 5 minuti per consentire all'agarosio di depositarsi sulla superficie cerebrale senza solidificarsi. - Smontare la camera e immergere il blocco di agarosio nel tampone NaBH4 per una notte a 4 °C.
- Sostituire il tampone in PB più volte nell'arco di 1-2 settimane a 4 °C.
NOTA: L'autofluorescenza tissutale diventa molto debole se lo scambio del tampone è incompleto. - Trasforma un palcoscenico in vetro scorrevole attaccando quattro magneti al neodimio con una miscela istantanea epossidica.
- Montare il blocco di agarosio sul tavolino con un adesivo forte (ad es. super colla) (Figura 1C).
3. Lavorazione dei tessuti
- Utilizzare l'intero sistema di imaging tissutale secondo le istruzioni del produttore. Viene pubblicato anche un protocollo dettagliato16. L'attuale procedura viene utilizzata principalmente per TissueCyte1000, ma può essere applicata ad altri modelli, come TissueCyte 1600FC.
- Il funzionamento è leggermente diverso tra i modelli, ma segui i punti sottostanti che sono comunemente importanti.
- L'intero cervello dell'uistitì può essere elaborato coronalmente dalle estremità anteriori a quelle posteriori senza alcuna modifica nell'hardware. Assicurarsi che i limiti di movimento del tavolino non vengano superati quando si posiziona il cervello.
- Regolare con precisione l'angolo delle lame per assicurarsi che le profondità della superficie su entrambe le estremità della lama siano entro 10 μm. A causa della mielinizzazione densa, anche una differenza di profondità di 10 μm potrebbe portare a diverse penetrazioni laser attraverso la sostanza bianca adulta dell'uistitì. Per lo stesso motivo, impostare il piano di imaging a circa 25-35 μm dalla superficie.
- Utilizzare una lama di ceramica (consigliata) per tagliare l'intero cervello a intervalli di 50 μm (~oltre 650 fette).
- Le meningi rimangono non tagliate e interferiscono con l'affettatura se non vengono rimosse correttamente (Figura 2). Alcune meningi (ad esempio, quelle che circondano l'ippocampo o il pulvinar) sono difficili da rimuovere. Estrarre manualmente queste meningi con una pinza fine quando notate.
4. Tecniche istologiche ausiliarie
- Recupero delle sezioni
- Recuperare le fette di tessuto per eseguire varie colorazioni istologiche. Allinea accuratamente queste sezioni in ordine di affettatura. Tracciare i vasi sanguigni attraverso gli strati corticali per l'allineamento (Figura 3).
- Tagliare l'agarosio in eccesso da ogni sezione per una migliore maneggevolezza.
NOTA: Questo processo non deve essere eccessivo. L'agarosio serve a mantenere in posizione i segmenti di tessuto senza interferire con l'elaborazione istologica. - Imaging retroilluminato
- Montare le sezioni su un vetrino scorrevole e asciugarle.
- Reidratare la sezione con PBS e posizionare il vetrino coprioggetti per l'imaging.
- Osservare un modello di mielinizzazione senza colorazione quando la sezione viene visualizzata da un microscopio a fluorescenza (Figura 4) in modalità campo chiaro. Regolare il tempo di esposizione in modo che il riflesso della luce possa essere visibile.
NOTA: Qualsiasi microscopio può essere utilizzato se può utilizzare l'illuminazione a campo scuro. - Rimuovere il vetrino coprioggetti e procedere alla colorazione Nissl o a qualsiasi altra colorazione. È sufficiente qualsiasi procedura di colorazione standard.
- Utilizzare questo metodo per confrontare direttamente diversi modelli di colorazione con i modelli di mielinizzazione (Figura 4).
- Imaging BDA
- Vedere la Tabella 1 per una procedura dettagliata.
- Per prima cosa, trattare le sezioni con la soluzione di Dent (20% DMSO, 80% metanolo). Il metanolo incluso nella soluzione di Dent migliora notevolmente i segnali BDA negli assoni.
- Utilizzare il metodo di amplificazione del segnale tiramide (TSA) per migliorare il segnale BDA da rilevare in fluorescenza17.
NOTA: La soluzione TSA-biotina può essere acquistata dal fornitore, ma il reagente interno è molto più economico ed efficace. - Per registrare l'immagine STPT, acquisire un'immagine retroilluminata dopo aver montato la sezione su un vetro scorrevole con il supporto di montaggio.
- Colorazione con proteina fluorescente spaghetti monster (smFP)
NOTA: Le proteine fluorescenti 'spaghetti monster' (smFP) sono una famiglia di varianti di GFP non fluorescenti con più tag epitopi18. Forniscono eccellenti segnali fluorescenti al momento dell'immunorilevazione e sono adatti come traccianti complementari ai traccianti fluorescenti. Vedere la Tabella 2 per i costrutti AAV disponibili da Addgene.- Seguire la procedura dettagliata fornita nella Tabella 1.
5. Elaborazione dei dati post-imaging
- Test di integrità dei dati
NOTA: STPT genera un vasto numero di file immagine. Quando il cervello di un uistitì adulto viene sezionato coronalmente a intervalli di 50 μm, sono in genere necessarie più di 650 sezioni per coprire l'intero cervello. I dati di ogni sezione sono costituiti da una serie di immagini di piastrellatura catturate in tre canali. Questi file di immagine vengono archiviati in un'unica cartella insieme a un file di metadati che registra la posizione di ogni riquadro. Per ridurre al minimo la quantità totale di dati e il tempo di elaborazione, l'imaging viene condotto in blocchi di esecuzioni, durante i quali il numero di passaggi X e Y per l'affiancamento cambia gradualmente. In genere, 20-30 blocchi di esecuzioni, ciascuno comprendente l'imaging di 30-50 sezioni, costituiscono il set di dati completo per un singolo cervello. Per utilizzare questi dati, sono necessarie diverse fasi di elaborazione delle immagini, tra cui lo stitching delle immagini affiancate, la segmentazione dei segnali fluorescenti e la registrazione dei dati volumetrici in un modello standard, tra le altre.- Eseguire le fasi di elaborazione delle immagini singolarmente utilizzando vari strumenti software o attraverso pipeline dedicate su misura per le esigenze specifiche dei ricercatori. I dettagli dell'elaborazione delle immagini esulano dallo scopo di questo articolo e possono essere trovati in altre pubblicazioni11,19.
- Indipendentemente dalla procedura utilizzata, assicurarsi che i dati siano organizzati in modo coerente.
NOTA: poiché l'intero processo di imaging prevede molti blocchi di esecuzioni e si estende per diversi giorni, non è raro che un'esecuzione venga annullata a metà e riavviata di nuovo. In questi casi, la struttura dei dati può essere interrotta, ostacolando la corretta esecuzione della pipeline di elaborazione delle immagini. - Per ridurre al minimo il rischio di interruzione della struttura dei dati, eseguire uno script Python per verificare l'integrità della struttura dei dati (kn_pipeline_check_mosaic.py) disponibile in GitHub (github.com/watkarbey/STPT_depo).
- Registrazione dell'immagine colorata nell'immagine STPT
NOTA: La trasformazione non lineare dell'immagine colorata nell'immagine STPT può essere eseguita utilizzando il plug-in bUnwarpJ di ImageJ.- Regolare le dimensioni delle immagini di destinazione (STPT) e delle immagini sorgente (colorate) in modo che siano approssimativamente uguali.
- Binarizza ogni immagine per visualizzare le forme delle sezioni.
- Esegui bUnwarpJ. Ricordati di selezionare l'opzione Salva trasformazioni .
- Avvia nuovamente bUnwarpJ . Selezionare l'immagine originale prima della binarizzazione come immagine di origine. Quindi fare clic su Carica trasformazione elastica e scegliere la trasformazione salvata.
Risultati
Nella configurazione tipica qui utilizzata, l'intero cervello di un uistitì adulto può essere ripreso (Figura 5) alla risoluzione di ~1,3 x 1,3 μm/pixel con un intervallo di sezione di 50 μm in circa 1 settimana. Ciò equivale a ~650 immagini coronali in tre canali dopo lo stitching dell'immagine. Con un obiettivo 16x (Nikon 16xW CFI75 LWD; NA = 0,80), il campo visivo di un singolo scatto è di circa 1 x 1 mm. L'immagine per l'intera superficie coronale è ottenuta unendo questi scatti (Figura 5A). L'allineamento in direzione Z è eccellente e una buona immagine 3D si ottiene semplicemente impilando i dati dell'immagine coronale (Figura 5B, E). Per la standardizzazione, il modello STPT per il cervello dell'uistitì11 può essere utilizzato per la registrazione 3D-3D (Figura 5F). Questa trasformazione dei dati è uno degli aspetti chiave dell'intera neuroanatomia cerebrale, in cui a una regione di interesse viene assegnata una coordinata spaziale assoluta indipendente dall'annotazione anatomica. Una volta che il cervello del campione è registrato nello spazio standard, si possono facilmente estrarre le sottoregioni di interesse per ulteriori analisi (Figura 5F, G). In particolare, le regioni corticali possono essere trasformate in una mappa piatta utilizzando un parametro predeterminato (Figura 5H).
Le sezioni generate durante l'imaging possono essere utilizzate per vari scopi istologici. Come mostrato nella Figura 4A, B, l'imaging retroilluminato senza colorazione fornisce un modello molto simile alla colorazione autentica della mielina. Questa può essere un'ottima alternativa alla colorazione della mielina. Inoltre, se l'immagine retroilluminata viene ottenuta prima della colorazione di Nissl, la stessa sezione può essere utilizzata per ottenere i pattern sia della mielina che della colorazione di Nissl, fornendo così informazioni utili per identificare aree e strati (Figura 5C-E). Queste sezioni possono essere utilizzate anche per la colorazione immunologica. Nella Figura 5J, la sezione intorno al centro di iniezione è stata controcolorata con anticorpo NeuN per stimare l'efficienza di trasduzione del virus AAV. È anche una buona strategia iniettare traccianti non fluorescenti oltre ai traccianti fluorescenti e rilevarli istologicamente dopo il prelievo della sezione. Nel nostro studio precedente, abbiamo combinato traccianti verdi anterogradi con il vettore retrogrado "cre", che è stato successivamente rilevato dall'anticorpo anti-cre10. In un esempio della Figura 6A-E, il BDA è stato iniettato nel lato controlaterale del tracciante verde (trifoglio) e lo ha rilevato in modo fluorescente. Si noti che i segnali rossi BDA possono essere registrati nell'immagine TissueCyte per essere localizzati nell'intera coordinata cerebrale. In un altro esempio della Figura 6F-I, smFP-myc è stato iniettato nell'area parietale (Figura 6G), mentre il tracciante verde è stato iniettato nell'area frontale. In questo modo, più traccianti possono essere iniettati nello stesso animale senza interferire con l'imaging. Un grande vantaggio dell'utilizzo delle sezioni STPT per ulteriori colorazioni è che la relazione tra i traccianti fluorescenti e non fluorescenti può essere determinata per lo stesso cervello. Pertanto, siamo stati in grado di determinare la reciprocità delle proiezioni corticocorticali ad alta precisione10. Un altro vantaggio è che le coordinate 3D delle sezioni colorate possono essere mappate nuovamente sui dati STPT e quindi sul modello standard. Pertanto, potrebbe non essere necessario utilizzare tutte le sezioni recuperate per la colorazione. Per una migliore interpretazione, è possibile selezionare le sezioni per la colorazione per aggiungere ulteriore contesto ai dati STPT.
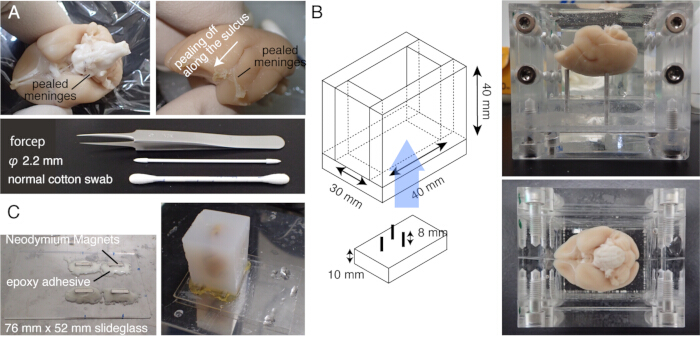
Figura 1: Preparazione del campione per STPT. (A) Rimozione della meninge mediante tamponi di cotone. Le meningi che circondano il tronco encefalico possono essere rimosse con una pinza a punta fine. Le meningi che circondano il cervello dell'uistitì vengono rimosse manualmente strofinando con tamponi di cotone. La foto mostra le meningi espulse dall'interno del solco laterale. (B) La scatola di acrilico utilizzata per l'inclusione dell'agarosio. I perni sono mobili e vengono utilizzati per regolare l'angolo del cervello in modo che sia vicino alla posizione stereotassicamente fissa20. (C) Una diapositiva magnetica in vetro diapositiva da 76 mm x 52 mm e quattro magneti al neodimio fissati con adesivo epossidico. Il blocco di agarosio è fissato al tavolino magnetico con una super colla. Clicca qui per visualizzare una versione più grande di questa figura.
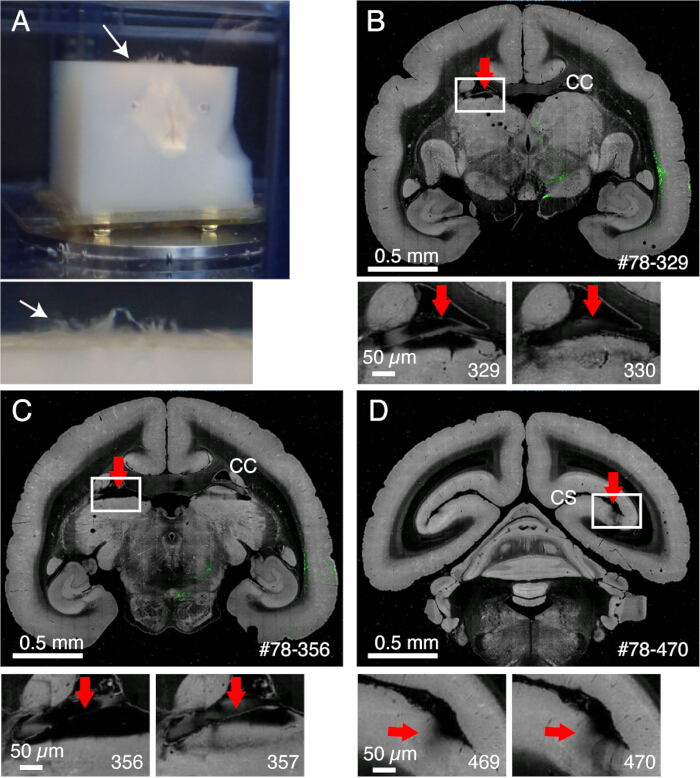
Figura 2: L'effetto delle meningi sull'imaging. (A) Le meningi che rimangono non tagliate galleggiano verso l'alto, come mostrato dalla freccia bianca. In questo esempio, si vede un'estesa sporgenza delle meningi perché non sono state rimosse prima dell'inclusione dell'agarosio. Di solito, le meningi rimangono intatte solo per alcune regioni difficili. (B) Un esempio di cattivo taglio dovuto alla presenza di meningi tra il corpo calloso e la parte superiore del talamo. In questo caso, un cattivo slicing ha portato all'alternanza di una fetta profonda (329) e una fetta relativamente normale (330). #78-329 sta per il campione n. 78 sezione n. 329 nel portale di dati Brain/MINDS (C) Un altro esempio di cattiva imaging. In un caso peggiore, il nucleo pulvinare mostrato dalla freccia rossa potrebbe staccarsi completamente. Barre di scala: 0,5 mm (pannello superiore), 0,5 μm (pannello inferiore). (D) Un altro esempio di cattiva imaging. Le meningi profonde all'interno del solco calcarino sono difficili da rimuovere. Le ombre viste nelle sezioni 469 e 470 sono causate dalle meningi fluttuanti che sono finite sotto l'obiettivo. Barre di scala: 0,5 mm (pannello superiore), 0,5 μm (pannello inferiore). Clicca qui per visualizzare una versione più grande di questa figura.
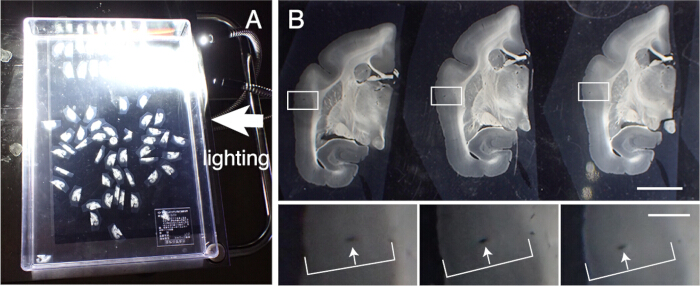
Figura 3: Allineamento delle sezioni di tessuto in ordine. (A) Fino a 50 sezioni coronali di uistitì possono essere allineate correttamente in ordine in un contenitore di plastica (31 cm x 22,5 cm). Per visualizzare le strutture dettagliate, il contenitore viene posizionato su carta nera e illuminato lateralmente. Queste sezioni vengono prima allineate approssimativamente in ordine e poi soggette a un allineamento preciso. (B) L'allineamento preciso utilizza il vaso sanguigno come marcatore. La corteccia cerebrale contiene molti vasi sanguigni che corrono verticalmente attraverso gli strati corticali. Sono identificati come fori allungati che cambiano sistematicamente la loro posizione all'interno degli strati corticali (frecce bianche). Con questo metodo, è possibile allineare con precisione anche sezioni con intervalli di 50 μm. Questi fori dei vasi sanguigni possono essere identificati nelle immagini della sezione SPTP per conferma. Barra scala: 5 mm (pannello superiore), 1 mm (pannello inferiore). Clicca qui per visualizzare una versione più grande di questa figura.
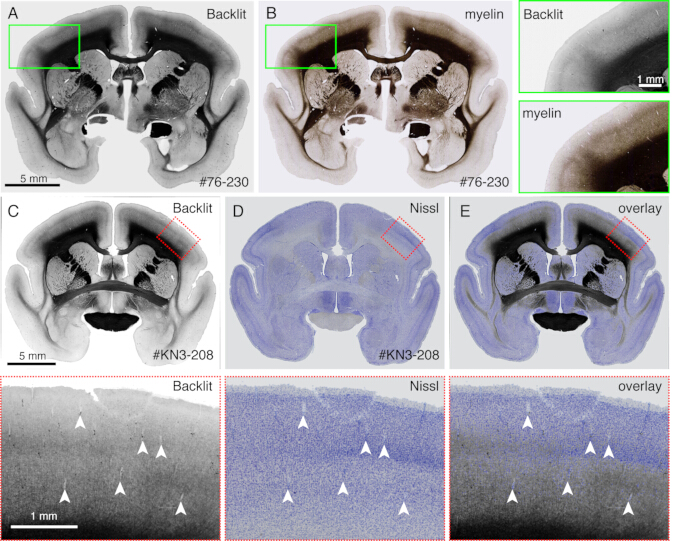
Figura 4: Immagine retroilluminata come sostituto della colorazione mielinica. (A,B) La sezione identica è stata utilizzata per l'imaging retroilluminato e la colorazione mielinica. In primo luogo, la sezione è stata montata sul vetrino, asciugata, reidratata con PBS, vetrino coprioggetti e ripresa con un microscopio ottico (Tabella dei materiali). Dopo aver rimosso il vetrino coprioggetti, la stessa sezione è stata utilizzata per la colorazione della mielina21 e ripresa con un microscopio a fluorescenza. L'immagine retroilluminata è stata registrata nell'immagine della mielina utilizzando il plug-in bUnwarpJ di ImageJ. Le caselle verdi mostrano le viste ingrandite di ogni immagine. Si noti che queste immagini mostrano modelli quasi identici, tranne per il fatto che la colorazione della mielina visualizza meglio le strutture fibrose. (C-E) La sezione identica è stata utilizzata per l'imaging retroilluminato e la colorazione Nissl. La maschera di soglia bassa per l'immagine retroilluminata è stata prima registrata con la maschera di soglia bassa per l'immagine Nissl utilizzando il plug-in bUnwarpJ e l'immagine originale è stata trasformata utilizzando lo stesso parametro. Si noti che i vasi sanguigni (punte di freccia bianche) corrispondono bene tra le due immagini. Poiché queste due immagini sono per la stessa sezione, la corrispondenza è quasi perfetta e si possono confrontare direttamente i modelli di mielina e Nissl per l'identificazione degli strati corticali. Barre di scala: 5 mm (pannelli A-E). Clicca qui per visualizzare una versione più grande di questa figura.
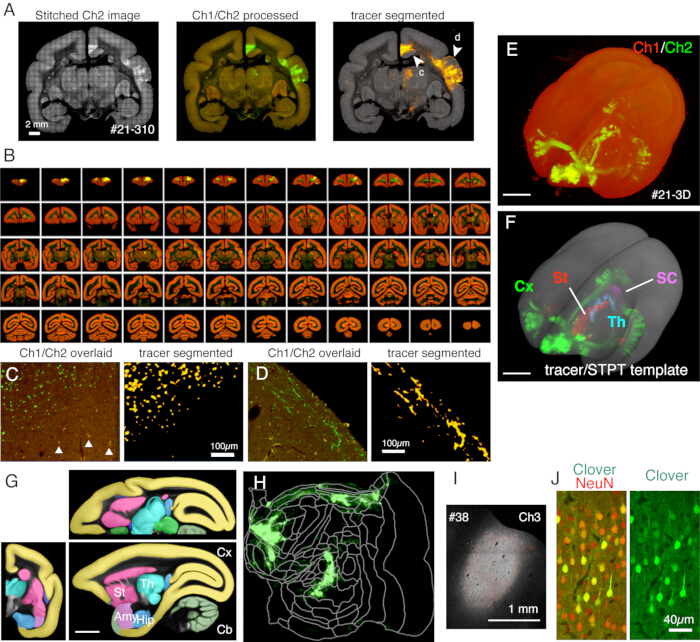
Figura 5: Risultato tipico dell'imaging STPT. (A) Una semplice affiancatura di immagini Ch2 (verdi) di una sezione di esempio (sezione 310 del campione #21). Senza la correzione dello sfondo, i bordi di ogni riquadro sono visibili. Nella colonna centrale, le tessere sono state unite con la correzione dello sfondo rispettivamente per Ch1 (rosso) e Ch2 (verde). Per ridurre i segnali della lipofuscina (vedi pannello C), i segnali Ch1 sono stati sottratti da Ch2 e mostrati in verde. Per ridurre i segnali di lipofuscina di Ch1, il comando "Rimuovi outilers.." è stato utilizzato prima della cucitura da ImageJ. Nella colonna di destra, i segnali traccianti sono stati segmentati in base alla pipeline di elaborazione delle immagini11. Le punte delle frecce (c, d) mostrano le posizioni in cui le viste ingrandite sono mostrate nei pannelli C e D. Barra di scala: 2 mm. (B) Panoramica delle sezioni seriali per il campione #21. STPT ha generato 635 immagini coronali ad alta risoluzione per questo campione. (C) Una vista ad alto ingrandimento mostrata dalla punta della freccia c nel pannello A. Si tratta di una semplice sovrapposizione di Ch1 (rosso) e Ch2 (verde) senza ulteriori elaborazioni. I triangoli mostrano segnali di fluorescenza della lipofuscina, che mostrano uno spettro diffuso. L'algoritmo di segmentazione del tracciante distingue accuratamente i segnali del tracciante dal fondo di lipofuscina nonostante una forma molto simile (pannello di destra). Barra della scala: 100 μm. (D) Un altro esempio di vista ad alto ingrandimento. Si noti che le fibre sottili degli assoni nello strato 1 possono essere ben visibili. Barra della scala: 100 μm. (E) La ricostruzione 3D delle immagini STPT originali. Le 635 immagini coronali a bassa risoluzione, come mostrato nel pannello B, sono state utilizzate come tiff-stack per la visualizzazione 3D utilizzando fluorender22. Barra della scala: 5 mm. (F) La ricostruzione 3D dei segnali traccianti segmentati registrati nel modello STPT (grigio). I segnali traccianti in diverse regioni del cervello sono stati mostrati da colori diversi. Queste regioni sono state ritagliate utilizzando l'annotazione mostrata nel pannello G. Barra della scala: 5 mm. (G) Modello STPT sovrapposto con l'annotazione di diverse regioni cerebrali. Barra di scala: 5 mm. (H) Il segnale tracciante corticale mostrato nel pannello F è stato mostrato sotto forma di mappa piatta. (I) Identificazione del sito di iniezione mediante la fluorescenza Ch3, che è meno sensibile alla fluorescenza del tracciante e rimane insaturo. Barra graduata: 1 mm. (J) La colorazione della sezione intorno al centro di iniezione con anticorpo NeuN ha mostrato che circa il 30% dei neuroni mostra una forte espressione di fluorescenza del verde trifoglio. Barra della scala: 40 μm. Abbreviazioni: Cx; corteccia, St; striato, Th; talamo, SC; collicolo superiore. Amy; amigdala, anca; ippocampo, Cb; cervelletto. Clicca qui per visualizzare una versione più grande di questa figura.
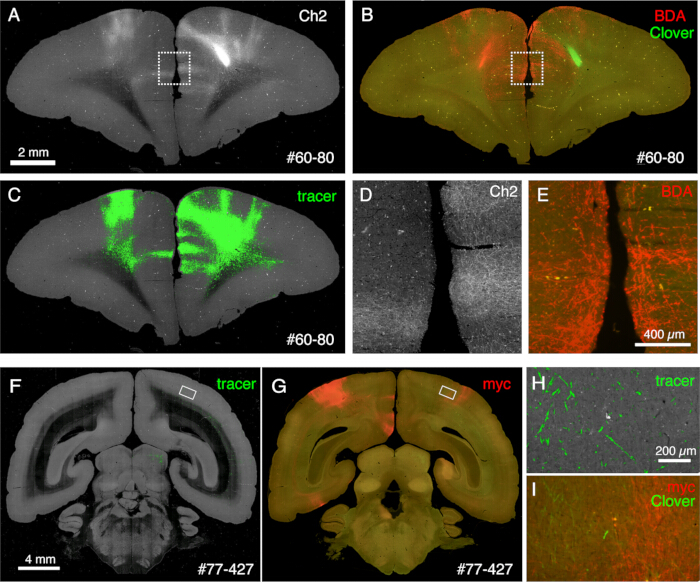
Figura 6: Istologia post-STPT che mostra traccianti multipli non fluorescenti. (A-C) Confronto dell'immagine STPT con l'immagine colorata con BDA. In questo campione, il BDA viene iniettato nel lato controlaterale dell'iniezione di trifoglio. I pannelli A e C mostrano l'immagine Ch2 e la segmentazione del tracciante dei dati STPT. Il pannello B mostra la colorazione post-STPT per BDA. La fluorescenza del trifoglio è diminuita a causa del trattamento con metanolo della sezione. Barra della scala: 2 mm. (D,E) Le caselle tratteggiate nei pannelli A e B sono ingrandite. Barra della scala: 100 μm. (F,G) Confronto dell'immagine STPT con la colorazione anticorpale con tag anti-myc. In questo campione, l'iniezione di trifoglio è nella PFC, mentre l'AAV-smFP-myc viene iniettato nella corteccia parietale controlaterale. I rettangoli bianchi sono ingranditi nei pannelli H e I. Barra della scala: 4 mm. (H) La segmentazione del tracciante è mostrata in verde. Barra graduata: 200 μm. (I) La colorazione myc è mostrata in rosso. La fluorescenza del trifoglio è mostrata in verde. Barra di scala: 200 μm. I segnali verdi nei pannelli H e I sono presenti in una posizione simile ma non identica perché STPT recupera solo una sezione ottica di ~10 μm. Clicca qui per visualizzare una versione più grande di questa figura.
| Protocollo di colorazione fluorescente BDA | |
| Rimuovere l'agarosio | |
| Lavaggio TBS | 10 minuti (2x) |
| 1% H2O2 nella soluzione di Dent | 10 minuti |
| Lavaggio TBS | Breve |
| 0,5% di blocco TNB | 1 ora |
| StAvHRP (1:4000) in TNB | 2 pernottamenti |
| Lavaggio TNT | 10 minuti (3x) |
| TSA Biotina (1:4000) in borato 0,1 M (pH8,5) + 0,003% H2O2 | 2 h |
| Lavaggio TNT | 10 minuti (3x) |
| Cy3-streptavidina (1:1000) in TNT | 3 h |
| Lavaggio TNT | 10 minuti (2x) |
| Lavaggio TBS | Conservare la sezione fino al montaggio |
| Montare la sezione su slideglass utilizzando un mezzo di montaggio antisbiadimento | |
| Protocollo di colorazione fluorescente anti-MYC | |
| Rimuovere l'agarosio | |
| Lavaggio TBS | 10 minuti (2x) |
| blocco in IB | 1 ora |
| Anti-Myc (1:4000) in IB | 2 durante la notte |
| Lavaggio TNT | 10 minuti (3x) |
| Anti-topo Cy3 (1:1000) in TNT | 3 h |
| Lavaggio TNT | 10 minuti (2x) |
| Lavaggio TBS | Conservare la sezione fino al montaggio |
| Montare la sezione su slideglass utilizzando un mezzo di montaggio antisbiadimento | |
| Tamponi/soluzioni | Composizione |
| 0,5% TNB | Reagente bloccante TSA allo 0,5% in TS7.5 |
| La soluzione di Dent | 20% DMSO, 80% metanolo |
| Tampone ad immersione (IB) | 10% FBS, 2% BSA 0,5% TritonX100 in TBS |
| TBS (soluzione salina con tampone Tris) | 25 mM Tris, 137 mM NaCl, 2,7 mM KCl (pH 7,4) |
| TNT | 0.05 % Tween20 in TS7.5 |
| TS7.5 | 0,1 m TRIS-HCl, pH 7,5, 0,15 m NaCl |
Tabella 1: Colorazione fluorescente BDA e protocollo di colorazione fluorescente anti-myc
| Plasmide per tracciante AAV | Addgene No. | Anticorpi raccomandati | Diluizione consigliata | Risultato atteso |
| pAAV-EF1_Cre | 201198 | Clone di Millipore 2D8 | 1:1000 | buono per le cellule |
| pAAVCam1.3_smFP_Myc | 201205 | MBL M192 My3 (mouse) | 1:4000 | Eccellente |
| pAAVCam1.3_smFP_HA | 201206 | CST C29F4 (coniglio) | 1:1000 | buono per le cellule |
| pAAVCam1.3_smFP_FLAG | 201207 | MBL PM020B (coniglio) | 1:1000 | OK |
| AAVTRE3_smFP_Myc | 201208 | |||
| AAVTRE3_smFP_HA | 201209 | |||
| AAVTRE3_smFP_FLAG | 201210 |
Tabella 2: Elenco dei plasmidi Addgene disponibili per la produzione di traccianti non fluorescenti. Il costrutto cre ha come bersaglio il nucleo ed è adatto per il tracciamento retrogrado avvolto da AAV2 retro. smFP_HA costrutto è buono per il rilevamento dei corpi cellulari (e retrogrado).
Video supplementare: Vista al microscopio della rimozione delle meningi. Clicca qui per scaricare questo video.
Discussione
Questo articolo ha spiegato le soluzioni pratiche per gestire i cervelli degli uistitì per l'elaborazione dell'intero cervello, nonché le tecniche istologiche ausiliarie che migliorano l'utilità della tecnica STPT. Il punto di forza della "neuroanatomia dell'intero cervello" utilizzando STPT è che è possibile ottenere le coordinate 3D di qualsiasi regione di interesse, indipendentemente dal fatto che sia anatomicamente annotata o meno. Grazie alla registrazione 3D-to-3D ad alta precisione, è possibile trasformare queste coordinate in un modello standard per la sovrapposizione di più set di dati. In questo modo, il modello standard funge da mezzo per l'integrazione dei dati. Questo è stato un aspetto essenziale del nostro progetto di mappatura della corteccia prefrontale (PFC)10, in cui sono stati analizzati i dati ottenuti da molti individui. Inoltre, le informazioni mappate sul modello standard possono essere confrontate con vari dati che sono già stati mappati, sia che si tratti di Nissl, pattern di mielina, dati traccianti, dati MRI (inclusa la risonanza magnetica di diffusione) o annotazioni anatomiche11. È importante sottolineare che può anche essere confrontato con i dati futuri che saranno ottenuti da una tecnologia ancora da emergere. Attualmente esistono più modelli per il cervello dell'uistitì, che si basano sulla colorazione Nissl23, sulla risonanza magnetica23, 24, 25, 26, 27, 28 e STPT11. Ma possono essere trasformati l'uno nelle coordinate dell'altro in base ai contrasti dell'immagine e ai parametri precalcolati11. L'integrazione dei dati su scala cerebrale tra gli studi contribuisce a una migliore comprensione del cervello come sistema. Il prerequisito per il funzionamento di questa strategia è l'acquisizione affidabile dei dati in tutto il cervello. Di seguito, vengono discussi i passaggi critici e i potenziali problemi associati al protocollo corrente.
Uno dei processi più vulnerabili dell'imaging STPT è il taglio dei tessuti. Come accennato in precedenza, le meningi spesso rimangono non tagliate e possono interferire con l'affettatura. In particolare, il nucleo pulvinare e il collicolo superiore sono le due regioni cerebrali più colpite: a seconda dell'accuratezza della rimozione della meninge e dell'inclusione nell'agarosio, possono essere rimossi dal blocco di tessuto durante l'affettatura. Queste regioni profonde sono difficili da avvicinare dall'esterno e possono facilmente rompersi durante la rimozione della meninge. Un'attenta ma accurata rimozione della meninge è fondamentale per il successo dell'imaging. Un'altra preoccupazione è il ribaltamento delle sezioni affettate sul blocco di tessuto, che a volte si verifica quando le sezioni rimangono attaccate anche dopo l'affettatura. Può essere ridotto al minimo modellando il blocco in modo che la lama tagli obliquamente all'estremità.
Eseguendo l'imaging in profondità nel blocco di tessuto, l'STPT evita le irregolarità della sua superficie. Mentre i segnali fluorescenti possono passare attraverso la regione corticale piuttosto facilmente, sono altamente diminuiti nelle regioni mielinizzate. Pertanto, la profondità dell'imaging deve essere determinata con attenzione per bilanciare l'imaging coerente su tutta la superficie del blocco e la luminosità dei segnali nella regione ricca di mielina, come la sostanza bianca. Nella configurazione qui utilizzata, di solito miriamo a 25-35 μm dalla superficie. È inoltre necessario notare che la superficie di taglio potrebbe ridursi in modo non uniforme dopo lunghe ore di conservazione. Per ridurre al minimo l'area di imaging, abbiamo suddiviso la sessione di imaging in 20-30 esecuzioni con diverse impostazioni del palco per 5-6 giorni. Facciamo in modo che l'intervallo tra le corse sia inferiore a 2 ore o confermiamo la profondità della superficie e regoliamo l'altezza del palco prima di ogni corsa.
In questo protocollo, il segnale BDA è stato amplificato dal metodo TSA. Questo metodo è altamente efficace e può rilevare segnali BDA trasportati anterogradamente anche a una risoluzione relativamente bassa (ad esempio, Figura 6B). La biotina TSA è disponibile in commercio da Akoya Biosciences, ma la soluzione fatta in casa mostra un miglioramento molto migliore. D'altra parte, la diluizione dell'anticorpo e della soluzione richiede un'attenta regolazione per ottenere il risultato ottimale. Il pretrattamento della sezione con una soluzione di metanolo è fondamentale. Senza pretrattamento, i segnali BDA sono a malapena rilevati negli assoni mielinizzati.
Quando si utilizza un tracciante anterogrado, è spesso difficile identificare il sito esatto di iniezione a causa della saturazione dei segnali di fluorescenza. Nella configurazione dell'intero sistema di imaging tissutale utilizzato in questo studio, utilizziamo il canale blu per identificare le cellule infette (Figura 5I). Anche quando i canali rosso e verde sono saturi, ogni singolo neurone infetto è solitamente rilevabile nel canale blu. Questo vale anche per l'Allen Mouse Brain Connectivity Atlas1. L'esame delle cellule di origine è importante perché l'infezione a volte coinvolge solo strati particolari. Abbiamo riscontrato tali infezioni parziali piuttosto frequentemente per il Mouse Brain Connectivity Atlas, forse a causa dell'utilizzo del metodo di ionoforesi29. La diffusione laterale dei traccianti virali potrebbe essere più o meno variabile a seconda delle iniezioni. Questa variabilità può potenzialmente influire sul risultato della traccia e richiede un'attenta normalizzazione.
La corretta registrazione dell'immagine 3D ottenuta nel modello standard è un processo chiave dell'intera neuroanatomia cerebrale. La registrazione dell'immagine STPT nel modello STPT può essere abbastanza accurata, e abbiamo osservato solo devianze di pochi voxel (50 μm isocubici) per i bordi con un alto contrasto dell'immagine10. Tuttavia, c'è un limite a ciò che la registrazione può fare. A causa del processo di rimozione della meninge, i campioni STPT presentano generalmente spazi vuoti tra gli emisferi e tra la corteccia e il mesencefalo/rombencefalo, mentre i tessuti cerebrali sono strettamente impacchettati nelle immagini MRI in vivo . Tali differenze sono difficili da regolare mediante registrazione. La corteccia dell'uistitì è per lo più priva di solchi, ma il solco intraparietale è molto profondo in alcuni individui. Tali solchi andranno persi (si verifica la fusione dall'alto verso l'alto) al momento della registrazione. Sebbene la registrazione sia una tecnica potente, è necessario risalire ai dati grezzi per la conferma del risultato ottenuto.
La generazione di enormi quantità di dati di immagini è sia un punto di forza che un limite di questa tecnica. Se da un lato migliora la completezza del set di dati, dall'altro richiede un'attenta gestione dei dati acquisiti e lo sviluppo di una pipeline di elaborazione automatizzata delle immagini per un'interpretazione efficiente dei dati. In futuro, l'applicazione dell'intelligenza artificiale generativa (AI) nella costruzione di pipeline di elaborazione delle immagini potrebbe semplificare significativamente questo processo. L'imaging sistematico dell'intero cervello è stato eseguito anche utilizzando metodi basati su scanner per vetrini21,30. Rispetto a tali metodi, l'STPT non richiede calcoli aggiuntivi per la ricostruzione 3D. In combinazione con il sezionamento ottico, abbiamo dimostrato che l'STPT ha il potenziale per ricostruire anche segmenti assonali attraverso le sezioni31. Con l'ulteriore combinazione di tecniche di clearance tissutale, Economo et al. hanno sviluppato un metodo per visualizzare la totalità dei neuroni scarsamente marcati32,33. Le ultime versioni di TissueCyte offrono ora opzioni per un laser aggiuntivo per migliorare l'eccitazione delle proteine fluorescenti rosse o un'unità di cattura delle sezioni per il recupero automatico delle sezioni. Con questi progressi, l'approccio dell'intero cervello diventerà più efficiente, fornendo una base per una comprensione completa del cervello dei primati, compreso quello degli esseri umani.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Ringraziamo profondamente lo staff tecnico del laboratorio e delle strutture per animali Yamamori (RRD) per il loro aiuto. Ringraziamo il RIKEN CBS-Olympus Collaboration Center per l'assistenza tecnica con l'acquisizione di immagini confocali. Questo lavoro è stato supportato dal programma per la ricerca scientifica sulle aree innovative (numero di sovvenzione 22123009) di MEXT, Giappone, da Brain/MINDS e Brain/MINDS2.0 di AMED, Giappone (JP15dm0207001, JP23wm0625001 e JP24wm0625218), e da JSPS KAKENHI Grant Number 24K09678 ad A.W.
Materiali
| Name | Company | Catalog Number | Comments |
| Agarose type I | Sigma | A6013 | |
| All-in-one fluorescence microscope | Keyence | BZ-X series | This study used BZ-X710 |
| anti-mouse Cy3 | Jackson Immuno Research Laboratories, Inc. | 115-165-003 | |
| anti-myc antibody | MBL | M192 | mouse monoclonal |
| Butorphanol | Meiji Animal Health | Vetorphale | |
| Ceramic blade | TissueVision | N/A | |
| Collagenase type I | Wako Chemical | #031-17601 | Aliquot into 100 µL x 10 of 100 mg/mL in PBS |
| Cotton swab (HUBY-340) | HUBY | BB-013SP | micro cotton swabs |
| Custom-made chamber | N/A | N/A | The chamber consist of four side plates and one bottom plates, all made of 1 cm thick acrylic plates, which can be securely fastened together using screw bolts. An additional acrylic plate is placed inside the chamber, with three metal poles inserted through it to hold the marmoset brain in an inverted position. |
| Cy3-Streptavidin | Jackson Immuno Research Laboratories, Inc. | 016-160-084 | |
| Dumont #5 forceps, Inox | N/A | N/A | |
| Epoxy instant mix | Locktite | N/A | |
| FIJI | NIH | https://imagej.net/software/fiji/downloads | |
| kn_pipeline_check_mosaic.py | N/A | N/A | github.com/watkarbey/STPT_depo; This is a checking script for TissueCyte data. |
| Medetomidine | Zenoaq | Domitor | |
| Midazolam | Sandoz | N/A | |
| NaBH4 | Sigma | #452882 | |
| NaIO4 | Sigma | S1878 | |
| Perfusion needle | Natsume Seisakusho Co., Ltd | KN-348, 20G-50 | |
| Slideglass (76 mm x 52 mm) | Matsunami | S9111 | |
| StAvHRP | Jackson Immuno Research Laboratories, Inc. | 016-030-084 | HRP-streptavidin (titration needs to be carefully determined) |
| TissueCyte1000 | TissueVision | https://www.tissuevision.com/tissuecyte | |
| TSA blocking reagent | Kiko Tech | FP1012 | Refer to TSA-biotin kit (Akoya Biosciences) |
| TSA-biotin | House-made | N/A | See Okamoto et al (ref. 16) |
| VECTASHIELD HardSet Mounting Medium | Vector laboratories Inc | N/A | Antifade mounting medium |
Riferimenti
- Oh, S. W., Harris, J. A., Ng, L., Winslow, B., Cain, N., et al. A mesoscale connectome of the mouse brain. Nature. 508 (7495), 207-214 (2014).
- Ragan, T., et al. Serial two-photon tomography for automated ex vivo mouse brain imaging. Nat Methods. 9 (3), 255-258 (2012).
- Liang, Z., Arefin, T. M., Lee, C. H., Zhang, J. Using mesoscopic tract-tracing data to guide the estimation of fiber orientation distributions in the mouse brain from diffusion MRI. Neuroimage. 270, 119999(2023).
- Watakabe, A., Hirokawa, J. Cortical networks of the mouse brain elaborate within the gray matter. Brain Struct Funct. 223 (8), 3633-3652 (2018).
- Ypma, R. J. F., Bullmore, E. T. Statistical analysis of tract-tracing experiments demonstrates a dense, complex cortical network in the mouse. PLoS Comput Biol. 12 (9), e1005104(2016).
- Wang, Q., et al. The Allen mouse brain common coordinate framework: A 3D reference atlas. Cell. 181 (4), 936-953.e20 (2020).
- Köbbert, C., Apps, R., Bechmann, I., Lanciego, J. L., Mey, J., Thanos, S. Current concepts in neuroanatomical tracing. Prog Neurobiol. 62 (4), 327-351 (2000).
- Lanciego, J. L., Wouterlood, F. G. Neuroanatomical tract-tracing techniques that did go viral. Brain Struct Funct. 225 (4), 1193-1224 (2020).
- Bakker, R., Wachtler, T., Diesmann, M. CoCoMac 2.0 and the future of tract-tracing databases. Front Neuroinform. 6, (2012).
- Watakabe, A., et al. Local and long-distance organization of prefrontal cortex circuits in the marmoset brain. Neuron. 111 (14), 2258-2273.e10 (2023).
- Skibbe, H., et al. The Brain/MINDS marmoset connectivity resource: An open-access platform for cellular-level tracing and tractography in the primate brain. PLoS Biol. 21 (6), e3002158(2023).
- Mitchell, J. F., Leopold, D. A. The marmoset monkey as a model for visual neuroscience. Neurosci Res. 93, 20-46 (2015).
- Okano, H. Current status of and perspectives on the application of marmosets in neurobiology. Annu Rev Neurosci. 44, 27-48 (2021).
- Watakabe, A., et al. Comparative analyses of adeno-associated viral vector serotypes 1, 2, 5, 8 and 9 in marmoset, mouse and macaque cerebral cortex. Neurosci Res. 93, 144-157 (2015).
- Hata, J., et al. Multi-modal brain magnetic resonance imaging database covering marmosets with a wide age range. Sci Data. 10, 221(2023).
- Liwang, J. K., Bennett, H. C., Pi, H. -J., Kim, Y. Protocol for using serial two-photon tomography to map cell types and cerebrovasculature at single-cell resolution in the whole adult mouse brain. STAR Protoc. 4 (1), 102048(2023).
- Okamoto, S., et al. Exclusive labeling of direct and indirect pathway neurons in the mouse neostriatum by an adeno-associated virus vector with Cre/lox system. STAR Protoc. 2 (1), 100230(2021).
- Viswanathan, S., et al. High-performance probes for light and electron microscopy. Nat Methods. 12 (6), 568-576 (2015).
- Kuan, L., et al. Neuroinformatics of the Allen mouse brain connectivity atlas. Methods. 73, 4-17 (2015).
- Paxinos, G., Watson, C., Petrides, M., Rosa, M., Tokuno, H. The Marmoset Brain in Stereotaxic Coordinates. , Academic Press. London, Waltham, MA. (2012).
- Abe, H., et al. 3D reconstruction of brain section images for creating axonal projection maps in marmosets. J Neurosci Methods. 286, 102-113 (2017).
- Wan, Y., et al. FluoRender: Joint freehand segmentation and visualization for many-channel fluorescence data analysis. BMC Bioinformatics. 18 (1), 280(2017).
- Majka, P., et al. Towards a comprehensive atlas of cortical connections in a primate brain: Mapping tracer injection studies of the common marmoset into a reference digital template. J Comp Neurol. 524 (11), 2161-2181 (2016).
- Liu, C., Yen, C. C. -C., Szczupak, D., Tian, X., Glen, D., Silva, A. C. Marmoset brain mapping V3: Population multi-modal standard volumetric and surface-based templates. Neuroimage. 226, 117620(2021).
- Liu, C., et al. A digital 3D atlas of the marmoset brain based on multi-modal MRI. Neuroimage. 169, 106-116 (2018).
- Liu, C., et al. A resource for the detailed 3D mapping of white matter pathways in the marmoset brain. Nat Neurosci. 23 (2), 271-280 (2020).
- Woodward, A., et al. The Brain/MINDS 3D digital marmoset brain atlas. Sci Data. 5, 180009(2018).
- Saleem, K. S., Avram, A. V., Glen, D., Schram, V., Basser, P. J. The subcortical atlas of the marmoset ("SAM") monkey based on high-resolution MRI and histology. Cereb Cortex. 34 (4), bhae120(2024).
- Watakabe, A., Hirokawa, J. Cortical networks of the mouse brain elaborate within the gray matter. Brain Struct Funct. 223 (8), 3633-3652 (2018).
- Lin, M. K., et al. A high-throughput neurohistological pipeline for brain-wide mesoscale connectivity mapping of the common marmoset. eLife. 8, e40042(2019).
- Skibbe, H., et al. PAT-probabilistic axon tracking for densely labeled neurons in large 3-D micrographs. IEEE Trans Med Imaging. 38 (1), 69-78 (2019).
- Economo, M. N., et al. A platform for brain-wide imaging and reconstruction of individual neurons. eLife. 5, e10566(2016).
- Winnubst, J., et al. Reconstruction of 1,000 projection neurons reveals new cell types and organization of long-range connectivity in the mouse brain. Cell. 179 (1), 268-281.e13 (2019).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon