Method Article
Tomographie à deux photons en série de l’ensemble du cerveau du ouistiti pour les analyses neuroanatomiques
Dans cet article
Résumé
L’imagerie par tomographie à deux photons en série (STPT) est une technique permettant d’imager une masse de tissu dans sa forme tridimensionnelle en combinant l’imagerie à deux photons avec le contrôle automatique de la platine et le tranchage par microtome. Nous décrivons ici un protocole permettant de l’implémenter pour les cerveaux de ouistitis afin de mieux comprendre leurs caractéristiques structurelles.
Résumé
La tomographie à deux photons en série (STPT) est une technique permettant d’imager une masse de tissu dans sa forme tridimensionnelle en combinant l’imagerie à deux photons avec le contrôle automatique de la platine et le découpage par microtome. Nous avons réussi à mettre en œuvre cette technique pour tracer les projections axonales dans le cerveau du ouistiti. Ici, les procédures expérimentales détaillées qui ont abouti à une imagerie volumétrique fiable de l’ensemble du cerveau du ouistiti sont décrites. L’un des processus clés d’une imagerie réussie a été l’ablation des méninges entourant le cerveau, qui interfèrent avec le découpage. Un grand avantage de cette méthodologie est que les sections tranchées peuvent être utilisées pour une coloration supplémentaire. Dans la configuration d’origine, les sections tranchées sont brouillées dans le bain-marie. Ces sections peuvent être correctement alignées dans leur ordre d’origine en fonction des modèles de vaisseaux sanguins dans le cortex. Un exemple d’histologie efficace est la visualisation de la structure de la myéline par simple réflexion de la lumière, qui peut être combinée avec la coloration de Nissl pour définir les limites anatomiques. Ces sections peuvent également être utilisées pour la détection immunologique de traceurs antérogrades et rétrogrades non fluorescents, qui peuvent être enregistrés dans les données STPT pour la superposition de plusieurs données.
Introduction
Dans les études neuroanatomiques, les chercheurs sont confrontés à la nécessité d’observer des structures d’ordre micrométrique (par exemple, des axones et des boutons) dans le contexte de l’ensemble du cerveau. Cette tâche difficile a généralement été abordée en inspectant visuellement les coupes en série et en recherchant la région d’intérêt pour une imagerie détaillée, des analyses et un enregistrement photo. Cependant, avec les progrès technologiques, il devient possible d’imager l’ensemble du cerveau à haute résolution pour l’analyse de l’ensemble du cerveau. Dans une étude révolutionnaire menée par Oh et al.1, des centaines de cerveaux de souris ont reçu des injections de traceur pour l’analyse connectomique, qui ont été traitées par la technique d’imagerie par tomographie à deux photons en série (STPT)2. La particularité de cette étude est d’avoir sélectionné des traceurs antérogrades pour quantifier la « connectivité ». Alors que les traceurs antérogrades fournissent des informations spatiales très détaillées sur la distribution axonale, les neuroanatomistes se sont appuyés sur une segmentation manuelle laborieuse pour son analyse. En automatisant diverses procédures analytiques, dont cette étape de segmentation, ils ont réussi la « production de masse » de données de traçage haute résolution prêtes à l’emploi pour des usages polyvalents. L’efficacité de leur approche est évidente, compte tenu d’une variété d’études qui ont utilisé leurs données de traçage 3,4,5 et leur cerveau standard6.
Historiquement, la connectivité neuronale des cerveaux de primates non humains a attiré l’attention de nombreux neuroanatomistes depuis le développement de la méthode de dégénérescence dans les années 1950, jusqu’aux méthodes de transport de substances antérogrades/rétrogrades dans les années 1970 jusqu’à la stratégie virale actuelle 7,8. En tant que tel, il existe de vastes documents qui étudient les connexions neuronales des primates. En particulier, de nombreux chercheurs ont étudié la connectivité cortico-corticorique complexe du cerveau du macaque, et leurs résultats ont été compilés (par exemple, CoCoMac9). Bien qu’utiles, ces études classiques présentaient plusieurs limites. Premièrement, parce que chaque étude ne se concentre que sur des régions cérébrales limitées, l’information obtenue devient inévitablement fragmentaire. Deuxièmement, chaque étude utilise des méthodes et des conditions différentes. Ainsi, l’évaluation quantitative entre les études devient compliquée. Troisièmement, la connectivité est généralement représentée soit sous la forme d’une camera lucida de sections représentatives, soit sous la forme d’un tableau/graphique semi-quantitatif pour les régions cérébrales définies par l’auteur. En d’autres termes, seules des informations très limitées provenant d’une structure cérébrale complexe sont extraites pour être présentées dans la littérature publiée. Avec le développement des techniques d’imagerie par résonance magnétique (IRM), les études du cerveau entier à basse résolution sont devenues importantes. Cependant, il existe un grand écart entre les niveaux de détail entre ce que nous savons des connexions neuronales chez les souris et celles chez les primates.
Avec ce contexte à l’esprit, nous avons entrepris d’effectuer un traçage antérograde complet des cerveaux de ouistitis communs10,11. Bien que beaucoup plus petit et plus lisse que le cerveau du macaque, l’homologue ouistiti présente des signes évidents de primates, tels que la présence de la zone MT et des zones préfrontales granulaires, qui ne sont pas clairement définies chez les rongeurs12,13. Ici, la petite taille était un grand avantage car même le cerveau du ouistiti pèse dix fois plus lourd que le cerveau de la souris. Heureusement, nous avons pu imager l’ensemble du cerveau du ouistiti avec des mises à jour minimales de la version originale du logiciel d’exploitation TissueCyte1000 (ci-après appelé système d’imagerie tissulaire complet), le microscope disponible dans le commerce pour l’imagerie STPT2. Cette mise à jour devait permettre un mouvement supplémentaire de la scène avant le découpage. La version actuelle est maintenant suffisante pour traiter le cerveau du ouistiti. Cet article présente un protocole permettant de manipuler le cerveau du ouistiti pour l’imagerie STPT. Le protocole post-imagerie qui améliore encore l’utilité de cette méthode est également fourni.
Protocole
Toutes les procédures expérimentales ont été réalisées conformément au Guide de l’Institut national de la santé pour le soin et l’utilisation des animaux de laboratoire (NIH Publications n° 80-23) révisé en 1996 et aux « Principes directeurs pour le soin et l’utilisation des animaux dans le domaine des sciences physiologiques » de la Société japonaise de physiologie, et ont été approuvés par le Comité des animaux de laboratoire du RIKEN (W2020-2-009(2)).
1. Injection de traceur
- Effectuer des injections de traceurs fluorescents et non fluorescents dans le cerveau du ouistiti conformément aux procédures précédemment rapportées14.
- En ce qui concerne la combinaison avec la méthode STPT, assurez-vous que la fluorescence des traceurs est suffisamment forte pour être détectée sans amélioration. Adoptez un système TET-Off à double vecteur10 pour une meilleure expression.
REMARQUE : Des traceurs non fluorescents peuvent également être injectés et détectés après la réalisation de STPT à l’aide des tranches produites. Parmi les exemples de traceurs non fluorescents, citons la dextran amine biotinylée (BDA), l’AAV exprimant la cre dans l’AAV2 rétro pour la détection rétrograde des noyaux de cellules d’entrée10, ainsi que les AAV smFP-tag basés sur la GFP (voir ci-dessous).
- En ce qui concerne la combinaison avec la méthode STPT, assurez-vous que la fluorescence des traceurs est suffisamment forte pour être détectée sans amélioration. Adoptez un système TET-Off à double vecteur10 pour une meilleure expression.
- Perfusion fixer et obtenir le cerveau du ouistiti après 4 semaines.
- Pour anesthésier le ouistiti, administrez par voie intramusculaire de la médétomidine (0,04 mg/kg), du midazolam (0,4 mg/kg) et du butorphanol (0,4 mg/kg), appelé MMB, suivi d’une injection intrapéritonéale de thiopental sodique (100 mg/kg).
- Après avoir confirmé que le réflexe de douleur est perdu, ouvrez la cavité thoracique pour exposer le cœur et coupez l’oreillette droite pour permettre au sang et au fixateur de sortir.
- Ensuite, coupez le ventricule gauche pour insérer l’aiguille de perfusion pour rincer d’abord le sang avec la solution de préfixe (250 mM de saccharose, 5 mM de MgCl2 dans un tampon de phosphate 0,02 M [PB, pH 7,4]) pendant quelques minutes (~50-100 mL), suivie d’une fixation avec un tampon de paraformaldéhyde à 4 %/phosphate à 0,1 M (PB ; 2 à 3 fois le poids de l’animal sur ~20 min).
- Une bonne perfusion est essentielle pour un bon résultat. Lors de l’insertion de l’aiguille de perfusion, avancez-la le long du septum vers l’aorte pour assurer un bon écoulement du fixateur. La pointe de l’aiguille devient visible lorsqu’elle atteint l’aorte. Rétractez-vous jusqu’à ce que l’extrémité soit à peine visible afin qu’elle ne contourne pas l’artère carotide.
- Maintenir le cerveau dans 4 % de paraformaldéhyde/0,1 M PB pendant 48 h à 4 °C et transférer à 50 mM PB. S’il n’est pas utilisé immédiatement, stockez le cerveau dans 0,75 % de glycine/0,1 M PB pour éviter l’autofluorescence due à une surfixation.
2. Préparation de l’échantillon (figure 1)
- Incuber le cerveau fixe avec de la collagénase (1 mg/mL dans 3 mM, CaCl2 dans 10 mL de solution saline tris-tamponnée [TBS])) à 37 °C pendant 1 h.
REMARQUE : Préchauffer le cerveau à 37 °C pendant 5 minutes avant l’incubation. - Frottez soigneusement la surface du cerveau avec un coton-tige pour enlever la matière de la pie, et les autres méninges (Figure 1A, vidéo supplémentaire). À l’aide d’une pince fine, pelez les méninges détachées.
REMARQUE : Il est important d’enlever les méninges entourant le mésencéphale, y compris le colliculus supérieur ou ceux au sommet du thalamus. Il est difficile d’exposer ces structures qui sont cachées au plus profond de l’intérieur. Cependant, enlevez les méninges autant que possible. Sinon, ils restent souvent non coupés, flottent vers le haut et interfèrent avec l’imagerie et le tranchage (Figure 2A). - Redoublez de prudence lorsque le cerveau est soumis à une IRM ex vivo avant l’intégration, au cours de laquelle le cerveau est immergé dans une solution de fluor15. Le fluor restant, s’il est intégré ensemble, pourrait former de minuscules bulles d’air sous l’objectif pendant l’imagerie. Pour éviter cela, gardez le cerveau en PB pendant 1 semaine avant de l’intégrer.
REMARQUE : Cette étape d’attente n’est généralement pas nécessaire mais est critique lorsqu’elle est associée à l’IRM ex vivo . - Préparez le tampon NaBH4 en dissolvant 0,2 g de NaBH4 dans 100 mL de tampon de borate de 50 mM (pH 9,2) chauffé à 40°C. Laissez le capuchon desserré pour laisser sortir le gaz CO2 . Conservez la solution toute la nuit avec le capuchon desserré. Enveloppez la bouteille dans du papier d’aluminium pour la protéger de la lumière jusqu’à l’utilisation.
- Préparez de l’agarose oxydé en mélangeant 2,25 g d’agarose et 0,21 g de NaIO4 dans 100 mL de PB pendant 2-3 h. Filtrez la solution avec une aspiration sous vide et lavez-la avec trois changements de PB. Remettre l’agarose en suspension dans 50 mL de PB.
- Faites fondre complètement l’agarose dans un four à micro-ondes et laissez-le refroidir à 60-65 °C.
- Placez le cerveau dans une chambre sur mesure et enfoncez le cerveau dans de l’agarose. Prenez soin d’introduire l’agarose dans la cavité située sous le corps calleux (Figure 1B).
REMARQUE : Préchauffez le cerveau d’abord à température ambiante, puis à 65 °C pendant 5 minutes pour permettre à l’agarose de se déposer à la surface du cerveau sans se solidifier. - Démontez la chambre et plongez le bloc d’agarose dans le tampon NaBH4 pendant une nuit à 4 °C.
- Changez le tampon en PB plusieurs fois pendant 1 à 2 semaines à 4 °C.
REMARQUE : L’autofluorescence tissulaire devient très faible si l’échange de tampon est incomplet. - Réalisez une scène en verre de diapositive en fixant quatre aimants en néodyme avec un mélange instantané d’époxy.
- Montez le bloc d’agarose sur la scène à l’aide d’un adhésif puissant (p. ex., de la super colle) (figure 1C).
3. Traitement des tissus
- Faites fonctionner l’ensemble du système d’imagerie tissulaire conformément aux instructions du fabricant. Un protocole détaillé est également publié16. La procédure actuelle est principalement utilisée pour TissueCyte1000 mais peut être appliquée à d’autres modèles, tels que TissueCyte 1600FC.
- Le fonctionnement est légèrement différent entre les modèles, mais suivez les points ci-dessous qui sont généralement importants.
- L’ensemble du cerveau du ouistiti peut être traité coronalement de l’extrémité antérieure à l’extrémité postérieure sans aucun changement dans le matériel. Assurez-vous que les limites de mouvement de la scène ne sont pas dépassées lors de la mise en place du cerveau.
- Ajustez l’angle des lames pour vous assurer que les profondeurs de surface aux deux extrémités de la lame sont inférieures à 10 μm. En raison de la myélinisation dense, même une différence de profondeur de 10 μm pourrait conduire à des pénétrations laser différentes à travers la substance blanche du ouistiti adulte. Pour la même raison, réglez le plan d’imagerie à environ 25-35 μm de la surface.
- À l’aide d’une lame en céramique (recommandée), coupez tout le cerveau à des intervalles de 50 μm (~plus de 650 tranches).
- Les méninges ne sont pas coupées et gênent le tranchage si elles ne sont pas correctement enlevées (figure 2). Certaines méninges (par exemple, celles qui entourent l’hippocampe ou le pulvinar) sont difficiles à enlever. Extrayez manuellement ces méninges à l’aide d’une pince fine lorsque vous vous en rendez compte.
4. Techniques histologiques auxiliaires
- Récupération de section
- Récupérer les coupes de tissu pour effectuer diverses colorations histologiques. Alignez avec précision ces sections dans l’ordre de tranchage. Tracez les vaisseaux sanguins à travers les couches corticales pour l’alignement (Figure 3).
- Coupez l’excès d’agarose de chaque section pour une meilleure manipulation.
REMARQUE : Ce processus n’a pas besoin d’être excessif. L’agarose sert à maintenir les segments de tissu en place sans interférer avec le traitement histologique. - Imagerie rétroéclairée
- Montez les sections sur une vitre coulissante et séchez-les.
- Réhydratez la section avec du PBS et placez la lamelle pour l’imagerie.
- Observez un motif de myélinisation sans coloration lorsque la coupe est imagée par un microscope à fluorescence (Figure 4) en mode fond clair. Ajustez le temps d’exposition de manière à ce que la réflexion de la lumière soit visible.
REMARQUE : N’importe quel microscope peut être utilisé s’il peut utiliser un éclairage en fond sombre. - Retirez la lamelle et procédez à la coloration Nissl ou à toute autre tache. N’importe quelle procédure de coloration standard est suffisante.
- Utilisez cette méthode pour comparer directement différents modèles de coloration avec les modèles de myélinisation (Figure 4).
- Imagerie BDA
- Voir le tableau 1 pour une procédure détaillée.
- Tout d’abord, traitez les sections avec la solution Dent’s (20 % DMSO, 80 % méthanol). Le méthanol inclus dans la solution de Dent améliore considérablement les signaux BDA dans les axones.
- Utilisez la méthode d’amplification du signal tyramide (TSA) pour améliorer le signal BDA à détecter par fluorescence17.
REMARQUE : La solution TSA-biotine peut être achetée auprès du fournisseur, mais le réactif interne est beaucoup moins cher et plus efficace. - Pour vous enregistrer sur l’image STPT, prenez une image rétroéclairée après avoir monté la section sur une vitre de diapositive avec le support de montage.
- Coloration de la protéine fluorescente Spaghetti Monster (smFP)
REMARQUE : Les protéines fluorescentes « monstre spaghetti » (smFPs) sont une famille de variantes GFP non fluorescentes avec plusieurs étiquettes d’épitopes18. Ils fournissent d’excellents signaux fluorescents lors de l’immunodétection et conviennent comme traceurs compagnons des traceurs fluorescents. Voir le tableau 2 pour les constructions AAV disponibles auprès d’Addgene.- Suivez la procédure détaillée fournie dans le Tableau 1.
5. Traitement des données post-imagerie
- Test d’intégrité des données
REMARQUE : STPT génère un grand nombre de fichiers image. Lorsque le cerveau d’un ouistiti adulte est sectionné coronalement à des intervalles de 50 μm, plus de 650 sections sont généralement nécessaires pour couvrir l’ensemble du cerveau. Les données de chaque section se composent d’une série d’images en mosaïque capturées dans trois canaux. Ces fichiers image sont stockés dans un seul dossier avec un fichier de métadonnées qui enregistre la position de chaque tuile. Pour minimiser la quantité totale de données et le temps de traitement, l’imagerie est réalisée par blocs d’exécutions, au cours desquels le nombre d’étapes X et Y pour la mosaïque change progressivement. En règle générale, 20 à 30 blocs d’analyses, chacun comprenant l’imagerie de 30 à 50 sections, constituent l’ensemble complet de données pour un seul cerveau. Pour utiliser ces données, plusieurs étapes de traitement d’image sont nécessaires, notamment l’assemblage des images en mosaïque, la segmentation des signaux fluorescents et l’enregistrement des données volumétriques dans un modèle standard, entre autres.- Effectuer les étapes de traitement d’images individuellement à l’aide de différents outils logiciels ou via des pipelines dédiés adaptés aux besoins spécifiques des chercheurs. Les détails du traitement d’image dépassent le cadre de cet article et peuvent être trouvés dans d’autres publications11,19.
- Quelle que soit la procédure utilisée, assurez-vous que les données sont organisées de manière cohérente.
REMARQUE : Étant donné que l’ensemble du processus d’imagerie implique de nombreux blocs de courses et s’étend sur plusieurs jours, il n’est pas rare qu’une série soit annulée à mi-chemin et recommencée à nouveau. Dans de tels cas, la structure des données peut être perturbée, ce qui entrave la bonne exécution du pipeline de traitement d’images. - Pour minimiser le risque d’interruption de la structure de données, exécutez un script Python pour vérifier l’intégrité de la structure de données (kn_pipeline_check_mosaic.py) disponible sur GitHub (github.com/watkarbey/STPT_depo).
- Recalage de l’image colorée à l’image STPT
REMARQUE : La transformation non linéaire de l’image colorée en image STPT peut être effectuée à l’aide du plugin bUnwarpJ d’ImageJ.- Ajustez la taille des images de la cible (STPT) et des images source (colorées) de manière à ce qu’elles soient à peu près égales.
- Binariser chaque image pour visualiser les formes des sections.
- Exécutez bUnwarpJ. N’oubliez pas de cocher l’option Enregistrer les transformations .
- Redémarrez bUnwarpJ . Sélectionnez l’image d’origine avant la binarisation comme image source. Cliquez ensuite sur Charger la transformation élastique et choisissez la transformation enregistrée.
Résultats
Dans la configuration typique utilisée ici, l’ensemble du cerveau d’un ouistiti adulte peut être imagé (Figure 5) à la résolution de ~1,3 x 1,3 μm/pixel avec un intervalle de section de 50 μm en environ 1 semaine. Cela équivaut à ~650 images coronales dans trois canaux après l’assemblage de l’image. Avec un objectif 16x (Nikon 16xW CFI75 LWD ; NA = 0,80), le champ de vision d’un seul tir est d’environ 1 x 1 mm. L’image de l’ensemble de la surface coronale est obtenue en assemblant ces clichés (Figure 5A). L’alignement dans la direction Z est excellent et une bonne image 3D est obtenue en empilant simplement les données de l’image coronale (Figure 5B,E). Pour la normalisation, le modèle STPT pour le cerveau du ouistiti11 peut être utilisé pour l’enregistrement 3D-3D (Figure 5F). Cette transformation des données est l’un des aspects clés de l’ensemble de la neuroanatomie du cerveau, dans laquelle une région d’intérêt se voit attribuer une coordonnée spatiale absolue indépendante de l’annotation anatomique. Une fois que le cerveau de l’échantillon est enregistré dans l’espace standard, il est facile de retirer les sous-régions d’intérêt pour une analyse plus approfondie (Figure 5F,G). En particulier, les régions corticales peuvent être transformées en une carte plate à l’aide d’un paramètre prédéterminé (Figure 5H).
Les coupes générées lors de l’imagerie peuvent être utilisées à diverses fins histologiques. Comme le montrent les figures 4A, B, l’imagerie rétroéclairée sans coloration fournit un motif très similaire à la coloration authentique de la myéline. Cela peut être une excellente alternative à la coloration à la myéline. De plus, si l’image rétroéclairée est obtenue avant la coloration Nissl, la même section peut être utilisée pour obtenir les motifs de la myéline et de la coloration Nissl, fournissant ainsi des informations utiles pour identifier les zones et les couches (Figure 5C-E). Ces sections peuvent également être utilisées pour la coloration immunologique. Dans la figure 5J, la section autour du centre d’injection a été contre-colorée avec de l’anticorps NeuN pour estimer l’efficacité de transduction du virus AAV. C’est aussi une bonne stratégie d’injecter des traceurs non fluorescents en plus des traceurs fluorescents et de les détecter histologiquement après le prélèvement de la section. Dans notre étude précédente, nous avons combiné des traceurs verts antérogrades avec un vecteur rétrograde « cre », qui a ensuite été détecté par l’anticorps anti-cre10. Dans un exemple de la figure 6A-E, le BDA a été injecté dans le côté controlatéral du traceur vert (trèfle) et l’a détecté par fluorescence. Notez que les signaux BDA rouges peuvent être enregistrés sur l’image TissueCyte pour être localisés dans toute la coordonnée du cerveau. Dans un autre exemple de la figure 6F-I, smFP-myc a été injecté dans la zone pariétale (figure 6G), tandis que le traceur vert a été injecté dans la zone frontale. De cette façon, plusieurs traceurs peuvent être injectés dans le même animal sans interférer avec l’imagerie. Un grand avantage de l’utilisation des sections STPT pour une coloration supplémentaire est que la relation entre les traceurs fluorescents et non fluorescents peut être déterminée pour le même cerveau. Ainsi, nous avons pu déterminer la réciprocité des projections cortico-artérielles à une haute précision10. Un autre avantage est que les coordonnées 3D des sections colorées peuvent être mappées à partir des données STPT, puis du modèle standard. Ainsi, il n’est peut-être pas nécessaire d’utiliser toutes les sections récupérées pour la coloration. Pour une meilleure interprétation, des sections peuvent être sélectionnées pour la coloration afin d’ajouter plus de contexte aux données STPT.
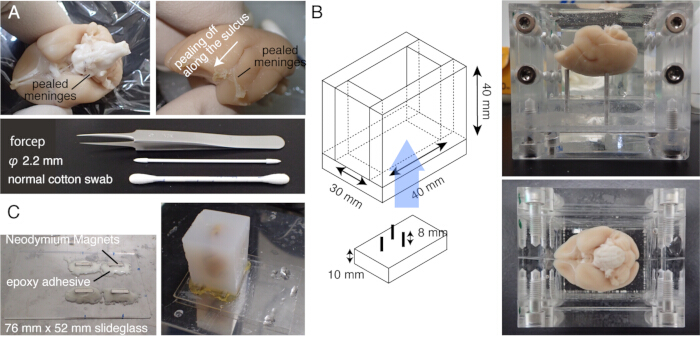
Figure 1 : Préparation de l’échantillon pour STPT. (A) Élimination de la méninge à l’aide de cotons-tiges. Les méninges entourant le tronc cérébral peuvent être enlevées à l’aide d’une pince à pointe fine. Les méninges entourant le cerveau du ouistiti sont enlevées manuellement en les frottant avec des cotons-tiges. La photo montre les méninges pelées à l’intérieur du sillon latéral. (B) La boîte en acrylique utilisée pour l’enrobage de l’agarose. Les broches sont mobiles et servent à ajuster l’angle du cerveau pour qu’il soit proche de la position stéréotaxique20. (C) Une diapositive magnétique composée de verre de diapositive de 76 mm x 52 mm et de quatre aimants en néodyme fixés par un adhésif époxy. Le bloc d’agarose est fixé à la platine magnétique avec de la super colle. Veuillez cliquer ici pour voir une version agrandie de cette figure.
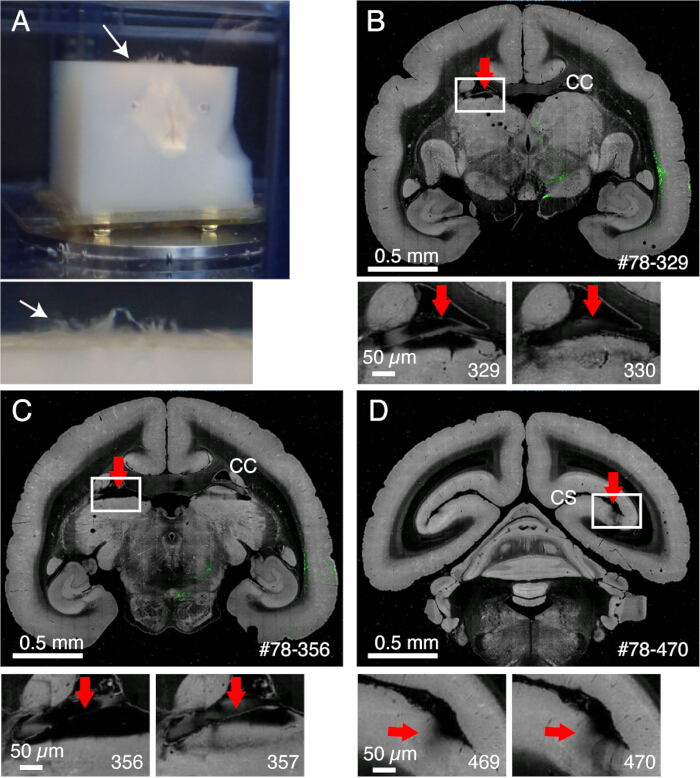
Figure 2 : L’effet des méninges sur l’imagerie. (A) Les méninges qui ne sont pas coupées flottent vers le haut, comme l’indique la flèche blanche. Dans cet exemple, on observe une saillie importante des méninges parce qu’elles n’ont pas été enlevées avant l’enrobage de l’agarose. Habituellement, les méninges ne restent non coupées que pour quelques régions difficiles. (B) Un exemple de mauvais tranchage dû à la présence de méninges entre le corps calleux et la partie supérieure du thalamus. Dans ce cas, un mauvais tranchage a conduit à l’alternance d’une tranche profonde (329) et d’une tranche relativement normale (330). #78-329 représente l’échantillon n° 78 section n° 329 dans le portail de données Brain/MINDS (C) Un autre exemple de mauvaise imagerie. Dans le pire des cas, le noyau pulvinar indiqué par la flèche rouge peut se détacher complètement. Barres d’échelle : 0,5 mm (panneau supérieur), 0,5 μm (panneau inférieur). (D) Un autre exemple de mauvaise imagerie. Les méninges profondes dans le sillon calcarin sont difficiles à enlever. Les ombres observées dans les sections 469 et 470 sont causées par les méninges flottantes qui se trouvaient sous l’objectif. Barres d’échelle : 0,5 mm (panneau supérieur), 0,5 μm (panneau inférieur). Veuillez cliquer ici pour voir une version agrandie de cette figure.
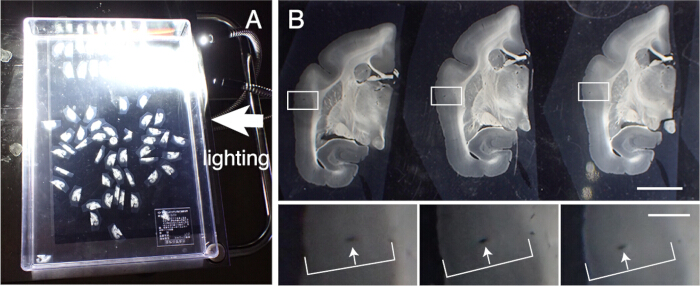
Figure 3 : Alignement des coupes de tissus dans l’ordre. (A) Jusqu’à 50 sections coronales de ouistitis peuvent être correctement alignées dans l’ordre dans un récipient en plastique (31 cm x 22,5 cm). Pour visualiser les structures détaillées, le conteneur est placé sur du papier noir et éclairé sur le côté. Ces sections sont d’abord grossièrement alignées dans l’ordre, puis soumises à un alignement précis. (B) L’alignement précis utilise le vaisseau sanguin comme marqueur. Le cortex cérébral contient de nombreux vaisseaux sanguins qui traversent verticalement les couches corticales. Ils sont identifiés comme des trous allongés qui changent systématiquement de position dans les couches corticales (flèches blanches). Grâce à cette méthode, même les sections avec des intervalles de 50 μm peuvent être alignées avec précision. Ces trous de vaisseaux sanguins peuvent être identifiés dans les images de la section SPTP pour confirmation. Barre d’échelle : 5 mm (panneau supérieur), 1 mm (panneau inférieur). Veuillez cliquer ici pour voir une version agrandie de cette figure.
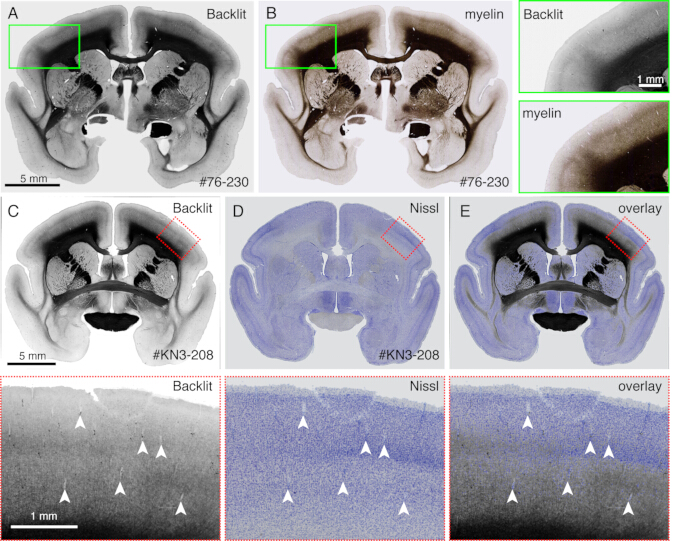
Figure 4 : Image rétroéclairée comme substitut de la coloration de la myéline. (A,B) La même section a été utilisée pour l’imagerie rétroéclairée et la coloration de la myéline. Tout d’abord, la section a été montée sur le verre de la lame, séchée, réhydratée avec du PBS, recouverte d’un lamelle et imagée à l’aide d’un microscope optique (Table des matériaux). Après avoir retiré la lamelle, la même section a été utilisée pour la coloration de la myéline21 et l’imagerie à l’aide d’un microscope à fluorescence. L’image rétroéclairée a été enregistrée sur l’image de myéline à l’aide du plugin bUnwarpJ d’ImageJ. Les cases vertes affichent les vues agrandies de chaque image. Notez que ces images montrent des motifs presque identiques, sauf que la coloration à la myéline visualise mieux les structures fibreuses. (C-E) La même section a été utilisée pour l’imagerie à contre-jour et la coloration Nissl. Le masque à bas seuil pour l’image rétroéclairée a d’abord été enregistré avec le masque à bas seuil pour l’image Nissl à l’aide du plugin bUnwarpJ, et l’image originale a été transformée à l’aide du même paramètre. Notez que les vaisseaux sanguins (pointes de flèches blanches) sont bien assortis entre les deux images. Parce que ces deux images sont pour la même section, l’appariement est presque parfait, et on peut comparer directement les motifs de myéline et de Nissl pour l’identification des couches corticales. Barres d’échelle : 5 mm (panneaux A-E). Veuillez cliquer ici pour voir une version agrandie de cette figure.
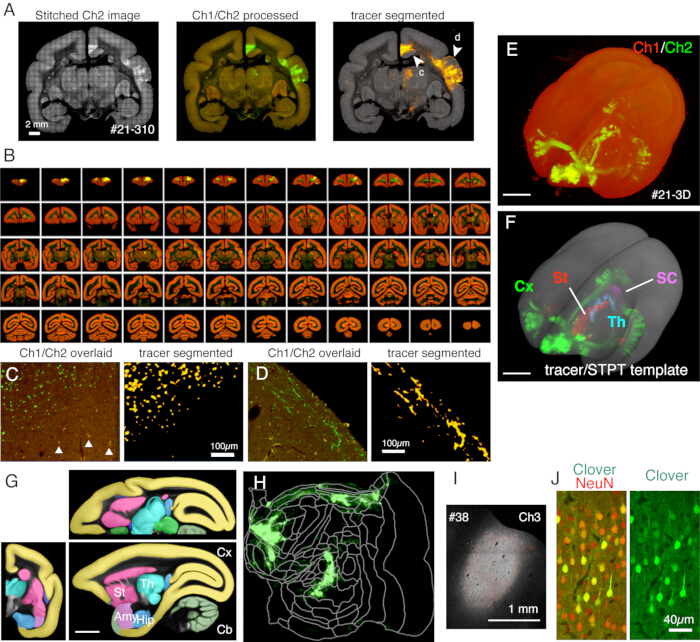
Figure 5 : Résultat typique de l’imagerie STPT. (A) Un simple pavage d’images Ch2 (en vert) d’une section d’exemple (section 310 de l’échantillon #21). Sans correction d’arrière-plan, les bordures de chaque tuile sont visibles. Dans la colonne du milieu, les tuiles ont été cousues avec une correction d’arrière-plan pour le Ch1 (rouge) et le Ch2 (vert), respectivement. Pour réduire les signaux de la lipofuscine (voir panneau C), les signaux Ch1 ont été soustraits de Ch2 et affichés en vert. Pour réduire les signaux de lipofuscine de Ch1, la commande « Remove Outilers.. » a été utilisée avant l’assemblage par ImageJ. Dans la colonne de droite, les signaux de suivi ont été segmentés par pipeline de traitement d’image11. Les pointes de flèches (c, d) indiquent les endroits où les vues agrandies sont affichées dans les panneaux C et D. Barre d’échelle : 2 mm. (B) Vue d’ensemble des coupes en série pour l’échantillon #21. STPT a généré 635 images coronales haute résolution pour cet échantillon. (C) Une vue à fort grossissement représentée par la pointe de flèche c dans le panneau A. Il s’agit d’une simple superposition de Ch1 (rouge) et Ch2 (vert) sans traitement supplémentaire. Les triangles montrent des signaux de fluorescence de lipofuscine, qui montrent un spectre étendu. L’algorithme de segmentation des traceurs distingue avec précision les signaux traceurs du fond de lipofuscine malgré une forme très similaire (panneau de droite). Barre d’échelle : 100 μm. (D) Un autre exemple de vue à fort grossissement. Notez que les fines fibres axonales de la couche 1 peuvent être bien visibles. Barre d’échelle : 100 μm. (E) La reconstruction 3D des images STPT originales. Les 635 images coronales à basse résolution présentées dans le panneau B ont été utilisées comme pile de fichiers pour la visualisation 3D à l’aide du fluorender22. Barre d’échelle : 5 mm. (F) La reconstruction 3D des signaux de traçage segmentés enregistrés dans le modèle STPT (gris). Les signaux de traçage dans différentes régions du cerveau étaient représentés par des couleurs différentes. Ces régions ont été découpées à l’aide de l’annotation illustrée dans le panneau G. Barre d’échelle : 5 mm. (G) Modèle STPT superposé avec des annotations de différentes régions du cerveau. Barre d’échelle : 5 mm. (H) Le signal de traçage cortical montré dans le panneau F a été montré sous la forme d’une carte plate. (I) Identification du site d’injection par la fluorescence Ch3, qui est moins sensible à la fluorescence du traceur et reste insaturée. Barre d’échelle : 1 mm. (J) La coloration de la section autour du centre d’injection avec l’anticorps NeuN a montré qu’environ 30 % des neurones présentent une forte expression de la fluorescence vert trèfle. Barre d’échelle : 40 μm. Abréviations : Cx ; cortex, St ; striatum, Th ; thalamus, SC ; colliculus supérieur. Amy; amygdale, hanche ; hippocampe, Cb ; cervelet. Veuillez cliquer ici pour voir une version agrandie de cette figure.
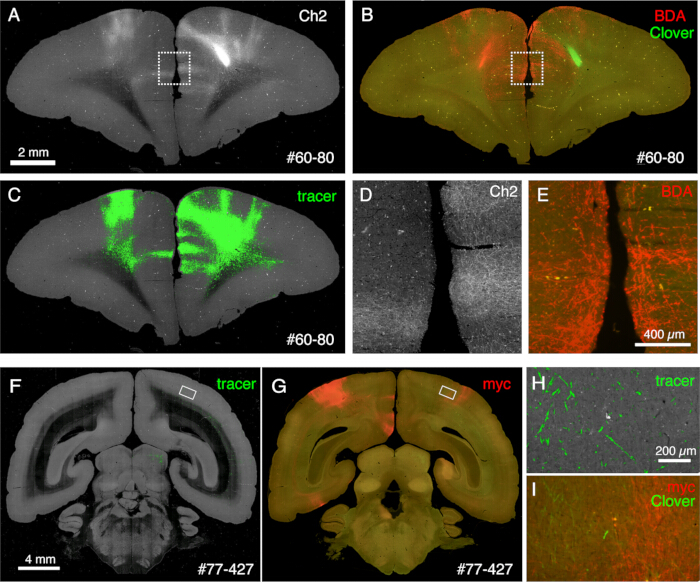
Figure 6 : Histologie post-STPT montrant plusieurs traceurs non fluorescents. (A-C) Comparaison de l’image STPT avec l’image colorée au BDA. Dans cet échantillon, le BDA est injecté dans le côté controlatéral de l’injection de trèfle. Les panneaux A et C montrent l’image Ch2 et la segmentation du traceur des données STPT. Le panneau B montre la coloration post-STPT pour le BDA. La fluorescence du trèfle est diminuée en raison du traitement au méthanol de la section. Barre d’échelle : 2 mm. (D,E) Les cases pointillées des panneaux A et B sont agrandies. Barre d’échelle : 100 μm. (F,G) Comparaison de l’image STPT avec coloration par anticorps anti-myc. Dans cet échantillon, l’injection de trèfle se fait dans le PFC, tandis que l’AAV-smFP-myc est injecté dans le cortex pariétal controlatéral. Les rectangles blancs sont agrandis dans les panneaux H et I. Barre d’échelle : 4 mm. (H) La segmentation du traceur est indiquée en vert. Barre d’échelle : 200 μm. (I) La coloration myc est représentée en rouge. La fluorescence du trèfle est représentée en vert. Barre d’échelle : 200 μm. Les signaux verts dans les panneaux H et I sont présents dans une position similaire mais pas identique car STPT ne récupère qu’une section optique de ~10 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Protocole de coloration fluorescente BDA | |
| Retirer l’agarose | |
| Lavage TBS | 10 min (2 fois) |
| 1 % H2O2 dans la solution de Dent | Durée : 10 minutes |
| Lavage TBS | Bref |
| 0,5 % de blocage des TNB | 1 h |
| StAvHRP (1:4000) en TNB | 2 nuit |
| Lavage TNT | 10 minutes (3x) |
| TSA Biotine (1:4000) dans 0,1 M borate (pH8,5) + 0,003 % H2O2 | 2 h |
| Lavage TNT | 10 minutes (3x) |
| Cy3-streptavidine (1:1000) dans la TNT | 3 h |
| Lavage TNT | 10 min (2 fois) |
| Lavage TBS | Conservez la section jusqu’au montage |
| Montez la section sur le slideglass à l’aide d’un support de montage anti-décoloration | |
| Protocole de coloration fluorescente anti-myc | |
| Retirer l’agarose | |
| Lavage TBS | 10 min (2 fois) |
| blocage dans IB | 1 h |
| Anti-Myc (1:4000) dans IB | 2 pendant la nuit |
| Lavage TNT | 10 minutes (3x) |
| Anti-souris Cy3 (1:1000) dans TNT | 3 h |
| Lavage TNT | 10 min (2 fois) |
| Lavage TBS | Conservez la section jusqu’au montage |
| Montez la section sur le slideglass à l’aide d’un support de montage anti-décoloration | |
| Tampons/solutions | Composition |
| 0,5 % TNB | 0,5 % de réactif bloquant TSA dans TS7.5 |
| La solution de Dent | 20 % DMSO, 80 % méthanol |
| Tampon d’immersion (IB) | 10 % FBS, 2 % BSA 0,5 % TritonX100 en TBS |
| TBS (solution saline tamponnée Tris) | 25 mM Tris, 137 mM NaCl, 2,7 mM KCl (pH 7,4) |
| TNT | 0,05 % Tween20 dans TS7.5 |
| TS7.5 | 0,1 M TRIS-HCl, pH 7,5, 0,15 M NaCl |
Tableau 1 : Protocole de coloration fluorescente BDA et de coloration fluorescente anti-myc
| Plasmide pour traceur AAV | Addgene : Non. | Anticorps recommandés | Dilution suggérée | Résultat attendu |
| pAAV-EF1_Cre | 201198 | Clone de Millipore 2D8 | 1:1000 | bon pour les cellules |
| pAAVCam1.3_smFP_Myc | 201205 | MBL M192 My3 (souris) | 1:4000 | Excellente |
| pAAVCam1.3_smFP_HA | 201206 | CST C29F4 (lapin) | 1:1000 | bon pour les cellules |
| pAAVCam1.3_smFP_FLAG | 201207 | MBL PM020B (lapin) | 1:1000 | D’ACCORD |
| AAVTRE3_smFP_Myc | 201208 | |||
| AAVTRE3_smFP_HA | 201209 | |||
| AAVTRE3_smFP_FLAG | 201210 |
Tableau 2 : Liste des plasmides Addgene disponibles pour la production de traceurs non fluorescents. La construction cre cible le noyau et convient au traçage rétrograde enveloppé par AAV2 rétro. smFP_HA construction est bonne pour la détection du corps cellulaire (et rétrograde).
Vidéo supplémentaire : Vue au microscope de l’ablation des méninges. Veuillez cliquer ici pour télécharger cette vidéo.
Discussion
Cet article expliquait les solutions pratiques pour manipuler les cerveaux de ouistitis pour le traitement du cerveau entier ainsi que les techniques histologiques auxiliaires qui améliorent l’utilité de la technique STPT. La force de la « neuroanatomie du cerveau entier » à l’aide de STPT est que vous pouvez obtenir les coordonnées 3D de n’importe quelle région d’intérêt, qu’elle soit anatomiquement annotée ou non. Grâce à un recalage 3D-3D de haute précision, il est possible de transformer ces coordonnées en un modèle standard pour la superposition de plusieurs ensembles de données. De cette façon, le modèle standard sert de support pour l’intégration des données. Il s’agissait d’un aspect essentiel de notre projet de cartographie du cortex préfrontal (PFC)10, où les données obtenues de nombreux individus ont été analysées. De plus, les informations mises en correspondance avec le modèle standard peuvent être comparées à diverses données qui ont déjà été cartographiées, qu’il s’agisse de Nissl, de motifs de myéline, de données de traçage, de données IRM (y compris l’IRM de diffusion) ou d’annotations anatomiques11. Il est important de noter qu’elles peuvent également être comparées aux données futures qui seront obtenues par la technologie qui n’a pas encore émergé. Il existe actuellement plusieurs modèles pour le cerveau du ouistiti, qui sont basés sur la coloration Nissl23, l’IRM23, 24, 25, 26, 27, 28 et STPT11. Mais ils peuvent être transformés aux coordonnées de l’autre en fonction des contrastes d’image et des paramètres précalculés11. L’intégration des données à l’échelle de l’ensemble du cerveau à travers les études contribue à une meilleure compréhension du cerveau en tant que système. La condition préalable au succès de cette stratégie est l’acquisition fiable de données dans tout le cerveau. Nous abordons ci-dessous les étapes critiques et les problèmes potentiels associés au protocole actuel.
L’un des processus les plus vulnérables de l’imagerie STPT est le tranchage tissulaire. Comme mentionné ci-dessus, les méninges restent souvent non coupées et peuvent interférer avec le tranchage. En particulier, le noyau pulvinar et le colliculus supérieur sont les deux régions cérébrales les plus touchées : en fonction de la minutie de l’élimination de la méninge et de l’enrobage dans l’agarose, elles peuvent être retirées du bloc tissulaire lors du tranchage. Ces régions profondes sont difficiles à approcher de l’extérieur et peuvent facilement se briser lors de l’ablation de la méninge. Une ablation minutieuse mais complète de la méninge est essentielle pour une imagerie réussie. Une autre préoccupation est le retournement des sections tranchées sur le bloc de tissu, ce qui se produit parfois lorsque les sections restent attachées même après le tranchage. Il peut être minimisé en façonnant le bloc de manière à ce que la lame coupe obliquement à la toute fin.
En imageant profondément dans le bloc tissulaire, STPT évite les bosses de sa surface. Alors que les signaux fluorescents peuvent traverser la région corticale assez facilement, ils sont fortement diminués dans les régions myélinisées. Par conséquent, la profondeur d’imagerie doit être soigneusement déterminée pour équilibrer l’imagerie cohérente sur toute la surface du bloc et la luminosité des signaux dans la région riche en myéline, comme la substance blanche. Dans la configuration utilisée ici, nous visons généralement à 25-35 μm de la surface. Il faut également noter que la surface coupée peut devenir inégalement rétrécie après de longues heures de stockage. Pour minimiser la zone d’imagerie, nous divisons la séance d’imagerie en 20 à 30 passages avec différents réglages de scène pendant 5 à 6 jours. Soit nous réduisons l’intervalle entre les descentes à moins de 2 h, soit nous confirmons la profondeur de la surface et ajustons la hauteur de la scène avant chaque descente.
Dans ce protocole, le signal BDA a été amplifié par la méthode TSA. Cette méthode est très efficace et permet de détecter les signaux BDA transportés antérogradement, même à une résolution relativement faible (p. ex., figure 6B). La biotine TSA est disponible dans le commerce auprès d’Akoya Biosciences, mais la solution maison montre une bien meilleure amélioration. D’autre part, la dilution de l’anticorps et de la solution nécessite un ajustement minutieux pour obtenir le résultat optimal. Le prétraitement de la section avec une solution de méthanol est essentiel. Sans prétraitement, les signaux BDA sont à peine détectés dans les axones myélinisés.
Lorsqu’un traceur antérograde est utilisé, il est souvent difficile d’identifier le site d’injection exact en raison de la saturation des signaux de fluorescence. Dans l’ensemble du système d’imagerie tissulaire utilisé dans cette étude, nous utilisons le canal bleu pour identifier les cellules infectées (Figure 5I). Même lorsque les canaux rouge et vert sont saturés, chaque neurone infecté est généralement détectable dans le canal bleu. C’est également le cas de l’Allen Mouse Brain Connectivity Atlas1. L’examen des cellules d’origine est important car l’infection n’implique parfois que des couches particulières. Nous avons rencontré de telles infections partielles assez fréquemment pour l’Atlas de connectivité cérébrale de souris, peut-être en raison de l’utilisation de la méthode d’ionophorèse29. La propagation latérale des traceurs viraux pourrait être plus ou moins variable en fonction des injections. Cette variabilité peut potentiellement affecter le résultat du traçage et nécessite une normalisation minutieuse.
L’enregistrement réussi de l’image 3D obtenue sur le modèle standard est un processus clé de l’ensemble de la neuroanatomie du cerveau. Le recalage de l’image STPT au modèle STPT peut être assez précis, et nous n’avons observé que des déviances de quelques voxels (isocubiques de 50 μm) pour les bordures avec un contraste d’image élevé10. Pourtant, il y a une limite à ce que l’inscription peut faire. En raison du processus d’élimination de la méninge, les échantillons STPT présentent généralement des espaces entre les hémisphères et entre le cortex et le mésencéphale/hérisse, tandis que les tissus cérébraux sont étroitement regroupés dans les images IRM in vivo . De telles différences sont difficiles à corriger par l’enregistrement. Le cortex du ouistiti est la plupart du temps dépourvu de sillons, mais le sillon intrapariétal est très profond chez certains individus. Un tel sillon sera perdu (fusion de haut en haut) lors de l’enregistrement. Bien que l’enregistrement soit une technique puissante, il est nécessaire de revenir sur les données brutes pour confirmer le résultat obtenu.
La génération de données d’images massives est à la fois une force et une limite de cette technique. Bien qu’il améliore l’exhaustivité de l’ensemble de données, il nécessite une gestion minutieuse des données acquises et le développement d’un pipeline de traitement d’images automatisé pour une interprétation efficace des données. À l’avenir, l’application de l’intelligence artificielle (IA) générative dans la construction de pipelines de traitement d’images pourrait simplifier considérablement ce processus. Une imagerie systématique du cerveau entier a également été réalisée à l’aide de méthodes basées sur un scanner de lames21,30. Par rapport à de telles méthodes, STPT ne nécessite pas de calcul supplémentaire pour la reconstruction 3D. Combiné à la coupe optique, nous avons démontré que STPT a le potentiel de reconstruire même des segments axonaux sur des sections31. Avec la combinaison supplémentaire de techniques d’élimination des tissus, Economo et al. ont développé une méthode pour imager l’intégralité des neurones peu marqués32,33. Les dernières versions de TissueCyte offrent désormais des options pour un laser supplémentaire afin d’améliorer l’excitation des protéines fluorescentes rouges ou une unité de capture de section pour la récupération automatique des sections. Grâce à ces progrès, l’approche du cerveau entier deviendra plus efficace, fournissant une base pour une compréhension complète du cerveau des primates, y compris celui des humains.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous remercions vivement le personnel technique du laboratoire et des animaleries de Yamamori (RRD) pour leur aide. Nous remercions le centre de collaboration RIKEN CBS-Olympus pour l’assistance technique fournie pour l’acquisition d’images confocales. Ce travail a été soutenu par le programme de recherche scientifique sur les domaines innovants (numéro de subvention 22123009) de MEXT, Japon, par Brain/MINDS et Brain/MINDS2.0 d’AMED, Japon (JP15dm0207001, JP23wm0625001 et JP24wm0625218), et par JSPS KAKENHI numéro de subvention 24K09678 à A.W.
matériels
| Name | Company | Catalog Number | Comments |
| Agarose type I | Sigma | A6013 | |
| All-in-one fluorescence microscope | Keyence | BZ-X series | This study used BZ-X710 |
| anti-mouse Cy3 | Jackson Immuno Research Laboratories, Inc. | 115-165-003 | |
| anti-myc antibody | MBL | M192 | mouse monoclonal |
| Butorphanol | Meiji Animal Health | Vetorphale | |
| Ceramic blade | TissueVision | N/A | |
| Collagenase type I | Wako Chemical | #031-17601 | Aliquot into 100 µL x 10 of 100 mg/mL in PBS |
| Cotton swab (HUBY-340) | HUBY | BB-013SP | micro cotton swabs |
| Custom-made chamber | N/A | N/A | The chamber consist of four side plates and one bottom plates, all made of 1 cm thick acrylic plates, which can be securely fastened together using screw bolts. An additional acrylic plate is placed inside the chamber, with three metal poles inserted through it to hold the marmoset brain in an inverted position. |
| Cy3-Streptavidin | Jackson Immuno Research Laboratories, Inc. | 016-160-084 | |
| Dumont #5 forceps, Inox | N/A | N/A | |
| Epoxy instant mix | Locktite | N/A | |
| FIJI | NIH | https://imagej.net/software/fiji/downloads | |
| kn_pipeline_check_mosaic.py | N/A | N/A | github.com/watkarbey/STPT_depo; This is a checking script for TissueCyte data. |
| Medetomidine | Zenoaq | Domitor | |
| Midazolam | Sandoz | N/A | |
| NaBH4 | Sigma | #452882 | |
| NaIO4 | Sigma | S1878 | |
| Perfusion needle | Natsume Seisakusho Co., Ltd | KN-348, 20G-50 | |
| Slideglass (76 mm x 52 mm) | Matsunami | S9111 | |
| StAvHRP | Jackson Immuno Research Laboratories, Inc. | 016-030-084 | HRP-streptavidin (titration needs to be carefully determined) |
| TissueCyte1000 | TissueVision | https://www.tissuevision.com/tissuecyte | |
| TSA blocking reagent | Kiko Tech | FP1012 | Refer to TSA-biotin kit (Akoya Biosciences) |
| TSA-biotin | House-made | N/A | See Okamoto et al (ref. 16) |
| VECTASHIELD HardSet Mounting Medium | Vector laboratories Inc | N/A | Antifade mounting medium |
Références
- Oh, S. W., Harris, J. A., Ng, L., Winslow, B., Cain, N., et al. A mesoscale connectome of the mouse brain. Nature. 508 (7495), 207-214 (2014).
- Ragan, T., et al. Serial two-photon tomography for automated ex vivo mouse brain imaging. Nat Methods. 9 (3), 255-258 (2012).
- Liang, Z., Arefin, T. M., Lee, C. H., Zhang, J. Using mesoscopic tract-tracing data to guide the estimation of fiber orientation distributions in the mouse brain from diffusion MRI. Neuroimage. 270, 119999 (2023).
- Watakabe, A., Hirokawa, J. Cortical networks of the mouse brain elaborate within the gray matter. Brain Struct Funct. 223 (8), 3633-3652 (2018).
- Ypma, R. J. F., Bullmore, E. T. Statistical analysis of tract-tracing experiments demonstrates a dense, complex cortical network in the mouse. PLoS Comput Biol. 12 (9), e1005104 (2016).
- Wang, Q., et al. The Allen mouse brain common coordinate framework: A 3D reference atlas. Cell. 181 (4), 936-953.e20 (2020).
- Köbbert, C., Apps, R., Bechmann, I., Lanciego, J. L., Mey, J., Thanos, S. Current concepts in neuroanatomical tracing. Prog Neurobiol. 62 (4), 327-351 (2000).
- Lanciego, J. L., Wouterlood, F. G. Neuroanatomical tract-tracing techniques that did go viral. Brain Struct Funct. 225 (4), 1193-1224 (2020).
- Bakker, R., Wachtler, T., Diesmann, M. CoCoMac 2.0 and the future of tract-tracing databases. Front Neuroinform. 6, (2012).
- Watakabe, A., et al. Local and long-distance organization of prefrontal cortex circuits in the marmoset brain. Neuron. 111 (14), 2258-2273.e10 (2023).
- Skibbe, H., et al. The Brain/MINDS marmoset connectivity resource: An open-access platform for cellular-level tracing and tractography in the primate brain. PLoS Biol. 21 (6), e3002158 (2023).
- Mitchell, J. F., Leopold, D. A. The marmoset monkey as a model for visual neuroscience. Neurosci Res. 93, 20-46 (2015).
- Okano, H. Current status of and perspectives on the application of marmosets in neurobiology. Annu Rev Neurosci. 44, 27-48 (2021).
- Watakabe, A., et al. Comparative analyses of adeno-associated viral vector serotypes 1, 2, 5, 8 and 9 in marmoset, mouse and macaque cerebral cortex. Neurosci Res. 93, 144-157 (2015).
- Hata, J., et al. Multi-modal brain magnetic resonance imaging database covering marmosets with a wide age range. Sci Data. 10, 221 (2023).
- Liwang, J. K., Bennett, H. C., Pi, H. -. J., Kim, Y. Protocol for using serial two-photon tomography to map cell types and cerebrovasculature at single-cell resolution in the whole adult mouse brain. STAR Protoc. 4 (1), 102048 (2023).
- Okamoto, S., et al. Exclusive labeling of direct and indirect pathway neurons in the mouse neostriatum by an adeno-associated virus vector with Cre/lox system. STAR Protoc. 2 (1), 100230 (2021).
- Viswanathan, S., et al. High-performance probes for light and electron microscopy. Nat Methods. 12 (6), 568-576 (2015).
- Kuan, L., et al. Neuroinformatics of the Allen mouse brain connectivity atlas. Methods. 73, 4-17 (2015).
- Paxinos, G., Watson, C., Petrides, M., Rosa, M., Tokuno, H. . The Marmoset Brain in Stereotaxic Coordinates. , (2012).
- Abe, H., et al. 3D reconstruction of brain section images for creating axonal projection maps in marmosets. J Neurosci Methods. 286, 102-113 (2017).
- Wan, Y., et al. FluoRender: Joint freehand segmentation and visualization for many-channel fluorescence data analysis. BMC Bioinformatics. 18 (1), 280 (2017).
- Majka, P., et al. Towards a comprehensive atlas of cortical connections in a primate brain: Mapping tracer injection studies of the common marmoset into a reference digital template. J Comp Neurol. 524 (11), 2161-2181 (2016).
- Liu, C., Yen, C. C. -. C., Szczupak, D., Tian, X., Glen, D., Silva, A. C. Marmoset brain mapping V3: Population multi-modal standard volumetric and surface-based templates. Neuroimage. 226, 117620 (2021).
- Liu, C., et al. A digital 3D atlas of the marmoset brain based on multi-modal MRI. Neuroimage. 169, 106-116 (2018).
- Liu, C., et al. A resource for the detailed 3D mapping of white matter pathways in the marmoset brain. Nat Neurosci. 23 (2), 271-280 (2020).
- Woodward, A., et al. The Brain/MINDS 3D digital marmoset brain atlas. Sci Data. 5, 180009 (2018).
- Saleem, K. S., Avram, A. V., Glen, D., Schram, V., Basser, P. J. The subcortical atlas of the marmoset ("SAM") monkey based on high-resolution MRI and histology. Cereb Cortex. 34 (4), bhae120 (2024).
- Watakabe, A., Hirokawa, J. Cortical networks of the mouse brain elaborate within the gray matter. Brain Struct Funct. 223 (8), 3633-3652 (2018).
- Lin, M. K., et al. A high-throughput neurohistological pipeline for brain-wide mesoscale connectivity mapping of the common marmoset. eLife. 8, e40042 (2019).
- Skibbe, H., et al. PAT-probabilistic axon tracking for densely labeled neurons in large 3-D micrographs. IEEE Trans Med Imaging. 38 (1), 69-78 (2019).
- Economo, M. N., et al. A platform for brain-wide imaging and reconstruction of individual neurons. eLife. 5, e10566 (2016).
- Winnubst, J., et al. Reconstruction of 1,000 projection neurons reveals new cell types and organization of long-range connectivity in the mouse brain. Cell. 179 (1), 268-281.e13 (2019).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon