Method Article
Tomografia seriada de dois fótons de todo o cérebro do sagui para análises neuroanatômicas
Neste Artigo
Resumo
A tomografia serial de dois fótons (STPT) é uma técnica para obter imagens de uma massa de tecido em sua forma tridimensional, combinando imagens de dois fótons com controle automático de estágio e corte de micrótomo. Aqui descrevemos um protocolo para implementá-lo para cérebros de saguis para entender melhor suas características estruturais.
Resumo
A tomografia serial de dois fótons (STPT) é uma técnica para obter imagens de uma massa de tecido em sua forma tridimensional, combinando imagens de dois fótons com controle automático de estágio e corte de micrótomo. Implementamos com sucesso essa técnica para traçar as projeções axonais no cérebro do sagui. Aqui, os procedimentos experimentais detalhados que resultaram em imagens volumétricas confiáveis de todo o cérebro do sagui são descritos. Um processo chave para o sucesso da imagem foi a remoção de meninges ao redor do cérebro, o que interfere no corte. Uma grande vantagem dessa metodologia é que as seções fatiadas podem ser usadas para coloração adicional. Na configuração original, as seções fatiadas são mexidas no banho-maria. Essas seções podem ser alinhadas corretamente em sua ordem original de acordo com os padrões dos vasos sanguíneos no córtex. Um exemplo de histologia eficaz é a visualização da estrutura da mielina por simples reflexão da luz, que pode ser combinada com a coloração de Nissl para definir as bordas anatômicas. Essas seções também podem ser usadas para detecção imunológica de traçadores anterógrados e retrógrados não fluorescentes, que podem ser registrados nos dados STPT para estratificação de vários dados.
Introdução
Em estudos neuroanatômicos, os pesquisadores se deparam com a necessidade de observar estruturas de ordem micrométrica (por exemplo, axônios e botões) no contexto de todo o cérebro. Essa tarefa difícil tem sido normalmente abordada inspecionando visualmente as seções de série e procurando a região de interesse para imagens detalhadas, análises e gravação de fotos. Com o avanço tecnológico, no entanto, está se tornando possível obter imagens de todo o cérebro em alta resolução para análise de todo o cérebro. Em um estudo inovador de Oh et al.1, centenas de cérebros de camundongos receberam injeções de traçadores para análise conectômica, que foram processadas pela técnica de imagem de tomografia serial de dois fótons (STPT)2. A característica deste estudo foi que eles selecionaram traçadores anterógrados para quantificar a "conectividade". Embora os traçadores anterógrados forneçam informações espaciais muito detalhadas sobre a distribuição axonal, os neuroanatomistas têm contado com a laboriosa segmentação manual para sua análise. Ao automatizar vários procedimentos analíticos, incluindo esta etapa de segmentação, eles conseguiram a "produção em massa" de dados traçadores de alta resolução prontos para uso para usos multifuncionais. A eficácia de sua abordagem é óbvia, dada uma variedade de estudos que usaram seus dados de rastreamento 3,4,5 e seu cérebro padrão6.
Historicamente, a conectividade neural de cérebros de primatas não humanos atraiu a atenção de muitos neuroanatomistas desde o desenvolvimento do método de degeneração na década de 1950, passando pelos métodos de transporte de substâncias anterógrados / retrógrados na década de 1970 até a presente estratégia viral 7,8. Como tal, existem vastas peças de literatura que investigam as conexões neurais dos primatas. Em particular, muitos pesquisadores investigaram a complexa conectividade corticocortical do cérebro do macaco e seus resultados foram selecionados para tabulação (por exemplo, CoCoMac9). Embora úteis, esses estudos clássicos tinham várias limitações. Primeiro, como cada estudo se concentra apenas em regiões cerebrais limitadas, as informações obtidas inevitavelmente se tornam fragmentadas. Em segundo lugar, cada estudo usa métodos e condições diferentes. Assim, a avaliação quantitativa entre os estudos torna-se complicada. Em terceiro lugar, a conectividade geralmente é mostrada como uma câmera lúcida de seções representativas ou como uma tabela/gráfico semiquantitativo para regiões cerebrais definidas pelo autor. Em outras palavras, apenas informações muito limitadas de uma estrutura cerebral complexa são extraídas para apresentação na literatura publicada. Com o desenvolvimento das técnicas de ressonância magnética (MRI), os estudos de todo o cérebro em baixa resolução tornaram-se proeminentes. No entanto, há uma grande lacuna entre os níveis de detalhe entre o que sabemos sobre conexões neurais em camundongos e em primatas.
Com esse histórico em mente, nos propusemos a realizar um rastreamento anterógrado abrangente dos cérebros de saguis comuns10,11. Embora muito menor e mais liso do que o cérebro do macaco, a contraparte do sagui exibe sinais claros de primatas, como a presença da área MT e as áreas pré-frontais granulares, nenhuma das quais é claramente definida em roedores12,13. Aqui, o tamanho pequeno foi uma grande vantagem porque até o cérebro do sagui pesa dez vezes mais do que o cérebro do camundongo. Felizmente, pudemos obter imagens de todo o cérebro do sagui com atualizações mínimas da versão original do software operacional TissueCyte1000 (doravante referido como um sistema de imagem de tecido inteiro), o microscópio disponível comercialmente para imagens STPT2. Esta atualização foi para permitir o movimento extra do estágio antes de fatiar. A versão atual agora é suficiente para processar o cérebro do sagui. Este artigo compartilha um protocolo para lidar com o cérebro do sagui para imagens de STPT. O protocolo de pós-imagem que aumenta ainda mais a utilidade desse método também é fornecido.
Protocolo
Todos os procedimentos experimentais foram realizados seguindo o Guia do Instituto Nacional de Saúde para o Cuidado e Uso de Animais de Laboratório (NIH Publications No. 80-23) revisado em 1996 e os "Princípios Orientadores para o Cuidado e Uso de Animais no Campo da Ciência Fisiológica" da Sociedade Japonesa de Fisiologia, e foram aprovados pelo Comitê de Animais Experimentais do RIKEN (W2020-2-009(2)).
1. Injeção de traçador
- Realizar injeções de marcadores fluorescentes e não fluorescentes no cérebro do sagui de acordo com procedimentos relatados anteriormente14.
- Em relação à combinação com o método STPT, certifique-se de que a fluorescência dos traçadores seja forte o suficiente para ser detectada sem aprimoramento. Adote um sistema TET-Off de vetor duplo10 para expressão aprimorada.
NOTA: Traçadores não fluorescentes também podem ser injetados e detectados após o STPT ser feito usando as fatias produzidas. Exemplos de traçadores não fluorescentes incluem Amina Dextrana Biotinilada (BDA), AAV que expressa cre em AAV2 retro para detecção retrógrada dos núcleos da célula de entrada10, bem como AAVs de tag smFP baseados em GFP (veja abaixo).
- Em relação à combinação com o método STPT, certifique-se de que a fluorescência dos traçadores seja forte o suficiente para ser detectada sem aprimoramento. Adote um sistema TET-Off de vetor duplo10 para expressão aprimorada.
- Correção de perfusão e obtenção do cérebro do sagui após 4 semanas.
- Para anestesiar o sagui, administrar Medetomidina (0,04 mg/kg), midazolam (0,4 mg/kg) e butorfanol (0,4 mg/kg), denominados MMB, por via intramuscular, seguidos de injeção intraperitoneal de Tiopental Sódico (100 mg/kg).
- Depois de confirmar que o reflexo da dor foi perdido, abra a cavidade torácica para expor o coração e corte o átrio direito para permitir que o sangue e o fixador saiam.
- Em seguida, corte o ventrículo esquerdo para inserir a agulha de perfusão para primeiro lavar o sangue com a solução de prefixo (250 mM de sacarose, 5 mM de MgCl2 em tampão fosfato 0,02 M [PB, pH 7,4]) por alguns minutos (~ 50-100 mL), seguido de fixação com paraformaldeído a 4% / tampão fosfato 0,1 M (PB; 2-3 vezes o peso do animal em ~ 20 min).
- Uma boa perfusão é fundamental para um bom resultado. Ao inserir a agulha de perfusão, avance-a ao longo do septo em direção à aorta para garantir um bom fluxo do fixador. A ponta da agulha torna-se visível quando atinge a aorta. Retraia até que a ponta fique pouco visível para não contornar a artéria carótida.
- Mantenha o cérebro em 4% de paraformaldeído / 0,1 M PB por 48 h a 4 ° C e transfira para 50 mM PB. Se não for usado imediatamente, armazene o cérebro em 0,75% de glicina / 0,1 M PB para evitar a autofluorescência devido à fixação excessiva.
2. Preparo da amostra (Figura 1)
- Incubar o cérebro fixo com colagenase (1 mg / mL em 3 mM CaCl2 em 10 mL de solução salina tamponada com tris [TBS]) a 37 ° C por 1 h.
NOTA: Pré-aqueça o cérebro a 37 ° C por 5 minutos antes da incubação. - Esfregue cuidadosamente a superfície do cérebro com um cotonete para retirar a matéria pia e outras meninges (Figura 1A, vídeo suplementar). Use uma pinça fina para descascar as meninges destacadas.
NOTA: É importante remover as meninges ao redor do mesencéfalo, incluindo o colículo superior ou as da parte superior do tálamo. É difícil expor essas estruturas que estão escondidas no fundo. No entanto, remova as meninges o máximo possível. Caso contrário, eles geralmente permanecem sem cortes, flutuam e interferem na imagem e no corte (Figura 2A). - Tome cuidado extra quando o cérebro for submetido a ressonância magnética ex vivo antes da incorporação, durante a qual o cérebro é imerso em solução de flúor15. O flúor restante, se incorporado, pode formar pequenas bolhas de ar sob a objetiva durante a imagem. Para evitar isso, mantenha o cérebro em PB por 1 semana antes de incorporar.
NOTA: Esta etapa de espera geralmente não é necessária, mas é crítica quando combinada com ressonância magnética ex vivo . - Prepare o tampão NaBH4 dissolvendo 0,2 g de NaBH4 em 100 mL de tampão borato 50 mM (pH 9,2) aquecido a 40 ° C. Deixe a tampa solta para deixar o gás CO2 sair. Mantenha a solução durante a noite com a tampa solta. Embrulhe a garrafa em papel alumínio para protegê-la da luz até o uso.
- Faça agarose oxidada mexendo 2,25 g de agarose e 0,21 g de NaIO4 em 100 mL de PB por 2-3 h. Filtrar a solução com aspiração a vácuo e lavá-la com três mudanças do PB. Ressuspenda a agarose em 50 mL de PB.
- Derreta completamente a agarose em um forno de micro-ondas e deixe esfriar a 60-65 °C.
- Coloque o cérebro em uma câmara feita sob medida e incorpore o cérebro em agarose. Tome cuidado para introduzir a agarose na cavidade abaixo do corpo caloso (Figura 1B).
NOTA: Pré-aqueça o cérebro primeiro à temperatura ambiente e depois a 65 ° C por 5 min para permitir que a agarose se deposite na superfície do cérebro sem solidificar. - Desmontar a câmara e mergulhar o bloco de agarose no tampão NaBH4 durante a noite a 4 °C.
- Alterar o tampão para PB várias vezes ao longo de 1-2 semanas a 4 °C.
NOTA: A autofluorescência do tecido torna-se muito fraca se a troca de tampão estiver incompleta. - Faça um palco de vidro deslizante anexando quatro ímãs de neodímio com mistura instantânea de epóxi.
- Monte o bloco de agarose no palco com um adesivo forte (por exemplo, super cola) (Figura 1C).
3. Processamento de tecidos
- Opere todo o sistema de imagem de tecido de acordo com as instruções do fabricante. Um protocolo detalhado também é publicado16. O procedimento atual é usado principalmente para o TissueCyte1000, mas pode ser aplicado a outros modelos, como o TissueCyte 1600FC.
- A operação é um pouco diferente entre os modelos, mas siga os pontos abaixo que são comumente importantes.
- Todo o cérebro do sagui pode ser processado coronalmente das extremidades anterior para posterior sem nenhuma alteração no hardware. Certifique-se de que os limites de movimento do palco não sejam excedidos ao colocar o cérebro.
- Ajuste o ângulo das lâminas para garantir que as profundidades da superfície em ambas as extremidades da lâmina estejam dentro de 10 μm. Por causa da mielinização densa, mesmo uma diferença de 10 μm na profundidade pode levar a diferentes penetrações de laser através da substância branca do sagui adulto. Pelo mesmo motivo, defina o plano de imagem em cerca de 25-35 μm da superfície.
- Use uma lâmina de cerâmica (recomendado) para cortar todo o cérebro em intervalos de 50 μm (~ mais de 650 fatias).
- As meninges permanecem sem corte e interferem no corte se não forem removidas adequadamente (Figura 2). Algumas meninges (por exemplo, aquelas ao redor do hipocampo ou pulvinar) são difíceis de remover. Extraia manualmente essas meninges com uma pinça fina quando notado.
4. Técnicas histológicas auxiliares
- Recuperação de seção
- Recupere as fatias de tecido para realizar várias colorações histológicas. Alinhe com precisão essas seções em ordem de fatiamento. Trace os vasos sanguíneos através das camadas corticais para alinhamento (Figura 3).
- Corte o excesso de agarose de cada seção para melhor manuseio.
NOTA: Este processo não precisa ser excessivo. A agarose serve para manter os segmentos de tecido no lugar sem interferir no processamento histológico. - Imagem retroiluminada
- Monte as seções em um vidro deslizante e seque.
- Reidrate a seção com PBS e coloque a lamínula para imagem.
- Observe um padrão de mielinização sem coloração quando a seção é visualizada por um microscópio de fluorescência (Figura 4) em um modo de campo claro. Ajuste o tempo de exposição para que o reflexo da luz possa ser visível.
NOTA: Qualquer microscópio pode ser usado se puder usar iluminação de campo escuro. - Remova a lamínula e prossiga para a coloração de Nissl ou qualquer outra coloração. Qualquer procedimento de coloração padrão é suficiente.
- Use este método para comparar diretamente diferentes padrões de coloração com os padrões de mielinização (Figura 4).
- Imagem BDA
- Consulte a Tabela 1 para um procedimento detalhado.
- Primeiro, trate as seções com solução de Dent (20% DMSO, 80% de metanol). O metanol incluído na solução de Dent aumenta drasticamente os sinais de BDA nos axônios.
- Use o método de amplificação de sinal de tiramida (TSA) para aumentar o sinal BDA a ser detectado fluorescentemente17.
NOTA: A solução de biotina TSA pode ser adquirida do fornecedor, mas o reagente interno é muito mais barato e eficaz. - Para registrar a imagem STPT, tire uma imagem retroiluminada depois de montar a seção em um vidro deslizante com a mídia de montagem.
- Coloração de proteína fluorescente de monstro espaguete (smFP)
NOTA: As proteínas fluorescentes do 'monstro espaguete' (smFPs) são uma família de variantes GFP não fluorescentes com várias tags de epítopos18. Eles fornecem excelentes sinais fluorescentes após a imunodetecção e são adequados como rastreadores complementares aos traçadores fluorescentes. Consulte a Tabela 2 para as construções AAV disponíveis no Addgene.- Siga o procedimento detalhado fornecido na Tabela 1.
5. Processamento de dados pós-imagem
- Teste de integridade de dados
NOTA: O STPT gera um grande número de arquivos de imagem. Quando o cérebro de um sagui adulto é seccionado coronalmente em intervalos de 50 μm, mais de 650 seções são normalmente necessárias para cobrir todo o cérebro. Os dados de cada seção consistem em uma série de imagens lado a lado capturadas em três canais. Esses arquivos de imagem são armazenados em uma única pasta junto com um arquivo de metadados que registra a posição de cada bloco. Para minimizar a quantidade total de dados e o tempo de processamento, a geração de imagens é realizada em blocos de execuções, durante os quais o número de etapas X e Y para ladrilhos muda gradualmente. Normalmente, 20 a 30 blocos de corridas, cada um compreendendo a imagem de 30 a 50 seções, constituem o conjunto de dados completo para um único cérebro. Para utilizar esses dados, são necessárias várias etapas de processamento de imagem, incluindo costura das imagens lado a lado, segmentação de sinais fluorescentes e registro dos dados volumétricos em um modelo padrão, entre outros.- Execute as etapas de processamento de imagem individualmente usando várias ferramentas de software ou por meio de pipelines dedicados adaptados às necessidades específicas dos pesquisadores. Detalhes do processamento das imagens estão além do escopo deste artigo e podem ser encontrados em outraspublicações11,19.
- Independentemente do procedimento usado, certifique-se de que os dados estejam organizados de forma consistente.
NOTA: Como todo o processo de imagem envolve muitos blocos de execuções e se estende por vários dias, não é incomum que uma execução seja cancelada no meio do caminho e reiniciada novamente. Nesses casos, a estrutura de dados pode ser interrompida, dificultando a execução adequada do pipeline de processamento de imagens. - Para minimizar o risco de interrupção da estrutura de dados, execute um script Python para verificar a integridade da estrutura de dados (kn_pipeline_check_mosaic.py) disponível no GitHub (github.com/watkarbey/STPT_depo).
- Registro da imagem corada na imagem STPT
NOTA: A transformação não linear da imagem manchada na imagem STPT pode ser realizada usando o plugin bUnwarpJ do ImageJ.- Ajuste o tamanho das imagens das imagens de destino (STPT) e de origem (manchadas) para que sejam aproximadamente iguais.
- Binarize cada imagem para visualizar as formas das seções.
- Execute o bUnwarpJ. Lembre-se de marcar a opção Salvar transformações .
- Inicie o bUnwarpJ novamente. Selecione a imagem original antes da binarização como a imagem de origem. Em seguida, clique em Load Elastic Transformation e escolha a transformação salva.
Resultados
Na configuração típica usada aqui, todo o cérebro de um sagui adulto pode ser fotografado (Figura 5) com resolução de ~ 1,3 x 1,3 μm / pixel com um intervalo de seção de 50 μm em cerca de 1 semana. Isso equivale a ~ 650 imagens coronais em três canais após a costura da imagem. Com uma lente objetiva de 16x (Nikon 16xW CFI75 LWD; NA = 0,80), o campo de visão de uma única foto é de cerca de 1 x 1 mm. A imagem de toda a superfície coronal é obtida costurando esses disparos (Figura 5A). O alinhamento na direção Z é excelente e uma boa imagem 3D é obtida simplesmente empilhando os dados da imagem coronal (Figura 5B, E). Para padronização, o modelo STPT para o cérebro do sagui11 pode ser usado para registro 3D-3D (Figura 5F). Essa transformação de dados é um dos principais aspectos da neuroanatomia cerebral inteira, na qual uma região de interesse recebe uma coordenada de espaço absoluto independente da anotação anatômica. Uma vez que o cérebro da amostra é registrado no espaço padrão, pode-se facilmente retirar as sub-regiões de interesse para análise posterior (Figura 5F, G). Em particular, as regiões corticais podem ser transformadas em um mapa plano usando um parâmetro predeterminado (Figura 5H).
Os cortes gerados durante a imagem podem ser usados para vários fins histológicos. Conforme mostrado na Figura 4A, B, a imagem retroiluminada sem coloração fornece um padrão muito semelhante à coloração autêntica de mielina. Esta pode ser uma excelente alternativa à coloração de mielina. Além disso, se a imagem retroiluminada for obtida antes da coloração de Nissl, a mesma seção pode ser usada para obter os padrões de coloração de mielina e Nissl, fornecendo informações úteis para identificar áreas e camadas (Figura 5C-E). Essas seções também podem ser usadas para coloração imunológica. Na Figura 5J, a seção ao redor do centro de injeção foi contracorada com anticorpo NeuN para estimar a eficiência de transdução do vírus AAV. Também é uma boa estratégia injetar traçadores não fluorescentes, além de traçadores fluorescentes, e detectá-los histologicamente após a recuperação da seção. Em nosso estudo anterior, combinamos traçadores verdes anterógrados com vetor "cre" retrógrado, que foi posteriormente detectado pelo anticorpo anti-cre10. Em um exemplo da Figura 6A-E, o BDA foi injetado no lado contralateral do traçador verde (trevo) e o detectou fluorescentemente. Observe que os sinais vermelhos de BDA podem ser registrados na imagem do TissueCyte para serem localizados em toda a coordenada cerebral. Em outro exemplo da Figura 6F-I, o smFP-myc foi injetado na área parietal (Figura 6G), enquanto o traçador verde foi injetado na área frontal. Dessa forma, vários traçadores podem ser injetados no mesmo animal sem interferir na imagem. Uma grande vantagem de usar as seções STPT para coloração adicional é que a relação entre os traçadores fluorescentes e não fluorescentes pode ser determinada para o mesmo cérebro. Dessa forma, foi possível determinar a reciprocidade das projeções corticocorticais com alta precisão10. Outra vantagem é que as coordenadas 3D das seções manchadas podem ser mapeadas de volta para os dados STPT e, em seguida, para o modelo padrão. Assim, pode não ser necessário usar todas as seções recuperadas para coloração. Para melhor interpretação, seções podem ser selecionadas para coloração para adicionar mais contexto aos dados STPT.
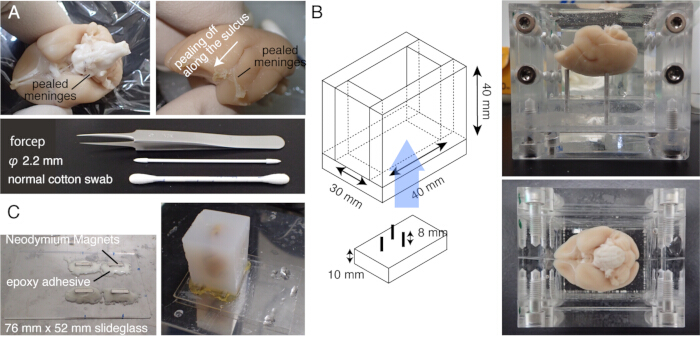
Figura 1: Preparação da amostra para STPT. (A) Remoção de meninge usando cotonetes. As meninges ao redor do tronco encefálico podem ser removidas por pinças de ponta fina. As meninges ao redor do cérebro do sagui são removidas manualmente esfregando com cotonetes. A foto mostra as meninges descascadas de dentro do sulco lateral. (B) A caixa de acrílico usada para incorporação de agarose. Os pinos são móveis e usados para ajustar o ângulo do cérebro para ficar próximo à posição fixa estereotaxicamente20. (C) Uma lâmina magnética feita de vidro deslizante de 76 mm x 52 mm e quatro ímãs de neodímio presos por adesivo epóxi. O bloco de agarose é fixado ao estágio magnético com super cola. Clique aqui para ver uma versão maior desta figura.
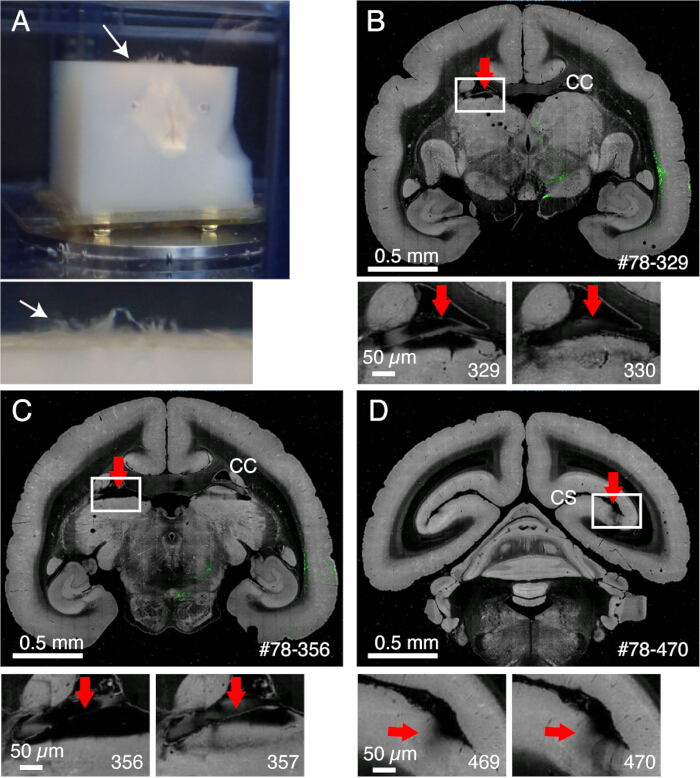
Figura 2: O efeito das meninges na imagem. (A) As meninges que permanecem não cortadas flutuam, como mostrado pela seta branca. Neste exemplo, a extensa protrusão das meninges é vista porque elas não foram removidas antes da incorporação de agarose. Normalmente, as meninges permanecem intactas apenas em várias regiões difíceis. (B) Um exemplo de corte ruim devido à presença de meninges entre o corpo caloso e a parte superior do tálamo. Nesse caso, o corte ruim levou à alternância de um corte profundo (329) e um corte relativamente normal (330). # 78-329 significa amostra nº 78 seção nº 329 no portal de dados Brain / MINDS (C) Outro exemplo de imagem ruim. Na pior das hipóteses, o núcleo pulvinar mostrado pela seta vermelha pode se soltar completamente. Barras de escala: 0,5 mm (painel superior), 0,5 μm (painel inferior). (D) Outro exemplo de imagem ruim. As meninges profundas no sulco calcarino são difíceis de remover. As sombras vistas nas seções 469 e 470 são causadas pelas meninges flutuantes que ficaram sob o objetivo. Barras de escala: 0,5 mm (painel superior), 0,5 μm (painel inferior). Clique aqui para ver uma versão maior desta figura.
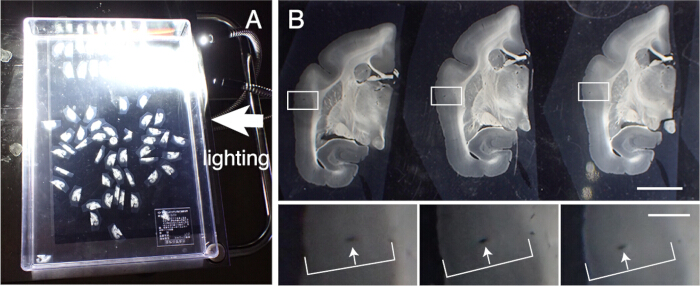
Figura 3: Alinhamento das seções de tecido em ordem. (A) Até 50 seções coronais de saguis podem ser alinhadas corretamente em ordem em um recipiente de plástico (31 cm x 22,5 cm). Para visualizar as estruturas detalhadas, o recipiente é colocado em papel preto e iluminado lateralmente. Essas seções são primeiro alinhadas aproximadamente em ordem e, em seguida, sujeitas a um alinhamento preciso. (B) O alinhamento preciso usa o vaso sanguíneo como marcador. O córtex cerebral contém muitos vasos sanguíneos que correm verticalmente através das camadas corticais. Eles são identificados como orifícios alongados que mudam sistematicamente suas posições dentro das camadas corticais (setas brancas). Usando este método, mesmo seções com intervalos de 50 μm podem ser alinhadas com precisão. Esses orifícios dos vasos sanguíneos podem ser identificados nas imagens da seção SPTP para confirmação. Barra de escala: 5 mm (painel superior), 1 mm (painel inferior). Clique aqui para ver uma versão maior desta figura.
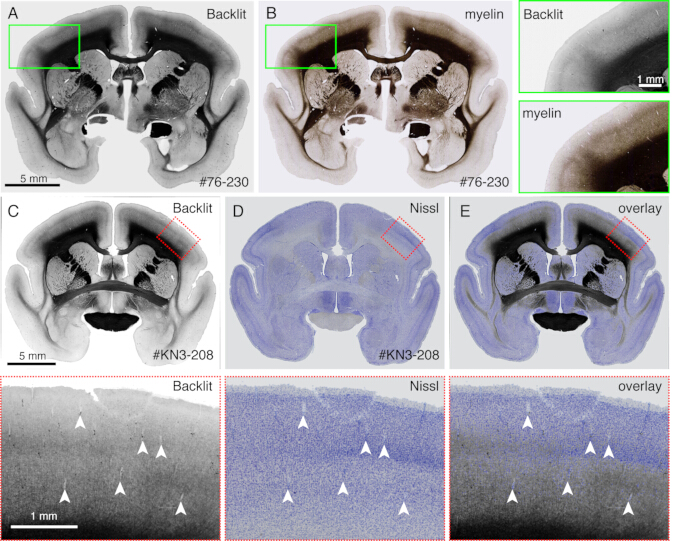
Figura 4: Imagem retroiluminada como substituto da coloração de mielina. (A,B) A seção idêntica foi usada para imagens retroiluminadas e coloração de mielina. Primeiro, a seção foi montada no vidro da lâmina, seca, reidratada com PBS, lamínula e fotografada usando um microscópio de luz (Tabela de Materiais). Após a remoção da lamínula, a mesma seção foi usada para coloração de mielina21 e fotografada usando um microscópio de fluorescência. A imagem retroiluminada foi registrada na imagem de mielina usando o plug-in bUnwarpJ do ImageJ. As caixas verdes mostram as visualizações ampliadas de cada imagem. Observe que essas imagens mostram padrões quase idênticos, exceto que a coloração de mielina visualiza melhor as estruturas fibrosas. (CE) A seção idêntica foi usada para imagens retroiluminadas e coloração de Nissl. A máscara de baixo limiar para a imagem retroiluminada foi registrada pela primeira vez com a máscara de baixo limiar para a imagem Nissl usando o plug-in bUnwarpJ, e a imagem original foi transformada usando o mesmo parâmetro. Observe que os vasos sanguíneos (pontas de setas brancas) correspondem bem entre as duas imagens. Como essas duas imagens são para a mesma seção, a correspondência é quase perfeita e pode-se comparar diretamente os padrões de mielina e Nissl para a identificação de camadas corticais. Barras de escala: 5 mm (painéis A-E). Clique aqui para ver uma versão maior desta figura.
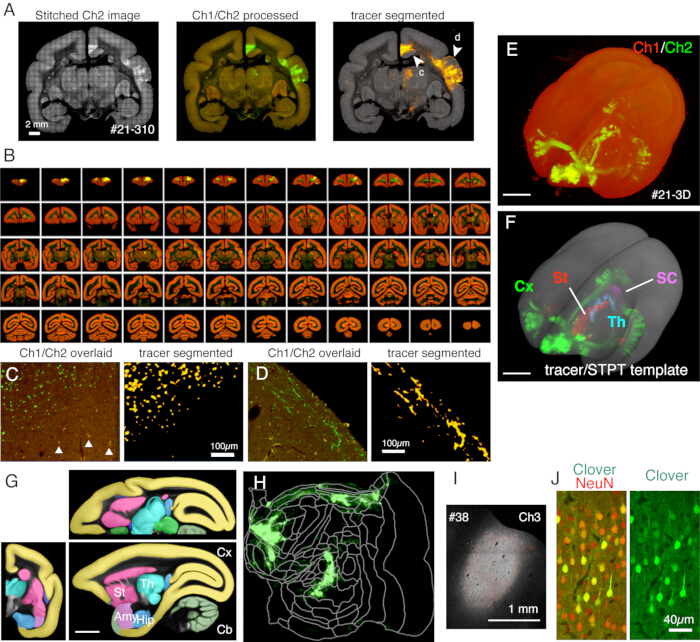
Figura 5: Resultado típico da imagem STPT. (A) Um simples ladrilho de imagens Ch2 (verde) de uma seção de exemplo (seção 310 da amostra # 21). Sem correção de plano de fundo, as bordas de cada bloco são visíveis. Na coluna do meio, os ladrilhos foram costurados com correção de fundo para Ch1 (vermelho) e Ch2 (verde), respectivamente. Para reduzir os sinais de lipofuscina (ver painel C), os sinais de Ch1 foram subtraídos de Ch2 e mostrados em verde. Para reduzir os sinais de lipofuscina de Ch1, o comando "Remover Utilitários.." foi usado antes da costura pelo ImageJ. Na coluna da direita, os sinais do traçador foram segmentados por pipeline de processamento de imagem11. As pontas das setas (c, d) mostram os locais onde as vistas ampliadas são mostradas nos painéis C e D. Barra de escala: 2 mm. (B) Visão geral das seções seriais para a amostra # 21. O STPT gerou 635 imagens coronais de alta resolução para esta amostra. (C) Uma vista de alta ampliação mostrada pela ponta da seta c no painel A. Esta é uma sobreposição simples de Ch1 (vermelho) e Ch2 (verde) sem processamento adicional. Os triângulos mostram sinais de fluorescência de lipofuscina, que mostram um espectro generalizado. O algoritmo de segmentação do traçador distingue com precisão os sinais do traçador do fundo da lipofuscina, apesar de uma forma muito semelhante (painel direito). Barra de escala: 100 μm. (D) Outro exemplo de uma vista de alta ampliação. Observe que as fibras finas do axônio na camada 1 podem ser bem visíveis. Barra de escala: 100 μm. (E) A reconstrução 3D das imagens STPT originais. As 635 imagens coronais de baixa resolução, conforme mostrado no painel B, foram usadas como tiff-stack para visualização 3D usando fluorender22. Barra de escala: 5 mm. (F) A reconstrução 3D dos sinais traçadores segmentados registrados no modelo STPT (cinza). Os sinais traçadores em diferentes regiões do cérebro foram mostrados por cores diferentes. Essas regiões foram cortadas usando a anotação mostrada no painel G. Barra de escala: 5 mm. (G) Modelo STPT sobreposto com anotação de diferentes regiões cerebrais. Barra de escala: 5 mm. (H) O sinal do traçador cortical mostrado no painel F foi mostrado na forma de um mapa plano. (I) Identificação do local de injeção pela fluorescência de Ch3, que é menos sensível à fluorescência do traçador e permanece insaturada. Barra de escala: 1 mm. (J) A coloração da secção em torno do centro de injecção com anticorpo NeuN mostrou que aproximadamente 30% dos neurónios apresentam uma forte expressão de fluorescência verde de trevo. Barra de escala: 40 μm. Abreviaturas: Cx; córtex, St; estriado, Th; tálamo, SC; colículo superior. Amy; amígdala, quadril; hipocampo, Cb; cerebelo. Clique aqui para ver uma versão maior desta figura.
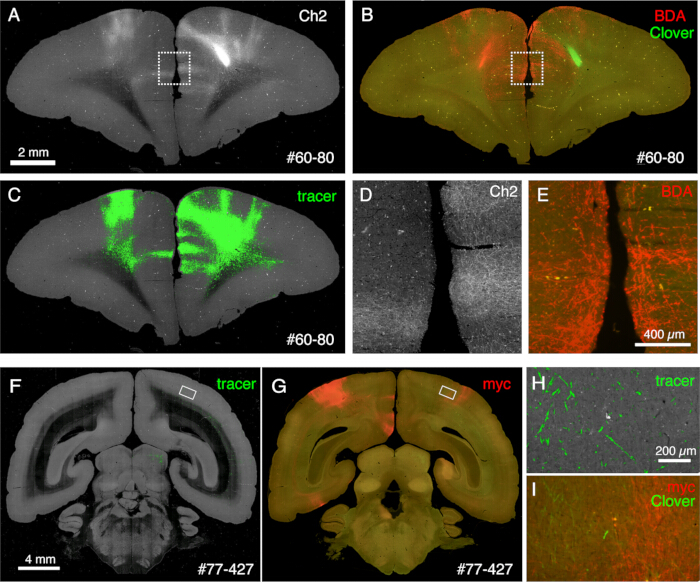
Figura 6: Histologia pós-STPT mostrando múltiplos traçadores não fluorescentes. (AC) Comparação da imagem STPT com a imagem corada com BDA. Nesta amostra, o BDA é injetado no lado contralateral da injeção de trevo. Os painéis A e C mostram a imagem Ch2 e a segmentação do traçador dos dados STPT. O painel B mostra a coloração pós-STPT para BDA. A fluorescência do trevo é diminuída devido ao tratamento com metanol da seção. Barra de escala: 2 mm. (D,E) As caixas pontilhadas nos painéis A e B são ampliadas. Barra de escala: 100 μm. (F,G) Comparação da imagem STPT com a coloração de anticorpos anti-myc tag. Nesta amostra, a injeção de trevo é no PFC, enquanto o AAV-smFP-myc é injetado no córtex parietal contralateral. Os retângulos brancos são ampliados nos painéis H e I. Barra de escala: 4 mm. (H) A segmentação do traçador é mostrada em verde. Barra de escala: 200 μm. (I) A coloração myc é mostrada em vermelho. A fluorescência do trevo é mostrada em verde. Barra de escala: 200 μm. Os sinais verdes nos painéis H e I estão presentes em uma posição semelhante, mas não idênticos, porque o STPT recupera apenas a seção óptica de ~ 10 μm. Clique aqui para ver uma versão maior desta figura.
| Protocolo de coloração fluorescente BDA | |
| Remover agarose | |
| Lavagem TBS | 10 min (2x) |
| 1% H2O2 em solução de Dent | 10 minutos |
| Lavagem TBS | Breve |
| 0,5% de bloqueio de TNB | 1 h |
| StAvHRP (1:4000) em TNB | 2 durante a noite |
| Lavagem TNT | 10 min (3x) |
| TSA Biotina (1:4000) em borato de 0,1 M (pH8,5) + 0,003% H2O2 | 2 h |
| Lavagem TNT | 10 min (3x) |
| Cy3-estreptavidina (1:1000) em TNT | 3 h |
| Lavagem TNT | 10 min (2x) |
| Lavagem TBS | Mantenha a seção até a montagem |
| Monte a seção no vidro deslizante usando um meio de montagem antidesbotamento | |
| Protocolo de coloração fluorescente anti-MYC | |
| Remover agarose | |
| Lavagem TBS | 10 min (2x) |
| bloqueio em IB | 1 h |
| Anti-Myc (1:4000) em IB | 2 durante a noite |
| Lavagem TNT | 10 min (3x) |
| Anti-mouse Cy3 (1:1000) em TNT | 3 h |
| Lavagem TNT | 10 min (2x) |
| Lavagem TBS | Mantenha a seção até a montagem |
| Monte a seção no vidro deslizante usando um meio de montagem antidesbotamento | |
| Buffers/soluções | Composição |
| 0,5% TNB | Reagente de bloqueio de 0,5% TSA em TS7.5 |
| Solução de Dent | 20% DMSO, 80% Metanol |
| Tampão de imersão (IB) | 10% FBS, 2% BSA 0,5% TritonX100 em TBS |
| TBS (solução salina tamponada com Tris) | 25 mM Tris, 137 mM NaCl, 2,7 mM KCl (pH 7,4) |
| TNT | 0,05% Tween20 em TS7.5 |
| TS7.5 | 0,1 M TRIS-HCl, pH 7,5, 0,15 M NaCl |
Tabela 1: Protocolo de coloração fluorescente BDA e coloração fluorescente anti-myc
| Plasmídeo para traçador AAV | Addgene No. | Anticorpo recomendado | Diluição sugerida | Resultado esperado |
| pAAV-EF1_Cre | 201198 | Clone Millipore 2D8 | 1:1000 | bom para as células |
| pAAVCam1.3_smFP_Myc | 201205 | MBL M192 My3 (mouse) | 1:4000 | Excelente |
| pAAVCam1.3_smFP_HA | 201206 | CST C29F4 (coelho) | 1:1000 | bom para as células |
| pAAVCam1.3_smFP_FLAG | 201207 | MBL PM020B (coelho) | 1:1000 | OKEY |
| AAVTRE3_smFP_Myc | 201208 | |||
| AAVTRE3_smFP_HA | 201209 | |||
| AAVTRE3_smFP_FLAG | 201210 |
Tabela 2: Lista de plasmídeos Addgene disponíveis para a produção de traçadores não fluorescentes. A construção cre tem como alvo o núcleo e é adequada para rastreamento retrógrado envolto por AAV2 retro. smFP_HA construção é boa para detecção de corpo celular (e retrógrado).
Vídeo Complementar: Vista microscópica da remoção de meninges. Clique aqui para baixar este vídeo.
Discussão
Este artigo explicou as soluções práticas para lidar com cérebros de saguis para processamento de todo o cérebro, bem como técnicas histológicas auxiliares que aumentam a utilidade da técnica STPT. A força da "neuroanatomia do cérebro inteiro" usando STPT é que você pode obter as coordenadas 3D de qualquer região de interesse, seja ela anatomicamente anotada ou não. Por meio do registro 3D para 3D de alta precisão, é possível transformar essas coordenadas em um modelo padrão para a sobreposição de vários conjuntos de dados. Dessa forma, o modelo padrão serve como meio de integração de dados. Este foi um aspecto essencial do nosso projeto de mapeamento do córtex pré-frontal (PFC)10, onde foram analisados dados obtidos de muitos indivíduos. Além disso, as informações mapeadas para o modelo padrão podem ser comparadas com vários dados que já foram mapeados, sejam Nissl, padrões de mielina, dados de traçadores, dados de ressonância magnética (incluindo ressonância magnética de difusão) ou anotações anatômicas11. É importante ressaltar que também pode ser comparado com dados futuros que serão obtidos por tecnologia ainda a surgir. Atualmente, existem vários modelos para o cérebro do sagui, que são baseados na coloração de Nissl23, ressonância magnética 23,24,25,26,27,28 e STPT11. Mas eles podem ser transformados nas coordenadas uns dos outros com base em contrastes de imagem e parâmetros pré-calculados11. A integração de dados em toda a escala cerebral entre os estudos contribui para uma melhor compreensão do cérebro como um sistema. O pré-requisito para que essa estratégia funcione é a aquisição confiável de dados em todo o cérebro. Abaixo, são discutidas as etapas críticas e os possíveis problemas associados ao protocolo atual.
Um dos processos mais vulneráveis da imagem de STPT é o corte de tecido. Como mencionado acima, as meninges geralmente permanecem sem cortes e podem interferir no corte. Em particular, o núcleo pulvinar e o colículo superior são as duas regiões cerebrais mais afetadas: dependendo da meticulosidade da remoção da meninge e da incorporação na agarose, elas podem ser removidas do bloco de tecido durante o corte. Essas regiões profundas são difíceis de abordar de fora e podem quebrar facilmente durante a remoção da meninge. Uma remoção cuidadosa, mas completa, da meninge é fundamental para um exame de imagem bem-sucedido. Outra preocupação é a inversão das seções fatiadas no bloco de tecido, o que às vezes ocorre quando as seções permanecem presas mesmo após o corte. Pode ser minimizado moldando o bloco de modo que a lâmina corte obliquamente no final.
Ao obter imagens profundas do bloco de tecido, o STPT evita a irregularidade de sua superfície. Enquanto os sinais fluorescentes podem passar pela região cortical com bastante facilidade, eles são altamente diminuídos nas regiões mielinizadas. Portanto, a profundidade da imagem precisa ser cuidadosamente determinada para equilibrar entre a imagem consistente em toda a superfície do bloco e o brilho dos sinais na região rica em mielina, como a substância branca. Na configuração usada aqui, geralmente visamos 25-35 μm da superfície. Também é preciso alertar que a superfície de corte pode ficar encolhida de forma desigual após longas horas de armazenamento. Para minimizar a área de imagem, dividimos a sessão de imagem em 20 a 30 execuções com diferentes configurações de estágio por 5 a 6 dias. Fazemos o intervalo entre as corridas inferior a 2 h ou confirmamos a profundidade da superfície e ajustamos a altura do palco antes de cada corrida.
Nesse protocolo, o sinal BDA foi amplificado pelo método TSA. Este método é altamente eficaz e pode detectar sinais BDA transportados anterógradamente, mesmo em resolução relativamente baixa (por exemplo, Figura 6B). A biotina TSA está disponível comercialmente na Akoya Biosciences, mas a solução caseira mostra um aprimoramento muito melhor. Por outro lado, a diluição do anticorpo e da solução requer um ajuste cuidadoso para obter o resultado ideal. O pré-tratamento da seção com uma solução de metanol é crítico. Sem pré-tratamento, os sinais de BDA mal são detectados nos axônios mielinizados.
Quando um traçador anterógrado é usado, muitas vezes é difícil identificar o local exato da injeção devido à saturação dos sinais de fluorescência. Na configuração do sistema de imagem de todo o tecido usado neste estudo, usamos o canal azul para identificar as células infectadas (Figura 5I). Mesmo quando os canais vermelho e verde estão saturados, cada neurônio infectado individual geralmente é detectável no canal azul. Isso também é verdade com o Allen Mouse Brain Connectivity Atlas1. O exame das células de origem é importante porque a infecção às vezes envolve apenas camadas específicas. Encontramos essas infecções parciais com bastante frequência para o Atlas de Conectividade do Cérebro de Camundongos, talvez por causa da utilização do método de iontoforese29. A disseminação lateral dos marcadores virais pode ser mais ou menos variável dependendo das injeções. Essa variabilidade pode afetar potencialmente o resultado do rastreamento e precisa de uma normalização cuidadosa.
O registro bem-sucedido da imagem 3D obtida no modelo padrão é um processo chave de toda a neuroanatomia cerebral. O registro da imagem STPT no modelo STPT pode ser bastante preciso, e observamos apenas desviâncias de alguns voxels (50 μm isocúbicos) para as bordas com alto contrastede imagem 10. Ainda assim, há um limite para o que o registro pode fazer. Devido ao processo de remoção da meninge, as amostras de STPT geralmente têm lacunas entre os hemisférios e entre o córtex e o mesencéfalo / rombencéfalo, enquanto os tecidos cerebrais são compactados nas imagens de ressonância magnética in vivo . Essas diferenças são difíceis de ajustar por registro. O córtex do sagui é principalmente desprovido de sulcos, mas o sulco intraparietal é muito profundo em alguns indivíduos. Esses sulcos serão perdidos (ocorre a fusão de topo a topo) no momento do registro. Embora o registro seja uma técnica poderosa, é necessário voltar aos dados brutos para confirmação do resultado obtido.
A geração de dados de imagem massivos é um ponto forte e uma limitação dessa técnica. Embora melhore a integridade do conjunto de dados, requer um gerenciamento cuidadoso dos dados adquiridos e o desenvolvimento de um pipeline de processamento de imagem automatizado para uma interpretação eficiente dos dados. No futuro, a aplicação de inteligência artificial generativa (IA) na construção de pipelines de processamento de imagens pode simplificar significativamente esse processo. Imagens sistemáticas de todo o cérebro também foram realizadas usando métodos baseados em scanner de lâminas21,30. Comparado a esses métodos, o STPT não requer computação adicional para reconstrução 3D. Combinado com o seccionamento óptico, demonstramos que o STPT tem o potencial de reconstruir até mesmo segmentos de axônio nas seções31. Com a combinação adicional de técnicas de depuração de tecidos, Economo et al. desenvolveram um método para obter imagens de todos os neurônios escassamente marcados32,33. As versões mais recentes do TissueCyte agora oferecem opções para um laser adicional para aumentar a excitação de proteínas fluorescentes vermelhas ou uma unidade de captura de seção para a recuperação automática de seções. Com esses avanços, a abordagem de todo o cérebro se tornará mais eficiente, fornecendo uma base para uma compreensão abrangente do cérebro dos primatas, incluindo o dos humanos.
Divulgações
Os autores não têm nada a divulgar.
Agradecimentos
Agradecemos profundamente à equipe técnica do laboratório e biotérios de Yamamori (RRD) por sua ajuda. Agradecemos ao Centro de Colaboração RIKEN CBS-Olympus pela assistência técnica na aquisição de imagens confocais. Este trabalho foi apoiado pelo programa de Pesquisa Científica em Áreas Inovadoras (número de concessão 22123009) do MEXT, Japão, por Brain/MINDS e Brain/MINDS2.0 da AMED, Japão (JP15dm0207001, JP23wm0625001 e JP24wm0625218), e pela JSPS KAKENHI Grant Number 24K09678 para A.W.
Materiais
| Name | Company | Catalog Number | Comments |
| Agarose type I | Sigma | A6013 | |
| All-in-one fluorescence microscope | Keyence | BZ-X series | This study used BZ-X710 |
| anti-mouse Cy3 | Jackson Immuno Research Laboratories, Inc. | 115-165-003 | |
| anti-myc antibody | MBL | M192 | mouse monoclonal |
| Butorphanol | Meiji Animal Health | Vetorphale | |
| Ceramic blade | TissueVision | N/A | |
| Collagenase type I | Wako Chemical | #031-17601 | Aliquot into 100 µL x 10 of 100 mg/mL in PBS |
| Cotton swab (HUBY-340) | HUBY | BB-013SP | micro cotton swabs |
| Custom-made chamber | N/A | N/A | The chamber consist of four side plates and one bottom plates, all made of 1 cm thick acrylic plates, which can be securely fastened together using screw bolts. An additional acrylic plate is placed inside the chamber, with three metal poles inserted through it to hold the marmoset brain in an inverted position. |
| Cy3-Streptavidin | Jackson Immuno Research Laboratories, Inc. | 016-160-084 | |
| Dumont #5 forceps, Inox | N/A | N/A | |
| Epoxy instant mix | Locktite | N/A | |
| FIJI | NIH | https://imagej.net/software/fiji/downloads | |
| kn_pipeline_check_mosaic.py | N/A | N/A | github.com/watkarbey/STPT_depo; This is a checking script for TissueCyte data. |
| Medetomidine | Zenoaq | Domitor | |
| Midazolam | Sandoz | N/A | |
| NaBH4 | Sigma | #452882 | |
| NaIO4 | Sigma | S1878 | |
| Perfusion needle | Natsume Seisakusho Co., Ltd | KN-348, 20G-50 | |
| Slideglass (76 mm x 52 mm) | Matsunami | S9111 | |
| StAvHRP | Jackson Immuno Research Laboratories, Inc. | 016-030-084 | HRP-streptavidin (titration needs to be carefully determined) |
| TissueCyte1000 | TissueVision | https://www.tissuevision.com/tissuecyte | |
| TSA blocking reagent | Kiko Tech | FP1012 | Refer to TSA-biotin kit (Akoya Biosciences) |
| TSA-biotin | House-made | N/A | See Okamoto et al (ref. 16) |
| VECTASHIELD HardSet Mounting Medium | Vector laboratories Inc | N/A | Antifade mounting medium |
Referências
- Oh, S. W., Harris, J. A., Ng, L., Winslow, B., Cain, N., et al. A mesoscale connectome of the mouse brain. Nature. 508 (7495), 207-214 (2014).
- Ragan, T., et al. Serial two-photon tomography for automated ex vivo mouse brain imaging. Nat Methods. 9 (3), 255-258 (2012).
- Liang, Z., Arefin, T. M., Lee, C. H., Zhang, J. Using mesoscopic tract-tracing data to guide the estimation of fiber orientation distributions in the mouse brain from diffusion MRI. Neuroimage. 270, 119999 (2023).
- Watakabe, A., Hirokawa, J. Cortical networks of the mouse brain elaborate within the gray matter. Brain Struct Funct. 223 (8), 3633-3652 (2018).
- Ypma, R. J. F., Bullmore, E. T. Statistical analysis of tract-tracing experiments demonstrates a dense, complex cortical network in the mouse. PLoS Comput Biol. 12 (9), e1005104 (2016).
- Wang, Q., et al. The Allen mouse brain common coordinate framework: A 3D reference atlas. Cell. 181 (4), 936-953.e20 (2020).
- Köbbert, C., Apps, R., Bechmann, I., Lanciego, J. L., Mey, J., Thanos, S. Current concepts in neuroanatomical tracing. Prog Neurobiol. 62 (4), 327-351 (2000).
- Lanciego, J. L., Wouterlood, F. G. Neuroanatomical tract-tracing techniques that did go viral. Brain Struct Funct. 225 (4), 1193-1224 (2020).
- Bakker, R., Wachtler, T., Diesmann, M. CoCoMac 2.0 and the future of tract-tracing databases. Front Neuroinform. 6, (2012).
- Watakabe, A., et al. Local and long-distance organization of prefrontal cortex circuits in the marmoset brain. Neuron. 111 (14), 2258-2273.e10 (2023).
- Skibbe, H., et al. The Brain/MINDS marmoset connectivity resource: An open-access platform for cellular-level tracing and tractography in the primate brain. PLoS Biol. 21 (6), e3002158 (2023).
- Mitchell, J. F., Leopold, D. A. The marmoset monkey as a model for visual neuroscience. Neurosci Res. 93, 20-46 (2015).
- Okano, H. Current status of and perspectives on the application of marmosets in neurobiology. Annu Rev Neurosci. 44, 27-48 (2021).
- Watakabe, A., et al. Comparative analyses of adeno-associated viral vector serotypes 1, 2, 5, 8 and 9 in marmoset, mouse and macaque cerebral cortex. Neurosci Res. 93, 144-157 (2015).
- Hata, J., et al. Multi-modal brain magnetic resonance imaging database covering marmosets with a wide age range. Sci Data. 10, 221 (2023).
- Liwang, J. K., Bennett, H. C., Pi, H. -. J., Kim, Y. Protocol for using serial two-photon tomography to map cell types and cerebrovasculature at single-cell resolution in the whole adult mouse brain. STAR Protoc. 4 (1), 102048 (2023).
- Okamoto, S., et al. Exclusive labeling of direct and indirect pathway neurons in the mouse neostriatum by an adeno-associated virus vector with Cre/lox system. STAR Protoc. 2 (1), 100230 (2021).
- Viswanathan, S., et al. High-performance probes for light and electron microscopy. Nat Methods. 12 (6), 568-576 (2015).
- Kuan, L., et al. Neuroinformatics of the Allen mouse brain connectivity atlas. Methods. 73, 4-17 (2015).
- Paxinos, G., Watson, C., Petrides, M., Rosa, M., Tokuno, H. . The Marmoset Brain in Stereotaxic Coordinates. , (2012).
- Abe, H., et al. 3D reconstruction of brain section images for creating axonal projection maps in marmosets. J Neurosci Methods. 286, 102-113 (2017).
- Wan, Y., et al. FluoRender: Joint freehand segmentation and visualization for many-channel fluorescence data analysis. BMC Bioinformatics. 18 (1), 280 (2017).
- Majka, P., et al. Towards a comprehensive atlas of cortical connections in a primate brain: Mapping tracer injection studies of the common marmoset into a reference digital template. J Comp Neurol. 524 (11), 2161-2181 (2016).
- Liu, C., Yen, C. C. -. C., Szczupak, D., Tian, X., Glen, D., Silva, A. C. Marmoset brain mapping V3: Population multi-modal standard volumetric and surface-based templates. Neuroimage. 226, 117620 (2021).
- Liu, C., et al. A digital 3D atlas of the marmoset brain based on multi-modal MRI. Neuroimage. 169, 106-116 (2018).
- Liu, C., et al. A resource for the detailed 3D mapping of white matter pathways in the marmoset brain. Nat Neurosci. 23 (2), 271-280 (2020).
- Woodward, A., et al. The Brain/MINDS 3D digital marmoset brain atlas. Sci Data. 5, 180009 (2018).
- Saleem, K. S., Avram, A. V., Glen, D., Schram, V., Basser, P. J. The subcortical atlas of the marmoset ("SAM") monkey based on high-resolution MRI and histology. Cereb Cortex. 34 (4), bhae120 (2024).
- Watakabe, A., Hirokawa, J. Cortical networks of the mouse brain elaborate within the gray matter. Brain Struct Funct. 223 (8), 3633-3652 (2018).
- Lin, M. K., et al. A high-throughput neurohistological pipeline for brain-wide mesoscale connectivity mapping of the common marmoset. eLife. 8, e40042 (2019).
- Skibbe, H., et al. PAT-probabilistic axon tracking for densely labeled neurons in large 3-D micrographs. IEEE Trans Med Imaging. 38 (1), 69-78 (2019).
- Economo, M. N., et al. A platform for brain-wide imaging and reconstruction of individual neurons. eLife. 5, e10566 (2016).
- Winnubst, J., et al. Reconstruction of 1,000 projection neurons reveals new cell types and organization of long-range connectivity in the mouse brain. Cell. 179 (1), 268-281.e13 (2019).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados