Method Article
신경 해부학적 분석을 위한 전체 마모셋 뇌의 연속 이광자 단층촬영(Serial Two-Photon Tomography of the whole Marmoset Brain)
요약
연속 이광자 단층촬영(STPT) 이미징은 이광자 이미징과 자동 스테이지 제어 및 마이크로톰 슬라이싱을 결합하여 조직 덩어리를 3차원 형태로 이미징하는 기술입니다. 여기에서는 마모셋 뇌가 구조적 특징을 더 잘 이해할 수 있도록 이를 구현하기 위한 프로토콜에 대해 설명합니다.
초록
연속 이광자 단층촬영(STPT)은 이광자 이미징과 자동 스테이지 제어 및 마이크로톰 슬라이싱을 결합하여 조직 덩어리를 3차원 형태로 이미지화하는 기술입니다. 우리는 마모셋 뇌의 축삭돌기를 추적하기 위해 이 기술을 성공적으로 구현했습니다. 여기에서는 마모셋 뇌 전체에 대한 신뢰할 수 있는 체적 영상화를 가능하게 한 상세한 실험 절차에 대해 설명합니다. 성공적인 이미징을 위한 핵심 프로세스는 슬라이싱을 방해하는 뇌 주변의 수막을 제거하는 것이었습니다. 이 방법론의 큰 장점은 얇게 썬 부분을 추가 염색에 사용할 수 있다는 것입니다. 원래 설정에서는 얇게 썬 부분이 수조에서 스크램블됩니다. 이 섹션은 피질의 혈관 패턴에 따라 원래 순서로 올바르게 정렬될 수 있습니다. 효과적인 조직학의 예로는 간단한 빛 반사에 의한 미엘린 구조의 시각화가 있으며, 이는 Nissl 염색과 결합하여 해부학적 경계를 정의할 수 있습니다. 이 절편은 비형광 전행성 및 역행성 추적자의 면역학적 검출에도 사용할 수 있으며, 여러 데이터의 계층화를 위해 STPT 데이터에 등록할 수 있습니다.
서문
신경 해부학 연구에서 연구자들은 전체 뇌의 맥락에서 마이크로미터 순서의 구조(예: 축삭 및 부톤)를 관찰해야 할 필요성에 직면해 있습니다. 이 어려운 작업은 일반적으로 연속 섹션을 육안으로 검사하고 자세한 이미징, 분석 및 사진 기록을 위한 관심 영역을 찾는 방식으로 접근했습니다. 그러나 기술 발전으로 전체 뇌 분석을 위해 전체 뇌를 고해상도로 이미지화하는 것이 가능해지고 있습니다. Oh 등1의 획기적인 연구에서 수백 개의 쥐 뇌가 커넥톰 분석을 위해 추적자 주사를 받았으며, 이는 연속 이광자 단층 촬영(STPT) 이미징 기술2로 처리되었습니다. 이 연구의 특징은 "연결성"을 정량화하기 위해 선행성 추적자를 선택했다는 것입니다. 선행성 추적자는 축삭 분포에 대한 매우 상세한 공간 정보를 제공하지만 신경 해부학자들은 분석을 위해 힘든 수동 세분화에 의존해 왔습니다. 이 세분화 단계를 포함한 다양한 분석 절차를 자동화함으로써 다용도 사용을 위해 즉시 사용할 수 있는 고해상도 추적 데이터의 "대량 생산"에 성공했습니다. 그들의 추적자 데이터(tracer data) 3,4,5 와 표준 뇌(standard brain)6를 사용한 다양한 연구를 감안할 때, 그들의 접근법의 효과는 명백하다.
역사적으로 인간이 아닌 영장류 뇌의 신경 연결성은 1950년대 퇴행법이 개발된 이래 1970년대의 전행성/역행성 물질 수송 방법, 현재의 바이러스 전략 7,8에 이르기까지 많은 신경해부학자들의 관심을 끌었습니다. 따라서 영장류의 신경 연결을 조사하는 방대한 문헌이 존재합니다. 특히, 많은 연구자들이 원숭이 뇌의 복잡한 피질피질 연결성을 조사했으며, 그 결과는 표로 작성하기 위해 선별되었습니다(예: CoCoMac9). 이러한 고전적 연구는 유용하기는 하지만 몇 가지 한계가 있었다. 첫째, 각 연구는 제한된 뇌 영역에만 초점을 맞추기 때문에 얻은 정보는 필연적으로 단편적이 됩니다. 둘째, 각 연구는 서로 다른 방법과 조건을 사용한다. 따라서 연구 전반에 걸친 정량적 평가는 복잡해집니다. 셋째, 연결성은 일반적으로 대표 섹션의 카메라 명료화로 표시되거나 저자가 정의한 뇌 영역에 대한 반정량적 표/그래프로 표시됩니다. 다른 말로 하면, 복잡한 뇌 구조에서 매우 제한된 정보만이 출판된 문헌에 발표되기 위해 추출됩니다. 자기공명영상(MRI) 기술의 발달로 저해상도의 전뇌 연구가 두각을 나타내게 되었습니다. 그러나 생쥐의 신경 연결에 대해 우리가 알고 있는 것과 영장류의 신경 연결에 대해 알고 있는 것 사이에는 큰 차이가 있습니다.
이러한 배경 지식을 염두에 두고, 우리는 일반적인 마모셋 뇌10,11에 대한 포괄적인 선행성 추적을 수행하기 시작했습니다. 원숭이의 뇌보다 훨씬 작고 매끄럽지만, 마모셋 뇌는 MT 영역과 과립형 전전두엽 영역의 존재와 같은 영장류의 명확한 징후를 보이며, 둘 다 설치류에서 명확하게 정의되지 않았습니다12,13. 여기서는, 마모셋의 뇌조차도 쥐의 뇌보다 10배 더 무겁기 때문에 작은 크기가 큰 장점이었다. 다행히도 STPT 이미징을 위한 상용 현미경인 TissueCyte1000(이하 전체 조직 이미징 시스템이라고 함) 운영 소프트웨어의 원래 버전을 최소한으로 업데이트하여 마모셋 뇌 전체를 이미지화할 수 있었습니다2. 이 업데이트는 슬라이싱하기 전에 무대를 추가로 이동할 수 있도록 하기 위한 것이었습니다. 이제 현재 버전은 마모셋 브레인을 처리하기에 충분합니다. 이 기사에서는 STPT 이미징을 위한 마모셋 브레인을 처리하는 프로토콜을 공유합니다. 이 방법의 유용성을 더욱 향상시키는 사후 이미징 프로토콜도 제공됩니다.
프로토콜
모든 실험 절차는 1996년 개정된 미국 국립보건원(National Institute of Health for the Care and Use of Laboratory Animals, NIH 간행물 No. 80-23)과 일본 생리학회(Japanese Physiological Society)의 "생리학 분야 동물 관리 및 사용을 위한 지침 원칙(Guiding Principles for the Care and Use of Animals in the Field of Physiological Science)"에 따라 수행되었으며, RIKEN 실험동물위원회(W2020-2-009(2))의 승인을 받았습니다.
1. 트레이서 주입
- 이전에 보고된 절차에 따라 마모셋 뇌에 형광 및 비형광 추적자를 주입합니다14.
- STPT 방법과의 조합과 관련하여 추적자의 형광이 향상 없이 검출될 수 있을 만큼 충분히 강한지 확인하십시오. 향상된 표현을 위해 이중 벡터 TET-Off 시스템10 을 채택합니다.
참고: 생산된 slice를 사용하여 STPT가 완료된 후 비형광 추적자를 주입하고 검출할 수도 있습니다. 비형광 추적자의 예로는 Biotinylated Dextran Amine(BDA), 입력 세포핵10의 역행 검출을 위한 AAV2 retro의 cre-expressing AAV, GFP 기반 smFP-tag AAV(아래 참조)가 있습니다.
- STPT 방법과의 조합과 관련하여 추적자의 형광이 향상 없이 검출될 수 있을 만큼 충분히 강한지 확인하십시오. 향상된 표현을 위해 이중 벡터 TET-Off 시스템10 을 채택합니다.
- 관류는 4주 후에 마모셋 뇌를 수정하고 얻습니다.
- 마모셋을 마취하기 위해 MMB라고 하는 메데토미딘(0.04mg/kg), 미다졸람(0.4mg/kg) 및 부토르파놀(0.4mg/kg)을 근육 주사한 다음 티오펜탈 나트륨(100mg/kg)을 복강내 주사합니다.
- 통증 반사가 소실된 것을 확인한 후 흉강을 절개하여 심장을 노출시키고 우심방을 절개하여 혈액과 고정제가 빠져나갈 수 있도록 합니다.
- 그런 다음 좌심실을 절단하여 먼저 접두사 용액(0.02M 인산염 완충액[PB, pH 7.4])으로 혈액을 먼저 세척하기 위한 관류 바늘을 삽입한 다음 몇 분(~50-100mL) 동안 4% 파라포름알데히드/0.1M 인산염 완충액(PB, ~20분 동안 동물 무게의 2-3배)으로 고정합니다.
- 좋은 관류는 좋은 결과를 위해 매우 중요합니다. 관류 바늘을 삽입할 때는 중격을 따라 대동맥 쪽으로 전진시켜 고정제의 양호한 흐름을 확보합니다. 바늘 끝은 대동맥에 도달하면 보이게 됩니다. 끝이 경동맥을 우회하지 않도록 끝이 거의 보이지 않을 때까지 집어넣습니다.
- 뇌를 4°C에서 48시간 동안 4% 파라포름알데히드/0.1M PB로 유지하고 50mM PB로 전환합니다. 즉시 사용하지 않을 경우 뇌를 0.75% 글리신/0.1M PB에 보관하여 과고정으로 인한 자가형광을 방지하십시오.
2. 시료 준비(그림 1)
- 콜라겐 분해 효소 (3mM CaCl 중 1 mg/mL, 트리스 완충 식염수[TBS] 10 mL 중2 )로 고정 뇌를 37 °C에서 1시간 동안 배양합니다.
참고: 배양 전에 37°C에서 5분 동안 뇌를 예열합니다. - 면봉으로 뇌 표면을 조심스럽게 문질러 피아 물질과 다른 수막을 벗겨냅니다(그림 1A, 보충 비디오). 가는 집게를 사용하여 분리된 수막을 벗겨냅니다.
참고: 상구(superior colliculus) 또는 시상(thalamus) 상단에 있는 수막을 포함하여 중뇌를 둘러싼 수막을 제거하는 것이 중요합니다. 내면 깊숙이 숨겨져 있는 이러한 구조를 드러내는 것은 어렵다. 그러나 수막은 가능한 한 많이 제거하십시오. 그렇지 않으면 종종 절단되지 않은 상태로 남아 있고 떠다니며 이미징 및 슬라이싱을 방해합니다(그림 2A). - 뇌가 불소 용액에 담그는 동안 뇌가 삽입되기 전에 생체 외 MRI를 받을 때 각별히 주의하십시오15. 남은 불소는 함께 삽입되면 이미징하는 동안 대물렌즈 아래에 작은 기포를 형성할 수 있습니다. 이를 피하려면 삽입하기 전에 1주일 동안 뇌를 PB에 유지하십시오.
참고: 이 대기 단계는 일반적으로 필요하지 않지만 생체 외 MRI와 결합할 때 중요합니다. - 40°C로 예열된 100mL의 50mM 붕산염 완충액(pH 9.2)에 0.2g의 NaBH4를 용해하여 NaBH4 완충액을 준비합니다. CO2 가스가 배출되도록 캡을 느슨하게 두십시오. 캡을 느슨하게 한 상태에서 용액을 밤새 보관하십시오. 병을 알루미늄 호일로 싸서 사용할 때까지 빛으로부터 보호하십시오.
- 2-3 시간 동안 PB 100 mL에 아가로 스 2.25 g과 NaIO4 0.21 g을 교반하여 산화 된 아가로 스를 만든다. 진공 흡입으로 용액을 여과하고 PB를 세 번 갈아서 세척합니다. 아가로스를 PB 50mL에 재현탁합니다.
- 아가로스를 전자레인지에 완전히 녹여 60-65°C로 식힌다.
- 뇌를 주문 제작한 방에 넣고 뇌를 아가로스에 삽입한다. 아가로스를 corpus callosum 아래의 구멍에 도입하도록 주의하십시오(그림 1B).
알림: 먼저 뇌를 실온에서 예열한 다음 65°C에서 5분 동안 예열하여 아가로스가 응고되지 않고 뇌 표면에 정착할 수 있도록 합니다. - 챔버를 분해하고 아가로스 블록을 NaBH4 버퍼에 4°C에서 하룻밤 동안 담그십시오.
- 4°C에서 1-2주에 걸쳐 버퍼를 PB로 여러 번 변경합니다.
참고: 완충액 교환이 불완전하면 조직 자가형광이 매우 약해집니다. - 에폭시 인스턴트 믹스와 함께 4 개의 네오디뮴 자석을 부착하여 슬라이드 유리 무대를 만드십시오.
- 아가로스 블록을 s에 장착합니다.tage 강력한 접착제(예: 슈퍼 접착제)로 (그림 1C).
3. 조직 가공
- 제조업체의 지침에 따라 전체 조직 이미징 시스템을 작동하십시오. 자세한 프로토콜도 발표되어있습니다 16. 현재 절차는 주로 TissueCyte1000에 사용되지만 TissueCyte 1600FC와 같은 다른 모델에도 적용할 수 있습니다.
- 작동은 모델마다 약간 다르지만 일반적으로 중요한 아래 사항을 따르십시오.
- 마모셋 뇌 전체는 하드웨어의 변화 없이 전방에서 후방까지 관상동맥으로 처리될 수 있습니다. 뇌를 배치할 때 단계 이동 한계를 초과하지 않는지 확인하십시오.
- 블레이드의 각도를 미세 조정하여 블레이드 양쪽 끝의 표면 깊이가 10μm 이내가 되도록 합니다. 조밀한 수초화로 인해 10μm의 깊이 차이만 있어도 성체 마모셋 백질을 통해 다른 레이저 침투를 유발할 수 있습니다. 같은 이유로, 이미징 평면을 표면에서 약 25-35 μm로 설정합니다.
- 세라믹 칼날(권장)을 사용하여 50μm 간격(~650개 이상)으로 뇌 전체를 자릅니다.
- 수막은 절단되지 않은 상태로 남아 있으며 제대로 제거되지 않으면 절단을 방해합니다(그림 2). 일부 수막(예: 해마 또는 pulvinar를 둘러싼 수막)은 제거하기 어렵습니다. 발견되면 미세한 집게로 이러한 수막을 수동으로 추출하십시오.
4. 보조 조직학 기법
- 섹션 검색
- 조직 절편을 회수하여 다양한 조직학적 염색을 수행합니다. 이러한 섹션을 슬라이싱 순서로 정확하게 정렬합니다. 정렬을 위해 피질층을 가로질러 혈관을 추적합니다(그림 3).
- 더 나은 취급을 위해 각 섹션에서 과도한 아가로스를 잘라냅니다.
참고: 이 프로세스는 과도할 필요가 없습니다. 아가로스는 조직학적 처리를 방해하지 않고 조직 분절을 제자리에 유지하는 역할을 합니다. - 백라이트 이미징
- 섹션을 슬라이드 유리에 장착하고 말리십시오.
- PBS로 섹션을 재수화하고 이미징을 위해 커버슬립을 놓습니다.
- 명시야 모드에서 형광 현미경(그림 4)으로 단면을 이미지화할 때 염색이 없는 수초화 패턴을 관찰합니다. 빛의 반사가 보이도록 노출 시간을 조정합니다.
알림: 암시야 조명을 사용할 수 있는 경우 모든 현미경을 사용할 수 있습니다. - 커버슬립을 제거하고 Nissl 염색 또는 기타 염색을 진행합니다. 모든 표준 염색 절차로 충분합니다.
- 이 방법을 사용하여 다양한 염색 패턴을 수초화 패턴과 직접 비교할 수 있습니다(그림 4).
- BDA 이미징
- 자세한 절차는 표 1 을 참조하십시오.
- 먼저 Dent's Solution(20% DMSO, 80% 메탄올)으로 절편을 처리합니다. Dent의 솔루션에 포함된 메탄올은 축삭돌기의 BDA 신호를 크게 향상시킵니다.
- 티라미드 신호 증폭(TSA) 방법을 사용하여 BDA 신호가 형광 검출되도록 강화합니다17.
참고: TSA-비오틴 용액은 공급업체에서 구입할 수 있지만 사내 시약이 훨씬 저렴하고 효과적입니다. - STPT 이미지에 등록하려면 장착 매체를 사용하여 슬라이드 유리에 섹션을 장착한 후 역광 이미지를 촬영합니다.
- 스파게티 몬스터 형광 단백질(smFP) 염색
참고: '스파게티 몬스터' 형광 단백질(smFP)은 여러 개의 에피토프 태그를 가진 비형광 GFP 변이체의 계열입니다18. 이 제품은 면역 검출 시 우수한 형광 신호를 제공하며 형광 추적기의 동반 추적자로 적합합니다. Addgene에서 사용할 수 있는 AAV 구문에 대해서는 표 2 를 참조하십시오.- 표 1에 제공된 자세한 절차를 따릅니다.
5. 이미징 후 데이터 처리
- 데이터 무결성 테스트
참고: STPT는 방대한 수의 이미지 파일을 생성합니다. 성인 마모셋의 뇌가 50μm 간격으로 관상동맥 절편을 이루면 일반적으로 전체 뇌를 덮기 위해 650개 이상의 절편이 필요합니다. 각 섹션의 데이터는 3개의 채널로 캡처된 일련의 타일링 이미지로 구성됩니다. 이러한 이미지 파일은 각 타일의 위치를 기록하는 메타데이터 파일과 함께 단일 폴더에 저장됩니다. 총 데이터 양과 처리 시간을 최소화하기 위해 이미징은 실행 블록에서 수행되며, 이 동안 타일링을 위한 X 및 Y 단계의 수가 점진적으로 변경됩니다. 일반적으로 각각 30-50개의 섹션 이미징으로 구성된 20-30개의 실행 블록이 단일 뇌에 대한 완전한 데이터 세트를 구성합니다. 이 데이터를 활용하려면 타일링된 이미지의 스티칭, 형광 신호의 분할, 표준 템플릿에 체적 데이터 등록 등을 포함한 여러 이미지 처리 단계가 필요합니다.- 다양한 소프트웨어 도구를 사용하거나 연구자의 특정 요구 사항에 맞는 전용 파이프라인을 통해 이미지 처리 단계를 개별적으로 수행합니다. 이미지 처리에 대한 자세한 내용은 이 논문의 범위를 벗어나며 다른 출판물11,19에서 확인할 수 있습니다.
- 사용된 절차에 관계없이 데이터가 일관되게 구성되었는지 확인합니다.
참고: 전체 이미징 프로세스에는 많은 실행 블록이 포함되고 며칠에 걸쳐 진행되기 때문에 실행이 중간에 취소되고 새로 다시 시작되는 것은 드문 일이 아닙니다. 이러한 경우 데이터 구조가 중단되어 이미지 처리 파이프라인의 적절한 실행을 방해할 수 있습니다. - 데이터 구조가 중단될 위험을 최소화하려면 Python 스크립트를 실행하여 GitHub(github.com/watkarbey/STPT_depo)에서 사용할 수 있는 데이터 구조(kn_pipeline_check_mosaic.py)의 무결성을 확인합니다.
- STPT 이미지에 스테인된 이미지의 정합
참고: 스테인된 이미지를 STPT 이미지로 비선형 변환은 ImageJ의 bUnwarpJ 플러그인을 사용하여 수행할 수 있습니다.- 대상(STPT) 이미지와 소스(스테인드) 이미지의 크기를 거의 같아지도록 조정합니다.
- 각 이미지를 이진화하여 섹션의 모양을 시각화합니다.
- bUnwarpJ를 실행합니다. Save Transformations 옵션을 선택하는 것을 잊지 마십시오.
- bUnwarpJ를 다시 시작합니다. 이진화 전의 원본 이미지를 소스 이미지로 선택합니다. 그런 다음 Load Elastic Transformation을 클릭하고 저장된 변환을 선택합니다.
결과
여기에 사용된 일반적인 설정에서, 성인 마모셋의 전체 뇌는 약 1주일 만에 50μm 절편 간격으로 ~1.3 x 1.3 μm/픽셀의 해상도로 이미지화할 수 있습니다(그림 5). 이는 이미지 스티칭 후 3개 채널에서 ~650개의 코로나 이미지에 해당합니다. 16x 대물 렌즈(Nikon 16xW, CFI75 LWD; NA = 0.80), 단일 샷의 시야는 약 1 x 1mm입니다. 전체 코로나 표면에 대한 이미지는 이러한 샷을 스티칭하여 얻습니다(그림 5A). Z 방향의 정렬이 우수하고 코로나 이미지 데이터를 적층하기만 하면 좋은 3D 이미지를 얻을 수 있습니다(그림 5B, E). 표준화를 위해 마모셋 브레인11 에 대한 STPT 템플릿을 3D-3D 정합에 사용할 수 있습니다(그림 5F). 이 데이터 변환은 전체 뇌 신경 해부학의 주요 측면 중 하나이며, 관심 영역에는 해부학적 주석과 독립적인 절대 공간 좌표가 할당됩니다. 샘플 브레인이 표준 공간에 등록되면 추가 분석을 위해 관심 있는 하위 영역을 쉽게 제거할 수 있습니다(그림 5F, G). 특히, 피질 영역은 미리 결정된 매개변수를 사용하여 플랫 맵으로 변환될 수 있습니다(그림 5H).
이미징 중에 생성된 절편은 다양한 조직학적 목적으로 사용될 수 있습니다. 그림 4A, B에서 볼 수 있듯이, 염색이 없는 역광 이미징은 실제 미엘린 염색과 매우 유사한 패턴을 제공합니다. 이것은 미엘린 염색에 대한 훌륭한 대안이 될 수 있습니다. 또한, Nissl 염색 전에 백라이트 이미지를 얻은 경우 동일한 섹션을 사용하여 미엘린 및 Nissl 염색 패턴을 모두 얻을 수 있으므로 영역과 층을 식별하는 데 유용한 정보를 제공할 수 있습니다(그림 5C-E). 이 절편은 면역학적 염색에도 사용할 수 있습니다. 그림 5J에서 주사 센터 주변 부분을 NeuN 항체로 대조염색하여 AAV 바이러스의 transduction efficiency를 추정했습니다. 또한 형광 추적자 외에 비형광 추적자를 주입하고 절편 검색 후 조직학적으로 검출하는 것도 좋은 전략입니다. 이전 연구에서는 선행성 녹색 추적자를 역행 "cre" 벡터와 결합했으며, 이는 나중에 anti-cre 항체10에 의해 검출되었습니다. 그림 6A-E의 예에서, BDA를 녹색 추적자(클로버)의 반대쪽 측에 주입하고 형광으로 검출했습니다. 적색 BDA 신호는 TissueCyte 이미지에 등록되어 전체 뇌 좌표에 국소화될 수 있습니다. 그림 6F-I의 또 다른 예에서, smFP-myc는 두정엽 영역(그림 6G)에 주입되었고, 녹색 추적자는 전두엽 영역에 주입되었습니다. 이러한 방식으로 이미징을 방해하지 않고 동일한 동물에 여러 추적자를 주입할 수 있습니다. 추가 염색을 위해 STPT 절편을 사용하는 것의 큰 장점은 동일한 뇌에 대해 형광 추적자와 비형광 추적자 간의 관계를 결정할 수 있다는 것입니다. 따라서, 우리는 고정밀10에서 피질 피질 돌출의 상호성을 결정할 수 있었습니다. 또 다른 장점은 염색된 단면의 3D 좌표를 STPT 데이터에 다시 매핑한 다음 표준 템플릿에 매핑할 수 있다는 것입니다. 따라서 검색된 모든 섹션을 염색에 사용할 필요가 없을 수도 있습니다. 더 나은 해석을 위해 STPT 데이터에 더 많은 컨텍스트를 추가하기 위해 염색할 섹션을 선택할 수 있습니다.
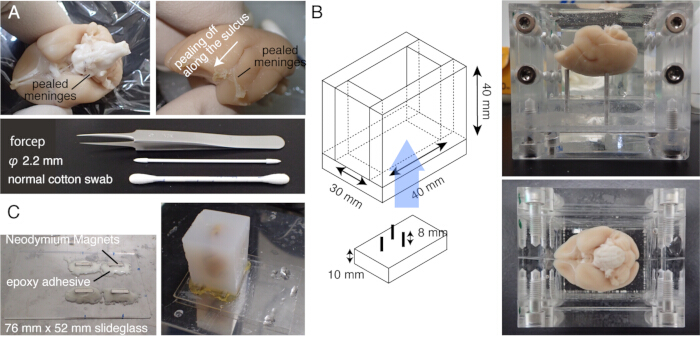
그림 1: STPT를 위한 시료 준비. (A) 면봉을 사용한 수막막 제거. 뇌간을 둘러싼 수막은 끝이 가는 집게로 제거할 수 있습니다. 마모셋 뇌를 둘러싼 수막은 면봉으로 문질러 수동으로 제거합니다. 사진은 측면 열구 내부에서 벗겨진 수막을 보여줍니다. (B) 아가로스 포매에 사용되는 아크릴 상자. 핀들은 움직일 수 있고, 입체택적으로 고정된 위치(20)에 가깝게 뇌의 각도를 조절하는데 사용된다. (C) 76mm x 52mm 슬라이드 유리와 에폭시 접착제로 부착된 4개의 네오디뮴 자석으로 만들어진 마그네틱 슬라이드. 아가로스 블록은 슈퍼 접착제로 마그네틱 스테이지에 부착됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
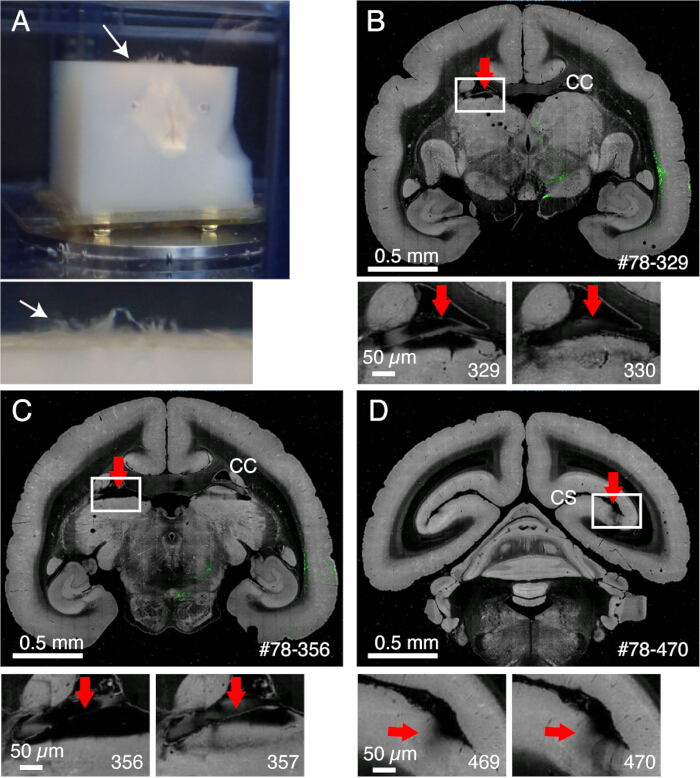
그림 2: 수막이 영상에 미치는 영향. (A) 절단되지 않은 상태로 남아 있는 수막은 흰색 화살표로 표시된 것처럼 위로 떠오릅니다. 이 예에서는 수막이 아가로스 포매 전에 제거되지 않았기 때문에 수막의 광범위한 돌출이 보입니다. 일반적으로 수막은 몇 가지 어려운 부위에 대해서만 절단되지 않은 상태로 남아 있습니다. (B) 뇌량(corpus callosum)과 시상(thalamus)의 상부 사이에 수막(meninges)의 존재로 인한 나쁜 절단의 예. 이 경우, 잘못된 슬라이싱으로 인해 딥 슬라이스(329)와 상대적으로 정상적인 슬라이스(330)가 번갈아 나타납니다. #78-329는 Brain/MINDS 데이터 포털의 샘플 번호 78 섹션 번호 329를 나타냅니다 (C) 나쁜 이미징의 또 다른 예입니다. 더 나쁜 경우, 빨간 화살표로 표시된 pulvinar nucleus가 완전히 떨어져 나갈 수 있습니다. 스케일 바: 0.5mm(상단 패널), 0.5μm(하단 패널). (D) 나쁜 영상의 또 다른 예. 칼카린 열구 깊숙한 곳에 있는 수막은 제거하기가 어렵습니다. 섹션 469와 470에서 보이는 그림자는 대물렌즈 아래에 떠 있는 수막에 의해 발생합니다. 스케일 바: 0.5mm(상단 패널), 0.5μm(하단 패널). 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
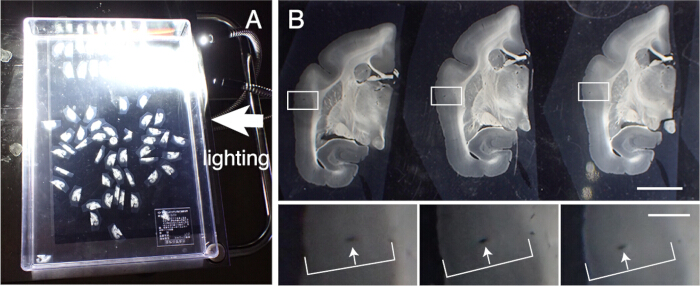
그림 3: 조직 절편을 순서대로 정렬 .(A) 최대 50개의 마모셋 관상 절편을 플라스틱 용기(31cm x 22.5cm)에 순서대로 올바르게 정렬할 수 있습니다. 세부적인 구조를 시각화하기 위해 용기를 검은 종이 위에 놓고 측면에서 조명을 비춥니다. 이러한 섹션은 먼저 대략적으로 순서대로 정렬된 다음 정확한 정렬이 적용됩니다. (B) 정확한 정렬은 혈관을 마커로 사용합니다. 대뇌 피질에는 피질층을 가로질러 수직으로 뻗어 있는 많은 혈관이 있습니다. 그것들은 피질층 내에서 체계적으로 위치를 변경하는 길쭉한 구멍(흰색 화살표)으로 식별됩니다. 이 방법을 사용하면 50μm 간격의 섹션도 정확하게 정렬할 수 있습니다. 이러한 혈관 구멍은 확인을 위해 SPTP 절편 이미지에서 확인할 수 있습니다. 눈금자: 5mm(상단 패널), 1mm(하단 패널). 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
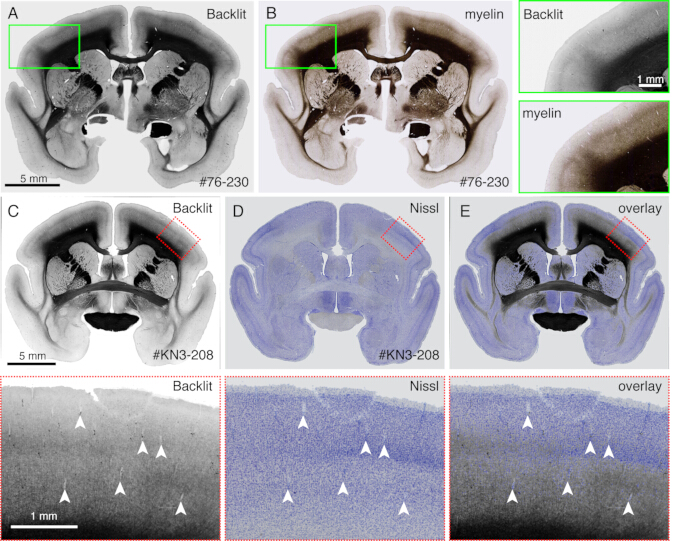
그림 4: 미엘린 염색을 대체하는 역광 이미지.(A,B) 동일한 단면이 역광 이미징 및 미엘린 염색에 사용되었습니다. 먼저, 단면을 슬라이드 유리에 장착하고, 건조하고, PBS로 재수화하고, 커버슬립하고, 광학 현미경을 사용하여 이미지화했습니다(Table of Materials). 커버슬립을 제거한 후, 동일한 절편을 미엘린 염색21에 사용하고 형광 현미경을 사용하여 이미지화했습니다. 백라이트 이미지는 ImageJ의 bUnwarpJ 플러그인을 사용하여 미엘린 이미지에 등록되었습니다. 녹색 상자는 각 이미지의 확대된 보기를 보여줍니다. 이 이미지는 미엘린 염색이 섬유질 구조를 더 잘 시각화한다는 점을 제외하고는 거의 동일한 패턴을 보여줍니다. (씨-E) 동일한 섹션이 백라이트 이미징 및 Nissl 염색에 사용되었습니다. 역광 이미지의 낮은 임계값 마스크는 bUnwarpJ 플러그인을 사용하여 Nissl 이미지의 낮은 임계값 마스크에 먼저 등록되었으며 원본 이미지는 동일한 매개변수를 사용하여 변환되었습니다. 혈관(흰색 화살촉)은 두 이미지 사이에서 잘 일치합니다. 이 두 이미지는 동일한 섹션에 대한 것이기 때문에 일치가 거의 완벽하며 피질층을 식별하기 위해 myelin 및 Nissl 패턴을 직접 비교할 수 있습니다. 스케일 바: 5mm(패널 AE). 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
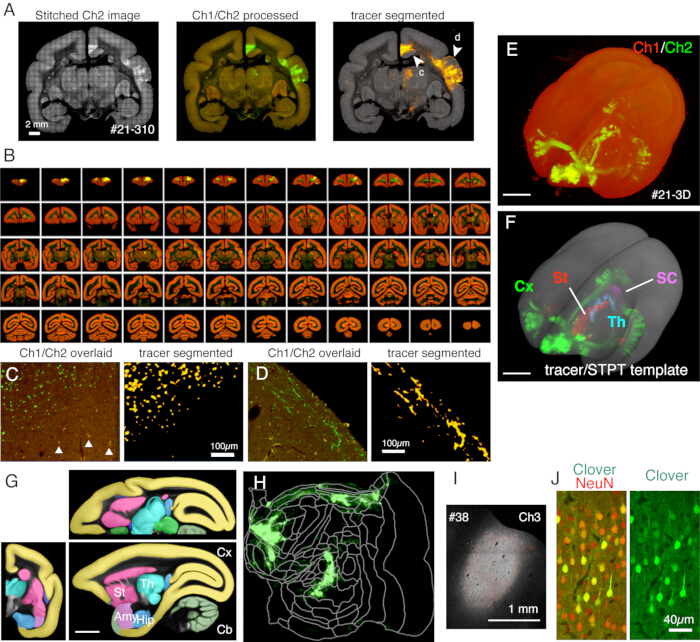
그림 5: STPT 이미징의 일반적인 결과. (A) 예시 섹션의 Ch2(녹색) 이미지의 간단한 타일링(샘플 #21의 섹션 310). 배경을 보정하지 않으면 각 타일의 테두리가 표시됩니다. 가운데 열에서 타일은 각각 Ch1(빨간색) 및 Ch2(녹색)에 대한 배경 보정으로 스티치되었습니다. 리포푸신(lipofuscin)의 신호를 줄이기 위해(패널 C 참조), Ch2에서 Ch1 신호를 빼고 녹색으로 표시했습니다. Ch1의 lipofuscin 신호를 줄이기 위해 ImageJ에 의해 스티칭하기 전에 "Remove Outilers.." 명령이 사용되었습니다. 오른쪽 열에서 트레이서 신호는 이미지 처리 파이프라인11에 의해 분할되었습니다. 화살촉 (c, d)은 확대 된 보기가 패널 C 및 D에 표시되는 위치를 보여줍니다. 스케일 바: 2mm. (B) 샘플 #21의 직렬 섹션 개요. STPT는 이 샘플을 위해 635개의 고해상도 코로나 이미지를 생성했습니다. (C) 패널 A에서 화살촉 c로 표시된 고배율 보기. 이것은 추가 처리가 없는 Ch1(빨간색) 및 Ch2(녹색)의 간단한 오버레이입니다. 삼각형은 광범위한 스펙트럼을 보여주는 리포푸신 형광 신호를 보여줍니다. 트레이서 분할 알고리즘은 매우 유사한 모양(오른쪽 패널)에도 불구하고 리포푸신 배경에서 트레이서 신호를 정확하게 구별합니다. 스케일 바 : 100 μm. (D) 고배율보기의 또 다른 예. 층 1의 미세한 축삭 섬유가 잘 보일 수 있습니다. 눈금 막대: 100 μm. (E) 원본 STPT 이미지의 3D 재구성. 패널 B에 표시된 635개의 저해상도 코로나 이미지는 fluorender22를 사용하여 3D 시각화를 위한 tiff 스택으로 사용되었습니다. 스케일 바: 5mm. (F) STPT 템플릿(회색)에 등록된 세그먼트 트레이서 신호의 3D 재구성. 서로 다른 뇌 영역의 추적 신호는 서로 다른 색상으로 표시되었습니다. 이 영역은 패널 G에 표시된 주석을 사용하여 잘라냈습니다. 스케일 바: 5mm. (G) 다른 뇌 영역의 주석으로 오버레이된 STPT 템플릿. 스케일 바: 5mm. (H) 패널 F에 표시된 피질 추적기 신호는 플랫맵 형태로 표시되었습니다. (I) 추적자 형광에 덜 민감하고 불포화 상태로 유지되는 Ch3 형광에 의한 주입 부위 식별. 스케일 바: 1mm. (J) 주사 센터 주변 부분을 NeuN 항체로 염색한 결과 약 30%의 뉴런이 클로버 그린 형광의 강한 발현을 보이는 것으로 나타났습니다. 기준자: 40 μm. 약어: Cx; 피질, 세인트; 선조체, 토; 시상, 사우스캐롤라이나; 우수한 colliculus. 에이미; 편도체, 엉덩이; 해마, Cb; 소뇌. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
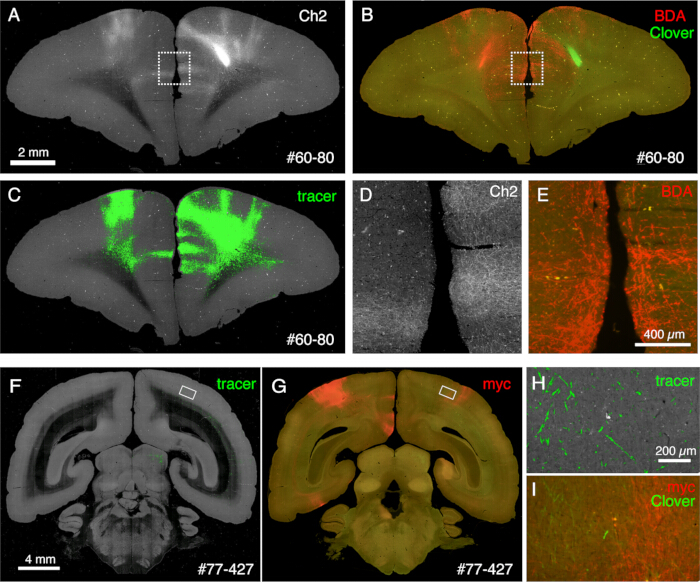
그림 6: 여러 개의 비형광 추적자를 보여주는 STPT 후 조직학. (에이씨) STPT 이미지와 BDA 염색 이미지의 비교. 이 샘플에서 BDA는 Clover 주입의 반대쪽 측에 주입됩니다. 패널 A와 C는 STPT 데이터의 Ch2 이미지 및 트레이서 분할을 보여줍니다. 패널 B는 BDA에 대한 STPT 염색 후 모습을 보여줍니다. 클로버 형광은 단면의 메탄올 처리로 인해 감소합니다. 눈금자: 2mm. (D,E) 패널 A와 B의 점선 상자가 확대됩니다. 스케일 바: 100 μm. (F,G) STPT 이미지와 anti-myc 태그 항체 염색의 비교. 이 샘플에서 클로버 주입은 PFC에 주입되는 반면 AAV-smFP-myc는 반대쪽 두정엽 피질에 주입됩니다. 흰색 사각형은 패널 H와 I에서 확대됩니다. 눈금 막대: 4mm. (H) 트레이서 분할은 녹색으로 표시됩니다. 눈금 막대 : 200 μm. (I) myc 염색은 빨간색으로 표시됩니다. 클로버 형광은 녹색으로 표시됩니다. 가뭄줄: 200 μm. 패널 H와 I의 녹색 신호는 유사한 위치에 존재하지만 STPT는 ~10μm 광학 섹션만 검색하기 때문에 동일하지는 않습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| BDA 형광 염색 프로토콜 | |
| 아가로스 제거 | |
| TBS 워시 | 10분(2회) |
| 덴트 용액의 1% H2O2 | 10분 |
| TBS 워시 | 짧다 |
| 0.5% TNB 차단 | 1시간 |
| TNB의 StAvHRP (1:4000) | 2 하룻밤 |
| TNT 워시 | 10분 (3회) |
| 0.1M 붕산염(pH8.5) + 0.003% H2O2의 TSA 비오틴(1:4000) | 2시간 |
| TNT 워시 | 10분 (3회) |
| TNT의 Cy3-streptavidin (1:1000) | 3시간 |
| TNT 워시 | 10분(2회) |
| TBS 워시 | 장착할 때까지 섹션을 유지하십시오. |
| 퇴색 방지 장착 매체를 사용하여 슬라이드 유리에 섹션을 장착합니다. | |
| Anti-MyC 형광 염색 프로토콜 | |
| 아가로스 제거 | |
| TBS 워시 | 10분(2회) |
| IB에서 차단 | 1시간 |
| IB의 Anti-Myc (1:4000) | 2 하룻밤 |
| TNT 워시 | 10분 (3회) |
| TNT의 안티 마우스 Cy3 (1:1000) | 3시간 |
| TNT 워시 | 10분(2회) |
| TBS 워시 | 장착할 때까지 섹션을 유지하십시오. |
| 퇴색 방지 장착 매체를 사용하여 슬라이드 유리에 섹션을 장착합니다. | |
| 버퍼/솔루션 | 구성 |
| 0.5% 티엔비 | TS7.5의 0.5% TSA 차단 시약 |
| Dent의 솔루션 | 20% DMSO, 80% 메탄올 |
| 침지 버퍼(IB) | 10% FBS, 2% BSA 0.5% TritonX100(TBS) |
| TBS (트리스 완충 식염수) | 25mM 트리스, 137mM NaCl, 2.7mM KCl(pH 7.4) |
| 티엔티 | TS7.5의 0.05% Tween20 |
| TS7.5 | 0.1 M 트리스-HCl, pH 7.5, 0.15 M NaCl |
표 1: BDA 형광 염색 및 anti-myc 형광 염색 프로토콜
| AAV 트레이서용 플라스미드 | 애드진: No. | 권장 항체 | 제안된 희석 | 예상 결과 |
| pAAV-EF1_Cre | 201198 | 밀리포어 클론 2D8 | 1:1000 | 세포에 좋습니다 |
| pAAVCam1.3_smFP_Myc | 201205 | MBL M192 My3 (마우스) | 1:4000 | 훌륭한 |
| pAAVCam1.3_smFP_HA | 201206 | CST C29F4 (토끼) | 1:1000 | 세포에 좋습니다 |
| pAAVCam1.3_smFP_FLAG | 201207 | MBL PM020B (토끼) | 1:1000 | 그래 |
| AAVTRE3_smFP_Myc | 201208 | |||
| AAVTRE3_smFP_HA | 201209 | |||
| AAVTRE3_smFP_FLAG | 201210 |
표 2: 비형광 추적자 생산에 사용할 수 있는 Addgene 플라스미드 목록. cre 구조체는 핵을 표적으로 하며 AAV2 retro로 둘러싸인 역행 추적에 적합합니다. smFP_HA 구조는 세포체 검출(및 역행)에 좋습니다.
보충 비디오: 수막 제거의 현미경 보기. 이 비디오를 다운로드하려면 여기를 클릭하십시오.
토론
이 기사에서는 전체 뇌 처리를 위해 마모셋 뇌를 처리하는 실용적인 솔루션과 STPT 기술의 유용성을 향상시키는 보조 조직학적 기술에 대해 설명했습니다. STPT를 사용하는 "전뇌 신경 해부학"의 강점은 해부학적으로 주석이 달렸는지 여부에 관계없이 모든 관심 영역의 3D 좌표를 얻을 수 있다는 것입니다. 고정밀 3D-3D 등록을 통해 이러한 좌표를 여러 데이터 세트의 오버레이를 위한 표준 템플릿으로 변환할 수 있습니다. 이러한 방식으로 표준 템플릿은 데이터 통합의 매체 역할을 합니다. 이것은 전전두엽 피질(PFC) 매핑 프로젝트10의 필수적인 측면이었으며, 많은 개인으로부터 얻은 데이터를 분석했습니다. 더욱이, 표준 템플릿에 매핑된 정보는 Nissl, 미엘린 패턴, 추적자 데이터, MRI(확산 MRI 포함) 데이터, 또는 해부학적 주석(11) 등 이미 매핑된 다양한 데이터와 비교할 수 있다. 중요한 것은 아직 등장하지 않은 기술로 얻을 수 있는 미래 데이터와도 비교할 수 있다는 것입니다. 현재 마모셋 뇌에는 Nissl staining23, MRI 23,24,25,26,27,28 및 STPT 11을 기반으로 하는 여러 템플릿이 존재합니다. 그러나 이미지 대비와 미리 계산된 매개변수11을 기반으로 서로의 좌표로 변환될 수 있습니다. 연구 전반에 걸친 전체 뇌 규모의 데이터 통합은 시스템으로서의 뇌를 더 잘 이해하는 데 기여합니다. 이 전략이 효과를 발휘하기 위한 전제 조건은 뇌 전반에 걸쳐 신뢰할 수 있는 데이터 수집입니다. 아래에서는 현재 프로토콜과 관련된 중요한 단계 및 잠재적인 문제에 대해 설명합니다.
STPT 이미징의 가장 취약한 프로세스 중 하나는 조직 슬라이싱입니다. 위에서 언급했듯이 수막은 종종 절단되지 않은 상태로 남아 있어 절단을 방해할 수 있습니다. 특히, pulvinar nucleus와 superior colliculus는 가장 영향을 많이 받는 두 개의 뇌 영역입니다: 수막을 제거하고 아가로스에 포매하는 정도에 따라 절단하는 동안 조직 블록에서 벗겨낼 수 있습니다. 이 깊은 영역은 외부에서 접근하기 어렵고 수막을 제거하는 동안 쉽게 끊어질 수 있습니다. 신중하지만 철저한 수막 제거는 성공적인 이미징을 위해 매우 중요합니다. 또 다른 우려 사항은 얇게 썬 부분이 조직 블록으로 뒤집히는 것인데, 이는 슬라이스 후에도 섹션이 부착된 상태로 유지될 때 때때로 발생합니다. 블레이드가 맨 끝에서 비스듬히 절단되도록 블록을 형성하여 최소화할 수 있습니다.
STPT는 조직 블록 깊숙이 이미징함으로써 표면의 울퉁불퉁함을 방지합니다. 형광 신호는 피질 영역을 다소 쉽게 통과할 수 있는 반면, 수초화된 영역에서는 크게 감소합니다. 따라서 이미징 깊이는 전체 블록 표면에 걸친 일관된 이미징과 백질과 같은 미엘린이 풍부한 영역에서 신호의 밝기 사이의 균형을 맞추기 위해 신중하게 결정해야 합니다. 여기에 사용된 설정에서는 일반적으로 표면에서 25-35μm를 조준합니다. 또한 절단면이 장시간 보관 후 고르지 않게 줄어들 수 있다는 점에 주의해야 합니다. 이미징 영역을 최소화하기 위해 이미징 세션을 5-6일 동안 서로 다른 스테이지 설정을 사용하여 20-30개의 실행으로 분할합니다. 우리는 주행 사이의 간격을 2시간 미만으로 만들거나 표면 깊이를 확인하고 각 실행 전에 스테이지 높이를 조정합니다.
이 프로토콜에서 BDA 신호는 TSA 방법에 의해 증폭되었습니다. 이 방법은 매우 효과적이며 상대적으로 낮은 분해능에서도 전방암으로 수송된 BDA 신호를 검출할 수 있습니다(예: 그림 6B). TSA 비오틴은 Akoya Biosciences에서 시판하고 있지만 홈메이드 솔루션이 훨씬 더 향상되었습니다. 반면에 항체와 용액의 희석은 최적의 결과를 얻기 위해 신중한 조정이 필요합니다. 메탄올 용액으로 단면을 전처리하는 것이 중요합니다. 전처리가 없으면 BDA 신호는 수초화된 축삭돌기에서 거의 감지되지 않습니다.
선행성 추적자를 사용하는 경우 형광 신호의 포화로 인해 정확한 주입 부위를 식별하기 어려운 경우가 많습니다. 이 연구에 사용된 전체 조직 이미징 시스템 설정에서 파란색 채널을 사용하여 감염된 세포를 식별합니다(그림 5I). 적색 및 녹색 채널이 포화 상태일 때에도 감염된 각 뉴런은 일반적으로 파란색 채널에서 검출될 수 있습니다. Allen Mouse Brain Connectivity Atlas1의 경우에도 마찬가지입니다. 기원 세포에 대한 검사가 중요한 이유는 감염이 때때로 특정 층만을 관련시키기 때문입니다. 우리는 Mouse Brain Connectivity Atlas에서 이러한 부분적 감염을 꽤 자주 접했는데, 아마도 iontophoresis 방법29의 활용 때문일 것입니다. 바이러스 추적자의 측면 확산은 주사에 따라 다소 다양할 수 있습니다. 이러한 가변성은 추적 결과에 영향을 줄 수 있으며 신중한 정규화가 필요합니다.
획득한 3D 이미지를 표준 템플릿에 성공적으로 등록하는 것은 전체 뇌 신경 해부학의 핵심 프로세스입니다. STPT 템플릿에 대한 STPT 이미지의 등록은 매우 정확할 수 있으며, 이미지 대비10이 높은 경계에 대해 몇 개의 복셀(50μm 등입방체)의 일탈만 관찰되었습니다. 그러나 등록이 할 수 있는 일에는 제한이 있습니다. 수막 제거 과정으로 인해 STPT 샘플은 일반적으로 반구 사이와 피질과 중뇌/후뇌 사이에 틈이 있는 반면, 뇌 조직은 생체 내 MRI 이미지에서 빽빽하게 채워져 있습니다. 이러한 차이는 등록으로 조정하기 어렵습니다. 마모셋 피질(Marmoset cortex)에는 대부분 설시(sulci)가 없지만, 두정내 열구(intraparietal sulcus)는 일부 개체에서 매우 깊습니다. 이러한 sulci는 등록 시 손실됩니다(하향식 융합 발생). 등록은 강력한 기술이지만 얻은 결과를 확인하기 위해 원시 데이터로 돌아가야 합니다.
방대한 이미지 데이터의 생성은 이 기술의 강점이자 한계입니다. 데이터 세트의 완전성을 향상시키는 동시에 수집된 데이터를 신중하게 관리하고 효율적인 데이터 해석을 위해 자동화된 이미지 처리 파이프라인을 개발해야 합니다. 미래에는 이미지 처리 파이프라인을 구축하는 데 생성형 인공 지능(AI)을 적용하면 이 프로세스가 크게 간소화될 수 있습니다. 또한 슬라이드 스캐너 기반 방법을 사용하여 체계적인 전뇌 영상이 수행되었습니다21,30. 이러한 방법과 비교하여 STPT는 3D 재구성을 위한 추가 계산이 필요하지 않습니다. 광학 절편과 결합하여 STPT가 섹션31에 걸쳐 균일한 축삭 분절을 재구성할 수 있는 잠재력이 있음을 입증했습니다. 조직 제거 기술의 추가 조합으로 Economo 등은 희박하게 표지된 뉴런 전체를 이미지화하는 방법을 개발했습니다32,33. 최신 버전의 TissueCyte는 이제 적색 형광 단백질의 여기를 향상시키기 위한 추가 레이저 또는 절편의 자동 복구를 위한 절편 캡처 장치 옵션을 제공합니다. 이러한 발전으로 전뇌 접근법은 더욱 효율적이 될 것이며, 인간을 포함한 영장류의 뇌에 대한 포괄적인 이해를 위한 토대를 제공할 것입니다.
공개
저자는 공개할 내용이 없습니다.
감사의 말
Yamamori lab and animal facilities(RRD)의 기술 직원들의 도움에 깊이 감사드립니다. 컨포칼 이미지 획득에 대한 기술 지원을 해주신 RIKEN CBS-Olympus 협업 센터에 감사드립니다. 이 작업은 일본 문부과학성의 혁신 분야에 대한 과학적 연구 프로그램(보조금 번호 22123009), 일본 AMED의 Brain/MINDS 및 Brain/MINDS2.0(JP15dm0207001, JP23wm0625001 및 JP24wm0625218), 그리고 JSPS KAKENHI 보조금 번호 24K09678(A.W.)의 지원을 받았습니다.
자료
| Name | Company | Catalog Number | Comments |
| Agarose type I | Sigma | A6013 | |
| All-in-one fluorescence microscope | Keyence | BZ-X series | This study used BZ-X710 |
| anti-mouse Cy3 | Jackson Immuno Research Laboratories, Inc. | 115-165-003 | |
| anti-myc antibody | MBL | M192 | mouse monoclonal |
| Butorphanol | Meiji Animal Health | Vetorphale | |
| Ceramic blade | TissueVision | N/A | |
| Collagenase type I | Wako Chemical | #031-17601 | Aliquot into 100 µL x 10 of 100 mg/mL in PBS |
| Cotton swab (HUBY-340) | HUBY | BB-013SP | micro cotton swabs |
| Custom-made chamber | N/A | N/A | The chamber consist of four side plates and one bottom plates, all made of 1 cm thick acrylic plates, which can be securely fastened together using screw bolts. An additional acrylic plate is placed inside the chamber, with three metal poles inserted through it to hold the marmoset brain in an inverted position. |
| Cy3-Streptavidin | Jackson Immuno Research Laboratories, Inc. | 016-160-084 | |
| Dumont #5 forceps, Inox | N/A | N/A | |
| Epoxy instant mix | Locktite | N/A | |
| FIJI | NIH | https://imagej.net/software/fiji/downloads | |
| kn_pipeline_check_mosaic.py | N/A | N/A | github.com/watkarbey/STPT_depo; This is a checking script for TissueCyte data. |
| Medetomidine | Zenoaq | Domitor | |
| Midazolam | Sandoz | N/A | |
| NaBH4 | Sigma | #452882 | |
| NaIO4 | Sigma | S1878 | |
| Perfusion needle | Natsume Seisakusho Co., Ltd | KN-348, 20G-50 | |
| Slideglass (76 mm x 52 mm) | Matsunami | S9111 | |
| StAvHRP | Jackson Immuno Research Laboratories, Inc. | 016-030-084 | HRP-streptavidin (titration needs to be carefully determined) |
| TissueCyte1000 | TissueVision | https://www.tissuevision.com/tissuecyte | |
| TSA blocking reagent | Kiko Tech | FP1012 | Refer to TSA-biotin kit (Akoya Biosciences) |
| TSA-biotin | House-made | N/A | See Okamoto et al (ref. 16) |
| VECTASHIELD HardSet Mounting Medium | Vector laboratories Inc | N/A | Antifade mounting medium |
참고문헌
- Oh, S. W., Harris, J. A., Ng, L., Winslow, B., Cain, N., et al. A mesoscale connectome of the mouse brain. Nature. 508 (7495), 207-214 (2014).
- Ragan, T., et al. Serial two-photon tomography for automated ex vivo mouse brain imaging. Nat Methods. 9 (3), 255-258 (2012).
- Liang, Z., Arefin, T. M., Lee, C. H., Zhang, J. Using mesoscopic tract-tracing data to guide the estimation of fiber orientation distributions in the mouse brain from diffusion MRI. Neuroimage. 270, 119999 (2023).
- Watakabe, A., Hirokawa, J. Cortical networks of the mouse brain elaborate within the gray matter. Brain Struct Funct. 223 (8), 3633-3652 (2018).
- Ypma, R. J. F., Bullmore, E. T. Statistical analysis of tract-tracing experiments demonstrates a dense, complex cortical network in the mouse. PLoS Comput Biol. 12 (9), e1005104 (2016).
- Wang, Q., et al. The Allen mouse brain common coordinate framework: A 3D reference atlas. Cell. 181 (4), 936-953.e20 (2020).
- Köbbert, C., Apps, R., Bechmann, I., Lanciego, J. L., Mey, J., Thanos, S. Current concepts in neuroanatomical tracing. Prog Neurobiol. 62 (4), 327-351 (2000).
- Lanciego, J. L., Wouterlood, F. G. Neuroanatomical tract-tracing techniques that did go viral. Brain Struct Funct. 225 (4), 1193-1224 (2020).
- Bakker, R., Wachtler, T., Diesmann, M. CoCoMac 2.0 and the future of tract-tracing databases. Front Neuroinform. 6, (2012).
- Watakabe, A., et al. Local and long-distance organization of prefrontal cortex circuits in the marmoset brain. Neuron. 111 (14), 2258-2273.e10 (2023).
- Skibbe, H., et al. The Brain/MINDS marmoset connectivity resource: An open-access platform for cellular-level tracing and tractography in the primate brain. PLoS Biol. 21 (6), e3002158 (2023).
- Mitchell, J. F., Leopold, D. A. The marmoset monkey as a model for visual neuroscience. Neurosci Res. 93, 20-46 (2015).
- Okano, H. Current status of and perspectives on the application of marmosets in neurobiology. Annu Rev Neurosci. 44, 27-48 (2021).
- Watakabe, A., et al. Comparative analyses of adeno-associated viral vector serotypes 1, 2, 5, 8 and 9 in marmoset, mouse and macaque cerebral cortex. Neurosci Res. 93, 144-157 (2015).
- Hata, J., et al. Multi-modal brain magnetic resonance imaging database covering marmosets with a wide age range. Sci Data. 10, 221 (2023).
- Liwang, J. K., Bennett, H. C., Pi, H. -. J., Kim, Y. Protocol for using serial two-photon tomography to map cell types and cerebrovasculature at single-cell resolution in the whole adult mouse brain. STAR Protoc. 4 (1), 102048 (2023).
- Okamoto, S., et al. Exclusive labeling of direct and indirect pathway neurons in the mouse neostriatum by an adeno-associated virus vector with Cre/lox system. STAR Protoc. 2 (1), 100230 (2021).
- Viswanathan, S., et al. High-performance probes for light and electron microscopy. Nat Methods. 12 (6), 568-576 (2015).
- Kuan, L., et al. Neuroinformatics of the Allen mouse brain connectivity atlas. Methods. 73, 4-17 (2015).
- Paxinos, G., Watson, C., Petrides, M., Rosa, M., Tokuno, H. . The Marmoset Brain in Stereotaxic Coordinates. , (2012).
- Abe, H., et al. 3D reconstruction of brain section images for creating axonal projection maps in marmosets. J Neurosci Methods. 286, 102-113 (2017).
- Wan, Y., et al. FluoRender: Joint freehand segmentation and visualization for many-channel fluorescence data analysis. BMC Bioinformatics. 18 (1), 280 (2017).
- Majka, P., et al. Towards a comprehensive atlas of cortical connections in a primate brain: Mapping tracer injection studies of the common marmoset into a reference digital template. J Comp Neurol. 524 (11), 2161-2181 (2016).
- Liu, C., Yen, C. C. -. C., Szczupak, D., Tian, X., Glen, D., Silva, A. C. Marmoset brain mapping V3: Population multi-modal standard volumetric and surface-based templates. Neuroimage. 226, 117620 (2021).
- Liu, C., et al. A digital 3D atlas of the marmoset brain based on multi-modal MRI. Neuroimage. 169, 106-116 (2018).
- Liu, C., et al. A resource for the detailed 3D mapping of white matter pathways in the marmoset brain. Nat Neurosci. 23 (2), 271-280 (2020).
- Woodward, A., et al. The Brain/MINDS 3D digital marmoset brain atlas. Sci Data. 5, 180009 (2018).
- Saleem, K. S., Avram, A. V., Glen, D., Schram, V., Basser, P. J. The subcortical atlas of the marmoset ("SAM") monkey based on high-resolution MRI and histology. Cereb Cortex. 34 (4), bhae120 (2024).
- Watakabe, A., Hirokawa, J. Cortical networks of the mouse brain elaborate within the gray matter. Brain Struct Funct. 223 (8), 3633-3652 (2018).
- Lin, M. K., et al. A high-throughput neurohistological pipeline for brain-wide mesoscale connectivity mapping of the common marmoset. eLife. 8, e40042 (2019).
- Skibbe, H., et al. PAT-probabilistic axon tracking for densely labeled neurons in large 3-D micrographs. IEEE Trans Med Imaging. 38 (1), 69-78 (2019).
- Economo, M. N., et al. A platform for brain-wide imaging and reconstruction of individual neurons. eLife. 5, e10566 (2016).
- Winnubst, J., et al. Reconstruction of 1,000 projection neurons reveals new cell types and organization of long-range connectivity in the mouse brain. Cell. 179 (1), 268-281.e13 (2019).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유