Method Article
Tomografía seriada de dos fotones de todo el cerebro del tití para análisis neuroanatómicos
En este artículo
Resumen
La tomografía de dos fotones en serie (STPT) es una técnica para obtener imágenes de una masa de tejido en su forma tridimensional mediante la combinación de imágenes de dos fotones con el control automático de la etapa y el corte de micrótomos. Aquí describimos un protocolo para implementarlo en cerebros de tití para comprender mejor sus características estructurales.
Resumen
La tomografía seriada de dos fotones (STPT) es una técnica para obtener imágenes de una masa de tejido en su forma tridimensional mediante la combinación de imágenes de dos fotones con el control automático de etapas y el corte de micrótomos. Implementamos con éxito esta técnica para trazar las proyecciones axonales en el cerebro del tití. Aquí, se describen los procedimientos experimentales detallados que dieron como resultado imágenes volumétricas confiables de todo el cerebro del tití. Un proceso clave para el éxito de las imágenes fue la extirpación de las meninges que rodean el cerebro, lo que interfiere con el corte. Una gran ventaja de esta metodología es que las secciones cortadas se pueden utilizar para tinciones adicionales. En la configuración original, las secciones cortadas en rodajas se mezclan en el baño de agua. Estas secciones se pueden alinear correctamente en su orden original de acuerdo con los patrones de los vasos sanguíneos en la corteza. Un ejemplo de histología eficaz es la visualización de la estructura de la mielina mediante una simple reflexión de la luz, que puede combinarse con la tinción de Nissl para definir los bordes anatómicos. Estas secciones también se pueden utilizar para la detección inmunológica de trazadores anterógrados y retrógrados no fluorescentes, que se pueden registrar en los datos STPT para la estratificación de múltiples datos.
Introducción
En los estudios neuroanatómicos, los investigadores se enfrentan a la necesidad de observar estructuras de orden micrométrico (por ejemplo, axones y botones) en el contexto de todo el cerebro. Por lo general, esta difícil tarea se ha abordado mediante la inspección visual de las secciones de serie y la búsqueda de la región de interés para obtener imágenes detalladas, análisis y registro fotográfico. Sin embargo, con el avance tecnológico, se está haciendo posible obtener imágenes de todo el cerebro en alta resolución para el análisis de todo el cerebro. En un estudio pionero realizado por Oh et al.1, cientos de cerebros de ratones recibieron inyecciones de trazadores para análisis conectómico, que se procesaron mediante la técnica de imagen de tomografía de dos fotones en serie (STPT)2. La característica de este estudio fue que seleccionaron trazadores anterógrados para cuantificar la "conectividad". Mientras que los trazadores anterógrados proporcionan información espacial muy detallada sobre la distribución axonal, los neuroanatomistas se han basado en una laboriosa segmentación manual para su análisis. Mediante la automatización de varios procedimientos analíticos, incluido este paso de segmentación, lograron la "producción en masa" de datos de trazadores de alta resolución listos para usar para usos múltiples. La efectividad de su enfoque es obvia, dada la variedad de estudios que utilizaron sus datos de trazador 3,4,5 y su cerebro estándar6.
Históricamente, la conectividad neuronal de los cerebros de los primates no humanos ha atraído la atención de muchos neuroanatomistas desde el desarrollo del método de degeneración en la década de 1950, pasando por los métodos de transporte de sustancias anterógrados/retrógrados en la década de 1970 hasta la estrategia viral actual 7,8. Como tal, existen vastas piezas de literatura que investigan las conexiones neuronales de los primates. En particular, muchos investigadores han investigado la compleja conectividad corticocortical del cerebro de los macacos, y sus resultados han sido seleccionados para la tabulación (por ejemplo, CoCoMac9). Aunque útiles, estos estudios clásicos tenían varias limitaciones. En primer lugar, debido a que cada estudio se enfoca solo en regiones cerebrales limitadas, la información obtenida inevitablemente se vuelve fragmentaria. En segundo lugar, cada estudio utiliza diferentes métodos y condiciones. Por lo tanto, la evaluación cuantitativa entre estudios se complica. En tercer lugar, la conectividad suele mostrarse como una cámara lúcida de secciones representativas o como una tabla/gráfico semicuantitativo para las regiones cerebrales definidas por el autor. En otras palabras, solo se extrae información muy limitada de una estructura cerebral compleja para su presentación en la literatura publicada. Con el desarrollo de las técnicas de imágenes por resonancia magnética (IRM), los estudios de todo el cerebro a baja resolución se hicieron prominentes. Sin embargo, existe una gran brecha entre los niveles de detalle entre lo que sabemos sobre las conexiones neuronales en ratones y las de los primates.
Con estos antecedentes, nos propusimos realizar un rastreo anterógrado exhaustivo de los cerebros de los titíes comunes10,11. Aunque mucho más pequeño y liso que el cerebro del macaco, el homólogo tití exhibe signos claros de primates, como la presencia de área MT y las áreas prefrontales granulares, ninguna de las cuales está claramente definida en los roedores12,13. Aquí, el pequeño tamaño era una gran ventaja porque incluso el cerebro de tití pesa diez veces más que el cerebro de ratón. Afortunadamente, pudimos obtener imágenes de todo el cerebro del tití con actualizaciones mínimas de la versión original del software operativo TissueCyte1000 (en adelante denominado sistema de imágenes de tejido completo), el microscopio disponible comercialmente para la obtención de imágenes STPT2. Esta actualización fue para permitir un movimiento adicional del escenario antes de cortar. La versión actual es ahora suficiente para procesar el cerebro del tití. Este artículo comparte un protocolo para manejar el cerebro del tití para la obtención de imágenes STPT. También se proporciona el protocolo de posimagen que mejora aún más la utilidad de este método.
Protocolo
Todos los procedimientos experimentales se llevaron a cabo siguiendo la Guía del Instituto Nacional de Salud para el Cuidado y Uso de Animales de Laboratorio (Publicaciones de los NIH Nº 80-23) revisada en 1996 y los "Principios rectores para el cuidado y uso de animales en el campo de la ciencia fisiológica" de la Sociedad Japonesa de Fisiología, y fueron aprobados por el Comité de Animales Experimentales de RIKEN (W2020-2-009(2)).
1. Inyección de trazadores
- Realizar inyecciones de trazadores fluorescentes y no fluorescentes en el cerebro del tití de acuerdo con los procedimientos previamente informados14.
- En cuanto a la combinación con el método STPT, asegúrese de que la fluorescencia de los trazadores sea lo suficientemente fuerte como para ser detectada sin mejora. Adopte un sistema TET-Off de doble vector10 para mejorar la expresión.
NOTA: Los trazadores no fluorescentes también se pueden inyectar y detectar después de realizar el STPT utilizando los cortes producidos. Ejemplos de trazadores no fluorescentes incluyen amina de dextrano biotinilada (BDA), AAV que expresa cre en AAV2 retro para la detección retrógrada de los núcleos de la célulade entrada 10, así como AAV de etiqueta smFP basados en GFP (ver más abajo).
- En cuanto a la combinación con el método STPT, asegúrese de que la fluorescencia de los trazadores sea lo suficientemente fuerte como para ser detectada sin mejora. Adopte un sistema TET-Off de doble vector10 para mejorar la expresión.
- Fijar la perfusión y obtener el cerebro de tití después de 4 semanas.
- Para anestesiar al tití, administrar Medetomidina (0,04 mg/kg), midazolam (0,4 mg/kg) y butorfanol (0,4 mg/kg), llamado MMB, por vía intramuscular, seguido de una inyección intraperitoneal de Tiopental Sódico (100 mg/kg).
- Después de confirmar que se ha perdido el reflejo del dolor, abra la cavidad torácica para exponer el corazón y corte la aurícula derecha para permitir que la sangre y el fijador salgan.
- A continuación, corte el ventrículo izquierdo para insertar la aguja de perfusión para lavar primero la sangre con la solución de prefijo (250 mM de sacarosa, 5 mM MgCl2 en tampón de fosfato 0,02 M [PB, pH 7,4]) durante unos minutos (~50-100 mL), seguido de la fijación con 4% de paraformaldehído/0,1 M de tampón de fosfato (PB; 2-3 veces el peso del animal durante ~20 min).
- Una buena perfusión es fundamental para un buen resultado. Al insertar la aguja de perfusión, acelere a lo largo del tabique hacia la aorta para asegurar un buen flujo del fijador. La punta de la aguja se hace visible cuando llega a la aorta. Retraiga hasta que la punta sea apenas visible para que no pase por alto la arteria carótida.
- Mantener el cerebro en paraformaldehído al 4%/0,1 M PB durante 48 h a 4 °C y transferir a 50 mM PB. Si no se usa inmediatamente, almacene el cerebro en 0,75% de glicina/0,1 M PB para evitar la autofluorescencia debido a la sobrefijación.
2. Preparación de la muestra (Figura 1)
- Incubar el cerebro fijo con colagenasa (1 mg/mL en 3 mM de CaCl,2 en 10 mL de solución salina tamponada tris [TBS]) a 37 °C durante 1 h.
NOTA: Precaliente el cerebro a 37 °C durante 5 minutos antes de la incubación. - Frote cuidadosamente la superficie del cerebro con un hisopo de algodón para despegar la materia de pia y otras meninges (Figura 1A, video complementario). Utilice pinzas finas para pelar las meninges desprendidas.
NOTA: Es importante extirpar las meninges que rodean el mesencéfalo, incluyendo el colículo superior o las que se encuentran en la parte superior del tálamo. Es difícil exponer estas estructuras que se esconden en lo más profundo de su interior. Sin embargo, elimine las meninges tanto como sea posible. De lo contrario, a menudo permanecen sin cortar, flotan hacia arriba e interfieren con la obtención de imágenes y el corte (Figura 2A). - Tenga especial cuidado cuando el cerebro se someta a una resonancia magnética ex vivo antes de la incorporación, durante la cual el cerebro se sumerge en una solución de flúor15. El flúor sobrante, si se incrusta, podría formar pequeñas burbujas de aire debajo del objetivo mientras se toman imágenes. Para evitar esto, mantenga el cerebro en PB durante 1 semana antes de incrustarlo.
NOTA: Este paso de espera generalmente no es necesario, pero es crítico cuando se combina con una resonancia magnética ex vivo . - Prepare el tampón de NaBH4 disolviendo 0,2 g de NaBH4 en 100 mL de tampón de borato de 50 mM (pH 9,2) calentado a 40 °C. Deje la tapa suelta para dejar salir el gas CO2 . Mantenga la solución toda la noche con la tapa aflojada. Envuelva la botella en papel de aluminio para protegerla de la luz hasta su uso.
- Hacer agarosa oxidada agitando 2,25 g de agarosa y 0,21 g de NaIO4 en 100 mL de PB durante 2-3 h. Filtrar la solución con aspiración al vacío y lavarla con tres cambios de PB. Vuelva a suspender la agarosa en 50 mL de PB.
- Derretir completamente la agarosa en un horno microondas y enfriar a 60-65 °C.
- Coloque el cerebro en una cámara hecha a medida e incruste el cerebro en agarosa. Tenga cuidado de introducir la agarosa en la cavidad debajo del cuerpo calloso (Figura 1B).
NOTA: Precaliente el cerebro primero a temperatura ambiente y luego a 65 °C durante 5 minutos para permitir que la agarosa se asiente en la superficie del cerebro sin solidificarse. - Desmontar la cámara y sumergir el bloque de agarosa en el tampón de NaBH4 durante la noche a 4 °C.
- Cambie el tampón a PB varias veces durante 1-2 semanas a 4 °C.
NOTA: La autofluorescencia tisular se vuelve muy débil si el intercambio de tampón es incompleto. - Haga una etapa de vidrio deslizante colocando cuatro imanes de neodimio con una mezcla instantánea de epoxi.
- Monte el bloque de agarosa en el escenario con un adhesivo fuerte (por ejemplo, superpegamento) (Figura 1C).
3. Procesamiento de tejidos
- Opere todo el sistema de imágenes de tejidos de acuerdo con las instrucciones del fabricante. También se publica un protocolo detallado16. El procedimiento actual se utiliza principalmente para TissueCyte1000, pero se puede aplicar a otros modelos, como TissueCyte 1600FC.
- El funcionamiento es ligeramente diferente entre los modelos, pero siga los puntos a continuación que suelen ser importantes.
- Todo el cerebro del tití se puede procesar coronalmente desde los extremos anterior a posterior sin ningún cambio en el hardware. Asegúrese de que no se excedan los límites de movimiento del escenario al colocar el cerebro.
- Ajuste el ángulo de las cuchillas para asegurarse de que las profundidades de la superficie en ambos extremos de la cuchilla estén dentro de los 10 μm. Debido a la mielinización densa, incluso una diferencia de profundidad de 10 μm podría provocar diferentes penetraciones láser a través de la sustancia blanca del tití adulto. Por la misma razón, establezca el plano de imagen a unos 25-35 μm de la superficie.
- Use una cuchilla de cerámica (recomendado) para cortar todo el cerebro a intervalos de 50 μm (~más de 650 rebanadas).
- Las meninges permanecen sin cortar e interfieren con el corte si no se retiran correctamente (Figura 2). Algunas meninges (por ejemplo, las que rodean el hipocampo o el pulvinar) son difíciles de extirpar. Extraiga manualmente estas meninges con pinzas finas cuando las note.
4. Técnicas histológicas auxiliares
- Recuperación de secciones
- Recuperar los cortes de tejido para realizar diversas tinciones histológicas. Alinee con precisión estas secciones en orden de corte. Traza los vasos sanguíneos a través de las capas corticales para alinearlos (Figura 3).
- Corta el exceso de agarosa de cada sección para un mejor manejo.
NOTA: Este proceso no tiene por qué ser excesivo. La agarosa sirve para mantener los segmentos de tejido en su lugar sin interferir con el procesamiento histológico. - Imágenes a contraluz
- Monta las secciones en un portaobjetos y sécalas.
- Vuelva a hidratar la sección con PBS y coloque el cubreobjetos para la obtención de imágenes.
- Observe un patrón de mielinización sin tinción cuando la sección se visualiza con un microscopio de fluorescencia (Figura 4) en un modo de campo claro. Ajuste el tiempo de exposición para que el reflejo de la luz pueda ser visible.
NOTA: Se puede utilizar cualquier microscopio si puede utilizar iluminación de campo oscuro. - Retire el cubreobjetos y proceda a la tinción de Nissl o cualquier otra mancha. Cualquier procedimiento de tinción estándar es suficiente.
- Utilice este método para comparar directamente diferentes patrones de tinción con los patrones de mielinización (Figura 4).
- Imágenes BDA
- Consulte la Tabla 1 para obtener un procedimiento detallado.
- Primero, trate las secciones con la solución de Dent (20% DMSO, 80% Metanol). El metanol incluido en la solución de Dent mejora drásticamente las señales de BDA en los axones.
- Utilice el método de amplificación de señal de tiramida (TSA) para mejorar la señal de BDA que se detectará fluorescentemente17.
NOTA: La solución de biotina TSA se puede comprar al proveedor, pero el reactivo interno es mucho más barato y efectivo. - Para registrarse en la imagen STPT, tome una imagen a contraluz después de montar la sección en un vidrio deslizante con el medio de montaje.
- Tinción de proteína fluorescente del monstruo de espagueti (smFP)
NOTA: Las proteínas fluorescentes del 'monstruo de espagueti' (smFPs) son una familia de variantes no fluorescentes de GFP con múltiples etiquetas de epítopos18. Proporcionan excelentes señales fluorescentes tras la inmunodetección y son adecuados como trazadores complementarios de los trazadores fluorescentes. Consulte la Tabla 2 para ver las construcciones AAV disponibles en Addgene.- Siga el procedimiento detallado que se proporciona en la Tabla 1.
5. Procesamiento de datos posterior a la obtención de imágenes
- Prueba de integridad de datos
NOTA: STPT genera un gran número de archivos de imagen. Cuando el cerebro de un tití adulto se secciona coronalmente a intervalos de 50 μm, normalmente se requieren más de 650 secciones para cubrir todo el cerebro. Los datos de cada sección consisten en una serie de imágenes de mosaico capturadas en tres canales. Estos archivos de imagen se almacenan en una sola carpeta junto con un archivo de metadatos que registra la posición de cada mosaico. Para minimizar la cantidad total de datos y el tiempo de procesamiento, la creación de imágenes se realiza en bloques de ejecuciones, durante los cuales el número de pasos X e Y para el mosaico cambia gradualmente. Por lo general, 20-30 bloques de carreras, cada uno compuesto por la imagen de 30-50 secciones, constituyen el conjunto de datos completo para un solo cerebro. Para utilizar estos datos, se requieren varios pasos de procesamiento de imágenes, incluida la unión de las imágenes en mosaico, la segmentación de señales fluorescentes y el registro de los datos volumétricos en una plantilla estándar, entre otros.- Realice los pasos de procesamiento de imágenes individualmente utilizando diversas herramientas de software o a través de tuberías dedicadas adaptadas a las necesidades específicas de los investigadores. Los detalles del procesamiento de imágenes están fuera del alcance de este trabajo y se pueden encontrar en otras publicaciones 11,19.
- Independientemente del procedimiento utilizado, asegúrese de que los datos estén organizados de forma coherente.
NOTA: Dado que todo el proceso de creación de imágenes implica muchos bloques de ejecuciones y abarca varios días, no es raro que una ejecución se cancele a mitad de camino y se reinicie de nuevo. En tales casos, la estructura de datos puede verse interrumpida, lo que dificulta la correcta ejecución de la canalización de procesamiento de imágenes. - Para minimizar el riesgo de interrupción de la estructura de datos, ejecute un script de Python para comprobar la integridad de la estructura de datos (kn_pipeline_check_mosaic.py) disponible en GitHub (github.com/watkarbey/STPT_depo).
- Registro de la imagen manchada en la imagen STPT
NOTA: La transformación no lineal de la imagen teñida a la imagen STPT se puede realizar utilizando el complemento bUnwarpJ de ImageJ.- Ajuste el tamaño de las imágenes de las imágenes de destino (STPT) y de las imágenes de origen (manchadas) para que sean aproximadamente iguales.
- Binariza cada imagen para visualizar las formas de las secciones.
- Ejecuta bUnwarpJ. Recuerde marcar la opción Guardar transformaciones .
- Inicie bUnwarpJ de nuevo. Seleccione la imagen original antes de la binarización como imagen de origen. A continuación, haga clic en Cargar transformación elástica y elija la transformación guardada.
Resultados
En la configuración típica utilizada aquí, se puede obtener una imagen de todo el cerebro de un tití adulto (Figura 5) con una resolución de ~1,3 x 1,3 μm/píxel con un intervalo de sección de 50 μm en aproximadamente 1 semana. Esto equivale a ~650 imágenes coronales en tres canales después de la unión de imágenes. Con un objetivo de 16x (Nikon 16xW CFI75 LWD; NA = 0,80), el campo de visión de un solo disparo es de aproximadamente 1 x 1 mm. La imagen de toda la superficie coronal se obtiene uniendo estas tomas (Figura 5A). La alineación en la dirección Z es excelente y se obtiene una buena imagen 3D simplemente apilando los datos de la imagen coronal (Figura 5B,E). Para la estandarización, la plantilla STPT para el cerebro de tití11 se puede utilizar para el registro 3D-3D (Figura 5F). Esta transformación de datos es uno de los aspectos clave de la neuroanatomía del cerebro completo, en la que a una región de interés se le asigna una coordenada de espacio absoluta independiente de la anotación anatómica. Una vez que la muestra de cerebro se registra en el espacio estándar, se pueden extraer fácilmente las subregiones de interés para su posterior análisis (Figura 5F, G). En particular, las regiones corticales se pueden transformar en un mapa plano utilizando un parámetro predeterminado (Figura 5H).
Las secciones generadas durante la toma de imágenes se pueden utilizar para diversos fines histológicos. Como se muestra en la Figura 4A, B, las imágenes retroiluminadas sin tinción proporcionan un patrón muy similar a la tinción de mielina auténtica. Esta puede ser una excelente alternativa a la tinción de mielina. Además, si la imagen retroiluminada se obtiene antes de la tinción de Nissl, se puede utilizar la misma sección para obtener los patrones de tinción tanto de mielina como de Nissl, proporcionando así información útil para identificar áreas y capas (Figura 5C-E). Estas secciones también se pueden utilizar para la tinción inmunológica. En la Figura 5J, la sección alrededor del centro de inyección se contratiñó con anticuerpo NeuN para estimar la eficiencia de transducción del virus AAV. También es una buena estrategia inyectar trazadores no fluorescentes además de los trazadores fluorescentes y detectarlos histológicamente después de la extracción de la sección. En nuestro estudio anterior, combinamos trazadores verdes anterógrados con vectores "cre" retrógrados, que luego fueron detectados por el anticuerpo anti-cre10. En un ejemplo de la Figura 6A-E, se inyectó BDA en el lado contralateral del trazador verde (trébol) y se detectó con fluorescencia. Tenga en cuenta que las señales rojas de BDA se pueden registrar en la imagen de TissueCyte para localizarlas en toda la coordenada del cerebro. En otro ejemplo de la Figura 6F-I, se inyectó smFP-myc en el área parietal (Figura 6G), mientras que el trazador verde se inyectó en el área frontal. De esta manera, se pueden inyectar múltiples trazadores en el mismo animal sin interferir con las imágenes. Una gran ventaja de usar las secciones STPT para tinción adicional es que la relación entre los trazadores fluorescentes y no fluorescentes se puede determinar para el mismo cerebro. De este modo, pudimos determinar la reciprocidad de las proyecciones corticocorticales con alta precisión10. Otra ventaja es que las coordenadas 3D de las secciones manchadas se pueden mapear a los datos STPT y luego a la plantilla estándar. Por lo tanto, puede que no sea necesario utilizar todas las secciones recuperadas para teñir. Para una mejor interpretación, se pueden seleccionar secciones para la tinción a fin de agregar más contexto a los datos STPT.
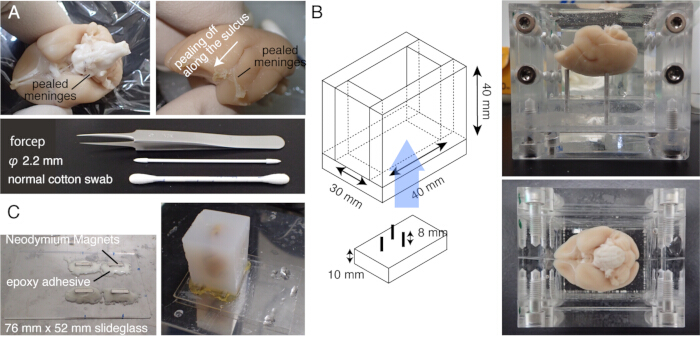
Figura 1: Preparación de la muestra para el STPT. (A) Eliminación de meninge con hisopos de algodón. Las meninges que rodean el tronco encefálico se pueden extirpar con pinzas de punta fina. Las meninges que rodean el cerebro del tití se extraen manualmente frotándolas con bastoncillos de algodón. La foto muestra las meninges peladas desde el interior del surco lateral. (B) La caja de acrílico utilizada para la incrustación de agarosa. Los pines son móviles y se utilizan para ajustar el ángulo del cerebro para que esté cerca de la posición fija estereotáxicamente20. (C) Un portaobjetos magnético hecho de vidrio deslizante de 76 mm x 52 mm y cuatro imanes de neodimio unidos por adhesivo epoxi. El bloque de agarosa se fija a la etapa magnética con superpegamento. Haga clic aquí para ver una versión más grande de esta figura.
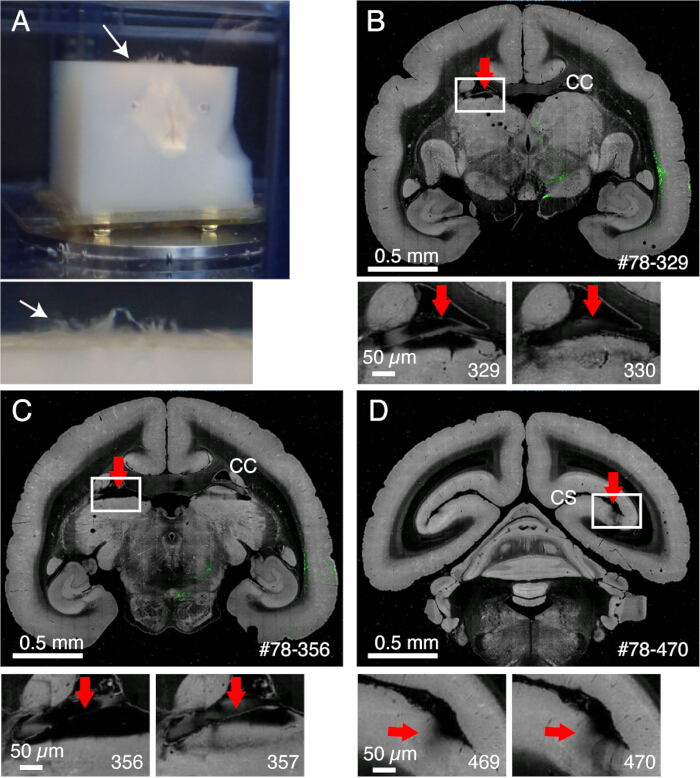
Figura 2: Efecto de las meninges en las imágenes. (A) Las meninges que permanecen sin cortar flotan hacia arriba, como lo muestra la flecha blanca. En este ejemplo, se observa una protuberancia extensa de las meninges porque no se eliminaron antes de la inclusión de la agarosa. Por lo general, las meninges permanecen sin cortar solo en varias regiones difíciles. (B) Un ejemplo de mal corte debido a la presencia de meninges entre el cuerpo calloso y la parte superior del tálamo. En este caso, un mal corte llevó a la alternancia de un corte profundo (329) y un corte relativamente normal (330). #78-329 representa la muestra n.º 78, sección n.º 329 en el portal de datos Brain/MINDS (C) Otro ejemplo de malas imágenes. En el peor de los casos, el núcleo pulvinar mostrado por la flecha roja puede desprenderse por completo. Barras de escala: 0,5 mm (panel superior), 0,5 μm (panel inferior). (D) Otro ejemplo de mala imagen. Las meninges profundas dentro del surco calcarino son difíciles de eliminar. Las sombras que se ven en las secciones 469 y 470 son causadas por las meninges flotantes que cayeron debajo del objetivo. Barras de escala: 0,5 mm (panel superior), 0,5 μm (panel inferior). Haga clic aquí para ver una versión más grande de esta figura.
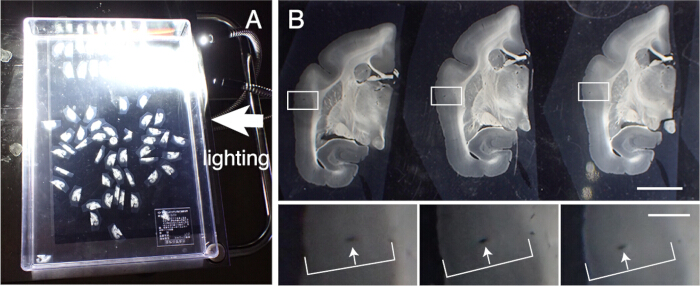
Figura 3: Alineación de las secciones de tejido en orden. (A) Hasta 50 secciones coronales de tití se pueden alinear correctamente en orden en un contenedor de plástico (31 cm x 22,5 cm). Para visualizar las estructuras detalladas, el contenedor se coloca sobre papel negro y se ilumina desde un lado. Estas secciones primero se alinean aproximadamente en orden y luego se someten a una alineación precisa. (B) La alineación precisa utiliza el vaso sanguíneo como marcador. La corteza cerebral contiene muchos vasos sanguíneos que atraviesan verticalmente las capas corticales. Se identifican como agujeros alargados que cambian sistemáticamente de posición dentro de las capas corticales (flechas blancas). Con este método, incluso las secciones con intervalos de 50 μm se pueden alinear con precisión. Estos orificios de los vasos sanguíneos se pueden identificar en las imágenes de la sección SPTP para su confirmación. Barra de escala: 5 mm (panel superior), 1 mm (panel inferior). Haga clic aquí para ver una versión más grande de esta figura.
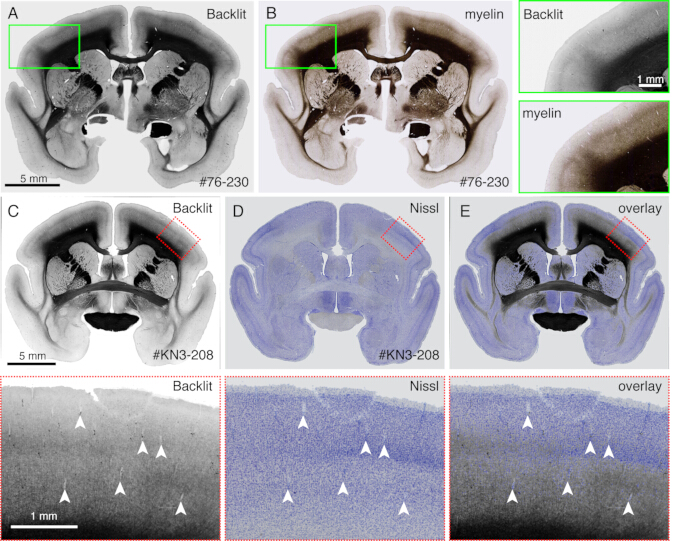
Figura 4: Imagen a contraluz como sustituto de la tinción de mielina. (A,B) La sección idéntica se utilizó para la imagen a contraluz y la tinción de mielina. En primer lugar, la sección se montó en el vidrio del portaobjetos, se secó, se rehidrató con PBS, se cubrió y se obtuvieron imágenes con un microscopio óptico (Tabla de materiales). Después de retirar el cubreobjetos, se utilizó la misma sección para la tinción de mielina21 y se obtuvieron imágenes con un microscopio de fluorescencia. La imagen retroiluminada se registró en la imagen de mielina utilizando el plugin bUnwarpJ de ImageJ. Los cuadros verdes muestran las vistas ampliadas de cada imagen. Tenga en cuenta que estas imágenes muestran patrones casi idénticos, excepto que la tinción de mielina visualiza mejor las estructuras fibrosas. (C-E) La sección idéntica se utilizó para la obtención de imágenes a contraluz y la tinción de Nissl. La máscara de umbral bajo para la imagen retroiluminada se registró por primera vez con la máscara de umbral bajo para la imagen de Nissl usando el complemento bUnwarpJ, y la imagen original se transformó usando el mismo parámetro. Tenga en cuenta que los vasos sanguíneos (puntas de flecha blancas) coinciden bien entre las dos imágenes. Debido a que estas dos imágenes son para la misma sección, la coincidencia es casi perfecta y se pueden comparar directamente los patrones de mielina y Nissl para la identificación de las capas corticales. Barras de escala: 5 mm (paneles A-E). Haga clic aquí para ver una versión más grande de esta figura.
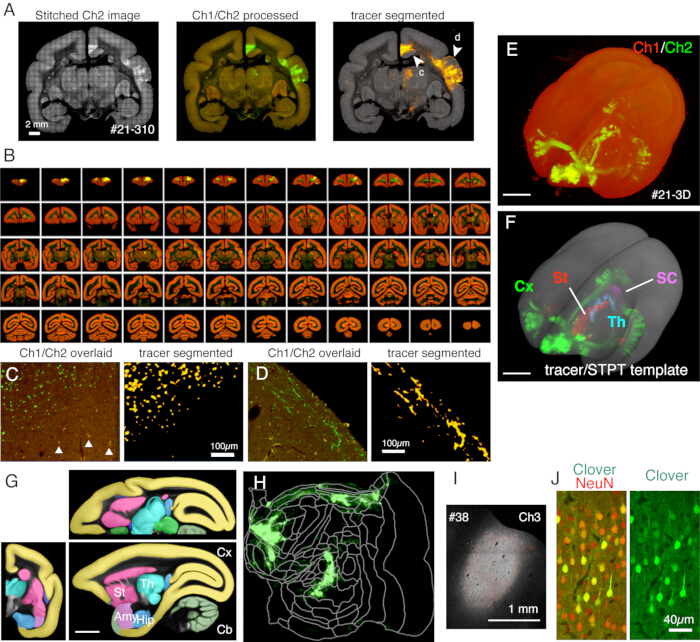
Figura 5: Resultado típico de las imágenes STPT. (A) Un mosaico simple de imágenes Ch2 (verde) de una sección de ejemplo (sección 310 de la muestra #21). Sin corrección de fondo, los bordes de cada mosaico son visibles. En la columna del medio, las baldosas se cosieron con corrección de fondo para Ch1 (rojo) y Ch2 (verde), respectivamente. Para reducir las señales de lipofuscina (ver panel C), las señales Ch1 se restaron de Ch2 y se mostraron en verde. Para reducir las señales de lipofuscina de Ch1, se utilizó el comando "Remove Outilers.." antes de coser por ImageJ. En la columna de la derecha, las señales trazadoras fueron segmentadas por la tubería de procesamiento de imágenes11. Las puntas de flecha (c, d) muestran las ubicaciones donde se muestran las vistas ampliadas en los paneles C y D. Barra de escala: 2 mm. (B) Descripción general de las secciones en serie para la muestra # 21. STPT generó 635 imágenes coronales de alta resolución para esta muestra. (C) Una vista de gran aumento mostrada por la punta de flecha c en el panel A. Se trata de una simple superposición de Ch1 (rojo) y Ch2 (verde) sin más procesamiento. Los triángulos muestran señales de fluorescencia de lipofuscina, que muestran un espectro amplio. El algoritmo de segmentación del trazador distingue con precisión las señales del trazador del fondo de lipofuscina a pesar de tener una forma muy similar (panel derecho). Barra de escala: 100 μm. (D) Otro ejemplo de una vista de gran aumento. Tenga en cuenta que las fibras finas de los axones en la capa 1 pueden ser bien visibles. Barra de escala: 100 μm. (E) La reconstrucción en 3D de imágenes STPT originales. Las 635 imágenes coronales de baja resolución que se muestran en el panel B se utilizaron como pila tiff para la visualización en 3D utilizando fluorandra22. Barra de escala: 5 mm. (F) La reconstrucción en 3D de las señales trazadoras segmentadas registradas en la plantilla STPT (gris). Las señales trazadoras en diferentes regiones del cerebro se mostraron con diferentes colores. Estas regiones se recortaron utilizando la anotación que se muestra en el panel G. Barra de escala: 5 mm. (G) Plantilla STPT superpuesta con anotación de diferentes regiones cerebrales. Barra de escala: 5 mm. (H) La señal trazadora cortical que se muestra en el panel F se mostró en forma de mapa plano. (I) Identificación del lugar de inyección por la fluorescencia de Ch3, que es menos sensible a la fluorescencia del trazador y permanece insaturada. Barra de escala: 1 mm. (J) La tinción de la sección alrededor del centro de inyección con el anticuerpo NeuN mostró que aproximadamente el 30 % de las neuronas muestran una fuerte expresión de fluorescencia verde de trébol. Barra de escala: 40 μm. Abreviaturas: Cx; corteza, St; cuerpo estriado, Th; tálamo, SC; Colículo superior. Amy; amígdala, cadera; hipocampo, Cb; cerebelo. Haga clic aquí para ver una versión más grande de esta figura.
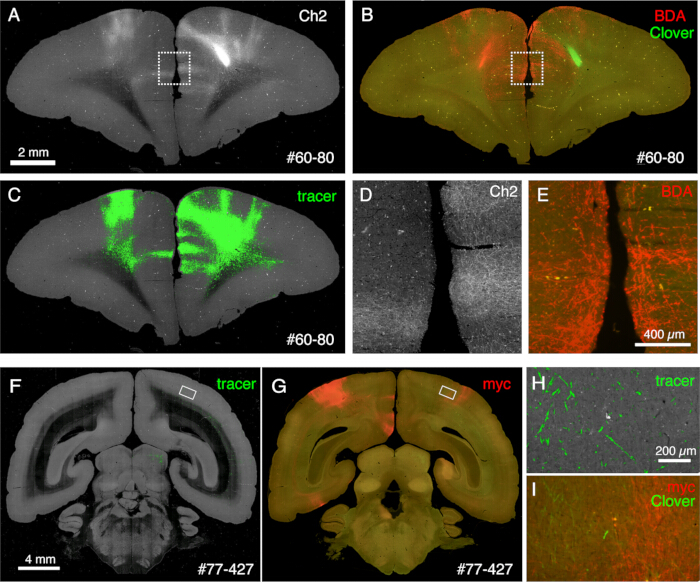
Figura 6: Histología post-STPT que muestra múltiples trazadores no fluorescentes. (A-C) Comparación de la imagen STPT con la imagen teñida con BDA. En esta muestra, se inyecta BDA en el lado contralateral de la inyección de Trébol. Los paneles A y C muestran la imagen Ch2 y la segmentación del trazador de los datos STPT. El panel B muestra la tinción posterior a STPT para BDA. La fluorescencia del trébol disminuye debido al tratamiento con metanol de la sección. Barra de escala: 2 mm. (D,E) Los cuadros punteados de los paneles A y B están ampliados. Barra de escala: 100 μm. (F,G) Comparación de la imagen STPT con la tinción de anticuerpos anti-myc tag. En esta muestra, la inyección de trébol se realiza en el PFC, mientras que el AAV-smFP-myc se inyecta en la corteza parietal contralateral. Los rectángulos blancos se amplían en los paneles H e I. Barra de escala: 4 mm. (H) La segmentación del trazador se muestra en verde. Barra de escala: 200 μm. (I) La tinción myc se muestra en rojo. La fluorescencia del trébol se muestra en verde. Barra de escala: 200 μm. Las señales verdes en los paneles H e I están presentes en una posición similar pero no idénticas porque STPT recupera solo una sección óptica de ~ 10 μm. Haga clic aquí para ver una versión más grande de esta figura.
| Protocolo de tinción fluorescente BDA | |
| Eliminar la agarosa | |
| Lavado TBS | 10 minutos (2x) |
| 1% H2O2 en la solución de Dent | 10 minutos |
| Lavado TBS | Breve |
| 0.5% de bloqueo de TNB | 1 h |
| StAvHRP (1:4000) en TNB | 2 noches |
| Lavado con TNT | 10 minutos (3x) |
| TSA Biotina (1:4000) en borato 0,1 M (pH8,5) + 0,003% H2O2 | 2 h |
| Lavado con TNT | 10 minutos (3x) |
| Cy3-estreptavidina (1:1000) en TNT | 3 h |
| Lavado con TNT | 10 minutos (2x) |
| Lavado TBS | Mantenga la sección hasta el montaje |
| Monte la sección en el vidrio deslizante utilizando un medio de montaje antidecoloración | |
| Protocolo de tinción fluorescente anti-MYC | |
| Eliminar la agarosa | |
| Lavado TBS | 10 minutos (2x) |
| Bloqueo en IB | 1 h |
| Anti-Myc (1:4000) en IB | 2 durante la noche |
| Lavado con TNT | 10 minutos (3x) |
| Anti-ratón Cy3 (1:1000) en TNT | 3 h |
| Lavado con TNT | 10 minutos (2x) |
| Lavado TBS | Mantenga la sección hasta el montaje |
| Monte la sección en el vidrio deslizante utilizando un medio de montaje antidecoloración | |
| Tampones/soluciones | Composición |
| 0.5% TNB | 0.5% Reactivo de bloqueo TSA en TS7.5 |
| La solución de Dent | 20% DMSO, 80% metanol |
| Tampón de inmersión (IB) | 10% FBS, 2% BSA 0.5% TritonX100 en TBS |
| TBS (solución salina tamponada con Tris) | 25 mM Tris, 137 mM NaCl, 2,7 mM KCl (pH 7,4) |
| TNT | 0.05 % Tween20 en TS7.5 |
| TS7.5 | 0,1 m TRIS-HCl, pH 7,5, 0,15 m NaCl |
Tabla 1: Protocolo de tinción fluorescente BDA y anti-myc fluorescente
| Plásmido para trazador AAV | Addgene No. | Anticuerpo recomendado | Dilución sugerida | Resultado esperado |
| pAAV-EF1_Cre | 201198 | Clon de Millipore 2D8 | 1:1000 | Bueno para las células |
| pAAVCam1.3_smFP_Myc | 201205 | MBL M192 My3 (ratón) | 1:4000 | Excelente |
| pAAVCam1.3_smFP_HA | 201206 | CST C29F4 (conejo) | 1:1000 | Bueno para las células |
| pAAVCam1.3_smFP_FLAG | 201207 | MBL PM020B (conejo) | 1:1000 | De acuerdo |
| AAVTRE3_smFP_Myc | 201208 | |||
| AAVTRE3_smFP_HA | 201209 | |||
| AAVTRE3_smFP_FLAG | 201210 |
Tabla 2: Lista de plásmidos Addgene disponibles para la producción de trazadores no fluorescentes. La construcción cre se dirige al núcleo y es adecuada para el trazado retrógrado envuelto por AAV2 retro. smFP_HA constructo es bueno para la detección de cuerpos celulares (y retrógrado).
Video complementario: Vista microscópica de la extracción de meninges. Haga clic aquí para descargar este video.
Discusión
En este artículo se explican las soluciones prácticas para el manejo de cerebros de tití para el procesamiento de todo el cerebro, así como las técnicas histológicas auxiliares que mejoran la utilidad de la técnica STPT. El punto fuerte de la "neuroanatomía del cerebro completo" utilizando STPT es que se pueden obtener las coordenadas 3D de cualquier región de interés, ya sea anatómicamente anotada o no. Mediante el registro de 3D a 3D de alta precisión, es posible transformar estas coordenadas en una plantilla estándar para la superposición de múltiples conjuntos de datos. De esta manera, la plantilla estándar sirve como medio de integración de datos. Este fue un aspecto esencial de nuestro proyecto de mapeo de la corteza prefrontal (PFC)10, donde se analizaron los datos obtenidos de muchos individuos. Además, la información asignada a la plantilla estándar se puede comparar con varios datos que ya se han mapeado, ya sea Nissl, patrones de mielina, datos de trazadores, datos de resonancia magnética (incluida la resonancia magnética de difusión) o anotaciones anatómicas11. Y lo que es más importante, también se puede comparar con los datos futuros que se obtendrán con la tecnología que aún está por surgir. Actualmente existen múltiples plantillas para el cerebro del tití, que se basan en la tinción23 de Nissl, la resonancia magnética 23,24,25,26,27,28 y STPT11. Pero se pueden transformar a las coordenadas de cada uno en función de los contrastes de la imagen y los parámetros precalculados11. La integración de datos a escala de todo el cerebro en todos los estudios contribuye a una mejor comprensión del cerebro como sistema. El requisito previo para que esta estrategia funcione es la adquisición confiable de datos en todo el cerebro. A continuación, se analizan los pasos críticos y los posibles problemas asociados con el protocolo actual.
Uno de los procesos más vulnerables de las imágenes STPT es el corte de tejido. Como se mencionó anteriormente, las meninges a menudo permanecen sin cortar y pueden interferir con el corte. En particular, el núcleo pulvinar y el colículo superior son las dos regiones cerebrales más afectadas: dependiendo de la minuciosidad de la extracción de la meninge y de la incrustación en la agarosa, pueden desprenderse del bloque de tejido durante el corte. Estas regiones profundas son difíciles de abordar desde el exterior y pueden romperse fácilmente durante la extracción de la meninge. Una extirpación cuidadosa pero completa de la meninge es fundamental para el éxito de la obtención de imágenes. Otra preocupación es el retroceso de las secciones cortadas en el bloque de tejido, que a veces ocurre cuando las secciones permanecen unidas incluso después de cortarlas. Se puede minimizar dando forma al bloque de modo que la hoja corte oblicuamente en el extremo.
Al obtener imágenes profundas en el bloque de tejido, STPT evita la irregularidad de su superficie. Mientras que las señales fluorescentes pueden pasar a través de la región cortical con bastante facilidad, están muy disminuidas en las regiones mielinizadas. Por lo tanto, la profundidad de la imagen debe determinarse cuidadosamente para equilibrar la imagen consistente en toda la superficie del bloque y el brillo de las señales en la región rica en mielina, como la materia blanca. En la configuración utilizada aquí, generalmente apuntamos a 25-35 μm de la superficie. También debe tenerse en cuenta que la superficie de corte puede encogerse de manera desigual después de largas horas de almacenamiento. Para minimizar el área de imagen, dividimos la sesión de imagen en 20-30 carreras con diferentes configuraciones de etapa durante 5-6 días. Hacemos el intervalo entre carreras menos de 2 h o confirmamos la profundidad de la superficie y ajustamos la altura de la etapa antes de cada carrera.
En este protocolo, la señal BDA fue amplificada por el método TSA. Este método es muy eficaz y puede detectar señales de BDA transportadas anterógradamente incluso a una resolución relativamente baja (por ejemplo, Figura 6B). La biotina TSA está disponible comercialmente en Akoya Biosciences, pero la solución casera muestra una mejora mucho mejor. Por otro lado, la dilución del anticuerpo y la solución requiere un ajuste cuidadoso para obtener el resultado óptimo. El tratamiento previo de la sección con una solución de metanol es fundamental. Sin tratamiento previo, las señales de BDA apenas se detectan en los axones mielinizados.
Cuando se utiliza un trazador anterógrado, a menudo es difícil identificar el sitio exacto de la inyección debido a la saturación de las señales de fluorescencia. En toda la configuración del sistema de imágenes de tejidos utilizada en este estudio, utilizamos el canal azul para identificar las células infectadas (Figura 5I). Incluso cuando los canales rojo y verde están saturados, cada neurona infectada individual suele ser detectable en el canal azul. Esto también es cierto con el Allen Mouse Brain Connectivity Atlas1. El examen de las células de origen es importante porque la infección a veces afecta solo a capas particulares. Encontramos este tipo de infecciones parciales con bastante frecuencia en el Atlas de Conectividad Cerebral del Ratón, tal vez debido a la utilización del método de iontoforesis29. La propagación lateral de los trazadores virales podría ser más o menos variable en función de las inyecciones. Esta variabilidad puede afectar potencialmente al resultado del rastreo y necesita una normalización cuidadosa.
El registro exitoso de la imagen 3D obtenida en la plantilla estándar es un proceso clave de toda la neuroanatomía del cerebro. El registro de la imagen STPT en la plantilla STPT puede ser bastante preciso, y solo observamos desviaciones de unos pocos vóxeles (50 μm isóculos) para los bordes con un alto contraste de imagen10. Aún así, hay un límite a lo que puede hacer el registro. Debido al proceso de extirpación de la meninge, las muestras de STPT generalmente tienen espacios entre los hemisferios y entre la corteza y el mesencéfalo/cerebro posterior, mientras que los tejidos cerebrales están muy apretados en las imágenes de resonancia magnética in vivo . Tales diferencias son difíciles de ajustar mediante el registro. La corteza del tití está en su mayoría desprovista de surcos, pero el surco intraparietal es muy profundo en algunos individuos. Dichos surcos se perderán (se produce una fusión de arriba a arriba) al registrarse. Aunque el registro es una técnica poderosa, es necesario volver a los datos brutos para confirmar el resultado obtenido.
La generación masiva de datos de imágenes es tanto una fortaleza como una limitación de esta técnica. Si bien mejora la integridad del conjunto de datos, requiere una gestión cuidadosa de los datos adquiridos y el desarrollo de una canalización de procesamiento de imágenes automatizada para una interpretación eficiente de los datos. En el futuro, la aplicación de la inteligencia artificial generativa (IA) en la construcción de tuberías de procesamiento de imágenes puede simplificar significativamente este proceso. También se han realizado imágenes sistemáticas de todo el cerebro utilizando métodos basados en escáner de portaobjetos21,30. En comparación con estos métodos, STPT no requiere cálculos adicionales para la reconstrucción 3D. Combinado con el corte óptico, hemos demostrado que STPT tiene el potencial de reconstruir incluso segmentos de axones a través de las secciones31. Con la combinación adicional de técnicas de depuración de tejidos, Economo et al. desarrollaron un método para obtener imágenes de la totalidad de las neuronas escasamente marcadas32,33. Las últimas versiones de TissueCyte ahora ofrecen opciones para un láser adicional para mejorar la excitación de proteínas fluorescentes rojas o una unidad de captura de secciones para la recuperación automática de secciones. Con estos avances, el enfoque de todo el cerebro será más eficiente, proporcionando una base para una comprensión integral del cerebro de los primates, incluido el de los humanos.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Agradecemos profundamente al personal técnico del laboratorio y las instalaciones para animales de Yamamori (RRD) por su ayuda. Agradecemos al Centro de Colaboración CBS-Olympus de RIKEN por la asistencia técnica con la adquisición de imágenes confocal. Este trabajo fue apoyado por el programa de Investigación Científica en Áreas Innovadoras (subvención número 22123009) del MEXT, Japón, por Brain/MINDS y Brain/MINDS2.0 de AMED, Japón (JP15dm0207001, JP23wm0625001 y JP24wm0625218), y por la subvención JSPS KAKENHI número 24K09678 a A.W.
Materiales
| Name | Company | Catalog Number | Comments |
| Agarose type I | Sigma | A6013 | |
| All-in-one fluorescence microscope | Keyence | BZ-X series | This study used BZ-X710 |
| anti-mouse Cy3 | Jackson Immuno Research Laboratories, Inc. | 115-165-003 | |
| anti-myc antibody | MBL | M192 | mouse monoclonal |
| Butorphanol | Meiji Animal Health | Vetorphale | |
| Ceramic blade | TissueVision | N/A | |
| Collagenase type I | Wako Chemical | #031-17601 | Aliquot into 100 µL x 10 of 100 mg/mL in PBS |
| Cotton swab (HUBY-340) | HUBY | BB-013SP | micro cotton swabs |
| Custom-made chamber | N/A | N/A | The chamber consist of four side plates and one bottom plates, all made of 1 cm thick acrylic plates, which can be securely fastened together using screw bolts. An additional acrylic plate is placed inside the chamber, with three metal poles inserted through it to hold the marmoset brain in an inverted position. |
| Cy3-Streptavidin | Jackson Immuno Research Laboratories, Inc. | 016-160-084 | |
| Dumont #5 forceps, Inox | N/A | N/A | |
| Epoxy instant mix | Locktite | N/A | |
| FIJI | NIH | https://imagej.net/software/fiji/downloads | |
| kn_pipeline_check_mosaic.py | N/A | N/A | github.com/watkarbey/STPT_depo; This is a checking script for TissueCyte data. |
| Medetomidine | Zenoaq | Domitor | |
| Midazolam | Sandoz | N/A | |
| NaBH4 | Sigma | #452882 | |
| NaIO4 | Sigma | S1878 | |
| Perfusion needle | Natsume Seisakusho Co., Ltd | KN-348, 20G-50 | |
| Slideglass (76 mm x 52 mm) | Matsunami | S9111 | |
| StAvHRP | Jackson Immuno Research Laboratories, Inc. | 016-030-084 | HRP-streptavidin (titration needs to be carefully determined) |
| TissueCyte1000 | TissueVision | https://www.tissuevision.com/tissuecyte | |
| TSA blocking reagent | Kiko Tech | FP1012 | Refer to TSA-biotin kit (Akoya Biosciences) |
| TSA-biotin | House-made | N/A | See Okamoto et al (ref. 16) |
| VECTASHIELD HardSet Mounting Medium | Vector laboratories Inc | N/A | Antifade mounting medium |
Referencias
- Oh, S. W., Harris, J. A., Ng, L., Winslow, B., Cain, N., et al. A mesoscale connectome of the mouse brain. Nature. 508 (7495), 207-214 (2014).
- Ragan, T., et al. Serial two-photon tomography for automated ex vivo mouse brain imaging. Nat Methods. 9 (3), 255-258 (2012).
- Liang, Z., Arefin, T. M., Lee, C. H., Zhang, J. Using mesoscopic tract-tracing data to guide the estimation of fiber orientation distributions in the mouse brain from diffusion MRI. Neuroimage. 270, 119999 (2023).
- Watakabe, A., Hirokawa, J. Cortical networks of the mouse brain elaborate within the gray matter. Brain Struct Funct. 223 (8), 3633-3652 (2018).
- Ypma, R. J. F., Bullmore, E. T. Statistical analysis of tract-tracing experiments demonstrates a dense, complex cortical network in the mouse. PLoS Comput Biol. 12 (9), e1005104 (2016).
- Wang, Q., et al. The Allen mouse brain common coordinate framework: A 3D reference atlas. Cell. 181 (4), 936-953.e20 (2020).
- Köbbert, C., Apps, R., Bechmann, I., Lanciego, J. L., Mey, J., Thanos, S. Current concepts in neuroanatomical tracing. Prog Neurobiol. 62 (4), 327-351 (2000).
- Lanciego, J. L., Wouterlood, F. G. Neuroanatomical tract-tracing techniques that did go viral. Brain Struct Funct. 225 (4), 1193-1224 (2020).
- Bakker, R., Wachtler, T., Diesmann, M. CoCoMac 2.0 and the future of tract-tracing databases. Front Neuroinform. 6, (2012).
- Watakabe, A., et al. Local and long-distance organization of prefrontal cortex circuits in the marmoset brain. Neuron. 111 (14), 2258-2273.e10 (2023).
- Skibbe, H., et al. The Brain/MINDS marmoset connectivity resource: An open-access platform for cellular-level tracing and tractography in the primate brain. PLoS Biol. 21 (6), e3002158 (2023).
- Mitchell, J. F., Leopold, D. A. The marmoset monkey as a model for visual neuroscience. Neurosci Res. 93, 20-46 (2015).
- Okano, H. Current status of and perspectives on the application of marmosets in neurobiology. Annu Rev Neurosci. 44, 27-48 (2021).
- Watakabe, A., et al. Comparative analyses of adeno-associated viral vector serotypes 1, 2, 5, 8 and 9 in marmoset, mouse and macaque cerebral cortex. Neurosci Res. 93, 144-157 (2015).
- Hata, J., et al. Multi-modal brain magnetic resonance imaging database covering marmosets with a wide age range. Sci Data. 10, 221 (2023).
- Liwang, J. K., Bennett, H. C., Pi, H. -. J., Kim, Y. Protocol for using serial two-photon tomography to map cell types and cerebrovasculature at single-cell resolution in the whole adult mouse brain. STAR Protoc. 4 (1), 102048 (2023).
- Okamoto, S., et al. Exclusive labeling of direct and indirect pathway neurons in the mouse neostriatum by an adeno-associated virus vector with Cre/lox system. STAR Protoc. 2 (1), 100230 (2021).
- Viswanathan, S., et al. High-performance probes for light and electron microscopy. Nat Methods. 12 (6), 568-576 (2015).
- Kuan, L., et al. Neuroinformatics of the Allen mouse brain connectivity atlas. Methods. 73, 4-17 (2015).
- Paxinos, G., Watson, C., Petrides, M., Rosa, M., Tokuno, H. . The Marmoset Brain in Stereotaxic Coordinates. , (2012).
- Abe, H., et al. 3D reconstruction of brain section images for creating axonal projection maps in marmosets. J Neurosci Methods. 286, 102-113 (2017).
- Wan, Y., et al. FluoRender: Joint freehand segmentation and visualization for many-channel fluorescence data analysis. BMC Bioinformatics. 18 (1), 280 (2017).
- Majka, P., et al. Towards a comprehensive atlas of cortical connections in a primate brain: Mapping tracer injection studies of the common marmoset into a reference digital template. J Comp Neurol. 524 (11), 2161-2181 (2016).
- Liu, C., Yen, C. C. -. C., Szczupak, D., Tian, X., Glen, D., Silva, A. C. Marmoset brain mapping V3: Population multi-modal standard volumetric and surface-based templates. Neuroimage. 226, 117620 (2021).
- Liu, C., et al. A digital 3D atlas of the marmoset brain based on multi-modal MRI. Neuroimage. 169, 106-116 (2018).
- Liu, C., et al. A resource for the detailed 3D mapping of white matter pathways in the marmoset brain. Nat Neurosci. 23 (2), 271-280 (2020).
- Woodward, A., et al. The Brain/MINDS 3D digital marmoset brain atlas. Sci Data. 5, 180009 (2018).
- Saleem, K. S., Avram, A. V., Glen, D., Schram, V., Basser, P. J. The subcortical atlas of the marmoset ("SAM") monkey based on high-resolution MRI and histology. Cereb Cortex. 34 (4), bhae120 (2024).
- Watakabe, A., Hirokawa, J. Cortical networks of the mouse brain elaborate within the gray matter. Brain Struct Funct. 223 (8), 3633-3652 (2018).
- Lin, M. K., et al. A high-throughput neurohistological pipeline for brain-wide mesoscale connectivity mapping of the common marmoset. eLife. 8, e40042 (2019).
- Skibbe, H., et al. PAT-probabilistic axon tracking for densely labeled neurons in large 3-D micrographs. IEEE Trans Med Imaging. 38 (1), 69-78 (2019).
- Economo, M. N., et al. A platform for brain-wide imaging and reconstruction of individual neurons. eLife. 5, e10566 (2016).
- Winnubst, J., et al. Reconstruction of 1,000 projection neurons reveals new cell types and organization of long-range connectivity in the mouse brain. Cell. 179 (1), 268-281.e13 (2019).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados