Method Article
神経解剖学的解析のためのマーモセット脳全体の連続2光子断層撮影法
要約
シリアル2光子断層撮影法(STPT)イメージングは、2光子イメージングと自動ステージ制御およびミクロトームスライスを組み合わせて、組織の塊を3次元形状でイメージングする技術です。ここでは、マーモセットの脳がその構造的特徴をよりよく理解するために、それを実装するためのプロトコルについて説明します。
要約
シリアル2光子断層撮影法(STPT)は、2光子イメージングと自動ステージ制御およびミクロトームスライスを組み合わせて、組織の塊を3次元形状でイメージングする技術です。私たちは、マーモセット脳の軸索突起をトレースするために、この技術を成功裏に実装しました。ここでは、マーモセットの脳全体の信頼性の高い体積イメージングにつながった詳細な実験手順について説明します。イメージングを成功させるための重要なプロセスは、スライスの妨げとなる脳の周囲の髄膜の除去でした。この方法の大きな利点は、スライスした切片を追加の染色に使用できることです。元のセットアップでは、スライスされたセクションはウォーターバスでスクランブルされます。これらの切片は、皮質の血管パターンに従って、元の順序で正しく位置合わせすることができます。効果的な組織学の一例は、単純な光反射によるミエリン構造の可視化であり、これをニッスル染色と組み合わせて解剖学的境界を定義することができます。これらの切片は、非蛍光性の前向性トレーサーおよび逆行性トレーサーの免疫学的検出にも使用でき、STPTデータに登録して複数のデータを階層化することができます。
概要
神経解剖学的研究では、研究者は脳全体の文脈でマイクロメートルオーダーの構造(軸索やブートンなど)を観察する必要性に直面しています。この困難な作業は、通常、シリアルセクションを目視検査し、詳細なイメージング、分析、写真記録のための関心領域を探すことによって取り組まれてきました。しかし、技術の進歩により、脳全体を高解像度で画像化し、全脳を解析することが可能になりつつあります。Ohらによる画期的な研究1では、数百のマウス脳にコネクトーム解析用のトレーサー注射を照射し、これを連続2光子断層撮影法(STPT)イメージング技術2で処理しました。この研究の特徴は、「接続性」を定量化するために前向性トレーサーを選択したことです。前向性トレーサーは軸索分布に関する非常に詳細な空間情報を提供しますが、神経解剖学者はその分析のために面倒な手動セグメンテーションに依存してきました。このセグメンテーションステップを含むさまざまな分析手順を自動化することで、多目的に使える高分解能トレーサーデータの「量産」に成功しました。彼らのアプローチの有効性は、トレーサーデータ3,4,5と標準的な脳6を使用したさまざまな研究を考えると明らかです。
歴史的に、1950年代の変性法の開発から、1970年代の前行性/逆行性物質輸送法、そして現在のウイルス戦略まで、非ヒト霊長類の脳の神経接続性は多くの神経解剖学者の注目を集めてきました7,8。そのため、霊長類の神経接続を調査する膨大な文献が存在します。特に、多くの研究者がマカクザルの脳の複雑な皮質結合性を調査しており、その結果は集計用にキュレーションされています(例:CoCoMac9)。これらの古典的な研究は有用ではありましたが、いくつかの制限がありました。まず、各研究は限られた脳の領域のみに焦点を当てているため、得られる情報は必然的に断片的になります。第二に、各研究は異なる方法と条件を使用します。そのため、研究をまたいで定量的な評価が複雑化します。第三に、接続性は通常、代表的なセクションのカメラルシーダとして、または著者が定義した脳領域の半定量的な表/グラフとして示されます。言い換えれば、複雑な脳構造から非常に限られた情報のみが抽出され、公開された文献に提示されます。磁気共鳴画像法(MRI)技術の発展により、低解像度での全脳研究が目立つようになりました。しかし、マウスの神経接続について私たちが知っていることと霊長類の神経接続について私たちが知っていることとの間には、詳細レベルの間に大きなギャップがあります。
このような背景を念頭に置いて、私たちは一般的なマーモセット脳の包括的な順行性トレースの実行に着手しました10,11。マカクザルの脳よりもはるかに小さく滑らかですが、マーモセットの対応物は、MT領域や顆粒状の前頭前野の存在など、霊長類の明確な兆候を示しますが、どちらもげっ歯類では明確に定義されていません12,13。ここでは、マーモセットの脳でさえマウスの脳の10倍の重さがあるため、サイズが小さいことが大きな利点でした。幸いなことに、STPTイメージング2用の市販顕微鏡であるTissueCyte1000(以下、全組織イメージングシステム)のオリジナルバージョン(以下、全組織イメージングシステム)のアップデートを最小限に抑えながら、マーモセットの脳全体をイメージングすることができました。このアップデートは、スライスする前にステージをさらに動かすことができるようにするためのものでした。現在のバージョンでは、マーモセットの脳を処理するのに十分です。この記事では、STPTイメージングのためにマーモセット脳を処理するためのプロトコルを共有しています。この方法の有用性をさらに高めるポストイメージングプロトコルも提供される。
プロトコル
すべての実験手順は、1996年に改訂された国立衛生研究所実験動物の管理と使用に関するガイド(NIH Publications No. 80-23)および日本生理学会の「生理科学分野における動物の管理と使用に関する指導原則」に従って行われ、理化学研究所実験動物委員会(W2020-2-009(2))で承認されました。
1.トレーサーインジェクション
- 以前に報告された手順に従って、マーモセット脳に蛍光および非蛍光トレーサーの注射を行う14。
- STPT法との組み合わせについては、トレーサーの蛍光が増強されずに検出できるほど強いことを確認してください。ダブルベクターTET-Offシステム10 を採用し、表現力を高めます。
注:非蛍光トレーサーは、STPTが完了した後に、生成されたスライスを使用して注入および検出することもできます。非蛍光トレーサーの例としては、ビオチン化デキストランアミン(BDA)、インプット細胞核10の逆行性検出のためのAAV2レトロ中のcre発現AAV、ならびにGFPベースのsmFPタグAAV(下記参照)が挙げられる。
- STPT法との組み合わせについては、トレーサーの蛍光が増強されずに検出できるほど強いことを確認してください。ダブルベクターTET-Offシステム10 を採用し、表現力を高めます。
- 灌流固定を行い、4週間後にマーモセット脳を取得します。
- マーモセットを麻酔するには、メデトミジン(0.04 mg / kg)、ミダゾラム(0.4 mg / kg)、MMBと呼ばれるブトルファノール(0.4 mg / kg)を筋肉内投与した後、チオペンタールナトリウム(100 mg / kg)を腹腔内注射します。.
- 疼痛反射が失われたことを確認した後、胸腔を切開して心臓を露出させ、右心房を切開して血液と固定液を排出します。
- 次に、左心室を切断して灌流針を挿入し、最初に接頭辞溶液(250 mMスクロース、5 mM MgCl2 0.02 Mリン酸緩衝液[PB、pH 7.4])で血液を数分間(~50-100 mL)洗い流し、続いて4%パラホルムアルデヒド/0.1 Mリン酸緩衝液(PB;動物の体重の2〜3倍~20分)で固定します。
- 良好な結果を得るためには、良好な灌流が重要です。灌流針を挿入するときは、固定剤の流れを良好に保つために、中隔に沿って大動脈に向かって進めます。針の先端は大動脈に達すると見えます。先端がかろうじて見えるまで引っ込めて、頸動脈を迂回しないようにします。
- 脳を4%パラホルムアルデヒド/0.1 M PBに4°Cで48時間保持し、50 mM PBに移します。すぐに使用しない場合は、脳を0.75%グリシン/0.1 M PBで保存し、過剰固定による自家蛍光を防ぎます。
2. サンプル調製(図1)
- 固定脳をコラゲナーゼ(3 mM CaCl 1 mg/mL、3 mM CaCl2 、10 mL トリス緩衝生理食塩水[TBS])と37°Cで1時間インキュベートします。
注:インキュベーション前に脳を37°Cで5分間温めてください。 - 綿棒で脳表面を丁寧にこすり、軟膜物質やその他の髄膜を剥がします(図1A、 補足ビデオ)。細い鉗子を使用して、剥離した髄膜を剥がします。
注:上丘や視床の上部にある髄膜など、中脳を取り巻く髄膜を取り除くことが重要です。これらの構造物は、奥深くに隠されているものを露出させることは困難です。ただし、髄膜はできるだけ取り除きます。そうしないと、多くの場合、切断されずに浮き上がり、イメージングやスライスの妨げになります(図2A)。 - 脳を埋め込む前に ex vivo MRIにさらすとき、その間に脳をフッ素溶液に浸すときは、特に注意が必要である15。残ったフッ素は、一緒に埋め込まれると、イメージング中に対物レンズの下に小さな気泡を形成する可能性があります。これを避けるためには、脳をPBに1週間置いてから埋め込んでください。
注:この待機ステップは通常必要ではありませんが、 ex vivo MRIと組み合わせると重要です。 - 40°Cに温めた50 mMホウ酸緩衝液(pH 9.2)100 mLに0.2 gのNaBH4を溶解して、NaBH4緩衝液を調製します。 キャップを緩めたままにして、CO2 ガスを排出します。キャップを緩めた状態で溶液を一晩保管します。ボトルをアルミホイルで包み、使用まで光から保護します。
- 2.25 gのアガロースと0.21 gのNaIO4 を100 mLのPB中に2〜3時間攪拌して、酸化アガロースを作成します。溶液を真空吸引でろ過し、PBを3回交換して洗浄します。アガロースを50mLのPBに再懸濁します。
- アガロースを電子レンジで完全に溶かし、60〜65°Cに冷まします。
- 脳をカスタムメイドのチャンバーに入れ、脳をアガロースに埋め込みます。アガロースを脳梁の下の空洞に導入するように注意してください(図1B)。
注:最初に室温で脳を温め、次に65°Cで5分間予温して、アガロースが固化せずに脳表面に沈殿できるようにします。 - チャンバーを分解し、アガロースブロックをNaBH4バッファーに4°Cで一晩浸します。
- バッファーをPBに数回、4°Cで1〜2週間交換します。
注:バッファー交換が不完全な場合、組織の自家蛍光は非常に弱くなります。 - エポキシインスタントミックスでネオジム磁石を4個取り付けてスライドガラスステージを作ります。
- アガロースブロックを強力な接着剤(瞬間接着剤など)でステージに取り付けます(図1C)。
3. ティッシュ加工
- 製造元の指示に従って、組織イメージングシステム全体を操作します。詳細なプロトコルも公開されています16。現在の手順は主にTissueCyte1000に使用されていますが、TissueCyte 1600FCなどの他のモデルにも適用できます。
- 操作はモデル間で若干異なりますが、一般的に重要な以下のポイントに従ってください。
- マーモセットの脳全体は、ハードウェアを変更することなく、前端から後端まで冠状に処理できます。ブレインを配置するときは、ステージの移動制限を超えないようにしてください。
- ブレードの角度を微調整して、ブレードの両端の表面深さが10μm以内になるようにします。髄鞘形成が密集しているため、深さが10μmの違いであっても、成体のマーモセット白質を介したレーザーの透過が異なる可能性があります。同じ理由で、結像面を表面から約25〜35μmに設定します。
- セラミックブレード(推奨)を使用して、50μm間隔で脳全体を切断します(~650スライス以上)。
- 髄膜は切断されず、適切に除去しないとスライスの妨げになります(図2)。一部の髄膜(例えば、海馬または肺の周囲の髄膜)は除去が困難です。気づいたら、細かい鉗子でこれらの髄膜を手動で抽出します。
4.補助的な組織学的技術
- セクションの取得
- 組織切片を回収して、さまざまな組織学的染色を行います。これらのセクションをスライス順に正確に位置合わせします。皮質層を横切って血管をトレースし、位置合わせを行います(図3)。
- 取り扱いを良くするために、各セクションから余分なアガロースを切り取ります。
注: このプロセスは過度にする必要はありません。アガロースは、組織学的処理を妨げることなく組織セグメントを所定の位置に保持する役割を果たします。 - バックライト付きイメージング
- セクションをスライドグラスに取り付けて乾かします。
- 切片をPBSで再水和し、イメージング用のカバースリップを配置します。
- 切片を蛍光顕微鏡(図4)の明視野モードでイメージングすると、染色のない髄鞘形成パターンが観察されます。光の反射が見えるように露光時間を調整してください。
注:暗視野照明を使用できる場合は、どの顕微鏡でも使用できます。 - カバースリップをはがし、ニッスル染色またはその他の染色に進みます。標準的な染色手順で十分です。
- この方法を使用して、異なる染色パターンと髄鞘形成パターンを直接比較します(図4)。
- BDAイメージング
- 詳細な手順については、 表 1 を参照してください。
- まず、切片をデント液(20%DMSO、80%メタノール)で処理します。デントの溶液に含まれるメタノールは、軸索のBDAシグナルを劇的に増強します。
- チラミドシグナル増幅(TSA)法を使用して、蛍光検出されるBDAシグナルを強化します17。
注:TSA-ビオチン溶液はベンダーから購入できますが、In-House試薬の方がはるかに安価で効果的です。 - STPTイメージに登録するには、封入メディアで切片をスライドガラスにマウントした後、バックライト画像を撮影します。
- スパゲッティモンスター蛍光タンパク質(smFP)染色
注:「スパゲッティモンスター」蛍光タンパク質(smFP)は、複数のエピトープタグ18を有する非蛍光GFP変異体のファミリーである。免疫検出時に優れた蛍光シグナルを提供し、蛍光トレーサーのコンパニオントレーサーとして適しています。Addgeneから入手可能なAAVコンストラクトについては、 表2 を参照してください。- 表 1 に示す詳細な手順に従います。
5. イメージング後のデータ処理
- データ整合性テスト
注: STPT は膨大な数の画像ファイルを生成します。成魚のマーモセットの脳を50μm間隔で冠状に切片化する場合、脳全体を覆うためには通常650以上の切片が必要になります。各セクションのデータは、3 つのチャネルでキャプチャされた一連のタイリング画像で構成されています。これらの画像ファイルは、各タイルの位置を記録するメタデータ ファイルと共に 1 つのフォルダーに格納されます。データの総量と処理時間を最小限に抑えるために、イメージングは実行のブロックで行われ、その間にタイリングのXステップとYステップの数が徐々に変化します。通常、20〜30ブロックの実行は、それぞれが30〜50のセクションのイメージングを含み、単一の脳の完全なデータセットを構成します。このデータを利用するには、タイル化された画像のスティッチング、蛍光シグナルのセグメンテーション、標準テンプレートへのボリュームデータの登録など、いくつかの画像処理手順が必要です。- 画像処理ステップは、さまざまなソフトウェアツールを使用するか、研究者の特定のニーズに合わせて調整された専用のパイプラインを使用して、個別に実行します。画像処理の詳細は、この論文の範囲を超えており、他の出版物11,19で見つけることができます。
- 使用する手順に関係なく、データが一貫して整理されていることを確認してください。
注:イメージングプロセス全体には多数の実行ブロックが含まれ、数日間にわたるため、実行が途中でキャンセルされ、新たに再開されることは珍しくありません。このような場合、データ構造が乱れ、画像処理パイプラインの適切な実行が妨げられる可能性があります。 - データ構造の中断のリスクを最小限に抑えるには、Python スクリプトを実行して、GitHub (github.com/watkarbey/STPT_depo) で入手可能なデータ構造 (kn_pipeline_check_mosaic.py) の整合性を確認します。
- STPT画像への染色画像の登録
注:染色された画像からSTPT画像への非線形変換は、ImageJのbUnwarpJプラグインを使用して実行できます。- ターゲット(STPT)画像とソース(染色)画像の画像サイズを調整して、ほぼ等しくなります。
- 各画像を二値化して、セクションの形状を視覚化します。
- bUnwarpJ を実行します。[変換の保存] オプションをオンにすることを忘れないでください。
- bUnwarpJを再度起動します。二値化前の元の画像をソース画像として選択します。次に、[Load Elastic Transformation]をクリックし、保存した変換を選択します。
結果
ここで使用される一般的なセットアップでは、成魚のマーモセットの全脳を~1.3 x 1.3 μm/ピクセルの解像度で、約1週間で50 μmの切片間隔でイメージングできます(図5)。これは、画像スティッチング後の3つのチャネルで~650個のコロナ画像に相当します。16倍対物レンズ(Nikon 16xW CFI75 LWD;NA=0.80)の場合、単発の視野は約1×1mmです。コロナ表面全体の画像は、これらのショットをステッチすることによって得られます(図5A)。Z方向のアライメントが優れており、冠状画像データを単純に積み重ねるだけで良好な3次元画像が得られます(図5B、E)。標準化のために、マーモセットブレイン11 用のSTPTテンプレートを3D−3Dレジストレーションに用いることができる(図5F)。このデータ変換は、全脳神経解剖学の重要な側面の1つであり、対象領域には解剖学的注釈に依存しない絶対空間座標が割り当てられます。サンプルの脳が標準空間に登録されると、さらに分析するために関心のあるサブ領域を簡単に取り出すことができます(図5F、G)。特に、皮質領域は、所定のパラメータを使用してフラットマップに変換することができる(図5H)。
イメージング中に生成された切片は、さまざまな組織学的目的に使用できます。図4A、Bに示すように、染色のないバックライトイメージングは、真正なミエリン染色と非常によく似たパターンを提供します。これは、ミエリン染色の優れた代替手段となります。さらに、Nissl染色前にバックライト画像を取得した場合、同じ切片を使用してミエリン染色とNissl染色の両方のパターンを取得できるため、領域と層を特定するための有用な情報が得られます(図5C-E)。これらの切片は、免疫学的染色にも使用できます。図5Jでは、注入中心の周囲をNeuN抗体で対比染色し、AAVウイルスの形質導入効率を推定しています。また、蛍光トレーサーに加えて非蛍光トレーサーを注入し、切片検索後に組織学的に検出することも良い戦略です。私たちの以前の研究では、前行性グリーントレーサーと逆行性「cre」ベクターを組み合わせ、後に抗cre抗体10によって検出されました。図6A-Eの例では、BDAをグリーントレーサー(クローバー)の反対側に注入し、蛍光で検出しました。なお、赤色のBDAシグナルは、TissueCyte画像に登録して、全脳座標に局在させることができます。図6F-Iの別の例では、smFP-mycを頭頂部に注入し(図6G)、緑色のトレーサーを前頭部に注入しました。このようにして、イメージングを妨げることなく、複数のトレーサーを同じ動物に注入することができます。STPT切片を追加染色に使用する大きな利点は、同じ脳の蛍光トレーサーと非蛍光トレーサーの関係を決定できることです。そのため、皮質突起の相反性を高精度10で決定することができました。もう1つの利点は、染色された切片の3D座標をSTPTデータにマッピングし直し、次に標準テンプレートにマッピングできることです。したがって、取得したすべての切片を染色に使用する必要がない場合があります。より解釈しやすくするために、染色用の切片を選択して、STPTデータにさらにコンテキストを追加することができます。
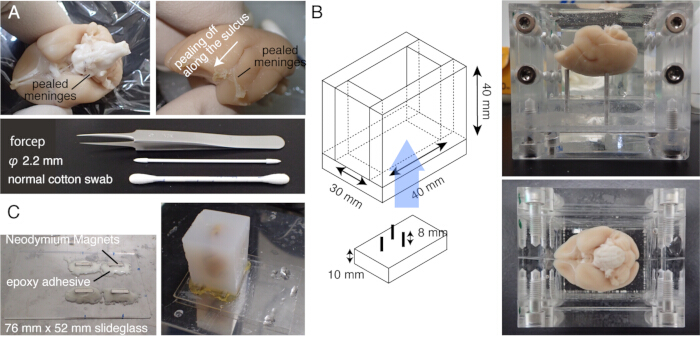
図1:STPTのサンプル調製 (A)綿棒を使用した髄膜の除去。脳幹を囲む髄膜は、先端の細い鉗子で取り除くことができます。マーモセットの脳を囲む髄膜は、綿棒でこすりながら手作業で取り除きます。写真は、外側溝の内側から髄膜が剥がれているところです。(B)アガロースの埋め込みに用いるアクリルボックス。ピンは可動式で、脳の角度を定位固定位置20に近づけるように調整するために使用される。(C)76mm×52mmのスライドガラスと4つのネオジム磁石をエポキシ接着剤で取り付けた磁気スライド。アガロースブロックは、瞬間接着剤で磁気ステージに取り付けられています。 この図の拡大版を表示するには、ここをクリックしてください。
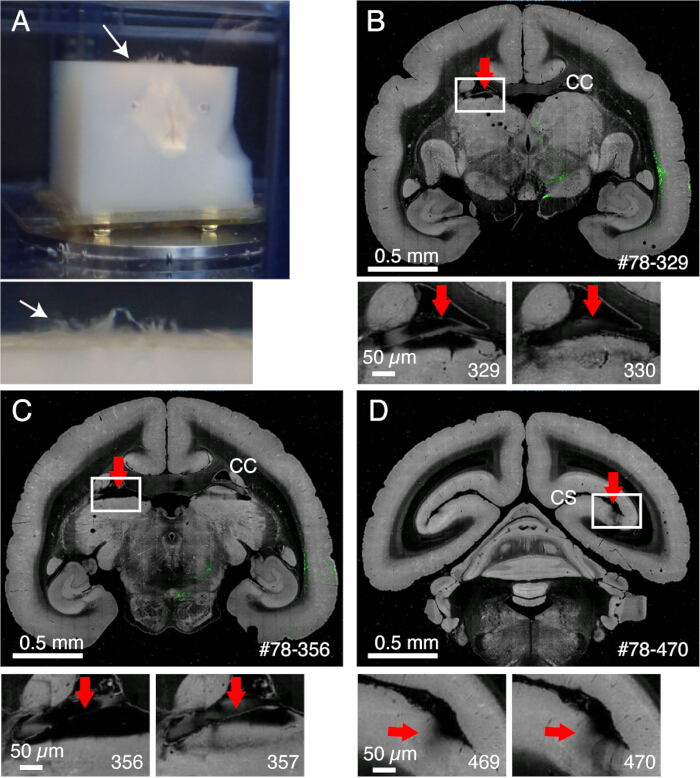
図2:髄膜のイメージングへの影響。 (A)白矢印のように、切り残した髄膜が浮き上がります。この例では、アガロース包埋前に髄膜が除去されなかったため、髄膜の広範な突出が見られます。通常、髄膜はいくつかの困難な領域でのみ未切断のままです。(B)脳梁と視床の上部との間に髄膜が存在することによる悪いスライスの例。この場合、スライシングが悪いと、深いスライス(329)と比較的正常なスライス(330)が交互に繰り返されることになった。#78-329は、Brain/MINDSデータポータルのサンプルNo.78セクションNo.329の略です(C)これも不良イメージングの一例です。最悪の場合、赤い矢印で示されているパルビナー核が完全に剥がれる可能性があります。スケールバー:0.5 mm(上部パネル)、0.5 μm(下部パネル)。(D)悪いイメージングの別の例。カルカリン溝の奥深くにある髄膜は除去が困難です。セクション469と470に見られる影は、目標の下に浮遊する髄膜によって引き起こされます。スケールバー:0.5 mm(上部パネル)、0.5 μm(下部パネル)。 この図の拡大版を表示するには、ここをクリックしてください。
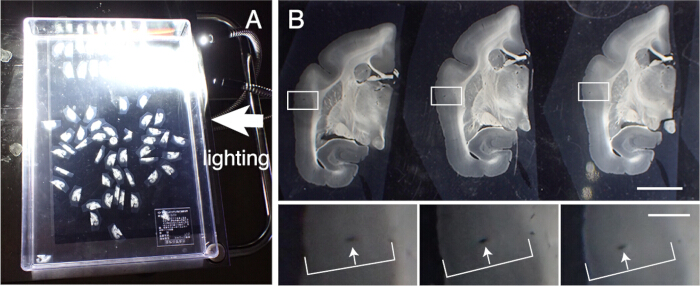
図3:組織切片の整列 。(A)プラスチック容器(31cm×22.5cm)内で、マーモセットの冠状切片を最大50個まで正しく整列させることができます。詳細な構造を視覚化するために、コンテナを黒い紙の上に置き、側面から照明を当てます。これらのセクションは、最初に大まかに整列され、次に正確な整列の対象となります。(B)正確なアライメントは、血管をマーカーとして使用します。大脳皮質には、皮質層を垂直に走る多くの血管が含まれています。それらは、皮質層(白い矢印)内で位置を体系的に変化する細長い穴として識別されます。この方法を用いると、50μm間隔の切片でも正確に位置合わせすることができます。これらの血管の穴は、SPTPセクションの画像で確認するために識別できます。スケールバー:5 mm(上部パネル)、1 mm(下部パネル)。 この図の拡大版を表示するには、ここをクリックしてください。
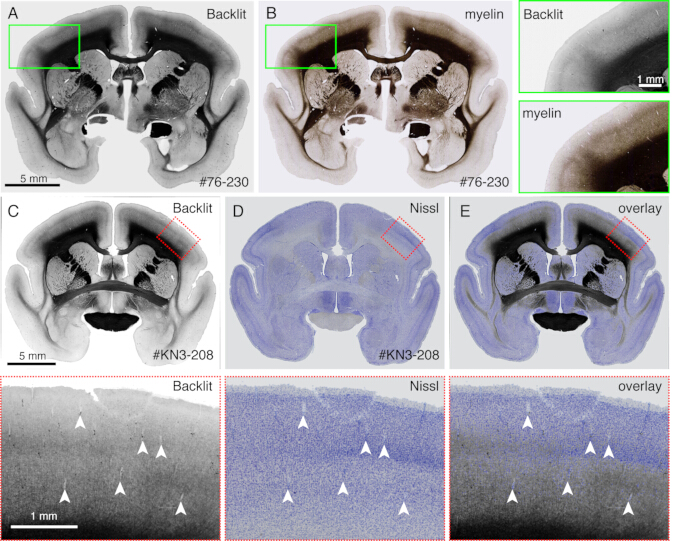
図4:ミエリン染色の代替としてのバックライト画像(A、B)同じ切片をバックライトイメージングとミエリン染色に使用しました。まず、切片をスライドガラスに載せ、乾燥させ、PBSで再水和させ、カバースリップを施し、光学顕微鏡で撮像しました(Table of Materials)。カバーガラスをはがした後、同じ切片をミエリン染色21に用い、蛍光顕微鏡を用いて画像化した。バックライト付き画像は、ImageJのbUnwarpJプラグインを使用してミエリン画像に登録しました。緑色のボックスは、各画像の拡大図を示しています。これらの画像は、ミエリン染色が繊維構造をよりよく視覚化することを除いて、ほぼ同じパターンを示していることに注意してください。(C-E)同一の切片をバックライトイメージングとニッスル染色に使用しました。バックライト付き画像の下限マスクは、まず bUnwarpJ プラグインを使用して Nissl 画像の下限マスクに登録され、元の画像は同じパラメータを使用して変換されました。血管 (白い矢印) が 2 つの画像間でよく一致していることに注意してください。これら2つの画像は同じセクションのものであるため、一致はほぼ完全であり、皮質層の識別のためにミエリンパターンとニッスルパターンを直接比較できます。スケールバー:5 mm(パネルAE)。この図の拡大版を表示するには、ここをクリックしてください。
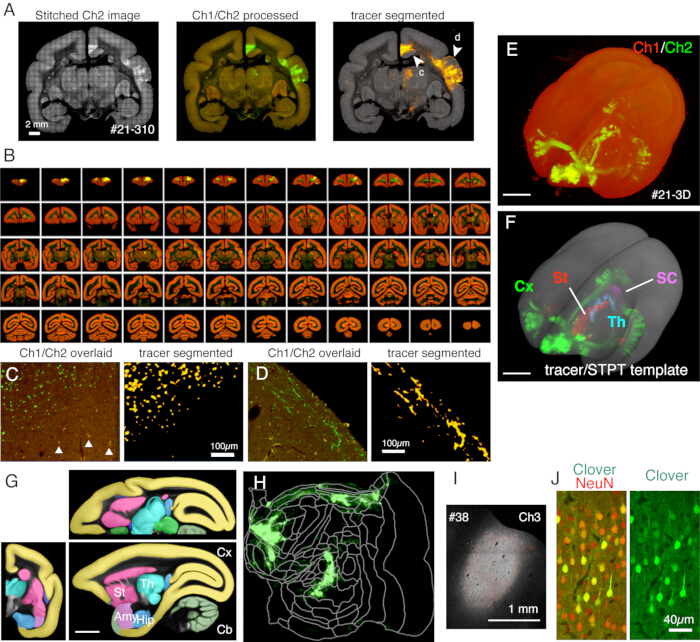
図5:STPTイメージングの典型的な結果 (A)サンプルセクション(サンプル#21のセクション310)のCh2(緑)画像の単純なタイリング。背景補正を行わないと、各タイルの境界線が表示されます。中央の列では、タイルにCh1(赤)とCh2(緑)の背景補正を施してステッチしました。リポフスチンのシグナルを減らすために(パネルCを参照)、Ch1シグナルをCh2から減算し、緑色で示しました。Ch1のリポフスチンシグナルを減らすために、ImageJによるスティッチングの前に「Remove Outilers..」コマンドを使用しました。右の列では、トレーサ信号は画像処理パイプライン11によってセグメント化されている。矢印(c、d)は、パネルCおよびDに拡大されたビューが示されている場所を示しています。 スケールバー:2 mm。 (B)サンプル#21のシリアルセクションの概要。STPTは、このサンプルのために635枚の高解像度コロナ画像を生成しました。(C)パネルAの矢印cで示された高倍率図。これは、Ch1 (赤) と Ch2 (緑) の単純なオーバーレイで、それ以上の処理は行われていません。三角形はリポフスチン蛍光シグナルを示しており、広範囲にわたるスペクトルを示しています。トレーサーセグメンテーションアルゴリズムは、形状が非常に似ているにもかかわらず、トレーサーシグナルをリポフスチンの背景から正確に区別します(右パネル)。スケールバー:100μm.(D)高倍率ビューの別の例。レイヤー1の細い軸索繊維はよく見えることに注意してください。スケールバー:100μm.(E)元のSTPT画像の3D再構成。パネルBに示されている635枚の低解像度コロナ画像は、fluorender22を使用した3D視覚化のtiffスタックとして使用されました。スケールバー:5 mm. (F) STPTテンプレートに登録されたセグメント化されたトレーサー信号の3D再構成(灰色)。異なる脳領域のトレーサー信号を異なる色で示しました。これらの領域は、パネルGに示されている注釈を使用して切り取られました。 スケールバー:5 mm.(G)STPTテンプレートに異なる脳領域の注釈を重ね合わせました。スケールバー:5mm.(H)パネルFに示されている皮質トレーサー信号は、フラットマップの形で示されました。(I)Ch3蛍光による注入部位の同定は、トレーサー蛍光に対する感度が低く、不飽和のままです。スケールバー:1 mm. (J) NeuN抗体で注入中心周囲の切片を染色すると、ニューロンの約30%がクローバーグリーン蛍光を強く発現していることが示されました。スケールバー:40μm.略語:Cx;皮質、セント;線条体、Th;視床、サウスカロライナ州;上コリキュラス。エイミー;扁桃体、ヒップ;海馬、Cb;小脳。 この図の拡大版を表示するには、ここをクリックしてください。
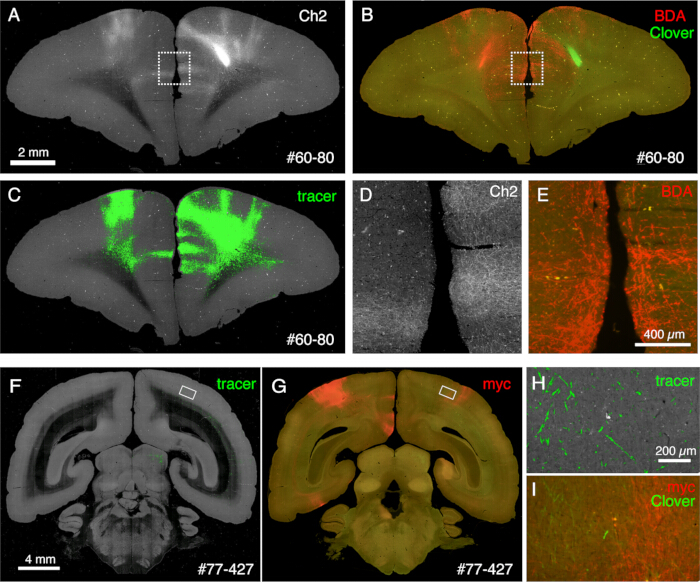
図6:複数の非蛍光トレーサーを示すSTPT後の組織型。 (A-C)STPT画像とBDA染色画像の比較。このサンプルでは、BDAをClover注入の反対側に注入します。パネルAとCは、STPTデータのCh2イメージとトレーサーセグメンテーションを示しています。パネルBは、BDAのSTPT後染色を示しています。クローバーの蛍光は、切片のメタノール処理により減少します。スケールバー:2 mm. (D,E) パネルAとBの点線ボックスが拡大されています。スケールバー:100 μm. (F,G) STPT画像と抗mycタグ抗体染色との比較。このサンプルでは、クローバー注射はPFCに対して行われますが、AAV-smFP-mycは反対側の頭頂葉皮質に注入されます。白い長方形はパネルHとIで拡大されています。 スケールバー:4 mm、(H)トレーサーのセグメンテーションは緑色で示されています。スケールバー:200μm.(I)myc染色は赤で示されています。クローバーの蛍光は緑色で示されています。スケールバー:200μm。パネルHとIの緑色信号は同様の位置に存在しますが、STPTは~10μmの光学セクションしか取得しないため、同一ではありません。この図の拡大版を表示するには、ここをクリックしてください。
| BDA蛍光染色プロトコール | |
| アガロースを取り除く | |
| TBSウォッシュ | 10 分 (2x) |
| デント溶液中の1%H2O2 | 10分間 |
| TBSウォッシュ | 短 |
| 0.5% TNBブロッキング | 1時間 |
| StAvHRP (1:4000) (TNB) | 2泊 |
| TNTウォッシュ | 10 分 (3x) |
| 0.1 M ホウ酸 (pH8.5) + 0.003% H2O2 に TSA ビオチン (1:4000) | 2時間 |
| TNTウォッシュ | 10 分 (3x) |
| TNT中のCy3-ストレプトアビジン(1:1000) | 3時間 |
| TNTウォッシュ | 10 分 (2x) |
| TBSウォッシュ | 取り付けるまでセクションを保管してください |
| 褪色防止封入剤を使用してスライドガラスにセクションをマウントします | |
| 抗MYC蛍光染色プロトコル | |
| アガロースを取り除く | |
| TBSウォッシュ | 10 分 (2x) |
| IBでのブロッキング | 1時間 |
| IBにおける抗Myc(1:4000) | 2泊 |
| TNTウォッシュ | 10 分 (3x) |
| TNT中の抗マウスCy3(1:1000) | 3時間 |
| TNTウォッシュ | 10 分 (2x) |
| TBSウォッシュ | 取り付けるまでセクションを保管してください |
| 褪色防止封入剤を使用してスライドガラスにセクションをマウントします | |
| バッファー/ソリューション | 組成 |
| 0.5%TNBの | TS7.5の0.5% TSAブロッキング試薬 |
| デントのソリューション | 20%DMSO、80%メタノール |
| イマージョンバッファー(IB) | TBSで10%FBS、2%BSA 0.5%TritonX100 |
| TBS(トリスバッファー生理食塩水) | 25 mM Tris、137 mM NaCl、2.7 mM KCl (pH 7.4) |
| TNTの | 0.05 % Tween20 (TS7.5) |
| TS7.5の | 0.1 M TRIS-HCl、pH 7.5、0.15 M NaCl |
表1:BDA蛍光染色および抗myc蛍光染色のプロトコール
| AAVトレーサー用プラスミド | Addgene番号 | 推奨抗体 | 推奨希釈率 | 期待される結果 |
| pAAV-EF1_Cre | 201198 | ミリポアクローン2D8 | 1:1000 | 細胞に良い |
| pAAVCam1.3_smFP_Myc | 201205 | MBL M192 My3 (マウス) | 1:4000 | たいへん良い |
| pAAVCam1.3_smFP_HA | 201206 | CST C29F4 (ウサギ) | 1:1000 | 細胞に良い |
| pAAVCam1.3_smFP_FLAG | 201207 | MBL PM020B(うさぎ) | 1:1000 | わかりました |
| AAVTRE3_smFP_Myc | 201208 | |||
| AAVTRE3_smFP_HA | 201209 | |||
| AAVTRE3_smFP_FLAG | 201210 |
表2:非蛍光トレーサーの作製に利用可能なAddgeneプラスミドのリスト。creコンストラクトは核を標的とし、AAV2レトロに包まれたレトログラードトレースに適しています。smFP_HAコンストラクトは、細胞体の検出(および逆行性)に適しています。
補足ビデオ:髄膜除去の顕微鏡ビュー。このビデオをダウンロードするには、ここをクリックしてください。
ディスカッション
この記事では、マーモセットの脳を全脳処理に扱うための実用的な解決策と、STPT技術の有用性を高める補助的な組織学的技術について説明しました。STPTを用いた「全脳神経解剖学」の強みは、解剖学的にアノテーションが付いているかどうかにかかわらず、任意の関心領域の3D座標を取得できることです。高精度な3D-to-3Dレジストレーションにより、これらの座標を複数のデータセットを重ね合わせるための標準テンプレートに変換することができます。このように、標準テンプレートはデータ統合の媒体として機能します。これは、多くの個人から得られたデータを分析した前頭前野(PFC)マッピングプロジェクト10の重要な側面でした。さらに、標準テンプレートにマッピングされた情報は、ニッスル、ミエリンパターン、トレーサーデータ、MRI(拡散MRIを含む)データ、または解剖学的アノテーション11であるかどうかにかかわらず、既にマッピングされている種々のデータと比較することができる。重要なのは、まだ出現していないテクノロジーによって得られる将来のデータと比較することもできることです。現在、マーモセット脳には、ニッスル染色23、24、25、26、27、28、およびSTPT11に基づく複数のテンプレートが存在します。しかし、それらは、画像のコントラストと事前に計算されたパラメータ11に基づいて、互いの座標に変換することができる。研究間での脳全体のスケールでのデータ統合は、システムとしての脳の理解を深めることに貢献します。この戦略が機能するための前提条件は、脳全体で信頼性の高いデータ取得を行うことです。以下では、現在のプロトコルに関連する重要な手順と潜在的な問題について説明します。
STPTイメージングの最も脆弱なプロセスの1つは、組織スライスです。前述のように、髄膜はしばしばカットされずに残り、スライスを妨げる可能性があります。特に、肺核と上丘は最も影響を受ける2つの脳領域であり、髄膜の除去とアガロースへの埋め込みの徹底性によっては、スライス中に組織ブロックから剥がすことができます。これらの深い領域は外部から近づくのが難しく、髄膜の除去中に簡単に壊れる可能性があります。慎重かつ徹底的な髄膜除去は、イメージングを成功させるために重要です。また、スライスした部分がティッシュブロックに反転するのも問題で、スライス後も切片がくっついたままの場合に発生することがあります。ブレードが最後に斜めに切れるようにブロックを成形することで最小限に抑えることができます。
組織ブロックの深部までイメージングすることで、STPTはその表面の凹凸を回避します。蛍光シグナルは皮質領域を比較的容易に通過できますが、有髄領域では大幅に減少します。したがって、イメージング深度は、ブロック表面全体にわたる一貫したイメージングと、白質などのミエリンリッチ領域の信号の明るさとのバランスをとるために慎重に決定する必要があります。ここで使用するセットアップでは、通常、表面から25〜35μmを目指します。また、長時間の保管では切断面が不均一に縮むことがあるので注意が必要です。イメージングエリアを最小限に抑えるために、イメージングセッションを20〜30回に分割し、5〜6日間、異なるステージ設定で実行します。実行間隔を2時間未満にするか、表面の深さを確認して、各実行前にステージの高さを調整します。
このプロトコルでは、BDA信号をTSA法によって増幅しました。この方法は非常に効果的で、比較的低い分解能でも順行性して輸送されたBDA信号を検出できます( 例:図6B)。TSAビオチンはアコヤバイオサイエンス社から市販されていますが、自家製の溶液の方がはるかに優れた強化を示しています。一方、抗体と溶液の希釈は、最適な結果を得るために慎重に調整する必要があります。メタノール溶液による切片の前処理は重要です。前処理を行わないと、BDA信号は有髄軸索でほとんど検出されません。
順行性トレーサーを使用する場合、蛍光シグナルが飽和しているため、正確な注入部位を特定することが困難になることがよくあります。この研究で使用した全組織イメージングシステムのセットアップでは、青色チャネルを使用して感染細胞を同定します(図5I)。赤と緑のチャネルが飽和している場合でも、個々の感染ニューロンは通常、青色チャネルで検出可能です。これは、Allen Mouse Brain Connectivity Atlas1にも当てはまります。感染は特定の層のみに関与する場合があるため、起源の細胞の検査は重要です。Mouse Brain Connectivity Atlasでは、このような部分感染がかなり頻繁に発生しましたが、これはおそらくイオントフォレーシス法29を利用したためでしょう。ウイルストレーサーの横方向の広がりは、注射によって多かれ少なかれ変動する可能性があります。この変動性は、トレースの結果に影響を与える可能性があるため、慎重な正規化が必要です。
取得した3D画像を標準テンプレートに正常に登録することは、全脳神経解剖学の重要なプロセスです。STPTテンプレートへのSTPT画像の登録はかなり正確であり、画像コントラストが高い境界10では、数ボクセル(50μm等立体)の逸脱のみが観察されました。それでも、登録でできることには限界があります。髄膜の除去プロセスにより、STPTサンプルは通常、半球間と皮質と中脳/後脳の間にギャップがありますが、 脳組織はin vivo MRI画像に密集しています。このような違いは、登録で調整するのが難しいです。マーモセット皮質にはほとんど溝がありませんが、一部の個体では頭頂内溝が非常に深いです。このような溝は、登録時に失われます(上部から上部への融合が発生します)。登録は強力な手法ですが、得られた結果を確認するためには、生データに戻る必要があります。
大量の画像データの生成は、この手法の強みであると同時に制限でもあります。データセットの完全性を向上させる一方で、取得したデータを慎重に管理し、効率的なデータ解釈のための自動画像処理パイプラインを開発する必要があります。将来的には、画像処理パイプラインの構築に生成人工知能(AI)を適用することで、このプロセスが大幅に簡素化される可能性があります。体系的な全脳イメージングも、スライドスキャナーベースの方法を使用して行われています21,30。このような方法と比較して、STPTは3D再構成のための追加計算を必要としません。光学セクショニングと組み合わせることで、STPTがセクション31全体で均一な軸索セグメントを再構築する可能性があることを実証しました。組織クリアランス技術のさらなる組み合わせにより、Economoらは、まばらに標識されたニューロン32,33の全体を画像化する方法を開発した。最新バージョンのTissueCyteでは、赤色蛍光タンパク質の励起を強化するためのレーザーの追加や、切片の自動回収のための切片キャプチャユニットのオプションが提供されています。これらの進歩により、全脳アプローチはより効率的になり、ヒトを含む霊長類の脳を包括的に理解するための基盤となります。
開示事項
著者は何も開示していません。
謝辞
山森研究室・動物施設(RRD)の技術スタッフの方々に深く感謝申し上げます。共焦点画像取得の技術支援をいただいた理研CBS-オリンパス連携センターに感謝いたします。本研究は、文部科学省の新学術領域研究推進事業(課題番号22123009)、AMEDのBrain/MINDSおよびBrain/MINDS2.0(JP15dm0207001、JP23wm0625001、JP24wm0625218)、およびJSPS科研費24K09678(A.W.)の支援を受けて行われました。
資料
| Name | Company | Catalog Number | Comments |
| Agarose type I | Sigma | A6013 | |
| All-in-one fluorescence microscope | Keyence | BZ-X series | This study used BZ-X710 |
| anti-mouse Cy3 | Jackson Immuno Research Laboratories, Inc. | 115-165-003 | |
| anti-myc antibody | MBL | M192 | mouse monoclonal |
| Butorphanol | Meiji Animal Health | Vetorphale | |
| Ceramic blade | TissueVision | N/A | |
| Collagenase type I | Wako Chemical | #031-17601 | Aliquot into 100 µL x 10 of 100 mg/mL in PBS |
| Cotton swab (HUBY-340) | HUBY | BB-013SP | micro cotton swabs |
| Custom-made chamber | N/A | N/A | The chamber consist of four side plates and one bottom plates, all made of 1 cm thick acrylic plates, which can be securely fastened together using screw bolts. An additional acrylic plate is placed inside the chamber, with three metal poles inserted through it to hold the marmoset brain in an inverted position. |
| Cy3-Streptavidin | Jackson Immuno Research Laboratories, Inc. | 016-160-084 | |
| Dumont #5 forceps, Inox | N/A | N/A | |
| Epoxy instant mix | Locktite | N/A | |
| FIJI | NIH | https://imagej.net/software/fiji/downloads | |
| kn_pipeline_check_mosaic.py | N/A | N/A | github.com/watkarbey/STPT_depo; This is a checking script for TissueCyte data. |
| Medetomidine | Zenoaq | Domitor | |
| Midazolam | Sandoz | N/A | |
| NaBH4 | Sigma | #452882 | |
| NaIO4 | Sigma | S1878 | |
| Perfusion needle | Natsume Seisakusho Co., Ltd | KN-348, 20G-50 | |
| Slideglass (76 mm x 52 mm) | Matsunami | S9111 | |
| StAvHRP | Jackson Immuno Research Laboratories, Inc. | 016-030-084 | HRP-streptavidin (titration needs to be carefully determined) |
| TissueCyte1000 | TissueVision | https://www.tissuevision.com/tissuecyte | |
| TSA blocking reagent | Kiko Tech | FP1012 | Refer to TSA-biotin kit (Akoya Biosciences) |
| TSA-biotin | House-made | N/A | See Okamoto et al (ref. 16) |
| VECTASHIELD HardSet Mounting Medium | Vector laboratories Inc | N/A | Antifade mounting medium |
参考文献
- Oh, S. W., Harris, J. A., Ng, L., Winslow, B., Cain, N., et al. A mesoscale connectome of the mouse brain. Nature. 508 (7495), 207-214 (2014).
- Ragan, T., et al. Serial two-photon tomography for automated ex vivo mouse brain imaging. Nat Methods. 9 (3), 255-258 (2012).
- Liang, Z., Arefin, T. M., Lee, C. H., Zhang, J. Using mesoscopic tract-tracing data to guide the estimation of fiber orientation distributions in the mouse brain from diffusion MRI. Neuroimage. 270, 119999 (2023).
- Watakabe, A., Hirokawa, J. Cortical networks of the mouse brain elaborate within the gray matter. Brain Struct Funct. 223 (8), 3633-3652 (2018).
- Ypma, R. J. F., Bullmore, E. T. Statistical analysis of tract-tracing experiments demonstrates a dense, complex cortical network in the mouse. PLoS Comput Biol. 12 (9), e1005104 (2016).
- Wang, Q., et al. The Allen mouse brain common coordinate framework: A 3D reference atlas. Cell. 181 (4), 936-953.e20 (2020).
- Köbbert, C., Apps, R., Bechmann, I., Lanciego, J. L., Mey, J., Thanos, S. Current concepts in neuroanatomical tracing. Prog Neurobiol. 62 (4), 327-351 (2000).
- Lanciego, J. L., Wouterlood, F. G. Neuroanatomical tract-tracing techniques that did go viral. Brain Struct Funct. 225 (4), 1193-1224 (2020).
- Bakker, R., Wachtler, T., Diesmann, M. CoCoMac 2.0 and the future of tract-tracing databases. Front Neuroinform. 6, (2012).
- Watakabe, A., et al. Local and long-distance organization of prefrontal cortex circuits in the marmoset brain. Neuron. 111 (14), 2258-2273.e10 (2023).
- Skibbe, H., et al. The Brain/MINDS marmoset connectivity resource: An open-access platform for cellular-level tracing and tractography in the primate brain. PLoS Biol. 21 (6), e3002158 (2023).
- Mitchell, J. F., Leopold, D. A. The marmoset monkey as a model for visual neuroscience. Neurosci Res. 93, 20-46 (2015).
- Okano, H. Current status of and perspectives on the application of marmosets in neurobiology. Annu Rev Neurosci. 44, 27-48 (2021).
- Watakabe, A., et al. Comparative analyses of adeno-associated viral vector serotypes 1, 2, 5, 8 and 9 in marmoset, mouse and macaque cerebral cortex. Neurosci Res. 93, 144-157 (2015).
- Hata, J., et al. Multi-modal brain magnetic resonance imaging database covering marmosets with a wide age range. Sci Data. 10, 221 (2023).
- Liwang, J. K., Bennett, H. C., Pi, H. -. J., Kim, Y. Protocol for using serial two-photon tomography to map cell types and cerebrovasculature at single-cell resolution in the whole adult mouse brain. STAR Protoc. 4 (1), 102048 (2023).
- Okamoto, S., et al. Exclusive labeling of direct and indirect pathway neurons in the mouse neostriatum by an adeno-associated virus vector with Cre/lox system. STAR Protoc. 2 (1), 100230 (2021).
- Viswanathan, S., et al. High-performance probes for light and electron microscopy. Nat Methods. 12 (6), 568-576 (2015).
- Kuan, L., et al. Neuroinformatics of the Allen mouse brain connectivity atlas. Methods. 73, 4-17 (2015).
- Paxinos, G., Watson, C., Petrides, M., Rosa, M., Tokuno, H. . The Marmoset Brain in Stereotaxic Coordinates. , (2012).
- Abe, H., et al. 3D reconstruction of brain section images for creating axonal projection maps in marmosets. J Neurosci Methods. 286, 102-113 (2017).
- Wan, Y., et al. FluoRender: Joint freehand segmentation and visualization for many-channel fluorescence data analysis. BMC Bioinformatics. 18 (1), 280 (2017).
- Majka, P., et al. Towards a comprehensive atlas of cortical connections in a primate brain: Mapping tracer injection studies of the common marmoset into a reference digital template. J Comp Neurol. 524 (11), 2161-2181 (2016).
- Liu, C., Yen, C. C. -. C., Szczupak, D., Tian, X., Glen, D., Silva, A. C. Marmoset brain mapping V3: Population multi-modal standard volumetric and surface-based templates. Neuroimage. 226, 117620 (2021).
- Liu, C., et al. A digital 3D atlas of the marmoset brain based on multi-modal MRI. Neuroimage. 169, 106-116 (2018).
- Liu, C., et al. A resource for the detailed 3D mapping of white matter pathways in the marmoset brain. Nat Neurosci. 23 (2), 271-280 (2020).
- Woodward, A., et al. The Brain/MINDS 3D digital marmoset brain atlas. Sci Data. 5, 180009 (2018).
- Saleem, K. S., Avram, A. V., Glen, D., Schram, V., Basser, P. J. The subcortical atlas of the marmoset ("SAM") monkey based on high-resolution MRI and histology. Cereb Cortex. 34 (4), bhae120 (2024).
- Watakabe, A., Hirokawa, J. Cortical networks of the mouse brain elaborate within the gray matter. Brain Struct Funct. 223 (8), 3633-3652 (2018).
- Lin, M. K., et al. A high-throughput neurohistological pipeline for brain-wide mesoscale connectivity mapping of the common marmoset. eLife. 8, e40042 (2019).
- Skibbe, H., et al. PAT-probabilistic axon tracking for densely labeled neurons in large 3-D micrographs. IEEE Trans Med Imaging. 38 (1), 69-78 (2019).
- Economo, M. N., et al. A platform for brain-wide imaging and reconstruction of individual neurons. eLife. 5, e10566 (2016).
- Winnubst, J., et al. Reconstruction of 1,000 projection neurons reveals new cell types and organization of long-range connectivity in the mouse brain. Cell. 179 (1), 268-281.e13 (2019).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved