Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Entwicklung eines sensitiven und spezifischen chromogenen Agar Medium zum Nachweis von
In diesem Artikel
Zusammenfassung
Detection and isolation of clinically relevant Vibrio species require selective and differential culture media. This study evaluated the ability of a new chromogenic medium to detect and identify V. parahaemolyticus and other related species. The new medium was found to have better sensitivity and specificity than the conventional medium.
Zusammenfassung
Lebensmittelbedingte Infektionen in der durch Vibrio - Spezies verursacht USA haben einen Aufwärtstrend gezeigt. In der Gattung Vibrio, V. parahaemolyticus verantwortlich für die meisten Vibrio -assoziierten Infektionen. So präzise Differenzierung zwischen Vibrio spp. und Detektion von V. parahaemolyticus ist von entscheidender Bedeutung für die Sicherheit unserer Nahrungsmittelversorgung zu gewährleisten. Obwohl molekulare Techniken immer häufiger sind, kultur je Methoden sind immer noch routinemäßig durchgeführt, und sie sind unter bestimmten Umständen von Standardmethoden in Betracht gezogen. Daher wurde eine neue chromogene Agarmedium mit dem Ziel untersucht , eine bessere Methode zur Isolierung und der Differenzierung von klinisch relevanten Vibrio spp bereitzustellen. Das Protokoll verglichen , um die Sensitivität, Spezifität und Nachweisgrenze für die Detektion von V. parahaemolyticus zwischen dem neuen chromogenes Medium und einem herkömmlichen Medium. Verschiedene V. parahaemolyticus - Stämme (n = 22) rediverse Serotypen und die Quelle der Herkunft präsentiert wurden verwendet. Sie waren zuvor von der Food and Drug Administration (FDA) und Centers for Disease Control and Prevention (CDC) identifiziert und weiter in unserem Labor von TLH -PCR verifiziert. In mindestens vier getrennten Versuchen wurden diese Stämme auf dem chromogenen Agar und Thiosulfat-Citrat-Gallensalze-Saccharose (TCBS) Agar beimpft, die die empfohlene Medium zum Kultivieren dieser Spezies ist, gefolgt von einer Inkubation bei 35-37 ° C für 24 -96 h. Drei V. parahaemolyticus - Stämme (13,6%) wuchs nicht optimal auf TCBS, dennoch zeigte grüne Kolonien , wenn es Wachstum war. Zwei Stämme (9,1%) brachte nicht die erwarteten Cyan Kolonien auf dem chromogenen Agar. Nicht V. parahaemolyticus - Stämme (n = 32) wurden auch die Spezifität des chromogenen Agar zu bestimmen getestet. Unter diesen Stämmen wuchsen, 31 nicht oder anderen Morphologien Kolonie ausgestellt. Die mittlere Rückgewinnung von V. parahaemolyticus auf der chromogenic Agar war ~ 96,4%, bezogen auf CASO-Agar mit 2% NaCl ergänzt. Im Ergebnis ist die neue chromogene Agar ein effektives Medium V. nachzuweisen parahaemolyticus und es von anderen Vibrionen zu unterscheiden.
Einleitung
Als Mitglied der Gattung Vibrio, V. parahaemolyticus ist ein Gram-negative, nicht-sporenbildende, gekrümmte, stabförmiges Bakterium. Es weist eine hohe Beweglichkeit in sowohl flüssigen als auch halbfesten Umgebungen. Die meisten V. parahaemolyticus Stämme sind nicht pathogen für den Menschen, aber die pathogenen Subtypen Epidemien und Pandemien verursacht, daher ist diese Spezies als 1,2 ein wichtiges Lebensmittel übertragene Erreger in vielen Ländern zu sein. Die Inzidenz von Vibrio - Infektion in den USA hat sich seit dem Jahr 2000 3 einen Aufwärtstrend gezeigt. Unter Vibrio spp., V. parahaemolyticus ist die am häufigsten Spezies berichtet Krankheiten in den USA 4,5 verursacht. Andere klinisch relevante Arten gehören V. alginolyticus, V. vulnificus, V. cholerae usw. Ein kleiner Prozentsatz der Erkrankungen wird gleichzeitig von mehreren Arten verursacht.
V. parahaemolyticus ist eine natürliche inhabitant von Meerwasser und damit in Meeresgewässern in der ganzen Welt, einschließlich der Mündungen weit verbreitet. Die Art wurde im Jahr 1950 nach einem Ausbruch der Lebensmittelvergiftung in Japan entdeckt. In den USA wurde die Art erstmals in Meerwasser, Sedimenten und Muscheln in der Region 6,7 Puget Sound isoliert. Filtrierer und in der marinen Lebensräume, wie Muscheln Muscheln, kann V. Hafen parahaemolyticus als Teil ihrer natürlichen Flora 8. Als solche V. parahaemolyticus Infektionen bei Mensch sind oft auf den Verzehr von kontaminierten Meeresfrüchten zurückzuführen sind, insbesondere rohen oder ungekochten Schalentiere. Eine weniger häufige Route der Eintrag erfolgt, wenn offene Wunde dem Meerwasser ausgesetzt ist, was zu einer Infektion der Haut. Die meisten V. parahaemolyticus - Stämme verursachen keine Krankheit beim Menschen, noch bestimmte Subtypen Virulenzfaktoren wie thermo direkte Hämolysin (TDH) sind beherbergen pathogen. Die häufigsten Symptome von Lebensmittelvergiftungen V. parahaemolyticus - InfektionDurchfall und Bauchschmerzen, gefolgt von Übelkeit, Erbrechen und Fieber. Kopfschmerzen und Schüttelfrost sind auch berichtet. Die mediane Inkubationszeit beträgt 15 Stunden, kann jedoch bis zu 96 Stunden nach dem Verzehr einer ausreichenden Menge an pathogenen Stämmen 9 sein. Die Krankheit dauert von zwei bis drei Tagen. Die Gastroenteritis Symptome verursacht durch V. parahaemolyticus sind weitgehend selbstlimitierend und deshalb Spezialbehandlung ist nicht erforderlich. Leichte Fälle von Gastroenteritis wirksam durch orale Rehydratation behandelt werden. Weitere schwere Erkrankungen können 10 von Antibiotika wie Tetracyclin oder Ciprofloxacin behandelt werden. Die Sterblichkeit liegt bei etwa 2% für Gastroenteritis Fällen, kann aber als 29% für die so hoch sein, die Blut-Infektion oder Sepsis zu entwickeln. Jede Person , die Meeresfrüchte verzehrt oder hat offene Wunde dem Meerwasser ausgesetzt ist gefährdet von V. parahaemolyticus Infektion. Die schwere Form von Krankheiten, lebensbedrohlichen Sepsis, ist häufiger in einer Subpopulation mit zugrunde liegenden medizinischen coBedingungen ausgebracht 11, der Alkoholismus, Lebererkrankungen, Diabetes, Nierenkrankheit, Bösartigkeit und andere Bedingungen zu einer geschwächten Immunantwort führt. Bemerkenswert ist , ist diese Gruppe von Individuen , auch zu einem höheren Risiko für schwere Vertrags von V. verursachte Krankheiten vulnificus, die in natürlichen Lebensräume ähnlich wie V. gefunden werden kann parahaemolyticus.
V. parahaemolyticus wird routinemäßig unter Verwendung von Thiosulfat-Citrat-Gallensalze-Saccharose (TCBS) Agar als selektiver und Differenzierungsmedium isoliert. Die Anreicherung in alkalischem Peptonwasser kann Isolation auf TCBS Agar vorangehen. Präsumptive Kolonien auf TCBS werden dann weiter in einer Reihe von biochemischen Tests getestet und / oder molekulare Assays, die die Anwesenheit von Spezies-spezifischen Gene Targeting. PCR-basierten Verfahren werden häufig verwendet , um die Identität von V. bestätigen parahaemolyticus durch das thermolabile Hämolysin - Gen verstärkt, tlh 12.
Unabhängig von der chStimm - Bestätigungsverfahren ist es wichtig , ein wirksames Medium zu haben , V. , zu isolieren und zu differenzieren parahaemolyticus von anderen Meeres Vibrionen in den ersten Platz. TCBS hat routinemäßig innerhalb der Vibrio Gattung Spezies zu unterscheiden , entsprechend ihren Fähigkeiten Saccharose 12 zu gären eingesetzt. Positive Fermentationsreaktion wird durch eine Farbänderung des pH - Indikators Bromthymolblau begleitet. V. parahaemolyticus Kolonien sind ziemlich unverwechselbar auf TCBS, Ausstellen blau zu grün. Jedoch kann dieses Medium nicht leicht unterscheiden V. alginolyticus und V. cholerae. Sucrose-Fermentation von Proteusarten gelbe Kolonien ähnlich V. produzieren kann cholerae oder V. alginolyticus 13. Bei der ersten Isolation auf TCBS, V. parahaemolyticus kann auch als Aeromonas hydrophila, Plesiomonas shigelloides falsch identifiziert werden, und Pseudomonas spp 14. Stämme mit verzögerter Saccharose fermsentation kann mit anderen Saccharose nonfermenting Vibrio 13, verwechselt werden , die V. umfassen parahaemolyticus. TCBS wurde als nicht empfindlich gegen Escherichia coli, Pseudomonas putrefaciens unter anderem gefunden. Mehrere andere Arten ergeben grün zu grau Kolonien , die mit V. möglicherweise verwechselt werden parahaemolyticus oder V. vulnificus 15. Als Ergebnis ist es wünschenswert , alternative Kulturmedien zu Erfassung mit einer besseren Empfindlichkeit und Spezifität und V. Isolierung zu entwickeln , parahaemolyticus und andere eng verwandte Arten.
Mehrere Medien Alternativen vor kurzem entwickelt wurden. Neben der Aufnahme von selektiven Agenzien, inkorporieren meisten chromogene Substrate Spezies zu unterscheiden, basierend auf ihrer unterschiedlichen enzymatischen Aktivitäten. Beispielsweise Indoxyl-β-glucosid und indoxyl-β-galactosid wurden als chromogene Substrate verwendet , zu unterscheiden V. parahaemolyticus Kolonien (die blaugrün erscheinen) von denen V. cholerae (lila) aufgrund ihrer unterschiedlichen Fähigkeiten β-Glucosidase und 16 β-Galactosidase zu produzieren. Verschiedene Formulierungen von chromogenen Agar von mehreren Gruppen entwickelt wurden bewertet und wurden zur Durchführung vergleichsweise gemeldet oder besser als TCBS 17,18,19. Ein Vorteil eines chromogenen Mediums besteht darin, dass die Färbung des umgebenden Mediums minimal ist, wodurch die Isolierung von bestimmten Kolonien zu erleichtern. In dieser Studie haben wir die Fähigkeit eines neu chromogenes Medium formuliert zu detektieren und zu isolieren , V. cholerae, V. parahaemolyticus und V. vulnificus; mit einem besonderen Fokus auf seine Fähigkeit , V. zu unterscheiden parahaemolyticus von anderen Spezies.
Protokoll
1. Medien und Anzucht von Bakterienstämmen
HINWEIS: Die Verwendung aseptischer Techniken in allen Experimenten. Verwenden Sie sterile Materialien. Sterilisieren alle Behälter, Geräte und Reagenzien vor der Verwendung. Autoklav alle Abfälle vor der Entsorgung, da sie biologisch gefährlichen betrachtet werden. Autoklaventemperatur und Zeitkombination ist ≥121 ° C x ≥15 min für alle folgenden Verfahren.
- Um ~ 1-L CASO-Agar (TSA), fügen Sie zuerst 1 L VE-Wasser in einem 2-L-Erlenmeyerkolben einen magnetischen Rührstab enthält. Verwenden einen Kolben, der mindestens zwei Mal größer ist als das Endvolumen ist. In 30 g tryptische Sojabrühe Pulver und 20 g Agar-Granulat in den Kolben.
HINWEIS: Die Verwendung von 2% Agar anstelle von 1,5% Schwärmen einiger Vibrio spp zu begrenzen.- Gut mischen, indem Sie auf das Rührwerk drehen. Unter Rühren, schalten Sie die Wärme der Mischung zum Kochen bringen. Man nimmt den Kolben von der Heizeinrichtung, sobald das Gemisch zu sieden beginnt. Lose Abdeckung the-Kolben mit Alufolie. Kleben Sie die Folie es in den Kolben vor dem Autoklaven zu sichern.
- Um tryptische Sojabrühe (TSB) machen, lassen Sie den Agar aus dem Rezept in Schritt 1.1.
HINWEIS: Flaschen anstelle von Erlenmeyer-Kolben verwenden. - Um tryptischen Soja-Agar mit 2% Natriumchlorid oder NaCl (TSAS) ergänzt, fügen Sie 20 g NaCl in dem Gemisch vor dem Rühren und Erwärmen. Um tryptischer Sojabrühe mit 2% NaCl (TSBS) ergänzt machen, lassen Sie den Agar, fügen Sie 20 g NaCl in dem Gemisch vor dem Rühren und Erwärmen.
- Um Hirn-Herz-Infusion (BHI) Agar, zu suspendieren 37 g BHI-Pulver und 15 g Agar-Granulat in 1 l gereinigtes Wasser zu machen. Erhitze unter häufigem Rühren das Pulver zu lösen. Autoklav. Lassen Sie den Agar zu BHI-Brühe zu machen.
- Um TCBS Agar, auszusetzen 89 g des TCBS Pulver in 1 l gereinigtes Wasser. Erhitze unter häufigem Rühren und kochen für 1 min vollständig um das Pulver aufzulösen. Sterilisieren nicht.
- Für alle Agar-Medien, kühlen die heißen Agar auf 45-50 ° C in einem Wasserbad. Ordnen Sie leere Petriplatten in Stapeln von fünf bis sechs Platten. Ausgehend von der Unterseite des Stapels, gießen Sie die geschmolzene Agar in jede Petrischale etwa halb voll zu erreichen. Schließen Sie die Petrischale Deckel nach dem Gießen. Lassen Sie den Agar, indem man die Platten sitzen bei Raumtemperatur zu verfestigen.
- Verwenden Sie die Agar-Platten am nächsten Tag oder nach 12 Stunden. Nicht gebrauchte Platten in einem Kühlschrank für bis zu zwei Wochen. Vor der Anwendung entfernen Platten aus dem Kühlschrank und äquilibrieren sie bei Raumtemperatur für mindestens 15 min.
HINWEIS: Ein-Liter-Agar macht ~ 45-Agar-Platten. Lassen Sie die Agarplatten ausreichend am Tag der Zubereitung zu trocknen, und äquilibrieren sie nach Kühllagerung auf Raumtemperatur, um wirksam die Ausbreitung der Kolonien reduzieren.
- Verwenden Sie die Agar-Platten am nächsten Tag oder nach 12 Stunden. Nicht gebrauchte Platten in einem Kühlschrank für bis zu zwei Wochen. Vor der Anwendung entfernen Platten aus dem Kühlschrank und äquilibrieren sie bei Raumtemperatur für mindestens 15 min.
- Erhalten Schokolade und chromogenen Agarplatten und äquilibrieren sie bei Raumtemperatur vor jedem Experiment.
- Subkultur alle 54 mikrobieller in Tabelle 1 alle paar da gezeigt Stämmeys.
- Verwenden Sie eine sterile Impföse Kulturen aus einem gefrorenen Lager oder einer früheren Charge zu nicht-selektiven Medien zu übertragen, wie BHI, TSB / TSA oder Schokolade-Agar. Wachsen halophilen Vibrio spp. auf TSBS / TSAS.
- Um zu überprüfen, um die Reinheit der Kultur, Streifen alle Stämme in einem Muster, das für die Beobachtung von isolierten Kolonien erlauben würde. Zum Beispiel verwenden ein Muster dreiphasigen Streifen eine große Menge an Bakterien zu kleineren Menge zu verdünnen, was schließlich isolierte Kolonien erhalten wurden.
- Inkubieren Sie die Platten upside-down bei 35-37 ° C bis zu 48 Stunden. Für Campylobacter spp., Röhrchen oder Platten in einem geschlossenen Deckel jar einen Gas Beutel enthält eine microaerophilic Umgebung zu erzeugen. Beobachten Koloniemorphologie nach der Inkubation. Reinkulturen sollten Kolonien ergeben, die ähnliche Kolonie Morphologie aufweisen.
HINWEIS: Inkubieren alle Platten upside-down kondensierte Wassertröpfchen auf der Unterseite des Deckels fallen auf dem col gebildet zu verhindernOnies.
2. Artbestimmung durch PCR
- Führen Sie tLH -PCR die Identität von V. zu bestätigen parahaemolyticus - Stämme. Verwendung Primern TLH -F (5 'AAA GCG GAT TAT GCA GAA GCA CTG 3') und TLH -R (5 'GCT ACT TTC TAG CAT TTT CTC TGC 3') eine 450-bp - Fragment des thermolabilen Hämolysin - Gens 20 zu amplifizieren .
- Verwenden Sie Schleife ein Sterile ein paar isolierte Kolonien von jeder V. zu übertragen parahaemolyticus Stamm von TSAS zu 5 ml TSBS. Inkubieren bei 35-37 ° C für 16-24 Std.
- Zentrifugen Kulturen bei 14.000 × g für 1 min. Überstand entfernen und wasche das Pellet zweimal mit phosphatgepufferter Salzlösung (PBS). Man kocht die Suspension 3 min Zelllysat zu erhalten.
HINWEIS: V. parahaemolyticus ist einfach aufgelöst werden. Daher Lysereagenz ist nicht erforderlich. Es ist auch möglich, ein Bit der kolonie direkt als Matrize zu verwenden. - Führen Sie die PCR in einem 25-ul-Reaktions volume. Bereiten eines Reaktionsgemisches , um eine Endkonzentration von 1 × PCR - Puffer, 1,5 mM MgCl 2, 100 & mgr; M von jedem dNTP, 1 & mgr; M jedes Primers, 1 U Taq - Polymerase und 1 & mgr; l Zelllysat enthält. Nach Vorinkubation bei 94 ° C für 5 min, laufen 35 Amplifikationszyklen bei 94 ° C für 30 sec, 58 ° C für 30 sec, und 72 ° C für 60 sec 21.
- Last Aliquots von 5 & mgr; l-Amplikon in 1,5% Agarose-Gelen. Stromversorgung einschalten Elektrophorese starten. Visualisieren der Anwesenheit oder Abwesenheit von Amplicons unter UV-Beleuchtung nach Ethidiumbromidfärbung.
3. Wachstum auf selektiven und differentiellen Medien
- Zwei bis vier Tage vor dem Experiment Streifen alle in der Tabelle gezeigt mikrobielle Stämme 1 auf nicht - selektiven Medium (TSAS, BHI oder Schokolade - Agar) zur Kolonieisolierung. Inkubiere Platten bei 35-37 ° C für 48 Stunden. Überprüfen Sie die Reinheit der Kulturen durch die Beobachtung Koloniemorphologie nach der Inkubation. Reinkulturen sollten Kolonien ergeben, die ähnliche Kolonie Morphologie aufweisen.
- Bringen Sie ein paar isolierte Kolonien aus Schritt 3.1 in 5 ml Brühe. Röhrchen bei 35-37 ° C für 16-24 Stunden.
HINWEIS: Verwenden Sie junge Kolonien, die weniger als vier Tage alt, um über Nacht Kulturen in allen Experimenten vorzubereiten. - Streak eine Öse voll über Nacht Kulturen auf selektiven und differentiellen Medien (TCBS und chromogenen Agar) zur Kolonieisolierung. Inkubiere Platten bei 35-37 ° C für bis zu 96 Stunden.
- Notieren Sie die Gesamt Wachstum aller Stämme, die durch sowohl die Kulturdichte auf der Platte zu prüfen und die Größe der isolierten Kolonien. Notieren Sie sich die Farbe der Kolonien unter der Umgebungs- und / oder UV-Licht. Hinweis andere Eigenschaften der isolierten Kolonie wie die Erhebung, Marge und Form.
4. Wiederherstellungs Assay
- Wählen Sie eine repräsentative Teilmenge von V. parahaemolyticus - Stämme (n = 14) , die verschiedenen Serotypen und Herkunft der Isolation umfassen 22. Beimpfen junge Kulturen von den Platten in 5 ml TSBS. Die Röhrchen bei 35-37 ° C für 16-24 Stunden.
- Führen Sie eine Standardplatte Zählweise der Nacht-Kulturen wie unten beschrieben.
- Vortex gut die Nacht-Kulturen zu mischen. Machen Sie eine 10-fache oder 10 -1 Verdünnung von 100 ul der Übernachtkultur in ein Röhrchen übertragen , enthaltend 900 & mgr; l PBS. Vortex gut mischen.
- Verwenden Sie eine neue Pipettenspitze 100 & mgr; l aus der 10 -1 Verdünnung Rohr in ein anderes Röhrchen zu übertragen , enthaltend 900 & mgr; l PBS. Vortex gut mischen. Das sind 10 -2 Verdünnung. Wiederholen Sie den Vorgang nacheinander 10 -7 Verdünnung zu erhalten.
- Unter Verwendung der 10 -4 bis 10 -7 Verdünnungsröhrchen, Platte 100 ul jeweils an den chromogenen, TCBS und TSAS Agarplatten.
HINWEIS: Der Verdünnungsfaktor (DF) auf der Platte wird 10 -5 bis 10 -8, respectively. - Verbreiten Sie die Aliquots gleichmäßig auf ter Agar-Oberfläche.
HINWEIS: Es ist in Ordnung denselben Spreader pro Stamm auf dem gleichen Medium zu verwenden, solange die meisten verdünnten Aliquot zuerst gespreizt wird (dh von 10 -8 bis 10 -5). Sie nicht den gleichen Spreader für unterschiedliche Medien verwenden. - Inkubiere Platten bei 35-37 ° C für bis zu 96 Stunden. Zählen Kolonien auf den Platten. Ignorieren Platten tragenden Kolonien, die zu zahlreich sind, zu zählen (TNTC) oder weniger als 25 berechnen CFU / ml nach der folgenden:
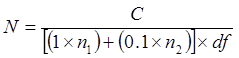
Woher
N die Anzahl der Zellen in der unverdünnten Rohr =, ausgedrückt in CFU / ml oder CFU / g
C = die Gesamtzahl der Kolonien auf den Platten gezählt , die 25-300 Kolonien ertragen
n 1 = die Anzahl der Platte (n) in dem gezählten Kolonien von der unteren df sind
n 2 = die Anzahl der Platte (n) in dem gezählten Kolonien aus der nachfolgenden 10-fache Verdünnung sind
df = die untere Verdünnungsfaktor (dh konzentrierter Verdünnung)
- Vergleichen CFU / ml unter den verschiedenen Medien. Verwenden Sie KBE / ml auf TSAS als 100%, berechnen die% Rückgewinnung von V. parahaemolyticus auf dem chromogenen und TCBS Agar gezüchtet.
5. Wettbewerb Assay
- Wählen Sie eine Teilmenge von Stämmen, die unterschiedliche Morphologien Kolonie auf dem chromogenen und TCBS Agar aufweisen.
HINWEIS: Auf diese Weise wird es möglich sein , Kolonien von V. entstanden zu zählen nur parahaemolyticus, trotz der Anwesenheit von anderen Spezies in dem Inokulum.- Wählen Sie eine V. parahaemolyticus - Stamm, der die erwarteten Türkis und Cyan - Kolonien auf TCBS und dem chromogenen Agar jeweils ergibt.
- Wählen Sie eine nicht V. parahaemolyticus und eine nicht Vibrio Spezies , die auf jedem dieser Medien nicht wächst, oder andere Koloniefarbe aufweisen.
HINWEIS: Zum Beispiel V. metschnikovii wächst sehr schwach on TCBS und wuchs nicht auf dem chromogenen Agar. Shigella sonnei wächst nicht auf TCBS sondern ergibt Magenta Kolonien auf dem chromogenen Agar.
- Nach der Auswahl der Stämme oben, bereiten über Nacht Brühekulturen unter Verwendung von isolierten Kolonien auf nicht-selektiven Medien gezüchtet.
- Machen Sie über Nacht Kulturen von V. parahaemolyticus und V. metschnikovii durch ein paar isolierte Kolonien von TSAS zu 5 ml TSBS übertragen. Inkubieren bei 35-37 ° C für 16-24 Std.
- Machen Nacht - Kulturen von Shigella sonnei durch ein paar isolierte Kolonien von BHI - Agar zu 5 ml BHI - Brühe zu übertragen. Inkubieren bei 35-37 ° C für 16-24 Std.
- Für jeden Stamm, führen Sie eine Verdünnungsreihe ähnlich den Schritten 4.2.1 und 4.2.2. Platte geeignete Verdünnungen auf TSAS oder BHI zu CFU / ml der Übernachtkultur zu bestimmen, wird die Gleichung in Schritt 4.2.5 dargestellt werden.
HINWEIS: In der Regel 100 & mgr; l von 10 -5 bis 10 -7 VerdünnungRöhren funktioniert für die meisten Kulturen. Verwenden Sie die KBE / ml Werte, die genaue Menge an Zellen im nächsten Schritt verwendet berechnen zu unterstützen. Die berechneten CFU / ml-Werte werden nach der Inkubation erhalten wird, obwohl die folgenden Schritte am selben Tag wie Schritt 5.3 durchgeführt werden. - Mit Hilfe der über Nacht Kulturen und Verdünnungsröhrchen in den Schritten 5.2 und 5.3, mischen verschiedene Mengen eines V. parahaemolyticus - Stamm und ein nicht V. parahaemolyticus Arten. Beispielsweise 500 & mgr; l der 10 -5 Verdünnungsröhrchen von V. Mischungs parahaemolyticus mit 500 ul der Übernachtkulturen von V. metschnikovii.
HINWEIS: Diese Mischung hohe Mikroflora Hintergrund simuliert. Um einen niedrigen Mikroflora Hintergrund simulieren, mischen 500 ul jeder der 10 -5 Verdünnungsröhrchen aus beiden Spezies. - Verbreiten Sie 100 ul der Mischung jeweils auf den chromogenen, TCBS und TSAS Agarplatten.
- Nach der Inkubation bei 35 bis 37 ° C bis zu 96 Stunden, zählen Kolonien von V. parahaemolyticus und die Nicht- V. parahaemolyticus Spezies auf der Grundlage ihrer Differenz in Wachstum und Morphologie der Kolonie auf dem chromogenen und TCBS Agar.
HINWEIS: Wenn beispielsweise die nicht V. parahaemolyticus Arten wächst nicht auf dem chromogenen und TCBS Agar, alle Kolonien von V. sein parahaemolyticus. Wenn der nicht V. parahaemolyticus Art wächst auf beiden Medien, nur die türkisfarbene Kolonien auf TCBS und Cyan - Kolonien auf chromogenen Agar wird von V. sein parahaemolyticus. Die nicht V. parahaemolyticus Arten aufweisen kann oder nicht ähnliche Morphologie der Kolonie bis V. parahaemolyticus auf TSAS.- Wenn der nicht V. parahaemolyticus Art wächst ähnlich wie V. parahaemolyticus in diesem nicht - selektiven Medium, teilen Sie die Koloniezahl durch zwei Zahlen für V zu erhalten parahaemolyticus nur. Vergleichen Sie die aktuelle Zählung der Kolonien mit dem erwarteten Zählwert abgeleitet aus Schritt 5.3.
6. Effjekte von Oyster Homogenate
- Wiegen ≥50 g Austernfleisch von ≥12 Weichtiere einschließlich Fleisch und Schnaps.
- In gleichen Menge PBS in die Auster Fleisch und Schnaps. Mischen Sie die Mischung bei hoher Geschwindigkeit für 90 Sekunden. Dies stellt 2 -1 verdünnt Auster Homogenat.
- Werden 100 g 2 -1 verdünnt Auster Homogenat zu 400 g PBS. Verwenden Sie eine Skala das Gewicht zu messen, nicht Volumen. Mischen Sie die Mischung bei hoher Geschwindigkeit 1 min. Autoclave die Auster Homogenat.
Hinweis: das wird die Auster Homogenat für Spick verwendet werden.
- Wiederholen Sie den Recovery-Test (Schritt 4) in Gegenwart von Auster Homogenat.
- Nach dem 500 g Austern Homogenat abkühlt, werden 100 & mgr; l V. Nacht - Kulturen parahaemolyticus in TSBS ihm gewachsen. Bestimmen Sie die tatsächliche Menge an V. parahaemolyticus Zellen in dem Inokulum durch die Standardzählverfahren berechnet 4.2 in Schritt beschrieben durch.
- Vertreteressen, um den Wettbewerb Test (Schritt 5) in Gegenwart von Auster Homogenat.
- Nachdem die 500 g kühlt Auster Homogenat, fügen Sie unten jeweils 100 ul der Übernachtkulturen von V. parahaemolyticus und nicht V. parahaemolyticus zu. Bestimmen Sie die tatsächliche Menge der Bakterien Zellen in dem Inokulum durch die Standardzählverfahren berechnet die Durchführung in Schritt 4.2 beschrieben
- Mischen Sie die Bakterienzellen mit Auster Homogenat gut unter Verwendung eines Homogenisators.
HINWEIS: Nach dem Mischen der Auster Homogenat die absichtlich zugesetzt Zellen enthält Auster Homogenat genannt wird versetzt. - Machen Verdünnungen der spiked Auster Homogenat zu erhalten 10 -1 bis 10 -3 Verdünnungsröhrchen nach den beschriebenen Verfahren in Schritt 4.2.2. Striches 100 ul jeder Verdünnung auf dem chromogenen, TCBS und TSAS Agar. Inkubiere Platten bei 35-37 ° C für bis zu 96 Stunden.
- Vergleichen Sie die aktuelle Zählung der Kolonien auf chromogenen und TCBS Agar mit dem Exteten Koloniezahl aus den Schritten 6.2.1 und 6.3.1 abgeleitet.
HINWEIS: Wenn beispielsweise ein Rohr aus V. parahaemolyticus Übernachtkultur enthält 10 8 CFU / ml, ein Inokulum von 100 & mgr; l bedeutet , dass 10 & sup7 ; Zellen in den 500-g Austern Homogenat zugesetzt werden, wodurch man 5 x 10 4 Zellen / g. Nach Verdünnung und Plattierung sollte die Platte df = 10 -2 500 Kolonien erhalten; während dieser mit df = 10 -3 sollte 50 Kolonien ergeben. Dies sind die erwarteten Koloniezahlen.
Ergebnisse
In dieser Studie wurden 54 Mikrobenstämme zusammengebaut, das in der V. 22 - Stämme enthalten parahaemolyticus Spezies Vibrio 19 andere Arten und 13 nicht Vibrio - Spezies (Tabelle 1). Die meisten V. parahaemolyticus Stämme wurden entweder von der FDA, CDC oder anderen staatlichen Gesundheitsbehörden erhalten. Sie stellen diverse Serotypen und Isolation Quellen. Diese Stämme wurden zuvor von den Aufsichtsbehörden identif...
Diskussion
Diese Studie konzentriert sich auf Kulturmedien Entwicklung und Evaluierung. Herkömmlicherweise TCBS ist die selektive und Medium zur Isolierung und Detektion V. Differential verwendet parahaemolyticus, V. cholerae und V. vulnificus 12. Es wurden jedoch Einschränkungen berichtet V. für dieses Medium, wie die Unfähigkeit , zu differenzieren cholerae aus anderen Vibrio - Spezies. Sucrose und pH-Indikator sind die Differenzierungsmittel von TCBS. Somit ve...
Offenlegungen
Some media were generously provided by Hardy Diagnostics, Santa Maria, CA. Thorsen conducted this study while a student at California Polytechnic State University. He is currently an employee of Hardy Diagnostics.
Danksagungen
Wir danken M. Channey, E. Chau, und K. Tomas für ihre Unterstützung für das Projekt. Projekt Lieferungen wurden zum Teil von der California Polytechnic State University finanziert.
Materialien
| Name | Company | Catalog Number | Comments |
| Reagent/Equipment | |||
| Agar | Fisher Scientific | DF0140-15-4 | may use other brands |
| Autoclave | Any | ||
| BHI powder | Fisher Scientific | DF0418-17-7 | may use other brands |
| Blender | Any | to blend oyster meat | |
| CampyGen gas generator | Hardy Diagnostics | CN035A | to provide a microaerophilic atmosphere; may use other brands |
| Chocolate agar plates | Hardy Diagnostics | E14 | may use other brands |
| Common PCR reagents (dNTPs, MgCl2, Taq Polymerase) | Any | or use PCR beads (Fisher Sci 46-001-014) | |
| Culture tubes | Fisher Scientific | S50712 | may use other brands |
| Eppendorf tubes | Fisher Scientific | S348903 | may use other brands |
| Gel doc | Any | ||
| HardyChrom Vibrio agar plates | Hardy Diagnostics | G319 | This study evaluates this medium |
| Incubator | Any | ||
| Inoculating loops | Fisher Scientific | 22-363-606 | 10 microliter-size was used in this study |
| NaCl | Fisher Scientific | BP358-212 | may use other brands |
| Oysters | Any | ||
| PBS | Fisher Scientific | R23701 | may use other brands |
| Petri dish | Fisher Scientific | FB0875713 | may use other brands |
| Pipette and tips | Any | Sterilized tips | |
| Primers for tlh | IDT DNA | ||
| Scale | Any | ||
| Spreader | Fisher Scientific | 08-100-11 | Beads may be used instead |
| Stomacher blender | Stomacher | 400 | Samples were homogenized at 200 rpm for 30 sec. Other homogenizer can be used. |
| Sterile filter bags for blenders | Fisher Scientific | 01-812-5 | |
| TCBS powder | Hardy Diagnostics | 265020 | This study evaluates this medium |
| Thermocycler | Any | ||
| TSB powder | Fisher Scientific | DF0370-07-5 | may use other brands |
| UV viewing cabinet | Any | Emit long-wave UV light | |
| Water bath | Any | | |
| Name | Sources | Catalog Number | Comments |
| Bacterial species and strains | |||
| Aeromonas hydrophila | ATCC | ||
| Candida albicans | ATCC | ||
| Campylobacter jejuni | ATCC | ||
| Escherichia coli | ATCC | ||
| Proteus mirabilis | ATCC | ||
| Pseudomonas aeruginosa | ATCC | ||
| Staphylococcus aureus | ATCC | ||
| Salmonella Choleraesuis | ATCC | ||
| Shigella boydii | ATCC | ||
| Shigella flexneri | ATCC | ||
| Shigella sonnei | ATCC | ||
| Vibrio alginolyticus | ATCC | ||
| V. cholerae (serotypes include O139, O1, non O1, El Tor biovars) | FDA, ATCC | ||
| V. damsela | FDA | ||
| V. fisheri | Environment | ||
| V. fluvialis | CDC | ||
| V. furnissii | CDC | ||
| V. hollisae | FDA | ||
| V. metschnikovii | ATCC | ||
| V. mimicus | FDA | ||
| V. parahaemolyticus (serotypes include O3:K6, O1:K56, O4:K8, O5:K15, O8, etc.) | ATCC, FDA, CDC, Environment | ||
| V. proteolyticus | FDA | ||
| V. vulnificus | FDA |
Referenzen
- Yeung, P. S., Boor, K. J. Epidemiology, pathogenesis, and prevention of foodborne Vibrio parahaemolyticus infections. Foodborne Pathog. Dis. 1 (2), 74-88 (2004).
- Yeung, P. S. M., Boor, K. J., Faruque, S. M. Epidemiology, molecular biology, and detection of foodborne Vibrio parahaemolyticus infections. Foodborne and Waterborne Bacterial pathogens: Epidemiology, Evolution and Molecular Biology. , 153-184 (2012).
- Centers for Disease Control and Prevention. Vital Signs: Incidence and Trends of Infection with Pathogens Transmitted Commonly Through Food - Foodborne Diseases Active Surveillance Network, 10 U.S. Sites, 1996-2010. Morbidity and Mortality Weekly Report. 10, 1996-2010 (2011).
- . Summary of human Vibrio cases reported to CDC, 2008 Available from: https://stacks.cdc.gov/view/cdc/21591 (2008)
- Scallan, E., et al. Foodborne illness acquired in the United States - major pathogens. Emerg. Infect. Dis. 17 (1), 7-15 (2011).
- Baross, J., Liston, J. Occurrence of Vibrio parahaemolyticus and related hemolytic vibrios in marine environments of Washington State. Appl. Microbiol. 20 (2), 179-186 (1970).
- Baross, J., Liston, J. Isolation of Vibrio parahaemolyticus from the Northwest Pacific. Nature. 217 (5135), 1263-1264 (1968).
- Kueh, C. S., Chan, K. Y. Bacteria in bivalve shellfish with special reference to the oyster. J. Appl. Bacteriol. 59 (1), 41-47 (1985).
- Food and Drug Administration. . Bad Bug Book, Foodborne Pathogenic Microorganisms and Natural Toxins. , (2012).
- Qadri, F., et al. Adaptive and inflammatory immune responses in patients infected with strains of Vibrio parahaemolyticus. J. Infect. Dis. 187 (7), 1085-1096 (2003).
- MacFaddin, J. F. . Media for isolation-cultivation-identification-maintenance of medical bacteria. 1, (1985).
- Bottone, E. J., Robin, T. Vibrio parahaemolyticus: suspicion of presence based on aberrant biochemical and morphological features. J. Clin. Microbiol. 8 (6), 760-763 (1978).
- Lotz, M. J., Tamplin, M. L., Rodrick, G. E. Thiosulfate-citrate-bile salts-sucrose agar and its selectivity for clinical and marine vibrio organisms. Ann. Clin. Lab. Sci. 13 (1), 45-48 (1983).
- Hara-Kudo, Y., Nishina, T., Nakagawa, H., Konuma, H., Hasegawa, J., Kumagai, S. Improved method for detection of Vibrio parahaemolyticus in seafood. Appl. Environ. Microbiol. 67 (12), 5819-5823 (2001).
- Eddabra, R., Piemont, Y., Scheftel, J. M. Evaluation of a new chromogenic medium, chromID Vibrio, for the isolation and presumptive identification of Vibrio choleare and Vibrio parahaemolyticus from human clinical specimens. Eur. J. Clin. Microbiol. Infect. Dis. 30 (6), 733-737 (2011).
- Kodaka, H., Teramura, H., Mizuochi, S., Saito, M., Matsuoka, H. Evaluation of the Compact Dry VP method for screening raw seafood for total Vibrio parahaemolyticus. J. Food. Prot. 72 (1), 169-173 (2009).
- Su, Y. C., Duan, J., Wu, W. H. Selectivity and specificity of a chromogenic medium for detecting Vibrio parahaemolyticus. J. Food Prot. 68 (7), 1454-1456 (2005).
- Bej, A. K., Patterson, D. P., Brasher, C. W., Vickery, M. C., Jones, D. D., Kaysner, C. A. Detection of total and hemolysin-producing Vibrio parahaemolyticus in shellfish using multiplex PCR amplification of tl, tdh and trh. J. Microbiol. Methods. 36 (3), 215-225 (1999).
- Yeung, P. S. M., DePaola, A., Kaysner, C. A., Boor, K. J. A PCR assay for specific detection of the pandemic Vibrio parahaemolyticus O3:K6 clone from shellfish. J. Food Sci. 68 (4), 1459-1466 (2003).
- Yeung, P. S. M., Hayes, M. C., DePaola, A., Kaysner, C. A., Kornstein, L., Boor, K. J. Comparative phenotypic, molecular, and virulence characterization of Vibrio parahaemolyticus O3:K6 isolates. Appl. Environ. Microbiol. 68 (6), 2901-2909 (2002).
- Duan, J., Su, Y. -. C. Comparison of a chromogenic medium with thiosulfate-citrate-bile salts-sucrose agar for detecting Vibrio parahaemolyticus. J. Food Sci. 70, M125-M128 (2005).
- Pinto, A. D., Terio, V., Novello, L., Tantillo, G. Comparison between thiosulphate-citrate-bile salt sucrose (TCBS) agar and CHROMagar Vibrio for isolating Vibrio parahaemolyticus. Food Control. 22 (1), 124-127 (2011).
- Canizalez-Roman, A., Flores-Villaseñor, H., Zazueta-Beltran, J., Muro-Amador, S., Leòn-Sicairos, N. Comparative evaluation of a chromogenic agar medium-PCR protocol with a conventional method for isolation of Vibrio parahaemolyticus strains from environmental and clinical samples. Can J Microbiol. 57 (2), 136-142 (2011).
- Kriem, M. R., et al. Prevalence of Vibrio spp. in raw shrimps (Parapenaeus longirostris) and performance of a chromogenic medium for the isolation of Vibrio strains. Lett Appl Microbiol. 61 (3), 224-230 (2015).
- Food Drug Administration. Statistical guidance on reporting results from studies evaluating diagnostic tests. http://www.fda.gov/RegulatoryInformation/Guidances/ucm071148.htm. , (2007).
- Burd, E. M. Validation of laboratory-developed molecular assays for infectious diseases. Clin Microbiol Rev. 23 (3), 550-576 (2010).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten