Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Développement d'un plus sensible et spécifique chromogénique Agar moyen pour la détection de
Dans cet article
Résumé
Detection and isolation of clinically relevant Vibrio species require selective and differential culture media. This study evaluated the ability of a new chromogenic medium to detect and identify V. parahaemolyticus and other related species. The new medium was found to have better sensitivity and specificity than the conventional medium.
Résumé
Les infections d'origine alimentaire aux États - Unis causée par les espèces de Vibrio ont montré une tendance à la hausse. Dans le genre Vibrio, V. parahaemolyticus est responsable de la majorité des infections -Associated Vibrio. Ainsi, la différenciation précise entre les Vibrio spp. et la détection de V. parahaemolyticus est d'une importance cruciale pour assurer la sécurité de notre approvisionnement alimentaire. Bien que les techniques moléculaires sont de plus en plus commun, des méthodes de culture-fonction sont toujours systématiquement fait et ils sont considérés comme des méthodes standard dans certaines circonstances. Par conséquent, un nouveau milieu de gélose chromogène a été testé avec le but de fournir une meilleure méthode pour l' isolement et la différenciation des Vibrio spp cliniquement pertinente. Le protocole par rapport à la limite de sensibilité, de spécificité et de détection pour la détection de V. parahaemolyticus entre le nouveau milieu chromogène et un milieu classique. Divers V. souches parahaemolyticus (n = 22) représentant divers sérotypes et source d'origine ont été utilisés. Ils ont déjà été identifiés par la Food and Drug Administration (FDA) et des Centers for Disease Control and Prevention (CDC), et en outre vérifié dans notre laboratoire par tlh -PCR. Au moins quatre essais séparés, ces souches ont été inoculées sur l'agar et du thiosulfate citrate sels biliaires chromogènes-saccharose (TCBS) d'agar-agar, ce qui est le support recommandé pour la culture de cette espèce, suivie d'une incubation à 35-37 ° C pendant 24 -96 h. Trois V. souches parahaemolyticus (13,6%) ne se développent pas de façon optimale sur TCBS, néanmoins exposées colonies vertes s'il y avait une croissance. Deux souches (9,1%) n'a pas donné les colonies cyan attendus sur la gélose chromogène. V. Non- souches parahaemolyticus (n = 32) ont également été testés pour déterminer la spécificité de la gélose chromogénique. Parmi ces souches, 31 ne poussent pas ou présentaient d'autres morphologies de colonies. La récupération moyenne de V. parahaemolyticus sur le chromogenic agar était d'environ 96,4% par rapport à la gélose trypticase soja complémenté avec 2% de NaCl. En conclusion, le nouveau gélose chromogénique est un moyen efficace pour détecter V. parahaemolyticus et pour le différencier des autres vibrions.
Introduction
En tant que membre du genre Vibrio, V. parahaemolyticus est une, courbe, bactérie en forme de bâtonnet à Gram négatif, non sporulé. Il présente une grande mobilité dans les deux milieux liquides et semi-solides. La plupart V. souches parahaemolyticus sont non pathogènes pour l' homme, mais les sous - types pathogènes ont provoqué des épidémies et des pandémies, d' où cette espèce est considérée comme une importante pathogène d'origine alimentaire dans de nombreux pays 1,2. L'incidence de l' infection par Vibrio aux États - Unis a montré une tendance à la hausse depuis 2000 3. Parmi Vibrio spp., V. parahaemolyticus est l'espèce la plus fréquemment rapportés causant des maladies aux États - Unis 4,5. D' autres espèces cliniquement pertinentes comprennent V. alginolyticus, V. vulnificus, V. cholerae, etc. Un petit pourcentage des maladies est due à la fois par de multiples espèces.
V. parahaemolyticus est un i naturelnhabitant de l'eau de mer et donc largement distribué dans les eaux marines à travers le monde, y compris les estuaires. L'espèce a été découverte en 1950 suite à une épidémie d'intoxication alimentaire au Japon. Aux Etats - Unis, l'espèce a été isolée d' abord dans l' eau de mer, des sédiments et des mollusques dans la région de Puget Sound 6,7. Filtreurs dans les habitats marins, tels que les mollusques bivalves, peuvent abriter V. parahaemolyticus dans le cadre de leur flore naturelle 8. En tant que tel, V. infections parahaemolyticus chez l' homme sont souvent liés à la consommation de fruits de mer contaminés, en particulier les fruits de mer crus ou pas assez cuits. Un itinéraire moins commun d'entrée se produit lorsque plaie ouverte est exposée à l'eau de mer, ce qui conduit à une infection de la peau. La plupart V. souches parahaemolyticus ne causent pas la maladie humaine, mais certains sous - types hébergeant des facteurs de virulence tels que hémolysine directe thermostable (TDH) sont pathogènes. Les symptômes les plus fréquents de V. d' origine alimentaire infection parahaemolyticus sontla diarrhée et des douleurs abdominales, suivie par des nausées, des vomissements et de la fièvre. Maux de tête et des frissons sont également signalés. La période d'incubation moyenne est de 15 heures, mais peut aller jusqu'à 96 heures après la consommation d' une quantité suffisante de souches pathogènes 9. La maladie dure de deux à trois jours. Les symptômes de gastro - entérite causée par V. parahaemolyticus sont en grande partie auto-limitation et le traitement spécial donc est pas nécessaire. Des cas bénins de la gastro-entérite peuvent être traités efficacement par réhydratation orale. Plus graves maladies peuvent être traitées par des antibiotiques tels que la tetracycline ou la ciprofloxacine 10. Le taux de mortalité est d'environ 2% pour les cas de gastro-entérite, mais peut être aussi élevé que 29% pour ceux qui développent une infection sanguine ou une septicémie. Toute personne qui consomme des fruits de mer ou a une plaie ouverte exposée à l' eau de mer est à risque de V. infection parahaemolyticus. La forme la plus grave des maladies, la septicémie mortelle, est plus fréquente chez une sous-population avec co médicale sous-jacentenditions 11, qui comprennent l' alcoolisme, une maladie hépatique, le diabète, une maladie rénale, une affection maligne, et d' autres conditions conduisant à une réponse immunitaire affaibli. Notamment, ce groupe de personnes est également à un risque plus élevé de contracter des maladies graves causées par V. vulnificus, qui peut être trouvé dans les habitats naturels similaires à V. parahaemolyticus.
V. parahaemolyticus est régulièrement isolé en utilisant des sels-saccharose thiosulfate-citrate-biliaires (TCBS) agar comme milieu sélectif et différentiel. Enrichissement en eau peptonée alcaline peut précéder l'isolement sur gélose TCBS. Les colonies sur TCBS sont ensuite testés dans une gamme de tests biochimiques et / ou des tests moléculaires ciblant la présence de gènes spécifiques à l'espèce. Les méthodes basées sur la PCR sont souvent utilisés pour confirmer l'identité de V. parahaemolyticus en amplifiant le gène hémolysine thermolabile, tlh 12.
Quelle que soit la choix des méthodes de confirmation, il est important de disposer d' un moyen efficace pour isoler et de différencier V. parahaemolyticus d'autres vibrions marins en premier lieu. TCBS a régulièrement été utilisé pour différencier les espèces dans le genre Vibrio selon leurs capacités à fermenter le saccharose 12. Réaction de fermentation positive est accompagnée par un changement de couleur de l'indicateur de pH bleu de bromothymol. V. colonies parahaemolyticus sont assez distinctif sur TCBS, présentant le bleu à la couleur verte. Cependant, ce moyen ne peut pas différencier facilement V. alginolyticus et V. cholerae. Proteus sucrose-fermentation peuvent produire des colonies jaunes ressemblant à V. cholerae ou V. 13 alginolyticus. Sur l' isolement initial sur TCBS, V. parahaemolyticus peut également être mal identifié comme Aeromonas hydrophila, Plesiomonas shigelloides et Pseudomonas spp 14. Les souches avec un retard ferm saccharoseentation peut être confondue avec d' autres saccharose fermentant Vibrio 13, qui comprennent V. parahaemolyticus. TCBS a été jugée non sensible contre Escherichia coli, Pseudomonas putrefaciens, entre autres. Plusieurs autres espèces donnent vert à des colonies grises qui sont potentiellement confondus avec V. parahaemolyticus ou V. vulnificus 15. Par conséquent, il est souhaitable de développer des milieux de culture alternative avec une meilleure sensibilité et une spécificité envers la détection et l' isolement de V. parahaemolyticus et d' autres espèces étroitement apparentées.
Plusieurs alternatives des médias ont été récemment mis au point. En plus de l'incorporation d'agents sélectifs, la plupart incorporent des substrats chromogènes pour différencier les espèces en fonction de leurs activités enzymatiques différentielles. Par exemple, l' indoxyl-β-glucoside et le β-indoxyl galactoside ont été utilisés comme substrats chromogènes pour différencier V. paracolonies de haemolyticus (qui apparaissent bleu-vert) à partir de ceux de V. cholerae (violet) en raison de leurs capacités différentielles pour produire β-glucosidase et β-galactosidase 16. Différentes formulations de gélose chromogène développées par plusieurs groupes ont été évalués et ont été signalés à effectuer comparable ou supérieure à TCBS 17,18,19. Un avantage d'utiliser un milieu chromogène est que la coloration du milieu environnant est minime, ce qui facilite l'isolement de colonies particulières. Dans cette étude, nous avons évalué la capacité d'un milieu chromogénique nouvellement formulé pour détecter et isoler V. cholerae, V. parahaemolyticus, et V. vulnificus; avec un accent particulier sur sa capacité à différencier V. parahaemolyticus provenant d' autres espèces.
Protocole
1. Médias et culture des souches microbiennes
NOTE: Utiliser des techniques aseptiques dans toutes les expériences. Utiliser des matériaux stériles. Stériliser tous les conteneurs, les outils et réactifs avant utilisation. Autoclave tous les déchets avant leur élimination, car ils sont considérés comme un risque biologique. la température et le temps Autoclave combinaison est ≥121 ° C x ≥15 min pour toutes les procédures suivantes.
- Pour faire ~ 1-L de gélose trypticase soja (TSA), ajouter d'abord 1 L d'eau déminéralisée dans un 2-L Erlenmeyer contenant un barreau d'agitation magnétique. Utiliser un flacon qui est au moins deux fois plus grand que le volume final. Ajouter 30 g de poudre de bouillon trypsique de soja et 20 g d'agar granulés dans le ballon.
REMARQUE: Utiliser 2% de gélose au lieu de 1,5% pour limiter l' essaimage de certains Vibrio spp.- Bien mélanger en tournant sur l'agitateur. Tout en agitant, allumer la chaleur pour faire bouillir le mélange. Retirer le ballon de l'appareil de chauffage dès que le mélange commence à bouillir. Couvrir légèrement the flacon avec une feuille d'étain. Collez la feuille pour le fixer dans le ballon avant de l'autoclave.
- Pour faire du bouillon de soja tryptique (TSB), omettre l'agar de la recette à l'étape 1.1.
NOTE: Peut utiliser des bouteilles au lieu de Erlenmeyer de. - Pour faire de la gélose trypticase soja complémenté avec du chlorure de sodium à 2% et du NaCl (TSAS), ajouter 20 g de NaCl dans le mélange avant l'agitation et le chauffage. Pour rendre le bouillon trypticase soja complémenté avec 2% de NaCl (TSBS), omettre la gélose, ajouter 20 g de NaCl dans le mélange avant l'agitation et le chauffage.
- Pour perfusion cardiaque du cerveau (BHI) agar, suspendre 37 g de poudre BHI et 15 g granulés de gélose dans 1 litre d'eau purifiée. Chauffer en agitant fréquemment pour dissoudre la poudre. Autoclave. Omettre la gélose à faire du bouillon BHI.
- Pour rendre la gélose TCBS, suspendre 89 g de la poudre TCBS dans 1 litre d'eau purifiée. Chauffer en agitant fréquemment et faire bouillir pendant 1 min pour dissoudre complètement la poudre. Ne pas autoclaver.
- Pour tous les médias agar, refroidir l'agar chaud pour 45-50 ° C dans un bain d'eau. Disposez vides boîtes de Petri dans des piles de cinq à six plaques. En partant du bas de la pile, verser la gélose fondue dans chaque boîte de Petri pour atteindre environ la moitié. Fermer le couvercle de boîte de Pétri après la coulée. Laisser la gélose se solidifier en laissant les plaques sont assis à la température ambiante.
- Utilisez les plaques d'agar le lendemain ou après 12 h. Stocker les plaques inutilisées dans un réfrigérateur jusqu'à deux semaines. Avant utilisation, enlever les plaques du réfrigérateur et équilibrer les à température ambiante pendant au moins 15 min.
REMARQUE: Un litre d'agar fait ~ 45 plaques de gélose. Laisser les plaques de gélose à sécher suffisamment le jour de leur préparation, et leur équilibrent à la température ambiante après un stockage à froid pour réduire efficacement la propagation des colonies.
- Utilisez les plaques d'agar le lendemain ou après 12 h. Stocker les plaques inutilisées dans un réfrigérateur jusqu'à deux semaines. Avant utilisation, enlever les plaques du réfrigérateur et équilibrer les à température ambiante pendant au moins 15 min.
- Obtenir des plaques de gélose chocolat et chromogénique et équilibrer les à température ambiante avant chaque expérience.
- Repiquer les 54 souches microbiennes indiquées dans le tableau 1 tout petit nombre days.
- Utilisez une boucle d'inoculation stérile pour transférer les cultures à partir d'un stock congelé ou un lot précédent aux médias non sélectifs tels que BHI, TSB / TSA ou gélose chocolat. Cultivez halophile Vibrio spp. sur TSBS / TSAS.
- Pour vérifier la pureté de la culture, strie toutes les souches dans un modèle qui permettrait l'observation des colonies isolées. Par exemple, utiliser un motif de stries en trois phases pour diluer une grande quantité de bactéries plus petite quantité, donnant finalement des colonies isolées.
- Incuber les plaques à l'envers à 35-37 ° C pendant jusqu'à 48 heures. Pour Campylobacter spp., Incuber des tubes ou des plaques dans un bocal couvercle fermé contenant une poche de gaz pour produire un environnement microaérophile. Observez la morphologie des colonies après incubation. Les cultures pures devraient produire des colonies qui présentent une colonie semblable morphologie.
REMARQUE: Incuber les plaques à l'envers pour empêcher des gouttelettes d'eau condensée formée sur la face inférieure du couvercle de tomber sur le colOnies.
2. Espèces Détermination par PCR
- Mener tlh -PCR pour confirmer l'identité de V. souches parahaemolyticus. Utilisez amorces Tlh -F (5 'AAA GCG GAT TAT GCA GAA GCA CTG 3') et tlh -R (5 'GCT ACT TTC TAG CAT TTT CTC TGC 3') pour amplifier un fragment de 450 pb du gène thermolabile hémolysine 20 .
- Utilisez une boucle d'inoculation stérile pour transférer quelques colonies isolées de chaque V. souche parahaemolyticus de TSAS à 5 ml de TSBS. On incube à 35-37 ° C pendant 16-24 h.
- cultures Centrifugeuse à 14.000 xg pendant 1 min. Retirer le surnageant et on lave le culot deux fois avec du tampon phosphate salin (PBS). Faire bouillir la suspension pendant 3 min pour obtenir un lysat cellulaire.
NOTE: V. parahaemolyticus est facile d'être lysées. Par conséquent, le réactif de lyse est pas nécessaire. Il est également possible d'utiliser un peu de colonies directement comme matrice. - Faire la PCR dans une réaction de 25 ul volume. Préparer un mélange réactionnel contenant une concentration finale de 1 x tampon PCR, MgCl2 1,5 mM, 100 uM de chaque dNTP, 1 pM de chaque amorce, 1 U de Taq polymérase et 1 pi de lysat cellulaire. Après une pré - incubation à 94 ° C pendant 5 min, course de 35 cycles d'amplification à 94 ° C pendant 30 secondes, 58 ° C pendant 30 secondes et 72 ° C pendant 60 s 21.
- aliquotes de charge de amplicon 5 pi à 1,5% de gels d'agarose. Allumez l'alimentation pour démarrer l'électrophorèse. Visualiser la présence ou l'absence d'amplicons sous illumination UV après coloration au bromure d'éthidium.
3. Croissance sur sélectif et différentiel des médias
- Deux à quatre jours avant l'expérience, strie toutes les souches microbiennes indiquées dans le tableau 1 sur un milieu non sélectif (TSAS, BHI ou gélose chocolat) pour l' isolement de la colonie. Incuber les plaques à 35-37 ° C pendant 48 heures. Vérifiez la pureté des cultures en observant la morphologie des colonies après incubation. Les cultures pures devraient produire des colonies qui présentent une colonie semblable morphologie.
- Transfert quelques colonies isolées de l'étape 3.1 dans 5 ml de bouillon. Incuber les tubes à 35-37 ° C pendant 16-24 h.
NOTE: Utiliser les jeunes colonies, qui sont âgés de moins de quatre jours, pour préparer cultures d'une nuit dans toutes les expériences. - Streak une anse de cultures d'une nuit sur les médias sélectif et différentiel (TCBS et gélose chromogénique) pour l'isolement de la colonie. Incuber les plaques à 35-37 ° C pendant jusqu'à 96 heures.
- Notez la croissance globale de toutes les souches en examinant à la fois la densité de la culture sur la plaque et la taille des colonies isolées. Notez la couleur des colonies sous la lumière ambiante et / ou UV. Notez d'autres caractéristiques de colonie isolée tels que l'élévation, la marge et la forme.
4. Essai de récupération
- Sélectionnez un sous - ensemble représentatif de V. souches parahaemolyticus (n = 14) qui englobent les différents sérotypes d'isolement et d' origines 22. Inoculer jeunes cultures des plaques dans 5 ml de TSBS. Incuber les tubes à 35-37 ° C pendant 16-24 h.
- Procéder à une méthode de comptage sur plaque standard des cultures d'une nuit comme décrit ci-dessous.
- Vortex à bien mélanger les cultures d'une nuit. Faire un facteur 10 ou 10 -1 dilution en transférant 100 pl de la culture pendant une nuit dans un tube contenant 900 ul de PBS. Vortex pour bien mélanger.
- Utiliser un nouvel embout de pipette pour transférer 100 ul du tube 10 -1 de dilution dans un autre tube contenant 900 ul de PBS. Vortex pour bien mélanger. Ceci constitue 10 -2 dilution. Répétez la séquence de processus pour obtenir 10 -7 dilution.
- En utilisant les 10 -4 à 10 -7 tubes de dilution, la plaque 100 ul chacun sur les, des TCB et TSAS plaques d' agar chromogènes.
NOTE: Le facteur de dilution (DF) sur la plaque devient 10 -5 à 10 -8, respectivement. - Répartir les aliquotes uniformément sur til Agar surface.
NOTE: Il est bon d'utiliser la même épandeur par souche sur le même milieu, aussi longtemps que la partie aliquote plus diluée est répartie en premier (ie, de 10 -8 à 10 -5). Ne pas utiliser le même épandeur pour différents médias. - Incuber les plaques à 35-37 ° C pendant jusqu'à 96 heures. Compter les colonies sur les plaques. Ignorer les plaques portant les colonies qui sont trop nombreuses pour être comptées (incomptable) ou inférieur à 25. Calculer CFU / ml selon les critères suivants:
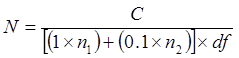
Où
N = le nombre de cellules dans le tube non dilué, exprimé en UFC / ml ou UFC / g
C = le nombre total de colonies comptées sur des plaques qui portent 25-300 colonies
n 1 = le nombre de plaque (s) où les colonies sont comptées à partir de la df inférieure
n 2 = le nombre de plaque (s) où les colonies sont comptées à partir de la dilution de 10 fois ultérieur
réf = le facteur de dilution plus faible ( par exemple, une dilution plus concentrée)
- Comparer UFC / ml entre les différents médias. Utilisez UFC / ml sur TSAS 100%, calculer la récupération% de V. parahaemolyticus cultivé sur la gélose chromogène et TCBS.
5. Concurrence Assay
- Choisissez un sous-ensemble des souches qui présentent des morphologies différentes de colonies sur la gélose chromogène et TCBS.
NOTE: De cette façon, il sera possible de compter les colonies provenaient de V. parahaemolyticus que, malgré la présence d'autres espèces dans l'inoculum.- Choisissez un V. souche parahaemolyticus qui donne les turquoises et cyan colonies attendues sur TCBS et la gélose chromogène, respectivement.
- Choisissez un V. non parahaemolyticus et un non - espèces de Vibrio qui ne poussent pas sur l' un de ces supports, ou présentent une couleur différente de la colonie.
NOTE: Par exemple, V. metschnikovii croît très faiblement on TCBS et n'a pas grandi sur la gélose chromogène. Shigella sonnei ne pousse pas sur TCBS mais donne des colonies magenta sur la gélose chromogène.
- Après sélection des souches ci-dessus, de préparer des bouillons de culture d'une nuit en utilisant des colonies isolées cultivées sur des milieux non sélectifs.
- Faire cultures d'une nuit de V. parahaemolyticus et V. metschnikovii en transférant quelques colonies isolées à partir TSAS à 5 ml de TSBS. On incube à 35-37 ° C pendant 16-24 h.
- Faire cultures d'une nuit de Shigella sonnei en transférant quelques colonies isolées de gélose BHI à 5 ml de bouillon BHI. On incube à 35-37 ° C pendant 16-24 h.
- Pour chaque souche, effectuer une série de dilution similaire aux étapes 4.2.1 et 4.2.2. Plaque des dilutions appropriées sur TSAS ou BHI pour déterminer UFC / ml de la culture pendant une nuit, en utilisant l'équation illustrée à l'étape 4.2.5.
NOTE: En règle générale, 100 ul de la 10 -5 à 10 -7 dilutiontubes fonctionne pour la plupart des cultures. Utilisez les valeurs / ml UFC à dos calculer le montant exact des cellules utilisées dans l'étape suivante. Les valeurs UFC / ml calculées sont obtenues après incubation, bien que les étapes suivantes sont exécutées à la même date que dans l'étape 5.3. - En utilisant les cultures de la nuit et les tubes de dilution dans les étapes 5.2 et 5.3, mélanger différentes quantités d'un V. souche parahaemolyticus et V. non espèces parahaemolyticus. Par exemple, mélanger 500 pi du tube 10 -5 dilution de V. parahaemolyticus avec 500 pi des cultures d'une nuit de V. metschnikovii.
NOTE: Ce mélange simule un bruit de fond de la microflore. Pour simuler un faible bruit de fond de la microflore, mélanger 500 pi de chacun des tubes 10 -5 dilution de ces deux espèces. - Étaler 100 pi du mélange chacun des, des TCB et TSAS plaques d'agar chromogènes.
- Après incubation à 35-37 ° C pendant jusqu'à 96 heures, compter les colonies de V. parahaemolyticus et non V. espèces parahaemolyticus en fonction de leur différence dans la croissance et la morphologie des colonies sur la gélose chromogène et TCBS.
NOTE: Par exemple, si le V. non espèces parahaemolyticus ne poussent pas sur la gélose chromogène et TCBS, toutes les colonies seront de V. parahaemolyticus. V. Si la non - espèces parahaemolyticus pousse sur les médias, que les colonies de turquoise sur TCBS et cyan colonies sur gélose chromogène seront de V. parahaemolyticus. V. non espèces parahaemolyticus peuvent ou non présenter une colonie semblable à la morphologie V. parahaemolyticus sur TSAS.- V. Si la non - les espèces parahaemolyticus croît de manière similaire à V. parahaemolyticus sur ce milieu non sélectif, diviser le nombre de colonies par deux pour obtenir des numéros pour V. parahaemolyticus seulement. Comparez le nombre de colonies réelle avec le nombre attendu dérivé de l'étape 5.3.
6. Effète de Oyster homogénats
- Peser ≥50 g huîtres viande de ≥12 mollusques, y compris la viande et l'alcool.
- Ajouter quantité égale de PBS à la viande d'huître et de la liqueur. Mélanger le mélange à grande vitesse pendant 90 secondes. Ceci constitue 2 -1 dilué homogénat d'huîtres.
- Ajouter 100 g de 2 -1 dilué homogénat d'huîtres à 400 g de PBS. Utilisez une échelle pour mesurer le poids, pas de volume. Mélanger le mélange à grande vitesse pendant 1 min. Autoclave le broyat d'huîtres.
NOTE: Ce sera le broyat d'huîtres utilisé pour dopage.
- Répétez le test de récupération (étape 4) en présence d'huîtres homogénat.
- Après les 500 g huîtres homogénat refroidit, ajouter 100 pi de V. parahaemolyticus cultures d'une nuit cultivées dans TSBS à elle. Déterminer le montant réel de V. cellules parahaemolyticus dans l'inoculum en effectuant les procédures de comptage des plaques standard décrites à l' étape 4.2.
- Représentantmanger le dosage de la concurrence (étape 5) en présence d'huîtres homogénat.
- Après les 500 g huîtres homogénat refroidit, ajouter 100 ul chacune des cultures d'une nuit de V. parahaemolyticus et V. non parahaemolyticus à elle. Déterminer la quantité réelle de bactéries cellules dans l'inoculum en effectuant les procédures de comptage des plaques standard décrit dans l'étape 4.2
- Mélanger les cellules bactériennes avec des huîtres homogénat bien à l'aide d'un homogénéisateur.
NOTE: Après le mélange, le broyat d'huîtres contenant les cellules ajoutées intentionnellement est appelé à pointes huîtres homogénat. - Faire des dilutions de l'huître homogénat à pointes pour obtenir 10 -1 à 10 -3 tubes de dilution selon les procédures décrites à l' étape 4.2.2. Étaler 100 pi de chaque dilution sur le, TCBS et TSAS agar chromogène. Incuber les plaques à 35-37 ° C pendant jusqu'à 96 heures.
- Comparez le nombre de colonies réelle sur gélose chromogène et TCBS avec l'exinatten- comptage des colonies déduit étapes 6.2.1 et 6.3.1.
NOTE: Par exemple, si un tube de V. culture d'une nuit parahaemolyticus contient 10 8 CFU / ml, un inoculum de 100 ul signifie que 10 7 cellules sont ajoutés à 500 g d'homogénat huître, ce qui donne 5 x 10 4 cellules / g. Après dilution et le placage, la plaque ayant df = 10 -2 devrait donner 500 colonies; tandis que d' avoir df = 10 -3 devrait produire 50 colonies. Ce sont les comptes de colonies attendues.
Résultats
Dans cette étude, 54 souches microbiennes ont été assemblés, qui comprenait 22 souches dans le V. espèces parahaemolyticus, 19 autres espèces de Vibrio et 13 espèces de Vibrio non (tableau 1). La plupart V. souches parahaemolyticus ont été soit reçu de la FDA, le CDC ou d' autres services de santé de l' Etat. Ils représentent divers sérotypes et sources d'isolement. Ces souches ont été ident...
Discussion
Cette étude se concentre sur le développement et l'évaluation des milieux de culture. Classiquement, TCBS est sélectif et différentiel moyen utilisé pour isoler et détecter V. parahaemolyticus, V. cholerae et V. vulnificus 12. Cependant, les limites ont été rapportées pour ce milieu, comme l'incapacité de différencier V. cholerae d'autres espèces de Vibrio. Le saccharose et un indicateur de pH sont les agents de différenciation de TCB. Ainsi, la pr...
Déclarations de divulgation
Some media were generously provided by Hardy Diagnostics, Santa Maria, CA. Thorsen conducted this study while a student at California Polytechnic State University. He is currently an employee of Hardy Diagnostics.
Remerciements
Nous remercions M. Channey, E. Chau, et K. Tomas pour leur aide sur le projet. fournitures de projets ont été financés en partie par California Polytechnic State University.
matériels
| Name | Company | Catalog Number | Comments |
| Reagent/Equipment | |||
| Agar | Fisher Scientific | DF0140-15-4 | may use other brands |
| Autoclave | Any | ||
| BHI powder | Fisher Scientific | DF0418-17-7 | may use other brands |
| Blender | Any | to blend oyster meat | |
| CampyGen gas generator | Hardy Diagnostics | CN035A | to provide a microaerophilic atmosphere; may use other brands |
| Chocolate agar plates | Hardy Diagnostics | E14 | may use other brands |
| Common PCR reagents (dNTPs, MgCl2, Taq Polymerase) | Any | or use PCR beads (Fisher Sci 46-001-014) | |
| Culture tubes | Fisher Scientific | S50712 | may use other brands |
| Eppendorf tubes | Fisher Scientific | S348903 | may use other brands |
| Gel doc | Any | ||
| HardyChrom Vibrio agar plates | Hardy Diagnostics | G319 | This study evaluates this medium |
| Incubator | Any | ||
| Inoculating loops | Fisher Scientific | 22-363-606 | 10 microliter-size was used in this study |
| NaCl | Fisher Scientific | BP358-212 | may use other brands |
| Oysters | Any | ||
| PBS | Fisher Scientific | R23701 | may use other brands |
| Petri dish | Fisher Scientific | FB0875713 | may use other brands |
| Pipette and tips | Any | Sterilized tips | |
| Primers for tlh | IDT DNA | ||
| Scale | Any | ||
| Spreader | Fisher Scientific | 08-100-11 | Beads may be used instead |
| Stomacher blender | Stomacher | 400 | Samples were homogenized at 200 rpm for 30 sec. Other homogenizer can be used. |
| Sterile filter bags for blenders | Fisher Scientific | 01-812-5 | |
| TCBS powder | Hardy Diagnostics | 265020 | This study evaluates this medium |
| Thermocycler | Any | ||
| TSB powder | Fisher Scientific | DF0370-07-5 | may use other brands |
| UV viewing cabinet | Any | Emit long-wave UV light | |
| Water bath | Any | ||
| Name | Sources | Catalog Number | Comments |
| Bacterial species and strains | |||
| Aeromonas hydrophila | ATCC | ||
| Candida albicans | ATCC | ||
| Campylobacter jejuni | ATCC | ||
| Escherichia coli | ATCC | ||
| Proteus mirabilis | ATCC | ||
| Pseudomonas aeruginosa | ATCC | ||
| Staphylococcus aureus | ATCC | ||
| Salmonella Choleraesuis | ATCC | ||
| Shigella boydii | ATCC | ||
| Shigella flexneri | ATCC | ||
| Shigella sonnei | ATCC | ||
| Vibrio alginolyticus | ATCC | ||
| V. cholerae (serotypes include O139, O1, non O1, El Tor biovars) | FDA, ATCC | ||
| V. damsela | FDA | ||
| V. fisherii | Environment | ||
| V. fluvialis | CDC | ||
| V. furnissii | CDC | ||
| V. hollisae | FDA | ||
| V. metschnikovii | ATCC | ||
| V. mimicus | FDA | ||
| V. parahaemolyticus(serotypes include O3:K6, O1:K56, O4:K8, O5:K15, O8, etc) | ATCC, FDA, CDC, Environment | ||
| V. proteolyticus | FDA | ||
| V. vulnificus | FDA |
Références
- Yeung, P. S., Boor, K. J. Epidemiology, pathogenesis, and prevention of foodborne Vibrio parahaemolyticus infections. Foodborne Pathog. Dis. 1 (2), 74-88 (2004).
- Yeung, P. S. M., Boor, K. J., Faruque, S. M. Epidemiology, molecular biology, and detection of foodborne Vibrio parahaemolyticus infections. Foodborne and Waterborne Bacterial pathogens: Epidemiology, Evolution and Molecular Biology. , 153-184 (2012).
- Centers for Disease Control and Prevention. Vital Signs: Incidence and Trends of Infection with Pathogens Transmitted Commonly Through Food - Foodborne Diseases Active Surveillance Network, 10 U.S. Sites, 1996-2010. Morbidity and Mortality Weekly Report. 10, 1996-2010 (2011).
- . Summary of human Vibrio cases reported to CDC, 2008 Available from: https://stacks.cdc.gov/view/cdc/21591 (2008)
- Scallan, E., et al. Foodborne illness acquired in the United States - major pathogens. Emerg. Infect. Dis. 17 (1), 7-15 (2011).
- Baross, J., Liston, J. Occurrence of Vibrio parahaemolyticus and related hemolytic vibrios in marine environments of Washington State. Appl. Microbiol. 20 (2), 179-186 (1970).
- Baross, J., Liston, J. Isolation of Vibrio parahaemolyticus from the Northwest Pacific. Nature. 217 (5135), 1263-1264 (1968).
- Kueh, C. S., Chan, K. Y. Bacteria in bivalve shellfish with special reference to the oyster. J. Appl. Bacteriol. 59 (1), 41-47 (1985).
- Food and Drug Administration. . Bad Bug Book, Foodborne Pathogenic Microorganisms and Natural Toxins. , (2012).
- Qadri, F., et al. Adaptive and inflammatory immune responses in patients infected with strains of Vibrio parahaemolyticus. J. Infect. Dis. 187 (7), 1085-1096 (2003).
- MacFaddin, J. F. . Media for isolation-cultivation-identification-maintenance of medical bacteria. 1, (1985).
- Bottone, E. J., Robin, T. Vibrio parahaemolyticus: suspicion of presence based on aberrant biochemical and morphological features. J. Clin. Microbiol. 8 (6), 760-763 (1978).
- Lotz, M. J., Tamplin, M. L., Rodrick, G. E. Thiosulfate-citrate-bile salts-sucrose agar and its selectivity for clinical and marine vibrio organisms. Ann. Clin. Lab. Sci. 13 (1), 45-48 (1983).
- Hara-Kudo, Y., Nishina, T., Nakagawa, H., Konuma, H., Hasegawa, J., Kumagai, S. Improved method for detection of Vibrio parahaemolyticus in seafood. Appl. Environ. Microbiol. 67 (12), 5819-5823 (2001).
- Eddabra, R., Piemont, Y., Scheftel, J. M. Evaluation of a new chromogenic medium, chromID Vibrio, for the isolation and presumptive identification of Vibrio choleare and Vibrio parahaemolyticus from human clinical specimens. Eur. J. Clin. Microbiol. Infect. Dis. 30 (6), 733-737 (2011).
- Kodaka, H., Teramura, H., Mizuochi, S., Saito, M., Matsuoka, H. Evaluation of the Compact Dry VP method for screening raw seafood for total Vibrio parahaemolyticus. J. Food. Prot. 72 (1), 169-173 (2009).
- Su, Y. C., Duan, J., Wu, W. H. Selectivity and specificity of a chromogenic medium for detecting Vibrio parahaemolyticus. J. Food Prot. 68 (7), 1454-1456 (2005).
- Bej, A. K., Patterson, D. P., Brasher, C. W., Vickery, M. C., Jones, D. D., Kaysner, C. A. Detection of total and hemolysin-producing Vibrio parahaemolyticus in shellfish using multiplex PCR amplification of tl, tdh and trh. J. Microbiol. Methods. 36 (3), 215-225 (1999).
- Yeung, P. S. M., DePaola, A., Kaysner, C. A., Boor, K. J. A PCR assay for specific detection of the pandemic Vibrio parahaemolyticus O3:K6 clone from shellfish. J. Food Sci. 68 (4), 1459-1466 (2003).
- Yeung, P. S. M., Hayes, M. C., DePaola, A., Kaysner, C. A., Kornstein, L., Boor, K. J. Comparative phenotypic, molecular, and virulence characterization of Vibrio parahaemolyticus O3:K6 isolates. Appl. Environ. Microbiol. 68 (6), 2901-2909 (2002).
- Duan, J., Su, Y. -. C. Comparison of a chromogenic medium with thiosulfate-citrate-bile salts-sucrose agar for detecting Vibrio parahaemolyticus. J. Food Sci. 70, M125-M128 (2005).
- Pinto, A. D., Terio, V., Novello, L., Tantillo, G. Comparison between thiosulphate-citrate-bile salt sucrose (TCBS) agar and CHROMagar Vibrio for isolating Vibrio parahaemolyticus. Food Control. 22 (1), 124-127 (2011).
- Canizalez-Roman, A., Flores-Villaseñor, H., Zazueta-Beltran, J., Muro-Amador, S., Leòn-Sicairos, N. Comparative evaluation of a chromogenic agar medium-PCR protocol with a conventional method for isolation of Vibrio parahaemolyticus strains from environmental and clinical samples. Can J Microbiol. 57 (2), 136-142 (2011).
- Kriem, M. R., et al. Prevalence of Vibrio spp. in raw shrimps (Parapenaeus longirostris) and performance of a chromogenic medium for the isolation of Vibrio strains. Lett Appl Microbiol. 61 (3), 224-230 (2015).
- Food Drug Administration. Statistical guidance on reporting results from studies evaluating diagnostic tests. http://www.fda.gov/RegulatoryInformation/Guidances/ucm071148.htm. , (2007).
- Burd, E. M. Validation of laboratory-developed molecular assays for infectious diseases. Clin Microbiol Rev. 23 (3), 550-576 (2010).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon