È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Sviluppo di un più sensibile e specifica Chromogenic Agar Media per la rilevazione di
In questo articolo
Riepilogo
Detection and isolation of clinically relevant Vibrio species require selective and differential culture media. This study evaluated the ability of a new chromogenic medium to detect and identify V. parahaemolyticus and other related species. The new medium was found to have better sensitivity and specificity than the conventional medium.
Abstract
Infezioni di origine alimentare negli Stati Uniti causata da specie Vibrio hanno mostrato una tendenza al rialzo. Nel genere Vibrio, V. parahaemolyticus è responsabile per la maggior parte delle infezioni -associated Vibrio. Così, la differenziazione precisa tra Vibrio spp. e rilevazione di V. parahaemolyticus è di fondamentale importanza per garantire la sicurezza del nostro approvvigionamento alimentare. Sebbene le tecniche molecolari sono sempre più comuni, i metodi di coltura-base sono ancora regolarmente fatte e sono considerati metodi standard in determinate circostanze. Pertanto, un nuovo mezzo agar cromogeno è stato testato con l'obiettivo di fornire un metodo migliore per l'isolamento e la differenziazione di clinicamente rilevante spp Vibrio. Il protocollo rispetto al limite di sensibilità, specificità e rilevamento per il rilevamento di V. parahaemolyticus tra il nuovo terreno cromogeno e un mezzo convenzionale. Vari V. ceppi parahaemolyticus (n = 22) ripresentando diversi sierotipi e fonte di origine sono stati utilizzati. Essi sono stati precedentemente individuati dalla Food and Drug Administration (FDA) e Centri per il Controllo e la Prevenzione delle Malattie (CDC), e ulteriormente verificati nel nostro laboratorio da tlh -PCR. In almeno quattro prove separate, questi ceppi sono stati inoculati sui cromogenici agar e tiosolfato-citrato-biliari sali-saccarosio (TCBS) agar, che è il mezzo consigliato per la coltura di questa specie, seguita da incubazione a 35-37 ° C per 24 -96 ore. Tre V. ceppi parahaemolyticus (13,6%) non sono cresciuti in modo ottimale su TCBS, comunque esposti colonie verdi se non ci fosse la crescita. Due ceppi (9,1%) non ha dato i colonie ciano attesi sul agar cromogenico. V. non- ceppi parahaemolyticus (n = 32) sono stati testati per determinare la specificità del agar cromogenico. Tra questi ceppi, 31 non sono cresciuti o esposte altre morfologie di colonia. Il recupero medio di V. parahaemolyticus sul chromogenic agar era ~ 96,4% rispetto al agar soia trittico integrato con il 2% di NaCl. In conclusione, il nuovo agar cromogeno è un mezzo efficace per rilevare V. parahaemolyticus e per differenziarlo da altri vibrioni.
Introduzione
In qualità di membro del genere Vibrio, V. parahaemolyticus è una curva, batterio a forma di bastoncello Gram-negativi, non sporigeni. Presenta elevata motilità sia in ambienti liquidi e semisolidi. La maggior parte V. ceppi parahaemolyticus non sono patogeni per l'uomo, ma i sottotipi patogeni hanno causato epidemie e pandemie, quindi questa specie è considerata un importante agente patogeno di origine alimentare in molti paesi 1,2. L'incidenza di infezione da Vibrio negli Stati Uniti ha mostrato una tendenza al rialzo dal 2000 3. Tra Vibrio spp., V. parahaemolyticus è la specie più frequentemente riportati causano malattie negli Stati Uniti 4,5. Altre specie clinicamente rilevanti includono V. alginolyticus, V. vulnificus, V. cholerae, ecc Una piccola percentuale delle malattie è causata da specie multiple simultaneamente.
V. parahaemolyticus è un ghiaccinhabitant di acqua marina e quindi ampiamente distribuita nelle acque marine in tutto il mondo, tra cui gli estuari. La specie è stata scoperta nel 1950 a seguito di un focolaio di intossicazione alimentare in Giappone. Negli Stati Uniti, la specie è stato isolato in acqua di mare, sedimenti, e frutti di mare nella regione di Puget Sound 6,7. Filtratori in habitat marini, come molluschi bivalvi, possono porto V. parahaemolyticus come parte della loro flora naturali 8. Come tale, V. infezioni parahaemolyticus in umano sono spesso legati al consumo di frutti di mare contaminati, in particolare frutti di mare crudi o poco cotti. Un percorso meno comune di entrata si verifica quando ferita aperta è esposto ad acqua di mare, che porta a infezioni della pelle. La maggior parte V. ceppi parahaemolyticus non causano malattie umane, ma alcuni sottotipi ospitano fattori di virulenza, come emolisina termostabile diretta (TDH) sono patogeni. I sintomi più frequenti di origine alimentare V. infezione parahaemolyticus sonodiarrea e dolori addominali, seguito da nausea, vomito e febbre. Mal di testa e brividi sono anche riportati. Il periodo di incubazione medio è di 15 ore, ma può essere fino a 96 ore dopo il consumo di una quantità sufficiente di ceppi patogeni 9. La malattia dura da due a tre giorni. I sintomi gastroenterite causata da V. parahaemolyticus sono largamente auto-limitante e quindi il trattamento speciale non è necessaria. I casi lievi di gastroenterite possono essere efficacemente trattati con reidratazione orale. Più gravi malattie possono essere trattati con antibiotici come la tetraciclina o ciprofloxacina 10. tasso di mortalità è circa il 2% per i casi di gastroenterite, ma può essere alto come il 29% per coloro che sviluppano infezione sangue o setticemia. Qualsiasi persona che consuma frutti di mare o ha ferita aperta esposta all'acqua di mare è a rischio di V. infezione parahaemolyticus. La forma più grave di malattie, la setticemia pericolosa per la vita, è più comune in una sottopopolazione con sottostante co medicalli operativi 11, che includono l'alcolismo, malattie del fegato, diabete, malattie renali, tumori maligni, e le altre condizioni che portano ad una risposta immunitario indebolito. In particolare, questo gruppo di individui è anche ad un elevato rischio di contrarre gravi malattie causate da V. vulnificus, che si trova in habitat naturali simili a V. parahaemolyticus.
V. parahaemolyticus è abitualmente isolato usando tiosolfato-citrato-biliari sali-saccarosio (TCBS) agar come un terreno selettivo e differenziale. Arricchimento in acqua peptonata alcalina può precedere isolamento su TCBS agar. presunte colonie su TCBS sono poi ulteriormente testati in una serie di test biochimici e / o saggi molecolari mirati la presenza di geni specie-specifici. Metodi basati sulla PCR sono spesso usati per confermare l'identità di V. parahaemolyticus amplificando il gene emolisina termolabile, tlh 12.
Indipendentemente dal choice dei metodi di conferma, è importante disporre di un mezzo efficace per isolare e differenziare V. parahaemolyticus da altri vibrioni marini, in primo luogo. TCBS è stato solitamente usata per differenziare le specie all'interno del genere Vibrio secondo le loro capacità di fermentare il saccarosio 12. Reazione di fermentazione positiva è accompagnato da un cambiamento di colore dell'indicatore pH bromotimolo blu. V. colonie parahaemolyticus sono abbastanza distintivo TCBS, esibendo blu al colore verde. Tuttavia, questo mezzo non può facilmente distinguere V. alginolyticus e V. cholerae. Proteus saccarosio-fermentazione possono produrre colonie gialle simili a V. cholerae o V. alginolyticus 13. Su isolamento iniziale TCBS, V. parahaemolyticus può anche essere erroneamente identificato come Aeromonas hydrophila, shigelloides Plesiomonas, e Pseudomonas spp 14. Ceppi con ferm saccarosio in ritardosentazione può essere confusa con altri saccarosio nonfermenting Vibrio 13, che comprendono V. parahaemolyticus. TCBS è risultato essere non sensibili contro Escherichia coli, putrefaciens Pseudomonas, tra gli altri. Diverse altre specie cedono verde di colonie grigie, che sono potenzialmente confondibili con V. parahaemolyticus o V. vulnificus 15. Di conseguenza, è desiderabile sviluppare mezzi di cultura alternativa con una migliore sensibilità e specificità verso individuare e isolare V. parahaemolyticus e di altre specie strettamente correlate.
Diverse alternative dei media sono stati recentemente sviluppati. Inoltre l'inclusione di agenti selettivi, più incorporano substrati cromogeni per differenziare le specie in base alle loro attività enzimatiche differenziali. Ad esempio, indoxyl-β-glucoside e indoxyl-β-galattoside sono stati utilizzati come substrati cromogeni differenziare V. paràcolonie haemolyticus (che appaiono blu-verde) da quelle di V. cholerae (viola) a causa della loro capacità di produrre differenziali β-glucosidasi e β-galattosidasi 16. Diverse formulazioni di cromogenico agar sviluppate da diversi gruppi sono stati valutati e sono stati segnalati per eseguire paragonabile o superiore a quella TCBS 17,18,19. Un vantaggio di utilizzare un terreno cromogeno è che la colorazione del mezzo circostante è minimo facilitando così l'isolamento di colonie particolari. In questo studio abbiamo valutato la capacità di un terreno cromogeno nuova formulazione per rilevare ed isolare V. cholerae, V. parahaemolyticus e V. vulnificus; con un focus particolare sulla sua capacità di differenziare V. parahaemolyticus da altre specie.
Protocollo
1. Media e coltura di ceppi microbici
NOTA: Utilizzare tecniche asettiche in tutti gli esperimenti. Utilizzare materiali sterili. Sterilizzare tutti i contenitori, strumenti e reagenti prima dell'uso. Autoclavare tutti i materiali di scarto prima dello smaltimento perché sono considerati a rischio biologico. la temperatura e il tempo Autoclave combinazione è ≥121 ° C x ≥15 min per tutte le seguenti procedure.
- Per rendere ~ 1-L Tryptic Soy Agar (TSA), prima aggiungere 1 L di acqua deionizzata in una beuta 2-L contenente una ancoretta magnetica. Utilizzare un pallone che è almeno due volte più grande del volume finale. Aggiungere 30 g di polvere di triptico brodo di soia e 20 g di agar granuli nel matraccio.
NOTA: Utilizzare 2% agar invece del 1,5% per limitare brulicare di qualche Vibrio spp.- Mescolare accuratamente accendendo l'agitatore. Mentre si agita, accendere il fuoco a bollire la miscela. Rimuovere la beuta dal riscaldatore non appena il composto inizia a bollire. Liberamente coprire °e pallone con una carta stagnola. Nastro il foglio per fissarlo al pallone prima autoclave.
- Per fare il brodo di soia trittico (TSB), omettere l'agar dalla ricetta al punto 1.1.
NOTA: Può usare bottiglie invece di beuta. - Per rendere triptico agar di soia integrato con cloruro di sodio 2% o NaCl (TSAS), aggiungere 20 g di NaCl nella miscela prima agitazione e riscaldamento. Per fare il brodo di soia trittico addizionato con 2% NaCl (TSBS), omettere l'agar, aggiungere 20 g di NaCl nella miscela prima agitazione e riscaldamento.
- Per rendere infuso cuore cervello (BHI) agar, sospendere 37 g di polvere di BHI e 15 g granulato di agar in 1 L di acqua purificata. Il calore sotto agitazione per sciogliere la polvere. Autoclave. Omettere l'agar per fare BHI brodo.
- Per rendere TCBS agar, sospendere 89 g della polvere TCBS in 1 L di acqua purificata. Di calore con agitazione e far bollire per 1 minuto per sciogliere completamente la polvere. Non sterilizzare in autoclave.
- Per tutti i supporti di agar, raffreddare l'agar caldo per 45-50 ° C in un bagno d'acqua. Disporre le piastre di Petri vuote in pile di cinque o sei piatti. Partendo dal fondo della pila, versare l'agar fuso in ciascuna piastra di Petri per raggiungere circa metà pieno. Chiudere il coperchio piatto Petri dopo avere versato. Lasciare che l'agar solidificare lasciando le piastre siedono a temperatura ambiente.
- Utilizzare le piastre di agar il giorno dopo o dopo 12 ore. Conservare le piastre inutilizzati in frigorifero per un massimo di due settimane. Prima dell'uso, rimuovere le piastre dal frigorifero e equilibrare a temperatura ambiente per almeno 15 min.
NOTA: agar Un litri rende ~ 45 piastre di agar. Lasciare le piastre di agar per asciugare sufficientemente il giorno di preparazione, e portare a temperatura ambiente dopo conservazione frigorifera per ridurre efficacemente la diffusione delle colonie.
- Utilizzare le piastre di agar il giorno dopo o dopo 12 ore. Conservare le piastre inutilizzati in frigorifero per un massimo di due settimane. Prima dell'uso, rimuovere le piastre dal frigorifero e equilibrare a temperatura ambiente per almeno 15 min.
- Ottenere piastre cioccolato e cromogenico agar e equilibrare a temperatura ambiente prima di ogni esperimento.
- Sottocultura tutti i 54 ceppi microbici indicati nella tabella 1 ogni pochi days.
- Utilizzare un ciclo inoculo sterile per trasferire le culture da uno stock congelati o un lotto precedente di terreni non selettivi, come BHI, TSB / TSA o agar cioccolato. Grow halophilic Vibrio spp. su TSBS / TSAS.
- Per controllare la purezza della cultura, consecutive tutti i ceppi in un modello che consenta di osservazione di colonie isolate. Ad esempio, utilizzare un modello striature trifase per diluire una grande quantità di batteri per piccole quantità, per ottenere alla fine colonie isolate.
- Incubare le piastre a testa in giù a 35-37 ° C per un massimo di 48 ore. Per campylobacter spp., Incubare i tubi o piastre in un vaso chiuso coperchio contenente una sacca di gas per produrre un ambiente microaerofili. Osservare morfologia delle colonie dopo l'incubazione. colture pure dovrebbero produrre colonie che presentano simili morfologia delle colonie.
NOTA: Incubare tutte le piastre capovolta per evitare goccioline d'acqua condensate formate sul lato inferiore del coperchio di cadere sul colleOnies.
2. Specie Determinazione mediante PCR
- Condurre tlh -PCR per confermare l'identità di V. ceppi parahaemolyticus. Utilizzare primer tlh -F (5 'AAA GCG GAT TAT GCA GCA GAA CTG 3') e tlh -R (5 'GCT ACT TTC TAG CAT TTT CTC TGC 3') per amplificare un frammento di 450 bp del gene termolabile hemolysin 20 .
- Utilizzare un ciclo inoculo sterile per trasferire alcune colonie isolate di ciascun V. parahaemolyticus ceppo da TSAS a 5 ml di TSBS. Incubare a 35-37 ° C per 16-24 ore.
- culture centrifugare a 14000 g per 1 min. Rimuovere il surnatante e lavare il pellet due volte con tampone fosfato (PBS). Far bollire la sospensione per 3 minuti a cedere lisato cellulare.
NOTA: V. parahaemolyticus è facile essere lisate. Pertanto reagente di lisi non è necessaria. È anche possibile usare un po 'di colonia direttamente come modello. - Eseguire la PCR in una reazione di 25 microlitri volume. Preparare una miscela di reazione contenente una concentrazione finale di 1x tampone PCR, 1,5 mM MgCl 2, 100 mM di ciascun dNTP, 1 mM di ciascun primer, 1 U Taq polimerasi e 1 ml di lisato cellulare. Dopo preincubazione a 94 ° C per 5 min, eseguire 35 cicli di amplificazione di 94 ° C per 30 sec, 58 ° C per 30 sec e 72 ° C per 60 sec 21.
- aliquote di carico di amplicone 5 ml a 1,5% gel di agarosio. Attivare alimentazione per iniziare l'elettroforesi. Visualizzare la presenza o l'assenza di ampliconi sotto illuminazione UV dopo colorazione con etidio bromuro.
3. La crescita su selettivo e differenziale media
- Da due a quattro giorni prima dell'esperimento, striscia tutti i ceppi microbici indicati nella tabella 1 su terreno non selettivo (TSAS, BHI o agar cioccolato) per l'isolamento delle colonie. Incubare le piastre a 35-37 ° C per 48 ore. Controllare la purezza delle culture osservando la morfologia delle colonie dopo incubazione. colture pure dovrebbero produrre colonie che presentano simili morfologia delle colonie.
- Trasferire alcune colonie isolate dal punto 3.1 in 5 ml di brodo. Incubare le provette a 35-37 ° C per 16-24 ore.
Nota: utilizzare giovani colonie, che sono meno di quattro giorni, per preparare le culture durante la notte in tutti gli esperimenti. - Streak un'ansata di culture durante la notte su terreni selettivi e differenziali (TCBS e cromogenico agar) per l'isolamento delle colonie. Incubare le piastre a 35-37 ° C per un massimo di 96 ore.
- Registrare la crescita complessiva di tutti i ceppi esaminando sia la densità coltura su piastra e la dimensione delle colonie isolate. Registrare il colore delle colonie sotto luce ambientale e / o UV. Nota altre caratteristiche della colonia isolata, come l'elevazione, il margine e la forma.
Assay 4. Recupero
- Selezionare un sottoinsieme rappresentativo di V. ceppi parahaemolyticus (n = 14) che comprendono diversi sierotipi e le origini di isolamento 22. Seminare giovani culture dalle piastre in 5 ml di TSBS. Incubare le provette a 35-37 ° C per 16-24 ore.
- Effettuare un metodo Plate Count standard delle culture overnight come descritto di seguito.
- Vortex per mescolare bene le culture durante la notte. Fare un 10 volte o 10 -1 diluizione con il trasferimento di 100 microlitri della cultura durante la notte in una provetta contenente 900 ml di PBS. Vortex per mescolare bene.
- Utilizzare un nuovo puntale per trasferire 100 ul dal tubo 10 -1 diluizione ad un altro tubo contenente 900 ml di PBS. Vortex per mescolare bene. Ciò costituisce 10 -2 diluizione. Ripetere il processo sequenziale di ottenere 10 -7 diluizione.
- Utilizzando i tubi di diluizione 10 -4 a 10 -7, piatto 100 ml ciascuno sulle cromogenici, TCBS e CST piastre di agar.
NOTA: Il fattore di diluizione (df) sulla piastra diventa 10 -5 a 10 -8, rispettivamente. - Stendere le aliquote in modo uniforme su tegli AGAR superficie.
NOTA: È bene utilizzare lo stesso spreader per ceppo sullo stesso supporto, purché il un'aliquota più diluito viene diffuso prima (cioè, da 10 -8 a 10 -5). Non usare la stessa spreader per diversi media. - Incubare le piastre a 35-37 ° C per un massimo di 96 ore. Contare colonie sulle piastre. Ignorare lastre portanti colonie che sono troppo numerosi per contare (TNTC) o minore di 25. Calcolare CFU / ml secondo il seguente:
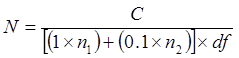
Dove
N = numero di cellule nel tubo non diluito, espressa come CFU / ml o CFU / g
C = il numero totale di colonie contate su piastre che portano 25-300 colonie
n 1 = il numero di targa (s) dove colonie contate sono da df minore
n 2 = il numero di targa (s) dove colonie contate sono dalla successiva diluizione di 10 volte
dF = fattore di diluizione più basso (vale a dire, la diluizione più concentrata)
- Confronto CFU / ml tra i diversi mezzi di comunicazione. Utilizzare CFU / ml su TSAS al 100%, calcolare il recupero% di V. parahaemolyticus cresciuto sul agar cromogenico e TCBS.
Assay 5. Concorrenza
- Scegliere un sottoinsieme di ceppi che mostrano diverse morfologie di colonia sul agar cromogenico e TCBS.
NOTA: In questo modo, sarà possibile contare le colonie originati da V. parahaemolyticus solo, nonostante la presenza di altre specie nella inoculo.- Scegliere un V. ceppo parahaemolyticus che produce i turchesi e ciano colonie attesi sulle TCBS e l'agar cromogenico, rispettivamente.
- Scegli un non V. parahaemolyticus e una specie non Vibrio che non crescono su uno di questi mezzi, o presentano diversi colori colonia.
NOTA: per esempio, V. metschnikovii cresce molto debolmente on TCBS e non crescono sulla agar cromogenico. Shigella sonnei non crescono sugli TCBS ma produce colonie magenta sul agar cromogenico.
- Dopo la selezione dei ceppi di cui sopra, preparare brodi di coltura durante la notte utilizzando colonie isolate cresciute su terreni non selettivi.
- Fare culture durante la notte di V. parahaemolyticus e V. metschnikovii trasferendo alcune colonie isolate da TSAS a 5 ml di TSBS. Incubare a 35-37 ° C per 16-24 ore.
- Fare culture durante la notte di Shigella sonnei con il trasferimento di alcune colonie isolate da BHI agar a 5 ml di brodo BHI. Incubare a 35-37 ° C per 16-24 ore.
- Per ogni ceppo, eseguire una serie di diluizioni simile ai punti 4.2.1 e 4.2.2. Piastra diluizioni appropriate sul TSAS o BHI per determinare CFU / ml della cultura durante la notte, utilizzando l'equazione mostrato al passaggio 4.2.5.
NOTA: in genere, 100 ml di diluizione 10 -5 a 10 -7tubi funziona per la maggior parte delle culture. Utilizzare i valori CFU / ml per eseguire il calcolo l'esatta quantità di cellule utilizzate nella fase successiva. I valori CFU / ml calcolati si ottengono dopo incubazione, anche se le seguenti operazioni vengono eseguite nella stessa data come punto 5.3. - Utilizzando le culture durante la notte e tubi di diluizione in passi 5.2 e 5.3, mescolare diverse quantità di un V. ceppo parahaemolyticus e un non V. Specie parahaemolyticus. Ad esempio, miscelare 500 ml di tubo 10 -5 diluizione V. parahaemolyticus con 500 ml di culture durante la notte di V. metschnikovii.
NOTA: Questa miscela simula fondo elevato microflora. Per simulare uno sfondo partire microflora, mescolare 500 ml ciascuna del tubo 10 -5 diluizione da entrambe le specie. - Distribuire 100 ml di miscela ciascuno sulla cromogenici, TCBS e CST piastre di agar.
- Dopo incubazione a 35-37 ° C per un massimo di 96 ore, contare le colonie di V. parahaemolyticus e la non V. Specie parahaemolyticus in base alla loro differenza di crescita e morfologia delle colonie su agar cromogenico e TCBS.
NOTA: Ad esempio, se la non V. specie parahaemolyticus non cresce sulla agar cromogenico e TCBS, tutte le colonie saranno di V. parahaemolyticus. Se la mancata V. Specie parahaemolyticus cresce su entrambi i supporti, solo le colonie turchese su colonie TCBS e ciano su agar cromogenico saranno di V. parahaemolyticus. La mancata V. Specie parahaemolyticus può o non può esibire simili morfologia delle colonie a V. parahaemolyticus su TSAS.- Se la mancata V. Specie parahaemolyticus cresce in modo simile a V. parahaemolyticus su questo terreno non selettivo, dividere il conteggio delle colonie per due per ottenere i numeri per V. parahaemolyticus solo. Confrontare il conteggio effettivo colonie con il conteggio atteso derivata dal punto 5.3.
6. Effetti di Oyster omogenati
- Pesare ≥50 g di carne di ostriche da ≥12 molluschi tra cui carne e liquori.
- Aggiungere la stessa quantità di PBS alla carne di ostriche e liquori. Miscela la miscela ad alta velocità per 90 sec. Ciò costituisce 2 -1 diluito omogeneizzato di ostriche.
- Aggiungere 100 g di 2 -1 diluito omogenato ostriche a 400 g di PBS. Utilizzare una scala per misurare il peso, non in volume. Frullare il composto ad alta velocità per 1 min. Autoclave l'omogeneizzato di ostriche.
NOTA: Questo sarà l'omogeneizzato ostrica utilizzato per la chiodare.
- Ripetere l'analisi di recupero (fase 4), in presenza di ostriche omogeneizzato.
- Dopo il 500 g di ostriche omogenato si raffredda, aggiungere 100 ml di V. parahaemolyticus culture durante la notte coltivate in TSBS ad esso. Determinare la quantità effettiva di V. cellule parahaemolyticus nel inoculo di gestione delle procedure di conteggio su piastra standard descritti al punto 4.2.
- rappresentantemangiare il saggio di concorrenza (punto 5) in presenza di ostriche omogeneizzato.
- Dopo il 500 g di ostriche omogenato si raffredda, aggiungere 100 ml ciascuna delle culture durante la notte di V. parahaemolyticus e non V. parahaemolyticus ad esso. Determinare la quantità effettiva di batteri cellule nel inoculo di gestione delle procedure di conteggio su piastra standard descritto al punto 4.2
- Mescolare le cellule batteriche con ostriche omogenato bene usando un omogeneizzatore.
NOTA: Dopo la miscelazione, l'omogeneizzato ostriche contenente le cellule aggiunti intenzionalmente è chiamato a spillo ostriche omogeneizzato. - Fare diluizioni del omogeneizzato ostrica a spillo per ottenere 10 -1 a 10 -3 tubi di diluizione secondo le modalità descritte al punto 4.2.2. Distribuire 100 ml di ogni diluizione sul cromogenico, TCBS e TSAS agar. Incubare le piastre a 35-37 ° C per un massimo di 96 ore.
- Confrontare la conta delle colonie su agar reale cromogenico e TCBS con l'expected conteggio delle colonie dedotta da Piazza 6.2.1 e 6.3.1.
NOTA: Ad esempio, se un tubo di V. parahaemolyticus coltura overnight contiene 10 8 UFC / ml, un inoculo di 100 microlitri significa che 10 7 cellule vengono aggiunti 500 g di ostriche omogenato, ottenendo 5 x 10 4 cellule / g. Dopo la diluizione e la placcatura, la piastra avendo df = 10 -2 dovrebbe produrre 500 colonie; mentre che avere df = 10 -3 dovrebbe cedere 50 colonie. Queste sono le conta delle colonie attesi.
Risultati
In questo studio, 54 ceppi microbici sono stati assemblati, che comprendeva 22 ceppi all'interno V. Specie parahaemolyticus, altre 19 specie Vibrio, e 13 specie Vibrio non (Tabella 1). La maggior parte V. ceppi parahaemolyticus erano o ricevute da parte della FDA, CDC o altri dipartimenti sanitari statali. Essi rappresentano diversi sierotipi e fonti di isolamento. Questi ceppi sono stati precedentemente identific...
Discussione
Questo studio si concentra sullo sviluppo terreni di coltura e di valutazione. Convenzionalmente, TCBS è selettivo e differenziale mezzo utilizzato per l'isolamento e la rilevazione V. parahaemolyticus, V. cholerae e V. vulnificus 12. Tuttavia, le limitazioni sono stati riportati per questo mezzo, come l'incapacità di differenziare V. cholerae da altre specie Vibrio. Il saccarosio e indicatore di pH sono gli agenti di differenziazione di TCBS. Così, la produzione...
Divulgazioni
Some media were generously provided by Hardy Diagnostics, Santa Maria, CA. Thorsen conducted this study while a student at California Polytechnic State University. He is currently an employee of Hardy Diagnostics.
Riconoscimenti
Ringraziamo M. Channey, E. Chau, e K. Tomas per la loro assistenza nel progetto. forniture di progetto sono stati in parte finanziati dalla California Polytechnic State University.
Materiali
| Name | Company | Catalog Number | Comments |
| Reagent/Equipment | |||
| Agar | Fisher Scientific | DF0140-15-4 | may use other brands |
| Autoclave | Any | ||
| BHI powder | Fisher Scientific | DF0418-17-7 | may use other brands |
| Blender | Any | to blend oyster meat | |
| CampyGen gas generator | Hardy Diagnostics | CN035A | to provide a microaerophilic atmosphere; may use other brands |
| Chocolate agar plates | Hardy Diagnostics | E14 | may use other brands |
| Common PCR reagents (dNTPs, MgCl2, Taq Polymerase) | Any | or use PCR beads (Fisher Sci 46-001-014) | |
| Culture tubes | Fisher Scientific | S50712 | may use other brands |
| Eppendorf tubes | Fisher Scientific | S348903 | may use other brands |
| Gel doc | Any | ||
| HardyChrom Vibrio agar plates | Hardy Diagnostics | G319 | This study evaluates this medium |
| Incubator | Any | ||
| Inoculating loops | Fisher Scientific | 22-363-606 | 10 microliter-size was used in this study |
| NaCl | Fisher Scientific | BP358-212 | may use other brands |
| Oysters | Any | ||
| PBS | Fisher Scientific | R23701 | may use other brands |
| Petri dish | Fisher Scientific | FB0875713 | may use other brands |
| Pipette and tips | Any | Sterilized tips | |
| Primers for tlh | IDT DNA | ||
| Scale | Any | ||
| Spreader | Fisher Scientific | 08-100-11 | Beads may be used instead |
| Stomacher blender | Stomacher | 400 | Samples were homogenized at 200 rpm for 30 sec. Other homogenizer can be used. |
| Sterile filter bags for blenders | Fisher Scientific | 01-812-5 | |
| TCBS powder | Hardy Diagnostics | 265020 | This study evaluates this medium |
| Thermocycler | Any | ||
| TSB powder | Fisher Scientific | DF0370-07-5 | may use other brands |
| UV viewing cabinet | Any | Emit long-wave UV light | |
| Water bath | Any | | |
| Name | Sources | Catalog Number | Comments |
| Bacterial species and strains | |||
| Aeromonas hydrophila | ATCC | ||
| Candida albicans | ATCC | ||
| Campylobacter jejuni | ATCC | ||
| Escherichia coli | ATCC | ||
| Proteus mirabilis | ATCC | ||
| Pseudomonas aeruginosa | ATCC | ||
| Staphylococcus aureus | ATCC | ||
| Salmonella Choleraesuis | ATCC | ||
| Shigella boydii | ATCC | ||
| Shigella flexneri | ATCC | ||
| Shigella sonnei | ATCC | ||
| Vibrio alginolyticus | ATCC | ||
| V. cholerae (serotypes include O139, O1, non O1, El Tor biovars) | FDA, ATCC | ||
| V. damsela | FDA | ||
| V. fisheri | Environment | ||
| V. fluvialis | CDC | ||
| V. furnissii | CDC | ||
| V. hollisae | FDA | ||
| V. metschnikovii | ATCC | ||
| V. mimicus | FDA | ||
| V. parahaemolyticus (serotypes include O3:K6, O1:K56, O4:K8, O5:K15, O8, etc.) | ATCC, FDA, CDC, Environment | ||
| V. proteolyticus | FDA | ||
| V. vulnificus | FDA |
Riferimenti
- Yeung, P. S., Boor, K. J. Epidemiology, pathogenesis, and prevention of foodborne Vibrio parahaemolyticus infections. Foodborne Pathog. Dis. 1 (2), 74-88 (2004).
- Yeung, P. S. M., Boor, K. J., Faruque, S. M. Epidemiology, molecular biology, and detection of foodborne Vibrio parahaemolyticus infections. Foodborne and Waterborne Bacterial pathogens: Epidemiology, Evolution and Molecular Biology. , 153-184 (2012).
- Centers for Disease Control and Prevention. Vital Signs: Incidence and Trends of Infection with Pathogens Transmitted Commonly Through Food - Foodborne Diseases Active Surveillance Network, 10 U.S. Sites, 1996-2010. Morbidity and Mortality Weekly Report. 10, 1996-2010 (2011).
- . Summary of human Vibrio cases reported to CDC, 2008 Available from: https://stacks.cdc.gov/view/cdc/21591 (2008)
- Scallan, E., et al. Foodborne illness acquired in the United States - major pathogens. Emerg. Infect. Dis. 17 (1), 7-15 (2011).
- Baross, J., Liston, J. Occurrence of Vibrio parahaemolyticus and related hemolytic vibrios in marine environments of Washington State. Appl. Microbiol. 20 (2), 179-186 (1970).
- Baross, J., Liston, J. Isolation of Vibrio parahaemolyticus from the Northwest Pacific. Nature. 217 (5135), 1263-1264 (1968).
- Kueh, C. S., Chan, K. Y. Bacteria in bivalve shellfish with special reference to the oyster. J. Appl. Bacteriol. 59 (1), 41-47 (1985).
- Food and Drug Administration. . Bad Bug Book, Foodborne Pathogenic Microorganisms and Natural Toxins. , (2012).
- Qadri, F., et al. Adaptive and inflammatory immune responses in patients infected with strains of Vibrio parahaemolyticus. J. Infect. Dis. 187 (7), 1085-1096 (2003).
- MacFaddin, J. F. . Media for isolation-cultivation-identification-maintenance of medical bacteria. 1, (1985).
- Bottone, E. J., Robin, T. Vibrio parahaemolyticus: suspicion of presence based on aberrant biochemical and morphological features. J. Clin. Microbiol. 8 (6), 760-763 (1978).
- Lotz, M. J., Tamplin, M. L., Rodrick, G. E. Thiosulfate-citrate-bile salts-sucrose agar and its selectivity for clinical and marine vibrio organisms. Ann. Clin. Lab. Sci. 13 (1), 45-48 (1983).
- Hara-Kudo, Y., Nishina, T., Nakagawa, H., Konuma, H., Hasegawa, J., Kumagai, S. Improved method for detection of Vibrio parahaemolyticus in seafood. Appl. Environ. Microbiol. 67 (12), 5819-5823 (2001).
- Eddabra, R., Piemont, Y., Scheftel, J. M. Evaluation of a new chromogenic medium, chromID Vibrio, for the isolation and presumptive identification of Vibrio choleare and Vibrio parahaemolyticus from human clinical specimens. Eur. J. Clin. Microbiol. Infect. Dis. 30 (6), 733-737 (2011).
- Kodaka, H., Teramura, H., Mizuochi, S., Saito, M., Matsuoka, H. Evaluation of the Compact Dry VP method for screening raw seafood for total Vibrio parahaemolyticus. J. Food. Prot. 72 (1), 169-173 (2009).
- Su, Y. C., Duan, J., Wu, W. H. Selectivity and specificity of a chromogenic medium for detecting Vibrio parahaemolyticus. J. Food Prot. 68 (7), 1454-1456 (2005).
- Bej, A. K., Patterson, D. P., Brasher, C. W., Vickery, M. C., Jones, D. D., Kaysner, C. A. Detection of total and hemolysin-producing Vibrio parahaemolyticus in shellfish using multiplex PCR amplification of tl, tdh and trh. J. Microbiol. Methods. 36 (3), 215-225 (1999).
- Yeung, P. S. M., DePaola, A., Kaysner, C. A., Boor, K. J. A PCR assay for specific detection of the pandemic Vibrio parahaemolyticus O3:K6 clone from shellfish. J. Food Sci. 68 (4), 1459-1466 (2003).
- Yeung, P. S. M., Hayes, M. C., DePaola, A., Kaysner, C. A., Kornstein, L., Boor, K. J. Comparative phenotypic, molecular, and virulence characterization of Vibrio parahaemolyticus O3:K6 isolates. Appl. Environ. Microbiol. 68 (6), 2901-2909 (2002).
- Duan, J., Su, Y. -. C. Comparison of a chromogenic medium with thiosulfate-citrate-bile salts-sucrose agar for detecting Vibrio parahaemolyticus. J. Food Sci. 70, M125-M128 (2005).
- Pinto, A. D., Terio, V., Novello, L., Tantillo, G. Comparison between thiosulphate-citrate-bile salt sucrose (TCBS) agar and CHROMagar Vibrio for isolating Vibrio parahaemolyticus. Food Control. 22 (1), 124-127 (2011).
- Canizalez-Roman, A., Flores-Villaseñor, H., Zazueta-Beltran, J., Muro-Amador, S., Leòn-Sicairos, N. Comparative evaluation of a chromogenic agar medium-PCR protocol with a conventional method for isolation of Vibrio parahaemolyticus strains from environmental and clinical samples. Can J Microbiol. 57 (2), 136-142 (2011).
- Kriem, M. R., et al. Prevalence of Vibrio spp. in raw shrimps (Parapenaeus longirostris) and performance of a chromogenic medium for the isolation of Vibrio strains. Lett Appl Microbiol. 61 (3), 224-230 (2015).
- Food Drug Administration. Statistical guidance on reporting results from studies evaluating diagnostic tests. http://www.fda.gov/RegulatoryInformation/Guidances/ucm071148.htm. , (2007).
- Burd, E. M. Validation of laboratory-developed molecular assays for infectious diseases. Clin Microbiol Rev. 23 (3), 550-576 (2010).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon