É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Desenvolvimento de uma mais sensível e específica Cromogênico Agar para a detecção de
Neste Artigo
Resumo
Detection and isolation of clinically relevant Vibrio species require selective and differential culture media. This study evaluated the ability of a new chromogenic medium to detect and identify V. parahaemolyticus and other related species. The new medium was found to have better sensitivity and specificity than the conventional medium.
Resumo
Infecções alimentares em os EUA causada por espécies de Vibrio têm mostrado uma tendência ascendente. No género Vibrio, V. parahaemolyticus é responsável pela maioria das infecções por Vibrio -associated. Assim, a diferenciação precisa entre Vibrio spp. e detecção de V. parahaemolyticus é extremamente importante para garantir a segurança do nosso abastecimento alimentar. Embora as técnicas moleculares são cada vez mais comuns, métodos, dependendo de cultura são ainda feito rotineiramente e são considerados métodos padrão em determinadas circunstâncias. Assim, um novo meio de agar cromogénico foi testado com o objectivo de proporcionar um melhor método para o isolamento e diferenciação de Vibrio spp clinicamente relevante. O protocolo de comparação do limite de sensibilidade, especificidade e de detecção para a detecção de V. parahaemolyticus entre o novo meio cromogénico e um meio convencional. Vários V. cepas parahaemolyticus (n = 22) reapresentando diversos sorotipos e fonte de origens foram utilizados. Eles foram previamente identificados pela Food and Drug Administration (FDA) e os Centros de Controle e Prevenção de Doenças (CDC), e ainda mais verificada em nosso laboratório por tlh-PCR. Em pelo menos quatro ensaios independentes, estas estirpes foram inoculadas sobre os sais de agar-sacarose e tiossulfato-citrato-biliares cromogénicos (TCBS) de agar, que é a forma recomendada para a cultura desta espécie, seguido de incubação a 35-37 ° C durante 24 hr -96. Três V. cepas parahaemolyticus (13,6%) não cresceu de forma otimizada em TCBS, no entanto exibiram colónias verdes se houve crescimento. Duas cepas (9,1%) não deu as colônias ciano esperados no ágar cromogênico. V. não estirpes parahaemolyticus (n = 32) foram também testados para determinar a especificidade do ágar cromogénico. Entre estas estirpes, 31 não crescer ou exibiu outras morfologias colônia. A recuperação média de V. parahaemolyticus na chromogenic ~ ágar foi de 96,4% em relação ao agar de soja tríptica suplementado com 2% de NaCl. Em conclusão, o novo ágar cromogénico é um meio eficaz para detectar V. parahaemolyticus e diferenciá-lo de outros vibrios.
Introdução
Como um membro do gênero Vibrio, V. parahaemolyticus é, curvo bactéria Gram-negativa, não-formadoras de esporos, em forma de haste. Ele exibe uma elevada motilidade em ambos os ambientes líquidos e semi-sólidos. A maioria V. cepas parahaemolyticus não são patogénicas para os seres humanos, mas os subtipos patogênicos têm causado epidemias e pandemias, portanto, esta espécie é considerada um importante patógeno de origem alimentar em muitos países 1,2. A incidência de infecção Vibrio em os EUA tem mostrado uma tendência ascendente desde 2000 3. Entre Vibrio spp., V. parahaemolyticus é a espécie mais frequentemente relatados que causam doenças em os EUA 4,5. Outras espécies clinicamente relevantes incluem V. alginolyticus, V. vulnificus, V. cholerae, etc. Uma pequena porcentagem das doenças é causada por várias espécies simultaneamente.
V. parahaemolyticus é uma i naturaisnhabitant de água do mar e, portanto, amplamente distribuídos em águas marinhas em todo o mundo, incluindo os estuários. A espécie foi descoberta em 1950 na sequência de um surto de intoxicação alimentar no Japão. Em os EUA, a espécie foi isolada pela primeira vez na água do mar, sedimentos, e marisco na região de Puget Sound 6,7. Filtro alimentadores em habitats marinhos, tais como moluscos bivalves, podem abrigar V. parahaemolyticus como parte de sua flora natural 8. Como tal, V. infecções parahaemolyticus em humanos são muitas vezes ligados ao consumo de mariscos contaminados, principalmente mariscos crus ou mal cozidos. Uma rota menos comum de entrada ocorre quando ferida aberta é exposta à água do mar, levando à infecção de pele. A maioria V. cepas parahaemolyticus não causam a doença humana, mas certos subtipos que abrigam fatores de virulência como hemolisina direta termoestável (TDH) são patogênicas. Os sintomas mais prevalentes de origem alimentar V. infecção parahaemolyticus sãodiarreia e dor abdominal, seguida de náuseas, vómitos e febre. Dor de cabeça e arrepios são também relatados. O período de incubação médio é de 15 horas, mas pode ser de até 96 horas após o consumo de uma quantidade suficiente de estirpes patogénicas 9. A doença dura de dois a três dias. Os sintomas de gastroenterite causados por V. parahaemolyticus são amplamente auto-limitada e, por conseguinte, um tratamento especial não é necessário. Os casos leves de gastroenterite podem ser eficazmente tratados por reidratação oral. Mais doenças graves podem ser tratados com antibióticos tais como tetraciclina ou ciprofloxacina 10. A mortalidade é de cerca de 2% para os casos de gastroenterite, mas pode ser tão elevado quanto 29% para os que desenvolvem infecção da corrente sanguínea ou a septicemia. Qualquer pessoa que consome frutos do mar ou tem ferida aberta exposta à água do mar está em risco de V. infecção parahaemolyticus. A forma mais grave da septicemia, doenças com risco de vida, é mais comum em uma subpopulação com o co médica subjacentenditions 11, que incluem alcoolismo, doença hepática, diabetes, doença renal, doença maligna, e outras condições que conduzem a uma resposta imune enfraquecido. Notavelmente, este grupo de indivíduos também está em maior risco de contrair doenças graves causadas por V. vulnificus, que pode ser encontrado em habitats naturais semelhantes a V. parahaemolyticus.
V. parahaemolyticus é rotineiramente isolado utilizando ágar sais de ácido sacarose-tiossulfato-citrato biliares (TCBS) como um meio selectivo e diferencial. Enriquecimento em água peptonada alcalina pode preceder o isolamento em agar TCBS. colónias presumivelmente em TCBS são então testados em uma série de testes bioquímicos e / ou ensaios moleculares visando a presença de genes específicos da espécie. Métodos baseados em PCR são muitas vezes utilizados para confirmar a identidade de V. parahaemolyticus por amplificação do gene da hemolisina termolábeis, tlh 12.
Independentemente do choice de métodos de confirmação, é importante dispor de um meio eficaz para isolar e diferenciar V. parahaemolyticus de outros vibrios marinhos em primeiro lugar. TCBS tem sido rotineiramente usado para diferenciar as espécies dentro do gênero Vibrio acordo com suas habilidades para fermentar sacarose 12. Reacção de fermentação positiva é acompanhada por uma mudança de cor do indicador de pH azul de bromotimol. V. colónias parahaemolyticus são bastante distinta em TCBS, exibindo azul para cor verde. No entanto, este meio não pode facilmente diferenciar V. alginolyticus e V. cholerae. espécies de Proteus-fermentação da sacarose podem produzir colónias amarelas se assemelham V. cholerae ou V. alginolyticus 13. No isolamento inicial em TCBS, V. parahaemolyticus também pode ser identificado erroneamente como Aeromonas hydrophila, Plesiomonas shigelloides e Pseudomonas spp 14. As cepas com ferm sacarose adiadaentação pode ser confundida com outra sacarose não-fermentadoras, Vibrio 13, que incluem V. parahaemolyticus. TCBS foi encontrado para ser não é sensível contra Escherichia coli, putrefaciens Pseudomonas, entre outros. Várias outras espécies render verde para colónias cinzentas que são potencialmente confundidos com V. parahaemolyticus ou V. vulnificus 15. Como resultado, é desejável desenvolver meios alternativos com melhor sensibilidade e especificidade para detectar e isolar V. parahaemolyticus e outras espécies estreitamente relacionadas.
Várias alternativas de mídia têm sido desenvolvidos recentemente. Para além da inclusão de agentes selectivos, mais incorporar substratos cromogénicos para diferenciar espécies com base nas suas actividades enzimáticas diferenciais. Por exemplo, o indoxil-β-glucósido e indoxil-β-galactósido têm sido usadas como os substratos cromogénicos para diferenciar V. páracolónias haemolyticus (que aparecem verde-azulado) dos de V. cholerae (roxo), devido às suas capacidades diferenciais para produzir β-glicosidase e β-galactosidase 16. Diferentes formulações de ágar cromogénico desenvolvidas por diversos grupos foram avaliados e foram relatados para executar comparável ou melhor do que TCBS 17,18,19. Uma vantagem de usar um meio cromogénico é que a coloração do meio circundante é mínima facilitando assim o isolamento de colónias particulares. Neste estudo, avaliou-se a capacidade de um meio cromogénico recentemente formulado para detectar e isolar V. cholerae, V. parahaemolyticus e V. vulnificus; com um foco especial sobre a sua capacidade para diferenciar V. parahaemolyticus a partir de outras espécies.
Protocolo
1. Mídia e Cultura de microbianas Cepas
NOTA: Use técnicas assépticas em todos os experimentos. Usar materiais estéreis. Esterilizar todos os recipientes, ferramentas e reagentes antes de usar. Autoclave todos os resíduos antes do descarte, porque eles são considerados de risco biológico. temperatura e tempo autoclave combinação é ≥121 ° C x ≥15 min durante todos os procedimentos a seguir.
- Para tornar ~ 1-L-agar de soja tríptica (TSA), primeiro adicionar 1 L de água desionizada num balão de Erlenmeyer de 2 L contendo uma barra de agitação magnética. Usar um balão que é pelo menos duas vezes maior do que o volume final. Adicionar 30 g de caldo de soja tríptica em pó e 20 g de agar grânulos para o frasco.
NOTA: Use 2% de agar em vez de 1,5% para limitar enxame de algum Vibrio spp.- Misturar bem, rodando sobre o agitador. Enquanto se agitava, ligue o fogo para ferver a mistura. Retirar o frasco do aquecedor, logo que a mistura começa a ferver. Vagamente cobrir the frasco com uma folha de estanho. Tape o papel alumínio para protegê-lo para o frasco antes de autoclave.
- Para fazer tríptico caldo de soja (TSB), omitir o agar da receita no passo 1.1.
NOTA: Pode usar garrafas em vez de Erlenmeyer. - Para fazer agar de soja tríptica suplementado com cloreto de sódio a 2% ou NaCl (CST), adicionar 20 g de NaCl na mistura antes da agitação e aquecimento. Para fazer caldo de soja tríptica suplementado com 2% de NaCl (TSBs), omitir o agar, adicionar 20 g de NaCl na mistura antes da agitação e aquecimento.
- Para tornar a infusão de coração e cérebro de agar (BHI), suspensão 37 g de BHI em pó e 15 g de grânulos de agar em 1 litro de água purificada. Aquecer com agitação frequente para dissolver o pó. Autoclave. Omitir o agar para fazer caldo BHI.
- Para fazer ágar TCBS, suspensão 89 g do pó em TCBS 1 L de água purificada. Aquecer com agitação frequente e fervura durante 1 min para dissolver completamente o pó. Não autoclave.
- Por todos os meios de agar, arrefecer o agar quente para 45-50 ° C num banho de água. Organizar placas de Petri vazios em pilhas de cinco a seis placas. A partir da parte inferior da pilha, verter o agar derretido em cada placa de Petri para atingir cerca de metade cheio. Feche a tampa placa de Petri após o vazamento. Permitir que o agar solidificar, permitindo que as placas de repouso à temperatura ambiente.
- Use as placas de ágar no dia seguinte ou após 12 horas. Armazenar placas não utilizados em uma geladeira por até duas semanas. Antes da utilização, remover as placas a partir do frigorífico e equilibrar-los à temperatura ambiente durante pelo menos 15 min.
NOTA: agar de um litro faz ~ 45 placas de ágar. Permitir que as placas de ágar para secar suficientemente no dia da preparação, e equilibrar-los à temperatura ambiente após o armazenamento a frio para reduzir eficazmente a propagação das colônias.
- Use as placas de ágar no dia seguinte ou após 12 horas. Armazenar placas não utilizados em uma geladeira por até duas semanas. Antes da utilização, remover as placas a partir do frigorífico e equilibrar-los à temperatura ambiente durante pelo menos 15 min.
- Obter chocolate e de ágar cromogênico placas e equilibrar-los à temperatura ambiente antes de cada experimento.
- Subcultura todos os 54 cepas microbianas apresentados na Tabela 1 a cada poucos days.
- Use um loop de inoculação estéril para transferir culturas a partir de um estoque congelado ou um lote anterior para mídia não-seletivos, como BHI, TSB / TSA ou agar chocolate. Crescer halophilic Vibrio spp. em TSBs / TSAS.
- Para verificar a pureza da cultura, raia todas as estirpes em um padrão que permita a observação das colônias isoladas. Por exemplo, usar um padrão de estrias de três fases para diluir uma grande quantidade de bactérias a quantidade menor, originando eventualmente colónias isoladas.
- Incubar as placas de cabeça para baixo a 35-37 ° C durante até 48 h. Para Campylobacter spp., Tubos ou placas incubar em um frasco de tampa fechada contendo uma bolsa de gás para produzir um ambiente microaerophilic. Observar morfologia das colónias após incubação. Culturas puras deve produzir colónias que exibem uma morfologia de colónia semelhante.
NOTA: Incubar todas as placas de cabeça para baixo para evitar que as gotículas de água condensada formadas no lado inferior da tampa de cair na colonies.
2. Espécies Determinação por PCR
- Realizar tlh-PCR para confirmar a identidade de V. cepas parahaemolyticus. Utilização iniciadores TLH -F (5 'AAA GCG TAT GAT GCA GAA GCA CTG 3') e -R TLH (5 'TCG ACT TTC CAT TAG TTT CTC TGC 3') para amplificar um fragmento de 450 pb do gene de hemolisina 20 termolábil .
- Use um loop de inoculação estéril para transferir algumas colônias isoladas de cada V. estirpe parahaemolyticus a partir TSAS a 5 ml de TSBs. Incubar a 35-37 ° C durante 16-24 hr.
- culturas centrifugar a 14.000 xg por 1 min. Remover o sobrenadante e lavar o sedimento duas vezes com solução salina tamponada com fosfato (PBS). Ferver a suspensão durante 3 minutos para se obter lisado celular.
NOTA: V. parahaemolyticus é fácil de ser lisadas. Portanto reagente de lise não é necessária. É também possível usar um pouco de colónia directamente como molde. - Executar PCR numa reacção de 25-ul volume. Prepara-se uma mistura reaccional contendo uma concentração final de tampão de PCR 1x, MgCl2 1,5 mM, 100 uM de cada dNTP, 1 pM de cada iniciador, 1 U de Taq polimerase e 1 ml de lisado celular. Após pré-incubação a 94 ° C durante 5 min, executar 35 ciclos de amplificação de 94 ° C durante 30 seg, 58 ° C durante 30 seg, e 72 ° C durante 60 seg 21.
- alíquotas de carga de amplicão 5-ul em géis de agarose a 1,5%. Ligue a fonte de alimentação para iniciar a electroforese. Visualizar a presença ou ausência de produtos de amplificação sob iluminação com UV, após coloração com brometo de etídio.
3. Crescimento na selectivo e diferencial de mídia
- Dois a quatro dias antes do experimento, raia todas as cepas microbianas apresentados na Tabela 1 no meio não selectivo (TSAS, BHI ou agar chocolate) para o isolamento de colónia. Incubar as placas a 35-37 ° C durante 48 h. Verifique a pureza das culturas através da observação da morfologia das colónias após incubação. Culturas puras deve produzir colónias que exibem uma morfologia de colónia semelhante.
- Transferir algumas colónias isoladas a partir do passo 3.1 em 5 mL de caldo. Incubar os tubos a 35-37 ° C durante 16-24 hr.
Nota: Use as colónias jovens, que são menos de quatro dias de idade, para preparar culturas durante a noite em todos os experimentos. - Streak uma ansa de culturas durante a noite em meio selectivo e diferencial (TCBS e agar cromogênico) para o isolamento de colónia. Incubar as placas a 35-37 ° C durante até 96 h.
- Grave o crescimento global de todas as estirpes, examinando tanto a densidade da cultura no prato e o tamanho de colônias isoladas. Grave a cor das colónias sob a luz ambiente e / ou UV. Nota outras características da colônia isolada como a altitude, a margem e forma.
Ensaio 4. Recuperação
- Seleccionar um subconjunto representativo de V. cepas parahaemolyticus (n = 14) que abrangem diferentes sorotipos e origens de isolamento 22. Inocular culturas jovens das placas em 5 ml de TSBs. Incubar os tubos a 35-37 ° C durante 16-24 hr.
- Realizar um método de contagem padrão em placa das culturas durante a noite, como descrito abaixo.
- Vortex para misturar as culturas durante a noite bem. Adicione uma dobra em 10 ou 10 -1 de diluição por transferência de 100 uL da cultura durante a noite para um tubo contendo 900 jil de PBS. Vortex para misturar bem.
- Usar uma nova ponta de pipeta para transferir 100 ul a partir do tubo 10 -1 de diluição para outro tubo contendo 900 ul de PBS. Vortex para misturar bem. Isto constitui 10 -2 diluição. Repetir o processo sequencial para se obter 10 -7 diluição.
- Usando os 10 -4 a 10 -7 tubos de diluição, placa 100 ul cada um no, TCBs e CST placas de ágar cromogênico.
NOTA: O factor de diluição (DF) sobre a placa torna-se 10 -5 a 10 -8, respectivamente. - Espalhe as alíquotas uniformemente em tele agar superfície.
NOTA: É bem usar o mesmo espalhador por pressão sobre o mesmo meio, enquanto a alíquota mais diluída é espalhado primeiro (ou seja, a partir de 10 -8 a 10 -5). Não use o mesmo espalhador para diferentes mídias. - Incubar as placas a 35-37 ° C durante até 96 h. Contagem das colónias nas placas. Ignorar placas de rolamento colónias que são demasiado numerosas para contar (TnTc) ou menos do que 25. Calcular CFU / ml de acordo com o seguinte:
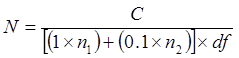
Onde
N = o número de células no tubo não diluído, expresso em CFU / ml ou CFU / g
C = o número total de colónias contadas em placas que dão 25-300 colónias
N 1 = o número de placa (s), onde as colónias são contadas a partir do DF inferior
n = 2 o número de placa (s), onde as colónias são contadas a partir da diluição de 10 vezes subsequente
dF = factor de diluição menor (isto é, a diluição mais concentrada)
- Compare UFC / ml entre os diferentes meios de comunicação. Use UFC / ml em TSAS como 100%, calcular a recuperação% de V. parahaemolyticus cultivadas em agar cromogénico e TCBS.
Ensaio 5. Concorrência
- Escolha um subconjunto de estirpes que exibem diferentes morfologias colônia na agar cromogênico e TCBS.
NOTA: Dessa forma, será possível a contagem das colónias originado V. única parahaemolyticus, apesar da presença de outras espécies no inoculo.- Escolha um V. estirpe parahaemolyticus que produz os turquesa e ciano colónias esperado em TCBS eo agar cromogênico, respectivamente.
- Escolha um V. não- parahaemolyticus e uma espécie não-Vibrio que não crescem em qualquer um destes meios de comunicação, ou exibem cor diferente colônia.
NOTA: Por exemplo, V. metschnikovii cresce muito pouco oN TCBS e não crescem em agar cromogénico. Shigella sonnei que não crescem em TCBS mas produz colónias magenta sobre o ágar cromogénico.
- Após a seleção das estirpes anteriores, preparar caldos de cultura durante a noite usando colônias isoladas cultivadas em meio não selectivo.
- Faça culturas durante a noite de V. parahaemolyticus e V. metschnikovii transferindo algumas colónias isoladas a partir de TSAS a 5 ml de TSBs. Incubar a 35-37 ° C durante 16-24 hr.
- Adicione culturas de uma noite de Shigella sonnei, transferindo-se algumas colónias isoladas a partir de agar de BHI para 5 ml de caldo BHI. Incubar a 35-37 ° C durante 16-24 hr.
- Para cada cepa, realizar uma série de diluição semelhante ao Passos 4.2.1 e 4.2.2. Placa diluições apropriadas em TSAS ou BHI para determinar UFC / ml da cultura durante a noite, utilizando-se a equação mostrado no Passo 4.2.5.
NOTA: Normalmente, 100 ul da diluição de 10 -5 a 10 -7tubos funciona para a maioria das culturas. Use os valores de UFC / mL para trás calcular a quantidade exata de células usadas na próxima etapa. Os valores de CFU / ml calculados são obtidos depois de incubação, embora os seguintes passos são executados na mesma data no passo 5.3. - Usando as culturas durante a noite e tubos de diluição nos Passos 5.2 e 5.3, misturar diferentes quantidades de um V. estirpe parahaemolyticus e V. não espécies parahaemolyticus. Por exemplo, misturar 500 ul da diluição -5 tubo 10 de V. parahaemolyticus com 500 ul das culturas durante a noite de V. metschnikovii.
NOTA: Esta mistura simula fundo alta microflora. Para simular um fundo baixo microflora, misturar 500 ul cada do tubo 10 -5 diluição de ambas as espécies. - Espalhe 100 uL da mistura de cada um no, TCBS e TSAS placas de agar cromogénicos.
- Após incubação a 35-37 ° C durante até 96 h, a contagem de colónias de V. parahaemolyticus e o V. não- espécies parahaemolyticus com base na sua diferença no crescimento e morfologia da colônia no ágar cromogênico e TCBS.
NOTA: Por exemplo, se o V. não- O parahaemolyticus espécies não crescem em agar cromogénico e TCBS, todas as colónias será de V. parahaemolyticus. Se o V. não- espécies parahaemolyticus cresce em ambos os meios, apenas as colónias azul-turquesa em TCBs e ciano colónias em agar cromogênico será de V. parahaemolyticus. O V. não- espécies parahaemolyticus podem ou não exibem uma morfologia de colónia semelhante a V. parahaemolyticus em TSAS.- Se o V. não- espécies parahaemolyticus cresce de modo semelhante a V. parahaemolyticus neste meio não selectivo, divida o número de colónias por dois para obter números de V. parahaemolyticus somente. Comparar o número de colónias real com a contagem esperada derivada da etapa 5.3.
6. EffECTS de ostra homogeneizados
- Pesar ≥50 g carne de ostra de ≥12 moluscos incluindo carne e bebidas alcoólicas.
- Adicionar uma quantidade igual de PBS à carne de ostra e bebidas alcoólicas. Mistura-se a mistura a alta velocidade durante 90 segundos. Isto constitui 2 -1 homogenato diluído de ostras.
- Adicionar 100 g de 2 -1 homogenato diluído de ostra a 400 g de PBS. Use uma escala para medir o peso, não volume. Mistura-se a mistura a alta velocidade durante 1 minuto. Autoclave a homogenato de ostras.
NOTA: Este será o homogeneizado ostra usado para spiking.
- Repetir o ensaio de recuperação (Passo 4), na presença de homogenato de ostras.
- Após a 500 g de ostra homogeneizado esfriar, adicione 100 ml de V. parahaemolyticus culturas durante a noite cultivadas em TSBs a ele. Determinar a quantidade real de V. células parahaemolyticus no inóculo por realização dos procedimentos de contagem padrão em placas descritas no Passo 4.2.
- Repcomer o ensaio de competição (Passo 5) na presença de homogenato de ostras.
- Após a 500 g de ostra homogeneizado esfria, adicionar 100 ul cada um culturas durante a noite de V. parahaemolyticus e V. não- parahaemolyticus a ele. Determinar a quantidade real de bactérias células do inóculo por realização dos procedimentos de contagem padrão em placas descrito no Passo 4.2
- Misturar as células bacterianas com homogenato de ostra bem utilizando um homogeneizador.
NOTA: Após a mistura, o homogeneizado ostra contendo as células intencionalmente adicionados é chamado cravado ostra homogeneizado. - Fazer diluições do homogeneizado ostra cravada para se obter 10 -1 a -3 10 tubos de diluição de acordo com os procedimentos descritos no Passo 4.2.2. Espalhe 100 ul de cada diluição para o cromogénico TCBS e TSAS ágar,. Incubar as placas a 35-37 ° C durante até 96 h.
- Compare o número de colónias real sobre agar cromogênico e TCBS com o experado contagem de colónias deduzida a partir de Passos 6.2.1 e 6.3.1.
NOTA: Por exemplo, se um tubo de V. parahaemolyticus cultura durante a noite contém 10 8 CFU / ml, um inoculo de 100 ul significa que 10 7 células são adicionadas a 500 g de homogeneizado de ostra, obtendo-se 5 x 10 4 células / g. Após a diluição e em placas, a placa com df = 10 -2 deve render 500 colónias; enquanto que com df = 10 -3 deve rendimento de 50 colónias. Estas são as contagens de colónias esperado.
Resultados
Neste estudo, 54 cepas microbianas foram montados, que incluiu 22 cepas dentro do V. espécies parahaemolyticus, 19 outras espécies de Vibrio, e 13 espécies de Vibrio não (Tabela 1). A maioria V. cepas parahaemolyticus ou foram recebidos de FDA, CDC ou de outros departamentos de saúde estaduais. Eles representam diversos sorotipos e fontes de isolamento. Estas estirpes foram previamente identificados pelos órgão...
Discussão
Este estudo centra-se no desenvolvimento e avaliação meios de cultura. Convencionalmente, TCBS é o selectivo e diferencial meio utilizado para isolar e detectar V. parahaemolyticus, V. cholerae e V. vulnificus 12. No entanto, limitações têm sido relatados para este meio, tais como a incapacidade para diferenciar V. cholerae a partir de outras espécies de Vibrio. Sacarose e indicador de pH são os agentes de diferenciação de TCBS. Assim, a produção de ácido por f...
Divulgações
Some media were generously provided by Hardy Diagnostics, Santa Maria, CA. Thorsen conducted this study while a student at California Polytechnic State University. He is currently an employee of Hardy Diagnostics.
Agradecimentos
Agradecemos a M. Channey, E. Chau, e K. Tomas por sua assistência no projeto. suprimentos do projeto foram parcialmente financiado pela California Polytechnic State University.
Materiais
| Name | Company | Catalog Number | Comments |
| Reagent/Equipment | |||
| Agar | Fisher Scientific | DF0140-15-4 | may use other brands |
| Autoclave | Any | ||
| BHI powder | Fisher Scientific | DF0418-17-7 | may use other brands |
| Blender | Any | to blend oyster meat | |
| CampyGen gas generator | Hardy Diagnostics | CN035A | to provide a microaerophilic atmosphere; may use other brands |
| Chocolate agar plates | Hardy Diagnostics | E14 | may use other brands |
| Common PCR reagents (dNTPs, MgCl2, Taq Polymerase) | Any | or use PCR beads (Fisher Sci 46-001-014) | |
| Culture tubes | Fisher Scientific | S50712 | may use other brands |
| Eppendorf tubes | Fisher Scientific | S348903 | may use other brands |
| Gel doc | Any | ||
| HardyChrom Vibrio agar plates | Hardy Diagnostics | G319 | This study evaluates this medium |
| Incubator | Any | ||
| Inoculating loops | Fisher Scientific | 22-363-606 | 10 microliter-size was used in this study |
| NaCl | Fisher Scientific | BP358-212 | may use other brands |
| Oysters | Any | ||
| PBS | Fisher Scientific | R23701 | may use other brands |
| Petri dish | Fisher Scientific | FB0875713 | may use other brands |
| Pipette and tips | Any | Sterilized tips | |
| Primers for tlh | IDT DNA | ||
| Scale | Any | ||
| Spreader | Fisher Scientific | 08-100-11 | Beads may be used instead |
| Stomacher blender | Stomacher | 400 | Samples were homogenized at 200 rpm for 30 sec. Other homogenizer can be used. |
| Sterile filter bags for blenders | Fisher Scientific | 01-812-5 | |
| TCBS powder | Hardy Diagnostics | 265020 | This study evaluates this medium |
| Thermocycler | Any | ||
| TSB powder | Fisher Scientific | DF0370-07-5 | may use other brands |
| UV viewing cabinet | Any | Emit long-wave UV light | |
| Water bath | Any | | |
| Name | Sources | Catalog Number | Comments |
| Bacterial species and strains | |||
| Aeromonas hydrophila | ATCC | ||
| Candida albicans | ATCC | ||
| Campylobacter jejuni | ATCC | ||
| Escherichia coli | ATCC | ||
| Proteus mirabilis | ATCC | ||
| Pseudomonas aeruginosa | ATCC | ||
| Staphylococcus aureus | ATCC | ||
| Salmonella Choleraesuis | ATCC | ||
| Shigella boydii | ATCC | ||
| Shigella flexneri | ATCC | ||
| Shigella sonnei | ATCC | ||
| Vibrio alginolyticus | ATCC | ||
| V. cholerae (serotypes include O139, O1, non O1, El Tor biovars) | FDA, ATCC | ||
| V. damsela | FDA | ||
| V. fisheri | Environment | ||
| V. fluvialis | CDC | ||
| V. furnissii | CDC | ||
| V. hollisae | FDA | ||
| V. metschnikovii | ATCC | ||
| V. mimicus | FDA | ||
| V. parahaemolyticus (serotypes include O3:K6, O1:K56, O4:K8, O5:K15, O8, etc.) | ATCC, FDA, CDC, Environment | ||
| V. proteolyticus | FDA | ||
| V. vulnificus | FDA |
Referências
- Yeung, P. S., Boor, K. J. Epidemiology, pathogenesis, and prevention of foodborne Vibrio parahaemolyticus infections. Foodborne Pathog. Dis. 1 (2), 74-88 (2004).
- Yeung, P. S. M., Boor, K. J., Faruque, S. M. Epidemiology, molecular biology, and detection of foodborne Vibrio parahaemolyticus infections. Foodborne and Waterborne Bacterial pathogens: Epidemiology, Evolution and Molecular Biology. , 153-184 (2012).
- Centers for Disease Control and Prevention. Vital Signs: Incidence and Trends of Infection with Pathogens Transmitted Commonly Through Food - Foodborne Diseases Active Surveillance Network, 10 U.S. Sites, 1996-2010. Morbidity and Mortality Weekly Report. 10, 1996-2010 (2011).
- . Summary of human Vibrio cases reported to CDC, 2008 Available from: https://stacks.cdc.gov/view/cdc/21591 (2008)
- Scallan, E., et al. Foodborne illness acquired in the United States - major pathogens. Emerg. Infect. Dis. 17 (1), 7-15 (2011).
- Baross, J., Liston, J. Occurrence of Vibrio parahaemolyticus and related hemolytic vibrios in marine environments of Washington State. Appl. Microbiol. 20 (2), 179-186 (1970).
- Baross, J., Liston, J. Isolation of Vibrio parahaemolyticus from the Northwest Pacific. Nature. 217 (5135), 1263-1264 (1968).
- Kueh, C. S., Chan, K. Y. Bacteria in bivalve shellfish with special reference to the oyster. J. Appl. Bacteriol. 59 (1), 41-47 (1985).
- Food and Drug Administration. . Bad Bug Book, Foodborne Pathogenic Microorganisms and Natural Toxins. , (2012).
- Qadri, F., et al. Adaptive and inflammatory immune responses in patients infected with strains of Vibrio parahaemolyticus. J. Infect. Dis. 187 (7), 1085-1096 (2003).
- MacFaddin, J. F. . Media for isolation-cultivation-identification-maintenance of medical bacteria. 1, (1985).
- Bottone, E. J., Robin, T. Vibrio parahaemolyticus: suspicion of presence based on aberrant biochemical and morphological features. J. Clin. Microbiol. 8 (6), 760-763 (1978).
- Lotz, M. J., Tamplin, M. L., Rodrick, G. E. Thiosulfate-citrate-bile salts-sucrose agar and its selectivity for clinical and marine vibrio organisms. Ann. Clin. Lab. Sci. 13 (1), 45-48 (1983).
- Hara-Kudo, Y., Nishina, T., Nakagawa, H., Konuma, H., Hasegawa, J., Kumagai, S. Improved method for detection of Vibrio parahaemolyticus in seafood. Appl. Environ. Microbiol. 67 (12), 5819-5823 (2001).
- Eddabra, R., Piemont, Y., Scheftel, J. M. Evaluation of a new chromogenic medium, chromID Vibrio, for the isolation and presumptive identification of Vibrio choleare and Vibrio parahaemolyticus from human clinical specimens. Eur. J. Clin. Microbiol. Infect. Dis. 30 (6), 733-737 (2011).
- Kodaka, H., Teramura, H., Mizuochi, S., Saito, M., Matsuoka, H. Evaluation of the Compact Dry VP method for screening raw seafood for total Vibrio parahaemolyticus. J. Food. Prot. 72 (1), 169-173 (2009).
- Su, Y. C., Duan, J., Wu, W. H. Selectivity and specificity of a chromogenic medium for detecting Vibrio parahaemolyticus. J. Food Prot. 68 (7), 1454-1456 (2005).
- Bej, A. K., Patterson, D. P., Brasher, C. W., Vickery, M. C., Jones, D. D., Kaysner, C. A. Detection of total and hemolysin-producing Vibrio parahaemolyticus in shellfish using multiplex PCR amplification of tl, tdh and trh. J. Microbiol. Methods. 36 (3), 215-225 (1999).
- Yeung, P. S. M., DePaola, A., Kaysner, C. A., Boor, K. J. A PCR assay for specific detection of the pandemic Vibrio parahaemolyticus O3:K6 clone from shellfish. J. Food Sci. 68 (4), 1459-1466 (2003).
- Yeung, P. S. M., Hayes, M. C., DePaola, A., Kaysner, C. A., Kornstein, L., Boor, K. J. Comparative phenotypic, molecular, and virulence characterization of Vibrio parahaemolyticus O3:K6 isolates. Appl. Environ. Microbiol. 68 (6), 2901-2909 (2002).
- Duan, J., Su, Y. -. C. Comparison of a chromogenic medium with thiosulfate-citrate-bile salts-sucrose agar for detecting Vibrio parahaemolyticus. J. Food Sci. 70, M125-M128 (2005).
- Pinto, A. D., Terio, V., Novello, L., Tantillo, G. Comparison between thiosulphate-citrate-bile salt sucrose (TCBS) agar and CHROMagar Vibrio for isolating Vibrio parahaemolyticus. Food Control. 22 (1), 124-127 (2011).
- Canizalez-Roman, A., Flores-Villaseñor, H., Zazueta-Beltran, J., Muro-Amador, S., Leòn-Sicairos, N. Comparative evaluation of a chromogenic agar medium-PCR protocol with a conventional method for isolation of Vibrio parahaemolyticus strains from environmental and clinical samples. Can J Microbiol. 57 (2), 136-142 (2011).
- Kriem, M. R., et al. Prevalence of Vibrio spp. in raw shrimps (Parapenaeus longirostris) and performance of a chromogenic medium for the isolation of Vibrio strains. Lett Appl Microbiol. 61 (3), 224-230 (2015).
- Food Drug Administration. Statistical guidance on reporting results from studies evaluating diagnostic tests. http://www.fda.gov/RegulatoryInformation/Guidances/ucm071148.htm. , (2007).
- Burd, E. M. Validation of laboratory-developed molecular assays for infectious diseases. Clin Microbiol Rev. 23 (3), 550-576 (2010).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados