Method Article
Isolation und Fluoreszenz-Imaging für Single-Particle-Rekonstruktion der Chlamydomonas Centriolen
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Wir haben eine Strategie, um zu reinigen und Bild eine große Anzahl von Centriolen in verschiedenen Ausrichtungen für Höchstauflösung Mikroskopie und Single-Teilchen im Durchschnitt entwickelt.
Zusammenfassung
Centriolen sind großen makromolekularen Versammlungen wichtig für die ordnungsgemäße Durchführung des grundlegenden biologischen Zellprozesse wie Zellteilung, Zelle Motilität oder Zelle signalisieren. Die Grünalge Chlamydomonas Reinhardtii erweist sich eine aufschlussreiche Modell in der Studie der Proteinzusammensetzung Zentriol Architektur und Funktionalität. Trotz der großen Fortschritte in Richtung Verständnis centriolar Architektur gehört zu den aktuellen Herausforderungen, die präzise Lokalisierung der centriolar Komponenten innerhalb der strukturellen Regionen die Zentriol festzustellen, um besser zu verstehen, ihre Rolle bei der Zentriol Biogenese. Eine große Einschränkung liegt in der Auflösung der Fluoreszenz-Mikroskopie, die die Interpretation der Protein-Lokalisierung in dieser Organellen mit Dimensionen in der Nähe der Beugungsgrenze erschwert. Um diese Frage zu lösen, bieten wir eine Methode, um zu reinigen und eine große Anzahl von C. Reinhardtii Centriolen mit unterschiedlichen Ausrichtungen Höchstauflösung Mikroskopie mit Bild. Diese Technik ermöglicht die Weiterverarbeitung von Daten durch fluoreszierende Single-Teilchen im Durchschnitt (Fluo-SPA) wegen der großen Zahl der Centriolen erworben. Fluo-SPA erzeugt Durchschnitte der gebeizt C. Reinhardtii Centriolen in verschiedenen Ausrichtungen, und erleichtert so die Lokalisierung von unterschiedlichen Proteinen in centriolar Sub-Regionen. Wichtig ist, kann diese Methode von anderen Spezies oder anderen großen makromolekularen Versammlungen auf Bild Centriolen angewendet werden.
Einleitung
Die Zentriol ist ein evolutionär konservierte Organell, das liegt an den Kern der Centrosome in tierischen Zellen und kann als eine Basaltemperatur (bezeichnet als Centriolen nachstehend) Vorlage Cilien oder Flagellen in vielen Eukaryoten1,2. Als solche sind Centriolen entscheidend für grundlegende Zelle biologischen Prozesse von Spindel Baugruppe zu Zelle signalisieren. Daher wurden Mängel in Zentriol Montage oder Funktion mit mehreren menschlichen Krankheiten einschließlich Ciliopathies und Krebserkrankungen3verbunden.
Centriolen sind multiplizieren, symmetrisch, Mikrotubuli Triplett-basierte zylindrische Strukturen, die sind, in der Regel ~ 450 nm lang und ~ 250 nm breite4,5,6,7. Konventionelle Elektronenmikroskopie und Cryo-Elektron Tomographie der Centriolen verschiedener Arten haben gezeigt, dass Centriolen entlang ihrer Längsachse mit drei verschiedene Regionen polarisiert sind: einen proximalen Bereich, einen zentralen Kern und einem distalen Region5 , 7 , 8 , 9 , 10 , 11. wichtig ist, jede dieser Regionen zeigt strukturelle Besonderheiten. Zunächst enthält das Lumen der 100 nm langen proximalen Bereich die Wagenrad-Struktur an die Mikrotubuli Triplett durch die Pinhead Element12angeschlossen. Zweitens enthält der zentralen Region von 300 – 400 nm langen faserige dichten in das Lumen und strukturelle Eigenschaften entlang der Innenseite der Mikrotubuli: der y-förmigen Linker, die C-Röhrchen Tail und die A-Röhrchen stub9. Schließlich weist der 50 – 100 nm distalen Region Sub distale und distale Gliedmaßen, die den distalen Teil des Zentriol5,13umgeben.
Die letzten zwei Jahrzehnte waren geprägt von der Entdeckung einer wachsenden Zahl von centriolar Proteinen, was zu einer aktuellen Schätzung etwa 100 verschiedene Proteine, die als Teil der Zentriol14,15,16, 17. Trotz dieser Fortschritte bleibt die präzise Lokalisierung dieser Proteine innerhalb der Zentriol schwer, vor allem in strukturellen Unterregionen. Wichtiger ist es entscheidend für ein besseres Verständnis ihrer Funktion, strukturelle Regionen die Zentriol eine präzise Lokalisierung zuweisen. In dieser Hinsicht wurden C. Reinhardtii Centriolen beide Aspekte maßgeblich durch erste Abgrenzung der verschiedenen strukturellen Merkmale entlang der Zylinder9,18,19, die dann erlaubt Forscher, die Lokalisierung einer Teilmenge von Proteinen mit Fluoreszenz-Mikroskopie zu einer Sub-strukturelle Region zu korrelieren. Dazu gehören beispielsweise die Proteine Bld12p und Bld10p, die in den proximalen Bereich und in der Wagenrad Struktur insbesondere lokalisieren20,21,22,23. Die Liste der Unterkonstruktion lokalisiert Proteine enthält auch POB15 und POC16, zwei neue Proteine durch Massenspektrometrie identifiziert, die die innere zentrale Kernregion C. Reinhardtii Centriolen17schmücken.
Dieses Dokument enthält eine vollständige Beschreibung der Methode entwickelt, um zu isolieren und Bild C. Reinhardtii Centriolen für nachfolgende Höchstauflösung Mikroskopie und Single-Teilchen im Durchschnitt. Um dieses Ziel zu erreichen, ist es wichtig, die technischen Grenzen abzugrenzen, die überwunden werden müssen. Erstens kann Zentriol Reinigung die Gesamtarchitektur, mit oft verloren werden, während die verschiedenen Schritte der Isolierung9Wagenrad-Struktur beeinflussen. Zweitens sind die Abmessungen der Zentriol sehr nah an die Beugungsgrenze in der optischen Mikroskopie. In der Tat ist die laterale Auflösung, die in der konfokalen Mikroskopie gewonnen werden können rund 200 nm24, ähnlich wie der Durchmesser der Zentriol und die Auflösung in der Z-Achse ist über 2 – 3 X niedriger, führt zu einem anisotropen Volumen. Drittens könnte die Heterogenität der Antikörper labeling und Zentriol Ausrichtung die Auslegung erforderlich, um ein Protein in einer bestimmten centriolar Sub-Region zu lokalisieren einschränken. Schließlich gibt es die Centriolen in nur zwei Kopien pro Zelle, macht es schwierig, eine große Anzahl von Bildern zu erwerben und eine eindeutige Zentriol Orientierung zu finden. Um diese technischen Probleme zu umgehen, haben wir eine Methode, die stützt sich auf die Anwendung Höchstauflösung Mikroskopie über die große Zahl der isolierten Centriolen, die verschiedenen Orientierungen annehmen. Zunächst beschreiben wir ein Protokoll C. Reinhardtii Centriolen zu reinigen, die Reinigung von strukturell intakt Centriolen und Procentrioles, enthält das Wagenrad ermöglicht. Dann beschreiben wir Schritt für Schritt Protokoll um die Centriolen auf Deckgläsern für Bildgebung durch konventionelle oder Höchstauflösung Fluoreszenz-Mikroskopie zu konzentrieren. Dieser wichtige Schritt ermöglicht zur Erhöhung der Zahl der Centriolen abgebildet in mehrere Richtungen. Zu guter Letzt werden wir beschreiben, ein Verfahren zum Ausführen, Single-Teilchen im Durchschnitt auf Daten, die auf Fluoreszenz Mikroskope, die erleichtert die Erkennung der Centriolen in verschiedenen Ausrichtungen. Insgesamt kann diese Methode von verschiedenen Arten oder anderen großen makromolekularen Versammlungen auf Bild Centriolen angewendet werden.
Protokoll
(1) Medien Vorbereitung für C. Reinhardtii Kultur und Zentriol Isolation
-
Vorbereitung von Medien auf Zellkulturen C. reinhardtii
Hinweis: Die folgenden Schritte beschrieben der Vorbereitung der Stammlösungen für 1 x TAP (Tris-Acetat Phosphat) Medium.- Bereiten Sie einen Phosphatpuffer (pH 7), durch Mischen 250 mL 1 M K2HPO4 (174,2 g K2HPO4 zu 1 L mit destilliertem Wasser ergänzt) mit ~ 170 mL 1 M KH2PO4 (136,09 g KH2PO4 in 1 L). Stellen Sie die Mischung um pH 7 zu erreichen.
- Ansetzen Sie Lösung (40 X) durch das Mischen von 96,8 g Tris, 40 mL Phosphatpuffer (pH 7) und 40 mL Essigsäure, und passen Sie die Lösung 1 L mit destilliertem Wasser.
- Bereiten Sie Lösung B (40 X) mit 16 g NH4Cl, CaCl22 g und 4 g MgSO4. Seien Sie vorsichtig, CaCl2 in destilliertem Wasser getrennt aufzulösen, bevor Sie die anderen Komponenten hinzufügen. Passen Sie die Lösung für 1 L mit destilliertem Wasser.
- Hutners Spurenelemente Puffer25 wie folgt vorbereiten.
- Für 1 L Puffer, jede Verbindung in das angegebene Volumen des Wassers auflösen: EDTA binatrium Salz (50 g in 250 mL), ZnSO4.7H2O (22 g in 100 mL), H3BO3 (11,4 g in 200 mL), MnCl2.4H2O (5,06 g in 50 mL) , CoCl2.6H2O (1,61 g in 50 mL), CuSO4.5h2O (1,57 g in 50 mL) (NH4) 6Mo7O24.4H2O (1,10 g in 50 mL) und FeSO4.7H2O (4,99 g in 50 mL).
Hinweis: EDTA sollten in kochendem Wasser aufgelöst und FeSO4 sollte letzten vorbereitet werden, um Oxidation zu vermeiden. - Alle Lösungen außer der EDTA vermischen und zum Kochen bringen. Fügen Sie dann die EDTA, und die Lösung sollte grün. Nach der Auflösung alles, Abkühlen der Lösung auf 70 ° C. Zugeben Sie bei dieser Temperatur 85 mL heiße 20 % KOH-Lösung (20 g in 100 mL). Passen Sie die Lösung für 1 L mit destilliertem Wasser bei Raumtemperatur (RT).
- Der Kolben einen Baumwoll-Stecker hinzu und lassen Sie die Lösung schütteln stehen für 1 oder 2 Wochen, 1 x pro Tag. Die Lösung sollte zunächst grün und dann violett, verlassen einen rostbraune Niederschlag; Entfernen Sie den Niederschlag mit Filterpapier, bis die Lösung klar ist. Die Aliquote Einfrieren und Lagerung bei-20 ° C.
- Für 1 L Puffer, jede Verbindung in das angegebene Volumen des Wassers auflösen: EDTA binatrium Salz (50 g in 250 mL), ZnSO4.7H2O (22 g in 100 mL), H3BO3 (11,4 g in 200 mL), MnCl2.4H2O (5,06 g in 50 mL) , CoCl2.6H2O (1,61 g in 50 mL), CuSO4.5h2O (1,57 g in 50 mL) (NH4) 6Mo7O24.4H2O (1,10 g in 50 mL) und FeSO4.7H2O (4,99 g in 50 mL).
- Bereiten Sie 1 x TAP Mittel-bis C. Reinhardtii Zellen wachsen durch Mischen der folgenden Komponenten: 25 mL der Lösung A (40 X) und 25 mL der Lösung B (40 X) mit 1 mL von Hutners Spurenelementen Puffer25, und passen Sie die Mischung auf 1 L mit destilliertem Wasser. Sterilisieren Sie die Mischung mit einem 0,4 µm-Filter.
-
Medien-Vorbereitung für Zentriol Reinigung
- Bereiten Sie einen Deflagellation-Puffer mit 5 % Saccharose in 10 mM HEPES (pH 7) zu einem Endvolumen von 500 mL mit destilliertem Wasser angepasst.
- 250 mL 0,5 M Essigsäure vorzubereiten.
- 250 mL einer 1 M K-Rohre-Stammlösung (pH 7,2) vom ersten Hinzufügen von 50 mL H2O, die Rohre Pulver aufzulösen vorbereiten, dann hinzufügen 10 N KOH bis die Lösung klar zu werden beginnt. Titrieren Sie bis pH 7,2 mit 10 N und 1 N KOH und passen Sie die Lösung zu einem Endvolumen von 250 mL mit H2O.
- 5 Saccharose-Lösungen (w/w) wie folgt vorbereiten.
Hinweis: Alle Saccharose-Lösungen müssen gefiltert werden, nachdem eine Spritze mit einem 0,4 µm Filter Solubilisierung gesteckt. Beachten Sie, dass die 60 % und 70 % Saccharose-Lösungen schwer sind zu lösen und sollten in einem Wasserbad bei 60 ° C zur Erleichterung der Solubilisierung vorgewärmt gelegt werden. Mischen Sie alle 10 Minuten, bis der Zucker vollständig aufgelöst ist.- Um 25 % Saccharose vorzubereiten, wiegen Sie 25 g Saccharose und stellen Sie das Gewicht 100 g durch Zugabe von 10 mM K-Rohre (pH 7,2).
- Zur Vorbereitung von 60 % Saccharose, wiegen 60 g Saccharose und stellen das Gewicht auf 100 g mit 10 mM K-Rohre (pH 7,2).
- Die Saccharose-Gradienten Saccharose-Lösungen vorbereiten. Bereiten Sie 40 % Saccharose, indem mit einem Gewicht von 40 g Saccharose und Anpassung der Lösung auf 100 g mit 10 mM K-Rohre (pH 7,2). In ähnlicher Weise bereiten Sie 50 % (w/w) und 70 % (w/w) Saccharose.
- Speichern Sie die Lösungen bei-20 ° C. Achten Sie darauf, die Saccharose-Lösungen nach dem Auftauen richtig aufzuwirbeln.
- Bereiten Sie 100 mL Lyse-Puffer durch Mischen 1 mM HEPES (pH 7), 0,5 mM MgCl2, und 1 % NP-40 und halten Sie es bei 4 ° C. Bereiten Sie dieser Puffer frisch immer am Tag des Experiments zu. Fügen Sie Anti-Protease Tabletten am Tag der Zentriol Isolation.
- Bereiten Sie 1 x Phosphat-Puffer Kochsalzlösung (PBS) (pH 7,4) durch Mischen 8 g NaCl, KCl, 1,44 g Na2HPO4, 2G-und 0,24 g KH2PO4 in 800 mL destilliertem H2O. Adjust pH-Wert 7,4 mit HCl. bringen das Volumen der Mischung auf 1 L mit destilliertem H2O.
2. Isolierung von C. Reinhardtii Centriolen
Hinweis: Siehe Abbildung 1.
-
Kultur und den Ausbau der C. Reinhardtii Zellen
- Am Abend am 1. Tag, impfen eine cw15 - Sorte aus einer festen Platte in eine Kultur Erlenmeyerkolben mit 10 mL 1 x tippen Sie auf. Wachsen Sie die Zellen unter weißen Leuchtstoffröhren (60 µE/m2s) für 2 – 3 Tage bei 23 ° C.
- Am 3. Tag, verdünnen die Kultur 10 X (bis 100 mL) 1 X tippen Sie auf und wachsen die Zellen unter Licht für 2 – 3 Tage bei 23 ° C.
- Am 6. Tag, verdünnen die Kultur 10 X in 1 X tippen, um 1 L Kultur zu erhalten. Wachsen Sie die Zellen unter Licht bei 23 ° C, bis die Kultur eine dunkelgrüne Farbe erreicht, die eine ungefähre Zelldichte von ~ 1 x 107 Zellen/mL26 (Tag 9 – 10) angibt.
-
Reinigung von C. Reinhardtii Centriolen
- Zentrifugieren Sie die cw15 - Zellen bei 600 X g für 10 min in 50 mL konische Röhrchen. Waschen Sie die Pellets von Zellen 1 X mit 50 mL 1 X PBS und drehen Sie es bei 600 X g für 10 min. Aufschwemmen das Pellet in 100 mL Raumtemperatur Deflagellation Puffer mit einer Pipette.
- Deflagellate die Zellen mit einem pH-Schock, indem Sie langsam Tropfen 0,5 M Essigsäure zu einem endgültigen pH-Wert von 4,5-4,7 auf einen Magnetrührer und inkubieren Sie die Zellen, für 2 min. langsam Tropfen 1 N KOH pH 7.0 wiederherstellen.
- Zentrifugieren Sie die Zellen bei 600 X g für 10 min, eine freistehenden Flagellen zu entfernen. Entfernen Sie den überstand zu und speichern Sie das Pellet auf Eis. Waschen Sie das Pellet 2 X mit 50 mL 1 X PBS bei 4 ° c vorgekühlt Dann drehen Sie das Pellet 600 X g für 10 min bei 4 ° c
- Aufschwemmen Sie das Pellet in 30 mL 1 X PBS und laden Sie der Suspension auf eine 25 %-Saccharose-Kissen von 20 mL langsam zu, ohne sich zu vermischen (Abbildung 1B).
- Drehen Sie mit 600 X g für 15 min bei 4 ° C, die restlichen Flagellen im überstand zu entfernen; die Zellen sind in 25 % Saccharose (Abbildung 1C) verteilt. Halten Sie nur die untersten 20 mL (roter Pfeil, Abbildung 1C) durch sorgfältig Absaugen des Überstands mit einer Absauganlage.
- Waschen Sie die restliche 20 mL durch Zugabe von 20 mL kalten 1 X PBS. Drehen Sie die Probe bei 600 X g für 10 min bei 4 ° C. Aufschwemmen Sie das Pellet in 10 mL kalten 1 X PBS (bei 4 ° C). Stellen Sie sicher, dass es keine Klumpen gibt, so dass die folgende Lyse der Zellen auf einmal trifft.
- Übertragen Sie die resuspendierte Pellet auf eine neue 250 mL Flasche. Fügen Sie 100 mL des Puffers Lyse ergänzt mit 5.000 Einheiten der DNase zu den Zellen hinzu. Es ist wichtig, um die Lyse-Puffer zu den Zellen und nicht die andere Weise herum hinzufügen. Inkubieren Sie die Mischung für 1 h bei 4 ° C und mischen Sie es sorgfältig durch Umdrehen der Flasche alle 15 min ohne Bläschen zu bilden.
- Zentrifugieren der lysierten Zellen bei 600 X g für 10 min bei 4 ° C in einem 50 mL konische Rohr, jede Zelle Ablagerungen zu entfernen. Wenn die Lyse korrekt durchgeführt wurde, sollte die Zelle Pellet weiß sein (Abbildung 1D). Sammeln Sie den Überstand mit einer Pipette zu und laden Sie es auf eine 30 mL Rundboden Röhrchen mit einem 60 % Saccharose-Kissen, auf Eis gelegt. Dann drehen Sie das Rohr auf 10.000 X g für 30 min bei 4 ° C.
Hinweis: Mehrere Rundboden Röhrchen (Table of Materials) können, je nach Volumen des Überstands erforderlich sein. - Aspirieren Sie Überstand bis zu 1 mL über die Saccharose-Kissen. Beachten Sie die Gelbe Schnittstelle zwischen 1 mL der restlichen überstand und 2 mL der Saccharose Kissen. Mischen Sie vorsichtig die Saccharose und die restlichen überstand mit einer geschnittenen P1000-Spitze. Nicht Vortex: bei diesem Schritt zu tun; Andernfalls kann die Procentrioles zu diesem Zeitpunkt verloren. Alle Saccharose Kissen zu bündeln und auf Eis aufbewahren.
- Bereiten Sie einen 40 % - 70 % Saccharose Farbverlauf in einem dünnwandigen 38,5 mL Polypropylen Röhrchen durch sanft Zugabe von 3 mL 70 % Saccharose (bei 4 ° C), gefolgt von 3 mL 50 % und schließlich 3 mL 40 % Saccharose. Laden Sie die gepoolten Schnittstellen auf der 40 % - 70 % Saccharose Gradient; tun Sie dies langsam, weil die kalten Saccharose sehr zähflüssig ist. Auszugleichen Sie die Rohre mit dem 10 mM K-PIPES-Puffer (pH 7,2) und Zentrifugieren sie 68.320 x g (z. B.mit SW32Ti Rotor) für 1 h und 15 min bei 4 ° C.
- Sammeln Sie 12 x 500 µL Brüche bei 4 ° C durch ein Loch in den Boden des Röhrchens mit einer 0,8 mm Nadel ohne zu stören die verschiedenen Saccharose-Schichten. Bereiten Sie mit einem Schnitt P200 PIPETTENSPITZE zusätzliche 10 µL-Aliquots aus jeder Fraktion, die für die folgenden Immunfluoreszenz verwendet werden. Snap-Freeze die Brüche in flüssigem Stickstoff.
Hinweis: Isolierte Centriolen können analysiert werden, mittels Elektronenmikroskopie um sicherzustellen, dass die gesamte Ultrastruktur der Centriolen erhalten bleibt.
3. Quantifizierung der isolierten Centriolen auf Deckgläsern: Zentrifugieren und Immunfluoreszenz
Hinweis: Siehe Abbildung 2.
-
Vorbereitung der Rohre und Deckgläsern
- Verwenden Sie 1 Rundboden Glasröhre (15 mL) pro angebrochene centriolar Fraktionen (12 Röhren insgesamt) zu analysieren.
- Setzen Sie einen benutzerdefinierten Deckglas Unterstützung Adapter (Adapter im folgenden genannt) in das Rundboden Röhrchen. Legen Sie ein steriles 12 mm Deckglas in Rundboden Röhrchen.
Hinweis: Hier haben wir 12 mm Deckgläsern verwendet, aber das Protokoll kann für 18 mm Deckgläsern mit 30 ml Rundboden Röhrchen angepasst werden. - Fügen Sie 5 ml vorgekühlte 10 mM K-Rohre (pH 7,2) bei 4 ° C. Stellen Sie sicher, dass das Deckglas nicht schwimmt und unten am Adapter bleibt. Legen Sie die Rohre auf Eis.
-
Zentrifugieren der Centriolen
- Verdünnen Sie jeweils 10 µL Bruch mit 100 µL kalt 10 mM K-Rohre (pH 7,2). Aufschwemmen der Verdünnung gut, bis das vollständige Verschwinden der Saccharose (Abb. 2A). Laden Sie jede verwässerte Fraktion in ein Rundboden Röhrchen.
- Drehen Sie das Rohr auf 10.000 X g für 10 min. (z.B.mit einem JS-13.1 schwingende Eimer Rotor) bei 4 ° C.
- Erholen Sie das Deckglas durch Einfügen einer handgefertigten angeschlossen Gerät in das Loch im gekerbtem Rand des Adapters zu und heben Sie es vorsichtig.
Hinweis: Das handgemachte angeschlossen Gerät kann mit Spritzennadel manuell eingehängt und geformt, auf einen hausgemachten Stick (Abbildungen 2 b-2D) erfolgen. - Fangen Sie nach dem Erreichen der Spitze des Rohres Rundboden den Rand des Adapters mit einem behandschuhten Finger und entfernen Sie das Deckglas mit einer Pinzette zu. Achten Sie darauf, an welcher Seite das Deckglas Centriolen enthält. Fahren Sie mit die Deckgläsern für Immunfluoreszenz zu behandeln.
-
Immunfluoreszenz-Färbung und Bildgebung des isolierten C. Reinhardtii Centriolen
Hinweis: Siehe Abbildung 2.- Bereiten Sie das Material für die Immunfluoreszenz-Färbung wie folgt.
- Bereiten Sie ein Deckglas Färbung Rack in einem Kristall Polystyrol Lab Getriebekasten (60 mm in der Länge von 50 mm in der Breite von 43 mm Höhe). Füllen Sie ihn mit 100 % Methanol und bei-20 ° c Lagern
- Bereiten Sie eine Feuchte Kammer. Hierzu montieren die Feuchte Kammer indem man einem Wasser befeuchteten Tuch neben dem inneren Rand eine quadratische Petrischale (Abb. 2E). Fügen Sie ein Stück des Labors Abdichtung Wrap (siehe Tabelle der Materialien) in die Mitte der Petrischale, auf dem die Antikörper-Mischungen bei der Immunfluoreszenz (Schritte 3.3.2–3.3.3) platziert werden. Decken Sie den Deckel und die Feuchte Kammer mit Alu-Folie vor Licht zu schützen.
- Immunostain isolierte Centriolen wie folgt.
- Befestigen Sie die Deckgläsern mit Centriolen direkt nach der Zentrifugation (Schritt 3.2.4) durch Inkubation sie für 5 Minuten in der Box mit-20 ° C Methanol (Schritt 3.3.1.1) gefüllt.
- Entfernen Sie die Deckgläsern mit Pinzette und Stelle, die Sie in einer transparenten Labor-Box gefüllt mit 50 mL 1 X PBS (siehe Tabelle der Materialien) und waschen Sie sie für 5 min bei Raumtemperatur.
- Pipette 60 µL Primärantikörper Mix [Primärantikörper verdünnt in 1 % Rinderserumalbumin (BSA) und 0,05 % Tween 20 in PBS] auf dem Stück des Labors Abdichtung in die Feuchte Kammer wickeln. Legen Sie vorsichtig Deckgläsern auf die Antikörper-Mischung mit der Centriolen direkt vor den Tropfen. Inkubieren Sie Deckgläsern für 45 min mit primären Antikörper.
Hinweis: Der primäre Antikörper, die verwendet wurden, um repräsentative Ergebnisse zu generieren sind Kaninchen polyklonalen Bld12 (1: 300) und Maus α-Tubulin (DM1A) (1: 300). - Entfernen Sie die Deckgläsern und waschen Sie sie für 5 min in 1 X PBS, wie in Schritt 3.3.2.2 beschrieben. Inkubieren Deckgläsern für 45 min mit Sekundärantikörper in PBS mit 1 % BSA und 0,05 % Tween-20.
Hinweis: Die sekundäre Antikörper, die verwendet wurden, um repräsentative Ergebnisse zu generieren sind Ziege Anti-Maus mit Alexa 488 (1:1 000) gekoppelt und Ziege Anti-Kaninchen mit Alexa 568 (1:1 000) gekoppelt. - Entfernen Sie die Deckgläsern und waschen Sie sie für 5 min in 1 X PBS, wie in Schritt 3.3.2.2 beschrieben.
- Montieren Sie die Deckgläsern auf einen Glasobjektträger durch Zugabe von 3 µL Montage Medium auf der Folie und stellen Sie die Deckgläsern vorsichtig nach oben (Centriolen mit Blick auf das Eindeckmittel). Versiegeln Sie den Deckglas Rand mit Nagellack.
- Bild der isolierten Centriolen auf einem confocal Mikroskop bei 63 X Öl-Ziel mit einer N.A von 1,4 während der Anwendung Dekonvolution27 (siehe Tabelle der Materialien).
Hinweis: Hier wurden die folgenden Einstellungen verwendet: 500 – 545 nm für Alexa 488 und 580 – 635 nm für Alexa 568.
- Bereiten Sie das Material für die Immunfluoreszenz-Färbung wie folgt.
4. Konzentration der Centriolen auf die Mitte der Deckgläsern
Hinweis: Siehe Abbildung 3.
-
Vorbereitung des Materials
- Bereiten Sie eine 15 mL Rundboden Glasrohr auf Eis, eine benutzerdefinierte Deckglas Unterstützung Adapter (so genannte Adapter im folgenden .stl-Datei zur Verfügung gestellt als ergänzende Datei 1), eine benutzerdefinierte Konzentrator (.stl-Datei als ergänzende Datei 2zur Verfügung gestellt) und 10 mM K-Rohre (pH 7.2) bei 4 ° C.
- Poly-D-Lysin (PDL) vorbereiten-Deckgläsern beschichtet. Verdünnen Sie 10 x 1 mg/mL-PDL-Stammlösung mit H2O. Erstens, waschen Sie die Deckgläsern mit 70 % EtOH, entfernen das Ethanol und lassen Sie die Deckgläsern trocknen. Bestreichen Sie die Deckgläsern mit PDL und inkubieren sie 30 min bei Raumtemperatur. Waschen Sie die Deckgläsern 3 X mit Wasser und trocknen lassen.
Hinweis: Mantel befestigt die Deckgläsern mit PDL zur Erhöhung der Zahl der isolierten Centriolen zu den Deckgläsern.
-
Zentrifugation
- Legen Sie ein steriles 12 mm Deckglas auf den eingelassenen, Ende des Konzentrators, halten die PDL Mantel verdeckt. Kappe das Deckglas, indem man den Adapter direkt an der Spitze. Invertieren der Rundboden-Röhre und legen Sie es über den Konzentrator Deckglas und Adapter.
- Vorsichtig drücken Sie das Ensemble mit einer Pinzette zu, bis es den Boden des Rohres Rundboden erreicht und invertieren Sie das Rohr zu. Rundboden-Rohr fügen Sie 10 mM K-PIPES-Puffer (pH 7,2 hinzu) bis es an die Spitze des Konzentrators kommt. Stellen Sie sicher, dass es keine Bläschen in den Zentralzylinder des Konzentrators bleiben.
- Eine Aliquote mit angereicherten Zentriol Bruchteil sanft 100 µL 10 mM K-PIPES-Puffer (pH 7,2) hinzu und mischen Sie die Lautstärke.
- Entfernen Sie 100 µL 10 mM K-PIPES-Puffer (pH 7,2) aus dem hohlen Zentrum des Konzentrators und fügen Sie 100 µL der angereicherten centriolar Bruch in 10 mM K-PIPES-Puffer (pH 7,2 hinzu) in die hohle Mitte des Konzentrators, darauf achten, dass die Inhalte in der hohle Mitte bleiben.
- Zentrifugieren Sie bei 10.000 X g für 10 min (z.B.mit einem JS-13.1 schwingende Eimer Rotor) in eine vorgekühlte Zentrifuge bei 4 ° c
- Entfernen Sie den Konzentrator mit einer Pinzette.
- Erholen Sie das Deckglas durch Einfügen einer handgefertigten angeschlossen Gerät in das Loch im gekerbtem Rand des Adapters zu und heben Sie es vorsichtig. Fangen Sie nach dem Erreichen der Spitze des Rohres Rundboden den Rand des Adapters mit einem behandschuhten Finger und entfernen Sie das Deckglas mit einer Pinzette zu. Achten Sie darauf, an welcher Seite das Deckglas Centriolen enthalten. Fahren Sie mit die Deckgläsern für Immunfluoreszenz zu behandeln.
Hinweis: Das handgemachte angeschlossen Gerät kann mit einer Spritzennadel manuell eingehängt und geformt, auf einen hausgemachten Stick (Abbildungen 2 b-D) erfolgen. - Durchführen Sie Fixierung und Immunfluoreszenz Färbung des konzentrierten Centriolen wie im Schritt 3.3.2–3.3.4.
(5) Single-Teilchen im Durchschnitt
Hinweis: Siehe Abbildung 4.
- Imaging für Single-Teilchen im Durchschnitt
- Montieren Sie das Deckglas auf einer Folie mit einem regelmäßigen Anti-Fading-Montage-Medium. Strukturierte Beleuchtung Mikroskopie (2D SIM) Bildgebung mit einem CFI Apochromat TIRF Ziel durchführen (100 X, NA 1,49, WD 0,12 mm) und eine Rückseite beleuchtet EM CCD-Kamera.
Hinweis: Die Erfassungszeit wurde auf 100 ms bei einer Kamera Auslesen von 3 MHz festgelegt. A 2.5 X-Objektiv wurde verwendet für die SIM-Bildgebung.
Hinweis: Das hier vorgestellte Dataset erwarb am 3-d-SIM-Mikroskop (siehe Tabelle der Materialien). - Bild der Centriolen durch den Erwerb eines großen Stapels von Bildern aus der gesamten Zentriol signalisieren durch die Einrichtung der oberen und unteren Position im Z-Stapel oben und unten das Zentriol Signal bzw.. Fahren Sie mit der Stack-Projekt und führen Sie Einzel-Teilchen im Durchschnitt nach Schritte 5.2 und 5.3.
- Montieren Sie das Deckglas auf einer Folie mit einem regelmäßigen Anti-Fading-Montage-Medium. Strukturierte Beleuchtung Mikroskopie (2D SIM) Bildgebung mit einem CFI Apochromat TIRF Ziel durchführen (100 X, NA 1,49, WD 0,12 mm) und eine Rückseite beleuchtet EM CCD-Kamera.
- Projektion eines Stapels
- Öffnen Sie den Bildstapel mit ImageJ. Klicken Sie auf "Bild → Stapel → Z Projekt". Geben Sie die Projektion alsMax Intensität'.
- Invertieren Sie das Bild durch Anklicken "Bearbeiten → invertieren'. Speichern Sie die Projektion, die generiert wird (TIF-Format).
- Einzelne Partikel Ausrichtung mit Scipion
- Erstellen Sie ein neues Projekt in Scipion durch drücken den roten "Schaltfläche"Create Project" " am oberen Rand der Seite. Doppelklicken Sie im linken Bereich auf "Importe → Import mikrographen'.
- Füllen Sie die "Verzeichnis' und 'Muster" Felder nach den Daten-Verzeichnis und Namen. Halten Sie die Standardparameter. Klicken Sie auf "Ausführen".
- Doppelklicken Sie auf "Partikel → Kommissionierung → xmipp3 - Handbuch Kommissionierung (Schritt 1)". Klicken Sie auf das Lupensymbol in der Nähe von Feld "Input mikrographen" und wählen Sie die importierten mikrographen Schritt 5.3.1. Klicken Sie auf "Ausführen".
- Wählen Sie in dem neu geöffneten Fenster Partikel aus den verschiedenen mikrographen indem Sie darauf klicken. Wenn jeder Schliffbild beendet wurde, klicken Sie auf die rote Taste "+ koordiniert".
- Doppelklicken Sie auf "Partikel → Extrakt → xmipp3 - Extrakt Partikel". Klicken Sie auf das Lupensymbol in der Nähe von Feld "Eingabe Koordinaten" und wählen Sie die ausgewählten Koordinaten aus Schritt 5.3.4. Füllen Sie die "Box Partikelgröße (px)" entsprechend den Abmessungen der Partikel. Legen Sie im Register "Preprocess" Entstaubung: keine (es kann Artefakte erzeugen); Kontrast zu invertieren: keine (schwarze Partikel, weißer Hintergrund); Phase zu kippen: Nein (in Verbindung mit CTF Korrektur); Normalisieren: Ja.
- Klicken Sie auf 'Ausführen'. Wenn die Arbeit getan ist, überprüfen Sie die extrahierten Teilchen Kontrollkästchen der Arbeit (Grenzen dicker geworden) und klicken Sie auf "Analyze Ergebnisse (unten links im Hauptmenü).
- Doppelklicken Sie auf "2D → → xmipp3 ausrichten: richtet mit cl2d". Klicken Sie auf das Lupensymbol in der Nähe von Feld "Input Partikel" und wählen Sie die extrahierten Teilchen aus Schritt 5.3.5. Verwenden Sie kein Referenzbild. Klicken Sie auf "Ausführen".
- Doppelklicken Sie auf ' 2D → → ausrichten mehr → xmipp3 - Ausrichtung gelten 2d'. Klicken Sie auf das Lupensymbol in der Nähe von Feld "Input Partikel" und wählen Sie die ausgerichtete Partikel aus Schritt 5.3.7.
- Klicken Sie auf 'Ausführen' , wie in Schritt 5.3.6. Die Ergebnisse können durch Klicken auf "Analyze Ergebnisse" nach der Auswahl des Feldes Arbeit überprüft werden.
- Doppelklicken Sie auf "Partikel → Maske → xmipp3 - 2d Maske anwenden".
- Klicken Sie auf das Lupensymbol in der Nähe von Feld "Input Partikel" und wählen Sie die ausgerichtete Partikel aus Schritt 5.3.9. Die "Maske Quelle" wird auf "Geometrie"festgelegt. Legen Sie die Parameter der Maske als Maskentyp: Rundschreiben; RADIUS (px): auf der Suche nach einem Teilchen (ein Zentriol) ohne alles um ihn herum, klicken Sie auf die "Zauberstab" -Symbol auf der linken Seite die öffnet sich ein Fenster um den perfekten Wert; finden Schicht-Center: Nein (wenn die Teilchen perfekt zentriert) oder Ja (wenn das Teilchen verschoben wird); X-Center Versatz: abhängig von der Position des Partikels; Y-Center Versatz: abhängig von der Position des Partikels. Klicken Sie auf "Ausführen".
- Verwenden Sie die Schaltfläche "Ergebnisse analysieren" um zu überprüfen, ob die Maske korrekt angewendet wird. Wenn dies nicht der Fall ist, auf dem Job "Übernehmen 2d Maske" mit der rechten Maustaste und wählen Sie "Bearbeiten". Ändern Sie die Parameter von der Maske (Größe und/oder Schichten) und führen Sie es erneut mit 'Ausführen' button.
- Doppelklicken Sie auf "2D → klassifizieren → xmipp3 - cl2d".
- Klicken Sie auf das Lupensymbol in der Nähe von Feld "Input Partikel" und wählen Sie die maskierten Partikel aus Schritt 5.3.11. Die Anzahl der Klassen sollen etwa 50 Partikel pro Klasse zu erhalten. Klicken Sie auf "Ausführen".
- Überprüfen Sie die Ergebnisse durch Klicken auf die Schaltfläche "Ergebnisse analysieren" . Klicken Sie im sich öffnenden Fenster auf das Symbol "Auge" direkt neben "Was zu zeigen".
- Das neue Fenster erlaubt die Überprüfung von den generierten Klassen durch Auswahl von "Classes2D" im Menü "Block" . Überprüfen Sie den Inhalt der einzelnen Klassen durch Auswahl von "Class00N_Particles" im gleichen Menü. Überprüfen Sie jede Klasse zu identifizieren, welche nur schlecht Partikel enthalten. Gehen Sie zurück zur Ansicht "Classes2D" und wählen Sie die Klassen mit guter Partikel durch Anklicken jeweils. Es ist möglich, mehrere Klassen durch halten der "Strg" -Taste gedrückt bei der Auswahl wählen.
- Wenn alle Klassen mit guter Partikel ausgewählt haben, klicken Sie auf "+ Partikel" eine Teilmenge mit diesen Partikeln zu erstellen.
- Wählen Sie im gleichen Fenster ein paar Sätze von Klassen, die unterschiedliche Ausrichtungen der Partikel zu vertreten. Erstellen Sie eine neue Untergruppe durch Klicken auf "+ Durchschnitte".
- Gehen Sie für jede Ausrichtung/durchschnittlich ausgewählt wie folgt vor.
- Doppelklicken Sie auf "2D → → xmipp3 ausrichten: richtet mit cl2d".
- Klicken Sie auf das Lupensymbol in der Nähe von Feld "Input Partikel" und wählen Sie die gute Partikel aus Schritt 5.3.17. Legen Sie "Use ein Referenzbild" auf "Ja". Klicken Sie auf das Lupensymbol in der Nähe von Feld "Referenzbild" und klicken Sie auf weißer Pfeil links auf den 'Create Teilmenge' Job Schritt 5.3.18 und wählen Sie das Bild, um es als Referenz zu verwenden. Doppelklicken Sie auf ein Objekt in einem separaten Fenster öffnen und überprüfen, welches Objekt ist. Klicken Sie auf "Ausführen".
- Doppelklicken Sie auf ' 2D → → ausrichten mehr → xmipp3 - Ausrichtung gelten 2d'. Klicken Sie auf das Lupensymbol in der Nähe von Feld "Input Partikel" und wählen Sie die ausgerichtete Partikel aus Schritt 5.3.19.2.
- Klicken Sie auf "Ausführen".
- Überprüfen Sie das Ergebnis der Ausrichtung ("Ergebnisse analysieren); Es zeigt die ausgerichtete Partikel und der Durchschnitt dieser Teilchen.
- Wenn der Durchschnitt gut ist und die Partikel werden auf die gleiche Weise orientiert, speichern im Durchschnitt durch Anklicken "Advanced → ImageJ" und speichern Sie das Bild mit ImageJ.
- Wenn der Durchschnitt verbessert werden kann, wählen Sie die gut ausgerichtete Bilder und erstellen Sie eine neue Untergruppe mit der Taste "+ Partikel" . Wiederholen Sie Schritte 5.3.19.1–5.3.19.4, bis die Teilmenge (alle schlechten Partikel entfernt) gereinigt wird. Jedes Mal, wenn die Ausrichtung ist durchgeführt (Schritt 5.3.19.2), der Verweis auf dem letzten generierten Durchschnitt (von Iteration zu Iteration, die durchschnittliche Qualität erhöht) festgelegt ist.
Ergebnisse
C. Reinhardtii Zentriol Isolation:
Centriolen, cw15- isolieren C. Reinhardtii Zellen waren für mehrere Tage unter Licht in Flüssigkultur angebaut und anschließend pelleted durch Zentrifugation bei 600 X g für 10 min. Pelleted Zellen 1 X mit PBS gewaschen wurden und resuspendierte in einem deflagellation Puffer vor der deflagellation durch die Durchführung eines pH-Schocks mit 0,5 M Essigsäure zu einem endgültigen pH-Wert von 4,5-4,7 für 2 min(Abbildung 1). Ein Zusatz von 1 N KOH wurde verwendet, um pH 7.0 wiederherstellen. Um freistehende Flagellen vom Zellkörper zu trennen, wurden deflagellated Zellen zuerst zentrifugiert, um den Großteil der Flagellen zu entfernen. Das Pellet wurde dann 2 X mit PBS gewaschen und Nukleinsäuretablette in 30 mL PBS vor dem laden es langsam auf einem 25 %-Saccharose-Kissen (Abbildung 1B). Das Rohr war 600 X g für 15 min bei 4 ° C, die meisten der freistehenden Flagellen entfernen gesponnen. Nach Zentrifugation, waren die Zellkörper in die Saccharose-Kissen (Abbildung 1C) zu verbreiten und erholt durch Entfernen der etwa 30 mL des Überstandes (bis der rote Pfeil in Abbildung 1C). Die daraus resultierende 20 mL gewaschenen Zellen wurden dann Nukleinsäuretablette in 20 mL kaltem PBS und 600 X g für 10 min zentrifugiert. Als nächstes der Überstand wurde verworfen, und die Zellen wurden komplett in 10 mL PBS Nukleinsäuretablette. Die Zellen wurden zu einem 250-mL-Flasche und der Lyse-Puffer wurde hinzugefügt, um die resuspendierte Zellen auf einmal. DNase wurde hinzugefügt, um die Lyse und für 1 h bei 4 ° c inkubiert Nachdem ein Zentrifugationsschritt, Zelle Ablagerungen zu entfernen (siehe die weiße Kugel in Abbildung 1D), der Überstand wurde gesammelt und sorgfältig verladen eine 2 mL 60 % Saccharose Kissen vor der Zentrifugation bei 10.000 X g für 30 min bei 4 ° C. Beachten Sie, dass für 100 mL Lyse, 8 Tuben von 15 mL wurden verwendet, um diesen Schritt entspricht 12,5 mL Lyse Puffer geladen, auf 2 mL Saccharose pro Röhre ausführen. Nach Zentrifugation, war die meisten des Überstands entfernt bis zu 1 mL über das Kissen. 1 mL der restlichen überstand wurde dann mit 2 mL Kissen gesammelt und dann gemischt und gebündelt zu einem Endvolumen von 24 mL. Der Pool wurde verladen auf einem 40 %, 50 %, 70 % Saccharose gradient und gesponnene 68.320 x g für 1 h und 15 min bei 4 ° c Isolierte Centriolen wurden schließlich durch ein Loch im Unterteil der Zentrifugation Röhre mit einer Nadel und die Tropfen wurden in 12 x 500 µL Fraktionen gesammelt. Aufgrund der hohen Dichte von den verschiedenen Saccharose gebildet die Tropfen sehr langsam am Anfang (70 % Saccharose) und dann mehr schnell (40 % Saccharose).
Immunfluoreszenz von isolierten Centriolen
Um die Qualität des Verfahrens isoliert zu beurteilen, wurde 10 µL der einzelnen gesammelten gradient Fraktionen dann auf einem deckgläschen mit einem Deckglas-Unterstützung-Adapter (Abbildung 2A-2D) zentrifugiert. Wichtig ist, um das Deckglas sicher zu entfernen, ein benutzerdefiniertes Gerät angeschlossen wurde (Abbildung 2B). Als nächstes wurden die Deckgläsern durch Immunfluoreszenz analysiert. Antikörper gegen CrSAS-6(Bld12p) wurden in dieser Studie verwendet, um das Vorhandensein der Wagenrad Struktur und α-Tubulin (DMA1) an die centriolar Wand markieren. Durch zählen der Centriolen, die für CrSAS-6 und α-Tubulin pro Gesichtsfeld positiv waren und dann berechnen die Gesamtzahl der Centriolen pro Brüche, war es möglich, festzustellen, welche Fraktionen für isolierte Centriolen ( angereichert waren Abbildung 2F-2 H). Interessanterweise wurden 6 Fraktionen Centriolen (Abbildung 2F und 2 G, Brüche #1 – 6), bereichert mit einem Spitzenwert für Bruchteil #3, während die letzten Fraktionen nicht, darauf hinweist waren, dass die Reinigung gearbeitet. Beachten Sie, dass in diesem besonderen Experiment, 95 % der gesamten Centriolen für CrSAS-6 und α-Tubulin in Bruchteil #3 positiv waren. Dies bedeutet, dass am stärksten isolierte Centriolen ihrer Wagenräder beibehalten. Wenn keine Bereicherung der Centriolen in den ersten Sekundenbruchteilen beobachtet wird, die Isolierung Verfahren funktionierte nicht und sollte wiederholt werden. Beachten Sie, dass einige flagellar Stücke meist in den Fraktionen frei von Centriolen beobachtet werden können.
Als nächstes sollte um die Gesamtzahl der Centriolen pro Mikroliter zu berechnen, die Anzahl der Zentriol pro Gesichtsfeld durch das Verhältnis wie in Abbildung 2Hpräsentiert multipliziert werden. Die sich daraus ergebende Zahl sollte dann durch das Volumen des Anteils für die Immunfluoreszenz verwendet, um die Anzahl der Centriolen pro Mikroliter erhalten unterteilt werden. In diesem Verfahren besondere Isolierung enthalten die meisten angereicherte Fraktion etwa 37 Centriolen für eine Fläche von 0,00846 mm2 (mit einem Sichtfeld von 92 x 92 µm2). Die Oberfläche des Rohrs war 7,5 mm Radius mit einer Gesamtfläche von 176 mm2. Das entsprechende Verhältnis war dann 176/0.00846 = 20,803.8, also insgesamt 769.740 Centriolen (37 x 20, 803.8) in 10 µL. Daher war die Anzahl der Centriolen in 1 µL 76.974.
Konzentration der isolierten Centriolen auf Deckgläsern:
Erhöhung der Zahl der Centriolen pro Feld erhöht die Chance auf eindeutige Zentriol Orientierung erkennen, als auch erhöhen die Wahrscheinlichkeit der Erkennung von ähnlichen Orientierungen, die für die weitere Teilchen durchschnittlich Vorgehensweise verwendet werden können. Centriolen aus konzentrierteren Fraktionen noch spärlich auf dem Deckglas sind, entwickelten wir eine Zentrifugation Zubehör Centriolen in der Mitte das Deckglas(Abbildung 3)benannt einen Konzentrator zu konzentrieren. Beachten Sie, dass eine .stl-Datei mit den genauen Maßnahmen für den 3D-Druck mit dieser Handschrift versehen ist.
Erstens ein Deckglas 12 mm wurde auf der Konzentrator (Abbildung 3B) montiert. Der Adapter wurde auf das Deckglas platziert, Rundboden Röhrchen wurde invertiert und über die montierte Adapter und Konzentrator gelegt. Rundboden-Rohr wurde dann sanft invertiert, so dass die Belastung der Probe (Protokoll Schritt 4.2). Die Centriolen wurden dann 10.000 x g für 10 min bei 4 ° c zentrifugiert. Danach wurden die Centriolen Immunfluoreszenz unterzogen und gebeizt für CrSAS-6/Bld12p und α-Tubulin (Abbildung 3-3E). Wichtig ist, wurden ohne Konzentrator, etwa 30 Centriolen pro Gesichtsfeld (Abb. 3C), gesehen, während 183 Centriolen pro Gesichtsfeld gesehen wurden, wenn der Konzentrator verwendet (Abbildung 3D-3F) war. Beachten Sie, dass die Centriolen nur eine Scheibe aus 4 mm im Durchmesser in der Mitte das Deckglas abgedeckt. Dieses Ergebnis zeigt, dass die Konzentration Schritt arbeitet und eine 6-fold Bereicherung der Centriolen in einer definierten Region von Deckgläsern erlaubt, damit Lockerung ihrer Entdeckung und imaging.
Fluoreszierende Single-Teilchen im Durchschnitt von isolierten C. Reinhardtii Centriolen:
Hier mittels SIM-Mikroskopie, die eine Auflösung von etwa 120 erreichen kann nm, Centriolen gebeizt für Monoglutamylated Tubulin (GT335, Abbildung 4), eine Tubulin-Änderung in centriolar Mikrotubuli vorhanden waren abgebildeten24. C. Reinhardtii Centriolen sind etwa 500 nm lang, immer paarweise, und oft fand mit verbundenen, neu Probasal Körper (bezeichnet als Procentrioles im folgenden) dupliziert und gekerbter Mikrotubuli-assoziierten Fasern19. Daher war diese Endmontage ca. 1 µm groß. Aus diesem Grund und um die Centriolen in ihrer Gesamtheit Bild empfehlen wir erwerben einen Z-Stack auf die isolierte Centriolen.
Hier wurde nach der Übernahme, eine endgültige Bild generiert, durch Ausführen einer Beihilfehöchstintensität Projektion mit ImageJ28 (Bild/Stapel/Z Projekt/Max Intensität Projektion, Abbildung 4B). Von solchen Bildern war eine Single-Partikelanalyse mit Cryo-Elektron Mikroskopie Software um Klassen der Centriolen mit ähnlicher Ausrichtung zu machen und dann durchschnittlich durchgeführt wurde. Hierzu wurde die Bildfarbe zunächst invertiert, um die Objekte (Abb. 4C) besser zu visualisieren. Centriolen abgeholt wurden manuell in einer Box zentriert über jedes Teilchen mit der frei verfügbaren Scipion Software29 , die mehrere Elektronen-Mikroskopie-Software-Programme wie Xmipp3 integriert (Abbildung 4D). Beachten Sie, dass die Größe des Kastens muss vom Benutzer definiert werden. Hier, Boxen von 50 x 50 Pixel für eine Pixelgröße von 31.84 nm verwendet wurden. Als nächstes wurden alle Partikel (Abbildung 4E) extrahiert und mit Xmipp3 ausgerichtet (Abbildung 4F). Als nächstes wurde eine kreisförmige Maske von 12 Pixel Radius angewandt, um jedes Zentriol aus dem centriolar-Paar (Abb. 4G) zu isolieren. Die Partikel wurden dann klassifiziert, mit Xmipp3, um mehrere Durchschnitte zu erzeugen. Nur wurden homogene Klasse Durchschnittswerte gehalten, was bedeutet, dass Partikel, die von der Klasse Durchschnitt abweichen manuell ausgeschlossen wurden. Dieser Schritt wurde wiederholt, um eine nahezu perfekte Durchschnitt für jede gewählte Ausrichtung zu generieren. Nach fünf Wiederholungen wurden zwei Klassen von Durchschnittswerten generiert: eine Draufsicht aus 63 Objekte (Abbildung 4H) und eine Seite Anzeigen von 98 Partikeln (Abbildung 4ich) des Monoglutamylated Centriolen. Die Abmessungen des Objekts wurden durch Messen des Abstands zwischen den Gipfeln der Intensität Plot Profil entlang des Monoglutamylation-Signals bestimmt.
Wichtig ist, ist die Länge der Seitenansicht Klassendurchschnitt 260 nm mit einem Durchmesser von 250 nm, vergleichbar mit den gemessenen Monoglutamylated-Tubulin-Signal, das gezeigt wurde, zu einer Region von 286 ± 33 nm Länge innerhalb des Kerns der Zentriol17zu lokalisieren.
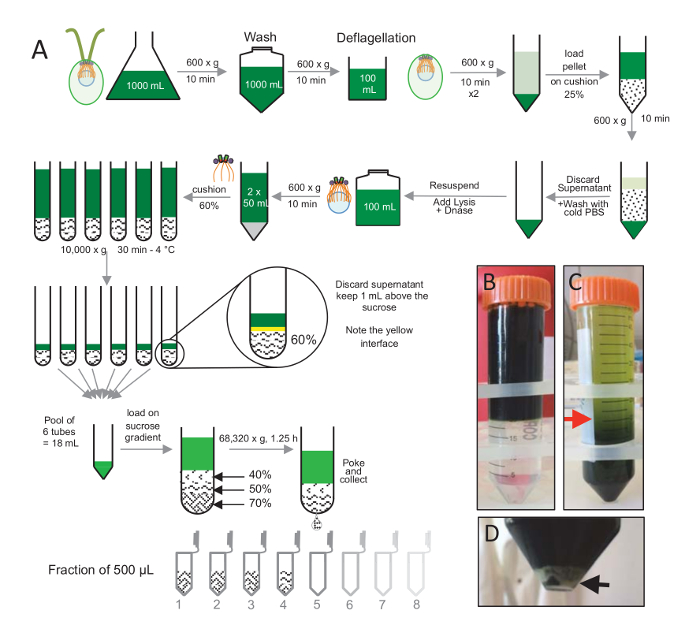
Abbildung 1 : Reinigung von C. Reinhardtii Centriolen. (A) Dies ist eine schematische Darstellung der einzelnen Schritte führen zur Isolation des C. Reinhardtii Centriolen. Freuen Sie sich auf repräsentative Schritte des Protokolls (B) vor und (C) nach Zentrifugation auf 25 % Saccharose Farbverlauf. Der rote Pfeil im Feld C gibt das minimale Volumen nach Zentrifugation zu halten. (D) dieses Panel zeigt die weiße Kugel lysierten Zellen nach Zentrifugation. Der schwarze Pfeil zeigt das Pellet. Bitte klicken Sie hier für eine größere Version dieser Figur.
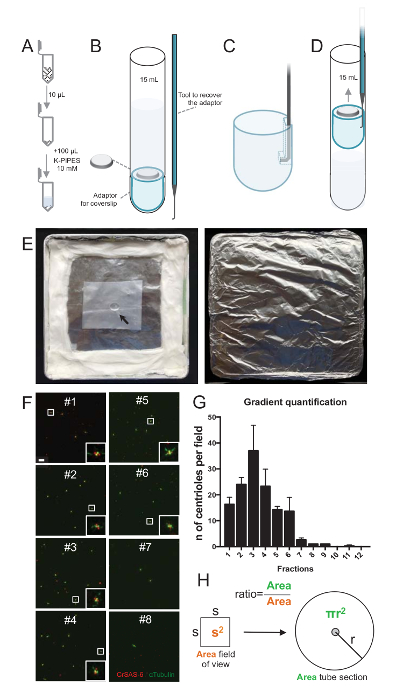
Abbildung 2 : Zentrifugation Setup durchführen Immunfluoreszenz auf isolierte Centriolen. (A) 10 µL der einzelnen gesammelten Fraktionen wird zuerst in 100 µL 10 mM K-Rohre (pH 7,2) verdünnt. (B) Dies ist eine schematische Darstellung der Zentrifugation Geräte, umfasst eine 15 mL Rundboden-Schlauch, ein 12 mm-Deckglas, einen Adapter für das Deckglas und eine maßgeschneiderte angeschlossen Gerät das Deckglas nach Zentrifugation zu erholen. Tafeln, C und D zeigen Zeichnungen erklären, wie man das Deckglas nach Zentrifugation zu erholen. (C) Ort das hakenförmige Werkzeug in das Loch im gekerbtem Rand des Adapters und (D) ziehen. (E) sind Bilder der feuchten Kammer Immunfluoreszenz Bildgebung durchführen musste. Der Pfeil zeigt die 12 mm-Deckglas. (F) sind repräsentative konfokale Bilder bei 63 X (zoom 2) gradient Brüche #1-8, gesammelt während der Reinigungsprozedur und gebeizt für CrSAS-6 (rot) und α-Tubulin (grün). Die Intarsien entsprechen der Region durch ein weißes Feld in der unteren Vergrößerung Ansichten angezeigt. Maßstabsleiste = 10 µm. (G) Dies ist ein Diagramm der Centriolen positiv für CrSAS-6 und α-Tubulin pro Gesichtsfeld in jede Fraktion darstellt. Beachten Sie die Bereicherung in Sekundenbruchteilen #1-6. Die durchschnittliche Anzahl der Centriolen pro Feld in jedem Bruchteil: #1 = 16,3 ± 4,7, #2 = 24,0 ± 4,6, #3 = 37,0 ± 17,0, #4 = 23,3 ± 11,4, #5 = 14,3 ± 2.1, #6 = 13,7 ± 9,3, #7 = 2,7 ± 1,2, #8 = 1,0 ± 0.0, #9 = 1,0 ± 0.0, #10 = 0.0 ± 0.0, #11 = 0,3 ± 0,6, #12 = 0.0 ± 0,0. Für jede Fraktion wurden 3 ZUFALLSFELDER abgebildet. Beachten Sie, dass bei den meisten angereicherten Bruch #3 zählen, wir fanden, dass 95 % der Centriolen für CrSAS-6 und α-Tubulin positiv (n = 205 Centriolen). (H) A Verhältnis der Anzahl der Centriolen im gemessenen Bereich von den mikrographen wird verwendet, um die Gesamtzahl der Centriolen in der Gegend des Rohres zu berechnen. Die Zentriol Anzahl pro Mikroliter kann aus 10 µL ursprünglich zentrifugiert Brüche berechnet werden. Bitte klicken Sie hier für eine größere Version dieser Figur.
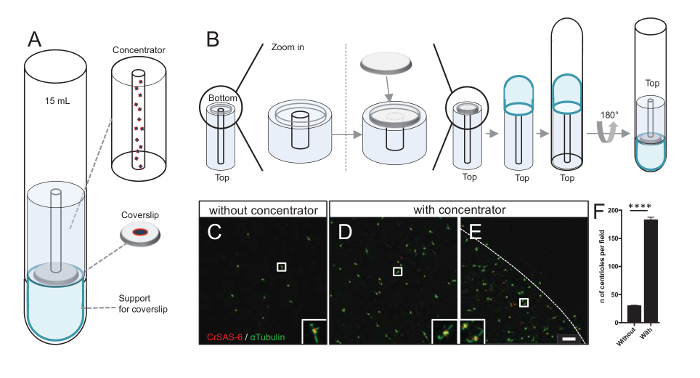
Abbildung 3 : Isolierte Centriolen sind zentrifugiert mit Konzentratoren. (A) dieses Panel zeigt die Zentrifugation Geräte Centriolen auf Deckgläsern vor Immunfluoreszenz, einschließlich eine 15 mL Rundboden-Tube, einem Konzentrator, ein 12 mm-Deckglas und eine Unterstützung Adapter Apparat konzentrieren musste. (B) dieses Panel zeigt die Schritte der Zentrifugation Gerät zusammenzubauen. Seitenteile C-E zeigen konfokale Bilder der Centriolen (D-E), CrSAS-6 (rot) oder α-Tubulin (grün) (C) ohne mit der Konzentrator befleckt. Die Intarsien entsprechen der Region durch ein weißes Feld in der unteren Vergrößerung Ansichten angezeigt. Der Maßstab ist 10 µm. Beachten Sie, dass die Centriolen sind in der Mitte das Deckglas (die gestrichelte Linie stellt die Grenze des Bereichs konzentriert) angereichert. (F) dieses Diagramm stellt die Anzahl der Centriolen pro Gesichtsfeld ohne und mit der Konzentrator. Fünf zufällige Gesichtsfelder wurden analysiert. Die durchschnittliche Anzahl der Centriolen ist, ohne Konzentrator, 29,8 ± 2,9, und mit Konzentrator, 182,6 ± 11,5, P < 0,0001. Die statistische Signifikanz wurde durch eine ungepaarte tbeurteilt-testen. Bitte klicken Sie hier für eine größere Version dieser Figur.
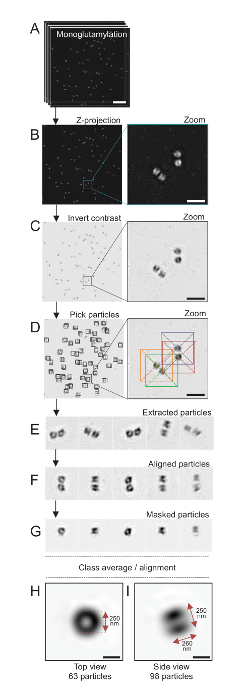
Abbildung 4 : Single-Teilchen im Durchschnitt auf isolierten C. Reinhardtii Centriolen. (A) dieses Panel zeigt Z Bilder Stapeln der Centriolen mit GT335 befleckt und erwarb mit einem SIM-Mikroskop. (B) zeigen diese Platten eine maximale Intensität Z Projektion der gestapelten Bilder. Maßstabsleiste = 1 µm. (C) Diese Tafeln zeigen ein repräsentatives Bild mit einer umgekehrten Kontrast. Der Inset repräsentiert eine Zoom-in, die Centriolen besser zu visualisieren. (D) Diese Tafeln zeigen Teilchen Kommissionierung. Der Inset zeigt an, wie die Partikel abgeholt wurden. (E) sind Beispiele für 5 extrahiert Partikel. (F) dieses Panel zeigt die Partikel nach dem ausrichten. (G) dieses Panel zeigt die Partikel nach dem Auftragen einer Maske. Panels H und ich zeigen zwei Klasse Durchschnitte: (H) ein Top View (63 Partikel) und (ich) eine Seitenansicht (98 Partikel). Die doppelte Pfeile zeigen die Dimensionen des GT335 Signals. Maßstabsleiste = 250 nm. Bitte klicken Sie hier für eine größere Version dieser Figur.
Ergänzende Datei 1. Bitte klicken Sie hier, um diese Datei herunterladen.
Ergänzende Datei 2. Bitte klicken Sie hier, um diese Datei herunterladen.
Diskussion
Eine der Herausforderungen in der Biologie ist die präzise Lokalisierung der Proteine im architektonischen Kontext zu entschlüsseln. Die Zentriol ist eine ideale Struktur, um diese Methoden anwenden, da seine Architektur mit Cryo-Elektron Tomographie, offenbart interessante Ultrastrukturforschung Features entlang seiner Länge untersucht worden ist. Jedoch wegen seiner Größe in der Nähe der Auflösungsgrenze in der optischen Mikroskopie ist es schwierig, ein Protein durch Immunfluoreszenz, eine strukturelle Teilbereich des Zentriol, die mit konventionellen Mikroskopen30genau zu lokalisieren.
Die Auflösung in der optischen Mikroskopie ist begrenzt durch die Beugung des Lichts, das gibt, grob gesagt, laterale Auflösungen von bis 200 nm in optischen Mikroskopie24. Diese Grenze wurde jedoch durch-geführt von einem der großen Durchbrüche der letzten 20 Jahre in der optischen Mikroskopie: die Erfindung der Höchstauflösung Methoden. Diese Ansätze können die Beugung Grenzen mit unterschiedlichen Auflösungen Bild: 120 nm für SIM, etwa 50 nm für stimulierte Emission Depletion (STED) und 20-40 nm für Einzelmolekül-Lokalisierung Mikroskopie (SMLM)24. Mit diesen neuen Entwicklungen der Höchstauflösung Mikroskopie sind die strukturellen Teilregionen der Zentriol erreichbar. In der Praxis ist es jedoch nach wie vor schwierig festzustellen genau die Lokalisation eines Proteins, ein tragendes Element für den Hauptgrund, dass Reife Centriolen sind in nur 2 Kopien pro Zelle vorhanden und zufällige Ausrichtungen haben, wodurch die Interpretation der Lokalisierung schwierig. Aus diesem Grund wurde ein Protokoll eingerichtet, die Forscher mit super-Resolution Imaging eine große Anzahl von Centriolen, erhöht die Chance, eindeutige Orientierungen zu beobachten zu können. Wichtig ist, da diese Methode stützt sich auf die Verwendung von isolierten Centriolen, bieten wir eine Methode, um intakte C. Reinhardtii reinigen Centriolen, die Reife Centriolen und Procentrioles enthalten.
Zu guter Letzt kann dank der Reihe von Zentriol Orientierungen, die mit diesem Protokoll abgebildet werden können, Single-Partikelanalyse angewendet werden mittels Elektronenmikroskopie Software. Dies führt zu der Generation von durchschnittlichen Klassen der Centriolen in einer bestimmten Ausrichtung. Wichtig ist, lässt diese entstehenden 2-D-Bilder dann die Lokalisierung eines spezifischen Proteins entlang der Zentriol zu beurteilen. In der Tat, diese Methode kann auf zweifarbige super-Resolution-Bilder angewendet werden, und eine Farbe kann zur offenbaren ein bestimmtes centriolar Protein Zentriol Skelett (z.B. Tubulin), während die andere Farbe zugeordnet werden kann. Durch Subtraktion der Mittelwerte bezogen mit ein oder zwei Farben, wird es einfacher, ein Protein entlang der Zentriol (proximalen, zentral oder distal) zu registrieren. Beachten Sie, dass die beiden Kanäle exakt ausgerichtet werden sollte, um irreführenden Auslegungen zu verhindern. Darüber hinaus hilft Durchschnitte von Draufsichten entschlüsseln, wenn ein Protein im Inneren der centriolar Lumen, entlang der Mikrotubuli-Wand oder außerhalb des Zentriol lokalisiert.
Diese Methode hat den Vorteil, zu prüfen, die Lokalisierung von bestimmten Proteinen, die sonst aufgrund heterogener Markierung Lokalisierung schwierig sein könnte. Beachten Sie, dass andere Methoden, um Proteine innerhalb der Centriolen Karte in korrelativen 3-d-SIM/SMLM mit, zum Beispiel Beurteilung der spezifischen Ausrichtung der Centriolen beschrieben wurden durch die Bestimmung der elliptischen Profils eines Markers bilden einen Torus um die Zentriol durch SIM-Bildgebung. Bei Verwendung dieses Parameters, ist es möglich, mit einer Genauigkeit von 4 – 5 nm30Protein zu lokalisieren. Beachten Sie auch, dass die beschriebene Methode hier isolierende Centriolen mit intakten Procentrioles, ein Zeichen verwendet, die die Zentriol Architektur am wahrscheinlichsten weitgehend erhalten geblieben ist. Jedoch können wir nicht ausschließen, die einige architektonischen Besonderheiten während der Reinigung, wie z. B. der Zentriol Durchmesser Variation mit der Konzentration von zweiwertigen kationen wie mit der Isolierung des menschlichen Centrosome5verstärkt gestört sind.
Einer der kritischen Schritte des hier vorgestellten Protokolls ist hinreichend konzentrierte isolierende Centriolen in verschiedenen Ausrichtungen Fluo-SPA zugänglich zu erhalten. Zu diesem Zweck zunächst sicherstellen Sie die Reinheit und die Effizienz des Verfahrens Zentriol isoliert. Eine niedrige Konzentration von isolierten Centriolen verhindert richtige Bildgebung und weitere Bildverarbeitung. Zu diesem Zweck bieten wir eine Methode, um die Anzahl der Centriolen pro Gesichtsfeld zu bereichern. Abhängig von der Anzahl der Centriolen in der Fraktion verwendet sollte die Lautstärke in der Konzentrator geladen mit einem maximalen Volumen von 250 µL angepasst werden.
Wichtig ist, wurde diese Methode für eine Zellwand abzüglich cw15 C. Reinhardtii Zellen entwickelt. In dieser Sorte ermöglicht die Zerbrechlichkeit der Zellwand eine ordnungsgemäße Lyse der Zellen und damit die Befreiung seines Inhalts. Dieses Protokoll ist nicht effizient für Wildtyp C. Reinhardtii Zellen, da die Zellwand verhindert, eine ordnungsgemäße Lyse dass. Alternativstrategien wie Beschallung oder einer Vorinkubation der Zellen mit Autolysin, ein Enzym, das die Zellwand31, beeinträchtigen kann müssten geschaffen werden, die Zellwand vor dem Aufbringen der Isolierung Protokoll hier vorgestellten zu ändern.
Diese Einrichtung kann mit verschiedenen Arten von Mikroskopen, von konventionellen konfokale Mikroskope bis hin zu hohen Durchsatz gewidmet Höchstauflösung Mikroskope verwendet werden. Beachten Sie, dass dabei SMLM, ein spezieller Puffer erforderlich für die ordnungsgemäße Bildgebung ist und somit eine Kammer für ein 12 mm-Deckglas mit dem Puffer auf dem Deckglas angepasst verwendet werden sollte. Nachträgliche Bildbearbeitung erfolgt mit umgekehrten Mikroskopen. Wenn das Mikroskop-Set-up ein 12 mm-Deckglas nicht zulässt, kann die hier vorgestellten Protokoll auf 18 mm Deckgläsern mit eine 30 mL Rundboden Röhrchen und einem modifizierten Adapter und Konzentrator angewendet werden. Es ist auch wichtig, beachten Sie, dass die Qualität der endgültige Wiederaufbau in SMLM hängt von der Qualität der Färbung und von den primären Antikörper verwendet, sowie die Art der Fixierung.
Zusammenfassend lässt sich sagen bieten wir eine Methode, die angewendet werden können, Bild zahlreiche Centriolen gefolgt von Fluo-SPA, die Durchschnitte der Centriolen in verschiedenen Ausrichtungen generieren wird damit zur centriolar Protein mit Genauigkeit zu lokalisieren. Wichtig ist, diese Methode im allgemeinen isoliert Centriolen von anderen Arten, andere Organellen oder großen makromolekularen Baugruppen einsetzbar. Zu guter Letzt Vorbereitung beispielhafte hier vorgestellten, kombiniert mit den letzten Algorithmenentwicklung für Single-Partikelanalyse von fluoreszierenden SMLM Daten32, konnte weitere Verbesserung in der molekularen Kartographie des großen makromolekularen öffnen Baugruppen.
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Wir danken Pierre Gönczy und BioImaging & Optik-Plattform (BIOP) an der École Polytechnique Fédérale de Lausanne (EPFL), Lausanne, Schweiz, wo die SIM-Bilder der Centriolen erworben wurden. Nikolai Klena und Davide Gambarotto werden vom European Research Council (ERC) Starting Grant (StG) 715289 (ACCENT) und Maeva Le Guennec, Paul Guichard und Virginie Hamel durch den Schweizerischen Nationalfonds (SNF) PP00P3_157517 unterstützt. Susanne Borgers wird unterstützt von der Universität Genf.
Materialien
| Name | Company | Catalog Number | Comments |
| Mouse monoclonal anti-apha tubulin (clone DM1A) | Abcam | ab7291 | dilution 1:300 |
| DNaseI | Roche | 10104159001 | |
| 12 mm coverslips | Roth | YX03.1 | |
| 18 mm coverslips | Roth | LH23.1 | |
| K2HPO4 | Fluka | 60355 | |
| KH2PO4 | Fluka | 60230 | |
| Tris base | Biosolve Chimie SARL | 0020092391BS | |
| acetic acid | Carlo Erba Reagents | 524520 | |
| NH4Cl | Sigma | A-4514 | |
| CaCl2 | Sigma | C-7902 | |
| MgSO4 | Sigma | 63140-500G-F | |
| steritop filter | Millipore | SCGPT05RE | |
| sucrose | Sigma | S7903-1KG | |
| HEPES | AppliChem PanReac | A3724,0250 | |
| PIPES | Sigma | P6757-500G | |
| MgCl2 | ACROS ORGANICS | 197530010 | |
| NP-40 | AppliChem PanReac | A1694,0250 | |
| Round-bottom (Kimble) tubes 15 mL | Fisherscientific | 09-500-34 | |
| Round-bottom (Kimble) tubes 30 mL | Fisherscientific | 09-500-37 | |
| cover glass staining rack | Thomas scientific | 8542E40 | |
| crystal polystyrene transmission lab box | FISHERS | 11712944 | |
| Methanol | VWR | 20864.32 | |
| BSA | Roche | 10735086001 | |
| Triton X100 | Roth | 3051.3 | |
| goat anti-mouse coupled to Alexa 488 | invitrogen | A11029 | dilution 1:1,000 |
| goat anti-rabbit coupled to Alexa 568 | invitrogen | A11036 | dilution 1:1,000 |
| mounting medium | abcam | ab188804 | |
| Tube, thinwall polypropylene | Beckman Coulter | 326823 | |
| Poly-D-Lysine 1 mg/mL | SIGMA | A-003-E | |
| Mouse monoclonal anti-Polyglutamylation modification mAb (GT335) | Adipogen | AG-20B-0020 | dilution 1:1,000 |
| glycerol mounting medium with DAPI and DABCO | Abcam | ab188804 | |
| 50 mL conical tubes | Falcon | 14-432-22 | |
| Eppendorf 5810R centrifuge | Eppendorf | 5811000622 | |
| Beckman JS-13.1 swinging bucket rotor | Beckman Coulter | 346963 | |
| Beckman SW 32Ti rotor | Beckman Coulter | 369694 | |
| parafilm | Bemis | 13-374-10 | |
| Leica TCS SP8 with Hyvolution mode | Leica | ||
| OSRAM L18W/954 LUMILUX | Luxe Daylight/ OSRAM | ||
| Whatman filter paper | Sigma | WHA1001325 | |
| CrSAS-6/Bld12 antibody | dilution 1:300 (Hamel el al., 2014) | ||
| Scipion | http://scipion.i2pc.es/ | ||
| EM CCD camera (Andor iXON DU897) | Andor |
Referenzen
- Bornens, M. The centrosome in cells and organisms. Science. 335 (6067), 422-426 (2012).
- Nigg, E. A., Holland, A. J. Once and only once: mechanisms of centriole duplication and their deregulation in disease. Nature Reviews Molecular Cell Biology. 19 (5), 297-312 (2018).
- Nigg, E. A., Raff, J. W. Centrioles, centrosomes, and cilia in health and disease. Cell. 139 (4), 663-678 (2009).
- Gönczy, P. Towards a molecular architecture of centriole assembly. Nature Reviews Molecular Cell Biology. 13 (7), 425-435 (2012).
- Paintrand, M., Moudjou, M., Delacroix, H., Bornens, M. Centrosome organization and centriole architecture: their sensitivity to divalent cations. Journal of Structural Biology. 108 (2), 107-128 (1992).
- Winey, M., O'Toole, E. Centriole structure. Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 369 (1650), (2014).
- Dippell, R. V. The development of basal bodies in paramecium. Proceedings of the National Academy of Sciences of the United States of America. 61 (2), 461 (1968).
- Allen, R. D. The morphogenesis of basal bodies and accessory structures of the cortex of the ciliated protozoan Tetrahymena pyriformis. The Journal of Cell Biology. 40 (3), 716-733 (1969).
- Li, S., Fernandez, J. -. J., Marshall, W. F., Agard, D. A. Three-dimensional structure of basal body triplet revealed by electron cryo-tomography. The EMBO Journal. 31 (3), 552-562 (2012).
- Guichard, P., Chrétien, D., Marco, S., Tassin, A. -. M. Procentriole assembly revealed by cryo-electron tomography. The EMBO Journal. 29 (9), 1565-1572 (2010).
- Guichard, P., et al. Native architecture of the centriole proximal region reveals features underlying its 9-fold radial symmetry. Current Biology. 23 (17), 1620-1628 (2013).
- Hirono, M. Cartwheel assembly. Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 369 (1650), (2014).
- Bornens, M., Paintrand, M., Berges, J., Marty, M. C., Karsenti, E. Structural and chemical characterization of isolated centrosomes. Cell Motility and the Cytoskeleton. 8 (3), 238-249 (1987).
- Keller, L. C., Romijn, E. P., Zamora, I., Yates, J. R., Marshall, W. F. Proteomic analysis of isolated chlamydomonas centrioles reveals orthologs of ciliary-disease genes. Current Biology. 15 (12), 1090-1098 (2005).
- Kilburn, C. L., et al. New Tetrahymena basal body protein components identify basal body domain structure. The Journal of Cell Biology. 178 (6), 905-912 (2007).
- Bauer, M., Cubizolles, F., Schmidt, A., Nigg, E. A Quantitative analysis of human centrosome architecture by targeted proteomics and fluorescence imaging. The EMBO Journal. 35 (19), 1-15 (2016).
- Hamel, V., et al. Identification of Chlamydomonas Central Core Centriolar Proteins Reveals a Role for Human WDR90 in Ciliogenesis. Current Biology. 27 (16), 2486-2498 (2017).
- Cavalier-Smith, T. basal body and flagellar development during the vegetative cell cycle and the sexual cycle of Chlamydomonas reinhardii. Journal of Cell Science. 16 (3), 529 (1974).
- Geimer, S., Melkonian, M. The ultrastructure of the Chlamydomonas reinhardtii basal apparatus: identification of an early marker of radial asymmetry inherent in the basal body. Journal of Cell Science. 117, 2663-2674 (2004).
- Nakazawa, Y., Hiraki, M., Kamiya, R., Hirono, M. SAS-6 is a cartwheel protein that establishes the 9-fold symmetry of the centriole. Current Biology. 17 (24), 2169-2174 (2007).
- Hamel, V., et al. Correlative multicolor 3D SIM and STORM microscopy. Biomedical Optics Express. 5 (10), 3326 (2014).
- Guichard, P., et al. Cell-free reconstitution reveals centriole cartwheel assembly mechanisms. Nature Communications. 8, 14813 (2017).
- Hiraki, M., Nakazawa, Y., Kamiya, R., Hirono, M. Bld10p constitutes the cartwheel-spoke tip and stabilizes the 9-fold symmetry of the centriole. Current Biology. 17 (20), 1778-1783 (2007).
- Sahl, S. J., Hell, S. W., Jakobs, S. Fluorescence nanoscopy in cell biology. Nature Reviews Molecular Cell Biology. 18 (11), 685-701 (2017).
- Hutner, S. H., Provasoli, L., Schatz, A., Haskins, C. P. Some Approaches to the Study of the Role of Metals in the Metabolism of Microorganisms. Proceedings of the American Philosophical Society. 94 (2), 152-170 (1950).
- Keller, L. C., Marshall, W. F. Isolation and proteomic analysis of chlamydomonas centrioles. Methods in Molecular Biology. 432 (1), 289-300 (2008).
- Borlinghaus, R. T., Kappel, C. HyVolution-the smart path to confocal super-resolution. Nature Methods. 13 (3), (2016).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nature Methods. 9 (7), 671-675 (2012).
- de la Rosa-Trevín, J. M., et al. Scipion: A software framework toward integration, reproducibility and validation in 3D electron microscopy. Journal of Structural Biology. 195 (1), 93-99 (2016).
- Gartenmann, L., et al. A combined 3D-SIM/SMLM approach allows centriole proteins to be localized with a precision of ∼4-5 nm. Current Biology. 27 (19), 1054-1055 (2017).
- Jaenicke, L., Kuhne, W., Spessert, R., Wahle, U., Waffenschmidt, S. Cell-wall lytic enzymes (autolysins) of Chlamydomonas reinhardtii are (hydroxy)proline-specific proteases. European Journal of Biochemistry. 170 (1-2), 485-491 (1987).
- Salas, D., et al. Angular reconstitution-based 3D reconstructions of nanomolecular structures from superresolution light-microscopy images. Proceedings of the National Academy of Sciences of the United States of America. 114 (35), 9273-9278 (2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten