Method Article
Isolamento e formazione immagine di fluorescenza per la ricostruzione di singola particella di Chlamydomonas centrioli
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Abbiamo sviluppato una strategia per purificare e un gran numero di centrioli in diversi orientamenti suscettibili per microscopia di Super-risoluzione e con una media di singola particella di immagine.
Abstract
Centrioli sono grandi assiemi macromolecolari importante per la corretta esecuzione dei processi biologici delle cellule fondamentali quali la divisione cellulare, della motilità cellulare o segnalazione delle cellule. Le alghe verdi Chlamydomonas reinhardtii ha dimostrato di essere un modello perspicace nello studio di architettura Centriolo, funzione e composizione proteica. Nonostante i grandi progressi verso concetti relativi all'architettura centriolari, una delle attuali sfide consiste nel determinare la localizzazione precisa dei componenti centriolari all'interno di regioni strutturali del Centriolo al fine di meglio comprendere il loro ruolo nel biogenesi Centriolo. Una limitazione importante si trova nella risoluzione di microscopia di fluorescenza, che complica l'interpretazione di localizzazione della proteina in questo organello con dimensioni vicino al limite di diffrazione. Per affrontare questa domanda, stiamo fornendo un metodo per purificare e l'immagine di un gran numero di c. reinhardtii centrioli con orientamenti diversi usando la microscopia di Super-risoluzione. Questa tecnica permette un'ulteriore elaborazione dei dati attraverso fluorescente singola particella in media (Fluo-SPA) a causa del gran numero di centrioli acquisito. Fluo-SPA genera medie di macchiato c. reinhardtii centrioli in diversi orientamenti, facilitando così la localizzazione delle proteine distinte nelle sub-regioni centriolari. D'importanza, questo metodo può essere applicato a centrioli immagine da altre specie o altri grandi assiemi macromolecolari.
Introduzione
Il Centriolo è un organulo evolutivamente conservato che si trova al centro del centrosoma in cellule animali e può agire come un corpo basale (denominato centrioli qui di seguito) per ciglia modello o flagelli in molti eucarioti1,2. Come tale, centrioli sono fondamentali per i processi biologici fondamentali delle cellule che vanno dall'assemblaggio mandrino alla segnalazione delle cellule. Di conseguenza, difetti di assemblaggio Centriolo o funzione sono stati associati con diverse patologie umane, tra cui ciliopatie e cancri3.
Centrioli sono possono, simmetrico, microtubule tripletto basato su strutture cilindriche che sono, in genere, ~ 450 nm lungo e ~ 250 nm ampia4,5,6,7. Microscopia elettronica convenzionale e la tomografia cryo-elettrone di centrioli da diverse specie hanno rivelato che i centrioli sono polarizzate lungo il loro asse con tre distinte regioni: una regione prossimale, un nucleo centrale e una regione distale5 , 7 , 8 , 9 , 10 , 11. importante, ciascuna di queste regioni Visualizza particolari caratteristiche strutturali. In primo luogo, il lume della regione prossimale nm-lungo 100 contiene la struttura di cartwheel collegata per la tripletta di microtubule attraverso l' elemento capocchia di spillo12. In secondo luogo, la regione centrale di 300 – 400 nm lunga contiene fibrosa densità nelle caratteristiche strutturali e lumen lungo la faccia interna dei microtubuli: il linker a forma di Y, la coda C-tubulo e tubulo A9di stub. Infine, la regione distale di 50 – 100 nm esibisce appendici Sub-distale e distale che circondano la parte distale del Centriolo5,13.
Gli ultimi due decenni sono stati contrassegnati dalla scoperta di un numero crescente di proteine centriolari, portando a una stima attuale di circa 100 proteine distinte, essendo parte del Centriolo14,15,16, 17. Nonostante questi progressi, la localizzazione precisa di queste proteine all'interno il Centriolo resta sfuggente, specialmente all'interno di sub-regioni strutturali. È importante assegnare una precisa localizzazione regioni strutturali del Centriolo è cruciale per una migliore comprensione della loro funzione. A questo proposito, c. reinhardtii centrioli sono stati strumentali in entrambi gli aspetti di primo delimitando le diverse caratteristiche strutturali lungo il cilindro9,18,19, che poi ha permesso ricercatori di correlare la localizzazione di un sottoinsieme delle proteine usando microscopia fluorescente ad una regione sub-strutturale. Questo include, per esempio, le proteine Bld12p e Bld10p, che si localizza nella regione prossimale e nella struttura del cartwheel in particolare20,21,22,23. L'elenco delle proteine localizzata sottostruttura include anche POB15 e POC16, due nuove proteine identificate mediante spettrometria di massa che decorano la regione di nucleo centrale interno di c. reinhardtii centrioli17.
Questo documento fornisce una descrizione completa del metodo sviluppato per isolare e immagine c. reinhardtii centrioli per microscopia di Super-risoluzione successiva e una media di singola particella. Per raggiungere questo obiettivo, è importante delineare le limitazioni tecniche che devono essere superati. In primo luogo, purificazione Centriolo può influenzare l'architettura generale, con la struttura del cartwheel spesso essere perdita durante le varie fasi di isolamento9. In secondo luogo, le dimensioni del Centriolo sono molto vicini al limite di diffrazione in microscopia ottica. Infatti, la risoluzione laterale che può essere ottenuta in microscopia confocale è circa 200 nm24, simile al diametro del Centriolo e la risoluzione nel z-asse è circa 2 – 3 volte inferiore, che conduce a un volume anisotropo. In terzo luogo, l'eterogeneità di orientamento di etichettatura e Centriolo anticorpo potrebbe limitare l'interpretazione necessario per localizzare una proteina in una specifica sub-regione centriolari. Infine, centrioli esistono solo due copie per cellula, rendendo difficile per acquisire un gran numero di immagini e trovare un orientamento Centriolo inequivocabili. Per ovviare a questi problemi tecnici, abbiamo sviluppato un metodo che si basa sull'applicazione di microscopia di Super-risoluzione su grandi numeri di centrioli isolati che adottano vari orientamenti. In primo luogo descriveremo un protocollo per purificare c. reinhardtii centrioli che consente la purificazione dei centrioli strutturalmente intatti e procentrioles contenenti il cartwheel. Quindi, descriveremo un protocollo dettagliato per concentrare i centrioli sulle lamelle per imaging di convenzionale o microscopia fluorescente super-risoluzione. Questo importante passo consente di aumentare il numero dei centrioli imaged in diverse posizioni. Infine, descriviamo una procedura per eseguire la singola particella in media sui dati acquisiti su microscopi fluorescenti che facilita la rilevazione di centrioli in diversi orientamenti. Complessivamente, questo metodo può essere applicato a centrioli immagine da varie specie o altri grandi assiemi macromolecolari.
Protocollo
1. Media preparazione per Centriolo isolamento e coltura di c. reinhardtii
-
Preparazione dei media alle cellule di c. reinhardtii cultura
Nota: Procedura riportata di seguito descrive la preparazione delle soluzioni madri per 1 x mezzo di rubinetto (fosfato di Tris-acetato).- Preparare un tampone fosfato (pH 7), con 250 mL di 1 M K2HPO4 (174,2 g di K2HPO4 completate da 1 L con acqua distillata) con ~ 170 mL di 1 M KH2PO4 (136,09 g di KH2PO4 in 1 L). Regolare la miscela per raggiungere pH 7.
- Preparare soluzione (40x) mescolando 96,8 g di Tris, 40 mL di tampone fosfato (pH 7) e 40 mL di acido acetico e regolare la soluzione a 1 L con acqua distillata.
- Preparare soluzione B (40x) utilizzando 16 g di NH4Cl, 2 g di CaCl2e 4 g di MgSO4. Fare attenzione a sciogliere separatamente CaCl2 in acqua distillata prima di aggiungerlo agli altri componenti. Regolare la soluzione a 1 L con acqua distillata.
- Preparare oligoelementi buffer25 di Hutner come segue.
- Per 1 L di tampone, sciogliere ogni composto nel volume indicato di acqua: EDTA disodico sale (50 g in 250 mL), ZnSO4.7h2O (22 g in 100 mL), H3BO3 (11,4 g in 200 mL), di MnCl2.4h2O (5,06 g in 50 mL) , CoCl2.6h2O (1,61 g in 50 mL), O di CuSO4.5h2(1,57 g in 50 mL), (NH4) 6Mo7O24.4h2O (1,10 g in 50 mL) e FeSO4.7h2O (4,99 g in 50 mL).
Nota: EDTA deve essere sciolto in acqua bollente, e il FeSO4 dovrebbe essere preparato Ultima per evitare l'ossidazione. - Mescolare insieme tutte le soluzioni tranne l'EDTA e portare ad ebollizione. Quindi aggiungere l'EDTA, e la soluzione dovrebbe diventare verde. Dopo la dissoluzione di tutto, raffreddare la soluzione a 70 ° C. A questa temperatura, aggiungere 85 mL di soluzione KOH calda 20% (20 g in 100 mL). Regolare la soluzione a 1 L con acqua distillata a temperatura ambiente (TA).
- Aggiungere un tappo di cotone al pallone e lasciar riposare per 1 o 2 settimane, agitando la soluzione 1 volta al giorno. La soluzione dovrebbe inizialmente essere verde e quindi diventare viola, lasciando un precipitato di ruggine-marrone; rimuovere il precipitato con carta da filtro fino a quando la soluzione è limpida. Le aliquote di congelare e conservare a-20 ° C.
- Per 1 L di tampone, sciogliere ogni composto nel volume indicato di acqua: EDTA disodico sale (50 g in 250 mL), ZnSO4.7h2O (22 g in 100 mL), H3BO3 (11,4 g in 200 mL), di MnCl2.4h2O (5,06 g in 50 mL) , CoCl2.6h2O (1,61 g in 50 mL), O di CuSO4.5h2(1,57 g in 50 mL), (NH4) 6Mo7O24.4h2O (1,10 g in 50 mL) e FeSO4.7h2O (4,99 g in 50 mL).
- Preparare 1 x rubinetto medio a crescere cellule di c. reinhardtii mescolando i seguenti componenti: 25 mL di soluzione (40x) e 25 mL di soluzione B (40x) con 1 mL di oligoelementi di Hutner buffer25e regolare la miscela a 1 L con acqua distillata. Sterilizzare la miscela utilizzando un filtro di 0,4 µm.
-
Preparazione di terreni per purificazione Centriolo
- Preparare un buffer di deflagellation utilizzando il 5% di saccarosio in 10 mM HEPES (pH 7), a un volume finale di 500 mL con acqua distillata.
- Preparare 250 mL di acido acetico 0,5 M.
- Preparare 250 mL di una soluzione di riserva 1 tubi K M (pH 7,2) dai primi aggiungendo 50 mL di H2O per sciogliere la polvere di tubi, quindi l'aggiunta di 10 N KOH finché la soluzione comincia a diventare chiaro. Titolare a pH 7,2 con 10 N e 1 N KOH e regolare la soluzione ad un volume finale di 250 mL con H2O.
- Preparare 5 soluzioni di saccarosio (w/w) come indicato di seguito.
Nota: Tutte le soluzioni di saccarosio devono essere filtrati dopo solubilizzazione usando un filtro di 0,4 µm collegato a una siringa. Si noti che le soluzioni di saccarosio % e 70% 60 sono difficili da solubilizzare e dovrebbero essere posto in un bagno di acqua pre-riscaldato a 60 ° C per facilitare la solubilizzazione. Mescolare ogni 10 min fino alla completa dissoluzione del saccarosio.- Per preparare il 25% di saccarosio, pesare 25 g di saccarosio e regolare il peso di 100 g con l'aggiunta di 10 mM tubi K (pH 7,2).
- Per preparare il 60% di saccarosio, pesare 60 g di saccarosio e regolare il peso di 100 g con 10 mM tubi K (pH 7,2).
- Preparare le soluzioni di saccarosio per il gradiente di saccarosio. Preparare 40% saccarosio 40 g di saccarosio di pesatura e regolando la soluzione di 100 g con 10 mM tubi K (pH 7,2). Allo stesso modo, preparare 50% (w/w) e saccarosio 70% (w/w).
- Memorizzare le soluzioni a-20 ° C. State attenti risospendere le soluzioni di saccarosio correttamente dopo lo scongelamento.
- Preparare 100 mL di tampone di lisi di miscelazione 1 mM HEPES (pH 7), 0,5 mM MgCl2e 1% NP-40 e conservare a 4 ° C. Sempre preparare questo tampone fresco il giorno dell'esperimento. Aggiungere anti-proteasi compresse il giorno dell'isolamento Centriolo.
- Preparare la soluzione salina tampone fosfato (PBS) (pH 7,4): 1x mescolando 8 g di NaCl, 2 g di KCl, 1,44 g di Na2HPO4e 0,24 g di KH2PO4 in 800 mL di acqua distillata H2O. Adjust pH 7.4 con HCl. Bring il volume della miscela di 1 L con acqua distillata H2O.
2. isolamento di c. reinhardtii centrioli
Nota: Vedere la Figura 1.
-
Cultura e l'espansione delle cellule di c. reinhardtii
- La sera il giorno 1, inoculare un ceppo cw15 - da una solida piastra in una cultura beuta contenente 10 mL di 1 x toccare. Crescere le cellule sotto luci fluorescenti bianche (60 µE/m2s) per 2 – 3 giorni a 23 ° C.
- Il giorno 3, diluire la cultura 10 x (per 100 mL) in 1 x Toccare e far crescere le cellule sotto la luce per 2 – 3 giorni a 23 ° C.
- Il giorno 6, diluire la cultura 10 x 1 x Toccare per ottenere 1 L di cultura. Crescere le cellule sotto la luce a 23 ° C fino a quando la cultura raggiunge un colore verde scuro che indica una densità approssimativa delle cellule di ~ 1 x 107 cellule/mL26 (giorno 9 – 10).
-
Purificazione di c. reinhardtii centrioli
- Centrifugare le cellule cw15 - a 600 x g per 10 min in provette coniche da 50 mL. Lavare il pellet di cellule 1x con 50 mL di PBS 1X e girarla a 600 x g per 10 min. Risospendere il pellet in 100 mL di tampone di deflagellation di temperatura con una pipetta.
- Deflagellate le cellule con una scossa di pH aggiungendo lentamente gocce di acido acetico 0,5 M per un pH finale di 4.5-4.7 su un agitatore magnetico e incubare le cellule per 2 min, aggiungere lentamente gocce di 1 N KOH per ripristinare il pH a 7.0.
- Centrifugare le cellule a 600 x g per 10 min rimuovere qualsiasi flagelli indipendenti. Rimuovere il supernatante e memorizzare il pellet sul ghiaccio. Lavare la pallina 2 x 50 ml di PBS 1X preraffreddata a 4 ° C. Poi, girare la pallina a 600 x g per 10 min a 4 ° C.
- Risospendere il pellet in 30 mL di PBS 1X e caricare lentamente la sospensione su un cuscino di saccarosio al 25%-di 20 mL senza miscelazione (Figura 1B).
- Spin a 600 x g per 15 min a 4 ° C per rimuovere i flagelli rimanenti nel surnatante; le cellule sono distribuite nel 25% di saccarosio (Figura 1C). Tenere solo i 20 mL di più in basso (freccia rossa, Figura 1C) accuratamente aspirando il supernatante utilizzando un aspiratore.
- Lavare i rimanenti 20 mL aggiungendo 20 mL di PBS 1X freddo. Girare il campione a 600 x g per 10 min a 4 ° C. Risospendere il pellet in 10 mL di PBS 1X freddo (a 4 ° C). Assicurarsi che non siano senza ciuffi affinché la seguente Lisi colpisce tutte le celle in una sola volta.
- Trasferire il sedimento risospeso in un nuovo flacone di 250 mL. Aggiungere 100 mL di tampone di lisi completati con 5.000 unità di DNasi per le cellule. È importante aggiungere il buffer di lisi delle cellule, e non l'altro senso intorno. Incubare la miscela per 1 h a 4 ° C e mescolare accuratamente capovolgendo il flacone ogni 15 min senza formare bolle.
- Centrifugare le cellule lisate a 600 x g per 10 min a 4 ° C in una provetta conica 50 mL per rimuovere eventuali detriti cellulari. Se la lisi è stata eseguita correttamente, il pellet cellulare dovrebbe essere bianco (Figura 1D). Raccogliere il surnatante con una pipetta e caricarlo su un tubo rotondo-fondo da 30 mL contenente un cuscino di 60%-saccarosio, posizionato su ghiaccio. Poi, girare il tubo a 10.000 x g per 30 min a 4 ° C.
Nota: I diversi tubi di turno-fondo (Tabella materiali) possono essere necessari, a seconda del volume del surnatante. - Aspirare il supernatante fino a 1 mL sopra il cuscino di saccarosio. Nota l'interfaccia giallo tra il 1 mL del surnatante rimanente e i 2 mL del cuscino del saccarosio. Mescolare delicatamente il saccarosio e il surnatante rimanente con un taglio punta P1000. Fare non vortice in questa fase; in caso contrario, il procentrioles può essere perso in questa fase. Piscina tutti i cuscini di saccarosio e memorizzarli sul ghiaccio.
- Preparare una sfumatura di 40% - 70% saccarosio in un tubo in polipropilene sottili 38,5 mL delicatamente aggiungere 3 mL di 70% di saccarosio (a 4 ° C), seguito da 3 mL di 50% e infine, 3 mL di saccarosio al 40%. Caricare le interfacce pool sul 40% - 70% saccarosio sfumatura; Questa operazione lentamente perché il saccarosio freddo è molto viscoso. Bilanciare i tubi con il buffer di 10 mM tubi K (pH 7,2) e li Centrifugare a 68.320 x g (ad es., con un rotore di SW32Ti) per 1 h e 15 min a 4 ° C.
- Raccogliere le frazioni di 12 x 500 µ l a 4 ° C praticando un foro nella parte inferiore del tubo con un ago di 0,8 mm senza disturbare gli strati differenti di saccarosio. Con un taglio puntale P200, preparare ulteriori aliquote di 10 µ l da ogni frazione che verrà utilizzato per l'immunofluorescenza seguente. Snap-congelare le frazioni in azoto liquido.
Nota: Centrioli isolati possono essere analizzati da microscopia elettronica per verificare che venga mantenuta l'ultrastruttura complessiva dei centrioli.
3. quantificazione dei centrioli isolati sulle lamelle: centrifugazione e immunofluorescenza
Nota: Vedere la Figura 2.
-
Preparare le provette e i vetrini coprioggetti
- Utilizzare 1 tubetto di vetro rotondo-basso (15 mL) per ogni frazione per analizzare le frazioni centriolari (12 tubi in totale).
- Mettere un adattatore di supporto personalizzato vetrino coprioggetti (chiamato in seguito adattatore) nel tubo tondo-fondo. Porre un coprivetrino sterile 12 mm nel tubo tondo-fondo.
Nota: Qui abbiamo usato vetrini coprioggetti 12 mm, ma il protocollo può essere adattato per vetrini coprioggetto 18 mm utilizzando tubi di turno-fondo di 30 ml. - Aggiungere 5 ml di pre-raffreddata 10 mM tubi K (pH 7,2) a 4 ° C. Assicurarsi che il vetrino coprioggetto non è fluttuante e rimane premuto sull'adattatore. Posizionare i tubi sul ghiaccio.
-
Centrifugazione dei centrioli
- Diluire ciascuna frazione 10 µ l con 100 µ l di freddo 10 mM tubi K (pH 7,2). Risospendere la diluizione bene fino alla completa scomparsa del saccarosio (Figura 2A). Caricare ogni frazione diluita in un tubo rotondo-fondo.
- Girare il tubo a 10.000 x g per 10 min (per esempio, con un rotore oscillante JS-13.1) a 4 ° C.
- Recuperare il coprioggetto con l'inserimento di un dispositivo collegato a mano nel foro presente nel bordo scanalato dell'adattatore e sollevarlo delicatamente.
Nota: Il dispositivo agganciato a mano può essere fatta con ago della siringa manualmente agganciato e modellato in una chiavetta fatti in casa (figure 2B-2D). - Una volta raggiunta la sommità del tubo rotondo-fondo, intercettare il bordo dell'adattatore con un dito guantato e rimuovere il vetrino coprioggetto con le pinzette. È necessario ricordare che parte del coverslip contiene i centrioli. Procedere per trattare i coprioggetti per immunofluorescenza.
-
Immunofluorescenza colorazione e di imaging di isolato c. reinhardtii centrioli
Nota: Vedere la Figura 2.- Preparare il materiale per l'immunofluorescenza che macchia come segue.
- Preparare un rack di colorazione di vetro di copertura in una scatola di laboratorio di trasmissione in polistirolo cristallo (60 mm di lunghezza di 50 mm di larghezza di 43 mm in altezza). Riempirlo con 100% di metanolo e conservare a-20 ° C.
- Preparare una camera umida. Per questo, montare la camera umida inserendo un fazzoletto umidificato di acqua a fianco l'interno i bordi di un quadrato di Petri (Figura 2E). Aggiungere un pezzo di laboratorio involucro di tenuta (Vedi Tabella materiali) al centro del piatto Petri su cui le miscele di anticorpo verranno inserite durante la procedura di immunofluorescenza (passi 3.3.2–3.3.3). Coprire il coperchio e la camera umida con foglio di alluminio per proteggerlo dalla luce.
- Immunostain i centrioli isolati come segue.
- Difficoltà lamelle con i centrioli direttamente dopo la centrifugazione (punto 3.2.4) incubando li per 5 min nella casella riempita con metanolo-20 ° C (punto 3.3.1.1).
- Rimuovere i vetrini coprioggetti con pinzette e posto in una scatola trasparente laboratorio (Vedi Tabella materiali) riempito con 50 mL di PBS 1X e lavarli per 5 min a temperatura ambiente.
- 60 µ l di miscela di anticorpo primario [anticorpi primari diluiti in 1% albumina di siero bovino (BSA) e 0.05% Tween-20 in PBS] sul pezzo di laboratorio tenuta avvolgere nella camera umida. Appoggiare delicatamente i coprioggetti sopra il mix di anticorpo con i centrioli direttamente di fronte la goccia. Incubare i vetrini coprioggetto per 45 min con gli anticorpi primari.
Nota: Gli anticorpi primari che sono stati utilizzati per generare i risultati rappresentativi sono Bld12 policlonali di coniglio (1: 300) e mouse α-tubulina (DM1A) (1: 300). - Rimuovere i coprioggetti e lavarli per 5 min in 1X PBS, come descritto nel passaggio 3.3.2.2. Incubare i vetrini coprioggetto per 45 min con anticorpi secondari in PBS contenente BSA 1% e lo 0,05% Tween-20.
Nota: Gli anticorpi secondari che sono stati utilizzati per generare i risultati rappresentativi sono anti-topo di capra accoppiato ad Alexa 488 (1:1, 000) e capra anti-coniglio accoppiato ad Alexa 568 (1:1, 000). - Rimuovere i coprioggetti e lavarli per 5 min in 1X PBS, come descritto nel passaggio 3.3.2.2.
- Montare i vetrini coprioggetti su una lastra di vetro con l'aggiunta di 3 µ l di soluzione di montaggio sulla diapositiva e posizionare accuratamente i coprioggetti sopra (centrioli rivolto verso il mezzo di montaggio). Sigillate il bordo del vetrino coprioggetti con smalto.
- I centrioli isolati su un microscopio confocale a un 63 X obiettivo di olio con un N.A. di 1,4 di immagine durante l'applicazione di deconvoluzione27 (Vedi Tabella materiali).
Nota: Qui, sono state utilizzate le seguenti impostazioni: 500 – 545 nm per Alexa 488 e 580 – 635 nm per Alexa 568.
- Preparare il materiale per l'immunofluorescenza che macchia come segue.
4. concentrazione di centrioli sul centro di lamelle
Nota: Vedere la Figura 3.
-
Preparazione del materiale
- Preparare una provetta di turno-fondo di vetro 15ml su ghiaccio, un adattatore per supporto personalizzato vetrino coprioggetti (chiamato adattatore d'ora in avanti, file. STL fornito come complementare File 1), un concentratore personalizzato (file. STL fornito come complementare File 2) e 10 mM tubi K (pH 7.2) a 4 ° C.
- Preparare poli-D-lisina (PDL)-rivestito coprioggetti. Diluire la soluzione di riserva di PDL 1 mg/mL con H2O.: 10x In primo luogo, lavare i vetrini coprioggetti con 70% EtOH, Rimuovi l'etanolo e lasciare che asciugare i vetrini coprioggetti. Rivestire i coprioggetti con PDL e li Incubare 30 min a temperatura ambiente. Lavare i vetrini coprioggetti 3 x con acqua e lasciarli asciugare.
Nota: Cappotto lamelle con PDL per aumentare il numero dei centrioli isolati associata a lamelle.
-
Centrifugazione
- Porre un coprivetrino sterile 12 mm sull'incasso, l'estremità inferiore del concentratore, mantenendo il PDL coat rivolto verso il basso. Tappo il coprioggetto inserendo l'adattatore direttamente sulla parte superiore. Capovolgere la provetta di turno-inferiore e posizionarlo sopra il concentratore, il vetrino coprioggetto e adattatore.
- Delicatamente spingere l'ensemble con le pinzette, finché raggiunge il fondo del tubo rotondo-fondo e capovolgere la provetta. Aggiungere 10 mM tubi K di tampone (pH 7,2) al tubo tondo-inferiore fino a quando arriva alla cima del concentratore. Assicurarsi che non rimangano bolle nel cilindro centrale del concentratore.
- Delicatamente aggiungere 100 µ l di tampone di 10 mM tubi K (pH 7,2) ad un'aliquota contenente la frazione arricchita Centriolo e mescolare bene il volume.
- Togliere il centro vuoto del concentratore di 100 µ l di tampone di K-tubi di 10 mM (pH 7,2) e aggiungere 100 µ l della frazione centriolari arricchita in tampone 10 mM tubi K (pH 7.2) per il centro vuoto del concentratore, avendo cura che il contenuto rimanga nel centro della cavo.
- Centrifugare a 10.000 x g per 10 min (per esempio, con un rotore oscillante JS-13.1) in una centrifuga pre-raffreddata a 4 ° C.
- Rimuovere il concentratore con le pinzette.
- Recuperare il coprioggetto con l'inserimento di un dispositivo collegato a mano nel foro presente nel bordo scanalato dell'adattatore e sollevarlo delicatamente. Una volta raggiunta la sommità del tubo rotondo-fondo, intercettare il bordo dell'adattatore con un dito guantato e rimuovere il vetrino coprioggetto con le pinzette. È necessario ricordare che parte del coprivetrino contenga centrioli. Procedere per trattare i coprioggetti per immunofluorescenza.
Nota: Il dispositivo agganciato a mano può essere fatta con un ago di siringa manualmente agganciato e modellato in una chiavetta fatti in casa (Figure 2B-D). - Eseguire la fissazione e colorazione di immunofluorescenza di centrioli concentrati come fatto nel passo 3.3.2–3.3.4.
5. singola particella in media
Nota: Vedere la Figura 4.
- Imaging per una media di singola particella
- Montare il vetrino coprioggetto su una diapositiva utilizzando un mezzo di montaggio anti-affievolimento regolari. Eseguire la formazione immagine di microscopia (2D SIM) illuminazione strutturata utilizzando un obiettivo CFI Apochromat TIRF (100 X, NA 1.49, WD 0,12 mm) e un retro illuminato telecamera CCD di EM.
Nota: Il tempo di acquisizione è stato impostato di 100 ms a una fotocamera leggere fuori 3 MHz. Un obiettivo X 2,5 è stata utilizzata per l'imaging di SIM.
Nota: Il set di dati qui presentati è stato acquisito su un microscopio SIM 3-d (Vedi Tabella materiali). - Immagine i centrioli acquisendo una grande pila di immagini che comprende il Centriolo totale del segnale, impostando la posizione superiore e inferiore dello Z-stack sopra e sotto il segnale di Centriolo, rispettivamente. Procedere per proiettare lo stack ed eseguire una media di singola particella secondo i punti 5.2 e 5.3.
- Montare il vetrino coprioggetto su una diapositiva utilizzando un mezzo di montaggio anti-affievolimento regolari. Eseguire la formazione immagine di microscopia (2D SIM) illuminazione strutturata utilizzando un obiettivo CFI Apochromat TIRF (100 X, NA 1.49, WD 0,12 mm) e un retro illuminato telecamera CCD di EM.
- Proiezione di uno stack
- Aprire lo stack di immagine con ImageJ. Quindi fare clic su 'immagine → pile → Z Project'. Impostare il tipo di proiezione comeIntensità massima'.
- Invertire l'immagine cliccando su 'Modifica → Inverti'. Salvare la proiezione che viene generata (formato TIF).
- Allineamento di singola particella con Scipione
- Creare un nuovo progetto in Scipion premendo il rosso 'Crea progetto' pulsante nella parte superiore della pagina. Nel riquadro sinistro, fare doppio clic su 'micrografi di importazione importazioni →'.
- Riempire il 'file directory' e 'Pattern' campi secondo i nomi e le directory dei dati. Mantenere i parametri di default. Fare clic su 'Esegui'.
- Fare doppio clic su 'Particelle → raccolta → xmipp3 - manuale raccolta (passaggio 1)'. Fare clic sull'icona della lente d'ingrandimento vicino al campo 'Input micrografie' e selezionare le micrografie importati dal punto 5.3.1. Fare clic su 'Esegui'.
- Nella finestra appena aperta, selezionare particelle dalle micrografie diversi facendo clic su di essi. Al termine di ogni microfotografia, fare clic sul pulsante rosso '+ coordinate'.
- Fare doppio clic su 'Particelle → Estratto → xmipp3 - Estratto di particelle'. Fare clic sull'icona della lente d'ingrandimento vicino al campo 'Input coordinate' e selezionare le coordinate raccolte dal punto 5.3.4. Riempire la 'Dimensione di scatola delle particelle (px)' secondo le dimensioni di particelle. Nella scheda 'Preprocess' , impostare la rimozione della polvere: nessuna (può generare artefatti); Inverti contrasto: nessuna (sfondo di particelle nere, bianco); Fase di capovolgimento: No (legata alla correzione di CTF); Normalizzare: Sì.
- Quindi, fare clic su 'Esegui'. Quando il lavoro è fatto, controllare le particelle estratte selezionando la casella del lavoro (i bordi diventati più spessi) e cliccare su 'analizza risultati (basso a sinistra del menu principale).
- Fare doppio clic su '2D → allinea → xmipp3: allinea con cl2d'. Fare clic sull'icona della lente d'ingrandimento vicino al campo 'Input particelle' e selezionare le particelle estratte dal punto 5.3.5. Non utilizzare un'immagine di riferimento. Fare clic su 'Esegui'.
- Fare doppio clic su ' allineare 2D → → altro → xmipp3 - applicare allineamento 2d'. Fare clic sull'icona della lente d'ingrandimento vicino al campo 'Input particelle' e selezionare le particelle allineate dal passaggio 5.3.7.
- Fare clic su 'Esegui' come al punto 5.3.6. I risultati possono essere controllati cliccando su 'Analizza risultati' dopo la selezione della finestra di lavoro.
- Fare doppio clic su 'Particelle → maschera → xmipp3 - applicare maschera 2d'.
- Fare clic sull'icona della lente d'ingrandimento vicino al campo 'Input particelle' e selezionare le particelle allineate dal passaggio 5.3.9. L' 'Origine della maschera' è impostata su 'Geometria'. Quindi, impostare i parametri della maschera come maschera tipo: circolare; Raggio (px): alla ricerca di una particella (un Centriolo) senza niente intorno ad esso, fare clic sui 'bacchetta magica ' icona sulla sinistra che apre una finestra per aiutare a trovare il valore perfetto; Centro di Shift: No (se la particella è perfettamente centrata) o Sì (se la particella è spostata); Spostamento X-centro: secondo la posizione della particella; Offset Y-centro: a seconda della posizione della particella. Fare clic su 'Esegui'.
- Utilizzare il pulsante ' analisi dei risultati ' per controllare se la maschera viene applicata correttamente. In caso contrario, fare clic destro sul lavoro 'Applica maschera 2d' e selezionare 'Modifica'. Modificare i parametri della maschera (dimensione e/o turni) ed Esegui nuovamente con i 'Esecuzione' pulsante.
- Fare doppio clic su '2D → classificare → xmipp3 - cl2d'.
- Fare clic sull'icona della lente d'ingrandimento vicino al campo 'Input particelle' e selezionare le particelle mascherate dal passaggio 5.3.11. Il numero delle classi dovrebbe essere per ottenere circa 50 particelle per classe. Fare clic su 'Esegui'.
- Verifica i risultati facendo clic sul pulsante ' analisi dei risultati ' . Nella finestra aperta, fare clic sull'icona 'occhio' accanto a "Cosa mostrare".
- La nuova finestra permette l'ispezione delle classi generate selezionando 'Classes2D' nel menu 'Blocco' . Controllare il contenuto di ciascuna classe selezionando 'Class00N_Particles' nello stesso menu. Ispezionare ogni classe per identificare quelli che contengono particelle solo male. Tornare alla visualizzazione di 'Classes2D' e selezionare le classi con buone particelle facendo clic su ciascuno. È possibile selezionare diverse classi tenendo premuto durante la selezione il tasto 'Ctrl' .
- Quando tutte le classi con buone particelle sono state selezionate, fare clic su '+ particelle' per creare un sottoinsieme con queste particelle.
- Nella stessa finestra, selezionare alcuni set di classi che rappresentano i diversi orientamenti delle particelle. Creare un nuovo sottogruppo facendo clic su '+ medie'.
- Per ogni orientamento/media selezionato, procedere come segue.
- Fare doppio clic su '2D → allinea → xmipp3: allinea con cl2d'.
- Fare clic sull'icona della lente d'ingrandimento vicino al campo 'Input particelle' e selezionare le particelle buone dal passaggio 5.3.17. Impostare 'Uso un'immagine di riferimento' 'Sì'. Fare clic sull'icona della lente d'ingrandimento vicino al campo 'Immagine di riferimento' e clicca sulla freccia bianca sinistra al lavoro 'Crea sottoinsieme' dal passaggio 5.3.18 e selezionare l'immagine da utilizzare come riferimento. Fare doppio clic su un oggetto per aprirlo in una finestra separata e controllare quale oggetto si tratta. Fare clic su 'Esegui'.
- Fare doppio clic su ' allineare 2D → → altro → xmipp3 - applicare allineamento 2d'. Fare clic sull'icona della lente d'ingrandimento vicino al campo 'Input particelle' e selezionare le particelle allineate dal passaggio 5.3.19.2.
- Fare clic su 'Esegui'.
- Controllare il risultato dell'allineamento ('Analisi dei risultati); mostrerà le particelle allineate e la media di queste particelle.
- Se la media è buona e le particelle sono tutti orientate allo stesso modo, è possibile salvare la media facendo clic sul ' avanzate → ImageJ' e salvare l'immagine con ImageJ.
- Se la media può essere migliorata, selezionare tutte le immagini ben orientate e creare un sottoinsieme di nuovo con il tasto '+ particelle' . Ribadire, passaggi 5.3.19.1–5.3.19.4 fino a quando il sottoinsieme è pulito (tutte le cattive particelle rimosse). Ogni volta che l'allineamento è eseguita (passo 5.3.19.2), il riferimento viene impostato alla media dell'ultima generata (da iterazione per iterazione, gli aumenti di qualità media).
Risultati
C. reinhardtii Centriolo isolamento:
Per isolare i centrioli, cw15- c. reinhardtii cellule erano cresciute in coltura liquida per diversi giorni sotto la luce e successivamente pellettate mediante centrifugazione a 600 x g per 10 min. Pelleted cellule erano lavate 1x con PBS e sedimento in un deflagellation prima del buffer deflagellation eseguendo una scossa di pH utilizzando acido acetico 0,5 M per un pH finale di 4.5-4.7 per 2 min(Figura 1). Un'aggiunta di 1 N KOH è stata utilizzata per riportare il pH a 7.0. Per separare i flagelli distaccati da corpi cellulari, cellule deflagellated sono stati prima centrifugate per rimuovere la maggior parte dei flagelli. Il pellet è stato poi lavati 2 volte con PBS e risospesi in 30 mL di PBS prima di essere caricate lentamente su un cuscino di saccarosio al 25% (Figura 1B). Il tubo era filato a 600 x g per 15 min a 4 ° C per rimuovere la maggior parte dei flagelli indipendente. Dopo la centrifugazione, i corpi cellulari erano diffuse il cuscino di saccarosio (Figura 1C) e recuperati rimuovendo i circa 30 mL di surnatante (finché la freccia rossa in Figura 1C). La risultante 20 mL di cellule lavate erano quindi risospeso in 20 mL di PBS freddo e centrifugato a 600 x g per 10 min. Successivamente, il surnatante è stato scartato, e le cellule erano completamente risospesi in 10 mL di PBS. Le cellule sono state trasferite in un flacone da 250 mL e il buffer di Lisi è stato aggiunto alle cellule sedimento in una sola volta. Dnasi è stato aggiunto per la lisi e incubata per 1 h a 4 ° C. Dopo una fase di centrifugazione per rimuovere i detriti cellulari (Vedi la pallina bianca in Figura 1D), il surnatante è stato raccolto e accuratamente caricato su un cuscino di saccarosio al 60% di 2 mL prima della centrifugazione a 10.000 x g per 30 min a 4 ° C. Si noti che per 100 mL di Lisi, 8 provette da 15 mL sono stati utilizzati per eseguire questo passaggio, corrispondente a 12,5 mL di tampone di lisi caricati su 2 mL di saccarosio per tubo. Dopo la centrifugazione, la maggior parte del surnatante è stato rimosso fino a 1 mL sopra il cuscino. I 1 mL di surnatante rimanenti era quindi raccolti con il cuscino di 2 mL e poi mescolato e riuniti per ottenere un volume finale di 24 mL. La piscina è stato poi caricata su un 40%, 50%, 70%-saccarosio gradienti e filata a 68.320 x g per 1 h e 15 min a 4 ° C. Infine, sono stati raccolti i centrioli isolati praticando un foro nella parte inferiore del tubo di centrifugazione mediante un ago e le gocce sono stati raccolti in frazioni di 12 x 500 µ l. Dovuto l'alta densità del saccarosio diverso, la goccia formato molto lentamente all'inizio (70% di saccarosio) e quindi più rapidamente (40% di saccarosio).
Immunofluorescenza di centrioli isolati
Per valutare la qualità della procedura di isolamento, 10 µ l di ciascuna frazione di gradiente raccolti è stato poi centrifugato su un vetrino coprioggetti utilizzando un adattatore per supporto vetrino coprioggetti (Figura 2A-2D). Cosa importante, per rimuovere in modo sicuro il vetrino coprioggetto, è stato progettato un dispositivo personalizzato agganciato (Figura 2B). Successivamente, le lamelle sono state analizzate dall'immunofluorescenza. Anticorpi CrSAS-6(Bld12p) sono stati utilizzati in questo studio per indicare la presenza della struttura del cartwheel e α-tubulina (DMA1) per evidenziare la parete centriolari. Contando il numero di centrioli che erano positive per CrSAS-6 e di α-tubulina al campo di vista e quindi calcolando il numero totale di centrioli per frazioni, è stato possibile determinare che le frazioni sono state arricchite per isolato centrioli ( Figura 2F-2 H). È interessante notare che, 6 frazioni sono state arricchite per centrioli (Figura 2F e 2G, frazioni #1 – 6), con un picco per frazione #3, mentre le frazioni Ultima erano non, che indica che la purificazione lavorato. Si noti che in questo particolare esperimento, 95% dei centrioli totale erano positive per CrSAS-6 e α-tubulina in frazione #3. Questo indica che più isolate centrioli mantenuto loro capriole. Se nessun arricchimento dei centrioli è osservato nelle prime frazioni, la procedura di isolamento non ha funzionato e dovrebbe essere ripetuta. Si noti che alcuni pezzi flagellari possono essere osservate principalmente nelle frazioni privo di centrioli.
Successivamente, per calcolare il numero totale di centrioli per µ l, il numero di Centriolo al campo di vista dovrebbe essere moltiplicato per il rapporto come illustrato nella Figura 2H. Il numero risultante dovrebbe poi essere diviso per il volume della frazione utilizzata per l'immunofluorescenza per ottenere il numero di centrioli per µ l. In questa procedura di isolamento particolare, la frazione più arricchita conteneva circa 37 centrioli per un'area di 0,00846 mm2 (con un campo visivo di 92x92 µm2). La superficie della sezione del tubo era 7,5 mm di raggio con una superficie totale di 176 mm2. Il rapporto corrispondente era quindi 176/0.00846 = 20,803.8, quindi un totale di 769.740 centrioli (37 x 20, 803.8) in 10 µ l. Di conseguenza, il numero dei centrioli in 1 µ l era 76.974.
Concentrazione di centrioli isolati sulle lamelle:
Aumentando il numero dei centrioli per campo aumenta la probabilità di rilevazione Centriolo inequivocabile orientamento, come pure aumentando la possibilità di rilevare simili orientamenti che possono essere utilizzati per ulteriori procedure di calcolo della media delle particelle. Come centrioli da frazioni concentrate sono ancora sparse sul vetrino coprioggetto, abbiamo sviluppato un accessorio di centrifugazione per concentrare centrioli al centro del vetrino coprioggetti (Figura 3A) denominato un concentratore. Si noti che un file. STL con le misure precise per la stampa 3D è dotato di questo manoscritto.
In primo luogo, una lamella di 12 mm è stato montato sul concentratore (Figura 3B). L'adattatore è stato posizionato sopra il vetrino coprioggetto e il tubo rotondo-fondo è stato invertito e posizionato sopra l'adattatore montato e concentratore. Il tubo rotondo-fondo era quindi delicatamente invertito, permettendo il caricamento del campione (protocollo passo 4.2). I centrioli sono stati poi centrifugati a 10.000 x g per 10 min a 4 ° C. Dopo di che, i centrioli sono stati sottoposti a immunofluorescenza e macchiati per CrSAS-6/Bld12p e α-tubulina (Figura 3-3E). Cosa importante, senza concentratore, circa 30 centrioli sono stati veduti al campo di vista (Figura 3C), mentre 183 centrioli sono stati veduti a campo visivo quando il concentratore è stato usato (Figura 3D-3F). Si noti che i centrioli coperto solo un disco di 4 mm di diametro al centro del vetrino coprioggetti. Questo risultato dimostra che il passaggio di concentrazione funziona e permette un arricchimento 6 volte di centrioli in un'area definita di lamelle, così facilitando la loro rilevazione e imaging.
Singola particella fluorescente con una media di isolato C. reinhardtii centrioli:
Qui, usando la microscopia SIM che può raggiungere una risoluzione di circa 120 nm, centrioli macchiati per monoglutamylated tubulina (GT335, Figura 4), una modifica di tubulina presente in centriolari microtubuli, erano imaged24. C. reinhardtii centrioli sono circa 500 nm lunga, sempre in coppia e spesso trovato con associati, appena duplicato probasal corpi (denominati procentrioles in appresso) e striati fibre associate ai microtubuli19. Pertanto, questa assemblea finale era circa 1 micron di grandi dimensioni. Per questo motivo e per i centrioli nella loro interezza di immagine, si consiglia di acquisire un Z-stack sui centrioli isolati.
Qui, dopo l'acquisizione, un'immagine finale è stata generata eseguendo una proiezione di massima intensità usando ImageJ28 (proiezione di intensità di progetto immagine/pile/Z/max, Figura 4B). Da tali immagini, un'analisi di singola particella utilizzando software di microscopia di cryo-elettrone è stata effettuata per rendere le classi di centrioli con orientamenti simili, e quindi in media è stata effettuata. A tale scopo, il colore dell'immagine in primo luogo è stato invertito in modo da visualizzare meglio gli oggetti (Figura 4C). Centrioli sono state raccolte manualmente in una casella centrata sopra ogni particella utilizzando il liberamente disponibile di software di Scipion29 che integra diversi programmi software microscopia elettronica come Xmipp3 (Figura 4D). Si noti che la dimensione della scatola deve essere definito dall'utente. , Scatole di 50 x 50 pixel per una dimensione di pixel di 31,84 nm sono stati usati. Successivamente, tutte le particelle sono state estratte (Figura 4E) e allineate utilizzando Xmipp3 (Figura 4F). Successivamente, è stata applicata una maschera circolare di 12 pixel nel raggio per isolare ciascun Centriolo dalla centriolari-coppia (Figura 4G). Le particelle sono stati poi classificate, utilizzando Xmipp3, per generare diverse medie. Solo classe omogenea medie erano tenute, che significa che le particelle che divergono dalle medie di classe sono stati esclusi manualmente. Questo passaggio è stato ripetuto al fine di generare una media di quasi perfetto per ogni orientamento scelto. Dopo cinque iterazioni, sono state generate due classi delle medie: una vista dall'alto da 63 oggetti (Figura 4H) e un lato Mostra da 98 particelle (Figura 4io) di monoglutamylated centrioli. Le dimensioni dell'oggetto sono state determinate misurando la distanza tra i picchi del profilo trama intensità lungo il segnale di monoglutamylation.
D'importanza, la lunghezza della media classe vista laterale è di 260 nm con un diametro di 250 nm, paragonabile al segnale misurato monoglutamylated tubulina che è stato indicato per localizzare a un'area di 286 ± 33 nm di lunghezza all'interno del nucleo del Centriolo17.
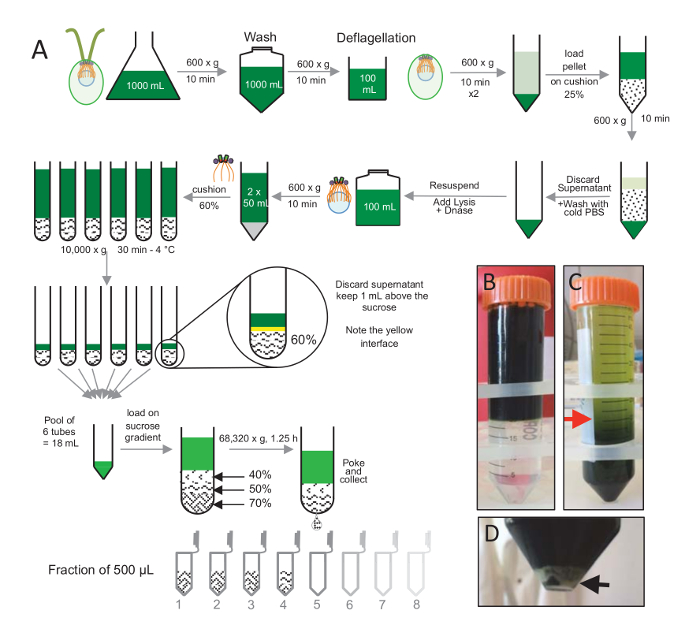
Figura 1 : Purificazione di C. reinhardtii centrioli. (A) si tratta di una rappresentazione schematica di ogni passo che porta all'isolamento di c. reinhardtii centrioli. Include passaggi rappresentativi del protocollo (B) prima e (C) dopo la centrifugazione su gradiente di saccarosio al 25%. La freccia rossa nel pannello C indica il volume minimo per mantenere dopo la centrifugazione. (D), questo pannello mostra il pellet bianco delle cellule lisate dopo centrifugazione. La freccia nera indica il pellet. Clicca qui per visualizzare una versione più grande di questa figura.
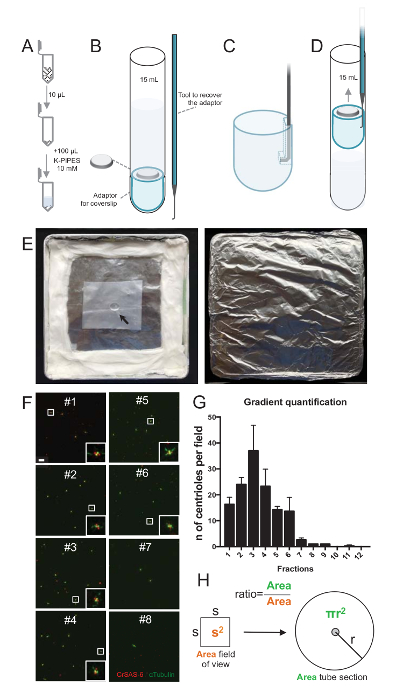
Figura 2 : Set-up di centrifugazione per eseguire immunofluorescenza su isolato centrioli. (A), 10 µ l di ciascuna frazione raccolti è diluito prima in 100 µ l di 10 mM tubi K (pH 7,2). (B) si tratta di una rappresentazione schematica dei dispositivi di centrifugazione, che comprende un tubo rotondo-fondo da 15 mL, una lamella di 12 mm, un adattatore per il vetrino coprioggetto e un dispositivo agganciato su misura per recuperare il coprioggetto dopo centrifugazione. Pannelli C e D mostrano disegni che spiega come recuperare il coprioggetto dopo la centrifugazione. (C) posto l'attrezzo a gancio nel foro presente nel bordo scanalato dell'adattatore e (D) tirare delicatamente. (E) Queste sono immagini di camera umida necessarie per eseguire imaging di immunofluorescenza. La freccia indica il coprioggetto di 12 mm. (F) questi sono immagini rappresentative confocale a 63 X (zoom 2) del gradiente frazioni #1-8, raccolti durante la procedura di purificazione e macchiato per CrSAS-6 (rosso) e α-tubulina (verde). Gli inserti corrispondono alla regione indicata da una casella bianca le viste di ingrandimento inferiore. Barra della scala = 10 µm. (G) si tratta di un grafico che rappresenta il numero di centrioli positivi per CrSAS-6 e di α-tubulina al campo di vista in ogni frazione. Si noti l'arricchimento in frazioni #1-6. Il numero medio di centrioli per ogni campo in ogni frazione: #1 = 16,3 ± 4.7, #2 = 24.0 ± 4.6, #3 = 37.0 ± 17.0, #4 = 23,3 ± 11,4, #5 = 14,3 ± 2.1, #6 = 13,7 ± 9.3, #7 = 2,7 ± 1.2, #8 = 1,0 ± 0,0, #9 = 1,0 ± 0,0, #10 = ± 0.0 0.0, #11 = ± 0,3 0,6, #12 = ± 0.0 0.0. Per ogni frazione, 3 campi casuali erano imaged. Si noti che, quando si contano la frazione più arricchita #3, abbiamo trovato che il 95% dei centrioli sono positivi per CrSAS-6 e α-tubulina (n = 205 centrioli). (H), A rapporto tra il numero dei centrioli presenti in zona misurata le micrografie viene utilizzato per calcolare il numero totale di centrioli presente nella zona del tubo. Il numero di Centriolo per µ l può essere calcolato da 10 frazioni µ l originariamente centrifugato. Clicca qui per visualizzare una versione più grande di questa figura.
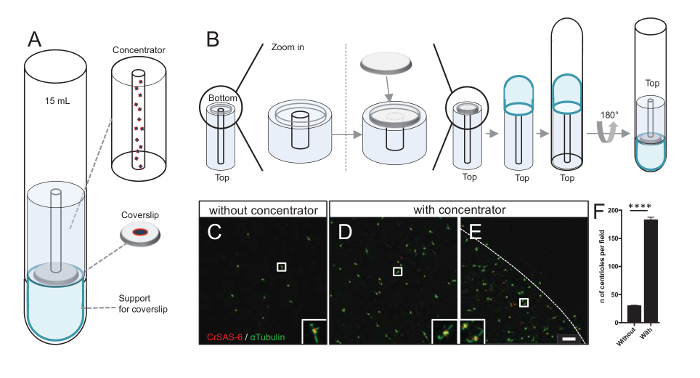
Figura 3 : Isolati centrioli sono centrifugate utilizzando concentratori. (A), questo pannello mostra i dispositivi di centrifugazione necessari a concentrarsi centrioli su vetrini coprioggetti prima dell'immunofluorescenza, tra cui un tubo rotondo-fondo da 15 mL, un concentratore, un coprioggetto di 12 mm e un apparato di supporto adattatore. (B), questo pannello mostra i passaggi per montare il dispositivo di centrifugazione. Pannelli C-E mostrano immagini confocal di centrioli macchiati per CrSAS-6 (rosso) e α-tubulina (verde) (C) senza o (D-E) con il concentratore. Gli inserti corrispondono alla regione indicata da una casella bianca le viste di ingrandimento inferiore. La barra della scala è di 10 µm. nota che i centrioli sono arricchite nel mezzo il coprioggetto (la linea tratteggiata rappresenta il confine dell'area concentrata). (F) questo grafico rappresenta il numero di centrioli al campo visivo senza e con il concentratore. Sono stati analizzati cinque casuale campi di vista. È il numero medio di centrioli, senza concentratore, 29,8 ± 2.9 e con il concentratore, 182,6 ± 11.5, P < 0,0001. La significatività statistica è stata valutata da un spaiati t-test. Clicca qui per visualizzare una versione più grande di questa figura.
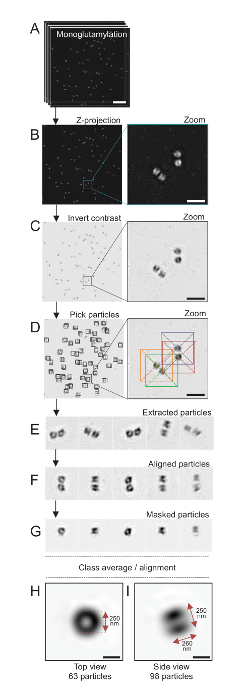
Figura 4 : Con una media di singola particella sull'isolato su C. reinhardtii centrioli. (A), questo pannello spettacoli Z stack immagini di centrioli macchiato con GT335 e acquisiti utilizzando un microscopio SIM. (B) questi pannelli mostrano una proiezione di Z di intensità massima delle immagini impilate. Barra della scala = 1 µm. (C), questi pannelli mostrano un'immagine rappresentativa con un contrasto invertito. L'inserto rappresenta uno zoom-in per visualizzare i centrioli meglio. (D), questi pannelli Visualizza particella picking. L'inserto indica come le particelle sono stati raccolti. (E) questi sono esempi di 5 estratte particelle. (F), questo pannello mostra le particelle dopo l'allineamento. (G), questo pannello mostra le particelle dopo l'applicazione di una maschera. Io e pannelli H Visualizza due classe medie: (H), un top view (63 particelle) e (mi) una vista laterale (98 particelle). Le doppie frecce indicano le dimensioni del segnale GT335. Barra della scala = 250 nm. Clicca qui per visualizzare una versione più grande di questa figura.
File supplementari 1. Per favore clicca qui per scaricare questo file.
File supplementari 2. Per favore clicca qui per scaricare questo file.
Discussione
Una delle sfide in biologia è di decifrare la precisa localizzazione delle proteine in un contesto architettonico. Il Centriolo è una struttura ideale per applicare questi metodi, come la sua architettura è stata studiata usando la tomografia di cryo-elettrone, rivelando interessanti caratteristiche ultrastrutturali lungo la sua lunghezza. Tuttavia, grazie alle sue dimensioni vicino al limite di risoluzione in microscopia ottica, è difficile da localizzare con precisione una proteina dall'immunofluorescenza una regione sub-strutturali del Centriolo usando microscopi convenzionali30.
La risoluzione in microscopia ottica è limitata dalla diffrazione della luce che dà, all'incirca, una risoluzione laterale massima di 200 nm in microscopia ottica24. Tuttavia, questo limite è stato aggirato da una delle innovazioni più importanti degli ultimi 20 anni in microscopia ottica: l'invenzione di metodi di Super-risoluzione. Questi approcci possono immagine oltre i limiti di diffrazione a diverse risoluzioni: 120 nm per SIM, circa 50 nm per svuotamento stimolato dell'emissione (STED) e 20 – 40 nm per localizzazione singola molecola microscopia (SMLM)24. Con questi nuovi sviluppi della microscopia di Super-risoluzione, il sub-regioni strutturali del Centriolo sono raggiungibili. Tuttavia, in pratica, è ancora difficile da determinare con precisione la localizzazione di una proteina ad un elemento strutturale per il motivo principale che i centrioli maturi esistano in solo 2 copie per cellula e hanno orientamenti casuali, che rende l'interpretazione di localizzazione difficile. Per questo motivo, un protocollo è stato istituito che permette ai ricercatori di immagine di Super-risoluzione di un gran numero di centrioli, aumentando la possibilità di osservare gli orientamenti non ambiguo. Cosa importante, come questo metodo si basa sull'uso di centrioli isolati, stiamo fornendo un metodo per purificare intatto c. reinhardtii centrioli contenenti centrioli maturi e procentrioles.
Infine, grazie alla gamma di orientamenti Centriolo che può essere imaged con questo protocollo, analisi di singola particella può essere applicata utilizzando software di microscopia elettronica. Questo determina la generazione di classi media di centrioli con un determinato orientamento. D'importanza, queste immagini 2D risultanti quindi utilizzabile per valutare la localizzazione di una proteina specifica lungo il Centriolo. Infatti, questo metodo può essere applicato alle immagini ad alta super-risoluzione doppio-colore, e un colore può essere utilizzato per rivelare lo scheletro Centriolo (ad es., tubulina), mentre l'altro colore può essere attribuito a una specifica proteina centriolari. Sottraendo le medie ottenute con un colore o due colori, diventa più facile registrare una proteina lungo il Centriolo (prossimale, centrale o distale). Si noti che i due canali devono essere allineati con precisione per evitare eventuali interpretazioni fuorvianti. Inoltre, medie di viste dall'alto verranno aiuterà a decifrare se una proteina si localizza all'interno del lume centriolari, lungo la parete dei microtubuli, o fuori il Centriolo.
Questo metodo ha il vantaggio di accertare la localizzazione delle proteine specifiche che potrebbero essere difficili da localizzare altrimenti a causa di etichettatura eterogenei. Si noti che altri metodi per mappare le proteine all'interno i centrioli sono state descritte in SIM/SMLM 3D correlativo con, per esempio, valutare gli orientamenti specifici dei centrioli determinando il profilo ellittico di un marcatore formando un toro intorno il Centriolo da formazione immagine SIM. Utilizzando questo parametro, è possibile localizzare la proteina con una precisione di 4 – 5 nm30. Si noti inoltre che il metodo descritto qui utilizza centrioli isolati con procentrioles intatto, un segno che l'architettura Centriolo è probabilmente in gran parte conservato. Tuttavia, non possiamo escludere che alcune caratteristiche architettoniche sono disturbati durante la purificazione, come ad esempio il diametro Centriolo varia con la concentrazione di cationi bivalenti come amplificato con l'isolamento del centrosoma umano5.
Una delle fasi critiche del protocollo presentato qui è ottenere sufficientemente concentrate centrioli isolati in diversi orientamenti suscettibili di Fluo-SPA. A tale scopo, in primo luogo garantire la purezza e l'efficienza della procedura di isolamento Centriolo. Una concentrazione bassa di centrioli isolati impedirà il corretto imaging e ulteriore elaborazione delle immagini. Per questo scopo, stiamo fornendo un metodo per arricchire il numero dei centrioli al campo di vista. A seconda del numero dei centrioli in frazione utilizzata, deve essere regolato il volume caricato nel concentratore, con un volume massimo di 250 µ l.
D'importanza, questo metodo è stato sviluppato per una parete cellulare meno cellule cw15-C. reinhardtii . In questo ceppo, la fragilità della parete cellulare consente un'adeguata lisi delle cellule e, quindi, la liberazione del suo contenuto. Questo protocollo non è efficiente per il selvaggio-tipo c. reinhardtii cellule, come la parete cellulare impedisce una corretta lisi. Strategie alternative quali sonicazione o una pre-incubazione delle cellule con autolysin, un enzima che può degradare la parete cellulare31, avrebbe dovuto essere messo in atto per alterare la parete cellulare prima di applicare il protocollo di isolamento presentato qui.
Questo set-up può essere utilizzato con diversi tipi di microscopi, che vanno da convenzionali microscopi confocali a microscopi di Super-risoluzione dedicata a resa elevata. Notare che quando si fa SMLM, un buffer speciale è richiesto per l'imaging corretta e, pertanto, deve essere utilizzata una camera adattata per un coprioggetto di 12mm con il buffer in cima il vetrino coprioggetti. La formazione immagine successiva verrà eseguita con microscopi rovesciati. Se il set-up del microscopio non consente un coprioggetto di 12mm, il protocollo presentato qui può essere applicato alle lamelle di 18 mm utilizzando un tubo da 30 mL rotondo-fondo e un adattatore modificate e concentratore. È anche importante nota che la qualità della ricostruzione finale in SMLM dipenderà la qualità della colorazione e dell'anticorpo primario utilizzato, nonché il metodo di fissazione.
In sintesi, stiamo fornendo un metodo che può essere applicato all'immagine centrioli numerosi seguiti da Fluo-SPA che genererà medie dei centrioli in diversi orientamenti, contribuendo così a localizzare centriolari proteina con precisione. D'importanza, questo metodo può essere applicato più generalmente centrioli isolati da altre specie, per altri organelli o alle grandi assemblee macromolecolari. Infine, l'approccio di preparazione del campione presentato qui, combinato con il recente sviluppo di un algoritmo per analisi di singola particella di fluorescente SMLM dati32, potrebbe aprire ulteriore miglioramento nella cartografia molecolare di grandi dimensioni macromolecolare assembly.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Ringraziamo Pierre Gönczy e bioimmagini & ottica piattaforma (BIOP) alla École Polytechnique Fédérale de Lausanne (EPFL), Lausanne, Svizzera, dove sono state acquisite le immagini SIM dei centrioli. Nikolai Klena e Davide Gambarotto sono supportati dall'European Research Council (ERC) a partire Grant (StG) 715289 (accento) e Maeva Le Guennec, Paul Guichard e Virginie Hamel dalla PP00P3_157517 di fondazione di scienza nazionale svizzero (FNS). Susanne Borgers è supportato dall'Università di Ginevra.
Materiali
| Name | Company | Catalog Number | Comments |
| Mouse monoclonal anti-apha tubulin (clone DM1A) | Abcam | ab7291 | dilution 1:300 |
| DNaseI | Roche | 10104159001 | |
| 12 mm coverslips | Roth | YX03.1 | |
| 18 mm coverslips | Roth | LH23.1 | |
| K2HPO4 | Fluka | 60355 | |
| KH2PO4 | Fluka | 60230 | |
| Tris base | Biosolve Chimie SARL | 0020092391BS | |
| acetic acid | Carlo Erba Reagents | 524520 | |
| NH4Cl | Sigma | A-4514 | |
| CaCl2 | Sigma | C-7902 | |
| MgSO4 | Sigma | 63140-500G-F | |
| steritop filter | Millipore | SCGPT05RE | |
| sucrose | Sigma | S7903-1KG | |
| HEPES | AppliChem PanReac | A3724,0250 | |
| PIPES | Sigma | P6757-500G | |
| MgCl2 | ACROS ORGANICS | 197530010 | |
| NP-40 | AppliChem PanReac | A1694,0250 | |
| Round-bottom (Kimble) tubes 15 mL | Fisherscientific | 09-500-34 | |
| Round-bottom (Kimble) tubes 30 mL | Fisherscientific | 09-500-37 | |
| cover glass staining rack | Thomas scientific | 8542E40 | |
| crystal polystyrene transmission lab box | FISHERS | 11712944 | |
| Methanol | VWR | 20864.32 | |
| BSA | Roche | 10735086001 | |
| Triton X100 | Roth | 3051.3 | |
| goat anti-mouse coupled to Alexa 488 | invitrogen | A11029 | dilution 1:1,000 |
| goat anti-rabbit coupled to Alexa 568 | invitrogen | A11036 | dilution 1:1,000 |
| mounting medium | abcam | ab188804 | |
| Tube, thinwall polypropylene | Beckman Coulter | 326823 | |
| Poly-D-Lysine 1 mg/mL | SIGMA | A-003-E | |
| Mouse monoclonal anti-Polyglutamylation modification mAb (GT335) | Adipogen | AG-20B-0020 | dilution 1:1,000 |
| glycerol mounting medium with DAPI and DABCO | Abcam | ab188804 | |
| 50 mL conical tubes | Falcon | 14-432-22 | |
| Eppendorf 5810R centrifuge | Eppendorf | 5811000622 | |
| Beckman JS-13.1 swinging bucket rotor | Beckman Coulter | 346963 | |
| Beckman SW 32Ti rotor | Beckman Coulter | 369694 | |
| parafilm | Bemis | 13-374-10 | |
| Leica TCS SP8 with Hyvolution mode | Leica | ||
| OSRAM L18W/954 LUMILUX | Luxe Daylight/ OSRAM | ||
| Whatman filter paper | Sigma | WHA1001325 | |
| CrSAS-6/Bld12 antibody | dilution 1:300 (Hamel el al., 2014) | ||
| Scipion | http://scipion.i2pc.es/ | ||
| EM CCD camera (Andor iXON DU897) | Andor |
Riferimenti
- Bornens, M. The centrosome in cells and organisms. Science. 335 (6067), 422-426 (2012).
- Nigg, E. A., Holland, A. J. Once and only once: mechanisms of centriole duplication and their deregulation in disease. Nature Reviews Molecular Cell Biology. 19 (5), 297-312 (2018).
- Nigg, E. A., Raff, J. W. Centrioles, centrosomes, and cilia in health and disease. Cell. 139 (4), 663-678 (2009).
- Gönczy, P. Towards a molecular architecture of centriole assembly. Nature Reviews Molecular Cell Biology. 13 (7), 425-435 (2012).
- Paintrand, M., Moudjou, M., Delacroix, H., Bornens, M. Centrosome organization and centriole architecture: their sensitivity to divalent cations. Journal of Structural Biology. 108 (2), 107-128 (1992).
- Winey, M., O'Toole, E. Centriole structure. Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 369 (1650), (2014).
- Dippell, R. V. The development of basal bodies in paramecium. Proceedings of the National Academy of Sciences of the United States of America. 61 (2), 461 (1968).
- Allen, R. D. The morphogenesis of basal bodies and accessory structures of the cortex of the ciliated protozoan Tetrahymena pyriformis. The Journal of Cell Biology. 40 (3), 716-733 (1969).
- Li, S., Fernandez, J. -. J., Marshall, W. F., Agard, D. A. Three-dimensional structure of basal body triplet revealed by electron cryo-tomography. The EMBO Journal. 31 (3), 552-562 (2012).
- Guichard, P., Chrétien, D., Marco, S., Tassin, A. -. M. Procentriole assembly revealed by cryo-electron tomography. The EMBO Journal. 29 (9), 1565-1572 (2010).
- Guichard, P., et al. Native architecture of the centriole proximal region reveals features underlying its 9-fold radial symmetry. Current Biology. 23 (17), 1620-1628 (2013).
- Hirono, M. Cartwheel assembly. Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 369 (1650), (2014).
- Bornens, M., Paintrand, M., Berges, J., Marty, M. C., Karsenti, E. Structural and chemical characterization of isolated centrosomes. Cell Motility and the Cytoskeleton. 8 (3), 238-249 (1987).
- Keller, L. C., Romijn, E. P., Zamora, I., Yates, J. R., Marshall, W. F. Proteomic analysis of isolated chlamydomonas centrioles reveals orthologs of ciliary-disease genes. Current Biology. 15 (12), 1090-1098 (2005).
- Kilburn, C. L., et al. New Tetrahymena basal body protein components identify basal body domain structure. The Journal of Cell Biology. 178 (6), 905-912 (2007).
- Bauer, M., Cubizolles, F., Schmidt, A., Nigg, E. A Quantitative analysis of human centrosome architecture by targeted proteomics and fluorescence imaging. The EMBO Journal. 35 (19), 1-15 (2016).
- Hamel, V., et al. Identification of Chlamydomonas Central Core Centriolar Proteins Reveals a Role for Human WDR90 in Ciliogenesis. Current Biology. 27 (16), 2486-2498 (2017).
- Cavalier-Smith, T. basal body and flagellar development during the vegetative cell cycle and the sexual cycle of Chlamydomonas reinhardii. Journal of Cell Science. 16 (3), 529 (1974).
- Geimer, S., Melkonian, M. The ultrastructure of the Chlamydomonas reinhardtii basal apparatus: identification of an early marker of radial asymmetry inherent in the basal body. Journal of Cell Science. 117, 2663-2674 (2004).
- Nakazawa, Y., Hiraki, M., Kamiya, R., Hirono, M. SAS-6 is a cartwheel protein that establishes the 9-fold symmetry of the centriole. Current Biology. 17 (24), 2169-2174 (2007).
- Hamel, V., et al. Correlative multicolor 3D SIM and STORM microscopy. Biomedical Optics Express. 5 (10), 3326 (2014).
- Guichard, P., et al. Cell-free reconstitution reveals centriole cartwheel assembly mechanisms. Nature Communications. 8, 14813 (2017).
- Hiraki, M., Nakazawa, Y., Kamiya, R., Hirono, M. Bld10p constitutes the cartwheel-spoke tip and stabilizes the 9-fold symmetry of the centriole. Current Biology. 17 (20), 1778-1783 (2007).
- Sahl, S. J., Hell, S. W., Jakobs, S. Fluorescence nanoscopy in cell biology. Nature Reviews Molecular Cell Biology. 18 (11), 685-701 (2017).
- Hutner, S. H., Provasoli, L., Schatz, A., Haskins, C. P. Some Approaches to the Study of the Role of Metals in the Metabolism of Microorganisms. Proceedings of the American Philosophical Society. 94 (2), 152-170 (1950).
- Keller, L. C., Marshall, W. F. Isolation and proteomic analysis of chlamydomonas centrioles. Methods in Molecular Biology. 432 (1), 289-300 (2008).
- Borlinghaus, R. T., Kappel, C. HyVolution-the smart path to confocal super-resolution. Nature Methods. 13 (3), (2016).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nature Methods. 9 (7), 671-675 (2012).
- de la Rosa-Trevín, J. M., et al. Scipion: A software framework toward integration, reproducibility and validation in 3D electron microscopy. Journal of Structural Biology. 195 (1), 93-99 (2016).
- Gartenmann, L., et al. A combined 3D-SIM/SMLM approach allows centriole proteins to be localized with a precision of ∼4-5 nm. Current Biology. 27 (19), 1054-1055 (2017).
- Jaenicke, L., Kuhne, W., Spessert, R., Wahle, U., Waffenschmidt, S. Cell-wall lytic enzymes (autolysins) of Chlamydomonas reinhardtii are (hydroxy)proline-specific proteases. European Journal of Biochemistry. 170 (1-2), 485-491 (1987).
- Salas, D., et al. Angular reconstitution-based 3D reconstructions of nanomolecular structures from superresolution light-microscopy images. Proceedings of the National Academy of Sciences of the United States of America. 114 (35), 9273-9278 (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon