Method Article
Isolement et imagerie de Fluorescence pour la Reconstruction de la particule de Chlamydomonas Centrioles
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Nous avons développé une stratégie visant à purifier et un grand nombre de centrioles dans différentes orientations se prête pour la microscopie de Super-résolution et particule avec une moyenne de l’image.
Résumé
Centrioles sont grands assemblages macromoléculaires importants pour la bonne exécution des processus biologiques fondamentaux cellule telles que la division cellulaire, la motilité cellulaire ou la signalisation de cellules. L’algue Chlamydomonas reinhardtii s’est avéré pour être un modèle perspicace dans l’étude de l’architecture de centriole, fonction et composition en protéines. Malgré les grands progrès vers architecture centriolaire compréhension, l’un des défis actuels est de déterminer la localisation précise des composants centriolaire dans des régions structurales de la centriole afin de mieux comprendre leur rôle dans biogenèse centriole. Une limitation majeure réside dans la résolution de la microscopie en fluorescence, ce qui complique l’interprétation de la localisation de la protéine dans cet organite avec des dimensions proches de la limite de diffraction. Pour s’attaquer à cette question, nous fournissons une méthode pour purifier et l’image d’un grand nombre de c. reinhardtii centrioles avec différentes orientations Super-résolution par microscopie. Cette technique permet le traitement ultérieur des données par le biais de fluorescent particule en moyenne (Fluo-SPA) en raison du grand nombre de centrioles acquis. Fluo-SPA génère des moyennes de teint c. reinhardtii centrioles dans des orientations différentes, facilitant ainsi la localisation des protéines distinctes dans les sous-régions centriole. Ce qui est important, cette méthode peut être appliquée aux centrioles image d’autres espèces ou d’autres grands assemblages macromoléculaires.
Introduction
Le centriole est un organite évolutivement conservé qui se trouve au cœur du centrosome dans les cellules animales et peut agir comme un corps basal (appelé centrioles ci-après) aux cils de modèle ou de flagelles dans nombreux eucaryotes1,2. Ainsi, les centrioles sont critiques pour les processus biologiques fondamentaux cellule allant de montage de la broche de signalisation cellulaire. Par conséquent, tout défaut de centriole assembly ou de la fonction ont été associés à plusieurs pathologies humaines, y compris les cancers et les ciliopathies3.
Centrioles sont neuf fois, symétrique, microtubule triplet axée sur les structures cylindriques, qui sont, en règle générale, ~ 450 nm de long et de ~ 250 nm de large4,5,6,7. La microscopie électronique conventionnelle et la tomographie cryo-électronique des centrioles provenant de différentes espèces ont révélé que les centrioles sont polarisés le long de leur axe longitudinal avec trois régions distinctes : une zone proximale, un noyau central et une partie distale5 , 7 , 8 , 9 , 10 , 11. ce qui est important, chacune de ces régions affiche des caractéristiques structurelles spécifiques. Tout d’abord, la lumière de la région proximale de 100 nm de long contient la structure de la roue de charrette connectée pour le triplet de microtubules grâce à l' élément de tête d’épingle12. En second lieu, la région centrale de 300 à 400 nm de long contient des densités fibreuses dans les caractéristiques structurelles et de la lumière le long de la face interne des microtubules : l’éditeur de liens en forme de Y, la queue C-tubule et les tubules A talon9. Enfin, la région distale de 50 – 100 nm pièces Sub distales et distales des appendices qui entourent la partie distale du centriole5,13.
Les deux dernières décennies ont été marquées par la découverte d’un nombre croissant de protéines centriolaire, conduisant à une estimation actuelle d’environ 100 protéines distinctes faisant partie du centriole14,15,16, 17. Malgré ces progrès, la localisation précise de ces protéines dans le centriole demeure difficile à atteindre, en particulier dans les sous-régions structurelles. Ce qui est important, assignant une localisation précise à des régions structurales du centriole est crucial pour une meilleure compréhension de leur fonction. À cet égard, c. reinhardtii centrioles ont grandement contribués à ces deux aspects de première délimitant les différentes caractéristiques structurelles le long du cylindre9,18,19, qui a alors permis chercheurs à établir une corrélation entre la localisation d’un sous-ensemble des protéines par microscopie fluorescente à une région infra structurelle. Cela inclut, par exemple, les protéines Bld12p et Bld10p, dont la localisation dans la région proximale et dans la structure de la roue de charrette en particulier20,,21,22,23. La liste des protéines de la sous-structure localisé comprend également POB15 et POC16, deux nouvelles protéines identifiées par spectrométrie de masse qui ornent la région intérieure centrale de c. reinhardtii centrioles17.
Cet article fournit une description complète de la méthode développée pour isoler et image c. reinhardtii centrioles pour microscopie Super-résolution ultérieures et une moyenne de particule. Pour atteindre cet objectif, il est important de délimiter les limites techniques qui doivent être surmontés. Tout d’abord, purification de centriole peut affecter l’architecture globale, avec la structure de la roue étant souvent perdue durant les différentes étapes de l’isolement,9. Deuxièmement, les dimensions de la centriole sont très proches de la limite de diffraction en microscopie optique. En effet, la résolution latérale qui peut être obtenue en microscopie confocale est environ 200 nm24, semblable au diamètre de la centriole et la résolution dans le z-axe est de 2 à 3 x plus faible, conduisant à un volume anisotrope. Troisièmement, l’hétérogénéité de l’orientation de l’étiquetage et le centriole anticorps pourrait limiter l’interprétation nécessaire pour localiser une protéine dans une sous-région centriolaire spécifique. Enfin, les centrioles existent dans seulement deux copies par cellule, rendant difficile d’acquérir un grand nombre d’images et de trouver une orientation claire centriole. Pour contourner ces problèmes techniques, nous avons développé une méthode qui repose sur l’application de la microscopie de Super-résolution sur un grand nombre de centrioles isolés qui adoptent diverses orientations. Nous décrirons tout d’abord un protocole pour purifier les centrioles c. reinhardtii qui permet la purification de centrioles structurellement intacts et procentrioles contenant la roue de la charrette. Ensuite, nous allons décrire un protocole étape par étape pour concentrer les centrioles sur lamelles couvre-objet pour l’imagerie conventionnelle ou microscopie fluorescente Super-résolution. Cette étape importante permet d’accroître le nombre des centrioles imagés à orientations multiples. Enfin, nous allons décrire une procédure pour effectuer une particule avec une moyenne sur les données acquises sur les microscopes fluorescents qui facilite la détection des centrioles dans différentes orientations. Au total, cette méthode peut être appliquée aux centrioles image de diverses espèces ou autres grands assemblages macromoléculaires.
Protocole
1. médias préparation pour c. reinhardtii Culture et isolement de Centriole
-
Préparation de milieux pour cellules de c. reinhardtii culture
Remarque : Les étapes ci-dessous décrivent la préparation des solutions mères pour 1 x support (phosphate de Tris-acétate) de robinet.- Préparer un tampon phosphate (pH 7), en mélangeant 250 mL de 1 M K2HPO4 (174,2 g K2HPO4 complété à 1 L avec de l’eau distillée) ~ 170 ml de 1 M KH2PO4 (136,09 g de KH2PO4 en 1 L). Ajuster le mélange pour atteindre le pH 7.
- Préparer la solution A (x 40) en mélangeant 96,8 g de Tris, 40 mL de tampon phosphate (pH 7) et 40 mL d’acide acétique et ajuster la solution à 1 L avec de l’eau distillée.
- Préparer la solution B (x 40) à l’aide de 16 g de NH4Cl, 2 g de CaCl2et 4 g de MgSO4. Veillez à dissoudre CaCl2 dans l’eau distillée séparément avant de l’ajouter aux autres composants. Ajuster la solution à 1 L avec de l’eau distillée.
- Préparer oligo-éléments tampon25 de Hutner comme suit.
- Pour 1 litre de tampon, dissoudre chaque composé dans le volume d’eau : 50 g dans 250 mL de sel de disodium EDTA ZnSO4.7H2O (22 g dans 100 mL), H3BO3 (11,4 g dans 200 mL), MnCl2.4H2O (5,06 g dans 50 mL) , CoCl2.6H2O (1,61 g dans 50 mL), CuSO4.5H2O (1,57 g dans 50 mL), (NH4) 6Mo7O24.4H2O (1,10 g dans 50 mL) et FeSO4.7H2O (4,99 g dans 50 mL).
NOTE : EDTA doit être dissous dans l’eau bouillante, et la FeSO4 doit être préparé dernière afin d’éviter l’oxydation. - Mélanger toutes les solutions sauf l’EDTA et amener à ébullition. Ajouter ensuite l’EDTA et la solution doit verdir. Après la dissolution de tout, laisser refroidir la solution à 70 ° C. À cette température, ajouter 85 mL de solution chaude de KOH 20 % (20 g dans 100 mL). Ajuster la solution à 1 L avec de l’eau distillée à température ambiante (RT).
- Ajouter une fiche de coton dans la fiole et laisser la solution pendant 1 ou 2 semaines, secouant 1 x par jour. La solution doit initialement être vert et puis deviennent violette, laissant un précipité brun-rouille ; Retirez le précipité à l’aide de papier filtre jusqu'à ce que la solution est claire. Geler les aliquotes et conserver à-20 ° C.
- Pour 1 litre de tampon, dissoudre chaque composé dans le volume d’eau : 50 g dans 250 mL de sel de disodium EDTA ZnSO4.7H2O (22 g dans 100 mL), H3BO3 (11,4 g dans 200 mL), MnCl2.4H2O (5,06 g dans 50 mL) , CoCl2.6H2O (1,61 g dans 50 mL), CuSO4.5H2O (1,57 g dans 50 mL), (NH4) 6Mo7O24.4H2O (1,10 g dans 50 mL) et FeSO4.7H2O (4,99 g dans 50 mL).
- Préparer 1 x robinet moyenne à la croissance de cellules de c. reinhardtii en mélangeant les éléments suivants : 25 mL de solution A (x 40) et 25 mL de solution B (x 40) avec 1 mL d’oligo-éléments de Hutner tampon25et ajuster le mélange dans 1 L d’eau distillée. Stériliser le mélange à l’aide d’un filtre de 0,4 µm.
-
Préparation des supports pour la purification de centriole
- Préparer un tampon de deflagellation à l’aide de 5 % de saccharose en 10 mM HEPES (pH 7) à un volume final de 500 mL ajusté avec de l’eau distillée.
- Préparez 250 mL d’acide acétique 0,5 M.
- Préparez 250 mL d’une solution mère à 1 M K-PIPES (pH 7,2) par première ajouter 50 mL d’H2O à dissoudre la poudre de PIPES, puis ajouter 10 N KOH jusqu'à la solution commence à devenir clair. Titrer à pH 7.2 avec 10 N et 1 N KOH et ajuster la solution pour un volume final de 250 mL avec H2O.
- Préparer ainsi 5 solutions de saccharose (w/w).
Remarque : Toutes les solutions de saccharose doivent être filtrée après que solubilisation filtre 0,4 µm branché dans une seringue. Notez que les solutions de saccharose % et 70 %-60 sont difficiles à solubiliser et doivent être placées dans un bain-marie préchauffé à 60 ° C pour faciliter la solubilisation. Mélanger toutes les 10 min jusqu'à ce que le saccharose est complètement dissous.- Pour préparer les 25 % de sucrose, peser 25 g de saccharose et ajuster le poids de 100 g en ajoutant 10 mM K-PIPES (pH 7,2).
- Pour préparer les 60 % de sucrose, peser 60 g de saccharose et ajuster le poids de 100 g avec 10 mM K-PIPES (pH 7,2).
- Préparer des solutions de saccharose pour le gradient de saccharose. Préparer les 40 % de saccharose en pesant 40 g de saccharose et en ajustant la solution à 100 g avec 10 mM K-PIPES (pH 7,2). De même, préparez 50 % (p/p) et saccharose de 70 % (p/p).
- Stocker les solutions à-20 ° C. Veillez à remettre en suspension les solutions de saccharose correctement après décongélation.
- Préparation de 100 mL de tampon de lyse par mélange 1 mM HEPES (pH 7), 0,5 mM MgCl2et 1 % NP-40 et conserver à 4 ° C. Toujours préparer ce tampon frais le jour de l’expérience. Ajouter anti-protéases comprimés le jour de l’isolement de centriole.
- Préparer 1 x solution saline le tampon au phosphate (PBS) (pH 7,4) en mélangeant 8 g de NaCl, 2 g de KCl, 1,44 g de Na2HPO4et 0,24 g de KH2PO4 dans 800 mL de distillée H2O. ajuster le pH à 7,4 avec HCl. apportez le volume du mélange à 1 L avec distillée H2O.
2. isolement de c. reinhardtii Centrioles
Remarque : Voir la Figure 1.
-
La culture et l’expansion des cellules de c. reinhardtii
- Dans la soirée le jour 1, inoculer une mission AC 15 - déformation d’une plaque solide dans une culture de l’erlenmeyer contenant 10 mL de 1 x robinet. Cultiver les cellules sous un éclairage fluorescent blanc (60 µE/m2s) 2 – 3 jours à 23 ° C.
- Le jour 3, diluer la culture 10 x (pour 100 mL) 1 x TAP et pousse les cellules sous la lumière pendant 2 à 3 jours à 23 ° C.
- Jour 6, diluer la culture 10 x 1 x robinet pour obtenir 1 L de la culture. Cultiver les cellules sous la lumière à 23 ° C jusqu'à ce que la culture atteint une couleur vert foncé qui indique une densité cellulaire approximative de ~ 1 x 107 cellules/mL26 (jour 9 – 10).
-
Purification des centrioles reinhardtii c.
- Centrifuger les mission AC 15 - cellules à 600 x g pendant 10 min dans les tubes coniques de 50 mL. Laver le culot de cellules 1 x avec 50 mL de PBS 1 x et le tourner à 600 x g pendant 10 min. remettre le culot dans 100 mL de tampon deflagellation à température ambiante avec une pipette.
- Deflagellate les cellules avec un choc de pH en ajoutant lentement des gouttes d’acide acétique de 0,5 M à un pH final de 4.5 – 4.7 sur un agitateur magnétique et incuber les cellules pendant 2 min. ajouter lentement des gouttes de 1 N KOH pour rétablir le pH à 7.0.
- Centrifuger les cellules à 600 x g pendant 10 min enlever toute flagelles détachés. Retirez le surnageant et stocker les granulés sur la glace. Laver le culot 2 x 50 ml de PBS 1 x prérefroidir à 4 ° C. Puis, faites tourner le culot à 600 x g pendant 10 min à 4 ° C.
- Resuspendre le culot dans 30 mL de PBS 1 x et charger lentement la suspension sur un coussin de 25 % du saccharose 20 ml sans mélange (Figure 1B).
- Tournent à 600 x g pendant 15 min à 4 ° C pour éliminer les flagelles restants dans le surnageant ; les cellules sont répartis dans le 25 % de sucrose (Figure 1C). Conservez seulement le plus bas 20 mL (flèche rouge, Figure 1C) par aspirer soigneusement le surnageant à l’aide d’un aspirateur.
- Laver le reste 20 mL en ajoutant 20 mL de PBS 1 x contre le rhume. Faire tourner l’échantillon à 600 x g pendant 10 min à 4 ° C. Resuspendre le culot dans 10 mL de PBS 1 x contre le rhume (à 4 ° C). S’assurer qu’il n’y a aucun amas afin que la lyse suivante frappe toutes les cellules à la fois.
- Transférer l’extrait concentré dans un nouveau flacon de 250 mL. Ajouter 100 mL de tampon lyse additionné de 5 000 unités de DNase aux cellules. Il est important d’ajouter le tampon de lyse les cellules, et non l’inverse. Incuber le mélange pendant 1 h à 4 ° C et mélanger soigneusement en retournant la bouteille toutes les 15 min sans former de bulles.
- Centrifuger les cellules lysées à 600 x g pendant 10 min à 4 ° C dans un tube conique de 50 mL pour enlever tous les débris cellulaires. Si la lyse a été effectuée correctement, le culot cellulaire doit être blanc (Figure 1D). Recueillir le liquide surnageant avec une pipette et le charger dans un tube à fond rond 30 mL contenant un coussin de 60 % du saccharose, mis dans la glace. Puis, faites tourner le tube à 10 000 x g pendant 30 min à 4 ° C.
Remarque : Plusieurs tubes fond rond (Table des matières) peuvent être nécessaires, selon le volume du liquide surnageant. - Aspirer le surnageant jusqu'à 1 mL le coussin de saccharose. Remarque l’interface jaune entre la 1 mL du surnageant restant et les 2 mL du coussin de saccharose. Mélanger doucement le saccharose et le surnageant restant avec une pointe de P1000 coupée. Ne pas vortexer à cette étape ; Sinon, le procentrioles peut se perdre à ce stade. Mettre en commun tous les coussins de saccharose et stockez-les sur la glace.
- Préparer un dégradé de 40 % - 70 % de sucrose dans un tube en polypropylène à paroi mince 38,5 mL en ajoutant doucement 3 mL de 70 % de saccharose (à 4 ° C), suivie de 3 mL de 50 % et enfin, 3 mL, 40 % de sucrose. Charger les interfaces regroupées sur le gradient de 40 % - 70 % de saccharose ; faire lentement parce que le saccharose froid est très visqueux. Équilibrer les tubes avec le tampon de 10 mM K-PIPES (pH 7,2) et les centrifuger à 68 320 g (par exemple, avec un rotor SW32Ti) pendant 1 h et 15 min à 4 ° C.
- Recueillir des fractions de 12 x 500 µL à 4 ° C en faisant un trou dans le fond du tube avec une aiguille de 0. 8 mm sans perturber les couches différentes de saccharose. Avec une coupe P200 de pipette, préparer supplémentaires 10 µL d’extraits de chaque fraction qui sera utilisé pour l’immunofluorescence suivant. Clin d’oeil-les fractions congélation dans l’azote liquide.
NOTE : Centrioles isolés peuvent être analysés par microscopie électronique pour s’assurer que l’ultrastructure globale des centrioles est préservée.
3. quantification des Centrioles isolés sur des lamelles couvre-objet : Centrifugation et Immunofluorescence
Remarque : Voir la Figure 2.
-
Préparation des tubes et lamelles couvre-objet
- Utiliser 1 tube fond rond en verre (15 mL) par fraction à analyser les fractions centriolaire (12 tubes au total).
- Mettre un adaptateur de prise en charge personnalisée lamelle couvre-objet (appelé adaptateur ci-après) dans le tube à fond rond. Placez un lamelle couvre-objet stérile 12 mm dans le tube à fond rond.
NOTE : Ici nous avons utilisé 12 mm lamelles couvre-objet, mais le protocole peut être adapté pour 18 mm lamelles couvre-objet à l’aide de tubes à fond rond 30 ml. - Ajouter 5 ml d’un refroidissement préalable 10 mM K-PIPES (pH 7,2) à 4 ° C. S’assurer que le couvre-objet n’est pas flottant et reste en bas du bloc. Placer les tubes sur la glace.
-
Centrifugation des centrioles
- Diluer chaque fraction de 10 µL à 100 µL de froid 10 mM K-PIPES (pH 7,2). Resuspendre la dilution bien jusqu'à la disparition complète de la saccharose (Figure 2A). Chargez chaque fraction diluée dans un tube à fond rond.
- Tourner le tube à 10 000 x g pendant 10 min (par exemple, avec un rotor de seau oscillant JS-13,1) à 4 ° C.
- Récupérer la lamelle couvre-objet en insérant un dispositif crocheté à la main dans le trou présent à l’extrémité fendue de l’adaptateur et soulevez-le doucement.
Remarque : L’appareil crocheté à la main peut être fait avec aiguille de seringue manuellement accroché et moulé sur une clé maison (Figures 2 b-2D). - En arrivant à l’extrémité du tube fond rond, piéger au bord de l’adaptateur avec un doigt ganté et enlever le couvre-objet avec des pincettes. Veillez à ne se souvenir de quel côté de la lamelle contient les centrioles. Continuer à traiter les lamelles couvre-objet pour l’immunofluorescence.
-
Immunofluorescence souillant et imagerie d’isolé c. reinhardtii centrioles
Remarque : Voir la Figure 2.- Préparer le matériel pour l’immunofluorescence souillant comme suit.
- Préparer une grille coloration de verre dans une boîte de laboratoire de transmission en polystyrène cristal (60 mm de longueur par 50 mm de largeur de 43 mm de hauteur). Remplissez-le avec 100 % de méthanol et conserver à-20 ° C.
- Préparer une chambre humide. Pour ce faire, monter la chambre humide en plaçant un tissu humidifié à l’eau aux côtés de l’intérieur les bords d’un carré de Pétri (Figure 2E). Ajouter un morceau de laboratoire, enveloppe d’étanchéité (voir Table des matières) jusqu’au milieu de la boîte de Pétri sur lequel les mélanges d’anticorps seront placés pendant la procédure d’immunofluorescence (mesures 3.3.2–3.3.3). Couvrir le couvercle et la chambre humide avec du papier d’aluminium pour protéger de la lumière.
- Réagissent les centrioles isolés comme suit.
- Fixer les lamelles avec les centrioles directement après que la centrifugation (étape 3.2.4) en les incubant pendant 5 min dans la zone remplie de méthanol de-20 ° C (étape 3.3.1.1).
- Enlever le couvre-objet avec la pince à épiler et place dans une boîte transparente (voir Table des matières) contenant 50 mL de PBS 1 x et les laver pendant 5 min à température ambiante.
- Distribuer 60 µL du mélange de l’anticorps primaire [anticorps primaires dilués à 1 % d’albumine sérique bovine (BSA) et 0,05 % Tween-20 dans du PBS] sur le morceau de laboratoire envelopper dans la chambre humide d’étanchéité. Posez avec précaution les lamelles sur le dessus le mélange d’anticorps avec les centrioles directement face à la baisse. Incuber les lamelles pendant 45 min avec l’anticorps primaires.
Remarque : Les anticorps primaires qui ont servi à produire des résultats représentatifs sont lapin polyclonal Bld12 (1 : 300) et de la souris α-tubuline (DM1A) (1 : 300). - Retirer les lamelles et les laver pendant 5 min dans du PBS 1 x, tel que décrit à l’étape 3.3.2.2. Incuber les lamelles pendant 45 min avec des anticorps secondaires dans du PBS contenant 1 % de BSA et 0,05 % Tween-20.
Remarque : Les anticorps secondaires qui ont servi à produire des résultats représentatifs sont chèvre anti-souris couplé à Alexa 488 (1:1, 000) et anti-lapin de chèvre couplé à Alexa 568 (1:1, 000). - Retirer les lamelles et les laver pendant 5 min dans du PBS 1 x, tel que décrit à l’étape 3.3.2.2.
- Monter les lamelles sur une lame de verre en ajoutant 3 µL de milieu de montage sur la lame et placer soigneusement les lamelles sur le dessus (centrioles vers le milieu de montage). Scellez le bord de la lamelle couvre-objet avec vernis à ongles.
- Les centrioles isolés sur un microscope confocal à un 63 X objectif d’huile avec un N.A. de 1.4 d’image tout en appliquant une déconvolution27 (voir Table des matières).
NOTE : Ici, les paramètres suivants ont été utilisés : 500 – 545 nm pour Alexa 488 et 580 – 635 nm pour Alexa 568.
- Préparer le matériel pour l’immunofluorescence souillant comme suit.
4. la concentration des Centrioles vers le centre les lamelles
Remarque : Voir la Figure 3.
-
Préparation du matériel
- Préparer un tube fond rond en verre 15 mL sur la glace, un adaptateur de prise en charge personnalisée lamelle couvre-objet (appelé adaptateur ci-après, fichier .stl fourni comme supplémentaire 1 fichier), un concentrateur personnalisé (fichier .stl fourni à titre complémentaire fichier 2) et 10 mM K-PIPES (pH 7.2) à 4 ° C.
- Préparation de poly-D-lysine (PDL)-enduit des lamelles couvre-objet. Diluer 10 x la solution mère de PDL 1 mg/mL avec H2O. Tout d’abord, lavez les lamelles couvre-objet avec 70 % EtOH, enlever l’éthanol et laissez que sécher les lamelles couvre-objet. Enrober les lamelles couvre-objet avec PDL et les incuber 30 min à température ambiante. Laver les lamelles 3 x avec de l’eau et laissez-les sécher.
NOTE : Enrober les lamelles couvre-objet avec PDL d’augmenter le nombre des centrioles isolés attaché à des lamelles couvre-objet.
-
Centrifugation
- Placez un lamelle couvre-objet stérile 12 mm sur le retrait, -bas du concentrateur, gardant la PDL manteau face vers le bas. Cap la lamelle couvre-objet en plaçant l’adaptateur directement sur le dessus. Renverser le tube fond rond et placez-le sur le concentrateur, lamelle couvre-objet et adaptateur.
- Doucement pousser l’ensemble avec des pincettes jusqu'à ce qu’il atteigne le fond du tube fond rond, puis retournez le tube. Ajouter un tampon 10 mM K-PIPES (pH 7,2) au tube fond rond jusqu'à ce qu’il s’agit de la partie supérieure du concentrateur. Assurez-vous qu’il ne restent pas les bulles dans le cylindre central du concentrateur.
- Doucement ajouter 100 µL de tampon de 10 mM K-PIPES (pH 7,2) une partie aliquote contenant la fraction enrichie centriole et mélanger soigneusement le volume.
- Retirer le centre creux du concentrateur de 100 µL de tampon de K-tuyaux de 10 mM (pH 7,2) et ajouter 100 µL de la fraction enrichie de centriole dans un tampon 10 mM K-PIPES (pH 7,2) au centre creux du concentrateur, en prenant soin que le contenu reste dans le centre creux.
- Centrifuger à 10 000 g pendant 10 min (par exemple, avec un rotor de seau oscillant JS-13,1) dans une centrifugeuse préalablement refroidie à 4 ° C.
- Retirer le concentrateur avec des pincettes.
- Récupérer la lamelle couvre-objet en insérant un dispositif crocheté à la main dans le trou présent à l’extrémité fendue de l’adaptateur et soulevez-le doucement. En arrivant à l’extrémité du tube fond rond, piéger au bord de l’adaptateur avec un doigt ganté et enlever le couvre-objet avec des pincettes. Veillez à ne se souvenir de quel côté de la lamelle contiennent des centrioles. Continuer à traiter les lamelles couvre-objet pour l’immunofluorescence.
Remarque : L’appareil crocheté à la main peut être fait avec une aiguille de seringue manuellement accroché et moulé sur une clé maison (Figures 2 b-D). - Effectuer la fixation et l’immunofluorescence souillant des centrioles concentrés comme fait dans l’étape 3.3.2–3.3.4.
5. particule en moyenne
Remarque : Voir la Figure 4.
- Imagerie pour la moyenne de la particule
- Monter la lamelle couvre-objet sur une diapositive à l’aide d’un support régulier montage anti-décoloration. Effectuer l’imagerie de microscopie (SIM 2D) illumination structurée à l’aide d’un objectif de FCI Apochromat FRBR (100 X, NA 1,49, WD 0,12 mm) et un rétro éclairé caméra CCD EM.
Remarque : Le temps d’acquisition a été réglé à 100 ms à une caméra lire sur 3 MHz. Une lentille X 2,5 a été utilisée pour l’imagerie de la SIM.
Remarque : L’ensemble de données présentée ici a été acquis sur un microscope 3D de la carte SIM (voir Table des matières). - Image les centrioles en acquérant une grande pile d’images comprenant le centriole totale du signal, en mettant en place la position haut et en bas de la pile-Z ci-dessus et en dessous le signal centriole, respectivement. Procéder à la pile du projet et effectuer la particule en moyenne selon les étapes 5.2 et 5.3.
- Monter la lamelle couvre-objet sur une diapositive à l’aide d’un support régulier montage anti-décoloration. Effectuer l’imagerie de microscopie (SIM 2D) illumination structurée à l’aide d’un objectif de FCI Apochromat FRBR (100 X, NA 1,49, WD 0,12 mm) et un rétro éclairé caméra CCD EM.
- Projection d’une pile
- Ouvrez la pile d’image avec ImageJ. Cliquez ensuite sur 'Image → Stacks → projet Z'. Définissez le type de projection commeIntensité Max'.
- Inverser l’image en cliquant sur 'édition → inverser'. Sauver la projection généré (format .tif).
- Alignement de la particule avec Scipion
- Créez un nouveau projet dans Scipion en appuyant sur le rouge "Créer un projet de' bouton en haut de la page. Sur le panneau gauche, double-cliquez sur ''desimportations → importer des micrographies.
- Remplir le 'répertoire Files' et 'Pattern' champs selon le répertoire de données et les noms. Conservez les paramètres par défaut. Cliquez sur « Exécuter ».
- Double-cliquez sur « Particules → cueillette → xmipp3 - manuel de cueillette (étape 1) ». Cliquez sur l’icône de loupe située à proximité du champ « Input micrographies » et sélectionnez les micrographies importés dans étape 5.3.1. Cliquez sur « Exécuter ».
- Dans la fenêtre nouvellement ouverte, sélectionnez des particules dans les micrographies différents en cliquant dessus. Lorsque vous avez terminé avec chaque micrograph, cliquez sur le bouton rouge « + coordonnées ».
- Double-cliquez sur « Particules → extrait → xmipp3 - extrait de particules ». Cliquez sur l’icône de loupe située à proximité du champ « Input coordonnées » et sélectionnez le point de coordonnées cueillies dans étape 5.3.4. Remplissez la « Granulométrie boîte (px) » selon les dimensions des particules. Dans l’onglet « Prétraiter » , définissez le dépoussiérage : aucun (il peut générer des artefacts) ; Inverser le contraste : aucun (fond de particules noires, blanches) ; Phase de retournement : No (liée à la correction de la FCT) ; Normaliser : Oui.
- Cliquez ensuite sur « Exécuter ». Lorsque le travail est terminé, vérifier les particules extraites en cochant la case de l’emploi (les frontières devenus plus épais) et cliquez sur "Analyze results' (bas à gauche du menu principal).
- Double-cliquez sur « 2D → aligner → xmipp3 - aligner avec cl2d ». Cliquez sur l’icône de loupe située à proximité du champ « Input particules » et sélectionnez les particules extraites de l’étape 5.3.5. Ne pas utiliser une image de référence. Cliquez sur « Exécuter ».
- Double-cliquez sur ' 2D → aligner → plus → xmipp3 - appliquer alignement 2d'. Cliquez sur l’icône de loupe située à proximité du champ « Input particules » et sélectionnez les particules alignées dans étape 5.3.7.
- Cliquez sur « Exécuter » comme à l’étape 5.3.6. Les résultats peuvent être vérifiés en cliquant sur « Analyze résultats » après la sélection de la zone de travail.
- Double-cliquez sur « Particules → masque → xmipp3 - appliquer le masque 2d ».
- Cliquez sur l’icône de loupe située à proximité du champ « Input particules » et sélectionnez les particules alignées dans étape 5.3.9. La « Source de masque » est réglée sur « Géométrie ». Ensuite, définissez les paramètres du masque comme type de masque : circulaire ; Rayon (px) : à la recherche d’une particule (un centriole) sans rien autour d’elle, cliquez sur le 'baguette magique ' icône à gauche qui ouvre une fenêtre pour aider à trouver la valeur idéale ; Centre de MAJ : No (si la particule est parfaitement centrée) ou Oui (si la particule est déplacée) ; Décalage X-Centre : selon la position de la particule ; Décalage Y-Centre : selon la position de la particule. Cliquez sur « Exécuter ».
- Utilisez le bouton « analyser les résultats » pour vérifier si le masque est appliqué correctement. Si ce n’est pas le cas, faites un clic droit sur le tas « Appliquer le masque 2d » et sélectionnez « Modifier ». Modifier les paramètres du masque (taille et/ou déplacements) et exécuter à nouveau avec « Exécuter » bouton.
- Double-cliquez sur « 2D → classer → xmipp3 - cl2d ».
- Cliquez sur l’icône de loupe située à proximité du champ « Input particules » et sélectionnez les particules masqués dans étape 5.3.11. Le nombre de classes doit être d’obtenir environ 50 particules par classe. Cliquez sur « Exécuter ».
- Vérifier les résultats en cliquant sur le bouton « analyser les résultats » . Dans la fenêtre ouverte, cliquez sur l’icône « œil » juste à côté de « Ce qu’il faut voir l’établissement ».
- La nouvelle fenêtre permet l’inspection des classes générées en sélectionnant « Classes2D » dans le menu « En bloc » . Vérifiez le contenu de chaque catégorie en sélectionnant « Class00N_Particles » dans le même menu. Inspecter chaque classe afin d’identifier ceux qui contiennent des particules seuls les mauvais. Revenir à l’affichage de « Classes2D » , puis sélectionnez les classes avec des particules bons en cliquant sur chacun. Il est possible de sélectionner plusieurs classes en gardant la touche « Ctrl » enfoncée pendant la sélection.
- Quand toutes les classes avec des particules bons ont été sélectionnés, cliquez sur « + particules » pour créer un sous-ensemble de ces particules.
- Dans la même fenêtre, sélectionnez quelques ensembles de classes qui représentent les différentes orientations des particules. Créer un nouveau sous-ensemble en cliquant sur « + moyennes ».
- Pour chaque orientation/moyenne sélectionnée, procédez comme suit.
- Double-cliquez sur « 2D → aligner → xmipp3 - aligner avec cl2d ».
- Cliquez sur l’icône de loupe située à proximité du champ « Input particules » et sélectionnez les particules bonnes étape 5.3.17. Régler « Utiliser une Image de référence » à « Oui ». Cliquez sur l’icône de loupe située à proximité du champ « Image de référence » et cliquer sur la gauche de la flèche blanche pour le travail de « Créer un sous-ensemble » de l’étape 5.3.18 et sélectionner l’image à utiliser comme une référence. Double-cliquez sur un objet pour l’ouvrir dans une fenêtre séparée et vérifiez de quel objet il est. Cliquez sur « Exécuter ».
- Double-cliquez sur ' 2D → aligner → plus → xmipp3 - appliquer alignement 2d'. Cliquez sur l’icône de loupe située à proximité du champ « Input particules » et sélectionnez les particules alignés de l’étape 5.3.19.2.
- Cliquez sur « Exécuter ».
- Vérifier le résultat de l’alignement («Analyse des résultats) ; il vous montrera les particules alignés et la moyenne de ces particules.
- Si la moyenne est bonne et les particules sont tous orientés de la même manière, enregistrez la moyenne en cliquant sur ' Advanced → ImageJ' et enregistrer l’image avec ImageJ.
- Si la moyenne peut être améliorée, sélectionnez toutes les images bien orientées et créer un sous-ensemble de nouveau avec le bouton « + particules » . Réitérer les étapes 5.3.19.1–5.3.19.4 jusqu'à ce que le sous-ensemble est nettoyé (tous les mauvaises particules enlevées). Chaque fois que l’alignement est effectué (opération 5.3.19.2), la référence se trouve à la dernière moyenne générée (d’itération d’itération, les augmentations de qualité moyenne).
Résultats
C. reinhardtii Centriole isolement :
Pour isoler les centrioles, mission AC 15- c. reinhardtii cellules ont été cultivés dans le liquide pendant plusieurs jours sous la lumière et granulés par la suite par centrifugation à 600 g pour 10 min. même cellules sont lavées 1 x avec du PBS et resuspendues dans un deflagellation avant le tampon deflagellation en effectuant un choc pH moyen de 0,5 M d’acide acétique à un pH final de 4.5 – 4,7 pendant 2 min (Figure 1A). Un ajout de 1 N KOH servait à rétablir le pH à 7.0. Pour séparer des flagelles détachés du corps cellulaires, cellules deflagellated ont été tout d’abord centrifugés pour éliminer la majeure partie des flagelles. Le culot a ensuite été lavé 2 x avec PBS et resuspendues dans 30 mL de PBS avant le chargement il lentement sur un coussin de 25 % du saccharose (Figure 1B). Le tube a été filé à 600 x g pendant 15 min à 4 ° C pour enlever la plupart des flagelles détachés. Après centrifugation, le corps des cellules ont été répartie dans le coussin de saccharose (Figure 1C) et récupéré en supprimant les environ 30 mL du surnageant (jusqu'à la flèche rouge dans la Figure 1C). La résultante de 20 mL de cellules lavées étaient alors resuspendues dans 20 mL de PBS froids et centrifugé à 600 x g pendant 10 min. Ensuite, le surnageant a été écarté, et les cellules ont été complètement remises en suspension dans 10 mL de PBS. Les cellules sont transférées à une bouteille de 250 mL et le tampon de lyse a été ajouté aux cellules remises en suspension à la fois. DNase a été ajouté à la lyse et incubé pendant 1 h à 4 ° C. Après une étape de centrifugation pour enlever les débris de la cellule (voir la pastille blanche dans la Figure 1D), le surnageant a été recueilli et soigneusement chargé sur un coussin de 60 % du saccharose 2 mL avant la centrifugation à 10 000 x g pendant 30 min à 4 ° C. Notez que pour 100 mL de lyse, 8 tubes de 15 mL ont été utilisés pour effectuer cette étape, correspondant à 12,5 mL de tampon de lyse chargé sur 2 mL de saccharose par tube. Après centrifugation, la majeure partie du liquide surnageant a été supprimé à 1 mL au-dessus du coussin. Le 1 mL du surnageant restant était alors recueillie avec le coussin de 2 mL et puis mélangé et mis en commun afin d’obtenir un volume final de 24 mL. La piscine a été ensuite chargée sur un 40 %, 50 %, 70 %-saccharose dégradé et filé à 68 320 x g pendant 1 h et 15 min à 4 ° C. Enfin, les centrioles isolés ont été recueillis en faisant un trou dans la partie inférieure du tube de centrifugation à l’aide d’une aiguille et les gouttes ont été prélevés dans les fractions de 12 x 500 µL. En raison de la forte densité de saccharose différente, la forme goutte très lentement au début (70 % de saccharose), puis plus rapidement (40 % de saccharose).
Immunofluorescence des Centrioles isolés
Pour évaluer la qualité de la technique d’isolation, 10 µL de chaque fraction de gradient recueillie a été ensuite centrifugé sur un lamelle couvre-objet à l’aide d’un adaptateur de prise en charge de lamelle couvre-objet (Figure 2 a-2D). Ce qui est important, pour retirer en toute sécurité la lamelle, un dispositif accroché personnalisé a été conçu (Figure 2B). Ensuite, les lamelles ont été analysés par immunofluorescence. Anticorps contre CrSAS-6(Bld12p) ont été utilisés dans cette étude pour indiquer la présence de la structure de la roue et les α-tubuline (DMA1) pour mettre en surbrillance le mur centriole. En comptant le nombre de centrioles qui se sont révélés positifs pour les CrSAS-6 et α-tubuline par champ de vue et puis en calculant le nombre total de centrioles par fractions, il a été possible de déterminer les fractions ont été enrichies pour isolé centrioles ( Figure 2F-2 H). Fait intéressant, 6 fractions ont été enrichies des centrioles (Figure 2F et 2 G, fractions #1 – 6), avec un pic pour fraction #3, tandis que les fractions derniers n’étaient pas, ce qui indique que la purification a travaillé. Notez que dans cette expérience particulière, 95 % des centrioles totales étaient positifs pour CrSAS-6 et α-tubuline en fraction #3. Cela signifie que plus isolées centrioles conservé leur roue. Si aucun enrichissement de centrioles n’est observée dans les premières fractions, la technique d’isolation ne fonctionnait pas et doit être répétée. Notez que quelques morceaux de flagelles peut être observées pour la plupart dans les fractions dépourvues de centrioles.
Ensuite, pour calculer le nombre total de centrioles par µL, le nombre de centriole par champ de vision doit être multiplié par le rapport tel que présenté dans la Figure 2H. Le nombre obtenu doit ensuite être divisé par le volume de la fraction utilisée pour l’immunofluorescence pour obtenir le nombre de centrioles par µL. Dans cette procédure particulière d’isolation, la fraction plus enrichie contient environ 37 centrioles pour une superficie de 0,00846 mm2 (avec un champ de vision de 92 x 92 µm2). La surface de la section du tube était 7,5 mm de rayon avec une surface totale de 176 mm2. Le ratio correspondant était alors 176/0.00846 = 20,803.8, donc un total de 769 740 centrioles (37 x 20, 803.8) à 10 µL. Par conséquent, le nombre de centrioles dans 1 µL était de 76 974.
Concentration des Centrioles isolés sur des lamelles couvre-objet :
La multiplication des centrioles par champ augmente les chances de détecter l’orientation centriole non ambiguë, mais aussi augmenter les chances de détecter des orientations similaires qui peuvent être utilisées pour les procédures d’autres particules de moyenne. Centrioles de fractions concentrées sont encore rares sur la lamelle couvre-objet, nous avons développé un accessoire de centrifugation pour concentrer les centrioles au milieu de la lamelle couvre-objet (Figure 3A) nommé un concentrateur. Notez qu’un fichier .stl avec les mesures précises pour l’impression 3D est fourni avec ce manuscrit.
Tout d’abord, une lamelle de 12 mm est monté sur le concentrateur (Figure 3B). L’adaptateur a été placé sur le dessus de la lamelle couvre-objet et le tube fond rond a été inversé et placé sur l’adaptateur assemblé et du concentrateur. Le tube fond rond a été ensuite doucement inversé, ce qui permet le chargement de l’échantillon (protocole étape 4.2). Les centrioles sont ensuite centrifugés à 10 000 x g pendant 10 min à 4 ° C. Après cela, les centrioles ont été soumis à l’immunofluorescence et colorés pour CrSAS-6/Bld12p et α-tubuline (Figure 3-3E). Ce qui est important, sans concentrateur, centrioles environ 30 ont été vus par le champ de vision (Figure 3C), tandis que les 183 centrioles ont été vus par le champ de vision lorsque le concentrateur a été utilisé (Figure 3D-3F). Notez que les centrioles couvert seulement un disque de 4 mm de diamètre au milieu de la lamelle. Ce résultat montre que l’étape de concentration fonctionne et permet un enrichissement 6 fois de centrioles dans une région donnée les lamelles, ainsi, faciliter leur détection et imagerie.
Particule fluorescente avec une moyenne d’isolé C. reinhardtii Centrioles :
Ici, en utilisant la microscopie SIM pouvant atteindre une résolution d’environ 120 nm, centrioles colorées pour monoglutamylated tubuline (GT335, Figure 4), une modification de la tubuline présente dans les microtubules centriolaire, étaient imagés24. C. reinhardtii centrioles sont environ 500 nm de long, toujours par paires et souvent trouvé avec associés, nouvellement dupliqué probasal organes (ci-après comme procentrioles) et strié de fibres associées aux microtubules19. Par conséquent, cet assemblage final était grande environ 1 µm. Pour cette raison et pour l’image les centrioles dans leur intégralité, nous vous recommandons d’acquérir une Z-pile sur les centrioles isolés.
Ici, après l’acquisition, une image finale a été générée en effectuant une projection d’intensité maximale à l’aide de ImageJ28 (projection intensité de l’image/Stacks/Z projet/max, Figure 4B). De telles images, une analyse de la particule à l’aide de logiciels de microscopie de cryo-électron a été réalisée pour créer des classes de centrioles ayant une orientation similaire, et ensuite en moyenne a été réalisée. Pour ce faire, la couleur de l’image a été tout d’abord inversée afin de mieux visualiser les objets (Figure 4C). Centrioles sont recueillis manuellement dans une boîte centrée sur chaque particule en utilisant le librement disponibles de logiciel Scipion29 qui intègre plusieurs logiciels de microscopie électronique tels que Xmipp3 (Figure 4D). Notez que la taille de la boîte doit être défini par l’utilisateur. Ici, les boîtes de 50 x 50 pixels pour une taille de pixel de 31.84 nm ont été utilisés. Ensuite, toutes les particules ont été extraites (Figure 4E) et alignés à l’aide de Xmipp3 (Figure 4F). Ensuite, un masque circulaire de 12 pixels de rayon a été appliqué pour isoler chaque centriole dans la centriole-paire (Figure 4G). Les particules ont ensuite été classées, à l’aide de Xmipp3, pour générer plusieurs moyennes. Seulement les moyennes de classe homogènes ont été détenus, ce qui signifie que les particules qui s’écartent de la classe moyenne étaient exclues manuellement. Cette étape est répétée afin de générer une moyenne de presque parfait pour chaque orientation choisie. Après cinq itérations, deux classes de moyennes ont été générés : une vue de dessus de 63 objets (Figure 4H) et un côté Découvre des 98 particules (Figure 4j’ai) des centrioles monoglutamylated. Les dimensions de l’objet ont été déterminées en mesurant la distance entre les pics du profil terrain intensité le long du signal de monoglutamylation.
Ce qui est important, la longueur de la moyenne de la classe de vue latérale est 260 nm avec un diamètre de 250 nm, comparable au signal de tubuline monoglutamylated mesurée qui a été montré à localiser dans une région de 286 ± 33 nm de longueur dans le noyau du centriole17.
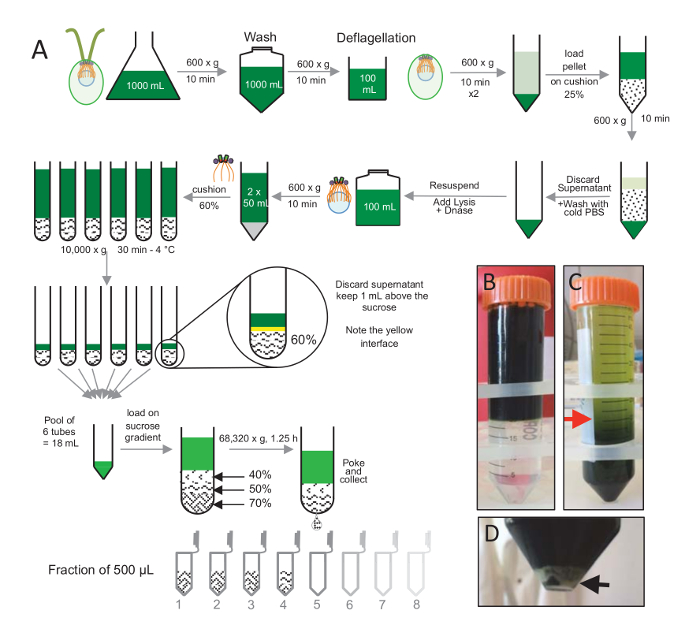
Figure 1 : Purification de C. reinhardtii centrioles. (A) il s’agit d’une représentation schématique de chacune des étapes menant à l’isolement de c. reinhardtii centrioles. Il comprend des mesures représentatives du protocole (B) avant et (C) après centrifugation sur la pente de 25 % du saccharose. La flèche rouge dans panneau C indique le volume minimal de garder après centrifugation. (D), ce panneau affiche la pastille blanche de cellules lysées après centrifugation. La flèche noire indique le culot. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
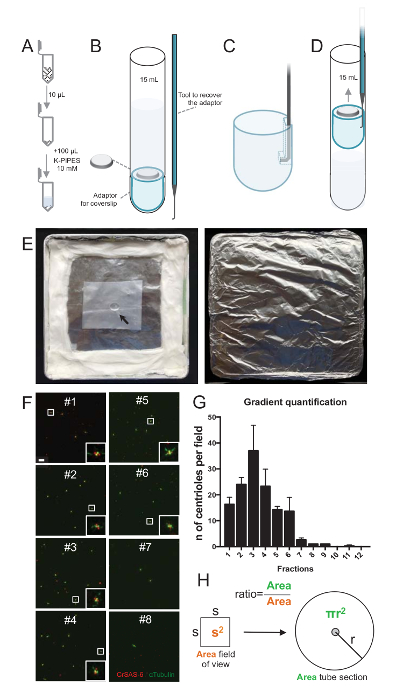
Figure 2 : Mise en place de centrifugation pour effectuer l’immunofluorescence sur isolé centrioles. (A), 10 µL de chacune des fractions collectées est tout d’abord dilué dans 100 µL de 10 mM K-PIPES (pH 7,2). (B) il s’agit d’une représentation schématique des dispositifs centrifugation, comprenant un tube de 15 mL fond rond, une lamelle de 12 mm, un adaptateur pour la lamelle couvre-objet et un dispositif accroché sur mesure pour récupérer la lamelle après centrifugation. Séries C et D afficher les dessins expliquant comment faire pour récupérer la lamelle après centrifugation. (C) placer l’outil dans le trou présent à l’extrémité fendue de l’adaptateur et (D) Tirez doucement. (E) Voici les photos de la chambre humide nécessaire pour réaliser l’imagerie immunofluorescence. La flèche indique la lamelle de 12 mm. (F), ce sont des images confocales représentant 63 X (zoom 2) fractions gradients #1-8, recueillies au cours de la procédure de purification et colorés pour CrSAS-6 (rouge) et α-tubuline (vert). Les EISN correspondent à la région indiquée par une boîte blanche dans le point de vue plus faible grossissement. Echelle = 10 µm. (G) il s’agit d’un graphique représentant le nombre de centrioles positives pour CrSAS-6 et α-tubuline par champ de vision dans chaque fraction. Notez l’enrichissement en fractions #1-6. Le nombre moyen de centrioles par champ dans chaque fraction : #1 = 16,3 ± 4,7, #2 = 24,0 ± 4,6, #3 = 37,0 ± 17,0, #4 = 23,3 ± 11,4, #5 = 14,3 ± 2.1, #6 = 13,7 ± 9.3, #7 = 2,7 ± 1.2, #8 = 1,0 ± 0,0, #9 = 1,0 ± 0,0, #10 = ± 0.0 0.0, #11 = ± 0,3 0,6, #12 = ± 0.0 0.0. Pour chaque fraction, 3 champs aléatoires ont été projetés. Notez que, lorsque vous comptez la fraction plus enrichie #3, nous avons constaté que 95 % des centrioles sont positifs pour CrSAS-6 et α-tubuline (n = 205 centrioles). (H), A rapport entre le nombre des centrioles présents dans la zone mesurée de la microscopie est utilisé pour calculer le nombre total de centrioles présents dans la zone du tube. Le nombre de centriole par µL peut être calculé de 10 fractions µL initialement centrifugé. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
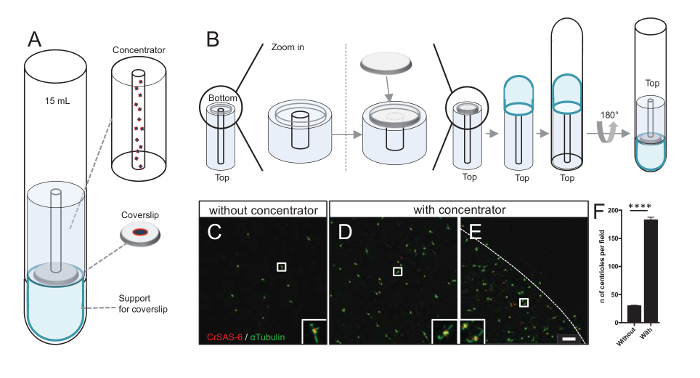
Figure 3 : Isolés centrioles sont centrifugés à l’aide de concentrateurs. (A), ce panneau affiche les périphériques de centrifugation pour concentrer les centrioles sur lamelles avant immunofluorescence, y compris un tube de 15 mL fond rond, un concentrateur, une lamelle de 12 mm et un appareil d’adaptateur de prise en charge. (B), ce panneau affiche les étapes pour assembler l’appareil de centrifugation. Panneaux C-E montrent des images confocales de centrioles colorées pour CrSAS-6 (rouge) et α-tubuline (vert) (C) sans ouD-Eavec le concentrateur. Les EISN correspondent à la région indiquée par une boîte blanche dans le point de vue plus faible grossissement. La barre d’échelle est de 10 µm. Note que les centrioles sont enrichis dans le milieu de la lamelle couvre-objet (la ligne pointillée représente la bordure de la zone concentrée). (F), ce graphique représente le nombre de centrioles par champ de vision sans et avec le concentrateur. Cinq champs aléatoires de vue ont été analysés. Le nombre moyen de centrioles est, sans concentrateur, 29.8 ± 2,9 et avec le concentrateur, 182,6 ± 11,5, P < 0,0001. La signification statistique a été évaluée par un non-appariés t-test. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
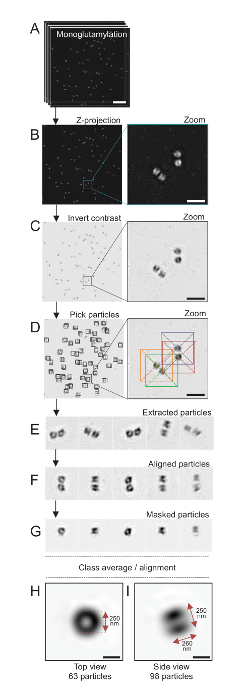
Figure 4 : Particule en moyenne isolé C. reinhardtii centrioles. (A), ce panneau montre Z empiler les images des centrioles colorées avec GT335 et acquis à l’aide d’un microscope SIM. (B) ces panneaux montrent une projection de Z d’intensité maximale de l’images superposées. Echelle = 1 µm. (C), ces panneaux montrent une image représentative offrant un contraste inversé. L’encart représente un zoom-in pour visualiser les centrioles mieux. (D), ces panneaux Voir la particule cueillette. L’encart indique comment les particules ont été cueillis. (E) ce sont des exemples de 5 extrait des particules. (F), ce panneau montre les particules après mise en conformité. (G), ce panneau montre les particules après avoir appliqué un masque. Panneaux H et j’ai montrent deux moyennes de classe : (H), un top view (63 particules) etj’aiune vue de côté (98 particules). Les doubles flèches indiquent les dimensions du signal GT335. Echelle = 250 nm. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Fichier complémentaire 1. S’il vous plaît cliquez ici pour télécharger ce fichier.
Fichier supplémentaire 2. S’il vous plaît cliquez ici pour télécharger ce fichier.
Discussion
Un des défis en biologie est de déchiffrer la localisation précise des protéines dans un contexte architectural. Le centriole est une structure idéale pour appliquer ces méthodes, car son architecture a été étudiée en utilisant la tomographie cryo-électronique, révélant des caractéristiques ultrastructurales intéressantes sur toute sa longueur. Toutefois, en raison de ses dimensions proches de la limite de résolution en microscopie optique, il est difficile de localiser avec précision une protéine par immunofluorescence à une sous-région structurelle de la centriole à l’aide de microscopes classiques30.
La résolution en microscopie optique est limitée par la diffraction de la lumière qui donne, en gros, une résolution latérale maximale de 200 nm en microscopie optique24. Toutefois, cette limite a été contournée par l’un des progrès majeurs de ces 20 dernières années en microscopie optique : l’invention des méthodes Super-résolution. Ces approches peuvent l’image au-delà des limites de diffraction à différentes résolutions : 120 nm pour SIM, environ 50 nm pour l’épuisement de l’émission stimulée (STED) et 20 à 40 nm pour la localisation de la molécule unique microscopie (SMLM)24. Avec ces nouveaux développements de la microscopie de Super-résolution, les sous-régions structurelles de la centriole sont réalisables. Cependant, en pratique, il est encore difficile de déterminer avec précision la localisation d’une protéine à un élément structurel pour la raison principale que les centrioles matures existent seulement 2 copies par cellule et ont des orientations aléatoires, ce qui rend l’interprétation des localisation difficile. Pour cette raison, un protocole a été mis en place qui permet aux chercheurs d’image par Super-résolution un grand nombre de centrioles, augmentant la chance d’observer des orientations non ambiguë. Ce qui est important, car cette méthode repose sur l’utilisation des centrioles isolés, nous fournissons une méthode pour purifier intact c. reinhardtii centrioles qui contiennent des centrioles matures et procentrioles.
Enfin, en raison de la diversité des orientations de centriole qui peut être photographiée avec ce protocole, analyse de la particule peut être appliqué à l’aide de logiciels de microscopie électronique. Cela se traduit par la génération des classes moyennes de centrioles dans une orientation particulière. Ce qui est important, ces images 2D résultantes puis permet d’évaluer la localisation d’une protéine spécifique le long du centriole. En effet, cette méthode peut être appliquée aux images de Super-résolution bicolores, et une couleur peut être utilisée pour révéler le squelette de centriole (p. ex., tubuline), tandis que l’autre couleur peut être attribué à une protéine spécifique du centriole. En soustrayant les moyennes obtenues avec une seule couleur ou deux couleurs, il devient plus facile d’enregistrer une protéine le long le centriole (centrale, proximale ou distale). Notez que les deux canaux devraient être alignés avec précision afin d’éviter toute interprétation trompeuse. En outre, les moyennes des vues haut aidera déchiffrer si une protéine se localise à l’intérieur de la lumière centriolaire, le long du mur de microtubules, ou à l’extérieur le centriole.
Cette méthode a l’avantage d’établir la localisation des protéines spécifiques qui pourraient être difficiles à localiser par ailleurs dû aux inscriptions hétérogènes. Notez qu’autres méthodes pour cartographier les protéines à l’intérieur les centrioles ont été décrites dans corrélatif SIM/SMLM 3D avec, par exemple, évaluer les orientations spécifiques des centrioles en déterminant le profil elliptique d’un marqueur formant un tore autour de la centriole par imagerie de SIM. Vous utilisez ce paramètre, il est possible de localiser des protéines avec une précision de 4 à 5 nm30. Notez également que la méthode décrite ici utilise centrioles isolés avec procentrioles intacte, un signe que l’architecture de centriole est probablement en grande partie conservée. Toutefois, nous ne pouvons exclure que certains éléments architecturaux sont dérangés pendant la purification, tels que le diamètre de centriole varie avec la concentration des cations divalents comme amplifié avec l’isolement du centrosome humain5.
Une des étapes critiques du protocole présenté ici est d’obtenir des centrioles isolés suffisamment concentrées dans des orientations différentes se prête au Fluo-SPA. Pour ce faire, tout d’abord assurer la pureté et l’efficacité de la technique d’isolation centriole. Une faible concentration de centrioles isolés n’empêchera imagerie appropriée et autre traitement de l’image. À cette fin, nous fournissons une méthode afin d’enrichir le nombre des centrioles par champ de vision. Selon le nombre de centrioles dans la fraction utilisée, le volume chargé dans le concentrateur doit être ajusté, avec un volume maximal de 250 µL.
Ce qui est important, cette méthode a été développée pour une paroi cellulaire moins mission AC 15-C. reinhardtii cellules. Cette souche, la fragilité de la paroi cellulaire permet une bonne lyse des cellules et, par conséquent, la libération de son contenu. Ce protocole n’est pas efficace pour sauvage c. reinhardtii cellules, car la paroi empêche une lyse adéquate. Des stratégies alternatives comme la sonication ou une pré-incubation des cellules avec autolysine, une enzyme qui peut dégrader la paroi cellulaire31, devra être mis en place pour modifier la paroi cellulaire avant d’appliquer le protocole présenté ici.
Cette configuration peut être utilisée avec différents types de microscopes, allant de microscopes confocaux classiques à haut débit dédié Super-résolution microscopes. Notez que lorsque vous faites des SMLM, un tampon spécial est requis pour l’imagerie appropriée et, par conséquent, une chambre adaptée pour une lamelle de 12 mm avec le tampon sur le dessus de la lamelle doit être utilisée. L’imagerie ultérieur est effectué avec des microscopes inversés. Si la mise en place de microscope ne permet pas une lamelle de 12 mm, le protocole présenté ici peut être appliqué à 18 mm lamelles couvre-objet à l’aide d’un tube de 30 mL fond rond et un adaptateur modifié et concentrateur. Il est également important de remarque que la qualité de la reconstruction finale en SMLM dépendra de la qualité de la coloration et de l’anticorps primaire utilisé, ainsi que la méthode de fixation.
En résumé, nous fournissons une méthode qui peut être appliquée à l’image de nombreux centrioles suivis par Fluo-SPA qui va générer des moyennes des centrioles dans des orientations différentes, ce qui permet de localiser des protéines centriolaire avec précision. Ce qui est important, cette méthode peut être appliquée plus généralement aux centrioles isolés des autres espèces, d’autres organites ou à gros assemblages macromoléculaires. Enfin, la démarche de préparation d’échantillon présenté ici, combiné avec le récent développement d’algorithmes pour l’analyse de la particule de fluorescent SMLM données32, pourrait ouvrir davantage amélioration dans la cartographie moléculaire du grand macromoléculaire assemblys.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous remercions Pierre Gönczy BioImaging et plate-forme optique (BIOP) à l’École Polytechnique Fédérale de Lausanne (EPFL), Lausanne (Suisse), où les images SIM des centrioles ont été acquis. Nikolai Klena et Davide Gambarotto sont pris en charge par le Conseil européen de la recherche (CER) Starting Grant (StG) 715289 (ACCENT) et Maeva Le Guennec, Paul Guichard et Virginie Hamel par la PP00P3_157517 de Swiss National (FNS). Susanne Borgers est pris en charge par l’Université de Genève.
matériels
| Name | Company | Catalog Number | Comments |
| Mouse monoclonal anti-apha tubulin (clone DM1A) | Abcam | ab7291 | dilution 1:300 |
| DNaseI | Roche | 10104159001 | |
| 12 mm coverslips | Roth | YX03.1 | |
| 18 mm coverslips | Roth | LH23.1 | |
| K2HPO4 | Fluka | 60355 | |
| KH2PO4 | Fluka | 60230 | |
| Tris base | Biosolve Chimie SARL | 0020092391BS | |
| acetic acid | Carlo Erba Reagents | 524520 | |
| NH4Cl | Sigma | A-4514 | |
| CaCl2 | Sigma | C-7902 | |
| MgSO4 | Sigma | 63140-500G-F | |
| steritop filter | Millipore | SCGPT05RE | |
| sucrose | Sigma | S7903-1KG | |
| HEPES | AppliChem PanReac | A3724,0250 | |
| PIPES | Sigma | P6757-500G | |
| MgCl2 | ACROS ORGANICS | 197530010 | |
| NP-40 | AppliChem PanReac | A1694,0250 | |
| Round-bottom (Kimble) tubes 15 mL | Fisherscientific | 09-500-34 | |
| Round-bottom (Kimble) tubes 30 mL | Fisherscientific | 09-500-37 | |
| cover glass staining rack | Thomas scientific | 8542E40 | |
| crystal polystyrene transmission lab box | FISHERS | 11712944 | |
| Methanol | VWR | 20864.32 | |
| BSA | Roche | 10735086001 | |
| Triton X100 | Roth | 3051.3 | |
| goat anti-mouse coupled to Alexa 488 | invitrogen | A11029 | dilution 1:1,000 |
| goat anti-rabbit coupled to Alexa 568 | invitrogen | A11036 | dilution 1:1,000 |
| mounting medium | abcam | ab188804 | |
| Tube, thinwall polypropylene | Beckman Coulter | 326823 | |
| Poly-D-Lysine 1 mg/mL | SIGMA | A-003-E | |
| Mouse monoclonal anti-Polyglutamylation modification mAb (GT335) | Adipogen | AG-20B-0020 | dilution 1:1,000 |
| glycerol mounting medium with DAPI and DABCO | Abcam | ab188804 | |
| 50 mL conical tubes | Falcon | 14-432-22 | |
| Eppendorf 5810R centrifuge | Eppendorf | 5811000622 | |
| Beckman JS-13.1 swinging bucket rotor | Beckman Coulter | 346963 | |
| Beckman SW 32Ti rotor | Beckman Coulter | 369694 | |
| parafilm | Bemis | 13-374-10 | |
| Leica TCS SP8 with Hyvolution mode | Leica | ||
| OSRAM L18W/954 LUMILUX | Luxe Daylight/ OSRAM | ||
| Whatman filter paper | Sigma | WHA1001325 | |
| CrSAS-6/Bld12 antibody | dilution 1:300 (Hamel el al., 2014) | ||
| Scipion | http://scipion.i2pc.es/ | ||
| EM CCD camera (Andor iXON DU897) | Andor |
Références
- Bornens, M. The centrosome in cells and organisms. Science. 335 (6067), 422-426 (2012).
- Nigg, E. A., Holland, A. J. Once and only once: mechanisms of centriole duplication and their deregulation in disease. Nature Reviews Molecular Cell Biology. 19 (5), 297-312 (2018).
- Nigg, E. A., Raff, J. W. Centrioles, centrosomes, and cilia in health and disease. Cell. 139 (4), 663-678 (2009).
- Gönczy, P. Towards a molecular architecture of centriole assembly. Nature Reviews Molecular Cell Biology. 13 (7), 425-435 (2012).
- Paintrand, M., Moudjou, M., Delacroix, H., Bornens, M. Centrosome organization and centriole architecture: their sensitivity to divalent cations. Journal of Structural Biology. 108 (2), 107-128 (1992).
- Winey, M., O'Toole, E. Centriole structure. Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 369 (1650), (2014).
- Dippell, R. V. The development of basal bodies in paramecium. Proceedings of the National Academy of Sciences of the United States of America. 61 (2), 461 (1968).
- Allen, R. D. The morphogenesis of basal bodies and accessory structures of the cortex of the ciliated protozoan Tetrahymena pyriformis. The Journal of Cell Biology. 40 (3), 716-733 (1969).
- Li, S., Fernandez, J. -. J., Marshall, W. F., Agard, D. A. Three-dimensional structure of basal body triplet revealed by electron cryo-tomography. The EMBO Journal. 31 (3), 552-562 (2012).
- Guichard, P., Chrétien, D., Marco, S., Tassin, A. -. M. Procentriole assembly revealed by cryo-electron tomography. The EMBO Journal. 29 (9), 1565-1572 (2010).
- Guichard, P., et al. Native architecture of the centriole proximal region reveals features underlying its 9-fold radial symmetry. Current Biology. 23 (17), 1620-1628 (2013).
- Hirono, M. Cartwheel assembly. Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 369 (1650), (2014).
- Bornens, M., Paintrand, M., Berges, J., Marty, M. C., Karsenti, E. Structural and chemical characterization of isolated centrosomes. Cell Motility and the Cytoskeleton. 8 (3), 238-249 (1987).
- Keller, L. C., Romijn, E. P., Zamora, I., Yates, J. R., Marshall, W. F. Proteomic analysis of isolated chlamydomonas centrioles reveals orthologs of ciliary-disease genes. Current Biology. 15 (12), 1090-1098 (2005).
- Kilburn, C. L., et al. New Tetrahymena basal body protein components identify basal body domain structure. The Journal of Cell Biology. 178 (6), 905-912 (2007).
- Bauer, M., Cubizolles, F., Schmidt, A., Nigg, E. A Quantitative analysis of human centrosome architecture by targeted proteomics and fluorescence imaging. The EMBO Journal. 35 (19), 1-15 (2016).
- Hamel, V., et al. Identification of Chlamydomonas Central Core Centriolar Proteins Reveals a Role for Human WDR90 in Ciliogenesis. Current Biology. 27 (16), 2486-2498 (2017).
- Cavalier-Smith, T. basal body and flagellar development during the vegetative cell cycle and the sexual cycle of Chlamydomonas reinhardii. Journal of Cell Science. 16 (3), 529 (1974).
- Geimer, S., Melkonian, M. The ultrastructure of the Chlamydomonas reinhardtii basal apparatus: identification of an early marker of radial asymmetry inherent in the basal body. Journal of Cell Science. 117, 2663-2674 (2004).
- Nakazawa, Y., Hiraki, M., Kamiya, R., Hirono, M. SAS-6 is a cartwheel protein that establishes the 9-fold symmetry of the centriole. Current Biology. 17 (24), 2169-2174 (2007).
- Hamel, V., et al. Correlative multicolor 3D SIM and STORM microscopy. Biomedical Optics Express. 5 (10), 3326 (2014).
- Guichard, P., et al. Cell-free reconstitution reveals centriole cartwheel assembly mechanisms. Nature Communications. 8, 14813 (2017).
- Hiraki, M., Nakazawa, Y., Kamiya, R., Hirono, M. Bld10p constitutes the cartwheel-spoke tip and stabilizes the 9-fold symmetry of the centriole. Current Biology. 17 (20), 1778-1783 (2007).
- Sahl, S. J., Hell, S. W., Jakobs, S. Fluorescence nanoscopy in cell biology. Nature Reviews Molecular Cell Biology. 18 (11), 685-701 (2017).
- Hutner, S. H., Provasoli, L., Schatz, A., Haskins, C. P. Some Approaches to the Study of the Role of Metals in the Metabolism of Microorganisms. Proceedings of the American Philosophical Society. 94 (2), 152-170 (1950).
- Keller, L. C., Marshall, W. F. Isolation and proteomic analysis of chlamydomonas centrioles. Methods in Molecular Biology. 432 (1), 289-300 (2008).
- Borlinghaus, R. T., Kappel, C. HyVolution-the smart path to confocal super-resolution. Nature Methods. 13 (3), (2016).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nature Methods. 9 (7), 671-675 (2012).
- de la Rosa-Trevín, J. M., et al. Scipion: A software framework toward integration, reproducibility and validation in 3D electron microscopy. Journal of Structural Biology. 195 (1), 93-99 (2016).
- Gartenmann, L., et al. A combined 3D-SIM/SMLM approach allows centriole proteins to be localized with a precision of ∼4-5 nm. Current Biology. 27 (19), 1054-1055 (2017).
- Jaenicke, L., Kuhne, W., Spessert, R., Wahle, U., Waffenschmidt, S. Cell-wall lytic enzymes (autolysins) of Chlamydomonas reinhardtii are (hydroxy)proline-specific proteases. European Journal of Biochemistry. 170 (1-2), 485-491 (1987).
- Salas, D., et al. Angular reconstitution-based 3D reconstructions of nanomolecular structures from superresolution light-microscopy images. Proceedings of the National Academy of Sciences of the United States of America. 114 (35), 9273-9278 (2017).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon