Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
SA--Galactosidase-basierter Screening-Test zur Identifizierung von Senotherapeutischen Arzneimitteln
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Zelluläre Seneszenz ist der Schlüsselfaktor bei der Entwicklung chronischer altersbedingter Pathologien. Die Identifizierung von Therapeutika, die auf seneszente Zellen abzielen, verspricht, eine gesunde Alterung zu verlängern. Hier präsentieren wir einen neuartigen Test zur Identifizierung von Senotherapeutika auf der Grundlage der Messung der seneszenzassoziierten Galactosidase-Aktivität in Einzelzellen.
Zusammenfassung
Zellseneszenz ist eines der Kennzeichen des Alterns bekannt, um eine gesunde Lebensdauer negativ beeinflussen. Medikamente, die in der Lage sind, seneszente Zellen speziell in der Zellkultur zu töten, als Sennolytika bezeichnet, können die seneszente Zellbelastung in vivo reduzieren und die Gesundheitsspanne verlängern. Mehrere Klassen von Senolytika wurden bisher identifiziert, einschließlich HSP90-Inhibitoren, Bcl-2-Familieninhibitoren, Piperlongumin, einem FOXO4-hemmenden Peptid und der Kombination von Dasatinib/Quercetin. Der Nachweis von SA-A-Gal bei einem erhöhten lysosomalen pH-Wert ist einer der am besten charakterisierten Marker für den Nachweis von seneszenten Zellen. Live-Zellmessungen der seneszenzassoziierten Aktivität von Seneszenz-assoziierten B-Galactosidase (SA---Gal) mit dem fluoreszierenden Substrat C12FDG in Kombination mit der Bestimmung der Gesamtzellzahl mittels eines DNA-interkalierenden Hoechst-Farbstoffs eröffnet Bildschirm auf senotherapeutische Medikamente, die entweder die gesamte SA-A-Gal-Aktivität durch Tötung von seneszenten Zellen (Senestik) reduzieren oder sA--Gal und andere Phänotypen von seneszenten Zellen (Senosozien) unterdrücken. Die Verwendung einer fluoreszierenden Bildaufnahme- und Analyseplattform mit hohem Gehalt ermöglicht das schnelle Screening von Medikamentenbibliotheken mit hohem Durchsatz auf Effekte auf SA--Gal, Zellmorphologie und Zellzahl.
Einleitung
Die Zellseneszenz wurde zum ersten Mal von Leonard Hayflick und Paul Moorhead beschrieben, die zeigten, dass normale Zellen eine begrenzte Fähigkeit hatten, sich in Kultur1zu vermehren. Seneszenzzellen vermehren sich trotz des Vorhandenseins von Nährstoffen, Wachstumsfaktoren und mangelnder Kontakthemmung nicht, bleiben aber metabolisch aktiv2. Dieses Phänomen ist als replizierende Seneszenz bekannt und wurde hauptsächlich auf die Telomerverkürzung zurückgeführt, zumindest in menschlichen Zellen3. Weitere Studien haben gezeigt, dass Zellen auch als Reaktion auf andere Reize, wie onkogenen Stress (Onkogeninduzierte Seneszenz, OIS), DNA-Schäden, zytotoxische Medikamente oder Bestrahlung (stressinduzierte Seneszenz, SIS) induziert werden können. , 5 , 6. Als Reaktion auf DNA-Schäden, einschließlich Telomererosion, seneszen die Zellen entweder, beginnen unkontrolliertes Zellwachstum oder erleiden Apoptose, wenn der Schaden nicht repariert werden kann. In diesem Fall scheint die Zellseneszenz vorteilhaft zu sein, da sie tumorsuppressiv wirkt2. Im Gegensatz dazu wird die Seneszenz mit dem Altern aufgrund der Anhäufung von Zellschäden einschließlich DNA-Schäden erhöht. Da seneszente Zellen Zytokine, Metalloproteinasen und Wachstumsfaktoren, die als seneszenzassoziierter sekretomlischer Phänotyp (SASP) bezeichnet werden, sezernieren können, trägt diese altersabhängige Zunahme der zellulären Seneszenz und SASP zu einer verminderten Gewebehomöostase und später altern. Auch ist diese altersabhängige Erhöhung der Seneszenzbelastung bekannt, um Stoffwechselerkrankungen, Stressempfindlichkeit, Progerie-Syndrome und beeinträchtigte Heilung7,8 zu induzieren und ist zum Teil verantwortlich für die zahlreichen altersbedingten Krankheiten wie Arteriosklerose, Arthrose, Muskeldegeneration, Geschwürbildung und Alzheimer-Krankheit9,10,11,12,13. Die Beseitigung seneszierender Zellen kann helfen, Gewebefunktionsstörungen zu verhindern oder zu verzögern und die Gesundheitsspanne14zu verlängern. Dies wurde in transgenen Mausmodellen14,15,16 sowie durch die Verwendung von senolytischen Medikamenten und Arzneimittelkombinationen gezeigt, die sowohl durch Arzneimittelscreenings als auch durch bioinformatische Analyse von Wege, die speziell in seneszierenden Zellen induziert werden17,18,19,20,21,22. Die Identifizierung optimaler senotherapeutischer Medikamente, die in der Lage sind, die seneszente Zellbelastung effektiver zu reduzieren, ist ein wichtiger nächster Schritt bei der Entwicklung von therapeutischen Ansätzen für ein gesundes Altern.
Seneszenzzellen weisen charakteristische phänotypische und molekulare Merkmale auf, sowohl in der Kultur als auch in vivo. Diese Seneszenzmarker können entweder die Ursache oder das Ergebnis der Seneszenzinduktion oder ein Nebenprodukt molekularer Veränderungen in diesen Zellen sein. Es wird jedoch kein einzelner Marker speziell in seneszierenden Zellen gefunden. Derzeit ist der Seneszenz-assoziierte Nachweis von Seneszenz-assoziierten '-Galactosidase (SA---Gal) eine der am besten charakterisierten und etablierten einzelzellbasierten Methoden zur Messung der Seneszenz in vitro und in vivo. SA-A-Gal ist eine lysosomale Hydrolase mit einer optimalen enzymatischen Aktivität bei pH 4. Die Messung der Aktivität am pH 6 ist möglich, da seneszentiver Zellen eine erhöhte lysosomale Aktivität zeigen23,24. Bei lebenden Zellen wird eine erhöhte lysosomale pH-Wert-Taste durch lysosomale Alkalinisierung mit dem Vacuolar H+-ATPase-Inhibitor Bafilomycin A1 oder dem endosomalen Versauerungsinhibitor Chloroquin25,26. 5-Dodecanoylaminofluorescein Di--D-Galactopyranosid (C12FDG) wird als Substrat in lebenden Zellen verwendet, da es das geballte Produkt in den Zellen aufgrund seiner 12 Kohlenstoff lipophilen Moiety25behält. Wichtig ist, dass die SA---Gal-Aktivität selbst nicht direkt mit einem in seneszenten Zellen identifizierten Pfad verbunden ist und nicht notwendig ist, um Seneszenz zu induzieren. Mit diesem Test können seneszente Zellen auch in den heterogenen Zellpopulationen und alternden Geweben identifiziert werden, wie Hautbiopsien von älteren Menschen. Es wurde auch verwendet, um eine Korrelation zwischen Zellseneszenz und Alterung23 zu zeigen, da es ein zuverlässiger Marker für seneszente Zelldetektion in mehreren Organismen und Bedingungenist 27,28,29, 30. Hier wird ein sA--Gal-Screening-Assay mit hohem Durchsatz auf Basis des fluoreszierenden Substrats C12FDG mit primären embryonalen Fibroblasten (MEFs) mit robust oxidativer Stressinduzierter Zellseneszenz beschrieben und seine Vor- und Nachteile werden diskutiert. Obwohl dieser Test mit verschiedenen Zelltypen durchgeführt werden kann, ermöglicht die Verwendung von Ercc1-mangelhaften, DNA-Reparatur beeinträchtigten MEFs eine schnellere Induktion der Seneszenz unter Bedingungen von oxidativem Stress. Bei Mäusen verursacht eine reduzierte Expression der DNA-Reparatur-Endonuklease ERCC1-XPF eine beeinträchtigte DNA-Reparatur, beschleunigte Anhäufung von endogenen DNA-Schäden, erhöhte ROS, mitochondriale Dysfunktion, erhöhte seneszente Zellbelastung, Verlust der Stammzellfunktion und vorzeitige Alterung, ähnlich wie natürliche Alterung31,32. In ähnlicher Weise werden Ercc1-mangelhafte MEFs in Kultur17schneller sensithenz. Ein wichtiges Merkmal des seneszenten MEF-Assays ist, dass jeder Brunnen eine Mischung aus seneszenten und nicht-seneszenten Zellen hat, was eine klare Demonstration von seneszenten zellspezifischen Wirkungen ermöglicht. Obwohl wir glauben, dass die Verwendung von oxidativem Stress in Primärzellen, um Seneszenz zu induzieren, eher physiologisch ist, kann dieser Assay auch mit Zelllinien verwendet werden, bei denen Seneszenz mit DNA-schädlichen Mitteln wie Etoposid oder Bestrahlung induziert wird.
Protokoll
Die Verwendung von Tieren wurde vom Scripps Florida Institutional Animal Care and Use Committee genehmigt.
1. Erzeugung von seneszierendem murinen embryonalen Fibroblasten (MEF) – 12-15 Tage
- Isolieren Sie wilden Typ und Ercc1-/- MEFs von trächtigen weiblichen Mäusen am embryonalen Tag 13 (E13) wie zuvor beschrieben33.
HINWEIS: Alle folgenden Schritte werden in einer Gewebekulturhaube unter aseptischen Bedingungen und mit sterilen Instrumenten durchgeführt. - Resect den Embryokopf über den Augen.
- Entfernen Sie das rote Gewebe (Herz und Leber) und verwenden Sie es für die Genotypisierung, falls erforderlich.
- Bereiten Sie 500 ml einer 1:1-Mischung aus Dulbecco es Modified Eagle es Medium (DMEM) und Ham es F10 mit 10% fetalem Rinderserum, 1x nicht essentiellen Aminosäuren, Penicillin und Streptomycin als Wachstumsmedium vor und erwärmen Sie es vor jedem Gebrauch bis zu 37 °C. Wachstumsmedium bei 4 °C lagern.
- Den Rest des Embryos mit 0,25% Trypsin/EDTA für 10 min inkubieren.
- Den Embryo in 1 mm Stücke zerkleinern und das Gewebe mehrmals nach oben und unten pipette.
- Fügen Sie 10 ml Wachstumsmedium und Plattengewebe eines Embryos pro 10 cm Durchmesser Zellkulturplatte (Passage 0) hinzu.
- Kultivieren Sie Zellen bei 37°C, 3% O2 , 5% CO2 .
HINWEIS: Unter diesen Bedingungen befestigen sich nur MEF-Zellen an nicht beschichteten Gewebekulturplatten. - Ändern Sie das Medium jeden Tag in Durchgang 0, um nicht angeschlossenes Gewebe und Zellfragmente zu entfernen.
HINWEIS: Abhängig von der Größe des Embryos und der Qualität der Isolierung erreichen die Zellen in der Regel nach 2 bis 3 Tagen die Konfluenz. - Trypsinisierung
- Entfernen Sie das Wachstumsmedium vorsichtig und waschen Sie Zellen mit 10 ml 1x PBS zweimal.
- 2 ml einer 0,025% Trypsin/EDTA-Lösung in Platten mit 10 cm Durchmesser hinzufügen und bei 37 °C für 2-3 min inkubieren.
- Stellen Sie sicher, dass zellenlos von der Oberfläche gelöst sind, indem Sie die Zellen unter dem Mikroskop untersuchen.
- Beenden Sie die Trypsin-Verdauung, indem Sie die gleiche Menge an Wachstumsmedium hinzufügen.
- Übertragen Sie die Zellen bei 200 x g für 3 min in ein konisches Rohr und Zentrifugenzellen und entsorgen Sie den Überstand.
- Zellen sorgfältig in frischem Wachstumsmedium aussetzen, Zellen zählen und in neuen Platten bei der projizierten Zelldichte aussaieren.
- Für nicht-seneszierende Subkultivierung spalten konfluente Zellen 1:4 und erstrecken sich für einen weiteren Durchgang bei 3%O2, 37 °C, um mehr Zellen zu ergeben (Durchgang 1).
HINWEIS: An dieser Stelle können die Zellen entweder in Kultur gehalten oder für die spätere Verwendung in flüssigem Stickstoff gespeichert werden, in Kryovials mit jeweils etwa 1 Million Zellen. Dieser Schritt bietet auch die Möglichkeit, eine gemischte Charge von Zellen von verschiedenen Tieren zu erzeugen, um die Variabilität zu reduzieren, die aus der Einzeltieranalyse stammt. - Um die Zellseneszenz zu induzieren, spaltet der Samen die konfluenten Zellen ausDurchgang 1 im Verhältnis 1:4 und inkubiert sie bei 20% O2, 37 °C, 5% CO2 für 3 Tage; diese Kulturbedingungen sind atmosphärisch für Sauerstoff.
HINWEIS: Die Kultivierung von Zellen und Blastozysten unter DenerUmgebungssauerstoffkonzentrationen kann Marker der zellulären Seneszenz erhöhen, insbesondere wenn die Reparatur von DNA-Schäden beeinträchtigt ist34,35,36. - Wiederholen Sie diesen Vorgang für 2 weitere Passagen.
- Um die zelluläre Seneszenz zu überwachen, messen Sie die allmähliche Zunahme des Zelldurchmessers und des Zellvolumens während jedes Trypsinisierungsschritts mit einem fortschrittlichen Coulter-Zellzählersystem.
- Bewerten Sie die Verringerung der Zellproliferation durch Bestimmung der Populationsverdoppelung (PD) mit der Gleichung
PDT = (t2-t1)/3,32 x (log n2 – log n1)
HINWEIS: Die Verdoppelungszeit der Bevölkerung wurde nur für nicht-seneszierende Zellen verwendet. - Verwenden Sie frühe Durchgangwildtyp oder Ercc1-/- MEF-Zellen, die bei 3% O2, 37 °C, 5% CO2 als nicht-seneszente Kontrollzellen gehalten wurden.
2. Senescent assoziierter Screening-Test – 2-3 Tage
- Bereiten Sie 10 mM Lagerlösungen in DMSO aller zu testenden Medikamente vor und lagern Sie Aliquots bei -80 °C. Einfrieren-Tau-Aktienlösungen, da dies die Aktivität von Medikamenten verringern kann.
HINWEIS: Hier wurde der HSP90-Hemmer 17DMAG als senolytisches Medikament eingesetzt, das in der Lage ist, seneszente Zellen gezielt abzutöten17. - Tauen Sie am Tag des Experiments das Aliquot, verdünnen Sie die Medikamente in einem frischen Kulturmedium und fügen Sie den Zellen, die konditioniertes Medium enthalten, im Verhältnis 1:1 hinzu, um die Endkonzentration im Wachstumsmedium zu ergeben.
- Verwenden Sie 96-Well-Vorverdünnungsplatten für serielle Verdünnungen und Arzneimittelkombinationen.
HINWEIS: Bei MEF-Zellen wurde empirisch festgestellt, dass die DMSO-Konzentrationen 2 % nicht überschreiten sollten und Kontrollzellen, die mit den höchsten DMSO-Konzentrationen behandelt wurden, sollten in jeden Durchlauf einbezogen werden. - Samen 5 x 103 Seneszenzzellen oder 3 x 103 nicht-seneszente Zellen pro Brunnen in 96 Brunnenplatten mindestens 6 h vor der Behandlung in 100 l Wachstumsmedium und inkubieren bei 20% O2 , 37 °C, 5% CO2 .
HINWEIS: Zellen sollten vor der Behandlung etwa 80 % konfluent sein. - Verwenden Sie schwarze Wand /klare Bodengewebekultur, behandelt 96 Brunnenplatten, um fluoreszierende Signal-Crosstalk und Hintergrund zu minimieren.
HINWEIS: Klare Platten wurden jedoch auch erfolgreich getestet. - Fügen Sie den MEF-Zellen Arzneimittelverdünnungen hinzu und brüten Sie 24 h bis 48 h unter 20%O2, 37 °C, 5% CO2-Bedingungen.
- Halten Sie nicht-seneszenzfreieZellen unter 3% O2 , 37 °C, 5% CO2-Bedingungen.
- Für die lysosomale Alkalinisierung eine 10 mM Bafilomycin A1 Lösung vorbereiten, aliquotisieren und bei -20 °C einfrieren lassen.
- Für die Fluoreszenzanalyse der SA-A-Gal-Aktivität eine 2 mM C12FDG-Lagerlösung vorbereiten, bei -20 °C lagern und vor Licht schützen.
- Für die Arbeitslösung am Experimenttag 100 m C12FDG im Wachstumsmedium vorbereiten.
HINWEIS: Alle (Inkubations-)Schritte mit C12FDG sollten im Dunkeln durchgeführt werden. - Entfernen Sie die Arzneimittellösung und waschen Sie die Zellen 1 Mal mit 100 l 1x PBS.
- Induzieren Sie die lysosomale Alkalinisierung durch Vorbehandlung von Zellen mit 90 l einer 100 nM Bafilomycin A1-Lösung, die in einem Frischzellkulturmedium für 1 Stunde bei 20%O2, 37 °C, 5% CO2 hergestellt wird.
- Fügen Sie dem Kulturmedium 10 l von 100 'M C12FDG-Arbeitslösung hinzu (Endkonzentration 10 'M).
- Inkubieren Sie Zellen für 2 h.
- Fügen Sie der Kultur 2 l eines Hoechst 33342 Farbstoffs (Endkonzentration 2 g/ml) hinzu und brüten 20 min.
- Entfernen Sie die Medien, und fügen Sie 100 L frisches Wachstumsmedium hinzu.
3. Quantitative fluoreszierende Bildanalyse mit hohem Gehalt
- Verwenden Sie eine fluoreszierende Bildaufnahme- und Analyseplattform mit hohem Anteil, um fluoreszierende Bilder der Zellen in den beiden Kanälen zu erfassen, die für die Erfassung von Hoechst- und C12FDG-Fluoreszenz (z. B. DAPI- bzw. FITC-Kanalvoreinstellungen) geeignet sind.
HINWEIS: Erfassungsprotokolle erfordern die Definition mehrerer Variablen, die für den Test spezifisch sind. Ziel eines Erfassungsprotokolls ist es, eine ausreichende Anzahl von in-Fokus-Fluoreszenzbildern einer ausreichenden Anzahl von Zellen für die nachgelagerte quantitative Analyse zu erfassen. - Entwickeln Sie ein geeignetes Analyseprotokoll, indem Sie jeden Kanal auswählen und durch Anpassen eines oder mehrerer selektiver Kriterien definieren, was für jeden Kanal als interessanter Merkmal gilt.
- Verwenden Sie für den Kern so Segmentierungsvorsätze (z. B. Kernsegmentierung, Vesikelsegmentierung, Zytoplasmasegmentierung), die die Identifizierung von Zellorganellen auf der Grundlage mehrerer Kriterien wie Morphologie, Größe und Signal ermöglichen. intensität. Passen Sie diese Kriterien so an, dass Kerne eingeschlossen werden, während nukleare Fragmente und Trümmer ausgeschlossen werden, die zwar ein Signal haben, aber beispielsweise zu groß oder zu klein sind, um Kerne zu sein.
- Überprüfen Sie das Signal im FITC-Kanal, das fluoreszenz aus cleaved C12FDG ist und die Menge der seneszenzassoziierten Aktivität in den Zellen darstellt.
ANMERKUNG: Seneszenzzellen haben eine höhere seneszenzassoziierte Aktivität von 'Galactosidase als nicht-seneszierende Zellen; C12FDG-Fluoreszenz wird jedoch nicht diskret und kontinuierlich sein, was die Festlegung eines Schwellenwerts zwischen dem, was als C12FDG-positiv angesehen wird, und einer C12FDG-negativen Zelle erforderlich macht. - Erzeugen Sie mit kommerziell erhältlicher Analysesoftware eine Anzahl von Instanzen, in denen sich eine definierte Region um den Kern (eine mutmaßliche Zelle) mindestens einmal mit einem überschwelligen C12FDG überlappt hat.
HINWEIS: Die Analysesoftware generiert automatisch eine Zählung mithilfe der Zielverknüpfung. Dies ist die praktische Definition einer seneszenten, C12FDG-positiven Zelle.
- Analysieren Sie alle Stichproben in dreifacher Ausfertigung mit 3-5 Feldern pro Brunnen und Mittelwerten und Standardabweichungen werden entsprechend berechnet.
- Berechnen Sie den Prozentsatz der seneszierenden Zellen mit der folgenden Formel:
Seneszierende Zellen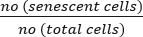 (%) = x 100
(%) = x 100
4. Assay-Validierungsparameter
- Für alle Stichproben wurden der Intra- und Inter-Assay-Variationskoeffizient (%CVs) nach folgender Formel berechnet:
Intra-Assay CV (n = 10 Wiederholungen in einem Experiment gemessen) = x 100 (%)
in einem Experiment gemessen) = x 100 (%)
Inter-Assay CV (n = 5 unabhängige Experimente) = x 100 (%)
unabhängige Experimente) = x 100 (%) - Bestimmen Sie zu Screening-Zwecken den Z-Wert, einen statistischen Parameter zur Bewertung der Qualität eines Assays, aus Zellen, diemit 200 nM Rapamycin für 24 h bei 20% O2, 37 °C, 5% CO2 (eine positive Kontrolle für senotherapeutische Arzneimittel) und unbehandelt behandelt wurden. seneszierenden Zellen (negative Kontrolle).
HINWEIS: Der Wert des Z wurde nach Zhang et al.37berechnet. Z'-Werte zwischen 0,5 und 1 deuten darauf hin, dass ein Assay für das Drogenscreening verwendet werden kann.
Ergebnisse
Die SA---Gal-Aktivität kann in Zellen nachgewiesen werden, die durch verschiedene Arten von replizierter Erschöpfung, genotoxischem und oxidativem Stress bis hin zur Onkogenaktivierung23,25,38zur Senesz induziert werden. Im aktuellen Modell mit Ercc1-mangelhaften mausembryonalen Fibroblastenzellen reichtennormoxische Wachstumsbedingungen (20% O2 ) aus, um die Zellseneszenz n...
Diskussion
SA-A-Gal ist ein klar definierter Biomarker für zelluläre Seneszenz, der ursprünglich von Dimri et al. entdeckt wurde. (1995) zeigt, dass seneszente menschliche Fibroblasten eine erhöhte Aktivität von SA-A-Gal haben, wenn sie bei pH 623 im Vergleich zu sich ausbreitenden Zellen gemessen werden. Inzwischen wurden In-vitro- und In-vivo-Assay für SA-A-Gal für verschiedene Zelltypen und Gewebe25,39,...
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Diese Arbeit wurde von der NIH Grants AG043376 (Projekt 2 und Core A, PDR; Projekt 1 und Core B, LJN) und AG056278 (Projekt 3 und Kern A, PDR; und Projekt 2, LJN) und ein Stipendium der Glenn Foundation (LJN).
Materialien
| Name | Company | Catalog Number | Comments |
| DMEM | Corning | 10-013-CV | medium |
| Ham's F10 | Gibco | 12390-035 | medium |
| fetal bovine serum | Tissue Culture Biologics | 101 | serum |
| 1x non-essential amino acids | Corning | 25-025-Cl | amino-acids |
| bafilomycin A1 | Sigma | B1793 | lysosomal inhibitor |
| C12FDG | Setareh Biotech | 7188 | b-Gal substrate |
| Hoechst 33342 | Life Technologies | H1399 | DNA intercalation agent |
| 17DMAG | Selleck Chemical LLC | 50843 | HSP90 inhibitor |
| InCell6000 Cell Imaging System | GE Healthcare | High Content Imaging System |
Referenzen
- Hayflick, L., Moorhead, P. S. The serial cultivation of human diploid cell strains. Experimental Cell Research. 25, 585-621 (1961).
- Campisi, J., di Fagagna, F. D. Cellular senescence: when bad things happen to good cells. Nature Reviews Molecular Cell Biology. 8 (9), 729-740 (2007).
- Harley, C. B., Futcher, A. B., Greider, C. W. Telomeres shorten during ageing of human fibroblasts. Nature. 345 (6274), 458-460 (1990).
- Zhu, J., Woods, D., McMahon, M., Bishop, J. M. Senescence of human fibroblasts induced by oncogenic Raf. Genes and Development. 12 (19), 2997-3007 (1998).
- Dimri, G. P., Itahana, K., Acosta, M., Campisi, J. Regulation of a senescence checkpoint response by the E2F1 transcription factor and p14(ARF) tumor suppressor. Molecular and Cellular Biology. 20 (1), 273-285 (2000).
- Michaloglou, C., et al. BRAFE600-associated senescence-like cell cycle arrest of human naevi. Nature. 436 (7051), 720-724 (2005).
- Niedernhofer, L. J. Tissue-specific accelerated aging in nucleotide excision repair deficiency. Mechanisms of Ageing and Development. 129 (78), 408-415 (2008).
- Gitenay, D., et al. Glucose metabolism and hexosamine pathway regulate oncogene-induced senescence. Cell Death & Disease. 5, e1089 (2014).
- Erusalimsky, J. D., Kurz, D. J. Cellular senescence in vivo: its relevance in ageing and cardiovascular disease. Experimental Gerontology. 40 (8-9), 634-642 (2005).
- Kassem, M., Marie, P. J. Senescence-associated intrinsic mechanisms of osteoblast dysfunctions. Aging Cell. 10 (2), 191-197 (2011).
- Campisi, J., Andersen, J. K., Kapahi, P., Melov, S. Cellular senescence: A link between cancer and age-related degenerative disease. Seminars in Cancer Biology. 21 (6), 354-359 (2011).
- Golde, T. E., Miller, V. M. Proteinopathy-induced neuronal senescence: a hypothesis for brain failure in Alzheimer's and other neurodegenerative diseases. Alzheimers Research & Therapy. 1 (2), 5 (2009).
- Telgenhoff, D., Shroot, B. Cellular senescence mechanisms in chronic wound healing. Cell Death & Differentiation. 12 (7), 695-698 (2005).
- Baker, D. J., et al. Clearance of p16Ink4a-positive senescent cells delays ageing-associated disorders. Nature. 479 (7372), 232-236 (2011).
- Baker, D. J., et al. Naturally occurring p16-positive cells shorten healthy lifespan. Nature. , (2016).
- Childs, B. G., et al. Senescent intimal foam cells are deleterious at all stages of atherosclerosis. Science. 354 (6311), 472-477 (2016).
- Fuhrmann-Stroissnigg, H., et al. Identification of HSP90 inhibitors as a novel class of senolytics. Nature Communications. 8 (1), 422 (2017).
- Zhu, Y., et al. New agents that target senescent cells: the flavone, fisetin, and the BCL-XL inhibitors, A1331852 and A1155463. Aging. 9 (3), 955-963 (2017).
- Zhu, Y., et al. Identification of a Novel Senolytic Agent, Navitoclax, Targeting the Bcl-2 Family of Anti-Apoptotic Factors. Aging Cell. , (2015).
- Zhu, Y., et al. The Achilles' heel of senescent cells: from transcriptome to senolytic drugs. Aging Cell. , (2015).
- Baar, M. P., et al. Targeted Apoptosis of Senescent Cells Restores Tissue Homeostasis in Response to Chemotoxicity and Aging. Cell. 169 (1), 132-147 (2017).
- Jeon, O. H., et al. Local clearance of senescent cells attenuates the development of post-traumatic osteoarthritis and creates a pro-regenerative environment. Nature. , (2017).
- Dimri, G. P., et al. A biomarker that identifies senescent human cells in culture and in aging skin in vivo. Proceedings of the National Academy of Sciences USA. 92 (20), 9363-9367 (1995).
- Itahana, K., Campisi, J., Dimri, G. P. Methods to detect biomarkers of cellular senescence: the senescence-associated beta-galactosidase assay. Methods in Molecular Biology. 371, 21-31 (2007).
- Debacq-Chainiaux, F., Erusalimsky, J. D., Campisi, J., Toussaint, O. Protocols to detect senescence-associated beta-galactosidase (SA-[beta]gal) activity, a biomarker of senescent cells in culture and in vivo. Nature Protocols. 4 (12), 1798-1806 (2009).
- Cahu, J., Sola, B. A sensitive method to quantify senescent cancer cells. Journal of Visualized Experiments. 78 (78), (2013).
- Collado, M., et al. Tumour biology: Senescence in premalignant tumours. Nature. 436 (7051), 642 (2005).
- Krishnamurthy, J., et al. Ink4a/Arf expression is a biomarker of aging. The Journal of Clinical Investigation. 114 (9), 1299-1307 (2004).
- Mishima, K., et al. Senescence-associated beta-galactosidase histochemistry for the primate eye. Investigative Ophthalmology, Visual Science. 40 (7), 1590-1593 (1999).
- Melk, A., et al. Expression of p16INK4a and other cell cycle regulator and senescence associated genes in aging human kidney. Kidney International. 65 (2), 510-520 (2004).
- Niedernhofer, L. J., et al. A new progeroid syndrome reveals that genotoxic stress suppresses the somatotroph axis. Nature. 444 (7122), 1038-1043 (2006).
- Wang, J., Clauson, C. L., Robbins, P. D., Niedernhofer, L. J., Wang, Y. The oxidative DNA lesions 8,5′-cyclopurines accumulate with aging in a tissue-specific manner. Aging Cell. 11 (4), 714-716 (2012).
- Jozefczuk, J., Drews, K., Adjaye, J. Preparation of mouse embryonic fibroblast cells suitable for culturing human embryonic and induced pluripotent stem cells. Journal of Visualized Experiments. (64), (2012).
- Jagannathan, L., Cuddapah, S., Costa, M. Oxidative stress under ambient and physiological oxygen tension in tissue culture. Current Pharmacology Reports. 2 (2), 64-72 (2016).
- Meuter, A., et al. Markers of cellular senescence are elevated in murine blastocysts cultured in vitro: molecular consequences of culture in atmospheric oxygen. J Assist Reprod Genet. 31 (10), 1259-1267 (2014).
- Robinson, A. R., et al. Spontaneous DNA damage to the nuclear genome promotes senescence, redox imbalance and aging. Redox Biology. 17, 259-273 (2018).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A Simple Statistical Parameter for Use in Evaluation and Validation of High Throughput Screening Assays. Journal Of Biomolecular Screening. 4 (2), 67-73 (1999).
- Zhao, H., Darzynkiewicz, Z. Biomarkers of cell senescence assessed by imaging cytometry. Methods in Molecular Biology. 965, 83-92 (2013).
- Yang, N. -. C., Hu, M. -. L. A fluorimetric method using fluorescein di-β-d-galactopyranoside for quantifying the senescence-associated β-galactosidase activity in human foreskin fibroblast Hs68 cells. Analytical Biochemistry. 325 (2), 337-343 (2004).
- Zhao, J., et al. Quantitative Analysis of Cellular Senescence in Culture and In Vivo. Current Protocols in Cytometry. 79, (2017).
- Capparelli, C., et al. CDK inhibitors (p16/p19/p21) induce senescence and autophagy in cancer-associated fibroblasts, "fueling" tumor growth via paracrine interactions, without an increase in neo-angiogenesis. Cell Cycle. 11 (19), 3599-3610 (2012).
- Coppe, J. P., Desprez, P. Y., Krtolica, A., Campisi, J. The senescence-associated secretory phenotype: the dark side of tumor suppression. Annual Review of Pathology. 5, 99-118 (2010).
- Mah, L. J., El-Osta, A., Karagiannis, T. C. GammaH2AX as a molecular marker of aging and disease. Epigenetics. 5 (2), 129-136 (2010).
- Hewitt, G., et al. Telomeres are favoured targets of a persistent DNA damage response in ageing and stress-induced senescence. Nature Communications. 3, 708 (2012).
- Narita, M., et al. Rb-mediated heterochromatin formation and silencing of E2F target genes during cellular senescence. Cell. 113 (6), 703-716 (2003).
- Georgakopoulou, E. A., et al. Specific lipofuscin staining as a novel biomarker to detect replicative and stress-induced senescence. A method applicable in cryo-preserved and archival tissues. Aging-Us. 5 (1), 37-50 (2013).
- Severino, J., Allen, R. G., Balin, S., Balin, A., Cristofalo, V. J. Is β-Galactosidase Staining a Marker of Senescence in Vitro and in Vivo?. Experimental Cell Research. 257 (1), 162-171 (2000).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten