Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Essai de dépistage basé sur sA-MD-Galactosidase pour l'identification des médicaments sérothérapeutiques
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
La sénescence cellulaire est le facteur clé dans le développement de pathologies chroniques liées à l'âge. L'identification des thérapies qui ciblent les cellules sénescentes s'avère prometteuse pour prolonger le vieillissement sain. Ici, nous présentons un essai nouveau pour l'identification des sénotherapeutics basés sur la mesure de l'activité associée de sénescence de 'Galactosidase dans les cellules simples.
Résumé
La sénescence cellulaire est l'une des caractéristiques du vieillissement connu pour influencer négativement une durée de vie saine. Les médicaments capables de tuer les cellules sénescentes spécifiquement dans la culture cellulaire, appelées sénolytiques, peuvent réduire le fardeau cellulaire sénescent in vivo et prolonger la durée de vie. Plusieurs classes de sénolytiques ont été identifiées à ce jour, y compris les inhibiteurs du HSP90, les inhibiteurs de la famille Bcl-2, la piperlongumine, un peptide inhibiteur FOXO4 et la combinaison de Dasatinib/Quercetin. La détection de SA-A-Gal à un pH lysosomal accru est l'un des marqueurs les mieux caractérisés pour la détection des cellules sénescentes. Mesures cellulaires vivantes de l'activité de la sénescence associée à la sénescence (SA-MD-Gal) à l'aide du substrat fluorescent C12FDG en combinaison avec la détermination du nombre total de cellules à l'aide d'un colorant Hoechst intercalant l'ADN ouvre la possibilité de dépistage des médicaments sénothérapeutiques qui réduisent l'activité globale de SA-A-Gal en tuant les cellules sénescentes (sénolytiques) ou en supprimant le SA-Gal et d'autres phénotypes des cellules sénescentes (sénomorphiques). L'utilisation d'une plate-forme d'acquisition et d'analyse d'images fluorescentes à haute teneur permet le dépistage rapide et à haut débit des bibliothèques de médicaments pour les effets sur sA-MD-Gal, la morphologie cellulaire et le numéro de cellulaire.
Introduction
La sénescence cellulaire a été décrite pour la première fois par Leonard Hayflick et Paul Moorhead, qui ont montré que les cellules normales avaient une capacité limitée à proliférer dans la culture1. Les cellules sénescentes ne prolifèrent pas malgré la présence de nutriments, les facteurs de croissance et le manque d'inhibition du contact, mais restent métaboliquement actives2. Ce phénomène est connu sous le nom de sénescence réplicative et a été principalement attribué au raccourcissement des télomères, au moins dans les cellules humaines3. D'autres études ont montré que les cellules peuvent également être induites à subir la sénescence en réponse à d'autres stimuli, tels que le stress oncogène (sénescence induite par l'oncogène, OIS), les dommages à l'ADN, les médicaments cytotoxiques, ou l'irradiation (sénescence induite par le stress, SIS)4 , 5 Annonces , 6. En réponse aux dommages à l'ADN, y compris l'érosion des télomères, les cellules séneesce, commencent la croissance cellulaire incontrôlée, ou subissent l'apoptose si les dommages ne peuvent pas être réparés. Dans ce cas, la sénescence cellulaire semble être bénéfique car elle agit d'une manière suppressive de tumeur2. En revanche, la sénescence est augmentée avec le vieillissement dû à l'accumulation des dommages cellulaires comprenant des dommages d'ADN. Puisque les cellules sénescentes peuvent sécrétiser des cytokines, des metalloproteinases et des facteurs de croissance, appelés phénotype sécréteur sénieux séniescence-associés (SASP), cette augmentation âge-dépendante de la sénescence cellulaire et sASP contribue à l'homéostasie diminuée de tissu et par la suite le vieillissement. En outre, cette augmentation dépendante de l'âge de la charge de sénescence est connue pour induire des maladies métaboliques, la sensibilité au stress, les syndromes de progeria, et la guérison altérée7,8 et est, en partie, responsable des nombreux âges liés maladies, telles que l'athérosclérose, l'arthrose, la dégénérescence musculaire, la formation d'ulcères, et la maladie d'Alzheimer9,10,11,12,13. L'élimination des cellules sénescentes peut aider à prévenir ou retarder le dysfonctionnement des tissus et à prolonger l'étendue de la santé14. Cela a été démontré dans les modèles de souris transgéniques14,15,16 ainsi qu'en utilisant des médicaments sénolytiques et des combinaisons de médicaments qui ont été découverts à la fois par les efforts de dépistage des médicaments et l'analyse bioinformatique de voies induites spécifiquement dans les cellules sénescentes17,18,19,20,21,22. L'identification de médicaments sénothérapeutiques plus optimaux, capables de réduire plus efficacement le fardeau cellulaire sénescent, est une prochaine étape importante dans le développement d'approches thérapeutiques pour le vieillissement en santé.
Les cellules sénescentes présentent des caractéristiques phénotypiques et moléculaires, tant en culture qu'in vivo. Ces marqueurs de sénescence pourraient être la cause ou le résultat de l'induction de sénescence ou un sous-produit des changements moléculaires dans ces cellules. Cependant, aucun marqueur unique n'est trouvé spécifiquement dans les cellules sénescentes. À l'heure actuelle, la détection de la sénescence associée à la sénescence est l'une des méthodes à base de cellules individuelles les mieux caractérisées et établies pour mesurer la sénescence in vitro et in vivo. SA-Gal est une hydrolase lysosomale avec une activité enzymatique optimale au pH 4. La mesure de son activité au pH 6 est possible parce que les cellules sénescentes montrent une activité lysosomale accrue23,24. Pour les cellules vivantes, l'augmentation du pH lysosomal est obtenue par alcalinisation lysosomale avec l'inhibiteur vacuolar H -ATPase Bafilomycin A1 ou l'inhibiteur de l'acidification endosomal chloroquine25,26. 5-Dodecanoylaminofluorescein Di-MD-D-galactopyranoside (C12FDG) est utilisé comme substrat dans les cellules vivantes car il conserve le produit clivé dans les cellules en raison de sa moiétologie lipophile de 12 carbone25. Fait important, l'activité de SA--Gal elle-même n'est pas directement liée à une voie identifiée dans les cellules sénescentes et n'est pas nécessaire pour induire la sénescence. Avec cet analyse, les cellules sénescentes peuvent être identifiées même dans les populations hétérogènes de cellules et les tissus vieillissants, tels que des biopsies de peau des individus plus âgés. Il a également été utilisé pour montrer une corrélation entre la sénescence cellulaire et le vieillissement23 car il est un marqueur fiable pour la détection des cellules sénescentes dans plusieurs organismes et les conditions27,28,29, 30. Ici, un test de dépistage à haut débit SA-M-Gal basé sur le substrat fluorescent C12FDG à l'aide de fibroblastes embryonnaires de souris primaires (MEFs) avec une sénescence cellulaire induite par le stress solidement oxydatif est décrit et ses avantages et inconvénients sont discutés. Bien que cet analyse puisse être effectué avec différents types de cellules, l'utilisation d'Ercc1-déficient, MEFs altérés de réparation d'ADN permet l'induction plus rapide de la sénescence dans des conditions de stress oxydatif. Chez la souris, l'expression réduite de l'endodocléère d'ENdonuclease d'ADN ERCC1-XPF cause la réparation altérée d'ADN, l'accumulation accélérée des dommages endogènes d'ADN, le ROS élevé, le dysfonctionnement mitochondrique, le fardeau sénescent accru de cellules, la perte de fonction de cellules souches et vieillissement prématuré, semblable au vieillissement naturel31,32. De même, Ercc1-déficients MEFs subissent une sénescence plus rapide dans la culture17. Une caractéristique importante de l'exemple sénescent MEF est que chaque puits a un mélange de cellules sénescentes et non-sénescentes, permettant la démonstration claire des effets sénescents de cellules spécifiques. Cependant, bien que nous croyons que l'utilisation du stress oxydatif dans les cellules primaires pour induire la sénescence est plus physiologique, cet essai peut également être employé avec des lignes cellulaires où la sénescence est induite avec des agents dommageables d'ADN comme l'étoposide ou l'irradiation.
Protocole
L'utilisation des animaux a été approuvée par le Scripps Florida Institutional Animal Care and Use Committee.
1. Génération de fibroblaste sénescent embryonnaire murine (MEF) - 12-15 jours
- Isoler le type sauvage et Ercc1-/- MEFs des souris femelles enceintes au jour embryonnaire 13 (E13) comme décrit précédemment33.
REMARQUE : Toutes les étapes suivantes sont effectuées dans une hotte de culture tissulaire dans des conditions aseptiques et à l'aide d'instruments stériles. - Reséquez la tête de l'embryon au-dessus des yeux.
- Enlever le tissu rouge (coeur et foie) et les utiliser pour le génotypage si nécessaire.
- Préparer 500 ml d'un mélange de 1:1 de dulbecco's Modified Eagle's Medium (DMEM) et de Ham's F10 avec 10% de sérum bovin fœtal, 1x acides aminés non essentiels, pénicilline et streptomycine comme milieu de croissance et réchauffez-le jusqu'à 37 oC pendant environ 15 minutes avant chaque utilisation. Stocker le milieu de croissance à 4 oC.
- Incuber le reste de l'embryon avec 0,25% de trypsine/EDTA pendant 10 min.
- Émincer l'embryon en morceaux de 1 mm et pipette le tissu de haut en bas à plusieurs reprises.
- Ajouter 10 ml de tissus moyens de croissance et de plaque d'un embryon par plaque de culture cellulaire de 10 cm de diamètre (passage 0).
- Cultiver les cellules à 37 oC, 3 % O2, 5 % CO2.
REMARQUE : Seules les cellules MEF se fixent aux plaques de culture tissulaire non enduites dans ces conditions. - Changer de milieu tous les jours dans le passage 0 pour enlever les tissus non attachés et les fragments de cellules.
REMARQUE : Selon la taille de l'embryon et la qualité de l'isolement, la cellule atteint habituellement la confluence après 2 à 3 jours. - Trypsinisation
- Enlever soigneusement le milieu de croissance et laver les cellules avec 10 ml de 1x PBS deux fois.
- Ajouter 2 ml d'une solution trypsine/EDTA de 0,025 % aux cellules dans des plaques de 10 cm de diamètre et incuber à 37 oC pendant 2 à 3 min.
- Assurez-vous que les cellules sont détachées de la surface en inspectant les cellules sous le microscope.
- Terminer la digestion de la trypsine en ajoutant la même quantité de milieu de croissance.
- Transférer les cellules dans un tube conique et des cellules centrifugeuses à 200 x g pendant 3 min et jeter le supernatant.
- Resuspendre soigneusement les cellules dans le milieu de croissance frais, compter les cellules et les semer dans de nouvelles plaques à la densité cellulaire projetée.
- Pour les sous-cultures non sénescentes, les cellules confluents fendues 1:4 et s'étendent pour un autre passage à 3 % O2, 37 oC pour produire plus de cellules (passage 1).
REMARQUE : À ce stade, les cellules peuvent être maintenues en culture ou stockées pour une utilisation ultérieure dans l'azote liquide, dans des cryovials contenant environ 1 million de cellules chacune. Cette étape offre également la possibilité de générer un lot mixte de cellules à partir de différents animaux pour réduire la variabilité provenant de l'analyse animale unique. - Pour induire la sénescence cellulaire, les cellules confluentes de graines fendues du passage 1 à un rapport de 1:4 et les incitent à 20 % O2, 37 oC, 5 % CO2 pendant 3 jours; ces conditions de culture sont atmosphériques pour l'oxygène.
REMARQUE : La culture des cellules et des blastocystes sous des concentrations ambiantes d'oxygène peut élever des marqueurs de sénescence cellulaire spécifiquement quand la réparation de dommages d'ADN est altérée34,35,36. - Répétez cette procédure pour 2 passages de plus.
- Pour surveiller la sénescence cellulaire, mesurez l'augmentation graduelle du diamètre cellulaire et du volume cellulaire pendant chaque étape de trypsinisation à l'aide d'un système avancé de compteur de cellules Coulter.
- Évaluer la réduction de la prolifération cellulaire en déterminé le doublement de la population (PD) à l'aide de l'équation
PDT (t2-t1)/3.32 x (log n2 - log n1)
REMARQUE : Le temps de doublement de la population n'a été utilisé que pour les cellules non sénescentes. - Utilisez un passage précoce de type sauvage ou Ercc1-/- cellules MEF qui ont été maintenues à 3% O2, 37 oC, 5% CO2 comme cellules témoins non-sénescentes.
2. Test de dépistage associé au Senescent - 2-3 jours
- Préparer des solutions de stock de 10 mM dans DMSO de tous les médicaments à tester et stocker les aliquots à -80 oC. Ne pas geler-dégeler les solutions de stock car cela peut diminuer l'activité des médicaments.
REMARQUE: Ici, l'inhibiteur HSP90 17DMAG a été utilisé comme un médicament sénolytique capable de tuer spécifiquement les cellules sénescentes17. - Le jour de l'expérience dégeler l'aliquot, diluer les médicaments dans le milieu de culture frais et ajouter aux cellules contenant le milieu conditionné à un rapport de 1:1 pour produire la concentration finale dans le milieu de croissance.
- Utilisez des plaques de prédilution de 96 puits pour les dilutions en série et les combinaisons de médicaments.
REMARQUE : Pour les cellules MEF, il a été empiriquement déterminé que les concentrations de DMSO ne devraient pas dépasser 2% et les cellules témoins traitées avec les concentrations les plus élevées de DMSO utilisées devraient être incluses dans chaque course. - Graines 5 x 103 cellules sénescentes ou 3 x 103 cellules non sénescentes par puits dans 96 plaques de puits d'au moins 6 h avant le traitement dans 100 l de milieu de croissance et incuber à 20% O2, 37 oC, 5% CO2.
REMARQUE : Les cellules doivent être environ 80 % de confluents avant le traitement. - Utilisez la culture de tissu de mur noir/fond clair, traité 96 plaques de puits pour réduire au minimum le crosstalk et le fond fluorescents de signal.
REMARQUE : Cependant, des plaques claires ont également été testées avec succès. - Ajouter les dilutions de médicaments aux cellules MEF et couver pendant 24 h à 48 h sous 20% O2, 37 oC, 5% CO2 conditions.
- Maintenir les cellules non sénescentes sous 3 % O2, 37 oC, 5 % co2.
- Pour l'alcalinisation lysosomique, préparer une solution de 10 mM de bafilomycine A1, aliquot et garder congelé à -20 oC.
- Pour l'analyse de la fluorescence de l'activité SA-MD-Gal, préparez une solution de stock FDG de 2 mM C12,entreposez-le à -20 oC et protégez-vous de la lumière.
- Pour la solution de travail, préparez 100 M C12FDG en milieu de croissance le jour de l'expérience.
REMARQUE : Toutes les étapes (d'incubation) impliquant C12FDG doivent être exécutées dans l'obscurité. - Retirez la solution médicamenteuses et lavez les cellules 1 fois avec 100 l de 1x PBS.
- Induire l'alcalinisation lysosomale en prétraitant les cellules avec 90 oL d'une solution de 100 nM de bafilomycine A1 préparée dans un milieu de culture cellulaire frais pendant 1 heure à 20 % O2, 37 oC, 5 % CO2.
- Ajouter 10 l de 100 M C12FDG solution de travail au milieu de culture (concentration finale 10 M).
- Incuber les cellules pendant 2 h.
- Ajouter 2 oL d'un colorant Hoechst 33342 de 100 g/mL (concentration finale de 2 g/mL) à la culture et incuber pendant 20 min.
- Retirez les supports et ajoutez 100 l de milieu de croissance frais.
3. Analyse quantitative d'image fluorescente à haute teneur
- Utilisez une plate-forme d'acquisition et d'analyse d'images fluorescentes à haute teneur pour acquérir des images fluorescentes des cellules dans les deux canaux appropriés pour la capture de la fluorescence Hoechst et C12FDG (p. ex., préréglages du canal DAPI et FITC, respectivement).
REMARQUE : Les protocoles d'acquisition exigent la définition de plusieurs variables spécifiques à l'assidu. Le but d'un protocole d'acquisition est de capturer un nombre suffisant d'images fluorescentes en cours d'élaboration d'un nombre suffisant de cellules pour l'analyse quantitative en aval. - Élaborer un protocole d'analyse approprié en sélectionnant chaque canal et en définissant, en ajustant un ou plusieurs critères sélectifs, ce qui constitue une caractéristique d'intérêt pour chaque canal.
- Pour le noyau, utiliser ainsi des pré-ensembles de segmentation (p. ex. segmentation nucléaire, segmentation des vésicules, segmentation du cytoplasme) qui permettront d'identifier les organites cellulaires sur la base de multiples critères, y compris la morphologie, la taille et le signal. intensité. Ajuster ces critères pour inclure les noyaux tout en excluant les fragments nucléaires et les débris qui peuvent avoir un signal, mais qui sont, par exemple, trop grands ou trop petits pour être des noyaux.
- Vérifiez le signal dans le canal FITC qui est la fluorescence du C12FDG clivé et représente la quantité d'activité sénescence-associée-galactosidase dans les cellules.
REMARQUE : Les cellules sénescentes ont une activité plus élevée de sénescence-associée à la galactosidase que les cellules non-sénescentes ; cependant, la fluorescence C12FDG sera non discrète et continue, nécessitant l'établissement d'un seuil entre ce qui est considéré comme un C12FDG-positif et une cellule C12FDG-négatif. - À l'aide d'un logiciel d'analyse disponible dans le commerce, générez un nombre de cas où une région définie entourant le noyau (une cellule présumée) s'est chevauchée au moins une fois avec un FDG C12au-dessus du seuil.
REMARQUE : Le logiciel d'analyse génère automatiquement un comptage à l'aide de liens cibles. C'est la définition pratique de l'assidu d'une cellule sénescente, C12FDG-positive.
- Analyser tous les échantillons en tripleavec 3-5 champs par puits et les valeurs moyennes et les écarts types étant calculés en conséquence.
- Calculez le pourcentage de cellules sénescentes à l'aide de la formule suivante :
Cellules sénescentes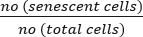 (%) x 100
(%) x 100
4. Paramètres de validation d'assise
- Pour tous les échantillons, le coefficient intra- et inter-analyse des variations (%CV) a été calculé à l'aide de la formule suivante :
CV intra-assay (n 10 répétitions mesurées dans une expérience) x 100 (%)
dans une expérience) x 100 (%)
CV d'inter-évaluation (n 5 expériences indépendantes) x 100 (%)
indépendantes) x 100 (%) - Aux fins du dépistage, déterminer la valeur du Z, un paramètre statistique pour évaluer la qualité d'un essai, à partir de cellules traitées avec 200 nM de rapamycine pendant 24 h à 20 % O2, 37 oC, 5 % CO2 (un contrôle positif pour les médicaments sénothérapeutiques) et non traitée cellules sénescentes (contrôle négatif).
REMARQUE : La valeur de Z a été calculée selon Zhang et coll.37. Les valeurs de Z entre 0,5 et 1 indiquent qu'un test peut être utilisé pour le dépistage des drogues.
Résultats
L'activité de SA--Gal peut être détectée dans les cellules qui sont induites à séneesce par diverses manières de l'épuisement réplicif, le stress génotoxique et oxydatif, à l'activation d'oncogène23,25,38. Dans le modèle actuel utilisant les cellules embryonnaires de fibroblaste d'Ercc1-déficientes de souris, les conditions normoxic de croissance (20% O2) étaient suffisa...
Discussion
SA-MD-Gal est un biomarqueur bien défini pour la sénescence cellulaire découvert à l'origine par Dimri et al. (1995) montrant que les fibroblastes humains sénescents ont augmenté l'activité de SA-A-Gal lorsqu'ils sont évalués au pH 623 par rapport aux cellules proliférantes. Pendant ce temps, l'analyse in vitro et in vivo pour SA--Gal ont été établis pour différents types de cellules et de tissus25,39,
Déclarations de divulgation
Les auteurs n'ont rien à révéler.
Remerciements
Ce travail a été soutenu par LES subventions des NIH AG043376 (Projet 2 et Core A, PDR; Projet 1 et Core B, LJN) et AG056278 (Projet 3 et Core A, PDR; et Projet 2, LJN) et une subvention de la Glenn Foundation (LJN).
matériels
| Name | Company | Catalog Number | Comments |
| DMEM | Corning | 10-013-CV | medium |
| Ham's F10 | Gibco | 12390-035 | medium |
| fetal bovine serum | Tissue Culture Biologics | 101 | serum |
| 1x non-essential amino acids | Corning | 25-025-Cl | amino-acids |
| bafilomycin A1 | Sigma | B1793 | lysosomal inhibitor |
| C12FDG | Setareh Biotech | 7188 | b-Gal substrate |
| Hoechst 33342 | Life Technologies | H1399 | DNA intercalation agent |
| 17DMAG | Selleck Chemical LLC | 50843 | HSP90 inhibitor |
| InCell6000 Cell Imaging System | GE Healthcare | High Content Imaging System |
Références
- Hayflick, L., Moorhead, P. S. The serial cultivation of human diploid cell strains. Experimental Cell Research. 25, 585-621 (1961).
- Campisi, J., di Fagagna, F. D. Cellular senescence: when bad things happen to good cells. Nature Reviews Molecular Cell Biology. 8 (9), 729-740 (2007).
- Harley, C. B., Futcher, A. B., Greider, C. W. Telomeres shorten during ageing of human fibroblasts. Nature. 345 (6274), 458-460 (1990).
- Zhu, J., Woods, D., McMahon, M., Bishop, J. M. Senescence of human fibroblasts induced by oncogenic Raf. Genes and Development. 12 (19), 2997-3007 (1998).
- Dimri, G. P., Itahana, K., Acosta, M., Campisi, J. Regulation of a senescence checkpoint response by the E2F1 transcription factor and p14(ARF) tumor suppressor. Molecular and Cellular Biology. 20 (1), 273-285 (2000).
- Michaloglou, C., et al. BRAFE600-associated senescence-like cell cycle arrest of human naevi. Nature. 436 (7051), 720-724 (2005).
- Niedernhofer, L. J. Tissue-specific accelerated aging in nucleotide excision repair deficiency. Mechanisms of Ageing and Development. 129 (78), 408-415 (2008).
- Gitenay, D., et al. Glucose metabolism and hexosamine pathway regulate oncogene-induced senescence. Cell Death & Disease. 5, e1089 (2014).
- Erusalimsky, J. D., Kurz, D. J. Cellular senescence in vivo: its relevance in ageing and cardiovascular disease. Experimental Gerontology. 40 (8-9), 634-642 (2005).
- Kassem, M., Marie, P. J. Senescence-associated intrinsic mechanisms of osteoblast dysfunctions. Aging Cell. 10 (2), 191-197 (2011).
- Campisi, J., Andersen, J. K., Kapahi, P., Melov, S. Cellular senescence: A link between cancer and age-related degenerative disease. Seminars in Cancer Biology. 21 (6), 354-359 (2011).
- Golde, T. E., Miller, V. M. Proteinopathy-induced neuronal senescence: a hypothesis for brain failure in Alzheimer's and other neurodegenerative diseases. Alzheimers Research & Therapy. 1 (2), 5 (2009).
- Telgenhoff, D., Shroot, B. Cellular senescence mechanisms in chronic wound healing. Cell Death & Differentiation. 12 (7), 695-698 (2005).
- Baker, D. J., et al. Clearance of p16Ink4a-positive senescent cells delays ageing-associated disorders. Nature. 479 (7372), 232-236 (2011).
- Baker, D. J., et al. Naturally occurring p16-positive cells shorten healthy lifespan. Nature. , (2016).
- Childs, B. G., et al. Senescent intimal foam cells are deleterious at all stages of atherosclerosis. Science. 354 (6311), 472-477 (2016).
- Fuhrmann-Stroissnigg, H., et al. Identification of HSP90 inhibitors as a novel class of senolytics. Nature Communications. 8 (1), 422 (2017).
- Zhu, Y., et al. New agents that target senescent cells: the flavone, fisetin, and the BCL-XL inhibitors, A1331852 and A1155463. Aging. 9 (3), 955-963 (2017).
- Zhu, Y., et al. Identification of a Novel Senolytic Agent, Navitoclax, Targeting the Bcl-2 Family of Anti-Apoptotic Factors. Aging Cell. , (2015).
- Zhu, Y., et al. The Achilles' heel of senescent cells: from transcriptome to senolytic drugs. Aging Cell. , (2015).
- Baar, M. P., et al. Targeted Apoptosis of Senescent Cells Restores Tissue Homeostasis in Response to Chemotoxicity and Aging. Cell. 169 (1), 132-147 (2017).
- Jeon, O. H., et al. Local clearance of senescent cells attenuates the development of post-traumatic osteoarthritis and creates a pro-regenerative environment. Nature. , (2017).
- Dimri, G. P., et al. A biomarker that identifies senescent human cells in culture and in aging skin in vivo. Proceedings of the National Academy of Sciences USA. 92 (20), 9363-9367 (1995).
- Itahana, K., Campisi, J., Dimri, G. P. Methods to detect biomarkers of cellular senescence: the senescence-associated beta-galactosidase assay. Methods in Molecular Biology. 371, 21-31 (2007).
- Debacq-Chainiaux, F., Erusalimsky, J. D., Campisi, J., Toussaint, O. Protocols to detect senescence-associated beta-galactosidase (SA-[beta]gal) activity, a biomarker of senescent cells in culture and in vivo. Nature Protocols. 4 (12), 1798-1806 (2009).
- Cahu, J., Sola, B. A sensitive method to quantify senescent cancer cells. Journal of Visualized Experiments. 78 (78), (2013).
- Collado, M., et al. Tumour biology: Senescence in premalignant tumours. Nature. 436 (7051), 642 (2005).
- Krishnamurthy, J., et al. Ink4a/Arf expression is a biomarker of aging. The Journal of Clinical Investigation. 114 (9), 1299-1307 (2004).
- Mishima, K., et al. Senescence-associated beta-galactosidase histochemistry for the primate eye. Investigative Ophthalmology, Visual Science. 40 (7), 1590-1593 (1999).
- Melk, A., et al. Expression of p16INK4a and other cell cycle regulator and senescence associated genes in aging human kidney. Kidney International. 65 (2), 510-520 (2004).
- Niedernhofer, L. J., et al. A new progeroid syndrome reveals that genotoxic stress suppresses the somatotroph axis. Nature. 444 (7122), 1038-1043 (2006).
- Wang, J., Clauson, C. L., Robbins, P. D., Niedernhofer, L. J., Wang, Y. The oxidative DNA lesions 8,5′-cyclopurines accumulate with aging in a tissue-specific manner. Aging Cell. 11 (4), 714-716 (2012).
- Jozefczuk, J., Drews, K., Adjaye, J. Preparation of mouse embryonic fibroblast cells suitable for culturing human embryonic and induced pluripotent stem cells. Journal of Visualized Experiments. (64), (2012).
- Jagannathan, L., Cuddapah, S., Costa, M. Oxidative stress under ambient and physiological oxygen tension in tissue culture. Current Pharmacology Reports. 2 (2), 64-72 (2016).
- Meuter, A., et al. Markers of cellular senescence are elevated in murine blastocysts cultured in vitro: molecular consequences of culture in atmospheric oxygen. J Assist Reprod Genet. 31 (10), 1259-1267 (2014).
- Robinson, A. R., et al. Spontaneous DNA damage to the nuclear genome promotes senescence, redox imbalance and aging. Redox Biology. 17, 259-273 (2018).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A Simple Statistical Parameter for Use in Evaluation and Validation of High Throughput Screening Assays. Journal Of Biomolecular Screening. 4 (2), 67-73 (1999).
- Zhao, H., Darzynkiewicz, Z. Biomarkers of cell senescence assessed by imaging cytometry. Methods in Molecular Biology. 965, 83-92 (2013).
- Yang, N. -. C., Hu, M. -. L. A fluorimetric method using fluorescein di-β-d-galactopyranoside for quantifying the senescence-associated β-galactosidase activity in human foreskin fibroblast Hs68 cells. Analytical Biochemistry. 325 (2), 337-343 (2004).
- Zhao, J., et al. Quantitative Analysis of Cellular Senescence in Culture and In Vivo. Current Protocols in Cytometry. 79, (2017).
- Capparelli, C., et al. CDK inhibitors (p16/p19/p21) induce senescence and autophagy in cancer-associated fibroblasts, "fueling" tumor growth via paracrine interactions, without an increase in neo-angiogenesis. Cell Cycle. 11 (19), 3599-3610 (2012).
- Coppe, J. P., Desprez, P. Y., Krtolica, A., Campisi, J. The senescence-associated secretory phenotype: the dark side of tumor suppression. Annual Review of Pathology. 5, 99-118 (2010).
- Mah, L. J., El-Osta, A., Karagiannis, T. C. GammaH2AX as a molecular marker of aging and disease. Epigenetics. 5 (2), 129-136 (2010).
- Hewitt, G., et al. Telomeres are favoured targets of a persistent DNA damage response in ageing and stress-induced senescence. Nature Communications. 3, 708 (2012).
- Narita, M., et al. Rb-mediated heterochromatin formation and silencing of E2F target genes during cellular senescence. Cell. 113 (6), 703-716 (2003).
- Georgakopoulou, E. A., et al. Specific lipofuscin staining as a novel biomarker to detect replicative and stress-induced senescence. A method applicable in cryo-preserved and archival tissues. Aging-Us. 5 (1), 37-50 (2013).
- Severino, J., Allen, R. G., Balin, S., Balin, A., Cristofalo, V. J. Is β-Galactosidase Staining a Marker of Senescence in Vitro and in Vivo?. Experimental Cell Research. 257 (1), 162-171 (2000).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon