È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Saggio di screening basato su SA-Galactosidase per l'identificazione dei farmaci senoterapici
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
La senescenza cellulare è il fattore chiave nello sviluppo di patologie croniche legate all'età. L'identificazione delle terapie che colpisce le cellule senescenti è promettente per estendere l'invecchiamento sano. Qui, presentiamo un nuovo saggio da vagliare per l'identificazione delle senoterapie sulla base della misurazione dell'attività di senescenza associata a z-Galactosidase in singole cellule.
Abstract
La senescenza cellulare è uno dei segni distintivi dell'invecchiamento noto per influenzare negativamente una vita sana. I farmaci in grado di uccidere le cellule senescenti specificamente nella coltura cellulare, chiamato senolitico, possono ridurre il carico di cellule senescenti in vivo ed estendere la salute. Diverse classi di senolitica sono state identificate fino ad oggi, tra cui inibitori HSP90, inibitori della famiglia Bcl-2, piperlongumine, un peptide inibitorio FOXO4 e la combinazione di Dasatinib/Quercetin. La rilevazione di SA-z-Gal in un pH lisonico aumentato è uno dei migliori marcatori caratterizzati per la rilevazione di cellule senescenti. Le misurazioni delle cellule vive dell'attività di senescenza associati alla galactosidasi (SA-z-Gal) utilizzando il substrato fluorescente C12FDG in combinazione con la determinazione del numero totale di cellule utilizzando un colorante Hoechst intercalante del DNA apre la possibilità di schermo per i farmaci senoteri che riducono l'attività complessiva di SA-z-Gal uccidendo cellule senescenti (senolytica) o sopprimendo SA-z-Gal e altri fenotipi delle cellule senescenti (senomorfici). L'uso di una piattaforma di acquisizione e analisi di immagini fluorescenti ad alto contenuto consente di uno screening rapido e ad alto consumo delle librerie di farmaci per gli effetti sulla SA-z-Gal, la morfologia cellulare e il numero di cellule.
Introduzione
La senescenza cellulare è stata descritta per la prima volta da Leonard Hayflick e Paul Moorhead, che hanno dimostrato che le cellule normali avevano una capacità limitata di proliferare nella coltura1. Le cellule senescenti non riescono a proliferare nonostante la presenza di nutrienti, fattori di crescita e mancanza di inibizione del contatto, ma rimangono metabolicamente attivi2. Questo fenomeno è noto come senescenza replicativa ed è stato attribuito principalmente all'accorciamento dei telomeri, almeno nelle cellule umane3. Ulteriori studi hanno dimostrato che le cellule possono anche essere indotte a subire senescenza in risposta ad altri stimoli, come lo stress oncogenico (senescenza indotta dall'oncogeno, OIS), danni al DNA, farmaci citotossici o irradiazione (senescenza indotta dallo stress, SIS)4 , 5 Del numero 3( , 6. In risposta al danno del DNA, compresa l'erosione dei telomeri, le cellule possono senescere, iniziano la crescita cellulare incontrollata o subiscono l'apoptosi se il danno non può essere riparato. In questo caso, la senescenza cellulare sembra essere utile in quanto agisce in modo repressivo tumorale2. Al contrario, la senescenza è aumentata con l'invecchiamento a causa dell'accumulo di danni cellulari tra cui il danno al DNA. Poiché le cellule senescenti possono secernere citochine, proteinasi di metallo e fattori di crescita, chiamato fenotipo secretoria associato alla senescenza (SASP), questo aumento dipendente dall'età della senescenza cellulare e sAS contribuisce alla diminuzione dell'omeostasi del tessuto e successivamente l'invecchiamento. Inoltre, questo aumento dipendente dall'età del carico di senescenza è noto per indurre malattie metaboliche, sensibilità allo stress, sindromi di progeria e guarigione alterata7,8 ed è, in parte, responsabile delle numerose età-correlati malattie, come l'aterosclerosi, l'osteoartrite, la degenerazione muscolare, la formazione di ulcere e il morbo di Alzheimer9,10,11,12,13. Eliminare le cellule senescenti può aiutare a prevenire o ritardare la disfunzione tissutale ed estendere la durata di salute14. Ciò è stato dimostrato nei modelli murini transgenici14,15,16, nonché utilizzando farmaci senolitici e combinazioni di farmaci che sono stati scoperti sia attraverso gli sforzi di screening farmacologico che l'analisi bioinformatica di percorsi indotti specificamente nelle cellule senescenti17,18,19,20,21,22. Identificare farmaci senoteri ottimali, in grado di ridurre in modo più efficace l'onere cellulare senescente, è un importante passo avanti nello sviluppo di approcci terapeutici per un invecchiamento sano.
Le cellule senescenti mostrano caratteristiche fenotipiche e molecolari caratteristiche, sia in coltura che in vivo. Questi marcatori di senescenza potrebbero essere la causa o il risultato dell'induzione della senescenza o un sottoprodotto dei cambiamenti molecolari in queste cellule. Tuttavia, nessun singolo marcatore si trova specificamente nelle cellule senescenti. Attualmente, il rilevamento associato alla senescenza con la galactosidase (SA-z-Gal) è uno dei metodi basati su una singola cellula più caratterizzati e consolidati per misurare la senescenza in vitro e in vivo. L'idrolasi lisosomia con un'attività enzimatica ottimale al pH 4 è un idrolasi lisoso. Misurare la sua attività al pH 6 è possibile perché le cellule senescenti mostrano una maggiore attività lisosomica23,24. Per le cellule viventi, l'aumento del pH lisosomiale si ottiene con alcalinizzazione lisosomica con il vacuolar H-L'inibitore di A!mycin A1 o l'inibitore dell'acidificazione endosomica cloroquine25,26. 5-Dodecanoylaminofluorescein Di-C12FDG) viene utilizzato come substrato nelle cellule viventi in quanto mantiene il prodotto fessurato nelle cellule a causa della sua moiety25lipofilica al carbonio. È importante sottolineare che l'attività SA-z-Gal stessa non è direttamente collegata ad alcuna via identificata nelle cellule senescenti e non è necessaria per indurre la senescenza. Con questo test, le cellule senescenti possono essere identificate anche nelle popolazioni cellulari eterogenee e nei tessuti di invecchiamento, come le biopsie cutanee di individui più anziani. E 'stato utilizzato anche per mostrare una correlazione tra senescenza cellulare e invecchiamento23 in quanto è un marcatore affidabile per il rilevamento delle cellule senescenti in diversi organismi e condizioni27,28,29, 30. In questo caso, viene descritto un saggio di screening SA-z-Gal ad alta produttività basato sul substrato fluorescente C12FDG che utilizza fibroblasti embrionali primari (MEF) con senescenza cellulare indotta dallo stress fortemente ossidativo e dei suoi vantaggi e svantaggi sono discussi. Anche se questo saggio può essere eseguito con diversi tipi di cellule, l'uso di Ercc1-deficient, DNA riparazione COMme compromessa consente una più rapida induzione di senescenza in condizioni di stress ossidativo. Nei topi, l'espressione ridotta dell'endonuclease del DNA endaccleatta ERCC1-XPF causa una riparazione compromessa del DNA, un accumulo accelerato di danni endogeni del DNA, ROS elevato, disfunzione mitocondriale, aumento del carico di cellule senescenti, perdita della funzione delle cellule staminali e invecchiamento precoce, simile all'invecchiamento naturale31,32. Allo stesso modo, i MEF carenti di Ercc1subiscono più rapidamente la senescenza nella cultura17. Una caratteristica importante del saggio SENescente MEF è che ogni pozzo ha una miscela di cellule senescenti e non senescenti, consentendo la chiara dimostrazione di effetti senescenti specifici delle cellule. Tuttavia, anche se crediamo che l'uso dello stress ossidativo nelle cellule primarie per indurre la senescenza sia più fisiologico, questo saggio può essere utilizzato anche con linee cellulari in cui la senescenza è indotta da agenti dannosi del DNA come l'etoposide o l'irradiazione.
Protocollo
L'uso degli animali è stato approvato dallo Scripps Florida Institutional Animal Care and Use Committee.
1. Generazione di fibroblasto embrionale murino senescente (MEF) – 12-15 giorni
- Isolare il tipo selvaggio e Ercc1-/- MEF da topi femmine in gravidanza alla giornata embrionale 13 (E13) come descritto in precedenza33.
NOTA: Tutti i seguenti passaggi vengono eseguiti in una cappa di coltura tissutale in condizioni asettiche e utilizzando strumenti sterili. - Rivedere la testa dell'embrione sopra gli occhi.
- Rimuovere il tessuto rosso (cuore e fegato) e utilizzarli per la genotipizzazione, se necessario.
- Preparare 500 mL di una miscela 1:1 di Dulbecco's Modified Eagle's Medium (DMEM) e Ham's F10 con 10% siero bovino fetale, 1 x aminoacidi non essenziali, penicillina e streptomicina come mezzo di crescita e riscaldarlo fino a 37 gradi centigradi per circa 15 min prima di ogni utilizzo. Riservare il mezzo di crescita a 4 gradi centigradi.
- Incubare il resto dell'embrione con 0,25% di metapsin/EDTA per 10 min.
- Tritate l'embrione in pezzi da 1 mm e pipette il tessuto su e giù più volte.
- Aggiungere 10 mL di mezzo di crescita e tessuti di lamiera di un embrione per 10 cm di diametro piastra di coltura cellulare (passaggio 0).
- Coltivare le cellule a 37 gradi centigradi, 3% O2, 5% CO2.
NOTA: solo le cellule MEF si attaccano a piastre di coltura di tessuti non rivestiti in queste condizioni. - Cambiare il mezzo ogni giorno nel passaggio 0 per rimuovere i frammenti di tessuto non attaccati e frammenti cellulari.
NOTA: A seconda delle dimensioni dell'embrione e della qualità dell'isolamento, le cellule di solito raggiungono la confluenza dopo 2 o 3 giorni. - Prova tossiazione
- Rimuovere con attenzione il mezzo di crescita e lavare le cellule con 10 mL di 1x PBS due volte.
- Aggiungere 2 mL di una soluzione di prova/EDTA 0,025% alle cellule in piastre di 10 cm di diametro e incubare a 37 gradi centigradi per 2-3 min.
- Assicurarsi che le cellule siano staccate dalla superficie ispezionando le cellule al microscopio.
- Terminare la digestione della trypsin aggiungendo la stessa quantità di mezzo di crescita.
- Trasferire le cellule su un tubo conico e centrifugare le cellule a 200 x g per 3 min e scartare il supernatante.
- Risospendere le cellule con attenzione nel mezzo di crescita fresco, contare le cellule e seme in nuove piastre alla densità delle cellule proiettate.
- Per la coltivazione secondaria non senescente diviso le cellule confluenti 1:4 ed estendere per un altro passaggio al 3% O2, 37 c per produrre più cellule (passaggio 1).
NOTA: A questo punto le cellule possono essere mantenute in coltura o conservate per un uso successivo in azoto liquido, in crioviali contenenti circa 1 milione di cellule ciascuna. Questa fase offre anche la possibilità di generare un lotto misto di cellule da diversi animali per ridurre la variabilità derivante dall'analisi di singoli animali. - Per indurre la senescenza cellulare, i semi dividono le cellule confluenti dal passaggio 1 con un rapporto di 1:4 e le incubano al 20% O2, 37 c, 5% di CO2 per 3 giorni; queste condizioni di coltura sono atmosferiche per l'ossigeno.
NOTA: La coltivazione di cellule e blastocisti in concentrazioni ambientali di ossigeno può elevare marcatori di senescenza cellulare in particolare quando la riparazione del danno al DNA è compromessa34,35,36. - Ripetere questa procedura per altri 2 passaggi.
- Per monitorare la senescenza cellulare, misurare l'aumento graduale del diametro cellulare e del volume delle cellule durante ogni fase di propsinizzazione utilizzando un sistema avanzato di contatore delle cellule Coulter.
- Valutare la riduzione della proliferazione cellulare mediante la determinazione del raddoppio della popolazione (PD)
PDT (t2-t 1)/3.32 x (log n2 – log n1)
NOTA: il tempo di raddoppio della popolazione è stato utilizzato solo per le cellule non senescenti. - Utilizzare cellule ercc1-/- MEF di passaggio precoce che sono state tenute al 3% O2, 37 gradi centigradi, 5% di CO2 come celle di controllo non senescenti.
2. Saggio di screening z-Gal associato a un senescente – 2-3 giorni
- Preparare soluzioni di magazzino da 10 mM in DMSO di tutti i farmaci da testare e immagazzinare aliquote a -80 gradi centigradi. Non congelare soluzioni di stock disgelo in quanto questo può diminuire l'attività dei farmaci.
NOTA: Qui, l'inibitore dell'HSP90 17DMAG è stato utilizzato come un farmaco senolitico in grado di uccidere specificamente le cellule senescenti17. - Il giorno dell'esperimento scongelare il, diluire i farmaci in mezzo di coltura fresca e aggiungere alle cellule contenenti aliquota media condizionata ad un rapporto 1:1 per produrre la concentrazione finale nel mezzo di crescita.
- Utilizzare piastre di pre-diluizione di 96 pozze per diluizioni seriali e combinazioni di farmaci.
NOTA: Per le cellule MEF, è stato empiricamente determinato che le concentrazioni di DMSO non devono superare il 2% e che le celle di controllo trattate con le più alte concentrazioni di DMSO utilizzate devono essere incluse in ogni corsa. - Seme 5 x 103 cellule senescenti o 3 x 103 cellule non senescenti per bene in 96 pozzetti almeno 6 h prima del trattamento in 100 : L di mezzo di crescita medio e incubare a 20% O2, 37 gradi C, 5% CO2.
NOTA: Le cellule devono essere circa l'80% confluenti prima del trattamento. - Utilizzare la coltura del tessuto nero a parete/tessuto limpido, trattata 96 lastre di pozzo per ridurre al minimo il crosstalk del segnale fluorescente e lo sfondo.
NOTA: Tuttavia, anche le piastre chiare sono state testate con successo. - Aggiungere le diluizioni farmacologiche alle cellule MEF e incubare per 24 h a 48 h sotto 20% O2, 37 gradi centigradi, 5% condizioni di CO2.
- Mantenere le cellule non senescenti sotto 3% O2, 37 C, 5% condizioni di CO2.
- Per l'alcalinizzazione lisosomica, preparare una soluzione di bafilomycin A1 da 10 mM e mantenere congelato l'aliquota .
- Per l'analisi della fluorescenza dell'attività SA-z-Gal, preparare una soluzione di stock 2 m C12FDG, conservare a -20 gradi centigradi e proteggere dalla luce.
- Per la soluzione di lavoro, preparare 100 FDG C12in mezzo di crescita il giorno dell'esperimento.
NOTA: Tutte le fasi (incubazione) che coinvolgono C12FDG devono essere eseguite al buio. - Rimuovere la soluzione farmacologica e lavare le cellule 1 tempo con 100 L di 1x PBS.
- Indurre l'alininizzazione lisosomica pretrattando le cellule con 90 -L di una soluzione a1 di bafilomycina da 100 nM preparata in coltura a cellule fresche medio per 1 ora al 20% O2, 37 c, 5% CO2.
- Aggiungete al mezzo di coltura una soluzione di lavoro 10L di 100 M C12(concentrazione finale 10 M).
- Incubano cellule per 2 h.
- Aggiungete alla coltura 2 -g/mL Hoechst 33342 (concentrazione finale 2 g/mL) e incubate per 20 min.
- Rimuovere i supporti e aggiungere 100 l di nuovo mezzo di crescita.
3. Analisi quantitativa delle immagini fluorescenti ad alto contenuto
- Utilizzare una piattaforma di analisi e acquisizione di immagini fluorescenti ad alto contenuto per acquisire immagini fluorescenti delle cellule nei due canali appropriati per l'acquisizione della fluorescenza di Hoechst e C12FDG (ad esempio, rispettivamente i preset di canale DAPI e FITC).
NOTA: i protocolli di acquisizione richiedono la definizione di diverse variabili specifiche per il saggio. Lo scopo di un protocollo di acquisizione è quello di catturare un numero adeguato di immagini fluorescenti a fuoco di un numero sufficiente di cellule per l'analisi quantitativa a valle. - Sviluppare un protocollo di analisi appropriato selezionando ogni canale e definendo, regolando uno o più criteri selettivi, ciò che si qualifica come caratteristica di interesse in ogni canale.
- Per il nucleo, utilizzare così pre-set di segmentazione (ad esempio, segmentazione nucleare, segmentazione vescica, segmentazione del citoplasma) che permetterà l'identificazione di organelli cellulari sulla base di più criteri tra cui morfologia, dimensione e segnale intensità. Regolare questi criteri per includere i nuclei escludendo frammenti nucleari e detriti che possono avere un segnale, ma che sono, ad esempio, troppo grandi o troppo piccoli per essere nuclei.
- Controllare il segnale nel canale FITC che è fluorescenza da cleaved C12FDG e rappresenta la quantità di attività di senescenza associata a z-galactosidase nelle cellule.
NOTA: le cellule senescenti hanno un'attività di senescenza maggiore associata alla z-galactosidasi rispetto alle cellule non senescenti; tuttavia, la fluorescenza C12FDG sarà non discreta e continua, rendendo necessaria l'istituzione di una soglia tra ciò che è considerato un C12FDG-positivo e una cellula C12FDG-negativo. - Utilizzando un software analitico disponibile in commercio, generare un conteggio delle istanze in cui una regione definita che circonda il nucleo (una cella presuntiva) si è sovrapposta almeno una volta con un FDG C12superiore alla soglia.
NOTA: il software analitico genera automaticamente un conteggio utilizzando il collegamento di destinazione. Questa è la definizione pratica del saggio di una cellula senescente, C12FDG-positiva.
- Analizzare tutti i campioni in triplice copia con 3-5 campi per pozzo e valori medi e deviazioni standard calcolati di conseguenza.
- Calcolare la percentuale di celle senescenti utilizzando la seguente formula:
Celle senescenti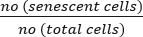 (%) : x 100
(%) : x 100
4. Parametri di convalida del test
- Per tutti i campioni il coefficiente di variazione intra-e inter-assay delle variazioni (%CV) è stato calcolato utilizzando la seguente formula:
CV intra-saggio (n - 10 ripetizioni misurate in un esperimento) x 100 (%)
100 (%)
CV inter-analisi (n : 5 esperimenti indipendenti) x 100 (%)
100 (%) - Ai fini dello screening, determinare il valore di , un parametro statistico per valutare la qualità di un saggio, da cellule trattate con 200 nM rapamicina per 24 h al 20% O2, 37 C, 5% CO2 (un controllo positivo per i farmaci senoteri) e non trattati celle senescenti (controllo negativo).
N.B.: Il valore di ' è stato calcolato in base a37. I valori di " n. tra 0,5 e 1 indicano che un saggio può essere utilizzato per lo screening farmacologico.
Risultati
L'attività di SA-z-Gal può essere rilevata in cellule che sono indotte a senescenza da vari modi da esaurimento replicativo, stress genotossico e ossidativo, all'attivazione dell'oncogene23,25,38. Nell'attuale modello che utilizza le cellule fibroblaste embrionali di Ercc1- carenti, le condizioni di crescita normossiche (20% O2) erano sufficienti a indurre la senescenza cell...
Discussione
È un biomarcatore ben definito per la senescenza cellulare originariamente scoperto da Dimri et al. (1995) dimostrando che i fibroblasti umani senescenti hanno aumentato l'attività di SA-z-Gal se sono stati analisi al pH 623 rispetto alle cellule che proliferano. Nel frattempo, in vitro e in vivo sono stati stabiliti saggi per SA-z-Gal per diversi tipi di cellule e tessuti25,39,40. Il metodo a ...
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo lavoro è stato sostenuto da NIH Grants AG043376 (Progetto 2 e Core A, PDR; Progetto 1 e Core B, LJN) e AG056278 (Progetto 3 e Core A, PDR; e Project 2, LJN) e una sovvenzione della Glenn Foundation (LJN).
Materiali
| Name | Company | Catalog Number | Comments |
| DMEM | Corning | 10-013-CV | medium |
| Ham's F10 | Gibco | 12390-035 | medium |
| fetal bovine serum | Tissue Culture Biologics | 101 | serum |
| 1x non-essential amino acids | Corning | 25-025-Cl | amino-acids |
| bafilomycin A1 | Sigma | B1793 | lysosomal inhibitor |
| C12FDG | Setareh Biotech | 7188 | b-Gal substrate |
| Hoechst 33342 | Life Technologies | H1399 | DNA intercalation agent |
| 17DMAG | Selleck Chemical LLC | 50843 | HSP90 inhibitor |
| InCell6000 Cell Imaging System | GE Healthcare | High Content Imaging System |
Riferimenti
- Hayflick, L., Moorhead, P. S. The serial cultivation of human diploid cell strains. Experimental Cell Research. 25, 585-621 (1961).
- Campisi, J., di Fagagna, F. D. Cellular senescence: when bad things happen to good cells. Nature Reviews Molecular Cell Biology. 8 (9), 729-740 (2007).
- Harley, C. B., Futcher, A. B., Greider, C. W. Telomeres shorten during ageing of human fibroblasts. Nature. 345 (6274), 458-460 (1990).
- Zhu, J., Woods, D., McMahon, M., Bishop, J. M. Senescence of human fibroblasts induced by oncogenic Raf. Genes and Development. 12 (19), 2997-3007 (1998).
- Dimri, G. P., Itahana, K., Acosta, M., Campisi, J. Regulation of a senescence checkpoint response by the E2F1 transcription factor and p14(ARF) tumor suppressor. Molecular and Cellular Biology. 20 (1), 273-285 (2000).
- Michaloglou, C., et al. BRAFE600-associated senescence-like cell cycle arrest of human naevi. Nature. 436 (7051), 720-724 (2005).
- Niedernhofer, L. J. Tissue-specific accelerated aging in nucleotide excision repair deficiency. Mechanisms of Ageing and Development. 129 (78), 408-415 (2008).
- Gitenay, D., et al. Glucose metabolism and hexosamine pathway regulate oncogene-induced senescence. Cell Death & Disease. 5, e1089 (2014).
- Erusalimsky, J. D., Kurz, D. J. Cellular senescence in vivo: its relevance in ageing and cardiovascular disease. Experimental Gerontology. 40 (8-9), 634-642 (2005).
- Kassem, M., Marie, P. J. Senescence-associated intrinsic mechanisms of osteoblast dysfunctions. Aging Cell. 10 (2), 191-197 (2011).
- Campisi, J., Andersen, J. K., Kapahi, P., Melov, S. Cellular senescence: A link between cancer and age-related degenerative disease. Seminars in Cancer Biology. 21 (6), 354-359 (2011).
- Golde, T. E., Miller, V. M. Proteinopathy-induced neuronal senescence: a hypothesis for brain failure in Alzheimer's and other neurodegenerative diseases. Alzheimers Research & Therapy. 1 (2), 5 (2009).
- Telgenhoff, D., Shroot, B. Cellular senescence mechanisms in chronic wound healing. Cell Death & Differentiation. 12 (7), 695-698 (2005).
- Baker, D. J., et al. Clearance of p16Ink4a-positive senescent cells delays ageing-associated disorders. Nature. 479 (7372), 232-236 (2011).
- Baker, D. J., et al. Naturally occurring p16-positive cells shorten healthy lifespan. Nature. , (2016).
- Childs, B. G., et al. Senescent intimal foam cells are deleterious at all stages of atherosclerosis. Science. 354 (6311), 472-477 (2016).
- Fuhrmann-Stroissnigg, H., et al. Identification of HSP90 inhibitors as a novel class of senolytics. Nature Communications. 8 (1), 422 (2017).
- Zhu, Y., et al. New agents that target senescent cells: the flavone, fisetin, and the BCL-XL inhibitors, A1331852 and A1155463. Aging. 9 (3), 955-963 (2017).
- Zhu, Y., et al. Identification of a Novel Senolytic Agent, Navitoclax, Targeting the Bcl-2 Family of Anti-Apoptotic Factors. Aging Cell. , (2015).
- Zhu, Y., et al. The Achilles' heel of senescent cells: from transcriptome to senolytic drugs. Aging Cell. , (2015).
- Baar, M. P., et al. Targeted Apoptosis of Senescent Cells Restores Tissue Homeostasis in Response to Chemotoxicity and Aging. Cell. 169 (1), 132-147 (2017).
- Jeon, O. H., et al. Local clearance of senescent cells attenuates the development of post-traumatic osteoarthritis and creates a pro-regenerative environment. Nature. , (2017).
- Dimri, G. P., et al. A biomarker that identifies senescent human cells in culture and in aging skin in vivo. Proceedings of the National Academy of Sciences USA. 92 (20), 9363-9367 (1995).
- Itahana, K., Campisi, J., Dimri, G. P. Methods to detect biomarkers of cellular senescence: the senescence-associated beta-galactosidase assay. Methods in Molecular Biology. 371, 21-31 (2007).
- Debacq-Chainiaux, F., Erusalimsky, J. D., Campisi, J., Toussaint, O. Protocols to detect senescence-associated beta-galactosidase (SA-[beta]gal) activity, a biomarker of senescent cells in culture and in vivo. Nature Protocols. 4 (12), 1798-1806 (2009).
- Cahu, J., Sola, B. A sensitive method to quantify senescent cancer cells. Journal of Visualized Experiments. 78 (78), (2013).
- Collado, M., et al. Tumour biology: Senescence in premalignant tumours. Nature. 436 (7051), 642 (2005).
- Krishnamurthy, J., et al. Ink4a/Arf expression is a biomarker of aging. The Journal of Clinical Investigation. 114 (9), 1299-1307 (2004).
- Mishima, K., et al. Senescence-associated beta-galactosidase histochemistry for the primate eye. Investigative Ophthalmology, Visual Science. 40 (7), 1590-1593 (1999).
- Melk, A., et al. Expression of p16INK4a and other cell cycle regulator and senescence associated genes in aging human kidney. Kidney International. 65 (2), 510-520 (2004).
- Niedernhofer, L. J., et al. A new progeroid syndrome reveals that genotoxic stress suppresses the somatotroph axis. Nature. 444 (7122), 1038-1043 (2006).
- Wang, J., Clauson, C. L., Robbins, P. D., Niedernhofer, L. J., Wang, Y. The oxidative DNA lesions 8,5′-cyclopurines accumulate with aging in a tissue-specific manner. Aging Cell. 11 (4), 714-716 (2012).
- Jozefczuk, J., Drews, K., Adjaye, J. Preparation of mouse embryonic fibroblast cells suitable for culturing human embryonic and induced pluripotent stem cells. Journal of Visualized Experiments. (64), (2012).
- Jagannathan, L., Cuddapah, S., Costa, M. Oxidative stress under ambient and physiological oxygen tension in tissue culture. Current Pharmacology Reports. 2 (2), 64-72 (2016).
- Meuter, A., et al. Markers of cellular senescence are elevated in murine blastocysts cultured in vitro: molecular consequences of culture in atmospheric oxygen. J Assist Reprod Genet. 31 (10), 1259-1267 (2014).
- Robinson, A. R., et al. Spontaneous DNA damage to the nuclear genome promotes senescence, redox imbalance and aging. Redox Biology. 17, 259-273 (2018).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A Simple Statistical Parameter for Use in Evaluation and Validation of High Throughput Screening Assays. Journal Of Biomolecular Screening. 4 (2), 67-73 (1999).
- Zhao, H., Darzynkiewicz, Z. Biomarkers of cell senescence assessed by imaging cytometry. Methods in Molecular Biology. 965, 83-92 (2013).
- Yang, N. -. C., Hu, M. -. L. A fluorimetric method using fluorescein di-β-d-galactopyranoside for quantifying the senescence-associated β-galactosidase activity in human foreskin fibroblast Hs68 cells. Analytical Biochemistry. 325 (2), 337-343 (2004).
- Zhao, J., et al. Quantitative Analysis of Cellular Senescence in Culture and In Vivo. Current Protocols in Cytometry. 79, (2017).
- Capparelli, C., et al. CDK inhibitors (p16/p19/p21) induce senescence and autophagy in cancer-associated fibroblasts, "fueling" tumor growth via paracrine interactions, without an increase in neo-angiogenesis. Cell Cycle. 11 (19), 3599-3610 (2012).
- Coppe, J. P., Desprez, P. Y., Krtolica, A., Campisi, J. The senescence-associated secretory phenotype: the dark side of tumor suppression. Annual Review of Pathology. 5, 99-118 (2010).
- Mah, L. J., El-Osta, A., Karagiannis, T. C. GammaH2AX as a molecular marker of aging and disease. Epigenetics. 5 (2), 129-136 (2010).
- Hewitt, G., et al. Telomeres are favoured targets of a persistent DNA damage response in ageing and stress-induced senescence. Nature Communications. 3, 708 (2012).
- Narita, M., et al. Rb-mediated heterochromatin formation and silencing of E2F target genes during cellular senescence. Cell. 113 (6), 703-716 (2003).
- Georgakopoulou, E. A., et al. Specific lipofuscin staining as a novel biomarker to detect replicative and stress-induced senescence. A method applicable in cryo-preserved and archival tissues. Aging-Us. 5 (1), 37-50 (2013).
- Severino, J., Allen, R. G., Balin, S., Balin, A., Cristofalo, V. J. Is β-Galactosidase Staining a Marker of Senescence in Vitro and in Vivo?. Experimental Cell Research. 257 (1), 162-171 (2000).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon