Method Article
Hepatischer Vorläufer aus pluripotenten Stammzellen unter Verwendung eines definierten Differenzierungssystems
In diesem Artikel
Zusammenfassung
Das Ziel dieses Artikels ist es, einen standardisierten Ansatz zur Induktion der Differenzierung des menschlichen Lebervorläufers von pluripotenten Stammzellen bereitzustellen. Die Entwicklung dieses Verfahrens mit gebrauchsfertigen Medienformulierungen bietet dem Anwender ein einfaches System zur Erzeugung menschlicher Leberzellen für die biomedizinische Forschung und Translation.
Zusammenfassung
Lebererkrankungen sind ein eskalierendes globales Gesundheitsproblem. Während die Lebertransplantation eine wirksame Therapieform ist, ist die Patientensterblichkeit aufgrund mangelnder Verfügbarkeit von Spenderorganen gestiegen. Organknappheit wirkt sich auch auf die routinemäßige Versorgung mit menschlichen Hepatozyten für die Grundlagenforschung und die Klinik aus. Daher ist die Entwicklung erneuerbarer Quellen menschlicher Lebervorläuferzellen wünschenswert und das Ziel dieser Studie. Um menschliche Lebervorläufer in großem Maßstab effektiv erzeugen und einsetzen zu können, wurde ein reproduzierbares hepatisches Vorläuferdifferenzierungssystem entwickelt. Dieses Protokoll unterstützt die experimentelle Reproduzierbarkeit zwischen Anwendern in einer Reihe von Zellkultur-Softwareformaten und ermöglicht Differenzierungen sowohl mit menschlichen embryonalen als auch mit induzierten pluripotenten Stammzelllinien. Dies sind wichtige Vorteile gegenüber aktuellen Differenzierungssystemen, die die Grundlagenforschung verbessern und den Weg zur klinischen Produktentwicklung ebnen können.
Einleitung
Lebererkrankungen stellen eine globale Gesundheitsherausforderung dar und verursachen weltweit etwa 2 Millionen Todesfälle pro Jahr1. Obwohl es eine Reihe von Modellsystemen gibt, um Lebererkrankungen zu untersuchen und klinisch einzugreifen, ist der routinemäßige Einsatz zellbasierter Systeme durch signifikante Nachteile begrenzt (für eine Überprüfung siehe Szkolnicka et al.2). Fortschrittliche Humanpluripotenten Stammzellkulturen (hPSC) und somatische Zelldifferenzierungsmethoden stellen vielversprechende Technologien dar, um Werkzeuge für die biomedizinische Grundlagenforschung und erneuerbare Quellen differenzierter Zellen für die Klinik zu entwickeln3,4.
Bis heute wurden mehrere Protokolle zur Hepatozyten-ähnlichen Zelldifferenzierung (HLC) entwickelt5,6,7,8. Diese Protokolle versuchen, Aspekte der menschlichen Leberentwicklung unter Verwendung einer Kombination von kleinen Molekülen und Wachstumsfaktoren9,10nachzubilden. Die meisten Protokolle bestehen aus einem schrittweisen Differenzierungsprozess, bei dem hPSCs auf das definitive Endoderm vorbereitet werden, gefolgt von der hepatischen Vorläuferspezifikation11,12,13und endet mit der HLC-Spezifikation. HLCs, die von diesen Protokollen produziert werden, zeigen eine Mischung aus fetalen und erwachsenen Phänotypen. Dazu gehören die Expression von Alpha-Fetoprotein (AFP), wie Hepatozytenmarker wie HNF4α und Albumin (ALB), sowie die Metabolisierungskapazität des Arzneimittels14,15,16. Zwischen den Laboren kann die HLC-Differenzierung variieren; daher ist die Entwicklung standardisierter Protokolle notwendig. Dies wird es den Forschern ermöglichen, stammzellbasierte HLCs in großem Maßstab effektiv für die Grundlagen- und klinische Forschung zu generieren und anzuwenden.
Es wurde ein hepatisches Vorläufer-Differenzierungssystem entwickelt, das sowohl auf humane embryonale als auch auf induzierte pluripotente Stammzelllinien unter Verwendung leicht zu befolgender Richtlinien angewendet werden kann. Dieses Verfahren liefert homogene Populationen von Lebervorläufern in unterschiedlichen Kulturmaterialformaten, die von Zellkulturkolben bis zu 96 Wellplatten reichen. Im Folgenden finden Sie das Protokoll zur Herstellung von stammzellbasierten leberischen Vorläufern in 24- und 96-Well-Formaten.
Die im folgenden Protokoll verwendete Zelldichte ist für eine Vertiefung einer 24- bzw. 96-Well-Platte spezifiziert (siehe Tabelle 1). Für die verschiedenen Zellkulturplattenformate und Zelllinien ist eine Optimierung der Ausgangszellnummer erforderlich. Die empfohlene Ausgangszelldichte für die Protokolloptimierung beträgt 2 x 105 Zellen/cm2. Zur Dichteoptimierung können mehrere Zelldichten getestet werden, indem ± 50.000Zellen/cm2 gleichzeitig hinzugefügt werden.
Protokoll
1. Aufrechterhaltung der humanen pluripotenten Stammzelle (hPSC) auf Laminin-521
- Halten Sie humane pluripotente Zellen (hPSCs) bei 37 °C und 5% CO2 in einer 6-Well-Platte auf Laminin-521 (LN-521). Füttern Sie die Zellen täglich mit 2 ml Stammzellerhaltungsmedium (d. H. mTeSR1-Medium) pro Vertiefung einer 6-Well-Platte bis zum gewählten Aussaattag zur Differenzierung (Tag 0).
- Stellen Sie sicher, dass die gewünschte Zellkonfluenz von 70-80% vor der Zellernte erreicht wird.
2. Laminin-521 Multiwell-Präparation und hPSC-Aussaat zur Differenzierung
HINWEIS: Für hPSCs, die nicht auf LN-521 gehalten werden (z. B. Matrigel oder Fibronektin), teilen Sie hPSCs auf LN-521 auf und kulturieren Sie sie für 1 Woche, bevor Sie eine Differenzierung vornehmen und eine Differenzierung hervorrufen, um die Effizienz des Prozesses zu verbessern15,17,18.
- Lamininbeschichtete Plattenzubereitung
- Auftauen Sie eine Durchstechflasche mit rekombinantem LN-521 (100 μg/ml) bei 4 °C für 2 h oder über Nacht.
- Eine 8 μg/ml Lösung wird durch Verdünnen des aufgetauten LN-521 in eiskaltem 1x DPBS mit Ca2+/Mg2+vorbereitet.
- 0,25 ml der 8 μg/ml LN-521-Lösung werden zu jeder Vertiefung einer 24-Well-Platte oder 0,05 ml zu jeder Vertiefung einer 96-Well-Platte hinzugefügt. Schaukeln Sie die Platte vorsichtig von einer Seite zur anderen, um die Vertiefungen gleichmäßig mit der LN-521-Lösung zu beschichten.
HINWEIS: Für das 96-Well-Plattenformat können Volumendosierung, Zellaussaat und Mediumwechsel mit einer halbautomatischen Rohrleitung durchgeführt werden. Für Details siehe Meseguer-Ripolles et al.19. - Versiegeln Sie die mit LN-521 beschichteten Platten mit einer halbtransparenten, flexiblen Folie und lagern Sie sie vor Gebrauch bei 4 °C über Nacht.
HINWEIS: LN-521 beschichtete Platten können bei Lagerung bei 4 °C bis zu 2 Wochen verwendet werden. Vermeiden Sie jegliches Austrocknen der mit Laminin beschichteten Vertiefungen.
- Am Tag der Zellaussaat die vorbeschichteten Platten in einem Zellkulturinkubator bei 37 °C für 30–60 min aufwärmen.
- Saugen Sie die LN-521-Lösung an.
HINWEIS: Vermeiden Sie den direkten Kontakt des Absaugers mit der Unterseite des Brunnens, um Schäden an der LN-521-Beschichtung zu vermeiden. - Geben Sie 0,5 ml Stammzellerhaltungsmedium mit frisch ergänztem 10 μM Rho-assoziiertem Kinase (ROCK)-Inhibitor Y27632 an jede Vertiefung einer 24-Well-Platte oder 0,05 ml an jede Vertiefung einer 96-Well-Platte. Legen Sie die Platte in den Inkubator, bis sie für die Zellaussaat bereit ist.
- Markieren Sie am geplanten Aussaattag (Tag 0) und mit einer hPSC-Konfluenz zwischen 70-80% alle Regionen mit spontaner Differenzierung auf der Unterseite der Vertiefungen der 6-Brunnenplatte.
HINWEIS: Spontane Differenzierung kann durch eine grobe Veränderung der Zellgröße und/oder das Vorhandensein verschiedener Zellmorphologien mittels Phasenkontrastmikroskopie visualisiert werden. - Saugen und verwerfen Sie die markierten Differenzierungsbereiche und das verbrauchte Medium aus den Vertiefungen. Waschen Sie jeden gut mit 1 ml DPBS ohne Ca2+/Mg2+ bei Raumtemperatur (RT).
- 1 ml enzymfreies Dissoziationsreagenz (siehe Materialtabelle)in jede Vertiefung geben und bei 37 °C für 8–10 min inkubieren, bis sich die Zellen sichtbar von der Platte lösen.
- Verwenden Sie einen Zellschaber, um die Zellen vorsichtig von den Vertiefungen zu lösen. Pipetten Sie den Inhalt jeder Vertiefung 2-4x mit einer P1000-Pipette nach oben und unten, um eine Einzelzellsuspension zu erhalten. Für jede Zelllinie werden Poolzellen aus allen Wartungsbrunnen in ein steriles 50-ml-Röhrchen eingeschmiert.
- Waschen Sie jedes entleerte gut mit 1 ml des Stammzellerhaltungsmediums. Geben Sie die Waschungen in das entsprechende Röhrchen, das die gepoolten Zellen aus der entsprechenden Zelllinie enthält.
- Führen Sie für jede Zelllinie drei lebensfähige Zellzählungen für die gepoolten Proben durch. Berechnen Sie die durchschnittliche Anzahl lebender Zellen (lebende Zellen/ml) für jede Zelllinie.
- Zentrifugieren Sie die gepoolten Proben bei 250 x g für 5 min bei RT. Saugen Sie den Überstand ab und resuspendieren Sie dann das Zellpellet in 1–3 ml RT-Stammzellerhaltungsmedium, frisch ergänzt mit 10 μM ROCK-Inhibitor Y27632.
- Berechnen Sie für jede Zelllinie die Zellzahl, die für die Anzahl der in Schritt 2.4 vorbereiteten Vertiefungen benötigt wird (siehe Tabelle 1). Resuspendieren Sie die erforderliche Zellzahl mit Stammzellerhaltungsmedium, das frisch mit 10 μM ROCK-Inhibitor Y27632 ergänzt wurde.
- Fügen Sie die berechneten Volumen in die Vertiefungen der vorbereiteten und vorbeschichteten Platten aus Schritt 2.4 ein, ohne das zuvor hinzugefügte Volumen zu entfernen. Das Gesamtvolumen pro Bohrung beträgt 1 ml für eine 24-Well-Platte und 0,1 ml für eine 96-Well-Platte. Schaukeln Sie die Platten vorsichtig von seite zu Seite und hin und her, um eine gleichmäßige Zelldispersion im gesamten Bohrbrunnen zu gewährleisten.
HINWEIS: Eine gleichmäßige Zellverteilung über den Brunnen ist der Schlüssel, um eine homogene Zellaussaat und eine erfolgreiche Differenzierung zu gewährleisten. - Legen Sie die ausgesäten Platten in den Inkubator und schaukeln Sie die ausgesäten Platten sofort sanft hin und her und von Seite zu Seite, um die Zellen gleichmäßig zu verteilen und die Kulturen bei 37 °C und 5%CO2zu halten.
3. Unterscheidung von hPSCs zu hepatischen Vorläufern auf Laminin-521
- Bereiten Sie Medien für die definitive Endoderm-Induktion (Stufe 1) mit dem Endoderm Basal Medium vor, das mit den richtigen Additiven ergänzt wird.
- Am Tag 0 die Flasche des Endoderm Basal Medium über Nacht bei 4 °C auftauen.
- Bereiten Sie Stufe 1 Medium 1 (für die Anwendung an Tag 1) nach Bedarf vor.
- Tauen Ergänzung MR und Ergänzung CJ auf Eis.
- Verdünnen Sie Supplement MR und Supplement CJ 1:100 im Endoderm Basal Medium.
- Bereiten Sie Stufe 1 Medium 2 für die Verwendung an den Tagen 2 bis 4 nach Bedarf vor.
- Verdünnen Sie Supplement CJ 1:100 im Endoderm Basal Medium.
- Bereiten Sie Medien für die anschließende Differenzierung der hepatischen Vorläuferzellenspezifikation (Stadium 2) mit dem Hepatic Progenitor Medium vor.
- Am 4. Tag die Flasche des Hepatic Progenitor Medium über Nacht bei 4 °C auftauen.
HINWEIS: Für dieses Experiment wurde 1% Penicillin/Streptomycin (Endkonzentrationen von 100 IE/ml bzw. 100 μg/ml) verwendet. Antibiotika sind nicht erforderlich; Die Verwendung von Antibiotika liegt im Ermessen des Benutzers.
- Am 4. Tag die Flasche des Hepatic Progenitor Medium über Nacht bei 4 °C auftauen.
- Entfernen Sie an Tag 1 der Differenzierung das verbrauchte Stammzellerhaltungsmedium mit 10 μM ROCKi Y-27632 Medium aus den Vertiefungen und ersetzen Sie es durch 0,5 ml komplettes Stadium 1 Medium 1 pro Vertiefung einer 24-Well-Platte und 0,1 ml pro Well einer 96-Well-Platte.
- Entfernen Sie an den Tagen 2, 3 und 4 das verbrauchte Medium und füttern Sie jede Vertiefung mit 0,5 ml Stufe 1 Medium 2 pro Vertiefung für eine 24-Well-Platte oder 0,1 ml pro Well einer 96-Well-Platte.
- Fixieren Sie an Tag 5 die Vertiefungen für die definitive Endodermdifferenzierungsanalyse mittels Immunzytochemie. Für die verbleibenden Vertiefungen entfernen Sie das verbrauchte Medium und füttern Sie jede Vertiefung mit 0,5 ml Hepatic Progenitor Differentiation Medium pro Well für eine 24-Well-Platte oder 0,1 ml pro Well einer 96-Well-Platte. Aktualisieren Sie das Medium an den Tagen 6, 7 und 9 erneut.
- Ernten Sie am 10. Tag Wells für die Hepatic Progenitor-Differenzierungsanalyse oder fahren Sie mit der weiteren hepatozytenähnlichen Zelldifferenzierung fort.
HINWEIS: Zu diesem Zeitpunkt wurden die Proben entweder mit 4% Paraformaldehyd (PFA) für die immunzytochemische Analyse fixiert oder Überstände für ELISA gesammelt und Zellen zur Proteinquantifizierung gesammelt.
4. Charakterisierung der hepatischen Vorläuferdifferenzierungskulturen, die aus hPSCs auf Laminin-521 erzeugt wurden
- Erkennen Sie an Tag 5 die Expression definitiver endodermspezifischer Marker mithilfe von Immunfärbung.
- Erkennen Sie am 10. Tag die Expression von hepatischen Vorläufer-spezifischen Markern mitHilfe von Immunfärbungen.
- Messen Sie an Tag 10 die AFP- und ALB-Sekretion über ELISA mit einem Kit gemäß den Anweisungen des Herstellers und normalisieren Sie pro mg Protein, wie durch einen Bicinchoninsäure (BCA) Proteinassay bestimmt.
- Beurteilen Sie die Variabilität des hepatischen Vorläufers der 96-Well-Platte durch Quantifizierung des Prozentsatzes der HNF4α-positiven Zellen pro Vertiefung.
5. Immunzytochemie und Bildaufnahme
- An den Tagen 5 und 10 der Differenzierung waschen Sie die Zellen 3x mit 1x DPBS, mit 0,5 ml pro Vertiefung einer 24-Well-Platte und 0,1 ml pro Well einer 96-Well-Platte. Inkubieren Sie die Platte mit sanftem Schütteln für 2-5 min bei RT.
HINWEIS: Verwenden Sie DPSB ohne Ca2+/Mg2+ für die Immunzytochemie. - Fixieren Sie die Zellen mit 4% Paraformaldehyd (PFA) bei RT für 15-30 min, indem Sie 0,3 ml PFA pro Vertiefung für eine 24-Well-Platte und 0,1 ml pro Well einer 96-Well-Platte hinzufügen.
- Waschen Sie 3x mit 1x DPBS wie in Schritt 5.1 beschrieben.
- Permeabilisieren Sie die Membran mit PBST unter Verwendung von 0,1% Tween, 1x DPBS und inkubieren Sie für 20 min bei RT, indem Sie 0,3 ml PBST pro Vertiefung einer 24-Well-Platte und 0,1 ml pro Well einer 96-Well-Platte hinzufügen.
- Führen Sie den Proteinblock durch, indem Sie die Zellen mit 10% BSA in PBST für 1 h inkubieren, 0,3 ml BSA pro Vertiefung einer 24-Well-Platte und 0,1 ml BSA pro Well einer 96-Well-Platte hinzufügen und sanft mit einem Plattenshatterer schütteln.
- Ersetzen Sie nach der Proteinblockierung die Blockierungslösung durch den primären Antikörper, der in 1% BSA in PBST verdünnt ist, und inkubieren Sie bei 4 °C mit sanftem Schütteln über Nacht.
HINWEIS: Waschen Sie nicht zwischen Proteinblock und Antikörperzugabe. - Nach 24 h Brunnen 3x mit PBST waschen.
- Fügen Sie den sekundären Antikörper in 1% BSA in PBST hinzu. 1 h bei RT im Dunkeln mit sanftem Schütteln inkubieren.
- Nach sekundärer Antikörperinkubation 3x mit 1x DPBS die Vertiefungen waschen und Hoechst Fleck gemäß Herstellerangaben für 10 min bei RT im Dunkeln mit sanftem Schütteln abgeben.
- 3x mit 1x DPBS waschen. Die Platten sind nun bereit für die Bildgebung.
HINWEIS: Lagern Sie die Platten bei 4 °C im Dunkeln bis zur Bildgebung. - Stellen Sie die Multiwell-Platte mit einem hochauflösenden Bildgebungsmikroskop nach der Immunhistochemie ab. Die Bildaufnahme mehrerer Sichtfelder wird empfohlen, um eine echte Darstellung des Bohrbrunnens zu erhalten. Die Expression der verschiedenen Marker wurde mittels Zellsegmentierungsanalyse mit kommerzieller Software bewertet (siehe Materialtabelle)(Abbildung 1).
HINWEIS: Die Zellsegmentierung kann auch mit einer Bildanalyse-Open-Source-Software wie CellProfiler oder Fiji20,21durchgeführt werden.
Ergebnisse
Die hepatische Vorläuferdifferenzierung von hESC (H9) und hiPSC (P106) Linien wurde nach dem in Abbildung 2beschriebenen schrittweisen Protokoll durchgeführt. Hier wurden pluripotente Stammzellen vor Beginn der Differenzierung als Einzelzellen in LN-521-beschichtete Platten eingesät. Zellkonfluenz ist der Schlüssel für eine robuste und reproduzierbare Differenzierung. Sobald die richtige Konfluenz erreicht war (Abbildung 2), wurde die Differenzierung eingeleitet. An Tag 5 wurde die definitive Endodermspezifikation anhand der Sox17-Expression beurteilt. In beiden Zelllinien war Sox17 mit 80% ± 0,5% bzw. 87,8% ± 0,5% REM von Sox17-positiven Zellen für H9 bzw. P106 hoch exprimiert(Abbildung 3). An Tag 10 zeigten hepatische Vorläufer eine kopfsteinpflasterartige Morphologie (Abbildung 2). Darüber hinaus wurde die Hepatische Vorläuferspezifikation für HNF4α-, AFP-, ALB- und Cytokeratin-19 (CK19)-Expression sowie AFP- und ALB-Proteinsekretion10,15,22 ( Abbildung4) bewertet. Sowohl H9- als auch P106-Stammstoffkulturen exprimierten fetale Lebermarker wie HNF4α (91% ± 0,5% und 90% ± 0,2%), AFP (89,7% ± 1,8% und 86% ± 1,2%) und CK19 (78,5% ± 3,2% und 83,6 ± 1,8%)(Abbildung 4). AFP-Sekretion wurde an Tag 10 in beiden Zelllinien nachgewiesen (32,4 ± 1,6 und 47,8 ± 5,9 ng/ml/mg/24 h)(Abbildung 5). Die Albuminsynthese wurde bei niedrigeren Konzentrationen (30,7% ± 1,8% und 27,2% ± 1,1%) beobachtet (Abbildung 4) und nicht über ELISA nachgewiesen (Abbildung 5).
Das Protokoll ermöglichte die standardisierte Produktion von Lebervorläufern von 24 Well- bis 96 Wellplatten. Eine halbautomatische Pipeline wurde verwendet, um 96 Wellplatten von hepatischen Vorläuferzellen aus H9- und P106-Zelllinien herzustellen, wie zuvor beschrieben17. Die Variabilität der Zellzahl und die Differenzierungseffizienz des hepatischen Vorläufers wurden durch Quantifizierung der HNF4α-Expression bewertet. Die Zellsegmentierung wurde zur Proteinquantifizierung mittels Immunfluoreszenz mit einem bildgebenden Instrument mit hohem Gehalt durchgeführt (Abbildung 1). An Tag 10 zeigten hepatische Vorläufer keine signifikante Variabilität über Reihen hinweg mit >94% der HNF4α-positiven Zellen pro Vertiefung für H9 und 97% HNF4α-positiven Zellen für P106 (Abbildung 6).

Abbildung 1: Übersicht über die Zellsegmentierungspipeline. (A) Unter Verwendung des Originalbildes wurde (B) kernige Färbung für die Kernsegmentierung verwendet. (C) Es wurde ein Qualitätskontrollschritt der Kernsegmentierung auf der Grundlage von Form und Größe durchgeführt, um nur klar segmentierte Kerne zu quantifizieren. (D) Anschließend wurden positive HNF4α-gefärbte Kerne quantifiziert. (E) Schließlich wurde ein intensitätsbasierter Schwellenwert verwendet, um HNF4α-exprimierende Zellen zu identifizieren. In C und Estellen grüne Kerne ausgewählte Zellen dar und magentafarbene Kerne zeigen verworfene Zellen an. Maßstabsleiste = 50 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Hepatische Vorläuferdifferenzierung von hPSCs. (A) Schematische Darstellung des hepatischen Vorläufer-Differenzierungsprotokolls. (B) Repräsentative Bilder, die die morphologischen Veränderungen während der Differenzierung hervorheben. An Tag 0 (D0) präsentierten hPSCs eine gepackte Monoschicht von Zellen. Anschließend wurden hPSCs am Tag 5 (D5) in das definitive Endoderm vorbereitet. Es folgte eine hepatische Vorläuferdifferenzierung am Tag 10 (D10). Hepatische Vorläufer zeigten eine kopfsteinsteinartigeZellmorphologie. Maßstabsleiste = 75 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
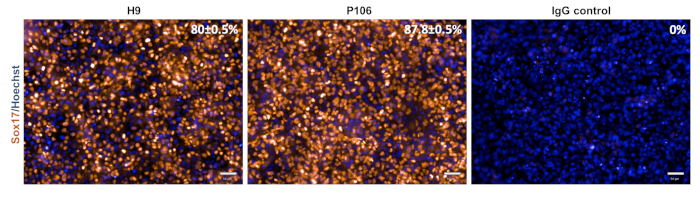
Abbildung 3: Charakterisierung der definitiven Endodermspezifikation. An Tag 5 wurden Die Zellen für Sox17, einen definitiven Endodermmarker, gefärbt. Der Prozentsatz der Sox17-positiven Zellen betrug 80 ± 0,5% für H9 und 87,8 ± 0,5% für P106. Die prozentuale Quantifizierung basierte auf 10 separaten Bohrfeldern mit 6 Sichtfeldern pro Bohrung. Die Daten werden als durchschnittliche ± REM angezeigt. Maßstabsbalken = 50 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: Charakterisierung des hepatischen Vorläufers. Am Tag 10 wurden hepatische Vorläufer für Lebermarker (A) HNF4α, (B) AFP und (C) ALB gefärbt. Für H9 betrug der Prozentsatz der positiven Zellen 91% ± 0,4%, 89,7% ± 1,8% und 30,7% ± 1,8% für HNF4α, AFP und ALB. Für P106 betrug der Prozentsatz der positiven Zellen 90% ± 0,2%, 86% +/- 1,2% und 27,2% ± 1,1% für HNF4α, AFP und ALB. (D) Das Cholangiozyten-Linienpotential wurde mittels CK19-Expression bewertet; H9-abgeleitete hepatische Vorläufer exprimierten 78,5% ± 3,2% CK19-positive Zellen, während 83,6% ± 1,8% der CK19-positiven Zellen für P106-Lebervorläufer beobachtet wurden. Immunglobulin G (IgG) Färbung wurde als Färbekontrolle verwendet. Die prozentuale Quantifizierung basierte auf 10 separaten Bohrfeldern mit 6 Sichtfeldern pro Bohrung. Die Daten werden als durchschnittliche ± REM angezeigt. Maßstabsbalken = 50 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 5: Analyse der Hepatischen Vorläuferproteinsekretion. Die Sekretion von Alpha-Fetoprotein (AFP) und Albumin (ALB) wurde in hepatischen Vorläuferkulturen an Tag 10 in H9 und P109 analysiert. Die Daten repräsentieren drei biologische Replikate und die Fehlerbalken repräsentieren die SD. Sezernierte Proteine wurden aus 24 h Kulturmedium als Nanogramm sezerniertes Protein pro ml pro mg Protein quantifiziert, n = 3; ND = nicht erkannt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
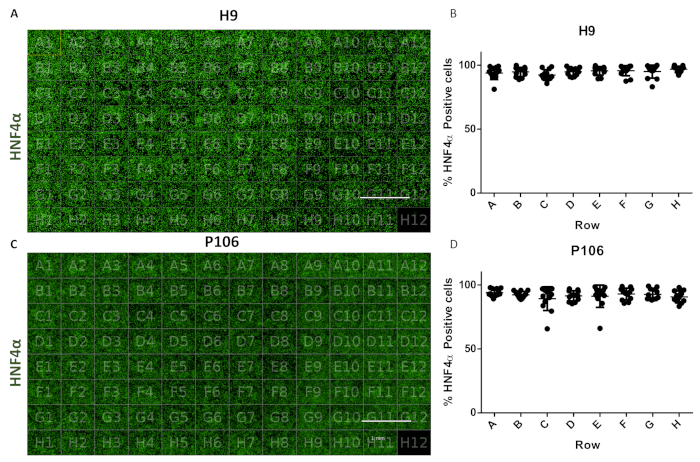
Abbildung 6: Bewertung der Well-to-Well-Variabilität in 96 Well-Platten. (A) Visualisierung einer 96-Well-Plattenansicht von H9-abgeleiteten leberartigen Vorläufern, die mit HNF4α gefärbt sind. (B) Quantifizierung der HNF4α-positiven Zellen. Durchschnitt der Zellzahl pro Vertiefung in Zeilen, aus sechs Sichtfeldern pro quantifiziertem Bohrfeld. Die durchschnittliche Zellzahl auf der Platte betrug 94,81% ± 0,22 SEM HNF4α-positiven Zellen pro Vertiefung. Es wurden keine statistisch signifikanten Unterschiede zwischen den Bohrungen beobachtet. (C) Visualisierung einer 96-Well-Plattenansicht von P106-abgeleiteten leberartigen Vorläufern, die mit HNF4α gefärbt sind. (D) Quantifizierung von HNF4α-positiven Zellen. Die durchschnittliche Zellzahl pro Vertiefung in Reihen, aus sechs Sichtfeldern pro Vertiefung und quantifiziert. Die durchschnittliche Zellzahl über die Platte betrug 97,7% ± 0,57 SEM HNF4α-positiven Zellen pro Vertiefung. Es wurden keine statistisch signifikanten Unterschiede zwischen den Zeilen beobachtet. Nun, H12 wurde als Immunglobulin G (IgG) Färbekontrolle verwendet. Maßstabsleiste = 1 mm. Es wurden Einweg-ANOVA mit Tukeys post-hoc-statistischen Tests verwendet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Plattenformat | Fläche (cm2) | Zellen pro cm2 | Gesamtzellen pro Vertiefung | Dosiervolumen (ml) | Zellkonzentration (Zellen/ml) |
| 24-Well-Platte | 1.9 | 210526 | 400000 | 0.5 | 800000 |
| 96-Well-Platte | 0.32 | 187500 | 60000 | 0.05 | 1200000 |
Tabelle 1: Empfohlene Zelldichte für die verschiedenen Plattenformate für die in diesem Protokoll verwendeten hPSC-Zelllinien.
Diskussion
Die Generierung von humanen leberischen Vorläuferzellen aus pluripotenten Stammzellen in großem Maßstab könnte eine vielversprechende Alternative zu Kadavermaterial darstellen. Protokollstandardisierung und Reproduzierbarkeit sind der Schlüssel, um die Technologieübersetzung und -wirkung für die biomedizinische Forschung sicherzustellen. Um dies zu beheben, konzentrierten sich frühere Arbeiten auf die Entwicklung eines schrittweisen Differenzierungsprotokolls von hESC und iPSCs unter Verwendung definierter Additive und Matrizen15,23,24,25,26,27,28. Auf diese Weise wurden der Hepatozytenphänotyp und die Reproduzierbarkeit verbessert, was eine Halbautomatisierung des Differenzierungsprozesses ermöglicht19. Das vorgestellte System wird durch die Kombination mit handelsüblichen Zellkulturmedien und einem einfachen Hepatozyten-Differenzierungssystem verstärkt.
Zuvor wurde die pluripotente Zelldichte vor Beginn des Differenzierungsprotokolls als Schlüsselvariable hervorgehoben, um eine homogene Population von hepatischen Vorläuferzellen zu erreichen26. Mit diesem verfeinerten Verfahren ist es möglich, eine große Anzahl von stammzellbasierten leberlichen Vorläufern schrittweise unter Verwendung einer Reihe von Ausgangszelldichten zu erzeugen (Tabelle 1). An Tag 5 wurde die definitive Endoderminduktion durch Sox17-Färbung validiert (Abbildung 3). Eine effiziente und robuste Differenzierung in definitives Endoderm wurde sowohl mit getesteten ESC- als auch mit iPSC-Linien erreicht, wobei mehr als 80% Sox17 exprimiert wurden (Abbildung 3). An Tag 10 zeigten hepatische Vorläufer eine einheitliche kopfsteinsteinartige Morphologie, und Leberstammzellmarker waren sowohl für AFP als auch für HNF4α stark angereichert (>86%, Abbildung 4). Mit einer Kombination aus manuellen und halbautomatischen Technologien war es möglich, in mehreren Plattenformaten zu differenzieren19.
In ihrer jetzigen Form eignet sich die Zelldifferenzierung für In-vitro-basierte Experimente. Eine Zellanreicherung wäre jedoch wahrscheinlich vor der klinischen Anwendung erforderlich, um sicherzustellen, dass eine homogene Population von Lebervorläufern für die Entbindung vorbereitet wird.
Zusammenfassend lässt sich sagen, dass das hier beschriebene Protokoll dem Feld einen standardisierten Ansatz bietet, um hepatische Vorläufer in großem Maßstab herzustellen. Zukünftige Arbeiten werden sich auf die Herstellung eines neuen Mediums für die anschließende HLC-Differenzierung, -Reifung und -Wartung konzentrieren.
Offenlegungen
David C. Hay ist Mitbegründer und Aktionär von Stemnovate Ltd. Der Rest der Autoren bestätigt, dass sie keine Interessenkonflikte in dem in diesem Artikel behandelten Thema oder Material haben.
Danksagungen
Diese Studie wurde mit Preisen der MRC Doctoral Training Partnership (MR/K501293/1), der UK Regenerative Medicine Platform (MRC MR/L022974/1 und MR/K026666/1), des Chief Scientist Office (TCS/16/37) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| DPBS with Calcium and Magnesium | ThermoFisher | 14040133 | |
| Gentle cell dissociation reagent | STEMCELL Technologies | 7174 | |
| Hoechst 33342 Ready Flow Reagent | thermofisher | R37165 | |
| Human Recombinant Laminin 521 | BioLamina | LN521-02 | |
| Human Serum Albumin ELISA | Alpha Diagnostics | 1190 | |
| Human Serum Alpha Fetoprotein ELISA | Alpha Diagnostics | 500 | |
| mTeSR1 medium | STEMCELL Technologies | 5850 | |
| Operetta High-Content Imaging System | PerkinElmer | HH12000000 | |
| PBS, no calcium, no magnesium | ThermoFisher | 14190250 | |
| Penicillin-Streptomycin (10,000 U/mL) | Life Technologies | 15140122 | |
| Rho-associated kinase (ROCK)inhibitor Y27632 | Sigma-Aldrich | Y0503-1MG | |
| STEMdiff Definitive Endoderm Supplement CJ | STEMCELL Technologies | ||
| STEMdiff Definitive Endoderm Supplement MR | STEMCELL Technologies | ||
| STEMdiff Endoderm Basal Medium | STEMCELL Technologies | ||
| STEMdiff Hepatic Progenitor Medium | STEMCELL Technologies | ||
| TWEEN 20 | Sigma-Aldrich | P9416 | |
| Antibodies | |||
| Albumin | Sigma-Aldrich | A6684 | 1:200 (mouse) |
| Alpha-fetoprotein | Sigma-Aldrich | A8452 | 1:400 (mouse) |
| HNF-4α | Santa Cruz | sc-8987 | 1:400 (rabbit) |
| IgG | DAKO | 1:400 | |
| Sox17 | R&D Systems, Inc. | AF1924 | 1:200 (Goat) |
| Software | |||
| Columbus Image Data Storage and Analysis system | PerkinElmer |
Referenzen
- Asrani, S. K., Devarbhavi, H., Eaton, J., Kamath, P. S. Burden of liver diseases in the world. Journal of Hepatology. 70 (1), 151-171 (2019).
- Szkolnicka, D., Hay, D. C. Concise Review: Advances in Generating Hepatocytes from Pluripotent Stem Cells for Translational Medicine. Stem Cells Dayton Ohio. 34 (6), 1421-1426 (2016).
- Heslop, J. A., Duncan, S. A. The Use of Human Pluripotent Stem Cells for Modeling Liver Development and Disease. Hepatology. 69 (3), 1306-1316 (2019).
- Alwahsh, S. M., Rashidi, H., Hay, D. C. Liver cell therapy: is this the end of the beginning. Cell and Molecular Life Sciences. 75 (8), 1307-1324 (2018).
- Hay, D. C., et al. Efficient differentiation of hepatocytes from human embryonic stem cells exhibiting markers recapitulating liver development in vivo. Stem Cells Dayton Ohio. 26 (4), 894-902 (2008).
- Si-Tayeb, K., et al. Highly efficient generation of human hepatocyte-like cells from induced pluripotent stem cells. Hepatology. 51 (1), 297-305 (2010).
- Hannan, N. R. F., Segeritz, C. -. P., Touboul, T., Vallier, L. Production of hepatocyte-like cells from human pluripotent stem cells. Nature Protocols. 8 (2), 430-437 (2013).
- Basma, H., et al. Differentiation and transplantation of human embryonic stem cell-derived hepatocytes. Gastroenterology. 136 (3), 990-999 (2009).
- Meseguer-Ripolles, J., Khetani, S. R., Blanco, J. G., Iredale, M., Hay, D. C. Pluripotent Stem Cell-Derived Human Tissue: Platforms to Evaluate Drug Metabolism and Safety. The AAPS Journal. 20 (1), 20 (2017).
- Si-Tayeb, K., Lemaigre, F. P., Duncan, S. A. Organogenesis and development of the liver. Developmental Cell. 18 (2), 175-189 (2010).
- D'Amour, K. A., et al. Efficient differentiation of human embryonic stem cells to definitive endoderm. Nature Biotechnology. 23 (12), 1534-1541 (2005).
- Shin, D., et al. Bmp and Fgf signaling are essential for liver specification in zebrafish. Development Cambridge England. 134 (11), 2041-2050 (2007).
- DeLaForest, A., et al. HNF4A is essential for specification of hepatic progenitors from human pluripotent stem cells. Development Cambridge England. 138 (19), 4143-4153 (2011).
- Baxter, M., et al. Phenotypic and functional analyses show stem cell-derived hepatocyte-like cells better mimic fetal rather than adult hepatocytes. Journal of Hepatology. 62 (3), 581-589 (2015).
- Cameron, K., et al. Recombinant Laminins Drive the Differentiation and Self-Organization of hESC-Derived Hepatocytes. Stem Cell Reports. 5 (6), 1250-1262 (2015).
- Szkolnicka, D., et al. Reducing Hepatocyte Injury and Necrosis in Response to Paracetamol Using Noncoding RNAs. Stem Cells Translational Medicine. 5 (6), 764-772 (2016).
- Domogatskaya, A., Rodin, S., Boutaud, A., Tryggvason, K. Laminin-511 but Not -332, -111, or -411 Enables Mouse Embryonic Stem Cell Self-Renewal In Vitro. Stem Cells. 26 (11), 2800-2809 (2008).
- Kanninen, L. K., et al. Laminin-511 and laminin-521-based matrices for efficient hepatic specification of human pluripotent stem cells. Biomaterials. 103, 86-100 (2016).
- Meseguer-Ripolles, J., Lucendo-Villarin, B., Wang, Y., Hay, D. C. Semi-automated Production of Hepatocyte Like Cells from Pluripotent Stem Cells. Journal of Visualized Experiments. (137), e57995 (2018).
- Carpenter, A. E., et al. CellProfiler: image analysis software for identifying and quantifying cell phenotypes. Genome Biology. 7, 100 (2006).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Raven, A., et al. Cholangiocytes act as Facultative Liver Stem Cells during Impaired Hepatocyte Regeneration. Nature. 547 (7663), 350-354 (2017).
- Hay, D. C., et al. Unbiased screening of polymer libraries to define novel substrates for functional hepatocytes with inducible drug metabolism. Stem Cell Research. 6 (2), 92-102 (2011).
- Medine, C. N., et al. Developing High-Fidelity Hepatotoxicity Models from Pluripotent Stem Cells. Stem Cells Translational Medicine. 2 (7), 505-509 (2013).
- Szkolnicka, D., et al. Accurate prediction of drug-induced liver injury using stem cell-derived populations. Stem Cells Translational Medicine. 3 (2), 141-148 (2014).
- Wang, Y., et al. Defined and Scalable Generation of Hepatocyte-like Cells from Human Pluripotent Stem Cells. Journal of Visualized Experiments. (121), e55355 (2017).
- Villarin, B. L., et al. Polymer Supported Directed Differentiation Reveals a Unique Gene Signature Predicting Stable Hepatocyte Performance. Advanced Healthcare Materials. 4 (12), 1820-1825 (2015).
- Wang, Y., et al. Multiomics Analyses of HNF4α Protein Domain Function during Human Pluripotent Stem Cell Differentiation. iScience. 16, 206-217 (2019).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten