Method Article
Spécification du progéniteur hépatique à partir de cellules souches pluripotentes à l’aide d’un système de différenciation défini
Dans cet article
Résumé
L’objectif de cet article est de fournir une approche standardisée pour induire la différenciation des progéniteurs hépatiques humains à partir de cellules souches pluripotentes. Le développement de cette procédure avec des formulations de milieux prêtes à l’emploi offre à l’utilisateur un système facile pour générer des cellules hépatiques humaines pour la recherche biomédicale et la traduction.
Résumé
La maladie du foie est un problème de santé mondial croissant. Bien que la transplantation hépatique soit un mode de traitement efficace, la mortalité des patients a augmenté en raison de la pénurie de disponibilité des organes des donneurs. La rareté des organes affecte également l’approvisionnement de routine en hépatocytes humains pour la recherche fondamentale et la clinique. Par conséquent, le développement de sources renouvelables de cellules progéniatrices hépatiques humaines est souhaitable et constitue l’objectif de cette étude. Pour pouvoir générer et déployer efficacement des progéniteurs hépatiques humains à grande échelle, un système de différenciation hépatique reproductible des progéniteurs a été développé. Ce protocole facilite la reproductibilité expérimentale entre les utilisateurs dans une gamme de formats de logiciels de culture cellulaire et permet des différenciations en utilisant à la fois des lignées de cellules souches embryonnaires humaines et pluripotentes induites. Ce sont des avantages importants par rapport aux systèmes de différenciation actuels qui amélioreront la recherche fondamentale et pourraient ouvrir la voie au développement de produits cliniques.
Introduction
Les maladies du foie représentent un défi sanitaire mondial, causant environ 2 millions de décès par an dans le monde1. Bien qu’il existe un certain nombre de systèmes modèles pour étudier les maladies hépatiques et intervenir cliniquement, l’utilisation systématique de systèmes cellulaires est limitée par des inconvénients importants (pour une revue, voir Szkolnicka et al.2). Les méthodes avancées de culture de cellules souches pluripotentes humaines (hPSC) et de différenciation des cellules somatiques représentent des technologies prometteuses pour développer des outils pour la recherche biomédicale fondamentale et des sources renouvelables de cellules différenciées pour la clinique3,4.
À ce jour, de multiples protocoles de différenciation des cellules de type hépatocytaire (HLC) ont étédéveloppés5,6,7,8. Ces protocoles tentent de recréer des aspects du développement du foie humain en utilisant une combinaison de petites molécules et de facteurs de croissance9,10. La plupart des protocoles consistent en un processus de différenciation par étapes, où les hPSC sont amorcés à l’endoderme définitif, suivi de la spécification du progéniteur hépatique11,12,13, et se terminant par la spécification HLC. Les HPC produits par ces protocoles présentent un mélange de phénotypes fœtaux et adultes. Cela inclut l’expression de l’alpha fœtoprotéine (AFP), tels que les marqueurs hépatocytaires tels que HNF4α et l’albumine (ALB), ainsi que la capacité de métabolisation desmédicaments 14,15,16. D’un laboratoire à l’autre, la différenciation des HLC peut varier; par conséquent, l’élaboration de protocoles normalisés est nécessaire. Cela permettra aux chercheurs de générer et d’appliquer efficacement des HPC dérivés de cellules souches à grande échelle pour la recherche fondamentale et clinique.
Un système de différenciation des progéniteurs hépatiques a été développé qui peut être appliqué à la fois à des lignées de cellules souches embryonnaires humaines et pluripotentes induites en utilisant des lignes directrices faciles à suivre. Cette procédure donne des populations homogènes de progéniteurs hépatiques dans différents formats de culture, allant des flacons de culture cellulaire aux plaques de 96 puits. Vous trouverez ci-dessous le protocole pour produire des progéniteurs hépatiques dérivés de cellules souches dans des formats de puits 24 et 96.
La densité cellulaire utilisée dans le protocole présenté ci-dessous est spécifiée pour un puits d’une plaque de 24 et 96 puits respectivement (voir tableau 1). L’optimisation du numéro de cellule de départ est nécessaire pour les différents formats de plaques de culture cellulaire et les lignées cellulaires. La densité de cellule de départ suggérée pour l’optimisation du protocole est de 2 x10 5 cellules/cm2. Pour l’optimisation de la densité, plusieurs densités de cellules peuvent être testées en ajoutant ± 50 000 cellules/cm2 à la fois.
Protocole
1. Maintien des cellules souches pluripotentes humaines (CSEh) sur la laminine-521
- Maintenir les cellules pluripotentes humaines (CSEh) à 37 °C et 5 % de CO2 dans une plaque de 6 puits sur la laminine-521 (LN-521). Nourrissez les cellules quotidiennement avec 2 mL de milieu d’entretien des cellules souches (c.-à-d. milieu mTeSR1) par puits d’une plaque de 6 puits jusqu’au jour d’ensemencement choisi pour la différenciation (jour 0).
- Assurez-vous que la confluence cellulaire souhaitée de 70 à 80 % est atteinte avant la récolte cellulaire.
2. Préparation multipuits de laminine-521 et ensemencement hPSC pour la différenciation
REMARQUE: Pour les CSEh non maintenus sur LN-521 (par exemple, matrigel ou fibronectine), divisez les HSCC sur LN-521 et les culturez pendant 1 semaine avant de passer et de provoquer une différenciation pour améliorer l’efficacité du processus15,17,18.
- Préparation de plaques revêtues de laminine
- Décongeler un flacon de LN-521 recombinant (100 μg/mL) à 4 °C pendant 2 h ou toute la nuit.
- Préparer une solution de 8 μg/mL en diluant le LN-521 décongelé dans du DPBS glacé 1x avec du Ca2+/Mg2+.
- Ajouter 0,25 mL de la solution de LN-521 de 8 μg/mL à chaque puits d’une plaque de 24 puits ou 0,05 mL à chaque puits d’une plaque de 96 puits. Bercez doucement la plaque d’un côté à l’autre pour recouvrir uniformément les puits avec la solution LN-521.
REMARQUE: Pour le format de plaque de 96 puits, la distribution de volume, l’ensemencement de cellules et les changements de milieu peuvent être effectués à l’aide d’un pipeline semi-automatisé. Pour plus de détails, voir Meseguer-Ripolles et al.19. - Scellez les plaques revêtues de LN-521 avec un film semi-transparent et flexible et conservez-les à 4 °C pendant la nuit avant utilisation.
REMARQUE: Les plaques revêtues de LN-521 peuvent être utilisées jusqu’à 2 semaines lorsqu’elles sont stockées à 4 ° C. Évitez tout séchage des puits recouverts de laminine.
- Le jour de l’ensemencement cellulaire, réchauffez les plaques prélaquées dans un incubateur de culture cellulaire à 37 °C pendant 30 à 60 min.
- Aspirez la solution LN-521.
REMARQUE: Évitez le contact direct de l’aspirateur avec le fond du puits pour éviter d’endommager le revêtement LN-521. - Distribuer 0,5 mL de milieu d’entretien des cellules souches avec un inhibiteur de la kinase rho-associée (ROCK) Y27632 fraîchement supplémenté de 10 μM à chaque puits d’une plaque de 24 puits ou 0,05 mL à chaque puits d’une plaque de 96 puits. Placez la plaque dans l’incubateur jusqu’à ce qu’elle soit prête pour l’ensemencement cellulaire.
- Le jour d’ensemencement prévu (jour 0) et avec une confluence hPSC comprise entre 70 et 80%, marquez toutes les régions de différenciation spontanée au fond des puits de la plaque de 6 puits.
REMARQUE: La différenciation spontanée peut être visualisée par un changement brut de la taille des cellules et / ou la présence de différentes morphologies cellulaires en utilisant la microscopie à contraste de phase. - Aspirer et écarter les régions marquées de différenciation et le milieu usé des puits. Laver chaque puits avec 1 mL de DPBS sans Ca2+/Mg2+ à température ambiante (RT).
- Ajouter 1 mL de réactif de dissociation sans enzyme (voir Tableau des matériaux)à chaque puits et incuber à 37 °C pendant 8 à 10 min jusqu’à ce que les cellules se détachent visiblement de la plaque.
- Utilisez un grattoir à cellules pour détacher doucement les cellules des puits. Pipettez le contenu de chaque puits de haut en bas de 2 à 4 fois avec une pipette P1000 pour obtenir une suspension à cellule unique. Pour chaque lignée cellulaire, regrouper les cellules de tous les puits d’entretien dans un tube stérile de 50 mL.
- Laver chaque puits vidé avec 1 mL du milieu d’entretien des cellules souches. Ajouter les lavages au tube correspondant contenant les cellules regroupées de la lignée cellulaire appropriée.
- Pour chaque lignée cellulaire, effectuez trois comptages de cellules viables sur les échantillons regroupés. Calculer le nombre moyen de cellules vivantes (cellules vivantes/mL) pour chaque lignée cellulaire.
- Centrifuger les échantillons regroupés à 250 x g pendant 5 min à RT. Aspirer le surnageant, puis ressuspend la pastille cellulaire dans 1 à 3 mL de milieu d’entretien des cellules souches RT, fraîchement complété par un inhibiteur de ROCK Y27632 de 10 μM.
- Pour chaque lignée cellulaire, calculer le nombre de cellules nécessaires en fonction du nombre de puits préparés à l’étape 2.4 (voir tableau 1). Resuspendez le nombre de cellules requis avec un milieu d’entretien de cellules souches fraîchement complété par un inhibiteur de ROCK Y27632 de 10 μM.
- Ajouter le(s) volume(s) calculé(s) dans les puits des plaques préparées et prélaquées à partir de l’étape 2.4 sans enlever le volume ajouté précédemment. Le volume total par puits sera de 1 mL pour une plaque de 24 puits et de 0,1 mL pour une plaque de 96 puits. Bercez doucement les plaques d’un côté à l’autre et d’avant en arrière pour assurer une dispersion uniforme des cellules dans tout le puits.
REMARQUE: Une distribution cellulaire uniforme à travers le puits est essentielle pour assurer un ensemencement cellulaire homogène et une différenciation réussie. - Placez les plaques ensemencées dans l’incubateur et bercez immédiatement les plaques ensemencées doucement d’avant en arrière et d’un côté à l’autre pour répartir uniformément les cellules et maintenir les cultures à 37 °C et 5% de CO2.
3. Différencier les CSEh des progéniteurs hépatiques sur la laminine-521
- Préparer le milieu pour l’induction définitive de l’endoderme (stade 1) en utilisant le milieu basal Endoderm complété par les additifs appropriés.
- Le jour 0, décongeler le flacon de l’Endoderm Basal Medium pendant la nuit à 4 °C.
- Préparez l’étape 1 Moyenne 1 (à utiliser le jour 1) au besoin.
- Supplément de décongélation MR et Supplément CJ sur glace.
- Diluer le supplément MR et le supplément CJ 1:100 dans le milieu basal endodermique.
- Préparez l’étape 1 Moyen 2 pour une utilisation aux jours 2 à 4 au besoin.
- Supplément dilué CJ 1:100 dans le milieu basal endodermique.
- Préparer le milieu pour la différenciation ultérieure de la spécification des cellules progénitaires hépatiques (stade 2) à l’aide du milieu progéniteur hépatique.
- Le jour 4, décongeler la bouteille du milieu progéniteur hépatique pendant la nuit à 4 °C.
NOTE : 1 % de pénicilline/streptomycine (concentrations finales de 100 UI/mL et 100 μg/mL, respectivement) a été utilisé pour cette expérience. Les antibiotiques ne sont pas nécessaires; l’utilisation d’antibiotiques est à la discrétion de l’utilisateur.
- Le jour 4, décongeler la bouteille du milieu progéniteur hépatique pendant la nuit à 4 °C.
- Au jour 1 de la différenciation, retirer du milieu d’entretien des cellules souches usé avec un milieu ROCKi Y-27632 de 10 μM et remplacer par 0,5 mL de milieu complet de stade 1 1 par puits d’une plaque de 24 puits et 0,1 mL par puits d’une plaque de 96 puits.
- Aux jours 2, 3 et 4, retirez le milieu usé et alimentez chaque puits avec 0,5 mL de milieu de stade 1 2 par puits pour une plaque de 24 puits ou 0,1 mL par puits d’une plaque de 96 puits.
- Le jour 5, fixez les puits pour une analyse définitive de la différenciation des endodermes via l’immunocytochimie. Pour les puits restants, retirer le milieu usé et alimenter chaque puits avec 0,5 mL de milieu de différenciation hépatique par puits pour une plaque de 24 puits ou 0,1 mL par puits d’une plaque de 96 puits. Actualisez à nouveau le support les jours 6, 7 et 9.
- Le jour 10, récoltez des puits pour une analyse de différenciation du progéniteur hépatique ou procédez à une différenciation cellulaire de type hépatocytaire.
REMARQUE: À ce stade, les échantillons ont été fixés avec 4% de paraformaldéhyde (PFA) pour l’analyse immunocytochimique ou un surnageant a été recueilli pour ELISA et des cellules ont été collectées pour la quantification des protéines.
4. Caractérisation des cultures de différenciation des progéniteurs hépatiques générées à partir de CSEh sur la laminine-521
- Le jour 5, détectez l’expression de marqueurs endodermiques définitifs à l’aide de l’immunocoloration.
- Le jour 10, détectez l’expression de marqueurs spécifiques du progéniteur hépatique à l’aide de l’immunocoloration.
- Le jour 10, mesurez la sécrétion d’AFP et d’ALB via ELISA à l’aide d’un kit en suivant les instructions du fabricant et normalisez par mg de protéines déterminées par un test de protéines d’acide bicinchoninique (BCA).
- Évaluer la variabilité hépatique du progéniteur de la plaque de 96 puits en quantifiant le pourcentage de cellules HNF4α positives par puits.
5. Immunocytochimie et acquisition d’images
- Les jours 5 et 10 de la différenciation, laver les cellules 3x avec 1x DPBS, avec 0,5 mL par puits d’une plaque de 24 puits et 0,1 mL par puits d’une plaque de 96 puits. Incuber la plaque en agitant doucement pendant 2 à 5 minutes à RT.
REMARQUE: Utilisez DPSB sans Ca2+/ Mg2 + pour l’immunocytochimie. - Fixer les cellules avec 4% de paraformaldéhyde (PFA) à RT pendant 15 à 30 min en ajoutant 0,3 mL de PFA par puits pour une plaque de 24 puits et 0,1 mL par puits d’une plaque de 96 puits.
- Lavez 3x avec 1x DPBS comme décrit à l’étape 5.1.
- Perméabiliser la membrane avec du PBST à l’aide de 0,1 % de interpolation, 1x DPBS et incuber pendant 20 min à RT en ajoutant 0,3 mL de PBST par puits d’une plaque de 24 puits et 0,1 mL par puits d’une plaque de 96 puits.
- Effectuer le bloc protéique en incubant les cellules avec 10% de BSA dans pbST pendant 1 h, en ajoutant 0,3 mL de BSA par puits d’une plaque de 24 puits et 0,1 mL de BSA par puits d’une plaque de 96 puits, en agitant doucement à l’aide d’un agitateur de plaque.
- Après blocage des protéines, remplacer la solution bloquante par l’anticorps primaire dilué dans 1% de BSA dans le PBST et incuber à 4 °C en agitant doucement pendant la nuit.
REMARQUE: Ne pas laver entre le bloc de protéines et l’ajout d’anticorps. - Après 24 h, laver les puits 3x avec PBST.
- Ajouter l’anticorps secondaire dans 1% de BSA dans le PBST. Incuber 1 h à RT dans l’obscurité en secouant doucement.
- Après l’incubation secondaire des anticorps, lavez les puits 3x avec 1x DPBS et distribuez la tache Hoechst selon les instructions du fabricant pendant 10 min à TA dans l’obscurité en secouant doucement.
- Laver 3x avec 1x DPBS. Les plaques sont maintenant prêtes pour l’imagerie.
REMARQUE: Conservez les plaques à 4 °C dans l’obscurité jusqu’à l’imagerie. - Imagez la plaque multipuit à l’aide d’un microscope d’imagerie à haute teneur après immunohistochimie. L’acquisition d’images de plusieurs champs de vision est recommandée pour obtenir une représentation fidèle du puits. L’expression des différents marqueurs a été évaluée par analyse de segmentation cellulaire à l’aide d’un logiciel commercial (voir Tableau des matériaux)(Figure 1).
REMARQUE: La segmentation cellulaire peut également être effectuée à l’aide d’un logiciel open source d’analyse d’images tel que CellProfiler ou Fiji20,21.
Résultats
La différenciation des progéniteurs hépatiques à partir des lignes hESC (H9) et hiPSC (P106) a été effectuée selon le protocole par étapes décrit à la figure 2. Ici, les cellules souches pluripotentes ont été ensemencées en tant que cellules uniques dans des plaques revêtues de LN-521 avant le début de la différenciation. La confluence cellulaire est la clé d’une différenciation robuste et reproductible. Une fois la bonne confluence atteinte(Figure 2), ladifférenciation a été initiée. Au jour 5, la spécification définitive de l’endoderme a été évaluée via l’expression de Sox17. Dans les deux lignées cellulaires, Sox17 a été fortement exprimé avec 80 % ± 0,5 % et 87,8 % ± 0,5 % de MEB de cellules Sox17 positives pour H9 et P106, respectivement(Figure 3). Au jour 10, les progéniteurs hépatiques présentaient une morphologie pavée (Figure 2). En outre, la spécification du progéniteur hépatique a été évaluée pour l’expression de HNF4α, AFP, ALB et cytokératine-19 (CK19) ainsi que pour la sécrétion de protéines AFP et ALB10,15,22 ( Figure4). Les cultures de progéniteurs hépatiques H9 et P106 ont exprimé des marqueurs hépatiques fœtaux tels que HNF4α (91 % ± 0,5 % et 90 % ± 0,2 %), AFP (89,7 % ± 1,8 % et 86 % ± 1,2 %) et CK19 (78,5 % ± 3,2 % et 83,6 ± 1,8 %)(Figure 4). La sécrétion d’AFP a été détectée au jour 10 dans les deux lignées cellulaires (32,4 ± 1,6 et 47,8 ± 5,9 ng/mL/mg/24 h)(Figure 5). La synthèse de l’albumine a été observée à des niveaux plus faibles (30,7 % ± 1,8 % et 27,2 % ± 1,1 %)(Figure 4)et n’a pas été détectée par ELISA (Figure 5).
Le protocole a permis la production standardisée de progéniteurs hépatiques de 24 puits à 96 plaques de puits. Un pipeline semi-automatisé a été utilisé pour produire 96 plaques de puits de progéniteurs hépatiques à partir de lignées cellulaires H9 et P106 comme décrit précédemment17. La variabilité du nombre de cellules et l’efficacité de la différenciation des progéniteurs hépatiques ont été évaluées par quantification de l’expression de HNF4α. La segmentation cellulaire a été réalisée pour la quantification des protéines par immunofluorescence à l’aide d’un instrument d’imagerie à haute teneur(Figure 1). Au jour 10, les progéniteurs hépatiques n’ont montré aucune variabilité significative entre les rangées avec >94 % de cellules HNF4α positives par puits pour H9 et 97 % de cellules HNF4α positives pour P106(Figure 6).

Figure 1: Vued’ensemble du pipeline de segmentation cellulaire. (A) En utilisant l’image originale, (B) la coloration nucléaire a été utilisée pour la segmentation des noyaux. (C) Une étape de contrôle de la qualité de la segmentation nucléaire basée sur la forme et la taille a été effectuée pour quantifier uniquement les noyaux clairement segmentés. (D) Par la suite, des noyaux colorés HNF4α positifs ont été quantifiés. (E) Enfin, un seuil basé sur l’intensité a été utilisé pour identifier les cellules exprimant HNF4α. En C et E,les noyaux verts représentent des cellules sélectionnées et les noyaux magenta indiquent des cellules rejetées. Barre d’échelle = 50 μm. Veuillez cliquer ici pour afficher une version agrandie de cette figure.

Figure 2: Différenciation du progéniteur hépatique par rapport aux HSPC. (A) Représentation schématique du protocole de différenciation du progéniteur hépatique. (B) Images représentatives mettant en évidence les changements morphologiques au cours de la différenciation. Au jour 0 (D0), les cSEh ont présenté une monocouche de cellules emballée. Par la suite, les CSEh ont été amorcés dans l’endoderme définitif le jour 5 (D5). Cela a été suivi d’une différenciation hépatique des progéniteurs au jour 10 (D10). Les progéniteurs hépatiques présentaient une morphologie cellulairepavée. Barre d’échelle = 75 μm. Veuillez cliquer ici pour voir une version plus grande de cette figure.
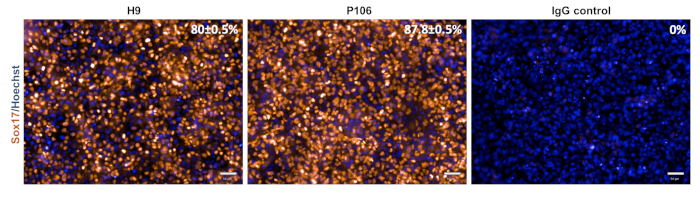
Figure 3 : Caractérisation de la spécification définitive de l’endoderme. Au jour 5, les cellules ont été colorées pour Sox17, un marqueur endodermique définitif. Le pourcentage de cellules Sox17 positives était de 80 ± 0,5 % pour H9 et de 87,8 ± 0,5 % pour P106. La quantification en pourcentage était basée sur 10 puits distincts avec 6 champs de vision par puits. Les données sont présentées comme la moyenne ± SEM. Barre d’échelle = 50 μm. Veuillez cliquer ici pour voir une version plus grande de cette figure.

Figure 4: Caractérisation des progéniteurs hépatiques. Au jour 10, les progéniteurs hépatiques ont été colorés pour les marqueurs hépatiques (A) HNF4α, (B) AFP et (C) ALB. Pour H9, le pourcentage de cellules positives était de 91 % ± 0,4 %, 89,7 % ± 1,8 % et 30,7 % ± 1,8 % pour HNF4α, AFP et ALB, respectivement. Pour P106, le pourcentage de cellules positives était de 90 % ± 0,2 %, 86 % +/- 1,2 % et 27,2 % ± 1,1 % pour HNF4α, AFP et ALB, respectivement. (D) Le potentiel de lignée cholangiocytes a été évalué par l’expression de CK19; Les progéniteurs hépatiques dérivés de H9 ont exprimé 78,5 % ± 3,2 % de cellules CK19 positives, tandis que 83,6 % ± 1,8 % des cellules CK19 positives ont été observées pour les progéniteurs hépatiques P106. La coloration à l’immunoglobuline G (IgG) a été utilisée comme témoin de la coloration. La quantification en pourcentage était basée sur 10 puits distincts avec 6 champs de vision par puits. Les données sont présentées comme la moyenne ± SEM. Barre d’échelle = 50 μm. Veuillez cliquer ici pour voir une version plus grande de cette figure.

Figure 5: Analyse de la sécrétion de protéines progénitaires hépatiques. La sécrétion d’alpha fœtoprotéine (AFP) et d’albumine (ALB) a été analysée dans des cultures de progéniteurs hépatiques au jour 10 chez H9 et P109. Les données représentent trois répliques biologiques et les barres d’erreur représentent le SD. Les protéines sécrétées ont été quantifiées à partir d’un milieu de culture de 24 h sous forme de nanogrammes de protéines sécrétées par mL par mg de protéine, n = 3; ND = non détecté. Veuillez cliquer ici pour voir une version agrandie de cette figure.
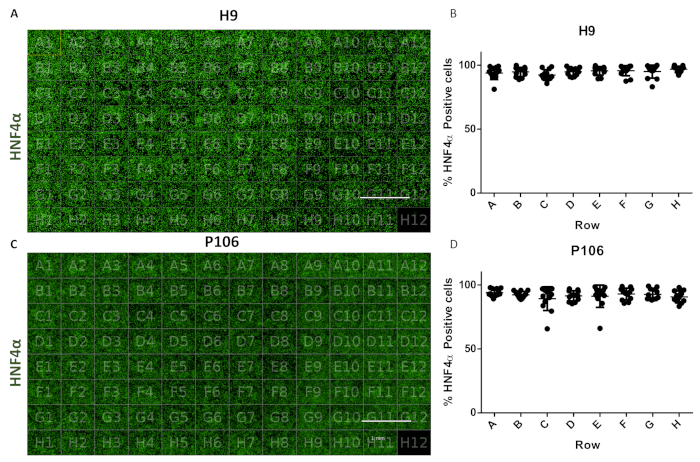
Figure 6: Évaluation de la variabilité de puits à puits dans 96 plaques de puits. (A) Visualisation d’une vue sur plaque de 96 puits de progéniteurs hépatiques dérivés de H9 colorés avec HNF4α. (B) Quantification des cellules HNF4α positives. Moyenne du nombre de cellules par puits en lignes, à partir de six champs de vision par puits quantifié. Le nombre moyen de cellules sur la plaque était de 94,81% ± 0,22 CELLULES SEM HNF4α positives par puits. Aucune différence statistiquement significative n’a été observée entre les puits. (C) Visualisation d’une vue sur plaque de 96 puits de progéniteurs hépatiques dérivés de P106 colorés avec HNF4α. (D) Quantification des cellules HNF4α-positives. Nombre moyen de cellules par puits en lignes, à partir de six champs de vision par puits et quantifié. Le nombre moyen de cellules sur la plaque était de 97,7% ± 0,57 SEM HNF4α-positives par puits. Aucune différence statistiquement significative n’a été observée entre les rangées. Eh bien H12 a été utilisé comme contrôle de la coloration de l’immunoglobuline G (IgG). Barre d’échelle = 1 mm. Une ANOVA unidirectionnelle avec les tests statistiques post-hoc de Tukey a été utilisée. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Format de plaque | Superficie (cm2) | Cellules par cm2 | Nombre total de cellules par puits | Volume de distribution (mL) | Concentration cellulaire (cellules/ml) |
| Plaque de 24 puits | 1.9 | 210526 | 400000 | 0.5 | 800000 |
| Plaque de 96 puits | 0.32 | 187500 | 60000 | 0.05 | 1200000 |
Tableau 1: Densité cellulaire recommandée pour les différents formats de plaques pour les lignées cellulaires hPSC utilisées dans ce protocole.
Discussion
La génération de cellules progéniatrices hépatiques humaines à partir de cellules souches pluripotentes à grande échelle pourrait représenter une alternative prometteuse au matériel dérivé du cadavre. La normalisation et la reproductibilité des protocoles sont essentielles pour assurer l’application de la technologie et l’impact de la recherche biomédicale. Pour y remédier, les travaux antérieurs se sont concentrés sur le développement d’un protocole de différenciation par étapes des CSEh et des CS IPS en utilisant des additifs définis et des matrices15,23,24 ,25,26,27,28. Ce faisant, le phénotype des hépatocytes et la reproductibilité ont été améliorés, permettant la semi-automatisation du processus de différenciation19. Le système présenté est renforcé par sa combinaison avec des milieux de culture cellulaire prêts à l’emploi et un système facile de différenciation des hépatocytes.
Auparavant, la densité des cellules pluripotentes avant le début du protocole de différenciation était mise en évidence comme une variable clé pour atteindre une population homogène de cellules progéniatrices hépatiques26. En utilisant cette procédure plus raffinée, il est possible de générer un grand nombre de progéniteurs hépatiques dérivés de cellules souches de manière progressive en utilisant une gamme de densités cellulaires de départ (Tableau 1). Au jour 5, l’induction définitive de l’endoderme a été validée par coloration Sox17(Figure 3). Une différenciation efficace et robuste en endoderme définitif a été obtenue avec les lignes ESC et iPSC testées, avec plus de 80% exprimant Sox17(Figure 3). Au jour 10, les progéniteurs hépatiques présentaient une morphologie pavée uniforme, et les marqueurs de cellules souches hépatiques étaient fortement enrichis pour l’AFP et le HNF4α (>86%, Figure 4). En utilisant une combinaison de technologies manuelles et semi-automatisées, il a été possible d’effectuer une différenciation dans plusieurs formats de plaques19.
Dans sa forme actuelle, la différenciation cellulaire convient à l’expérimentation in vitro. Cependant, l’enrichissement cellulaire serait probablement nécessaire avant l’application clinique pour s’assurer qu’une population homogène de progéniteurs hépatiques est préparée pour l’administration.
En conclusion, le protocole décrit ici fournit au domaine une approche standardisée pour produire des progéniteurs hépatiques à grande échelle. Les travaux futurs se concentreront sur la production d’un nouveau support pour la différenciation, la maturation et la maintenance ultérieures des HLC.
Déclarations de divulgation
David C. Hay est cofondateur et actionnaire de Stemnovate Ltd. Les autres auteurs certifient qu’ils n’ont aucun conflit d’intérêts dans le sujet ou les documents abordés dans cet article.
Remerciements
Cette étude a été soutenue par des bourses du MRC Doctoral Training Partnership (MR/K501293/1), de la UK Regenerative Medicine Platform (MRC MR/L022974/1 et MR/K026666/1), du Chief Scientist Office (TCS/16/37).
matériels
| Name | Company | Catalog Number | Comments |
| DPBS with Calcium and Magnesium | ThermoFisher | 14040133 | |
| Gentle cell dissociation reagent | STEMCELL Technologies | 7174 | |
| Hoechst 33342 Ready Flow Reagent | thermofisher | R37165 | |
| Human Recombinant Laminin 521 | BioLamina | LN521-02 | |
| Human Serum Albumin ELISA | Alpha Diagnostics | 1190 | |
| Human Serum Alpha Fetoprotein ELISA | Alpha Diagnostics | 500 | |
| mTeSR1 medium | STEMCELL Technologies | 5850 | |
| Operetta High-Content Imaging System | PerkinElmer | HH12000000 | |
| PBS, no calcium, no magnesium | ThermoFisher | 14190250 | |
| Penicillin-Streptomycin (10,000 U/mL) | Life Technologies | 15140122 | |
| Rho-associated kinase (ROCK)inhibitor Y27632 | Sigma-Aldrich | Y0503-1MG | |
| STEMdiff Definitive Endoderm Supplement CJ | STEMCELL Technologies | ||
| STEMdiff Definitive Endoderm Supplement MR | STEMCELL Technologies | ||
| STEMdiff Endoderm Basal Medium | STEMCELL Technologies | ||
| STEMdiff Hepatic Progenitor Medium | STEMCELL Technologies | ||
| TWEEN 20 | Sigma-Aldrich | P9416 | |
| Antibodies | |||
| Albumin | Sigma-Aldrich | A6684 | 1:200 (mouse) |
| Alpha-fetoprotein | Sigma-Aldrich | A8452 | 1:400 (mouse) |
| HNF-4α | Santa Cruz | sc-8987 | 1:400 (rabbit) |
| IgG | DAKO | 1:400 | |
| Sox17 | R&D Systems, Inc. | AF1924 | 1:200 (Goat) |
| Software | |||
| Columbus Image Data Storage and Analysis system | PerkinElmer |
Références
- Asrani, S. K., Devarbhavi, H., Eaton, J., Kamath, P. S. Burden of liver diseases in the world. Journal of Hepatology. 70 (1), 151-171 (2019).
- Szkolnicka, D., Hay, D. C. Concise Review: Advances in Generating Hepatocytes from Pluripotent Stem Cells for Translational Medicine. Stem Cells Dayton Ohio. 34 (6), 1421-1426 (2016).
- Heslop, J. A., Duncan, S. A. The Use of Human Pluripotent Stem Cells for Modeling Liver Development and Disease. Hepatology. 69 (3), 1306-1316 (2019).
- Alwahsh, S. M., Rashidi, H., Hay, D. C. Liver cell therapy: is this the end of the beginning. Cell and Molecular Life Sciences. 75 (8), 1307-1324 (2018).
- Hay, D. C., et al. Efficient differentiation of hepatocytes from human embryonic stem cells exhibiting markers recapitulating liver development in vivo. Stem Cells Dayton Ohio. 26 (4), 894-902 (2008).
- Si-Tayeb, K., et al. Highly efficient generation of human hepatocyte-like cells from induced pluripotent stem cells. Hepatology. 51 (1), 297-305 (2010).
- Hannan, N. R. F., Segeritz, C. -. P., Touboul, T., Vallier, L. Production of hepatocyte-like cells from human pluripotent stem cells. Nature Protocols. 8 (2), 430-437 (2013).
- Basma, H., et al. Differentiation and transplantation of human embryonic stem cell-derived hepatocytes. Gastroenterology. 136 (3), 990-999 (2009).
- Meseguer-Ripolles, J., Khetani, S. R., Blanco, J. G., Iredale, M., Hay, D. C. Pluripotent Stem Cell-Derived Human Tissue: Platforms to Evaluate Drug Metabolism and Safety. The AAPS Journal. 20 (1), 20 (2017).
- Si-Tayeb, K., Lemaigre, F. P., Duncan, S. A. Organogenesis and development of the liver. Developmental Cell. 18 (2), 175-189 (2010).
- D'Amour, K. A., et al. Efficient differentiation of human embryonic stem cells to definitive endoderm. Nature Biotechnology. 23 (12), 1534-1541 (2005).
- Shin, D., et al. Bmp and Fgf signaling are essential for liver specification in zebrafish. Development Cambridge England. 134 (11), 2041-2050 (2007).
- DeLaForest, A., et al. HNF4A is essential for specification of hepatic progenitors from human pluripotent stem cells. Development Cambridge England. 138 (19), 4143-4153 (2011).
- Baxter, M., et al. Phenotypic and functional analyses show stem cell-derived hepatocyte-like cells better mimic fetal rather than adult hepatocytes. Journal of Hepatology. 62 (3), 581-589 (2015).
- Cameron, K., et al. Recombinant Laminins Drive the Differentiation and Self-Organization of hESC-Derived Hepatocytes. Stem Cell Reports. 5 (6), 1250-1262 (2015).
- Szkolnicka, D., et al. Reducing Hepatocyte Injury and Necrosis in Response to Paracetamol Using Noncoding RNAs. Stem Cells Translational Medicine. 5 (6), 764-772 (2016).
- Domogatskaya, A., Rodin, S., Boutaud, A., Tryggvason, K. Laminin-511 but Not -332, -111, or -411 Enables Mouse Embryonic Stem Cell Self-Renewal In Vitro. Stem Cells. 26 (11), 2800-2809 (2008).
- Kanninen, L. K., et al. Laminin-511 and laminin-521-based matrices for efficient hepatic specification of human pluripotent stem cells. Biomaterials. 103, 86-100 (2016).
- Meseguer-Ripolles, J., Lucendo-Villarin, B., Wang, Y., Hay, D. C. Semi-automated Production of Hepatocyte Like Cells from Pluripotent Stem Cells. Journal of Visualized Experiments. (137), e57995 (2018).
- Carpenter, A. E., et al. CellProfiler: image analysis software for identifying and quantifying cell phenotypes. Genome Biology. 7, 100 (2006).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Raven, A., et al. Cholangiocytes act as Facultative Liver Stem Cells during Impaired Hepatocyte Regeneration. Nature. 547 (7663), 350-354 (2017).
- Hay, D. C., et al. Unbiased screening of polymer libraries to define novel substrates for functional hepatocytes with inducible drug metabolism. Stem Cell Research. 6 (2), 92-102 (2011).
- Medine, C. N., et al. Developing High-Fidelity Hepatotoxicity Models from Pluripotent Stem Cells. Stem Cells Translational Medicine. 2 (7), 505-509 (2013).
- Szkolnicka, D., et al. Accurate prediction of drug-induced liver injury using stem cell-derived populations. Stem Cells Translational Medicine. 3 (2), 141-148 (2014).
- Wang, Y., et al. Defined and Scalable Generation of Hepatocyte-like Cells from Human Pluripotent Stem Cells. Journal of Visualized Experiments. (121), e55355 (2017).
- Villarin, B. L., et al. Polymer Supported Directed Differentiation Reveals a Unique Gene Signature Predicting Stable Hepatocyte Performance. Advanced Healthcare Materials. 4 (12), 1820-1825 (2015).
- Wang, Y., et al. Multiomics Analyses of HNF4α Protein Domain Function during Human Pluripotent Stem Cell Differentiation. iScience. 16, 206-217 (2019).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon