Method Article
Eine effiziente Methode für die Adenovirus-Produktion
In diesem Artikel
Zusammenfassung
Hier stellen wir ein Protokoll für die Adenovirus-Produktion mit dem pAdEasy-System vor. Die Technologie umfasst die Rekombination der Plasmide pAdTrack und pAdEasy-1, die Adenovirus-Verpackung und -Verstärkung, die Reinigung der adenoviralen Partikel aus Zelllysat und Kulturmedium, die virale Titration und die funktionelle Prüfung des Adenovirus.
Zusammenfassung
Die adenovirale Transduktion hat den Vorteil einer starken und vorübergehenden Induktion der Expression des Gens von Interesse in eine Vielzahl von Zelltypen und Organen. Die rekombinante adenovirale Technologie ist jedoch mühsam, zeitaufwändig und teuer. Hier präsentieren wir ein verbessertes Protokoll mit dem pAdEasy-System, um gereinigte adenovirale Partikel zu erhalten, die eine starke grüne fluoreszierende Proteinexpression (GFP) in transduzierten Zellen induzieren können. Die Vorteile dieser verbesserten Methode sind eine schnellere Vorbereitung und geringere Produktionskosten im Vergleich zur ursprünglichen Methode, die bert Vogelstein entwickelt hat. Die wichtigsten Schritte der adenoviralen Technologie sind: (1) die Rekombination von pAdTrack-GFP mit dem pAdEasy-1 Plasmid in BJ5183 Bakterien; (2) die Verpackung der adenoviralen Partikel; (3) die Amplifikation des Adenovirus in AD293-Zellen; (4) die Reinigung der adenoviralen Partikel aus Zelllysat und Kulturmedium; und (5) die virale Titration und funktionelle Tests des Adenovirus. Die Verbesserungen der ursprünglichen Methode bestehen aus (i) der Rekombination in BJ5183-haltigen pAdEasy-1 durch chemische Umwandlung von Bakterien; ii) die Auswahl rekombinanter Klone durch "negative" und "positive" PCR; iii) die Transfektion von AD293-Zellen unter Verwendung des Transfektionssystems K2 für adenovirale Verpackungen; iv) die Ausfällung der von AD293-Zellen im Zellkulturmedium freigesetzten Viruspartikel mit Ammoniumsulfat; und (v) die Reinigung des Virus durch einstufige Cäsiumchlorid-Diskontinuierliche Gradienten-Ultrazentrifugation. Eine starke Expression des Gens von Interesse (in diesem Fall GFP) wurde in verschiedenen Arten von transduzierten Zellen (wie Hepatozyten, Endothelzellen) aus verschiedenen Quellen (Mensch, Rind, Murin) erhalten. Der adenoviral-vermittelte Gentransfer ist eines der wichtigsten Instrumente für die Entwicklung moderner Gentherapien.
Einleitung
Adenoviren sind nicht umhüllte Viren, die ein Nukleocapsid und ein doppelsträngiges lineares DNA-Genom1,2,3enthalten. Adenoviren können eine breite Palette von Zelltypen infizieren und Infektionen sind nicht von der aktiven Wirtszellteilung abhängig. Nach der Infektion führt das Adenovirus seine genomische DNA in den Zellzellkern ein, wo es epichromosomal bleibt und zusammen mit den Genen des Wirts transkribiert wird. Somit wird ein minimales potenzielles Risiko für Insertionsmutagenese oder Onkogenes-Regulierung erreicht4,5,6. Das adenovirale Genom wird nicht zusammen mit dem Wirtsgenom repliziert und somit werden die adenoviralen Gene in einer teilenden Zellpopulation verdünnt. Zu den Vorteilen der adenoviralen Transduktion gehören: (i) hohe Transgenexpression; ii) verringerte Risiken im Zusammenhang mit der Integration der viralen DNA in das Wirtsgenom aufgrund episomaler Expression; iii) Transduktion einer Vielzahl von teilenden und nicht trennenden Zelltypen. Die meisten Adenoviren, die in der biomedizinischen Forschung verwendet werden, sind nicht reproduzierbar, ohne die E1-Region7,8,9. Für ihre Produktion ist eine Zellleitung erforderlich, die die E1-Sequenz (z. B. HEK293) liefert. Außerdem wurde eine nicht-essentielle Region für den viralen Lebenszyklus (E3) gestrichen, um die Einfügung eines Transgens in das virale Genom zu ermöglichen; andere Regionen (E2 und E4) wurden bei einigen Adenoviren weiter gestrichen, aber in diesen Fällen wurde ein geringer Ertrag der adenoviralen Produktion und eine geringe Expression des Transgens berichtet7.
Hier stellen wir ein verbessertes Protokoll zum Erstellen, Verpacken und Reinigen der Adenoviren mit dem AdEasy-System vor. Diese Verbesserungen ermöglichten die Verpackung des Adenovirus in einer schnelleren und wirtschaftlicheren Weise im Vergleich zu der ursprünglichen Methode von Bert Vogelstein2,10entwickelt , aufgrund der folgenden Vorteile: (i) die Rekombination in BJ5183-haltigen pAdEasy-1 durch chemische Umwandlung von Bakterien; ii) die Auswahl der rekombinanten Klone nach PCR; iii) die Transfektion von AD293-Zellen unter Verwendung des Transfektionssystems K2 für adenovirale Verpackungen; iv) die Ausfällung adenoviraler Partikel aus kulturmedium nach viraler Verpackung und Amplifikation; v) die adenovirale Reinigung mit einstufigem Cäsiumchlorid (CsCl) Gradienten-Ultrazentrifugation.
Das Protokoll für die Adenovirus-Produktion mit dem AdEasy-System (Abbildung 1) umfasst die folgenden Schritte:
(1) Rekombination von pAdTrack-GFP mit pAdEasy-1 in BJ5183 Bakterien
(2) Verpackung der adenoviralen Partikel
(3) Verstärkung des Adenovirus
(4) Reinigung der adenoviralen Partikel aus Zelllysat und Kulturmedium
(5) Adenovirus-Titration.
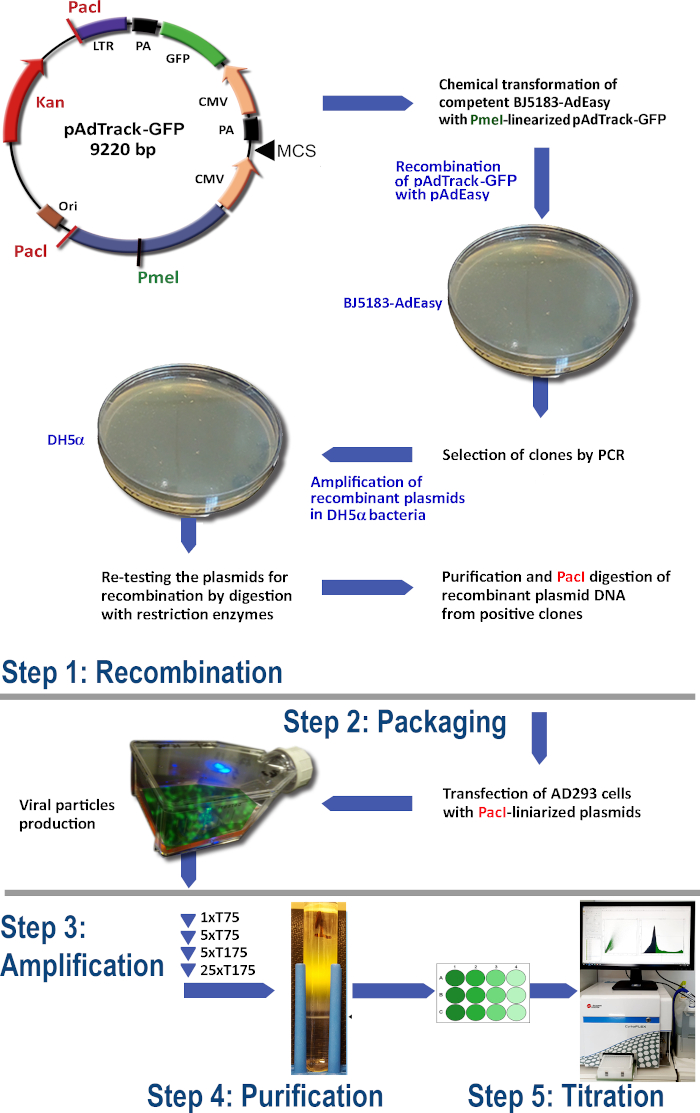
Abbildung 1: Die Adenovirus-Produktionstechnologie. Die wichtigsten Schritte der adenoviralen Technologie sind: (1) Die Rekombination des pAdTrack-GFP mit dem pAdEasy-1 Plasmid in BJ5183 Bakterien. Die ausgewählten rekombinierten Plasmide werden in DH5-Bakterien verstärkt und anschließend gereinigt; (2) Die Verpackung der adenoviralen Partikel in AD293-Zellen, die Adeno-E1-Proteine produzieren; (3) Die Amplifikation des Adenovirus in AD293-Zellen; (4) die Reinigung der adenoviralen Partikel aus dem Zelllysat und des Kulturmediums durch Ultrazentrifugation auf einem CsCl-Dichtegradienten; (5) Die Titration des Adenovirus und die funktionellen Tests. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
In diesem Protokoll haben wir die Technologie zur Herstellung des Adenovirus exemplarisch dargestellt, die die Expression von GFP in den Wirtszellen induzieren kann. GFP wird bereits im Backbone des pAdTrack-CMV-Shuttlevektors (Addgene #16405) unter einem zweiten CMV-Promotor eingesetzt und wird als Reportergen verwendet (Abbildung 1). Aus diesem Grund haben wir hier den pAdTrack-CMV-Vektor als pAdTrack-GFP bezeichnet und die Expression von GFP für Demonstrativzwecke bewertet. Neben der GFP-Expression kann das System verwendet werden, um ein Gen von Interesse zu überprimieren, das in den mehrfachen Klonstellen der pAdTrack-CMV geklont werden kann. Ein im pAdTrack-CMV geklontes Gen oder Minigen ist in der Regel effizienter für die Expressionsinduktion als die cDNA11. Die Daten zeigten eine starke GFP-Expression in transduzierten Zellen (wie Hepatozyten, Endothelzellen) aus verschiedenen Quellen (Mensch, Rind, Murin). Der adenoviral-vermittelte Gentransfer ist eines der wichtigsten Instrumente für die Entwicklung moderner Gentherapien.
Protokoll
Sicherheitshinweis: Im Allgemeinen werden Adenoviren als Organismen der Biosicherheitsstufe 2 eingestuft, so dass alle Manipulationen in einem Biosicherheitsschrank der Klasse II von einer geschulten Person durchgeführt werden müssen, die Biohazard-Schutzausrüstung (einschließlich Handschuhe, Gesichtsmaske für biologische Aerosole, Labormantel usw.) trägt. Alle mit dem Adenovirus kontaminierten festen Materialien müssen mit einer 10%igen Bleichlösung für 30 min desinfiziert und 30 min bei 121 °C und 1 bar autoklaviert werden. Je nach eingesetztem Gen kann das erzeugte Adenovirus gefährliches Potenzial haben und in andere Biosicherheitsstufen eingeteilt werden.
1. Experimentelle Zubereitung
- Verwenden Sie eine separate Zellkulturhaube für adenovirale Manipulationen und einen separaten Inkubator für jeden Adenovirus-Typ. T-Flaschen mit Filterkappen für virale Verpackung und Verstärkung verwenden; so weit wie möglich Transduktionsexperimente in belüfteten Petriplatten zu vermeiden.
- Leeren Sie die Zellkulturhaube nach jedem Gebrauch und setzen Sie sie 15 min UV aus.
- Autoklav periodisch die Pipettenhilfe, Pipetten und andere Utensilien. Wenn möglich, Kultur in einem separaten Zellkulturlabor/Haube die Zellen für adenovirale Verpackung (AD293 Zellen) und die Zellen in Transduktionsexperimenten verwendet werden. Chargen verschiedener Adenoviren, die im selben Zeitraum verstärkt wurden, sollten auf Kreuzkontamination durch PCR überprüft werden.
- Bereiten Sie die folgenden Lösungen vor.
- SOB (Super Optimale Brühe) mittel vorbereiten: 20 g Trypton, 5 g Hefeextrakt, 0,5 g NaCl (10 mM Endkonzentration), 2,5 ml 1 M KCl (2,5 mM Endkonzentration), amd H2O bis 1 L. Nach dem Autoklavieren bei 121 °C die folgenden sterilen Lösungen hinzufügen: 5 ml von 1 M MgCl2 und 5 ml von 1 M MgSO4.
- SoC (Super Optimal Brühe mit Katabolitenrepression) Medium vorbereiten: in 1 L steriles SOB die folgenden sterilen Lösungen hinzufügen: 20 ml 1 M Glukose, 5 ml von 1 M MgCl2 und 5 ml von 1 M MgSO4.
- Niederschlagslösung vorbereiten: 29,5 g Kaliumacetat in 60 mlH2O auflösen, 11,5 ml Essigsäure und H2O bis 100 ml hinzufügen.
- Resuspensionspuffer vorbereiten: 95 ml 20% Glukose, 5 ml 1 M Tris-Cl pH 8, 4 ml mit 0,5 M EDTA pH 8, und H2O bis 200 ml hinzufügen.
- Vorbereiten der Lyselösung: 4,8 ml 8,3 M NaOH, 10 ml 20%SDS und H2O bis 200 ml.
2. Rekombination des pAdTrack-GFP-Virusvektors mit pAdEasy-1-Plasmid in BJ5183-Bakterien
- Linearisierung von pAdTrack-GFP und Reinigung des linearisierten Plasmids.
- Bereiten Sie die folgende Verdauungsmischung auf Eis vor:
10 g pAdTrack-GFP
5 l mit 10x farblosem Puffer
2 L von Pme I
H2O bis zu einem Endvolumen von 50 l. - 3 Stunden lang bei 37 °C in einem Wasserbad inkubieren.
- Bei 65 °C für 20 min inaktivieren.
- Überprüfen Sie die Effizienz der Verdauung von pAdTrack-GFP mit Pme I: führen Sie 1 g des verdauten Plasmids parallel zu 1 g unverdautem Plasmid auf einem 0,8% Agarose-Gel durch.
- Bereiten Sie die folgende Verdauungsmischung auf Eis vor:
- Isolierung und Reinigung der DNA
HINWEIS: Die Schritte 1-6 müssen in einer Dunstabzugshaube ausgeführt werden.- Fügen Sie ein gleiches Volumen phenol/chloroform/isoamylalkohol (25:24:1) über das Verdauungsgemisch hinzu und invertieren Sie das Rohr, bis das Gemisch homogen ist.
- Zentrifugieren Sie 3 min bei 16.200 x gund übertragen Sie dann die obere wässrige Phase in ein Sammelrohr.
- Fügen Sie ein gleiches Volumen an Phenol/Chloroform/Isoamylalkohol (25:24:1) über die untere organische Phase und Wirbel hinzu.
- Zentrifuge für 3 min bei 16.200 x g, und dann die obere Phase in das gleiche Sammelrohr übertragen.
- Fügen Sie ein gleiches Volumen Chloroform über die wässrige Phase in der Sammlung Röhre und Wirbel geerntet.
- Zentrifugieren Sie 3 min bei 16.200 x gund übertragen Sie dann die obere wässrige Phase in ein neues Sammelrohr.
- Fügen Sie ein 1/10 Volumen von 3 M Natriumacetat und 2 Volumen von kaltem 100% Ethanol und Wirbel hinzu.
- 1 Stunde bei -70 °C oder über Nacht bei -20 °C inkubieren.
- Die Probe auf Eis auftauen und 10 min bei 16.200 x g und 4 °C zentrifugieren.
- Entfernen Sie den Überstand und fügen Sie 750 l 75% Ethanol hinzu.
- Zentrifuge für 3 min bei 16.200 x g und 4 °C und entfernen Sie den Überstand.
- Drehen Sie das Rohr kurz, um den ganzen Überstand zu entfernen und das Pellet in der Haube zu trocknen. Trocknen Sie das DNA-Pellet nicht lange, da es schwierig ist, sich aufzulösen.
- Lösen Sie das Pellet in 15 l von H2O auf.
- Messen Sie die DNA-Konzentration mit einem Spektralphotometer (z. B. Nanodrop).
- Transformation von AdEasier-1-Bakterien mit pAdTrack-GFP
HINWEIS: In diesem Schritt erfolgt die Rekombination von pAdTrack-GFP mit pAdEasy-1 Plasmid.- Bereiten Sie AdEasier-1 (BJ5183-haltige pAdEasy-1, Addgene #16399) chemisch kompetente Bakterien mithilfe eines kommerziellen Transformationskits vor, das den Anweisungen des Herstellers folgt. Halten Sie Aliquots von 100 l kompetenten Bakterien bei -80 °C.
- Thaw ein Aliquot von kompetenten AdEasier-1 Bakterien auf Eis und fügen Sie 1 g gereinigte Pme I -verdaut pAdTrack-GFP. Mischen Sie sanft durch Streichen des Rohres (nicht Pipette die Mischung). 10 min auf Eis bebrüten.
- Fügen Sie 900 l SOC-Medium hinzu und brüten Sie 1 Stunde bei 37 °C mit Schütteln.
- Mikrofuge für 5 min bei 600 x g.
- Entfernen Sie 900 l des Überstandes, mischen Sie das Pellet und den Überstand und säen Sie die transformierten Bakterien auf LB-Agar-Platten mit Kanamycin.
- 16 Stunden bei 37 °C inkubieren (nicht mehr als 18 Stunden).
- Auswahl der möglichen positiven Klone per PCR
- Geben Sie Zahnstocher in zwei Hälften auf und sterilisieren Sie die Halbzahnstocher durch Autoklavieren.
- Nehmen Sie kleine und transluzente Kolonien mit sterilen Halbzahnstochern auf.
- Kurz umdrehen Sie den Halbzahnstocher mit Bakterien in 10 l Wasser (in einem PCR-Rohr) und legen Sie den Halbzahnstocher dann in ein 1,5 ml Eppendorf-Rohr, das 100 l SOC-Medium mit Kanamycin enthält. Inkubieren Sie für 4-6 Stunden bei 37 °C, während Sie die Klone mit "negativ" und "positiv" PCR testen.
- Inkubieren Sie die PCR-Röhren, die 10 l Wasser enthalten, für 5 min bei 95 °C mit Bakterien, um die bakterielle Probe zu erhalten und parallel die "negative" und "positive" PCR auszuführen.
- "Negative" PCR - um die pAdTrack-GFP-Integrität zu testen: Bereiten Sie den folgenden PCR-Mix für die negative PCR auf Eis vor.
5 L der Bakteriellen Probe
0,1 l Primer Vorwärts (4631 F: 5'-CAGTAGTCGGTGCTCGTCCAG)
Grundierung umgedreht (5616 R: 5'-TATGGGGGCTGTAATGTTGTCTC)
0,1 l dNTP 10 mM
3 l 5x Puffer
MgCl2 25mM
GoTaq-Polymerase 0,1 l
H2O bis zu einem Endvolumen von 15 l
HINWEIS: Die positive Kontrolle, in der die DNA-Vorlage der pAdTrack-GFP-Vektor ist, muss enthalten sein. - "Positive" PCR - um das Vorhandensein des Gens von Interesse zu testen. Verwenden Sie spezifische Primer für das eingefügte Gen und bereiten Sie die Mischung wie im vorherigen Schritt vor. Die Grundierungen für GFP waren die folgenden:
F: 5'-CAAGGACGACGGCAACTACA
R: 5'-ATGGGGGTGTTCTGCTGGTA - Führen Sie parallel die "negative" und die "positive" PCR aus. Das PCR-Programm ist: 5 min, 95 °C; 40 Zyklen der folgenden Schritte: 30 Sek., 95 °C; 30 Sek., 68 °C; 1 min, 72 °C; Enddehnung: 10 min, 72 °C.
HINWEIS: Passen Sie die Glühtemperatur für die Verstärkung des Gens von Interesse an. - Bewerten Sie die PCR-Produkte auf einem 1% Agarose-Gel und treffen Sie die Auswahl der Klone.
- Berücksichtigen Sie die weitere Verarbeitung der Klone, die keine PCR-Produkte für die "negative PCR" und das spezifische PCR-Produkt nach der "positiven PCR" geben.
- Wachsen Sie die Bakterienkulturen ausgewählter rekombinanter Klone
- Verdünnen Sie die Kulturen der vermuteten positiven Klone (führte zum Schritt 2.4.3.) in 4 ml SOC-Medium mit Kanamycin und inkubieren Sie sie über Nacht bei 37°C mit Schütteln.
- Isolierung der Plasmid-DNA aus AdEasier-1-Bakterien (Miniprep mit alkalischer Lyse)
- 1,5 ml Bakterienkultur in Mikrozentrifugenröhren, Zentrifuge für 1 min bei 16.200 x gübertragen und den Überstand entfernen.
- Übertragen Sie eine weitere 1,5 ml Bakterienkultur in der gleichen Röhre, wiederholen Zentrifugation, und entfernen Sie den Überstand.
- Fügen Sie 200 l Resuspensionspuffer hinzu (50 mM Glukose, 10 mM EDTA, 25 mM Tris-HCl pH 8).
- Fügen Sie 200 l Lyselösung (0,2 N NaOH, 1% SDS) hinzu, mischen Sie vorsichtig, indem Sie das Rohr invertieren.
- Fügen Sie 200 l Niederschlagslösung (60 ml 5 M Kaliumacetat, 11,5 ml Eisessigsäure, fügen Sie H2O bis 100 ml), und mischen Sie vorsichtig, indem Sie das Rohr invertieren.
- Zentrifuge für 3 min bei 16.200 x g.
- Übertragen Sie den Überstand in ein neues Mikrozentrifugenrohr, fügen Sie 500 L Isopropanol, mischen und inkubieren für 20 min auf Eis.
- Zentrifuge für 15 min bei 16.200 x g und 500 l 75% Ethanol hinzufügen.
- Zentrifuge für 10 min bei 16.200 x g und entfernen Sie den Überstand.
- Zentrifuge für 3 min bei 16.200 x g, entfernen Sie den Überstand und fügen Sie 15 lH2O hinzu.
- Verstärkung, Isolierung und erneute Prüfung der rekombinierten Plasmide
- Transformation von DH5-Bakterien mit der DNA, die aus AdEasier-1-Zellen isoliert ist.
- Bereiten Sie DH5- kompetente Bakterien mithilfe des kommerziellen Transformationskits vor, und folgen Sie den Anweisungen des Herstellers.
- 100 L DH5-fähiger Bakterien auf Eis auftauen, die rekombinante DNA hinzufügen und 10 min auf Eis brüten. Dann säen Sie die Bakterien auf LB-Agar-Platten mit Kanamycin.
- Bei 37 °C über Nacht inkubieren.
- Nehmen Sie mehrere Kolonien auf und wachsen Sie jeweils in 2 ml LB-Medium mit Kanamycin, bei 37 °C, über Nacht, mit Schütteln.
- Isolieren Sie die DNA (Miniprep mit alkalischer Lyse) und setzen Sie die erhaltene DNA in 25 LH2O wieder auf.
- Bestätigen Sie die positiven Klone durch enzymatische Verdauung.
- Bereiten Sie die folgende Mischung auf Eis:
5 L rekombinante DNA
1,5 l mit 10x farblosem Puffer
0,5 l Hind III oder Pst I
H2O bis zu einem Endvolumen von 15 l - Bei 37 °C für 30 min inkubieren.
HINWEIS: Als Kontrolle verdauen Sie auch die plasmide pAdTrack-GFP und pAdEasy-1. - Fügen Sie in jeder Probe 3 L Sx6-Ladepuffer mit RNase A hinzu (wenn RNase A in den Miniprep-Puffern nicht vorhanden ist).
- Führen Sie die verdauten DNA-Fragmente auf 1% Agarose-Gel-Elektrophorese.
HINWEIS: Das Verdauungsmuster eines positiven Klons enthält die Mehrheit der Fragmente des verdauten pAdEasy-Plasmids, was eine pAdEasy-Rekombination mit dem pAdTrack-Vektor offenbart. Das Gen von Interesse sollte durch Dieverdauung mit den Restriktionsenzymen nachgewiesen werden, die zum Klonen verwendet werden.
- Bereiten Sie die folgende Mischung auf Eis:
- Herstellung von Plasmid-DNA (Transfektions-Grade) für Adenovirus-Verpackungen.
- Wachsen Sie eine 200 ml Kultur von Bakterien aus einem positiven Klon, um die Plasmid-DNA zu isolieren.
- Isolieren Sie die Plasmid-DNA mit einem kommerziellen Kit für Plasmid-DNA Midiprep (z. B. Qiagen Plasmid Midi Kit) nach den Anweisungen des Herstellers.
- Transformation von DH5-Bakterien mit der DNA, die aus AdEasier-1-Zellen isoliert ist.
3. Verpackung der adenoviralen Partikel
- Tag 1. Samen der AD293-Zellen
- Waschen Sie die AD293-Zellen mit PBS und inkubieren Sie sie mit 0,125% Trypsin für 2-5 min bei 37 °C.
- Sammeln Sie die Zellen in kaltem Medium mit Serum.
- Zentrifuge für 5 min bei 400 x g bei 4 °C.
- Setzen Sie die Zellen im Medium mit Serum aus und säen Sie die Zellen mit einer Dichte von 2 x 106/T25 Kolben. Vorzugsweise verwenden Sie einen Kolben mit einem Filter.
- Tag 1. Verdauen Sie die rekombinante DNA mit Pac I
- Bereiten Sie die folgende Mischung vor:
6 l rekombinante DNA (1 g/l)
2 L von Pac I
2,5 l mit 10x farblosem Puffer
H2O bis zu einem Endvolumen von 25 l - 3 h (oder über Nacht) bei 37 °C inkubieren und dann das Enzym bei 65 °C für 20 min inaktivieren.
- DNA-Fällung mit Ethanol: Fügen Sie 2,5 l (1/10 v/v) 3 M Natriumacetat und 2-3 Volumen von 100% Ethanol hinzu. Inkubation für 30 min bei -70 °C oder über Nacht bei -20 °C.
- Zentrifuge bei 16.200 x g für 30 min bei 4 °C und das Pellet in sterilem Wasser wieder aufsetzen.
- Bereiten Sie die folgende Mischung vor:
- Tag 2: Transfektion von AD293-Zellen mit K2-Reagenz
- Fügen Sie 40 L K2-Multiplikator über die Zellen, zwei Stunden vor der Transfektion.
- Vorbereiten von A- und B-Lösungen:
Lösung A: Fügen Sie 6 g Pac l-linearisierte DNA in 260 l Opti-MEM hinzu.
Lösung B: Fügen Sie 21,6 l K2-Reagenz in 248,4 l Opti-MEM hinzu. - Fügen Sie Lösung A over Lösung B hinzu und mischen Sie vorsichtig durch Pipettieren.
- Inkubieren Sie die Mischung für 20 min bei Raumtemperatur. Fügen Sie den Zellen tropfenweise A- und B-Mischung hinzu.
- Tag 3-11: Überwachung der GFP-Expression durch Fluoreszenzmikroskopie
HINWEIS: Zellen sollten in der Fluoreszenzmikroskopie grün erscheinen und sich nach und nach lösen. - Tag 11: Ernte der adenoviralen F1-Partikel
- Sammeln Sie die abgetrennten Zellen und das Medium in einem 50 ml Rohr, kratzen Sie die anhaftenden Zellen und fügen Sie sie in der gleichen Röhre hinzu.
- Zentrifuge für 5 min bei 400 x g, den Überstand in einem neuen Rohr sammeln und das Zellpellet in 0,5 ml PBS wieder aufhängen.
- Zellunterbrechung
- Übertragen Sie die Zellsuspension in ein Mikrozentrifugenrohr.
- Führen Sie drei Gefrier-/Tauzyklen durch (Einfrieren in flüssigem Stickstoff oder bei -80 °C /Taubei bei 37 °C für maximal 7 min).
- Passieren Sie die gebrochenen Zellen dreimal durch eine 23 G Spritzennadel.
- Entfernen Sie die Zellablagerungen durch Zentrifugation bei 9.600 x g für 12 min.
- Übertragen Sie den Überstand mit dem gesammelten Medium auf das 50 ml-Rohr.
4. Verstärkung des Adenovirus
HINWEIS: Wenn die AD293-Zellen nicht den erforderlichen Zusammenfluss erreicht haben, können die Aliquots der adenoviralen Bestände (Lysat, das aus den virusproduzierenden Zellen gewonnen wird) zur Infektion bei -80 °C gelagert werden.
- Bereiten Sie die F2 adenoviralen Partikel vor.
- Säen Sie die AD293-Zellen in einem T75-Kolben (5 x 106 Zellen/Kolben).
- Infizieren Sie 90 % konfluente AD293-Zellen mit den adenoviralen F1-Partikeln: Fügen Sie das Zellhomogenat und das Medium aus dem T25-Kolben über die im T75-Kolben angebauten Zellen hinzu.
- Überwachen Sie die GFP-Expression durch Fluoreszenzmikroskopie.
- Ernten Sie die virusproduzierenden Zellen, wenn 90 % des transducierten AD293 abgetrennt sind (am5. Tag nach der Transduktion). Halten Sie das Zellkulturmedium bei 4 °C.
- Disruptieren Sie die Zellen (ähnlich wie bei F1) in 1 ml PBS.
- Bereiten Sie die Adenoviralpartikel F3 vor.
- Infizieren Sie 90 % konfluente AD293-Zellen, die in T175-Kolben mit F2-Adenoviralpartikeln und dem Zellkulturmedium aus den adenoviralen F2-Partikeln ausgesät sind.
- Ernte der Zellen (ca. 5 Tage nach der Transduktion).
- Disruptieren Sie die Zellen (ähnlich wie bei F1) in 2 ml PBS.
- Bereiten Sie die Adenoviralpartikel F4 vor.
- Infizieren Sie 5 T175-Kolben mit einem Konfluentad293-Zellen von 90 % mit F3-Adenoviralpartikeln und Zellkulturmedium.
- Ernte der Zellen (ca. 5 Tage nach der Transduktion).
- Disruptieren Sie die Zellen (ähnlich wie bei F1) in 3 ml PBS.
- Bereiten Sie die Adenoviralpartikel F5 vor.
- Infizieren Sie 25 T175-Kolben mit einem Konfluent ad293-Zellen von 90 % mit F4-Adenoviral-Bestand und Zellkulturmedium.
5. Reinigung des Adenovirus aus Zelllysat und Kulturmedium
- Ernte der virusproduzierenden Zellen und des Kulturmediums.
- Ernte der AD293-Zellen von F5 nach 5 Tagen nach Deruktion.
- Speichern Sie das Medium in einer sterilen Flasche für die Ausfällung der adenoviralen Partikel.
HINWEIS: Bewahren Sie das Medium bis zur Reinigung des Adenovirus im Kühlschrank auf. - Zentrifugieren Sie die Zellen bei 400 x g, für 5 min, bei 4 oC.
- Das endende Pellet in 5 ml von 10 mM Tris HCl, pH 8 mit 2 mM MgCl2wieder aufhängen.
- Aliquot die Suspension in 1,5 ml Rohre.
- Stören Sie die Zellen (ähnlich wie bei F1): drei Zyklen des Einfrierens/Auftauens.
HINWEIS: Wenn die Ultrazentrifugation nicht sofort durchgeführt werden kann, halten Sie die Proben bei -80 °C. - Passieren Sie die Zellsuspension dreimal durch eine 23G Spritzennadel.
- Zentrifugieren Sie das Homogenat bei 9 600 x g, für 12 min.
- Speichern Sie den Überstand für die Adenovirus-Reinigung durch CsCl Gradienten-Ultrazentrifugation.
- Niederschlag des Adenovirus im Kulturmedium freigesetzt.
- Bringen Sie die Flasche mit gespeichertem Zellkulturmedium bei Raumtemperatur.
- Fügen Sie 121 g Ammoniumsulfat zu jedem 500 ml Zellkulturmedium hinzu (Sättigung der Lösung sollte zwischen 40 - 42%) liegen.
- Sorgfältig mischen, bis das Ammoniumsulfat vollständig aufgelöst ist.
- Mindestens 2,5 Stunden bei Raumtemperatur inkubieren.
- Zentrifuge bei 1600 x g, für 15 min, bei 22 oCund das Pellet speichern.
- Das Pellet in 4 ml von 10mTris HCl pH 8 mit 2mM MgCl2wieder aufsetzen; Diese Suspension sollte sofort durch CsCl Gradienten-Ultrazentrifugation gereinigt werden.
HINWEIS: Wenn der Reinigungsschritt nicht nachträglich durchgeführt werden kann, dialysieren Sie über Nacht das resuspendierte Pellet gegen 10mM Tris HCl, pH 8 mit 2mM MgCl2.
- Adenovirus-Reinigung durch Ultrazentrifugation.
- Bereiten Sie einen diskontinuierlichen CsCl-Gradienten in Polypropylenrohren für den SW41Ti-Rotor vor. Fügen Sie 3 ml von 765 mg/ml CsCl (hohe Dichte: 1,4 g/L) an der Unterseite des Rohres hinzu. Fügen Sie langsam 3 ml von 288,5 mg/ml CsCl (niedrige Dichte: 1,2 g/L) auf die erste CsCl-Schicht.
- Überlagerung von 3 - 4 ml adenoviraler Partikelsuspension, die aus den Zellen freigesetzt oder aus dem Zellkulturmedium (wie zuvor beschrieben) auf dem Gradienten ausgefällt wird.
- Füllen Sie die Rohre mit Mineralöl und legen Sie die Rohre in die kalten SW41Ti Eimer.
- Gleichgewichten Sie die Rohre aus. Stellen Sie sicher, dass die gefüllten Polypropylenrohre symmetrisch in den Rotor geladen werden. Setzen Sie den Rotor in die Ultrazentrifuge.
- Zentrifuge bei 210.000 x g und 4 °C, 18 Stunden lang ohne Bremse.
- Legen Sie die Ultrazentrifugenrohre auf einen Ständer mit einem schwarzen Papier dahinter, um die Bänder zu bekommen.
- Entsorgen Sie die klare obere Phase, den Zellabfall und das obere Band in einem Abfallbehälter mit der Bleichlösung.
- Ernten Sie das niedrigste Band, das das komplette Adenovirus (ca. 700 l - 1 ml) in einem sterilen 1,5 ml-Rohr enthält, und halten Sie es auf Eis.
- Vorbenetzung einer Dialysekassette im Dialysepuffer (10 mM Tris-Cl Puffer pH 8, 2 mM MgCl2).
- Injizieren Sie das gereinigte Adenovirus mit einer 2 ml Spritze in die Dialysekassette.
- Dialyse über Nacht gegen 10 mM Tris-Cl Puffer pH 8, 2 mM MgCl2 (Den Dialysepuffer 3 - 4 Mal ändern).
- Ernten Sie den adenoviralen Vorrat aus der Dialysekassette in Aliquots von 10 - 100 l.
- Saccharose zu 4% Endkonzentration zu viralen Aliquots hinzufügen (zum Kryoschutz).
- Aliquots bei -80 °C lagern.
6. Adenovirus-Titration
- Tag 1: Plattieren der Zellen
- Samen Sie die AD293-Zellen mit einer Dichte von 2,5 × 105 Zellen pro Brunnen (in einer 12-Well-Kulturplatte) in 1 ml vollständigem Wachstumsmedium, wie in Abbildung 2dargestellt. Stellen Sie sicher, dass die Zellen gleichmäßig in jedem Brunnen verteilt sind, um eine genaue Titerbestimmung zu erhalten.
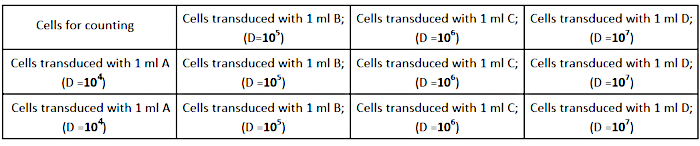
Abbildung 2: Titrationsplattendesign. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
- Tag 2: Transduktion von Zellen
- Lösen Sie die Zellen von einem Brunnen mit Trypsin und zählen Sie sie. Beachten Sie diese Zahl, da sie zur Berechnung des viralen Titers verwendet wird.
- Führen Sie serielle Verdünnungen (1/104; 1/105; 1/106; 1/107) des Virusbestandes in 1 ml vollständigem Wachstumsmedium wie folgt durch:
- 1/103: Verdünnung des Virusbestands - 2 L Virusbestand zu 1998 l des gesamten Mediums hinzufügen.
- 1/104: 1:10 Verdünnung von 1/103 durch Verdünnung von 120 l auf 1080 l des kompletten Mediums (A) vornehmen.
- 1/105: 1:10 Verdünnung von B durch Verdünnung von 120 l A bis 1080 l des kompletten Mediums (B) vornehmen.
- 1/106: 1:10 Verdünnung von C durch Verdünnung von 120 l B bis 1080 l des kompletten Mediums (C) vornehmen.
- 1/107: 1:10 Verdünnung von D durch Verdünnung von 120 l C bis 1080 l des kompletten Mediums (D) vornehmen.
HINWEIS: Bereiten Sie 3 Röhren jeder Verdünnung (A, B, C, D) vor, um das Experiment in Triplicaten durchzuführen. - Entfernen Sie das Zellkulturmedium aus den Brunnen und fügen Sie die vorbereiteten Verdünnungen des Virus hinzu, wie in Abbildung 2dargestellt.
- Tag 3: Überwachung der GFP-Expression
- Überprüfen Sie die Brunnen auf das Vorhandensein von grünen Zellen mit einem Fluoreszenzmikroskop.
- Tag 4: Durchflusszytometrieanalyse von GFP-positiven Zellen
- Zwölf 1,5 ml-Rohre vorbereiten und beschriften.
- Sammeln Sie das Zellkulturmedium (zusammen mit den freistehenden Zellen) in 1,5 ml-Röhren und halten Sie es auf Eis.
- Fügen Sie in jedem Brunnen 200 l Trypsin hinzu.
- Inkubieren Sie die Platte für 2 - 3 min bei 37 °C im CO2-Inkubator.
- Ernten Sie die Zellen in den gleichen Eppendorf-Röhren mit dem Zellkulturmedium. Halten Sie die Rohre auf Eis.
- Pellet die Zellen bei 400 x g, für 5 min, bei 4 °C.
- Entfernen Sie den Überstand; die Rohre auf Eis halten.
- Das Pellet in 250 l PBS + 2% FBS wieder aufhängen; die Rohre auf Eis halten.
- Übertragen Sie die Zellsuspension in Durchflusszytometrieröhrchen oder Platten.
- Führen Sie die Proben auf einem Durchflusszytometer aus, das die Fluoreszenz der GFP-exezierenden Zellen aufzeichnet.
Titer-Berechnung: Die Proben mit 5 - 20% GFP-positiven Zellen aus der Stammpopulation sollten bei der Berechnung des viralen Titers nach folgender Formel berücksichtigt werden:
Titer (TU/mL) = D x F/100 x C/V
D = Verdünnungsfaktor
F = Prozent der positiven Zellen / 100
C = Anzahl der Zellen / gut
V = Volumen des viralen Inokulums
7. Adenovirale Transduktion von Zielzellen und Prüfung der induzierten Proteinexpression
- Tag 1: Aussaat der Zellen
- Samen Sie die Zielzellen, um sicherzustellen, dass sie gleichmäßig in den Brunnen verteilt werden.
- Tag 2: Transduktion der Zellen
- Lösen Sie die Zielzellen von einem Brunnen und zählen Sie sie.
- Berechnen Sie das geeignete Volumen der adenoviralen Suspension, das erforderlich ist, um die Zellen mit der gewünschten Anzahl von infektiösen Partikeln pro Zelle zu transducieren.
- Fügen Sie die entsprechende Menge an viraler Suspension zu den Zielzellen hinzu.
- Tag 3: Entfernung der viralen Suspension und Überprüfung auf GFP-Expression
- Ersetzen Sie das Zellkulturmedium, das adenovirale Partikel enthält, durch frisches Medium.
- Überprüfen Sie die GFP-Expression am Fluoreszenzmikroskop.
Ergebnisse
Wir haben das ursprüngliche Vogelstein-Protokoll modifiziert und verbessert, um eine schnellere und effizientere Adenovirus-Produktion zu erreichen. Zunächst haben wir die Methodik überarbeitet, um eine einfachere Auswahl von Rekombinanten zu erreichen. Nach der Rekombination wurden die BJ5183 bakteriellen Klone mit "negativer PCR" getestet, um die Integrität von pAdTrack-GFP als Indikator für die fehlende Rekombination zu bewerten (Abbildung 3A), oder durch "positive PCR", um das Gen von Interesse zu identifizieren, in unserem Fall gfP assimiliert (Abbildung 3B). Sowohl in "negativen" als auch in "positiven" PCRs verwendeten wir pAdTrack-GFP als Steuervorlage, die ein Band von 986 bp für pAdTrack-Integrität(Abbildung 3A, Spur 1) und ein Band von 264 bp für GFP(Abbildung 3B, Spur 3) gab. Die für die "negative PCR" verwendeten Primer wurden entwickelt, um ein Fragment von 986 bp zu verstärken, das die PmeI-Site in pAdTrack-GFP enthält. Dieses DNA-Fragment wird nach der Rekombination drastisch vergrößert und nicht in den positiven rekombinanten Klonen verstärkt. Negative Klone zur Rekombination, bei denen pAdTrack-GFP intakt blieb, sind in Abbildung 3A, Bahnen 3, 4 und 6 dargestellt. Die Primer glühen auf den DNA-Sequenzen neben der Rekombinationsstelle. Potenzielle positive rekombinante Klone(Abbildung 3A, Bahnen 2 und 5) ausgedrückt GFP wie in Abbildung 3B, Spur 1 und 2 dargestellt. Plasmid-DNA aus diesen Klonen wurde isoliert und für die DH5-Transformation verwendet, um eine höhere Menge an DNA zu erhalten. Diese vorgewählten rekombinanten Plasmide, die in DH5 verstärkt wurden, wurden dann durch enzymatische Verdauung getestet. In Abbildung 3c-E sind die Ergebnisse der enzymatischen Verdauung eines rekombinant-positiven Klons dargestellt, der mit Hind III, PstI, BamHI-Restriktionsenzymen verdaut wurde (Abbildung 3C, D, E Lane 2). Die HindIII- und PstI-Verdauungsmuster des rekombinanten Klons ähnelten denen, die für pAdEasy-1 erhalten wurden, da HindIII und PstI das pAdEasy-1-Plasmid 24 bzw. 25 Mal geschnitten haben(Abbildung 3C und D,Spur 3); HindIII einmal geschnitten und PstI viermal den pAdTrack-GFP-Vektor geschnitten(Abbildung 3C und D, Spur 1). BamHI schnitt zweimal pAdEasy-1 Vektor (Abbildung 3C, Spur 3), und einmal pAdTrack-GFP (Abbildung 3C, Spur 1).
PacI schnitt ein Fragment von 4,5 kb aus dem rekombinanten Plasmid (Abbildung 3F, Spur 2), ein Fragment von 2863 bp aus pAdTrack-GFP (Abbildung 3F, Spur 1) und linearisierte den pAdEasy-1-Vektor (Abbildung 3F, Spur 3). Die DNA-Leiter ist in Abbildung 3C-Fin Bahnen 4 dargestellt. Das rekombinante Plasmid wurde mit Pac I für die weitere Verwendung zur AD293-Transfektion verdaut.
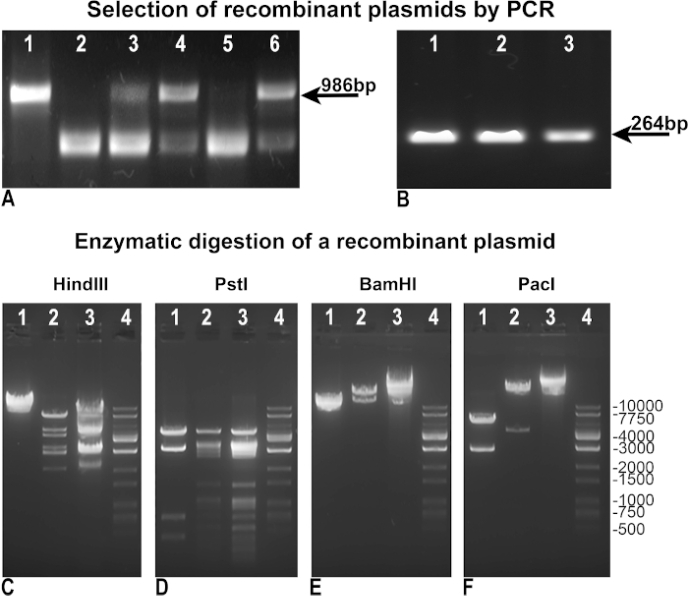
Abbildung 3: Die Rekombination von pAdTrack-GFP mit dem pAdEasy-1-Plasmid. Die nach der Rekombination von pAdTrack-GFP und pAdEasy-1 erhaltenen Plasmiden wurden mit "negativer" PCR auf die pAdTrack-GFP-Integrität (A) getestet. Die nicht rekombinanten Klone wurden durch das Vorhandensein eines 986 bp-Bands nachgewiesen, das der Sequenz entspricht, die aus dem pAdTrack-GFP-Plasmid verstärkt wird (A, Bahnen 3, 4 und 6). Die klonenpotenziell positiv für die Rekombination (A, Bahnen 2 und 5) wurden auch erhalten. Als der pAdTrack-GFP-Vektor als Vorlage verwendet wurde, wurde ein Band von 986 bp für pAdTrack-GFP (A, Lane 1) erhalten. Die potenziell positiven rekombinanten Klone wurden auf GFP-Expression mit "positiver" PCR (B) getestet; ein Band von 264 bp erscheint sowohl für potenziell rekombinierte Klone (B, Lane 1 und 2), als auch für das pAdTrack-GFP-Plasmid. Die DNA eines potenziellen rekombinanten Klons wurde mit HindIII, PstI, BamHI und PacI-Restriktionsenzym (C-F, Bahnen 2) getestet. In den Kontrollen wurden der pAdEasy-1-Vektor (C-F, Bahnen 3) und das pAdTrack-GFP-Plasmid (C-F, Bahnen 1) mit den gleichen Enzymen verdaut. Die DNA-Leiter ist in Der C-F-Spur 4 dargestellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Die adenovirale Verpackung und Amplifikation wurde in AD293-Zellen durchgeführt. Die adenoviralen Partikel (AdV-GFP) wurden aus dem AD293-Zelllysat sowie aus dem Zellkulturmedium gereinigt, wo sie von den infizierten Zellen freigesetzt worden waren. Um das im Zellkulturmedium gefundene Adenovirus zu konzentrieren, wurden die Partikel mit Ammoniumsulfat ausgefällt und dann in 10 mM Tris HCl pH 8 mit 2 mM MgCl2,dem gleichen Puffer wie bei der Zelllyse, resuspendiert. Anschließend wurden die adenoviralen Partikel aus dem Zelllysat und aus dem Kulturmedium durch Diskontinuierliche Gradienten-Ultrazentrifugation von CsCl gereinigt. Nach der Ultrazentrifugation wurde ein starkes Band von gereinigtem AdV-GFP erhalten, wie in Abbildung 4dargestellt.

Abbildung 4: Die adenovirale Reinigung durch Ultrazentrifugation bei diskontinuierlichem CsCl-Gradienten. Das Zellhomogenat und das aus dem Medium ausgefällte Adenovirus wurden einer Ultrazentrifugation auf einem diskontinuierlichen Gradienten ausgesetzt, der durch CsCl-Lösungen mit niedriger und hoher Dichte gebildet wurde. Starke Bänder von GFP- Adenovirus wurden in beiden Fällen nachgewiesen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Zur Bestimmung des viralen Titers, der in Transducing Units pro mL (TU/mL) exprimiert wurde, wurden die AD293-Zellen mit seriellen Verdünnungen des AdV-GFP infiziert. Nach 48 Stunden exprimierte die infizierten Zellen GFP, in umgekehrter Korrelation mit dem Verdünnungsfaktor der viralen Suspension. Dies wurde durch Fluoreszenzmikroskopie beobachtet und der Prozentsatz der GFP-positiven Zellen wurde durch Durchflusszytometrie bestimmt (Abbildung 5). Zur Berechnung des Titers wurde die virale Verdünnung berücksichtigt, die 5 - 20% der GFP-positiven Zellen induzierte (Abbildung 5C). In der Regel erhalten wir einen viralen Titer von 1010 DOLLAR (TU/ml) für das GFP-Adenovirus.
Im Folgenden stellen wir ein Beispiel für eine adenovirale Titerberechnung für eine bestimmte adenovirale Charge dar, bei der 300000 Zellen (C) mit 1 ml adenoviraler Lösung (V) bei einem Verdünnungsfaktor von 106 (D) transduziert wurden, für die 6% GFP-positive Zellen (F) erhalten wurden:
Titer (TU/mL) = D x F/100 x C/V = 106 x 6/100 x 300000/1 = 1,8 x 1010 TU/mL
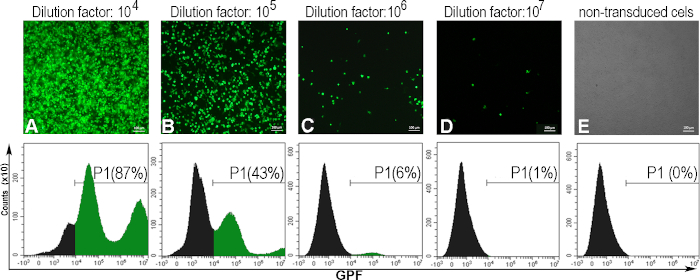
Abbildung 5: Die Bewertung des adenoviralen Titers. AD293-Zellen wurden mit verschiedenen adenoviralen Verdünnungen infiziert. Achtundvierzig Stunden später wurden die Zellen durch Fluoreszenzmikroskopie beobachtet und durch Durchflusszytometrie analysiert, um den Prozentsatz der GFP-positiven Zellen zu bestimmen, der durch verschiedene adenovirale Verdünnungen (A-D) induziert wurde. Um das Tor für die Durchflusszytometrie zu etablieren, wurden auch nicht-transducierte Zellen analysiert (E). Der für den Verdünnungsfaktor 106berechnete Titer, wenn 6% der Zellen GFP-positiv waren, betrug 1,8 x 1010 TU/ml. Für Paneele A-E, Balken: 100 m. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Um das Transduktionspotential des präparierten Adenovirus zu testen, wurden vier Zelllinien verwendet: menschliche Endothelzellen (EA.hy926), Rinderaorten-Endothelzellen (BAEC), murine Hepatozyten (Hepa 1-6) und murine mesenchymale Stromalzellen (MSC). Endothelzellen (EA.hy926 und BAEC) wurden mit 25 TU/Zelle transduziert, die Hepatozyten mit 5 TU/Zelle und MSC mit 250 TU/Zelle transduziert.
Hier ist ein Beispiel dafür, wie das Volumen der adenoviralen Suspension berechnet wurde, die benötigt wurde, um 3 x 106 Zellen mit 25 TU/Zelle mit der adenoviralen Suspension mit 1,8 x 1010 TU/ml zu infizieren.
Für 1 Zelle .............. 25 TU
3 x 106 Zellen .............. x TU  x=75 x 106 TU
x=75 x 106 TU
Wenn der virale Bestand
1.8 x 1010 TU .............. 1 ml
75 x 106 TU .............. y mL  y= 4,2 x 10-3 ml = 4,2 l Virusbestand
y= 4,2 x 10-3 ml = 4,2 l Virusbestand
Achtundvierzig Stunden nach der Transduktion wurden die Zellen mittels Fluoreszenzmikroskopie analysiert. Wie in Abbildung 6dargestellt, wurden menschliche oder rinderische Endothelzellen mit guter Effizienz transduziert (ca. 50%) für 25 TU/Zelle(Abbildung 6 EA.hy926 und BAEC). Murine Hepatozyten (Hepa 1-6) wurden durch das Adenovirus effizient bei einer geringen Menge an Adenovirus-Partikeln (5 TU/Zelle) transduziert, aber sie sind auch empfindlich gegenüber dem Adenovirus, da ein höherer Prozentsatz abgestorbener Zellen (PI-positive Zellen) aufgezeichnet wurde (16 %) im Vergleich zu den anderen Zelltypen. Mesenchymale Stromalzellen waren aufgrund des Fehlens spezifischer adenoviraler Rezeptoren (unveröffentlichte Daten) am schwierigsten zu transduzieren (Abbildung 6).
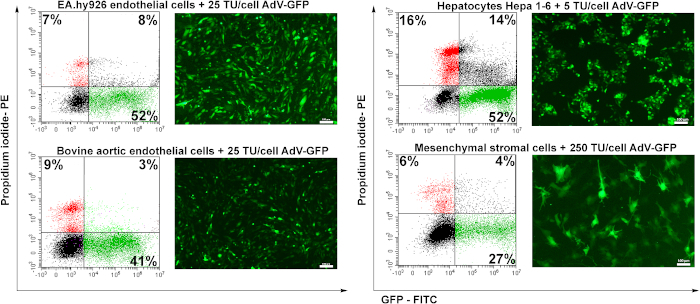
Abbildung 6: Die Infektiosität des Adenovirus und die Induktion der GFP-Expression in transduzierten Zellen. Menschliche Endothelzellen (EA.hy926), Rinderaorten-Endothelzellen (BAEC), murine Hepatozyten (Hepa 1-6) und murine mesenchymale Stromalzellen (MSC) wurden mit der angegebenen Menge an Adenovirus transduziert. GfP wurde durch Fluoreszenzmikroskopie nachgewiesen und der Prozentsatz der GFP-positiven Zellen wurde durch Durchflusszytometrie analysiert. PI-positive Zellen, die durch Durchflusszytometrie bestimmt werden, zeigen die zellmortale Zellsterblichkeit, die durch virale Transduktion bestimmt wird. EA.hy926 Zellen, Rinderaorten-Endothelzellen und Hepa 1-6-Zellen wurden durch das Adenovirus hochtransduziert, die Ausbeute der Transduktion reichte von 41 - 52%. Bei MSC induzierte eine höhere Virusmenge (250 TU/Zellen) nur 27% GFP-positiv der transduzierten Zellen. Balken: 100 m. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Rekombinante Adenoviren sind ein vielseitiges Werkzeug für die Genabgabe und Expression12,13,14. Um eine starke Proteinexpression durch adenovirale Transduktion zu induzieren, wird die Kodierungssequenz des Gens von Interesse in das Genom des Adenovirus eingefügt. Das adEasy adenovirale System, das im Labor von Bert Vogelstein entwickelt wurde, besteht aus einem Backbone-Plasmid (pAdEasy-1), das den größten Teil des Wilden-Adenovirus-Serotyp-5-Genoms enthält, und einem Shuttle-Vektor (pAdTrack), der für das Klonen vonGenen2,10. Die Deletion der adenoviralen Gene E1 (zuständig für die Montage von infektiösen Viruspartikeln) und E3 (kodierungsproteine Proteine, die an der Umgehung der Wirtsimmunität beteiligt sind) schuf einen Raum im adenoviralen Genom, in dem ein Gen von Interesse von 6,5-7,5 kb eingefügt werden kann2,3. Diese Größe ist ausreichend für viele Gene, vor allem für diejenigen mit kürzeren Introns15,16,17. Es gibt auch Forscher, die über die Produktion von Adenoviren berichten, die die cDNA eines Transgens18,19,20tragen. Wir erhielten jedoch eine geringere Ausbeute an transgener Expression für cDNA-tragende Adenoviren als für ihre Gegenstücke, die ein Gen oder ein Mini-Gen tragen (Daten nicht gezeigt).
Die Verbesserung und Anpassung der bisherigen Methoden2,10,14,18,21, die Technologie für die adenovirale Produktion erfordert eine kürzere Zeit, geringere Kosten und weniger Aufwand. Die adenovirale DNA in voller Länge wird durch Rekombination zwischen dem Shuttle-Vektor und dem pAdEasy-1-Plasmid in der homologen Rekombination anfälligen E. coli-Stamm, BJ5183, erhalten. Das Protokoll impliziert die chemische Umwandlung von AdEasier-1-Zellen (BJ5183-Bakterien, die pAdEasy-1 enthalten). Diese Technik erfordert keinen Elektroporator, der in einigen Laboratorien möglicherweise nicht verfügbar ist, sehr einfach ist, die Rekombinationsausbeute erhöht und die Zeit reduziert, die erforderlich ist, um kompetente Zellen zu erhalten und die Transformation durchzuführen. Die Vorauswahl rekombinanter Klone, die von PCR durchgeführt werden, verkürzt die Zeit weiter und erleichtert den gesamten Ablauf. Ein ähnliches Verfahren wurde von Zhao und Kollegen22verwendet, aber im Protokoll optimierten wir die Sequenzen der Primer.
Für die GFP-Adenovirus-Verpackung und -Verstärkung wurde eine HEK293-Derivatzelllinie verwendet, nämlich AD293-Zellen, die stärker an der Kulturplatte haften. Andere Zelllinien, die häufig für die adenovirale Produktion verwendet werden, sind die folgenden: 911, 293FT, pTG6559 (A549 Derivat), PER. C6 (HER-Derivat), GH329 (HeLa-Derivat), N52. E6 und HeLa-E123,24,25,26. In unseren Händen wurde keine Verbesserung der adenoviralen Produktion erzielt, als 911 Zellen verwendet wurden (Daten nicht gezeigt). Die Transfektion von AD293-Zellen mit dem rekombinanten Plasmid mit K2-Reagenz steigerte die Effizienz des viralen Verpackungsschritts stark. Nach der Adenovirus-Produktion befinden sich bis zu 70 % des Adenovirus noch in den Zellen und werden durch drei Gefrier- und Auftauzyklen freigesetzt. Die Erhöhung der Anzahl der Zyklen ist nicht geeignet, weil es das Adenovirus zerstört.
Während des routinemäßigen adenoviralen Produktionsprozesses werden zahlreiche virusvirale Partikel im Zellkulturmedium freigesetzt. Die Verwirft dieses Zellkulturmediums während der Ernte der infizierten AD293-Zellen würde zu einem wichtigen viralen Verlust führen. Wir haben das von Schagen und Kollegen beschriebene Protokoll optimiert, um die adenoviralen Partikel aus dem Zellkulturmedium durch Niederschlag mit Ammoniumsulfat27zu reinigen. Diese Methode hat eine höhere Effizienz in adenovirus Erholung aus dem Zellkulturmedium im Vergleich zu der Methode mit Polyethylenglykol28. Das gefällte Adenovirus sollte sofort durch Ultrazentrifugation gereinigt oder für ein paar Tage im Kühlschrank aufbewahrt werden, aber nur nach der Dialyse, um den Salzüberschuss zu entfernen. Den Niederschlag länger als ein paar Stunden ohne Dialyse zu halten, ist schädlich für das Virus.
Die Reinigung der adenoviralen Partikel durch ultrazentrifugierende Partikel in einem Schritt reduziert die Manipulation des adenoviralen Bestands und erleichtert das Verfahren im Vergleich zu den Protokollen mit aufeinanderfolgenden Ultrazentrifugationsschritten14,29. Die Dialyse des gereinigten Adenovirus ist notwendig, um Cäsiumchlorid zu entfernen, das die Transduktion weiter beeinträchtigen kann. Im Protokoll verwendeten wir Tris-Puffer, der MgCl2, aber nicht Saccharose für die Dialyse enthält, da es eine riesige, ungerechtfertigte Menge Saccharose erfordert, die sonst als Konservierungsmittel zum Einfrieren benötigt wird. So fügten wir Saccharose später direkt in die adenoviralen Bestände ein, die zum Einfrieren vorbereitet wurden. Um ein häufiges Einfrieren und Auftauen des gereinigten Adenovirus zu vermeiden, ist es ratsam, die adenoviralen Bestände zu aliquotieren und bei -80 °C zu lagern. Der adenovirale Titer wurde durch Durchflusszytometrie unter Berücksichtigung des GFP-Reportergens und des Prozentsatzes der transduzierten Zellen für eine bestimmte virale Verdünnung bewertet. Diese Methode ist schneller im Vergleich zum klassischen "Plaque-Assay" und ist vertrauensvoller als die Auswertung der Kapsidproteine (durch verschiedene Methoden wie ELISA oder Durchflusszytometrie), die nicht die Fähigkeit der Infektion der adenoviralen Partikel offenbaren. ELISA-basierte Quantifizierung, Q-PCR oder Plaque-Assay mit kommerziell erhältlichen Kits sind jedoch alternative Methoden, die besonders nützlich für die Titration von Adenoviren sind, die keinen fluoreszierenden Tracer enthalten.
In Anbetracht der Tatsache, dass pAdTrack-Adenoviren von menschlichen Adenoviren Serotyp 5 abgeleitet sind, der durch Coxsackievirus und Adenovirus-Rezeptoren (CAR) erkannt wird, haben wir die Fähigkeit des GFP-Adenovirus demonstriert, Zellen menschlichen Ursprungs (Endothelzellen), aber auch Zellen anderer Herkunft zu transduzieren, aber auch Zellen anderer Herkunft: Rinder (Endothelzellen) und Murine (mesenchymale Stromzellen). Die Daten zeigten, dass das GFP-Adenovirus eine hohe Expression eines Transgens induzieren kann.
Zusammenfassend haben wir diese mühsame Technologie optimiert, um die Zeit, die Kosten und den Aufwand zu reduzieren, der erforderlich ist, um die adenoviralen Partikel zu erhalten. Das hergestellte Adenovirus ist in der Lage, verschiedene Zelltypen zu infizieren und die Expression des Gens von Interesse zu induzieren. Dieses Protokoll kann in einer Vielzahl von Experimenten verwendet werden, da der adenoviral-vermittelte Gentransfer eines der wichtigsten Werkzeuge für die Entwicklung moderner Gentherapien darstellt.
ABBREVIATIONS: AdV-GFP, adenovirale Partikel; BAEC, aortenatotäre Endothelzellen; CsCl, Cäsiumchlorid; GFP, grünes fluoreszierendes Protein; MSC, mesenchymale Stromalzellen; TU, Messeinheiten.
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Diese Arbeit wurde durch ein Projekt unterstützt, das aus dem Europäischen Fonds für regionale Entwicklung aus dem operationellen Programm für Wettbewerbsfähigkeit 2014-2020 (POC-A.1-A.1.1.4-E-2015, ID: P_37_668; Akronym DIABETER), ein Stipendium des rumänischen Ministeriums für Forschung und Innovation PCCDI- UEFISCDI, Projektnummer PN-III-P1-1.2-PCCDI-2017-0697 innerhalb der PNCD III und der Rumänischen Akademie. Die Autoren danken Kyriakos Kypreos (Universität Patras, Griechenland) für seine großzügige und sachdienliche Beratung, Ovidiu Croitoru (Universität der Schönen Künste, Bukarest, Rumänien) für Dreharbeiten, Filmschnitt und grafisches Design und Mihaela Bratu für technische Hilfe.
Materialien
| Name | Company | Catalog Number | Comments |
| AD293 cells | Agilent Technologies | 240085 | |
| AdEasier-1 cells | Addgene | 16399 | |
| Agarose I (for electrophoresis) | Thermo Scientific | 17850 | |
| Ammonium sulfate | Sigma | A4418 | |
| Ampicillin sodium salt | Sigma | A0166 | |
| BamH I | Thermo Scientific | FD0054 | |
| Cell culture plates 100 mm | Eppendorf | 30702115 | |
| Cesium chloride | Sigma | L4036 | |
| DH5alpha bacteria | Thermo Scientific | 18265017 | |
| DMEM (GlutaMAX, 4.5g/L D-Glucose) | Gibco | 3240-027 | |
| EA.hy926 cells | ATCC | CRL-2922 | |
| EDTA | Sigma | E5134 | |
| Ethanol (99.8%) | Roth | 5054.2 | |
| Fetal Bovine Serum | Sigma | F7524 | |
| Flasks T25, T75, T175 | Eppendorf | 30712129 | |
| Glucose | Sigma | G7021 | |
| Hepa 1-6 murine hepatocytes | ATCC | CRL-1830 | |
| Hind III | Thermo Scientific | FD0504 | |
| Kanamycin Sulfate | Thermo Scientific | 15160054 | |
| K2 Transfection System | Biontex | T060-5.0 | |
| LB medium | Formedium | LBx0102 | |
| LB-agar | Formedium | LBx0202 | |
| Mix & Go E. coli Transformation kit | Zymo Research | T3001 | |
| Midori Green Advanced DNA stain | Nippon Genetics Europe | MG-04 | |
| NaOH | Sigma | S8045 | |
| Opti-MEM | Thermo Scientific | 31985070 | |
| Pac I | Thermo Scientific | FD2204 | |
| pAdEasy-1 | Addgene | 16400 | |
| pAdTrack-CMV | Addgene | 16405 | |
| Phenol:chloroform:isoamyl alcohol (24:24:1) | Invitrogen | 15593-031 | |
| Polymerase GoTaq | Promega | M3005 | |
| Pme I (Mss I) | Thermo Scientific | FD1344 | |
| Potassium acetate | VWR Chemicals | 43065P | |
| Pst I | Thermo Scientific | FD0614 | |
| Qiagen Midi Prep kit | Qiagen | 12125 | |
| Cell Scraper | TPP | 99003 | |
| SDS | Thermo Scientific | 28365 | |
| Slide-A-Lyzer dialysis cassettes | Thermo Scientific | 66330 | |
| Sodium pyruvate | SIGMA | P5280-100G | |
| Syringe with 23G neeedle | B Braun | 464BR | |
| Tris HCl | Sigma | 1185-53-1 | |
| Trypan blue | Roth | CN76.1 | |
| Tubes 50ml | TPP | 91050 | |
| Ultra-Clear Tubes (14x89 mm) | Beckman Coulter | 344059 | |
| Centrifuge (refrigerated) | Sigma Sartorius | 3-19KS | |
| HeraeusFresco 17 Microcentrifuge | Thermo Scientific | 75002420 | |
| Ultracentrifuge with SW41Ti rotor | Beckman Coulter | Optima L-80 XP | |
| Culture Hood | Thermo Scientific | Class II | |
| Pipettes (0-2µl, 1-10µl, 2-20µl, 10-100µl, 20-200µl, 100-1000µl) | Thermo Scientific | ||
| Dry Block Heating Thermostat | Biosan | TDB-120 | |
| Thermocycle | SensoQuest | 012-103 | |
| Water Bath | Memmert | WNB 14 |
Referenzen
- Lee, C. S., et al. Adenovirus-Mediated Gene Delivery: Potential Applications for Gene and Cell-Based Therapies in the New Era of Personalized Medicine. Genes and Diseases. 4 (2), 43-63 (2017).
- He, T. C., et al. A simplified system for generating recombinant adenoviruses. Proceedings of the National Academy of Sciences of the United States of America. 95 (5), 2509-2514 (1998).
- Russell, W. C. Update on adenovirus and its vectors. The Journal of General Virology. 81, 2573-2604 (2000).
- Rauschhuber, C., Noske, N., Ehrhardt, A. New insights into stability of recombinant adenovirus vector genomes in mammalian cells. European Journal of Cell Biology. 91 (1), 2-9 (2012).
- Saha, B., Wong, C. M., Parks, R. J. The adenovirus genome contributes to the structural stability of the virion. Viruses. 6 (9), 3563-3583 (2014).
- Kreppel, F., Kochanek, S. Modification of adenovirus gene transfer vectors with synthetic polymers: a scientific review and technical guide. Molecular Therapy: the Journal of the American Society of Gene Therapy. 16 (1), 16-29 (2008).
- Dormond, E., Perrier, M., Kamen, A. From the first to the third generation adenoviral vector: what parameters are governing the production yield. Biotechnol Advances. 27 (2), 133-144 (2009).
- Parks, R. J., et al. A helper-dependent adenovirus vector system: removal of helper virus by Cre-mediated excision of the viral packaging signal. Proceedings of the National Academy of Sciences of the United States of America. 93 (24), 13565-13570 (1996).
- Jager, L., Ehrhardt, A. Emerging adenoviral vectors for stable correction of genetic disorders. Current Gene Therapy. 7 (4), 272-283 (2007).
- Luo, J., et al. A protocol for rapid generation of recombinant adenoviruses using the AdEasy system. Nature Protocols. 2 (5), 1236-1247 (2007).
- Dumitrescu, M., et al. Adenovirus-Mediated FasL Minigene Transfer Endows Transduced Cells with Killer Potential. International Journal of Molecular Sciences. 21 (17), (2020).
- Campos, S. K., Barry, M. A. Current advances and future challenges in Adenoviral vector biology and targeting. Current Gene Therapy. 7 (3), 189-204 (2007).
- Khare, R., Chen, C. Y., Weaver, E. A., Barry, M. A. Advances and future challenges in adenoviral vector pharmacology and targeting. Current Gene Therapy. 11 (4), 241-258 (2011).
- Jager, L., et al. A rapid protocol for construction and production of high-capacity adenoviral vectors. Nature Protocols. 4 (4), 547-564 (2009).
- Zvintzou, E., et al. Pleiotropic effects of apolipoprotein C3 on HDL functionality and adipose tissue metabolic activity. Journal of Lipid Research. 58 (9), 1869-1883 (2017).
- Karavia, E. A., et al. Apolipoprotein A-I modulates processes associated with diet-induced nonalcoholic fatty liver disease in mice. Molecular Medicine. 18, 901-912 (2012).
- Lampropoulou, A., Zannis, V. I., Kypreos, K. E. Pharmacodynamic and pharmacokinetic analysis of apoE4 [L261A, W264A, F265A, L268A, V269A], a recombinant apolipoprotein E variant with improved biological properties. Biochemical Pharmacology. 84 (11), 1451-1458 (2012).
- Zheng, S. Y., Li, D. C., Zhang, Z. D., Zhao, J., Ge, J. F. Adenovirus-mediated FasL gene transfer into human gastric carcinoma. World Journal of Gastroenterology. 11 (22), 3446-3450 (2005).
- Ambar, B. B., et al. Treatment of experimental glioma by administration of adenoviral vectors expressing Fas ligand. Human Gene Therapy. 10 (10), 1641-1648 (1999).
- Okuyama, T., et al. Efficient Fas-ligand gene expression in rodent liver after intravenous injection of a recombinant adenovirus by the use of a Cre-mediated switching system. Gene Therapy. 5 (8), 1047-1053 (1998).
- van Dijk, K. W., Kypreos, K. E., Fallaux, F. J., Hageman, J. Adenovirus-mediated gene transfer. Methods in Molecular Biology. 693, 321-343 (2011).
- Zhao, Y. D., Li, T., Huang, G. A simple negative selection method to identify adenovirus recombinants using colony PCR. Electronic Journal of Biotechnology, North America. 17 (1), 46-49 (2014).
- Kovesdi, I., Hedley, S. J. Adenoviral producer cells. Viruses. 2 (8), 1681-1703 (2010).
- Lin, X. Construction of new retroviral producer cells from adenoviral and retroviral vectors. Gene Therapy. 5 (9), 1251-1258 (1998).
- Fallaux, F. J., et al. Characterization of 911: a new helper cell line for the titration and propagation of early region 1-deleted adenoviral vectors. Human Gene Therapy. 7 (2), 215-222 (1996).
- Altaras, N. E., et al. Production and formulation of adenovirus vectors. Advances in Biochemical Engineering/ Biotechnology. 99, 193-260 (2005).
- Schagen, F. H., et al. Ammonium sulphate precipitation of recombinant adenovirus from culture medium: an easy method to increase the total virus yield. Gene Therapy. 7 (18), 1570-1574 (2000).
- Colombet, J., et al. Virioplankton 'pegylation': use of PEG (polyethylene glycol) to concentrate and purify viruses in pelagic ecosystems. Journal of Microbiological Methods. 71 (3), 212-219 (2007).
- Kypreos, K. E., van Dijk, K. W., van Der Zee, A., Havekes, L. M., Zannis, V. I. Domains of apolipoprotein E contributing to triglyceride and cholesterol homeostasis in vivo. Carboxyl-terminal region 203-299 promotes hepatic very low density lipoprotein-triglyceride secretion. Journal of Biological Chemistry. 276 (23), 19778-19786 (2001).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten