Method Article
Un metodo efficiente per la produzione di adenovirus
In questo articolo
Riepilogo
Qui, presentiamo un protocollo per la produzione di adenovirus utilizzando il sistema pAdEasy. La tecnologia include la ricombinazione dei plasmidi pAdTrack e pAdEasy-1, l'imballaggio e l'amplificazione dell'adenovirus, la purificazione delle particelle adenovirali dal litorato cellulare e dal mezzo di coltura, la titolazione virale e il test funzionale dell'adenovirus.
Abstract
La trasduzione adenovirale ha il vantaggio di un'induzione forte e transitoria dell'espressione del gene di interesse in un'ampia varietà di tipi cellulari e organi. Tuttavia, la tecnologia adenovirale ricombinante è laboriosa, dispendiosa in termini di tempo e costosa. Qui, presentiamo un protocollo migliorato utilizzando il sistema pAdEasy per ottenere particelle adenovirali purificate che possono indurre una forte espressione di proteine fluorescenti verdi (GFP) nelle cellule trasdotta. I vantaggi di questo metodo migliorato sono una preparazione più rapida e una riduzione dei costi di produzione rispetto al metodo originale sviluppato da Bert Vogelstein. I passaggi principali della tecnologia adenovirale sono: (1) la ricombinazione di pAdTrack-GFP con il plasmide pAdEasy-1 nei batteri BJ5183; 2) l'imballaggio delle particelle adenovirali; 3) l'amplificazione dell'adenovirus nelle cellule AD293; 4) la purificazione delle particelle adenovirali dal liquido cellulare e dal mezzo di coltura; e (5) la titolazione virale e il test funzionale dell'adenovirus. I miglioramenti al metodo originale consistono (i) nella ricombinazione in pAdEasy-1 contenente BJ5183 mediante trasformazione chimica dei batteri; — la selezione dei cloni ricombinanti mediante PCR "negativo" e "positivo"; — la trasfezione delle celle AD293 mediante il sistema di trasfezione K2 per l'imballaggio adenovirale; — la precipitazione con solfato di ammonio delle particelle virali rilasciate dalle cellule AD293 nel mezzo di coltura cellulare; e v) la purificazione del virus mediante ultracentrifugazione del cloruro di cesio in una fase. Una forte espressione del gene di interesse (in questo caso, GFP) è stata ottenuta in diversi tipi di cellule trasdotte (come epatociti, cellule endoteliali) da varie fonti (umane, bovine, murine). Il trasferimento genico mediato da adenovirale rappresenta uno dei principali strumenti per lo sviluppo di moderne terapie geniche.
Introduzione
Gli adenovirus sono virus non ineveloped contenenti un nucleocapside e un genoma lineare del DNA a doppio filamento1,2,3. Gli adenovirus possono infettare un'ampia gamma di tipi di cellule e l'infezione non dipende dalla divisione attiva delle cellule ospiti. Dopo l'infezione, l'adenovirus introduce il suo DNA genomico nel nucleo della cellula ospite, dove rimane epicromosomiale e viene trascritto insieme ai geni dell'ospite. Pertanto, un rischio potenziale minimo di mutagenesi inserimento o regolazione degli oncogeni vieneraggiunto 4,5,6. Il genoma adenovirale non viene replicato insieme al genoma ospite e quindi i geni adenovirali vengono diluiti in una popolazione di cellule divisorie. Tra i vantaggi della trasduzione adenovirale, ci sono: (i) alti livelli di espressione transgena; ii riduzione dei rischi connessi all'integrazione del DNA virale nel genoma ospite, dovuta all'espressione episomale; — trasduzione di un'ampia varietà di tipi di cellule divisorie e non divisorie. La maggior parte degli adenovirus utilizzati nella ricerca biomedica non sono replicativi, mancando della regione E17,8,9. Per la loro produzione, è necessaria una linea cellulare che fornisca la sequenza E1 (come HEK293). Inoltre, una regione non essenziale per il ciclo di vita virale (E3) è stata eliminata per consentire l'inserimento di un transgene nel genoma virale; altre regioni (E2 ed E4) sono state ulteriormente eliminate in alcuni adenovirus, ma in questi casi è stata segnalata una diminuzione della resa della produzione adenoovirale e una bassa espressione del transgene7.
Qui presentiamo un protocollo migliorato per la costruzione, l'imballaggio e la purificazione degli adenovirus utilizzando il sistema AdEasy. Questi miglioramenti hanno permesso l'imballaggio dell'adenovirus in modo più veloce ed economico rispetto al metodo originale sviluppato da Bert Vogelstein2,10,a causa dei seguenti vantaggi: (i) la ricombinazione in pAdEasy-1 contenente BJ5183 mediante trasformazione chimica di batteri; — la selezione dei cloni ricombinanti mediante PCR; — la trasfezione delle celle AD293 mediante il sistema di trasfezione K2 per l'imballaggio adenovirale; — la precipitazione delle particelle adenovirali dal mezzo di coltura dopo l'imballaggio e l'amplificazione virali; — la purificazione adenovirale mediante ultracentrifugazione sfumato del cloruro di cesio in un solo passaggio (CsCl).
Il protocollo per la produzione di adenovirus mediante il sistema AdEasy (Figura 1) comprende i seguenti passaggi:
(1) Ricombinazione di pAdTrack-GFP con pAdEasy-1 nei batteri BJ5183
2) Imballaggio delle particelle adenovirali
(3) Amplificazione dell'adenovirus
(4) Purificazione delle particelle adenovirali dal litorato cellulare e dal mezzo di coltura
(5) Titolazione da adenovirus.
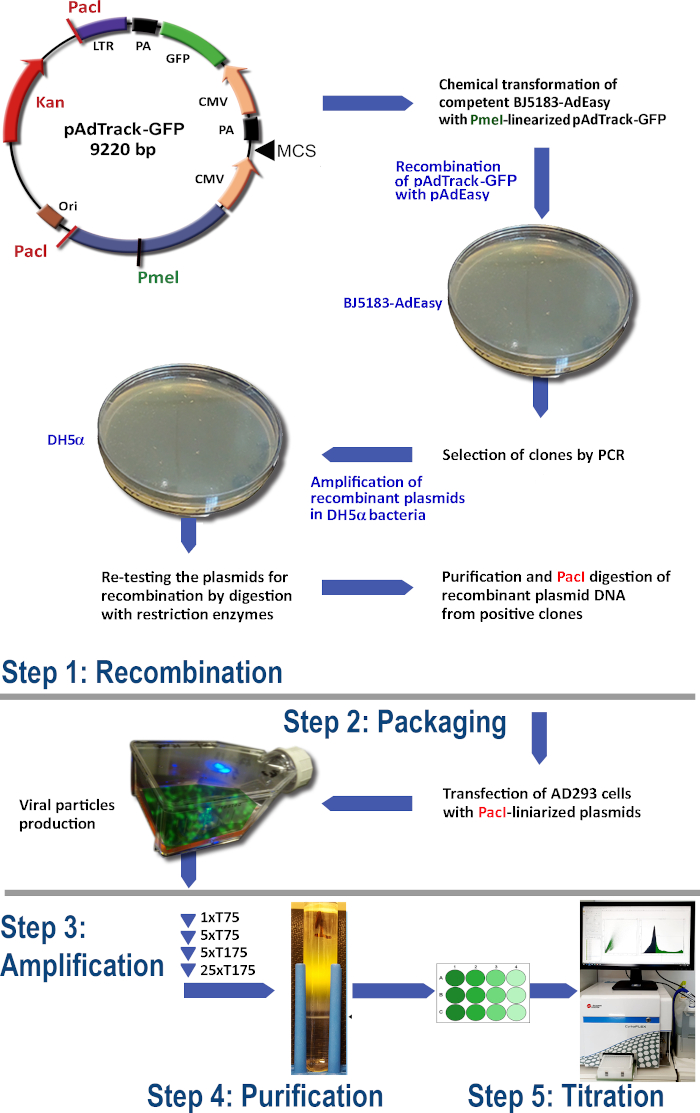
Figura 1: La tecnologia di produzione dell'adenovirus. I passaggi principali della tecnologia adenovirale sono: (1) La ricombinazione del pAdTrack-GFP con il plasmide pAdEasy-1 nei batteri BJ5183. I plasmidi ricombinati selezionati vengono amplificati nei batteri DH5α e quindi purificati; (2) l'imballaggio delle particelle adenovirali nelle cellule AD293 che producono proteine adeno-E1; (3) L'amplificazione dell'adenovirus nelle cellule AD293; (4) La purificazione delle particelle adenovirali dal litorato cellulare e dal mezzo di coltura mediante ultracentrifugazione su un gradiente di densità cscl; (5) La titolazione dell'adenovirus e la prova funzionale. Clicca qui per visualizzare una versione più grande di questa figura.
In questo protocollo, abbiamo esemplificato la tecnologia per la produzione dell'adenovirus, che può indurre l'espressione di GFP nelle cellule ospiti. GFP è già inserito nella spina dorsale del vettore shuttle pAdTrack-CMV (Addgene #16405), sotto un secondo promotore CMV ed è utilizzato come gene reporter (Figura 1). Per questo motivo, qui abbiamo designato il vettore pAdTrack-CMV come pAdTrack-GFP e abbiamo valutato l'espressione di GFP per scopi dimostrativi. Oltre all'espressione GFP, il sistema può essere utilizzato per sovraesprimere un gene di interesse, che può essere clonato nei siti di clonazione multipla del pAdTrack-CMV. Un gene o un minigene clonato nel pAdTrack-CMV è solitamente più efficiente per l'induzione dell'espressione rispetto al cDNA11. I dati hanno mostrato una forte espressione di FPG nelle cellule trasdotte (come epatociti, cellule endoteliali) da varie fonti (umane, bovine, murine). Il trasferimento genico mediato da adenovirale rappresenta uno dei principali strumenti per lo sviluppo di moderne terapie geniche.
Protocollo
Nota di sicurezza: In generale, gli adenovirus sono classificati come organismi di livello 2 di biosicurezza e quindi tutte le manipolazioni devono essere fatte in un armadietto di biosicurezza di classe II da una persona addestrata, indossando dispositivi di protezione a rischio biologico (compresi guanti, maschera facciale per aerosol biologici, camice da laboratorio, ecc.). Tutti i materiali solidi contaminati dall'adenovirus devono essere disinfettati con una soluzione di candeggina al 10% per 30 minuti e autoclavati per 30 minuti a 121 °C e 1 bar. A seconda del gene inserito, l'adenovirus creato può avere un potenziale pericoloso e può essere classificato in altri livelli di biosicurezza.
1. Preparazione sperimentale
- Utilizzare una cappa di coltura cellulare separata per le manipolazioni adenovirali e un incubatore separato per ogni tipo di adenovirus. Utilizzare flaconi a T con tappi filtranti per il confezionamento e l'amplificazione virali; evitare il più possibile esperimenti di trasduzione in piastre di Petri ventilate.
- Svuotare il cappuccio di coltura cellulare dopo ogni utilizzo ed esporlo ai raggi UV per 15 minuti.
- Autoclave periodicamente l'ausilio pipetta, pipette e altri utensili. Se possibile, coltura in un laboratorio di coltura cellulare separato/cappa le cellule per l'imballaggio adenovirale (cellule AD293) e le cellule da utilizzare negli esperimenti di trasduzione. Lotti di adenovirus diversi amplificati nello stesso periodo devono essere controllati per verificare la contaminazione incrociata da PCR.
- Preparare le soluzioni seguenti.
- Preparare sob (Super Optimal Broth) medio: 20 g di triptono, 5 g di estratto di lievito, 0,5 g di NaCl (concentrazione finale di 10 mM), 2,5 mL di 1 M KCl (concentrazione finale di 2,5 mM), amd H2O a 1 L. Dopo l'autoclave a 121 °C, aggiungere le seguenti soluzioni sterili: 5 mL di 1 M MgCl2 e 5 mL di 1 M MgSO4.
- Preparare SOC (Super Optimal broth with Catabolite repression) medium: in 1 L sob sterile aggiungere le seguenti soluzioni sterili: 20 mL di 1 M di glucosio, 5 mL di 1 M MgCl2 e 5 mL di 1 M MgSO4.
- Preparare lasoluzionedi precipitazione: sciogliere 29,5 g di acetato di potassio in 60 mL di H 2 O, aggiungere 11,5 mL di acido acetico e Hda 2O a 100 mL.
- Preparare tampone di resuspensione: 95 mL di glucosio al 20%, 5 mL di 1 M Tris-Cl pH 8, 4 mL di 0,5 M EDTA pH 8 e aggiungere H2O a 200 mL.
- Preparare la soluzione di lysis: 4,8 mL di 8,3 M NaOH, 10 mL di 20%SDS e Hda 2O a 200 mL.
2. Ricombinazione del vettore virale pAdTrack-GFP con pAdEasy-1 plasmide nei batteri BJ5183
- Linearizzazione di pAdTrack-GFP e purificazione del plasmide linearizzato.
- Preparare il seguente mix di digestione sul ghiaccio:
10 μg di pAdTrack-GFP
5 μL di tampone incolore 10x
2 μL di Pme I
H2O di ad un volume finale di 50 μL. - Incubare a 37 °C per 3 ore in un bagno d'acqua.
- Inattivare a 65 °C per 20 min.
- Controllare l'efficienza della digestione di pAdTrack-GFP con Pme I: eseguire 1 μg del plasmide digerito in parallelo con 1 μg di plasmide non digerito su un gel di agarosio dello 0,8%.
- Preparare il seguente mix di digestione sul ghiaccio:
- Isolamento e purificazione del DNA
NOTA: I passaggi da 1 a 6 devono essere eseguiti in una cappa aspirante.- Aggiungere un volume uguale di fenolo/cloroformio/alcol isoamil (25:24:1) sulla miscela di digestione e invertire il tubo fino a quando la miscela non è omogenea.
- Centrifugare per 3 minuti a 16.200 x g,quindi trasferire la fase acquosa superiore in un tubo di raccolta.
- Aggiungere un volume uguale di fenolo/cloroformio/alcol isoamile (25:24:1) sulla fase organica inferiore e sul vortice.
- Centrifugare per 3 min a 16.200 x g,quindi trasferire la fase superiore allo stesso tubo di raccolta.
- Aggiungere un volume uguale di cloroformio sulla fase acquosa raccolta nel tubo di raccolta e nel vortice.
- Centrifugare per 3 min a 16.200 x g,quindi trasferire la fase acquosa superiore in un nuovo tubo di raccolta.
- Aggiungere un volume 1/10 di acetato di sodio 3 M e 2 volumi di etanolo e vortice freddi al 100%.
- Incubare per 1 ora a -70 °C o durante la notte a -20 °C.
- Scongelare il campione sul ghiaccio e centrifugarlo per 10 minuti a 16.200 x g e 4 °C.
- Rimuovere il supernatante e aggiungere 750 μL di 75% di etanolo.
- Centrifugare per 3 min a 16.200 x g e 4 °C e rimuovere il supernatante.
- Ruotare brevemente il tubo per rimuovere tutto il supernatante e asciugare il pellet nel cappuccio. Non asciugare il pellet di DNA per molto tempo perché è difficile da sciogliere.
- Sciogliere il pellet in 15 μL di H2O.
- Misurare la concentrazione di DNA usando uno spettrofotometro (ad esempio, Nanodrop).
- Trasformazione dei batteri AdEasier-1 con pAdTrack-GFP
NOTA: In questo passaggio, avviene la ricombinazione di pAdTrack-GFP con pAdEasy-1 plasmide.- Preparare batteri chimici competenti AdEasier-1 (bj5183-containing pAdEasy-1, Addgene #16399), utilizzando un kit di trasformazione commerciale, seguendo le istruzioni dei produttori. Mantenere aliquote di batteri competenti a -80 °C.
- Scongelare un'aliquota di batteri AdEasier-1 competenti sul ghiaccio e aggiungere 1 μg di pAdTrack-GFP purificato di pme I. Mescolare delicatamente sfarfallando il tubo (non pipettare la miscela). Incubare per 10 minuti sul ghiaccio.
- Aggiungere 900 μL di soc medio e incubare per 1 ora a 37 °C con scuotimento.
- Microfugo per 5 min a 600 x g.
- Rimuovere 900 μL del supernatante, mescolare il pellet e il supernatante e seminare i batteri trasformati su piastre LB-agar con kanamicina.
- Incubare per ~ 16 ore a 37 °C (non superare le 18 ore).
- Selezione dei possibili cloni positivi da parte della PCR
- Dividere gli stuzzicadenti in due metà e sterilizzare gli stuzzicadenti con l'autoclave.
- Raccogli colonie piccole e traslucide usando sgombro sterile.
- In breve, ruotare lo stuzzicadenti con batteri in acqua da 10 μL (in un tubo PCR) e quindi mettere lo stuzzicadenti in un tubo Eppendorf da 1,5 ml contenente mezzo SOC da 100 μL con kanamicina. Incubare per 4-6 ore a 37 °C, mentre si testano i cloni con PCR "negativo" e "positivo".
- Incubare i tubi PCR contenenti acqua da 10 μL con batteri per 5 min a 95 °C per ottenere il campione batterico ed eseguire in parallelo la PCR "negativa" e "positiva".
- PCR "negativo" - per testare l'integrità pAdTrack-GFP: preparare il seguente mix PCR per la PCR negativa sul ghiaccio.
5 μL del campione batterico
0,1 μL di primer Forward (4631 F: 5'-CAGTAGTCGGTGCTCGTCCAG)
0,1 μL di primer Reverse (5616 R: 5'-TATGGGGGCTGTAATGTTGTCTC)
0,1 μL di dNTP 10 mM
3 μL di 5x Tampone
1,5 μL di MgCl2 25mM
0,1 μL di GoTaq Polimerasi
H2O ad un volume finale di 15 μL
NOTA: Il controllo positivo in cui il modello di DNA è il vettore pAdTrack-GFP deve essere incluso. - PCR "positiva" - per testare la presenza del gene di interesse. Utilizzare primer specifici per il gene inserito e preparare il mix come nella fase precedente. I primer utilizzati per la GFP erano i seguenti:
F: 5'-CAAGGACGACGGCAACTACA
R: 5'-ATGGGGGTGTTCTGCTGGTA - Eseguire in parallelo il PCR "negativo" e "positivo". Il programma PCR è: 5 min, 95 °C; 40 cicli dei seguenti passaggi: 30 sec, 95 °C; 30 sec, 68 °C; 1 min, 72 °C; allungamento finale: 10 min, 72 °C.
NOTA: Adattare la temperatura di ricottura per amplificare il gene di interesse. - Valuta i prodotti PCR su un gel di agarosio all'1% ed effettuare la selezione dei cloni.
- Considerare per l'ulteriore elaborazione i cloni che non danno prodotti PCR per la "PCR negativa" e lo specifico prodotto PCR dopo la "PCR positiva".
- Far crescere le colture batteriche di cloni ricombinanti selezionati
- Diluire le colture dei presunti cloni positivi (ha portato al passo 2.4.3.) in 4 mL di mezzo SOC con kanamicina, e incubarli durante la notte a 37 °C con scuotimento.
- Isolamento del DNA plasmide dai batteri AdEasier-1 (Miniprep con lisi alcalina)
- Trasferire 1,5 ml di coltura batterica in tubi di microcentrifugo, centrifugare per 1 min a 16.200 x ge rimuovere il supernatante.
- Trasferire un'altra coltura batterica da 1,5 ml nello stesso tubo, ripetere la centrifugazione e rimuovere il supernatante.
- Aggiungere 200 μL di tampone di resopensione (50 mM di glucosio, 10 mM EDTA, 25 mM Tris-HCl pH 8).
- Aggiungere 200 μL di soluzione dilisi (0,2 N NaOH, 1% SDS), mescolare delicatamente invertendo il tubo.
- Aggiungere 200 μL di soluzione di precipitazione (60 ml di acetato di potassio da 5 M, 11,5 ml di acido acetico glaciale, aggiungere H2O fino a 100 mL) e mescolare delicatamente invertendo il tubo.
- Centrifuga per 3 min a 16.200 x g.
- Trasferire il supernatante in un nuovo tubo di microcentrifugo, aggiungere isopropanolo da 500 μL, mescolare e incubare per 20 minuti sul ghiaccio.
- Centrifugare per 15 min a 16.200 x g e aggiungere 500 μL di 75% di etanolo.
- Centrifugare per 10 min a 16.200 x g e rimuovere il supernatante.
- Centrifugare per 3 min a 16.200 x g, rimuovere il supernatante e aggiungere 15 μL di H2O.
- Amplificazione, isolamento e ri-test dei plasmidi ricombinati
- Trasformazione dei batteri DH5α con il DNA isolato dalle cellule di AdEasier-1.
- Preparare i batteri competenti DH5α utilizzando il kit di trasformazione commerciale, seguendo le istruzioni dei produttori.
- Scongelare 100 μL di batteri competenti DH5α sul ghiaccio, aggiungere il DNA ricombinante e incubare 10 minuti sul ghiaccio. Quindi seminare i batteri su piastre LB-agar con kanamicina.
- Incubare a 37 °C durante la notte.
- Raccogli diverse colonie e cresci ognuna in 2 mL di mezzo LB con kanamicina, a 37 °C, durante la notte, con tremori.
- Isolare il DNA (Miniprep usando lisi alcalina) e rimescolare il DNA ottenuto in 25 μL H2O.
- Confermare i cloni positivi mediante digestione enzimatica.
- Preparare il seguente mix sul ghiaccio:
5 μL di DNA ricombinante
1,5 μL di tampone incolore 10x
0,5 μL di Hind III o Pst I
H2O ad un volume finale di 15 μL - Incubare a 37 °C per 30 min.
NOTA: Come controllo, digerire anche i plasmidi pAdTrack-GFP e pAdEasy-1. - In ogni campione aggiungere 3 μL di tampone di carico Sx6 con RNasi A (se la RNasi A non è presente nei buffer miniprep).
- Eseguire i frammenti di DNA digeriti sull'elettroforesi del gel di agarosio all'1%.
NOTA: Il modello di digestione di un clone positivo include la maggior parte dei frammenti del plasmide pAdEasy digerito, rivelando la ricombinazione pAdEasy con il vettore pAdTrack. Il gene di interesse dovrebbe essere evidenziato dalla digestione con gli enzimi di restrizione utilizzati per la clonazione.
- Preparare il seguente mix sul ghiaccio:
- Preparazione del DNA plasmide (grado di trasfezione) per l'imballaggio dell'adenovirus.
- Far crescere una coltura di batteri da 200 mL da un clone positivo per isolare il DNA plasmide.
- Isolare il DNA plasmide utilizzando un kit commerciale per il DNA plasmide Midiprep (ad esempio, Qiagen Plasmid Midi Kit) seguendo le istruzioni del produttore.
- Trasformazione dei batteri DH5α con il DNA isolato dalle cellule di AdEasier-1.
3. Confezionare le particelle adenovirali
- Giorno 1. Seminare le cellule AD293
- Lavare le cellule AD293 con PBS e incubarle con 0,125% di tripside per 2-5 min a 37 °C.
- Raccogliere le cellule in mezzo freddo con siero.
- Centrifuga per 5 min a 400 x g a 4 °C.
- Rimorsiva le cellule in mezzo con siero e semina le cellule ad una densità di ~2 x 106/T25 pallone. Preferibilmente, utilizzare un pallone con un filtro.
- Giorno 1. Digerire il DNA ricombinante con Pac I
- Preparare la combinazione seguente:
6 μL di DNA ricombinante (1 μg/μL)
2 μL di Pac I
2,5 μL di tampone incolore 10x
H2O ad un volume finale di 25 μL - Incubare per 3 ore (o durante la notte) a 37 °C, quindi inattivare l'enzima a 65 °C per 20 minuti.
- Precipitazione del DNA con etanolo: aggiungere 2,5 μL di (1/10 v/v) 3 M acetato di sodio e 2-3 volumi di etanolo al 100%. Incubazione per 30 min a -70 °C o durante la notte a -20 °C.
- Centrifugare a 16.200 x g per 30 min a 4 °C e rimosogliere il pellet in acqua sterile.
- Preparare la combinazione seguente:
- Giorno 2: Trasfezione di cellule AD293 con reagente K2
- Aggiungere 40 μL di moltiplicatore K2 sulle cellule, due ore prima della trasfezione.
- Preparare soluzioni A e B:
Soluzione A: Aggiungere 6 μg di DNA pacificamente linearizzato in 260 μL di Opti-MEM.
Soluzione B: Aggiungere 21,6 μL di Reagente K2 in 248,4 μL di Opti-MEM. - Aggiungere la soluzione A sulla soluzione B e mescolare delicatamente con pipettazione.
- Incubare la miscela per 20 minuti a temperatura ambiente. Aggiungere la combinazione A e B dropwise alle celle.
- Giorno 3-11: Monitorare l'espressione della GFP mediante microscopia a fluorescenza
NOTA: Le cellule devono apparire verdi nella microscopia a fluorescenza e dovrebbero gradualmente staccarsi. - Giorno 11: Raccogliere le particelle adenovirali di F1
- Raccogliere le celle staccate e il mezzo in un tubo da 50 ml, raschiare le cellule aderenti e aggiungerle nello stesso tubo.
- Centrifugare per 5 minuti a 400 x g, raccogliere il supernatante in un nuovo tubo e rimescolare il pellet di cella in 0,5 ml di PBS.
- Interruzione cellulare
- Trasferire la sospensione cellulare in un tubo di microcentrifugo.
- Eseguire tre cicli di congelamento/disgelo (congelare in azoto liquido o a -80 °C /scongelamento a 37 °C per un massimo di 7 min).
- Passare le cellule rotte attraverso un ago da siringa da 23 G tre volte.
- Rimuovere i detriti cellulari mediante centrifugazione a 9.600 x g per 12 min.
- Trasferire il supernatante sul tubo da 50 ml con il mezzo raccolto.
4. Amplificazione dell'adenovirus
NOTA: Se le cellule AD293 non hanno raggiunto la confluenza necessaria, le aliquote delle scorte adenovirali (lisato ottenuto dalle cellule produttrici di virus) da utilizzare per l'infezione possono essere immagazzinate a -80 °C.
- Preparare le particelle adenovirali F2.
- Seminare le cellule AD293 in un pallone T75 (5 x 106 cellule/pallone).
- Infettare ~90% di cellule AD293 confluenti usando le particelle adenovirali F1: aggiungere l'omogeneato cellulare e il mezzo dal pallone T25 sopra le cellule coltivate nel pallone T75.
- Monitorare l'espressione della GFP mediante microscopia a fluorescenza.
- Raccogliere le cellule produttrici di virus quando ~90% dell'AD293 trasdotto viene staccato (~ il 5 °giorno dopo la trasduzione). Mantenere il mezzo di coltura cellulare a 4 °C.
- Interrompere le cellule (in modo simile a quelle per F1) in 1 mL PBS.
- Preparare le particelle adenovirali F3.
- Infettare ~90% di cellule AD293 confluenti sementi in mascella T175 con particelle adenovirali F2 e il mezzo di coltura cellulare dalle particelle adenovirali F2.
- Raccogliere le cellule (~ 5 giorni dopo la trasduzione).
- Interrompere le cellule (in modo simile a quelle per F1) in 2 mL di PBS.
- Preparare le particelle adenovirali F4.
- Infettare 5 mastri T175 contenenti circa il 90% di cellule AD293 confluenti con particelle adenovirali F3 e mezzo di coltura cellulare.
- Raccogliere le cellule (~ 5 giorni dopo la trasduzione).
- Interrompere le cellule (in modo simile a quelle per F1) in 3 mL di PBS.
- Preparare le particelle adenovirali F5.
- Infettare 25 mastri T175 contenenti circa il 90% di cellule AD293 confluenti con stock adenovirale F4 e mezzo di coltura cellulare.
5. Purificazione dell'adenovirus dal llysato cellulare e dal mezzo di coltura
- Raccolta delle cellule produttrici di virus e del mezzo di coltura.
- Raccogliere le cellule AD293 di F5 dopo 5 giorni dalla trasduzione.
- Salvare il mezzo in una bottiglia sterile per la precipitazione delle particelle adenovirali.
NOTA: Conservare il mezzo in frigorifero fino alla purificazione dell'adenovirus. - Centrifugare le cellule a 400 x g, per 5 min, a 4 oC.
- Resuspend il pellet finale in 5 mL di 10 mM Tris HCl, pH 8 con 2 mM MgCl2.
- Aliquota la sospensione in tubi da 1,5 ml.
- Interrompere le cellule (in modo simile a quelle per F1): tre cicli di congelamento/scongelamento.
NOTA: Se l'ultracentrifugazione non può essere eseguita immediatamente, mantenere i campioni a -80 °C. - Passare la sospensione cellulare attraverso un ago per siringhe 23G per tre volte.
- Centrifugare l'omogeneato a 9 600 x g, per 12 min.
- Salvare il supernatante per la purificazione dell'adenovirus mediante ultracentrifugazione gradiente CsCl.
- Precipitazione dell'adenovirus rilasciato nel mezzo di coltura.
- Portare la bottiglia con un mezzo di coltura cellulare salvato a temperatura ambiente.
- Aggiungere 121 g di solfato di ammonio a ogni 500 mL di mezzo di coltura cellulare (la saturazione della soluzione deve essere compresa tra il 40 e il 42%).
- Mescolare con cura fino a quando il solfato di ammonio non è completamente sciolto.
- Incubare per un minimo di 2,5 ore a temperatura ambiente.
- Centrifuga a 1600 x g, per 15 min, a 22 oC e salva il pellet.
- Resuspend il pellet in 4 mL di 10mM Tris HCl pH 8 con 2mM MgCl2; questa sospensione deve essere immediatamente purificata dall'ultracentrifugazione gradiente CsCl.
NOTA: Se la fase di purificazione non può essere successivamente eseguita, dializzare durante la notte il pellet rimescolato contro 10mM Tris HCl, pH 8 con 2mM MgCl2.
- Purificazione dell'adenovirus mediante ultracentrifugazione.
- Preparare un gradiente CsCl discontinuo in tubi in polipropilene per il rotore SW41Ti. Aggiungere 3 ml di 765 mg/mL CsCl (alta densità: 1,4 g/L) nella parte inferiore del tubo. Aggiungere lentamente 3 mL di 288,5 mg/mL CsCl (bassa densità: 1,2 g/L) sopra il primo strato cscl.
- Sovrapporre delicatamente 3 - 4 mL della sospensione di particelle adenovirale rilasciate dalle cellule o precipitate dal mezzo di coltura cellulare (come descritto in precedenza) sopra il gradiente.
- Riempire i tubi con olio minerale e mettere i tubi nei secchi FREDDI SW41Ti.
- Equilibrare i tubi. Assicurarsi che i tubi riempiti in polipropilene siano caricati simmetricamente nel rotore. Metti il rotore nell'ultracentrifugo.
- Centrifuga a 210.000 x g e 4 °C, per 18 ore, nessun freno.
- Posizionare i tubi ultracentrifugo su un supporto con una carta nera dietro per ottenere le bande.
- Scartare la fase superiore chiara, i detriti cellulari e la banda superiore in un contenitore di rifiuti con la soluzione di sbiancamento.
- Raccogliere la banda più bassa che contiene l'adenovirus completo (~700 μL - 1 ml) in un tubo sterile da 1,5 ml e tenerlo sul ghiaccio.
- Pre-bagnare una cassetta di dialisi nel tampone di dialisi (tampone Tris-Cl da 10 mM pH 8, 2 mM MgCl2).
- Iniettare l'adenovirus purificato nella cassetta di dialisi utilizzando una siringa da 2 ml.
- Dializzare durante la notte contro 10 mM Tris-Cl tampone pH 8, 2 mM MgCl2 (cambiare il buffer di dialisi 3 - 4 volte).
- Raccogliere il materiale adenovirale dalla cassetta di dialisi in aliquote da 10 a 100 μL.
- Aggiungere saccarosio al 4% di concentrazione finale alle aliquote virali (per la crioprotezione).
- Conservare le aliquote a -80 °C.
6. Titolazione adenovirus
- Giorno 1: Placcare le cellule
- Sementire le cellule AD293 ad una densità di 2,5 × 105 cellule per pozzo (in una piastra di coltura di 12 pozzi) in un mezzo di crescita completo di 1 mL, come mostrato nella figura 2. Assicurarsi che le cellule siano distribuite uniformemente in ogni pozzo per una determinazione accurata del formicolio.
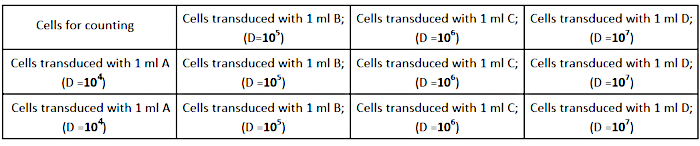
Figura 2: Design della piastra di titolazione. Clicca qui per visualizzare una versione più grande di questa figura.
- Giorno 2: Trasduzione delle cellule
- Staccare le cellule da un pozzo con tripina e contarle. Nota questo numero perché verrà utilizzato per calcolare il titer virale.
- Eseguire diluizioni seriali (1/104; 1/105; 1/106; 1/107) dello stock virale in 1 mL di mezzo di crescita completo come segue:
- 1/103: diluizione dello stock virale - aggiungere 2 μL di stock virale al 1998 μL di mezzo completo.
- 1/104: Effettuare una diluizione di 1:10 di 1/103 diluindo da 120 μL a 1080 μL di mezzo completo (A).
- 1/105: Effettuare la diluizione 1:10 di B diluindo da 120 μL di A a 1080 μL di mezzo completo (B).
- 1/106: Effettuare la diluizione 1:10 di C diluire da 120 μL di B a 1080 μL di mezzo completo (C).
- 1/107: Effettuare la diluizione 1:10 di D diluire da 120 μL di C a 1080 μL di mezzo completo (D).
NOTA: Preparare 3 tubi di ogni diluizione (A, B, C, D) per eseguire l'esperimento in triplicati. - Rimuovere il supporto di coltura cellulare dai pozzi e aggiungere le diluizioni preparate del virus, come illustrato nella figura 2.
- Giorno 3: Monitoraggio dell'espressione GFP
- Controllare i pozzi per la presenza di cellule verdi utilizzando un microscopio a fluorescenza.
- Giorno 4: Analisi della citometria del flusso delle cellule positive alla GFP
- Preparare ed etichettare dodici tubi da 1,5 ml.
- Raccogliere il mezzo di coltura cellulare (insieme alle celle staccate) in tubi da 1,5 ml e tenerli sul ghiaccio.
- Aggiungere 200 μL di triptina in ogni pozzo.
- Incubare la piastra per 2 - 3 minuti a 37 °C nell'incubatore di CO2.
- Raccogliere le cellule negli stessi tubi Eppendorf con il mezzo di coltura cellulare. Tieni i tubi sul ghiaccio.
- Pellettizzare le cellule a 400 x g, per 5 minuti, a 4 °C.
- Rimuovere il supernatante; tenere i tubi sul ghiaccio.
- Resuspend il pellet in 250 μL di PBS + 2% FBS; tenere i tubi sul ghiaccio.
- Trasferire la sospensione cellulare in tubi o piastre di citometria a flusso.
- Eseguire i campioni su un citometro a flusso che registra la fluorescenza delle cellule di esprimezione GFP.
Calcolo del titer: I campioni con cellule positive 5 - 20% GFP della popolazione madre dovrebbero essere presi in considerazione per il calcolo del titer virale utilizzando la seguente formula:
Titer (TU/mL) = D x F/100 x C/V
D = fattore di diluizione
F = percentuale di cellule positive / 100
C = numero di celle / pozzo
V = volume di inoculo virale
7. Trasduzione adenovirale delle cellule bersaglio e test dell'espressione proteica indotta
- Giorno 1: Seminare le cellule
- Seminare le cellule bersaglio assicurando che siano distribuite uniformemente nei pozzi.
- Giorno 2: Trasduzione delle cellule
- Staccare le celle di destinazione da un pozzo e contarle.
- Calcolare il volume appropriato di sospensione adenovirale necessario per trasdurre le cellule con il numero desiderato di particelle infettive per cellula.
- Aggiungere la quantità corrispondente di sospensione virale alle cellule bersaglio.
- Giorno 3: Rimozione della sospensione virale e controllo dell'espressione GFP
- Sostituire il mezzo di coltura cellulare contenente particelle adenovirali con mezzo fresco.
- Controllare l'espressione GFP al microscopio a fluorescenza.
Risultati
Abbiamo modificato e migliorato il protocollo originale di Vogelstein al fine di ottenere una produzione di adenovirus più veloce ed efficiente. In primo luogo, abbiamo rivisto la metodologia per ottenere una selezione più semplice dei ricombinanti. Dopo la ricombinazione, i cloni batterici BJ5183 sono stati testati da "PCR negativo" per valutare l'integrità di pAdTrack-GFP come indicatore della mancanza di ricombinazione (Figura 3A), o da "PCR positivo" per identificare il gene di interesse, assimilato nel nostro caso alla GFP (Figura 3B). Sia nei PCR "negativi" che in "positivi", abbiamo usato pAdTrack-GFP come modello di controllo, che ha dato una banda di 986 bp per l'integrità di pAdTrack(Figura 3A, corsia 1) e una banda di 264 bp per GFP(Figura 3B,corsia 3). I primer utilizzati per la "PCR negativa" sono stati progettati per amplificare un frammento di 986 bp contenente il sito PmeI in pAdTrack-GFP. Questo frammento di DNA viene drasticamente ingrandito dopo la ricombinazione e non viene amplificato nei cloni ricombinanti positivi. I cloni negativi per la ricombinazione, in cui pAdTrack-GFP è rimasto intatto, sono rappresentati nella figura 3A, corsie 3, 4 e 6. I primer ricottura sulle sequenze di DNA adiacenti al sito di ricombinazione. I potenziali cloni ricombinanti positivi(figura 3A, corsie 2 e 5) hanno espresso GFP come mostrato nella figura 3B, corsia 1 e 2. Il DNA plasmide di questi cloni è stato isolato e utilizzato per la trasformazione di DH5α per ottenere una maggiore quantità di DNA. Questi plasmidi ricombinanti preselezionati amplificati in DH5α sono stati quindi testati mediante digestione enzimatica. Nella figura 3C-E sono illustrati i risultati della digestione enzimatica di un clone recombinant-positivo digerito con enzimi di restrizione Hind III, PstI, BamHI(Figura 3C, D, E corsia 2). I modelli di digestione HindIII e PstI del clone ricombinante erano simili a quelli ottenuti per pAdEasy-1 poiché HindIII e PstI tagliavano il plasmide pAdEasy-1 rispettivamente 24 e 25 volte(figura 3C e D,corsia 3); HindIII tagliato una volta e PstI tagliato quattro volte il vettore pAdTrack-GFP (Figura 3C e D, corsia 1). BamHI ha tagliato due volte il vettore pAdEasy-1(Figura 3C, corsia 3) e una volta pAdTrack-GFP (Figura 3C, corsia 1).
PacI ritagliare un frammento di 4,5 kb dal plasmide ricombinante (Figura 3F, corsia 2), un frammento di 2863 bp da pAdTrack-GFP(Figura 3F, corsia 1), e linearizzato il vettore pAdEasy-1(Figura 3F, corsia 3). La scala del DNA è rappresentata nella figura 3C-F, nelle corsie 4. Il plasmide ricombinante è stato digerito con Pac I per un ulteriore utilizzo per la trasfezione AD293.
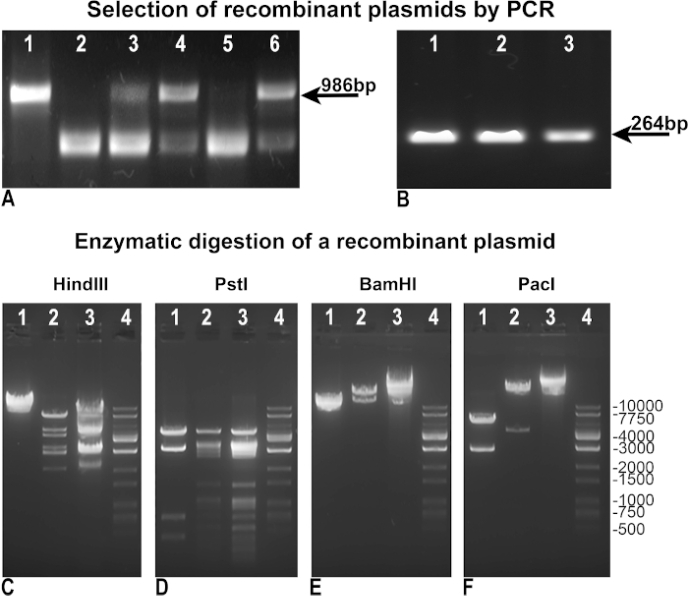
Figura 3: Ricombinazione di pAdTrack-GFP con il plasmide pAdEasy-1. I plasmidi ottenuti dopo la ricombinazione di pAdTrack-GFP e pAdEasy-1 sono stati testati da PCR "negativo" per l'integrità pAdTrack-GFP (A). I cloni non ricombinanti sono stati evidenziati dalla presenza di una banda di 986 bp corrispondente alla sequenza amplificata dal plasmide pAdTrack-GFP (A, corsie 3, 4 e 6). Sono stati ottenuti anche i cloni potenzialmente positivi per la ricombinazione (A, corsie 2 e 5). Quando il vettore pAdTrack-GFP è stato utilizzato come modello, è stata ottenuta una banda di 986 bp per pAdTrack-GFP (A, corsia 1). I cloni ricombinanti potenzialmente positivi sono stati testati per l'espressione GFP da PCR (B" "positivo"); una banda di 264 bp appare sia per i cloni potenzialmente ricombinati (B, corsia 1 e 2), sia per il plasmide pAdTrack-GFP. Il DNA di un potenziale clone ricombinante è stato testato con l'enzima di restrizione HindIII, PstI, BamHI e PacI (C-F, corsie 2). Nei controlli, il vettore pAdEasy-1 (C-F, corsie 3) e il plasmide pAdTrack-GFP (C-F, corsie 1) sono stati digeriti con gli stessi enzimi. La scala del DNA è rappresentata nella corsia C-F 4. Clicca qui per visualizzare una versione più grande di questa figura.
L'imballaggio e l'amplificazione addenovirale sono stati eseguiti in celle AD293. Le particelle addenovirali (AdV-GFP) sono state purificate dal lisato cellulare AD293 e dal mezzo di coltura cellulare, dove erano state rilasciate dalle cellule infette. Per concentrare l'adenovirus presente nel mezzo di coltura cellulare, le particelle sono state precipitate con solfato di ammonio e poi rimescolate in 10 mM Tris HCl pH 8 con 2 mM MgCl2, lo stesso tampone utilizzato per la lisi cellulare. Successivamente, le particelle adenovirali del lisato cellulare e del mezzo di coltura sono state purificate dall'ultracentrifugazione gradiente discontinuo cscl. Dopo l'ultracentrifugazione, è stata ottenuta una forte banda di AdV-GFP purificata, come mostrato nella figura 4.

Figura 4: La purificazione adenovirale mediante ultracentrifugazione su un gradiente CsCl discontinuo. L'omogeneato cellulare e l'adenovirus precipitato dal mezzo sono stati sottoposti ad ultracentrifugazione su un gradiente discontinuo formato da soluzioni CsCl a bassa e alta densità. Forti bande di GFP- adenovirus sono state evidenziate in entrambi i casi. Clicca qui per visualizzare una versione più grande di questa figura.
Per determinare il formicolio virale espresso in unità trasduttivi per un mL (TU/mL), le cellule AD293 sono state infettate da diluizioni seriali dell'AdV-GFP. Dopo 48 ore, le cellule infette hanno espresso GFP, in una correlazione inversa con il fattore di diluizione della sospensione virale. Ciò è stato osservato dalla microscopia a fluorescenza e la percentuale di cellule positive alla GFP è stata determinata dalla citometria del flusso (Figura 5). Per calcolare il timone, è stata presa in considerazione la diluizione virale che ha indotto il 5-20% delle cellule positive alla GFP (Figura 5C). Di solito, otteniamo un formicolio virale di ~10 10 (TU / mL) per GFP-adenovirus.
Di seguito, forniamo un esempio di calcolo del titer adenovirale per uno specifico lotto adenovirale in cui 300000 cellule (C) sono state trasdotta con soluzione adenovirale da 1 mL (V), con un fattore di diluizione di 106 (D), per il quale sono state ottenute cellule positive alla GFP (F):
Titer (TU/mL) = D x F/100 x C/V = 106 x 6/100 x 300000/1 = 1,8 x 1010 TU/mL
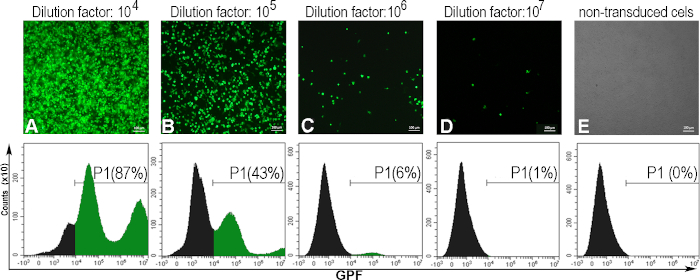
Figura 5: La valutazione del titer adenovirale. Le cellule AD293 sono state infettate da varie diluizioni adenovirali. Quarantotto ore dopo, le cellule sono state osservate mediante microscopia a fluorescenza e analizzate dalla citometria del flusso per determinare la percentuale di cellule positive GFP indotte da diverse diluizioni adenovirali (A-D). Per stabilire il gate per la citometria del flusso, sono state analizzate anche cellule non trasdotto (E). Il timone calcolato per il fattore di diluizione 106, quando il 6% delle cellule erano positive alla GFP era 1,8 x 1010 TU/mL. Per i pannelli A-E, barre: 100μm. Fare clic qui per visualizzare una versione più grande di questa figura.
Per testare il potenziale di trasduzione dell'adenovirus preparato, sono state utilizzate quattro linee cellulari: cellule endoteliali umane (EA.hy926), cellule endoteliali aortiche bovine (BAEC), epatociti murini (Hepa 1-6) e cellule stromali mesenchimali murine (MSC). Le cellule endoteliali (EA.hy926 e BAEC) sono state trasdotte con 25 TU/cellula, gli epatociti sono stati trasdotti con 5 TU/cellula e MSC sono stati trasdotti con 250 TU/cellula.
Ecco un esempio di come è stato calcolato il volume della sospensione adenovirale necessaria per infettare 3 x10 6 celle con 25 TU/cella, utilizzando la sospensione adenovirale con 1,8 x10 10 TU/mL.
Per 1 cella .............. 25 TU
3 x 106 celle .............. x TU  x=75 x 106 TU
x=75 x 106 TU
Se il patrimonio virale contiene
1,8 x 1010 TU .............. 1 mL
75 x 106 TU .............. y mL  y= 4,2 x 10-3 mL = 4,2μL di stock virale
y= 4,2 x 10-3 mL = 4,2μL di stock virale
Quarantotto ore dopo la trasduzione, le cellule sono state analizzate mediante microscopia a fluorescenza. Come mostrato nella figura 6, le cellule endoteliali umane o bovine sono state trasdotto con una buona efficienza (~50%) per 25 TU/cella(figura 6 EA.hy926 e BAEC). Gli epatociti murini (Hepa 1-6) sono stati trasdutti in modo efficiente dall'adenovirus a una bassa quantità di particelle di adenovirus (5 TU/cellula), ma sono anche sensibili all'adenovirus poiché è stata registrata una percentuale più alta di cellule morte (cellule PI-positive) (~16%) rispetto agli altri tipi di cellule. Le cellule stromali mesenchimali erano le più difficili da trasdurre (Figura 6), a causa della mancanza di specifici recettori adenovirali (dati inediti).
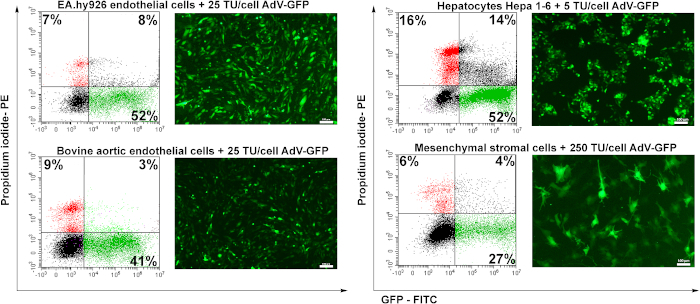
Figura 6: L'infettività dell'adenovirus e l'induzione dell'espressione della FPG nelle cellule trasdutte. Le cellule endoteliali umane (EA.hy926), le cellule endoteliali aortiche bovine (BAEC), gli epatociti murini (Hepa 1-6) e le cellule stromali mesenchimali murine (MSC) sono state trasdotte con la quantità indicata di adenovirus. La GFP è stata rilevata dalla microscopia a fluorescenza e la percentuale delle cellule positive della GFP è stata analizzata dalla citometria del flusso. Le cellule pi-positive determinate dalla citometria del flusso mostrano la mortalità cellulare determinata dalla trasduzione virale. Le cellule EA.hy926, le cellule endoteliali aortiche bovine e le cellule Hepa 1-6 sono state altamente trasdutite dall'adenovirus, con una resa di trasduzione compresa tra il 41 e il 52%. Per MSC, una maggiore quantità di virus (250 TU/cellule) ha indotto solo il 27% di GFP positivo delle cellule trasdutte. Barre: 100μm. Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
Gli adenovirus ricombinanti sono uno strumento versatile per l'erogazione el'espressione genica 12,13,14. Per indurre una forte espressione proteica mediante trasduzione adenovirale, la sequenza di codifica del gene di interesse è inserita nel genoma dell'adenovirus. Il sistema adenovirale AdEasy, sviluppato nel laboratorio di Bert Vogelstein, comprende un plasmide dorsale (pAdEasy-1) contenente la maggior parte del genoma del sierotipo 5 adenovirus di tipo selvatico e un vettore navetta (pAdTrack), progettato per la clonazione genica2,10. La cancellazione dei geni adenovirali E1 (responsabile dell'assemblaggio di particelle di virus infettivi) ed E3 (codificare proteine coinvolte nell'evadere l'immunità ospite) ha creato uno spazio nel genoma adenovirale, in cui può essere inserito un gene di interesse di 6,5-7,5 kb2,3. Questa dimensione è sufficiente per molti geni, specialmente per quelli con introni piùcorti 15,16,17. Ci sono anche ricercatori che riportano la produzione di adenovirus che trasportano il cDNA di un transgene18,19,20. Tuttavia, abbiamo ottenuto una resa inferiore di espressione transgena per gli adenovirus portatori di cDNA rispetto alle loro controparti che trasportano un gene o un mini-gene (dati non mostrati).
Migliorando e adattando i metodiprecedenti 2,10,14,18,21, la tecnologia per la produzione adenovirale richiede un tempo più breve, costi inferiori e meno sforzo. Il DNA adenovirale a tutta lunghezza è ottenuto ricombinando tra il vettore shuttle e il plasmide pAdEasy-1 nel ceppo E. coli incline alla ricombinazione omologa, BJ5183. Il protocollo implica la trasformazione chimica delle cellule AdEasier-1 (batteri BJ5183 contenenti pAdEasy-1). Questa tecnica non richiede un elettroporatore che potrebbe non essere disponibile in alcuni laboratori, è molto semplice, aumenta la resa di ricombinazione e riduce il tempo necessario per ottenere cellule competenti ed eseguire la trasformazione. La preselezione dei cloni ricombinanti eseguiti da PCR riduce ulteriormente i tempi e facilita l'intera procedura. Una procedura simile è stata utilizzata da Zhao e colleghi22, tuttavia, nel protocollo, abbiamo ottimizzato le sequenze dei primer.
Per l'imballaggio e l'amplificazione del GFP-adenovirus, è stata utilizzata una linea cellulare derivata HEK293, vale a dire cellule AD293, che sono più aderenti alla piastra di coltura. Altre linee cellulari comunemente utilizzate per la produzione adenovirale sono le seguenti: 911, 293FT, pTG6559 (derivato A549), PER. C6 (derivata HER), GH329 (derivata di HeLa), N52. E6, e HeLa-E123,24,25,26. Nelle nostre mani, non è stato ottenuto alcun miglioramento nella produzione adenovirale quando sono state utilizzate 911 cellule (dati non mostrati). La trasfezione delle cellule AD293 con il plasmide ricombinante utilizzando il reagente K2 ha aumentato notevolmente l'efficienza della fase di confezionamento virale. Dopo la produzione di adenovirus, fino a ~ 70% dell'adenovirus è ancora all'interno delle cellule e viene rilasciato da tre cicli di congelamento e scongelamento. Aumentare il numero di cicli non è adatto perché distrugge l'adenovirus.
Durante tutto il processo di produzione adenovirale di routine, numerose particelle virali vengono rilasciate nel mezzo di coltura cellulare. Scartare questo mezzo di coltura cellulare durante la raccolta delle cellule AD293 infette comporterebbe un'importante perdita virale. Abbiamo ottimizzato il protocollo descritto da Schagen e colleghi per purificare le particelle adenovirali dal mezzo di coltura cellulare per precipitazione con solfato di ammonio27. Questo metodo ha una maggiore efficienza nel recupero dell'adenovirus dal mezzo di coltura cellulare rispetto al metodo utilizzando polietilene glicole28. L'adenovirus precipitato deve essere purificato immediatamente con ultracentrifugation o conservato in frigorifero per un paio di giorni ma solo dopo la dialisi, per rimuovere l'eccesso di sale. Mantenere il precipitato più a lungo di poche ore senza dialisi è dannoso per il virus.
La purificazione delle particelle adenovirali mediante ultracentrifugazione eseguita in un solo passaggio riduce la manipolazione del materiale adenovirale e facilita la procedura rispetto ai protocolli utilizzando successivi passaggi di ultracentrifugazione14,29. La dialisi dell'adenovirus purificato è necessaria per rimuovere il cloruro di cesio che può influenzare ulteriormente la trasduzione. Nel protocollo, abbiamo usato il tampone Tris contenente MgCl2 ma non saccarosio per la dialisi, poiché richiede un'enorme quantità ingiustificata di saccarosio che è altrimenti necessaria come conservante per il congelamento. Così, abbiamo aggiunto saccarosio in seguito, direttamente nelle scorte adenovirali preparate per il congelamento. Per evitare frequenti congelamento e scongelamento dell'adenovirus purificato, è consigliabile aliquotare le scorte adenovirali e conservarle a -80 °C. Il titer adenovirale è stato valutato dalla citometria del flusso considerando il gene reporter della GFP e la percentuale di cellule trasdotta per una specifica diluizione virale. Questo metodo è più veloce rispetto al classico "saggio della placca" ed è più fiducioso rispetto alla valutazione delle proteine capsidi (con vari metodi come ELISA o citometria del flusso) che non rivela la capacità di infezione delle particelle adenovirali. Tuttavia, la quantificazione basata su ELISA, il Q-PCR o il test della placca utilizzando kit disponibili in commercio sono metodi alternativi, particolarmente utili per la titolazione di adenovirus che non contengono un tracciante fluorescente.
Considerando che gli adenovirus pAdTrack derivano dal sierotipo 5 di adenovirus umano riconosciuto da Coxsackievirus e Adenovirus Receptors (CAR), abbiamo dimostrato la capacità del GFP-adenovirus di trasdurre cellule di origine umana (cellule endoteliali), ma anche cellule di altre origini: bovine (cellule endoteliali) e murine (cellule stromali mesenchimali ed epatociti). I dati hanno mostrato che il GFP-adenovirus può indurre un alto livello di espressione di un transgene.
In conclusione, abbiamo ottimizzato questa laboriosa tecnologia per ridurre il tempo, i costi e lo sforzo necessario per ottenere le particelle adenovirali. L'adenovirus preparato è in grado di infettare vari tipi di cellule e di indurre l'espressione del gene di interesse. Questo protocollo può essere utilizzato in una varietà di esperimenti poiché il trasferimento genico mediato da adenovirale rappresenta uno dei principali strumenti per lo sviluppo di moderne terapie geniche.
ABBREVIAZIONI: AdV-GFP, particelle adenovirali; BAEC, cellule endoteliali aortiche bovine; CsCl, cloruro di cesio; GFP, proteina fluorescente verde; MSC, cellule stromali mesenchimali; TU, unità trasdutnti.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo lavoro è stato sostenuto da un Progetto cofinanziato dal Fondo europeo di sviluppo regionale attraverso il Programma operativo per la competitività 2014-2020 (POC-A.1-A.1.1.4-E-2015, ID: P_37_668; acronimo DIABETER), sovvenzione del Ministero rumeno della Ricerca e dell'Innovazione PCCDI- UEFISCDI, Numero di progetto PN-III-P1-1.2-PCCDI-2017-0697 all'interno di PNCD III e dall'Accademia Rumena. Gli autori ringraziano Kyriakos Kypreos (Università di Patrasso, Grecia) per i suoi consigli generosi e pertinenti, Ovidiu Croitoru (Università di Belle Arti, Bucarest, Romania) per le riprese, il montaggio cinematografico e il design grafico e Mihaela Bratu per l'assistenza tecnica.
Materiali
| Name | Company | Catalog Number | Comments |
| AD293 cells | Agilent Technologies | 240085 | |
| AdEasier-1 cells | Addgene | 16399 | |
| Agarose I (for electrophoresis) | Thermo Scientific | 17850 | |
| Ammonium sulfate | Sigma | A4418 | |
| Ampicillin sodium salt | Sigma | A0166 | |
| BamH I | Thermo Scientific | FD0054 | |
| Cell culture plates 100 mm | Eppendorf | 30702115 | |
| Cesium chloride | Sigma | L4036 | |
| DH5alpha bacteria | Thermo Scientific | 18265017 | |
| DMEM (GlutaMAX, 4.5g/L D-Glucose) | Gibco | 3240-027 | |
| EA.hy926 cells | ATCC | CRL-2922 | |
| EDTA | Sigma | E5134 | |
| Ethanol (99.8%) | Roth | 5054.2 | |
| Fetal Bovine Serum | Sigma | F7524 | |
| Flasks T25, T75, T175 | Eppendorf | 30712129 | |
| Glucose | Sigma | G7021 | |
| Hepa 1-6 murine hepatocytes | ATCC | CRL-1830 | |
| Hind III | Thermo Scientific | FD0504 | |
| Kanamycin Sulfate | Thermo Scientific | 15160054 | |
| K2 Transfection System | Biontex | T060-5.0 | |
| LB medium | Formedium | LBx0102 | |
| LB-agar | Formedium | LBx0202 | |
| Mix & Go E. coli Transformation kit | Zymo Research | T3001 | |
| Midori Green Advanced DNA stain | Nippon Genetics Europe | MG-04 | |
| NaOH | Sigma | S8045 | |
| Opti-MEM | Thermo Scientific | 31985070 | |
| Pac I | Thermo Scientific | FD2204 | |
| pAdEasy-1 | Addgene | 16400 | |
| pAdTrack-CMV | Addgene | 16405 | |
| Phenol:chloroform:isoamyl alcohol (24:24:1) | Invitrogen | 15593-031 | |
| Polymerase GoTaq | Promega | M3005 | |
| Pme I (Mss I) | Thermo Scientific | FD1344 | |
| Potassium acetate | VWR Chemicals | 43065P | |
| Pst I | Thermo Scientific | FD0614 | |
| Qiagen Midi Prep kit | Qiagen | 12125 | |
| Cell Scraper | TPP | 99003 | |
| SDS | Thermo Scientific | 28365 | |
| Slide-A-Lyzer dialysis cassettes | Thermo Scientific | 66330 | |
| Sodium pyruvate | SIGMA | P5280-100G | |
| Syringe with 23G neeedle | B Braun | 464BR | |
| Tris HCl | Sigma | 1185-53-1 | |
| Trypan blue | Roth | CN76.1 | |
| Tubes 50ml | TPP | 91050 | |
| Ultra-Clear Tubes (14x89 mm) | Beckman Coulter | 344059 | |
| Centrifuge (refrigerated) | Sigma Sartorius | 3-19KS | |
| HeraeusFresco 17 Microcentrifuge | Thermo Scientific | 75002420 | |
| Ultracentrifuge with SW41Ti rotor | Beckman Coulter | Optima L-80 XP | |
| Culture Hood | Thermo Scientific | Class II | |
| Pipettes (0-2µl, 1-10µl, 2-20µl, 10-100µl, 20-200µl, 100-1000µl) | Thermo Scientific | ||
| Dry Block Heating Thermostat | Biosan | TDB-120 | |
| Thermocycle | SensoQuest | 012-103 | |
| Water Bath | Memmert | WNB 14 |
Riferimenti
- Lee, C. S., et al. Adenovirus-Mediated Gene Delivery: Potential Applications for Gene and Cell-Based Therapies in the New Era of Personalized Medicine. Genes and Diseases. 4 (2), 43-63 (2017).
- He, T. C., et al. A simplified system for generating recombinant adenoviruses. Proceedings of the National Academy of Sciences of the United States of America. 95 (5), 2509-2514 (1998).
- Russell, W. C. Update on adenovirus and its vectors. The Journal of General Virology. 81, 2573-2604 (2000).
- Rauschhuber, C., Noske, N., Ehrhardt, A. New insights into stability of recombinant adenovirus vector genomes in mammalian cells. European Journal of Cell Biology. 91 (1), 2-9 (2012).
- Saha, B., Wong, C. M., Parks, R. J. The adenovirus genome contributes to the structural stability of the virion. Viruses. 6 (9), 3563-3583 (2014).
- Kreppel, F., Kochanek, S. Modification of adenovirus gene transfer vectors with synthetic polymers: a scientific review and technical guide. Molecular Therapy: the Journal of the American Society of Gene Therapy. 16 (1), 16-29 (2008).
- Dormond, E., Perrier, M., Kamen, A. From the first to the third generation adenoviral vector: what parameters are governing the production yield. Biotechnol Advances. 27 (2), 133-144 (2009).
- Parks, R. J., et al. A helper-dependent adenovirus vector system: removal of helper virus by Cre-mediated excision of the viral packaging signal. Proceedings of the National Academy of Sciences of the United States of America. 93 (24), 13565-13570 (1996).
- Jager, L., Ehrhardt, A. Emerging adenoviral vectors for stable correction of genetic disorders. Current Gene Therapy. 7 (4), 272-283 (2007).
- Luo, J., et al. A protocol for rapid generation of recombinant adenoviruses using the AdEasy system. Nature Protocols. 2 (5), 1236-1247 (2007).
- Dumitrescu, M., et al. Adenovirus-Mediated FasL Minigene Transfer Endows Transduced Cells with Killer Potential. International Journal of Molecular Sciences. 21 (17), (2020).
- Campos, S. K., Barry, M. A. Current advances and future challenges in Adenoviral vector biology and targeting. Current Gene Therapy. 7 (3), 189-204 (2007).
- Khare, R., Chen, C. Y., Weaver, E. A., Barry, M. A. Advances and future challenges in adenoviral vector pharmacology and targeting. Current Gene Therapy. 11 (4), 241-258 (2011).
- Jager, L., et al. A rapid protocol for construction and production of high-capacity adenoviral vectors. Nature Protocols. 4 (4), 547-564 (2009).
- Zvintzou, E., et al. Pleiotropic effects of apolipoprotein C3 on HDL functionality and adipose tissue metabolic activity. Journal of Lipid Research. 58 (9), 1869-1883 (2017).
- Karavia, E. A., et al. Apolipoprotein A-I modulates processes associated with diet-induced nonalcoholic fatty liver disease in mice. Molecular Medicine. 18, 901-912 (2012).
- Lampropoulou, A., Zannis, V. I., Kypreos, K. E. Pharmacodynamic and pharmacokinetic analysis of apoE4 [L261A, W264A, F265A, L268A, V269A], a recombinant apolipoprotein E variant with improved biological properties. Biochemical Pharmacology. 84 (11), 1451-1458 (2012).
- Zheng, S. Y., Li, D. C., Zhang, Z. D., Zhao, J., Ge, J. F. Adenovirus-mediated FasL gene transfer into human gastric carcinoma. World Journal of Gastroenterology. 11 (22), 3446-3450 (2005).
- Ambar, B. B., et al. Treatment of experimental glioma by administration of adenoviral vectors expressing Fas ligand. Human Gene Therapy. 10 (10), 1641-1648 (1999).
- Okuyama, T., et al. Efficient Fas-ligand gene expression in rodent liver after intravenous injection of a recombinant adenovirus by the use of a Cre-mediated switching system. Gene Therapy. 5 (8), 1047-1053 (1998).
- van Dijk, K. W., Kypreos, K. E., Fallaux, F. J., Hageman, J. Adenovirus-mediated gene transfer. Methods in Molecular Biology. 693, 321-343 (2011).
- Zhao, Y. D., Li, T., Huang, G. A simple negative selection method to identify adenovirus recombinants using colony PCR. Electronic Journal of Biotechnology, North America. 17 (1), 46-49 (2014).
- Kovesdi, I., Hedley, S. J. Adenoviral producer cells. Viruses. 2 (8), 1681-1703 (2010).
- Lin, X. Construction of new retroviral producer cells from adenoviral and retroviral vectors. Gene Therapy. 5 (9), 1251-1258 (1998).
- Fallaux, F. J., et al. Characterization of 911: a new helper cell line for the titration and propagation of early region 1-deleted adenoviral vectors. Human Gene Therapy. 7 (2), 215-222 (1996).
- Altaras, N. E., et al. Production and formulation of adenovirus vectors. Advances in Biochemical Engineering/ Biotechnology. 99, 193-260 (2005).
- Schagen, F. H., et al. Ammonium sulphate precipitation of recombinant adenovirus from culture medium: an easy method to increase the total virus yield. Gene Therapy. 7 (18), 1570-1574 (2000).
- Colombet, J., et al. Virioplankton 'pegylation': use of PEG (polyethylene glycol) to concentrate and purify viruses in pelagic ecosystems. Journal of Microbiological Methods. 71 (3), 212-219 (2007).
- Kypreos, K. E., van Dijk, K. W., van Der Zee, A., Havekes, L. M., Zannis, V. I. Domains of apolipoprotein E contributing to triglyceride and cholesterol homeostasis in vivo. Carboxyl-terminal region 203-299 promotes hepatic very low density lipoprotein-triglyceride secretion. Journal of Biological Chemistry. 276 (23), 19778-19786 (2001).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon