Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Werkzeuge zur Echtzeitbewertung eines Pseudomonas aeruginosa-Infektionsmodells
In diesem Artikel
Zusammenfassung
Synthetisches Mukoviszidose-Sputummedium (SCFM2) kann in Kombination mit konfokaler Laserscanning-Mikroskopie und fluoreszenzaktivierter Zellsortierung verwendet werden, um bakterielle Aggregate mit hoher Auflösung zu beobachten. Dieser Artikel beschreibt Methoden zur Bewertung aggregierter Populationen während der antimikrobiellen Behandlung als Plattform für zukünftige Studien.
Zusammenfassung
Pseudomonas aeruginosa (Pa) ist einer der häufigsten opportunistischen Erreger im Zusammenhang mit Mukoviszidose (CF). Sobald die Pa-Besiedlung etabliert ist, bildet ein großer Teil der infizierenden Bakterien Biofilme im Atemwegssputum. Pa-Biofilme, die aus CF-Sputum isoliert wurden, wachsen nachweislich in kleinen, dichten Aggregaten von ~ 10-1.000 Zellen, die räumlich organisiert sind und klinisch relevante Phänotypen wie antimikrobielle Toleranz aufweisen. Eine der größten Herausforderungen bei der Untersuchung, wie Pa-Aggregate auf die sich verändernde Sputumumgebung reagieren, ist das Fehlen ernährungsrelevanter und robuster Systeme, die die Aggregatbildung fördern. Mit einem synthetischen CF-Sputummedium (SCFM2) kann die Lebensgeschichte von Pa-Aggregaten mittels konfokaler Laserscanning-Mikroskopie (CLSM) und Bildanalyse mit der Auflösung einer einzelnen Zelle beobachtet werden. Dieses In-vitro-System ermöglicht die Beobachtung von Tausenden von Aggregaten unterschiedlicher Größe in Echtzeit, drei Dimensionen und im Mikrometerbereich. Auf Individueller und Populationsebene ermöglicht die Fähigkeit, Aggregate nach Phänotyp und Position zu gruppieren, die Beobachtung von Aggregaten in verschiedenen Entwicklungsstadien und deren Reaktion auf Veränderungen in der Mikroumgebung, wie z.B. Antibiotikabehandlung, präzise zu differenzieren.
Einleitung
Pseudomonas aeruginosa (Pa) ist ein opportunistischer Erreger, der chronische Infektionen bei immungeschwächten Personen hervorruft. Bei Menschen mit der Erbkrankheit Mukoviszidose (CF) können diese Infektionen den Verlauf eines Lebens umfassen. CF verursacht den Aufbau eines viskosen, nährstoffreichen Auswurfs in den Atemwegen, der im Laufe der Zeit von einer Vielzahl mikrobieller Krankheitserreger besiedelt wird. Pa ist einer der häufigsten CF-Erreger, der die Atemwege in der frühen Kindheit besiedelt und schwer zu behandelnde Infektionen etabliert1. Pa bleibt ein signifikantes klinisches Problem und gilt als eine der häufigsten Todesursachen bei Menschen mit CF, trotz verbesserter Therapieschemata in den letzten Jahren2,3. Dieser Persistenzphänotyp und die zunehmende Antibiotikatoleranz haben Pa einen Platz in einer Gruppe von Krankheitserregern eingebracht, die sowohl von den Centers for Disease Control (CDC) als auch von der Weltgesundheitsorganisation (WHO) als Forschungsprioritäten für die Entwicklung neuer therapeutischer Strategien identifiziert wurden - die ESKAPE-Erreger4.
Wie bei anderen ESKAPE-Erregern ist die erworbene Antibiotikaresistenz bei Paüblich, aber es gibt auch viele intrinsische Eigenschaften, die zur antimikrobiellen Toleranz von Pa beitragen. Dazu gehört die Fähigkeit von Pa, Aggregate zu bilden - hochdichte Cluster von ~ 10-1.000 Zellen, die bei mehreren Infektionen beobachtet werden können, einschließlich CF-Patienten-Sputum5,6. Ähnlich wie Pa, das in anderen Biofilmsystemen untersucht wurde, zeigen Pa-Aggregate klinisch relevante Phänotypen wie erhöhte Antibiotikaresistenz und Aktivierung der Zell-Zell-Kommunikation (Quorum Sensing (QS)). Zum Beispiel wurde gezeigt, dass Aggregate von Pa QS-reguliertes Verhalten verwenden, um andere Mikroben zu bekämpfen und antimikrobielle Behandlungen wie die Produktion von Pyocyanin7zu tolerieren. Die Fähigkeit, solche Verhaltensweisen zu untersuchen, bietet einen spannenden Einblick in bakterielle Ökosysteme in einer Umgebung, die derjenigen ähnelt, in der sie im menschlichen Körper vorkommen.
Eine der größten Herausforderungen bei der Untersuchung, wie Pa-Aggregate auf die sich verändernde Sputumumgebung reagieren, ist das Fehlen ernährungsrelevanter und robuster Systeme, die die Aggregatbildung fördern. Vieles von dem, was über Pa bekannt ist, wurde mit In-vitro-Systemen entdeckt, in denen Zellen planktonisch oder in einer charakteristischen oberflächengebundenen "Pilz"-Architektur wachsen, die in vivo8nicht beobachtet wurde. Während klassische Biofilm-Wachstumsmodelle wie Flusszellen oder fester Agar umfangreiches und wertvolles Wissen über bakterielles Verhalten und Mechanismen der Antibiotikatoleranz gewonnen haben, lassen sich diese Ergebnisse nicht immer in vivo übersetzen. Viele In-vitro-Modelle haben eine begrenzte Fähigkeit, die Wachstumsumgebung der menschlichen Infektionsstelle nachzuahmen, was kostspielige In-vivo-Studien erfordert. Im Gegenzug fehlt vielen In-vivo-Modellen die Flexibilität und Auflösung, die In-vitro-Techniken bieten.
Synthetischer Mukoviszidose-Sputum (SCFM2) wurde entwickelt, um eine Umgebung für das Pa-Wachstum zu schaffen, die der bei chronischen Infektionen in der CF-Lunge ähnelt. SCFM2 umfasst Nährstoffquellen, die in schleimlöserter CF-Sputa zusätzlich zu Mucin, Lipiden und DNA identifiziert wurden. Das Pa-Wachstum in SCFM2 erfordert ein nahezu identisches Gen, das für das Wachstum im tatsächlichen Sputum erforderlich ist, und unterstützt die natürlichePa-Aggregatbildung 9,10. Nach der Impfung bilden planktonische Zellen Aggregate, die durch Expansion an Größe zunehmen. Einzelne Zellen (als Migranten bezeichnet) werden aus Aggregaten freigesetzt, wandern in unbesilumisierte Gebiete und bilden neue Aggregate10. Diese Lebensgeschichte kann mittels CLSM und Bildanalyse bei der Auflösung einer einzelnen Zelle beobachtet werden. Die in SCFM2 gebildeten Pa-Aggregate sind ähnlich groß wie die in der CF-Lunge10beobachteten. Dieses Modell ermöglicht die Beobachtung mehrerer Aggregate unterschiedlicher Größe in Echtzeit und in drei Dimensionen im Mikrometerbereich. Die Zeitraffermikroskopie ermöglicht die Verfolgung von Tausenden (~ 50.000) aggregaten in einem Experiment. Die Verwendung von Bildanalysesoftware ermöglicht die Quantifizierung von aggregierten Phänotypen aus Mikroaufnahmen, einschließlich Aggregatvolumen, Oberfläche und Position in drei Dimensionen auf die nächsten 0,1 μm, sowohl auf der Ebene des einzelnen Aggregats als auch der Population. Die Fähigkeit, Aggregate nach Phänotyp und Position zu gruppieren, ermöglicht die präzise Differenzierung von Aggregaten in verschiedenen Entwicklungsstadien sowie ihre Reaktion auf eine sich verändernde Mikroumgebung6,11.
Die Anwendung von SCFM2 zur Untersuchung von Pa-Aggregaten in Assays mit geringem Volumen und hohem Durchsatz macht es zu einem flexiblen, kostengünstigen Modell. Als definiertes Medium bietet SCFM2 Einheitlichkeit und Reproduzierbarkeit über mehrere Plattformen hinweg und bietet eine ernährungsphysiologisch und physikalisch relevante Methode zur Untersuchung von Pa-Aggregaten in vitro9. Zu den Anwendungen gehört die Verwendung in Kombination mit CLSM zur Beobachtung der räumlichen Organisation und antibiotikatoleranz bei hoher Auflösung (wie in diesem Methodenpapier beschrieben). Die Fähigkeit, Experimente durchzuführen, die Echtzeitdaten im Mikrometerbereich liefern, ermöglicht die Untersuchung von Interaktionen innerhalb und zwischen Spezies, wie sie in vivoauftreten können. Zum Beispiel wurde SCFM2 zuvor verwendet, um die räumliche Dynamik der Zell-Zell-Kommunikation in aggregierten Populationen über ein Netzwerk von Systemen zu untersuchen, die von Pa verwendet werden, um mehrere Gene zu regulieren, die zur Virulenz und Pathogenese beitragen6.

Abbildung 1: Grafische Darstellung der wichtigsten experimentellen Schritte. (A) SCFM2 wird mit Pa-Zellen geimpft und darf in einer Kulturschale mit Glasboden Aggregate bilden. (B) Aggregate werden auf das konfokale Mikroskop übertragen und Antibiotika hinzugefügt. Dargestellt sind drei technische Replikate (Kammern 1-3) und eine Kontrollbohrung (4) von geimpften SCFM2 ohne antibiotikafreie Behandlung. Aggregate werden mit CLSM im Laufe von 18 h abgebildet. (C) Nach der ersten 18-h-Bildgebung werden Aggregate mit Propidieniodid behandelt, um tote Zellen zu visualisieren, und mit CLSM abgebildet (D) Aggregate mit gewünschtem Phänotyp werden mitHILFE VON FACS von SCFM2 getrennt. Abkürzungen: SCFM2 = synthetisches Mukoviszidose-Sputummedium; Pa = Pseudomonas aeruginosa; CLSM = konfokale Laserscanning-Mikroskopie; FACS = fluoreszenzaktivierte Zellsortierung. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Hier wird der Nutzen von SCFM2 zur Untersuchung der Auswirkungen der Antibiotikabehandlung auf Pa-Aggregate in Echtzeit demonstriert, gefolgt von der Verwendung eines Zellsortierungsansatzes zur Isolierung von Populationen von Aggregaten mit unterschiedlichen Phänotypen für die nachgelagerte Analyse (Abbildung 1).
Access restricted. Please log in or start a trial to view this content.
Protokoll
1. Synthetisches Mukoviszidosemedium (SCFM2) vorbereiten
HINWEIS: Die Vorbereitung von SCFM2 umfasst drei Hauptphasen, die unten beschrieben werden (Abbildung 2). Ausführliche Informationen und Referenzen finden Sie unter9,10,12.
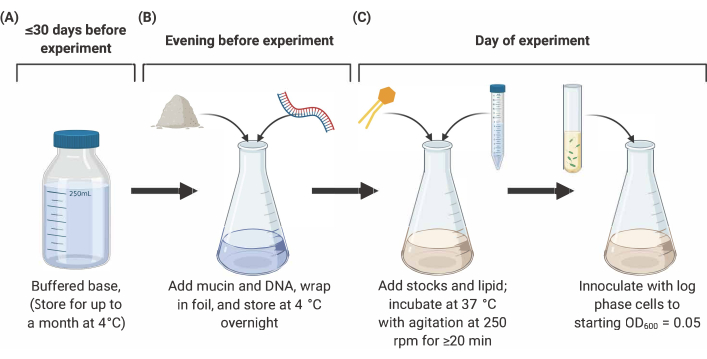
Abbildung 2: Vorbereitung und Impfung des Mediums SCFM2. (A) Gepufferte Base wird unter Verwendung der in Tabelle 1 und Tabelle 2 aufgeführten Salze und Aminosäuren hergestellt. Gepufferte Basis kann bis zu 30 Tage bei 4 °C gelagert werden, muss aber vor Lichteinwirkung geschützt werden. (B) Mucin und DNA werden einem Aliquot aus gepufferter Base zugesetzt und über Nacht bei 4 °C in Lösung gelöst. (C) Lipid und zusätzliche Bestände werden der Übernachtlösung zugesetzt und bei 37 °C unter Rühren bei 250 U / min für 20 min inkubiert. SFCM2 wird dann mit gewaschenen Log-Phasenzellen bei einem OD600 = 0,05 geimpft. Abkürzungen: SCFM2 = synthetisches Mukoviszidose-Sputummedium. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
- Sterilisation von Schweinemucin
- Steriles Mucin in einer Endkonzentration von 5 mg/ml in SCFM2 vorbereiten. Zum Beispiel, für ein 5 ml Volumen von SCFM2, wiegen Sie 25 mg Typ II Mucin in einer sterilen Petrischale und legen Sie es in einen ultravioletten (UV) Sterilisator für 4 h, sanft jede Stunde rühren.
- Nach 4 h das UV-behandelte Mucin unter sterilen Bedingungen in autoklavierte 1,7 ml Röhrchen geben und bei -20 °C lagern.
- Um die vollständige Sterilisation zu bestätigen, lösen Sie eine Mucinprobe in einer sterilen Flüssigkeit wie Wasser oder Luria Bertani (LB) Brühe auf und beobachten Sie sie unter einem Mikroskop.
HINWEIS: Sterilisiertes Mucin kann bei -20 °C bis zu 6 Monate gelagert werden.
- Vorbereitung der gepufferten Basis
- Salz- und Aminosäure-Stammlösungen durch Zugabe der entsprechenden Mengen nach Gewicht zu entionisiertem Wasser gemäß Tabelle 1 zubereiten. Filtersterilisieren Sie alle Lagerlösungen mit einem 0,22 μm-Filter, wickeln Sie sie zum Schutz vor Lichtabbau in Folie und lagern Sie sie bis zu einem Monat bei 4 °C.
- Die gepufferte Base wird durch Kombinieren von 190 ml entionisiertem Wasser mit Aminosäuren und Salzstocklösungen nach den in Tabelle 1aufgeführten Volumina zubereitet. Stellen Sie die Lösung auf pH 6,8 ein und erhöhen Sie sie auf ein Endvolumen von 250 ml. Filtersterilisieren Sie mit einem 0,22 μm-Filter und lagern Sie es bei 4 °C für bis zu 30 Tage.
- Am Abend vor dem Experiment aliquotieren Sie die gewünschte Menge gepufferter Base in einen Glaskulturkolben und fügen Mucin (5 mg / ml wie in Schritt 1.1.1 beschrieben) und gereinigte Lachssperma-DNA (0,6 mg / ml) hinzu. Vorsichtig rühren, in Folie wickeln und über Nacht bei 4 °C belassen, damit sich Mucin und DNA in Lösung auflösen können.
HINWEIS: Lachssperma-DNA-Aliquoten sollten auf Eis aufgetaut, vortexiert und zu gepufferter Base und Mucin hinzugefügt werden. Tryptophan-, Asparagin- und Tyrosin-Stammlösungen müssen in Lösungen von NaOH (Konzentrationen siehe Tabelle 1) anstelle von entionisiertem Wasser hergestellt werden. Bewahren Sie die gepufferte Basis und alle Lagerlösungen in Folie eingewickelt auf, um sie vor Lichteinwirkung zu schützen. Die meisten Aktien werden bis zu einem Monat stabil sein. Bestände, die sich verfärben, sollten nicht verwendet und vor Gebrauch ersetzt werden.
- Hinzufügung von Zusatzbeständen
- Am Tag des Experiments werden die in Tabelle 2 aufgeführten Bestände der gepufferten Basis, die Mucin und DNA enthält, hinzugefügt.
HINWEIS: Bereiten Sie am Tag des Experiments eine frische FeSO4-Lösung vor, aber alle anderen Bestände können im Voraus hergestellt und 30 Tage bei 4 °C gelagert werden. 1,2-Dioleoyl-sn-glycero-3-phosphocholin (DOPC) enthält Chloroform. Behandeln Sie mit Vorsicht und verwenden Sie keine offenen Flammen. Nach Zugabe von DOPC SCFM2 bei 37 °C unter Schütteln (250 U/min) für mindestens 20 min (für 5 mL Kultur) inkubieren. Diese Inkubationszeit lässt das Chloroform im DOPC verdampfen. Der Kolben sollte nicht luftdicht sein; Stattdessen die Kolbenöffnung locker mit Folie abdecken.
- Am Tag des Experiments werden die in Tabelle 2 aufgeführten Bestände der gepufferten Basis, die Mucin und DNA enthält, hinzugefügt.
2. Echtzeitbewertung der antimikrobiellen Toleranz in bakteriellen Aggregaten
- Bereiten Sie Kulturen über Nacht vor
- Am Abend vor dem Experiment impfen Sie 5 ml LB-Brühe mit mehreren Kolonien Pa PAO1-pMRP9-113 aus einer LB-Agarplatte, die ein Antibiotikum (Carbenicillin 300 μg / ml) enthält. Über Nacht bei 37 °C bei Rühren bei 250 U/min wachsen.
HINWEIS: Züchten Sie Übernachtkulturen mit dem Zusatz von Antibiotika, die für die Auswahl der erforderlichen Plasmide erforderlich sind (hier das grün fluoreszierende Protein (GFP) Expressionsplasmid, pMRP9-1). Beachten Sie, dass Pa-Zellen gewaschen werden müssen, bevor SCFM2 geimpft wird. LB kann durch andere reichhaltige Labormedien für Nachtkulturen ersetzt werden. Alle Bakterienisolate sollten in diesem Protokoll unter Verwendung geeigneter BSL-2-Richtlinien behandelt werden.
- Am Abend vor dem Experiment impfen Sie 5 ml LB-Brühe mit mehreren Kolonien Pa PAO1-pMRP9-113 aus einer LB-Agarplatte, die ein Antibiotikum (Carbenicillin 300 μg / ml) enthält. Über Nacht bei 37 °C bei Rühren bei 250 U/min wachsen.
- SCFM2 impfen
- Am Tag des Experiments über Nacht Kulturen von Pa 1:10 (Kultur: flüssige Medien) verdünnen, indem Sie 500 μL in 5 ml frische LB-Brühe impfen. Züchten Sie die Zellen bis zur Log-Phase (60-90 min) bei 37 °C mit Rührung bei 250 U /min.
- Zentrifugen-Log-Phasenkulturen bei 10.000 × g für 5 min. Waschen Sie die Zellen, indem Sie den Überstand entfernen und in 3 ml filtersterilisierter phosphatgepufferter Kochsalzlösung (PBS, pH 7,0) wieder auffüllen. Wiederholen Sie dies zweimal und stellen Sie das Pellet in einem Endvolumen von 1 ml PBS wieder ein.
- Messen Sie die Absorption der gewaschenen Zellen mit einem Spektralphotometer bei 600 nm (OD600)und berechnen Sie das Kulturvolumen, das für einen Start-OD600 von 0,05 in 5 ml SCFM2 erforderlich ist. Impfen Sie Pa in das SCFM2 und wirbeln Sie sanft, um die Zellen überall zu verteilen. 1 ml des geimpften SCFM2 in jede Kammer eines 4-Well-Glasbodens, einer optischen Schüssel pipetten und 4 h statisch bei 37 °C inkubieren.
HINWEIS: Die Verdopplungszeit von Pa-Kulturen ist abhängig von der Belastung und der Verfügbarkeit von Sauerstoff. In SCFM2 beträgt die Verdopplungszeit von Pa unter den hier beschriebenen Bedingungen ~1,4 h10.
3. Visualisierung von Aggregaten während der Antibiotikabehandlung mit konfokaler Laserscanning-Mikroskopie (CLSM)
HINWEIS: In diesem Abschnitt wird die Verwendung von konfokalen Laserscanning-Mikroskopen und Bilderfassungssoftware für die Bildgebung von Pa-Aggregaten in SCFM2 beschrieben. Ziel ist es, die verbleibende (tolerante) bakterielle Biomasse nach der Behandlung mit Antibiotika zu beobachten und zu charakterisieren. Die beschriebenen Schritte können mit Erfolg an anderen konfokalen Mikroskopen durchgeführt werden, obwohl die Bedienungsanleitung des Instruments für eine spezifische Anleitung referenziert werden sollte.
- Bild Pa-Kulturen entweder mit einer beheizten Kammer oder einer beheizten Mikroplatte, die auf dem Mikroskopstand angebracht ist, um eine Umgebungstemperatur von 37 °C aufrechtzuerhalten. Starten Sie das Inkubationsmodul mindestens 2 h vor Beginn des Experiments, damit alle Geräte die gewünschte Temperatur erreichen und die weitere Ausdehnung und Bewegung während der Datenerfassung reduzieren können.
- Nach 4 h SCFM2-Kulturen, die Pa-Zellen enthalten, in den beheizten Mikroskopstadium überführen. Bezeichnen Sie 3 von 4 Vertiefungen als technische Replikate für die Antibiotikabehandlung und betrachten Sie die4. Gut als eine Kontrolle ohne Behandlung, die nur Pa-Zellen in SCFM2 ohne Antibiotikum enthält. Identifizieren Sie Aggregate mithilfe der Hellfeldmikroskopie auf der Registerkarte Lokalisieren vor der Anregung von fluoreszierenden Reportern. Definieren Sie einen Bereich für die Bildgebung innerhalb jeder Vertiefung und speichern Sie ihre Position (x-y-z-Koordinaten) mit dem Positionsmodul in der Bildgebungssoftware.
- Verwenden Sie ein 63-faches Ölimmersionsobjektiv, um Pa-Kulturen zu visualisieren, die das GFP-Expressionsplasmid pMRP9-1 in SCFM2 mit einer Anregungswellenlänge von 488 nm und einer Emissionswellenlänge von 509 nm enthalten. Nehmen Sie Bilder mit der Z-Stack-Option im Erfassungsmodul in Intervallen von 1 μm auf (insgesamt 60 Slices). Verwenden Sie das Line-Averaging-Modul, um die Hintergrundfluoreszenz im GFP-Kanal innerhalb des Gesamtvolumens der 60 μm z-Stack-Bilder (1.093,5 mm3)zu reduzieren. Nehmen Sie Kontrollbilder von uninokuliertem SCFM2 auf, indem Sie identische Einstellungen verwenden, um die Hintergrundfluoreszenz für die Bildanalyse zu bestimmen.
HINWEIS: Bilder werden durch Erzeugen von 512 Pixel x 512 Pixel (0,26 μm x 0,26 μm Pixel) 8-Bit-Z-Stack-Bildern erfasst, die 60 μm von der Basis des Coverlips entfernt sind. - Verwenden Sie die Zeitreihenoption in der Bildgebungssoftware, um 60 Scheiben an jeder Position (Vertiefung) in 15-Minuten-Intervallen über einen Zeitraum von 18 Stunden zu erfassen. Verwenden Sie die definitive Fokusstrategie innerhalb der Software, um für jede Position eine Fokusebene zu speichern, die zu jedem Zeitpunkt während des Experiments zurückgegeben wird.
- Nach insgesamt 4,5 Stunden Inkubation können Sie jede Position mit den oben genannten Einstellungen abbilden, um die aggregierte Biomasse in jeder der vier Vertiefungen vor der Zugabe von Antibiotika zu bestimmen.
- Nach 6 h Gesamtinkubation ein Antibiotikum mit einer minimalen inhibitorischen Konzentration (MIC) von 2x unter der minimalen inhibitorischen Konzentration (MIC) zu jedem Replikat hinzufügen. Direkt und sanft in die Mitte des Brunnens, direkt unterhalb der Luft-Flüssig-Interphase, pipetten. Bewahren Sie alle Kulturen in der beheizten konfokalen Kammer auf.
HINWEIS: Hier wurde Colistinsulfat in einer Konzentration von 140 μg/ml verwendet. - Beginnen Sie mit der bildgebenden behandlung nach einem Antibiotikum, indem Sie im Bildgebungsprogramm auf die Option Experiment starten klicken.
HINWEIS: Die verwendete Antibiotikakonzentration hängt vom Antibiotikum, dem Pa-Isolat, ab und davon, ob der Benutzer Tötungs- oder Toleranzeffekte untersuchen möchte. Dieses Experiment verwendet eine Einzeldosis. Zusätzliche Dosen können bei Bedarf hinzugefügt werden, ohne die Kulturen zu stören.
4. Propidieniodidfärbung von Pa-Aggregaten
HINWEIS: Propidieniodid (PI) wird häufig als Färbereagenz verwendet, um nicht lebensfähige (tote) Bakterienzellen in Kultur zu identifizieren. Hier wird es verwendet, um Aggregate zu identifizieren, die empfindlich auf die in Abschnitt 3 angewendete Antibiotikabehandlung reagieren. In diesem Protokoll wird die Expression und der Nachweis von GFP in Pa-Zellen als Hauptproxy für die Zelllebensfähigkeit verwendet. In diesem letzten Schritt kann die konfokale Bildgebung erneut verwendet werden, um die räumliche Positionierung von lebenden/toten Aggregaten in Relation zueinander zu identifizieren. Zusätzlich werden Aggregate für die weitere nachgelagerte Zellsortierung in Abschnitt 5 als lebend/tot identifiziert.
- Nach 18 h PI in jede Vertiefung der vierkammerigen optischen Bodenschale mit SCFM2-Kulturen geben. Befolgen Sie die Anweisungen des Herstellers für das Volumen des PI und die Inkubationszeit(z. B.~ 2 μL pro ml Kultur, ~ 20-30 min).
5. Isolierung lebender Zellen aus Aggregaten mit einem FACS-Ansatz
HINWEIS: FACS bietet eine leistungsstarke Plattform, um Zellgruppen nach einem fluoreszierend markierten Phänotyp zu sortieren und zu isolieren. Hier wird FACS verwendet, um lebende (antibiotikatolerante) Aggregate aus nicht lebensfähigen Aggregaten zu isolieren.
- Nach dem Färben mit Propidieniodid die Kulturen aus der Inkubation entfernen und in einem isolierten Behälter auf ein FACS-Instrument übertragen, um 37 °C zu halten.
- Führen Sie 1 ml Aliquoten von SCFM2 mit Pa-Aggregaten bei der niedrigsten Durchflussrate aus.
HINWEIS: Jedes Aliquot enthält ~15.000 Aggregate. - Um GFP zu detektieren, beleuchten Sie die Zellen mit einem 488-nm-Laser und zeichnen Sie die fluoreszierende Signalhöhe bei 530/30 nm auf. Visualisieren Sie die PI-Färbung durch Anregung mit einem 561 nm Laser und zeichnen Sie die fluoreszierende Signalhöhe bei 610/20 nm auf. Führen Sie die Sortierung mit einer 70-u-Düse durch.
HINWEIS: Sortierte Aggregate können je nach Anwendung des Benutzers auf verschiedene Arten gepoolt werden. In diesem Fall wurde FACS verwendet, um lebensfähige Pa-Aggregate für die nachgeschaltete RNA-Sequenzierung zu sortieren. Alternative Anwendungen werden im Folgenden erläutert.
6. Bildanalyse
HINWEIS: Die Zeitraffermikroskopie erzeugt große Datenmengen. Ein 18-Stunden-Experiment zur Beobachtung von Pa-Aggregaten in SCFM2 identifiziert im Laufe der Zeit ~50.000 Aggregate, die das Potenzial haben, für Volumen und räumliche Positionierung charakterisiert zu werden. Verwenden Sie eine Bildanalysesoftware, um die aggregierte Dynamik in SCFM2 zu quantifizieren:
- Für aggregierte Studien in SCFM2 quantifizieren Sie die GFP-Hintergrundfluoreszenz, indem Sie ein Histogramm der Zählungen im GFP-Kanal erstellen, das für uninokulierte SCFM2 und SCFM2 erzeugt wird, die mit Pa-Stamm PAO1 geimpft sind, der pMRP9-1 trägt. Um sicherzustellen, dass detektierte GFP-Voxel mit Pa-Biomasse korrelieren, definieren Sie einen GFP+-Voxel als ≥1,5-fachen des GFP-Hintergrundzählwerts.
HINWEIS: Die Hintergrundfluoreszenz ist definiert als der höchste Voxelwert von drei zufällig ausgewählten Positionen, gemittelt. Hintergrundzählungen werden als Standardmaß von allen Pixeln in experimentellen Bildern durch die Bildanalysesoftware subtrahiert. - Erzeugen Sie nach der Hintergrundsubtraktion im Surpass-Modul Isoflächen für alle verbleibenden Voxel.
- Um einzelne Aggregate zu erkennen, aktivieren Sie die Option Objekte teilen und definieren Sie Aggregate als Objekte mit Volumina von ≥5 μm3. Verwenden Sie das Vantage-Modul in der Bildanalysesoftware, um das Volumen x-y-z und die Summe der GFP-Voxel für jedes einzelne Objekt zu berechnen. Exportieren Sie diese Daten auf eine externe Statistikplattform.
HINWEIS: Einige Bildanalysesoftware ermöglicht den Export mehrerer quantitativer Phentoypes gleichzeitig, so dass Korrelationen berechnet werden können. - Filtern Sie die aus dem Vantage-Modul exportierten Daten nach Größe, um sicherzustellen, dass keine Objekte mit <0,5 μm3 (dispergierte Biomasse) zurückbleiben. Berechnen Sie für jedes einzelne Objekt innerhalb des Bildes den Abstand von sich selbst im Verhältnis zu anderen Objekten (Aggregaten) mit dem Vantage-Modul der Bildanalysesoftware oder manuell mit der folgenden Gleichung.
d = sqrt((x2− x1)2 + (y2− y1)2 + (z2− z1)2) (1) - Verwenden Sie SUM- und AVERAGE-Berechnungen, um die Gesamtexmasse und das durchschnittliche Aggregatvolumen zu ermitteln. Alternativ können Sie Daten in andere statistische Plattformen oder Skripte exportieren, z. B.die Verteilung von Aggregaten über Biomasse, wie in den repräsentativen Ergebnissen diskutiert (Skript unveröffentlicht in Zusammenarbeit mit dem Whiteley-Labor, Georgia Institute of Technology).
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Diese Arbeit beschreibt Methoden zur Beobachtung von Pa-Aggregaten mit hoher Auflösung und in einer Umgebung, die der chronischen Infektion der CF-Lunge9,10,12ähnelt. SCFM2 bietet ein In-vitro-System, das die natürliche Aggregation von Pa-Zellen in Größen fördert, die denen ähneln, die während der tatsächlichen Infektion beobachtetwurden 10. Die Anpassungsfähigkeit von...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Diese Arbeit hat Methoden eingeführt, die kombiniert werden können, um bakterielle Aggregatpopulationen in Gegenwart und Abwesenheit von Antibiotikabehandlung zu untersuchen. Hochauflösendes CLSM ermöglicht die Visualisierung von Veränderungen der aggregierten Biomasse und der strukturellen Orientierung von Aggregaten in Echtzeit, wenn sie Antibiotika ausgesetzt sind. Darüber hinaus können physikalische und strukturelle Merkmale der Biomasse, die nach der Behandlung mit Antibiotika übrig bleiben, quantifiziert we...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren haben keine Interessenkonflikte.
Danksagungen
S.E.D wird durch Start-up-Fonds des Department of Molecular Medicine, der University of South Florida, sowie ein CFF-Forschungsstipendium (DARCH19G0), das N.I.H (5R21AI147654 - 02 (PI, Chen)) und das USF Institute on Microbiomes unterstützt. Wir danken dem Whiteley-Labor für die kontinuierliche Zusammenarbeit mit Datensätzen zu diesem Manuskript. Wir danken Dr. Charles Szekeres für die Erleichterung der FACS-Sortierung. Die Figuren wurden von A.D.G und S.E.D mit Biorender.com erstellt.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| Amino acids | |||
| Alanine | Acros Organics | 56-41-7 | |
| Arginine HCl | MP | 1119-34-2 | |
| Asparagine | Acros Organics | 56-84-8 | Prepared in 0.5 M NaOH |
| Cystine HCl | Alfa Aesar | L06328 | |
| Glutamic acid HCl | Acros Organics | 138-15-8 | |
| Glycine | Acros Organics | 56-40-6 | |
| Histidine HCl H2O | Alfa Aesar | A17627 | |
| Isoleucine | Acros Organics | 73-32-5 | |
| Leucine | Alfa Aesar | A12311 | |
| Lysine HCl | Alfa Aesar | J62099 | |
| Methionine | Acros Organics | 63-68-3 | |
| Ornithine HCl | Alfa Aesar | A12111 | |
| Phenylalanine | Acros Organics | 63-91-2 | |
| Proline | Alfa Aesar | A10199 | |
| Serine | Alfa Aesar | A11179 | |
| Threonine | Acros Organics | 72-19-5 | |
| Tryptophan | Acros Organics | 73-22-3 | Prepared in 0.2 M NaOH |
| Tyrosine | Alfa Aesar | A11141 | Prepared in 1.0 M NaOH |
| Valine | Acros Organics | 72-18-4 | |
| Antibiotic | |||
| Carbenicillin | Alfa Aesar | J6194903 | |
| Day-of Stocks | |||
| CaCl2 * 2H2O | Fisher Chemical | C79-500 | |
| Dextrose (D-glucose) | Fisher Chemical | 50-99-7 | |
| 1,2-dioleoyl-sn-glycero-3-phosphocholine (DOPC) | Fisher (Avanti Polar Lipids) | 4235-95-4 | shake 15-20 min at 37 °C to evaporate chloroform |
| FeSO4 * 7H2O | Acros Organics | 7782-63-0 | this stock equals 1 mg/mL, MUST make fresh |
| L-lactic acid | Alfa Aesar | L13242 | pH stock to 7 with NaOH |
| MgCl2 * 6H2O | Acros Organics | 7791-18-6 | |
| N-acetylglucosamine | TCI | A0092 | |
| Prepared solids | |||
| Porcine mucin | Sigma | M1778-100G | UV-sterilize |
| Salmon sperm DNA | Invitrogen | 15632-011 | |
| Stain | |||
| Propidium iodide | Alfa Aesar | J66764MC | |
| Salts | |||
| K2SO4 | Alfa Aesar | A13975 | |
| KCl | Alfa Aesar | J64189 | add solid directly to buffered base |
| KNO3 | Acros Organics | 7757-79-1 | |
| MOPS | Alfa Aesar | A12914 | add solid directly to buffered base |
| NaCl | Fisher Chemical | S271-500 | add solid directly to buffered base |
| Na2HPO4 | RPI | S23100-500.0 | |
| NaH2PO4 | RPI | S23120-500.0 | |
| NH4Cl | Acros Organics | 12125-02-9 | add solid directly to buffered base |
| Consumables | |||
| Conical tubes (15 mL) | Olympus plastics | 28-101 | |
| Conical tubes (50 mL) | Olympus plastics | 28-106 | |
| Culture tubes w/air flow cap | Olympus plastics | 21-129 | |
| 35 mm four chamber glass-bottom dish | CellVis | NC0600518 | |
| Luria Bertani (LB) broth | Genessee Scientific | 11-118 | |
| Phosphate-buffered saline (PBS) | Fisher Bioreagents | BP2944100 | |
| Pipet tips (p200) | Olympus plastics | 23-150RL | |
| Pipet tips (p1000) | Olympus plastics | 23-165RL | |
| Serological pipets (5 mL) | Olympus plastics | 12-102 | |
| Serological pipets (25 mL) | Olympus plastics | 12-106 | |
| Serological pipets (50 mL) | Olympus plastics | 12-107 | |
| Ultrapure water (RNase/DNase free); nanopure water | Genessee Scientific | 18-194 | Nanopure water used for preparation of solutions in Table 1 |
| Syringes (10 mL) | BD | 794412 | |
| Syringes (50 mL) | BD | 309653 | |
| 0.22 mm PES syringe filter | Olympus plastics | 25-244 | |
| PS cuvette semi-mico | Olympus plastics | 91-408 | |
| Software | |||
| Biorender | To prepare the figures | ||
| FacsDiva6.1.3 | Becton Dickinson, San Jose, CA | ||
| Imaris | Bitplane | version 9.6 | |
| Zen Black | |||
| Equipment | |||
| FacsAriallu | Becton Dickinson, San Jose, CA | ||
| LSM 880 confocal laser scanning microscope | Zeiss |
Referenzen
- Ramsay, K. A., et al. The changing prevalence of pulmonary infection in with fibrosis: A longitudinal analysis. Journal of Cystic Fibrosis. 16 (1), 70-77 (2017).
- Bessonova, L., et al. Data from the US and UK cystic fibrosis registries support disease modification by CFTR modulation with ivacaftor. Thorax. 73 (8), 731-740 (2018).
- Breuer, O., et al. Changing prevalence of lower airway infections in young children with cystic fibrosis. American Journal of Respiratory and Critical Care Medicine. 200 (5), 590-599 (2019).
- O'Donnell, J. N., Bidell, M. R., Lodise, T. P. Approach to the treatment of patients with serious multidrug-resistant Pseudomonas aeruginosa infections. Pharmacotherapy. 40 (9), 952-969 (2020).
- Bjarnsholt, T., et al. The in vivo biofilm. Trends in Microbiology. 21 (9), 466-474 (2013).
- Darch, S. E., et al. Spatial determinants of quorum signaling in a Pseudomonas aeruginosa infection model. Proceedings of the National Academy of Sciences of the United States of America. 115 (18), 4779-4784 (2018).
- Zhu, K., Chen, S., Sysoeva, T. A., You, L. Universal antibiotic tolerance arising from antibiotic-triggered accumulation of pyocyanin in Pseudomonas aeruginosa. PLoS Biology. 17 (12), 3000573(2019).
- Ciofu, O., Tolker-Nielsen, T. Tolerance and resistance of Pseudomonas aeruginosa biofilms to antimicrobial agents-how P. aeruginosa can escape antibiotics. Frontiers in Microbiology. 10, 913(2019).
- Turner, K. H., Wessel, A. K., Palmer, G. C., Murray, J. L., Whiteley, M. Essential genome of Pseudomonas aeruginosa in cystic fibrosis sputum. Proceedings of the National Academy of Sciences of the United States of America. 112 (13), 4110-4115 (2015).
- Darch, S. E., et al. Phage inhibit pathogen dissemination by targeting bacterial migrants in a chronic infection model. MBio. 8 (2), 00240(2017).
- Jorth, P., et al. Regional isolation drives bacterial diversification within cystic fibrosis lungs. Cell Host & Microbe. 18 (3), 307-319 (2015).
- Palmer, K. L., Aye, L. M., Whiteley, M. Nutritional cues control Pseudomonas aeruginosa multicellular behavior in cystic fibrosis sputum. Journal of Bacteriology. 189 (22), 8079-8087 (2007).
- Davies, D. G., et al. The involvement of cell-to-cell signals in the development of a bacterial biofilm. Science. 280 (5361), 295-298 (1998).
- Hartmann, R., et al. Quantitative image analysis of microbial communities with BiofilmQ. Nature Microbiology. 6 (2), 151-156 (2021).
- Stacy, A., et al. Bacterial fight-and-flight responses enhance virulence in a polymicrobial infection. Proceedings of the National Academy of Sciences of the United States of America. 111 (21), 7819-7824 (2014).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten