Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Инструменты для оценки в режиме реального времени модели инфекции Pseudomonas aeruginosa
В этой статье
Резюме
Синтетическая среда мокроты при муковисцидозе (SCFM2) может быть использована в сочетании как с конфокальной лазерной сканирующей микроскопией, так и с флуоресцентно-активированной сортировкой клеток для наблюдения бактериальных агрегатов с высоким разрешением. В этой статье подробно описываются методы оценки совокупных популяций во время антимикробного лечения в качестве платформы для будущих исследований.
Аннотация
Pseudomonas aeruginosa (Pa) является одним из наиболее распространенных условно-патогенных микроорганизмов, связанных с муковисцидозом (МВ). Как только колонизация Па установлена, большая часть инфекционных бактерий образует биопленки в мокроте дыхательных путей. Было показано, что биопленки Па, выделенные из мокроты муковисцидного средства, растут в небольших, плотных агрегатах ~ 10-1000 клеток, которые пространственно организованы и демонстрируют клинически значимые фенотипы, такие как устойчивость к противомикробным препаратам. Одной из самых больших проблем при изучении того, как агрегаты Pa реагируют на изменение среды мокроты, является отсутствие питательных и надежных систем, которые способствуют образованию агрегатов. Используя синтетическую среду мокроты CF (SCFM2), историю жизни агрегатов Pa можно наблюдать с помощью конфокальной лазерной сканирующей микроскопии (CLSM) и анализа изображений с разрешением одной клетки. Эта система in vitro позволяет наблюдать тысячи агрегатов различного размера в режиме реального времени, трех измерениях и в микроном масштабе. На индивидуальном и популяционном уровнях способность группировать агрегаты по фенотипу и положению облегчает наблюдение агрегированных агрегатов на разных стадиях развития и их реакцию на изменения в микросреде, такие как лечение антибиотиками, которые должны быть дифференцированы с точностью.
Введение
Pseudomonas aeruginosa (Pa) является оппортунистическим патогеном, который устанавливает хронические инфекции у людей с ослабленным иммунитетом. Для людей с генетическим заболеванием муковисцидоз (МВ) эти инфекции могут охватывать всю жизнь. Муковисцидоз вызывает накопление вязкой, богатой питательными веществами мокроты в дыхательных путях, которая со временем колонизируется различными микробными патогенами. Па является одним из наиболее распространенных патогенов муковисцидоза, колонизируя дыхательные пути в раннем детстве и устанавливая трудноизуемые инфекции1. Па остается значительной клинической проблемой и считается ведущей причиной смертности у лиц с муковисцидозом, несмотря на улучшение схем терапии в последниегоды2,3. Этот фенотип персистенции и повышение толерантности к антибиотикам принесли Па место в группе патогенов, идентифицированных как Центрами по контролю и профилактике заболеваний (CDC), так и Всемирной организацией здравоохранения (ВОЗ) в качестве исследовательских приоритетов для разработки новых терапевтических стратегий - патогенов ESKAPE4.
Как и другие патогены ESKAPE, приобретенная устойчивость к антибиотикам распространена в Pa,но есть также много внутренних свойств, которые способствуют антимикробной толерантности Pa. Среди них способность Па образовывать агрегаты — высокоплотные кластеры из ~10-1000 клеток, которые могут наблюдаться при множественных инфекциях, в том числе при МВ мокроте больного5,6. Подобно Pa, изученным в других системах биопленки, агрегаты Pa демонстрируют клинически значимые фенотипы, такие как повышенная устойчивость к антибиотикам и активация клеточно-клеточной коммуникации (кворум-зондирование (QS)). Например, было показано, что агрегаты Па используют QS-регулируемое поведение для борьбы с другими микробами, а также переносят антимикробные методы лечения, такие как производство пиоцианина7. Способность изучать такое поведение дает захватывающее представление о бактериальных экосистемах в среде, подобной той, в которой они существуют в организме человека.
Одной из самых больших проблем при изучении того, как агрегаты Pa реагируют на изменение среды мокроты, является отсутствие питательных и надежных систем, которые способствуют образованию агрегатов. Многое из того, что известно о Па, было обнаружено с использованием систем in vitro, в которых клетки растут планктонально или в характерной поверхностно-прикрепленной, «грибной» архитектуре, которая не наблюдалась in vivo8. В то время как классические модели роста биопленки, такие как прототочные клетки или твердый агар, дали обширные и ценные знания о поведении бактерий и механизмах толерантности к антибиотикам, эти результаты не всегда переводятся in vivo. Многие модели in vitro имеют ограниченную способность имитировать среду роста места инфекции человека, что требует дорогостоящих исследований in vivo. В свою очередь, многим моделям in vivo не хватает гибкости и разрешения, предоставляемых методами in vitro.
Синтетический муковисцидоз мокроты (SCFM2) предназначен для обеспечения среды для роста Па, аналогичной той, которая наблюдается во время хронической инфекции в легких муковисцидоза. SCFM2 включает источники питания, идентифицированные в отхаркиваемой смуте CF в дополнение к муцину, липидам и ДНК. Рост Pa в SCFM2 требует почти идентичного набора генов, необходимых для роста фактической мокроты, и поддерживает естественное образование агрегата Pa 9,10. После инокуляции планктонные клетки образуют агрегаты, которые увеличиваются в размерах за счет расширения. Отдельные клетки (называемые мигрантами) высвобождаются из агрегатов, мигрируют в неколонизированные районы и образуют новые агрегаты10. Эту историю жизни можно наблюдать с помощью CLSM и анализа изображений с разрешением одной клетки. Агрегаты Па, образующиеся в SCFM2, имеют размеры, аналогичные тем, которые наблюдаются в легкомCF 10. Эта модель позволяет наблюдать несколько агрегатов различного размера в режиме реального времени и в трех измерениях в микроновом масштабе. Покадровая микроскопия позволяет отслеживать тысячи (~50 000) агрегатов в одном эксперименте. Использование программного обеспечения для анализа изображений позволяет количественно оценить совокупные фенотипы по микроснимкам, включая совокупный объем, площадь поверхности и положение в трех измерениях до ближайших 0,1 мкм, как на индивидуальном агрегированном, так и на популяционном уровнях. Наличие способности группировать агрегаты по фенотипу и положению позволяет с точностью дифференцировать агрегаты на разных стадиях развития, а также их реакцию на изменениемикросреды 6,11.
Применение SCFM2 для изучения агрегатов Pa в анализах с низким объемом и высокой пропускной способностью делает его гибкой и экономически эффективной моделью. В качестве определенной среды SCFM2 обеспечивает однородность и воспроизводимость на нескольких платформах, обеспечивая питательную и физически релевантную методику изучения агрегатов Pa in vitro9. Приложения включают его использование в сочетании с CLSM для наблюдения пространственной организации и толерантности к антибиотикам при высоком разрешении (как описано в этой статье о методах). Возможность проводить эксперименты, которые предоставляют данные в микроновом масштабе в режиме реального времени, позволяет изучать внутривидовые и межвидовые взаимодействия, поскольку они могут происходить in vivo. Например, SCFM2 ранее использовался для изучения пространственной динамики клеточно-клеточной коммуникации в совокупных популяциях через сеть систем, используемых Pa для регулирования нескольких генов, которые способствуют вирулентности и патогенезу6.

Рисунок 1:Графическое изображение основных экспериментальных этапов. (A)SCFM2 инокулируют Па-клетками и позволяют формировать агрегаты в чашке со стеклянным дном. (B) Агрегаты переносятся в конфокальный микроскоп и добавляют антибиотик. Изображены три технические реплики (камеры 1-3) и контрольная скважина (4) инокулированного SCFM2 без лечения антибиотиками. Агрегаты визуализируются с использованием CLSM в течение 18 ч. (C) После первоначальной 18-ч визуализации агрегаты обрабатывают йодидом пропидия для визуализации мертвых клеток и визуализируют с помощью CLSM (D) Агрегаты с желаемым фенотипом отделяют от SCFM2 с помощью FACS. Сокращения: SCFM2 = синтетический муковисцидоз мокроты; Pa = Синегнойная палочка; CLSM = конфокальная лазерная сканирующая микроскопия; FACS = флуоресцентная сортировка клеток. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Здесь демонстрируется полезность SCFM2 для изучения влияния лечения антибиотиками на агрегаты Pa в режиме реального времени, за которым следует использование клеточного подхода для выделения популяций агрегатов с различными фенотипами для последующего анализа(рисунок 1).
Access restricted. Please log in or start a trial to view this content.
протокол
1. Подготовьте синтетическую среду муковисцидоза (SCFM2)
ПРИМЕЧАНИЕ: Подготовка SCFM2 состоит из трех основных этапов, описанных ниже(рисунок 2). Для получения полной информации и ссылок см.9,10,12.
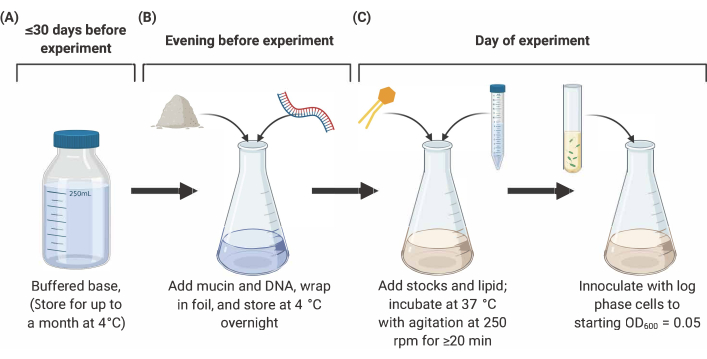
Рисунок 2:Подготовка и инокуляция среды SCFM2. (А)Буферное основание получают с использованием солей и аминокислот, перечисленных в Таблице 1 и Таблице 2. Буферизованная основа может храниться при 4 °C до 30 дней, но должна быть защищена от воздействия света. (B)Муцин и ДНК добавляют к аликвоте буферного основания и растворяют в растворе на ночь при 4 °C.(C)Липиды и дополнительные запасы добавляют в ночной раствор и инкубируют при 37 °C с перемешиванием при 250 об/мин в течение 20 мин. Затем SFCM2 инокулируют промытыми, логарифматическими клетками при OD600 = 0,05. Сокращения: SCFM2 = синтетический муковисцидоз мокроты среды. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
- Стерилизация муцина свиней
- Готовят стерильный муцин в конечной концентрации 5 мг/мл в SCFM2. Например, для объема SCFM2 объемом 5 мл взвесьте 25 мг муцина типа II в стерильной чашке Петри и поместите в ультрафиолетовый (УФ) стерилизатор на 4 ч, осторожно перемешивая каждый час.
- Через 4 ч переведите обработанный УФ-излучение муцин в аутоклавные пробирки по 1,7 мл в стерильных условиях и храните при -20 °C.
- Чтобы подтвердить полную стерилизацию, растворите образец муцина в стерильной жидкости, такой как вода или бульон Luria Bertani (LB), и наблюдайте под микроскопом.
ПРИМЕЧАНИЕ: Стерилизованный муцин можно хранить при -20 °C до 6 месяцев.
- Подготовка буферного основания
- Приготовьте растворы солей и аминокислот, добавив соответствующие количества по весу в деионизированную воду, как указано в таблице 1. Фильтруйте и стерилизуйте все стандартные растворы с помощью фильтра 0,22 мкм, заверните в фольгу для защиты от легкой деградации и храните при 4 °C до одного месяца.
- Готовят буферное основание путем объединения 190 мл деионизированной воды с растворами аминокислот и солей по объемам, перечисленным в таблице 1. Отрегулируйте раствор до рН 6,8 и увеличьте до конечного объема 250 мл. Фильтруют-стерилизуют с помощью фильтра 0,22 мкм и хранят при 4 °C до 30 дней.
- Вечером перед экспериментом аликвотируйте нужное количество буферного основания в стеклянную культурную колбу и добавляйте муцин (5 мг/мл, как описано на этапе 1.1.1) и очищенную ДНК сперматозоидов лосося (0,6 мг/мл). Осторожно перемешать, завернуть в фольгу и оставить при 4 °C на ночь, чтобы муцин и ДНК растворились в растворе.
ПРИМЕЧАНИЕ: Аликвоты ДНК сперматозоидов лосося следует размораживать на льду, вихрь и добавлять в буферное основание и муцин. Растворы триптофана, аспарагина и тирозина необходимо готовить в растворах NaOH (см. таблицу 1 для концентраций) вместо деионизированной воды. Храните буферизованный фундамент и все склады растворов, обернутые в фольгу для защиты от воздействия света. Большинство акций будут стабильными до месяца. Запасы, которые обесцвечиваются, не должны использоваться и должны быть заменены перед использованием.
- Добавление дополнительных запасов
- В день эксперимента добавьте запасы, перечисленные в таблице 2, к буферной основе, содержащей муцин и ДНК.
ПРИМЕЧАНИЕ: Готовят свежий раствор FeSO4 в день эксперимента, но все остальные запасы можно сделать заранее и хранить в течение 30 дней при 4 °C. 1,2-диолеоил-sn-глицеро-3-фосфохолин (DOPC) содержит хлороформ. Относитесь с осторожностью и не используйте близкое открытое пламя. После добавления DOPC инкубируют SCFM2 при 37 °C с встряхиванием (250 об/мин) в течение не менее 20 мин (для культуры 5 мл). Этот инкубационный период позволяет хлороформу в DOPC испаряться. Колба не должна быть герметичной; вместо этого накройте отверстие колбы свободно фольгой.
- В день эксперимента добавьте запасы, перечисленные в таблице 2, к буферной основе, содержащей муцин и ДНК.
2. Оценка толерантности к противомикробным препаратам в бактериальных агрегатах в режиме реального времени
- Подготовка культур на ночь
- Вечером перед экспериментом привить 5 мл бульона LB несколькими колониями Pa PAO1-pMRP9-113 из пластины агара LB, содержащей антибиотик (карбенициллин 300 мкг/мл). Расти в течение ночи при 37 °C с перемешиванием при 250 об/мин.
ПРИМЕЧАНИЕ: Выращивать ночные культуры с добавлением антибиотиков, необходимых для отбора необходимых плазмид (здесь плазмида экспрессии зеленого флуоресцентного белка (GFP), pMRP9-1). Обратите внимание, что Па-клетки должны быть промыты перед инокуляцией SCFM2. LB может быть заменен другими богатыми лабораторными средами для ночных культур. Все бактериальные изоляты должны обрабатываться с использованием соответствующих руководящих принципов BSL-2 на протяжении всего этого протокола.
- Вечером перед экспериментом привить 5 мл бульона LB несколькими колониями Pa PAO1-pMRP9-113 из пластины агара LB, содержащей антибиотик (карбенициллин 300 мкг/мл). Расти в течение ночи при 37 °C с перемешиванием при 250 об/мин.
- Инокулировать SCFM2
- В день эксперимента обратно разбавляют ночные культуры Па 1:10 (культура: жидкие среды) путем инокуляции 500 мкл в 5 мл свежего бульона LB. Выращивают клетки до логарифмической фазы (60-90 мин) при 37 °C с перемешиванием при 250 об/мин.
- Центрифуга log phase культуры при 10 000 × г в течение 5 мин. Промывайте клетки путем удаления супернатанта и повторного использования в 3 мл стерилизованного фильтром фосфатно-буферного физиологического раствора (PBS, pH 7,0). Повторите дважды и повторно суспендируют гранулу в конечном объеме 1 мл PBS.
- Измерьте абсорбируемость промытых клеток с помощью спектрофотометра при 600 нм (OD600)и рассчитайте объем культуры, необходимый для начального OD600 0,05 в 5 мл SCFM2. Инокулируют Па в SCFM2 и осторожно вихрь распределяют клетки по всему. Пипетка 1 мл инокулированного SCFM2 в каждую камеру 4-х скважин, стеклянного дна, оптической чашки и инкубировать в течение 4 ч статически при 37 °C.
ПРИМЕЧАНИЕ: Время удвоения па-культур зависит от штамма и доступности кислорода. В SCFM2, в условиях, описанных здесь, время удвоения Pa составляет ~1,4 ч10.
3. Визуализация агрегатов во время лечения антибиотиками с помощью конфокальной лазерной сканирующей микроскопии (CLSM)
ПРИМЕЧАНИЕ: В этом разделе описывается использование конфокального лазерного сканирующего микроскопа и программного обеспечения для захвата изображений для визуализации агрегатов Pa в SCFM2. Цель состоит в том, чтобы наблюдать и охарактеризовать оставшуюся (толерантную) бактериальную биомассу после лечения антибиотиками. Описанные шаги могут быть успешно выполнены на других конфокальных микроскопах, хотя руководство по эксплуатации прибора должно быть упомянуто для конкретного руководства.
- Изображение культур Pa с использованием либо нагретой камеры, либо нагретой микропластины, установленной на сцене микроскопа, для поддержания температуры окружающей среды 37 °C. Запустите инкубационный модуль по меньшей мере за 2 ч до начала эксперимента, чтобы позволить всему аппарату достичь желаемой температуры и уменьшить дальнейшее расширение и движение во время сбора данных.
- Через 4 ч перекладывать культуры SCFM2, содержащие Па-клетки, на стадию нагретого микроскопа. Назначьте 3 из 4 скважин в качестве технических реплик для лечения антибиотиками, а4-ю скважину рассмотрите как необочевой контроль, содержащую только Па-клетки в SCFM2, без антибиотика. Идентифицируйте агрегаты с помощью микроскопии яркого поля на вкладке «Местоположение» перед любым возбуждением флуоресцентных репортеров. Определите область для визуализации в пределах каждой скважины и сохраните ее положение (координаты x-y-z) с помощью модуля «Позиции» в программном обеспечении для визуализации.
- Используйте 63-кратный масляный объектив для визуализации культур Pa, содержащих плазмиду экспрессии GFP pMRP9-1 в SCFM2 с длиной волны возбуждения 488 нм и длиной волны излучения 509 нм. Снимайте снимки с помощью опции z-stack в модуле Acquisition с интервалом 1 мкм (всего 60 фрагментов). Используйте модуль линейного усреднения для уменьшения фоновой флуоресценции в канале GFP в пределах общего объема изображений z-стека 60 мкм (1 093,5 мм3). Получение контрольных изображений неинокулированного SCFM2 с использованием идентичных настроек для определения фоновой флуоресценции для анализа изображений.
ПРИМЕЧАНИЕ: Изображения получены путем создания 512 пикселей x 512 пикселей (0,26 мкм x 0,26 мкм пикселей) 8-битных изображений z-стека, которые находятся на 60 мкм от основания крышки. - Используйте опцию временных рядов в программном обеспечении для визуализации, чтобы захватить 60 срезов в каждом положении (скважине) с интервалом 15 минут в течение 18 часов. Используйте определенную стратегию фокусировки в программном обеспечении для хранения фокальной плоскости для каждой позиции, которая возвращается в каждую точку времени на протяжении всего эксперимента.
- После в общей сложности 4,5 ч инкубации визуйте каждую позицию, используя вышеуказанные настройки, чтобы определить совокупную биомассу в каждой из четырех скважин до добавления антибиотика.
- После 6 ч полной инкубации добавляют антибиотик в 2 раза ниже минимальной ингибирующей концентрации (МИК) к каждой реплике. Пипетка прямо и осторожно в середину колодца, чуть ниже воздушно-жидкой межфазы. Храните все культуры в отапливаемой конфокальной камере.
ПРИМЕЧАНИЕ: Здесь использовался колистина сульфат в концентрации 140 мкг/мл. - Начните визуализацию после лечения антибиотиками, нажав на опцию Начать эксперимент в программе визуализации.
ПРИМЕЧАНИЕ: Используемая концентрация антибиотика зависит от антибиотика, изолята Па, и от того, хочет ли пользователь изучить убийственные или переносимые эффекты. В этом эксперименте используется одна доза. Дополнительные дозы могут быть добавлены без разрушения культур, если это необходимо.
4. Окрашивание йодида пропидия агрегатов Pa
ПРИМЕЧАНИЕ: Йодид пропидия (PI) обычно используется в качестве окрашивающего реагента для идентификации нежизнеспособных (мертвых) бактериальных клеток в культуре. Здесь он используется для выявления агрегатов, чувствительных к лечению антибиотиками, применяемым в разделе 3. На протяжении всего этого протокола экспрессия и обнаружение GFP в Па-клетках используется в качестве основного прокси для жизнеспособности клеток. Этот заключительный этап позволяет еще раз использовать конфокальную визуализацию для определения пространственного позиционирования живых/мертвых агрегатов по отношению друг к другу. Кроме того, агрегаты идентифицируются как живые/мертвые для дальнейшей последующей сортировки ячеек в разделе 5.
- Через 18 ч добавляют PI в каждую скважину четырехкамерной оптической донных тарелок, содержащей культуры SCFM2. Следуйте инструкциям производителя по объему ПИ и времени инкубации(например,~2 мкл на мл культуры, ~20-30 мин).
5. Изоляция живых клеток от агрегатов с использованием подхода FACS
ПРИМЕЧАНИЕ: FACS представляет собой мощную платформу для сортировки и изоляции групп клеток в соответствии с флуоресцентно помеченным фенотипом. Здесь FACS используется для выделения живых (устойчивых к антибиотикам) агрегатов из нежизностных агрегатов.
- После окрашивания йодидом пропидия удаляют культуры из инкубации и переносят на прибор FACS в изолированном контейнере для поддержания 37 °C.
- Запустите 1 мл аликвот SCFM2, содержащих агрегаты Pa, с наименьшим расходом.
ПРИМЕЧАНИЕ: Каждая аликвота будет содержать ~15 000 агрегатов. - Чтобы обнаружить GFP, осветите ячейки лазером 488 нм и запишите высоту флуоресцентного сигнала на уровне 530/30 нм. Визуализируйте окрашивание PI путем возбуждения с помощью лазера 561 нм и запишите высоту флуоресцентного сигнала на уровне 610/20 нм. Выполните сортировку с помощью 70-еу сопла.
ПРИМЕЧАНИЕ: Отсортированные агрегаты могут быть объединены в пул несколькими способами в зависимости от приложения пользователя. В этом случае FACS использовался для сортировки жизнеспособных агрегатов Pa для последующего секвенирования РНК. Альтернативные приложения обсуждаются ниже.
6. Анализ изображений
ПРИМЕЧАНИЕ: Покадровая микроскопия генерирует большие объемы данных. 18-часовой эксперимент по наблюдению агрегатов Па в SCFM2 выявил ~50 000 агрегатов с течением времени, которые могут быть охарактеризованы для объемного и пространственного позиционирования. Используйте программное обеспечение для анализа изображений для количественной оценки совокупной динамики в SCFM2:
- Для агрегированных исследований в SCFM2 количественно оцените фоновую флуоресценцию GFP путем создания гистограммы количеств в канале GFP, которая производится для неинокуляционных SCFM2 и SCFM2, привитых штаммом Pa PAO1, несущим pMRP9-1. Чтобы убедиться, что обнаруженные вокселы GFP коррелируют с биомассой Pa, определите воксель GFP+ как ≥1,5x значения фонового количества GFP.
ПРИМЕЧАНИЕ: Фоновая флуоресценция определяется как наибольшее значение вокселя из трех случайно выбранных позиций, усредненное. Количество фона, как стандартная мера, вычитается из всех пикселей экспериментальных изображений программным обеспечением для анализа изображений. - После вычитания фона в модуле Surpass создайте изоповерхности для всех оставшихся вокселей.
- Чтобы обнаружить отдельные агрегаты, включите параметр «Разделенные объекты» и определите агрегаты как объекты с объемами ≥5 мкм3. Используйте модуль Vantage в программном обеспечении для анализа изображений для расчета объема, x-y-z и суммы вокселей GFP для каждого отдельного объекта. Экспортируйте эти данные на внешнюю статистическую платформу.
ПРИМЕЧАНИЕ: Некоторые программы для анализа изображений позволяют экспортировать несколько количественных фентоайпов одновременно, что позволяет рассчитывать корреляции. - Фильтруйте данные, экспортируемые из модуля Vantage, по размеру, чтобы гарантировать, что не останется объектов <0,5мкм3 (дисперсная биомасса). Для каждого отдельного объекта в пределах изображения рассчитайте расстояние от себя по отношению к другим объектам (агрегатам) с помощью модуля Vantage программного обеспечения для анализа изображений или вручную с помощью следующего уравнения.
d = sqrt((x2− x1)2 + (y2− y1)2 + (z2− z1)2) (1) - Используйте расчеты SUM и AVERAGE, чтобы найти общую биомассу и средний совокупный объем. В качестве альтернативы можно экспортировать данные в другие статистические платформы или сценарии, например,распределение агрегатов по биомассе, как обсуждается в репрезентативных результатах (сценарий не опубликован в сотрудничестве с лабораторией Уайтли, Технологический институт Джорджии).
Access restricted. Please log in or start a trial to view this content.
Результаты
В этой работе подробно описываются методы наблюдения агрегатов Па в высоком разрешении и в среде, аналогичной хронической инфекциилегкого МВ 9,10,12. SCFM2 обеспечивает систему in vitro, которая способствует естественной агрегации
Access restricted. Please log in or start a trial to view this content.
Обсуждение
Эта работа представила методологии, которые могут быть объединены для изучения бактериальных совокупных популяций при наличии и отсутствии лечения антибиотиками. CLSM с высоким разрешением позволяет визуализировать изменения в совокупной биомассе и структурной ориентации агрегатов ?...
Access restricted. Please log in or start a trial to view this content.
Раскрытие информации
У авторов нет конфликта интересов.
Благодарности
S.E.D поддерживается стартовыми фондами, предоставляемыми Департаментом молекулярной медицины Университета Южной Флориды, а также исследовательским грантом CFF (DARCH19G0) N.I.H (5R21AI147654 - 02 (PI, Chen)) и Институтом микробиомов USF. Мы благодарим лабораторию Уайтли за постоянное сотрудничество с наборами данных, связанных с этой рукописью. Мы благодарим д-ра Чарльза Секереса за содействие в сортировке FACS. Рисунки были созданы A.D.G и S.E.D с использованием Biorender.com.
Access restricted. Please log in or start a trial to view this content.
Материалы
| Name | Company | Catalog Number | Comments |
| Amino acids | |||
| Alanine | Acros Organics | 56-41-7 | |
| Arginine HCl | MP | 1119-34-2 | |
| Asparagine | Acros Organics | 56-84-8 | Prepared in 0.5 M NaOH |
| Cystine HCl | Alfa Aesar | L06328 | |
| Glutamic acid HCl | Acros Organics | 138-15-8 | |
| Glycine | Acros Organics | 56-40-6 | |
| Histidine HCl H2O | Alfa Aesar | A17627 | |
| Isoleucine | Acros Organics | 73-32-5 | |
| Leucine | Alfa Aesar | A12311 | |
| Lysine HCl | Alfa Aesar | J62099 | |
| Methionine | Acros Organics | 63-68-3 | |
| Ornithine HCl | Alfa Aesar | A12111 | |
| Phenylalanine | Acros Organics | 63-91-2 | |
| Proline | Alfa Aesar | A10199 | |
| Serine | Alfa Aesar | A11179 | |
| Threonine | Acros Organics | 72-19-5 | |
| Tryptophan | Acros Organics | 73-22-3 | Prepared in 0.2 M NaOH |
| Tyrosine | Alfa Aesar | A11141 | Prepared in 1.0 M NaOH |
| Valine | Acros Organics | 72-18-4 | |
| Antibiotic | |||
| Carbenicillin | Alfa Aesar | J6194903 | |
| Day-of Stocks | |||
| CaCl2 * 2H2O | Fisher Chemical | C79-500 | |
| Dextrose (D-glucose) | Fisher Chemical | 50-99-7 | |
| 1,2-dioleoyl-sn-glycero-3-phosphocholine (DOPC) | Fisher (Avanti Polar Lipids) | 4235-95-4 | shake 15-20 min at 37 °C to evaporate chloroform |
| FeSO4 * 7H2O | Acros Organics | 7782-63-0 | this stock equals 1 mg/mL, MUST make fresh |
| L-lactic acid | Alfa Aesar | L13242 | pH stock to 7 with NaOH |
| MgCl2 * 6H2O | Acros Organics | 7791-18-6 | |
| N-acetylglucosamine | TCI | A0092 | |
| Prepared solids | |||
| Porcine mucin | Sigma | M1778-100G | UV-sterilize |
| Salmon sperm DNA | Invitrogen | 15632-011 | |
| Stain | |||
| Propidium iodide | Alfa Aesar | J66764MC | |
| Salts | |||
| K2SO4 | Alfa Aesar | A13975 | |
| KCl | Alfa Aesar | J64189 | add solid directly to buffered base |
| KNO3 | Acros Organics | 7757-79-1 | |
| MOPS | Alfa Aesar | A12914 | add solid directly to buffered base |
| NaCl | Fisher Chemical | S271-500 | add solid directly to buffered base |
| Na2HPO4 | RPI | S23100-500.0 | |
| NaH2PO4 | RPI | S23120-500.0 | |
| NH4Cl | Acros Organics | 12125-02-9 | add solid directly to buffered base |
| Consumables | |||
| Conical tubes (15 mL) | Olympus plastics | 28-101 | |
| Conical tubes (50 mL) | Olympus plastics | 28-106 | |
| Culture tubes w/air flow cap | Olympus plastics | 21-129 | |
| 35 mm four chamber glass-bottom dish | CellVis | NC0600518 | |
| Luria Bertani (LB) broth | Genessee Scientific | 11-118 | |
| Phosphate-buffered saline (PBS) | Fisher Bioreagents | BP2944100 | |
| Pipet tips (p200) | Olympus plastics | 23-150RL | |
| Pipet tips (p1000) | Olympus plastics | 23-165RL | |
| Serological pipets (5 mL) | Olympus plastics | 12-102 | |
| Serological pipets (25 mL) | Olympus plastics | 12-106 | |
| Serological pipets (50 mL) | Olympus plastics | 12-107 | |
| Ultrapure water (RNase/DNase free); nanopure water | Genessee Scientific | 18-194 | Nanopure water used for preparation of solutions in Table 1 |
| Syringes (10 mL) | BD | 794412 | |
| Syringes (50 mL) | BD | 309653 | |
| 0.22 mm PES syringe filter | Olympus plastics | 25-244 | |
| PS cuvette semi-mico | Olympus plastics | 91-408 | |
| Software | |||
| Biorender | To prepare the figures | ||
| FacsDiva6.1.3 | Becton Dickinson, San Jose, CA | ||
| Imaris | Bitplane | version 9.6 | |
| Zen Black | |||
| Equipment | |||
| FacsAriallu | Becton Dickinson, San Jose, CA | ||
| LSM 880 confocal laser scanning microscope | Zeiss |
Ссылки
- Ramsay, K. A., et al. The changing prevalence of pulmonary infection in with fibrosis: A longitudinal analysis. Journal of Cystic Fibrosis. 16 (1), 70-77 (2017).
- Bessonova, L., et al. Data from the US and UK cystic fibrosis registries support disease modification by CFTR modulation with ivacaftor. Thorax. 73 (8), 731-740 (2018).
- Breuer, O., et al. Changing prevalence of lower airway infections in young children with cystic fibrosis. American Journal of Respiratory and Critical Care Medicine. 200 (5), 590-599 (2019).
- O'Donnell, J. N., Bidell, M. R., Lodise, T. P. Approach to the treatment of patients with serious multidrug-resistant Pseudomonas aeruginosa infections. Pharmacotherapy. 40 (9), 952-969 (2020).
- Bjarnsholt, T., et al. The in vivo biofilm. Trends in Microbiology. 21 (9), 466-474 (2013).
- Darch, S. E., et al. Spatial determinants of quorum signaling in a Pseudomonas aeruginosa infection model. Proceedings of the National Academy of Sciences of the United States of America. 115 (18), 4779-4784 (2018).
- Zhu, K., Chen, S., Sysoeva, T. A., You, L. Universal antibiotic tolerance arising from antibiotic-triggered accumulation of pyocyanin in Pseudomonas aeruginosa. PLoS Biology. 17 (12), 3000573(2019).
- Ciofu, O., Tolker-Nielsen, T. Tolerance and resistance of Pseudomonas aeruginosa biofilms to antimicrobial agents-how P. aeruginosa can escape antibiotics. Frontiers in Microbiology. 10, 913(2019).
- Turner, K. H., Wessel, A. K., Palmer, G. C., Murray, J. L., Whiteley, M. Essential genome of Pseudomonas aeruginosa in cystic fibrosis sputum. Proceedings of the National Academy of Sciences of the United States of America. 112 (13), 4110-4115 (2015).
- Darch, S. E., et al. Phage inhibit pathogen dissemination by targeting bacterial migrants in a chronic infection model. MBio. 8 (2), 00240(2017).
- Jorth, P., et al. Regional isolation drives bacterial diversification within cystic fibrosis lungs. Cell Host & Microbe. 18 (3), 307-319 (2015).
- Palmer, K. L., Aye, L. M., Whiteley, M. Nutritional cues control Pseudomonas aeruginosa multicellular behavior in cystic fibrosis sputum. Journal of Bacteriology. 189 (22), 8079-8087 (2007).
- Davies, D. G., et al. The involvement of cell-to-cell signals in the development of a bacterial biofilm. Science. 280 (5361), 295-298 (1998).
- Hartmann, R., et al. Quantitative image analysis of microbial communities with BiofilmQ. Nature Microbiology. 6 (2), 151-156 (2021).
- Stacy, A., et al. Bacterial fight-and-flight responses enhance virulence in a polymicrobial infection. Proceedings of the National Academy of Sciences of the United States of America. 111 (21), 7819-7824 (2014).
Access restricted. Please log in or start a trial to view this content.
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены