Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Bildbasierte Methoden zur Untersuchung von Membrantransportereignissen in stomatalen Zellen
In diesem Artikel
Zusammenfassung
Hier werden mehrere häufig verwendete Methoden vorgestellt, um die Membrantransportereignisse einer Plasmamembranrezeptorkinase zu untersuchen. Dieses Manuskript beschreibt detaillierte Protokolle, einschließlich der Vorbereitung des Pflanzenmaterials, der pharmakologischen Behandlung und der konfokalen Bildgebung.
Zusammenfassung
In eukaryotischen Zellen werden Membrankomponenten, darunter Proteine und Lipide, innerhalb des Endomembransystems räumlich und zeitlich an ihren Bestimmungsort transportiert. Dazu gehören der sekretorische Transport von neu synthetisierten Proteinen an die Zelloberfläche oder die Außenseite der Zelle, der endozytäre Transport von extrazellulären Ladungen oder Plasmamembrankomponenten in die Zelle und das Recycling oder der Shuttletransport von Ladungen zwischen den subzellulären Organellen usw. Membrantransportereignisse sind entscheidend für die Entwicklung, das Wachstum und die Anpassung an die Umwelt aller eukaryotischen Zellen und unterliegen daher einer strengen Regulierung. Zelloberflächenrezeptorkinasen, die Ligandensignale aus dem extrazellulären Raum wahrnehmen, durchlaufen sowohl sekretorischen als auch endozytären Transport. Häufig verwendete Ansätze zur Untersuchung der Membrantransportereignisse unter Verwendung einer Plasmamembran-lokalisierten Leucin-Rich-Repeat-Rezeptorkinase, ERL1, werden hier beschrieben. Zu den Ansätzen gehören die Vorbereitung des Pflanzenmaterials, die pharmakologische Behandlung und die konfokale Bildgebung. Um die räumlich-zeitliche Regulation von ERL1 zu überwachen, beschreibt diese Arbeit die Kolokalisationsanalyse zwischen ERL1 und einem multivesikulären Körpermarkerprotein, RFP-Ara7, die Zeitreihenanalyse dieser beiden Proteine und die Z-Stapel-Analyse von ERL1-YFP, die mit den Membrantransportinhibitoren Brefeldin A und Wortmannin behandelt wurde.
Einleitung
Der Membrantransport ist ein konservierter zellulärer Prozess, der Membrankomponenten (auch bekannt als Frachten), einschließlich Proteine, Lipide und andere biologische Produkte, zwischen verschiedenen Organellen innerhalb einer eukaryotischen Zelle oder über die Plasmamembran zum und vom extrazellulären Raum verteilt1. Dieser Prozess wird durch eine Ansammlung von Membranen und Organellen erleichtert, die als Endomembransystem bezeichnet werden und aus der Kernmembran, dem endoplasmatischen Retikulum, dem Golgi-Apparat, den Vakuolen/Lysosomen, der Plasmamembran und mehreren Endosomen bestehen1. Das Endomembransystem ermöglicht die Modifikation, Verpackung und den Transport von Membrankomponenten mithilfe dynamischer Vesikel, die zwischen diesen Organellen pendeln. Membrantransportereignisse sind entscheidend für die Zellentwicklung, das Wachstum und die Anpassung an die Umwelt und unterliegen daher einer strengen und komplexen Regulierung2. Derzeit wurden mehrere Ansätze in der Molekularbiologie, der chemischen Biologie, der Mikroskopie und der Massenspektrometrie entwickelt und auf das Gebiet des Membrantransports angewendet und haben das Verständnis der raumzeitlichen Regulation des Endomembransystems erheblich vorangebracht 3,4. Die Molekularbiologie wird für klassische genetische Manipulationen der mutmaßlichen Akteure verwendet, die am Membrantransport beteiligt sind, wie z. B. die Veränderung der Genexpression des interessierenden Proteins oder die Markierung des interessierenden Proteins mit bestimmten Markierungen. Zu den Werkzeugen in der chemischen Biologie gehört die Verwendung von Molekülen, die spezifisch in den Verkehr bestimmter Routen eingreifen 4,5. Die Massenspektrometrie ist leistungsfähig für die Identifizierung der Komponenten in einer Organelle, die durch biochemische Ansätze mechanisch isoliert wurde 3,4. Der Membranverkehr ist jedoch ein dynamischer, vielfältiger und komplexer biologischer Prozess1. Um den Prozess des Membrantransports in lebenden Zellen unter verschiedenen Bedingungen sichtbar zu machen, ist die Lichtmikroskopie ein wesentliches Werkzeug. Kontinuierliche Fortschritte wurden bei fortschrittlichen Mikroskoptechniken erzielt, um die Herausforderungen bei der Messung der Effizienz, Kinetik und Vielfalt der Ereignisse zu bewältigen4. Hier konzentriert sich diese Studie auf die weit verbreiteten Methoden in der chemischen/pharmakologischen Biologie, Molekularbiologie und Mikroskopie, um Membrantransportereignisse in einem natürlich vereinfachten und experimentell zugänglichen System, dem stomatären Entwicklungsprozess, zu untersuchen.
Spaltöffnungen sind Mikroporen auf pflanzlichen Luftoberflächen, die sich öffnen und schließen, um den Gasaustausch zwischen den inneren Zellen und der Umgebung zu erleichtern 6,7,8. Daher sind Spaltöffnungen essentiell für die Photosynthese und Transpiration, zwei Ereignisse, die für das Überleben und Wachstum von Pflanzen entscheidend sind. Die Stomaentwicklung wird dynamisch durch Umwelteinflüsse angepasst, um die Anpassung der Pflanze an die Umgebung zu optimieren9. Die Identifizierung des Rezeptorproteins Too Many Mouths (TMM) aus dem Jahr 2002 eröffnete eine neue Ära der Erforschung der molekularen Mechanismen der Stomataentwicklung in der Modellpflanze Arabidopsis thaliana10. Bereits nach wenigen Jahrzehnten wurde ein klassischer Signalweg identifiziert. Von Upstream zu Downstream umfasst dieser Signalweg eine Gruppe von sekretorischen Peptidliganden aus der Familie der epidermalen Musterungsfaktoren (EFP), mehrere Zelloberflächen-Leucin-Rich-Repeat (LRR)-Rezeptorkinasen in der EREECTA (ER)-Familie, das LRR-Rezeptorprotein TMM, eine MAPK-Kaskade und mehrere bHLH-Transkriptionsfaktoren, darunter SPEECHLESS (SPCH), MUTE, FAMA und SCREAM (SCRM)11,12,13,14, 15,16,17,18,19,20,21,22,23,24,25,26. Frühere Arbeiten deuten darauf hin, dass eine der Rezeptorkinasen, ER-LIKE 1 (ERL1), aktives subzelluläres Verhalten bei EPF-Wahrnehmung zeigt20. ERL2 transportiert auch dynamisch zwischen der Plasmamembran und einigen intrazellulären Organellen27. Die Blockierung der Membrantransportschritte führt zu einer abnormen Stomatamusterbildung, die zu Stomataclustern auf der Blattoberflächeführt 28. Diese Ergebnisse deuten darauf hin, dass der Membranverkehr eine wesentliche Rolle bei der Entwicklung der Spaltöffnungen spielt. Diese Studie beschreibt ein Protokoll zur räumlichen und zeitlichen Untersuchung der ERL1-Dynamik mittels Protein-Protein-Subzellulär-Kolokalisationsanalyse in Kombination mit pharmakologischer Behandlung mit einigen Membrantransportinhibitoren.
Protokoll
1. Vorbereitung der Lösungen
- Bereiten Sie die Saatgutsterilisationslösung vor, indem Sie 15 ml Bleichmittel mit 35 ml destilliertem Wasser und 50 μl Triton X-100 mischen.
- Bereiten Sie Brefeldin A (BFA)-Lösung vor, indem Sie das BFA-Pulver in Ethanol bis zu einer Endkonzentration von 10 mM (Stamm) auflösen. Bereiten Sie die Wortmannin (Wm)-Lösung vor, indem Sie das Wm-Pulver in DMSO auf eine Endkonzentration von 10 mM (Stamm) auflösen.
2. Aussaat der Samen
- Aliquotieren Sie 10-50 Samen von jeder der erforderlichen transgenen Pflanzen in 1,5 ml Mikrozentrifugenröhrchen. Geben Sie 1 ml Saatgutsterilisationslösung in jedes Röhrchen und mischen Sie es gründlich, indem Sie das Röhrchen 10 Minuten lang vorsichtig auf einem Schüttler umdrehen.
- Entsorgen Sie die Samensterilisationslösung und waschen Sie die Samen fünfmal mit 1 ml autoklaviertem destilliertem Wasser.
- Geben Sie 300 μl autoklaviertes destilliertes Wasser in jedes Röhrchen und säen Sie die Samen auf halbstarken MS-Medien mit 1 % (w/v) Saccharose und 0,75 % (w/v) Agar. Ergänzen Sie das Medium bei Bedarf mit den entsprechenden Antibiotika.
- Bewahren Sie die Platte 2 Tage lang kopfüber bei 4 °C im Dunkeln auf, um die Keimung zu synchronisieren.
- Nach 2 Tagen wird die Platte in einen Wachstumsraum mit einem 16 h Licht/8 h Dunkelzyklus (80 μmol/m2/s1) bei 22 °C überführt. Dies gilt als der erste Tag nach der Keimung (1 dpg).
- Verpflanze die Sämlinge mit 10 dpg in die Erde, um weiter wachsen zu können.
3. Herstellung von zweifarbigen transgenen F1-Pflanzen
- In einem Wachstumsraum mit einem Zyklus von 16 h Licht / 8 h (80 μmol/m2/s1) bei 22 °C werden homozygote transgene Pflanzen, die ein RFP-markiertes Markerprotein RFP-Ara7 (Pflanze B)20 tragen, und homozygote transgene Pflanzen, die ein RFP-markiertes Markerprotein RFP-Ara7 (Pflanze B)20 tragen, bis zur Blüte nebeneinander gezüchtet.
- Wähle einen jungen Blütenstand von Pflanze A. Behalte eine Blüte, die sich gerade auf dem Blütenstand öffnet, für genetische Kreuzungen. Entferne alle älteren Blüten und Siliques. Entfernen Sie außerdem vorsichtig die jüngeren Blüten und Blütenmeristeme, um zukünftige Verwechslungen zu vermeiden.
- Zerlege die ungeöffnete Blüte vorsichtig, indem du die Kelchblätter, Blütenblätter und Staubblätter mit einer scharfen Pinzette entfernst. Lassen Sie den Stempel nur am Blütenstand.
- Nimm ein reifes Staubblatt von einer geöffneten Blüte auf Pflanze B und lege die Pollenkörner auf die Narbe des präparierten Stempels von Pflanze A.
- Beschriften Sie diese manuell gekreuzte Blume, indem Sie ihren Vater, ihre Mutter und das Datum der genetischen Kreuzung angeben. Lassen Sie die Blüte reifen und ernten Sie die F1-Samen, wenn die Kieselsäure gelb/braun wird (~20 Tage nach der Kreuzung). Prüfen Sie, ob es sich um eine erfolgreiche F1-Anlage handelt, die sowohl YFP- als auch RFP-Signale enthält.
4. Pharmakologische Behandlung
- Für die BFA-Behandlung werden die Keimblätter von 7 Tage alten Keimlingen entfernt, der Rest der Keimlinge entweder in eine Scheinlösung (0,3 % Ethanol) oder eine 30 μM BFA-Lösung getaucht, 1 Minute lang Vakuum angelegt und die Probe vor der Bildgebung 30 Minuten lang eingetaucht.
- Für die Wortmannin-Behandlung entfernen Sie die Keimblätter von 7 Tage alten Sämlingen, tauchen Sie den Rest der Keimlinge entweder in 0,25%ige DMSO-Lösung oder 25 mM Wortmannin, legen Sie 1 Minute lang Vakuum an und lassen Sie die Probe 30 Minuten lang eingetaucht, bevor Sie sie bebildern.
- Für die Vakuumbehandlung von Proben werden in einem 1,5-ml-Mikrozentrifugenröhrchen 500 μl der entsprechenden Arzneimittellösung hinzugefügt und die präparierten Keimlinge in die Lösung getaucht. Befestigen Sie eine 10-ml-Spritze fest am Mikrozentrifugenröhrchen und legen Sie 1 Minute lang Vakuum an (Abbildung 1). Entfernen Sie die Spritze und bewahren Sie die Probe für die erforderliche Zeit in der Lösung auf. Nimm die Sämlinge vorsichtig heraus, um sie bildgebend vorzubereiten.

Abbildung 1: Einfaches Vakuumgerät. Für die Vakuumbehandlung wird eine 10-ml-Spritze an ein 1,5-ml-Mikrozentrifugenröhrchen angeschlossen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
5. Probenvorbereitung für die Bildgebung
- Sezieren Sie das echte Blatt der behandelten Probe mit einer scharfen Rasierklinge. Lege das echte Blatt vorsichtig in einen Wassertropfen auf einen Objektträger und halte die abaxiale Seite nach oben. Decken Sie das echte Blatt langsam mit einem Deckglas ab und vermeiden Sie es, Blasen einzufangen.
6. Konfokale Bildgebung
HINWEIS: In dieser Arbeit wurde ein inverses konfokales Scanning-Mikroskop Leica SP8 verwendet, um das Fluoreszenzsignal der Proben abzubilden.
- Einrichtung des Strahlwegs
- Wählen Sie einen 514-nm-Laser für die YFP-Anregung. Verwenden Sie eine hohe Laserleistung, um die Signalintensität zu erhöhen und so eine hohe Bildqualität zu erzielen. Bei Laserleistungen über 5 % besteht jedoch die Gefahr eines Photobleichens, und die Gesundheit der Probe kann beeinträchtigt werden. Wenn das Probensignal nicht zu schwach ist, beginnen Sie mit niedriger Laserintensität.
- Schalten Sie PMT/HyD für die Detektion ein und definieren Sie die oberen und unteren Emissionsbandschwellen basierend auf dem Spektrum für den YFP-Fluorophor. Stellen Sie ein Detektionsfenster von 530-570 nm ein, um das YFP-Signal zu erfassen.
- Sequenzielle Bildgebung für die zweite Farbe: Klicken Sie auf die Schaltfläche Seq und fügen Sie einen neuen Kanal hinzu. Standardmäßig ist der zuvor entworfene YFP-Strahlengang Seq 1. Wählen Sie in Seq 2 einen 561-nm-Laser für die RFP-Anregung aus und stellen Sie ein Detektionsfenster von 570-630 nm ein, um das RFP-Signal zu erfassen.
- Einrichten der Scanbedingungen (Abbildung 2)
- Um die Aktivität des subzellulären Membranverkehrs in den Stomata-Vorläuferzellen abzubilden, wählen Sie eine 63x/1,2-W-Corr-Linse auf dem System für die Bildgebung.
- Das Format bezieht sich auf die Bildgröße in Pixeln. Beginnen Sie mit 1.024 x 1.024 Pixeln und optimieren Sie diese dann anhand des Zoomfaktors und der Objektiveinstellungen für eine gute Auflösung der Publikationsqualität.
- Die Geschwindigkeit bezieht sich auf die Geschwindigkeit des Scankopfes, wenn der Laser über jedes Pixel fährt. Obwohl langsame Scangeschwindigkeiten oft zu einem besseren Signal-Rausch-Verhältnis führen, können sie die sich schnell ändernden Membranverkehrsereignisse möglicherweise nicht effizient erfassen. Beginnen Sie mit einer Geschwindigkeit von 400 Hz und optimieren Sie diese basierend auf der spezifischen Situation der Proben.
- Der Zoomfaktor wird verwendet, um einen Interessenbereich zu vergrößern, ohne das Objektiv zu wechseln. Beginnen Sie mit einem Zoomfaktor von 1 und optimieren Sie diesen basierend auf den spezifischen Anforderungen des Experiments.
- Der Zeilendurchschnitt bezieht sich darauf, wie oft jede X-Linie gescannt wird, um ein durchschnittliches Ergebnis zu erhalten. Ein größerer Linienmittelwert reduziert das Rauschen im resultierenden Bild, erhöht aber auch die Scanzeit und die Belichtungszeit für das Laserlicht. Verwenden Sie zum Abbilden von Membrantransportereignissen keinen hohen Liniendurchschnitt. Beginnen Sie mit einem 2-fachen Liniendurchschnitt und optimieren Sie nach Bedarf.
- Erfassen einer Zeitreihe
HINWEIS: Bei der Untersuchung von Membrantransportereignissen wird häufig eine Zeitreihe benötigt, um die schnelle Bewegung der subzellulären Endosomen aufzuzeichnen.- Wählen Sie im Erfassungsmodus den xyt-Scanmodus aus, um das Zeitreihendienstprogramm zu aktivieren.
- Legen Sie unter Zeitintervall eine Wartezeit zwischen den Zeitpunkten fest. Alternativ können Sie auch auf Minimieren klicken, um die Bilder sofort nacheinander aufzunehmen. Im folgenden Zeitreihenexperiment wurde ein Zeitintervall von 7 s verwendet.
- Wählen Sie Dauer aus, und geben Sie die Gesamtzeit für die Ausführung des Experiments ein. Alternativ können Sie die Anzahl der zu erfassenden Frames definieren, indem Sie Frames auswählen (Abbildung 3).
- Um die dreidimensionalen Informationen über das Membrantransportereignis innerhalb der gesamten Zelle zu sammeln, verwenden Sie den Z-Stapel-Scan-Modus. Für die Analyse wird häufig die Projektion mit maximaler Intensität der Z-Stapelbilder verwendet.
- Wählen Sie den xyz-Scanmodus im Erfassungsmodus aus, und dann wird auf das Dienstprogramm Z-Stapel zugegriffen.
- Wählen Sie die Option Z-breit aus. Definieren Sie im Scan-Modus das obere und das untere Bild des Z-Stapels mit den Schaltflächen " Anfang " und " Ende" .
- Definieren Sie die Dicke des z-Schritts, indem Sie auf z-Schrittgröße klicken. Alternativ können Sie festlegen, wie viele Bilder aufgenommen werden sollen, um den gesamten Bereich des Z-Stapels abzudecken. Um die Konsistenz zwischen den Proben sicherzustellen, definieren Sie die z-Schrittweite (Abbildung 4).
- Bildverarbeitung
- Die maximale Intensitätsprojektion der Z-Stack-Bilder wird von der konfokalen Software (http://www.leica-microsystems.com) erzeugt. Wählen Sie auf der primären Registerkarte "Prozess " zunächst die gewünschte Z-Stack-Datei unter der Registerkarte " Projekte öffnen" ganz links aus. Wechseln Sie dann zur mittleren Registerkarte mit dem Namen Prozesswerkzeuge, wählen Sie die Projektionsfunktion , wählen Sie Maximum im Dropdown-Bereich von Methode und klicken Sie auf die Schaltfläche Anwenden . Ein Z-Stapel-Projektionsbild wird auf der Registerkarte "Projekte öffnen" generiert.
- Das Video der Zeitreihenbilder stammt von Fidschi (https://imagej.net/Fiji). Verwenden Sie auf der Registerkarte Datei die Funktion Öffnen , um alle Zeitreihenbilder in der richtigen Reihenfolge zu öffnen. Suchen Sie auf der Registerkarte Bild die Funktion Stapeln , und wählen Sie Zu stapelnde Bilder aus , um ein Video zu generieren. Speichern Sie die Datei im .avi-Format mit der gewünschten Bildrate (5 Bilder/s für Video 1).
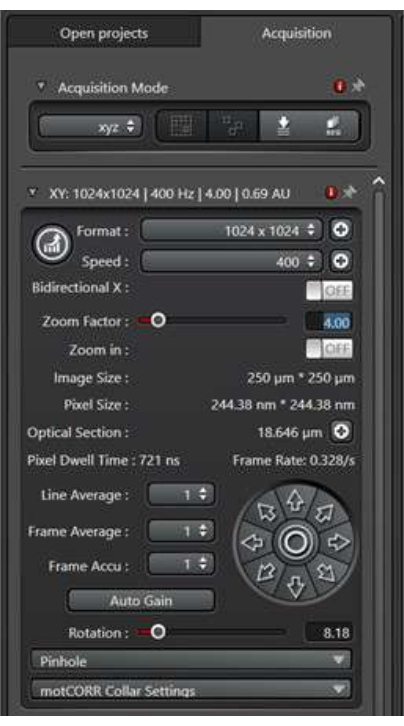
Abbildung 2: Der Scan-Modus-Bereich der XY-Bemaßung. Das Bedienfeld "Scanmodus" wird verwendet, um die Bedingungen für das Scannen von Bildern einzurichten. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
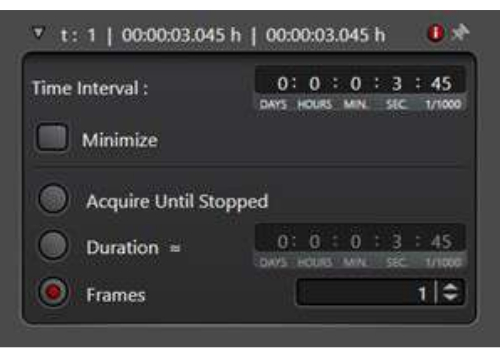
Abbildung 3: Das Zeitreihendienstprogramm im xyt-Scan-Modus. Das Zeitreihendienstprogramm wird verwendet, um die Bildverarbeitungsbedingungen einzurichten, um eine Reihe von Bildern nacheinander zu erfassen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
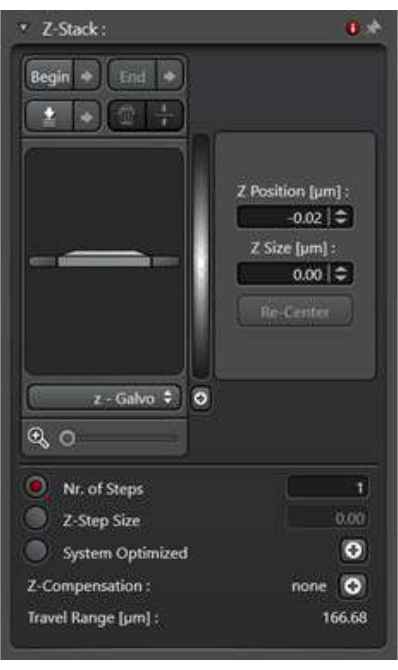
Abbildung 4: Das Z-Stack-Dienstprogramm im xyz-Scan-Modus. Das Z-Stapel-Dienstprogramm wird verwendet, um die Bildgebungsbedingungen einzurichten, um eine Reihe von Bildern auf der Z-Achse zu erfassen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergebnisse
Eine frühere Studie deutete darauf hin, dass ERL1 eine aktive Rezeptorkinase ist, die dynamische Membrantransportereignisse durchläuft20. ERL1 ist eine Transmembran-LRR-Rezeptorkinase auf der Plasmamembran. Neu synthetisiertes ERL1 im endoplasmatischen Retikulum wird in den Golgi-Körpern prozessiert und weiter zur Plasmamembran transportiert. Die ERL1-Moleküle auf der Plasmamembran können EPF-Liganden mit Hilfe ihrer extrazellulären LRR-Domäne18 wahrnehmen. Nach der ...
Diskussion
Das Endomembransystem trennt das Zytoplasma einer eukaryotischen Zelle in verschiedene Kompartimente, was die spezialisierte biologische Funktion dieser Organellen ermöglicht. Um Frachtproteine und Makromoleküle zum richtigen Zeitpunkt an ihren endgültigen Bestimmungsort zu bringen, werden zahlreiche Vesikel zwischen diesen Organellen hin- und hergeleitet. Hochregulierte Membrantransportereignisse spielen eine grundlegende Rolle für die Lebensfähigkeit, Entwicklung und das Wachstum von Zellen. Der Mechanismus, der d...
Offenlegungen
Die Autoren erklären, dass keine Interessenkonflikte bestehen.
Danksagungen
Diese Arbeit wurde von der National Science Foundation (IOS-2217757) (X.Q.) und dem Bronson Foundation Award (H.Z.) der University of Arkansas for Medical Sciences (UAMS) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 10 mL syringes | VWR | BD309695 | Vacuum samples |
| Brefeldin A (BFA) | Sigma | B7651 | membrane trafficking drug |
| Confocal Microscope | Leica | Lecia SP8 TCS with LAS-X software package | Imaging |
| Dissecting Forceps | VWR | 82027-402 | Genetic cross |
| Fiji | NIH | https://imagej.net/Fiji | Image processing |
| Leica LAS AF software | Leica | http://www.leica-microsystems.com | Image processing |
| transgenic seeds of ERL1-YFP | Qi, X. et al. The manifold actions of signaling peptides on subcellular dynamics of a receptor specify stomatal cell fate. Elife. 9, doi:10.7554/eLife.58097, (2020). | ||
| transgenic seeds of RFP-Ara7 | Ebine, K. et al. A membrane trafficking pathway regulated by the plant-specific RAB GTPase ARA6. Nat Cell Biol. 13 (7), 853-859, doi:10.1038/ncb2270, (2011). | ||
| Wortmannin (Wm) | Sigma | W1628 | membrane trafficking drug |
Referenzen
- Aniento, F., Sanchez de Medina Hernandez, V., Dagdas, Y., Rojas-Pierce, M., Russinova, E. Molecular mechanisms of endomembrane trafficking in plants. Plant Cell. 34 (1), 146-173 (2022).
- Sigismund, S., et al. Endocytosis and signaling: Cell logistics shape the eukaryotic cell plan. Physiological Reviews. 92 (1), 273-366 (2012).
- Lyu, Z., Genereux, J. C. Methodologies for measuring protein trafficking across cellular membranes. ChemPlusChem. 86 (10), 1397-1415 (2021).
- Rodriguez-Furlan, C., Raikhel, N. V., Hicks, G. R. Merging roads: Chemical tools and cell biology to study unconventional protein secretion. Journal of Experimental Botany. 69 (1), 39-46 (2017).
- Foissner, I., Sommer, A., Hoeftberger, M., Hoepflinger, M. C., Absolonova, M. Is wortmannin-induced reorganization of the trans-Golgi network the key to explain charasome formation. Frontiers in Plant Science. 7, 756 (2016).
- Qi, X., Torii, K. U. Hormonal and environmental signals guiding stomatal development. BMC Biology. 16 (1), 21 (2018).
- Han, S. K., Kwak, J. M., Qi, X. Stomatal lineage control by developmental program and environmental cues. Frontiers in Plant Science. 12, 751852 (2021).
- Bharath, P., Gahir, S., Raghavendra, A. S. Abscisic acid-induced stomatal closure: An important component of plant defense against abiotic and biotic stress. Frontiers in Plant Science. 12, 615114 (2021).
- Becklin, K. M., Ward, J. K., Way, D. A. . Photosynthesis, Respiration, and Climate Change., 1st edition. , (2021).
- Yang, M., Sack, F. D. The too many mouths and four lips mutations affect stomatal production in Arabidopsis. Plant Cell. 7 (12), 2227-2239 (1995).
- Hara, K., Kajita, R., Torii, K. U., Bergmann, D. C., Kakimoto, T. The secretory peptide gene EPF1 enforces the stomatal one-cell-spacing rule. Genes & Development. 21 (14), 1720-1725 (2007).
- Hara, K., et al. Epidermal cell density is autoregulated via a secretory peptide, EPIDERMAL PATTERNING FACTOR 2 in Arabidopsis leaves. Plant & Cell Physiology. 50 (6), 1019-1031 (2009).
- Hunt, L., Gray, J. E. The signaling peptide EPF2 controls asymmetric cell divisions during stomatal development. Current Biology. 19 (10), 864-869 (2009).
- Sugano, S. S., et al. Stomagen positively regulates stomatal density in Arabidopsis. Nature. 463 (7278), 241-244 (2010).
- Kondo, T., et al. Stomatal density is controlled by a mesophyll-derived signaling molecule. Plant & Cell Physiology. 51 (1), 1-8 (2010).
- Hunt, L., Bailey, K. J., Gray, J. E. The signalling peptide EPFL9 is a positive regulator of stomatal development. New Phytologist. 186 (3), 609-614 (2010).
- Shpak, E. D., McAbee, J. M., Pillitteri, L. J., Torii, K. U. Stomatal patterning and differentiation by synergistic interactions of receptor kinases. Science. 309 (5732), 290-293 (2005).
- Lin, G., et al. A receptor-like protein acts as a specificity switch for the regulation of stomatal development. Genes & Development. 31 (9), 927-938 (2017).
- Lee, J. S., et al. Direct interaction of ligand-receptor pairs specifying stomatal patterning. Genes & Development. 26 (2), 126-136 (2012).
- Qi, X., et al. The manifold actions of signaling peptides on subcellular dynamics of a receptor specify stomatal cell fate. Elife. 9, e58097 (2020).
- MacAlister, C. A., Ohashi-Ito, K., Bergmann, D. C. Transcription factor control of asymmetric cell divisions that establish the stomatal lineage. Nature. 445 (7127), 537-540 (2007).
- Pillitteri, L. J., Sloan, D. B., Bogenschutz, N. L., Torii, K. U. Termination of asymmetric cell division and differentiation of stomata. Nature. 445 (7127), 501-505 (2007).
- Ohashi-Ito, K., Bergmann, D. C. Arabidopsis FAMA controls the final proliferation/differentiation switch during stomatal development. Plant Cell. 18 (10), 2493-2505 (2006).
- Kanaoka, M. M., et al. SCREAM/ICE1 and SCREAM2 specify three cell-state transitional steps leading to Arabidopsis stomatal differentiation. Plant Cell. 20 (7), 1775-1785 (2008).
- Bergmann, D. C., Lukowitz, W., Somerville, C. R. Stomatal development and pattern controlled by a MAPKK kinase. Science. 304 (5676), 1494-1497 (2004).
- Wang, H., Ngwenyama, N., Liu, Y., Walker, J. C., Zhang, S. Stomatal development and patterning are regulated by environmentally responsive mitogen-activated protein kinases in Arabidopsis. Plant Cell. 19 (1), 63-73 (2007).
- Ho, C. M., Paciorek, T., Abrash, E., Bergmann, D. C. Modulators of stomatal lineage signal transduction alter membrane contact sites and reveal specialization among ERECTA kinases. Developmental Cell. 38 (4), 345-357 (2016).
- Le, J., et al. Auxin transport and activity regulate stomatal patterning and development. Nature Communications. 5, 3090 (2014).
- Geldner, N., et al. The Arabidopsis GNOM ARF-GEF mediates endosomal recycling, auxin transport, and auxin-dependent plant growth. Cell. 112 (2), 219-230 (2003).
- Qi, X., et al. Autocrine regulation of stomatal differentiation potential by EPF1 and ERECTA-LIKE1 ligand-receptor signaling. Elife. 6, 24102 (2017).
- Heilemann, M., et al. Subdiffraction-resolution fluorescence imaging with conventional fluorescent probes. Angewandte Chemie. 47 (33), 6172-6176 (2008).
- Leighton, R. E., Alperstein, A. M., Frontiera, R. R. Label-free super-resolution imaging techniques. Annual Review of Analytical Chemistry. 15 (1), 37-55 (2022).
- Oreopoulos, J., Berman, R., Browne, M. Spinning-disk confocal microscopy: Present technology and future trends. Methods in Cell Biology. 123, 153-175 (2014).
- Gao, R., et al. Cortical column and whole-brain imaging with molecular contrast and nanoscale resolution. Science. 363 (6424), (2019).
- Nwaneshiudu, A., et al. Introduction to confocal microscopy. Journal of Investigative Dermatology. 132 (12), (2012).
- Sanderson, J. Multi-photon microscopy. Current Protocols. 3 (1), 634 (2023).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten