Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
In vivo Visualisierung der spontanen Aktivität im sensorischen Kortex von Neonatalen Mäusen mit einer Auflösung von einzelnen Neuronen
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Primäre sensorische Areale im Neokortex zeigen während der Entwicklung einzigartige spontane Aktivitäten. In diesem Artikel wird beschrieben, wie einzelne Neuronenaktivitäten und primäre sensorische Areale visualisiert werden können, um bereichsspezifische synchrone Aktivitäten bei neugeborenen Mäusen in vivo zu analysieren.
Zusammenfassung
Das Gehirn von Säugetieren durchläuft während der pränatalen und postnatalen Periode dynamische Entwicklungsveränderungen sowohl auf zellulärer als auch auf Schaltkreisebene. Nach der Entdeckung zahlreicher Gene, die zu diesen Entwicklungsveränderungen beitragen, ist nun bekannt, dass auch die neuronale Aktivität diese Prozesse wesentlich moduliert. In der sich entwickelnden Großhirnrinde weisen Neuronen synchronisierte Aktivitätsmuster auf, die auf jeden primären sensorischen Bereich spezialisiert sind. Diese Muster unterscheiden sich deutlich von denen, die in der reifen Hirnrinde beobachtet werden, was ihre Rolle bei der Regulierung bereichsspezifischer Entwicklungsprozesse unterstreicht. Defizite in der neuronalen Aktivität während der Entwicklung können zu verschiedenen Erkrankungen des Gehirns führen. Diese Ergebnisse unterstreichen die Notwendigkeit, die Regulationsmechanismen zu untersuchen, die den Aktivitätsmustern in der neuronalen Entwicklung zugrunde liegen. Diese Arbeit fasst eine Reihe von Protokollen zusammen, um primäre sensorische Areale und neuronale Aktivität bei neonatalen Mäusen zu visualisieren, die Aktivität einzelner Neuronen innerhalb der kortikalen Teilfelder mittels Zwei-Photonen-Mikroskopie in vivo abzubilden und teilfeldbezogene Aktivitätskorrelationen zu analysieren. Wir zeigen repräsentative Ergebnisse der Patchwork-artigen synchronen Aktivität innerhalb einzelner Fässer im somatosensorischen Kortex. Wir diskutieren auch verschiedene mögliche Anwendungen und einige Einschränkungen dieses Protokolls.
Einleitung
Die Großhirnrinde umfasst mehrere sensorische Areale mit unterschiedlichen Funktionen. Die Areale erhalten Eingaben, die von den entsprechenden Sinnesorganen ausgehen und meist über das Rückenmark oder den Hirnstamm weitergeleitet und über den Thalamus 1,2 weitergeleitet werden. Bemerkenswert ist, dass Neuronen in jedem primären sensorischen Bereich während der frühen Entwicklungsstadien eine einzigartig synchronisierte Aktivität aufweisen, die ebenfalls von den Sinnesorganen oder den unteren Nervenzentren ausgeht, sich aber im Wesentlichen von den Aktivitäten unterscheidet, die im reifen Kortex beobachtet werden3.
Bei neonatalen Nagetieren beispielsweise zeigt der primäre Sehbereich (V1) eine wellenartige Aktivität, die von der Netzhaut ausgeht (Netzhautwelle) und sich über die gesamte Sehbahn ausbreitet, während die Retinotopieerhalten bleibt 4. Der primäre auditorische Bereich (A1) zeigt eine synchrone Aktivität, die in bandförmigen Subregionen organisiert ist, die den Isofrequenzbändern im reifen Gehirn entsprechen. Die Aktivität geht von den inneren Haarzellen der Cochlea aus 5,6. Der Fasskortex im primären somatosensorischen Bereich (S1) zeigt ein patchworkartiges Aktivitätsmuster, in dem Neuronen der Schicht 4 innerhalb einzelner Fässer, nämlich Neuronen, die auf einzelne Schnurrhaare reagieren, synchron aktiviert werden7. Obwohl vermutet wird, dass sie vom Trigeminusganglion ausgeht, ist die Quelle der Aktivität unbekannt7. Folglich sind die Aktivitätsmuster von Neugeborenen sowohl innerhalb jedes primären sensorischen Bereichs als auch innerhalb der intra-räumlichen Teilbereiche spezialisiert. Die gleichzeitige Visualisierung der neuronalen Aktivität und der Struktur primärer sensorischer Areale kann eine Untersuchung des Beitrags dieser Aktivitätsmuster zur Entwicklung sensorischer Systeme erleichtern.
In diesem Artikel haben wir eine Reihe von Protokollen zusammengefasst: (1) zur Visualisierung einzelner neuronaler Aktivitäten unter Verwendung der spärlichen Markierung von GCaMP und primären sensorischen Bereichen unter Verwendung von TCA-RFP-Mäusen, die rot fluoreszierendes Protein in thalamokortikalen Axonen exprimieren7, (2) zur Abbildung der Aktivität auf Einzelzellebene bei neonatalen Mäusen mittels Zwei-Photonen-Mikroskopie in vivound (3) die Aktivitätskorrelationen innerhalb des S1-Fasskortex zu analysieren. Die repräsentativen Ergebnisse zeigen eine Patchwork-artig synchronisierte Aktivität innerhalb einzelner Fässer einer postnatalen Tag (P)6 Maus. Trotz einiger Einschränkungen kann diese Technik für die chronische Bildgebung, die Weitfeldbildgebung über mehrere sensorische Bereiche und verschiedene Manipulationsexperimente verwendet werden. Die facettenreiche Analyse der neuronalen Aktivität während der Entwicklung wird unser Verständnis der Mechanismen der Bildung von Schaltkreisen im Gehirn bereichern.
Protokoll
Alle Versuche wurden in Übereinstimmung mit den Richtlinien für Tierversuche der Universität Kumamoto und des Nationalen Instituts für Genetik durchgeführt und von den Tierversuchskommissionen genehmigt.
1. In-utero-Elektroporation (IUE)
- Verpaaren Sie männliche TCA-RFP-Mäuse mit ICR-Hintergrund mit weiblichen Wildtyp-ICR-Mäusen. Beobachten Sie den Vaginalpfropfen, um am frühen Morgen des nächsten Tages nach Paarung zu suchen. Beobachten Sie den Bauch, um 2 Wochen später nach einer Schwangerschaft zu suchen.
- Bereiten Sie eine Plasmidlösung her, die 5 ng/μl TRE-nCre, 1 μg/μl CAG-loxP-stop-loxP-GCaMP6s-ires-tTA-WPRE und 0,02 % Trypanblau enthält, um Neuronen mit GCaMP unter Verwendung des Supernova-Systems8 spärlich zu markieren.
- Bereiten Sie eine analgetische Lösung vor, die 0,5 mg/ml Carprofen und 0,01 mg/ml Buprenorphin enthält. Schützen Sie es vor Licht und bewahren Sie es bei Raumtemperatur auf.
- Am embryonalen Tag (E)14 ist die IUE wie folgt durchzuführen. Die IUE-Methoden sind im Wesentlichen die gleichen wie in den vorangegangenen Berichten 9,10,11.
- Wischen Sie den Labortisch mit 70%iger Ethanol- oder Glutaraldehydlösung ab. Sterilisieren Sie alle chirurgischen Instrumente durch Autoklavieren. Tragen Sie eine Maske und einen Laborkittel, um das Infektionsrisiko für die Maus zu verringern.
- Bereiten Sie Mikropipetten aus Glas mit einem Mikropipettenabzieher vor. Nehmen Sie die Plasmidmischungslösung mit einem Aspiratorröhrchen in die Glasmikropipette.
- Induzieren Sie die Anästhesie der trächtigen Mäuse bei E14 mit Isofluran (2,0 % an der Luft). Halten Sie die Mäuse während des Eingriffs mit Isofluran (1,2% an der Luft) anästhesiert. Kneifen Sie den Zeh zusammen, um sicherzustellen, dass die Narkose tief genug ist. Verwenden Sie eine Augensalbe, um Trockenheit der Augen vorzubeugen.
- Desinfizieren Sie den Bauchbereich mindestens dreimal abwechselnd mit Peeling auf Jodbasis und 70 % Ethanol. Legen Sie ein steriles Abdecktuch an, um den Operationsbereich abzudecken. Machen Sie einen Schnitt in der Mittellinie und legen Sie die Gebärmutter auf dem Tuch frei.
- Wiederholen Sie das Beträufeln von warmer Kochsalzlösung auf die Gebärmutter, um zu verhindern, dass sie bis zum Bauchverschluss austrocknet oder abkühlt (Schritt 1.4.8).
- Injizieren Sie die Plasmidlösung nacheinander mit einer Mikropipette und einem Aspiratorröhrchen in eine Seite des Seitenventrikels der Embryonen.
- Die Embryoköpfe werden mit einer Pinzettenelektrode eingeklemmt und mit einem Elektroporator fünfmal im Abstand von 1 s quadratische elektrische Impulse (40 V, 50 ms) abgegeben.
- Bringen Sie die Gebärmutter wieder in die Bauchhöhle zurück. Tragen Sie ~3 ml warme Kochsalzlösung in den Hohlraum auf. Vernähen Sie die Peritonealmembran und die Bauchhaut. Die analgetische Lösung mit 10 μl/g Gewicht (Carprofen mit 5 μg/g Gewicht und Buprenorphin mit 0,1 μg/g Gewicht) unter der Haut im Nacken verabreichen.
- Legen Sie die Maus in einen Aufwachkäfig auf einem Heizkissen (37 °C), um sich von der Narkose zu erholen. Lege die Maus auf den Bauch, um zu verhindern, dass ihre Zunge und ihr Speichel den Hals ersticken. Halten Sie die Maus von anderen Mäusen getrennt und überwachen Sie sie, bis sie sich wieder normal bewegt. Setzen Sie die Maus wieder in den Käfig ein.
- Führen Sie nach der Geburt eine Genotypisierung durch, um nach RFP-Allel zu suchen, und euthanasieren Sie die Welpen ohne Allel. Dieser Schritt wird empfohlen, insbesondere wenn die Bildgebung in der zweiten postnatalen Woche durchgeführt wird, wenn das TCA-RFP-Signal schwach und schwer zu überprüfen ist (Schritt 2.2.7).
2. Schädelfenster-OP
- Bereiten Sie den Cortex-Puffer mit 125 mM NaCl, 5 mM KCl, 10 mM Glukose, 10 mM HEPES, 2 mM CaCl2 und 2 mM MgSO4 in ddH2O (pH-Wert eingestellt auf 7,4 mit 1 M NaOH)12 vor dem Tag der Operation vor. Sterilisieren Sie den Puffer mit einem Vakuumfilter.
HINWEIS: Der Puffer kann bis zu 3 Monate bei 4 °C aufbewahrt werden. Das erforderliche Volumen beträgt 5-10 mL pro Welpe. - Führen Sie die folgenden Schritte auf P3-12 aus. Weitere Informationen finden Sie auch in den vorherigen Berichten, in denen dieses Verfahren beschrieben wurde13,14.
- Mischen Sie 50 mg Agarose mit 5 mL Cortex-Puffer und lösen Sie die Agarose durch Erhitzen vollständig auf. Geben Sie einen Teil der Lösung in ein 1,5-ml-Röhrchen und halten Sie es bei 42 °C.
- Geben Sie etwas Cortex-Puffer in ein konisches 50-ml-Röhrchen und bewahren Sie es bei Raumtemperatur auf. Geben Sie etwas Kochsalzlösung in ein Gefäß (z. B. eine Kappe mit einem konischen 50-ml-Röhrchen) und bewahren Sie es bei Raumtemperatur auf.
- Bereiten Sie eine analgetische Lösung vor, die 0,01 mg/ml Buprenorphin enthält. Schützen Sie es vor Licht und bewahren Sie es bei Raumtemperatur auf.
- Sterilisieren Sie alle chirurgischen Instrumente durch Autoklavieren. Desinfizieren Sie das Fluoreszenz-Stereomikroskop mit 70%igem Ethanol.
- Verwenden Sie einen Isofluran-Verdampfer, um beim Welpen eine Anästhesie mit Isofluran (2,0% in Luft) einzuleiten. Achten Sie auf den Welpen und nehmen Sie ihn heraus, wenn sich seine Atmung leicht verlangsamt.
HINWEIS: Wenn die Anästhesie verlängert wird, kann die Atmung des Welpen für einige Sekunden aufhören. Selbst wenn die Atmung des Welpen wieder aufgenommen wird, kann eine längere Narkose den zerebralen Blutfluss des Welpen verringern und irreversible Hirnschäden verursachen. - Desinfizieren Sie den Welpenkopf mindestens dreimal abwechselnd mit Peeling auf Jodbasis und 70 % Ethanol. Legen Sie den Welpen unter ein Fluoreszenz-Stereomikroskop auf das Heizkissen (35 °C). Halten Sie den Welpen immer mit Isofluran (1,5%-2,5% an der Luft) betäubt.
- Wählen Sie die Welpen aus, die TCA-RFP und GCaMP exprimieren, indem Sie die Fluoreszenz durch den Schädel beobachten. Euthanasiere die Welpen, die nicht beides exprimieren.
- Entfernen Sie die Kopfhaut oberhalb der Gehirnhälften so weit wie möglich vorsichtig, um keine Blutungen zu verursachen. Reiben Sie den Schädel mit einem sterilen, mit Kochsalzlösung getränkten Wattestäbchen ab, um das Bindegewebe zu entfernen.
- Nach dem Trocknen des Schädels kleben Sie die eingeschnittene Kopfhautoberfläche mit einem Gewebekleber auf den Schädel.
- Übertragen Sie den Welpen auf ein 37 °C heißes Heizkissen, um sich von der Narkose zu erholen. Warten Sie mindestens 15 Minuten, bis sich der Klebstoff verfestigt hat.
HINWEIS: Pausieren Sie bis zu 1 Stunde, bevor Sie mit dem nächsten Schritt fortfahren. Bereiten Sie andere Welpen in dieser Zeit bei Bedarf auf ähnliche Weise vor. - Betäuben Sie den Welpen mit Isofluran. Legen Sie den Welpen unter Betäubung (1,5 %-2,5 % Isofluran an der Luft) unter ein Fluoreszenz-Stereomikroskop (37 °C).
HINWEIS: Wenn die Anästhesie länger dauert als die Zeit bis zum Endpunkt (60 Minuten), euthanasieren Sie den Welpen durch Enthauptung während der Narkose mit Isofluran. - Markieren Sie die GCaMP-exprimierende Stelle auf dem Schädel mit einem sterilen Permanentmarker. Wenden Sie Cortex-Puffer auf die Stelle an.
- Führe die Ecke einer Rasierklinge in den Schädel ein. Schiebe die Klinge langsam an, um den Schädel abzurasieren und ein Loch zu machen. Kneifen Sie den rissigen Schädel mit einer Pinzette ab und entfernen Sie ihn.
- Überprüfen Sie, ob das Schädelloch erfolgreich hergestellt wurde, indem Sie die Blutgefäße in dem Loch beobachten. Wenn Blutungen auftreten, spülen Sie das Loch schnell mit Cortex-Puffer mit einer Mikropipette aus. Wiederholen Sie das Spülen, bis die Blutung vollständig aufhört.
- Tragen Sie einen Tropfen Cortex-Puffer auf das Schädelloch auf und legen Sie es in ein steriles rundes Deckglas mit einem Durchmesser von 3 mm über das Loch. Wischen Sie den überschüssigen Puffer mit Vliesstoff ab. Warten Sie, bis der Bereich um das Glas herum getrocknet ist.
- Tragen Sie mit einer Mikropipette warme Agaroselösung um den Glasrand herum auf. Da zu viel Agarose die Fluoreszenzsignale verringern kann, entfernen Sie die überschüssige Lösung unter dem Glas, indem Sie das Glas vorsichtig von oben drücken.
- Entfernen Sie die Agarose auf dem Glas oder entfernt vom Glasrand mit einer Pinzette. Lassen Sie Agarose nur um den äußeren Glasumfang.
- Warten Sie, bis die Agarose fest wird. Wenn durch die Agaroseschrumpfung ein Raum unter dem Glas entsteht, fügen Sie Agaroselösung von der Seite hinzu, um den gesamten Glasrand zu bedecken. Entfernen Sie jegliche Flüssigkeit mit einem Vliesstoff von der Schädeloberfläche.
- Mischen Sie Acrylharzpulver und Flüssigkeit in einem Gummibehälter. Aspirieren Sie die Mischung mit einer Mikropipette und gießen Sie sie auf, um die Agarose, die den Glasrand umgibt, mit Harz zu bedecken.
HINWEIS: Da sich das Harz kurz nach dem Mischen von Pulver und Flüssigkeit verfestigt, müssen sie vor dem Auftragen wiederholt gemischt werden. Die Mengen, die bei jedem Mischen benötigt werden, betragen ~500 μl für Flüssigkeit und ~0,15 g für Pulver. - Befestigen Sie einen Titanstab mit Harz auf der kontralateralen Hemisphäre. Halten Sie den Stangenwinkel parallel zum Deckglas. Fixiere die gesamte Schädeloberfläche mit Harz.
- Die analgetische Lösung mit 10 μl/g Gewicht (Buprenorphin mit 0,1 μg/g Gewicht) unter die Haut im Nacken verabreichen. Legen Sie den Welpen zur Wiederherstellung der Anästhesie wieder auf ein 37 °C heißes Heizkissen. Warten Sie >60 Minuten, bis sich das Harz verfestigt hat, bevor Sie mit der Bildgebung beginnen.
HINWEIS: Pause für 1-5 Stunden vor der Aufnahme. Führen Sie während dieser Zeit auf ähnliche Weise Operationen an anderen Welpen durch.
3. Zwei-Photonen-Kalzium-Bildgebung
- Befestigen Sie ein zweiachsiges Goniometer mit einer Titanplatte an einer Tischplatte mit XY-Positionierung unter dem Mikroskop. Stellen Sie ein Heizkissen (35 °C) auf die Bühne.
- Schalten Sie die Scan-Software unter den folgenden Bedingungen ein: Pixel, 512 x 512; Bidirektional, EIN; Mittelwertbildung, keine; Bildgebungsbereich, 600 x 600 μm mit einem 20-fach-Objektiv. Stellen Sie die Einstellungen so ein, dass die Scanrate schneller als 1 Hz ist.
- Lege den Welpen auf das Heizkissen und befestige den Titanstab mit Schrauben an der Titanplatte. Halten Sie den Welpen betäubt, indem Sie einen Schlauchanschluss für Isofluran (1,5-2,0% an der Luft) platzieren.
- Stellen Sie den Fensterwinkel horizontal mit dem Goniometer ein. Schalten Sie die Hintergrundbeleuchtung ein, beobachten Sie die Gehirnoberfläche mit einem 5-fach-Objektiv und wählen Sie den Bildbereich durch XY-Positionierung aus.
- Geben Sie Augentropfen auf das Schädelfenster. Schalten Sie das Objektiv auf eine 20-fache Wasserimmersionslinse um. Beobachten Sie die kortikale Oberfläche, um zu bestätigen, dass der Blutfluss auf der Gehirnoberfläche zu sehen ist.
- Schalten Sie die Hintergrundbeleuchtung aus und scannen Sie die Gehirnoberfläche im Ein-Photonen-Modus. Erhöhen Sie die Laserleistung, um die grüne Autofluoreszenz des Glases und der Gehirnoberfläche sichtbar zu machen.
- Senken Sie die Isoflurankonzentration auf 1,0 % bis 1,5 %. Decken Sie das Mikroskop ab, um Lichtlecks zu vermeiden. Schalten Sie die Scansoftware in den Zwei-Photonen-Modus.
- Passen Sie die Laserleistung und die Detektorverstärkung für das Scannen von GCaMP- und RFP-Signalen an. Finden Sie die Tiefe, in der TCA-RFP-Signale zu sehen sind. Stellen Sie sicher, dass die Tiefe Schicht 4 ist, ~300 μm tiefer als die Gehirnoberfläche bei P6. Wählen Sie den Bildgebungsbereich aus, in dem viele GCaMP-exprimierende Neuronen zu sehen sind.
- Starten Sie die Zeitraffer-Bildgebung von GCaMP- und RFP-Signalen. Wenn das gleichzeitige Scannen über zwei Kanäle nicht anwendbar ist, erfassen Sie GCaMP- und TCA-RFP-Bilder vor der Bildgebung.
- Stoppen Sie Isofluran, um die Anästhesie zu schwächen, und beobachten Sie spontane Aktivitäten für ~20 Minuten. Überwachen Sie die Bewegungen des Welpen während der Bildgebung mit einer Infrarotkamera. Setzen Sie die Isofluran-Anästhesie (2 % in der Luft) sofort fort, wenn eine Reaktion auf Stress beobachtet wird.
- Nachdem sich der Welpe nicht mehr bewegt, wiederholen Sie die Bildgebung aus Schritt 3.9. Ändern Sie bei Bedarf den Bildgebungsbereich.
- Euthanasiere den Welpen mit einer Überdosis Isofluran. Fixieren Sie das Gehirn durch transkardiale Perfusion von Kochsalzlösung und 4 % PFA, gefolgt von einer Nachfixierung in 4 % PFA über Nacht, um tangentiale Schnitte vorzubereiten und Immunhistochemie durchzuführen. Andernfalls schläfern Sie den Welpen mit einer Überdosis Betäubungsmittel ein, gefolgt von einer Enthauptung.
- Wenn das Muttertier keine weiteren Jungen hat, schläfern Sie die Muttermaus mit einer Überdosis Betäubungsmittel ein, gefolgt von einer Gebärmutterhalsluxation.
4. Würdigung
- Starten Sie MATLAB und führen Sie EZcalcium toolbox15 aus, um eine grafische Benutzeroberfläche (GUI) "Initial GUI" zu öffnen.
- Kompensieren Sie die Drifts des Bildrahmens aufgrund von Mausbewegungen oder anderen Ursachen.
- Klicken Sie in der ersten GUI auf Bewegungskorrektur , um die Bewegungskorrektur-GUI zu öffnen. Klicken Sie auf "Dateien hinzufügen...", um eine TIF-Datei mit den Bilddaten zu laden.
- Stellen Sie die Einstellungen wie folgt ein: Nicht starre Bewegungskorrektur, leer; Upsampling-Faktor, 50; Maximale Schicht, 15; Anfängliche Losgröße, 200; Behälterbreite, 200. Klicken Sie auf "Bewegungskorrektur ausführen", um die Korrektur auszuführen. Bewegungskorrigierte Bilddaten werden automatisch gespeichert.
HINWEIS: Die Einstellungen sollten an die Eigenschaften der Bilddaten angepasst werden. Wenn die Drifts einiger Frames aufgrund von nichtlinearen Frame-Verzerrungen oder Bewegungen des Kortex in Tiefenrichtung nicht kompensiert werden, öffnen Sie die ursprünglichen Bilddaten ohne Korrektur in ImageJ Fiji, entfernen Sie die Frames und starten Sie Schritt 4.2 neu.
- Erkennen Sie Neuronen und zeichnen Sie Regionen von Interesse (ROIs).
- Klicken Sie in der ersten GUI auf Automatisierte ROI-Erkennung , um die GUI für die ROI-Erkennung zu öffnen. Klicken Sie auf Dateien hinzufügen... , um die bewegungskorrigierten Bilddaten zu laden.
- Legen Sie die Einstellungen wie folgt fest: Initialisierung, Gierig; Suchmethode, Ellipse; Dekonvolution, eingeschränktes FOOPSI-SPGL1; Autoregression, Zerfall; Geschätzte # der ROIs, 60 (mehr als das Doppelte der visuell erkannten Anzahl wird empfohlen); Geschätzter ROI für die Breite, 17 (~20 μm); Merge-Schwellenwert, 0,95; Fudge-Faktor, 0,95; Räumliches Downsampling, 1; Temp. Downsampling, 1; Temporäre Iterationen, 5.
HINWEIS: Die Einstellungen sollten an die Eigenschaften der Bilddaten angepasst werden. - Klicken Sie auf ROI-Erkennung ausführen , um die Erkennung auszuführen. ROI-Daten werden automatisch gespeichert.
- Klicken Sie in der ersten GUI auf ROI-Verfeinerung , um die ROI-Verfeinerungs-GUI zu öffnen. Klicken Sie auf Daten laden , um die ROI-Daten zu laden. Wählen Sie die ROIs aus, die eine niedrige Aktivitätsfrequenz (<1 Hz) aufwiesen, sich unter dem Schädel befanden oder die Neuriten anderer Neuronen enthielten. Klicken Sie auf ROI ausschließen , um die ROIs aus der Analyse auszuschließen.
- Wählen Sie das Datenexportformat in XLSX aus und klicken Sie auf Daten exportieren , um eine Excel-Datei mit dF/F-Rohwerten zu erhalten. dF/F verwendet Gleichung (1), wobei F die durchschnittliche Intensität der Pixel in jedem Frame, F0 die Signalintensität der Grundlinie und Fb die Hintergrundfluoreszenz ist.
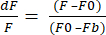 (1)
(1)
- Berechnen Sie den Pearson-Korrelationskoeffizienten von dF/F zwischen ROIs und erstellen Sie eine Korrelationskoeffizientenmatrix. Verwenden Sie dF/F erst, nachdem die Anästhesie nachgelassen hat und spontane Aktivität begonnen hat (~10 Minuten nach Absetzen von Isofluran).
- Definieren Sie die Zylinderkanten aus dem TCA-RFP-Bild mit Fiji. Kategorisieren Sie die ROIs nach ihren jeweiligen Fässern oder Septen. Vergleichen Sie die paarweise Korrelation innerhalb desselben Laufs mit der zwischen verschiedenen Läufen.
- Generieren Sie 1.000 bis 10.000 Surrogatdaten, indem Sie die Korrespondenz zwischen ROI- und Ca2+ -Traces nach dem Zufallsprinzip mischen. Berechnen Sie den durchschnittlichen Korrelationskoeffizienten innerhalb einzelner Fässer in jedem Surrogatdaten und bewerten Sie die statistische Signifikanz der Korrelation in den realen Daten.
HINWEIS: Wenn eine von 10.000 Surrogaten einen Wert hat, der höher ist als der reale Wert, beträgt die statistische Signifikanz 0,0001. Die in den Schritten 4.5 und 4.6 beschriebenen Analysen können für gepoolte Daten von mehreren Tieren durchgeführt werden, wie sie an anderer Stelle 7,16 durchgeführt wurden.
Ergebnisse
Abbildung 1 zeigt die repräsentativen Ergebnisse der Neuronenaktivitäten der Schicht 4 im Fasskortex eines P6-Welpen, die mit dem vorliegenden Protokoll visualisiert wurden. Zwei-Photonen-Bilder des grünen Kanals (GCaMP) und des roten Kanals (TCA-RFP) wurden zeitlich gemittelt und in Abbildung 1A dargestellt. Da die TCA-RFP-Fluoreszenz viel schwächer war als die GCaMP-Fluoreszenz, sickerte das GCaMP-Signal in den roten Kanal...
Diskussion
Angesichts der Tatsache, dass die spontanen Aktivitäten aus dem Sinnesorgan oder dem unteren Nervensystem hervorgehen und über einen Weg, der dem eines reifen Nervensystems3 entspricht, zum primären sensorischen Bereich gelangen, ist es entscheidend, den primären sensorischen Bereich und die Position der abgebildeten Neuronen innerhalb des Bereichs zu definieren. In diesem Protokoll haben wir diese Anforderung erfüllt, indem wir transgene Mäuse eingesetzt ha...
Offenlegungen
Die Autoren haben keine konkurrierenden finanziellen Interessen zu deklarieren.
Danksagungen
Diese Arbeit wurde unterstützt von der Japan Society for the Promotion of Science Grants-in-Aid for Transformative Research Areas (B) (22H05092, 22H05094) und für Scientific Research Grants 20K06876, AMED unter der Fördernummer 21wm0525015, der Takeda Science Foundation, der Naito Foundation, der Kato Memorial Bioscience Foundation, der Kowa Life Science Foundation, NIG-JOINT (24A2021) (an H.M.); und der Japan Society for the Promotion of Science Grants-in-Aid für wissenschaftliche Forschungsstipendien 19K06887 und 22K06446, dem Kodama Memorial Fund for Medical Research, der Uehara Memorial Foundation, der Kato Memorial Bioscience Foundation und der Takeda Science Foundation (an N.N-T). Wir danken Dr. Takuji Iwasato für die TCA-RFP-Mäuse.
Materialien
| Name | Company | Catalog Number | Comments |
| 20× objective lens (water immersion) | |||
| 250 mL Vacuum Filter/Storage Bottle System | Corning | 431096 | |
| 4%-paraformaldehyde phosphate buffer solution (4% PFA) | Nacalai | 09154-85 | |
| Acrylic resin (UNIFAST II) | GC | N/A | |
| Agarose | Sigma | A9793 | |
| Aspirator tube assembly | Drummond | 2-040-000 | |
| CaCl2•2H2O | Nacalai | 06731-05 | |
| Electroporator | BEX | GEB14 | |
| Eye drop (Scopisol) | Senju Pharmaceutical | N/A | |
| Fluorescence stereo microscope | Leica | M165FC | |
| Glucose | Nacalai | 16806-25 | |
| Heating pad | Muromachi Kikai | FHC-HPS | |
| HEPES | Gibco | 15630-080 | |
| Isoflurane | Pfizer | N/A | |
| KCl | Nacalai | 28514-75 | |
| MgSO4•7H2O | Wako | 131-00405 | |
| Micropipette puller | Narishige | PC-100 | |
| Multiphoton laser | Spectra-Physics | Mai Tai eHP DeepSee | |
| Multiphoton microscope | Zeiss | LSM 7MP | |
| NaCl | Nacalai | 31320-05 | |
| Non-woven fabric (Kimwipe) | Kimberly Clark | S-200 | |
| Phosphate buffered saline (PBS) | Nacalai | 27575-31 | |
| Plasmid: CAG-loxP-STOP-loxP-GCaMP6s-ires-tTA-WPRE | Addgene | pK175 | |
| Plasmid: TRE-nCre | Addgene | pK031 | |
| Precision calibrated micropipets | Drummond | 2-000-050 | |
| Razor blade | Feather | FA-10 | |
| Rimadyl (50 mg/mL Carprofen) | Zoetis JP | N/A | |
| Round cover glass, 3-mm-diameter | Matsunami | CS01078 | |
| Saline | Otsuka | 035175315 | |
| Sodium pentobarbital | Nacalai | 26427-72 | |
| Stage for imaging living pup (two single-axis translation stage for XY positioning, two-axis goniometer, base plate, adjustable pillar for z positioning) | ThorLabs | LT1/M, GN2/M, BM2060/M, MLP01/M | |
| TCA-RFP mouse | N/A | N/A | Mizuno et al., 2018a |
| Tissue adhesive (Vetbond) | 3M | 1469SB | |
| Titanium bar | Endo Scientific Instrument | N/A | Custom made (Mizuno et al., 2018b) |
| Titanium bar fixing plate | N/A | Custom made (Mizuno et al., 2018b) | |
| Trypan blue | Sigma | T8154 | |
| Tweezers with platinum plate electrode, 5 mm diameter | BEX | CUY650P5 | |
| Wild-type ICR mouse | Nihon SLC | Slc:ICR |
Referenzen
- Rao, M. S., Mizuno, H. Elucidating mechanisms of neuronal circuit formation in layer 4 of the somatosensory cortex via intravital imaging. Neuroscience Research. 167, 47-53 (2021).
- Iwasato, T., Erzurumlu, R. S. Development of tactile sensory circuits in the CNS. Current Opinion in Neurobiology. 53, 66-75 (2018).
- Martini, F. J., Guillamón-Vivancos, T., Moreno-Juan, V., Valdeolmillos, M., López-Bendito, G. Spontaneous activity in developing thalamic and cortical sensory networks. Neuron. 109 (16), 2519-2534 (2021).
- Ackman, J. B., Burbridge, T. J., Crair, M. C. Retinal waves coordinate patterned activity throughout the developing visual system. Nature. 490 (7419), 219-225 (2012).
- Tritsch, N. X., Yi, E., Gale, J. E., Glowatzki, E., Bergles, D. E. The origin of spontaneous activity in the developing auditory system. Nature. 450 (7166), 50-55 (2007).
- Babola, T. A., et al. Homeostatic control of spontaneous activity in the developing auditory system. Neuron. 99 (3), 511-524.e5 (2018).
- Mizuno, H., et al. Patchwork-type spontaneous activity in neonatal barrel cortex layer 4 transmitted via thalamocortical projections. Cell Reports. 22 (1), 123-135 (2018).
- Mizuno, H., et al. NMDAR-regulated dynamics of layer 4 neuronal dendrites during thalamocortical reorganization in neonates. Neuron. 82 (2), 365-379 (2014).
- Tabata, H., Nakajima, K. Efficient in utero gene transfer system to the developing mouse brain using electroporation: visualization of neuronal migration in the developing cortex. Neuroscience. 103 (4), 865-872 (2001).
- Fukuchi-Shimogori, T., Grove, E. A. Neocortex patterning by the secreted signaling molecule FGF8. Science. 294 (5544), 1071-1074 (2001).
- Saito, T., Nakatsuji, N. Efficient gene transfer into the embryonic mouse brain using in vivo electroporation. Developmental Biology. 240 (1), 237-246 (2001).
- Holtmaat, A., et al. Long-term, high-resolution imaging in the mouse neocortex through a chronic cranial window. Nature Protocols. 4 (8), 1128-1144 (2009).
- Mizuno, H., Nakazawa, S., Iwasato, T. In vivo two-photon imaging of cortical neurons in neonatal mice. Journal of Visualized Experiments. 140, e58340 (2018).
- Egashira, T., et al. In vivo two-photon calcium imaging of cortical neurons in neonatal mice. STAR Protocols. 4 (2), 102245 (2023).
- Cantu, D. A., et al. EZcalcium: Open-source toolbox for analysis of calcium imaging data. Frontiers in Neural Circuits. 14, 25 (2020).
- Maruoka, H., et al. Lattice system of functionally distinct cell types in the neocortex. Science. 358 (6363), 610-615 (2017).
- Antón-Bolaños, N., et al. Prenatal activity from thalamic neurons governs the emergence of functional cortical maps in mice. Science. 364 (6444), 987-990 (2019).
- Guillamón-Vivancos, T., et al. Input-dependent segregation of visual and somatosensory circuits in the mouse superior colliculus. Science. 377 (6608), 845-850 (2022).
- Cardin, J. A., Crair, M. C., Higley, M. J. Mesoscopic imaging: Shining a wide light on large-scale neural dynamics. Neuron. 108 (1), 33-43 (2020).
- Chen, J. L., Voigt, F. F., Javadzadeh, M., Krueppel, R., Helmchen, F. Long-range population dynamics of anatomically defined neocortical networks. eLife. 5, e14679 (2016).
- Ota, K., et al. cell-resolution, contiguous-wide two-photon imaging to reveal functional network architectures across multi-modal cortical areas. Neuron. 109 (11), 1810-1824 (2021).
- Zariwala, H. A., et al. A Cre-dependent GCaMP3 reporter mouse for neuronal imaging in vivo. The Journal of Neuroscience. 32 (9), 3131-3141 (2012).
- Murakami, T., Matsui, T., Uemura, M., Ohki, K. Modular strategy for development of the hierarchical visual network in mice. Nature. 608 (7923), 578-585 (2022).
- Pnevmatikakis, E. A., et al. Simultaneous denoising, deconvolution, and demixing of Calcium imaging data. Neuron. 89 (2), 285-299 (2016).
- Shemesh, O. A., et al. Precision calcium imaging of dense neural populations via a cell-body-targeted calcium indicator. Neuron. 107 (3), 470-486 (2020).
- Giovannucci, A., et al. CaImAn an open source tool for scalable calcium imaging data analysis. Elife. 8, e38173 (2019).
- Pachitariu, M., et al. Suite2p: beyond 10,000 neurons with standard two-photon microscopy. BioRxiv. , (2017).
- Sitdikova, G., et al. Isoflurane suppresses early cortical activity. Annals of Clinical and Translational Neurology. 1 (1), 15-26 (2014).
- Marques-Smith, A., et al. A Transient translaminar GABAergic interneuron circuit connects thalamocortical recipient layers in neonatal somatosensory cortex. Neuron. 89 (3), 536-549 (2016).
- Tuncdemir, S. N., et al. Early somatostatin interneuron connectivity mediates the maturation of deep layer cortical circuits. Neuron. 89 (3), 521-535 (2016).
- Nakazawa, S., Yoshimura, Y., Takagi, M., Mizuno, H., Iwasato, T. Developmental phase transitions in spatial organization of spontaneous activity in postnatal barrel cortex layer 4. The Journal of Neuroscience. 40 (40), 7637-7650 (2020).
- Yu, Y. -. C., et al. Preferential electrical coupling regulates neocortical lineage-dependent microcircuit assembly. Nature. 486 (7401), 113-117 (2012).
- Siegel, F., Heimel, J. A., Peters, J., Lohmann, C. Peripheral and central inputs shape network dynamics in the developing visual cortex in vivo. Current Biology. 22 (3), 253-258 (2012).
- Nakagawa, N., Hosoya, T. Slow dynamics in microcolumnar gap junction network of developing neocortical pyramidal neurons. Neuroscience. 406, 554-554 (2019).
- Valiullina, F., et al. Developmental changes in electrophysiological properties and a transition from electrical to chemical coupling between excitatory layer 4 neurons in the rat barrel cortex. Frontiers in Neural Circuits. 10, 1 (2016).
- Avitan, L., et al. Spontaneous and evoked activity patterns diverge over development. Elife. 10, e61942 (2021).
- Mölter, J., Avitan, L., Goodhill, G. J. Detecting neural assemblies in calcium imaging data. BMC Biology. 16 (1), 143 (2018).
- Nakazawa, S., Mizuno, H., Iwasato, T. Differential dynamics of cortical neuron dendritic trees revealed by long-term in vivo imaging in neonates. Nature Communications. 9 (1), 3106 (2018).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
ISSN 1940-087X
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten
Wir verwenden Cookies, um Ihre Erfahrung auf unserer Website zu verbessern.
Indem Sie unsere Website weiterhin nutzen oder auf „Weiter“ klicken, stimmen Sie zu, unsere Cookies zu akzeptieren.