È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Visualizzazione in vivo dell'attività spontanea nella corteccia sensoriale del topo neonatale con risoluzione di un singolo neurone
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Le aree sensoriali primarie nella neocorteccia mostrano attività spontanee uniche durante lo sviluppo. Questo articolo descrive come visualizzare le attività dei singoli neuroni e le aree sensoriali primarie per analizzare le attività sincrone area-specifiche nei topi neonatali in vivo.
Abstract
Il cervello dei mammiferi subisce cambiamenti dinamici dello sviluppo sia a livello cellulare che circuitale durante i periodi prenatale e postnatale. A seguito della scoperta di numerosi geni che contribuiscono a questi cambiamenti dello sviluppo, è ora noto che anche l'attività neuronale modula sostanzialmente questi processi. Nella corteccia cerebrale in via di sviluppo, i neuroni mostrano modelli di attività sincronizzati che sono specializzati per ciascuna area sensoriale primaria. Questi modelli differiscono notevolmente da quelli osservati nella corteccia matura, sottolineando il loro ruolo nella regolazione dei processi di sviluppo area-specifici. Le carenze nell'attività neuronale durante lo sviluppo possono portare a varie malattie cerebrali. Questi risultati evidenziano la necessità di esaminare i meccanismi regolatori alla base dei modelli di attività nello sviluppo neuronale. Questo articolo riassume una serie di protocolli per visualizzare le aree sensoriali primarie e l'attività neuronale nei topi neonati, per visualizzare l'attività dei singoli neuroni all'interno dei sottocampi corticali utilizzando la microscopia a due fotoni in vivo e per analizzare le correlazioni dell'attività correlata al sottocampo. Mostriamo risultati rappresentativi di un'attività sincrona simile a un patchwork all'interno di singoli barili nella corteccia somatosensoriale. Discutiamo anche varie potenziali applicazioni e alcune limitazioni di questo protocollo.
Introduzione
La corteccia cerebrale contiene diverse aree sensoriali con funzioni distinte. Le aree ricevono input provenienti dai loro organi sensoriali corrispondenti, per lo più convogliati attraverso il midollo spinale o il tronco encefalico e trasmessi attraverso il talamo 1,2. In particolare, i neuroni in ciascuna area sensoriale primaria mostrano un'attività sincronizzata in modo univoco durante le prime fasi di sviluppo, che originano anche dagli organi sensoriali o dai centri nervosi inferiori, ma differiscono essenzialmente dalle attività osservate nella corteccia matura3.
Nei roditori neonatali, ad esempio, l'area visiva primaria (V1) mostra un'attività ondulatoria, che ha origine nella retina (onda retinica) e si propaga attraverso l'intera via visiva conservando la retinotopia4. L'area uditiva primaria (A1) mostra un'attività sincrona organizzata in sottoregioni a forma di banda che corrispondono alle bande di isofrequenza nel cervello maturo. L'attività emana dalle cellule ciliate interne della coclea 5,6. La corteccia del barilotto nell'area somatosensoriale primaria (S1) mostra un modello di attività simile a un patchwork in cui i neuroni dello strato 4 all'interno dei singoli barili, vale a dire i neuroni sensibili ai singoli baffi, sono attivati in modo sincrono7. Sebbene si proponga che abbia origine dal ganglio del trigemino, la fonte dell'attività rimane sconosciuta7. Di conseguenza, i modelli di attività neonatale sono specializzati sia all'interno di ciascuna area sensoriale primaria che all'interno di sottocampi intra-areali. La visualizzazione simultanea dell'attività neuronale e della struttura delle aree sensoriali primarie può facilitare un'indagine sul contributo di questi modelli di attività allo sviluppo dei sistemi sensoriali.
In questo articolo, abbiamo riassunto una serie di protocolli: (1) visualizzare le singole attività neuronali utilizzando la marcatura sparsa di GCaMP e aree sensoriali primarie utilizzando topi TCA-RFP che esprimono la proteina fluorescente rossa negli assoni talamocorticali7, (2) visualizzare l'attività a livello di singola cellula in topi neonatali utilizzando la microscopia a due fotoni in vivo, e (3) analizzare le correlazioni di attività all'interno della corteccia cilindrica S1. I risultati rappresentativi mostrano un'attività sincronizzata simile a un patchwork all'interno dei singoli barili di un topo (P)6 del giorno postnatale. Nonostante alcune limitazioni, questa tecnica può essere utilizzata per l'imaging cronico, l'imaging ad ampio campo in più aree sensoriali e vari esperimenti di manipolazione. L'analisi multiforme dell'attività neuronale durante lo sviluppo arricchirà la nostra comprensione dei meccanismi di formazione dei circuiti cerebrali.
Access restricted. Please log in or start a trial to view this content.
Protocollo
Tutti gli esperimenti sono stati condotti in conformità con le linee guida per la sperimentazione animale dell'Università di Kumamoto e dell'Istituto Nazionale di Genetica e approvati dai comitati per la sperimentazione animale.
1. Elettroporazione in utero (IUE)
- Accoppiare topi maschi TCA-RFP di background ICR con topi ICR femmina wild-type. Osservare il tappo vaginale per verificare l'accoppiamento la mattina presto del giorno successivo. Osservare l'addome per verificare la gravidanza 2 settimane dopo.
- Preparare una soluzione plasmidica contenente 5 ng/μL di TRE-nCre, 1 μg/μL di CAG-loxP-stop-loxP-GCaMP6s-ires-tTA-WPRE e 0,02 % di Trypan Blue in ddH2O per marcare scarsamente i neuroni mediante GCaMP utilizzando il sistema Supernova8.
- Preparare una soluzione analgesica contenente 0,5 mg/mL di carprofene e 0,01 mg/mL di buprenorfina. Proteggerlo dalla luce e tenerlo a temperatura ambiente.
- Al giorno embrionale (E)14, eseguire l'IUE come segue. I metodi IUE sono essenzialmente gli stessi dei precedenti rapporti 9,10,11.
- Pulire il banco da laboratorio con una soluzione di etanolo al 70% o glutaraldeide. Sterilizzare tutti gli strumenti chirurgici in autoclave. Indossare una maschera e un camice da laboratorio per ridurre il rischio di infezione per il topo.
- Preparare le micropipette di vetro utilizzando un estrattore per micropipette. Prelevare la soluzione della miscela plasmidica nella micropipetta di vetro utilizzando un tubo aspiratore.
- Indurre l'anestesia alle tope gravide all'E14 utilizzando isoflurano (2,0% nell'aria). Mantenere i topi anestetizzati durante la procedura con isoflurano (1,2% nell'aria). Pizzica la punta del piede per assicurarti che l'anestesia sia abbastanza profonda. Usa un unguento oftalmico per prevenire la secchezza oculare.
- Disinfettare la zona addominale almeno tre volte con cicli alternati di scrub a base di iodio ed etanolo al 70%. Posizionare un telo sterile per coprire l'area chirurgica. Praticare un'incisione sulla linea mediana ed esporre l'utero sul telo.
- Ripetere il gocciolamento di soluzione salina calda sull'utero per evitare che si secchi o si raffreddi fino alla chiusura dell'addome (Passaggio 1.4.8).
- Iniettare la soluzione plasmidica in un lato del ventricolo laterale degli embrioni uno per uno utilizzando una micropipetta e un tubo aspiratore.
- Utilizzare un elettrodo a pinza per pizzicare le teste dell'embrione e fornire impulsi elettrici quadrati (40 V, 50 ms) cinque volte a intervalli di 1 s utilizzando un elettroporatore.
- Riportare l'utero nella cavità addominale. Applicare ~3 ml di soluzione salina calda nella cavità. Sutura la membrana peritoneale e la pelle addominale. Somministrare la soluzione analgesica a 10 μL/g di peso (carprofene a 5 μg/g di peso e buprenorfina a 0,1 μg/g di peso) sotto la pelle nella parte posteriore del collo.
- Posizionare il mouse in una gabbia di recupero su un termoforo (37 °C) per riprendersi dall'anestesia. Appoggia il topo sulla pancia per evitare che la lingua e la saliva soffochino la gola. Tieni il mouse lontano dagli altri mouse e monitoralo fino a quando non si riprende a muoversi normalmente. Rimetti il mouse nella gabbia.
- Dopo la nascita, eseguire la genotipizzazione per verificare la presenza di allele RFP e sopprimere i cuccioli senza l'allele. Questo passaggio è consigliato, soprattutto se l'imaging viene eseguito durante la seconda settimana postnatale, quando il segnale TCA-RFP è debole e difficile da controllare al passaggio 2.2.7.
2. Chirurgia della finestra cranica
- Preparare il tampone cortex contenente 125 mM di NaCl, 5 mM di KCl, 10 mM di glucosio, 10 mM di HEPES, 2 mM di CaCl2 e 2 mM di MgSO4 in ddH2O (pH regolato a 7,4 con 1 M di NaOH)12 prima del giorno dell'intervento. Sterilizzare il tampone utilizzando un filtro sottovuoto.
NOTA: Il tampone può essere conservato a 4 °C per un massimo di 3 mesi. Il volume richiesto è di 5-10 ml per cucciolo. - Eseguire i seguenti passaggi in P3-12. Si vedano anche i rapporti precedenti che hanno descritto questa procedura13,14.
- Mescolare 50 mg di agarosio con 5 ml di tampone cortex e sciogliere completamente l'agarosio riscaldandolo. Assumere una parte della soluzione in una provetta da 1,5 mL e mantenerla a 42 °C.
- Prendi un po' di tampone cortex in una provetta conica da 50 ml e tienila a temperatura ambiente. Prelevare un po' di soluzione fisiologica in un contenitore (ad esempio, un tappo di una provetta conica da 50 ml) e conservarlo a temperatura ambiente.
- Preparare una soluzione analgesica contenente 0,01 mg/mL di buprenorfina. Proteggerlo dalla luce e tenerlo a temperatura ambiente.
- Sterilizzare tutti gli strumenti chirurgici in autoclave. Disinfettare lo stereomicroscopio a fluorescenza con etanolo al 70%.
- Usa un vaporizzatore di isoflurano per indurre l'anestesia nel cucciolo con isoflurano (2,0% nell'aria). Presta attenzione al cucciolo e tiralo fuori quando il suo respiro rallenta leggermente.
NOTA: Se l'anestesia è prolungata, la respirazione del cucciolo può cessare per alcuni secondi. Anche se la respirazione del cucciolo viene ripresa, l'anestesia prolungata può ridurre il flusso sanguigno cerebrale del cucciolo e causare danni cerebrali irreversibili. - Disinfettare la testa del cucciolo almeno tre volte con cicli alternati di scrub a base di iodio ed etanolo al 70%. Posizionare il cucciolo sul termoforo (35 °C) sotto uno stereomicroscopio a fluorescenza. Tenere sempre il cucciolo anestetizzato con isoflurano (1,5%-2,5% nell'aria).
- Seleziona i cuccioli che esprimono TCA-RFP e GCaMP osservando la fluorescenza attraverso il cranio. Eutanasia i cuccioli che non esprimono entrambi.
- Rimuovere il cuoio capelluto sopra gli emisferi cerebrali il più ampiamente possibile con attenzione per non causare sanguinamento. Strofina il cranio con un batuffolo di cotone sterile imbevuto di soluzione salina per rimuovere il tessuto connettivo.
- Dopo che il cranio si è asciugato, far aderire la superficie incisa del cuoio capelluto al cranio con un adesivo per tessuti.
- Trasferisci il cucciolo su un termoforo a 37 °C per riprenderlo dall'anestesia. Attendere almeno 15 minuti per far solidificare l'adesivo.
NOTA: Mettere in pausa per un massimo di 1 ora prima di passare al passaggio successivo. Prepara gli altri cuccioli in modo simile, se necessario, durante questo periodo. - Anestetizzare il cucciolo con isoflurano. Posizionare il cucciolo sul termoforo (37 °C) sotto uno stereomicroscopio a fluorescenza mentre è anestetizzato (1,5%-2,5% di isoflurano nell'aria).
NOTA: Se l'anestesia si prolunga più del tempo per il punto finale (60 minuti), sopprimere il cucciolo mediante decapitazione durante l'anestetizzazione con isoflurano. - Contrassegnare la posizione che esprime GCaMP sul cranio con un pennarello indelebile sterile. Applicare il tampone cortex sulla posizione.
- Inserisci l'angolo di una lama di rasoio nel cranio. Spingi lentamente la lama per radere il cranio e fare un buco. Pizzica il cranio rotto con una pinzetta e rimuovilo.
- Verificare che il foro cranico sia stato praticato correttamente osservando i vasi sanguigni nel foro. In caso di sanguinamento, sciacquare rapidamente il foro con il tampone cortex utilizzando una micropipetta. Ripetere il risciacquo fino a quando l'emorragia non si ferma completamente.
- Applicare una goccia di tampone cortex sul foro cranico e posizionarlo in un bicchiere di copertura rotondo sterile di 3 mm di diametro sopra il foro. Asciugare il tampone in eccesso con tessuto non tessuto. Attendere che l'area intorno al vetro si asciughi.
- Applicare una soluzione calda di agarosio attorno al bordo del vetro utilizzando una micropipetta. Poiché una quantità eccessiva di agarosio può ridurre i segnali fluorescenti, rimuovere la soluzione in eccesso sotto il vetro spingendo delicatamente il vetro dall'alto.
- Rimuovere l'agarosio sul vetro o lontano dal bordo del bicchiere utilizzando una pinzetta. Lasciare l'agarosio solo attorno al perimetro esterno del vetro.
- Attendere che l'agarosio si solidifichi. Se il restringimento dell'agarosio crea uno spazio sotto il vetro, aggiungere la soluzione di agarosio lateralmente per coprire l'intero bordo del vetro. Rimuovere qualsiasi liquido dalla superficie del cranio con tessuto non tessuto.
- Mescolare la polvere e il liquido di resina acrilica in un contenitore di gomma. Aspirare il composto con una micropipetta e versarlo fino a coprire di resina l'agarosio che circonda il bordo del bicchiere.
NOTA: Poiché la resina si solidifica subito dopo aver miscelato la polvere e il liquido, è necessario miscelarli ripetutamente prima dell'applicazione. Le quantità necessarie in ogni miscelazione sono ~500 μl per il liquido e ~0,15 g per la polvere. - Fissare una barra di titanio con resina sull'emisfero controlaterale. Mantenere l'angolo della barra parallelo al vetro di copertura. Fissare l'intera superficie del cranio con la resina.
- Somministrare la soluzione analgesica a 10 μL/g di peso (buprenorfina a 0,1 μg/g di peso) sotto la pelle nella parte posteriore del collo. Riportare il cucciolo su un termoforo a 37 °C per il recupero dell'anestesia. Attendere >60 minuti per la solidificazione della resina prima di eseguire l'imaging.
NOTA: Mettere in pausa per 1-5 ore prima dell'imaging. Esegui un intervento chirurgico su altri cuccioli in modo simile durante questo periodo.
3. Imaging del calcio a due fotoni
- Collegare un goniometro a due assi con una piastra in titanio a una piastra tavolino con posizionamento XY sotto il microscopio. Installare un termoforo (35 °C) sul tavolino.
- Attivare il software di scansione con le seguenti condizioni: Pixel, 512 x 512; Bidirezionale, ON; Media, nessuna; Area di imaging, 600 x 600 μm con obiettivo 20x. Impostare le impostazioni in modo che la velocità di scansione sia superiore a 1 Hz.
- Posiziona il cucciolo sul termoforo e fissa la barra in titanio montata sulla testa alla piastra in titanio con le viti. Mantieni il cucciolo anestetizzato posizionando una porta del tubo per l'isoflurano (1,5-2,0% nell'aria).
- Regolare l'angolo della finestra orizzontalmente con il goniometro. Accendi la retroilluminazione e osserva la superficie del cervello con una lente dell'obiettivo 5x, quindi seleziona l'area di imaging posizionando XY.
- Metti un collirio sulla finestra cranica. Passa l'obiettivo a una lente a immersione in acqua 20x. Osservare la superficie corticale per confermare che il flusso sanguigno è visibile sulla superficie del cervello.
- Spegni la retroilluminazione e scansiona la superficie del cervello utilizzando la modalità a un fotone. Aumenta la potenza del laser per rendere visibile l'autofluorescenza verde del vetro e della superficie cerebrale.
- Abbassare la concentrazione di isoflurano all'1,0%-1,5%. Coprire il microscopio per evitare perdite di luce. Passare il software di scansione alla modalità a due fotoni.
- Regolare la potenza del laser e il guadagno del rivelatore in modo appropriato per la scansione di segnali GCaMP e RFP. Trova la profondità in cui vengono visti i segnali TCA-RFP. Assicurarsi che la profondità sia di strato 4, ~300 μm inferiore alla superficie cerebrale a P6. Selezionare l'area di imaging in cui sono visibili molti neuroni che esprimono GCaMP.
- Avvia l'imaging timelapse dei segnali GCaMP e RFP. Se la scansione simultanea a due canali non è applicabile, acquisire le immagini GCaMP e TCA-RFP prima dell'imaging.
- Interrompere l'isoflurano per indebolire l'anestesia e osservare le attività spontanee per ~20 minuti. Monitora i movimenti del cucciolo durante l'imaging utilizzando una fotocamera a infrarossi. Riprendere immediatamente l'anestesia con isoflurano (2% nell'aria) se si osserva una risposta che indica sofferenza.
- Dopo che il cucciolo ha smesso di muoversi, ripetere l'imaging dal passaggio 3.9. Se necessario, modificare l'area di imaging.
- Eutanasia del cucciolo con un sovradosaggio di isoflurano. Fissare il cervello mediante perfusione transcardica di soluzione fisiologica e PFA al 4%, seguita da post-fissazione in PFA al 4% durante la notte per preparare fette tangenziali ed eseguire l'immunoistochimica. In caso contrario, sopprimere il cucciolo con un'overdose di anestetico seguita da decapitazione.
- Se la madre non ha altri cuccioli, sopprimere la madre topo con un'overdose di anestetico seguita da lussazione cervicale.
4. Analisi
- Avvia MATLAB ed esegui EZcalcium toolbox15 per aprire un'interfaccia utente grafica (GUI) "Initial GUI".
- Compensa le deviazioni del fotogramma dell'immagine dovute ai movimenti del mouse o ad altre cause.
- Fare clic su Correzione movimento nella GUI iniziale per aprire la GUI di Correzione movimento. Fare clic su "Aggiungi file..." per caricare un file TIF dei dati di imaging.
- Impostare le impostazioni come segue: Correzione movimento non rigida, vuoto; fattore di sovracampionamento, 50; Spostamento massimo, 15; Dimensione iniziale del lotto, 200; Larghezza del contenitore, 200. Fare clic su "Esegui correzione movimento" per eseguire la correzione. I dati di imaging corretti per il movimento verranno salvati automaticamente.
NOTA: Le impostazioni devono essere regolate in base alle proprietà dei dati di imaging. Se le derive di alcuni fotogrammi non vengono compensate a causa di distorsioni non lineari del fotogramma o del movimento della corteccia nella direzione della profondità, aprire i dati di imaging originali senza correzione in ImageJ Fiji e rimuovere i fotogrammi, quindi riavviare il passaggio 4.2.
- Rileva i neuroni e disegna le regioni di interesse (ROI).
- Fare clic su Rilevamento automatico ROI nella GUI iniziale per aprire la GUI di rilevamento ROI. Fare clic su Aggiungi file... per caricare i dati di imaging corretti per il movimento.
- Impostare le impostazioni come segue: Inizializzazione, Avido; Metodo di ricerca, ellisse; Deconvoluzione, FOOPSI-SPGL1 vincolato; Autoregressione, decadimento; # stimato di ROI, 60 (si consiglia più del doppio del numero rilevato visivamente); Larghezza ROI stimata, 17 (~20 μm); Soglia di fusione, 0,95; fattore di caramellismo, 0,95; Downsampling spaziale, 1; Temp. Downsampling, 1; Temp. Iterazioni, 5.
NOTA: Le impostazioni devono essere regolate in base alle proprietà dei dati di imaging. - Fare clic su Esegui rilevamento ROI per eseguire il rilevamento. I dati sul ROI verranno salvati automaticamente.
- Fare clic su Perfezionamento ROI nella GUI iniziale per aprire la GUI di Perfezionamento ROI. Fai clic su Carica dati per caricare i dati sul ROI. Seleziona le ROI che avevano una bassa frequenza di attività (<1 Hz), situate sotto il cranio o che contenevano neuriti di altri neuroni. Fare clic su Escludi ROI per escludere le ROI dall'analisi.
- Selezionare il formato di esportazione dei dati in XLSX e fare clic su Esporta dati per ottenere un file Excel con valori dF/F non elaborati. Il dF/F utilizza l'equazione (1), dove F è l'intensità media dei pixel in ogni fotogramma, F0 è l'intensità del segnale di base e Fb è la fluorescenza di fondo.
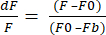 (1)
(1)
- Calcola il coefficiente di correlazione di Pearson di dF/F tra i ROI e crea una matrice del coefficiente di correlazione. Utilizzare dF/F solo dopo che l'anestesia si è indebolita e ha iniziato a verificarsi un'attività spontanea (~10 minuti dopo l'interruzione dell'isoflurano).
- Definisci i bordi del barilotto dall'immagine TCA-RFP utilizzando Fiji. Categorizza le ROI nei rispettivi barili o setti. Confronta la correlazione a coppie all'interno della stessa canna e quella tra canne diverse.
- Genera da 1.000 a 10.000 dati surrogati mescolando in modo casuale la corrispondenza tra le tracce ROI e Ca2+ . Calcola il coefficiente di correlazione medio all'interno dei singoli barili in ogni dato surrogato e valuta la significatività statistica della correlazione nei dati reali.
NOTA: Se uno dei 10.000 surrogati ha un valore superiore al valore reale, la significatività statistica è 0,0001. Le analisi descritte nelle fasi 4.5 e 4.6 possono essere condotte per dati aggregati provenienti da più animali, come eseguito altrove 7,16.
Access restricted. Please log in or start a trial to view this content.
Risultati
La Figura 1 mostra i risultati rappresentativi delle attività neuronali di livello 4 nella corteccia cilindrica di un cucciolo di P6 visualizzati utilizzando il presente protocollo. Le immagini a due fotoni del canale verde (GCaMP) e del canale rosso (TCA-RFP) sono state mediate temporalmente e mostrate nella Figura 1A. Poiché la fluorescenza TCA-RFP era molto più debole della fluorescenza GCaMP, il segnale GCaMP è fuoriusci...
Access restricted. Please log in or start a trial to view this content.
Discussione
Dato che le attività spontanee emergono dall'organo sensoriale o sistema nervoso inferiore e viaggiano verso l'area sensoriale primaria attraverso un percorso equivalente a quello di un sistema nervoso maturo3, è fondamentale definire l'area sensoriale primaria e la posizione dei neuroni ripresi all'interno dell'area. In questo protocollo, abbiamo affrontato questo requisito impiegando topi transgenici che visualizzano gli assoni talamocorticali e il sistema Sup...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno interessi finanziari concorrenti da dichiarare.
Riconoscimenti
Questo lavoro è stato sostenuto dalla Japan Society for the Promotion of Science Grants-in-Aid for Transformative Research Areas (B) (22H05092, 22H05094) e per le sovvenzioni per la ricerca scientifica 20K06876, AMED con il numero di sovvenzione 21wm0525015, dalla Takeda Science Foundation, dalla Naito Foundation, dalla Kato Memorial Bioscience Foundation, dalla Kowa Life Science Foundation, NIG-JOINT (24A2021) (a H.M.); e la Japan Society for the Promotion of Science Grants-in-Aid for Scientific Research Grants 19K06887 e 22K06446, il Kodama Memorial Fund for Medical Research, la Uehara Memorial Foundation, la Kato Memorial Bioscience Foundation e la Takeda Science Foundation (a N.N-T.). Ringraziamo il Dr. Takuji Iwasato per i topi TCA-RFP.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| 20× objective lens (water immersion) | |||
| 250 mL Vacuum Filter/Storage Bottle System | Corning | 431096 | |
| 4%-paraformaldehyde phosphate buffer solution (4% PFA) | Nacalai | 09154-85 | |
| Acrylic resin (UNIFAST II) | GC | N/A | |
| Agarose | Sigma | A9793 | |
| Aspirator tube assembly | Drummond | 2-040-000 | |
| CaCl2•2H2O | Nacalai | 06731-05 | |
| Electroporator | BEX | GEB14 | |
| Eye drop (Scopisol) | Senju Pharmaceutical | N/A | |
| Fluorescence stereo microscope | Leica | M165FC | |
| Glucose | Nacalai | 16806-25 | |
| Heating pad | Muromachi Kikai | FHC-HPS | |
| HEPES | Gibco | 15630-080 | |
| Isoflurane | Pfizer | N/A | |
| KCl | Nacalai | 28514-75 | |
| MgSO4•7H2O | Wako | 131-00405 | |
| Micropipette puller | Narishige | PC-100 | |
| Multiphoton laser | Spectra-Physics | Mai Tai eHP DeepSee | |
| Multiphoton microscope | Zeiss | LSM 7MP | |
| NaCl | Nacalai | 31320-05 | |
| Non-woven fabric (Kimwipe) | Kimberly Clark | S-200 | |
| Phosphate buffered saline (PBS) | Nacalai | 27575-31 | |
| Plasmid: CAG-loxP-STOP-loxP-GCaMP6s-ires-tTA-WPRE | Addgene | pK175 | |
| Plasmid: TRE-nCre | Addgene | pK031 | |
| Precision calibrated micropipets | Drummond | 2-000-050 | |
| Razor blade | Feather | FA-10 | |
| Rimadyl (50 mg/mL Carprofen) | Zoetis JP | N/A | |
| Round cover glass, 3-mm-diameter | Matsunami | CS01078 | |
| Saline | Otsuka | 035175315 | |
| Sodium pentobarbital | Nacalai | 26427-72 | |
| Stage for imaging living pup (two single-axis translation stage for XY positioning, two-axis goniometer, base plate, adjustable pillar for z positioning) | ThorLabs | LT1/M, GN2/M, BM2060/M, MLP01/M | |
| TCA-RFP mouse | N/A | N/A | Mizuno et al., 2018a |
| Tissue adhesive (Vetbond) | 3M | 1469SB | |
| Titanium bar | Endo Scientific Instrument | N/A | Custom made (Mizuno et al., 2018b) |
| Titanium bar fixing plate | N/A | Custom made (Mizuno et al., 2018b) | |
| Trypan blue | Sigma | T8154 | |
| Tweezers with platinum plate electrode, 5 mm diameter | BEX | CUY650P5 | |
| Wild-type ICR mouse | Nihon SLC | Slc:ICR |
Riferimenti
- Rao, M. S., Mizuno, H. Elucidating mechanisms of neuronal circuit formation in layer 4 of the somatosensory cortex via intravital imaging. Neuroscience Research. 167, 47-53 (2021).
- Iwasato, T., Erzurumlu, R. S. Development of tactile sensory circuits in the CNS. Current Opinion in Neurobiology. 53, 66-75 (2018).
- Martini, F. J., Guillamón-Vivancos, T., Moreno-Juan, V., Valdeolmillos, M., López-Bendito, G. Spontaneous activity in developing thalamic and cortical sensory networks. Neuron. 109 (16), 2519-2534 (2021).
- Ackman, J. B., Burbridge, T. J., Crair, M. C. Retinal waves coordinate patterned activity throughout the developing visual system. Nature. 490 (7419), 219-225 (2012).
- Tritsch, N. X., Yi, E., Gale, J. E., Glowatzki, E., Bergles, D. E. The origin of spontaneous activity in the developing auditory system. Nature. 450 (7166), 50-55 (2007).
- Babola, T. A., et al. Homeostatic control of spontaneous activity in the developing auditory system. Neuron. 99 (3), 511-524.e5 (2018).
- Mizuno, H., et al. Patchwork-type spontaneous activity in neonatal barrel cortex layer 4 transmitted via thalamocortical projections. Cell Reports. 22 (1), 123-135 (2018).
- Mizuno, H., et al. NMDAR-regulated dynamics of layer 4 neuronal dendrites during thalamocortical reorganization in neonates. Neuron. 82 (2), 365-379 (2014).
- Tabata, H., Nakajima, K. Efficient in utero gene transfer system to the developing mouse brain using electroporation: visualization of neuronal migration in the developing cortex. Neuroscience. 103 (4), 865-872 (2001).
- Fukuchi-Shimogori, T., Grove, E. A. Neocortex patterning by the secreted signaling molecule FGF8. Science. 294 (5544), 1071-1074 (2001).
- Saito, T., Nakatsuji, N. Efficient gene transfer into the embryonic mouse brain using in vivo electroporation. Developmental Biology. 240 (1), 237-246 (2001).
- Holtmaat, A., et al. Long-term, high-resolution imaging in the mouse neocortex through a chronic cranial window. Nature Protocols. 4 (8), 1128-1144 (2009).
- Mizuno, H., Nakazawa, S., Iwasato, T. In vivo two-photon imaging of cortical neurons in neonatal mice. Journal of Visualized Experiments. 140, e58340(2018).
- Egashira, T., et al. In vivo two-photon calcium imaging of cortical neurons in neonatal mice. STAR Protocols. 4 (2), 102245(2023).
- Cantu, D. A., et al. EZcalcium: Open-source toolbox for analysis of calcium imaging data. Frontiers in Neural Circuits. 14, 25(2020).
- Maruoka, H., et al. Lattice system of functionally distinct cell types in the neocortex. Science. 358 (6363), 610-615 (2017).
- Antón-Bolaños, N., et al. Prenatal activity from thalamic neurons governs the emergence of functional cortical maps in mice. Science. 364 (6444), 987-990 (2019).
- Guillamón-Vivancos, T., et al. Input-dependent segregation of visual and somatosensory circuits in the mouse superior colliculus. Science. 377 (6608), 845-850 (2022).
- Cardin, J. A., Crair, M. C., Higley, M. J. Mesoscopic imaging: Shining a wide light on large-scale neural dynamics. Neuron. 108 (1), 33-43 (2020).
- Chen, J. L., Voigt, F. F., Javadzadeh, M., Krueppel, R., Helmchen, F. Long-range population dynamics of anatomically defined neocortical networks. eLife. 5, e14679(2016).
- Ota, K., et al. cell-resolution, contiguous-wide two-photon imaging to reveal functional network architectures across multi-modal cortical areas. Neuron. 109 (11), 1810-1824 (2021).
- Zariwala, H. A., et al. A Cre-dependent GCaMP3 reporter mouse for neuronal imaging in vivo. The Journal of Neuroscience. 32 (9), 3131-3141 (2012).
- Murakami, T., Matsui, T., Uemura, M., Ohki, K. Modular strategy for development of the hierarchical visual network in mice. Nature. 608 (7923), 578-585 (2022).
- Pnevmatikakis, E. A., et al. Simultaneous denoising, deconvolution, and demixing of Calcium imaging data. Neuron. 89 (2), 285-299 (2016).
- Shemesh, O. A., et al. Precision calcium imaging of dense neural populations via a cell-body-targeted calcium indicator. Neuron. 107 (3), 470-486 (2020).
- Giovannucci, A., et al. CaImAn an open source tool for scalable calcium imaging data analysis. Elife. 8, e38173(2019).
- Pachitariu, M., et al. Suite2p: beyond 10,000 neurons with standard two-photon microscopy. BioRxiv. , (2017).
- Sitdikova, G., et al. Isoflurane suppresses early cortical activity. Annals of Clinical and Translational Neurology. 1 (1), 15-26 (2014).
- Marques-Smith, A., et al. A Transient translaminar GABAergic interneuron circuit connects thalamocortical recipient layers in neonatal somatosensory cortex. Neuron. 89 (3), 536-549 (2016).
- Tuncdemir, S. N., et al. Early somatostatin interneuron connectivity mediates the maturation of deep layer cortical circuits. Neuron. 89 (3), 521-535 (2016).
- Nakazawa, S., Yoshimura, Y., Takagi, M., Mizuno, H., Iwasato, T. Developmental phase transitions in spatial organization of spontaneous activity in postnatal barrel cortex layer 4. The Journal of Neuroscience. 40 (40), 7637-7650 (2020).
- Yu, Y. -C., et al. Preferential electrical coupling regulates neocortical lineage-dependent microcircuit assembly. Nature. 486 (7401), 113-117 (2012).
- Siegel, F., Heimel, J. A., Peters, J., Lohmann, C. Peripheral and central inputs shape network dynamics in the developing visual cortex in vivo. Current Biology. 22 (3), 253-258 (2012).
- Nakagawa, N., Hosoya, T. Slow dynamics in microcolumnar gap junction network of developing neocortical pyramidal neurons. Neuroscience. 406, 554-554 (2019).
- Valiullina, F., et al. Developmental changes in electrophysiological properties and a transition from electrical to chemical coupling between excitatory layer 4 neurons in the rat barrel cortex. Frontiers in Neural Circuits. 10, 1(2016).
- Avitan, L., et al. Spontaneous and evoked activity patterns diverge over development. Elife. 10, e61942(2021).
- Mölter, J., Avitan, L., Goodhill, G. J. Detecting neural assemblies in calcium imaging data. BMC Biology. 16 (1), 143(2018).
- Nakazawa, S., Mizuno, H., Iwasato, T. Differential dynamics of cortical neuron dendritic trees revealed by long-term in vivo imaging in neonates. Nature Communications. 9 (1), 3106(2018).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon