Method Article
Organoide des menschlichen Oberflächenepithels der Eierstöcke als Plattform zur Untersuchung der Geweberegeneration
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt die Etablierung dreidimensionaler (3D) Gewebeorganoide aus primären humanen Zellen des Oberflächenepithels der Eierstöcke (hOSE). Das Protokoll umfasst die Isolierung von hOSE aus frisch entnommenen Eierstöcken, die zelluläre Expansion des hOSE, Kryokonservierungs- und Auftauverfahren und die Ableitung von Organoiden. Immunfluoreszenz, quantitative Analyse und die Präsentation des Nutzens als Screening-Plattform sind enthalten.
Zusammenfassung
Das Oberflächenepithel der Eierstöcke (OSE), die äußerste Schicht des Eierstocks, erfährt bei jedem Eisprung einen Riss und spielt eine entscheidende Rolle bei der Wundheilung der Eierstöcke bei der Wiederherstellung der Integrität der Eierstöcke. Darüber hinaus kann der OSE als Quelle für epithelialen Eierstockkrebs dienen. Obwohl die regenerativen Eigenschaften von OSE bei Mäusen gut untersucht wurden, wird das Verständnis des genauen Mechanismus der Gewebereparatur im menschlichen Eierstock durch den begrenzten Zugang zu menschlichen Eierstöcken und geeignete In-vitro-Kulturprotokolle behindert. Gewebespezifische Organoide, miniaturisierte In-vitro-Modelle , die sowohl strukturelle als auch funktionelle Aspekte des ursprünglichen Organs nachbilden, bieten neue Möglichkeiten für die Untersuchung der Organphysiologie, die Modellierung von Krankheiten und die Erprobung von Medikamenten.
Hier beschreiben wir eine Methode, um primäres humanes OSE (hOSE) aus ganzen Eierstöcken zu isolieren und hOSE-Organoide zu etablieren. Wir beziehen eine morphologische und zelluläre Charakterisierung ein, die die Heterogenität zwischen den Spendern zeigt. Darüber hinaus zeigen wir die Fähigkeit dieser Kulturmethode, hormonelle Effekte auf das Wachstum von OSE-Organoiden über einen Zeitraum von 2 Wochen zu bewerten. Diese Methode könnte die Entdeckung von Faktoren ermöglichen, die zur OSE-Regeneration beitragen, und patientenspezifische Wirkstoffscreenings auf maligne OSE erleichtern.
Einleitung
Der Eierstock gilt als eines der dynamischsten Organe des Körpers und durchläuft während der gesamten reproduktiven Lebensspanne des Individuums ständige Zyklen der Wundheilung und des Umbaus. Ein Hauptakteur, der an der Regeneration des Eierstockgewebes nach jedem Ovulationszyklus beteiligt ist, ist das Oberflächenepithel der Eierstöcke (OSE)1. Das OSE ist eine aus Mesothel gewonnene Einzelschicht, die flache, quaderförmige und säulenförmige Epithelzellen enthält, die die gesamte Oberfläche der Eierstöcke bedecken2. Vor dem Eisprung wird das ovarielle Stromagewebe auf der Oberfläche des Ovulationsfollikels proteolytisch gestört, um die Freisetzung des Kumulus-Eizell-Komplexes zu ermöglichen. Der verwundete Bereich, der als ovulatorisches Stigma bekannt ist, wird dann repariert, wobei der vollständige Verschluss der Eierstockoberfläche bei Mäusen in weniger als 72 Stunden erreichtwird 3. Die hocheffiziente Fähigkeit des OSE, die ovulatorische Wunde zu proliferieren und zu schließen, unterstreicht die mutmaßliche Existenz einer residenten Stammzellpopulation4. Aufgrund der begrenzten Verfügbarkeit menschlicher Eierstöcke von Spenderinnen im gebärfähigen Alter stammt der größte Teil des Wissens über die Mechanismen der OSE-Reparatur aus Tiermodellen. Artspezifische Merkmale erschweren jedoch die Translation von der tierischen Eierstockforschung auf den Menschen5.
In vitro-Studien wurde aufgrund der Kosteneffizienz und der einfachen Kultivierung überwiegend 2-dimensionale (2D) Zellkulturen von humanen OSE verwendet, bei denen Zellen in einer Monoschicht wuchsen, die an der Oberfläche einer Kulturplatte befestigt war 6,7,8. Nichtsdestotrotz hat dieser Ansatz Grenzen, die die Komplexität der Dynamik des ovariellen Gewebes reproduzieren9. In diesem Zusammenhang haben 3D-Zellkulturplattformen mit besonderem Fokus auf Eierstockorganoide die Eierstockforschung revolutioniert10. Gewebe-Organoide sind miniaturisierte In-vitro-Darstellungen des Organs, von dem sie abgeleitet sind, die eine 3D-Selbstorganisationsfähigkeit aufweisen und Schlüsselfunktionen und -strukturen ihrer In-vivo-Gegenstücke nachahmen11. Diese Technologie bietet die Möglichkeit, grundlegende Fragen zur Entwicklung, Regeneration und Gewebereparatur im menschlichen Eierstock zu beleuchten10. In den letzten Jahren haben Forscher ihr Wissen über Eierstock-Organoide auch für die Generierung von patientenspezifischen Organoiden für Eierstockkrebs (OC) für die Krankheitsmodellierung und personalisierte Medizin angewendet 12,13,14.
Basierend auf verschiedenen Methoden, die zur Erzeugung von Maus-OSE-Organoiden und Eileiter-Organoiden15,16 sowie humanen OSE-Organoiden12 und FT-Organoiden17 verwendet werden, beschreiben wir hier ein Protokoll zur Gewinnung humaner OSE-Organoide aus humanen Eierstöcken mit potenziellen Anwendungen in OSE-Regenerationsstudien. Dieses Protokoll isoliert effizient primäre OSE-Zellen aus ganzen menschlichen Eierstöcken und enthält eine Schritt-für-Schritt-Beschreibung der 2D-Zellexpansion und der Erzeugung von 3D-hOSE-Organoiden. Die hOSE-Organoide zeigten eine (spenderspezifische) Variabilität in Morphologie und Wachstum, was ihren Nutzen für personalisierte Studien unterstreicht. Darüber hinaus umfasst dieses Protokoll die Erhaltung, Passage und Immunfluoreszenz von hOSE-Organoiden innerhalb derselben Kulturplatte. Darüber hinaus wird eine Beschreibung der unterschiedlichen Morphologien geliefert, die hOSE-Organoide annehmen können, und es werden Veränderungen des Immunphänotyps während der Kultur charakterisiert. Schließlich zeigt es seinen Nutzen, indem es den Einfluss von Umwelteinflüssen, wie z. B. Eierstockhormonen, auf die Bildung und das Wachstum von hOSE-Organoiden basierend auf der Anzahl und Größe der hOSE-Organoide untersucht.
Die Anwendung der hOSE-Organoid-Technologie wird unser Verständnis des Eierstocks verbessern, wobei der Schwerpunkt auf den Mechanismen liegt, die für seine bemerkenswerte Regenerationsfähigkeit verantwortlich sind. Mit der Weiterentwicklung menschlicher 3D-Ovarialmodelle wird die Abhängigkeit von Tiermodellen in der Eierstockforschung abnehmen, was zu innovativen Therapien im Bereich der regenerativen Medizin führenwird 18.
Protokoll
Die Studie wurde nach den Richtlinien der Deklaration von Helsinki durchgeführt. Das Studiendesign wurde bei der Medizinischen Ethikkommission des Leiden University Medical Center (LUMC) eingereicht, und vor der Studie wurde eine Unbedenklichkeitserklärung (B18.029) eingeholt. Primäres menschliches Ovarialgewebe wurde von transmaskulinen Personen entnommen, die sich im VUmc-Krankenhaus (Amsterdam, Niederlande) einer geschlechtsangleichenden Operation unterzogen haben. Von allen Spendern wurde eine unterschriebene Einverständniserklärung eingeholt. Alle Materialien, die in diesem Protokoll verwendet werden, sind in der Materialtabelle aufgeführt.
1. Isolierung von primären OSE-Zellen des Menschen
- Legen Sie die Eierstöcke nach der Oophorektomie in 0,9%iges NaCl oder eine ähnliche sterile Kochsalzlösung und transportieren Sie sie auf Eis ins Labor.
- Übertragen Sie die einzelnen Eierstöcke in ein konisches 50-ml-Röhrchen mit 2-3 ml Verdauungsmedium (Tabelle 1) oder genug, um den Eierstock zu bedecken.
ACHTUNG: Wenn der Eierstock nicht intakt ist (ein Teil des Organs wurde für die histologische Analyse geschnitten), ist es wichtig, diesen Teil nicht mit dem Verdauungsmedium zu bedecken. - Legen Sie den Schlauch für 30 Minuten in ein vorgewärmtes Perlenbad (oder Wasserbad) bei 37 °C.
- Übertragen Sie den Eierstock vorsichtig in eine 60-mm-Petrischale, die 10 ml Entnahmemedium enthält (Tabelle 1).
- Kratzen Sie die Oberfläche der Eierstöcke, die die hOSE-Zellen enthält, vorsichtig mit einem Zellschaber ab. Waschen Sie den Schaber im Medium und wiederholen Sie diesen Schritt mindestens dreimal (Abbildung 1).
ACHTUNG: Wenn der Eierstock nicht intakt ist, vermeiden Sie es, den beschädigten Bereich sowohl abzukratzen als auch einzutauchen, um die Kontamination mit unerwünschten Zelltypen zu minimieren. - Übertragen Sie die hOSE-Zellen im Sammelmedium in ein 15-ml-Röhrchen.
- Schleudern Sie hOSE-Zellen bei 240 x g für 5 min.
- Wenn das Pellet eine rote Farbe zeigt, die auf eine Kontamination mit roten Blutkörperchen (RBC) hinweist, resuspendieren Sie das Pellet mit 1 ml RBC-Lysepuffer. 3 min bei Raumtemperatur (RT) mit gelegentlichem Pipettieren inkubieren, 4 mL PBS mit Calcium und Magnesium (PBS+/+) zugeben und 5 min bei 240 x g zentrifugieren. Wenn RBC weiterhin besteht, wiederholen Sie diesen Schritt.
- Kryokonservieren Sie das hOSE-Zellpellet (Abschnitt 2), verwenden Sie es für 2D-Kulturen (Abschnitt 3) oder 3D-Organoide (Abschnitt 4) (Abbildung 1).
2. Kryokonservierung-Auftauen von hOSE-Zellen
- Kryokonservierung
- Resuspendieren Sie das hOSE-Zellpellet in 1 ml Gefriermedium.
- Zellsuspension in Kryoröhrchen überführen (2x Kryoröhrchen pro Eierstock).
- Stellen Sie die Kryoröhrchen in einen Gefrierbehälter und stellen Sie ihn über Nacht bei -80 °C auf.
- Füllen Sie die gefrorenen Kryofläschchen zur Langzeitlagerung in einen Flüssigstickstofftank um.
- Auftauen:
- Entfernen Sie das Kryofläschchen aus dem flüssigen Stickstoff.
- Stellen Sie das Kryofläschchen für 5 Minuten in ein vorgewärmtes Perlbad (oder Wasserbad) bei 37 °C oder bis nur noch ein kleiner gefrorener Kern im Kryofläschchen sichtbar ist.
- Pipettieren Sie die hOSE-Zellsuspension in ein 15-ml-Röhrchen mit 10 mL Sammelmedium (Tabelle 1).
- Bei 240 x g 5 min zentrifugieren.
- Verwenden Sie das hOSE-Zellpellet für 2D-Kulturen (Abschnitt 3) oder 3D-Organoide (Abschnitt 4).
3. 2D-hOSE-Kultur in einer Monoschicht
- Resuspendieren Sie das hOSE-Pellet in 1 mL OSE_2D Medium (Tabelle 1) und überführen Sie es in eine Vertiefung aus einer 12-Well-Platte.
- Geben Sie 1 ml zusätzlichen OSE_2D Mediums in die Vertiefung und kultivieren Sie ihn 72 Stunden lang bei 37 °C in einem befeuchteten Inkubator (5 % CO2), um sicherzustellen, dass sich die hOSE-Zellen vor dem ersten Medienwechsel anheften.
- Aktualisieren Sie das Medium alle 2-3 Tage, bis die hOSE-Zellen eine Konfluenz von 70 % bis 90 % (P0) erreichen (Abbildung 2A).
HINWEIS: Wenn noch Erythrozyten in der Kultur vorhanden sind, werden sie beim ersten Medienwechsel entfernt, und nur die hOSE-Zellen bleiben an der Vertiefung befestigt. - Führen Sie zum Passieren von hOSE-Zellen die unten beschriebenen Schritte aus.
- Entfernen Sie die Nährmedien aus der Vertiefung.
- Mit 1 ml sterilem PBS waschen.
- Entfernen Sie PBS und fügen Sie genügend 0,05 % Trypsin/EDTA hinzu, um die Zellen zu bedecken.
- Stellen Sie die Platte bei 37 °C für 4-7 Minuten in einen befeuchteten Inkubator (5 % CO2), bis Sie bemerken, dass sich die Zellen zusammenballen und aus der Vertiefung lösen.
- Stoppen Sie die enzymatische Reaktion, indem Sie 1 ml Sammelmedium hinzufügen.
- Sammeln Sie die hOSE-Zellen und zentrifugieren Sie sie bei 240 x g für 5 min.
- Säen Sie die Zellen in einer neuen Vertiefung mit der gewünschten Dichte, aktualisieren Sie das Medium alle 2-3 Tage, bis die hOSE-Zellen eine Konfluenz von 70 % bis 90 % erreicht haben, und wiederholen Sie Schritt 3.4.
HINWEIS: Primäre hOSE-Zellen können mit bis zu drei Durchgängen kultiviert werden. Danach werden die meisten Zellen seneszent (Abbildung 2A).
- Verwenden Sie die expandierten hOSE-Zellen für die weitere Charakterisierung mittels Immunfluoreszenz (Abbildung 2B), um die Wirkung von Kulturmedien (Abbildung 2C) oder für 3D-Organoide (Abschnitt 4) zu testen.
4. 3D hOSE-Organoid-Kultur
- Multiwell-Objektträger mit Kammer bei 37 °C vorwärmen.
- Zählen Sie (frisch isoliert oder kryokonserviert-aufgetaut) hOSE-Zellen mit einem automatisierten Zellzähler oder manuell mit einem Hämozytometer.
- Resuspendieren Sie die gewünschte Anzahl von hOSE-Zellen in 1 ml eiskaltes OSE-Organoid-Basismedium (Tabelle 1) in einem 1,5-ml-Röhrchen.
- Bei 240 x g 5 min zentrifugieren.
- Resuspendieren Sie das hOSE-Pellet in der gewünschten Menge eiskalter, unverdünnter Basalmembran-Extrakt-Lösung (BME), um eine Zellkonzentration von 1 x 104 Zellen/10 μl BME zu erhalten.
- Pipettieren Sie die hOSE-BME-Lösung auf und ab, um eine homogene Verteilung zu gewährleisten.
- Stellen Sie 10 μl Tröpfchen hOSE-BME-Lösung pro Vertiefung in dem vorgewärmten Objektträger mit mehreren Vertiefungen her.
HINWEIS: Stellen Sie sicher, dass sich jedes Tröpfchen in der Mitte der Vertiefung befindet, um eine Tropfenform zu erhalten. - Stellen Sie die Platte mit den Tröpfchen kopfüber bei 37 °C für 15 min in einen befeuchteten Inkubator (5 % CO2), damit sich das Gel verfestigen kann.
- 100 μl OSE_3D Medium (Tabelle 1) zugeben und bei 37 °C in einem befeuchteten Inkubator (5 % CO2) kultivieren.
- Frischen Sie das Medium alle 3-4 Tage auf.
HINWEIS: hOSE-Organoide wachsen in Kultur mindestens bis zu 28 Tagen weiter (Abbildung 3A, B), aber eine Passage alle 14-28 Tage (die Wachstumsrate ist spenderabhängig, frisch im Vergleich zu Kryo-hOSE) wird empfohlen, um die Zellproliferation und das Überleben der Organoide zu stimulieren. Drei unabhängige hOSE-Organoidlinien wurden mindestens 4 Mal ohne Anzeichen von Seneszenz durchquert. hOSE-Organoide weisen unterschiedliche Morphologien auf (Abbildung 3C). - Für die Passage von hOSE-Organoiden:
- Entfernen Sie das Medium und geben Sie 100 μl eiskaltes Advanced DMEM/F12 in jede Vertiefung.
- Kratzen Sie den Boden der Vertiefungen mit einer P1000-Pipettenspitze ab, um die Geltröpfchen zu lösen.
- Übertragen Sie jedes schwimmende Geltröpfchen in ein 1,5-ml-Röhrchen.
- Zentrifugieren Sie die Röhrchen mit den Geltröpfchen bei 240 x g für 5 min.
- Entfernen Sie den Überstand, resuspendieren Sie das Pellet in 300 μl Zelldissoziationspuffer und legen Sie die Röhrchen für 5-10 Minuten in ein vorgewärmtes Bead-Bad (oder Wasserbad) bei 37 °C.
HINWEIS: Wenn einige Organoide intakt geblieben sind, pipettieren Sie einige Male mit einer mit fötalem Rinderserum beschichteten, eiskalten Pipettenspitze auf und ab, um sie mechanisch aufzubrechen. Das Überziehen der Pipettenspitzen mit fötalem Rinderserum verhindert, dass die Organoide an der Spitze haften bleiben. - 300 μl hOSE-Organoid-Basismedium zugeben und 5 min bei 240 x g zentrifugieren.
- Resuspendieren Sie das Pellet in der gewünschten Menge unverdünnter BME-Lösung, säen Sie es in einem geeigneten Verhältnis erneut aus (verteilen Sie den Inhalt von 1 Tropfen auf 1-4 Tröpfchen) und kultivieren Sie es wie oben erwähnt.
HINWEIS: Die Organoide werden nicht in einzelne Zellen dissoziiert, so dass die Zellen für die weitere Passage nicht genau gezählt werden können.
5. Immunfluoreszenz von 3D-hOSE-Organoiden
HINWEIS: Die Immunfluoreszenz für den gesamten Mount kann in derselben Kulturvertiefung (in De-Droplet) durchgeführt werden, wenn Mehrwell-Objektträger verwendet werden, die für Mikroskopietechniken geeignet sind.
- Fixieren Sie die hOSE-Organoide in den Tröpfchen in 4% Paraformaldehyd (PFA) für 20 min bei RT.
- Zweimal mit PBS für 5 min bei RT auf einer rotierenden/schüttelnden Plattform waschen.
- Permeabilisieren Sie die hOSE-Organoide in den Tröpfchen mit 100 μl Permeabilisierungspuffer (Tabelle 1) für 15 min bei RT.
- Dreimal mit 100 μl Blockierungspuffer (Tabelle 1) für 15 min bei RT waschen und über Nacht (o/n) bei 4 °C auf einer rotierenden/schüttelnden Plattform blockieren.
- Inkubieren Sie mit 100 μl Primärantikörpermix, verdünnt in Blockierungspuffer bei 4 °C o/n auf einer rotierenden/schüttelnden Plattform.
HINWEIS: In der Materialtabelle finden Sie eine Liste der verwendeten Primärantikörper, des markierten Zelltyps und des Tiermodells, das zur Validierung der Expression 12,19,20,21,22,23,24,25 verwendet wurde. - Bringen Sie die Platte auf RT und waschen Sie sie dreimal mit 100 μl Blocking-Puffer für 15 min bei RT auf einer rotierenden/schüttelnden Plattform.
- Inkubieren Sie mit 100 μl Sekundärantikörpermix, verdünnt in Blockierungspuffer für 2 h bei RT auf einer rotierenden/schüttelnden Plattform, aber lichtgeschützt.
HINWEIS: Eine Liste der verwendeten Sekundärantikörper finden Sie in der Materialtabelle . Zum Schutz vor Licht legen Sie die Platte entweder in eine dunkle Box oder decken Sie sie mit Alufolie ab. - Dreimal 15 Minuten lang in 100 μl PBS bei RT auf einer rotierenden/schüttelnden Plattform waschen.
- Lagern Sie die Platte bis zur Bildgebung bei 4 °C mit Aluminiumfolie bedeckt (Abbildung 4).
6. Quantifizierung der Größe von hOSE-Organoiden
- Nehmen Sie 10x Hellfeldbilder (TIFF-Bild) mit einem Mikroskop auf.
- Laden Sie Fiji (https://imagej.net/software/fiji/)26 herunter und installieren Sie es.
- Messen Sie den Bildbereich von hOSE-Organoiden mit der ImageJ-Software Fidschi:
- Laden Sie TIFF-Bilddateien in Fidschi.
- Erstellen Sie einen Schwellenwert für die Software, um die hOSE-Organoide (dunklerer Bereich) im hochgeladenen Hellfeldbild zu erkennen, indem Sie auf Bild > Schwellenwert anpassen > klicken. Passen Sie die Werte unten und oben an, bis der größte Teil des Hintergrunds entfernt ist und einzelne Organoide beibehalten werden.
- Öffnen Sie das ROI-Tool, indem Sie auf > Tool analysieren > ROI klicken.
- Wählen Sie das Zauberstab-Werkzeug aus und klicken Sie auf den Bereich mit einem hOSE-Organoid, bis Sie eine gelbe Linie sehen, die den gesamten Organoidumfang umgibt. Klicken Sie im ROI-Bereich auf Hinzufügen und Messen , um den Flächenwert dieses OSE-Organoids zu erhalten.
- Wiederholen Sie Schritt 6.3.4. für jedes zu messende hOSE-Organoid.
Ergebnisse
2D-hOSE-Kultur
Frisch isolierte hOSE-Zellen wurden auf einer 12-Well-Platte plattiert und 3 Wochen lang kultiviert. Während dieser Zeit wurden die Zellen dreimal durchgelassen (Abbildung 2A). Primäre hOSE-Zellen in Kultur zeigten bis zur Passage 3 (P3) eine kopfsteinpflasterartige Morphologie, zeigten danach aber erste Anzeichen von Seneszenz, in Übereinstimmung mit früheren Ergebnissen, die berichteten, dass die begrenzte Zeitspanne hOSE passaget werden kann27. Darüber hinaus wurde gezeigt, dass murine OSE in vitro eine epitheliale zu mesenchymale Transition (EMT) durchlaufen und fibroblastenähnliche Merkmale wie die Umlagerung des Aktin-Zytoskeletts und die Ablagerung von Kollagen I19 aufweisen. In Übereinstimmung mit ACTA2+CD44+ hOSE zeigte sich eine ausgeprägte Hochregulation von COL1A1 zwischen P2 und P3 (Abbildung 2B), was darauf hindeutet, dass hOSE-Zellen während der Kultur einer EMT unterzogen werden.
Der TGF-β-Signalweg ist ein wichtiger Regulator von EMT28 und kann EMT in verschiedenen Epithelzelltypeninduzieren 29. Obwohl TGF-β den OSE_2D Medien nicht zugesetzt wurde, könnte das zugegebene FBS nachweisbare Spiegel dieses Zytokins enthalten30,31. Aus diesem Grund haben wir getestet, ob die Zugabe des TGF-β-Inhibitors Typ I Rezeptor SB-431542 eine EMT verhindern kann, was eine verlängerte 2D-Kultur ermöglicht. Überraschenderweise hemmte die Supplementierung der OSE_2D Medien mit 10 μM SB-43154232die Zellproliferation (Abbildung 2C). Wir kamen zu dem Schluss, dass der TGF-β-Signalweg für die OSE-Proliferation essentiell ist, und weitere 2D-Kulturexperimente wurden ohne den Inhibitor durchgeführt.
3D-hOSE-Organoid-Kultur
Basierend auf veröffentlichten Kulturbedingungen zur Züchtung humaner OSE-Organoide12 haben wir hOSE-Organoide abgeleitet, die in BME-Tröpfchen eingebettet und in OSE_3D Medium (mit einer Standardkonzentration von 100 nM Östradiol) für bis zu 28 Tage gezüchtet wurden. Innerhalb von 7 Tagen waren viele hOSE-Organoide, die aus frisch isolierten OSE-Zellen gewonnen wurden, zystisch mit einem durchschnittlichen Durchmesser von 130 mm (Abbildung 3A,B). Diese Ergebnisse ähnelten denen, die von Kopper und Kollegen über die Gewinnung von hOSE-Organoiden aus gehacktem und enzymatisch verdautem menschlichem Eierstockgewebe berichtetwurden 12. Interessanterweise wurden hOSE-Organoide, die aus frisch isolierten OSE-Zellen gewonnen wurden, nach 14 Tagen in Kultur größer (durchschnittlicher Durchmesser von 160 mm) als hOSE-Organoide aus 2D-expandierten OSE-Zellen (durchschnittlicher Durchmesser von 100 mm) (Abbildung 3B), wahrscheinlich aufgrund der Neigung der 2D-expandierten OSE-Zellen, die Proliferation zu verringern. Während viele 3D-hOSE-Organoide aus einer Monoschicht flacher Epithelzellen bestanden (Abbildung 3C oben links), wiesen einige eine quaderförmige Monoschicht von Zellen auf (Abbildung 3C oben rechts), andere waren mehrschichtig (Abbildung 3C unten rechts) oder bildeten einen nicht lumenisierten Zellcluster (Abbildung 3C unten links).
Um zu testen, ob sich hOSE-Organoide auch mit OSE_2D Medium ableiten lassen, wurden frisch isolierte hOSE-Zellen in Tröpfchen aus BME eingebettet und 12 Tage lang in OSE_2D Medium gezüchtet. Nach 6 Tagen waren spindelartige Zellen sichtbar, was darauf hindeutet, dass hOSE-Zellen in den Tröpfchen einer EMT unterzogen wurden. Wichtig ist, dass unter Verwendung OSE_2D Mediums keine hOSE-Organoide gebildet wurden (Abbildung 3D).
Zelluläre Eigenschaften der hOSE-Organoide
Im menschlichen adulten Eierstock markiert Keratin 8 (KRT8) spezifisch die OSE-Population (Abbildung 4A), und dementsprechend behielten hOSE-Organoide die KRT8-Expression bei, was ihre Zellidentität bestätigt (Abbildung 4B). In OSE-Zellen der Maus war die nukleäre Lokalisation des Yes1-assoziierten Proteins (YAP1) spezifisch mit OSE-Stamm-/Vorläuferzellen assoziiert, die in der Lage waren, den verwundeten Bereich nach dem Eisprung zu erweitern und zu heilen24,33. YAP zeigte in der Mehrzahl der Zellen in hOSE-Organoiden eine nukleäre Lokalisation unabhängig von ihrer Größe (Abbildung 4B). Andere Marker, von denen berichtet wurde, dass sie in OSE-Zellen der Maus exprimiert werden, wie CD4420 und LGR522, wurden ebenfalls untersucht. Interessanterweise wurden sowohl CD44 als auch LGR5 in kleinen hOSE-Organoiden (<100 μm Durchmesser) exprimiert, während CD44 und LGR5 in größeren Organoiden herunterreguliert schienen (Abbildung 4B).
Als nächstes untersuchten wir, ob hOSE-Organoide die apikal-basale Polarität aufwiesen, die typischerweise bei BME-eingebetteten Organoiden (apikal-in) beobachtet wird34. Das basolaterale Protein Integrin beta 1 (ITGB1) und das apikale Protein Podocalyxin (PODXL) wurden verwendet, um die Zellpolarität in den hOSE-Organoiden zu zeigen, was eine klare apikale In-Polarität unabhängig von der Größe des hOSE-Organoids bestätigt (Abbildung 4B).
Es ist bekannt, dass das humane OSE den mesenchymalen Marker N-Cadherin (CDH2) exprimiert, während die Expression des epithelialen Markers E-Cadherin (CDH1) auf säulenförmige OSE-Zellen beschränkt ist (Abbildung 4A)2. Interessanterweise exprimierten die in dieser Arbeit abgeleiteten hOSE-Organoide die mesenchymalen Marker CDH2 und Vimentin (VIM), und große zystische hOSE-Organoide mit quaderförmigen/säulenförmigen Epithelien waren sowohl CDH1+ als auch CDH2+VIM+ (Abbildung 4C). Es bleibt unklar, ob CDH1+ hOSE-Organoide von primären CDH1+ OSE-Zellen oder von CDH1-OSE-Zellen abgeleitet wurden, die in vitro eine epitheliale Differenzierung oder (neoplastische) Transformation durchlaufen haben 25,35.
Präsentation der Verwendung von hOSE-Organoiden: Wirkung von ovulatorischen Signalen auf hOSE-Organoide
Die Hormone follikelstimulierendes Hormon und humanes Choriongonadotropin können verwendet werden, um das Follikelwachstum und den Eisprung zu induzieren, während nach dem Eisprung ein deutlicher Anstieg der Konzentration von Eierstöcken produzierten Hormonen wie Progesteron und Östradiol auftritt36. Um die Verwendbarkeit von hOSE-Organoiden als Screening-Plattform zu demonstrieren, untersuchten wir die Wirkung dieser Hormone auf hOSE-Organoide, wobei wir die Anzahl und Größe der Organoide als Quantifizierungsausgabe verwendeten (unter Verwendung von Fidschi). Zu diesem Zweck haben wir hOSE-Organoide in OSE_3D Medien abgeleitet, denen Östradiol (kein Hormon) fehlt, und in OSE_3D Medien, die FSH, hCG, Östradiol oder Progesteron in unterschiedlichen Konzentrationen enthalten (Abbildung 5A).
Die Anzahl der hOSE-Organoide, die aus kryokonservierten-aufgetauten (nicht expandierten) hOSE-Zellen von 3 verschiedenen Spendern (n=3) gewonnen wurden, wurde pro Tröpfchen nach 7-tägiger Kultur quantifiziert (Abbildung 5A,B). Das Nährmedium wurde alle 3 Tage gewechselt. Es wurden keine signifikanten Unterschiede in der Gesamtzahl der hOSE-Organoide pro Tröpfchen beobachtet, die sich in mediumhaltigen Hormonen im Vergleich zu Medium ohne Hormone bildeten (Abbildung 5A,B). Um die Wirkung der Hormone auf die Organoidgröße zu quantifizieren, wurde nach 14 Tagen in Kultur jedes Tröpfchen (von jeder Bedingung) abgebildet und die Fläche der 4 größten hOSE-Organoide mit Fidschi gemessen. Interessanterweise führte die Erzeugung von hOSE-Organoiden aus zwei verschiedenen Donatoren (n = 2) in Gegenwart von Progesteron (1000 nM) oder Östradiol (200 nM) zu mindestens einem sehr großen Organoid (etwa 700 μm Durchmesser) pro Tröpfchen (Abbildung 5C,D).
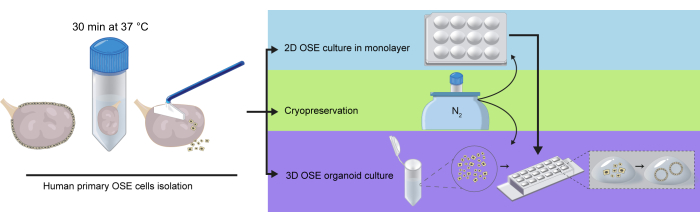
Abbildung 1: Schematische Darstellung der Isolierung von hOSE-Zellen aus ganzen Eierstöcken. Die Eierstöcke wurden 30 Minuten lang bei 37 °C in Aufschlusslösung inkubiert. hOSE-Zellen wurden durch vorsichtiges Schaben von der Ovarialoberfläche gelöst und anschließend kryokonserviert, direkt auf einer 2D-OSE-Kultur in einer Monoschicht plattiert oder in Basalmembranextrakt (BME) für die 3D-hOSE-Organoidkultur eingebettet. Kryokonservierte, aufgetaute hOSE-Zellen können für 2D-Kulturen oder die Bildung von 3D-Organoiden verwendet werden, und 2D-expandierte hOSE-Zellen können zur Erzeugung von 3D-hOSE-Organoiden verwendet werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: 2D-Primärkultur von hOSE-Zellen. (A) Hellfeldbilder von hOSE-Zellen an den Passagen 0 (P0), 2 (P2) und 3 (P3), die eine typische epitheliale Kopfsteinpflastermorphologie darstellen. Die Maßstabsleisten betragen 750 μm auf den oberen Platten und 125 μm auf den unteren Platten. (B) Immunfluoreszenz für ACTA2, CD44, COL1A1 auf 2D-hOSE-Zellen an P2 und P3. Die Maßstabsbalken betragen 50 μm. (C) Hellfeldbilder von hOSE-Zellen, die mit 10 μM SB-431542 an P0, P2 und P3 kultiviert wurden. Die Zellen zeigten Anzeichen einer Seneszenz von P2 und erreichten während der Kultur keine hohe Konfluenz. Die Maßstabsleisten betragen 750 μm auf den oberen Platten und 125 μm auf den unteren Platten. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
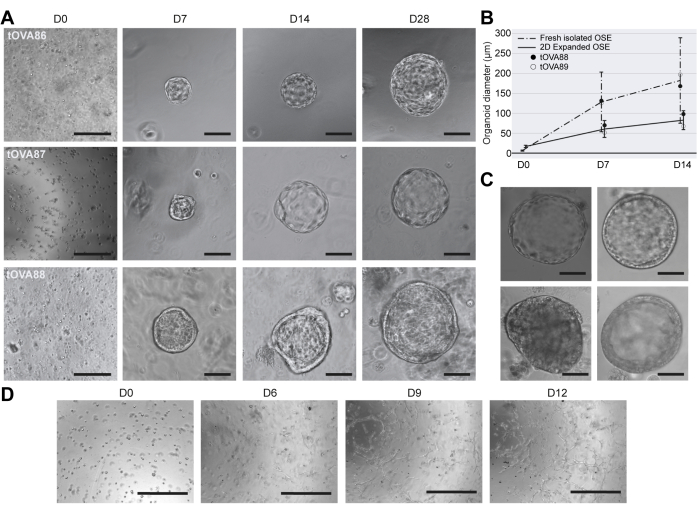
Abbildung 3: Morphologische Charakterisierung von hOSE-Organoiden. (A) Hellfeldbilder von drei unabhängigen hOSE-Organoid-Ableitungen von drei verschiedenen Donoren (tOVA86, tOVA87, tOVA88). Die Bilder wurden nach der Zelleinbettung (D0), Tag 7 (D7), Tag 14 (D14) und Tag 28 (D28) der Kultur aufgenommen. Die Maßstabsbalken betragen 750 μm bei D0 und 50 μm für den Rest. (B) Durchmesser von hOSE-Organoiden, die aus frisch isolierten hOSE-Zellen (gestrichelte Linie) und 2D-expandierten hOSE-Zellen (durchgezogene Linie) stammen. Dargestellt ist die durchschnittliche Größe ± Standardabweichung, gemessen bei D0, D7 und D14. Es werden Ergebnisse von zwei verschiedenen Spendern (tOVA88 und tOVA89) gezeigt. (C) Hellfeldbilder von hOSE-Organoiden mit unterschiedlichen Morphologien: einzelne flache Schicht (oben links), einzelne spaltenförmige Schicht (oben rechts), mehrschichtig (unten rechts) und nicht-lumenisiertes Zellaggregat (unten links). Die Maßstabsbalken betragen 100 μm. (D) Hellfeldbilder von hOSE-Zellen, die in BME eingebettet und 12 Tage lang mit OSE_2D Medien kultiviert wurden. Die Bilder wurden nach der Zelleinbettung (D0), Tag 6 (D6), Tag 9 (D9) und Tag 12 (D12) aufgenommen. Fibroblasten-ähnliche Zellen überholten die Kultur, und es bildeten sich keine organoidartigen Strukturen. Die Maßstabsstäbe betragen 750 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
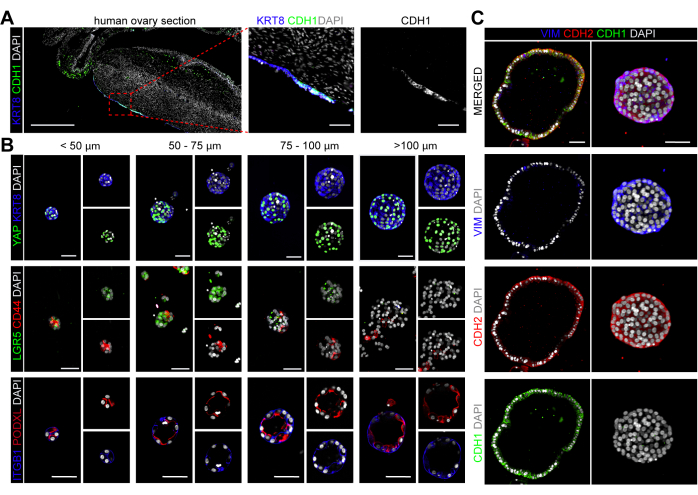
Abbildung 4: Zelluläre Charakterisierung von hOSE-Organoiden. (A) Immunfluoreszenz für KRT8 und CDH1 im menschlichen Ovarialschnitt. Der Maßstabsbalken auf dem linken Feld beträgt 500 μm, der mittlere und der rechte Bereich 50 μm. (B) Immunfluoreszenz für KRT8 und YAP, LGR5 und CD44 sowie ITGB1 und PODXL in hOSE-Organoiden unterschiedlicher Größe (<50 μm, 50-75 μm, 75-100 μm, >100 μm). Die Maßstabsbalken betragen 50 μm. (C) Immunfluoreszenz für VIM, CDH1, CDH2 in hOSE-Organoiden. Die Maßstabsstäbe sind 50 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
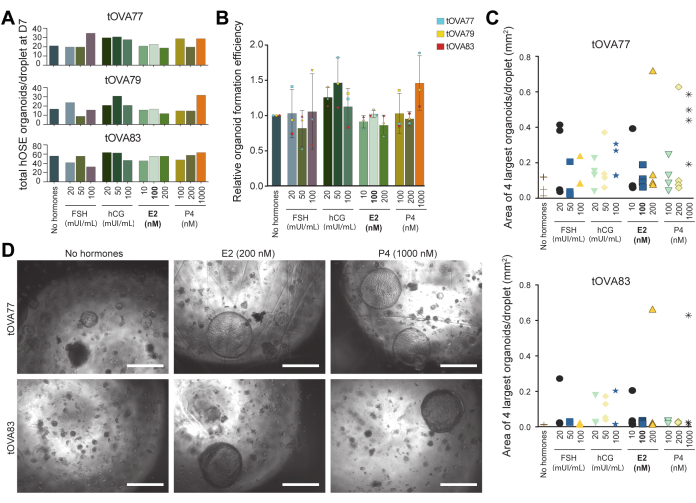
Abbildung 5: Einfluss von ovulatorischen Signalen auf die Bildung von hOSE-Organoiden. (A) Gesamtanzahl der hOSE-Organoide pro Tröpfchen, die an Tag 7 in verschiedenen Kulturmedien gebildet wurden. Die hOSE-Organoide wurden von drei verschiedenen Spendern (tOVA77, tOVA79, tOVA83) abgeleitet. Fett gedruckt ist das OSE_3D Medium, das für die Organoid-Ableitung verwendet wird. (B) Relative Organoidbildung im Vergleich zu Medium ohne Hormone. Die gepoolten Werte von hOSE-Organoiden wurden von drei verschiedenen Spendern (tOVA77, tOVA79, tOVA83) abgeleitet. (C) Grafik, die den Bildbereich der 4 größten hOSE-Organoide an Tag 14 unter jedem der getesteten Versuchsbedingungen von zwei verschiedenen Spendern (tOVA77, tOVA83) darstellt. (D) Hellfeldbilder, die hOSE-Organoide an Tag 14 in Kultur mit Medium ohne Hormone, 200 nM E2 und 1000 nM P4 von zwei verschiedenen Spendern (tOVA77, tOVA83) zeigen. Die Maßstabsstäbe betragen 750 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Tabelle 1: Zusammensetzung der in der Studie verwendeten Arbeitslösungen. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Diskussion
Die 3D-Organoid-Technologie entwickelt sich zu einem unverzichtbaren Werkzeug für die medizinische Forschung. Einerseits bietet diese in vitro Plattform die Möglichkeit, grundlegende mechanistische Fragen zur Geweberegeneration, Wundheilung und -entwicklung zu untersuchen18. Auf der anderen Seite ermöglichen 3D-Organoide, die aus Patientenproben gewonnen werden, personalisierte medizinische Studien, einschließlich Diagnostik, Arzneimitteltests und Zelltherapie 12,13,14,37,38. Im Bereich der Ovarialforschung hat das hOSE seit seiner Implikation als Ursprung der epithelialen Ovarialkarzinome erhebliches Interesse geweckt39. Obwohl angenommen wird, dass das meiste hochgradige seröse Ovarialkarzinom (HGSOC), einer der häufigsten epithelialen Eierstockkrebsarten, aus den Eileitern entsteht40, hat die aktuelle Forschung an 3D-Ovarialorganoiden von Mäusen einen möglichen dualen Ursprung von HGSOC aus OSE und Eileiter vorgeschlagen15,16.
In dieser Arbeit haben wir ein Protokoll für die Derivation von hOSE 3D-Organoiden beschrieben und seine Anwendung skizziert, um neue mechanistische Erkenntnisse in der Regeneration von Eierstockgewebe zu gewinnen. Dieses Protokoll beinhaltet eine Schritt-für-Schritt-Methode zur Isolierung primärer hOSE-Zellen aus menschlichen Eierstöcken und zur Erzeugung von 3D-hOSE-Organoiden. Um eine effiziente Ableitung von hOSE-Organoiden zu gewährleisten, ist es entscheidend, die Manipulation der Eierstöcke zu minimieren. Aufgrund seiner Lage auf der Oberfläche der Eierstöcke und der Organisation der Monoschicht ist das hOSE anfällig für Schäden und Verluste während der Oophorektomie und Organmanipulation. Aus diesem Grund haben wir eine enzymatische und schabende Methode bevorzugt, die auf den gesamten Eierstock angewendet wird, um hOSE 2,8 zu isolieren. Im vorliegenden Protokoll wurde eine milde enzymatische Behandlung angewendet, um die interzellulären hOSE-Verbindungen zu stören, gefolgt von einem sanften Abkratzen der Ovarialoberfläche.
Beim Vergleich von 2D- mit 3D-hOSE-Kulturen ist es wichtig zu beachten, dass sich trotz der anfänglich hohen Proliferationsrate von hOSE-Zellen in 2D-Kulturen ihre zellulären Eigenschaften durch EMT verändert haben, was darauf hindeutet, dass die angewandten 2D-Kulturbedingungen nicht geeignet sind, eine epitheliale Morphologie aufrechtzuerhalten. Im Gegensatz dazu konnten 3D-hOSE-Organoide mindestens 4 Mal ohne Anzeichen von Seneszenz durchquert werden. Die OSE_3D verwendeten Organoid-Kulturmedien basierte auf denen, die von Kopper und Kollegen für die Ableitung von OC und gesunden hOSE-Organoiden12 und von Kessler und Kollegen für die Ableitung von humanen FT-Organoiden17 verwendet wurden. Der Hauptunterschied bestand darin, dass humane Wnt3a- und R-Spondin-1-konditionierte Medien durch kommerziell erhältliche rekombinante Proteine ersetzt wurden, um die Reproduzierbarkeit zu erleichtern.
Bei Immunfluoreszenztechniken wird in der Regel die Gewebeprobe aus der Kulturplatte entnommen und für Paraffin- oder Kryoschnitte verarbeitet. Bei der Arbeit mit sehr kleinen Strukturen ist das Risiko, diese bei der Probenverarbeitung zu verlieren, hoch. In diesem Protokoll erfolgt die Gewinnung von hOSE-Organoiden in Zellkulturplatten, die eine direkte mikroskopische Bildgebung ermöglichen, ohne dass die hOSE-Organoide aus der BME-Matrix entfernt werden müssen. Darüber hinaus ermöglichte die hier verwendete Whole-Mount-Immunfluoreszenzmethode, die von Rezanejad und Kollegen für duktale Organoide der Bauchspeicheldrüsebeschrieben wurde 41, die In-situ-Beobachtung der Proteinlokalisation in morphologisch intakten Organoiden. Wir haben gezeigt, dass bei der Durchführung dieses Immunfluoreszenzprotokolls an hOSE-Organoiden, die in Objektträgern mit mehreren Wells gewonnen wurden, eine hocheffiziente Antikörperpenetration mit einem sehr geringen Hintergrundsignal erfolgt.
Obwohl die meisten der mit dieser Methode gewonnenen hOSE-Organoide keine CDH1-Expression aufwiesen, bildeten sich einige CDH1+ hOSE-Organoide, die im Vergleich zu CDH1-hOSE-Organoiden größere Größen erreichten. Die Expression von CDH1 wurde mit den neoplastischen hOSE-Phänotypenassoziiert 2,35. Die Eierstöcke, die für die hOSE-Isolierung verwendet wurden, wurden von gesunden transmaskulinen Spenderinnen im gebärfähigen Alter (27,1 ± 5 Jahre) gespendet. Diese Spenderinnen wurden vor der Oophorektomie über einen Zeitraum von 38 ± 15 Monaten mit Testosteron behandelt. Wir können die Möglichkeit nicht ausschließen, dass die CDH1+ hOSE-Zellen auf der Oberfläche der Eierstöcke auf die Testosteronbehandlung zurückgeführt werden könnten. Obwohl die Androgenbehandlung mit Veränderungen der Eierstöcke in Verbindung gebracht wurde, wie z. B. Anovulation42, Hyperplasie des kortikalen Bereichs43 und erhöhter kortikaler Steifigkeit44, bleibt die allgemeine ovarielle Pathologie während der Einnahme von Testosterongutartig 45.
Zusammenfassend unterstreicht dieses Protokoll das Potenzial der Generierung von hOSE 3D-Organoiden, um mechanistische Fragen zur Regeneration von Eierstockgewebe zu entschlüsseln. Wichtig ist, dass diese Methode auch für den Nachweis von bösartigen Zellen in Eierstockbiopsien von Patientinnen mit Krebsrisiko eingesetzt werden könnte. Insgesamt unterstützt diese Methode potenzielle Anwendungen dieser innovativen In-vitro-Plattform sowohl für grundlegende Studien zur Funktion der Eierstöcke als auch für klinische Anwendungen für individualisierte medizinische Behandlungen.
Offenlegungen
Nichts.
Danksagungen
Wir danken allen Patienten, die Gewebe für diese Studie gespendet haben, den Mitgliedern der Gruppe Chuva de Sousa Lopes für die nützlichen Diskussionen und I. De Poorter für die Gestaltung der in Abbildung 1 verwendeten Cartoons. Diese Forschung wurde vom Europäischen Forschungsrat unter der Fördernummer ERC-CoG-2016- 725722 (OVOGROWTH) für J.S.D.V. und S.M.C.d.S.L. finanziert; und die Novo Nordisk Foundation (reNEW), Fördernummer NNF21CC0073729 für J.S.D.V. und S.M.C.d.S.L.
Materialien
| Name | Company | Catalog Number | Comments |
| 0.05% Trypsin/EDTA | Invitrogen | 25200-056 | |
| 12-well Culture Plate | Corning | 3336 | Sterile |
| 15 mL tubes | Greiner | 188271 | Sterile |
| 28cm Cell Scraper | Greiner Bio-One | 541070 | |
| 50 mL tubes | Greiner | 227261 | Sterile |
| 60 mm Petri dish | Greiner Bio-One | 628160 | |
| A83-01 | Stem Cell Technologies | 72024 | |
| Advanced DMEM/F12 | Gibco | 12634-010 | |
| B27 supplement (50x) | ThermoFisher Scientific | 17504-044 | |
| Bead bath | M714 | ||
| Bovine serum albumin (BSA) | Sigma Aldrich | 10735086001 | |
| Cell Dissociation Buffer | ThermoFisher Scientific | 13151014 | |
| Cryo-container "Mr. Frosty" | BD Falcom | 479-3200 | |
| DMEM Medium | ThermoFisher Scientific | 41966-029 | |
| Donkey anti-Goat IgG Alexa Fluor 647 | Invitrogen | A-21447 | |
| Donkey anti-Mouse IgG Alexa Fluor 488 | Invitrogen | A-21202 | |
| Donkey anti-Mouse IgG Alexa Fluor 647 | Invitrogen | A-31571 | |
| Donkey anti-Rabbit IgG Alexa Fluor 488 | Invitrogen | A-21206 | |
| Donkey anti-Rabbit IgG Alexa Fluor 594 | Invitrogen | A-21207 | |
| Donkey anti-Sheep IgG Alexa Flour 647 | Invitrogen | A-21448 | |
| Fetal Bovine Serum (FBS) | ThermoFisher Scientific | A4736401 | |
| Follicle Stimulating Hormone (FSH) | Sigma Aldrich | F4021 | |
| Forskolin | Peprotech | 6652995 | |
| Glutamax (100x) | Gibco | 35050-038 | |
| Goat anti-CDH2 (N/R-cadherin) | Santa Cruz | SC-1502 | Mesenchymal Cells; Wong et al 1999 (human)25 |
| Goat anti-PODXL (podocalyxin of GP135) | R&D Systems | AF1658 | Apical Polarity; Bryant et al 2014 (canine)21 |
| Goat anti-Rat IgG Alexa Fluor 555 | Invitrogen | A-21434 | |
| hEGF | R&D Systems | 263-EG | |
| HEPES | Gibco | 15630-056 | |
| Hydrocortisone | Sigma Aldrich | H0888 | |
| Insulin-Transferrin-Selenium-Ethanolamine (ITS-X; 100x) | ThermoFisher Scientific | 51500-056 | |
| Liberase DH Research Grade | Sigma Aldrich | A4736401 | |
| Luna-II cell counter | Logos Biosystems | L40001 | |
| Matrigel | Sigma Aldrich | 354277 | |
| McCoy’s 5A Medium | ThermoFisher Scientific | 26600-023 | |
| Mouse anti-ITGB1 (integrin beta 1) | Santa Cruz | SC-53711 | Basolateral Polarity; Bryant et al 2014 (canine)21 |
| Mouse anti-KRT8 (cytokeratin 8) | Santa Cruz | SC-101459 | OSE Cells; Kopper et al 2019 (human)12 |
| Mouse anti-VIM (vimentin) | Abcam | AB0809 | Mesenchymal Cells; Abedini et al 2020 (mouse)19 |
| Mycozap Plus-CL | Lonza | V2A-2011 | |
| N-Acetyl-L-cysteine | Sigma Aldrich | A9165 | |
| Nicotinamide | Sigma Aldrich | N0636-100G | |
| OVITRELLE-Choriogonadotropin alfa (hCG) | Merk | G03GA08 | |
| Progesterone (P4) | Sigma Aldrich | P8783 | |
| Rabbit anti-ACTA2 (alpha smooth muscle actin) | Abcam | AB5694 | Mesenchymal Cells; Abedini et al 2020 (mouse)19 |
| Rabbit anti-CDH1 (E-cadherin) | Cell Signaling | CST 3195S | Epithelial Cells; Wong et al 1999 (human)25 |
| Rabbit anti-LGR5 | Abcam | AB75850 | OSE Progenitor Cells; Flesken-Nikitin et al 2013 (mouse)22 |
| Rabbit anti-YAP | Cell Signaling | 14074S | Proliferative OSE; Wang et al 2022 (mouse)24 |
| Rat anti-CD44 PE-conjugated | eBioscience | 12-0441-81 | OSE Progenitor Cells; Bowen et al 2009 (human)20 |
| Recombinant Human Heregulinβ-1 | Peprotech | 100-03 | |
| Recombinant Human Noggin | Peprotech | 120-10C | |
| Recombinant Human Wnt3a | R&D Systems | 5036-WN-010 | |
| Recombinant Rspondin-1 | Peprotech | 120-38 | |
| Red blood cells lysis buffer | eBiosciences | 00-4333-57 | |
| Revitacell Supplement (100x) | ThermoFisher Scientific | A26445-01 | |
| RNAse free DNAse | Qiagen | 79254 | |
| SB-431542 | Tocris Bioscience | 1624/10 | |
| Sheep anti-COL1A1 (pro-collagen 1 alpha 1) | R&D Systems | AF6220 | Mesenchymal Cells; Hosper et al 2013 (human)23 |
| Y-27632 | StemCell Technologies | 72304 | |
| β-Estradiol (E2) | Sigma-Aldrich | E8875 | |
| μ-Slide 18-well culture plate | Ibidi | 8181 | Sterile |
Referenzen
- Ng, A., Barker, N. Ovary and fimbrial stem cells: biology, niche and cancer origins. Nat Rev Mol Cell Biol. 16 (10), 625-638 (2015).
- Auersperg, N., Wong, A. S., Choi, K. C., Kang, S. K., Leung, P. C. Ovarian surface epithelium: biology, endocrinology, and pathology. Endocr Rev. 22 (2), 255-288 (2001).
- Tan, O. L., Fleming, J. S. Proliferating cell nuclear antigen immunoreactivity in the ovarian surface epithelium of mice of varying ages and total lifetime ovulation number following ovulation. Biol Reprod. 71 (5), 1501-1507 (2004).
- Carter, L. E., et al. Transcriptional heterogeneity of stemness phenotypes in the ovarian epithelium. Commun Biol. 4 (1), 527 (2021).
- Chumduri, C., Turco, M. Y. Organoids of the female reproductive tract. J Mol Med (Berl). 99 (4), 531-553 (2021).
- Edmondson, R. J., Monaghan, J. M., Davies, B. R. The human ovarian surface epithelium is an androgen responsive tissue. Br J Cancer. 86 (6), 879-885 (2002).
- Karlan, B. Y., Jones, J., Greenwald, M., Lagasse, L. D. Steroid hormone effects on the proliferation of human ovarian surface epithelium in vitro. Am J Obstet Gynecol. 173 (1), 97-104 (1995).
- Nakamura, M., Katabuchi, H., Ohba, T., Fukumatsu, Y., Okamura, H. Isolation, growth and characteristics of human ovarian surface epithelium. Virchows Arch. 424 (1), 59-67 (1994).
- Horvath, P., et al. Screening out irrelevant cell-based models of disease. Nat Rev Drug Discov. 15 (11), 751-769 (2016).
- Del Valle, J. S., Chuva de Sousa Lopes, S. M. Bioengineered 3D ovarian models as paramount technology for female health management and reproduction. Bioengineering (Basel). 10 (7), 832 (2023).
- Clevers, H. Modeling development and disease with organoids. Cell. 165 (7), 1586-1597 (2016).
- Kopper, O., et al. An organoid platform for ovarian cancer captures intra- and interpatient heterogeneity. Nat Med. 25 (5), 838-849 (2019).
- Maenhoudt, N., et al. Developing organoids from ovarian cancer as experimental and preclinical models. Stem Cell Reports. 14 (4), 717-729 (2020).
- Senkowski, W., et al. A platform for efficient establishment and drug-response profiling of high-grade serous ovarian cancer organoids. Dev Cell. 58 (12), 1106-1121.e7 (2023).
- Lohmussaar, K., et al. Assessing the origin of high-grade serous ovarian cancer using CRISPR-modification of mouse organoids. Nat Commun. 11 (1), 2660 (2020).
- Zhang, S., et al. Both fallopian tube and ovarian surface epithelium are cells-of-origin for high-grade serous ovarian carcinoma. Nat Commun. 10 (1), 5367 (2019).
- Kessler, M., et al. The Notch and Wnt pathways regulate stemness and differentiation in human fallopian tube organoids. Nat Commun. 6, 8989 (2015).
- Kim, J., Koo, B. K., Knoblich, J. A. Human organoids: model systems for human biology and medicine. Nat Rev Mol Cell Biol. 21 (10), 571-584 (2020).
- Abedini, A., Sayed, C., Carter, L. E., Boerboom, D., Vanderhyden, B. C. Non-canonical WNT5a regulates Epithelial-to-Mesenchymal Transition in the mouse ovarian surface epithelium. Sci Rep. 10 (1), 9695 (2020).
- Bowen, N. J., et al. Gene expression profiling supports the hypothesis that human ovarian surface epithelia are multipotent and capable of serving as ovarian cancer-initiating cells. BMC Med Genomics. 2, 71 (2009).
- Bryant, D. M., et al. A molecular switch for the orientation of epithelial cell polarization. Dev Cell. 31 (2), 171-187 (2014).
- Flesken-Nikitin, A., et al. Ovarian surface epithelium at the junction area contains a cancer-prone stem cell niche. Nature. 495 (7440), 241-245 (2013).
- Hosper, N. A., et al. Epithelial-to-mesenchymal transition in fibrosis: collagen type I expression is highly upregulated after EMT, but does not contribute to collagen deposition. Exp Cell Res. 319 (19), 3000-3009 (2013).
- Wang, J., et al. Selective YAP activation in Procr cells is essential for ovarian stem/progenitor expansion and epithelium repair. Elife. 11, e75449 (2022).
- Wong, A. S., et al. Constitutive and conditional cadherin expression in cultured human ovarian surface epithelium: influence of family history of ovarian cancer. Int J Cancer. 81 (2), 180-188 (1999).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Shepherd, T. G., Theriault, B. L., Campbell, E. J., Nachtigal, M. W. Primary culture of ovarian surface epithelial cells and ascites-derived ovarian cancer cells from patients. Nat Protoc. 1 (6), 2643-2649 (2006).
- Xu, J., Lamouille, S., Derynck, R. TGF-beta-induced epithelial to mesenchymal transition. Cell Res. 19 (2), 156-172 (2009).
- Miettinen, P. J., Ebner, R., Lopez, A. R., Derynck, R. TGF-beta induced transdifferentiation of mammary epithelial cells to mesenchymal cells: involvement of type I receptors. J Cell Biol. 127 (6 Pt 2), 2021-2036 (1994).
- Danielpour, D., et al. Sandwich enzyme-linked immunosorbent assays (SELISAs) quantitate and distinguish two forms of transforming growth factor-beta (TGF-beta 1 and TGF-beta 2) in complex biological fluids. Growth Factors. 2 (1), 61-71 (1989).
- Oida, T., Weiner, H. L. Depletion of TGF-beta from fetal bovine serum. J Immunol Methods. 362 (1-2), 195-198 (2010).
- Halder, S. K., Beauchamp, R. D., Datta, P. K. A specific inhibitor of TGF-beta receptor kinase, SB-431542, as a potent antitumor agent for human cancers. Neoplasia. 7 (5), 509-521 (2005).
- Wang, J., Wang, D., Chu, K., Li, W., Zeng, Y. A. Procr-expressing progenitor cells are responsible for murine ovulatory rupture repair of ovarian surface epithelium. Nat Commun. 10 (1), 4966 (2019).
- Kawata, M., et al. Polarity switching of ovarian cancer cell clusters via SRC family kinase is involved in the peritoneal dissemination. Cancer Sci. 113 (10), 3437-3448 (2022).
- Davies, B. R., Worsley, S. D., Ponder, B. A. Expression of E-cadherin, alpha-catenin and beta-catenin in normal ovarian surface epithelium and epithelial ovarian cancers. Histopathology. 32 (1), 69-80 (1998).
- Skory, R. M., Xu, Y., Shea, L. D., Woodruff, T. K. Engineering the ovarian cycle using in vitro follicle culture. Hum Reprod. 30 (6), 1386-1395 (2015).
- Boretto, M., et al. Patient-derived organoids from endometrial disease capture clinical heterogeneity and are amenable to drug screening. Nat Cell Biol. 21 (8), 1041-1051 (2019).
- Phan, N., et al. A simple high-throughput approach identifies actionable drug sensitivities in patient-derived tumor organoids. Commun Biol. 2, 78 (2019).
- Ducie, J., et al. Molecular analysis of high-grade serous ovarian carcinoma with and without associated serous tubal intra-epithelial carcinoma. Nat Commun. 8 (1), 990 (2017).
- Lee, Y., et al. A candidate precursor to serous carcinoma that originates in the distal fallopian tube. J Pathol. 211 (1), 26-35 (2007).
- Rezanejad, H., Lock, J. H., Sullivan, B. A., Bonner-Weir, S. Generation of pancreatic ductal organoids and whole-mount immunostaining of intact organoids. Curr Protoc Cell Biol. 83 (1), e82 (2019).
- Asseler, J. D., et al. One-third of amenorrheic transmasculine people on testosterone ovulate. Cell Rep Med. 5 (3), 101440 (2024).
- Ikeda, K., et al. Excessive androgen exposure in female-to-male transsexual persons of reproductive age induces hyperplasia of the ovarian cortex and stroma but not polycystic ovary morphology. Hum Reprod. 28 (2), 453-461 (2013).
- De Roo, C., et al. Texture profile analysis reveals a stiffer ovarian cortex after testosterone therapy: a pilot study. J Assist Reprod Genet. 36 (9), 1837-1843 (2019).
- Grimstad, F. W., et al. Ovarian histopathology in transmasculine persons on testosterone: A multicenter case series. J Sex Med. 17 (9), 1807-1818 (2020).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten