Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Identifizierung ruhender Zellen in einem Zebrafisch-T-Zell-Modell für akute lymphatische Leukämie mittels Zellproliferationsfärbung
In diesem Artikel
Zusammenfassung
Wir verwendeten die Zellproliferationsfärbung, um ruhende Zellen im Zebrafisch-T-Modell für akute lymphatische Leukämie zu identifizieren. Die Färbung wird in nicht teilenden Zellen zurückgehalten und während der Zellproliferation reduziert, was die Selektion ruhender Zellen für die weitere Befragung ermöglicht. Dieses Protokoll bietet ein funktionales Werkzeug, um die Selbsterneuerung im Kontext der zellulären Ruhe zu untersuchen.
Zusammenfassung
Zelluläre Ruhe ist ein Zustand des Wachstumsstillstands oder der verlangsamten Proliferation, der bei normalen und Krebsstammzellen (CSCs) beschrieben wird. Quiescence kann CSCs vor antiproliferativen Chemotherapeutika schützen. In T-Zell-Mausmodellen für akute lymphatische Leukämie (T-ALL) mit patientenabgeleitetem Xenotransplantat (PDX) sind ruhende Zellen mit Behandlungsresistenz und Stammzellen assoziiert. Zellproliferationsfarbstoffe sind beliebte Werkzeuge zur Verfolgung der Zellteilung. Der Fluoreszenzfarbstoff ist kovalent in Amingruppen auf der Membran und Makromolekülen im Inneren der Zelle verankert. Dies ermöglicht die Verfolgung markierter Zellen für bis zu 10 Teilungen, die durch Durchflusszytometrie aufgelöst werden können.
Letztendlich haben Zellen mit den höchsten Proliferationsraten eine geringe Farbstoffretention, da sie bei jeder Zellteilung verdünnt wird, während ruhende, sich langsamer teilende Zellen die höchste Retention aufweisen. Die Verwendung von Zellproliferationsfarbstoffen zur Isolierung ruhender Zellen wurde in T-ALL-Mausmodellen optimiert und beschrieben. Ergänzend zu den bestehenden Mausmodellen bietet das rag2:Myc-abgeleitete Zebrafisch-T-ALL-Modell aufgrund der hohen Häufigkeit leukämischer Stammzellen (LSCs) und der Bequemlichkeit des Zebrafisches für groß angelegte Transplantationsexperimente einen hervorragenden Ort, um die Selbsterneuerung bei T-ALL zu untersuchen.
Hier beschreiben wir den Arbeitsablauf für die Färbung von Zebrafisch-T-ALL-Zellen mit einem Zellproliferationsfarbstoff, die Optimierung der Konzentration des Farbstoffs für Zebrafischzellen, die Passage erfolgreich gefärbter Zellen in vivo und die Entnahme von Zellen mit unterschiedlicher Farbstoffretention durch Sortierung von lebenden Zellen aus transplantierten Tieren. Angesichts des Fehlens gut etablierter Zelloberflächenhersteller für LSCs in T-ALL bietet dieser Ansatz ein funktionelles Mittel, um ruhende Zellen in vivo zu befragen. Für repräsentative Ergebnisse beschreiben wir die Transplantationseffizienz und die LSC-Häufigkeit von Zellen mit hoher und niedriger Farbstoffretention. Diese Methode kann helfen, zusätzliche Eigenschaften ruhender Zellen zu untersuchen, einschließlich des Wirkstoffansprechens, der Transkriptionsprofile und der Morphologie.
Einleitung
Adulte Stammzellen sind für die Regeneration differenzierter Zelltypen in einem bestimmten Organ verantwortlich und liegen überwiegend in einem ruhenden, sich nicht teilenden Zustand vor 1,2. So bleiben beispielsweise hämatopoetische Stammzellen (HSCs), die das Blut erhalten, weitgehend ruhig, und nur ein kleiner Teil gelangt in den Zellzyklus, um sich selbst zu erneuern oder zu differenzieren, um reife Blutbestandteile zu erzeugen3. In ähnlicher Weise besitzt bei Krebserkrankungen eine seltene Subpopulation von Zellen, die als Krebsstammzellen (CSCs) bezeichnet werden, die Fähigkeit zur Selbsterneuerung und sind für die langfristige Aufrechterhaltung der Malignität verantwortlich4. Krebsstammzellen existieren in vivo in einem Zustand der Ruhe oder des langsamen Wachstums, was es ihnen ermöglichen könnte, den antiproliferativen Krebsbehandlungen zu entkommen5, sich der Beseitigung durch das Immunsystem zu entziehen6, oxidativen Stress zu reduzieren und ihre DNA-Reparaturwegezu verbessern 7. Selbst eine geringe Anzahl von CSCs, die nach der Behandlung zurückbleiben, kann den Tumor möglicherweise wieder besiedeln, was zu einem Rückfall des Patienten führt8. Dementsprechend ist das Verständnis der zellulären Stille vielversprechend für die Identifizierung potenzieller Schwachstellen von CSCs und die Entwicklung neuer Wege, diese zu bekämpfen.
Zellproliferationsfarbstoffe wie der Carboxyfluorescein-Succinimidylester (CFSE)-Farbstoff und seine Derivate werden häufig verwendet, um die Häufigkeit von Zellteilungen zu verfolgen9. Der Farbstoff dringt in die Zellmembran ein und wird im Inneren der Zelle durch intrazelluläre Esterasen zu einem fluoreszierenden Produkt aktiviert. Die resultierende fluoreszierende Verbindung wird durch die kovalenten Amidbindungen, die zwischen der Succinimidyleinheit und den funktionellen Amingruppen intrazellulärer Proteine gebildet werden, in der Zelle zurückgehalten10. Bei jeder Zellteilung wird die fluoreszierende Verbindung gleichmäßig auf die beiden resultierenden Zellen aufgeteilt, was zu einer zweifachen Signalverdünnung führt. Dieser Farbstoff ermöglicht den Nachweis von bis zu 10 Zellteilungen durch durchflusszytometrische Analyse11.
Dieser Ansatz wurde bereits verwendet, um CSC-Populationen in vitro anzureichern, indem langsam zyklische Populationen von Zellen mit hoher Retention des Farbstoffs identifiziertwurden 11,12. Bei der T-ALL wurde CFSE verwendet, um das Tumorwachstum in vivo in patienteneigenen Xenotransplantaten bei Mäusen zu verfolgen. Nach der Zellmarkierung und einer dreiwöchigen Transplantation zeigte die Durchflusszytometrie-Analyse eine seltene Population von Zellen, die noch CFSE-Fluoreszenz behielten. Diese Population war bei Patienten mit Stammzellen, Behandlungsresistenz und hoher Ähnlichkeit mit schubverursachenden Zellen assoziiert13. Dementsprechend stellt dieser Farbstoff ein nützliches Werkzeug für die Untersuchung von Leukämie-Stammzell-Phänotypen (LSC) bei T-ALL dar.
Ziel der Arbeit ist es, die Anwendung des Zellproliferationsfarbstoffs auf die Untersuchung der Stille in vivo mit Hilfe eines Zebrafisch-T-ALL-Modells auszuweiten. Insbesondere das rag2:Myc-getriebene Zebrafisch-T-ALL-Modell14 bietet aufgrund der hohen Häufigkeit von LSCs im Vergleich zu Mausmodellen und menschlichen Krankheiten15 einen hervorragenden Ort für die Untersuchung der Selbsterneuerung. Darüber hinaus ermöglicht die Verwendung von Zebrafischen groß angelegte Transplantationsstudien, die im Vergleich zu ihren Gegenstücken bei Mäusen mit wesentlich geringeren Pflege- und Wartungskosten durchgeführt werdenkönnen 16. Zebrafische eignen sich auch hervorragend für Live-Imaging-Anwendungen, da fluoreszenzmarkierte Tumorzellen mit einem einfachen Fluoreszenzmikroskop leicht betrachtet werden können, um die Rate der Tumorentwicklung abzuschätzen16.
In diesem Protokoll beschreiben wir den Arbeitsablauf für die Färbung von T-ALL-Zellen von Zebrafischen mit Zellproliferationsfarbstoff, gefolgt von der in vivo Vermehrung von gefärbten Zellen in syngenen CG1-Zebrafischen. Bei der Entwicklung von Leukämie beschreiben wir die Sortierung der Zellen, die den Farbstoff zurückhielten, und ihre Verwendung für ein anschließendes Transplantationsexperiment zur Begrenzung der Verdünnung, um die Raten der LSC-Selbsterneuerung zu quantifizieren. Dieses Protokoll kann für weitere Anwendungen erweitert werden, einschließlich des In-vivo-Wirkstoffscreenings potenzieller Verbindungen für das Targeting von ruhenden LSCs. Darüber hinaus können gesammelte Zellen für verschiedene nachgelagerte Analysen verwendet werden, wie z. B. Transkriptomisches Profiling, Proteomik und Metabolomik, was einzigartige Einblicke in das Verhalten ruhender LSCs bei T-ALL bietet.
Protokoll
In diesem Protokoll verwenden wir GFP-markierte Zebrafisch-T-ALL-Zellen, die zuvor im CG1-Stamm erzeugt wurden und daher direkt in den syngenen CG1-Zebrafisch15 des Empfängers injiziert werden können. Kurz gesagt, Leukämie wurde durch DNA-Mikroinjektion von rag2:Myc und rag2:GFP in einzellige CG1-Zebrafischembryonen erzeugt. Die Tiere wurden ab 3 Wochen nach der Injektion mittels Fluoreszenzmikroskopie auf die Entwicklung von Leukämie überwacht. GFP-positive Leukämiezellen wurden FACS isoliert und seriell in den Empfänger-CG1-Zebrafisch transplantiert, um Klone mit einer hohen LSC-Häufigkeit zu erzeugen. Einzelheiten des gesamten Protokolls werden von Blackburn et al.15 beschrieben.
Alternativ kann primäres rag2:Myc-abgeleitetes T-ALL durch DNA-Mikroinjektion von Zebrafischembryonen erzeugt werden17. Die DNA-Mikroinjektion von rag2:Myc mit einem rag2-gesteuerten fluoreszierenden Reporter wie GFP kann zur Entwicklung von B-Zellen, T-Zellen und gemischtem ALL18 führen. Die in diesem Protokoll verwendeten Leukämie-Klone wurden zuvor als T-ALL15 verifiziert. Alle Versuchsverfahren mit Zebrafischen wurden vom Institutional Animal Care and Use Committee der University of Kentucky, Protokoll 2019-3399, überprüft und genehmigt.
1. Vermehrung von Zebrafisch-T-ALL-Zellen in vivo
- Tauen Sie eine Durchstechflasche mit 1 ml gefrorener GFP-markierter Zebrafisch-T-ALL-Zellen in einem 37 °C Wasserbad unter leichtem Schütteln auf, bis sie aufgetaut ist. Nach dem Auftauen auf Eis legen.
- Pipettieren Sie den Inhalt des Röhrchens langsam in ein konisches 15-ml-Röhrchen mit 4 mL Fischmedium (0,9x PBS + 5 % FBS), um das Gefriermedium zu verdünnen.
- 5 min bei 2.500 × g bei 4 °C runterschleudern. Entfernen Sie das Medium aus dem Pellet. In 0,5 ml Fischmedium resuspendieren und die Zellen auf Eis halten.
- Zählen Sie die Zellen und verdünnen Sie sie bei Bedarf auf 100.000 Zellen/5 μl.
- Betäuben Sie den erwachsenen CG1-Zebrafisch, indem Sie 200 μl 4 mg/ml-Anästhetikum (MS-222) zu 25 ml Fischsystemwasser in einer Petrischale hinzufügen. Halten Sie den CG1-Fisch mit der Bauchseite nach oben und injizieren Sie 5-6 μl mit einer Hamilton-Mikroliterspritze in den intraperitonealen (IP) Raum. Bringen Sie die Tiere zurück in ihre Becken und überwachen Sie, ob sie wieder normal schwimmen können, bevor Sie sie wieder in das System setzen.
HINWEIS: Für die in diesem Protokoll beschriebenen Transplantationsstudien wurden adulte Zebrafische, die 60 Tage oder älter waren, verwendet. - Überwachen Sie das Leukämiewachstum einmal pro Woche mit einem Fluoreszenzmikroskop. Einzelheiten zum Screening-Verfahren finden Sie in Abschnitt 2 unten.
2. Ernte von fluoreszenzmarkierten Leukämiezellen
- Etwa 21-28 Tage nach der Transplantation betäuben Sie den erwachsenen CG1-Zebrafisch, indem Sie 200 μl (4-5 Tropfen) 4 mg/ml Anästhetikum zu 25 ml Wasser aus dem Fischsystem in einer Petrischale hinzufügen.
- Untersuchen Sie den CG1-Zebrafisch auf Leukämielast, indem Sie den Prozentsatz der GFP-markierten Leukämiezellen im Tier mit einem Epifluoreszenzmikroskop bestimmen und den entsprechenden Filter zum Nachweis der GFP-Fluoreszenz verwenden. Die GFP-positive Leukämie ist erntereif, wenn sie sich über >70 % des Tieres ausgebreitet hat.
- Euthanasieren Sie das Tier, indem Sie 1 ml 4 mg/ml Anästhetikum in eine Petrischale mit 9 ml Wasser aus dem Fischsystem geben. Lass alle Zeichen des Lebens aufhören; Beobachten Sie zum Beispiel den Fisch auf die Beendigung der Operkulärbewegung (~5 min).
- Setzen Sie den Zebrafisch in eine neue Petrischale und fügen Sie 1 ml Fischmedium hinzu, das als Puffer für die Zellen dient. Mit einer neuen Rasierklinge zuerst den Zebrafisch enthaupten, dann das Gewebe mit der gleichen Rasierklinge mazerieren. Pipettieren Sie auf und ab, um große Zellklumpen zu entfernen.
- Die Zellen werden durch ein 40-μm-Zellsieb in ein konisches 50-ml-Röhrchen geleitet, um in eine Einzelzellsuspension zu dissoziieren. Halten Sie die Zellen auf Eis.
3. Optimierung der Konzentration des Zellproliferationsfarbstoffs (CellTrace Far Red) für die Färbung und Lebensfähigkeit der Zebrafischzellen
HINWEIS: Für dieses Protokoll und da die Tumorzellen GFP-markiert sind, haben wir einen Proliferationsfarbstoff für fernrote Blutkörperchen (CellTrace Far Red) verwendet, um eine spektrale Überlappung zu vermeiden, der als CT-FR bezeichnet wird.
- Bereiten Sie unmittelbar vor der Verwendung eine 1 mM Lösung des Fluoreszenzfarbstoffs vor, indem Sie die entsprechende Menge DMSO gemäß den Empfehlungen des Herstellers hinzufügen. Arbeitslösungen von 1 mM, 0,5 mM, 0,1 mM und 0,01 mM; Bei Bedarf in Fischmedien verdünnen, um die Lebensfähigkeit der Zellen nach der Färbung zu testen.
- Zählen Sie die Zellen und sammeln Sie 1 × 106 Zellen in 1,5-ml-Mikrozentrifugenröhrchen.
- Bei 2.500 × g bei 4 °C 5 min zentrifugieren. Entfernen Sie die Flüssigkeit und resuspendieren Sie die Zellen in 1 mL 0,9x PBS.
- Die Zellsuspension wird für die Behandlung mit CT-FR auf vier Röhrchen à 250 μl aufgeteilt.
- Fügen Sie 250 μl 0,9x PBS hinzu, um das Volumen auf jeweils 500 μl mit ca. 250.000 Zellen/Röhrchen zu erhöhen.
- Geben Sie die erforderlichen Mengen der oben beschriebenen Arbeitslösungen in jedes der vier Röhrchen, um die folgenden Endkonzentrationen zu erreichen: 1 μM, 0,5 μM, 0,1 μM und 0,05 μM. Inkubieren Sie 20 Minuten lang bei 37 °C unter Lichtschutz.
- Pelletieren Sie die Zellen, indem Sie sie 5 Minuten lang bei Raumtemperatur bei 2.500 × g herunterschleudern. Entfernen Sie die Flüssigkeit und waschen Sie die Zellen mit 500 μl Fischmedium, um den überschüssigen Farbstoff zu entfernen.
- Erneut pelletieren, die Flüssigkeit entfernen und in 25 μl Fischmedium resuspendieren. Verwenden Sie 1 μl, um Zellen in einer Verdünnung von 1:10 in 50 % Trypanblau zu zählen.
- Untersuchen Sie die Zellen unter dem Mikroskop auf Zellviabilität; Lebende Zellen behalten das Trypanblau nicht bei und erscheinen unter dem Mikroskop hell, während tote Zellen den Farbstoff aufnehmen und dunkel erscheinen. Untersuchen Sie die Zellen zunächst unter dem entsprechenden Fluoreszenzkanal, um Tumorzellen zu identifizieren, und verwenden Sie dann den Hellfeldkanal, um die Lebensfähigkeit der Zellen zu beurteilen.
- Wählen Sie die Konzentration, die zu einer erfolgreichen Färbung bei gleichzeitiger Aufrechterhaltung der Zellviabilität führt (>90 % Trypanblau-negative Zellen).
HINWEIS: Für die in diesem Experiment getesteten Zebrafisch-T-ALL-Zellen wurde eine Konzentration von 1 μM für eine erfolgreiche Färbung und >90 % der Zellviabilität verwendet.
4. Vermehrung von CT-FR-gefärbten T-ALL-Zebrafischzellen in vivo
- Mit der optimierten CT-FR-Konzentration aus Sektion 3 werden die Zebrafischzellen gefärbt. Lassen Sie einige Tumorzellen für die Erzeugung des GFP-markierten T-ALL-Tumors ungefärbt, als GFP-positive Kontrolle während der FACS-Sortierung. Verdünnen Sie sowohl gefärbte als auch ungefärbte Zellen auf 50.000 Zellen/5 μl für die Transplantation.
- Betäuben Sie den erwachsenen CG1-Zebrafisch, indem Sie 200 μl 4 mg/ml-Anästhetikum zu 25 ml Fischsystemwasser in einer Petrischale hinzufügen.
- Injizieren Sie 5 μl der Zellsuspension in den IP-Hohlraum des Zebrafisches, wie in Abschnitt 1 beschrieben, sowohl für die gefärbten als auch für die ungefärbten Zellen. Injizieren Sie 5-6 Zebrafische pro Gruppe, um die potenzielle Sterblichkeit nach der Injektion zu berücksichtigen.
- Untersuchen Sie die Tiere durch Fluoreszenzmikroskopie unter Sedierung 2x wöchentlich auf die Entwicklung von Leukämien.
HINWEIS: Nach ~10-14 Tagen sollte die Leukämie etwa 30-40% dieses Körpers einnehmen. Planen Sie die Leukämieernte und die anschließende Sortierung innerhalb dieses Zeitrahmens. Das Warten auf die Ausdehnung des Tumors auf eine große Größe kann zu einer stärkeren Verdünnung des fluoreszierenden Zellfarbstoffs und zu Schwierigkeiten beim Nachweis seines Signals führen. Erwägen Sie die Optimierung des Zeitpunkts der Tumorentnahme für das gewählte Tumormodell. Darüber hinaus empfiehlt sich hier ein häufigeres Tumormonitoring, um die Erfassung der gewünschten Tumorgröße zu gewährleisten.
5. Vorbereitung der Sortierung von CT-FR-gefärbten Zebrafisch-T-ALL-Zellen
- Bereiten Sie die folgenden Kontrollen für die Zellsortierung vor: Keine Farbkontrolle (ungefärbte, nicht-transgene, einzellige Suspension, von einem Wildtyp-Zebrafisch), GFP-markierte T-ALL-Zellen und neu gefärbte CT-FR WT-Zebrafischzellen (zum Festlegen eines maximalen Schwellenwerts für das fernrote Signal).
- Vorbereitung von ungefärbten und CT-FR-Kontrollen
- Ernten Sie Wildtyp-Fischzellen (WT), indem Sie einen nicht transplantierten CG1-Zebrafisch einschläfern, indem Sie 1 ml 4 mg/ml Anästhetikum in eine Petrischale mit 9 ml Wasser des Fischsystems geben.
- Setzen Sie den Zebrafisch in eine neue Petrischale und fügen Sie 1 ml Fischmedium hinzu, das als Puffer für die Zellen dient. Verwenden Sie eine neue Rasierklinge, um den Zebrafisch zuerst zu enthaupten und dann mit derselben Rasierklinge zu mazerieren. Pipettieren Sie auf und ab, um große Zellklumpen zu entfernen.
- Die Zellen werden durch ein 40-μm-Zellsieb in ein konisches 50-ml-Röhrchen geleitet, um in eine Einzelzellsuspension zu dissoziieren. Halten Sie die Zellen auf Eis.
- Für die CT-FR-Steuerung werden 250.000 WT-Zellen in ein Mikrozentrifugenröhrchen überführt und die Zellen durch Zentrifugation bei 2.500 × g für 5 min bei Raumtemperatur pelletiert. Entfernen Sie das Fischmedium und fügen Sie 500 μl 0,9x PBS + 1% FBS hinzu.
- Fügen Sie 0,5 μl der 1 mM CT-FR Farbstoff-Stammlösung hinzu, um eine Endkonzentration von 1 μM zu erreichen. Pipettieren Sie vorsichtig mit 1.000 μl Pipette zum Mischen. Bei 37 °C 20 min lichtgeschützt inkubieren.
- Entfernen Sie die Flüssigkeit und waschen Sie die Zellen mit 500 μl Fischmedium, um den überschüssigen Farbstoff zu entfernen.
- Pelletieren Sie die Zellen erneut durch Zentrifugation, entfernen Sie die Flüssigkeit und resuspendieren Sie die Zellen in 500 μl 0,9x PBS+ 1% FBS und geben Sie sie durch eine 35 μm Filterkappe in ein FACS-Röhrchen. Bleiben Sie auf Eis.
- Vorbereitung der Spenderplatte
HINWEIS: Bei der Spenderplatte handelt es sich um eine 96-Well-V-Bodenplatte, in die die CT-FR-gefärbten T-ALL-Zellen von transplantierten Zebrafischen einsortiert werden. Jede Vertiefung der Platte enthält 5.000 ungefärbte WT CG1-Zellen in 50 μl Fischmedium, die als Träger dienen, um die sortierten Zellen in den nachfolgenden Schritten zu pelletieren.- Zählen Sie 500.000 WT CG1-Zellen und überführen Sie sie in ein konisches 15-ml-Röhrchen.
- Verdünnen Sie diese Zellbrühe auf 100 Zellen/μl, indem Sie 5 mL Fischmedium hinzufügen.
- Geben Sie 50 μl der Zellsuspension in jede Vertiefung der 96-Well-Platte. Mischen Sie das Röhrchen aus dem Zellsuspensionsmaterial nach jeder Reihe, um die Homogenität der Zellsuspension zu erhalten. Bewahren Sie die Platte bis zum Sortieren bei 4 °C auf.
- Aufbereitung der transplantierten Fischproben
- Wählen Sie umgepflanzte Fische mit 30-40% T-ALL-Belastung aus. Sammeln Sie Fische, die sowohl mit den CT-FR-gefärbten Zellen als auch mit ungefärbten GFP-Tumorzellen transplantiert wurden.
- Ernten Sie Zellen wie in Abschnitt 2 beschrieben.
HINWEIS: Da davon auszugehen ist, dass die Farbstoff-Retentionszellen in geringer Menge vorhanden sind, bereiten Sie sich darauf vor, eine große Anzahl von Zellen aus der CT-FR GFP-Probe zu sortieren. - In geeigneter Konzentration für die Sortierung von ~3-5 × 106 Zellen/ml in 0,9x PBS + 1% FBS verdünnen und durch eine 35 μm Filterkappe in ein FACS-Röhrchen geben. Fügen Sie DAPI (1 mg/ml) in einer Verdünnung von 1:1.000 als lebenswichtigen Farbstoff hinzu, um abgestorbene Zellen auszuschließen. Bleiben Sie auf Eis.
- Für die ungefärbten GFP-Tumorzellen werden 250.000 Zellen in ein Mikrozentrifugenröhrchen überführt und bei 2.500 × g für 5 min bei Raumtemperatur pelletiert. Entfernen Sie das Fischmedium, fügen Sie 500 μl PBS hinzu und führen Sie es durch eine 35-μm-Filterkappe in ein FACS-Röhrchen. Bleiben Sie auf Eis.
6. Sortierung
HINWEIS: Bewahren Sie alle Röhrchen und die Spenderplatte die ganze Zeit auf Eis auf, außer wenn sie zum Sortieren verwendet werden.
- Beginnen Sie zum Sortieren von Parametern und Gates mit der Anwendung des Vorwärts- und Seitenstreu-Gatings, um die interessierenden Zellen anhand ihrer Größe zu identifizieren und Zelltrümmer zu eliminieren. Verwenden Sie als Nächstes die Vorwärts-Streuhöhe im Vergleich zum Vorwärts-Streubereich, um doppelte Zellen auszuschließen und nur Singuletts beizubehalten. Verwenden Sie abschließend die No-Color- und Single-Color-Steuerelemente, um die Quadranten zu zeichnen, um GFP-positive und CT-FR-positive Zellpopulationen zu identifizieren.
HINWEIS: Es wird erwartet, dass die CT-FR-Kontrolle viel heller ist als die zu sortierenden Proben, da die Färbung für die Kontrolle frisch und ohne Zellteilung durchgeführt wurde. Es wird erwartet, dass Zellen aus den CT-FR-GFP-Proben am unteren Ende des positiven CT-FR-Gates erscheinen. - Sortieren Sie die Anzahl der Zellen, die in jeden Fisch transplantiert werden sollen, in eine Vertiefung der Spenderplatte.
HINWEIS: Für dieses Experiment haben wir einen limitierenden Verdünnungsassay verwendet, um die Häufigkeit von Leukämiestammzellen in CT-FR High und CT-FR Low abzuschätzen. Bei diesem Assay wurde eine Dosis von 25 Zellen verwendet, die in 3 Tiere transplantiert wurden, und eine Dosis von 10 Zellen, die in 10 Tiere transplantiert wurden. Ein typischer limitierender Verdünnungsassay mit dem Zebrafisch rag2:Myc-abgeleiteten T-ALL würde eine Gruppe mit einer höheren Anzahl von Zellen (500 oder 1.000) einbeziehen, da die LSC-Häufigkeit in diesem Modell auf 1 LSC in Hunderten von Zellen geschätzt wird. In diesem Assay verwenden wir jedoch einen der T-ALL-Klone, die zuvor von Blackburn et al.15 beschrieben wurden. Die LSC-Frequenz in der hier verwendeten Probe beträgt ~1 LSC in Dutzenden von Zellen; Wir prognostizieren, dass Leukämie bei Tieren nachgewiesen wird, die in der 25-Zell-Gruppe transplantiert wurden. Bei der Optimierung dieses Assays für Ihr Krankheitsmodell sollte bei der Auswahl der Gruppe mit höherer Zellzahl darauf geachtet werden, dass die Transplantation des Tumors sichergestellt ist. - Bewahren Sie die Platte nach dem Sortieren auf Eis auf, bis sie zum Umpflanzen bereit ist.
HINWEIS: Sortiervorgänge können stressig sein und die Lebensfähigkeit der Zellen beeinträchtigen. Vor der Durchführung des eigentlichen Verdünnungsversuchs wurde ein Pilot-Sortierversuch durchgeführt. Sortierte Zellen wurden transplantiert und die Leukämieentwicklung überwacht, um festzustellen, ob die Sortierung der Lebensfähigkeit der Zellen abträglich ist. Alternativ kann die Zellviabilität beurteilt werden, indem die sortierten Zellen mit Trypanblau gefärbt und vor der Transplantation unter dem Mikroskop untersucht werden.
7. Transplantation sortierter Zellen zur Begrenzung der Verdünnung und Verfolgung des Tumorbeginns
- Schleudern Sie die Platte mit einer großen Tischzentrifuge bei 2.500 × g bei 4 °C herunter.
- Entfernen Sie vorsichtig 45 μl des Überstands aus jeder Vertiefung, lassen Sie 5 μl Flüssigkeit zurück und achten Sie darauf, das Zellpellet am Boden der Vertiefung nicht zu berühren.
- Resuspendieren Sie die Zellen mit einer 20-μl-Pipette. Resuspendieren Sie 2-3 Vertiefungen mit der Pipette und injizieren Sie dann mit der Hamilton-Mikroliterspritze die Zellsuspensionen aus jeder Vertiefung per IP-Transplantation in die gewünschte Anzahl von CG1-Zebrafischen. Suspendieren Sie dann die Zellen in der nächsten Well-Gruppe, um ein Wiederansiedeln der Zellen zu vermeiden.
- Untersuchen Sie den Zebrafisch durch Fluoreszenzmikroskopie unter Sedierung einmal pro Woche für mindestens 6 Wochen auf die T-ALL-Entwicklung.
8. Bestimmung der Leukämie-Stammzellfrequenz
- Notieren Sie die Anzahl positiver Tiere pro Zelldosis und Behandlungsgruppe 6 Wochen nach der Transplantation, wobei Tiere mit einer beliebigen Menge an GFP-Signalen als positiv betrachtet werden. Betrachten Sie Tiere als signifikant leukämielastig, wenn das GFP-Signal >50 % des Körpers einnimmt.
- Verwenden Sie die webbasierte Statistiksoftware ELDA (Extreme Limiting Dilution Analysis) (http://bioinf.wehi.edu.au/software/elda/)19, um die Häufigkeit von LSCs innerhalb jeder Zellpopulation zu berechnen.
HINWEIS: Eine schematische Darstellung des Arbeitsablaufs ist in Abbildung 1 dargestellt.
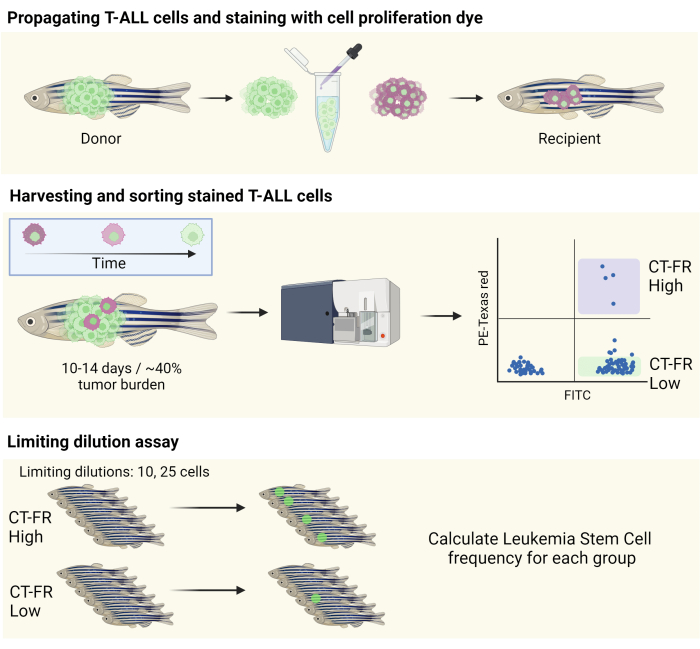
Abbildung 1: Arbeitsablauf für die Verwendung des Zellverfolgungsfärbes zur Isolierung ruhender Zellen im Zebrafisch-T-ALL-Modell. Schematische Darstellung der Färbung von Zebrafisch-T-ALL-Zellen mit der Zellproliferationsfärbung, der Vermehrung in einen CG1-Zebrafisch (oberes Bild) und der Sortierung der Zellen basierend auf der Retention des Zellproliferationsflecks (mittleres Bild). Die gesammelten Zellen wurden für einen limitierenden Verdünnungsassay verwendet, um die Häufigkeit von LSCs zu bestimmen (unteres Bild). Abkürzungen: T-ALL = T-Zell-akute lymphatische Leukämie; LSCs = leukämische Stammzellen; CT-FR = CellTrace Far Red; FITC = Fluorescein-Isothiocyanat; PE = Phycoerythrin. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergebnisse
Wir folgten dem oben beschriebenen Protokoll, um Zellen zu sortieren, die den Zellproliferationsfarbstoff CT-FR beibehalten haben, und verwendeten sie für einen Limiting Dilution Assay (LDA) zur Abschätzung der LSC-Häufigkeit in den CT-FR High- und CT-FR Low-Populationen. Um das Gating für das Durchflusszytometrie-Experiment einzustellen, verwendeten wir zusätzlich zu den einfarbigen Kontrollen eine Kontrolle ohne Fluorophor (ohne Farbe ) (Abbildung 2A
Diskussion
Es ist bekannt, dass LSCs resistent gegen konventionelle, antiproliferative Chemotherapien sind, und die Suche nach gezielten Therapien gegen diese Zellen ist vielversprechend, um das Auftreten von Rückfällen zu reduzieren und die Patientenprognose zu verbessern20. Frühere Forschungen beschrieben die Verwendung von fluoreszierenden Zellproliferationsfärbungen zur Identifizierung einer kleinen Population ruhender Zellen, die mit Arzneimittelresistenz und Stammz...
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Die Finanzierung dieser Forschung erfolgte durch das National Cancer Institute (R37CA227656 to JSB). Diese Forschung wurde auch durch die gemeinsamen Ressourcen für Durchflusszytometrie und Immunüberwachung des Markey Cancer Center (P30CA177558) der University of Kentucky unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 26 G/2” micro-syringe | Hamilton | 87930 | NA |
| 35 µm filter cap FACS tubes | Falcon | 352235 | NA |
| 40 µm cell strainer | CELLTREAT | 229482 | NA |
| 96-well skirted PCR plate | Thermo Fisher Scientific | AB0800 | NA |
| Cell sorter | Sony Biotechnology | SY3200 | NA |
| CellTrace Far Red | Thermo Fisher Scientific | C34564 | NA |
| Conical tubes | VWR | 10026-078 | NA |
| DAPI | Thermo Fisher Scientific | 62248 | NA |
| DMSO | Sigma-Aldrich | D4818 | NA |
| Dulbecco'sPhosphate-buffered saline (PBS) | Caisson Labs | 22110001 | NA |
| Epifluorescence stereo microscope | Nikon | SMZ25 | NA |
| Fetal Bovine Serum (FBS) | Sigma-Aldrich | 12306C | NA |
| Fish system water | N/A | N/A | 0.03-0.05% salinity, pH 6.5-8, buffered with sodium bicarbonate |
| Microcentrifuge tubes | Thermo Fisher Scientific | C2171 | NA |
| MS-222 | Pentaire | TRS-1 | tricaine mesylate, an anesthetic |
| Petri dishes | Corning | 07-202-011 | NA |
| Razor blades | American Line | 66-0089 | NA |
| Trypan Blue | Thermo Fisher Scientific | T10282 | NA |
Referenzen
- Reya, T., Morrison, S. J., Clarke, M. F., Weissman, I. L. Stem cells, cancer, and cancer stem cells. Nature. 414 (6859), 105-111 (2001).
- Arai, F., et al. Tie2/angiopoietin-1 signaling regulates hematopoietic stem cell quiescence in the bone marrow niche. Cell. 118 (2), 149-161 (2004).
- Li, L., Bhatia, R. Stem cell quiescence. Clin. Cancer Res. 17 (15), 4936-4941 (2011).
- Kreso, A., Dick, J. E. Evolution of the cancer stem cell model. Cell Stem Cell. 14 (3), 275-291 (2014).
- Chen, W., Dong, J., Haiech, J., Kilhoffer, M. -. C., Zeniou, M. Cancer stem cell quiescence and plasticity as major challenges in cancer therapy. Stem Cells Int. 2016, 1740936 (2016).
- Kleffel, S., Schatton, T. Tumor dormancy and cancer stem cells: Two sides of the same coin. Adv Exp Med Biol. 734, 145-179 (2013).
- Tuy, K., Rickenbacker, L., Hjelmeland, A. B. Reactive oxygen species produced by altered tumor metabolism impacts cancer stem cell maintenance. Redox Biol. 44, 101953 (2021).
- Zhou, B. -. B. S., et al. Tumour-initiating cells: Challenges and opportunities for anticancer drug discovery. Nat Rev Drug Discov. 8 (10), 806-823 (2009).
- Lyons, A. B., Blake, S. J., Doherty, K. V. Flow cytometric analysis of cell division by dilution of cfse and related dyes. Curr Protoc Cytom. 64 (1), 11-12 (2013).
- Lyons, A. B. Analysing cell division in vivo and in vitro using flow cytometric measurement of cfse dye dilution. J Immunol Methods. 243 (1-2), 147-154 (2000).
- Azari, H., Deleyrolle, L. P., Reynolds, B. A. Using carboxy fluorescein succinimidyl ester (cfse) to identify quiescent glioblastoma stem-like cells. Methods Mol Biol. 1686, 59-67 (2018).
- Deleyrolle, L. P., Rohaus, M. R., Fortin, J. M., Reynolds, B. A., Azari, H. Identification and isolation of slow-dividing cells in human glioblastoma using carboxy fluorescein succinimidyl ester (cfse). J Vis Exp. (62), e3918 (2012).
- Ebinger, S., et al. Characterization of rare, dormant, and therapy-resistant cells in acute lymphoblastic leukemia. Cancer Cell. 30 (6), 849-862 (2016).
- Langenau, D. M., et al. Myc-induced t cell leukemia in transgenic zebrafish. Science. 299 (5608), 887-890 (2003).
- Blackburn, J. S., et al. Clonal evolution enhances leukemia-propagating cell frequency in t cell acute lymphoblastic leukemia through akt/mtorc1 pathway activation. Cancer Cell. 25 (3), 366-378 (2014).
- Al-Hamaly, M. A., Turner, L. T., Rivera-Martinez, A., Rodriguez, A., Blackburn, J. S. Zebrafish cancer avatars: A translational platform for analyzing tumor heterogeneity and predicting patient outcomes. Int J Mol Sci. 24 (3), 2288 (2023).
- Blackburn, J. S., Liu, S., Langenau, D. M. Quantifying the frequency of tumor-propagating cells using limiting dilution cell transplantation in syngeneic zebrafish. J Vis Exp. (53), e2790 (2011).
- Borga, C., et al. Simultaneous b and t cell acute lymphoblastic leukemias in zebrafish driven by transgenic myc: Implications for oncogenesis and lymphopoiesis. Leukemia. 33 (2), 333-347 (2019).
- Hu, Y., Smyth, G. K. Elda: Extreme limiting dilution analysis for comparing depleted and enriched populations in stem cell and other assays. J Immunol Methods. 347 (1-2), 70-78 (2009).
- Bhojwani, D., Pui, C. -. H. Relapsed childhood acute lymphoblastic leukaemia. Lancet Oncol. 14 (6), e205-e217 (2013).
- Zon, L. I., Peterson, R. T. In vivo drug discovery in the zebrafish. Nat Rev Drug Discov. 4 (1), 35-44 (2005).
- Al-Hamaly, M. A., et al. Zebrafish drug screening identifies erlotinib as an inhibitor of wnt/β-catenin signaling and self-renewal in t-cell acute lymphoblastic leukemia. Biomed Pharmacother. 170, 116013 (2024).
- Yan, C., et al. Visualizing engrafted human cancer and therapy responses in immunodeficient zebrafish. Cell. 177 (7), 1903-1914 (2019).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten