É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Identificação de células quiescentes em um modelo de leucemia linfoblástica aguda de células T de peixe-zebra usando coloração de proliferação celular
Neste Artigo
Resumo
Usamos a coloração de proliferação celular para identificar células quiescentes no modelo de leucemia linfoblástica aguda T de peixe-zebra. A coloração é retida em células que não se dividem e reduzida durante a proliferação celular, permitindo a seleção de células dormentes para interrogação posterior. Este protocolo fornece uma ferramenta funcional para estudar a auto-renovação no contexto da quiescência celular.
Resumo
A quiescência celular é um estado de parada de crescimento ou proliferação lenta que é descrito em células-tronco normais e cancerígenas (CSCs). A quiescência pode proteger as CSCs de drogas quimioterápicas antiproliferativas. Em modelos de camundongos com xenoenxerto derivado de pacientes (PDX) de leucemia linfoblástica aguda de células T (T-ALL), as células quiescentes estão associadas à resistência ao tratamento e à estaminalidade. Os corantes de proliferação celular são ferramentas populares para o rastreamento da divisão celular. O corante fluorescente é covalentemente ancorado em grupos amina na membrana e macromoléculas dentro da célula. Isso permite o rastreamento de células marcadas para até 10 divisões, que podem ser resolvidas por citometria de fluxo.
Em última análise, as células com as maiores taxas de proliferação terão baixa retenção de corante, pois serão diluídas a cada divisão celular, enquanto as células dormentes e de divisão mais lenta terão a maior retenção. O uso de corantes de proliferação celular para isolar células dormentes foi otimizado e descrito em modelos de camundongos T-ALL. Complementar aos modelos de camundongos existentes, o modelo T-ALL de peixe-zebra derivado de rag2: Myc fornece um excelente local para interrogar a auto-renovação em T-ALL devido à alta frequência de células-tronco leucêmicas (LSCs) e à conveniência do peixe-zebra para experimentos de transplante em larga escala.
Aqui, descrevemos o fluxo de trabalho para a coloração de células T-ALL de peixe-zebra com um corante de proliferação celular, otimizando a concentração do corante para células de peixe-zebra, passando células coradas com sucesso in vivo e a coleta de células com níveis variados de retenção de corante por classificação de células vivas de animais transplantados. Dada a ausência de fabricantes de superfície celular bem estabelecidos para LSCs em T-ALL, essa abordagem fornece um meio funcional para interrogar células quiescentes in vivo. Para resultados representativos, descrevemos a eficiência do enxerto e a frequência de LSC de células de alta e baixa retenção de corante. Este método pode ajudar a investigar propriedades adicionais de células quiescentes, incluindo resposta a medicamentos, perfis transcricionais e morfologia.
Introdução
As células-tronco adultas são responsáveis pela regeneração de tipos celulares diferenciados em um determinado órgão e estão predominantemente presentes em um estado dormente e sem divisão 1,2. Por exemplo, as células-tronco hematopoiéticas (HSCs), que mantêm o sangue, permanecem em grande parte quiescentes, e apenas uma pequena fração entra no ciclo celular para se auto-renovar ou se diferenciar para gerar componentes sanguíneos maduros3. Da mesma forma, nos cânceres, uma subpopulação rara de células chamadas células-tronco cancerígenas (CSCs) possui a capacidade de autorrenovação e é responsável pela manutenção a longo prazo da malignidade4. As células-tronco cancerígenas existem in vivo em um estado de quiescência ou crescimento lento, o que pode permitir que elas escapem dos tratamentos antiproliferativos contra o câncer5, evitem a depuração pelo sistema imunológico6, reduzam o estresse oxidativo e melhorem suas vias de reparo do DNA7. Mesmo um baixo número de CSCs deixadas para trás após o tratamento pode potencialmente repovoar o tumor, resultando na recaída do paciente8. Assim, a compreensão da quiescência celular é uma grande promessa para a identificação de vulnerabilidades potenciais das CSCs e o desenvolvimento de novas maneiras de direcioná-las.
Corantes de proliferação celular, como a coloração de éster carboxifluoresceína succinimidil (CFSE) e seus derivados, são comumente usados para rastrear a frequência das divisões celulares9. O corante permeia a membrana celular e, uma vez dentro da célula, sofre ativação por esterases intracelulares em um produto fluorescente. O composto fluorescente resultante é retido dentro da célula através das ligações amida covalentes formadas entre a fração succinimidil e os grupos funcionais amina das proteínas intracelulares10. Com cada divisão celular, o composto fluorescente é dividido igualmente entre as duas células resultantes, causando uma diluição de sinal dupla. Este corante permite a detecção de até 10 divisões celulares por meio da análise de citometria de fluxo11.
Essa abordagem foi utilizada anteriormente para enriquecer populações de CSC in vitro, identificando populações de células de ciclo lento com alta retenção do corante11,12. Na LLA-T, o CFSE tem sido usado para rastrear o crescimento tumoral in vivo em xenoenxertos derivados de pacientes em camundongos. Após a marcação celular e três semanas de transplante, a análise de citometria de fluxo mostrou uma população rara de células que ainda retinham a fluorescência do CFSE. Essa população foi associada a stemness, resistência ao tratamento e alta semelhança com células causadoras de recidiva em pacientes13. Assim, este corante fornece uma ferramenta útil para o estudo dos fenótipos de células-tronco de leucemia (LSC) em T-ALL.
O objetivo do trabalho é estender a aplicação do corante de proliferação celular para estudar a quiescência in vivo usando um modelo T-ALL de peixe-zebra. Em particular, o modeloT-ALL de peixe-zebra acionado por rag2: Myc fornece um excelente local para o estudo da auto-renovação devido à alta frequência de LSCs em comparação com modelos de camundongos e doenças humanas15. Além disso, o uso de peixe-zebra permite estudos de transplante em larga escala, que podem ser feitos a um custo muito menor de cuidados e manutenção em comparação com seus equivalentes de camundongos16. O peixe-zebra também é excelente para aplicações de imagem ao vivo, pois as células tumorais marcadas com fluorescência podem ser facilmente visualizadas usando um microscópio de fluorescência simples para estimar a taxa de desenvolvimento do tumor16.
Neste protocolo, descrevemos o fluxo de trabalho para coloração de células T-ALL de peixe-zebra com corante de proliferação celular seguido de propagação in vivo de células coradas em peixe-zebra CG1 singênico. Após o desenvolvimento da leucemia, descrevemos a classificação das células que retiveram o corante e seu uso para um experimento de transplante de diluição limitante subsequente para quantificar as taxas de auto-renovação do LSC. Este protocolo pode ser estendido para aplicações adicionais, incluindo triagem de drogas in vivo de compostos potenciais para o direcionamento de LSCs quiescentes. Além disso, as células coletadas podem ser usadas para diferentes análises a jusante, como perfil transcriptômico, proteômica e metabolômica, oferecendo insights exclusivos sobre o comportamento de LSCs quiescentes em T-ALL.
Protocolo
Neste protocolo, estamos usando células T-ALL de peixe-zebra marcadas com GFP que foram geradas anteriormente na cepa CG1 e, portanto, podem ser injetadas diretamente no peixe-zebra CG1 singênico do receptor15. Resumidamente, a leucemia foi gerada por microinjeção de DNA de rag2:Myc e rag2:GFP em embriões de peixe-zebra CG1 de célula única. Os animais foram monitorados quanto ao desenvolvimento de leucemia a partir de 3 semanas após a injeção, usando microscopia de fluorescência. As células de leucemia GFP-positivas foram isoladas com FACS e transplantadas em série no peixe-zebra receptor CG1 para gerar clones com alta frequência de LSC. Os detalhes de todo o protocolo são descritos por Blackburn et al.15.
Alternativamente, a LLA-T primária derivada de rag2:Myc pode ser gerada por microinjeção de DNA de embriões de peixe-zebra17. A microinjeção de DNA de rag2:Myc com um repórter fluorescente acionado por rag2, como GFP, pode resultar no desenvolvimento de células B, células T e LLA18 mista. Os clones de leucemia utilizados neste protocolo foram previamente verificados como T-ALL15. Todos os procedimentos experimentais envolvendo peixe-zebra foram revisados e aprovados pelo Comitê Institucional de Cuidados e Uso de Animais da Universidade de Kentucky, protocolo 2019-3399.
1. Propagação de células T-ALL de peixe-zebra in vivo
- Descongele um frasco contendo 1 mL de células T-ALL de peixe-zebra congeladas com rótulo GFP em banho-maria a 37 °C com agitação suave, até descongelar. Depois de descongelado, transfira para o gelo.
- Pipete lentamente o conteúdo do tubo em um tubo cônico de 15 mL com 4 mL de meio de peixe (0,9x PBS + 5% FBS) para diluir o meio de congelamento.
- Gire para baixo por 5 min a 2.500 × g a 4 °C. Remova a mídia do pellet. Ressuspenda em 0,5 mL de meio de peixe e mantenha as células no gelo.
- Conte as células e dilua-as, se necessário, para 100.000 células/5 μL.
- Anestesiar o peixe-zebra CG1 adulto adicionando 200 μL de anestésico 4 mg/mL (MS-222) a 25 mL de água do sistema de peixes em uma placa de Petri. Segure o peixe CG1, com o lado ventral para cima, e injete 5-6 μL no espaço intraperitoneal (IP) usando uma seringa de microlitro Hamilton. Mova os animais de volta para seus tanques e monitore o retorno à natação normal antes de colocá-los de volta no sistema.
NOTA: Peixes-zebra adultos, com 60 dias ou mais, foram usados para os estudos de transplante descritos neste protocolo. - Monitore o crescimento da leucemia uma vez por semana usando um microscópio de fluorescência. Consulte a Seção 2 abaixo para obter detalhes sobre o procedimento de triagem.
2. Colheita de células de leucemia marcadas com fluorescência
- Aproximadamente 21-28 dias após o transplante, anestesiar o peixe-zebra CG1 adulto adicionando 200 μL (4-5 gotas) de 4 mg/mL de anestésico a 25 mL de água do sistema de peixes em uma placa de Petri.
- Examine o peixe-zebra CG1 quanto à carga de leucemia, avaliando a porcentagem de células de leucemia marcadas com GFP no animal com um microscópio epifluorescente, usando o filtro apropriado para detectar a fluorescência GFP. A leucemia positiva para GFP está pronta para ser colhida se tiver se espalhado por >70% do animal.
- Eutanasiar o animal adicionando 1 mL de anestésico 4 mg/mL em uma placa de Petri contendo 9 mL de água do sistema de peixes. Permita que todos os sinais de vida cessem; Por exemplo, observe o peixe quanto à cessação do movimento opercular (~ 5 min).
- Mova o peixe-zebra para uma nova placa de Petri e adicione 1 mL de meio de peixe para servir como um tampão para as células. Usando uma nova lâmina de barbear, primeiro decapite o peixe-zebra e, em seguida, macere o tecido usando a mesma lâmina de barbear. Pipet para cima e para baixo para desalojar grandes aglomerados de células.
- Passe as células através de um filtro de células de 40 μm para um tubo cônico de 50 mL para dissociar em uma suspensão de célula única. Mantenha as células no gelo.
3. Otimização da concentração do corante de proliferação celular (CellTrace Far Red) para coloração e viabilidade celular do peixe-zebra
NOTA: Para este protocolo, e uma vez que as células tumorais são marcadas com GFP, usamos um corante de proliferação de glóbulos vermelhos distantes (CellTrace Far Red) para evitar sobreposição espectral, que será referido como CT-FR.
- Prepare uma solução de 1 mM do corante fluorescente imediatamente antes de usar, adicionando o volume apropriado de DMSO de acordo com as recomendações do fabricante. Ter soluções de trabalho de 1 mM, 0,5 mM, 0,1 mM e 0,01 mM; Diluir conforme necessário em meio de peixe para testar a viabilidade celular após a coloração.
- Conte as células e colete 1 × 106 células em tubos de microcentrífuga de 1,5 mL.
- Centrifugar a 2.500 × g a 4 °C durante 5 min. Remova o líquido e ressuspenda as células em 1 mL de 0,9x PBS.
- Divida a suspensão celular entre quatro tubos de 250 μL cada para tratamento com CT-FR.
- Adicione 250 μL de 0,9x PBS para aumentar o volume para 500 μL cada um com aproximadamente 250.000 células/tubo.
- Adicione as quantidades necessárias das soluções de trabalho descritas acima a cada um dos quatro tubos para atingir as seguintes concentrações finais de 1 μM, 0,5 μM, 0,1 μM e 0,05 μM. Incubar por 20 min a 37 ° C, protegido da luz.
- Pulverize as células girando a 2.500 × g por 5 min em temperatura ambiente. Remova o líquido e lave as células com 500 μL de meio de peixe para remover o excesso de corante.
- Pellet novamente, remova o líquido e ressuspenda em 25 μL de meio de peixe. Use 1 μL para contar as células em uma diluição de 1:10 em 50% de azul de tripano.
- Examine as células ao microscópio quanto à viabilidade celular; as células vivas não reterão o azul de Trypan e parecerão brilhantes ao microscópio, enquanto as células mortas absorverão o corante e parecerão escuras. Examine as células sob o canal de fluorescência apropriado primeiro para identificar células tumorais e, em seguida, use o canal de campo claro para avaliar a viabilidade celular.
- Selecione a concentração que resulta em uma coloração bem-sucedida, mantendo a viabilidade celular (>90% de células negativas para azul de tripano).
NOTA: Para as células T-ALL de peixe-zebra testadas neste experimento, a concentração de 1 μM foi usada para uma coloração bem-sucedida e >90% da viabilidade celular.
4. Propagação de células de peixe-zebra T-ALL coradas com CT-FR in vivo
- Usando a concentração otimizada de CT-FR da Seção 3, core as células do peixe-zebra. Deixe algumas células tumorais sem coloração para a geração do tumor T-ALL marcado com GFP, como um controle positivo para GFP durante a classificação FACS. Dilua as células coradas e não coradas para 50.000 células/5 μL para transplante.
- Anestesiar o peixe-zebra CG1 adulto adicionando 200 μL de anestésico 4 mg/mL a 25 mL de água do sistema de peixes em uma placa de Petri.
- Injectar 5 μL da suspensão celular na cavidade IP do peixe-zebra, conforme descrito na secção 1, tanto para as células coradas como para as não coradas. Injete 5-6 peixes-zebra por grupo para contabilizar a mortalidade potencial após a injeção.
- Examinar os animais por microscopia fluorescente sob sedação 2x semanalmente para o desenvolvimento de leucemia.
NOTA: Após ~ 10-14 dias, a leucemia deve ocupar cerca de 30-40% deste corpo. Planeje a colheita da leucemia e a subsequente classificação dentro desse prazo. Esperar que o tumor se expanda para um tamanho grande pode resultar em uma maior diluição do corante de células fluorescentes e dificuldade em detectar seu sinal. Considere a otimização do tempo de colheita do tumor para o modelo de tumor escolhido. Além disso, o monitoramento mais frequente do tumor é recomendado aqui para garantir a captura do tamanho desejado do tumor.
5. Preparação para a classificação de células T-ALL de peixe-zebra coradas com CT-FR
- Prepare os seguintes controles para a classificação de células: Sem controle de cor (suspensão de célula única, não corada, não transgênica, de um peixe-zebra do tipo selvagem), células T-ALL marcadas com GFP e células de peixe-zebra CT-FR WT recém-coradas (para definir um limite máximo de sinal vermelho distante).
- Preparação de controles não corados e CT-FR
- Colha células de peixes do tipo selvagem (WT) sacrificando um peixe-zebra CG1 não transplantado, adicionando 1 mL de anestésico 4 mg / mL em uma placa de Petri contendo 9 mL de água do sistema de peixes.
- Mova o peixe-zebra para uma nova placa de Petri e adicione 1 mL de meio de peixe para servir como um tampão para as células. Use uma nova lâmina de barbear para primeiro decapitar e depois macerar o peixe-zebra usando a mesma lâmina de barbear; pipetar para cima e para baixo para desalojar grandes aglomerados de células.
- Passe as células através de um filtro de células de 40 μm para um tubo cônico de 50 mL para dissociar em uma suspensão de célula única. Mantenha as células no gelo.
- Para o controle CT-FR, transfira 250.000 células WT para um tubo de microcentrífuga e peletize as células por centrifugação a 2.500 × g por 5 min em temperatura ambiente. Remova o meio de peixe e adicione 500 μL de 0,9x PBS + 1% FBS.
- Adicione 0,5 μL da solução estoque de corante CT-FR de 1 mM para uma concentração final de 1 μM. Pipete suavemente com 1.000 μL de pipeta para misturar. Incubar a 37 °C durante 20 min, protegido da luz.
- Remova o líquido e lave as células com 500 μL de meio de peixe para remover o excesso de corante.
- Pulverize as células por centrifugação novamente, remova o líquido e ressuspenda as células em 500 μL de 0,9x PBS + 1% FBS e passe por uma tampa de filtro de 35 μm em um tubo FACS. Mantenha no gelo.
- Preparando a placa doadora
NOTA: A placa doadora é uma placa de fundo em V de 96 poços na qual as células T-ALL coradas com CT-FR do peixe-zebra transplantado serão classificadas. Cada poço da placa contém 5.000 células WT CG1 não coradas em 50 μL de meio de peixe para servir como um transportador para ajudar a peletar as células classificadas nas etapas subsequentes.- Conte 500.000 células WT CG1 e transfira-as para um tubo cônico de 15 mL.
- Dilua esse estoque de células para 100 células / μL adicionando 5 mL de meio de peixe.
- Dispense 50 μL da suspensão celular em cada poço da placa de 96 poços. Misture o tubo de estoque de suspensão celular após cada linha para manter a homogeneidade da suspensão celular. Manter a placa a 4 °C até separar.
- Preparando as amostras de peixes transplantados
- Selecione peixes transplantados com 30-40% de carga T-ALL. Colete peixes transplantados com células coradas com CT-FR e células tumorais GFP não coradas.
- Colher células conforme descrito na Seção 2.
NOTA: Como se espera que as células retentoras de corante estejam presentes em baixa abundância, prepare-se para classificar um grande número de células da amostra CT-FR GFP. - Diluir na concentração apropriada para classificação ~ 3-5 × 106 células / mL em 0,9x PBS + 1% FBS e passar por uma tampa de filtro de 35 μm em um tubo FACS. Adicione DAPI (1 mg / mL) na diluição de 1:1.000 como um corante vital para excluir células mortas. Mantenha no gelo.
- Para as células tumorais GFP não coradas, transfira 250.000 células para um tubo de microcentrífuga e pellet a 2.500 × g por 5 min em temperatura ambiente. Remova o meio de peixe, adicione 500 μL de PBS e passe por uma tampa de filtro de 35 μm em um tubo FACS. Mantenha no gelo.
6. Classificação
NOTA: Mantenha todos os tubos e a placa doadora no gelo o tempo todo, exceto quando usados para triagem.
- Para classificar parâmetros e portas, comece aplicando o gating de dispersão direta e lateral para identificar as células de interesse com base em seu tamanho e eliminar os detritos celulares. Em seguida, use a altura de dispersão direta versus a área de dispersão direta para excluir células duplicadas e manter apenas singlets. Finalmente, use os controles sem cor e de cor única para desenhar os quadrantes para identificar populações de células positivas para GFP e CT-FR.
NOTA: Espera-se que o controle CT-FR seja muito mais brilhante do que as amostras para triagem, uma vez que a coloração para o controle foi feita fresca, sem divisões celulares; espera-se que as células das amostras CT-FR GFP apareçam na extremidade inferior da porta positiva CT-FR. - Classifique o número de células a serem transplantadas para cada peixe em um poço da placa doadora.
NOTA: Para este experimento, usamos um ensaio de diluição limitante para estimar a frequência de células-tronco de leucemia em CT-FR High e CT-FR Low. Este ensaio utilizou uma dose de 25 células, transplantadas em 3 animais, e uma dose de 10 células, transplantadas em 10 animais. Um ensaio típico de diluição limitante usando o T-ALL derivado de peixe-zebra 2: Myc envolveria um grupo com um número maior de células (500 ou 1.000), uma vez que a frequência de LSC neste modelo é estimada em 1 LSC em centenas de células. No entanto, neste ensaio, estamos usando um dos clones T-ALL descritos anteriormente por Blackburn et al.15. A frequência de LSC na amostra usada aqui é ~ 1 LSC em dezenas de células; Prevemos que a leucemia seja detectada em animais transplantados no grupo de 25 células. Ao otimizar este ensaio para o seu modelo de doença, deve-se tomar cuidado ao selecionar o grupo de maior número de células para garantir o enxerto do tumor. - Após a classificação, mantenha o prato no gelo até que esteja pronto para o transplante.
NOTA: Os procedimentos de classificação podem ser estressantes e afetar a viabilidade celular. Antes de executar o experimento de diluição limitante real, um experimento piloto de classificação foi feito. As células selecionadas foram transplantadas e o desenvolvimento da leucemia foi monitorado para determinar se a classificação era prejudicial à viabilidade celular. Alternativamente, a viabilidade celular pode ser avaliada colorindo as células selecionadas com azul de tripano e examinando-as ao microscópio antes do transplante.
7. Transplante de células classificadas na diluição limitante e no rastreamento do início do tumor
- Gire a placa usando uma grande centrífuga de bancada a 2.500 × g a 4 °C.
- Remova cuidadosamente 45 μL do sobrenadante de cada poço, deixando 5 μL de líquido para trás e evitando tocar no pellet de célula no fundo do poço.
- Ressuspenda as células com uma pipeta de 20 μL. Ressuspenda 2-3 poços com a pipeta e, em seguida, usando a seringa de microlitro Hamilton, injete as suspensões de células de cada poço no número desejado de peixe-zebra CG1 via transplante IP. Em seguida, ressuspenda as células no próximo grupo de poços para evitar o reassentamento das células.
- Examine o peixe-zebra por microscopia de fluorescência sob sedação uma vez por semana para o desenvolvimento de T-ALL por pelo menos 6 semanas.
8. Determinação da frequência de células-tronco leucêmicas
- Registre o número de animais positivos por dose de célula e grupo de tratamento 6 semanas após o transplante, considerando os animais com qualquer quantidade de sinal de GFP como positivos. Considere que os animais têm carga significativa de leucemia quando o sinal GFP ocupa >50% do corpo.
- Use o software estatístico ELDA (Extreme Limiting Dilution Analysis) baseado na web (http://bioinf.wehi.edu.au/software/elda/)19 para calcular a frequência de LSCs dentro de cada população de células.
NOTA: Uma ilustração esquemática do fluxo de trabalho é fornecida na Figura 1.
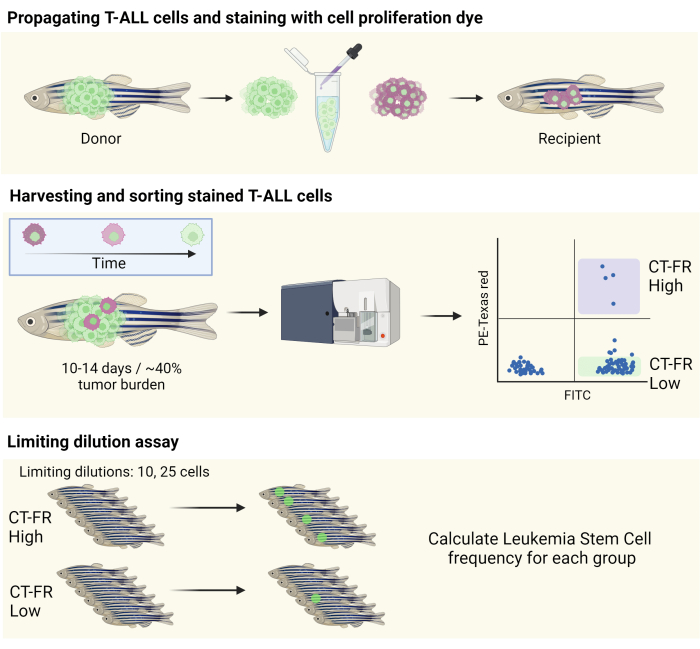
Figura 1: Fluxo de trabalho para o uso da coloração de rastreamento celular para isolar células quiescentes no modelo T-ALL de peixe-zebra. Ilustração esquemática da coloração de células T-ALL de peixe-zebra com a coloração de proliferação celular, propagação em um peixe-zebra CG1 (painel superior) e classificação de células com base na retenção da coloração de proliferação celular (painel do meio). As células coletadas foram usadas para um ensaio de diluição limitante para determinar a frequência de LSCs (painel inferior). Abreviaturas: LLA-T = leucemia linfoblástica aguda de células T; LSCs = células-tronco leucêmicas; CT-FR = CellTrace Vermelho Distante; FITC = isotiocianato de fluoresceína; PE = ficoeritrina. Clique aqui para ver uma versão maior desta figura.
Resultados
Seguimos o protocolo descrito acima para classificar as células que retiveram o corante de proliferação celular, CT-FR, e as usamos para um ensaio de diluição limitante (LDA) para estimar a frequência de LSC nas populações CT-FR High e CT-FR Low. Para definir o gating para o experimento de citometria de fluxo, usamos um controle sem fluoróforo (sem cor), além de controles de cor única (Figura 2A). Esses quadrantes foram usados par...
Discussão
Os LSCs são conhecidos por serem resistentes aos tratamentos convencionais de quimioterapia antiproliferativa, e encontrar terapias direcionadas contra essas células é uma grande promessa na redução da ocorrência de recidiva e na melhoria do prognóstico do paciente20. Pesquisas anteriores descreveram o uso de colorações de proliferação de células fluorescentes para identificar uma pequena população de células quiescentes associadas à resistência a...
Divulgações
Os autores não têm conflitos de interesse a divulgar.
Agradecimentos
O financiamento para esta pesquisa foi fornecido pelo Instituto Nacional do Câncer (R37CA227656 ao JSB). Esta pesquisa também foi apoiada pelos Recursos Compartilhados de Citometria de Fluxo e Monitoramento Imunológico do Centro de Câncer Markey da Universidade de Kentucky (P30CA177558).
Materiais
| Name | Company | Catalog Number | Comments |
| 26 G/2” micro-syringe | Hamilton | 87930 | NA |
| 35 µm filter cap FACS tubes | Falcon | 352235 | NA |
| 40 µm cell strainer | CELLTREAT | 229482 | NA |
| 96-well skirted PCR plate | Thermo Fisher Scientific | AB0800 | NA |
| Cell sorter | Sony Biotechnology | SY3200 | NA |
| CellTrace Far Red | Thermo Fisher Scientific | C34564 | NA |
| Conical tubes | VWR | 10026-078 | NA |
| DAPI | Thermo Fisher Scientific | 62248 | NA |
| DMSO | Sigma-Aldrich | D4818 | NA |
| Dulbecco'sPhosphate-buffered saline (PBS) | Caisson Labs | 22110001 | NA |
| Epifluorescence stereo microscope | Nikon | SMZ25 | NA |
| Fetal Bovine Serum (FBS) | Sigma-Aldrich | 12306C | NA |
| Fish system water | N/A | N/A | 0.03-0.05% salinity, pH 6.5-8, buffered with sodium bicarbonate |
| Microcentrifuge tubes | Thermo Fisher Scientific | C2171 | NA |
| MS-222 | Pentaire | TRS-1 | tricaine mesylate, an anesthetic |
| Petri dishes | Corning | 07-202-011 | NA |
| Razor blades | American Line | 66-0089 | NA |
| Trypan Blue | Thermo Fisher Scientific | T10282 | NA |
Referências
- Reya, T., Morrison, S. J., Clarke, M. F., Weissman, I. L. Stem cells, cancer, and cancer stem cells. Nature. 414 (6859), 105-111 (2001).
- Arai, F., et al. Tie2/angiopoietin-1 signaling regulates hematopoietic stem cell quiescence in the bone marrow niche. Cell. 118 (2), 149-161 (2004).
- Li, L., Bhatia, R. Stem cell quiescence. Clin. Cancer Res. 17 (15), 4936-4941 (2011).
- Kreso, A., Dick, J. E. Evolution of the cancer stem cell model. Cell Stem Cell. 14 (3), 275-291 (2014).
- Chen, W., Dong, J., Haiech, J., Kilhoffer, M. -. C., Zeniou, M. Cancer stem cell quiescence and plasticity as major challenges in cancer therapy. Stem Cells Int. 2016, 1740936 (2016).
- Kleffel, S., Schatton, T. Tumor dormancy and cancer stem cells: Two sides of the same coin. Adv Exp Med Biol. 734, 145-179 (2013).
- Tuy, K., Rickenbacker, L., Hjelmeland, A. B. Reactive oxygen species produced by altered tumor metabolism impacts cancer stem cell maintenance. Redox Biol. 44, 101953 (2021).
- Zhou, B. -. B. S., et al. Tumour-initiating cells: Challenges and opportunities for anticancer drug discovery. Nat Rev Drug Discov. 8 (10), 806-823 (2009).
- Lyons, A. B., Blake, S. J., Doherty, K. V. Flow cytometric analysis of cell division by dilution of cfse and related dyes. Curr Protoc Cytom. 64 (1), 11-12 (2013).
- Lyons, A. B. Analysing cell division in vivo and in vitro using flow cytometric measurement of cfse dye dilution. J Immunol Methods. 243 (1-2), 147-154 (2000).
- Azari, H., Deleyrolle, L. P., Reynolds, B. A. Using carboxy fluorescein succinimidyl ester (cfse) to identify quiescent glioblastoma stem-like cells. Methods Mol Biol. 1686, 59-67 (2018).
- Deleyrolle, L. P., Rohaus, M. R., Fortin, J. M., Reynolds, B. A., Azari, H. Identification and isolation of slow-dividing cells in human glioblastoma using carboxy fluorescein succinimidyl ester (cfse). J Vis Exp. (62), e3918 (2012).
- Ebinger, S., et al. Characterization of rare, dormant, and therapy-resistant cells in acute lymphoblastic leukemia. Cancer Cell. 30 (6), 849-862 (2016).
- Langenau, D. M., et al. Myc-induced t cell leukemia in transgenic zebrafish. Science. 299 (5608), 887-890 (2003).
- Blackburn, J. S., et al. Clonal evolution enhances leukemia-propagating cell frequency in t cell acute lymphoblastic leukemia through akt/mtorc1 pathway activation. Cancer Cell. 25 (3), 366-378 (2014).
- Al-Hamaly, M. A., Turner, L. T., Rivera-Martinez, A., Rodriguez, A., Blackburn, J. S. Zebrafish cancer avatars: A translational platform for analyzing tumor heterogeneity and predicting patient outcomes. Int J Mol Sci. 24 (3), 2288 (2023).
- Blackburn, J. S., Liu, S., Langenau, D. M. Quantifying the frequency of tumor-propagating cells using limiting dilution cell transplantation in syngeneic zebrafish. J Vis Exp. (53), e2790 (2011).
- Borga, C., et al. Simultaneous b and t cell acute lymphoblastic leukemias in zebrafish driven by transgenic myc: Implications for oncogenesis and lymphopoiesis. Leukemia. 33 (2), 333-347 (2019).
- Hu, Y., Smyth, G. K. Elda: Extreme limiting dilution analysis for comparing depleted and enriched populations in stem cell and other assays. J Immunol Methods. 347 (1-2), 70-78 (2009).
- Bhojwani, D., Pui, C. -. H. Relapsed childhood acute lymphoblastic leukaemia. Lancet Oncol. 14 (6), e205-e217 (2013).
- Zon, L. I., Peterson, R. T. In vivo drug discovery in the zebrafish. Nat Rev Drug Discov. 4 (1), 35-44 (2005).
- Al-Hamaly, M. A., et al. Zebrafish drug screening identifies erlotinib as an inhibitor of wnt/β-catenin signaling and self-renewal in t-cell acute lymphoblastic leukemia. Biomed Pharmacother. 170, 116013 (2024).
- Yan, C., et al. Visualizing engrafted human cancer and therapy responses in immunodeficient zebrafish. Cell. 177 (7), 1903-1914 (2019).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados