Method Article
Hochauflösende Bildgebung von C. elegans in allen Larvenstadien
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt die mikrofluidische Zeitraffer-Bildgebung von C. elegans über die gesamte postembryonale Entwicklung.
Zusammenfassung
Caenorhabditis elegans hat sich zu einem der am besten untersuchten und am besten verstandenen Tiermodelle in der Biologie entwickelt. Drei Merkmale sind entscheidend für den Erfolg von C. elegans als Modellorganismus: seine unveränderliche Zelllinie, seine Transparenz und seine genetische Rückverfolgbarkeit. Damit eignet es sich ideal für eine Vielzahl von mikroskopiebasierten Studien direkt in vivo. Lebende Larven und adulte Tiere von C. elegans müssen während der Bildaufnahme oft immobilisiert werden. Herkömmliche Immobilisierungsmethoden wirken sich negativ auf die Entwicklung der Tiere aus, insbesondere bei Zeitraffer-Bildgebungsanwendungen. Hier wird ein detailliertes Aufbau- und Betriebsprotokoll für ein neuartiges mikrofluidisches Bildgebungsverfahren vorgestellt, das die Einschränkungen der traditionellen Agar-Pad-basierten Immobilisierung und anderer mikrofluidischer Strategien berücksichtigt. Dieser Ansatz ermöglicht die gleichzeitige Live-Bildgebung in verschiedenen Larvenstadien unter Beibehaltung der Orientierung und Identität des Wurms über die Zeit. Um dies zu erreichen, wird ein mikrofluidisches Fallenkanal-Array verwendet, dessen Geometrie genau so ausgelegt ist, dass eine stabile Wurmausrichtung bei gleichzeitiger Berücksichtigung von Wachstum und Häutung gewährleistet ist. Die Immobilisierung wird durch ein aktives Hydraulikventil erleichtert, das Druck ausübt, um die Würmer nur während der Bildaufnahme am Deckglas zu sichern. Dieses Design ermöglicht eine hochauflösende Bildgebung mit minimalen Auswirkungen auf die Lebensfähigkeit des Wurms oder den Entwicklungszeitpunkt.
Einleitung
Die Bildgebung von C. elegans kann je nach Anwendung, gewünschtem Durchsatz und Auflösung auf verschiedene Arten durchgeführt werden. Wenn eine hohe Auflösung erforderlich ist, verwendet das Standardverfahren die Agar-Pads 1,2,3, eine einfache Konstruktion aus Glasobjektträger, Agarose und Deckglas. Die Tiere auf diesen Objektträgern werden durch den Druck eines Deckglases bewegungsunfähig. Die Immobilisierung kann durch die Zugabe von Anästhesiemitteln, z. B. Levamisol oder Natriumazid 1,2, weiter verbessert werden, so dass Tiere auf Agar-Pads vollkommen ruhig sind und mit hoher Auflösung abgebildet werden können, einschließlich verschiedener superauflösender Methoden2. Leider ist bekannt, dass Agar-Pads den Entwicklungsfortschritt beeinflussen, wobei die Kombination aus hohem Druck und Betäubungsmitteln die Entwicklung verzögert oder einen vollständigen Entwicklungsstillstand verursacht 4,5.
Neben diesen Standardmethoden sind in den letzten Jahren mehrere mikrofluidische Methoden entstanden, jede mit ihren Vor- und Nachteilen oder spezialisierten Anwendungsfällen 6,7,8,9,10,11,12,13,14,15,16,17,18,19. So führten Gritti et al.20 eine Methode ein, bei der Würmer in großen Kammern eingesperrt werden. Embryonen, die in diese Kammern gelegt werden, entwickeln sich und bewegen sich frei, während sie auf einem Mikroskop in der dafür vorgesehenen Region bleiben. Obwohl dieser Ansatz im Allgemeinen effektiv ist, beschränkt er sich auf die Verwendung von hellen Fluoreszenzmarkern. In jüngerer Zeit schlugen Keil et al.21 eine modifizierte Version dieser Methode vor, die ein On-Chip-Hydraulikventil zur Fixierung der Tiere während der Bildaufnahme enthielt und damit einige Einschränkungen des ursprünglichen Ansatzes beseitigte. Da jedoch bei beiden Methoden größere Kammern verwendet werden als bei gefangenen Würmern, bewegen und drehen sich die Tiere während des gesamten Versuchs weiter. Infolgedessen wird die Verfolgung von Entwicklungsprozessen zu einer Herausforderung und erfordert eine umfangreiche Bildnachbearbeitung, um Tierbewegungen zu korrigieren.
Um diese Einschränkungen zu beheben, wird der von Berger et al.18 (Abbildung 1) vorgestellte Ansatz angepasst, bei dem die Tiere nicht in einer großen Kammer gehalten werden, sondern in einem Fallenkanal untergebracht werden, der so konzipiert ist, dass die Tiere in ein kürzeres Entwicklungsfenster (bis zu 2,5 Larvenstadien) passen. Im Gegensatz zu bestehenden mikrofluidischen Methoden bewahrt dieser Ansatz die Orientierung und Identität der Tiere während eines Experiments und ermöglicht die Aufnahme hochauflösender Bilder mehrerer Würmer parallel und über mehrere Larvenstadien hinweg innerhalb eines einzigen Geräts.
Die Tiere werden mit einer speziellen Kanalgeometrie gefangen, bei der die Kanalhöhe nahe der Dicke eines Tieres zu Beginn eines Versuchs liegt, wodurch eine Rotation verhindert wird. Die Kanalbreiten und -längen sind so gewählt, dass sich die Tiere im Laufe der Zeit bewegen und wachsen können, was die Häutung und den Übergang von einem Larvenstadium zum nächsten erleichtert. Die Parallelisierung wird dann einfach erreicht, indem mehrere Fallenkanäle nebeneinander platziert werden, so dass mehrere Tiere in einem einzigen Sichtfeld sichtbar sind (FOV, Abbildung 1A') und bis zu 41 Tiere in einer einzigen Geräteeinheit abgebildet werden können (Abbildung 1A). Die Tiere auf dem Chip werden über einen Kanal mit geringer Höhe kontinuierlich mit einer hochkonzentrierten bakteriellen Futtersuspension versorgt, die gleichzeitig dazu beiträgt, die Tiere am Ende des Fallenkanal-Arrays einzusperren (Höhenstufe, Abbildung 1A, rot). Ähnlich wie bei Keil et al.21 wird ein großes hydraulisches On-Chip-Ventil implementiert, das während der Bildaufnahme aufgeblasen werden kann, wodurch die Tiere gegen das Deckglas gedrückt werden, wodurch die Immobilisierung der Tiere verbessert wird (Abbildung 1B). Die Beschränkung der hydraulischen Ventilbetätigung auf den Bildaufnahmezeitraum minimiert jegliche nachteiligen Auswirkungen auf die Entwicklung der Tiere und gewährleistet eine zuverlässige Entwicklung in allen Larvenstadien. Im Gegensatz zu den von Gritti et al.20 und Keil et al.21 beschriebenen Methoden können Tiere nicht über die gesamte postembryonale Entwicklung innerhalb desselben Geräts abgebildet werden. Stattdessen werden fünf verschiedene Geräte eingeführt, die als L1, L1-4, L2-A, L3-A und L4-A bezeichnet werden und das gesamte L1-Stadium und das frühe L2-Stadium, das mittlere bis späte L1-Stadium bis zum mittleren L4-Stadium, das mittlere bis späte L2-Stadium bis zum jungen Erwachsenenalter, das mittlere bis späte L3 bis zum jungen Erwachsenenalter und das späte L4 bis zum jungen Erwachsenenalter abdecken (Abbildung 1C). die eine ununterbrochene Bildgebung großer Teile der postembryonalen Entwicklung ermöglichen. Dieser Ansatz wird aufgrund der starken Zunahme der Tiergröße von L1 bis zum Erwachsenenalter gewählt, so dass ein Kanal, der für L1-Tiere geeignet ist, für Erwachsene viel zu klein wäre, was zu einem Entwicklungsstillstand führt, und ein Kanal, der zu Erwachsenen passt, viel zu groß für L1 wäre, was zu übermäßigen Bewegungen, Rotationen und Tieren führt, die aus den Fallenkanälen entkommen.
Mit diesem Ansatz wird eine zuverlässige Entwicklungsprogression für eine Vielzahl von Geweben erreicht18, z.B. die postembryonale Entwicklung der Hypodermis (L1- bis L4-Stadium), die Induktion des Vulvazellschicksals (L2- und L3-Stadium), die Vulva-Morphogenese (L3 bis zum Erwachsenenalter19) und die Ankerzellinvasion. Seit ihrer Einführung wurden diese Langzeitbildgebungsgeräte zur Untersuchung einer Vielzahl von Prozessen verwendet22,23, wobei die Plattform sogar mit Techniken wie der hochauflösenden AiryScan-Mikroskopie24 kombiniert wurde, was zahlreiche Entdeckungen ermöglichte, die zuvor unmöglich waren.
Im Folgenden werden die Bedienungsaspekte dieser Langzeitbildgebungsgeräte in einem Schritt-für-Schritt-Protokoll vorgestellt und die zu erwartenden Ergebnisse und einige mögliche Herausforderungen bei der Einrichtung des Systems hervorgehoben.
Protokoll
Die Einzelheiten zu den Reagenzien und der Ausrüstung, die in dieser Studie verwendet wurden, sind in der Materialtabelle aufgeführt.
1. Herstellung von Geräten
HINWEIS: Alle Geräte wurden unter Verwendung des unten aufgeführten Protokolls hergestellt, die Geräte sind jedoch direkt bei den Autoren erhältlich. Für jeden Bauelementtyp wird ein separater Wafer hergestellt, wobei jeder Bauelementwafer aus zwei unterschiedlich hohen SU8-Schichten (d. h. Lebensmittelschicht und Trap-Schicht) hergestellt wird. Der Ventilwafer wird auf einem separaten Wafer mit einer einzigen Höhe von SU-8 hergestellt, wobei die Schritte 1.3-1.6 weggelassen werden. Die Silane sind giftig und setzen korrosive Dämpfe frei. Dieser Schritt muss daher in einem Abzug durchgeführt werden. Für Details zum Herstellungsverfahren siehe Berger et al.18.
- Reinigen Sie Wafer mit Luftplasma.
- Die erste SU8-Schicht schleudern und dann bei 95 °C weich backen.
- Die erste Schicht belichten, gefolgt von einem Nachbacken bei 95 °C und einer Entwicklungsphase.
- Führen Sie die Plasmareinigung nach der Waferentwicklung und -trocknung erneut durch.
- Die zweite SU8-Schicht schleudern und anschließend bei 95 °C weich backen (ggf. bei 65 °C vorbacken).
- Richten Sie die zweite Maske mithilfe von Ausrichtungsmarkierungen auf beiden Seiten des Wafers an den vorhandenen Merkmalen aus und legen Sie dann die zweite Ebene frei.
- Die Waffel nachbacken und weiterentwickeln, dann ein hartes Backen bei 200 °C durchführen.
- Behandeln Sie den Wafer mindestens 2 h lang mit Chlortrimethylsilan.
- Bereiten Sie eine Charge von 20 g PDMS-Präpolymer (Teil A) vor und mischen Sie es gründlich mit den 1 g des Vernetzers (Verhältnis 20:1, Teil B) (siehe Materialtabelle).
- Entgasen Sie 10 Minuten lang oder bis alle Blasen verschwunden sind.
- Entfernen Sie 2 mL des PDMS-Gemisches und stellen Sie es für Schritt 1.13 bei.
- Weitere 3 g Vernetzer zur restlichen PDMS-Mischung geben und gründlich vermischen (Endverhältnis ca. 5:1).
- Mit der Charge von 20:1 PDMS wird der Bauteilwafer 30 s lang bei 750 U/min geschleudert.
- Legen Sie den Ventilwafer in eine Aluminiumschale und gießen Sie das 5:1 PDMS darauf. Entgasen Sie den Gerätewafer für ca. 10 min. Entfernen Sie nach der Entgasung alle verbleibenden Blasen mit einer Pipette.
- Sowohl die Ventil- als auch die Geräteschicht bei 70 °C ca. 15/20 min backen.
- Beurteilen Sie die Aushärtung der Geräte- und Ventilschichten, indem Sie sie vorsichtig mit einer Pipettenspitze oder einer Pinzette antasten. Die Ventilschicht sollte fest sein, während die Geräteschicht leicht klebrig bleiben sollte.
- Lösen Sie die Ventilschicht und schneiden Sie sie mit einem Skalpell oder einer Rasierklinge auf die gewünschte Größe zu.
- In den Ventilkanal (Spur 20) ein Zugangsloch stanzen.
- Positionieren Sie den Gerätewafer unter einem Stereomikroskop, das mit einer Top-Beleuchtung ausgestattet ist.
- Platzieren Sie das PDMS-Stück mit der Ventilschicht vorsichtig auf dem Gerätewafer.
- Richten Sie die Ventil- und Gerätefunktionen genau aus und stellen Sie sicher, dass die gesamte eingeschlossene Luft zwischen den Schichten entfernt wird.
- Stellen Sie das zusammengebaute Gerät in einen Ofen und backen Sie es über Nacht bei 70 °C.
- Trennen Sie das Gerät vom Wafer.
- Stanzen Sie alle restlichen Zugangslöcher (Feinheit 20).
- Verkleben Sie das PDMS mit Luftplasma auf ein Deckglas und stellen Sie das geklebte Gerät dann für einige Stunden in einen Ofen bei 70 °C.
2. Vorbereitung der Wurm
HINWEIS: In diesem Abschnitt wird die Wurmpräparation durch Bleichen beschrieben, die die Erzeugung einer großen Anzahl (Hunderte) von Tieren ermöglicht. Andere Synchronisationsmethoden, wie z. B. die manuelle Kommissionierung, sind möglich, aber in der Regel zeit- und arbeitsintensiver. Alle Würmer, die im Protokoll verwendet werden, werden gemäß den Standardprotokollen bei 20 °C und auf NGM-Platten gehalten, die mit E. coli OP50 besiedelt sind.
- Angefangen bei Platten mit vielen trächtigen Tieren.
- Die Tiere mit M9-Puffer vom Teller waschen.
- 5 % NaClO und 5 M NaOH zur Wurmsuspension geben (200 μl/100 μl pro 1 mL Wurmsuspension).
- Schütteln Sie die Bleichmischung vorsichtig, bis die Tiere anfangen auseinanderzubrechen (~10 min).
- Bleichmischung bei Raumtemperatur zentrifugieren, 1300 x g für 1 min.
- Entfernen Sie den Überstand mit einer Pipette und fügen Sie eine gleiche Menge frischen M9-Puffer hinzu.
- Nochmals bei Raumtemperatur zentrifugieren, 1300 x g für 1 min.
- Entfernen Sie den Überstand und geben Sie die pelletierten Würmer in ein 15-ml-Röhrchen mit 5 mL frischem M9-Puffer.
- Über Nacht bei 20 °C schütteln.
- Die Wurmsuspension wird durch ein 10 μm Zellsieb geleitet.
- Die Schneckensuspension wird bei 1300 × g 1 min bei Raumtemperatur zentrifugiert.
- Der Überstand wird verworfen und die Würmer werden in einem gleichen Volumen M9-Puffer resuspendiert.
- Die Wurmmischung erneut bei 1300 × g für 1 min bei Raumtemperatur zentrifugieren.
- Entfernen Sie den Überstand und übertragen Sie die Würmer auf NGM-Platten. Wenn L1-Larven benötigt werden, verwenden Sie diese sofort.
- Sobald die Würmer das gewünschte Entwicklungsstadium erreicht haben, waschen Sie sie mit frischem S-Basal-Puffer, der durch einen 0,2-μm-Filter gefiltert wird, von der Platte.
- Lassen Sie die Würmer durch die Schwerkraft sedimentieren oder pelletieren Sie sie durch Zentrifugation bei 750 × g für 1 min bei Raumtemperatur.
- Den Überstand verwerfen und das gleiche Volumen frischen S-Basalpuffer hinzufügen.
- Wiederholen Sie die Sedimentations- oder Zentrifugationsschritte (Schritte 2.16 und 2.17)
- Entfernen Sie den Überstand und fügen Sie das gleiche Volumen frischen S-Basalpuffer hinzu.
- Führen Sie die Sedimentations- oder Zentrifugationsschritte ein letztes Mal durch.
- Entfernen Sie den größten Teil des Überstands.
- Bewahren Sie die Würmer in der Tube auf, bis das Experiment beginnt.
3. Vorbereitung der Bakterien
HINWEIS: Auf dem Chip können verschiedene Bakterienstämme (z. B. OP50, NA22, HT114 usw.) verwendet werden. Passen Sie den Anteil des Dichtegradientenmediums an, das der Lebensmittelmischung zugesetzt wird, um Dichteunterschiede auszugleichen. Alle Puffer werden durch einen 0,2-μm-Filter geleitet, um Partikel zu entfernen.
- Bereiten Sie 40 mL LBroth vor, entweder in 2 x 20 mL in Zentrifugenröhrchen, sterilen Erlenmeyerkolben usw.
- Beimpfen Sie LB mit Bakterien, die von einer Lagerplatte entnommen wurden.
- Züchten Sie die Bakterien unter Schütteln über Nacht bei 37 °C (~16 h) (OD600 = 1,9).
- Bakterien bei 3000 x g für 10 min zentrifugieren.
- Entfernen Sie den Überstand und geben Sie 2 ml frischen S-Basalpuffer in jedes Röhrchen. Kombinieren Sie alle Tuben.
- Suspendieren Sie die Bakterien mechanisch mit einer Pipette.
- Zentrifugieren Sie die Bakterien bei 3000 x g für 5 min.
- Entfernen Sie den Überstand und fügen Sie erneut 2 ml frischen S-Basalpuffer hinzu.
- Suspendieren Sie die Bakterien mechanisch mit einer Pipette.
- Zentrifugieren Sie die Bakterien bei 3000 x g für 5 min.
- Entfernen Sie so viel Überstand wie möglich und fügen Sie 1 ml frischen S-Basalpuffer hinzu, bevor Sie alle Bakterien mechanisch resuspendieren.
- Kombinieren Sie 0,60 mL Dichtegradientenmedium, 0,38 mL S-Basal + 1 Gew.-% Pluronic F-127 (ein nichtionisches Triblock-Copolymer, hier dargestellt als PF) mit 1 mL OP50-Bakteriensuspension oder 0,65 mL Dichtegradientenmedium, 0,33 mL S-Basal + 1 Gew.-% PF mit 1 mL NA22- oder HT114-Bakteriensuspension.
- Mischen Sie die Zubereitung der Speisen mit einem Vortex-Mixer gründlich durch.
- Filtrieren Sie das Futter durch ein 5 μm oder 10 μm Zellsieb. Ein kleineres Sieb ist besonders bei der Verwendung kleinerer Geräte, z. B. L1-Geräte, zu empfehlen.
HINWEIS: Lebensmittel können bis zu 2 Tage bei Raumtemperatur aufbewahrt werden.
4. Vorbereitung der Späne
HINWEIS: In Abbildung 2 finden Sie eine schematische Übersicht über das Gerät, die Schläuche und die Anschlüsse. Alle Puffer werden durch einen 0,2 μm Filter geleitet, um Staub zu entfernen. Der in den folgenden Schritten erwähnte Stahlstift bezieht sich auf ein kurzes, hohles Rohr, das verwendet wird, um das Rohr mit größerem Außendurchmesser (1/16") mit den in der PDMS-Vorrichtung hergestellten Löchern mit kleinem Durchmesser zu verbinden. Diese Stahlstifte werden einfach in den Innendurchmesser des Rohrs gesteckt und dann in das PDMS-Material geschoben, wodurch die beiden effektiv verbunden werden.
- Beginnen Sie damit, eine Spritze mit deionisiertem (DI) Wasser zu füllen.
- Befestigen Sie eine 23 G Nadel und ein langes Stück 1/16" Schlauch mit einem hohlen Stahlstift (im 90°-Winkel gebogen) am Ende. Der Schlauch sollte lang genug sein, um vom Magneten bis zum Mikroskoptisch zu reichen.
- Füllen Sie den Schlauch mit DI-Wasser aus der Spritze und verbinden Sie ihn mit dem Ventileinlass, indem Sie den Stahlstift in das gestanzte Loch einführen (Abbildung 2A,J).
- Entfernen Sie die Spritze und die Nadel und befestigen Sie den Schlauch am Off-Chip-Magneten (Abbildung 2A,J).
- Schalten Sie mit der Bildgebungssoftware das Magnetventil ein und setzen Sie das Gerät einige Minuten lang unter Druck, um die gesamte Luft aus dem Ventil auszustoßen. Überprüfen Sie den Abschluss, indem Sie die Luft-Wasser-Grenzfläche auf dem Chip visuell überprüfen, die dunkel erscheinen und im PDMS-Material verschwinden sollte (Abbildung 2B).
- Schalten Sie den Magneten aus (Abbildung 2C).
- Füllen Sie eine 1-ml-Spritze mit der gefilterten Bakterienlösung (~0,5 mL Futter ist ausreichend).
- Befestigen Sie eine 30 G Nadel und ein langes Stück 1/32" Schlauch an der Nadel.
- Drücken Sie den Kolben, um sowohl die Nadel als auch den angeschlossenen Schlauch zu füllen, und stellen Sie sicher, dass keine Luft in der Spritze oder dem Schlauch verbleibt.
- Führen Sie den 1/32"-Schlauch direkt in den Lebensmitteleinlass des Mikrofluidikgeräts ein und verwenden Sie eine Pinzette, um eine Beschädigung des Schlauchs zu vermeiden (eine SMD-Pinzette wird empfohlen) (Abbildung 2D,K).
- Setzen Sie die Spritze auf die Spritzenpumpe (Abbildung 2D).
- Drücken Sie den Spritzenkolben mit der Rändelschraube auf der Rückseite, um das Gerät mit Flüssigkeit zu füllen. An jeder offenen Verbindung sollte ein Tropfen Flüssigkeit erscheinen (Abbildung 2D, Pfeil).
- Blockieren Sie sowohl den Einlass als auch den Auslass der Schnecke mit einem abgedichteten Stahlstift (ein Stahlstift mit einem kurzen Stück 1/16"-Schlauch, an dem ein Schlauch befestigt und durch Brennen abgedichtet ist) (Abbildung 2E, L).
- Üben Sie mit der Rändelschraube zusätzlichen Druck aus, um die verbleibende Luft im Gerät zu entfernen (Abbildung 2E, Pfeil).
- Entfernen Sie den blockierten Stift am Auslass und befestigen Sie den Abfallbehälter (hergestellt aus einem Kryoröhrchen oder Mikrozentrifugenröhrchen, mit zwei Stahlstiften, die durch den Deckel gesteckt und mit Klebstoff gesichert sind). Der Abfallbehälter wird mit einem kurzen Stück 1/16"-Schlauch verbunden, wobei ein Ende mit dem Behälter und das andere Ende mit einem hohlen Stahlstift (um 90° gebogen) verbunden ist (Abbildung 2F,G,M).
- Drücken Sie auf die Spritze, um sicherzustellen, dass der Abfallbehälter richtig angeschlossen ist und keine Verstopfungen im System vorhanden sind. Schieben Sie dazu eine kleine Menge Flüssigkeit durch das System (drehen Sie die Rändelschraube an der Spritzenpumpe), bis die Flüssigkeit durch den Abfallschlauch fließt (Abbildung 2G, Pfeil).
- Entfernen Sie den zweiten blockierten Stahlstift.
- Schieben Sie die Spritze, bis ein kleiner Tropfen Flüssigkeit am Wurmeinlass erscheint (Abbildung 2H,N).
- Befestigen Sie ein längeres Stück 1/16"-Schlauch (15-20 cm) mit einer 23-G-Nadel an einer 1-ml-Spritze, die mit S-Basalpuffer gefüllt ist (Abbildung 2H,N).
- Befestigen Sie einen geraden 23-G-Stahlstift am anderen Ende des Schlauchs.
- Füllen Sie die Nadel und den Schlauch mit Puffer aus der Spritze und stellen Sie sicher, dass keine Luft in der Spritze oder dem Schlauch verbleibt.
- Führe den Stahlstift am Ende des Schlauchs in das Rohr ein, das die Würmer enthält.
- Drücken Sie eine kleine Menge Flüssigkeit durch den Schlauch und stellen Sie sicher, dass keine Luft zurückbleibt.
- Ziehen Sie die Würmer in den Schlauch, aber nicht in die Spritze.
- Schieben Sie die mit den Würmern verbundene Spritze, bis ein kleiner Flüssigkeitstropfen auf dem Stahlstift erscheint (Abbildung 2H).
- Setzen Sie den Stahlstift in den Schneckeneinlass ein (Abbildung 2I).
HINWEIS: Die Gerätevorbereitung kann vor der endgültigen Wurmpräparation durchgeführt und am Mikroskop belassen werden, bis die Würmer fertig sind.
5. Wurmbelastung und Bildgebung
- Stellen Sie das Gerät auf ein Mikroskop mit geringer Vergrößerung (5x oder 10x) oder auf ein Präpariermikroskop.
- Identifizieren Sie die aktuell verwendete Geräteeinheit und suchen Sie den Schneckeneinlass mithilfe der Hellfeldbeleuchtung.
- Positionieren Sie das Gerät so, dass der Einlass auf einer Seite des Sichtfeldes sichtbar ist und auf der anderen Seite die Rückseite des Einlasses des Siphonkanals sichtbar ist.
- Drücke vorsichtig auf den Kolben der Wurmspritze. Die Flüssigkeit fließt spürbar vom Einlass durch das Kanalarray in Richtung Auslass. Das Vorhandensein von Bakterien und der Unterschied im Brechungsindex aufgrund der Zugabe des Dichtegradientenmediums machen die Flüssigkeitsströmung sichtbar.
- Stellen Sie sicher, dass die Würmer aus dem Einlass erscheinen und zur Rückseite des Kanal-Arrays fließen.
- Schieben Sie die Tiere vorsichtig in Richtung der Kanalanordnung. Sobald ein Tier dem Kanal zugewandt ist, schiebe es in den Kanal und wiederhole den Vorgang für weitere Tiere.
HINWEIS: Die Tiere müssen mit dem Kopf zum Auslass ausgerichtet werden, um einen einfachen Zugang zum Futter zu haben. Sie können durch Drücken und Ziehen am Kolben und durch Schwimmen orientiert werden. - Sobald genügend Tiere gefangen wurden, legen Sie die Spritze, die noch am Wurmeinlass befestigt ist, auf den Mikroskoptisch, wo sie während des gesamten Versuchs verbleibt. Es kann erforderlich sein, den Wurmschlauch kurz von der Spritze zu lösen, um den Druckaufbau abzubauen, und ihn dann vorsichtig wieder an der stumpfen Nadel anzubringen.
- Wenn ein Präpariermikroskop geladen wurde, übertragen Sie das Gerät auf das bildgebende Mikroskop. Der Schlauch kann am Chip befestigt bleiben, und die Spritze wird mit der Pumpe verbunden.
- Schalten Sie die Spritzenpumpe ein und lassen Sie sie mit der voreingestellten Geschwindigkeit von 1 μl/h für 0,5 μl laufen, erhöhen Sie die Geschwindigkeit um 100 μl/h für 0,5 μl und reduzieren Sie sie wieder auf 1 μl/h. Dieser Zyklus wird automatisch für das gesamte Experiment wiederholt. Informationen zur Programmierung der Pumpe finden Sie in der Bedienungsanleitung des Geräts.
- Stellen Sie das Gerät auf den Mikroskoptisch und stellen Sie sicher, dass es fest sitzt (Vorschläge für Montagemechanismen finden Sie in Abbildung 3A).
- Wenn das Bildgebungsmikroskop nicht geladen wurde, identifizieren Sie die gewünschte Geräteeinheit bei geringer Vergrößerung.
- Wechseln Sie zur gewünschten Bildvergrößerung.
HINWEIS: Die Geräte sind mit allen Objektivtypen kompatibel. - Identifizieren Sie die Tiere und Regionen von Interesse innerhalb des Fallenkanal-Arrays und richten Sie die gewünschten Bildgebungsbedingungen ein.
- Das Bild wird unter den gewünschten Bildgebungsbedingungen aufgenommen, wobei das On-Chip-Ventil 10 s vor der Bildaufnahme durch den Magneten betätigt wird, so dass die Tiere an Ort und Stelle gehalten werden.
HINWEIS: Die Betätigung des On-Chip-Ventils führt zu einer Verlängerung und Abflachung der Tiere, was möglicherweise Anpassungen des interessierenden Bildbereichs (ROI) erfordert.
6. Vorbereitung der Einrichtung
HINWEIS: Dieser Abschnitt bezieht sich auf die anfängliche Vorbereitung des Gerätesteuerungsmechanismus und muss nicht vor jedem Versuch wiederholt werden. Siehe Abbildung 3B,C. Da viele Magnete eine höhere Spannung benötigen, wird der Magnet höchstwahrscheinlich nicht direkt mit dem Mikrocontroller, sondern über ein Relais oder einen MOSFET verbunden sein (siehe Abbildung 3C).
- Identifizieren Sie eine Druckluftquelle (z. B. Wandinstallation oder Gasflasche).
- Schließen Sie die Druckquelle über einen 6-mm-Schlauch (Wandquelle) an einen geeigneten Druckregler (0-2 bar Druckbereich) an oder befestigen Sie einen geeigneten Regler direkt an der Gasflasche.
- Verbinden Sie den Druckregler mit dem Eingang des Magnetventils (6 mm Schlauch, siehe Abbildung 3B).
- Platzieren Sie den Magneten so nah wie möglich am Mikroskoptisch (~0,5-1 m vom Tisch wird empfohlen, obwohl bei Bedarf größere Entfernungen möglich sind).
- Verbinden Sie das Magnetventil elektronisch mit der gewählten Steuerung. Es stehen verschiedene Optionen zur Verfügung, wie z. B. die Steuerung über eine separate Mikrocontroller-Karte (z. B. Arduino) oder eine vorhandene DAq-Karte.
7. Reinigung und Lagerung
- Sobald das Experiment abgeschlossen ist, nehmen Sie das Gerät aus dem Mikroskop.
- Entfernen Sie die Tauchflüssigkeit. Immersionsöl kann mit organischen Lösungsmitteln (z. B. Aceton, Methanol usw.) entfernt werden.
- Stellen Sie das Gerät auf eine ebene Fläche und trennen Sie alle Schläuche außer den Ventilschläuchen. Um jeden Schlauch sicher zu trennen, drücken Sie auf das PDMS in der Nähe und ziehen Sie vorsichtig. Heben Sie das Gerät nicht an und biegen Sie es nicht, da dies zum Bruch des Deckglases führen kann.
- Reinigen Sie die PDMS-Oberfläche mit 70% Ethanol.
- Lagern Sie das Gerät bei Raumtemperatur mit angeschlossenem Ventilschlauch.
HINWEIS: Das Gerät kann wiederverwendet werden, bis alle Geräte verwendet wurden, wahrscheinlich ohne dass der Ventilschlauch nachgefüllt werden muss. Das Wasser in den Ventilschläuchen verdunstet jedoch über einen längeren Zeitraum und muss möglicherweise nachgefüllt werden.
Ergebnisse
Aufbau und Abmessungen des Geräts
Fünf verschiedene Geräte wurden entwickelt, um alle vier Larvenstadien aufzunehmen. Diese Vorrichtungen werden wie folgt bezeichnet: L1 für L1-Larven, die unmittelbar nach dem Schlüpfen geladen und bis zum L2-Stadium abgebildet werden; L1-L4, fähig, Larven vom mittleren bis späten L1-Stadium bis zum frühen bis mittleren L4 zu halten; L2-A, geeignet für Tiere etwa ab dem mittleren L2-Stadium bis zum jungen Erwachsenenalter; L3-A, für Tiere etwa ab dem mittleren L3-Stadium bis zum jungen Erwachsenenalter; und L4-A, das Tiere etwa vom späten L4-Stadium bis zum jungen Erwachsenenalter beherbergt.
Alle Geräte folgen dem gleichen Layout, bestehend aus einem Wurmeinlass, 1 (oder 2 im Falle eines Medienwechsels) bakteriellen Nahrungseinlässen und einem gemeinsamen Abfallauslass. Zwischen dem Einlass- und Auslassteil befindet sich eine Anordnung von 41 parallelen Siphonkanälen, wobei die Einlass- und Auslassseite effektiv durch den Nahrungszufuhrkanal mit niedrigerer Höhe getrennt sind (Abbildung 1A, A). Die Kanalabmessungen wurden so gewählt, dass sie deutlich länger und breiter sind als die zu Beginn eines Experiments geladenen Schnecken, mit einer Länge/Breite von 400/15 μm für L1-Bauelemente, 575/22 μm für L1-L4-Bauelemente, 800/27 μm für L2-A-Bauelemente, 900/30 μm für L3-A-Bauelemente und 1000/65 μm für L4-A-Bauelemente (Zusatzdatei 1, Zusatzdatei 2, Zusatzakte 3, Zusatzakte 4 und Zusatzakte 5).
Die Kanallänge wurde so gewählt, dass sie der Größe entsprach, die die Tiere am Ende des Versuchs erreichten, während die Breite so gewählt wurde, dass die Würmer zu Beginn des Versuchs etwa zwei Drittel der Kanalbreite einnahmen. Diese Konstruktion ermöglicht ausreichend Platz für Wachstum und Bewegung während der Häutung und verhindert gleichzeitig, dass sich die Würmer drehen oder drehen. Im Gegensatz zur Breite wurde die Kanalhöhe zu Beginn des Versuchs nahe an der Dicke der Würmer festgelegt: 8 μm für frühe L1-Larven, 12 μm für mittlere/späte L1-Larven, 15 μm für mittlere L2-Larven, 17,5 μm für mittlere L3-Larven und 22,5 μm für späte L4-Larven, entsprechend den Larven L1, L1-L4, L2-A, L3-A, bzw. L4-A-Geräte. Die reduzierte Kanalhöhe in Kombination mit einer sorgfältig ausgewählten Breite (unter Beibehaltung eines Höhen-Breiten-Verhältnisses von 0,5-0,6) stellt sicher, dass die Schnecken in einer festen Ausrichtung bleiben. Bemerkenswert ist, dass bei einer ausreichend kleinen Kanalhöhe alle Schnecken konstant in der gewünschten Querausrichtung positioniert sind.
Der Futterzufuhrkanal wurde in einer Höhe von 3 μm für L1 und 5 μm für alle anderen Geräte hergestellt, um sicherzustellen, dass Bakterien die Tiere leicht erreichen können, ohne dass die Tiere aus den Fangkanälen entweichen. Alle Geräte verwenden die gleiche Lebensmittelversorgungsschicht (Zusatzdatei 6) und die gleiche Ventilschicht (Zusatzdatei 7). Beachten Sie, dass die Ventilschicht auf einem separaten Wafer in einer Höhe von 20 μm hergestellt und erst bei der PDMS-Bauelementherstellung (Schritt 1.21) auf die Bauteilschicht aufgesetzt wird.
Diese stadienspezifischen geometrischen Einschränkungen, die unerlässlich sind, um zu verhindern, dass sich Würmer vom Kopf bis zum Schwanz drehen oder umkehren, erfordern die Verwendung von drei verschiedenen Gerätetypen, um alle Larvenstadien zu berücksichtigen. Würden L1-Larven in einen Kanal gesetzt, der für junge erwachsene Tiere ausgelegt ist, würden sie nicht stabil bleiben und den Fallenkanal schnell verlassen.
Zubereitung von C. elegans
Sofern nicht anders angegeben, wurden die Würmer gemäß dem Standardprotokoll25 auf NGM-Platten gehalten, die mit E. coli OP50 bei 20 °C besiedelt waren. Der Abschnitt zur Wurmvorbereitung beschreibt explizit die Vorbereitung einer synchronisierten Wurmpopulation, die in einem bildgebenden Experiment verwendet wird. Die Synchronisation erfolgte gemäß den Standardprotokollen unter Verwendung von Natriumhypochloritbleiche26, modifiziert mit einem zusätzlichen Filtrationsschritt, um Schmutz und nicht geschlüpfte Tiere nach dem Hungern über Nacht zu entfernen. Ziel ist es, viele synchronisierte L1-Larven zu erhalten, die dann auf NGM-Platten ausgesät oder direkt in das Bildgebungsgerät geladen werden. Andere Synchronisationsmethoden, wie die manuelle Kommissionierung oder die kontrollierte Eiablage, sind ebenso geeignet. Sie sind jedoch mühsamer, wenn es darum geht, genügend Tiere zu erzeugen (empfohlen werden 200 pro Zustand).
Sobald die Würmer das gewünschte Stadium erreicht haben, können sie mit sauberem S-Basal (idealerweise gefiltert durch einen 0,2 μm-Filter) vorsichtig von der NGM-Platte abgewaschen werden. Ziel ist es, so viele Tiere wie möglich zu sammeln, ohne dass sich Bakterien oder Schmutz von der Platte ansammeln. Die Tiere werden in einem kleinen Zentrifugenröhrchen gesammelt und 2-3 Mal mit frischem S-Basal-Puffer gewaschen, um schließlich so viel Flüssigkeit wie möglich zu entfernen. Die Tiere werden dann direkt aus dem konzentrierten Volumen entnommen. Wenn die Tierkonzentration zu niedrig ist, erfolgt die Beladung langsamer, sodass ein größeres Flüssigkeitsvolumen durch das Gerät fließen muss und das Risiko steigt, die Fallenkanäle mit Schmutz zu blockieren.
Bakterielle Lebensmittelzubereitung
Das Bakterienwachstum erfolgt nach Standardprotokollen, wobei die anfängliche Bakterienkonzentration (OD600 = 1,9) in S-Basalpuffer (filtriert durch einen 0,2-μm-Filter) um das 40-fache erhöht und mit dem Dichtegradientenmedium und dem S-Basalpuffer mit PF (gefiltert durch einen 0,2-μm-Filter) gemischt wird. Das erwartete Ergebnis dieses Prozessschrittes ist die Herstellung einer sehr dichten Bakteriensuspension und die Verhinderung der Bakteriensedimentation während des bildgebenden Experiments. Die hohe Dichte ist notwendig, um die Tiere bei den niedrigen Durchflussraten der Medienförderung (1 μL/h) auf dem Chip zu füttern. Der Zweck des Dichtegradientenmediums besteht darin, die Ausfällung von Bakterien während des Experiments zu verhindern, und der Zweck von PF besteht darin, zu verhindern, dass Bakterien an den Kanalwänden haften bleiben. Sowohl das Dichtegradientenmedium als auch das PF sind ungiftig und beeinträchtigen die Lebensfähigkeit der Tiere nicht.
Wenn in einem Experiment eine andere Bakterienart verwendet wird, muss die erforderliche Konzentration des Dichtegradientenmediums bestimmt werden. Dies kann erreicht werden, indem die Bakteriensuspension mit unterschiedlichen Mengen des Mediums gemischt und anschließend zentrifugiert wird. Wenn die Konzentration des Dichtegradienten zu niedrig ist, sedimentieren Bakterien; Ist die Konzentration zu hoch, schwimmen Bakterien. Sobald eine geeignete Konzentration gefunden wurde, wird empfohlen, das Ergebnis zu validieren, indem die Bakterienmischung etwa 48 Stunden lang bei Raumtemperatur belassen wird, um anzuzeigen, wie sich das Lebensmittel während des gesamten Versuchs verhalten wird. Nach dem Experiment empfiehlt es sich ebenfalls, die Spritze, durch die die Bakterien zugeführt werden, zu überprüfen und sicherzustellen, dass die Bakterien nicht sedimentieren oder schwimmen. Der Volumenunterschied, der sich aus der Zugabe unterschiedlicher Mengen Optiprep ergibt, kann durch Zugabe von mehr oder weniger S-Basal + 1 Gew.-% PF ausgeglichen werden. In ähnlicher Weise können zusätzliche Medien oder Verbindungen (z. B. Auxin) zu der Suspension hinzugefügt werden, und das zusätzliche Volumen wird von dem Volumen von S-Basal + 1 Gew.-% PF subtrahiert.
Vorbereitung der Einrichtung
Stellen Sie das Druckversorgungssystem so nah wie möglich am Mikroskop auf, so dass es bequem bedient werden kann. Alle Rohrverläufe, insbesondere der 1/16-Zoll-Schlauch, der mit dem Chip verbunden ist, sollten kurz bleiben (weniger als 0,5 m für den Schlauch mit einem Außendurchmesser von 1/16 Zoll) (Abbildung 2 und Abbildung 3B). Wie im obigen Abschnitt angedeutet, kann die Computersteuerung des On-Chip-Ventils auf verschiedene Weise erreicht werden. Dies sollte in jeder Mikroskopsoftware (getestet in Micro-Manager, Metamorph, NIS und ZenBlue) möglich sein, entweder durch den Anschluss einer dedizierten Mikrocontroller-Platine (z. B. Arduino) oder durch den Anschluss des Systems an eine vorhandene Datenerfassungskarte (DAq) über einen digitalen Ausgang. Der ausgewählte Platinenausgang sollte dann als Shutter konfiguriert werden, der sich 10 s vor der Bildaufnahme einschaltet oder während der Geräteeinrichtung manuell betätigt wird. Wie bereits erwähnt, wird es nicht empfohlen, den Magneten direkt an den Mikrocontroller anzuschließen. Stattdessen erfolgt der Anschluss über einen MOSFET- oder Relaisschalter, so dass der Magnet und der Mikrocontroller elektronisch isoliert bleiben (Abbildung 3C).
Ähnlich wie bei den Druckzuleitungen sollten alle anderen Schlauchverläufe zum Chip (1/16" AD und 1/32" AD) so kurz wie möglich gehalten werden. Halten Sie die Schnecke und die Abfallschläuche (1/16" AD) unter 2,5 cm bzw. 20 cm. Der Bakterienzufuhrschlauch (1/32" AD) muss lang genug sein, um von der Spritzenpumpe mit dem Gerät verbunden zu werden, sobald er auf den Mikroskoptisch gestellt wird. Es wird jedoch dringend empfohlen, diese so weit wie möglich zu verkürzen und die Spritzenpumpe in der Nähe des Mikroskoptisches zu platzieren, z. B. neben dem Tisch, über ein Regal oder auf einem Inkubatorgehäuse. Jede Länge von Schläuchen, die dem System hinzugefügt werden, erhöht den Gegendruck, der beim Laden von Tieren in das Gerät zu spüren ist, und erhöht den Gesamtdruck auf die gefangenen Tiere.
Bedienung des Geräts
Zu diesem Zeitpunkt können alle Schläuche vorbereitet und mit der jeweiligen stumpfen Nadel, dem Stahlstift und der Spritze verbunden werden (Beginn von Protokollschritt 4). Halten Sie, wie oben erwähnt, die Schlauchlänge so kurz wie möglich (Abbildung 2J-N). Wichtig ist, dass darauf geachtet wird, dass keine Luftblasen in den Spritzen oder Schläuchen verbleiben, da diese die Beladung der Tiere beeinträchtigen und dazu führen können, dass die Tiere während des Versuchs aus dem Fallenkanal entweichen. Luftblasen können auf verschiedene Arten aus der Spritze entfernt werden, z. B. durch Befüllen der Spritze, Umdrehen und leichtes Schütteln, so dass die Luftblasen an die Oberseite der Spritze steigen (ein Schnippen der Spritze wird nicht empfohlen, da dies dazu führen kann, dass zusätzliche Luftblasen eingeschlossen werden). Alternativ kann die Spritze befüllt werden, indem sie in den Puffer gesteckt und der Kolben schnell auf und ab bewegt wird, um die meisten Blasen schnell zu entfernen.
Sobald die Spritze gefüllt und blasenfrei ist, werden die stumpfe Nadel und der Schlauch angebracht und mit Puffer gefüllt. Auch beim Befüllen der Spritze ist besondere Vorsicht geboten, da im Kunststoffteil der stumpfen Nadel leicht Luft eingeschlossen werden kann. Wenn Luft eingeschlossen ist, setzen Sie die Nadel wieder ein. Alternativ kann das Vorbefüllen der Nadel mit Flüssigkeit, z. B. mit einer Pipette, das Problem lindern.
Wenn alle Komponenten an Ort und Stelle sind, kann der Schlauch am Gerät befestigt werden, beginnend mit dem Schlauch, der das Magnetventil und das On-Chip-Ventil verbindet (Abbildung 2A-C,J). Dieser Schlauch wird zunächst mit entionisiertem Wasser gefüllt, und das System wird unter Druck gesetzt, um die gesamte Luft aus dem Ventilkanal in der Sackgasse zu entfernen. PDMS ist gasdurchlässig; Somit drückt der erhöhte Druck die gesamte Luft aus dem Kanal in das PDMS, wodurch der Ventilkanal mit Wasser gefüllt wird. Dieser Vorgang kann am Mikroskop verfolgt werden, wobei sichergestellt wird, dass der gesamte Ventilkanal gefüllt ist. Nach dem Befüllen des Ventils kann die Bakterienzufuhr angeschlossen werden, und das Gerät kann befüllt und unter Druck gesetzt werden, indem alle offenen Ein- und Ausgänge blockiert werden (Abbildung 2D, E, K, L). Luft wird verdrängt und der Prozess kann am Mikroskop überwacht werden. Sobald die gesamte Luft verdrängt ist, sollten die Bakterien das Gerät gleichmäßig füllen. Die Absperrstifte werden entfernt und das Abfallsammelrohr angeschlossen (Abbildung 2F,G,M). Zu diesem Zeitpunkt sollte eine kleine Menge Flüssigkeit durch das Gerät in das Abfallsammelrohr fließen, wobei darauf zu achten ist, dass alle Verbindungen zum Rohr offen sind. Dies kann visuell bestätigt werden, bevor der Schlauch mit dem Sammelrohr verbunden wird. Wenn keine Flüssigkeit in das Abfallsammelrohr fließt, ist es wahrscheinlich verstopft. Dies würde zu einem Druckaufbau über dem Versuch führen, der sich negativ auf die gefangenen Tiere auswirken würde. Wenn der Schlauch oder seine Anschlüsse verstopft sind, entfernen Sie ihn und reinigen Sie alle Verbindungen mit einer Nadel oder spülen Sie sie mit einer wassergefüllten Spritze aus.
Abschließend kann der Schneckenschlauch angeschlossen werden (Bild 2H,N). Würmer werden aus dem Zentrifugenröhrchen aufgenommen, indem sie einfach in den Schlauch gesaugt werden, der an der S-Basal-gefüllten Spritze befestigt ist. Die Tiere sollten nur in den Schlauch gezogen werden, nicht in die Spritze. Der Schlauch kann dann mit dem Gerät verbunden werden. Zuvor wird eine kleine Menge Flüssigkeit aus dem Gerät und dem Wurmschlauch herausgedrückt und die beiden Tröpfchen so verbunden, dass beim Beladen keine Luft in das Gerät gedrückt wird.
Laden von Schnecken
Wenn alle Verbindungen hergestellt sind, können die Tiere durch Drücken und Ziehen am Kolben der Wurmspritze geladen werden. Die Tiere werden aus dem Einlass in Richtung des Fallenkanals gespült. Sie schwimmen jedoch von Natur aus gegen den Strom, den sie erleben. Die Tiere müssen daher so ausgerichtet werden, dass ihr Kopf zum Fallenkanal zeigt. Die korrekte Ausrichtung der Tiere wird erreicht, indem man den Kolben drückt und zieht und den Tieren Zeit gibt, sich in die gewünschte Ausrichtung zu drehen. Dies kann fast auf der Basis eines einzelnen Wurms erreicht werden, erfordert aber etwas Übung. Sobald die Tiere richtig ausgerichtet sind, können sie in den Fallenkanal geschoben werden und bis zum Ende des Kanals gleiten, wo sie durch eine Höhenstufe gestoppt werden (Abbildung 1A, B). Wenn Tiere nicht in den Kanal gelangen können, sind sie zu groß für das ausgewählte Gerät und müssen in einem größeren Gerät abgebildet werden. Wenn sich Tiere drehen oder drehen können, wenn sie in den Fallenkanal gesetzt werden, sind sie zu jung und es muss ein kleineres Gerät verwendet werden, oder die Tiere müssen noch einige Stunden wachsen gelassen werden.
Wichtig ist, dass sich Tiere in verschiedenen Kanälen nicht gegenseitig beeinflussen. Daher wirken sich falsch verladene Tiere nicht auf die korrekt verladenen Tiere aus, haben jedoch keinen ausreichenden Zugang zu Futter und wachsen daher langsamer oder stoppen die Entwicklung.
Wenn die Tiere im richtigen Alter geladen werden, sollte eine minimale Bewegung entlang der Längsachse des Kanals auftreten. Die Tiere können jedoch frei wackeln (Abbildung 1B) und beginnen innerhalb weniger Minuten mit der Fütterung. Wenn Tiere eine signifikante Bewegung entlang der Längsachse des Kanals zeigen, wird dies wahrscheinlich durch einen Druckaufbau im Gerät oder durch eine Luftblase verursacht, die sich irgendwo im Schlauch, der stumpfen Nadel oder der Spritze festgesetzt hat. Der Druckaufbau im Inneren des Gerätes kann abgebaut werden, indem der Wurmschlauch vorsichtig von der stumpfen Nadel gelöst und vorsichtig wieder angebracht wird. Wenn trotz der oben genannten Schritte und Vorsichtsmaßnahmen eine Luftblase im System geblieben ist, muss möglicherweise die gesamte Wurmspritze entfernt und durch eine neue ohne Luftblasen ersetzt werden. Luftblasen im System fungieren als Federn, die während der Beladung zusammengedrückt und nach dem Verladen der Tiere langsam gelöst werden. Die Strömung, die durch die Ausdehnung der Luftblasen entsteht, kann zu Systeminstabilitäten führen, die die Tiere aus den Fallenkanälen drücken.
Bilderfassung und -entwicklung auf dem Chip
Sobald die Tiere verladen sind, kann die Bildaufnahme beginnen. Prinzipiell sind die Geräte mit den meisten bildgebenden Verfahren kompatibel, Hellfeld, Epifluoreszenz 18,19,22,23 (Abbildung 4), konfokale Spinning-Disk 19 und sogar superauflösende Modalitäten (AiryScan24), da die gesamte Bildgebung durch ein 170 μm dickes Deckglas erfolgt. Folglich zeigt das mikrofluidische Gerät wenig bis gar keinen Einfluss auf die erzielbare Bildqualität auf dem Chip. Unabhängig von der Bildgebungsmodalität müssen jedoch geeignete Bildgebungsbedingungen ausgewählt werden, die die Lebensfähigkeit der Tiere nicht beeinträchtigen. Hohe Anregungsintensitäten führen aufgrund der hohen Energie und Wärme, die auf die Probe ausgeübt wird, leicht zu einer Fluorophorbleiche und einem Entwicklungsstillstand. Kurze Belichtungszeiten (~10 ms) und niedrige Anregungsintensitäten (weniger als 10 % einer typischen Fluoreszenz-LED-Quelle) werden empfohlen, um ein brauchbares Bild bei einer Kontrasteinstellung von min = 0 und max = 1000 (bei Verwendung einer 16-Bit-Kamera) zu erhalten (Abbildung 4).
Repräsentative Daten, die mit den Geräten L1, L1-4 und L2-A erhalten wurden, sind in Abbildung 4 dargestellt. Darstellung der Entwicklung der Epithelzellen von C . elegans vom Schlüpfen/Hungern über Nacht bis zum mittleren L2-Larvenstadium (Abbildung 4A, Ergänzender Film 1), Induktion der 1°-schicksalhaften Vulva-Vorläuferzellen und deren anschließende Teilung vom späten L1- bis zum frühen L4-Larvenstadium (Abbildung 4B, Ergänzender Film 2) und schließlich die Bildung der C . elegans-Vulva vom frühen L3-Larvenstadium bis zur Eversion beim Übergang zum Erwachsenenalter (Abbildung 4C, Ergänzungsfilm 3).
Jede Anwendung wird von einer Quantifizierung verschiedener Entwicklungszeitmetriken begleitet. Die Bilder in Abbildung 4A,B werden ohne Nachbearbeitung angezeigt, d.h. es wurde keine Dekonvolution oder Bildregistrierung angewendet. Die Bilder in Abbildung 4C hingegen wurden entfaltet27 und registriert, was die Verbesserungen der Bildqualität hervorhebt, die bei der Verwendung von Langzeitbildgebungsgeräten möglich sind.
Zunächst wird der Beginn jeder Nahtzellteilung bei Tieren in Abbildung 4A identifiziert (siehe Ergänzender Film 1 für einen Vollzeitkurs), was auf die konsistente und rechtzeitige Teilung aller Zellen bei 12 Tieren hinweist (Abbildung 4D). Anhand der Nahtzellen wurde auch der allgemeine Entwicklungszeitpunkt über alle vier Larvenstadien hinweg bewertet und es wurde festgestellt, dass alle Nahtzellteilungen nach 12,0 h ± 1,7 h (mittlere ± SD) in L1 (n = 22), 10,2 h ± 1,0 h in L2 (n = 27), 10,9 h ± 1,4 h in L3 (n = 27) und 14,6 h ± 2,2 h bei L4-Tieren (n = 19) abgeschlossen waren. Diese Daten stimmen mit den Literaturwerten 2,28 sowie mit den Werten überein, die an Tieren gemessen wurden, die auf Platte gezüchtet wurden, ergaben mediane Entwicklungszeiten für L1 15 h (n = 21), L2 10 h (n = 15), L3 12 h (n = 21) und L4 12,5 h (n = 61) (Adaptiert von Berger et al.18). Die beobachteten leichten Verzögerungen sind wahrscheinlich eine Folge der Bildaufnahme und der daraus resultierenden Phototoxizität oder der erhöhten Gefangenschaft, sobald die Tiere herangewachsen sind und einen Fangkanal bilden.
Zweitens verfolgten wir das Wachstum der Tiere in den Stadien L2 und L3 und quantifizierten die Länge der Keimdrüsen als hervorragenden Indikator für den Entwicklungsfortschritt (Abbildung 4E, siehe Ergänzungsvideo 2 für einen Vollzeitkurs). Wie bei der Teilung der Nahtzellen konnte dank der geraden Tierorientierung die genaue Gonadenlänge gemessen werden, und es wurde ein konsistentes Gonadenwachstum bei allen abgebildeten Tieren festgestellt (n = 19). Die erste VPC-Teilung in diesen Experimenten wurde 14,26 h ± 2,47 h nach Beginn des Experiments (~30 h nach der Aussaat) beobachtet, die zweite 1,46 h ± 0,13 h später (n = 19). Diese Daten stimmen gut mit den Literaturwerten überein, die auf NGM-Platten ermittelt wurden29,30. Euling und Ambros30 stellten fest, dass die erste Teilung nach etwa 29 Stunden und die zweite etwa 3 Stunden später stattfand.
Schließlich wurde die Zeit, bis zu der bestimmte Stadien der Vulvaentwicklung erreicht sind, quantifiziert (Abbildung 4F, siehe Ergänzungsfilm 3 für den Vollzeitkurs), wobei speziell die Zeit bewertet wurde, die benötigt wird, um von der Einstülpung (L4.0, t1) bis zum mittleren L4-Stadium (L4.5, t4) gemäß den von Mok et al.31 definierten Unterstadien fortzuschreiten. Die durchschnittliche Progressionszeit zwischen t1 und t2 betrug 3,8 h ± 1,2 h, zwischen t2 und t3 3,5 h ± 1,2 h und zwischen t3 und t4 2,8 h ± 1,6 h (n = 7), was auf einen annähernd linearen Entwicklungsverlauf hinweist. Ähnlich wie beim Entwicklungs-Timing-Assay erscheinen diese Zeiten, insbesondere t1-t2, im Vergleich zu den berichteten Literaturwerten (t1-t2: 0,7 h, t2-t3: 3 h, t3-t4: 2,3 h31) leicht verzögert, möglicherweise aufgrund der schmalen Geräteabmessungen, die die Häutung verlangsamen. Nichtsdestotrotz bleibt der Entwicklungszeitpunkt bei den Tieren sehr konstant, und alle schreiten erfolgreich ins Erwachsenenalter voran.
Kompensation von Tierbewegungen und Mikroskopdrift
Die Bewegung des Tieres in einem einzelnen Z-Stapel ist in der Regel minimal, wenn das On-Chip-Ventil betätigt wird. Es wird jedoch nicht erwartet, dass die Tiere völlig still stehen. Der verbleibende Jitter, der in einem einzelnen Stapel beobachtet wird, kann kompensiert werden, indem der Druck zur Betätigung des On-Chip-Ventils erhöht, die verwendete Belichtungszeit verringert oder der ausgewählte Z-Schritt erhöht wird, um die Stapelerfassung zu beschleunigen. Die Erfassungszeit des Stacks kann weiter reduziert werden, indem ein Piezo-Z-Antrieb verwendet wird, was zu einer deutlich schnelleren Z-Bewegung im Vergleich zu einem herkömmlichen Mikroskop-Fokusantrieb führt, oder durch die Verwendung von Multiband-Filtern (z. B. GFP/mCherry), so dass separate Farben ohne zeitaufwändige Filterwechsel erfasst werden können. Als Referenz gilt: In den bereitgestellten Beispielen (Abbildung 4 und Ergänzender Film 1, Ergänzender Film 2 und Ergänzender Film 3) lag die Stapelerfassungszeit in der Regel zwischen 3 und 8 Sekunden. Restbewegungen können bei Bedarf auch durch verschiedene Bildregistrierungsmethoden entfernt werden.
Es wird erwartet, dass die Anzahl der Tiere auf dem Chip stetig wachsen wird. Daher verschieben sich die interessierenden Merkmale unweigerlich entlang der longitudinalen Kanalachse (Abbildung 4A, B, Ergänzender Film 1 und Ergänzender Film 2). Daher wird empfohlen, das Bildfeld so einzustellen, dass das interessierende Merkmal in das Sichtfeld hineinwachsen kann und im Verlauf eines Experiments nicht verloren geht (Abbildung 1B und Abbildung 4A,B). Alternativ können Tiere in mehreren nebeneinander liegenden FOVs abgebildet werden, die den gesamten Tierkörper abdecken. Dies kann jedoch zu zusätzlicher Phototoxizität und Stress führen. Wie bei der Z-Bewegung können verschiedene Registrierungsverfahren die Bewegung der Tiere im Kanal über die Zeit ausgleichen. Wenn die Tiere eine unregelmäßige Bewegung entlang der Längsachse zeigen, die größer ist als das, was auf das Wachstum zurückzuführen ist, ist dies wahrscheinlich auf eine Luftblase zurückzuführen.
Jedes Langzeitbildgebungsgerät enthält sechs einzelne Bildgebungseinheiten, die jeweils für ein separates Experiment verwendet werden können. Jede Einheit kann bis zu 41 Tiere beherbergen, und die Fallenkanäle sind so angeordnet, dass mehrere Tiere in einem einzigen Sichtfeld abgebildet werden können. Darüber hinaus können mehrere FOVs in einem einzigen Gerät oder Gerät abgebildet werden. Wie bei allen Mikroskopproben kann es durch Bewegung über die Probenoberfläche aufgrund des Verlusts von Immersionsmedien zu Fokusdrift kommen. Diese Drift kann durch die Verwendung eines Immersionsöls mit niedrigerer Viskosität oder durch verschiedene Autofokus-Modalitäten kompensiert werden. Einige Hardware-Autofokus-Systeme funktionieren jedoch möglicherweise nicht, wenn sie mit einem PDMS-Mikrofluidikgerät verwendet werden, da diese den Übergang von Glas zu einem wässrigen Medium als Referenzpunkt (Probenseite des Deckglases) verwenden. Ein Großteil des Sichtfelds ist im Inneren des Geräts mit Silikon gefüllt, das einen höheren Brechungsindex als Wasser hat. Dies wirkt sich zwar nicht auf die Bildqualität aus, kann sich aber negativ auf den Autofokus auswirken. Bei der Abbildung eines einzelnen FOV ist die im Gerät beobachtete Fokusdrift minimal (weniger als 2 μm). Wenn eine übermäßige Fokusdrift beobachtet wird, kann dies auf die thermische Instabilität des Mikroskopsystems oder der Umgebung, auf übermäßige XY-Bewegungen, die mehrere FOVs erzeugen, oder auf eine unsachgemäße Montage des Geräts am Mikroskop zurückzuführen sein (siehe Abbildung 3A für empfohlene Montagemechanismen).
In ähnlicher Weise können auch übermäßige XY-Bewegungen und unsachgemäße Montage zu XY-Drift führen. Da das Gerät und das Mikroskopobjektiv über die Tauchflüssigkeit verbunden sind, können schnelle Bewegungen in X, Y oder Z dazu führen, dass Kräfte auf das Gerät wirken. Wenn die Geräte korrekt montiert sind und keine äußeren Kräfte auf sie einwirken, sind sie außergewöhnlich stabil mit minimaler Fokusdrift und ohne XY-Drift.
Wenn schließlich bestimmte Systemparameter ungeeignet sind, können sich die mikrofluidischen PDMS-Geräte negativ auf die Entwicklung der Tiere auswirken. Die Lebensfähigkeit wird durch übermäßigen Ventildruck, hohe Phototoxizität aufgrund hoher Anregungsintensitäten, lange Belichtungszeiten oder kurze Zeitintervalle zwischen den Stack-Erfassungen beeinträchtigt. Insbesondere bei der ersten Verwendung dieses Systems wird dringend empfohlen, die Lebensfähigkeit der Tiere mit einem hellen Fluoreszenzmarker oder einer Hellfeldbildgebung zu beurteilen, um sicherzustellen, dass die Bildgebungsbedingungen das Wachstum der Tiere nicht beeinflussen. Sobald sich die Tiere zuverlässig entwickeln, können die Bildgebungsparameter je nach Bedarf variiert werden.
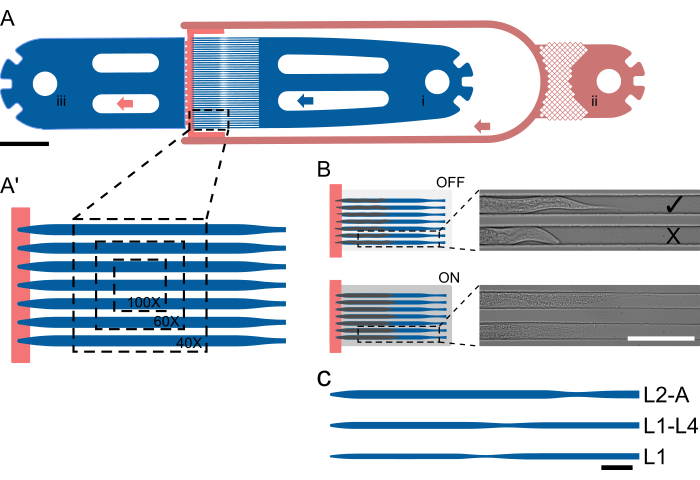
Abbildung 1: Gerätelayout und schematische Bedienung. (A) Geräteübersicht zeigt eine einzelne Einheit mit dem Wurmeinlass (i), dem bakteriellen Nahrungseinlass (ii) und dem allgemeinen Auslass (iii). (A') Vergrößerte Ansicht des Fallenkanals in einem L2-A-Gerät, die die Anzahl der Würmer hervorhebt, die in einem einzigen FOV bei unterschiedlichen Vergrößerungen (40x-100x) sichtbar sind. (B) Funktionsprinzip des On-Chip-Ventils. Wenn AUS, wird die Kanalhöhe so gewählt, dass die Tiere bequem in den Fallenkanal passen. Im eingeschalteten Zustand verringert sich die Kanalhöhe, wodurch das Tier auf das Deckglas gedrückt wird, wodurch die gefangenen Tiere reversibel bewegungsunfähig werden. Die Bilder zeigen zwei gefangene Tiere, die mit ausgeschaltetem On-Chip-Ventil und eingeschaltetem Ventil aufgenommen wurden, wobei sich die Tiere unter Druck sichtbar aufrichteten und streckten. Das obere Tier wurde korrekt geladen und zeigt zur Futterquelle (), das untere zur Futterquelle ( X). (C-C") Schaltplanmäßige Geräteeinrichtung. (C) Verfügbare Gerätegrößen. L1 ist für Tiere vom L1- bis zum mittleren L2-Stadium, L1-4 für Tiere vom mittleren L2- bis zum mittleren L4-Larvenstadium und L2-A für Tiere vom mittleren L2-Larvenstadium bis zum Erwachsenenalter geeignet. Maßstabsleisten sind (A) 1000 μm, (B) 100 μm und (C) 50 μm. Die Abbildung ist teilweise von Berger et al.18 übernommen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
X). (C-C") Schaltplanmäßige Geräteeinrichtung. (C) Verfügbare Gerätegrößen. L1 ist für Tiere vom L1- bis zum mittleren L2-Stadium, L1-4 für Tiere vom mittleren L2- bis zum mittleren L4-Larvenstadium und L2-A für Tiere vom mittleren L2-Larvenstadium bis zum Erwachsenenalter geeignet. Maßstabsleisten sind (A) 1000 μm, (B) 100 μm und (C) 50 μm. Die Abbildung ist teilweise von Berger et al.18 übernommen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
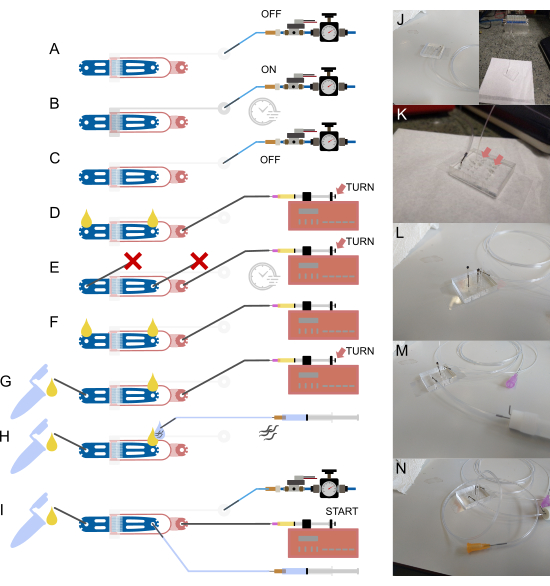
Abbildung 2: Geräteeinrichtung und Verbindungen. (A-I) Schematische Übersicht über den Einrichtungsprozess. (A) Anschluss eines 1/16" wassergefüllten Schlauchs an den Ventileinlass mit einem hohlen Stahlstift (grau), wobei das andere Ende am Druckversorgungssystem befestigt ist. (B) Befüllen des On-Chip-Hydraulikventils über den Off-Chip-Magneten und Beaufschlagen des Geräts für einige Minuten. (C) Deaktivierung des Drucks, bevor Sie mit den nächsten Schritten fortfahren. (D) Anschluss einer Spritze mit bakteriellem Futter an den Lebensmitteleinlass mit einem 1/32"-Schlauch und einer stumpfen 30-G-Nadel, anschließendes Befüllen des Geräts, bis an beiden offenen Anschlüssen Flüssigkeit austritt. (E) Abdichtung des Schneckeneinlasses und des Abfallauslasses mit Stahlstiften, die durch ein Stück 1/16"- Schlauch () blockiert sind, und Druckbeaufschlagung des Geräts mit der Lebensmittelspritze und der Pumpe (über die Einstellung der Rändelschraube), wobei der Druck einige Minuten lang aufrechterhalten wird. (F) Entfernen der Stopfen von (E), um sicherzustellen, dass sich an jeder offenen Verbindung Flüssigkeitströpfchen bilden. (G) Anschluss des Abfallbehälters über ein kurzes Stück 1/16" Schlauch und einen hohlen Stahlstift. H) Befestigen einer mit Puffer gefüllten Spritze an der Vorrichtung mit einem hohlen Stahlstift und einer stumpfen 23-G-Nadel. Würmer werden in den Schlauch gezogen, bevor er mit dem Gerät verbunden wird, wodurch sichergestellt wird, dass keine Luft eingetragen wird, indem Flüssigkeitströpfchen am Einlass- und Schlauchende ausgerichtet werden. (I) Endgültige Einrichtung, wobei alle Geräteverbindungen hergestellt sind. (J) Bild des Geräts mit Ventilschlauch, der mit dem Einlass und dem Magneten verbunden ist (Hintergrund), entsprechend (A-C). (K) Vergrößerte Ansicht des Geräts mit angebrachtem Lebensmittelzufuhrschlauch, der zwei Flüssigkeitströpfchen (Pfeile) am Schneckeneinlass und Abfallauslass zeigt, entsprechend (D). (L) Vergrößerte Ansicht des Geräts mit angeschlossenen Lebensmittelzufuhrschläuchen und blockiertem Schneckeneinlass und Abfallauslass, entsprechend (E). (M) Vergrößerte Ansicht des Geräts mit angebrachtem Abfallschlauch und Behälter, entsprechend (G). (N) Endmontage mit allen Rohrverbindungen an Ort und Stelle, entsprechend (I). Teilweise adaptiert von Berger et al.18. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Schlauch () blockiert sind, und Druckbeaufschlagung des Geräts mit der Lebensmittelspritze und der Pumpe (über die Einstellung der Rändelschraube), wobei der Druck einige Minuten lang aufrechterhalten wird. (F) Entfernen der Stopfen von (E), um sicherzustellen, dass sich an jeder offenen Verbindung Flüssigkeitströpfchen bilden. (G) Anschluss des Abfallbehälters über ein kurzes Stück 1/16" Schlauch und einen hohlen Stahlstift. H) Befestigen einer mit Puffer gefüllten Spritze an der Vorrichtung mit einem hohlen Stahlstift und einer stumpfen 23-G-Nadel. Würmer werden in den Schlauch gezogen, bevor er mit dem Gerät verbunden wird, wodurch sichergestellt wird, dass keine Luft eingetragen wird, indem Flüssigkeitströpfchen am Einlass- und Schlauchende ausgerichtet werden. (I) Endgültige Einrichtung, wobei alle Geräteverbindungen hergestellt sind. (J) Bild des Geräts mit Ventilschlauch, der mit dem Einlass und dem Magneten verbunden ist (Hintergrund), entsprechend (A-C). (K) Vergrößerte Ansicht des Geräts mit angebrachtem Lebensmittelzufuhrschlauch, der zwei Flüssigkeitströpfchen (Pfeile) am Schneckeneinlass und Abfallauslass zeigt, entsprechend (D). (L) Vergrößerte Ansicht des Geräts mit angeschlossenen Lebensmittelzufuhrschläuchen und blockiertem Schneckeneinlass und Abfallauslass, entsprechend (E). (M) Vergrößerte Ansicht des Geräts mit angebrachtem Abfallschlauch und Behälter, entsprechend (G). (N) Endmontage mit allen Rohrverbindungen an Ort und Stelle, entsprechend (I). Teilweise adaptiert von Berger et al.18. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
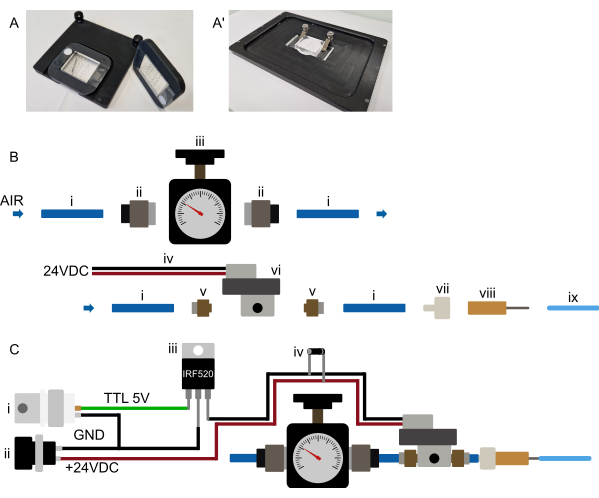
Abbildung 3: Hardware-Setup. (A-A') Empfohlene Gerätemontage. (A) Mögliche Halterung für ein aufrechtes Mikroskop. Die Geräte werden an einem rechteckigen, 3D-gedruckten Rahmen mit einem großen Mittelausschnitt befestigt. Zwei kleine Streifen doppelseitiges Klebeband werden zunächst auf dem Deckglas auf beiden Seiten des PDMS-Teils des Geräts platziert. Der 3D-gedruckte Rahmen wird dann über das Gerät gelegt, so dass der PDMS-Teil durch den Ausschnitt ragt und der Rahmen fest auf das doppelseitige Klebeband gedrückt wird, wodurch das Gerät dauerhaft mit dem Rahmen verbunden wird. Der Rahmen hält das Gerät also sicher und wird selbst mit Hilfe von Magneten, die in den Rahmen und den Tischeinsatz eingelassen sind, auf dem Mikroskoptisch gehalten. Dies gewährleistet eine stabile Gerätehalterung und verhindert eine Kollision des Mikroskopobjektivs mit jeglichem Montagematerial. Alle Schläuche werden zur Vorderseite des Geräts geführt. (A') Mögliche Montage für ein inverses Mikroskop. Ein Satz Klemmen hält das Gerät fest. Stellen Sie sicher, dass die Klemmen das Gerät auf den Tischeinsatz drücken, um eine XY-Bewegung während des Betriebs zu verhindern. (B) Ansicht der Teile des Drucksystems. i) Polyurethanschlauch mit einem Außendurchmesser von 6 mm, der an eine Druckquelle angeschlossen ist. (ii) G1/8-auf-6-mm-Push-in-Adapter, der den Schlauch (i) mit dem Druckregler (iii) verbindet. Der Schlauch wird dann über einen M5-auf-6-mm-Push-in-Adapter (v) mit einem Magneten (vi) verbunden, und schließlich wird der Schlauch mit einem Luer-Lock-to-Barb-Anschluss (vii) und einer stumpfen 23-G-Nadel (viii) mit dem 1/16" OD-Schlauch verbunden, der mit dem mikrofluidischen Gerät verbunden ist. (C) Schematische Übersicht über die elektronischen Anschlüsse und das aufgebaute Drucksystem. (i) Der von einem Arduino, einer DAq-Karte oder einem anderen Mikrocontroller erzeugte TTL-Trigger (+3,3-5 V) wird zusammen mit einer geeigneten Stromversorgung (ii, 24 VDC) mit einem MOSFET (iii) und den elektronischen Anschlüssen des Magneten, wie im Schaltplan gezeigt, verbunden, wobei eine Flyback-Diode (iv) über die Stromanschlüsse der Magnetspulen angeschlossen ist. Sobald ein TTL-Signal vom Mikrocontroller an das Gate des MOSFET gesendet wird, werden +24 VDC an den normalerweise geschlossenen Magneten angelegt und das On-Chip-Hydraulikventil wird aufgeblasen. Sobald das TTL-Signal entfernt wird, wird der Druck abgelassen und das On-Chip-Hydraulikventil entleert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
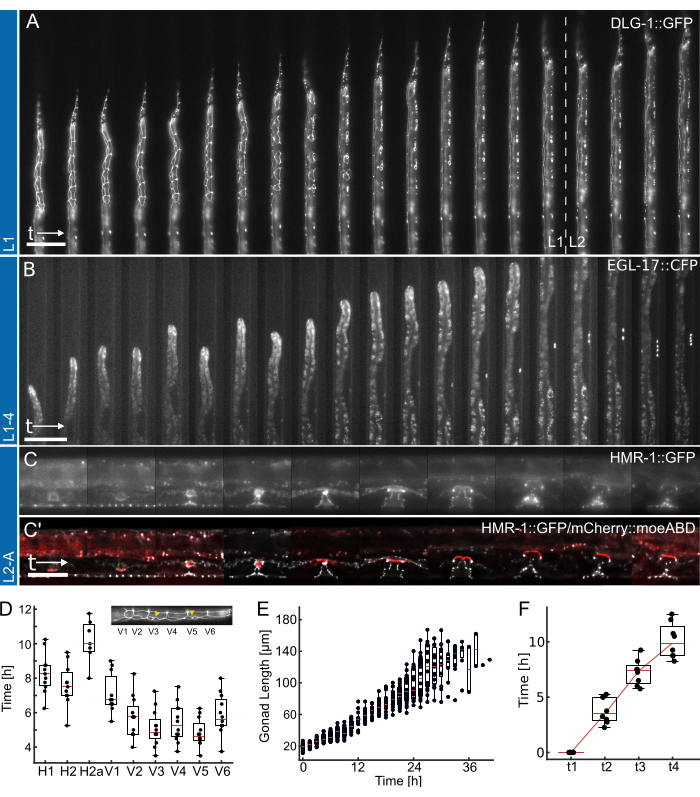
Abbildung 4: Repräsentative Ergebnisse. (A) Tier mit einem epithelialen Junction-Maker (ML2615, dlg-1(mc103[dlg-1::gfp])), das unter Verwendung des L1-Geräts vom nächtlichen Hunger bis zum frühen/mittleren L2-Larvenstadium wächst. Zu sehen sind die sich teilenden Nahtzellen, sowie die P-Zellen, die in Richtung des späten L1-Larvenstadiums wandern und sich teilen. (B) Tiere, die vom späten L1-Larvenstadium bis zum frühen L4-Stadium unter Verwendung des L1-4-Geräts heranwachsen. Sichtbar ist ein Marker für die 1°-schicksalhafte Vulva-Vorläuferzelle (VPC) (AH1187, arIs92[egl-17::cfp]), wobei eine einzelne Zelle zu Beginn des Experiments sichtbar ist, gefolgt von zwei Runden sichtbarer Zellteilungen im L3-Larvenstadium. (C) Tiere, die vom späten L2-Larvenstadium bis zum frühen Erwachsenenalter heranwachsen, unter Verwendung des L2-A-Geräts. Zu sehen ist ein epithelialer Junction-Maker (AH5786, hmr-1(cp21[hmr-1::gfp + LoxP])), der das sich entwickelnde Vulvagewebe hervorhebt, das ausgeprägte toroidale Strukturen bildet, und ein kombiniertes Bild mit einem Marker für die uterine Ankerzelle (AC) (AH5786, qyIs50[Pcdh-3>mCherry::moeABD, unc-119(+)]), die gegen Ende des L3-Larvenstadiums in das Vulvagewebe eindringt und das Gewebe verbindet. Bei allen Versuchen (A-C) wird das FOV zunächst so eingestellt, dass nur ein Teil der Tiere sichtbar ist. Im Laufe des Experiments füllt das Tier einen größeren Teil des Kanals aus, wobei sich die Merkmale als Folge des Tierwachstums entlang der Kanalachse verschieben. (D) Zeitpunkt, zu dem die einzelnen Nahtzellteilungen während des L1-Larvenstadiums mit der Teilung beginnen (n = 12). Insert hebt die Identitäten der Nahtzellen (H1-V6) entlang der anterior-posterioren Körperachse hervor. (E) Zeigt das Wachstum der Gonaden im Laufe der Zeit für Tiere in Abbildung 4B (n = 19). (F) Zeigt die Zeit, die benötigt wird, um 4 verschiedene Entwicklungszeitpunkte in der Vulva-Morphogenese (t1-t4) zu erreichen, was auf einen konsistenten Übergang für alle abgebildeten Tiere hinweist (n = 7). Maßstabsleisten sind (A,B) 50 μm und (C) 25 μm. Siehe Ergänzungsfilm 1, Ergänzungsfilm 2 und Ergänzungsfilm 3 für den Vollzeitkurs. Die Boxplots zeigen die Medianwerte (rote Linien) mit oberen und unteren Quartilen und Balken, die die Extreme der Verteilung anzeigen, ohne Ausreißer. Die Abbildung ist eine Adaption von Berger et al.18. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzender Film 1: Einzelne Larve von C. elegans entwickelt sich vom Hungertod über Nacht bis zum frühen L2-Stadium. Angezeigt werden projizierte Epifluoreszenzbilder mit maximaler Intensität (Gesamthöhe der Projektion beträgt 1,5 μm) in ansonsten unbearbeitetem Format. Sichtbar sind alle Nahtzellteilungs- und Fusionsereignisse, die in der angegebenen Phase (oben) stattfinden. Die Merkmale werden unter Verwendung des DLG-1::GFP-Markers (Epithelübergang) in 15-Minuten-Intervallen für insgesamt 25 Stunden skizziert. Der Film ist eine Wiederverwendung von Berger et al.18. Bitte klicken Sie hier, um diesen Film herunterzuladen.
Ergänzender Film 2: Einzelne Larve, die sich vom späten L1-Stadium bis zum frühen L4-Stadium entwickelt und einen EGL-17::CFP-Marker exprimiert (1°-fated vulval precursor cell (VPC)). In der frühen L2 (~400 min) manifestiert sich die EGL-17::CFP-Fluoreszenz in P6.p und nimmt kontinuierlich an Intensität zu. Während L3 durchläuft P6.p zwei aufeinanderfolgende Teilungsrunden, wobei es die 1-Zell-, 2-Zell- und 4-Zell-Stadien der Vulva-Entwicklung bildet. Die Bilder werden in 30-Minuten-Intervallen für insgesamt 34 Stunden gezeigt. Der Film ist eine Wiederverwendung von Berger et al.18. Bitte klicken Sie hier, um diesen Film herunterzuladen.
Ergänzender Film 3: Draufsicht: Z-Projektion der sich entwickelnden Vulva vom L3-Stadium bis zum Übergang L4/Erwachsener. Ansicht unten: X-Projektion der gleichen Vulva im Zeitverlauf. Beide Ansichten zeigen die anfängliche Teilung der VPCs, gefolgt von Invagination, Bildung und Vergrößerung der Toroide, gefolgt von einem Kollaps während der Eversion. Epifluoreszenzbilder werden nach dem Zuschneiden, Dekonvolution, Registrierung und Projektion angezeigt. Die Merkmale werden durch den HMR-1::GFP-Marker (Hypodermis und VPCs) und den mCherry::moeABD-Marker (der den AC umreißt) umrissen. Der Film ist eine Wiederverwendung von Berger et al.18. Bitte klicken Sie hier, um diesen Film herunterzuladen.
Ergänzende Datei 1: CAD-Datei für das L1-Gerät. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 2: CAD-Datei für das Gerät L1-4. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 3: CAD-Datei für das L2-A-Gerät. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 4: CAD-Datei für das Gerät L3-A. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzungsdatei 5: CAD-Datei für das Gerät L4-A. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzungsdatei 6: CAD-Datei für die Struktur der Lebensmittelverteilung. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 7: CAD-Datei für die Ventilschicht. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
In diesem Artikel wurde der Betrieb einer einzigartigen mikrofluidischen Langzeitbildgebungsplattform beschrieben, die für die hochauflösende Bildgebung und Verfolgung verschiedener Entwicklungsprozesse bei C. elegans während aller vier Larvenstadien bis ins frühe Erwachsenenalter geeignet ist18. Die Möglichkeit, Entwicklungsprozesse im Laufe der Zeit in vivo mit Hilfe dieser und anderer bildgebender Verfahren zu verfolgen, ist in vielen Bereichen der C. elegans-Forschung unverzichtbar geworden und hat es ermöglicht, bisher unzugängliche Fragen zu beantworten 23,24,29.
Im Gegensatz zu herkömmlichen bildgebenden Verfahren mit Agar-Pads und zuvor veröffentlichten mikrofluidikbasierten Langzeitbildgebungsstrategien behält dieser Ansatz die Identität und Orientierung des Wurms während des gesamten Experiments bei und ermöglicht so die Verfolgung komplexer Entwicklungsprozesse über die Zeit. Unabhängig vom Entwicklungsstadium sind die Tiere auf einen von 41 parallelen Fallenkanälen beschränkt, die jeweils mit einer On-Chip-Futterversorgung verbunden und von einem großen Hydraulikventil abgedeckt werden. Die Immobilisierung aller gefangenen Würmer erfolgt durch das Aufblasen des Hydraulikventils, wobei die Würmer nur während der Bildaufnahme auf das Deckglas gedrückt werden. Dank des eng beieinander liegenden Fallenkanal-Arrays ist eine Bildgebung mit hohem Durchsatz möglich, bei der mehrere Tiere in einem einzigen Sichtfeld sichtbar sind. Die Langzeitbildgebungsgeräte sind mit den meisten Mikroskop-Setups kompatibel und erfordern nur minimale zusätzliche Hardware (weniger als 1000 US-Dollar) und keine Änderungen an vorhandener Hardware. Die Bedienung der Geräte ist absichtlich einfach, so dass alle Benutzer schnell in der Lage sein sollten, sie zu erlernen, indem sie das obige Protokoll und die skizzierten Ergebnisse befolgen.
Kurz gesagt, Tiere können über bis zu 2,5 Larvenstadien hinweg auf dem Chip abgebildet werden (Abbildung 1C). Die Tiere werden zunächst synchronisiert und entweder direkt auf den Chip geladen (L1-Gerät) oder bis zum gewünschten Entwicklungsstadium auf die NGM-Platte ausgesät (L1-L4- und L2-A-Geräte). Die Tiere werden mit einer hochkonzentrierten Bakteriensuspension gefüttert und mit einem On-Chip-Hydraulikventil, das von der Bildgebungssoftware betätigt wird, immobilisiert (Abbildung 1B). Die Tiere werden mit einer Spritze in die Fallenkanäle manövriert, wobei die Tiere vorsichtig mit dem Kopf zum Futtervorrat ausgerichtet werden und schließlich die Tiere am Ende des Fallenkanals gefangen werden (Abbildung 1A und Abbildung 2). Es muss darauf geachtet werden, dass keine Luft in den Spritzen, stumpfen Nadeln und Schläuchen eingeschlossen wird, da Luftblasen die Position des Tieres leicht stören. Die Bauelemente sind mit den meisten Bildgebungsmodalitäten18,19,24 kompatibel, da das PDMS-Material hochtransparent, nicht fluoreszierend und gegen ein dünnes Deckglas abgedichtet ist (Abbildung 4, Ergänzender Film 1, Ergänzender Film 2 und Ergänzender Film 3). Bei der Auswahl der Bildgebungsbedingungen ist jedoch Vorsicht geboten, da lange Belichtungszeiten und hohe Anregungsintensitäten die Lebensfähigkeit der Tiere leicht beeinträchtigen und zu Photobleaching führen.
Neben der vorgestellten Version dieses bildgebenden Verfahrens sind mehrere Modifikationen des Protokolls möglich und bereits verfügbar. Die einfachste Modifikation besteht darin, die Standard-Bakterienstämme, z. B. durch RNAi-exprimierende Bakterien18 oder Bakterien mit unterschiedlichen Nährwerten32, zu ersetzen. Bakterienstämme können leicht ausgetauscht werden, indem der Optiprep-Gehalt angepasst wird, um Dichteunterschiede zu berücksichtigen. In ähnlicher Weise kann eine beliebige Anzahl von Verbindungen zu der bakteriellen Nahrung hinzugefügt werden, z. B. Auxin, Nährstoffe oder niedermolekulare Wirkstoffverbindungen, und ihre Wirkung wird direkt in vivo beobachtet, ohne dass Änderungen am Protokoll erforderlich sind. Insbesondere wurde eine Medienaustauschvorrichtung für den Auxin-induzierten Proteinabbau oder jede Anwendung, die eine zeitgesteuerte oder reversible Exposition erfordert, entwickelt, indem der Anordnung der Vorrichtung ein Lebensmitteleinlass hinzugefügt wird, ohne die Geometrie oder den Betrieb zu ändern. Der zusätzliche Einlass ermöglicht ein einfaches Umschalten zwischen zwei verschiedenen Bedingungen, z. B. Auxin und kein Auxin, die auf die gleiche Weise an das Gerät angeschlossen sind, wie hier für eine einzelne Nahrungsquelle gezeigt, und die daraus resultierenden Veränderungen werden direkt beobachtet. Es wurden auch Gerätevarianten entwickelt, bei denen das Hydraulikventil nicht von allen Einheiten eines Geräts gemeinsam genutzt wird, sondern für jede Einheit getrennt ist, so dass mehrere Bedingungen innerhalb desselben Geräts abgebildet und einzeln experimentiert werden können, wodurch die potenziell schädlichen Auswirkungen langer Ventilbetätigungs- und Erfassungszeiten reduziert werden. Schließlich wurden neben den ursprünglichen Gerätevarianten, die in Berger et al.18 beschrieben wurden, zwei neue Gerätevarianten entwickelt, L3-A und L4-A (hier vorgestellt), die eine Bildgebung ab dem mittleren L3- oder mittleren L4-Stadium bis zum Erwachsenenalter ermöglichen. Der Zweck dieser Modifikationen besteht darin, den Startzeitpunkt eines Versuchs auf ein späteres Larvenstadium zu verzögern, so dass größere Tiere in Anwendungen geladen werden können, bei denen die früheren Entwicklungsstadien nicht von Interesse sind.
Offenlegungen
Die Autoren erklären keine konkurrierenden oder finanziellen Interessen.
Danksagungen
Wir danken den Mitgliedern des Laboratoriums Hajnal für die kritische Diskussion und die Kommentare zum Manuskript. Wir danken auch dem C. elegans Genetics Center CGC, das vom NIH Office of Research Infrastructure Programs (P40 OD010440) finanziert wird. Darüber hinaus möchten wir den Mitgliedern des Galli-Labors (Hubrecht-Institut), des Conradt-Labors (University College London) und des Van-den-Heuvel-Labors (Universität Utrecht) für ihren wertvollen Beitrag zu dem vorgestellten Protokoll danken. Diese Arbeit wurde durch Zuwendungen des Schweizerischen Nationalfonds zur Förderung der wissenschaftlichen Forschung Nr. 31003A-166580 an AH, der Schweizerischen Krebsliga Nr. 4377-02-2018 an AH und der ETH Zürich an AdM unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 3/2 solenoid valve | Distrelec | 154-22-898 and 154-22-899 | Alternative to above. Solenoid valve connecting the pressure regulator to the on-chip valve. |
| Arduino | Distrelec | 301-01-956 | Optional microcontroller depending on the selected controller and interface. |
| Bacto-Tryptone | Thermo Fisher Scientific | 211705 | For L Broth production. |
| Bacto-Yeast | Thermo Fisher Scientific | 212750 | For L Broth production. |
| Blunt needle 23G | Gonano Dosiertechnik | GGA723050 | Blunt needle connecting the 1/16" tubing to a syringe. |
| Blunt needle 30G | Gonano Dosiertechnik | IG-TE730050 | Blunt needle connecting the 1/32" tubing to a syringe. |
| C. elegans Construct 1 | NA | NA | AH5786 LGI: hmr-1(cp21[hmr-1::gfp + LoxP]) Marston, D.J. et al. MRCK-1 drives apical constriction in C. elegans by linking developmental patterning to force generation. Curr. Biol. 26 (16), 2079-2089. 2016. |
| C. elegans Construct 2 | NA | NA | AH1187 LGII: arIs92[egl-17::cfp] Yoo, A.S., Bais, C., Greenwald, I. Crosstalk between the EGFR and LIN-12/Notch pathways in C. elegans vulval development. Science. 303 (5658), 663-666. 2004. |
| C. elegans Construct 3 | NA | NA | AH5786 LGV: qyIs50[Pcdh-3>mCherry::moeABD, unc-119(+)] Ziel, J.W., Hagedorn, E.J., Audhya, A., Sherwood, D.R. UNC-6 (netrin) orients the invasive membrane of the anchor cell in C. elegans. Nat. Cell Biol., 11 (2), 183-189. 2009. |
| C. elegans Construct 4 | NA | NA | ML2615 LGX: dlg-1(mc103[dlg-1::gfp]) Vuong-Brender, T.T.K., Suman, S.K., Labouesse, M. The apical ECM preserves embryonic integrity and distributes mechanical stress during morphogenesis. Development. 144 (23), 4336-4349. 2017. |
| Cable | Distrelec | 143-46-644 | Needed if selecting the solenoid from distrelec. Cable connecting the solenoid to the MOSFET. |
| Chlorotrimethyl silane | Sigma Aldrich | 386529-25ML | Silane for wafer passivation. |
| Cholesterol | Sigma Aldrich | C8667 | For S-Basal production. |
| Flyback Diode | Distrelec | 110-52-628 | Flyback diode protecting the microcontroller. |
| Hollow steel pin | Gonano Dosiertechnik | GGA7R23050 | 23G hollow steel pin connecting the 1/16" tubing to the device. |
| L Broth | NA | NA | Prepared in house following standard recipe. 10 g Bacto-tryptone, 5 g Bacto-yeast, 5 g NaCl, H2O to 1 litre, pH to 7.0 using 1 M NaOH |
| Long-term Imaging Device L1 | NA | NA | Long-term imaging device for L1 to early/mid L2 larvae. Made inhouse, available on request. Also available as media exchange variant. |
| Long-term Imaging Device L1-4 | NA | NA | Long-term imaging device for mid L1 to mid L4 larvae. Made inhouse, available on request. Also available as media exchange variant. |
| Long-term Imaging Device L2-A | NA | NA | Long-term imaging device for mid L2 to adulthood (eversion). Made inhouse, available on request. Also available as media exchange variant. |
| Long-term Imaging Device L3-A | NA | NA | Long-term imaging device for mid L3 to adulthood (eversion). Made inhouse, available on request. Also available as media exchange variant. |
| Long-term Imaging Device L4-A | NA | NA | Long-term imaging device for mid L4 to adulthood (eversion). Made inhouse, available on request. Also available as media exchange variant. |
| M9 Buffer | NA | NA | Prepared in house following standard recipe. |
| MOSFET | Distrelec | 303-41-391 | Metal-oxide-semiconductor field-effect transistor, used to switch the 24VDC required for the solenoid. |
| NGM plates | NA | NA | Prepared in house following standard recipe. |
| Nylon Luer-Fitting | Fisher Scientific | 11776048 | Adapter from OD 6mm tubing to 23G blunt needle. |
| Optiprep | Sigma Aldrich | D1556 | Density matching liquid. |
| PDMS | Ameba | Elastosil RT601A/B | PDMS elastomer kit. |
| Pluronic F-127 | Sigma Aldrich | P2443 | Non-ionic surfactant. |
| Potassium dihydrogen phosphate | Sigma Aldrich | 1.37039 | For S-Basal production. |
| Potassium hydrogen phosphate | Sigma Aldrich | 1.05099 | For S-Basal production. |
| Power Supply 24VDC | Distrelec | 301-63-304 | 24VDC power supply. Select a power supply suitable for the solenoid selected. |
| Precision pressure regulator | Distrelec | 154-23-578 | Alternativ to above. Pressure regulator. |
| Pressure Gauge G1/8 | Distrelec | 154-23-643 | Pressure gauge attaches to the pressure regulator. |
| PU Tubing OD 6mm | Distrelec | 154-23-866 | Tubing connecting the pressure source and regulator, as well as regulator and solenoid valve. |
| Push-in fitting G1/8 to 6mm | Distrelec | 301-69-330 | Alternativ to above. 2x Push-in fitting attaches to the pressure regulator and connects to the 6mm tubing. |
| Push-in fitting M5 to 6 mm | Distrelec | 301-69-459 | 2x Push-in fitting attaches to the solenoid and the 6mm tubing. |
| S-Basal Buffer | NA | NA | Prepared in house following standard recipe. 5.85 g NaCl, 1 g K2HPO4, 6 g KH2PO4, 1 ml cholesterol (5 mg/ml in ethanol), H2O to 1 litre. Sterilize by autoclaving. |
| Sodium Chloride | Sigma Aldrich | S9625 | For L Broth and S-Basal production. |
| Sodium hydroxite (5M) | Sigma Aldrich | 567530 | Sodium hydroxide used when bleaching C. elegans (prepare solution at 5M concentration) |
| Sodium hypochlorite (5%) | Sigma Aldrich | 1.05614 | Sodium hypochlorite used when bleaching C. elegans (prepare solution at 5% concentration) |
| Strainer 10 µm | pluriSelect | 43-10010-40 | Alternative for bacteria food preparation, especially if smaller devices are used. |
| Strainer 5 µm | pluriSelect | 43-10005-40 | Strainer used to filter L1 larvae after overnight starvation and bacteria after completed food preparation. |
| SU-8 | Gersteltec | GM1050 | SU-8 for height from 1-10 µm. |
| SU-8 | Gersteltec | GM1060 | SU-8 for height from 5-30 µm. |
| SU-9 | Gersteltec | GM1070 | SU-8 for height from 15-200 µm. |
| Syringe 1 mL | Fisher Scientific | 11338763 | 1mL syringe. If using a different type ensure that the syringe body and plunger are stiff, such that they won't be deformed by the syringe pump. |
| Syringe Pump | WPI | Al1000-220 | Syringe pump (https://www.wpiinc.com/var-2300-aladdin-single-syringe-pump.html). |
| Tube 15 mL | Fisher Scientific | 50-809-220 | Tube used for worm bleaching and bacteria preparation. |
| Tube 2 mL | Fisher Scientific | NC1186931 | Tube used for worm preparation and bacteria preparation. |
| Tube 50 mL | Fisher Scientific | 50-465-232 | Tube used for bacteria preparation and growth. |
| Tweezer SMD | Brütsch Rüegg Werkzeuge | 448980 | Tweezers used to plug the 1/32" tubing into the device. |
| Tygon tubing 1/16" | Fisher Scientific | 642002 | Flexible 1/16" OD tubing connecting most components to the device. |
| Tygon tubing 1/32" | Fisher Scientific | 641900 | Flexible 1/32" OD tubing connecting the bacteria supply to the device. |
| UV KUB-3 | Kloe | NA | Mask aligner and UV exposure system. |
| Wafer | Siegert Wafer | 4P0/>1/525±25/SSP/TTV<10 | Silicon Wafer. |
| Waste Container | NA | NA | Prepared in house using a 2mL tube or a cryo vial and hollow steel pins. |
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten