Method Article
Imaging ad alta risoluzione di C. elegans in tutti gli stadi larvali
In questo articolo
Riepilogo
Questo protocollo descrive l'imaging time-lapse basato su microfluidica di C. elegans durante l'intero sviluppo post-embrionale.
Abstract
Caenorhabditis elegans è diventato uno dei modelli animali più studiati e meglio compresi in biologia. Tre caratteristiche sono fondamentali per il successo di C. elegans come organismo modello: la sua linea cellulare invariante, la trasparenza e la trattabilità genetica. Questi lo rendono ideale per una vasta gamma di studi basati sulla microscopia direttamente in vivo. Le larve e gli adulti vivi di C. elegans spesso devono essere immobilizzati durante l'acquisizione dell'immagine. I metodi di immobilizzazione tradizionali influiscono negativamente sullo sviluppo degli animali, soprattutto nelle applicazioni di imaging time-lapse. Qui, viene introdotto un protocollo dettagliato di configurazione e funzionamento per un nuovo metodo di imaging microfluidico, che affronta i limiti associati all'immobilizzazione tradizionale basata su agar pad e ad altre strategie microfluidiche. Questo approccio consente l'imaging simultaneo dal vivo in vari stadi larvali, preservando l'orientamento e l'identità del verme nel tempo. Per raggiungere questo obiettivo, viene impiegato un array di canali trappola microfluidica, la cui geometria è stata progettata con precisione per mantenere un orientamento stabile del verme, assecondando al contempo la crescita e la muta. L'immobilizzazione è facilitata da una valvola idraulica attiva che applica una pressione per fissare le viti senza fine contro il vetro di copertura solo durante l'acquisizione dell'immagine. Questo design consente l'imaging ad alta risoluzione con effetti minimi sulla vitalità del verme o sui tempi di sviluppo.
Introduzione
L'imaging di C. elegans può essere eseguito in diversi modi a seconda dell'applicazione, della produttività desiderata e della risoluzione. Quando è necessaria un'alta risoluzione, il metodo standard utilizza i tamponi di agar 1,2,3, una semplice costruzione di vetrino in vetro, agarosio e vetro di copertura. Gli animali su questi scivoli sono immobilizzati dalla pressione esercitata da un vetro di copertura. L'immobilizzazione può essere ulteriormente migliorata con l'aggiunta di agenti anestetizzanti, ad esempio levamisolo o sodio azide 1,2, in modo tale che gli animali sui cuscinetti di agar siano perfettamente fermi e possano essere riprodotti ad alta risoluzione, compresi vari metodi di super-risoluzione2. Sfortunatamente, è noto che i cuscinetti di agar influenzano la progressione dello sviluppo, con la combinazione di alte pressioni applicate e agenti anestetizzanti che ritardano lo sviluppo o causano l'arresto completo dello sviluppo 4,5.
Oltre a questi metodi standard, negli ultimi anni sono emerse diverse metodologie microfluidiche, ognuna con i suoi vantaggi e svantaggi o casi d'uso specializzati 6,7,8,9,10,11,12,13,14,15,16,17,18,19. Ad esempio, Gritti et al.20 hanno introdotto un metodo in cui i vermi sono confinati all'interno di grandi camere. Gli embrioni posti in queste camere si sviluppano e si muovono liberamente rimanendo all'interno della regione designata di interesse su un microscopio. Sebbene generalmente efficace, questo approccio è limitato all'uso di marcatori fluorescenti luminosi. Più recentemente, Keil et al.21 hanno proposto una versione modificata di questo metodo, incorporando una valvola idraulica su chip per trattenere gli animali durante l'acquisizione dell'immagine, affrontando così alcune limitazioni dell'approccio originale. Tuttavia, poiché entrambi i metodi utilizzano camere più grandi di quelle utilizzate per i vermi intrappolati, gli animali continuano a muoversi e ruotare durante l'esperimento. Di conseguenza, il monitoraggio dei processi di sviluppo diventa impegnativo e richiede un'ampia post-elaborazione delle immagini per correggere il movimento degli animali.
Per affrontare queste limitazioni, viene adattato l'approccio introdotto da Berger et al.18 (Figura 1), in cui gli animali non sono tenuti in una grande camera ma alloggiati in un canale trappola progettato per adattarsi agli animali all'interno di una finestra di sviluppo più breve (fino a 2,5 stadi larvali). A differenza dei metodi microfluidici esistenti, questo approccio preserva l'orientamento e l'identità degli animali durante l'esperimento e consente l'acquisizione di immagini ad alta risoluzione di più vermi in parallelo e attraverso più stadi larvali all'interno di un singolo dispositivo.
Gli animali vengono intrappolati utilizzando una geometria del canale specializzata in cui l'altezza del canale è vicina allo spessore di un animale all'inizio di un esperimento, impedendo così la rotazione. Le larghezze e le lunghezze dei canali sono selezionate in modo che gli animali possano muoversi e crescere nel tempo, facilitando la muta e il passaggio da uno stadio larvale all'altro. La parallelizzazione si ottiene quindi semplicemente posizionando più canali di trappola uno accanto all'altro, in modo tale che più animali siano visibili in un unico campo visivo (FOV, Figura 1A') e che sia possibile visualizzare fino a 41 animali in un'unica unità di dispositivo (Figura 1A). Agli animali sul chip viene continuamente fornita una sospensione di cibo batterico altamente concentrata attraverso un canale a bassa altezza, che allo stesso tempo aiuta a confinare gli animali all'estremità dell'array di canali della trappola (gradino di altezza, Figura 1A, rosso). Simile all'approccio adottato da Keil et al.21, viene implementata una grande valvola idraulica on-chip, che può essere gonfiata durante l'acquisizione dell'immagine, premendo gli animali contro il vetro di copertura, migliorando così l'immobilizzazione degli animali (Figura 1B). Limitando l'azionamento della valvola idraulica al solo periodo di acquisizione dell'immagine, si riducono al minimo gli effetti negativi sullo sviluppo degli animali, garantendo uno sviluppo affidabile in tutti gli stadi larvali. A differenza dei metodi descritti da Gritti et al.20 e Keil et al.21, gli animali non possono essere visualizzati durante l'intero sviluppo post-embrionale all'interno dello stesso dispositivo. Invece, vengono introdotti cinque dispositivi distinti, denominati L1, L1-4, L2-A, L3-A e L4-A, che coprono rispettivamente l'intero stadio L1 e lo stadio L2 precoce, lo stadio L1 medio-tardivo fino allo stadio L4 medio-tardivo, L2 medio-tardivo fino alla giovane età adulta, L3 medio-tardivo fino alla giovane età adulta e il tardo L4 fino alla giovane età adulta (Figura 1C), che consentono l'imaging ininterrotto di gran parte dello sviluppo post-embrionale. Questo approccio è stato adottato a causa del grande aumento delle dimensioni degli animali da L1 all'età adulta, in modo tale che un canale adatto agli animali L1 sarebbe troppo piccolo per gli adulti, con conseguente arresto dello sviluppo, e un canale adatto agli adulti sarebbe troppo grande per gli L1, con conseguente movimento eccessivo, rotazione e animali che fuggono dai canali trappola.
Utilizzando questo approccio, si ottiene una progressione affidabile dello sviluppo per una varietà di tessuti18, ad esempio, lo sviluppo post-embrionale dell'ipoderma (stadio da L1 a L4), l'induzione dei destini delle cellule vulvare (stadio L2 e L3), la morfogenesi vulvare (da L3 all'età adulta19) e l'invasione delle cellule ancoranti. Sin dalla loro introduzione, questi dispositivi di imaging a lungo termine sono stati utilizzati per studiare una varietà di processi22,23, con la piattaforma che è stata persino combinata con tecniche come la microscopia a super-risoluzione AiryScan24, consentendo numerose scoperte precedentemente impossibili.
Di seguito, gli aspetti operativi di questi dispositivi di imaging a lungo termine vengono introdotti in un protocollo passo-passo, evidenziando i risultati attesi e alcune potenziali sfide durante la configurazione del sistema.
Protocollo
I dettagli dei reagenti e delle attrezzature utilizzate in questo studio sono elencati nella tabella dei materiali.
1. Fabbricazione del dispositivo
NOTA: Tutti i dispositivi sono stati fabbricati utilizzando il protocollo seguente, tuttavia, i dispositivi sono direttamente disponibili presso gli autori. Per ogni tipo di dispositivo viene fabbricato un wafer separato, con ogni wafer del dispositivo fabbricato da due strati di SU8 di diverse altezze (ad esempio, strato di cibo e strato di trappola). Il wafer della valvola è fabbricato su un wafer separato, con un'altezza singola di SU-8, omettendo i passaggi 1.3-1.6. I silani sono tossici e rilasciano vapori corrosivi. Questo passaggio, quindi, deve essere eseguito in una cappa aspirante. Per i dettagli sulla procedura di fabbricazione, fare riferimento a Berger et al.18.
- Pulire i wafer utilizzando il plasma ad aria.
- Centrifugare il primo strato di SU8, quindi eseguire una cottura morbida a 95 °C.
- Esporre il primo strato, seguito da una post-cottura a 95 °C e sviluppo.
- Eseguire nuovamente la pulizia al plasma dopo lo sviluppo e l'asciugatura del wafer.
- Centrifugare il secondo strato di SU8, quindi cuocere delicatamente a 95 °C (se necessario, effettuare una prima cottura a 65 °C).
- Allinea la seconda maschera alle caratteristiche esistenti utilizzando i marcatori di allineamento su entrambi i lati del wafer, quindi esponi il secondo strato.
- Post-cottura e sviluppo della cialda, quindi eseguire una cottura dura a 200 °C.
- Trattare il wafer con clorotrimetilsilano per almeno 2 ore.
- Preparare un lotto di 20 g di prepolimero PDMS (Parte A) e mescolarlo accuratamente con 1 g di reticolante (rapporto 20:1, Parte B) (vedere la Tabella dei Materiali).
- Degasare per 10 minuti o fino a quando tutte le bolle scompaiono.
- Rimuovere 2 mL della miscela PDMS e riservare per il passaggio 1.13.
- Aggiungere altri 3 g di reticolante alla miscela PDMS rimanente e mescolare accuratamente (rapporto finale circa 5:1).
- Utilizzando il lotto di PDMS 20:1, centrifugare il wafer del dispositivo a 750 giri/min per 30 s.
- Posizionare il wafer della valvola in un piatto di alluminio e fondere il PDMS 5:1 su di esso. Degassare il wafer del dispositivo per circa 10 minuti. Dopo il degasaggio, rimuovere le bolle rimanenti con una pipetta.
- Cuocere sia gli strati della valvola che quelli del dispositivo a 70 °C rispettivamente per circa 15/20 minuti.
- Valutare l'indurimento del dispositivo e degli strati della valvola sondandoli delicatamente con una punta di pipetta o una pinzetta. Lo strato della valvola deve essere solido, mentre lo strato del dispositivo deve rimanere leggermente appiccicoso.
- Staccare lo strato della valvola e tagliarlo alla dimensione richiesta usando un bisturi o una lama di rasoio.
- Praticare un foro di accesso nel canale della valvola (manometro 20).
- Posizionare il wafer del dispositivo sotto uno stereomicroscopio dotato di illuminazione superiore.
- Posizionare con cautela il pezzo PDMS contenente lo strato della valvola sul wafer del dispositivo.
- Allineare con precisione le caratteristiche della valvola e del dispositivo, garantendo la rimozione di tutta l'aria intrappolata tra gli strati.
- Trasferire l'apparecchio assemblato in forno e cuocere a 70 °C per una notte.
- Staccare il dispositivo dal wafer.
- Praticare tutti i fori di accesso rimanenti (calibro 20).
- Incollare il PDMS a un vetro di copertura utilizzando plasma ad aria, quindi posizionare il dispositivo incollato in un forno a 70 °C per alcune ore.
2. Preparazione dei vermi
NOTA: Questa sezione descrive la preparazione dei vermi mediante sbiancamento, che consente la generazione di un gran numero (centinaia) di animali. Altri metodi di sincronizzazione, come il prelievo manuale, sono possibili, ma in genere richiedono più tempo e manodopera. Tutti i vermi utilizzati nel protocollo sono mantenuti secondo protocolli standard a 20 °C e su piastre NGM seminate con E. coli OP50.
- A partire dalle placche con molti animali gravidi.
- Lavare gli animali dal piatto utilizzando il tampone M9.
- Aggiungere il 5% di NaClO e 5 M di NaOH alla sospensione a vite senza fine (200 μL/100 μL per ogni 1 mL di sospensione a vite senza fine).
- Agitare delicatamente la miscela decolorante fino a quando gli animali iniziano a sfaldarsi (~10 min).
- Centrifugare la miscela decolorante a temperatura ambiente, 1300 x g per 1 min.
- Rimuovere il surnatante utilizzando una pipetta e aggiungere una quantità uguale di tampone M9 fresco.
- Anche in questo caso, centrifugare a temperatura ambiente, 1300 x g per 1 min.
- Rimuovere il surnatante e trasferire i vermi pellettati in una provetta da 15 ml con 5 ml di tampone M9 fresco.
- Agitare per una notte a 20 °C.
- Far passare la sospensione a vite senza fine attraverso un filtro cellulare da 10 μm.
- Centrifugare la sospensione a vite senza fine a 1300 × g per 1 minuto a temperatura ambiente.
- Scartare il surnatante e risospendere i vermi in un volume uguale di tampone M9.
- Centrifugare nuovamente la miscela di vermi a 1300 × g per 1 minuto a temperatura ambiente.
- Rimuovere il surnatante e trasferire i vermi su piastre NGM. Se sono necessarie larve L1, utilizzarle immediatamente.
- Una volta che i vermi raggiungono lo stadio di sviluppo desiderato, lavarli via dalla piastra utilizzando un tampone S-Basal fresco filtrato attraverso un filtro da 0,2 μm.
- Lasciare sedimentare i vermi per gravità o pellettarli mediante centrifugazione a 750 × g per 1 minuto a temperatura ambiente.
- Scartare il surnatante e aggiungere un volume uguale di tampone S-Basal fresco.
- Ripetere le fasi di sedimentazione o centrifugazione (fasi 2.16 e 2.17)
- Rimuovere il surnatante e aggiungere un volume uguale di tampone S-Basal fresco.
- Eseguire un'ultima volta le fasi di sedimentazione o centrifugazione.
- Rimuovere la maggior parte del surnatante.
- Tenete i vermi nel tubo fino all'inizio dell'esperimento.
3. Preparazione dei batteri
NOTA: Diversi ceppi batterici (ad es. OP50, NA22, HT114, ecc.) possono essere utilizzati sul chip. Regolare la proporzione del mezzo del gradiente di densità aggiunto alla miscela alimentare per compensare le differenze di densità. Tutti i tamponi vengono fatti passare attraverso un filtro da 0,2 μm per rimuovere il particolato.
- Preparare 40 mL di LBroth, in 2 x 20 mL in provette da centrifuga, in matracci sterili di Erlenmeyer, ecc.
- Inoculare LB con batteri raccolti da una piastra di riserva.
- Far crescere i batteri agitando per una notte a 37 °C (~16 h) (OD600 = 1,9).
- Centrifugare i batteri a 3000 x g per 10 min.
- Rimuovere il surnatante e aggiungere 2 mL di tampone S-Basal fresco a ciascuna provetta. Combina tutti i tubi.
- Risospendere meccanicamente i batteri utilizzando una pipetta.
- Centrifugare i batteri a 3000 x g per 5 min.
- Rimuovere il surnatante e aggiungere nuovamente 2 mL di tampone S-Basal fresco.
- Risospendere meccanicamente i batteri utilizzando una pipetta.
- Centrifugare i batteri a 3000 x g per 5 min.
- Rimuovere la maggior parte possibile del surnatante e aggiungere 1 mL di tampone S-Basal fresco prima di risospendere meccanicamente tutti i batteri.
- Combinare 0,60 mL di terreno a gradiente di densità, 0,38 mL di S-Basal + 1% in peso di Pluronic F-127 (un copolimero triblocco non ionico, qui rappresentato come PF) con 1 mL di sospensione batterica OP50, o 0,65 mL di terreno a gradiente di densità, 0,33 mL di S-Basal + 1% in peso PF con 1 mL di sospensione batterica NA22 o HT114.
- Mescolare accuratamente la preparazione del cibo utilizzando un mixer a vortice.
- Filtrare il cibo attraverso un colino cellulare da 5 μm o 10 μm. Un filtro più piccolo è particolarmente consigliato quando si utilizzano dispositivi più piccoli, ad esempio dispositivi L1.
NOTA: Il cibo può essere conservato a temperatura ambiente per un massimo di 2 giorni.
4. Preparazione dei trucioli
NOTA: Fare riferimento alla Figura 2 per una panoramica schematica del dispositivo, dei tubi e delle connessioni. Tutti i tamponi vengono fatti passare attraverso un filtro da 0,2 μm per rimuovere la polvere. Il perno in acciaio menzionato nei passaggi seguenti si riferisce a un tubo cavo corto che viene utilizzato per interfacciare il tubo con diametro esterno più grande (1/16") ai fori di piccolo diametro fabbricati nel dispositivo PDMS. Questi perni in acciaio vengono semplicemente inseriti nel diametro interno del tubo e quindi spinti nel materiale PDMS, collegando efficacemente i due.
- Inizia riempiendo una siringa con acqua deionizzata (DI).
- Attacca un ago da 23 G e un lungo pezzo di tubo da 1/16", con un perno d'acciaio cavo (piegato a un angolo di 90°) all'estremità. Il tubo deve essere sufficientemente lungo da estendersi dal solenoide al tavolino del microscopio.
- Riempire il tubo con acqua deionizzata dalla siringa e collegarlo all'ingresso della valvola inserendo il perno d'acciaio nel foro perforato (Figura 2A, J).
- Rimuovere la siringa e l'ago, quindi collegare il tubo al solenoide off-chip (Figura 2A, J).
- Utilizzando il software di imaging, accendere il solenoide e pressurizzare il dispositivo per diversi minuti per espellere tutta l'aria dalla valvola. Verificare il completamento controllando visivamente l'interfaccia aria-acqua sul chip, che dovrebbe apparire scura e scomparire nel materiale PDMS (Figura 2B).
- Spegnere il solenoide (Figura 2C).
- Riempire una siringa da 1 ml con la soluzione batterica filtrata (~0,5 ml di cibo è sufficiente).
- Collegare un ago da 30 G e un lungo pezzo di tubo da 1/32" all'ago.
- Premere lo stantuffo per riempire sia l'ago che il tubo collegato, assicurandosi che non rimanga aria nella siringa o nel tubo.
- Inserire il tubo da 1/32" direttamente nell'ingresso del cibo del dispositivo microfluidico, utilizzando una pinzetta per evitare danni al tubo (si consigliano pinzette SMD) (Figura 2D, K).
- Posizionare la siringa sulla pompa a siringa (Figura 2D).
- Premere lo stantuffo della siringa utilizzando la vite a testa zigrinata sul retro per riempire il dispositivo di liquido. Una goccia di liquido dovrebbe apparire ad ogni connessione aperta (Figura 2D, freccia).
- Bloccare sia l'ingresso che l'uscita della vite senza fine con un perno d'acciaio sigillato (un perno d'acciaio con un breve pezzo di tubo da 1/16" attaccato e sigillato mediante combustione) (Figura 2E, L).
- Applicare una pressione aggiuntiva utilizzando la vite a testa zigrinata per rimuovere l'aria residua nel dispositivo (Figura 2E, freccia).
- Rimuovere il perno bloccato all'uscita e fissare il contenitore dei rifiuti (costituito da una provetta crioviale o da microcentrifuga, con due perni d'acciaio infilati nel coperchio e fissati con colla). Il contenitore dei rifiuti è collegato utilizzando un breve pezzo di tubo da 1/16", con un'estremità attaccata al contenitore e l'altra estremità collegata a un perno cavo in acciaio (piegato a 90°) (Figura 2F, G, M).
- Spingere la siringa per assicurarsi che il contenitore dei rifiuti sia collegato correttamente e che non vi siano ostruzioni nel sistema. A tal fine, spingere una piccola quantità di liquido attraverso il sistema (ruotare la vite a testa zigrinata sulla pompa a siringa) fino a quando il liquido scorre attraverso il tubo di scarico (Figura 2G, freccia).
- Rimuovere il secondo perno d'acciaio bloccato.
- Spingere la siringa fino a quando non appare una piccola goccia di liquido all'ingresso della vite senza fine (Figura 2H,N).
- Collegare un pezzo più lungo di tubo da 1/16" (15-20 cm) a una siringa da 1 ml riempita con tampone S-Basal, utilizzando un ago da 23 G (Figura 2H,N).
- Fissare un perno d'acciaio dritto da 23 G all'altra estremità del tubo.
- Riempire l'ago e il tubo con il tampone della siringa, assicurandosi che non rimanga aria nella siringa o nel tubo.
- Inserire il perno d'acciaio all'estremità del tubo nel tubo contenente le viti senza fine.
- Spingere una piccola quantità di liquido attraverso il tubo, assicurandosi che non rimanga aria.
- Tirare i vermi nel tubo, ma non tirarli nella siringa.
- Spingere la siringa collegata alle viti senza fine fino a quando una piccola goccia di liquido appare sul perno d'acciaio (Figura 2H).
- Inserire il perno in acciaio nell'ingresso della vite senza fine (Figura 2I).
NOTA: La preparazione del dispositivo può essere eseguita prima della preparazione finale della verme e lasciata sul microscopio fino a quando le vermi non sono pronte.
5. Caricamento e imaging di worm
- Posizionare il dispositivo su un microscopio a basso ingrandimento (5x o 10x) o su un microscopio da dissezione.
- Identificare l'unità del dispositivo attualmente in uso e trovare l'ingresso della vite senza fine utilizzando l'illuminazione a campo chiaro.
- Posizionare il dispositivo in modo che l'ingresso sia visibile su un lato del campo visivo e la parte posteriore dell'ingresso del canale di sifone sia visibile sull'altro lato.
- Spingere delicatamente lo stantuffo della siringa a vite senza fine. Il liquido scorrerà notevolmente dall'ingresso attraverso l'array di canali verso l'uscita. La presenza di batteri e la differenza di indice di rifrazione dovuta all'aggiunta del mezzo gradiente di densità rendono evidente il flusso del liquido.
- Assicurarsi che le viti senza fine appaiano dall'ingresso e fluiscano verso la parte posteriore dell'array di canali.
- Spingi delicatamente gli animali verso l'array di canali. Una volta che un animale è rivolto verso il canale, spingilo nel canale e ripeti per altri animali.
NOTA: Gli animali devono essere orientati con la testa verso l'uscita per avere un facile accesso al cibo. Possono essere orientati spingendo e tirando lo stantuffo e nuotando. - Una volta che un numero sufficiente di animali è stato intrappolato, posizionare la siringa, ancora attaccata all'ingresso del verme, sul tavolino del microscopio, dove rimarrà per tutta la durata dell'esperimento. Potrebbe essere necessario staccare brevemente il tubo della vite senza fine dalla siringa per rilasciare eventuali accumuli di pressione, quindi ricollegarlo con cura all'ago smussato.
- Se il caricamento è stato eseguito su un microscopio da dissezione, trasferire il dispositivo al microscopio per immagini. Il tubo può rimanere attaccato al chip e la siringa è collegata alla pompa.
- Accendere la pompa a siringa e farla funzionare alla velocità preimpostata di 1 μL/h per 0,5 μL, aumentare la velocità di 100 μL/h per 0,5 μL e ridurla a 1 μL/h. Questo ciclo viene ripetuto automaticamente per l'intero esperimento. Per informazioni su come programmare la pompa, consultare il manuale utente dell'apparecchiatura.
- Posizionare il dispositivo sul tavolino del microscopio e assicurarsi che sia tenuto saldamente (suggerimenti per i meccanismi di montaggio sono disponibili nella Figura 3A).
- Se il caricamento non è stato eseguito sul microscopio per immagini, identificare l'unità di interesse del dispositivo a basso ingrandimento.
- Passa all'ingrandimento dell'immagine desiderato.
NOTA: I dispositivi sono compatibili con tutti i tipi di obiettivi. - Identifica gli animali e le regioni di interesse all'interno dell'array di canali trappola e imposta le condizioni di imaging desiderate.
- Immagine alle condizioni di imaging desiderate, con la valvola on-chip, azionata attraverso il solenoide 10 s prima dell'acquisizione dell'immagine, in modo che gli animali siano tenuti in posizione.
NOTA: L'azionamento della valvola sul chip allungherà e appiattirà gli animali, richiedendo eventualmente regolazioni della regione di imaging di interesse (ROI).
6. Preparazione dell'installazione
NOTA: Questa sezione si riferisce alla preparazione iniziale del meccanismo di controllo del dispositivo e non deve essere ripetuta prima di ogni esperimento. Fare riferimento alla Figura 3B, C. Poiché molti solenoidi richiedono una tensione più elevata, molto probabilmente il solenoide non sarà collegato direttamente al microcontrollore, ma tramite un relè o un MOSFET (vedere la Figura 3C).
- Identificare una fonte d'aria pressurizzata (ad es. installazione di alimentazione a parete o bombola del gas).
- Collegare la fonte di pressione a un regolatore di pressione adatto (campo di pressione 0-2 bar) utilizzando un tubo da 6 mm (sorgente a parete) o collegare direttamente un regolatore adatto alla bombola del gas.
- Collegare il regolatore di pressione all'ingresso dell'elettrovalvola (tubo da 6 mm, vedere Figura 3B).
- Posizionare il solenoide il più vicino possibile al tavolino del microscopio (si consiglia una distanza di ~0,5-1 m dal tavolino, anche se sono possibili distanze maggiori se necessario).
- Collegare elettronicamente l'elettrovalvola al controller selezionato. Sono disponibili diverse opzioni, come il controllo tramite una scheda microcontrollore separata (ad esempio, Arduino) o una scheda DAq esistente.
7. Pulizia e stoccaggio
- Una volta completato l'esperimento, rimuovere il dispositivo dal microscopio.
- Rimuovere il liquido di immersione. L'olio da immersione può essere rimosso con solventi organici (ad es. acetone, metanolo, ecc.).
- Posizionare il dispositivo su una superficie piana e scollegare tutti i tubi tranne il tubo della valvola. Per scollegare in sicurezza ciascun tubo, premere il PDMS vicino ad esso e tirare delicatamente. Non sollevare e piegare il dispositivo, poiché ciò potrebbe causare la rottura del vetro di copertura.
- Pulire la superficie del PDMS con etanolo al 70%.
- Conservare il dispositivo a temperatura ambiente con il tubo della valvola collegato.
NOTA: Il dispositivo può essere riutilizzato fino a quando tutte le unità non sono state utilizzate, probabilmente senza dover riempire il tubo della valvola. Tuttavia, l'acqua nel tubo della valvola evaporerà per un periodo prolungato e potrebbe essere necessario riempirla nuovamente.
Risultati
Design e dimensioni del dispositivo
Cinque dispositivi distinti sono stati progettati per ospitare tutti e quattro gli stadi larvali. Questi dispositivi sono designati come segue: L1, per le larve L1 caricate immediatamente dopo la schiusa e sottoposte a imaging fino allo stadio L2; L1-L4, in grado di trattenere le larve dallo stadio L1 medio-tardivo fino a L4 precoce-medio; L2-A, adatto ad animali dallo stadio L2 circa medio alla giovane età adulta; L3-A, per animali dalla metà dello stadio L3 circa alla giovane età adulta; e L4-A, che ospita animali dal tardo stadio L4 fino alla giovane età adulta.
Tutti i dispositivi seguono lo stesso layout essenziale, costituito da un ingresso a vite senza fine, 1 (o 2 in caso di scambio di fluidi) ingresso di cibo batterico e uno scarico comune. Tra la parte di ingresso e quella di uscita, è posizionata una serie di 41 canali di sifone paralleli, con il lato di ingresso e quello di uscita efficacemente separati dal canale di alimentazione alimentare di altezza inferiore (Figura 1A, A). Le dimensioni del canale sono state scelte per essere significativamente più lunghe e larghe rispetto alle viti senza fine caricate all'inizio di un esperimento, con una lunghezza/larghezza di 400/15 μm per i dispositivi L1, 575/22 μm per i dispositivi L1-L4, 800/27 μm per i dispositivi L2-A, 900/30 μm per i dispositivi L3-A e 1000/65 μm per i dispositivi L4-A (File supplementare 1, File supplementare 2, Fascicolo supplementare 3, Fascicolo supplementare 4 e Fascicolo supplementare 5).
La lunghezza del canale è stata selezionata in modo che corrispondesse alle dimensioni raggiunte dagli animali alla fine dell'esperimento, mentre la larghezza è stata scelta in modo che i vermi all'inizio dell'esperimento occupassero circa due terzi della larghezza del canale. Questo design consente spazio sufficiente per la crescita e il movimento durante la muta, impedendo al contempo ai vermi di girare o ruotare. A differenza della larghezza, l'altezza del canale è stata impostata vicino allo spessore dei vermi all'inizio dell'esperimento: 8 μm per le larve L1 precoci, 12 μm per le larve L1 medio/tardive, 15 μm per le larve L2 medie, 17,5 μm per le larve L3 medie e 22,5 μm per le larve L4 tardive, corrispondenti alle larve L1, L1-L4, L2-A, L3-A, e dispositivi L4-A, rispettivamente. L'altezza ridotta del canale, combinata con una larghezza accuratamente selezionata (mantenendo un rapporto altezza-larghezza di 0,5-0,6), assicura che le viti senza fine rimangano in un orientamento fisso. In particolare, con un'altezza del canale sufficientemente piccola, tutte le viti senza fine sono posizionate in modo coerente nell'orientamento laterale desiderato.
Il canale di alimentazione del cibo è stato fabbricato a un'altezza di 3 μm per L1 e 5 μm per tutti gli altri dispositivi, garantendo che i batteri possano raggiungere facilmente gli animali senza che gli animali fuoriescano dai canali della trappola. Tutti i dispositivi utilizzano lo stesso strato di alimentazione alimentare (File supplementare 6) e lo stesso strato di valvole (File supplementare 7). Si noti che lo strato della valvola è fabbricato su un wafer separato ad un'altezza di 20 μm e posizionato sullo strato del dispositivo solo durante la fabbricazione del dispositivo PDMS (passaggio 1.21).
Questi vincoli geometrici specifici dello stadio, essenziali per impedire ai vermi di ruotare o invertire la testa verso la coda, richiedono l'uso di tre tipi di dispositivi distinti per adattarsi a tutti gli stadi larvali. Se le larve L1 fossero collocate in un canale delle dimensioni di giovani animali adulti, non rimarrebbero stabili e uscirebbero rapidamente dal canale della trappola.
Preparazione di C. elegans
Salvo diversa indicazione, i vermi sono stati mantenuti secondo i protocolli standard25 su piastre NGM seminate con E. coli OP50 a 20 °C. La sezione relativa alla preparazione dei worm descrive in modo esplicito la preparazione di una popolazione di worm sincronizzata utilizzata in un esperimento di imaging. La sincronizzazione è stata effettuata secondo protocolli standard utilizzando candeggina di ipoclorito di sodio26, modificata con una fase di filtrazione aggiuntiva per rimuovere i detriti e gli animali non schiusi dopo la fame notturna. L'obiettivo è quello di ottenere molte larve di L1 sincronizzate, che vengono poi seminate su piastre NGM o caricate direttamente nel dispositivo di imaging. Altri metodi di sincronizzazione, come la raccolta manuale o la deposizione controllata delle uova, sono altrettanto adatti. Tuttavia, sono più laboriosi nel generare un numero sufficiente di animali (si raccomandano 200 per condizione).
Una volta che i vermi hanno raggiunto lo stadio desiderato, possono essere lavati delicatamente via dalla piastra NGM utilizzando S-Basal pulito (idealmente filtrato attraverso un filtro da 0,2 μm). L'obiettivo è quello di raccogliere il maggior numero possibile di animali senza raccogliere batteri o detriti dalla piastra. Gli animali vengono raccolti in una piccola provetta da centrifuga e lavati 2-3 volte con tampone S-Basal fresco, rimuovendo infine quanto più liquido possibile. Gli animali vengono quindi prelevati direttamente dal volume concentrato. Se la concentrazione degli animali è troppo bassa, il caricamento sarà più lento, richiedendo un volume maggiore di liquido per fluire attraverso il dispositivo e aumentando il rischio di ostruire i canali delle trappole con detriti.
Preparazione di alimenti batterici
La crescita dei batteri viene effettuata seguendo protocolli standard, con la concentrazione iniziale di batteri (OD600 = 1,9) aumentata di circa 40 volte nel tampone S-basale (filtrato attraverso un filtro da 0,2 μm) e miscelato con il mezzo del gradiente di densità e il tampone S-basale contenente PF (filtrato attraverso un filtro da 0,2 μm). Il risultato atteso di questa fase del processo è la preparazione di una sospensione batterica molto densa e la prevenzione della sedimentazione batterica durante l'esperimento di imaging. L'alta densità è necessaria per alimentare gli animali on-chip alle basse portate impiegate nell'erogazione dei terreni (1 μL/h). Lo scopo del mezzo del gradiente di densità è quello di prevenire la precipitazione dei batteri durante l'esperimento e lo scopo del PF è quello di impedire ai batteri di attaccarsi alle pareti del canale. Sia il mezzo di gradiente di densità che il PF non sono tossici e non influiscono sulla vitalità degli animali.
Se in un esperimento viene utilizzato un tipo di batterio diverso, sarà necessario determinare la concentrazione necessaria del mezzo del gradiente di densità. Ciò può essere ottenuto mescolando la sospensione batterica con quantità variabili del mezzo seguita dalla centrifugazione. Se la concentrazione del gradiente di densità è troppo bassa, i batteri si sedimentano; Se la concentrazione è troppo alta, i batteri galleggeranno. Una volta trovata una concentrazione adeguata, si consiglia di convalidare il risultato lasciando la miscela batterica a temperatura ambiente per circa 48 ore per indicare come si comporterà il cibo durante l'esperimento. Dopo l'esperimento, si consiglia di controllare la siringa attraverso la quale vengono alimentati i batteri e assicurarsi che i batteri non si sedimentino o galleggino. La differenza di volume risultante dall'aggiunta di diverse quantità di Optiprep può essere compensata aggiungendo più o meno S-Basal + 1% in peso PF. Allo stesso modo, è possibile aggiungere ulteriori mezzi o composti (ad es. auxina) alla sospensione e il volume extra viene sottratto dal volume di S-Basal + 1% in peso PF.
Preparazione dell'installazione
Posizionare il sistema di alimentazione a pressione il più vicino possibile al microscopio, in modo che possa essere utilizzato comodamente. Tutti i tratti dei tubi, in particolare il tubo da 1/16" che si collega al chip, devono rimanere corti (meno di 0,5 m per il tubo da 1/16" OD) (Figura 2 e Figura 3B). Come indicato nella sezione precedente, il controllo computerizzato della valvola su chip può essere ottenuto in vari modi. Dovrebbe essere possibile in qualsiasi software per microscopio (testato in Micro-Manager, Metamorph, NIS e ZenBlue), sia collegando una scheda microcontrollore dedicata (ad esempio, Arduino) sia collegando il sistema a una scheda di acquisizione dati (DAq) esistente, utilizzando un'uscita digitale. L'uscita della scheda selezionata deve quindi essere configurata come un otturatore, che si accende 10 s prima dell'acquisizione dell'immagine o viene azionato manualmente durante la configurazione del dispositivo. Come accennato in precedenza, si sconsiglia di collegare il solenoide direttamente al microcontrollore; invece, collegandosi tramite un MOSFET o un interruttore a relè in modo che il solenoide e il microcontrollore rimangano isolati elettronicamente (Figura 3C).
Simile al tubo di alimentazione della pressione, mantenere tutti gli altri tubi verso il chip (1/16" OD e 1/32" OD) il più corti possibile. Mantenere la vite senza fine e i tubi di scarico (1/16" OD) rispettivamente al di sotto di 2.5 cm e 20 cm. Il tubo di alimentazione dei batteri (1/32" OD) deve essere sufficientemente lungo da poter essere collegato dalla pompa a siringa al dispositivo una volta posizionato sul tavolino del microscopio. Tuttavia, si consiglia vivamente di accorciare il più possibile questa funzione e di posizionare la pompa a siringa vicino al tavolino del microscopio, ad esempio accanto al tavolino, utilizzando uno scaffale o sopra l'involucro di un incubatore. Qualsiasi lunghezza di tubo aggiunta al sistema aumenterà la contropressione avvertita durante il caricamento degli animali nel dispositivo e aumenterà la pressione totale sugli animali intrappolati.
Funzionamento del dispositivo
In questa fase, tutti i tubi possono essere preparati e collegati al rispettivo ago smussato, al perno d'acciaio e alla siringa (inizio della fase 4 del protocollo). Come accennato in precedenza, mantenere la lunghezza del tubo il più corta possibile (Figura 2J-N). È importante prestare attenzione che non rimangano bolle d'aria nelle siringhe o nei tubi, poiché queste interferiranno con il carico degli animali e potrebbero causare la fuga degli animali dal canale della trappola durante l'esperimento. Le bolle d'aria possono essere rimosse dalla siringa in diversi modi, ad esempio riempiendo la siringa, capovolgendola e scuotendola delicatamente in modo che le bolle d'aria salgano verso l'alto della siringa (non è consigliabile muovere la siringa, in quanto ciò può causare l'intrappolamento di ulteriori bolle d'aria). In alternativa, la siringa può essere riempita infilandola nel tampone e muovendo rapidamente lo stantuffo su e giù per rimuovere rapidamente la maggior parte delle bolle.
Una volta che la siringa è piena e priva di bolle, l'ago smussato e il tubo vengono collegati e riempiti con il tampone. Particolare attenzione deve essere prestata anche durante il riempimento della siringa, poiché l'aria può essere facilmente intrappolata nella parte in plastica dell'ago smussato. Se l'aria è intrappolata, sostituire l'ago. In alternativa, il preriempimento dell'ago con liquido, ad esempio utilizzando una pipetta, può alleviare il problema.
Con tutti i componenti in posizione, il tubo può essere collegato al dispositivo, a partire dal tubo che collega il solenoide e la valvola on-chip (Figura 2A-C,J). Questo tubo viene inizialmente riempito con acqua deionizzata e il sistema viene pressurizzato per rimuovere tutta l'aria dal canale della valvola senza uscita. Il PDMS è permeabile ai gas; quindi, la pressione elevata spingerà tutta l'aria dal canale nel PDMS, lasciando il canale della valvola pieno d'acqua. Questo processo può essere seguito su un microscopio, assicurandosi che l'intero canale valvolare sia riempito. Dopo aver riempito la valvola, è possibile collegare l'alimentazione dei batteri e il dispositivo può essere riempito e pressurizzato bloccando tutti gli ingressi/uscite aperti (Figura 2D, E, K, L). L'aria viene spostata e il processo può essere monitorato al microscopio. Una volta che tutta l'aria è stata spostata, i batteri dovrebbero riempire il dispositivo in modo uniforme. I perni di bloccaggio vengono rimossi e il tubo di raccolta dei rifiuti viene collegato (Figura 2F,G,M). A questo punto, una piccola quantità di liquido dovrebbe fluire attraverso il dispositivo nel tubo di raccolta dei rifiuti, assicurandosi che tutti i collegamenti al tubo siano aperti. Ciò può essere confermato visivamente prima di collegare il tubo al tubo di raccolta. Se il liquido non scorre verso il tubo di raccolta dei rifiuti, è probabile che sia bloccato. Ciò comporterebbe un accumulo di pressione sull'esperimento, influenzando negativamente gli animali intrappolati. Se il tubo o le sue connessioni sono ostruite, rimuoverlo e pulire tutte le connessioni con un ago o sciacquarle con una siringa piena d'acqua.
Infine, è possibile collegare il tubo a vite senza fine (Figura 2H,N). I vermi vengono prelevati dalla provetta da centrifuga semplicemente aspirandoli nel tubo collegato alla siringa riempita di S-Basal. Gli animali devono essere inseriti solo nel tubo, non nella siringa. Il tubo può quindi essere collegato al dispositivo. Prima di ciò, una piccola quantità di liquido viene espulsa dal dispositivo e dal tubo a vite senza fine e le due goccioline sono collegate in modo tale che non venga spinta aria nel dispositivo durante il caricamento.
Caricamento a vite senza fine
Con tutti i collegamenti effettuati, gli animali possono essere caricati spingendo e tirando lo stantuffo della siringa a vite senza fine. Gli animali verranno scaricati dall'ingresso verso l'array di canali della trappola; Tuttavia, nuoteranno naturalmente contro il flusso che sperimentano. Gli animali, quindi, devono essere riorientati con la testa rivolta verso il canale della trappola. Il corretto orientamento dell'animale si ottiene spingendo e tirando lo stantuffo e dando agli animali il tempo di ruotare nell'orientamento desiderato. Questo può essere ottenuto quasi su una base a verme singolo, ma richiederà un po' di pratica. Una volta che gli animali sono orientati correttamente, possono essere spinti nel canale della trappola e scivolare fino alla fine del canale, dove un gradino in altezza li ferma (Figura 1A, B). Se gli animali non possono entrare nel canale, sono troppo grandi per il dispositivo selezionato e devono essere ripresi in un dispositivo più grande. Se gli animali possono ruotare o girarsi quando vengono inseriti nel canale della trappola, sono troppo giovani e deve essere utilizzato un dispositivo più piccolo, oppure gli animali devono essere lasciati crescere per qualche ora in più.
È importante sottolineare che gli animali in canali diversi non si influenzeranno a vicenda. Pertanto, gli animali caricati in modo errato non influenzeranno quelli caricati correttamente, tuttavia, non avranno sufficiente accesso al cibo e quindi cresceranno più lentamente o arresteranno lo sviluppo.
Se gli animali vengono caricati all'età corretta, dovrebbe verificarsi un movimento minimo lungo l'asse del canale longitudinale. Tuttavia, gli animali sono liberi di dimenarsi (Figura 1B) e iniziare a nutrirsi entro un paio di minuti. Se gli animali mostrano un movimento significativo lungo l'asse del canale longitudinale, ciò è probabilmente causato dall'accumulo di pressione nel dispositivo o da una bolla d'aria depositata da qualche parte nel tubo, nell'ago smussato o nella siringa. L'accumulo di pressione all'interno del dispositivo può essere rilasciato staccando delicatamente il tubo a vite senza fine dall'ago smussato e riattaccandolo delicatamente. Se una bolla d'aria è rimasta depositata all'interno del sistema nonostante i passaggi e le precauzioni sopra menzionati, potrebbe essere necessario rimuovere l'intera siringa del verme e sostituirla con una nuova senza bolle d'aria. Le bolle d'aria nel sistema funzionano come molle che vengono compresse durante il carico e rilasciate lentamente dopo il caricamento degli animali. Il flusso creato dall'espansione delle bolle d'aria può causare instabilità del sistema, spingendo gli animali fuori dai canali della trappola.
Acquisizione e sviluppo di immagini su chip
Infine, una volta caricati gli animali, può iniziare l'acquisizione delle immagini. In linea di principio, i dispositivi sono compatibili con la maggior parte dei metodi di imaging, campo chiaro, epifluorescenza 18,19,22,23 (Figura 4), disco rotante confocale19 e persino modalità di super-risoluzione (AiryScan24), poiché tutto l'imaging viene eseguito attraverso un vetro di copertura spesso 170 μm. Di conseguenza, il dispositivo microfluidico mostra un effetto minimo o nullo sulla qualità dell'immagine ottenibile sul chip. Tuttavia, devono essere selezionate condizioni di imaging appropriate che non influiscano sulla vitalità degli animali indipendentemente dalla modalità di imaging. Elevate intensità di eccitazione provocano facilmente lo sbiancamento dei fluorofori e l'arresto dello sviluppo a causa dell'elevata energia e del calore impartiti al campione. Si consigliano tempi di esposizione brevi (~10 ms) e basse intensità di eccitazione (meno del 10% di una tipica sorgente LED a fluorescenza), che producono un'immagine utilizzabile con un'impostazione di contrasto di min = 0 e max = 1000 (quando si utilizza una fotocamera a 16 bit) (Figura 4).
I dati rappresentativi ottenuti utilizzando i dispositivi L1, L1-4 e L2-A sono mostrati nella Figura 4. Mostra lo sviluppo delle cellule epiteliali di C. elegans dalla schiusa/fame notturna allo stadio larvale medio L2 (Figura 4A, Filmato supplementare 1), l'induzione delle cellule precursori della vulva 1°-fatate e le loro successive divisioni dalla fine di L1 allo stadio larvale precoce di L4 (Figura 4B, Filmato supplementare 2) e infine la formazione della vulva di C. elegans dallo stadio larvale precoce di L3 fino all'eversione al passaggio all'età adulta (Figura 4C, Filmato supplementare 3).
Ogni applicazione è accompagnata da una quantificazione di diverse metriche di temporizzazione dello sviluppo. Le immagini nella Figura 4A, B sono mostrate senza alcuna post-elaborazione applicata, ovvero non è stata applicata alcuna deconvoluzione o registrazione dell'immagine. Le immagini nella Figura 4C, d'altra parte, sono state deconvolute27 e registrate, evidenziando i miglioramenti nella qualità dell'immagine possibili quando si utilizzano dispositivi di imaging a lungo termine.
In primo luogo, identificare l'inizio di ciascuna divisione delle cellule di cucitura per gli animali mostrati nella Figura 4A (vedere il filmato supplementare 1 per un corso a tempo pieno), indicando la divisione coerente e tempestiva di tutte le cellule in 12 animali (Figura 4D). Utilizzando le cellule di cucitura, è stata valutata anche la tempistica generale dello sviluppo in tutti e quattro gli stadi larvali e si è scoperto che tutte le divisioni delle cellule di cucitura sono state completate dopo 12,0 ore ± 1,7 ore (media ± DS) in L1 (n = 22), 10,2 ore ± 1,0 ore in L2 (n = 27), 10,9 ore ± 1,4 ore in L3 (n = 27) e 14,6 ore ± 2,2 ore negli animali L4 (n = 19). Questi dati sono coerenti con i valori della letteratura 2,28 e con i valori misurati negli animali allevati su piastra, hanno prodotto tempi di sviluppo mediani per L1 15 h (n = 21), L2 10 h (n = 15), L3 12 h (n = 21) e L4 12,5 h (n = 61) (adattato da Berger et al.18). I lievi ritardi osservati sono probabilmente una conseguenza dell'acquisizione dell'immagine e della conseguente fototossicità o dell'aumento del confinamento una volta che gli animali sono cresciuti e saranno un canale di trappola.
In secondo luogo, abbiamo seguito la crescita degli animali attraverso gli stadi L2 e L3, quantificando la lunghezza delle gonadi come un eccellente indicatore della progressione dello sviluppo (Figura 4E, vedi Filmato supplementare 2 per un corso a tempo pieno). Come per la divisione delle cellule di cucitura, la lunghezza precisa delle gonadi può essere misurata grazie all'orientamento dritto dell'animale ed è stata riscontrata una crescita costante delle gonadi in tutti gli animali ripresi (n = 19). La prima divisione di VPC in questi esperimenti è stata osservata 14,26 ore ± 2,47 ore dopo l'inizio dell'esperimento (~30 ore dopo la semina), con la seconda che si è verificata 1,46 ore ± 0,13 ore dopo (n = 19). Questi dati sono in buon accordo con i valori di letteratura stabiliti sulle lastre NGM29,30. Euling e Ambros30 hanno scoperto che la prima divisione si è verificata dopo circa 29 ore e la seconda circa 3 ore dopo.
Infine, è stato quantificato il tempo entro il quale vengono raggiunte specifiche fasi dello sviluppo vulvare (Figura 4F, vedi Filmato supplementare 3 per il corso a tempo pieno), valutando in particolare il tempo necessario per passare dall'invaginazione (L4.0, t1) allo stadio medio di L4 (L4.5, t4) secondo le sottofasi definite da Mok et al.31. Il tempo medio di progressione tra t1-t2 è stato di 3,8 ore ± 1,2 ore, tra t2-t3 è stato di 3,5 ore ± 1,2 ore e tra t3-t4 è stato di 2,8 ore ± 1,6 ore (n = 7), indicando una progressione dello sviluppo approssimativamente lineare. Analogamente al test della tempistica dello sviluppo, questi tempi, in particolare t1-t2, appaiono leggermente ritardati rispetto ai valori riportati in letteratura (t1-t2: 0,7 h, t2-t3: 3 h, t3-t4: 2,3 h31), probabilmente a causa delle dimensioni ridotte del dispositivo che rallentano la muta. Tuttavia, i tempi di sviluppo rimangono altamente coerenti tra gli animali e tutti progrediscono con successo verso l'età adulta.
Compensazione del movimento degli animali e della deriva del microscopio
Il movimento degli animali in un singolo Z-stack è in genere minimo quando viene azionata la valvola on-chip. Tuttavia, non ci si aspetta che gli animali rimangano completamente fermi. Il jitter residuo osservato in un singolo stack può essere compensato aumentando la pressione per azionare la valvola on-chip, diminuendo il tempo di esposizione utilizzato o aumentando il passo Z selezionato per accelerare l'acquisizione dello stack. Il tempo di acquisizione dello stack può essere ulteriormente ridotto utilizzando un azionamento Z piezoelettrico, con conseguente movimento Z significativamente più veloce rispetto a un azionamento di messa a fuoco convenzionale del microscopio, o utilizzando filtri multibanda (ad esempio, GFP/mCherry) in modo tale da poter acquisire colori separati senza la necessità di lunghe sostituzioni del filtro. A titolo di riferimento, negli esempi forniti (Figura 4 e Filmato supplementare 1, Filmato supplementare 2 e Filmato supplementare 3), il tempo di acquisizione dello stack era in genere compreso tra 3 e 8 secondi. Se necessario, il movimento residuo può anche essere rimosso attraverso vari metodi di registrazione dell'immagine.
Si prevede che gli animali su chip cresceranno costantemente. Pertanto, le caratteristiche di interesse si sposteranno inevitabilmente lungo l'asse longitudinale del canale (Figura 4A, B, Filmato supplementare 1 e Filmato supplementare 2). Pertanto, si consiglia di impostare il FOV di imaging in modo tale che la caratteristica di interesse possa crescere nel FOV e non venga persa nel corso di un esperimento (Figura 1B e Figura 4A, B). In alternativa, gli animali possono essere ripresi in più FOV adiacenti, coprendo l'intero corpo animale. Tuttavia, questo può conferire ulteriore fototossicità e stress. Come per Z-motion, varie procedure di registrazione possono compensare il movimento degli animali nel canale nel tempo. Se gli animali mostrano un movimento irregolare lungo l'asse longitudinale, più esteso di quello che può essere attribuito alla crescita, è probabile che ciò sia causato da una bolla d'aria.
Ogni dispositivo di imaging a lungo termine contiene sei unità di imaging individuali, ognuna delle quali può essere utilizzata per un esperimento separato. Ogni unità può ospitare fino a 41 animali e i canali delle trappole sono distanziati in modo tale da poter riprendere più animali in un unico FOV. Inoltre, è possibile acquisire immagini di più FOV all'interno di una singola unità o dispositivo. Come per tutti i campioni da microscopio, il movimento sulla superficie del campione può introdurre una deriva della messa a fuoco dovuta alla perdita di mezzi di immersione. Questa deriva può essere compensata utilizzando un olio da immersione a bassa viscosità o attraverso varie modalità di messa a fuoco automatica. Tuttavia, alcuni sistemi hardware di messa a fuoco automatica potrebbero non funzionare se utilizzati con un dispositivo microfluidico PDMS, poiché utilizzano la transizione dal vetro a un mezzo acquoso come punto di riferimento (lato campione del vetro di copertura). Gran parte del FOV è riempito con silicone all'interno del dispositivo, che ha un indice di rifrazione più elevato rispetto all'acqua; Sebbene ciò non influisca sulla qualità dell'immagine, potrebbe influire negativamente sull'autofocus. Quando si esegue l'imaging di un singolo FOV, la deriva della messa a fuoco osservata all'interno del dispositivo è minima (meno di 2 μm). Se si osserva un'eccessiva deriva della messa a fuoco, ciò può derivare dall'instabilità termica del sistema o dell'ambiente del microscopio, dall'eccessivo movimento XY che acquisisce più FOV o da un montaggio errato del dispositivo sul microscopio (vedere la Figura 3A per i meccanismi di montaggio suggeriti).
Allo stesso modo, un movimento XY eccessivo e un montaggio improprio possono causare una deriva XY. Poiché il dispositivo e l'obiettivo del microscopio sono collegati tramite il liquido di immersione, movimenti rapidi in X, Y o Z possono causare l'applicazione di forze al dispositivo. Se i dispositivi sono montati correttamente e non agiscono forze esterne su di essi, sono eccezionalmente stabili con una deriva minima della messa a fuoco e nessuna deriva XY.
Infine, se specifici parametri di sistema sono inappropriati, i dispositivi microfluidici PDMS possono influire negativamente sullo sviluppo animale. La vitalità è influenzata da un'eccessiva pressione della valvola, da un'elevata fototossicità derivante da elevate intensità di eccitazione, da lunghi tempi di esposizione o da brevi intervalli di tempo tra le acquisizioni in pila. Soprattutto quando si utilizza questo sistema per la prima volta, si consiglia vivamente di valutare la vitalità degli animali utilizzando un marcatore fluorescente luminoso o l'imaging in campo chiaro, assicurandosi che le condizioni di imaging non influenzino la crescita degli animali. Una volta che gli animali si sviluppano in modo affidabile, i parametri di imaging possono essere variati in base alle esigenze.
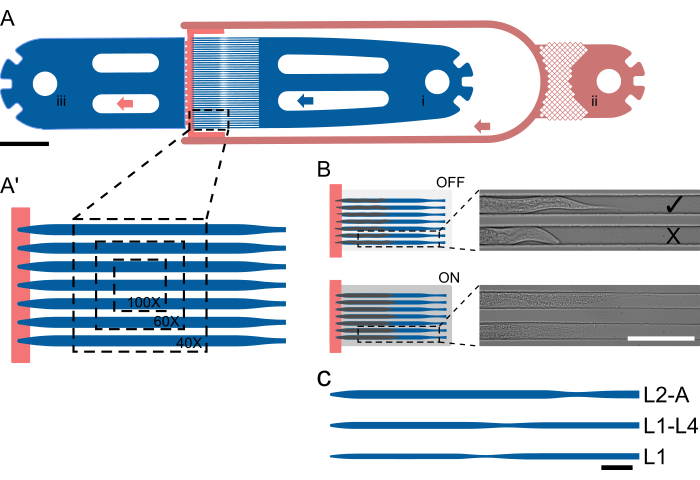
Figura 1: Layout del dispositivo e funzionamento schematico. (A) Panoramica del dispositivo che mostra una singola unità con l'ingresso della vite senza fine (i), l'ingresso del cibo batterico (ii) e l'uscita generale (iii). (A') Vista ingrandita del canale della trappola in un dispositivo L2-A, evidenziando il numero di vermi visibili in un singolo FOV a diversi ingrandimenti (40x-100x). (B) Principio di funzionamento della valvola on-chip. Quando è OFF, l'altezza del canale viene selezionata in modo che gli animali possano entrare comodamente nel canale della trappola. Quando è acceso, l'altezza del canale diminuisce, premendo l'animale sul vetro di copertura, immobilizzando così in modo reversibile gli animali intrappolati. Le immagini mostrano due animali intrappolati sul chip con la valvola sul chip OFF e con la valvola ON, con gli animali che si raddrizzano e si allungano visibilmente sotto pressione. L'animale superiore caricato correttamente e rivolto verso la fonte di cibo ( ), e quello inferiore rivolto verso l'esterno (X). (C-C") Configurazione schematica del dispositivo. (C) Dimensioni disponibili del dispositivo. L1 è adatto per animali dallo stadio L1 allo stadio L2 medio, L1-4 per animali dallo stadio larvale L2 L2 medio a L4 medio e L2-A per animali dallo stadio larvale L2 medio all'età adulta. Le barre della scala sono (A) 1000 μm, (B) 100 μm e (C) 50 μm. La figura è parzialmente adattata da Berger et al.18. Clicca qui per visualizzare una versione più grande di questa figura.
), e quello inferiore rivolto verso l'esterno (X). (C-C") Configurazione schematica del dispositivo. (C) Dimensioni disponibili del dispositivo. L1 è adatto per animali dallo stadio L1 allo stadio L2 medio, L1-4 per animali dallo stadio larvale L2 L2 medio a L4 medio e L2-A per animali dallo stadio larvale L2 medio all'età adulta. Le barre della scala sono (A) 1000 μm, (B) 100 μm e (C) 50 μm. La figura è parzialmente adattata da Berger et al.18. Clicca qui per visualizzare una versione più grande di questa figura.
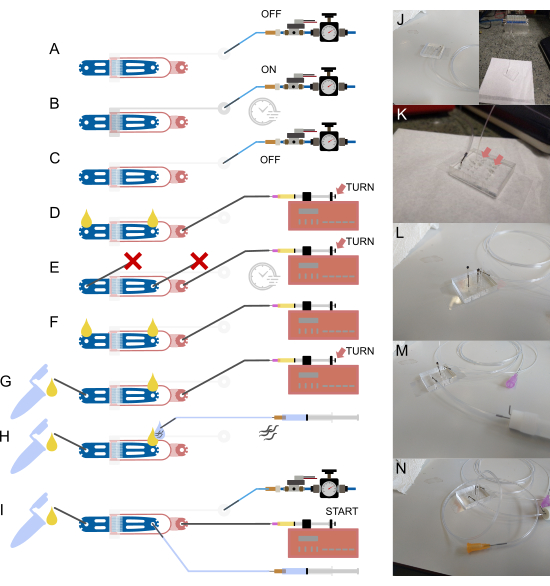
Figura 2: Configurazione e connessioni del dispositivo. (A-I) Panoramica schematica del processo di configurazione. (A) Collegamento di un tubo riempito d'acqua da 1/16" all'ingresso della valvola utilizzando un perno cavo in acciaio (grigio), con l'altra estremità collegata al sistema di alimentazione della pressione. (B) Riempimento della valvola idraulica on-chip tramite il solenoide off-chip e pressurizzazione del dispositivo per alcuni minuti. (C) Disattivazione della pressione prima di procedere alle fasi successive. (D) Collegamento di una siringa contenente cibo batterico all'ingresso del cibo utilizzando un tubo da 1/32" e un ago smussato da 30 G, quindi riempimento del dispositivo fino a quando non appare del liquido su entrambe le connessioni aperte. (E) Sigillatura dell'ingresso della vite senza fine e dell'uscita di scarico con perni in acciaio bloccati da un pezzo di tubo da 1/16" ( ) e pressurizzazione del dispositivo utilizzando la siringa per alimenti e la pompa (tramite regolazione a vite a testa zigrinata), mantenendo la pressione per alcuni minuti. (F) Rimozione delle spine da (E), assicurandosi che si formino goccioline di liquido ad ogni connessione aperta. (G) Collegamento del contenitore dei rifiuti tramite un breve pezzo di tubo da 1/16" e un perno cavo in acciaio. (H) Fissaggio di una siringa riempita di tampone al dispositivo utilizzando un perno cavo in acciaio e un ago smussato da 23 G. Le viti senza fine vengono aspirate nel tubo prima di collegarlo al dispositivo, assicurando che non venga introdotta aria allineando le goccioline di liquido all'ingresso e all'estremità del tubo. (I) Configurazione finale con tutte le connessioni del dispositivo stabilite. (J) Immagine del dispositivo con tubo della valvola collegato all'ingresso e al solenoide (sfondo), corrispondente a (A-C). (K) Vista ingrandita del dispositivo con il tubo di alimentazione del cibo collegato, che mostra due goccioline di liquido (frecce) sull'ingresso della vite senza fine e sull'uscita di scarico, corrispondenti a (D). (L) Vista ingrandita del dispositivo con il tubo di alimentazione del cibo collegato e l'ingresso della vite senza fine e l'uscita dei rifiuti ostruiti, corrispondente a (E). (M) Vista ingrandita del dispositivo con il tubo di scarico e il contenitore collegati, corrispondente a (G). (N) Assemblaggio finale con tutti i collegamenti dei tubi in posizione, corrispondenti a (I). Parzialmente adattato da Berger et al.18. Clicca qui per visualizzare una versione più grande di questa figura.
) e pressurizzazione del dispositivo utilizzando la siringa per alimenti e la pompa (tramite regolazione a vite a testa zigrinata), mantenendo la pressione per alcuni minuti. (F) Rimozione delle spine da (E), assicurandosi che si formino goccioline di liquido ad ogni connessione aperta. (G) Collegamento del contenitore dei rifiuti tramite un breve pezzo di tubo da 1/16" e un perno cavo in acciaio. (H) Fissaggio di una siringa riempita di tampone al dispositivo utilizzando un perno cavo in acciaio e un ago smussato da 23 G. Le viti senza fine vengono aspirate nel tubo prima di collegarlo al dispositivo, assicurando che non venga introdotta aria allineando le goccioline di liquido all'ingresso e all'estremità del tubo. (I) Configurazione finale con tutte le connessioni del dispositivo stabilite. (J) Immagine del dispositivo con tubo della valvola collegato all'ingresso e al solenoide (sfondo), corrispondente a (A-C). (K) Vista ingrandita del dispositivo con il tubo di alimentazione del cibo collegato, che mostra due goccioline di liquido (frecce) sull'ingresso della vite senza fine e sull'uscita di scarico, corrispondenti a (D). (L) Vista ingrandita del dispositivo con il tubo di alimentazione del cibo collegato e l'ingresso della vite senza fine e l'uscita dei rifiuti ostruiti, corrispondente a (E). (M) Vista ingrandita del dispositivo con il tubo di scarico e il contenitore collegati, corrispondente a (G). (N) Assemblaggio finale con tutti i collegamenti dei tubi in posizione, corrispondenti a (I). Parzialmente adattato da Berger et al.18. Clicca qui per visualizzare una versione più grande di questa figura.
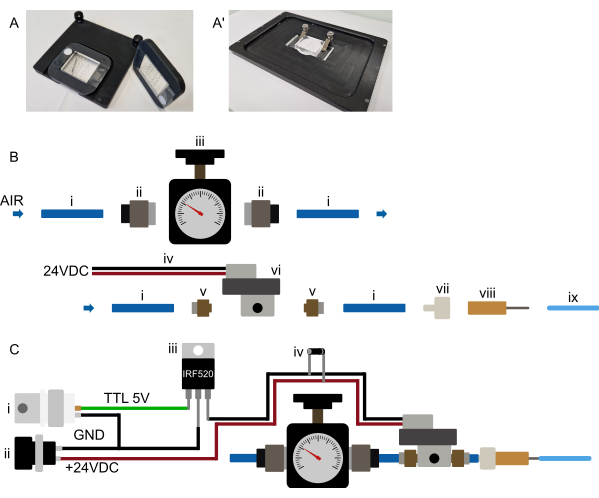
Figura 3: Configurazione hardware. (A-A') Montaggio del dispositivo suggerito. (A) Possibile montaggio per un microscopio verticale. I dispositivi sono fissati a un telaio rettangolare stampato in 3D con un grande ritaglio centrale. Due piccole strisce di nastro biadesivo vengono inizialmente posizionate sul vetro di copertura, su entrambi i lati della parte PDMS del dispositivo. Il telaio stampato in 3D viene quindi posizionato sopra il dispositivo, in modo che la parte PDMS aderisca attraverso il ritaglio e il telaio venga premuto saldamente sul nastro biadesivo, fissando in modo permanente il dispositivo al telaio. Il telaio, quindi, tiene il dispositivo in modo sicuro e viene tenuto sul tavolino del microscopio tramite magneti incorporati nel telaio e nell'inserto del tavolino. Ciò garantisce un montaggio stabile del dispositivo e previene la collisione dell'obiettivo del microscopio con qualsiasi hardware di montaggio. Tutti i tubi sono instradati verso la parte anteriore del dispositivo. (A') Possibilità di montaggio per un microscopio invertito. Una serie di morsetti tiene saldamente il dispositivo. Assicurarsi che i morsetti premano il dispositivo sul tavolino per evitare qualsiasi movimento XY durante il funzionamento. (B) Vista delle parti del sistema di pressione. i) tubo in poliuretano con diametro esterno di 6 mm collegato a una fonte di pressione. (ii) Adattatore push-in da G1/8 a 6 mm che collega il tubo (i) al regolatore di pressione (iii). Il tubo viene quindi collegato a un solenoide (vi) tramite un adattatore push-in da M5 a 6 mm (v) e, infine, il tubo viene collegato a un connettore luer-lock-to-barb (vii) e a un ago smussato da 23 G (viii) al tubo da 1/16" OD collegato al dispositivo microfluidico. (C) Panoramica schematica dei collegamenti elettronici e del sistema di pressione assemblato. (i) Il trigger TTL (+3,3-5 V) generato da un Arduino, una scheda DAq o qualsiasi altro microcontrollore, insieme a un alimentatore adeguato (ii, 24 VDC) è collegato a un MOSFET (iii) e alle connessioni elettroniche del solenoide come mostrato nello schema, con un diodo flyback (iv) collegato attraverso le connessioni di alimentazione del solenoide. Una volta che un segnale TTL viene inviato dal microcontrollore al gate del MOSFET, +24 VDC viene applicato al solenoide normalmente chiuso e la valvola idraulica on-chip viene gonfiata. Una volta rimosso il segnale TTL, la pressione viene rilasciata e la valvola idraulica on-chip si sgonfia. Clicca qui per visualizzare una versione più grande di questa figura.
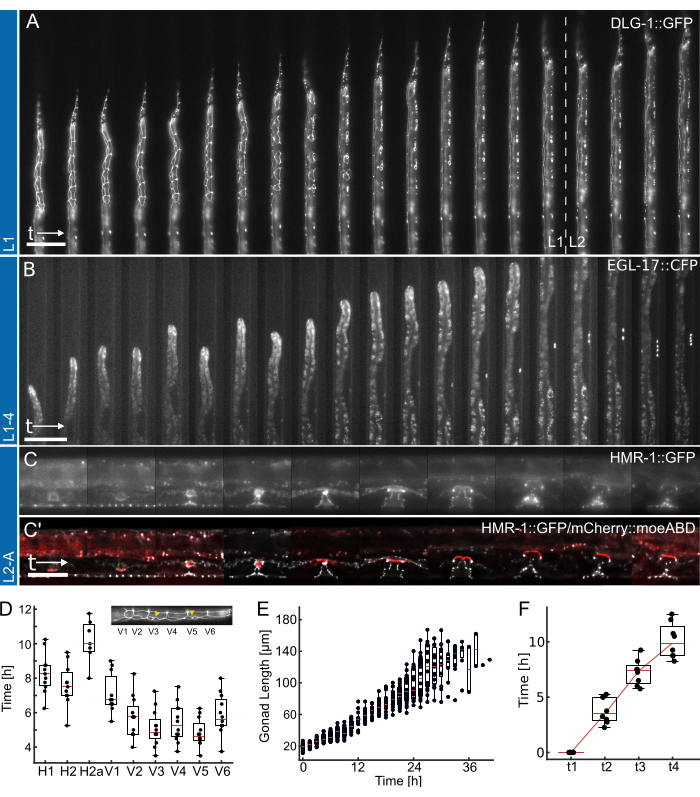
Figura 4: Risultati rappresentativi. (A) Animale che mostra un fattore di giunzione epiteliale (ML2615, dlg-1(mc103[dlg-1::gfp])) che cresce dalla fame notturna allo stadio larvale L2 iniziale/medio, utilizzando il dispositivo L1. Sono visibili le cellule di cucitura che si dividono, così come le cellule P, che migrano e si dividono verso il tardo stadio larvale L1. (B) Allevamento di animali dallo stadio larvale L1 tardivo fino allo stadio L4 iniziale, utilizzando il dispositivo L1-4. È visibile un marcatore per la cellula precursore della vulva (VPC) di 1° fato (AH1187, arIs92[egl-17::cfp]), con una singola cellula visibile all'inizio dell'esperimento, seguita da due cicli di divisioni cellulari visibili nello stadio larvale L3. (C) Animali che crescono dalla fase larvale L2 tardiva fino alla prima età adulta, utilizzando il dispositivo L2-A. È visibile un creatore di giunzione epiteliale (AH5786, hmr-1 (cp21[hmr-1::gfp + LoxP])), che evidenzia lo sviluppo del tessuto vulvare formando strutture toroidali distinte, e un'immagine combinata con un marcatore per la cellula di ancoraggio uterina (AC) (AH5786, qyIs50[Pcdh-3>mCherry::moeABD, unc-119(+)]) che invade il tessuto vulvare verso la fine dello stadio larvale L3, collegando i tessuti. Per tutti gli esperimenti (A-C), il FOV è inizialmente impostato in modo che solo una parte degli animali sia visibile. Man mano che l'esperimento procede, l'animale riempirà più del canale con le caratteristiche che si spostano lungo l'asse del canale come conseguenza della crescita dell'animale. (D) Tempo in cui le singole divisioni delle cellule di cucitura iniziano a dividersi durante lo stadio larvale L1 (n = 12). L'inserto evidenzia le identità delle cellule di cucitura (H1-V6) lungo l'asse antero-posteriore del corpo. (E) Mostra la crescita delle gonadi con il tempo per gli animali nella Figura 4B (n = 19). (F) Mostra il tempo necessario per raggiungere 4 distinti punti temporali di sviluppo nella morfogenesi vulvare (t1-t4) indicando una transizione coerente per tutti gli animali ripresi (n = 7). Le barre della scala sono (A,B) 50 μm e (C) 25 μm. Vedi Film supplementare 1, Film supplementare 2 e Film supplementare 3 per il corso a tempo pieno. I box plot mostrano i valori mediani (linee rosse) con quartili superiore e inferiore e barre che indicano gli estremi della distribuzione, esclusi i valori anomali. La figura è adattata da Berger et al.18. Clicca qui per visualizzare una versione più grande di questa figura.
Filmato supplementare 1: Singola larva di C. elegans che si sviluppa dalla fame notturna fino allo stadio iniziale di L2. Vengono visualizzate immagini a epifluorescenza proiettate a massima intensità (l'altezza totale della proiezione è di 1,5 μm), in formato altrimenti non elaborato. Sono visibili tutti gli eventi di divisione e fusione delle cellule di giunzione che si verificano nella fase specificata (in alto). Le caratteristiche sono delineate utilizzando il marcatore DLG-1::GFP (giunzione epiteliale) a intervalli di 15 minuti per un totale di 25 ore. Il film è stato riutilizzato da Berger et al.18. Clicca qui per scaricare questo filmato.
Filmato supplementare 2: Singola larva che si sviluppa dallo stadio L1 tardivo fino allo stadio L4 precoce, esprimendo un marcatore EGL-17::CFP (cellula precursore vulvare (VPC) 1°-fatale). All'inizio di L2 (~400 min), la fluorescenza EGL-17::CFP si manifesta in P6.p, aumentando continuamente di intensità. Durante L3, P6.p subisce due cicli consecutivi di divisione, formando gli stadi di sviluppo della vulva a 1, 2 e 4 cellule. Le immagini vengono mostrate a intervalli di 30 minuti per un totale di 34 ore. Il film è stato riutilizzato da Berger et al.18. Clicca qui per scaricare questo filmato.
Filmato supplementare 3: Vista dall'alto: Proiezione Z della vulva in via di sviluppo dallo stadio L3 fino alla transizione L4/adulto. Vista in basso: Proiezione X della stessa vulva nel tempo. Entrambe le opinioni mostrano la divisione iniziale dei VPC, seguita dall'invaginazione, dalla formazione e dall'allargamento dei toroidi, seguita dal collasso durante l'eversione. Le immagini in epifluorescenza vengono visualizzate dopo il ritaglio, la deconvoluzione, la registrazione e la proiezione. Le caratteristiche sono delineate attraverso il marcatore HMR-1::GFP (ipoderma e VPC) e il marcatore mCherry::moeABD (che delinea la AC). Il film è stato riutilizzato da Berger et al.18. Clicca qui per scaricare questo filmato.
File supplementare 1: file CAD per il dispositivo L1. Clicca qui per scaricare questo file.
File supplementare 2: file CAD per il dispositivo L1-4. Clicca qui per scaricare questo file.
File supplementare 3: file CAD per il dispositivo L2-A. Clicca qui per scaricare questo file.
File supplementare 4: file CAD per il dispositivo L3-A. Clicca qui per scaricare questo file.
File supplementare 5: file CAD per il dispositivo L4-A. Clicca qui per scaricare questo file.
File supplementare 6: file CAD per la struttura di distribuzione alimentare. Clicca qui per scaricare questo file.
File supplementare 7: file CAD per lo strato valvolare. Clicca qui per scaricare questo file.
Discussione
Questo articolo ha descritto il funzionamento di un'esclusiva piattaforma di imaging microfluidico a lungo termine, adatta per l'imaging ad alta risoluzione e il monitoraggio di vari processi di sviluppo in C. elegans durante tutti e quattro gli stadi larvali fino alla prima età adulta18. La capacità di seguire i processi di sviluppo nel tempo in vivo, utilizzando questo metodo di imaging e altri, è diventata indispensabile in molti campi della ricerca su C. elegans e ha permesso di rispondere a domande precedentemente inaccessibili 23,24,29.
A differenza dei metodi di imaging tradizionali che utilizzano tamponi di agar e strategie di imaging a lungo termine basate su microfluidica pubblicate in precedenza, questo approccio preserva l'identità e l'orientamento del verme durante l'esperimento, consentendo il monitoraggio di processi di sviluppo complessi nel tempo. Indipendentemente dallo stadio di sviluppo, gli animali sono confinati in uno dei 41 canali di trappola paralleli, ciascuno collegato a una fornitura di cibo su chip e coperto da una grande valvola idraulica. L'immobilizzazione di tutte le viti senza fine intrappolate avviene attraverso il gonfiaggio della valvola idraulica, premendo le viti senza fine sul vetro di copertura solo durante l'acquisizione dell'immagine. L'imaging ad alta produttività è possibile grazie all'array di canali trappola a spaziatura ristretta, con più animali visibili all'interno di un singolo campo visivo. I dispositivi di imaging a lungo termine sono compatibili con la maggior parte delle configurazioni di microscopio, richiedendo un hardware aggiuntivo minimo (meno di $ 1000) e nessuna modifica all'hardware esistente. Il funzionamento dei dispositivi è intenzionalmente semplice, quindi tutti gli utenti dovrebbero essere in grado di apprenderlo rapidamente seguendo il protocollo di cui sopra e i risultati delineati.
In breve, gli animali possono essere ripresi su chip fino a 2,5 stadi larvali (Figura 1C). Gli animali vengono inizialmente sincronizzati e caricati direttamente sul chip (dispositivo L1) o seminati sulla piastra NGM fino allo stadio di sviluppo desiderato (dispositivi L1-L4 e L2-A). Gli animali on-chip vengono alimentati utilizzando una sospensione batterica altamente concentrata e immobilizzati utilizzando una valvola idraulica on-chip azionata dal software di imaging (Figura 1B). Gli animali vengono manovrati nei canali della trappola utilizzando una siringa, orientando attentamente gli animali con la testa verso l'alimentazione del cibo e infine intrappolando gli animali all'estremità del canale della trappola (Figura 1A e Figura 2). È necessario prestare attenzione in modo che l'aria non rimanga intrappolata nelle siringhe, negli aghi smussati e nei tubi, poiché le bolle d'aria disturberanno facilmente la posizione dell'animale. I dispositivi sono compatibili con la maggior parte delle modalità di imaging 18,19,24, poiché il materiale PDMS è altamente trasparente, non fluorescente e sigillato contro un sottile vetro di copertura (Figura 4, Filmato supplementare 1, Filmato supplementare 2 e Filmato supplementare 3). Tuttavia, è necessario prestare attenzione quando si selezionano le condizioni di imaging, poiché lunghi tempi di esposizione e un'elevata intensità di eccitazione influenzeranno facilmente la vitalità degli animali e provocheranno il fotosbiancamento.
Oltre alla versione presentata di questo metodo di imaging, sono possibili e già disponibili diverse modifiche al protocollo. La modifica più semplice consiste nel sostituire i ceppi batterici standard, ad esempio con batteri che esprimono RNAi18 o batteri con valori nutrizionali diversi32. I ceppi batterici possono essere facilmente sostituiti regolando il contenuto di Optiprep per tenere conto delle differenze di densità. Allo stesso modo, un numero qualsiasi di composti può essere aggiunto al cibo batterico, ad esempio auxina, nutrienti o composti farmacologici a piccole molecole, e il loro effetto viene osservato direttamente in vivo, senza alcuna necessità di modifiche al protocollo. In particolare, è stato sviluppato un dispositivo di scambio multimediale per la degradazione delle proteine indotta dall'auxina o per qualsiasi applicazione che richieda un'esposizione temporizzata o reversibile aggiungendo un ingresso di cibo al layout del dispositivo senza modificare la geometria o il funzionamento. L'ingresso aggiuntivo consente di passare facilmente da una condizione all'altra, ad esempio auxina e assenza di auxina, collegate al dispositivo nello stesso modo mostrato qui per una singola fonte di cibo, e le variazioni risultanti vengono osservate direttamente. Sono state inoltre sviluppate varianti di dispositivo in cui la valvola idraulica non è condivisa tra tutte le unità di un dispositivo, ma separata per ciascuna unità, in modo tale che più condizioni possano essere visualizzate all'interno dello stesso dispositivo e sperimentate individualmente, riducendo gli effetti potenzialmente dannosi dei lunghi tempi di azionamento e acquisizione della valvola. Infine, oltre alle varianti originali del dispositivo descritte in Berger et al.18, sono state sviluppate due nuove varianti del dispositivo, L3-A e L4-A (introdotte qui), che consentono l'imaging dallo stadio medio L3 o medio L4 fino all'età adulta. Lo scopo di queste modifiche è quello di ritardare l'ora di inizio di un esperimento a uno stadio larvale successivo, in modo tale che gli animali più grandi possano essere caricati in applicazioni in cui le prime fasi di sviluppo non sono di interesse.
Divulgazioni
Gli autori dichiarano di non avere interessi concorrenti o finanziari.
Riconoscimenti
Desideriamo ringraziare i membri del laboratorio Hajnal per la discussione critica e i commenti sul manoscritto. Siamo anche grati al C. elegans Genetics Center CGC, finanziato dall'Office of Research Infrastructure Programs (P40 OD010440) del NIH. Inoltre, desideriamo ringraziare i membri del laboratorio Galli (Hubrecht Institute), del laboratorio Conradt (University College London) e del van den Heuvel lab (Università di Utrecht) per il prezioso contributo al protocollo presentato. Questo lavoro è stato sostenuto da sovvenzioni del Fondo nazionale svizzero per la ricerca scientifica n. 31003A-166580 ad AH, dalla Lega svizzera contro il cancro n. 4377-02-2018 ad AH e da finanziamenti dell'ETH di Zurigo ad AdM.
Materiali
| Name | Company | Catalog Number | Comments |
| 3/2 solenoid valve | Distrelec | 154-22-898 and 154-22-899 | Alternative to above. Solenoid valve connecting the pressure regulator to the on-chip valve. |
| Arduino | Distrelec | 301-01-956 | Optional microcontroller depending on the selected controller and interface. |
| Bacto-Tryptone | Thermo Fisher Scientific | 211705 | For L Broth production. |
| Bacto-Yeast | Thermo Fisher Scientific | 212750 | For L Broth production. |
| Blunt needle 23G | Gonano Dosiertechnik | GGA723050 | Blunt needle connecting the 1/16" tubing to a syringe. |
| Blunt needle 30G | Gonano Dosiertechnik | IG-TE730050 | Blunt needle connecting the 1/32" tubing to a syringe. |
| C. elegans Construct 1 | NA | NA | AH5786 LGI: hmr-1(cp21[hmr-1::gfp + LoxP]) Marston, D.J. et al. MRCK-1 drives apical constriction in C. elegans by linking developmental patterning to force generation. Curr. Biol. 26 (16), 2079-2089. 2016. |
| C. elegans Construct 2 | NA | NA | AH1187 LGII: arIs92[egl-17::cfp] Yoo, A.S., Bais, C., Greenwald, I. Crosstalk between the EGFR and LIN-12/Notch pathways in C. elegans vulval development. Science. 303 (5658), 663-666. 2004. |
| C. elegans Construct 3 | NA | NA | AH5786 LGV: qyIs50[Pcdh-3>mCherry::moeABD, unc-119(+)] Ziel, J.W., Hagedorn, E.J., Audhya, A., Sherwood, D.R. UNC-6 (netrin) orients the invasive membrane of the anchor cell in C. elegans. Nat. Cell Biol., 11 (2), 183-189. 2009. |
| C. elegans Construct 4 | NA | NA | ML2615 LGX: dlg-1(mc103[dlg-1::gfp]) Vuong-Brender, T.T.K., Suman, S.K., Labouesse, M. The apical ECM preserves embryonic integrity and distributes mechanical stress during morphogenesis. Development. 144 (23), 4336-4349. 2017. |
| Cable | Distrelec | 143-46-644 | Needed if selecting the solenoid from distrelec. Cable connecting the solenoid to the MOSFET. |
| Chlorotrimethyl silane | Sigma Aldrich | 386529-25ML | Silane for wafer passivation. |
| Cholesterol | Sigma Aldrich | C8667 | For S-Basal production. |
| Flyback Diode | Distrelec | 110-52-628 | Flyback diode protecting the microcontroller. |
| Hollow steel pin | Gonano Dosiertechnik | GGA7R23050 | 23G hollow steel pin connecting the 1/16" tubing to the device. |
| L Broth | NA | NA | Prepared in house following standard recipe. 10 g Bacto-tryptone, 5 g Bacto-yeast, 5 g NaCl, H2O to 1 litre, pH to 7.0 using 1 M NaOH |
| Long-term Imaging Device L1 | NA | NA | Long-term imaging device for L1 to early/mid L2 larvae. Made inhouse, available on request. Also available as media exchange variant. |
| Long-term Imaging Device L1-4 | NA | NA | Long-term imaging device for mid L1 to mid L4 larvae. Made inhouse, available on request. Also available as media exchange variant. |
| Long-term Imaging Device L2-A | NA | NA | Long-term imaging device for mid L2 to adulthood (eversion). Made inhouse, available on request. Also available as media exchange variant. |
| Long-term Imaging Device L3-A | NA | NA | Long-term imaging device for mid L3 to adulthood (eversion). Made inhouse, available on request. Also available as media exchange variant. |
| Long-term Imaging Device L4-A | NA | NA | Long-term imaging device for mid L4 to adulthood (eversion). Made inhouse, available on request. Also available as media exchange variant. |
| M9 Buffer | NA | NA | Prepared in house following standard recipe. |
| MOSFET | Distrelec | 303-41-391 | Metal-oxide-semiconductor field-effect transistor, used to switch the 24VDC required for the solenoid. |
| NGM plates | NA | NA | Prepared in house following standard recipe. |
| Nylon Luer-Fitting | Fisher Scientific | 11776048 | Adapter from OD 6mm tubing to 23G blunt needle. |
| Optiprep | Sigma Aldrich | D1556 | Density matching liquid. |
| PDMS | Ameba | Elastosil RT601A/B | PDMS elastomer kit. |
| Pluronic F-127 | Sigma Aldrich | P2443 | Non-ionic surfactant. |
| Potassium dihydrogen phosphate | Sigma Aldrich | 1.37039 | For S-Basal production. |
| Potassium hydrogen phosphate | Sigma Aldrich | 1.05099 | For S-Basal production. |
| Power Supply 24VDC | Distrelec | 301-63-304 | 24VDC power supply. Select a power supply suitable for the solenoid selected. |
| Precision pressure regulator | Distrelec | 154-23-578 | Alternativ to above. Pressure regulator. |
| Pressure Gauge G1/8 | Distrelec | 154-23-643 | Pressure gauge attaches to the pressure regulator. |
| PU Tubing OD 6mm | Distrelec | 154-23-866 | Tubing connecting the pressure source and regulator, as well as regulator and solenoid valve. |
| Push-in fitting G1/8 to 6mm | Distrelec | 301-69-330 | Alternativ to above. 2x Push-in fitting attaches to the pressure regulator and connects to the 6mm tubing. |
| Push-in fitting M5 to 6 mm | Distrelec | 301-69-459 | 2x Push-in fitting attaches to the solenoid and the 6mm tubing. |
| S-Basal Buffer | NA | NA | Prepared in house following standard recipe. 5.85 g NaCl, 1 g K2HPO4, 6 g KH2PO4, 1 ml cholesterol (5 mg/ml in ethanol), H2O to 1 litre. Sterilize by autoclaving. |
| Sodium Chloride | Sigma Aldrich | S9625 | For L Broth and S-Basal production. |
| Sodium hydroxite (5M) | Sigma Aldrich | 567530 | Sodium hydroxide used when bleaching C. elegans (prepare solution at 5M concentration) |
| Sodium hypochlorite (5%) | Sigma Aldrich | 1.05614 | Sodium hypochlorite used when bleaching C. elegans (prepare solution at 5% concentration) |
| Strainer 10 µm | pluriSelect | 43-10010-40 | Alternative for bacteria food preparation, especially if smaller devices are used. |
| Strainer 5 µm | pluriSelect | 43-10005-40 | Strainer used to filter L1 larvae after overnight starvation and bacteria after completed food preparation. |
| SU-8 | Gersteltec | GM1050 | SU-8 for height from 1-10 µm. |
| SU-8 | Gersteltec | GM1060 | SU-8 for height from 5-30 µm. |
| SU-9 | Gersteltec | GM1070 | SU-8 for height from 15-200 µm. |
| Syringe 1 mL | Fisher Scientific | 11338763 | 1mL syringe. If using a different type ensure that the syringe body and plunger are stiff, such that they won't be deformed by the syringe pump. |
| Syringe Pump | WPI | Al1000-220 | Syringe pump (https://www.wpiinc.com/var-2300-aladdin-single-syringe-pump.html). |
| Tube 15 mL | Fisher Scientific | 50-809-220 | Tube used for worm bleaching and bacteria preparation. |
| Tube 2 mL | Fisher Scientific | NC1186931 | Tube used for worm preparation and bacteria preparation. |
| Tube 50 mL | Fisher Scientific | 50-465-232 | Tube used for bacteria preparation and growth. |
| Tweezer SMD | Brütsch Rüegg Werkzeuge | 448980 | Tweezers used to plug the 1/32" tubing into the device. |
| Tygon tubing 1/16" | Fisher Scientific | 642002 | Flexible 1/16" OD tubing connecting most components to the device. |
| Tygon tubing 1/32" | Fisher Scientific | 641900 | Flexible 1/32" OD tubing connecting the bacteria supply to the device. |
| UV KUB-3 | Kloe | NA | Mask aligner and UV exposure system. |
| Wafer | Siegert Wafer | 4P0/>1/525±25/SSP/TTV<10 | Silicon Wafer. |
| Waste Container | NA | NA | Prepared in house using a 2mL tube or a cryo vial and hollow steel pins. |
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon