Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Seguimiento temporal de la progresión del ciclo celular El uso de citometría de flujo sin la necesidad de sincronización
En este artículo
Resumen
Este protocolo describe el uso de bromodesoxiuridina (BrdU) para permitir la absorción de la seguimiento temporal de las células que estaban en fase S en un punto específico en el tiempo. La adición de colorantes de ADN y el etiquetado de anticuerpos facilita el análisis detallado del destino de las células en fase S en épocas posteriores.
Resumen
This protocol describes a method to permit the tracking of cells through the cell cycle without requiring the cells to be synchronized. Achieving cell synchronization can be difficult for many cell systems. Standard practice is to block cell cycle progression at a specific stage and then release the accumulated cells producing a wave of cells progressing through the cycle in unison. However, some cell types find this block toxic resulting in abnormal cell cycling, or even mass death. Bromodeoxyuridine (BrdU) uptake can be used to track the cell cycle stage of individual cells. Cells incorporate this synthetic thymidine analog, while synthesizing new DNA during S phase. By providing BrdU for a brief period it is possible to mark a pool of cells that were in S phase while the BrdU was present. These cells can then be tracked through the remainder of the cell cycle and into the next round of replication, permitting the duration of the cell cycle phases to be determined without the need to induce a potentially toxic cell cycle block. It is also possible to determine and correlate the expression of both internal and external proteins during subsequent stages of the cell cycle. These can be used to further refine the assignment of cell cycle stage or assess effects on other cellular functions such as checkpoint activation or cell death.
Introducción
La evaluación de las características del ciclo celular y los cambios que se producen en las células durante la progresión del ciclo celular es fundamental para la comprensión de muchos aspectos de la biología, en particular la biología del cáncer. Muchos agentes en desarrollo para el tratamiento de tumores malignos tienen efectos profundos sobre la progresión del ciclo celular o inducir la muerte celular a través del ciclo celular dependiente de mecanismos. Con el fin de estudiar la dinámica del ciclo celular o células en una fase particular del ciclo celular, es habitual para sincronizar las células. Sin embargo métodos de sincronización pueden tener efectos perjudiciales sobre las células que se estudian, potencialmente confundiendo a los resultados obtenidos. 1 análisis Recientemente, el uso de proteínas etiquetadas con fluorescencia que sólo están presentes en fases particulares del ciclo de las células han permitido de la progresión del ciclo celular en las células individuales a través del tiempo 2, sin embargo las células a ser estudiadas necesidad de ser manipulados genéticamente para expresar estas proteínas etiquetadas, lo que limita su uso a sistemas en los que esto puede ser leidoslia logrado.
El ciclo celular consiste de dos fases activas: la fase de síntesis (S), donde se replica el ADN y la mitosis (M), donde la división celular tiene lugar. Estas fases están separadas por tres fases gap, G 0, G 1 y G 2. G 0 o quiescencia, es una fase de reposo donde la célula ha dejado el ciclo, G 1 es donde las células aumentan de tamaño antes de la replicación del ADN y G 2, donde el crecimiento celular continúa entre la terminación de la replicación del ADN pero antes de la división celular. La progresión a través del ciclo celular está controlada por un número de puntos de control. El G 1 puesto de control se activa cuando las condiciones ambientales no son de apoyo de la síntesis de ADN e impide la entrada en fase S. El puesto de control de fase intra-S o el retraso pueden ser provocados por daños en el ADN que puede resultar en las horquillas de replicación estancado. Durante G 2 la fidelidad de la ADN replicado se confirma y si se detecta daño, entonces el G 2 puesto de control se activa permitiendo la reparación del ADN antes de la división celular. Un punto de comprobación final durante la mitosis asegura que cromátidas se han alineado correctamente en la placa mitótico de modo que la división celular se puede completar con éxito. 3 La activación de estos puntos de control se utiliza comúnmente para sincronizar las poblaciones de células. Los puntos de control del ciclo celular pueden ser activados por un número de factores, pero en la biología del cáncer el más común es la detección de daños en el ADN. La respuesta al daño del ADN se inicia por la PI3-quinasa quinasas ataxia y telangiectasia Rad3 relacionados (ATR) y ataxia telangiectasia mutada (ATM) que activan las quinasas efectoras aguas abajo Chk1 y Chk2, respectivamente. 3 Una serie de eventos se activa Chk1 incluyendo estancado horquillas de replicación, entrecruzamientos de ADN, y el daño por radiación ultravioleta, mientras que Chk2 se activa principalmente por roturas de doble hebra.
El método usual para estudiar el efecto de las condiciones alteradas de la longitud de la i ciclo celulars para sincronizar las células en una fase particular del ciclo celular. 1 Esto puede lograrse a través de varios métodos. Las células pueden separarse, físicamente basan en el tamaño, densidad, dispersión lateral (granularidad), y marcadores de expresión de la superficie celular. Más prácticamente, las células pueden ser sincronizados por medios químicos. Varios agentes tales como la timidina, la hidroxiurea y arabinósido de citosina se pueden utilizar para inhibir la síntesis de ADN en la fase S del ciclo celular que resulta en una acumulación de células en fase S que siguen ciclismo después se eliminan los agentes. Las células tratadas con nocodazol, que previene la formación del huso mitótico, arresto con un G 2 - o M-fase el contenido de ADN. Eliminación de suero a partir de los resultados de medio de cultivo en la acumulación de células en G 0 fase. El re-adición de los nutrientes dentro de las séricos cultura re-inicia el ciclo normal de las células. Sin embargo, todos estos métodos de sincronización interfieren con el ciclo normal y crecimiento de las células y puede Result en la muerte celular significativa.
Sincronización de las células de la leucemia linfoblástica aguda es particularmente difícil y estas células no son susceptibles a la manipulación genética. El método aquí descrito permite la evaluación de la dinámica del ciclo celular y el estudio de células en fases particulares del ciclo celular sin sincronización tradicional o modificación genética. Este método también puede ser útil para otros tipos de células que no se consigue fácilmente la modificación genética y los procedimientos de sincronización tradicionales. El método se basa en el uso a largo establecido de bromodesoxiuridina (BrdU) incorporación, que tiene muy poco impacto en el crecimiento a corto plazo y la proliferación de las células. 4 protocolos establecidos BrdU toman ventaja de la incorporación de BrdU en el ADN recién sintetizado durante la fase S . Esto marca permanentemente las células como haber estado en la fase S durante la exposición BrdU. Esta población puede ser identificado en los puntos de tiempo posteriores por tinción para incor BrdUación y por lo tanto actúan como una población sincronizada que puede ser seguido y evaluado con el tiempo lo permite el estudio de los efectos del fármaco sobre el tránsito del ciclo celular. BrdU necesita ser expuesta antes de la tinción de anticuerpos, por lo general realizados a raíz de DNasa o tratamiento ácido 6,7. El uso de la citometría de flujo para detectar BrdU incorporado permite la inclusión de marcadores adicionales. El más importante es el uso de tintes para medir el contenido de ADN, lo que permite la evaluación de la distribución de fase del ciclo celular de las células que estaban en fase S en el inicio del estudio. 8 de superficie o intracelulares antígenos Además adicionales también pueden ser estudiados. 9 Estos pueden estar relacionados con los eventos del ciclo celular, como Ki67 o funciones celulares para aparentemente no relacionados, tales como marcadores de apoptosis como exfoliados caspasa-3. Las posibles aplicaciones están limitadas por la imaginación del investigador.
Access restricted. Please log in or start a trial to view this content.
Protocolo
El protocolo descrito aquí utiliza la línea celular de leucemia linfoblástica aguda NALM6 pero se puede aplicar a otros tipos de células.
1. Soluciones y Reactivos
- RPMI completo
- Añadir 56 ml de suero de ternera fetal (FCS) y 5,5 ml de 200 mM de L-glutamina a una botella de 500 ml de medio RPMI-1640.
- BrdU de la Solución
- Preparar 32,5 mM BrdU (10 mg / ml) en fosfato de Dulbecco Buffered Saline (DPBS).
- BrdU completo RPMI
- Añadir 6,2 l de solución madre de BrdU a 10 ml de RPMI completo.
- Solución DNasa
- Preparar 1 mg de DNasa / ml en DPBS.
- La tinción Buffer
- Preparar 3% de FCS inactivado por calor y 0,09% de azida de sodio en DPBS.
- Consulte la Lista de materiales para las definiciones de Fijación Buffer, permeabilización Buffer y el tampón de lavado.
2. Las células
NoTe: Las células no fueron cultivadas por más de 6 meses. Este método es directamente adaptable a cualquier línea celular no adherente con ajustes a la densidad de células y medios de cultivo. Utilice células que están creciendo exponencialmente a la iniciación del experimento.
- Mantener células NALM6 en frascos de cultivo T-75 en RPMI completo. Realice todos los pasos en condiciones estériles utilizando una Clase II Bioseguridad gabinete.
- Mantener células NALM6 entre 2.1 x 10 6 células por ml mediante el fraccionamiento de la cultura tres veces por semana.
- Se incuba a 37 ° C en 5% de CO 2 en el aire.
3. Pulso Etiquetado de células con BrdU
PRECAUCIÓN: Manipule BrdU con cuidado, ya que es un mutágeno potencial y teratógeno.
- Centrifugar las células a 150 xg durante 5 min. Nota: La transferencia de células en medio fresco mejora la reproducibilidad de los resultados.
- Realizar un recuento de células y las células volver a suspender en RPMI completo a 2 x 10 6 canas / ml.
- Diluir las células en 1 2 con BrdU RPMI completo produciendo una concentración final de células de 1 x 10 6 células / ml.
- Incubar a 37 ° C con 5% de CO2 durante 45 min, luego se diluye células 1 en 10 con RPMI completo. Centrifugar las células a 150 xg durante 5 min y cuidadosamente descartan todo el sobrenadante.
- Resuspender las células en un volumen pequeño (~ 100 l) de RPMI completo, realizar un recuento de células y se ajustan a 1 x 10 6 células / ml.
- Pipetear 1 ml de las células en los pocillos de una placa de 48 pocillos. Pipeta 1 ml de DPBS en cualquier pozo desocupadas para obtener resultados más reproducibles.
- Incubar a 37 ° C en 5% de CO2 en aire para puntos de tiempo deseados, aquí 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17 , 18, 19, 20, 21, 22, 23 y 24 h. Nota: La cantidad de tiempo dependerá de lo que tiene como objetivo el diseño experimental para medir.
- Transferencia de todas las células en tubos de FACS utilizando una pipeta. Enjuague la secuencialmente bien con 1 ml volumes de PBS a un volumen total final de 5 ml.
- Centrifugar a 150 xg durante 5 minutos y retirar con cuidado todo el sobrenadante. Las células están listas para la tinción, realizan esta (sección 4) inmediatamente.
4. La tinción celular
Nota: Si se requiere tinción de la superficie de las células que realizar antes de la fijación, asegurando que las células se mantienen a 4 ° C durante todo.
- Resuspender las células en 100 l de tampón de tinción (para la tinción de superficie opcional, añadir el volumen recomendado de anticuerpo a la superficie antígenos y se incuba durante 30 min a 4 ° C).
- Añadir 1 ml de tampón de tinción, de centrifugación durante 5 min a 150 xg y descartar el sobrenadante.
Nota: El anticuerpo específico, concentración, tiempo de incubación etc. variará dependiendo de las metas experimentales específicas. - Fijación y permeabilización
- Resuspender las células en 100 l de tampón de fijación y se incuba durante 15 min a temperatura ambiente.
- Añadir 1 ml otampón de lavado f, centrifugar durante 5 min a 150 xg y desechar el sobrenadante.
- Resuspender las células en 100 l de tampón de permeabilización e incubar las células durante 10 min en hielo.
- Añadir 1 ml de tampón de lavado, de centrifugación durante 5 min a 150 xg, y descartar el sobrenadante.
- Resuspender las células en 100 l de tampón de fijación por tubo y se incuba durante 5 min a temperatura ambiente.
- Añadir 1 ml de tampón de lavado, de centrifugación durante 5 min a 150 xg, y descartar el sobrenadante.
Nota: El protocolo puede ser una pausa aquí, si es necesario. Las células fijadas son estables durante varios días a 4 ° C si se resuspendieron en tampón de tinción. Retire el tampón de tinción después de la centrifugación antes de continuar.
- DNasa tratamiento
- Resuspender las células en 100 l de solución de DNasa (30 g de DNasa / 10 6 células) y se incuban las células durante 1 hora a 37 ° C.
- Añadir 1 ml de tampón de lavado, centrifugar a 150 xg durante 5 min y desechar el sobrenadante.
- Antibody tinción
Nota: La tinción para marcadores intracelulares distintos de BrdU se puede realizar simultáneamente con la tinción de BrdU.- IMPORTANTE: Preparar los controles de compensación que constan de células no teñidas y células marcadas con cada solo fluorocromo. Lo ideal es utilizar los mismos anticuerpos para los controles de compensación como los utilizados en los tubos experimentales. Sin embargo, si esto no es factible, los anticuerpos a los antígenos sustitutos altamente expresados conjugarse con el mismo fluorocromo.
- Resuspender las células en 50 l de tampón de lavado y añadir 1 l / 10 6 células de anticuerpo BrdU. Nota: anticuerpos conjugado directamente a otros antígenos intracelulares específicas también se pueden añadir.
NOTA:. Los anticuerpos frente a la histona H3 fosforilada en Ser10 se pueden utilizar para discriminar entre células en G2 y M, la histona H3 es fosforilada en Ser10 durante la mitosis 10 Los anticuerpos frente a cdc2 fosforilados en Tyr15 puede ser utilizado para detectar las células que have comprometida con la mitosis. 11 - Se incuban las células durante 20 min a temperatura ambiente.
- Añadir 1 ml de tampón de lavado, las células de centrifugación a 150 xg durante 5 min y desechar el sobrenadante.
- ADN de manchas de Análisis de Ciclo Celular
- Aflojar pellet y añadir 20 l de la solución de 7-AAD (0,25 g). Nota: Es importante utilizar una cantidad constante de 7-AAD / célula.
- Resuspender las células en 1 ml de tampón de tinción.
5. Recogida de Citometría de Flujo de Datos
Nota: La máquina requerida dependerá del número y naturaleza de los fluorocromos usados.
- Recoge los siguientes parámetros: FSC-A, SSC-A, FSC-H (FSC-W se pueden utilizar en lugar de FSC-H) y 7-AAD fluorescencia en una escala lineal. Recoger el canal de APC en una escala logarítmica. Recoge los canales adicionales que se requieren para la evaluación de la superficie o internos etiquetas utilizando una escala logarítmica.
- Realizar comcompensación de las señales en los espectros de emisión observada entre diferentes fluorocromos antes de analizar las muestras superpuestas. Nota: La mayoría de citómetros de flujo realizarán automáticamente.
- Recoge al menos 10.000 eventos en cada muestra.
6. Análisis de Citometría de Flujo de Datos
Nota: FlowJo se utilizó en este estudio para el análisis de citometría de flujo de datos, pero otros paquetes de software también pueden ser utilizados. La estrategia gating se ilustra en la Figura 1.
- Identificar la población de células viables utilizando parámetros FSC-A y SSC-A.
- Dentro de esta población excluir dobletes y agregados utilizando FSC-A y FSC-H (FSC-W también puede ser utilizado aquí).
- Dentro de esta población establecer un gráfico de puntos usando 7-AAD en el eje x y BrdU-APC en el eje y.
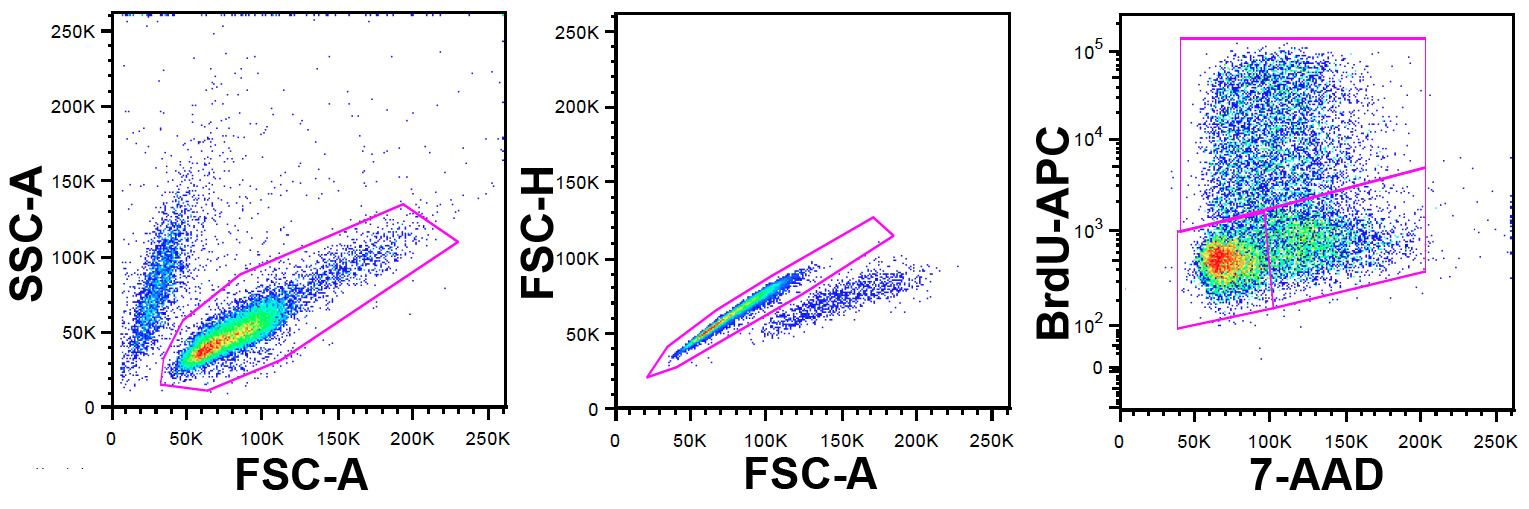
Figura 1: Estrategia de apertura de puerta izquierda pa.nel: células ungated se muestran en una FCS-A vs SSC-Un gráfico de puntos. La población de células viable se identifica por la puerta se muestra. Panel central: células cerradas desde el panel de la izquierda se muestran en una FSC-A vs FSC-H gráfico de puntos (FSC-W se puede utilizar en lugar de la altura). Dobletes y agregados se identifican y excluidos por la puerta se muestra. Panel derecho: células cerradas a partir de la fecha de exclusión doblete en el panel central se muestran en un 7-AAD vs. APC-Un gráfico de puntos. El anticuerpo BrdU se etiqueta con APC que permite la identificación de las células que han incorporado BrdU durante el etiquetado pulso. 7-AAD proporciona información sobre el contenido de ADN. La puerta superior define células positivas para BrdU y por lo tanto en la fase S en el momento del impulso de BrdU, la puerta inferior izquierda, las células en G 0/1 y la puerta inferior derecha los de G 2 / M. Por favor, haga clic aquí para ver una versión más grande de esta cifra.
- Ciclo Celular A nálisis
- Abra la primera archivo de datos y la puerta a las células en la puerta de exclusión doblete.
- Analizar esta población para la distribución del ciclo celular (que se encuentra bajo plataformas de software FloJo) y utilizar el modelo de Dean-Jett-Fox.
- Obtener las posiciones del G 0/1 y G 2 / M picos utilizando crear puertas.
- Puerta en la BrdU células positivas y sujetos a esta población con el mismo análisis del ciclo celular.
- Proporcionar las posiciones para el G 0/1 y G 2 / M picos mediante la aplicación de las mismas puertas de crear puertas y el establecimiento de restricciones (utilizando las puertas creadas) para las posiciones del G 0/1 y G 2 / M picos. Esto se ilustra en los primeros 2 paneles de la Figura 2.
Nota: Otro software también se puede utilizar para analizar los datos y las instrucciones podría variar en consecuencia.
840 / 52840fig2highres.jpg "width =" 700 "/>
Figura 2:. Progresión El primer panel de Ciclo Celular (Todas las células) es cerrada en la población de células definida por la puerta de la exclusión doblete. Esta población se muestra en el histograma con 7-AAD en el eje X. El pico de la 0/1 pico G está indicado por la flecha por debajo del eje. En los paneles posteriores células BrdU positivas han sido cerrada en como se muestra en la Figura 1. Se aplica el valor para la posición G 0/1 obtenida cuando gating de la puerta de exclusión doblete a las células BrdU positivas cerradas dentro de software FlowJo ciclo celular. Cada panel posterior fue cerrada en la población BrdU positivo como se muestra en la Figura 1 y la posición del pico G 0/1 basado en el valor obtenido en el análisis de toda la población como se muestra en los dos primeros paneles. Uso de la fracción negativa BrdU para identificar la ubicación de la población G 0/1 para las células BrdU positivas en la misma sample controla de ningún ligeras diferencias en la intensidad de la mancha de ADN entre muestras. El número que aparece en cada panel representa el tiempo desde que terminó el pulso de BrdU. Las fases del ciclo celular calculados se muestran en verde sombra. Por favor, haga clic aquí para ver una versión más grande de esta figura.
Access restricted. Please log in or start a trial to view this content.
Resultados
Esta metodología se puede utilizar para obtener una gama de información. Algunas aplicaciones se describen aquí.
Evaluación de la duración del ciclo celular
Para determinar el tiempo requerido para que las células tránsito a través del ciclo celular, las células se cosecharon a diversos puntos de tiempo tras el pulso de BrdU. Los intervalos entre las evaluaciones pueden ser adaptadas a las células particulares que se analiza. Líneas celulares hematopoy...
Access restricted. Please log in or start a trial to view this content.
Discusión
La capacidad de analizar el ciclo celular es importante para la comprensión de la biología del cáncer y el mecanismo de acción de ambos fármacos y genes que influyen en la proliferación celular y el crecimiento. Mientras que hay una multitud de ensayos que miden la proliferación de células según se informa, la mayoría sólo proporciona una medida que indica las células número presente. Estos incluyen ensayos que miden el número de células por visualización directa y el recuento, la actividad metabólica o...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
The authors have nothing to disclose.
Agradecimientos
The work was funded by the Leukemia and Lymphoma Society of the USA (6105-08), a Cancer Council NSW grant (13-02), an NHMRC Senior Research Fellowship (LJB) (1042305) and project grant (1041614).
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| APC BrdU Flow Kit | BD Biosciences | 552598 | Contains BrdU antibody, 7-AAD and BD Cytofix/Cytoperm Buffer (referred to as Fixation Buffer) |
| BD Cytoperm Permeabilization Buffer Plus | BD Biosciences | 561651 | Referred to as Permeabilization buffer |
| BD Perm/Wash Buffer | BD Biosciences | 554723 | Referred to as Wash buffer |
| DNase | Sigma | D-4513 | |
| BD Falcon 12 x 75 mm FACS tubes | BD Biosciences | 352008 | |
| BD Pharmingen Stain Buffer | BD Biosciences | 554656 | |
| BD LSR FORTESSA flow cytometer | BD Biosciences | FORTESSA | |
| Pipetman | Gilson | P2, P20, P100, P1000 | |
| RPMI 1,640 w/o L-Gln 500 ml | Lonza | 12-167F | |
| DPBS | Lonza | 17-512F | |
| Fetal Bovine Serum | FisherBiotec | FBS-7100113 | |
| L-Glutamine | Sigma | G7513-100ML | |
| 5-Bromo-2′-deoxyuridine | Sigma | B5002-1G | |
| Falcon TC 150 cm2 vented Flasks | BD Biosciences | 355001 | |
| Pipettes 25 ml | Greiner | 760180 | |
| Aersol Pipettes 200 µl | Interpath | 24700 | |
| Aersol Pipettes 1 ml | Interpath | 24800 | |
| Centrifuge | Spintron | GT-175R | |
| CO2 incubator | Binder | C 150 | |
| AF488 anti-Histone H3 Phospho (Ser10) Antibody | Cell Signalling | 9708S | |
| Phospho-Chk2 (Thr68) (C13C1) Rabbit mAb | Cell Signalling | 2197S | |
| Phospho-Chk1 (Ser345) (133D3) Rabbit mAb | Cell Signalling | 2348S | |
| NALM6 | DSMZ | ACC-128 |
Referencias
- Banfalvi, G. Methods Mol Biol. Banfalvi, G. 761, Humana Press. New York, Dordrecht, Heidelberg, London. 1-23 (2011).
- Sakaue-Sawano, A., et al. Visualizing spatiotemporal dynamics of multicellular cell-cycle progression. Cell. 132, 487-498 (2008).
- Harper, J. W., Elledge, S. J. The DNA damage response: ten years after. Molecular Cell. 28, 739-745 (2007).
- Latt, S. A., George, Y. S., Gray, J. W. Flow cytometric analysis of bromodeoxyuridine-substituted cells stained with 33258 Hoechst. The Journal of Histochemistry and Cytochemistry. 25, 927-934 (1977).
- Gratzner, H. G. Monoclonal antibody to 5-bromo- and 5-iododeoxyuridine: A new reagent for detection of DNA replication. Science. 218, 474-475 (1982).
- Carayon, P., Bord, A. Identification of DNA-replicating lymphocyte subsets using a new method to label the bromo-deoxyuridine incorporated into the DNA. Journal of Immunological Methods. 147, 225-230 (1992).
- Gonchoroff, N. J., et al. S-phase detection with an antibody to bromodeoxyuridine. Role of DNase pretreatment. Journal of Immunological Methods. 93, 97-101 (1986).
- Rabinovitch, P. S., Torres, R. M., Engel, D. Simultaneous cell cycle analysis and two-color surface immunofluorescence using 7-amino-actinomycin D and single laser excitation: applications to study of cell activation and the cell cycle of murine Ly-1 B cells. Journal of Immunology. 136, 2769-2775 (1986).
- Saunders, P. O., et al. RAD001 (everolimus) induces dose-dependent changes to cell cycle regulation and modifies the cell cycle response to vincristine. Oncogene. 32, 4789-4797 (2013).
- Hendzel, M. J., et al. Mitosis-specific phosphorylation of histone H3 initiates primarily within pericentromeric heterochromatin during G2 and spreads in an ordered fashion coincident with mitotic chromosome condensation. Chromosoma. 106, 348-360 (1997).
- Draetta, G., Beach, D. Activation of cdc2 protein kinase during mitosis in human cells: cell cycle-dependent phosphorylation and subunit rearrangement. Cell. 54, 17-26 (1988).
- Saunders, P. O., et al. RAD001 (everolimus) induces dose-dependent changes to cell cycle regulation and modifies the cell cycle response to vincristine. Oncogene. , (2012).
- Knudsen, R. C., Ahmed, A. A., Sell, K. W. An in vitro microassay for lymphotoxin using microculture plates and the multiple automated sample harvester. Journal of Immunological Methods. 5, 55-63 (1974).
- Beck, H. P. Proliferation kinetics of perturbed cell populations determined by the bromodeoxyuridine-33258 technique: radiotoxic effects of incorporated [3H]thymidine. Cytometry. 2, 170-174 (1981).
- Miller, M. J., Wei, S. H., Parker, I., Cahalan, M. D. Two-photon imaging of lymphocyte motility and antigen response in intact lymph node. Science. 296, 1869-1873 (2002).
- Collins, J. M., Berry, D. E., Bagwell, C. B. Different rates of DNA synthesis during the S phase of log phase HeLa S3, WI-38, and 2RA cells. Journal of Biological Chemistry. 255, 3585-3590 (1980).
- Schwarting, R., Gerdes, J., Niehus, J., Jaeschke, L., Stein, H. Determination of the growth fraction in cell suspensions by flow cytometry using the monoclonal antibody Ki-67. Journal of Immunological Methods. 90, 65-70 (1986).
- Danova, M., et al. Cell cycle-related proteins: a flow cytofluorometric study in human tumors. Biology of the Cell. 64, 23-28 (1988).
- Juan, G., et al. Histone H3 phosphorylation and expression of cyclins A and B1 measured in individual cells during their progression through G2 and mitosis. Cytometry. 32, 71-77 (1998).
- Langan, T. J., Chou, R. C. Synchronization of mammalian cell cultures by serum deprivation. Methods in Molecular Biology. 761, 75-83 (2011).
- Kues, W. A., et al. Cell cycle synchronization of porcine fetal fibroblasts: effects of serum deprivation and reversible cell cycle inhibitors. Biology of Reproduction. 62, 412-419 (2000).
- Urbani, L., Sherwood, S. W., Schimke, R. T. Dissociation of nuclear and cytoplasmic cell cycle progression by drugs employed in cell synchronization. Experimental Cell Research. 219, 159-168 (1995).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados