É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Acompanhamento Temporal de progressão do ciclo celular utilizando citometria de fluxo, sem a necessidade de sincronização
Neste Artigo
Resumo
Este protocolo descreve o uso de bromodesoxiuridina (BrdU) para permitir a absorção de seguimento temporais de células que estavam na fase S de um determinado ponto no tempo. A adição de corantes de ADN e rotulagem anticorpo facilita a análise detalhada do destino das células em fase S em momentos posteriores.
Resumo
This protocol describes a method to permit the tracking of cells through the cell cycle without requiring the cells to be synchronized. Achieving cell synchronization can be difficult for many cell systems. Standard practice is to block cell cycle progression at a specific stage and then release the accumulated cells producing a wave of cells progressing through the cycle in unison. However, some cell types find this block toxic resulting in abnormal cell cycling, or even mass death. Bromodeoxyuridine (BrdU) uptake can be used to track the cell cycle stage of individual cells. Cells incorporate this synthetic thymidine analog, while synthesizing new DNA during S phase. By providing BrdU for a brief period it is possible to mark a pool of cells that were in S phase while the BrdU was present. These cells can then be tracked through the remainder of the cell cycle and into the next round of replication, permitting the duration of the cell cycle phases to be determined without the need to induce a potentially toxic cell cycle block. It is also possible to determine and correlate the expression of both internal and external proteins during subsequent stages of the cell cycle. These can be used to further refine the assignment of cell cycle stage or assess effects on other cellular functions such as checkpoint activation or cell death.
Introdução
A avaliação das características do ciclo celular e alterações que ocorrem nas células durante a progressão do ciclo celular é fundamental para a compreensão de diversos aspectos da biologia, particularmente biologia do cancro. Muitos agentes em desenvolvimento para o tratamento de doenças malignas têm efeitos profundos sobre a progressão do ciclo celular ou induzir a morte de células através do ciclo celular dependente de mecanismos. A fim de estudar a dinâmica do ciclo celular ou células numa fase específica do ciclo celular, é usual para sincronizar as células. No entanto os métodos de sincronização pode ter efeitos prejudiciais sobre as células a ser estudada, potencialmente confundindo os resultados obtidos. 1 análise Recentemente, a utilização de proteínas marcadas com fluorescência que só estão presentes em fases específicas do ciclo de células têm permitido da progressão do ciclo celular em células individuais ao longo do tempo 2, no entanto as células a serem estudadas necessidade de ser geneticamente manipuladas para expressar estas proteínas marcadas, o que limita a sua utilização para sistemas em que este pode ser lidoily alcançado.
O ciclo celular consiste de duas fases activas: a fase de síntese (s), onde o DNA é replicado e mitose (M), onde ocorre a divisão celular. Estas fases são separadas por três fases de hiato, 0 G, G 1 e G 2. G 0 ou quiescência, é uma fase de repouso, onde a célula tem deixado o ciclo, L é um onde as células aumentam de tamanho antes da replicação de ADN e L 2, onde o crescimento celular continua entre a conclusão da replicação de ADN, mas antes da divisão celular. A progressão através do ciclo celular é controlado por um número de pontos de verificação. O G 1 ponto de verificação é ativada quando as condições ambientais não são favoráveis a síntese de DNA e impede a entrada na fase S. O intra-S fase checkpoint ou atraso pode ser desencadeada por danos no DNA que pode resultar em forquilhas de replicação paralisadas. Durante G 2 a fidelidade do DNA replicado é confirmada e se o dano for detectado, em seguida, o G2 ponto de verificação é ativada permitindo o reparo do DNA antes da divisão celular. Um ponto de verificação final durante a mitose assegura que os cromatídeos foram correctamente alinhado com a placa mitótico modo que a divisão celular pode ser completada com êxito. 3 A activação destes pontos de verificação é normalmente usado para sincronizar as populações de células. Checkpoints do ciclo celular pode ser activada por um número de factores, mas na biologia do cancro a mais comum é a detecção de danos no DNA. A resposta a danos no ADN é iniciada pela PI3-quinase-quinases como ataxia e telangiectasia Rad3 relacionado (ATR) e a ataxia telangiectasia mutada (ATM) que activam as cinases efectoras a jusante Chk1 e Chk2, respectivamente. 3 Uma série de eventos activa Chk1 incluindo parado forquilhas de replicação, ligações cruzadas de DNA e danos da radiação ultravioleta enquanto Chk2 é ativado principalmente por quebras de cadeia dupla.
O método usual para estudar o efeito de condições alteradas no comprimento do ciclo celular is para sincronizar as células numa fase específica do ciclo celular. 1 Isto pode ser alcançado através de vários métodos. As células podem ser fisicamente separadas com base no tamanho, densidade, dispersão lateral (granulosidade), e expressão de marcadores de superfície celular. Mais praticamente, as células podem ser sincronizado por meios químicos. Vários agentes, tais como timidina, hidroxiureia e arabinósido de citosina podem ser utilizados para inibir a síntese de ADN em fase S do ciclo celular, resultando numa acumulação de células em fase S que continuam ciclismo após os agentes são removidos. As células tratadas com nocodazole, o que impede a formação do fuso mitótico, parada com uma L 2 - ou M-fase teor de ADN. Eliminação do soro do meio de cultura resulta na acumulação de células na fase G 0. A re-adição de nutrientes dentro das séricos cultura re-inicia o ciclo normal das células. No entanto, todos esses métodos de sincronização interferir com ciclos normais e o crescimento de células e pode Result em morte celular significativa.
A sincronização das células de leucemia linfoblástica aguda é particularmente difícil e estas células não são passíveis de manipulação genética. O método aqui descrito permite a avaliação da dinâmica do ciclo celular e o estudo de células em fases específicas do ciclo celular, sem sincronização tradicional ou modificação genética. Este método também pode ser útil para outros tipos de células em que a modificação genética e procedimentos de sincronização tradicionais não são prontamente obtidos. O método baseia-se no uso há muito estabelecida de bromodesoxiuridina (BrdU) a incorporação, o que tem muito pouco impacto sobre o crescimento a curto prazo e a proliferação de células. 4 protocolos BrdU Estabelecida tirar vantagem da incorporação de BrdU no ADN sintetizado de novo durante a fase S . Isto marca permanentemente células como estando em fase S durante a exposição BrdU. Esta população pode ser identificado em pontos de tempo mais tardios por coloração para BrdU Incorporção e, assim, agir como uma população sincronizados que podem ser seguidos e avaliados ao longo do tempo permitindo o estudo dos efeitos da droga sobre o trânsito do ciclo celular. BrdU precisa de ser exposto antes da coloração do anticorpo, geralmente atingidos após a ADNase ou tratamento ácido. 6,7 utilizando citometria de fluxo para detectar BrdU incorporada possibilita a inclusão de marcadores adicionais. O mais importante é a utilização de corantes para medir o teor de ADN, permitindo a avaliação da distribuição de fases do ciclo celular das células que estavam na fase S no início do estudo. 8 antigénios de superfície ou intracelulares adicionais Além disso, também pode ser estudada. 9 Estes pode referir-se os eventos do ciclo celular, tais como Ki67 ou características celulares para aparentemente não relacionadas, tais como marcadores de apoptose como a caspase-3 clivada. As aplicações potenciais são limitadas pela imaginação do investigador.
Access restricted. Please log in or start a trial to view this content.
Protocolo
O protocolo aqui descrito utiliza a linha celular de leucemia linfoblástica aguda NALM6 mas pode ser aplicada a outros tipos de células.
1. Soluções e Reagentes
- RPMI completo
- Adicionar 56 ml de soro fetal de vitelo (FCS) e 5,5 ml de 200 mM de L-glutamina a um frasco de 500 ml de meio RPMI-1640.
- BrdU da Solução
- Prepare 32,5 mM de BrdU (10 mg / ml) em fosfato de Dulbecco tamponado com solução salina (DPBS).
- BrdU completo RPMI
- Adicionar 6,2 ml de solução de estoque de BrdU para 10 ml de meio RPMI completo.
- Solução DNase
- Prepare 1 mg de ADNase / ml em DPBS.
- Tampão de Coloração
- Prepare a 3% de FCS inactivado pelo calor e azida de sódio 0,09% em DPBS.
- Consulte a Lista de Materiais para definições de Fixação Buffer, Permeabilização Tampão e tampão de lavagem.
2. Células
NãoTe: As células não foram cultivadas durante mais de 6 meses. Este método é directamente adaptável a qualquer linha de células não-aderentes com os ajustes para a densidade de células e meios de cultura. Use de células que estão a crescer exponencialmente no início da experiência.
- Manter as células NALM6 em T-75 frascos de cultura em RPMI completo. Execute todas as etapas, em condições estéreis, utilizando uma classe II biossegurança gabinete.
- Manter células NALM6 entre 1-2 x 10 6 culas por ml, dividindo a cultura de três vezes por semana.
- Incubar a 37 ° C em 5% de CO2 em ar.
3. Pulso marcação das células com BrdU
CUIDADO: Manusear com cuidado BrdU, pois é um agente mutagênico e teratogênico potencial.
- Centrifugar células a 150 xg durante 5 minutos. Nota: A transferência de células em meio fresco melhora a reprodutibilidade dos resultados.
- Realizar uma contagem de células e as células ressuspender em completo RPMI a 2 x 10 6 cells / ml.
- Dilui-se as células em 1 2 com BrdU RPMI completo produzindo uma concentração celular final de 1 x 10 6 células / ml.
- Incubar a 37 ° C com 5% de CO 2 durante 45 min, depois dilui-se em 1 10 células com meio RPMI completo. Células centrifugar a 150 xg durante 5 min e cuidadosamente descartar todo o sobrenadante.
- Ressuspender as células em um pequeno volume (~ 100 mL) de meio RPMI completo, executar uma contagem das células e ajusta-se 1 x 10 6 células / ml.
- Pipetar 1 mL de células nos poços de uma placa de 48 poços. Pipeta de 1 ml de DPBS em todos os poços desocupadas para obter resultados mais reproduzíveis.
- Incubar a 37 ° C em 5% de CO 2 no ar para timepoints desejados, aqui 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17 , 18, 19, 20, 21, 22, 23 e 24 horas. Nota: O período de tempo vai depender do que o projeto experimental tem como objetivo medir.
- Transferir todas as células em tubos de FACS utilizando uma pipeta. Lavar o bem sequencialmente com 1 ml volumes de PBS para um volume total final de 5 ml.
- Centrifugar a 150 xg durante 5 min e cuidadosamente remover todo o sobrenadante. As células estão prontas para a coloração, executar esta (secção 4) imediatamente.
4. A coloração celular
Nota: Se for necessária a coloração da superfície das células realizá-la antes da fixação, assegurando que as células são mantidas a 4 ° C durante toda.
- Ressuspender as células em 100 ul de tampão de coloração (por coloração da superfície opcional, adicionar o volume recomendado de anticorpo para antigénios de superfície e incuba-se durante 30 min a 4 ° C).
- Adicionar 1 ml de tampão de coloração, centrifugar durante 5 minutos a 150 xg e desprezar o sobrenadante.
Nota: anticorpo específico, a concentração, tempo de incubação, etc variará dependendo objectivos específicos experimentais. - Fixação e permeabilização
- Ressuspender as células em 100 ul de tampão de fixação e incubar durante 15 min à temperatura ambiente.
- Adicionar 1 ml de of tampão de lavagem, centrifuga-se durante 5 min a 150 xg e desprezar o sobrenadante.
- Ressuspender as células em 100 ul de tampão de permeabilização e incubar as células durante 10 minutos em gelo.
- Adicionar 1 ml de tampão de lavagem, centrifugar durante 5 minutos a 150 xg, e desprezar o sobrenadante.
- Ressuspender as células em 100 ul de tampão de fixação por tubo e incubar durante 5 min à temperatura ambiente.
- Adicionar 1 ml de tampão de lavagem, centrifugar durante 5 minutos a 150 xg, e desprezar o sobrenadante.
Nota: O protocolo pode ser pausado aqui se necessário. As células fixadas são estáveis durante vários dias a 4 ° C, se ressuspensa em tampão de coloração. Remover o tampão de coloração após a centrifugação antes de prosseguir.
- O tratamento DNase
- Ressuspender as células em 100 ul de solução de ADNase (30 mg de ADNase / 10 6 células) e incubar as células durante 1 h a 37 ° C.
- Adicionar 1 ml de tampão de lavagem, centrifugar a 150 xg durante 5 minutos e descartar o sobrenadante.
- Coloração de anticorpos
Nota: A coloração para outros marcadores de BrdU intracelulares pode ser realizada em simultâneo com a coloração BrdU.- IMPORTANTE: Prepare os controles de compensação consistem de células não coradas e as células marcadas com cada único fluorocromo. Idealmente, usar os mesmos anticorpos para os controlos de compensação, como os utilizados nos tubos experimentais. No entanto, se isso não for possível, os anticorpos para antigénios substitutos altamente expressos conjugado com o mesmo fluorocromo.
- Ressuspender as células em 50 ul de tampão de lavagem e adicionar 1 mL / 10 6 células de anticorpo BrdU. Nota: pode também ser adicionado directamente conjugado anticorpos para outros antigénios intracelulares específicos.
NOTA:. Os anticorpos para histona H3 fosforilada em Ser10 pode ser utilizado para discriminar entre células em G2 e M, histona H3 é fosforilada em Ser10 durante a mitose 10 Os anticorpos para cdc2 fosforilada em Tyr15 pode ser usado para detectar células que o VHAe compromete-se a mitose. 11 - Incubam-se as células durante 20 minutos à temperatura ambiente.
- Adicionar 1 ml de tampão de lavagem, as células de centrifugação a 150 xg durante 5 minutos e descartar o sobrenadante.
- DNA Stain para análise do ciclo celular
- Soltar sedimento e adicionar 20 uL da solução de 7-AAD (0,25 ug). Nota: É crítico para utilizar uma quantidade constante de 7-AAD / célula.
- Ressuspender as células em 1 ml de tampão de coloração.
5. Recolha de Citometria de Fluxo de Dados
Nota: A máquina necessária vai depender da quantidade e natureza dos fluorocromos utilizados.
- Recolher os seguintes parâmetros: o FSC-A, SSC-A, o FSC-H (FSC-W pode ser utilizado em vez do FSC-H) e 7-AAD fluorescência numa escala linear. Recolhe-se o canal de APC numa escala logarítmica. Colete quaisquer canais adicionais necessários para a avaliação dos rótulos superficiais ou internos utilizando uma escala logarítmica.
- Execute comcompensação de sobreposição de sinais em espectros de emissão observada entre diferentes fluorocromos antes de analisar as amostras. Nota: A maioria dos citómetros de fluxo fará isso automaticamente.
- Recolher pelo menos 10.000 eventos para cada amostra.
6. Análise de citometria de fluxo de dados
Nota: FlowJo foi utilizado neste estudo de citometria de fluxo para a análise de dados, mas outros pacotes de software também pode ser usado. A estratégia de propagação é ilustrado na Figura 1.
- Identificar a população de células viáveis utilizando parâmetros FSC-A e SSC-A.
- Dentro desta população excluir dupletos e agregados utilizando FSC-A e FSC-H (FSC-W também podem ser usados aqui).
- Dentro desta população definir um gráfico de pontos utilizando 7-AAD no eixo dos x e BrdU-APC no eixo dos y.
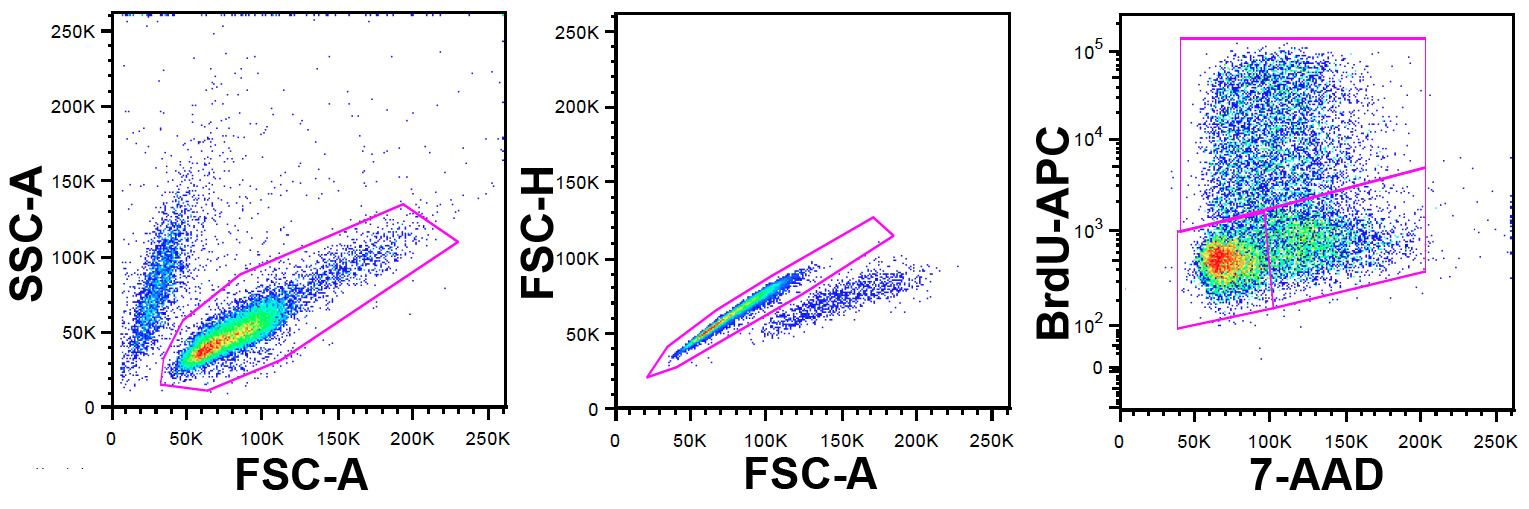
Figura 1: Estratégia Gating Esquerda pa.nel: células ungated são mostrados em um FCS-A vs. SSC-A gráfico de pontos. A população de células viáveis é identificado pela porta mostrado. Centro painel: células fechadas a partir do painel esquerdo são mostrados em uma FSC-A vs. FSC-H gráfico de pontos (FSC-W pode ser usado em vez de altura). Parelhas e agregados são identificados e excluídos pelo portão mostrado. Painel direito: células fechadas a partir da data de exclusão gibão no painel central são mostrados em um 7-AAD vs. APC-A gráfico de pontos. O anticorpo é marcado com BrdU APC permitindo a identificação de células que incorporaram BrdU durante a marcação por pulsos. 7-AAD fornece informações sobre o conteúdo de DNA. O portão superior define células positivas para BrdU e, portanto, na fase S no momento do pulso de BrdU, o portão inferior esquerdo, as células em G 0/1 ea porta inferior direito aqueles em G2 / M. Por favor clique aqui para ver uma versão maior desta figura.
- Um ciclo celular nálise
- Abra o ficheiro de dados e primeira porta nas células do portão exclusão dupleto.
- Analisar essa população para distribuição do ciclo celular (localizado sob plataformas em software Flojo) e usar o modelo de Dean-Jett-Fox.
- Obter as posições do G 0/1 e G 2 / M picos utilizando criar portões.
- Porta nas células BrdU positivas e sujeita essa população a mesma análise do ciclo celular.
- Forneça as posições para o G 0/1 e G 2 / M picos aplicando os mesmos portões de criar portas e definir restrições (usando as portas criadas) para as posições do G 0/1 e G 2 / M picos. Isto é ilustrado nos primeiros dois painéis da Figura 2.
Nota: Outro software também pode ser usado para analisar os dados e as instruções iria variar em conformidade.
840 / 52840fig2highres.jpg "width =" 700 "/>
Figura 2:. Progressão do Ciclo Celular O primeiro painel (todas as células) é fechado na população de células definida pela porta exclusão dupleto. Esta população foi exibida num histograma com 7-AAD no eixo dos X. O pico do pico G 0/1 é indicado pela seta por baixo do eixo. Em subsequentes painéis de células positivas para BrdU foram fechado em, como mostrado na Figura 1. O valor para a posição de 0/1 L obtido quando gating da porta exclusão dupleto é aplicada às células positivas para BrdU fechado dentro FlowJo software do ciclo celular. Cada painel posterior foi fechado sobre a população de BrdU positiva, como mostrado na Figura 1 e a posição do pico G 0/1 com base no valor obtido na análise de toda a população, como mostrado nos primeiros dois painéis. Utilizando a fracção negativa BrdU para identificar a localização da população L 0/1 para as células positivas para BrdU no mesmo SAMPle controla por quaisquer ligeiras diferenças na intensidade da mancha de ADN entre as amostras. O número mostrado em cada painel representa o tempo que o pulso de BrdU terminou. As fases do ciclo celular calculados são mostrados em verde com sombra. Por favor clique aqui para ver uma versão maior desta figura.
Access restricted. Please log in or start a trial to view this content.
Resultados
Esta metodologia pode ser utilizada para obter uma gama de informações. Alguns aplicativos são descritas aqui.
Avaliação da duração do ciclo celular
Para determinar o tempo necessário para que as células de trânsito através do ciclo celular, as células são colhidas em vários pontos de tempo após o pulso de BrdU. Os intervalos entre as avaliações pode ser adaptado para as células particulares a serem analisadas. Linhas de células hematopoiética...
Access restricted. Please log in or start a trial to view this content.
Discussão
A capacidade de analisar o ciclo celular é importante para a compreensão da biologia do cancro e o mecanismo de acção de ambas as drogas e os genes que influenciam a proliferação das células e o crescimento. Embora haja um grande número de ensaios que medem a proliferação celular supostamente, a maioria só fornecem uma medida que indica as células Número presente. Estes incluem ensaios que medem o número de células por visualização direta e contando, atividade metabólica ou concentração de ATP. A pri...
Access restricted. Please log in or start a trial to view this content.
Divulgações
The authors have nothing to disclose.
Agradecimentos
The work was funded by the Leukemia and Lymphoma Society of the USA (6105-08), a Cancer Council NSW grant (13-02), an NHMRC Senior Research Fellowship (LJB) (1042305) and project grant (1041614).
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| APC BrdU Flow Kit | BD Biosciences | 552598 | Contains BrdU antibody, 7-AAD and BD Cytofix/Cytoperm Buffer (referred to as Fixation Buffer) |
| BD Cytoperm Permeabilization Buffer Plus | BD Biosciences | 561651 | Referred to as Permeabilization buffer |
| BD Perm/Wash Buffer | BD Biosciences | 554723 | Referred to as Wash buffer |
| DNase | Sigma | D-4513 | |
| BD Falcon 12 x 75 mm FACS tubes | BD Biosciences | 352008 | |
| BD Pharmingen Stain Buffer | BD Biosciences | 554656 | |
| BD LSR FORTESSA flow cytometer | BD Biosciences | FORTESSA | |
| Pipetman | Gilson | P2, P20, P100, P1000 | |
| RPMI 1,640 w/o L-Gln 500 ml | Lonza | 12-167F | |
| DPBS | Lonza | 17-512F | |
| Fetal Bovine Serum | FisherBiotec | FBS-7100113 | |
| L-Glutamine | Sigma | G7513-100ML | |
| 5-Bromo-2′-deoxyuridine | Sigma | B5002-1G | |
| Falcon TC 150 cm2 vented Flasks | BD Biosciences | 355001 | |
| Pipettes 25 ml | Greiner | 760180 | |
| Aersol Pipettes 200 µl | Interpath | 24700 | |
| Aersol Pipettes 1 ml | Interpath | 24800 | |
| Centrifuge | Spintron | GT-175R | |
| CO2 incubator | Binder | C 150 | |
| AF488 anti-Histone H3 Phospho (Ser10) Antibody | Cell Signalling | 9708S | |
| Phospho-Chk2 (Thr68) (C13C1) Rabbit mAb | Cell Signalling | 2197S | |
| Phospho-Chk1 (Ser345) (133D3) Rabbit mAb | Cell Signalling | 2348S | |
| NALM6 | DSMZ | ACC-128 |
Referências
- Banfalvi, G. Methods Mol Biol. Banfalvi, G. 761, Humana Press. New York, Dordrecht, Heidelberg, London. 1-23 (2011).
- Sakaue-Sawano, A., et al. Visualizing spatiotemporal dynamics of multicellular cell-cycle progression. Cell. 132, 487-498 (2008).
- Harper, J. W., Elledge, S. J. The DNA damage response: ten years after. Molecular Cell. 28, 739-745 (2007).
- Latt, S. A., George, Y. S., Gray, J. W. Flow cytometric analysis of bromodeoxyuridine-substituted cells stained with 33258 Hoechst. The Journal of Histochemistry and Cytochemistry. 25, 927-934 (1977).
- Gratzner, H. G. Monoclonal antibody to 5-bromo- and 5-iododeoxyuridine: A new reagent for detection of DNA replication. Science. 218, 474-475 (1982).
- Carayon, P., Bord, A. Identification of DNA-replicating lymphocyte subsets using a new method to label the bromo-deoxyuridine incorporated into the DNA. Journal of Immunological Methods. 147, 225-230 (1992).
- Gonchoroff, N. J., et al. S-phase detection with an antibody to bromodeoxyuridine. Role of DNase pretreatment. Journal of Immunological Methods. 93, 97-101 (1986).
- Rabinovitch, P. S., Torres, R. M., Engel, D. Simultaneous cell cycle analysis and two-color surface immunofluorescence using 7-amino-actinomycin D and single laser excitation: applications to study of cell activation and the cell cycle of murine Ly-1 B cells. Journal of Immunology. 136, 2769-2775 (1986).
- Saunders, P. O., et al. RAD001 (everolimus) induces dose-dependent changes to cell cycle regulation and modifies the cell cycle response to vincristine. Oncogene. 32, 4789-4797 (2013).
- Hendzel, M. J., et al. Mitosis-specific phosphorylation of histone H3 initiates primarily within pericentromeric heterochromatin during G2 and spreads in an ordered fashion coincident with mitotic chromosome condensation. Chromosoma. 106, 348-360 (1997).
- Draetta, G., Beach, D. Activation of cdc2 protein kinase during mitosis in human cells: cell cycle-dependent phosphorylation and subunit rearrangement. Cell. 54, 17-26 (1988).
- Saunders, P. O., et al. RAD001 (everolimus) induces dose-dependent changes to cell cycle regulation and modifies the cell cycle response to vincristine. Oncogene. , (2012).
- Knudsen, R. C., Ahmed, A. A., Sell, K. W. An in vitro microassay for lymphotoxin using microculture plates and the multiple automated sample harvester. Journal of Immunological Methods. 5, 55-63 (1974).
- Beck, H. P. Proliferation kinetics of perturbed cell populations determined by the bromodeoxyuridine-33258 technique: radiotoxic effects of incorporated [3H]thymidine. Cytometry. 2, 170-174 (1981).
- Miller, M. J., Wei, S. H., Parker, I., Cahalan, M. D. Two-photon imaging of lymphocyte motility and antigen response in intact lymph node. Science. 296, 1869-1873 (2002).
- Collins, J. M., Berry, D. E., Bagwell, C. B. Different rates of DNA synthesis during the S phase of log phase HeLa S3, WI-38, and 2RA cells. Journal of Biological Chemistry. 255, 3585-3590 (1980).
- Schwarting, R., Gerdes, J., Niehus, J., Jaeschke, L., Stein, H. Determination of the growth fraction in cell suspensions by flow cytometry using the monoclonal antibody Ki-67. Journal of Immunological Methods. 90, 65-70 (1986).
- Danova, M., et al. Cell cycle-related proteins: a flow cytofluorometric study in human tumors. Biology of the Cell. 64, 23-28 (1988).
- Juan, G., et al. Histone H3 phosphorylation and expression of cyclins A and B1 measured in individual cells during their progression through G2 and mitosis. Cytometry. 32, 71-77 (1998).
- Langan, T. J., Chou, R. C. Synchronization of mammalian cell cultures by serum deprivation. Methods in Molecular Biology. 761, 75-83 (2011).
- Kues, W. A., et al. Cell cycle synchronization of porcine fetal fibroblasts: effects of serum deprivation and reversible cell cycle inhibitors. Biology of Reproduction. 62, 412-419 (2000).
- Urbani, L., Sherwood, S. W., Schimke, R. T. Dissociation of nuclear and cytoplasmic cell cycle progression by drugs employed in cell synchronization. Experimental Cell Research. 219, 159-168 (1995).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados