Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Zeitliche Verfolgung von Zellzyklus mittels Durchflusszytometrie, ohne die Notwendigkeit für die Synchronisation
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt die Verwendung von Bromdesoxyuridin (BrdU) -Aufnahme, die zeitliche Verfolgung von Zellen, die in der S-Phase auf einen bestimmten Zeitpunkt waren ermöglichen. Die Zugabe von DNA-Farbstoffen und Antikörpermarkierung erleichtert die detaillierte Analyse des Schicksals der S-Phasen-Zellen zu späteren Zeitpunkten.
Zusammenfassung
This protocol describes a method to permit the tracking of cells through the cell cycle without requiring the cells to be synchronized. Achieving cell synchronization can be difficult for many cell systems. Standard practice is to block cell cycle progression at a specific stage and then release the accumulated cells producing a wave of cells progressing through the cycle in unison. However, some cell types find this block toxic resulting in abnormal cell cycling, or even mass death. Bromodeoxyuridine (BrdU) uptake can be used to track the cell cycle stage of individual cells. Cells incorporate this synthetic thymidine analog, while synthesizing new DNA during S phase. By providing BrdU for a brief period it is possible to mark a pool of cells that were in S phase while the BrdU was present. These cells can then be tracked through the remainder of the cell cycle and into the next round of replication, permitting the duration of the cell cycle phases to be determined without the need to induce a potentially toxic cell cycle block. It is also possible to determine and correlate the expression of both internal and external proteins during subsequent stages of the cell cycle. These can be used to further refine the assignment of cell cycle stage or assess effects on other cellular functions such as checkpoint activation or cell death.
Einleitung
Die Beurteilung der Zellzyklus-Funktionen und Änderungen, die in den Zellen auftreten, während der Zellzyklusprogression ist grundlegend für das Verständnis vieler Aspekte der Biologie, insbesondere der Krebsbiologie. Viele Agenten in Entwicklung für die Behandlung von malignen Erkrankungen haben tief greifende Auswirkungen auf die Zellzyklusprogression oder Zelltod durch Zellzyklus abhängige-Mechanismen. Um Zellzyklus Dynamik oder Zellen in einer bestimmten Phase des Zellzyklus zu untersuchen, ist es üblich, Zellen zu synchronisieren. Synchronisationsverfahren kann jedoch nachteilige Auswirkungen auf die Zellen untersucht, die möglicherweise verwirrende die erhaltenen Ergebnisse. 1 Kürzlich wurde die Verwendung von fluoreszenzmarkierten Proteinen, die nur in bestimmten Phasen des Zyklus Zellen vorliegen erlaubt hätte Analyse der Zellzyklus-Progression in einzelnen Zellen über die Zeit 2, jedoch sind die Zellen, die untersucht werden müssen, um die genetisch manipuliert werden können, um diese markierten Proteine exprimieren, was ihre Anwendung auf Systeme, in denen diese gelesen werden könnenily erreicht.
Der Zellzyklus besteht aus zwei aktiven Phasen: die Synthese (S) Phase, in der DNA repliziert und Mitose (M), wo die Zellteilung erfolgt. Diese Phasen werden durch drei Spaltphasen, G 0, G 1 und G 2 voneinander getrennt. G 0 oder Ruhe ist eine Ruhephase, in der die Zelle den Zyklus verlassen, G 1, wo die Zellen an Größe vor der DNA-Replikation und G 2, wo das Zellwachstum weiterhin zwischen Beendigung der DNA-Replikation, jedoch vor der Zellteilung. Die Progression durch den Zellzyklus wird durch eine Reihe von Kontrollpunkten gesteuert. Der G1 Kontrollpunkt wird aktiviert, wenn die Umweltbedingungen nicht unterstützend der DNA-Synthese und verhindert den Eintritt in die S-Phase. Die intra-S-Phasen-Kontrollpunkt oder Verzögerung kann durch DNA-Schädigung, die in ins Stocken geraten Replikationsgabeln führen kann ausgelöst werden. Während G 2 die Wiedergabetreue des replizierten DNA bestätigt wird und wenn ein Schaden wird dann detektiert die G 2 Checkpoint aktiviert ermöglicht DNA-Reparatur vor der Zellteilung. Eine endgültige Prüfpunkt während der Mitose sichergestellt, dass Chromatiden wurden korrekt an der mitotischen Platte ausgerichtet, so dass die Zellteilung erfolgreich abgeschlossen werden kann. 3 Aktivierung dieser Kontroll wird häufig verwendet, um Zellpopulationen zu synchronisieren. Zellzyklus-Kontrollpunkte können von einer Reihe von Faktoren ab, sondern in der Krebsbiologie die häufigste ist die Detektion von DNA-Schäden aktiviert wird. Die DNA-Schadensantwort wird durch die PI3-Kinase-like Kinasen Ataxie Teleangiektasien und Rad3 bezogenen (ATR) initiiert und Ataxie Teleangiektasie mutiert (ATM), die den nachgeschalteten Effektor-Kinasen Chk1 und Chk2 aktivieren sind. 3 Eine Reihe von Ereignissen aktiviert Chk1 einschließlich, ins Stocken geraten Replikationsgabeln, DNA-Vernetzungen und UV-Strahlung Schäden während Chk2 wird vor allem von Doppelstrangbrüchen aktiviert.
Das übliche Verfahren für die Untersuchung der Wirkung der veränderten Bedingungen auf die Länge des Zellzyklus is, um die Zellen zu synchronisieren in einer bestimmten Phase des Zellzyklus. 1 Dieses kann über verschiedene Verfahren erreicht werden. Die Zellen können physikalisch getrennt basieren auf Größe, Dichte, Seitenstreuung (Granularität) und Zelloberflächenexpression Marker. Praktischer ausgedrückt können Zellen durch chemische Mittel synchronisiert werden. Mehrere Mittel, wie Thymidin, Hydroxyharnstoff und Cytosinarabinosid kann zur DNA-Synthese in der S-Phase des Zellzyklus, was zu einer Anhäufung von Zellen in S-Phase, die auch nach cycling die Mittel entfernt zu hemmen. Oder M-Phase der DNA-Gehalt - Zellen mit Nocodazol, die die Bildung der mitotischen Spindel, Festnahme mit einem G 2 verhindert, behandelt. Beseitigung von Serum aus dem Kulturmedium führt zur Akkumulation von Zellen in G 0 -Phase. Die erneute Zugabe der Nährstoffe in der Kultur Serum führt zum Start des normalen Fahrradfahren der Zellen. Jedoch sind alle diese Synchronisationsverfahren mit normalen Radfahren und Wachstum der Zellen stören und Result in signifikanten Zelltod.
Synchronisation der akuten lymphoblastischen Leukämiezellen, ist eine besondere Herausforderung und diese Zellen nicht zugänglich sind Genmanipulation. Das hier beschriebene Verfahren ermöglicht die Beurteilung von Zellzyklusdynamik und die Untersuchung von Zellen, die in bestimmten Phasen des Zellzyklus ohne traditionelle Synchronisation oder genetischen Veränderung. Dieses Verfahren kann auch für andere Zelltypen, bei denen genetische Veränderung und traditionelle Synchronisationsverfahren nicht leicht erreicht werden. Die Methode basiert auf der seit langem etablierten Einsatz von Bromdesoxyuridin (BrdU) Einbau, die sehr wenig Einfluss auf die kurzfristige Wachstum und die Vermehrung von Zellen hat basierend. 4 Gegründet BrdU-Protokolle nutzen den Einbau von BrdU in die neu synthetisierte DNA während der S-Phase . Dies markiert dauerhaft Zellen mit BrdU während der Belichtung in der S-Phase gewesen. Diese Population kann zu späteren Zeitpunkten durch Färbung auf BrdU incorpor identifiziert werdenation und damit zu handeln als Synchron Bevölkerung, die folgten und mit der Zeit zu beurteilen erlaubt die Untersuchung von Arzneimittelwirkungen auf die Zellzyklustransit werden kann. BrdU muss vor der Antikörperfärbung ausgesetzt sind, werden in der Regel folgende DNase oder Säurebehandlung erreicht. 6,7 mittels Durchflusszytometrie zu erkennen eingebautem BrdU ermöglicht die Aufnahme von zusätzlichen Markern. Die wichtigste ist die Verwendung von Farbstoffen auf den DNA-Gehalt zu messen, so dass die Beurteilung der Zellzyklusphasenverteilung der Zellen, in die S-Phase zu Beginn der Studie waren. 8 ferner zusätzliche Oberfläche oder intrazelluläre Antigene können ebenfalls untersucht werden. 9 Dies kann zu Zellzyklusereignisse wie Ki67 beziehen oder auf scheinbar unabhängige Zellfunktionen wie Apoptose-Marker wie gespalten Caspase-3. Die Einsatzmöglichkeiten sind durch die Phantasie des Forschers begrenzt.
Access restricted. Please log in or start a trial to view this content.
Protokoll
Das hier beschriebene Protokoll verwendet die akute lymphatische Leukämie-Zelllinie NALM6 sondern kann auch auf andere Zelltypen angewendet werden.
1. Lösungen und Reagenzien
- Komplettes RPMI
- Hinzuzufügen 56 ml fötales Kälberserum (FCS) und 5,5 ml von 200 mM L-Glutamin in einer 500 ml Flasche mit RPMI-1640-Medium.
- BrdU-Stammlösung
- Vorbereitung 32,5 mM BrdU (10 mg / ml) in Dulbeccos phosphatgepufferter Salzlösung (DPBS).
- BrdU komplettem RPMI
- In 6,2 ul der BrdU-Stammlösung zu 10 ml vollständigem RPMI.
- DNase-Lösung
- Bereiten Sie 1 mg DNase / ml in DPBS.
- Färbepuffer
- Vorbereitung 3% hitzeinaktiviertem FCS und 0,09% Natriumazid in DPBS.
- Siehe Materialliste für Definitionen der Fixation Buffer, Permeabilisierung Puffer und Waschpuffer.
2. Zellen
Neinte: Die Zellen waren nicht für mehr als 6 Monate kultiviert. Dieses Verfahren ist direkt anwendbar zu jeder nicht-adhärenten Zelllinie mit Anpassungen der Zelldichte und Kulturmedien. Zellen einzusetzen, die exponentiell zu Beginn des Experiments wachsen.
- Pflegen NALM6 Zellen in T-75 Kulturflaschen in komplettem RPMI. Führen Sie alle Schritte unter sterilen Bedingungen mit einem Klasse II Biosicherheitswerkbank.
- Aufrechtzuerhalten NALM6 Zellen zwischen 1-2 x 10 6 Zellen pro ml durch Aufteilung der Kultur dreimal wöchentlich.
- Inkubieren bei 37 ° C in 5% CO 2 in Luft.
3. Pulse Markierung von Zellen mit BrdU
VORSICHT: Gehen Sie BrdU mit Sorgfalt, da es eine potentielle mutagen und teratogen.
- Zentrifuge Zellen bei 150 g für 5 min. Hinweis: Übertragen von Zellen in frischem Medium verbessert die Reproduzierbarkeit der Ergebnisse.
- Durchführen einer Zellzahl und resuspendieren Zellen in komplettem RPMI mit 2 × 10 6 cEllen / ml.
- Verdünnte Zellen 1 in 2 mit BrdU komplettem RPMI Herstellung einer endgültigen Zellkonzentration von 1 x 10 6 Zellen / ml.
- Inkubieren bei 37 ° C mit 5% CO 2 für 45 min, dann verdünnt 1 in 10 Zellen mit komplettem RPMI. Zentrifuge Zellen bei 150 g für 5 min und sorgfältig berücksichtigen keines der überstehenden Flüssigkeit.
- Die Zellen in einem kleinen Volumen (~ 100 & mgr; l) von kompletten RPMI Führen einer Zellzahl und anpassen, um 1 x 10 6 Zellen / ml.
- Pipette 1 ml der Zellen in die Vertiefungen einer 48 Well-Platte. Pipette 1 ml DPBS in jede unbesetzte Brunnen, mehr reproduzierbare Ergebnisse zu erhalten.
- Inkubieren bei 37 ° C in 5% CO 2 in Luft für die gewünschten Zeitpunkte, hier 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17 , 18, 19, 20, 21, 22, 23 und 24 Stunden. Anmerkung: Die Länge der Zeit ab, was die Versuchsanordnung zielt darauf ab, zu messen abhängen.
- Übertragen Sie alle Zellen in FACS-Röhrchen mit einer Pipette. Die auch nacheinander mit 1 ml volu Spülenmes von PBS zu einem endgültigen Gesamtvolumen von 5 ml.
- Zentrifuge bei 150 xg für 5 min und entfernen Sie vorsichtig alle den Überstand. Zellen sind bereit für die Färbung, führen Sie diese (Abschnitt 4) sofort.
4. Zellfärbung
Anmerkung: Wenn eine Oberflächenfärbung von Zellen erforderlich zuführen es vor der Fixierung, so dass die Zellen bei 4 ° C durchgehend beibehalten.
- Die Zellen in 100 ul Färbepuffer (für optionale Oberflächenfärbung, fügen Sie die empfohlenen Volumen von Antikörper gegen Oberflächenantigene und Inkubation für 30 min bei 4 ° C).
- 1 ml der Färbepuffer, Zentrifuge für 5 Minuten bei 150 xg und den Überstand verwerfen.
Hinweis: Spezifische Antikörper, Konzentration, Inkubationszeit usw. in Abhängigkeit von bestimmten experimentellen Ziele variieren. - Fixierung und Permeabilisierung
- Die Zellen in 100 ul Puffer Fixierung und Inkubation für 15 min bei Raumtemperatur.
- 1 ml of Waschpuffer, Zentrifuge für 5 Minuten bei 150 xg und den Überstand verwerfen.
- Die Zellen in 100 ul Permeabilisierung Puffer und Inkubation der Zellen für 10 Minuten auf Eis.
- 1 ml Waschpuffer, Zentrifuge für 5 Minuten bei 150 · g, und den Überstand verwerfen.
- Die Zellen in 100 ul Puffer pro Röhrchen Fixierung und Inkubation für 5 min bei Raumtemperatur.
- 1 ml Waschpuffer, Zentrifuge für 5 Minuten bei 150 · g, und den Überstand verwerfen.
Hinweis: Das Protokoll kann hier bei Bedarf angehalten werden. Die fixierten Zellen werden bei 4 ° C mehrere Tage lang stabil, wenn sie in Färbepuffer resuspendiert. Entfernen Sie die Färbepuffer nach der Zentrifugation, bevor Sie fortfahren.
- DNase-Behandlung
- Die Zellen in 100 ul DNase-Lösung (30 ug DNase / 10 6 Zellen) und inkubieren Zellen für 1 h bei 37 ° C.
- 1 ml Waschpuffer, Zentrifuge bei 150 xg für 5 min und Überstand verwerfen.
- Antikörper-Färbung
Anmerkung: Die Färbung für andere als BrdU intrazellulären Marker können gleichzeitig mit der BrdU-Färbung durchgeführt werden.- WICHTIG: Bereiten Kompensationskontrollen, bestehend aus ungefärbten Zellen und Zellen, die mit jedem einzelnen Fluorochrom markiert. Idealerweise verwenden die gleichen Antikörper für die Entschädigung Kontrollen, wie sie in der experimentellen Röhren verwendet. Wenn dies jedoch nicht möglich ist, ersatz Antikörper stark exprimierten Antigene auf den gleichen Fluorochrom konjugiert ist.
- Die Zellen in 50 ul Waschpuffer und fügen 1 ul / 10 6 Zellen von BrdU-Antikörpers. Anmerkung: direkt konjugierten Antikörpern gegen andere spezifische intrazelluläre Antigene können ebenfalls zugegeben werden.
HINWEIS:. Antikörper gegen Histon H3 an Ser10 phosphoryliert kann zwischen den Zellen in der G2 und M zu unterscheiden ist phosphoryliert Histon H3 an Ser10 während der Mitose 10 Antikörper gegen cdc2 phosphoryliert am Tyr15 verwendet, um Zellen zu detektieren have Mitose verpflichtet. 11 - Inkubieren der Zellen für 20 Minuten bei Raumtemperatur.
- 1 ml Waschpuffer, Zentrifuge Zellen bei 150 xg für 5 min und Überstand verwerfen.
- Stain DNA für Zellzyklusanalyse
- Lösen Pellet und fügen Sie 20 ul der 7-AAD-Lösung (0,25 ug). Anmerkung: Es ist wichtig, eine konstante Menge von 7-AAD / Zelle zu verwenden.
- Resuspendieren der Zellen in 1 ml Färbepuffer.
5. Sammlung von Durchflusszytometrie Daten
Der benötigte hängt von der Anzahl und Art der verwendeten Fluorochromen abhängen Maschine.
- Sammeln Sie die folgenden Parameter: FSC-A, SSC-A, FSC-H und 7-AAD-Fluoreszenz auf einer linearen Skala (FSC-W kann anstelle von FSC-H verwendet werden). Sammeln Sie die APC-Kanal auf einer logarithmischen Skala. Sammeln Sie alle zusätzlichen Kanäle für die Beurteilung der Oberfläche und im Inneren von Etiketten mit einer logarithmischen Skala erforderlich.
- Führen comKompensation von überlappenden Signalen in vor der Analyse der Proben zwischen verschiedenen Fluorochromen Emissionsspektren beobachtet. Hinweis: Die meisten Durchflusszytometer wird dies automatisch durchführen.
- Sammeln Sie mindestens 10.000 Ereignisse für jede Probe.
6. Analyse der Durchflusszytometrie Daten
Anmerkung: FlowJo wurde in dieser Studie für die Strömung verwendet Zytometrie Datenanalyse, sondern auch andere Softwarepakete können ebenfalls verwendet werden. Die Gating-Strategie ist in Figur 1 veranschaulicht.
- Identifizieren Sie die Lebendzellpopulation mit FSC-A und SSC-A-Parameter.
- Innerhalb dieser Population auszuschließen Dubletten und Aggregate mit FSC-A und FSC-H (FSC-W kann natürlich auch verwendet werden).
- Innerhalb dieser Population eingestellt ein Punkt-Diagramm mit 7-AAD auf der x-Achse und BrdU-APC auf der y-Achse.
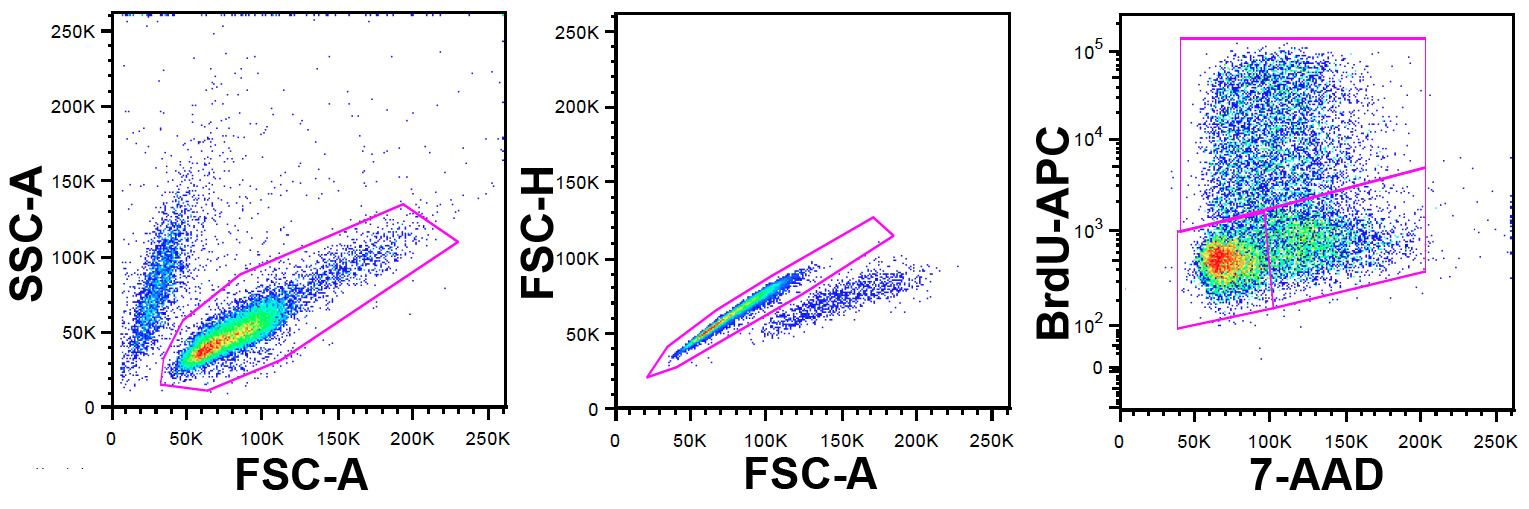
Abbildung 1: Gating-Strategie Left pa.nel: ungated Zellen auf einem FCS-A vs. SSC-ein Punkt-Diagramm dargestellt. Die lebensfähige Zellpopulation wird durch die gezeigte Gate identifiziert. Mitteltafel: Zellen aus dem linken Fenster gated auf einer FSC-A vs. FSC-H-Punktediagramm (FSC-W kann anstelle der Höhe verwendet werden) gezeigt. Dubletts und Aggregate werden identifiziert und durch die gezeigte Gate ausgeschlossen. Rechts: Zellen aus dem Wams Ausschlussdatum in der Mittelplatte gated sind auf einem 7-AAD vs. APC-ein Punkt-Diagramm dargestellt. Die BrdU-Antikörper wird mit APC, die die Identifizierung von Zellen, die BrdU während des Puls-Markierung inkorporiert haben markiert. 7-AAD bietet Informationen über die DNA-Gehalt. Die obere Gate definiert Zellen positiv für BrdU und damit in die S-Phase zu der Zeit des BrdU Puls, der unteren linken Tor, Zellen in G 0/1 und der rechten unteren Gate jene in G 2 / M. Bitte klicken Sie hier, um einen Blick Größere Version der Figur.
- Cell Cycle A nalyse
- Öffnen der ersten Datendatei und Gate auf die Zellen in dem Dublett Ausschluss Gate.
- Analysieren Sie diese Bevölkerung für Zellzyklus-Verteilung (unter Plattformen in FloJo Software befindet) und verwenden Sie die Dean-Jett-Fox-Modell.
- Besorgen Sie sich die Positionen der G 0/1 und G 2 / M mit Spitzen erstellen Tore.
- Tor auf der BrdU-positiven Zellen und unterliegen diese Bevölkerung zu gleichen Zellzyklusanalyse.
- Stellen die Positionen der G 0/1 und G 2 / M-Spitzen, die durch die Anwendung der gleichen Toren aus schaffen Gatter und Einstellen Bedingungen (mit den Gates angelegt) für die Positionen der G 0/1 und G 2 / M Spitzen. Dies wird in den ersten 2 Platten der Fig. 2 dargestellt
Hinweis: andere Software kann auch verwendet werden, um die Daten zu analysieren, und die Anweisungen würden entsprechend variieren.
840 / 52840fig2highres.jpg "width =" 700 "/>
Fig. 2: Zellzyklusprogression Die erste Platte (alle Zellen) auf der Zellpopulation durch das Dublett Ausschluss Gate definiert sucht. Diese Population wurde in einem Histogramm auf der X-Achse 7-AAD angezeigt. Der Peak der G 0/1 Peak durch den Pfeil unter der Achse angegeben. In den Folgeplatten BrdU-positiven Zellen wurden auf gated wie in Abbildung 1 dargestellt. Der Wert für das G 0/1 Position erreicht, wenn Gating des Dubletts Ausschluss Gate mit der BrdU positive Zellen in gated FlowJo Zellzyklus-Software angewendet. Jeder nachfolgende Tafel wurde auf der BrdU-positiven Population gated wie in Figur 1 und der Position der G 0/1 Peak, basierend auf der erhaltenen bei der Analyse der Gesamtbevölkerung in den ersten zwei Platten gezeigt Wert dargestellt. Verwendung des BrdU negative Fraktion, um die Position des G 0/1 Population für die BrdU-positiven Zellen in demselben samp identifizierenle steuert für leichte Unterschiede in der Intensität der DNA-Färbung zwischen den Proben. Die Zahl auf jeder Tafel gezeigt ist, stellt die Zeit seit dem BrdU puls beendet. Die berechneten Zellzyklus-Phasen werden in schattierten grün dargestellt. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Diese Methode kann verwendet werden, um eine Reihe von Informationen zu erhalten. Einige Anwendungen sind hier aufgeführt.
Ermittlung der Dauer des Zellzyklus
Die Zeit für die Zellen durch den Zellzyklus erforderlich ist, um Durchgangs bestimmen, werden die Zellen zu verschiedenen Zeitpunkten nach der BrdU puls geerntet. Die Intervalle zwischen den Bewertungen können auf die jeweiligen Zellen analysiert angepasst werden. Hämatopoetischen Zelllinien wurden st?...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Die Fähigkeit, den Zellzyklus zu analysieren ist wichtig für das Verständnis der Krebsbiologie und den Wirkmechanismus beider Medikamente und Gene, die die Zellproliferation und das Wachstum beeinflussen. Zwar gibt es eine Vielzahl von Assays, welche angeblich die Zellproliferation zu messen, wird nur die Mehrheit ein Maß, daß die Anzahl Zellen vorhanden angibt. Dazu gehören Tests, die Zellzahl durch direkte Visualisierung und Zählen, Stoffwechselaktivität oder ATP-Konzentration zu messen. Der Hauptvorteil bei v...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
The authors have nothing to disclose.
Danksagungen
The work was funded by the Leukemia and Lymphoma Society of the USA (6105-08), a Cancer Council NSW grant (13-02), an NHMRC Senior Research Fellowship (LJB) (1042305) and project grant (1041614).
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| APC BrdU Flow Kit | BD Biosciences | 552598 | Contains BrdU antibody, 7-AAD and BD Cytofix/Cytoperm Buffer (referred to as Fixation Buffer) |
| BD Cytoperm Permeabilization Buffer Plus | BD Biosciences | 561651 | Referred to as Permeabilization buffer |
| BD Perm/Wash Buffer | BD Biosciences | 554723 | Referred to as Wash buffer |
| DNase | Sigma | D-4513 | |
| BD Falcon 12 x 75 mm FACS tubes | BD Biosciences | 352008 | |
| BD Pharmingen Stain Buffer | BD Biosciences | 554656 | |
| BD LSR FORTESSA flow cytometer | BD Biosciences | FORTESSA | |
| Pipetman | Gilson | P2, P20, P100, P1000 | |
| RPMI 1,640 w/o L-Gln 500 ml | Lonza | 12-167F | |
| DPBS | Lonza | 17-512F | |
| Fetal Bovine Serum | FisherBiotec | FBS-7100113 | |
| L-Glutamine | Sigma | G7513-100ML | |
| 5-Bromo-2′-deoxyuridine | Sigma | B5002-1G | |
| Falcon TC 150 cm2 vented Flasks | BD Biosciences | 355001 | |
| Pipettes 25 ml | Greiner | 760180 | |
| Aersol Pipettes 200 µl | Interpath | 24700 | |
| Aersol Pipettes 1 ml | Interpath | 24800 | |
| Centrifuge | Spintron | GT-175R | |
| CO2 incubator | Binder | C 150 | |
| AF488 anti-Histone H3 Phospho (Ser10) Antibody | Cell Signalling | 9708S | |
| Phospho-Chk2 (Thr68) (C13C1) Rabbit mAb | Cell Signalling | 2197S | |
| Phospho-Chk1 (Ser345) (133D3) Rabbit mAb | Cell Signalling | 2348S | |
| NALM6 | DSMZ | ACC-128 |
Referenzen
- Banfalvi, G. Methods Mol Biol. Banfalvi, G. 761, Humana Press. New York, Dordrecht, Heidelberg, London. 1-23 (2011).
- Sakaue-Sawano, A., et al. Visualizing spatiotemporal dynamics of multicellular cell-cycle progression. Cell. 132, 487-498 (2008).
- Harper, J. W., Elledge, S. J. The DNA damage response: ten years after. Molecular Cell. 28, 739-745 (2007).
- Latt, S. A., George, Y. S., Gray, J. W. Flow cytometric analysis of bromodeoxyuridine-substituted cells stained with 33258 Hoechst. The Journal of Histochemistry and Cytochemistry. 25, 927-934 (1977).
- Gratzner, H. G. Monoclonal antibody to 5-bromo- and 5-iododeoxyuridine: A new reagent for detection of DNA replication. Science. 218, 474-475 (1982).
- Carayon, P., Bord, A. Identification of DNA-replicating lymphocyte subsets using a new method to label the bromo-deoxyuridine incorporated into the DNA. Journal of Immunological Methods. 147, 225-230 (1992).
- Gonchoroff, N. J., et al. S-phase detection with an antibody to bromodeoxyuridine. Role of DNase pretreatment. Journal of Immunological Methods. 93, 97-101 (1986).
- Rabinovitch, P. S., Torres, R. M., Engel, D. Simultaneous cell cycle analysis and two-color surface immunofluorescence using 7-amino-actinomycin D and single laser excitation: applications to study of cell activation and the cell cycle of murine Ly-1 B cells. Journal of Immunology. 136, 2769-2775 (1986).
- Saunders, P. O., et al. RAD001 (everolimus) induces dose-dependent changes to cell cycle regulation and modifies the cell cycle response to vincristine. Oncogene. 32, 4789-4797 (2013).
- Hendzel, M. J., et al. Mitosis-specific phosphorylation of histone H3 initiates primarily within pericentromeric heterochromatin during G2 and spreads in an ordered fashion coincident with mitotic chromosome condensation. Chromosoma. 106, 348-360 (1997).
- Draetta, G., Beach, D. Activation of cdc2 protein kinase during mitosis in human cells: cell cycle-dependent phosphorylation and subunit rearrangement. Cell. 54, 17-26 (1988).
- Saunders, P. O., et al. RAD001 (everolimus) induces dose-dependent changes to cell cycle regulation and modifies the cell cycle response to vincristine. Oncogene. , (2012).
- Knudsen, R. C., Ahmed, A. A., Sell, K. W. An in vitro microassay for lymphotoxin using microculture plates and the multiple automated sample harvester. Journal of Immunological Methods. 5, 55-63 (1974).
- Beck, H. P. Proliferation kinetics of perturbed cell populations determined by the bromodeoxyuridine-33258 technique: radiotoxic effects of incorporated [3H]thymidine. Cytometry. 2, 170-174 (1981).
- Miller, M. J., Wei, S. H., Parker, I., Cahalan, M. D. Two-photon imaging of lymphocyte motility and antigen response in intact lymph node. Science. 296, 1869-1873 (2002).
- Collins, J. M., Berry, D. E., Bagwell, C. B. Different rates of DNA synthesis during the S phase of log phase HeLa S3, WI-38, and 2RA cells. Journal of Biological Chemistry. 255, 3585-3590 (1980).
- Schwarting, R., Gerdes, J., Niehus, J., Jaeschke, L., Stein, H. Determination of the growth fraction in cell suspensions by flow cytometry using the monoclonal antibody Ki-67. Journal of Immunological Methods. 90, 65-70 (1986).
- Danova, M., et al. Cell cycle-related proteins: a flow cytofluorometric study in human tumors. Biology of the Cell. 64, 23-28 (1988).
- Juan, G., et al. Histone H3 phosphorylation and expression of cyclins A and B1 measured in individual cells during their progression through G2 and mitosis. Cytometry. 32, 71-77 (1998).
- Langan, T. J., Chou, R. C. Synchronization of mammalian cell cultures by serum deprivation. Methods in Molecular Biology. 761, 75-83 (2011).
- Kues, W. A., et al. Cell cycle synchronization of porcine fetal fibroblasts: effects of serum deprivation and reversible cell cycle inhibitors. Biology of Reproduction. 62, 412-419 (2000).
- Urbani, L., Sherwood, S. W., Schimke, R. T. Dissociation of nuclear and cytoplasmic cell cycle progression by drugs employed in cell synchronization. Experimental Cell Research. 219, 159-168 (1995).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten