Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Suivi temporel de la progression du cycle cellulaire par cytométrie en flux, sans la nécessité pour la synchronisation
Dans cet article
Résumé
Ce protocole décrit l'utilisation de bromodésoxyuridine (BrdU) absorption pour permettre le suivi temporel des cellules qui étaient en phase S à un moment précis dans le temps. Addition de colorants d'ADN et de l'étiquetage d'anticorps facilite l'analyse détaillée du sort des cellules en phase S au temps plus tard.
Résumé
This protocol describes a method to permit the tracking of cells through the cell cycle without requiring the cells to be synchronized. Achieving cell synchronization can be difficult for many cell systems. Standard practice is to block cell cycle progression at a specific stage and then release the accumulated cells producing a wave of cells progressing through the cycle in unison. However, some cell types find this block toxic resulting in abnormal cell cycling, or even mass death. Bromodeoxyuridine (BrdU) uptake can be used to track the cell cycle stage of individual cells. Cells incorporate this synthetic thymidine analog, while synthesizing new DNA during S phase. By providing BrdU for a brief period it is possible to mark a pool of cells that were in S phase while the BrdU was present. These cells can then be tracked through the remainder of the cell cycle and into the next round of replication, permitting the duration of the cell cycle phases to be determined without the need to induce a potentially toxic cell cycle block. It is also possible to determine and correlate the expression of both internal and external proteins during subsequent stages of the cell cycle. These can be used to further refine the assignment of cell cycle stage or assess effects on other cellular functions such as checkpoint activation or cell death.
Introduction
L'évaluation des caractéristiques du cycle cellulaire et les changements qui se produisent dans les cellules au cours de la progression du cycle cellulaire est fondamentale pour comprendre de nombreux aspects de la biologie, en particulier la biologie du cancer. De nombreux agents de développement pour le traitement de tumeurs malignes ont des effets profonds sur la progression du cycle cellulaire ou induire la mort cellulaire par l'intermédiaire de mécanismes dépendants-du cycle cellulaire. Afin d'étudier la dynamique du cycle cellulaire ou des cellules dans une phase particulière du cycle cellulaire, il est usuel de synchroniser les cellules. Cependant, les méthodes de synchronisation peuvent avoir des effets néfastes sur les cellules étudiées, potentiellement confondant les résultats obtenus. 1 analyse Récemment, l'utilisation de protéines marquées par fluorescence qui ne sont présents à des phases particulières du cycle de cellules ont permis la progression du cycle cellulaire dans des cellules individuelles au fil du temps 2, cependant les cellules à étudier besoin d'être génétiquement manipulée pour exprimer ces protéines marquées, ce qui limite leur utilisation à des systèmes dans lesquels il peut être luily atteint.
Le cycle cellulaire est constitué de deux phases actives: la phase de synthèse (S), où l'ADN est répliqué et la mitose (M) où la division cellulaire a lieu. Ces phases sont séparées par trois phases de l'écart, G 0, G 1 et G 2. G 0 ou quiescence, est une phase de repos où la cellule a laissé le cycle, G 1 est où les cellules augmentent de taille avant replication de l'ADN et G 2 dans laquelle la croissance cellulaire continue entre l'achèvement de la replication de l'ADN, mais avant la division cellulaire. La progression à travers le cycle cellulaire est régulée par un certain nombre de points de contrôle. Le G 1 point de contrôle est activé lorsque les conditions environnementales ne sont pas favorables à la synthèse d'ADN et empêche l'entrée en phase S. La phase checkpoint ou de retard intra-S peuvent être déclenchées par des dommages de l'ADN qui peuvent entraîner des fourches de réplication bloquées. Pendant G 2 de la fidélité de l'ADN répliqué est confirmée et si le dommage est détecté, le G 2 checkpoint est activée en permettant réparation de l'ADN avant la division cellulaire. Un point de contrôle finale lors de la mitose assure que chromatides ont été correctement alignés à la plaque mitotique sorte que la division cellulaire peut être complété avec succès. 3 L'activation de ces points de contrôle est communément utilisé pour synchroniser des populations cellulaires. points de contrôle du cycle cellulaire peuvent être activés par un certain nombre de facteurs, mais dans la biologie du cancer le plus commun est la détection des lésions de l'ADN. La réponse aux dommages de l'ADN est initiée par la PI3-kinase comme le télangiectasie kinases de l'ataxie et Rad3 liées (ATR) et protéine atm (ATM) qui activent les kinases effectrices aval Chk1 et Chk2, respectivement. 3 Une gamme d'événements active Chk1 y compris l'impasse fourches de réplication, réticulations d'ADN, et ultraviolet dégâts d'irradiation tout Chk2 est principalement activés par cassures double-brin.
La méthode habituelle pour étudier l'effet de conditions modifiées de la durée du cycle cellulaire is pour synchroniser les cellules dans une phase particulière du cycle cellulaire. 1 Ceci peut être réalisé par plusieurs procédés. Les cellules peuvent être physiquement séparés en fonction de la taille, la densité, la diffusion latérale (granularité), et des marqueurs d'expression de surface cellulaire. Plus concrètement, les cellules peuvent être synchronisées par des moyens chimiques. Plusieurs agents tels que la thymidine, l'hydroxyurée et la cytosine arabinoside peuvent être utilisés pour inhiber la synthèse d'ADN dans la phase S du cycle cellulaire résulte en une accumulation de cellules en phase S qui continuent après cyclage les agents sont enlevés. Les cellules traitées avec le nocodazole, ce qui empêche la formation du fuseau mitotique, avec un arrêt G 2 - phase M ou la teneur en ADN. Élimination de sérum à partir des résultats de milieu de culture dans l'accumulation de cellules en G 0 phase. Le rajout des éléments nutritifs dans le sérum de culture relance le vélo normal des cellules. Cependant, toutes ces méthodes de synchronisation normale interférer avec le vélo et la croissance des cellules et peut Result dans la mort cellulaire significative.
Synchronisation des cellules de leucémie lymphoblastique aiguë est particulièrement difficile et ces cellules ne se prêtent pas à la manipulation génétique. Le procédé décrit ici permet l'évaluation de la dynamique du cycle cellulaire et l'étude des cellules en phases particulières du cycle cellulaire sans synchronisation traditionnel ou une modification génétique. Cette méthode peut également être utile pour d'autres types lorsque la modification génétique et les procédures de synchronisation traditionnels ne sont pas facilement obtenus cellulaires. La méthode est basée sur l'utilisation de longue date de bromodésoxyuridine (BrdU) l'incorporation, qui a très peu d'impact sur la croissance à court terme et la prolifération des cellules. 4 protocoles de BrdU établis profitent de l'incorporation de BrdU dans l'ADN nouvellement synthétisé pendant la phase S . Ceci marque en permanence des cellules comme ayant été en phase S pendant l'exposition au BrdU. Cette population peut être identifié à des temps plus tard par coloration pour BrdU Incorporation et ainsi agir comme une population synchronisé qui peut être suivie et évaluée dans le temps permettant l'étude des effets des médicaments sur le transit du cycle cellulaire. BrdU besoin d'être exposée avant la coloration des anticorps, généralement obtenus après traitement à l'acide ou DNase. 6,7 En utilisant la cytométrie en flux pour détecter la BrdU incorporée permet l'inclusion de marqueurs supplémentaires. Le plus important est l'utilisation de colorants pour mesurer la teneur en ADN, ce qui permet l'évaluation de la distribution de phase du cycle cellulaire des cellules qui étaient en phase S au début de l'étude. 8 surface ou intracellulaires des antigènes outre supplémentaires peuvent également être étudiés. 9 Ces peuvent se rapporter à des événements du cycle cellulaire telles que Ki67 ou fonctions cellulaires à apparemment indépendants tels que des marqueurs de l'apoptose tels que la caspase-3 clivée. Les applications potentielles sont limitées par l'imagination de l'enquêteur.
Protocole
Le protocole décrit ici utilise la lignée cellulaire lymphoblastique leucémie aiguë NALM6 mais peut être appliquée à d'autres types de cellules.
1. Les solutions et réactifs
- RPMI complet
- Ajouter 56 ml de sérum de veau foetal (FCS) et 5,5 ml de 200 mM de L-glutamine à une bouteille de milieu RPMI-1640 500 ml.
- BrdU Stock Solution
- Préparer 32,5 mM BrdU (10 mg / ml) dans une solution saline tamponnée au phosphate de Dulbecco (DPBS).
- BrdU complet RPMI
- Ajouter 6,2 ul de BrdU solution stock à 10 ml de RPMI complet.
- Solution de DNase
- Préparez 1 mg DNase / ml dans du DPBS.
- La coloration du tampon
- Préparer 3% de FCS inactivé par la chaleur et de l'azoture de sodium à 0,09% dans du DPBS.
- Reportez-vous à la liste de matériaux pour les définitions de Fixation Buffer, Perméabilisation tampon et le tampon de lavage.
2. Cellules
NonTe: Les cellules ont été cultivées pendant pas plus de 6 mois. Cette méthode est directement adaptable à toute lignée de cellules non-adhérentes avec des ajustements à la densité des cellules et des milieux de culture. Utiliser les cellules qui croissent de façon exponentielle au début de l'expérience.
- Maintenir cellules NALM6 en flacons T-75 de la culture dans RPMI complet. Effectuez toutes les étapes dans des conditions stériles en utilisant une classe II Cabinet prévention des risques biotechnologiques.
- Maintenir cellules NALM6 entre 1-2 x 10 6 cellules par ml en divisant la culture trois fois par semaine.
- Incuber à 37 ° C dans 5% de CO2 dans l'air.
3. Pulse marquage des cellules avec BrdU
ATTENTION: Manipulez BrdU avec précaution car il est un mutagène potentiel et tératogène.
- Centrifuger les cellules à 150 g pendant 5 min. Remarque: Transfert de cellules dans un milieu frais améliore la reproductibilité des résultats.
- Effectuer une numération cellulaire et remettre les cellules en RPMI complet à 2 x 10 6 cElls / ml.
- Diluer les cellules 1 à 2 avec du milieu RPMI complet BrdU production d'une concentration cellulaire finale de 1 x 10 6 cellules / ml.
- Incuber à 37 ° C avec 5% de CO 2 pendant 45 minutes, puis diluer les cellules dans une 10 avec du milieu RPMI complet. Centrifuger les cellules à 150 g pendant 5 min et soigneusement jeter tout le surnageant.
- Resuspendre les cellules dans un petit volume (~ 100 ul) de RPMI complet, effectuer une numération des cellules et ajuster à 1 x 10 6 cellules / ml.
- Pipette 1 ml de cellules dans les puits d'une plaque de 48 puits. Pipette de 1 ml de DPBS dans tous les puits inoccupés pour obtenir des résultats plus reproductibles.
- Incuber à 37 ° C dans 5% de CO2 dans l'air pendant points temporels souhaités, ici 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17 , 18, 19, 20, 21, 22, 23, et 24 heures. Remarque: La longueur de temps dépendra de ce que la conception expérimentale vise à mesurer.
- Transférez toutes les cellules dans des tubes FACS aide d'une pipette. Rincez le bien séquentiellement avec 1 ml volumes de PBS à un volume total final de 5 ml.
- Centrifuger à 150 g pendant 5 min et retirez soigneusement tous les surnageant. Cellules sont prêtes pour la coloration, effectuer cette (section 4) immédiatement.
4. coloration des cellules
Remarque: si la coloration de surface des cellules est nécessaire exécuter avant la fixation, en veillant à ce que les cellules sont maintenues à 4 ° C tout au long.
- Resuspendre les cellules dans 100 ul de tampon de coloration (pour la coloration en surface en option, ajouter le volume recommandé de l'anticorps à des antigènes de surface et incuber pendant 30 min à 4 ° C).
- Ajouter 1 ml de tampon de coloration, centrifuger pendant 5 min à 150 xg et jeter le surnageant.
Nota: Les anticorps spécifiques, la concentration, le temps d'incubation, etc. variera en fonction des objectifs expérimentaux spécifiques. - Fixation et Perméabilisation
- Resuspendre les cellules dans 100 ul de tampon de fixation et incuber pendant 15 min à température ambiante.
- Ajouter 1 ml otampon de lavage de f, centrifugeuse pendant 5 min à 150 xg et jeter le surnageant.
- Resuspendre les cellules dans 100 ul de tampon de perméabilisation et incuber les cellules pendant 10 min sur de la glace.
- Ajouter 1 ml de tampon de lavage, centrifuger pendant 5 min à 150 xg, et jeter le surnageant.
- Resuspendre les cellules dans 100 ul de tampon de fixation par tube et incuber pendant 5 minutes à température ambiante.
- Ajouter 1 ml de tampon de lavage, centrifuger pendant 5 min à 150 xg, et jeter le surnageant.
Remarque: Le protocole peut être mis en pause ici si nécessaire. Les cellules fixées sont stables pendant plusieurs jours à 4 ° C si on remet en suspension dans du tampon de coloration. Retirez le tampon de coloration après centrifugation avant de procéder.
- Traitement DNase
- Resuspendre les cellules dans 100 ul de solution de DNase (30 pg de DNase / 10 6 cellules) et incuber les cellules pendant 1 heure à 37 ° C.
- Ajouter 1 ml de tampon de lavage, centrifuger à 150 g pendant 5 min et éliminer le surnageant.
- Anticorps Coloration
Note: La coloration de marqueurs intracellulaires autres que BrdU peut être réalisée simultanément avec la coloration de BrdU.- IMPORTANT: Préparer des témoins de compensation comprenant des cellules non colorées et les cellules marquées avec chaque fluorochrome unique. Idéalement, utilisez les mêmes anticorps pour les contrôles de rémunération que celles utilisées dans les tubes expérimentaux. Cependant, si cela est possible, des anticorps contre des antigènes de substitution hautement exprimés conjugués à la même fluorochrome.
- Remettre en suspension les cellules dans 50 ul de tampon de lavage et ajouter 1 pl / 10 6 cellules d'anticorps BrdU. Remarque: les anticorps directement conjugués à d'autres antigènes intracellulaires spécifiques peuvent également être ajoutés.
REMARQUE:. Les anticorps anti-histone H3 phosphorylée sur Ser10 peuvent être utilisées pour établir une discrimination entre les cellules dans G2 et M, l'histone H3 est phosphorylée sur Ser10 lors de la mitose 10 Les anticorps anti-cdc2 phosphorylés sur Tyr15 peut être utilisé pour détecter des cellules qui HAVe engagée à la mitose. 11 - Incuber les cellules pendant 20 min à température ambiante.
- Ajouter 1 ml de tampon de lavage, les cellules de centrifugation à 150 g pendant 5 min et éliminer le surnageant.
- Stain ADN pour l'analyse du cycle cellulaire
- Desserrer pastille et ajouter 20 ul de la solution de 7-AAD (0,25 ug). Remarque: Il est essentiel d'utiliser une quantité constante de 7-AAD / cellule.
- Remettre en suspension les cellules dans 1 ml de tampon de coloration.
5. Collecte de données de cytométrie en flux
Remarque: La machine requis dépendra du nombre et de la nature des fluorochromes utilisés.
- Recueillir les paramètres suivants: FSC-A, SSC-A, FSC-H (FSC-W peuvent être utilisés à la place de FSC-H) et 7-AAD fluorescence sur une échelle linéaire. Recueillir le canal APC sur une échelle logarithmique. Recueillir tous les canaux supplémentaires nécessaires pour l'évaluation de surface ou internes des étiquettes en utilisant une échelle logarithmique.
- Effectuer comcompensation de chevauchement des signaux dans les spectres d'émission observée entre les différents fluorochromes avant d'analyser les échantillons. Note: La plupart des cytomètres de flux effectueront automatiquement.
- Recueillir au moins 10 000 événements pour chaque échantillon.
6. Analyse de cytométrie en flux de données
Remarque: FlowJo a été utilisé dans cette étude pour cytométrie de flux de données, mais d'autres logiciels peuvent également être utilisés. La stratégie de déclenchement est illustré sur la figure 1.
- Identifier la population de cellules viables en utilisant les paramètres FSC-A et SSC-A.
- Dans cette population exclure doublets et d'agrégats à l'aide de FSC-A et FSC-H (FSC-W peut également être utilisée ici).
- Dans cette population définir un tracé de points en utilisant 7-AAD sur l'axe des x et BrdU-APC sur l'axe-y.
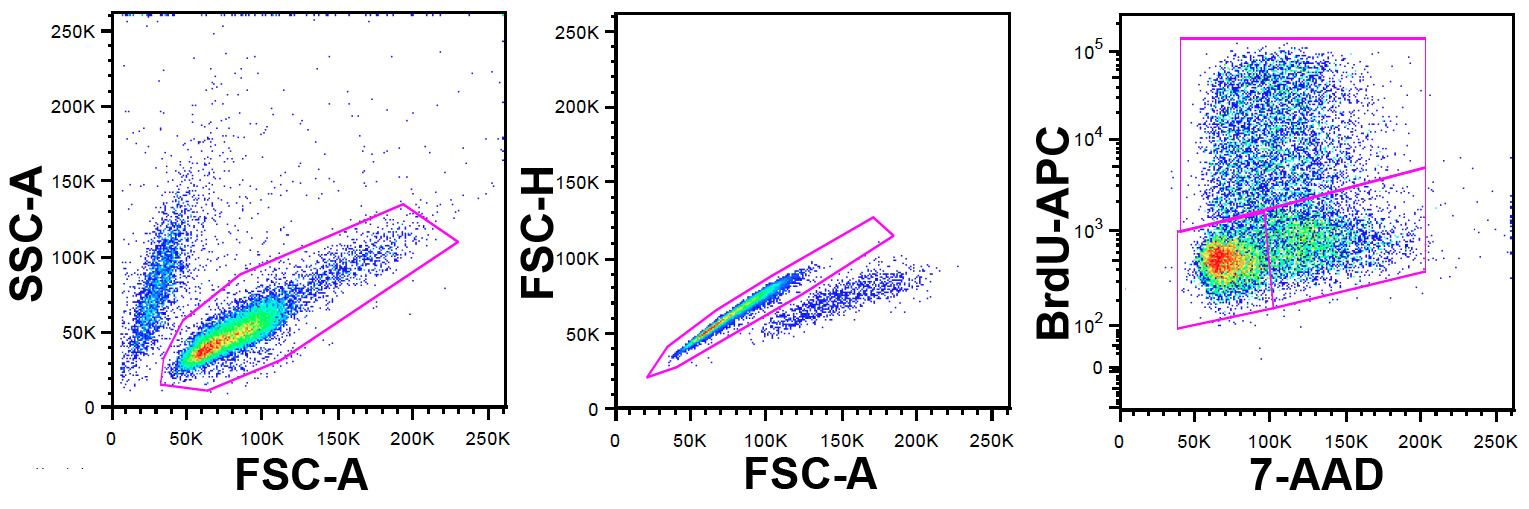
Figure 1: Stratégie Gating Gauche pa.nel: cellules non synchronisées sont présentés sur une FCS-A vs SSC-A dot plot. La population de cellules viables est identifié par la grille présentée. Panneau central: cellules bloquées à partir du panneau gauche sont indiqués sur une FSC-A vs FSC-H tracé de points (FSC-W peut être utilisé à la place de la hauteur). Doublets et les agrégats sont identifiés et exclus par la porte montré. Panneau de droite: cellules gated partir de la date de l'exclusion de doublet dans le panneau central sont présentés sur un 7-AAD vs APC-A dot plot. L'anticorps est marqué avec BrdU APC permettant l'identification des cellules qui ont incorporé la BrdU au cours de l'étiquetage d'impulsions. 7-AAD fournit des informations sur la teneur en ADN. La porte supérieure définit cellules positives pour BrdU et donc en phase S au moment de l'impulsion de BrdU, la porte en bas à gauche, les cellules en G 0/1 et la porte en bas à droite ceux de G 2 / M. S'il vous plaît cliquer ici pour voir une plus grande version de ce chiffre.
- Cell Cycle A nalyse
- Ouvrez le premier fichier de données et porte sur les cellules dans la porte de l'exclusion de doublet.
- Analyser cette population pour la distribution du cycle cellulaire (situé sous les plates-formes de logiciels FloJo) et utiliser le modèle de Dean-Jett-Fox.
- Procurez-vous les positions du G 0/1 et G 2 / M pics en utilisant créer portes.
- Porte sur la BrdU des cellules positives et sous cette même population à l'analyse du cycle cellulaire.
- Fournir les positions pour le G 0/1 et G 2 / M pics en appliquant les mêmes portes de créer des portes et la mise contraintes (en utilisant les portails créés) pour les positions du G 0/1 et G 2 / M pics. Ceci est illustré dans les deux premiers panneaux de la figure 2.
Remarque: Autre logiciel peut également être utilisé pour analyser les données et les instructions varie en conséquence.
840 / 52840fig2highres.jpg "width =" 700 "/>
Figure 2:. Progression du cycle cellulaire Le premier panneau (toutes les cellules) est fermée sur la population de cellules défini par la porte d'exclusion de doublet. Cette population a été affichée dans un histogramme avec 7-AAD sur l'axe-X. Le pic du pic G 0/1 est indiqué par la flèche en dessous de l'axe. Dans les panneaux ultérieures des cellules positives à la BrdU ont été fermée comme sur la figure 1. La valeur de la position de 0/1 G obtenue lorsque gating de la porte d'exclusion de doublet est appliquée aux cellules BrdU positives gated logiciel au sein du cycle cellulaire FlowJo. Chaque panneau a été fermée subséquent sur la population BrdU positives comme représenté sur la figure 1 et la position du pic G 0/1 sur la base de la valeur obtenue dans l'analyse de l'ensemble de la population comme indiqué dans les deux premiers panneaux. Utilisation de la fraction BrdU négatif pour identifier l'emplacement de la population G 0/1 pour les cellules positives à la BrdU dans la même sample contrôle des différences légères dans l'intensité de la tache d'ADN entre les échantillons. Le numéro indiqué sur chaque panneau représente le temps écoulé depuis l'impulsion BrdU terminé. Les phases du cycle cellulaire calculées sont indiquées en vert ombragé. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
Résultats
Cette méthodologie peut être utilisée pour obtenir une série d'informations. Quelques applications sont décrites ici.
L'évaluation de la durée du cycle cellulaire
Pour déterminer le temps nécessaire pour les cellules de transit à travers le cycle cellulaire, les cellules sont récoltées à divers points de temps suivants l'impulsion de BrdU. Les intervalles entre les évaluations peuvent être adaptés à des cellules particulières en cour...
Discussion
La capacité d'analyse du cycle cellulaire est importante pour la compréhension de la biologie du cancer et le mécanisme d'action de ces deux médicaments et les gènes qui influencent la prolifération cellulaire et la croissance. Bien qu'il existe une multitude de dosages qui mesurent la prolifération cellulaire aurait, dont la majorité ne fournissent une mesure qui indique les cellules numériques présents. Ceux-ci comprennent des dosages qui mesurent le nombre de cellules par visualisation directe e...
Déclarations de divulgation
The authors have nothing to disclose.
Remerciements
The work was funded by the Leukemia and Lymphoma Society of the USA (6105-08), a Cancer Council NSW grant (13-02), an NHMRC Senior Research Fellowship (LJB) (1042305) and project grant (1041614).
matériels
| Name | Company | Catalog Number | Comments |
| APC BrdU Flow Kit | BD Biosciences | 552598 | Contains BrdU antibody, 7-AAD and BD Cytofix/Cytoperm Buffer (referred to as Fixation Buffer) |
| BD Cytoperm Permeabilization Buffer Plus | BD Biosciences | 561651 | Referred to as Permeabilization buffer |
| BD Perm/Wash Buffer | BD Biosciences | 554723 | Referred to as Wash buffer |
| DNase | Sigma | D-4513 | |
| BD Falcon 12 x 75 mm FACS tubes | BD Biosciences | 352008 | |
| BD Pharmingen Stain Buffer | BD Biosciences | 554656 | |
| BD LSR FORTESSA flow cytometer | BD Biosciences | FORTESSA | |
| Pipetman | Gilson | P2, P20, P100, P1000 | |
| RPMI 1,640 w/o L-Gln 500 ml | Lonza | 12-167F | |
| DPBS | Lonza | 17-512F | |
| Fetal Bovine Serum | FisherBiotec | FBS-7100113 | |
| L-Glutamine | Sigma | G7513-100ML | |
| 5-Bromo-2′-deoxyuridine | Sigma | B5002-1G | |
| Falcon TC 150 cm2 vented Flasks | BD Biosciences | 355001 | |
| Pipettes 25 ml | Greiner | 760180 | |
| Aersol Pipettes 200 µl | Interpath | 24700 | |
| Aersol Pipettes 1 ml | Interpath | 24800 | |
| Centrifuge | Spintron | GT-175R | |
| CO2 incubator | Binder | C 150 | |
| AF488 anti-Histone H3 Phospho (Ser10) Antibody | Cell Signalling | 9708S | |
| Phospho-Chk2 (Thr68) (C13C1) Rabbit mAb | Cell Signalling | 2197S | |
| Phospho-Chk1 (Ser345) (133D3) Rabbit mAb | Cell Signalling | 2348S | |
| NALM6 | DSMZ | ACC-128 |
Références
- Banfalvi, G., Banfalvi, G. . Methods Mol Biol. 761, 1-23 (2011).
- Sakaue-Sawano, A., et al. Visualizing spatiotemporal dynamics of multicellular cell-cycle progression. Cell. 132, 487-498 (2008).
- Harper, J. W., Elledge, S. J. The DNA damage response: ten years after. Molecular Cell. 28, 739-745 (2007).
- Latt, S. A., George, Y. S., Gray, J. W. Flow cytometric analysis of bromodeoxyuridine-substituted cells stained with 33258 Hoechst. The Journal of Histochemistry and Cytochemistry. 25, 927-934 (1977).
- Gratzner, H. G. Monoclonal antibody to 5-bromo- and 5-iododeoxyuridine: A new reagent for detection of DNA replication. Science. 218, 474-475 (1982).
- Carayon, P., Bord, A. Identification of DNA-replicating lymphocyte subsets using a new method to label the bromo-deoxyuridine incorporated into the DNA. Journal of Immunological Methods. 147, 225-230 (1992).
- Gonchoroff, N. J., et al. S-phase detection with an antibody to bromodeoxyuridine. Role of DNase pretreatment. Journal of Immunological Methods. 93, 97-101 (1986).
- Rabinovitch, P. S., Torres, R. M., Engel, D. Simultaneous cell cycle analysis and two-color surface immunofluorescence using 7-amino-actinomycin D and single laser excitation: applications to study of cell activation and the cell cycle of murine Ly-1 B cells. Journal of Immunology. 136, 2769-2775 (1986).
- Saunders, P. O., et al. RAD001 (everolimus) induces dose-dependent changes to cell cycle regulation and modifies the cell cycle response to vincristine. Oncogene. 32, 4789-4797 (2013).
- Hendzel, M. J., et al. Mitosis-specific phosphorylation of histone H3 initiates primarily within pericentromeric heterochromatin during G2 and spreads in an ordered fashion coincident with mitotic chromosome condensation. Chromosoma. 106, 348-360 (1997).
- Draetta, G., Beach, D. Activation of cdc2 protein kinase during mitosis in human cells: cell cycle-dependent phosphorylation and subunit rearrangement. Cell. 54, 17-26 (1988).
- Saunders, P. O., et al. RAD001 (everolimus) induces dose-dependent changes to cell cycle regulation and modifies the cell cycle response to vincristine. Oncogene. , (2012).
- Knudsen, R. C., Ahmed, A. A., Sell, K. W. An in vitro microassay for lymphotoxin using microculture plates and the multiple automated sample harvester. Journal of Immunological Methods. 5, 55-63 (1974).
- Beck, H. P. Proliferation kinetics of perturbed cell populations determined by the bromodeoxyuridine-33258 technique: radiotoxic effects of incorporated [3H]thymidine. Cytometry. 2, 170-174 (1981).
- Miller, M. J., Wei, S. H., Parker, I., Cahalan, M. D. Two-photon imaging of lymphocyte motility and antigen response in intact lymph node. Science. 296, 1869-1873 (2002).
- Collins, J. M., Berry, D. E., Bagwell, C. B. Different rates of DNA synthesis during the S phase of log phase HeLa S3, WI-38, and 2RA cells. Journal of Biological Chemistry. 255, 3585-3590 (1980).
- Schwarting, R., Gerdes, J., Niehus, J., Jaeschke, L., Stein, H. Determination of the growth fraction in cell suspensions by flow cytometry using the monoclonal antibody Ki-67. Journal of Immunological Methods. 90, 65-70 (1986).
- Danova, M., et al. Cell cycle-related proteins: a flow cytofluorometric study in human tumors. Biology of the Cell. 64, 23-28 (1988).
- Juan, G., et al. Histone H3 phosphorylation and expression of cyclins A and B1 measured in individual cells during their progression through G2 and mitosis. Cytometry. 32, 71-77 (1998).
- Langan, T. J., Chou, R. C. Synchronization of mammalian cell cultures by serum deprivation. Methods in Molecular Biology. 761, 75-83 (2011).
- Kues, W. A., et al. Cell cycle synchronization of porcine fetal fibroblasts: effects of serum deprivation and reversible cell cycle inhibitors. Biology of Reproduction. 62, 412-419 (2000).
- Urbani, L., Sherwood, S. W., Schimke, R. T. Dissociation of nuclear and cytoplasmic cell cycle progression by drugs employed in cell synchronization. Experimental Cell Research. 219, 159-168 (1995).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon